Method Article
Proteom-weite Quantifizierung des Benennens Homogenität auf der Ebene des einzelnen Moleküls
In diesem Artikel
Zusammenfassung
Hier präsentieren wir ein Protokoll, um die Kennzeichnung Homogenität für jede Art von Protein in einer komplexen Protein-Probe auf der Einzelmolekül-Ebene zu bewerten.
Zusammenfassung
Zelle Proteome zeichnen sich oft mit Elektrophorese-Assays, wo alle Arten von Proteinen in den Zellen sind unspezifisch mit einer Leuchtstofffärbung beschriftet und sind von einem Photodetektor nach ihrer Trennung entdeckt. Einzelmolekül-Fluoreszenz-Bildgebung bieten Gromer Proteindetektion mit seiner Fähigkeit zur Visualisierung von einzelnen fluoreszierende Moleküle. Allerdings wird die Anwendung dieser leistungsstarke bildgebende Methode Elektrophorese-Assays durch den Mangel an Möglichkeiten, um die Homogenität der fluoreszierenden Kennzeichnung der einzelnen Protein-Arten über das Proteom charakterisieren behindert. Hier haben wir eine Methode zur Evaluierung der Kennzeichnung Homogenität über das Proteom basierend auf ein einzelnes Molekül Fluoreszenz imaging Assay entwickelt. In unserer Messung mit Hilfe einer Stichprobe von HeLa-Zelle, der Anteil der Proteine, die mit mindestens einem Farbstoff, der wir genannt "Kennzeichnung Belegung" beschriftet (LO), war entschlossen, Auswahl von 50 % bis 90 %, das hohe Potenzial der Anwendung der Einzelmolekül-Bildgebung zu unterstützen sensibel und präzise Proteomanalyse.
Einleitung
Proteomanalyse, deren Ziel es ist, die Gesamtheit der Protein-Moleküle in die Zelle ausgedrückt zu quantifizieren, ist ein wertvoller Ansatz an aktuellen biologischen und medizinischen Studien. Diese Analyse basiert häufig auf Massenspektrometrie, die Protein-Arten anhand der Spektren erzeugt durch Protein Ionisation1,2,3identifiziert. Eine alternative Methode für Proteomanalyse ist Elektrophorese, einschließlich Polyacrylamid-Gelelektrophorese (PAGE), Kapillarelektrophorese und zweidimensionale (2D) Gelelektrophorese. Diese Methode beruht auf unspezifischen fluoreszierende Kennzeichnung der Protein-Moleküle in den analysierten Zellen, gefolgt von elektrophoretischen Trennung und Erkennung und Quantifizierung der einzelnen Protein-Arten. Zur Erreichung der erforderlichen unspezifische Protein-Kennzeichnung ist eine Strategie, fluoreszierende Farbstoffe zu verwenden, die an Proteine über elektrostatische und hydrophobe Interaktionen, wie Coomassie Blau und Sypro-Ruby4,5,6 binden können . Eine alternative Strategie ist mit kovalente Kennzeichnung mit Farbstoffen mit N-Hydroxysuccinimide (NHS) Ester oder Maleimide, die kovalent an Proteine durch gemeinsame Rückstände wie primäre Amine und Thiole binden kann, bzw.7, 8.
Unterdessen ist die Nachweisempfindlichkeit Fluoreszenz ideal für Low-Fülle Proteine und kleine Anzahl von Zellen zu analysieren. Einzelmolekül-Fluoreszenz-Bildgebung ist eines der empfindlichsten Methoden, die ermöglicht die Erkennung von einzelnen fluoreszierende Farbstoffe und markierte Proteine in Vitro und in vivo9,10,11,12, 13,14,15. Anwendung dieser bildgebenden Methode Elektrophorese basierenden Proteomanalyse wird voraussichtlich hochsensiblen und quantitativen Assays ermöglichen durch individuelle Fluoreszent-markierten Proteinen zählen. Es bleibt unklar, ob Beschriftung mit Fluoreszenzfarbstoffen homogen genug über die Proteinmoleküle ist, und wie diese Homogenität von verschiedenen Protein-Arten (Abbildung 1) betroffen ist. Einfache Masse Lösung Messungen können verwendet werden, um einem Molverhältnis von Fluoreszenzfarbstoffen, Proteine, sogenannte "Kupplung Effizienz"8 oder "Beschriftung Effizienz" zu erhalten, aber diese Eigenschaft liefert keine Informationen über die Homogenität der Beschriftung unter Protein-Moleküle.
Hier beschreiben wir das Protokoll für eine Probe zu untersuchen, die Kennzeichnung Homogenität für alle Arten von Protein in der Zelle (Abbildung 2)16. Die zwei wichtigsten Schritte des Assays sind Proteinreinigung und Bildgebung. Im ersten Schritt alle Proteine in den Zellen sind Eindringmittel beschriftet und biotinylierte, dann extrahiert separat mit Gelelektrophorese gefolgt von Electroelution. Im zweiten Schritt sind Fluoreszenzeigenschaften von einzelnen Protein-Moleküle in den entnommenen Proben anhand des Einzelmolekül-Bildgebung. Aus diesen Daten, Parameter, die wichtig für die Zählung Analyse, wie z. B. der Anteil der Proteine mit mindestens einem beschriftet färben, die wir fordern Kennzeichnung Auslastung (LO)16, und die durchschnittliche Anzahl von Fluoreszenzfarbstoffen gebunden an ein einziges Protein-Molekül (N̄ Farbstoff), charakterisiert werden können. In das Protokoll ein optimiertes Verfahren zur Kennzeichnung der HeLa-Zelle Proteome mit NHS Ester-basierte Cyanin 3 (Cy3) Farbstoff wird exemplarisch dargestellt und kann mit anderen Kennzeichnung Verfahren nach gewünschten Forschungsziele geändert werden.
Protokoll
1. Zelle-Vorbereitung
- HeLa-Zellen in einer 10 cm Petrischale bei 37 ° C kultivieren unter 5 % CO2 in Dulbecco modifizierten Adlerhorst 10 % fötalen Rinderserum-haltigem Medium.
- Exponentiell wachsende Zellen zu sammeln, sobald sie 70 % Konfluenz, nach ATCC Anweisungen17erreicht haben.
- Spülen Sie die Zellen mit 5 mL 1 x Phosphat gepufferte Kochsalzlösung (PBS) (pH 7,4). PBS zu entfernen.
- Fügen Sie 1 mL 0,1 % Trypsin-Ethylenediaminetetraacetic Säure (EDTA). 5 min bei 37 ° c inkubieren
- Überwachen Sie das Fortschreiten der Zelle Dissoziation von Mikroskopie.
- Zellen werden Runde und aus der Schale gelöst sind, fügen Sie 4 mL Wachstumsmedium und brechen Sie Zelle Agglomeration durch Pipettieren zu.
- Die Anzahl der Zellen, die Zelle Zähler.
- 150 X g für 10 min in ein Zentrifugenröhrchen zentrifugiert. Entfernen Sie das Medium und Aufschwemmen der Zelle Pellet mit 5 mL 1 X PBS (pH 7,4).
- Wiederholen Sie Schritt 1.2.6. Aufzuwirbeln Sie Zellen mit 1 X PBS-Puffer (pH 7,4), eine Endkonzentration von 106 Zellen/mL mit der Anzahl in Schritt 1.2.5.
Hinweis: Die Zellsuspension kann regelmÄÑig und bei-80 ° C gelagert mehrere Monate sein. Einen Frost/Tau-Zyklus wiederholen sollte vermieden werden.
(2) Zell-Lyse und fluoreszierende Kennzeichnung
- 10 mL Lyse Puffer zu machen (0,1 M Borat, 1 % Natrium-Dodecyl-Sulfat (SDS) und 1 % Tween 20). Passen Sie auf pH 12 mit 2 M NaOH.
Achtung: Konzentrierte NaOH ist ätzend. Geeignete Handschuhe und Brille zu tragen, wenn Sie diese Lösung vorbereiten.
Hinweis: Lysiereinrichtung Puffer kann bis zu einer Woche bei Raumtemperatur aufbewahrt werden. - Mix 100 μL der Lyse Puffer mit 1 μL 1 M Dithiothreitol (DTT) und 4 μL des 50 %-3-[(3-cholamidopropyl)-Dimethylammonio] -1-Propanesulfonate (CHAPS).
- Mix 1 μL der Zellkultur (ca. 1.000 Zellen) mit dem gemischten Lysiereinrichtung Puffer aus Schritt 2.2. Pipettieren langsam um Luftblasen zu vermeiden, um die Lösung zu homogenisieren. Inkubieren Sie im Dunkeln bei Raumtemperatur für 5 min mit sanften Agitation. Neu die Lösung durch Pipettieren langsam zu homogenisieren.
- Fügen Sie 1 μL von 2 μg/mL Cy3 NHS-Ester Farbstoff. Pipettieren langsam um Luftblasen zu vermeiden, um die Lösung zu homogenisieren. 5 min im Dunkeln bei Raumtemperatur unter schonenden rühren inkubieren.
Hinweis: Der hohe pH-Wert dieses Puffers führt Neutralisierung der Lysin-Reste in Proteinen, was zu einer höheren Reaktivität. - Wiederholen Sie die Zugabe von 1 μl 2 μg/ml Cy3 NHS-Ester Farbstoff. Pipettieren langsam um Luftblasen zu vermeiden, um die Lösung zu homogenisieren. 10 min im Dunkeln bei Raumtemperatur unter schonenden rühren inkubieren.
Hinweis: Dieses wiederholte Zugabe von NHS-Ester Farbstoff kann die Kennzeichnung Effizienz zu steigern, weil einige der Farbstoff Cy3 NHS-Ester hydrolysiert und durch den hohen pH-Wert des Puffers Reaktion deaktiviert. - Passen Sie auf pH 7,2 durch Zugabe von 100 μl von 0,8 M 4-(2-hydroxyethyl)-1-Piperazineethanesulfonic Säure (HEPES)-NaOH pH 7,2 um die Reaktion zu stillen. Pipettieren langsam um Luftblasen zu vermeiden, um die Lösung zu homogenisieren.
- Fügen Sie 20 μL 19 mg/mL Biotin-(Polyethylenglykol (PEG))2-Amine und 5 μL von 20 mg/mL 1-Ethyl - 3-(3-Dimethylaminopropyl) Carbodiimide (EDC). Pipettieren langsam um Luftblasen zu vermeiden, um die Lösung zu homogenisieren. 1 h im Dunkeln bei Raumtemperatur unter schonenden rühren inkubieren.
Hinweis: Dieser Schritt Biotinylierungen ist notwendig, die Proteinmoleküle auf ein Mikroskop-Deckglas bei einer festen Dichte und mit keine Vorliebe für Protein-Arten zu beheben. - Umgesetzte Kennzeichnung Reagenzien zu entfernen und die Probe mit einer Ultrafiltration Spalte mit 10 kDa Cutoff, nach den Anweisungen des Herstellers konzentrieren.
Hinweis: Das Protokoll kann hier angehalten werden. Die Probe kann für mehrere Wochen bei-80 ° C gelagert werden.
(3) Proteom Trennung und Verwertung
- 4 x SDS-Probenpuffer (200 mM Tris-HCl pH 6,8, 4 % SDS, 4 % Glycerin und 0,4 % Bromophenol blue) hinzufügen.
Hinweis: Zur Vermeidung von Beschädigungen der Farbstoff die Protein-Probe sollte nicht erhitzt werden, wie im standard SDS-PAGE und bei Raumtemperatur aufbewahrt werden. - Durchführen Sie standard-SDS-PAGE für die Protein-Probe und eine molekulare Leiter, mit 20 % Acrylamid Gel. Migrieren Sie die Probe in das Gel in eine konstante Spannung (160 V) bis die Migration beendet vorne nur das Gel.
- Bild das Gel um die Position der Protein-Bands (Abbildung 3) identifizieren.
- Schneiden Sie die Gel-Teile einschließlich Proteinfraktionen von Interesse mit einer scharfen Klinge, basierend auf den Band-Positionen. Fraktionierung, 16, 23, 55 und 120 kDa Gipfeln und 19-25, 25-32, 32 – 42, 42 – 54, 54 – 69, 69 / 97, 97-136 und 136 – 226 kDa Regionen sind in Abbildung 3dargestellt.
- Proteine mit einem Dialysator durch Electroelution mit einer Porengröße von 6 – 8 kDa, nach den Anweisungen des Herstellers zu extrahieren.
Hinweis: Die extrahierte Probe kann für mehrere Wochen bei-80 ° C gelagert werden.
(4) Mikroskop-Deckglas-Vorbereitung
- Bereiten Sie 200 μL des Avidin Puffer (5 mM HEPES-NaOH pH 7,2, 10 ng/mL Avidin und Rinderserumalbumin (BSA) 2 mg/mL) für jede Probe.
- Bereiten Sie Deckgläsern 22 mm x 22 mm und 0,15 mm Dicke. Setzen Sie die Deckgläsern um Luft-Plasma mit einem Plasma cleaner für 1 min zu reinigen und aktivieren ihre Oberflächen.
- Mantel der Deckgläsern mit Avidin durch Spin-Coating 200 μL der Avidin-Puffer mit einem Spin Coater für 5 Sekunden bei 500 u/min, gefolgt von 30 Sekunden bei 1.000 u/min. Inkubieren Sie Deckgläsern für 15 min bei Raumtemperatur trocknen lassen.
- Die Protein-Probe Deckgläsern vorbereiten.
- Verdünnen Sie die Protein-Probe (ab 3.5) 100 Mal in 5 mM HEPES-NaOH pH 7,2.
- Platz 100 μL der verdünnten Probe auf der Oberfläche in der Mitte des Avidin beschichtete Deckglas (aus Schritt 4.3).
- In der Dunkelheit für 15 min bei Raumtemperatur inkubieren.
- Bereiten Sie die Positivkontrolle.
- Platz 100 μL von 10 ng/mL gereinigt fluoreszierende Biotin in 5 mM HEPES-NaOH pH 7,2 in der Mitte des das Avidin beschichtete Deckglas (aus Schritt 4.3).
- In der Dunkelheit für 15 min bei Raumtemperatur inkubieren.
Hinweis: Diese Positivkontrolle Deckglas stellt die Dichte des Avidin Bindung Spots, dient zum Berechnen der LO.
- Bereiten Sie die negative-Kontrolle.
- Spin-Mantel das Plasma gereinigt Deckglas mit 200 μl 5 mm HEPES-NaOH und 2 mg/mL BSA (ohne Avidin).
- Inkubieren Sie für 15 min bei Raumtemperatur trocknen lassen.
- Fügen Sie 100 μL von 10 ng/mL gereinigt fluoreszierende Biotin in 5 mM HEPES-NaOH pH 7,2.
- In der Dunkelheit für 15 min bei Raumtemperatur inkubieren.
Hinweis: Diese Negativkontrolle Deckglas stellt die Menge der unspezifischen Bindung von fluoreszierenden Biotin, das Deckglas ohne Avidin.
- Spülen Sie jedes Deckglas mit 200 μl destilliertes Wasser durch an den Rändern des Deckglases um die Mitte des Deckglases mit der Spitze berühren nicht zu pipettieren. Wiederholen Sie diese waschen dreimal.
Hinweis: Nach dem Absaugen von Wasser auf das Deckglas, sollte Wasser sofort wieder auf das Deckglas zu verhindern, dass es völlig austrocknen platziert werden. - Luft-Plasma auf die gleiche Anzahl von Deckgläsern wie in Schritt 4.2 aussetzen.
- Legen Sie die gereinigten Deckgläschen auf Probe-gebundenen Deckglas ab Schritt 4.7 um Austrocknen zu vermeiden.
5. Beobachtung mit Einzelmolekül-Fluoreszenz-Mikroskopie
- Starten Sie ein Fluoreszenzmikroskop Weitfeld- oder evaneszenten Feld.
Achtung: Direkte oder verstreuten Laserstrahlung kann dazu führen, dass Auge oder Hautschäden. Geeignete Schutzbrille und schirmen den Lichtweg Laser.
Hinweis: Das Mikroskop-Setup finden Sie auf unserer früheren Publikation16. Kurz gesagt, kann ein Weitfeld- oder evaneszenten Feld Fluoreszenzmikroskop, ausgestattet mit einer Hochleistungs-488 nm Laseranregung, ein hoher numerischer Apertur-Objektiv und eine hochempfindliche Kamera für diese Messung verwendet werden. Darüber hinaus sind für eine automatisierte Messung eine computergesteuerte XY translationale Probentisch, mechanische Fensterläden für Laser Erregungen und ein Autofokus-System empfohlen, installiert werden. - Das Deckglas auf dem Mikroskop und finden im Mittelpunkt.
- Führen Sie einen Kachel-Scan um mindestens 100 Bilder zu erhalten.
Hinweis: Die Laser-Beleuchtung sollte mit mechanischen Fensterläden zum Beispiel ausgesetzt werden, nur dann, wenn Bilder mit der Kamera zur Minimierung von Immunofluoreszenz Aufnahme gesteuert werden. Es gibt viele Beispiele, empfiehlt automatisierte Messung mit Hilfe eines Computerprogramms die Mikroskop-Geräte steuern. Jedes Bild umfasst in der Regel 100 – 500 Einzelmolekül Spots, wenn ein 60 X Objektiv verwenden.
6. Bild-Analyse und Gewinnung von Informationen
- Wenn das Bild Unebenheiten durch die Laser-Beleuchtung-Muster hat, durchführen Sie Bildverarbeitung zu18korrigieren.
- Erwerben Sie ein Referenzbild durch Messung mit einem Deckglas Stichprobe aus gleichmäßig verteilt Farbstoffe mit Hilfe einer Kamera Einstellung die gleiche wie in Schritt 5.3.
- Erwerben Sie ein Offset Bild durch Messung mit keine Laseranregung mit der gleichen Kameraeinstellung.
- Subtrahieren Sie jeder Pixelwert in jedem Beispiel-Bild und die Referenz-Bild mit dem Wert des Offset Bildes.
- Jeder Pixelwert in die subtrahierten Beispielbilder durch den Wert im subtrahierten Referenzbild zu teilen.
- Sammeln Sie ordnungsgemäß erworbenen Bilder zu und entfernen Sie den Bildhintergrund.
- Bilder mit großen fluoreszierende Aggregaten manuell zu entfernen.
- Entfernen Sie den Bildhintergrund durch die Anwendung der rollenden Kugel Algorithmus19 mit einem Ball-Radius von 50 Pixeln mit ImageJ20.
- Schärfen der Bilder durch Subtraktion unscharfe Bilder mit ImageJ (schärfen Bild-Funktion).
- Finden Sie und analysieren Sie Einzelmolekül-Spots in Bildern.
- Einzelmolekül-Spots mit dem Yen Schwelle Algorithmus21 mit ImageJ zu identifizieren.
- Filtern Sie Plätze mit weniger als zwei Pixel auszuschließende Dunkelrauschen der Kamera und stellen mehr als 20 Pixel um Aggregationen von Proteinen mit ImageJ (ROI-Manager) auszuschließen.
- Die Anzahl der Plätze in jedem Bild. Mit der Beleuchtung korrigierten Bilder (Schritt 6.1.4), analysieren total Intensitäten innerhalb von Pixeln zu jedem Spot mit ImageJ.
- Berechnen Sie die Kennzeichnung Auslastung (LO) mithilfe der folgenden Formel:
LO = (Anzahl der zählt in der Probe / Anzahl der Grafen in die Positivkontrolle) X 100 - Berechnen Sie die durchschnittliche Anzahl von Fluoreszenzfarbstoffen gesprungen, um ein einziges Eiweißmolekül (N̄Farbstoff).
- Analysieren Sie ein Histogramm der Intensitäten von jedem Ort.
Hinweis: Das Histogramm zeigt in der Regel mehrere Gipfel, und jede Spitze steht eine unterschiedliche Anzahl von Farbstoffmoleküle an ein Protein gebunden. Die erste, zweite und dritte Gipfel im Histogramm entspricht 1, 2 und 3 Moleküle, beziehungsweise. - Berechnen Sie die durchschnittliche Intensität aus dem Histogramm. Auch berechnen Sie der Peak-Intensität des ersten Peaks durch Anwendung ein "glockenförmig" Fitting.
- Berechnen Sie N̄Farbstoff , indem man die durchschnittliche Intensität mit der Peak-Intensität.
- Analysieren Sie ein Histogramm der Intensitäten von jedem Ort.
Ergebnisse
Abbildung 4 stellt raw-Bilddaten für Bruchteile von unterschiedlichem Molekulargewicht der Proteine von HeLa-Zelle lysate, sowie die positive und negative Kontrolle. Während der Protein-Probe und Positive Ausstellung 100 – 500 Punkte pro Bild steuern, zeigt die negative-Kontrolle keine oder wenige stellen, zeigen, dass das Protokoll ausreichend unspezifische Bindung der Farbstoffe auf der Oberfläche des Deckglases hemmt. Vor Ort Intensität Histogramme für die Proteinproben und die Positivkontrolle präsentieren mehrere Gipfel, stochastische Bindung der Farbstoffe auf primäre Amine an Proteinen und Avidin Tetramer Strukturen22, bzw. vertreten. Alle Spots zeigten blinkende und schrittweisen Immunofluoreszenz mit kontinuierlichen Laseranregung, Hervorhebung Beobachtung auf der Einzelmolekül-Ebene (Abbildung 5AB).
Die LO errechnet sich aus dem Verhältnis der Anzahl der Spots in jedes Protein Sample dazu in der positiv-Kontrolle. Die LO aus der HeLa-Zelle lysate Probe reichte von 50 % für die kleineren Molekulargewicht (16 kDa) Bruch zu 90 % für den höheren Molekulargewicht (120 kDa) Bruch gemessen und lag bei 72 % für die gesamte Proteom-Probe ohne Trennung (Abbildung 6). Entsprechend tendenziell N̄Farbstoff zugenommen mit steigendem Molekulargewicht. Diese steigende Tendenzen gelten auftreten, aufgrund der höheren Anzahl von Lysin Rückstände, führt zu erhöhten NHS-Ester Bindung zu färben. Beispielsweise hat der 23 kDa-Anteil einen hohen LO Wert wegen der großen Zahl an Lysin Rückstände in Histonproteine, die in dieser Fraktion enthalten.
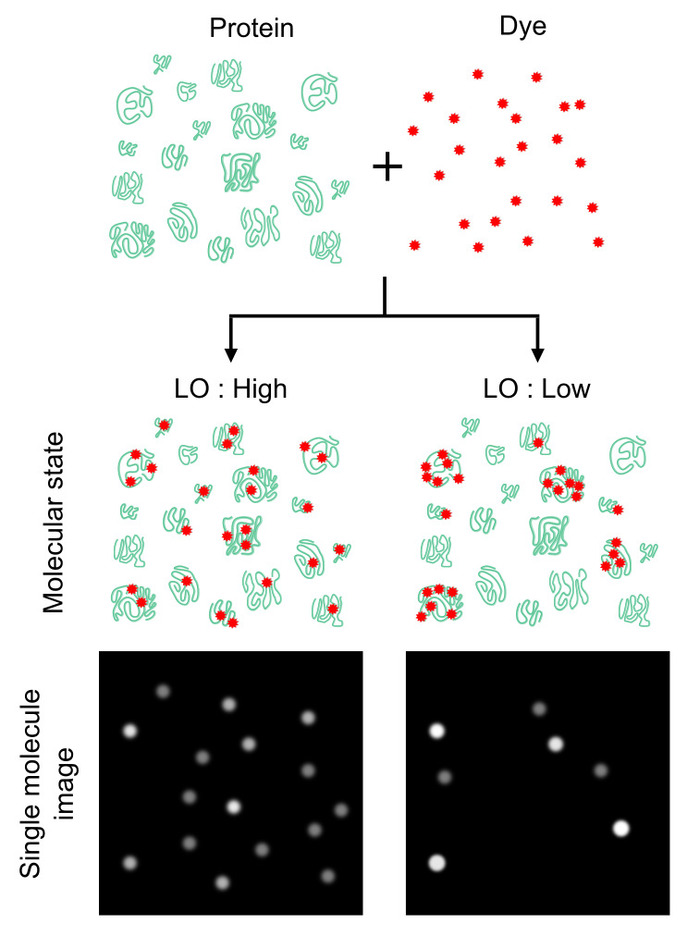
Abbildung 1: Effekt des Proteoms Kennzeichnung Homogenität auf Protein Zahl zählen. Die Protein-Anzahl ist stark abhängig wie stark und Proteinmoleküle sind homogen beschriftet, anstatt der Nummer Verhältnis von Proteinen, Farbstoffe. Die Homogenität gewertet mit einem Parameter bezeichnet Kennzeichnung Auslastung (LO), die die Wahrscheinlichkeit einer beschrifteten Proteinmoleküle gegen Gesamt-Protein-Moleküle definiert. Dieser Wert stellt die Effizienz des Proteins zu zählen (d. h. LO Mehrertrag höher zählen, Zahlen (links) und Vice Versa (rechts)). LO-Wert von 100 % ist ideal, LO von < 100 % können vorsehen, dass ein Dämpfungsfaktor Schätzung absolute Protein zahlen. Diese Zahl wurde von Leclerc Et Al.16 Copyright 2018 American Chemical Society geändert. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 2: Assay Workflow. Erstens sind Avidin, eine feste Dichte zu erreichen Deckgläsern (ein) und (b) behandelt. Zweite, Eindringmittel beschriftet Probe Proteine sind biotinylierte und Deckglas (ein) über die Avidin-Biotin-Interaktion an. Gleichzeitig ist fast 100 % Eindringmittel beschriftet gereinigten Biotin auf das Deckglas (b) immobilisiert. Drittens sind die Deckgläsern Einzelmolekül-Fluoreszenz-Mikroskopie, die Anzahl und Helligkeit Verteilung der Fluoreszenz-Spots zu erhalten abgebildet. Schließlich wird Parameters Homogenität, LO, aus dem Verhältnis der vor Ort zahlen in (ein), (b) berechnet. Diese Zahl wurde von Leclerc Et Al.16 Copyright 2018 American Chemical Society geändert. Bitte klicken Sie hier für eine größere Version dieser Figur.
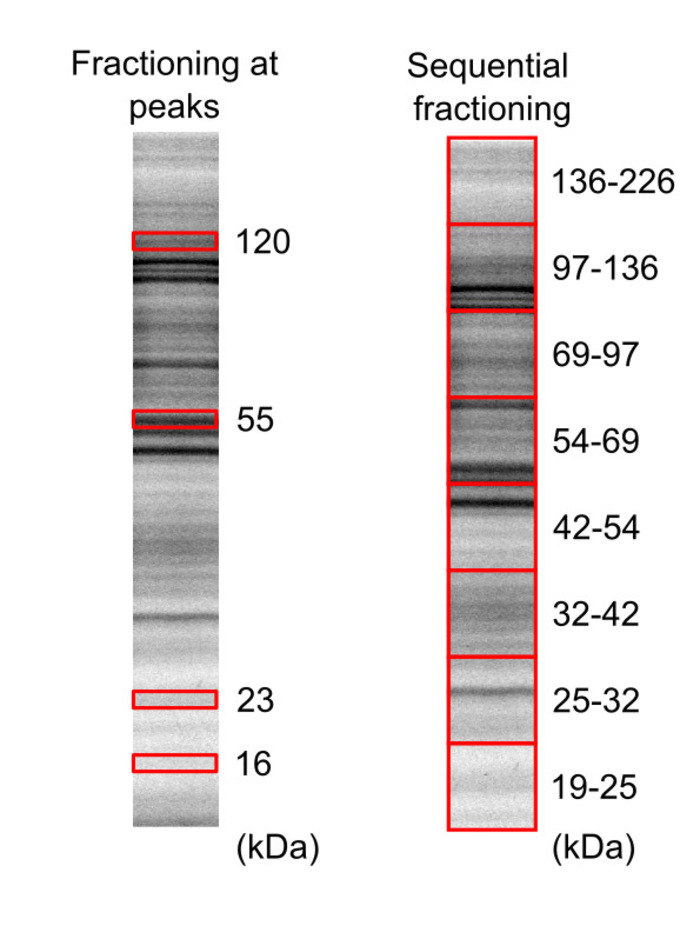
Abbildung 3: Trennung von einem HeLa Proteom Zellprobe SDS-PAGE mit. Fluoreszent markiert biotinylierte Zelle lysate wurden auf zwei verschiedene Gele (20 % Acrylamid) migriert und Proteine visualisiert mit dem Gel-Viewer mit 5 min und 10 min Belichtungszeit für gel 1 und 2, bzw. gel. Die roten Felder stellen die Gel-Regionen, die ausgeschnitten und extrahiert wurden. Diese Zahl wurde von Leclerc Et Al.16 Copyright 2018 American Chemical Society geändert. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 4: einzelne Molekül Bilder Proteome Samples. Fluoreszenz erkennen Bilder (links) und Histogramme (rechts) vor Ort Intensitäten von unterschiedlichem Molekulargewicht Proteom Fraktionen erhalten. Der Maßstab ist 10 μm. Pfeile in die Positivkontrolle zeigen unterschiedlichen Kennzeichnung Schritt des Avidin. Diese Zahl wurde von Leclerc Et Al.16 Copyright 2018 American Chemical Society geändert. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 5: Immunofluoreszenz Fluoreszenz Spots. (A) Zeitraffer Bilder von fluoreszierenden Flecken unter kontinuierlicher Laseranregung. (B) Zeit Spuren der Fluoreszenz-Intensität an verschiedenen Orten. Die Spuren zeigen entweder eins oder zwei Schritt Immunofluoreszenz, darauf hinweist, dass die jeweilige Zahl der Farbstoffe an das Protein an der Stelle des gereinigten BSA Probe gekoppelt waren. Diese Zahl wurde von Leclerc Et Al.16 Copyright 2018 American Chemical Society geändert. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 6: Kennzeichnung Homogenität der Proteome Samples. LO (blau) und N̄Farbstoff (dunkelrot) von unterschiedlichem Molekulargewicht Fraktionen erhalten. Daten werden ausgedrückt als ± s.e. 98, 46, 27, 77, 44, 39, 62, 101, 65, 62, 82, 61 und 70 Bilder bedeuten für die ganze Zelle lysate, analysiert wurden 16, 23, 55, 120, 19-25, 25-32, 32 – 42, 42 – 54, 54 – 69, 69 / 97 97-136 und 136 – 226 kDa-Fraktionen , beziehungsweise. Diese Zahl wurde von Leclerc Et Al.16 Copyright 2018 American Chemical Society geändert. Bitte klicken Sie hier für eine größere Version dieser Figur.
Diskussion
Dieses Whitepaper beschreibt ein Protokoll, um die Kennzeichnung Homogenität der einzelnen markierten Protein-Arten in den Zellen nach der Trennung mit SDS-PAGE (Schritt 3) zu quantifizieren. Das Trennverfahren kann mit anderen Methoden wie flüssige Chromatographie oder Kapillarelektrophorese, ersetzt werden, die Trennung und Fraktionierung von zellulären Proteinen mit hoher Auflösung erfordert spezielle Ausrüstung23ermöglichen. Die Kennzeichnung Methode mit NHS-Ester in das aktuelle Protokoll kann mit anderen Methoden, z.B. durch Maleimide-Ester oder Antikörper, ersetzt werden.
Die Forderung nach Kennzeichnung Homogenität Analyse hängt wieviel die Kennzeichnung von einer einfachen Zufallsprozess abweicht, wie in der Kupplung Effizienzanalyse angenommen. Unsere aktuelle Studie16 darauf hingewiesen, dass der NHS-Ester Kennzeichnung der Proteine von ein zufälliger Prozess je nach Reaktionsbedingungen wie pH-Wert und Reinigungsmittel abweicht. Wir beobachten, dass einige Bedingungen Heterogenität durch kooperative Bindung der Farbstoffe, Proteine oder die Existenz eines Bruchteils des nicht-reaktiven Proteine aufgrund unvollständiger Solubilisierung oder niedrigere Affinität für den Farbstoff induziert. Im Gegensatz dazu reduziert andere Bedingungen Heterogenität durch die vollständige Bindung reaktiver Rückstände innerhalb eines Proteins, der Farbstoff.
Man bedenkt, dass pH ist einer der wichtigsten Einflussfaktoren auf die Kennzeichnung Homogenität. Hoher pH-Wert kann Neutralisierung der Lysin-Reste in Proteinen, was zu einer höheren Reaktivität mit dem NHS-Ester-Farbstoff verursachen. Darüber hinaus hoher pH-Wert Zelle Lysis auslösen kann und kann Proteine denaturieren. Da hohe pH-Wert die Ladung der Proteine negative macht und eignet sich nicht für einige Experimente wie der isoelektrischen Elektrophorese oder 2D Elektrophorese, muss jedoch der pH-Wert Zustand sorgfältig geprüft werden, je nach dem Zweck der Forschung.
Um die Kennzeichnung Homogenität in der Probe genau zu messen, ist es wichtig, genügend biotinylierte Protein Probe verwenden. Und zwar deshalb, weil der Test setzt voraus, dass Proteine mit Sättigung an jedes Avidin-Molekül auf das Deckglas, und nicht genügend Protein Konzentration führt zu Unterschätzung der LO binden. Unsere früheren Studie16 hat gezeigt, dass in der Regel mehr als 10 Pg des Proteins notwendig für die gesättigten Bindung ist, und dies werden rund 1.000 HeLa Zellen24, auch nach der Fraktionierung erfragt kann. Es empfiehlt sich, erstellen Sie eine Verdünnungsreihe der Protein-Probe, die Sättigung des Avidin Flecken zu bestätigen. Wir beachten auch, dass die Avidin-Dichte auf dem Deckglas kann so optimiert werden nicht verursachen zwischen den Spots in aufgezeichneten Bildern, indem man vor Ort in ausreichender Zahl für die statistische Analyse überschneidet.
Dieser Test wird davon ausgegangen, dass man vierwertigen Avidin Molekül22 auf das Deckglas kann ein Protein binden. In der Tat zeigte die Positivkontrolle Daten in unseren früheren Studie bis zu vier Eindringmittel beschrifteten Biotins, aber die Mehrheit (55 %), dass ein Molekül von Avidin gebunden werden kann Avidin Moleküle binden Sie nur ein oder zwei Biotin-Moleküle. Da Proteine Biotin größer sind, sollte ihre sterische Effekte weniger Proteine gebunden zu einem Avidin führt. Unsere Annahme wird weiter durch die Tatsache unterstützt, dass N̄Farbstoff bleibt konstant bei der Messung von Mischproben beschriftet und unmarkiertem Proteine mit unterschiedlichen Seitenverhältnissen16.
Je nach Tierart Protein kann unspezifische Bindung von Proteinen an das Deckglas deutlich sogar mit einer BSA-Beschichtung auftreten. Wenn dies der Fall ist, kann eine Option sein, ein Steuerelement zu messen wo biotinylierte Proteine auf dem Deckglas ohne Avidin, anstelle von fluoreszierenden Biotin bei Schritt 4.6.3 beschichtet. Die Wirkung der unspezifischen Bindung auf LO kann mathematisch durch subtrahieren die Anzahl der Plätze dieses Steuerelements von derjenigen der Protein-Probe beseitigt werden.
Dieses Protokoll wird für Proteom-weite Einzelmolekül-Protein zählen Analysen von unschätzbarem Wert sein. Die Einzelmolekül-Empfindlichkeit ermöglicht Analyse von Protein-Mengen für jedes Protein-Arten unabhängig von ihrer Fülle in der Zelle25. Dies könnte durch Proteom Trennung gefolgt von Quantifizierung der Anzahl der Fluoreszenz Flecken in Verbindung mit Proteinen realisiert werden. Darüber hinaus die hohe Empfindlichkeit können auch einzelne Zellanalyse, Forscher, um Heterogenität über Zell-Populationen zu untersuchen und um clustering von Mobilfunk zu tun heißt es25,26.
Offenlegungen
Die Autoren erklären die folgenden konkurrierenden finanziellen vorgibt: RIKEN hat auf diese Ergebnisse mit S.L. und Y.T genannt als Miterfinder eine Patentanmeldung eingereicht.
Danksagungen
Die Autoren danken Masae Ohno und Kazuya Nishimura für experimentelle Unterstützung und Beratung. Diese Arbeit wurde von PRESTO (JPMJPR15F7), Japan Science and Technology Agency, Grants-in-aid für junge Wissenschaftler (A) (24687022), anspruchsvolle Pionierforschung (26650055) und wissenschaftliche Forschung an innovativen Bereichen (23115005), Japan Society for unterstützt. die Förderung der Wissenschaft und durch Zuschüsse von der Takeda Science Foundation und der Mochida Memorial Foundation für medizinische und pharmazeutische Forschung. S.l. räumt Unterstützung aus dem Programm von RIKEN International Programm zuordnen (IPA).
Materialien
| Name | Company | Catalog Number | Comments |
| 22x22x0.15 mm coverslip | VWR | 470019-004 | |
| 488 nm Argon laser | Coherent | Innova 70C | |
| 488 nm dichroic mirror | Semrock | FF495-Di03 | |
| 488 nm emission filter | Semrock | FF02-520/28 | |
| 560 nm dichroic filter | Semrock | Di02-R561 | |
| 560 nm emission filter | Semrock | FF02-617/73 | |
| 560 nm fiber laser | MBP Communications | F-04306-2 | |
| 60x oil immersion lens | Olympus | PLAPON 60x | |
| Avidin | Nacalai-tesque | 03553-64 | |
| Biotin-PEG-amine | Thermo Scientific | 21346 | |
| Biotinylated Alexa Fluor 488 | Nanocs | ||
| Borate | Nacalai-tesque | ||
| BSA | Sigma-Aldrich | A9547 | |
| CHAPS | Dojindo | C008-10 | |
| Cy3 NHS-ester dye | GE Healthcare | PA13101 | |
| Dialyzer - D-tube, 6-8 kDa | Merck Millipore | 71507-M | |
| DMEM | Sigma-Aldrich | ||
| DTT | Nacalai-tesque | 14112-94 | |
| EDC | Nacalai-tesque | ||
| EMCCD camera | Andor | IiXon 897 | |
| Epi fluorescence microscope | Olympus | IX81 | |
| Gel viewer | GE Healthcare | ImageQuant LAS 4000 | |
| Penicillin, streptomycine and Amphoterecin mix | Gibco | ||
| Plasma cleaner | Diener Electronic | ||
| SDS | Wako | NC0792960 | |
| Size purification column 10K | Merck Millipore | UFC5010 | |
| Tween 20 | Sigma-Aldrich | P9416 |
Referenzen
- Wilhelm, M., et al. Mass-spectrometry-based draft of the human proteome. Nature. 509 (7502), 582-587 (2014).
- Guo, A., et al. Immunoaffinity enrichment and mass spectrometry analysis of protein methylation. Molecular & Cellular Proteomics. 13 (1), 372-387 (2014).
- Tyanova, S., Temu, T., Cox, J. The MaxQuant computational platform for mass spectrometry-based shotgun proteomics. Nature Protocols. 11 (12), 2301-2319 (2016).
- Neuhoff, V., Arold, N., Taube, D., Ehrhardt, W. Improved staining of proteins in polyacrylamide gels including isoelectric focusing gels with clear background at nanogram sensitivity using Coomassie Brilliant Blue G-250 and R-250. Electrophoresis. 9 (6), 255-262 (1988).
- Berggren, K., et al. Background-free, high sensitivity staining of proteins in one- and two-dimensional sodium dodecyl sulfate-polyacrylamide gels using a luminescent ruthenium complex. Electrophoresis. 21 (12), 2509-2521 (2000).
- Butt, R. H., Coorssen, J. R. Coomassie blue as a near-infrared fluorescent stain: a systematic comparison with Sypro Ruby for in-gel protein detection. Molecular & cellular proteomics. 12 (12), 3834-3850 (2013).
- Nanda, J. S., Lorsch, J. R. Labeling a protein with fluorophores using NHS ester derivitization. Methods in Enzymology. , 87-94 (2014).
- Kim, Y., et al. Efficient site-specific labeling of proteins via cysteines. Bioconjugate chemistry. 19 (3), 786-791 (2008).
- Dickson, R. M., Cubitt, A. B., Tsien, R. Y., Moerner, W. E. On/off blinking and switching behaviour of single molecules of green fluorescent protein. Nature. 388 (6640), 355-358 (1997).
- Penna, A., et al. The CRAC channel consists of a tetramer formed by Stim-induced dimerization of Orai dimers. Nature. 456 (7218), 116-120 (2008).
- Ji, W., et al. Functional stoichiometry of the unitary calcium-release-activated calcium channel. Proceedings of the National Academy of Sciences of the United States of America. 105 (36), 13668-13673 (2008).
- Ulbrich, M. H., Isacoff, E. Y. Subunit counting in membrane-bound proteins. Nature Methods. 4 (4), 319-321 (2007).
- Sugiyama, Y., Kawabata, I., Sobue, K., Okabe, S. Determination of absolute protein numbers in single synapses by a GFP-based calibration technique. Nature Methods. 2 (9), 677-684 (2005).
- Puchner, E. M., Walter, J. M., Kasper, R., Huang, B., Lim, W. A. Counting molecules in single organelles with superresolution microscopy allows tracking of the endosome maturation trajectory. Proceedings of the National Academy of Sciences of the United States of America. 110 (40), 16015-16020 (2013).
- Grassart, A., et al. Actin and dynamin2 dynamics and interplay during clathrin-mediated endocytosis. The Journal of Cell Biology. 205 (5), 721-735 (2014).
- Leclerc, S., Arntz, Y., Taniguchi, Y. Extending Single Molecule Imaging to Proteome Analysis by Quantitation of Fluorescent Labeling Homogeneity in Complex Protein Samples. Bioconjugate chemistry. 29 (8), 2541-2549 (2018).
- Ian Freshney, R. Culture of Animal Cells: A Manual of Basic Technique and Specialized Applications. , John Wiley & Sons. (2015).
- Taniguchi, Y., et al. Quantifying E. coli proteome and transcriptome with single-molecule sensitivity in single cells. Science. 329 (5991), 533-538 (2010).
- Sternberg, Biomedical Image Processing. Computer. 16 (1), 22-34 (1983).
- Rueden, C. T., et al. ImageJ2: ImageJ for the next generation of scientific image data. BMC Bioinformatics. 18 (1), 529(2017).
- Yen, J. C., Chang, F. J., Chang, S. A new criterion for automatic multilevel thresholding. IEEE Transactions on Image Processing. 4 (3), 370-378 (1995).
- Livnah, O., Bayer, E. A., Wilchek, M., Sussman, J. L. Three-dimensional structures of avidin and the avidin-biotin complex. Proceedings of the National Academy of Sciences of the United States of America. 90 (11), 5076-5080 (1993).
- Mostovenko, E., Hassan, C., Rattke, J., Deelder, A. M., van Veelen, P. A., Palmblad, M. Comparison of peptide and protein fractionation methods in proteomics. EuPA Open Proteomics. 1, 30-37 (2013).
- Volpe, P., Eremenko-Volpe, T. Quantitative studies on cell proteins in suspension cultures. European Journal of Biochemistry. 12 (1), 195-200 (1970).
- Huang, B., et al. Counting low-copy number proteins in a single cell. Science. 315 (5808), 81-84 (2007).
- Johnson, A. C., Bowser, M. T. H. igh- High-Speed, Comprehensive, Two Dimensional Separations of Peptides and Small Molecule Biological Amines Using Capillary Electrophoresis Coupled with Micro Free Flow Electrophoresis. Analytical Chemistry. 89 (3), 1665-1673 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten