Method Article
Quantificazione di tutto il proteoma di etichettatura omogeneità a livello di singola molecola
In questo articolo
Riepilogo
Qui, presentiamo un protocollo per valutare l'omogeneità d'etichettatura per ogni specie di proteine in un campione di proteina complessa a livello di singola molecola.
Abstract
Proteomi cellulari sono spesso caratterizzati utilizzando dosaggi di elettroforesi, dove tutte le specie delle proteine nelle cellule non specifico sono marcate con un colorante fluorescente e sono individuate da una cellula fotoelettrica dopo la loro separazione. Formazione immagine di fluorescenza di singola molecola può fornire rilevamento ultrasensibile della proteina con la sua capacità per la visualizzazione di singole molecole fluorescenti. Tuttavia, l'applicazione di questo potente metodo di imaging a saggi di elettroforesi è ostacolata dalla mancanza di modi per caratterizzare l'omogeneità dell'etichettatura fluorescente di ogni specie di proteine attraverso il proteoma. Qui, abbiamo sviluppato un metodo per valutare l'omogeneità d'etichettatura attraverso il proteoma basato su un'analisi di formazione immagine di fluorescenza singola molecola. Nella nostra misura utilizzando un campione di cellule HeLa, la proporzione di proteine etichettate con almeno un colorante, che abbiamo chiamato 'etichettatura occupazione' (LO), è stato determinato per gamma dal 50% al 90%, sostenere l'elevato potenziale dell'applicazione di imaging di singola molecola di analisi del proteoma sensibile e precisa.
Introduzione
Analisi del proteoma, che mira a quantificare l'intero set di molecole di proteina espressa nella cella, è un valido approccio negli attuali studi biologici e medicinali. Questa analisi si basa comunemente sulla spettrometria di massa, che individua le specie di proteine basato su spettri generati attraverso proteine ionizzazione1,2,3. Un metodo alternativo per analisi del proteoma è l'elettroforesi, tra cui l'elettroforesi del gel di poliacrilammide (PAGE), elettroforesi capillare ed elettroforesi bidimensionale (2D). Questo metodo si basa sull'etichettatura fluorescente non specifici di tutte le molecole di proteina in cellule analizzate, seguite da separazione elettroforetica e la individuazione e la quantificazione di ogni specie di proteina. Per ottenere il contrassegno richiesta non specifico proteina, una strategia è quella di utilizzare tinture fluorescenti che possono legarsi a proteine tramite interazioni elettrostatiche e idrofobiche, come blu di Coomassie e Sypro Ruby4,5,6 . Una strategia alternativa consiste nell'utilizzare etichettatura covalente con coloranti contenenti N-Idrossisuccinimide (NHS) estere o maleimide, che può legare covalentemente alle proteine attraverso comuni residui come primarie ammine e tioli, rispettivamente7, 8.
Nel frattempo, la sensibilità di rilevazione della fluorescenza è ideale per l'analisi di basso-abbondanza proteine e un piccolo numero di cellule. Formazione immagine di fluorescenza di singola molecola è uno dei metodi più sensibili che permette la rilevazione dei singoli coloranti fluorescenti e con etichetta delle proteine in vitro e in vivo9,10,11,12, 13,14,15. Applicazione di questo metodo di imaging per analisi del proteoma basati su elettroforesi è previsto per consentire analisi altamente sensibili e quantitative contando singole proteine fluorescente etichettati. Tuttavia, rimane poco chiaro se etichettatura con coloranti fluorescenti è abbastanza omogenea in tutte le molecole di proteina, e come questa omogeneità è influenzata da specie differenti della proteina (Figura 1). Rilievi relativi alla soluzione semplice massa possono essere utilizzati per ottenere un rapporto molare di coloranti fluorescenti per proteine chiamate 'efficienza di accoppiamento'8 o 'etichettatura efficienza', ma questa proprietà non fornisce informazioni sull'omogeneità di etichettatura tra molecole di proteina.
Qui, descriviamo il protocollo per un'analisi per indagare l'omogeneità d'etichettatura per tutte le specie di proteine nella cellula (Figura 2)16. I due passaggi chiavi di questo test sono imaging e purificazione della proteina. Nel primo passaggio, tutte le proteine nelle cellule fluorescente sono etichettate e biotinilato, quindi estratta separatamente utilizzando l'elettroforesi del gel seguita da electroelution. Nel secondo passaggio, proprietà di fluorescenza di molecole singole proteine nei campioni estratti vengono valutate in base su formazione immagine singola molecola. Da questi dati, parametri importanti per l'analisi di conteggio, come la percentuale di proteine etichettate con un'almeno una tingono, che noi chiamiamo etichettatura occupazione (LO)16, e il numero medio di coloranti fluorescenti legati ad una molecola singola proteina (n̄ colorante), può essere caratterizzato. Nel protocollo, una procedura ottimizzata per l'etichettatura del proteoma delle cellule HeLa con tintura di cianina 3 (Cy3) a base di estere NHS è presentata come esempio e può essere modificata con altre procedure di etichettatura secondo obiettivi di ricerca desiderato.
Protocollo
1. cella preparazione
- Coltivare le cellule HeLa in un piatto di 10 cm a 37 ° C inferiore al 5% di CO2 in Dulbecco s modified mezzo di Eagle contenente 10% siero bovino fetale.
- Raccogliere le cellule in crescita esponenziale una volta che hanno raggiunto il 70% confluency, seguendo le istruzioni di ATCC17.
- Sciacquare le cellule con 5 mL di 1x tamponato fosfato salino (PBS) (pH 7.4). Rimuovere PBS.
- Aggiungere 1 mL di acido di 0,1% tripsina-etilendiamminotetraacetico (EDTA). Incubare per 5 minuti a 37 ° C.
- Monitorare la progressione della dissociazione delle cellule da microscopia.
- Una volta che le cellule diventano rotonde e sono staccate dal piatto, aggiungere 4 mL di terreno di coltura e rompere l'agglomerato di cellule pipettando.
- Contare il numero di celle utilizzando un contatore di cellule.
- Centrifugare a 150 x g per 10 min in una provetta da centrifuga. Rimuovere il mezzo e risospendere il pellet cellulare con 5 mL di 1X PBS (pH 7.4).
- Ripetere il punto 1.2.6. Risospendere le cellule in 1X PBS (pH 7.4) ad una concentrazione finale di 106 cellule/mL utilizzando il numero di conteggio nel passaggio 1.2.5.
Nota: La sospensione delle cellule può essere aliquotati e conservati a-80 ° C per diversi mesi. Ripetizione di un ciclo di congelamento/scongelamento dovrebbe essere evitato.
2. cellula Lisi e Contrassegno fluorescente
- Fare 10 mL di tampone di lisi (borato 0,1 M, 1% sodio dodecil solfato (SDS) e 1% Tween 20). Regolare il pH 12 utilizzando 2 M NaOH.
Attenzione: NaOH concentrato è corrosivo. Indossare occhiali e guanti adeguati durante la preparazione di questa soluzione.
Nota: Il buffer di Lisi possono essere memorizzati fino a una settimana a temperatura ambiente. - Mix 100 μL di lisante buffer con 1 μL di 1m ditiotreitolo (DTT) e 4 μL di 50% 3-[(3-cholamidopropyl) dimethylammonio] -1-propanesulfonate (CHAPS).
- Mescolare 1 μL di coltura delle cellule (circa 1.000 cellule) con il buffer di lisante mista dal punto 2.2. Omogeneizzare la soluzione pipettando lentamente per evitare bolle. Incubare al buio a temperatura ambiente per 5 minuti con agitazione delicata. Ri-omogeneizzare la soluzione pipettando lentamente.
- Aggiungere 1 μL di 2 μg/mL tintura Cy3 NHS-estere. Omogeneizzare la soluzione pipettando lentamente per evitare bolle. Incubare per 5 minuti al buio a temperatura ambiente con agitazione delicata.
Nota: Il pH elevato di questo buffer provoca la neutralizzazione dei residui di lisina in proteine, portando a una maggiore reattività. - Ripetere l'aggiunta di 1 μL di 2 μg/mL tintura Cy3 NHS-estere. Omogeneizzare la soluzione pipettando lentamente per evitare bolle. Incubare per 10 minuti al buio a temperatura ambiente con agitazione delicata.
Nota: Questo ripetuto aggiunta di tintura di NHS-estere può aumentare l'efficienza d'etichettatura perché alcuni dei coloranti Cy3 NHS-estere è idrolizzato e disattivato dal alto pH del tampone di reazione. - Regolare il pH 7.2 aggiungendo 100 μL di 0,8 M 4-(2-hydroxyethyl)-1-piperazineethanesulfonic acido (HEPES)-NaOH a pH 7,2 per placare la reazione. Omogeneizzare la soluzione pipettando lentamente per evitare bolle.
- Aggiungere 20 μL di 19 mg/mL (polietilenglicole (PEG)) biotina-2-ammina e 5 μL di 20 mg/mL 1-etil - 3-(3-dimetetilpropile) carbodiimide (EDC). Omogeneizzare la soluzione pipettando lentamente per evitare bolle. Incubare per 1 h al buio a temperatura ambiente con agitazione delicata.
Nota: Questo passaggio biotinylation è necessario fissare le molecole di proteina su un vetrino coprioggetti microscopio ad una densità fissa e con nessun pregiudizio per specie proteiche. - Rimuovere i reagenti d'etichettatura non reagiti e concentrare il campione utilizzando una colonna di ultrafiltrazione con 10 cut-off di kDa, seguendo le istruzioni del produttore.
Nota: Il protocollo può essere messo in pausa qui. Il campione può essere conservato a-80 ° C per diverse settimane.
3. Proteome separazione e recupero
- Aggiungere 4 x tampone campione SDS (200 mM Tris-HCl pH 6.8, SDS 4%, 4% glicerolo e 0,4% bromofenolo).
Nota: Per evitare danni di tintura, il campione della proteina non deve essere riscaldato come fatto in standard SDS-PAGE e dovrebbe essere conservato a temperatura ambiente. - Eseguire standard SDS-PAGE per il campione della proteina e una scala molecolare, utilizzando 20% gel dell'acrilamide. Migrare il campione nel gel a una tensione costante (160 V) fino a quando la migrazione anteriore appena esce il gel.
- Immagine del gel per identificare la posizione delle bande proteiche (Figura 3).
- Tagliare porzioni gel comprende le frazioni di proteine di interesse con una lama affilata, in base alle posizioni di banda. Frazionamento a 16, 23, 55 e 120 kDa picchi, che a 19 – 25, 25 – 32, 32 – 42, 42 – 54, 54 – 69, 69-97, 97 – 136 e 136 – 226 kDa regioni sono mostrate in Figura 3.
- Estrarre le proteine usando un dialyzer di electroelution con un diametro dei pori di 6 – 8 kDa, seguendo le istruzioni del produttore.
Nota: Il campione estratto può essere conservato a-80 ° C per diverse settimane.
4. preparazione del vetrino coprioggetto di microscopio
- Preparare 200 μL di tampone avidina (5 mM HEPES-NaOH pH 7.2, 10 avidina ng/mL e 2 mg/mL albumina di siero bovino (BSA)) per ogni campione.
- Preparare vetrini coprioggetto di 22 x 22 mm e 0,15 mm di spessore. Esporre i coprioggetti per aria plasma usando un plasma cleaner per 1 min per pulire e attivare le loro superfici.
- Rivestire i coprioggetti con avidina di rivestimento per rotazione 200 μL di tampone di avidina usando una macchina a rotazione per 5 secondi a 500 giri/min, seguito da 30 secondi a 1.000 giri/min. Incubare i vetrini coprioggetto per 15 min a temperatura ambiente per farli asciugare.
- Preparare vetrini coprioggetti per il campione della proteina.
- Diluire il campione di proteina (da 3,5) 100 volte in 5 mM HEPES-NaOH a pH 7,2.
- Posto 100 μL del campione diluito sulla superficie al centro del coprivetrino rivestite con avidina (dal punto 4.3).
- Incubare al buio per 15 min a temperatura ambiente.
- Preparare il controllo positivo.
- Posto 100 μL di 10 ng/mL purificato fluorescente biotina in 5 mM HEPES-NaOH pH 7.2 al centro dell'avidina rivestito vetrino coprioggetti (dal punto 4.3).
- Incubare al buio per 15 min a temperatura ambiente.
Nota: Questo controllo positivo vetrino coprioggetti fornisce la densità di avidina macchie di associazione, che viene utilizzato per calcolare il LO.
- Preparare il controllo negativo.
- Spin-cappotto plasma pulito coprivetrino con 200 μL di 5 mM HEPES-NaOH e 2 mg/mL BSA (senza avidina).
- Incubare per 15 min a temperatura ambiente per asciugare.
- Aggiungere 100 μL di 10 ng/mL purificato fluorescente biotina in 5 mM HEPES-NaOH a pH 7,2.
- Incubare al buio per 15 min a temperatura ambiente.
Nota: Questo controllo negativo vetrino coprioggetti fornisce la quantità di legame non specifico di biotina fluorescente per il coprioggetto senza avidina.
- Sciacquare ogni vetrino coprioggetti con 200 μL di acqua distillata pipettando ai bordi del coprivetrino per non toccare la metà del coprivetrino con la punta. Ripetere questo lavaggio tre volte.
Nota: Dopo avere aspirato acqua sul vetrino coprioggetto, acqua dovrebbe essere immediatamente reimmessi sul coprioggetto per evitare che secchino completamente. - Esporre il plasma ad aria per lo stesso numero di vetrini coprioggetti come descritto al punto 4.2.
- Posizionare il vetrino coprioggetti puliti il coprivetrino associato a campione da passo 4.7 per evitare la disidratazione.
5. osservazione con microscopia di fluorescenza di singola molecola
- Avviare un microscopio a fluorescenza Campo grandangolari o evanescente.
Attenzione: Diretta o radiazione laser sparse può causare occhio o danneggiare la pelle. Occhiali di protezione adatti e scudo il percorso della luce laser.
Nota: Per l'installazione di microscopio, fare riferimento alla nostra precedente pubblicazione16. Brevemente, un microscopio a fluorescenza grandangolari o evanescente campo dotato di un'eccitazione di laser ad alta potenza 488 nm, un obiettivo di apertura numerica elevata e una telecamera ad alta sensibilità può essere utilizzato per questa misura. Inoltre, per una misurazione automatica, un palco di campione traslazionale XY controllati dal computer, meccanico persiane per eccitazioni laser e un sistema di messa a fuoco automatica sono raccomandati per essere installato. - Impostare il coprioggetto sul microscopio e trovare la messa a fuoco.
- Eseguire una scansione di mattonelle per ottenere almeno 100 immagini.
Nota: L'illuminazione laser dovrebbe essere controllata con gli otturatori meccanici per essere esposti al campione solo quando si registrano immagini con la fotocamera, per ridurre al minimo photobleaching. Se ci sono molti campioni, usando un programma per computer per controllare i dispositivi di microscopio di misura automatici è raccomandato. Ogni immagine in genere include 100 – 500 punti di singola molecola quando si utilizza un obiettivo 60x.
6. analisi e l'estrazione di informazioni di immagine
- Se l'immagine ha irregolarità dovuta al modello di illuminazione laser, effettuare elaborazioni per correggerlo18.
- Acquisire un'immagine di riferimento misurando con un vetrino coprioggetti campione composto da coloranti uniformemente diffuso usando una fotocamera impostando lo stesso come descritto al punto 5.3.
- Acquisire un'immagine offset misurando con nessuna eccitazione laser utilizzando la stessa impostazione della fotocamera.
- Sottrarre ogni valore di pixel in ogni immagine di esempio e l'immagine di riferimento con il valore dell'offset immagine.
- Dividere ogni valore di pixel nelle immagini di esempio sottratto dal valore nell'immagine di riferimento sottratto.
- Raccogliere immagini acquisite correttamente e rimuovere lo sfondo dell'immagine.
- Rimuovere immagini contenenti grandi aggregati fluorescenti manualmente.
- Rimuovere lo sfondo dell'immagine applicando l' algoritmo di sfera19 con un raggio della sfera di 50 pixel usando ImageJ20.
- Affilare le immagini sottraendo immagini sfocate utilizzando ImageJ (nitidezza immagine funzione).
- Trovare e analizzare i punti di singola molecola in immagini.
- Individuare i punti di singola molecola con l' algoritmo soglia di Yen21 usando ImageJ.
- Filtrare i punti con meno di due pixel per escludere rumori oscuri della telecamera e macchie più di 20 pixel per escludere le aggregazioni delle proteine usando ImageJ (gestore di ROI).
- Contare il numero di punti in ogni immagine. Utilizzando l'illuminazione analizzare immagini corrette (punto 6.1.4), totale intensità all'interno di pixel per ogni punto usando ImageJ.
- Calcolare l'occupazione d'etichettatura (LO) utilizzando la seguente formula:
LO = (numero di conteggi nel campione / numero di conteggi nel controllo positivo) x 100 - Calcolare il numero medio di coloranti fluorescenti legati ad una molecola singola proteina (n̄tintura).
- Analizzare un istogramma dell'intensità di ogni spot.
Nota: L'istogramma ha solitamente parecchi picchi, e ogni picco rappresenta un diverso numero di molecole di colorante legata a una proteina. Il primo, secondo e terzo picco nell'istogramma corrisponde alle molecole di 1, 2 e 3, rispettivamente. - Calcolare l'intensità media dall'istogramma. Inoltre è possibile calcolare l'intensità di picco del primo picco applicando un raccordo gaussiano.
- N̄colorante viene calcolato dividendo l'intensità media con l'intensità di picco.
- Analizzare un istogramma dell'intensità di ogni spot.
Risultati
Figura 4 rappresenta i dati di immagine raw per frazioni di diverso peso molecolare delle proteine da lysate delle cellule HeLa, come pure il controllo positivo e negativo. Mentre il campione della proteina e il positivo controllo esposizione 100 – 500 punti per ogni immagine, viene visualizzato il controllo negativo nessuno o pochi punti, mostrando che il protocollo sufficientemente inibisce il legame non specifico di coloranti alla superficie del vetrino coprioggetti. Gli istogrammi di intensità spot per i campioni della proteina ed il controllo positivo presentano picchi multipli, che rappresenta associazione stocastico di coloranti di ammine primarie in proteine e avidina tetramero strutture22, rispettivamente. Tutte le macchie hanno mostrato photobleaching lampeggiante e graduale con eccitazione laser continuo, evidenziando l'osservazione a livello di singola molecola (Figura 5AB).
Il LO viene calcolato dal rapporto del numero di punti in ogni campione della proteina che nel controllo positivo. Il LO misurata dal campione lisato cellulare HeLa variato dal 50% per la più piccola frazione di peso molecolare (16 kDa) al 90% per il più alto peso molecolare (120 kDa) frazione ed era del 72% per l'intero proteoma esempio senza separazione (Figura 6). Corrispondentemente, n̄tintura ha teso ad aumentare con l'aumento del peso molecolare. Queste tendenze in aumento sono considerate si verificano a causa di più alti numeri di residui di lisina, che conduce a una maggiore NHS-estere tingere associazione. Ad esempio, la frazione di 23 kDa ha un alto valore LO a causa del gran numero di residui di lisina in proteine istoniche contenute in questa frazione.
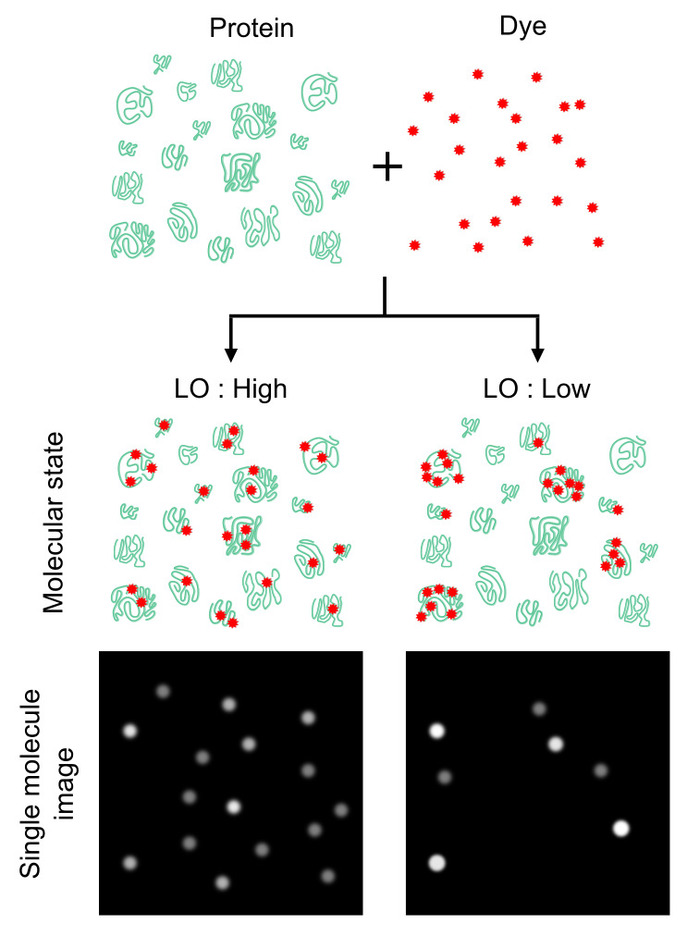
Figura 1: effetto del proteoma etichettatura omogeneità sul conteggio numero proteina. Il numero del numero di proteine è fortemente dipendente come fortemente e in modo omogeneo molecole proteiche sono etichettati, piuttosto che il rapporto del numero di proteine per coloranti. L'omogeneità può essere ottenuta utilizzando un parametro definito occupazione d'etichettatura (LO), che definisce la probabilità di molecole di proteina marcata contro le molecole di proteina totale. Questo valore fornisce l'efficienza di conteggio di proteina (cioè, più alto LO rese più elevate contano numeri (a sinistra) e vice versa (a destra)). Mentre un valore LO del 100% è l'ideale, LO di < 100% può fornire un fattore di attenuazione per stimare i numeri assoluti della proteina. Questa figura è stata modificata da Leclerc et al.16 Copyright 2018 American Chemical Society. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: flusso di lavoro test. In primo luogo, vetrini coprioggetti (un) e (b) sono trattati con avidina per ottenere una densità di fisso. Campione secondo, fluorescente etichettato proteine sono biotinilati e collegato il vetrino coprioggetti (un) tramite l'interazione di avidina-biotina. In parallelo, quasi 100% con etichetta fluorescente purificata biotina è immobilizzata il coprivetrino (b). In terzo luogo, le lamelle sono ripreso da microscopia di fluorescenza di singola molecola per ottenere la distribuzione di luminosità e numero dei punti di fluorescenza. Infine, il parametro di omogeneità, LO, è calcolato dal rapporto dei numeri di posto in (un) a quello (b). Questa figura è stata modificata da Leclerc et al.16 Copyright 2018 American Chemical Society. Clicca qui per visualizzare una versione più grande di questa figura.
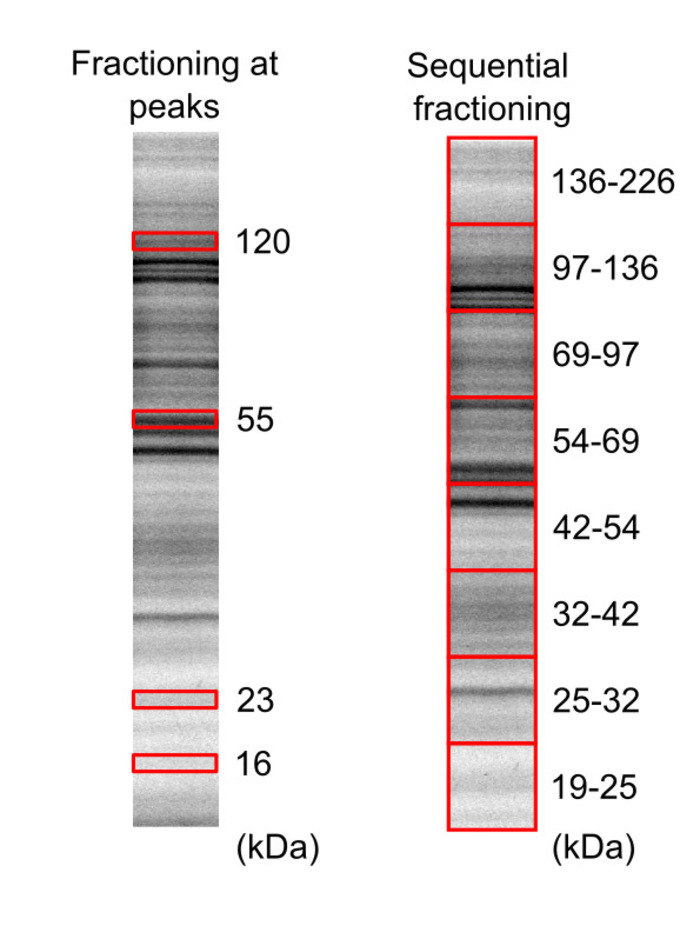
Figura 3: separazione di un campione di proteoma delle cellule HeLa mediante SDS-PAGE. Fluorescente etichettati e biotinilati lysate delle cellule sono stati migrati su due gel differenti (20% di acrilammide), e proteine visualizzati utilizzando il Visualizzatore di gel, con un tempo di esposizione di 5 min e 10 min per gel 1 e gel 2, rispettivamente. Le caselle rosse rappresentano le regioni di gel che sono state tagliate ed estratti. Questa figura è stata modificata da Leclerc et al.16 Copyright 2018 American Chemical Society. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4: singole immagini di molecola del proteoma campioni. Fluorescenza spot immagini (a sinistra) e istogrammi di spot intensità (a destra) ottenuti da frazioni del proteoma di diverso peso molecolare. La barra della scala è di 10 μm. Le frecce nel controllo positivo indicano il passo d'etichettatura differente dell'avidina. Questa figura è stata modificata da Leclerc et al.16 Copyright 2018 American Chemical Society. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 5: Photobleaching di macchie fluorescenti. (A) time-lapse immagini di macchie fluorescenti sotto eccitazione laser continuo. (B) tempo tracce di intensità di fluorescenza in punti diversi. Le tracce mostrano entrambi photobleaching a uno o due step, indicando i rispettivi numeri di coloranti sono stati accoppiati alla proteina al posto del campione purificato di BSA. Questa figura è stata modificata da Leclerc et al.16 Copyright 2018 American Chemical Society. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 6: etichettatura omogeneità dei campioni proteome. LO (blu) e n̄colorante (rosso scuro) ottenuti da frazioni di diverso peso molecolare. I dati sono espressi come media ± immagini s.e. 98, 46, 27, 77, 44, 39, 62, 65, 101, 62, 82, 61 e 70 sono stati analizzati per l'intera cellula lisato, 16, 23, 55, 120, 19 – 25, 25 – 32, 32 – 42, 42 – 54, 54 – 69, 69-97, 97 – 136 e 136 – 226 frazioni di kDa , rispettivamente. Questa figura è stata modificata da Leclerc et al.16 Copyright 2018 American Chemical Society. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Questo articolo descrive un protocollo per quantificare l'omogeneità d'etichettatura di ogni specie di proteina marcata nelle cellule dopo la separazione con SDS-PAGE (passaggio 3). Il metodo di separazione possa essere sostituito con altri metodi come la cromatografia liquida o elettroforesi capillare, che permettono la separazione e frazionamento delle proteine cellulari con alta risoluzione, mentre che richiedono attrezzature speciali23. Il metodo d'etichettatura utilizzando NHS-estere nel protocollo corrente può essere sostituito con altri metodi utilizzando maleimide-estere o anticorpi, per esempio.
Obbligo di etichettatura analisi di omogeneità dipende quanto l'etichettatura si discosta da un processo casuale semplice, come ipotizzato nell'analisi di efficienza di accoppiamento. Il nostro recente studio16 indicato che l'etichettatura di NHS-estere di proteine si discosta da un processo casuale a seconda delle condizioni di reazione quali pH e detergenti. Abbiamo osservato che alcune condizioni indotte da eterogeneità di legame cooperativo di coloranti di proteine o l'esistenza di una frazione delle proteine non reattivo a causa di solubilizzazione incompleta o affinità abbassata per la tintura. Al contrario, altre condizioni ridotta eterogeneità dall'associazione completa dei residui reattivi all'interno di una proteina alla tintura.
Consideriamo che il pH è uno dei fattori più influenti sull'omogeneità d'etichettatura. PH elevato può causare la neutralizzazione dei residui di lisina in proteine, portando a una maggiore reattività con il colorante di NHS-estere. Inoltre, il pH elevato può indurre la lisi delle cellule e possa denaturare le proteine. Tuttavia, poiché pH elevato rende la carica delle proteine negativo e non è adatto per alcuni esperimenti, come l'elettroforesi isoelettrico o elettroforesi 2D, la condizione di pH deve essere attentamente valutata a seconda dello scopo di ricerca.
Per misurare con precisione l'omogeneità d'etichettatura nell'analisi, è importante utilizzare abbastanza campione di proteine biotinilate. Questo è perché il dosaggio presuppone che proteine si legano con saturazione per ogni molecola di avidina sul vetrino coprioggetto e risultati di concentrazione di proteina insufficiente nella sottovalutazione di LO. Il nostro precedente studio16 ha dimostrato che in genere più di 10 pg della proteina è necessaria per l'associazione saturi, e questo può essere ottenuto da circa 1.000 di cellule HeLa24, anche dopo il frazionamento. Si consiglia di creare una serie di diluizioni del campione della proteina per confermare la saturazione delle macchie di avidina. Notiamo anche che la densità di avidina il coprivetrino potrebbe essere necessario essere ottimizzato così da non causare sovrapposizioni tra macchie nelle immagini registrate, mentre dando un numero posto sufficiente per l'analisi statistica.
Questa analisi presuppone che uno tetravalente avidina molecola22 il coprivetrino possibile associare una proteina. Infatti, i dati di controllo positivo nel nostro studio precedente hanno indicato che una molecola di avidina possibile associare fino a quattro fluorescente contrassegnati biotina, ma la maggioranza (55%) dell'avidina molecole si legano solo uno o due molecole di biotina. Perché le proteine sono più grandi di biotina, loro effetti sterici dovrebbero comportare meno proteine legate a una avidina. La nostra ipotesi sono ulteriormente supportata dal fatto che n̄tintura rimane costante quando misurare i campioni misti di etichettati e senza etichetta di proteine differenti rapporti16.
A seconda della specie di proteine, legame aspecifici delle proteine per il vetrino coprioggetto potrebbe verificarsi significativamente anche con un rivestimento di BSA. Se questo è il caso, un'opzione possibile per misurare un controllo dove proteine biotinilate sono rivestite sul coprioggetto senza avidina, invece di biotina fluorescente al punto 4.6.3. L'effetto dell'associazione aspecifiche su LO può eliminarsi matematicamente sottraendo il numero di spot di questo controllo da quello del campione della proteina.
Questo protocollo sarà prezioso per tutto il proteoma singola molecola proteica analisi di conteggio. La sensibilità di singola molecola permetterà analisi di quantità di proteina per ogni specie di proteina indipendentemente dalla loro abbondanza nella cella25. Ciò ha potuto essere realizzata tramite la separazione del proteoma seguita da quantificazione del numero di punti di fluorescenza accoppiati a proteine. Inoltre, l'alta sensibilità permetterà anche analisi unicellulare, consentendo ai ricercatori di esaminare eterogeneità tra le popolazioni delle cellule e per fare clustering di cellulare dichiara25,26.
Divulgazioni
Gli autori dichiarano l'interesse finanziario concorrenti seguenti: RIKEN ha depositato una domanda di brevetto su questi risultati con S.L. e Y.T. denominato come co-inventori.
Riconoscimenti
Gli autori ringraziano Masae Ohno e Kazuya Nishimura per consulenza e assistenza sperimentale. Questo lavoro è stato supportato da PRESTO (JPMJPR15F7), Japan Science and Technology Agency, localizzativi per giovani scienziati (A) (24687022), sfidando ricerca esplorativa (26650055) e ricerca scientifica in settori innovativi (23115005), Japan Society for la promozione della scienza e di sovvenzioni da parte della Takeda Science Foundation e la Fondazione Memorial Mochida per il medico e ricerca farmaceutica. S.L. riconosce sostegno dal programma RIKEN associare programma internazionale (IPA).
Materiali
| Name | Company | Catalog Number | Comments |
| 22x22x0.15 mm coverslip | VWR | 470019-004 | |
| 488 nm Argon laser | Coherent | Innova 70C | |
| 488 nm dichroic mirror | Semrock | FF495-Di03 | |
| 488 nm emission filter | Semrock | FF02-520/28 | |
| 560 nm dichroic filter | Semrock | Di02-R561 | |
| 560 nm emission filter | Semrock | FF02-617/73 | |
| 560 nm fiber laser | MBP Communications | F-04306-2 | |
| 60x oil immersion lens | Olympus | PLAPON 60x | |
| Avidin | Nacalai-tesque | 03553-64 | |
| Biotin-PEG-amine | Thermo Scientific | 21346 | |
| Biotinylated Alexa Fluor 488 | Nanocs | ||
| Borate | Nacalai-tesque | ||
| BSA | Sigma-Aldrich | A9547 | |
| CHAPS | Dojindo | C008-10 | |
| Cy3 NHS-ester dye | GE Healthcare | PA13101 | |
| Dialyzer - D-tube, 6-8 kDa | Merck Millipore | 71507-M | |
| DMEM | Sigma-Aldrich | ||
| DTT | Nacalai-tesque | 14112-94 | |
| EDC | Nacalai-tesque | ||
| EMCCD camera | Andor | IiXon 897 | |
| Epi fluorescence microscope | Olympus | IX81 | |
| Gel viewer | GE Healthcare | ImageQuant LAS 4000 | |
| Penicillin, streptomycine and Amphoterecin mix | Gibco | ||
| Plasma cleaner | Diener Electronic | ||
| SDS | Wako | NC0792960 | |
| Size purification column 10K | Merck Millipore | UFC5010 | |
| Tween 20 | Sigma-Aldrich | P9416 |
Riferimenti
- Wilhelm, M., et al. Mass-spectrometry-based draft of the human proteome. Nature. 509 (7502), 582-587 (2014).
- Guo, A., et al. Immunoaffinity enrichment and mass spectrometry analysis of protein methylation. Molecular & Cellular Proteomics. 13 (1), 372-387 (2014).
- Tyanova, S., Temu, T., Cox, J. The MaxQuant computational platform for mass spectrometry-based shotgun proteomics. Nature Protocols. 11 (12), 2301-2319 (2016).
- Neuhoff, V., Arold, N., Taube, D., Ehrhardt, W. Improved staining of proteins in polyacrylamide gels including isoelectric focusing gels with clear background at nanogram sensitivity using Coomassie Brilliant Blue G-250 and R-250. Electrophoresis. 9 (6), 255-262 (1988).
- Berggren, K., et al. Background-free, high sensitivity staining of proteins in one- and two-dimensional sodium dodecyl sulfate-polyacrylamide gels using a luminescent ruthenium complex. Electrophoresis. 21 (12), 2509-2521 (2000).
- Butt, R. H., Coorssen, J. R. Coomassie blue as a near-infrared fluorescent stain: a systematic comparison with Sypro Ruby for in-gel protein detection. Molecular & cellular proteomics. 12 (12), 3834-3850 (2013).
- Nanda, J. S., Lorsch, J. R. Labeling a protein with fluorophores using NHS ester derivitization. Methods in Enzymology. , 87-94 (2014).
- Kim, Y., et al. Efficient site-specific labeling of proteins via cysteines. Bioconjugate chemistry. 19 (3), 786-791 (2008).
- Dickson, R. M., Cubitt, A. B., Tsien, R. Y., Moerner, W. E. On/off blinking and switching behaviour of single molecules of green fluorescent protein. Nature. 388 (6640), 355-358 (1997).
- Penna, A., et al. The CRAC channel consists of a tetramer formed by Stim-induced dimerization of Orai dimers. Nature. 456 (7218), 116-120 (2008).
- Ji, W., et al. Functional stoichiometry of the unitary calcium-release-activated calcium channel. Proceedings of the National Academy of Sciences of the United States of America. 105 (36), 13668-13673 (2008).
- Ulbrich, M. H., Isacoff, E. Y. Subunit counting in membrane-bound proteins. Nature Methods. 4 (4), 319-321 (2007).
- Sugiyama, Y., Kawabata, I., Sobue, K., Okabe, S. Determination of absolute protein numbers in single synapses by a GFP-based calibration technique. Nature Methods. 2 (9), 677-684 (2005).
- Puchner, E. M., Walter, J. M., Kasper, R., Huang, B., Lim, W. A. Counting molecules in single organelles with superresolution microscopy allows tracking of the endosome maturation trajectory. Proceedings of the National Academy of Sciences of the United States of America. 110 (40), 16015-16020 (2013).
- Grassart, A., et al. Actin and dynamin2 dynamics and interplay during clathrin-mediated endocytosis. The Journal of Cell Biology. 205 (5), 721-735 (2014).
- Leclerc, S., Arntz, Y., Taniguchi, Y. Extending Single Molecule Imaging to Proteome Analysis by Quantitation of Fluorescent Labeling Homogeneity in Complex Protein Samples. Bioconjugate chemistry. 29 (8), 2541-2549 (2018).
- Ian Freshney, R. Culture of Animal Cells: A Manual of Basic Technique and Specialized Applications. , John Wiley & Sons. (2015).
- Taniguchi, Y., et al. Quantifying E. coli proteome and transcriptome with single-molecule sensitivity in single cells. Science. 329 (5991), 533-538 (2010).
- Sternberg, Biomedical Image Processing. Computer. 16 (1), 22-34 (1983).
- Rueden, C. T., et al. ImageJ2: ImageJ for the next generation of scientific image data. BMC Bioinformatics. 18 (1), 529(2017).
- Yen, J. C., Chang, F. J., Chang, S. A new criterion for automatic multilevel thresholding. IEEE Transactions on Image Processing. 4 (3), 370-378 (1995).
- Livnah, O., Bayer, E. A., Wilchek, M., Sussman, J. L. Three-dimensional structures of avidin and the avidin-biotin complex. Proceedings of the National Academy of Sciences of the United States of America. 90 (11), 5076-5080 (1993).
- Mostovenko, E., Hassan, C., Rattke, J., Deelder, A. M., van Veelen, P. A., Palmblad, M. Comparison of peptide and protein fractionation methods in proteomics. EuPA Open Proteomics. 1, 30-37 (2013).
- Volpe, P., Eremenko-Volpe, T. Quantitative studies on cell proteins in suspension cultures. European Journal of Biochemistry. 12 (1), 195-200 (1970).
- Huang, B., et al. Counting low-copy number proteins in a single cell. Science. 315 (5808), 81-84 (2007).
- Johnson, A. C., Bowser, M. T. H. igh- High-Speed, Comprehensive, Two Dimensional Separations of Peptides and Small Molecule Biological Amines Using Capillary Electrophoresis Coupled with Micro Free Flow Electrophoresis. Analytical Chemistry. 89 (3), 1665-1673 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon