Method Article
재조합 핵심 히스톤에서 뉴 클레오 배열과 뉴 클레오 솜 위치 DNA의 조립
요약
방법은 재조합 핵심 히스톤에서 모델 뉴 클레오 어레이의 재구성 및 tandemly 반복 뉴 클레오 솜 위치의 DNA에 표시됩니다. 또한 분석에서 초 원심 침강 속도 실험, 및 원자 힘 현미경 (AFM)을 재구성 후의 뉴 클레오 배열 포화 정도를 모니터링하는 데 사용되는 방법을 설명한다.
초록
반복적 DNA 분자를 따라 간격이 핵심 히스톤 octamers는 뉴 클레오 배열이라고합니다. 뉴 클레오 배열은 두 가지 방법 중 하나를 얻을 수있다 : 생체 소스의 정제, 또는 재조합 핵심 히스톤에서 체외에서 재구성 및 tandemly 반복 뉴 클레오 솜 위치의 DNA. 후자의 방법은보다 구조적으로 균일 한 조립 및 정밀 위치 뉴 클레오 배열을 허용하는 이점을 갖고있다. 그들이 원심력 하에서 용액을 통해 이주하는 비율을 분석함으로써 고분자의 크기 및 형상에 대한 분석 초 원심 수율 정보에 침강 속도 실험. 이 기술은, 원자력 현미경과 함께 DNA 템플릿의 대다수는 재구성 후 뉴 클레오로 포화되는 것을 보장하는, 품질 관리에 사용될 수있다. 여기에서 우리는 길이 밀리그램 수량을 재구성 할 필요가 조성이 D 프로토콜을 설명염색질의 구조와 기능의 생화학 및 생물 물리학 연구에 적합한 efined 뉴 클레오 배열.
서문
진핵 생물의 게놈은 벌거 벗은 DNA가 존재하지 않고, 압축 및 결합 단백질에 의해 구성됩니다. DNA와 단백질의이 단지는 염색질로 알려져 있습니다. 염색질의 기본 반복 단위는 뉴 클레오입니다. 뉴 클레오 히스톤 옥타 약 1.6 배 1 옥타 머 히스톤 주위에 감싸 인 DNA의 146 개의 염기쌍으로 구성되어 있습니다. 히스톤 옥타는 두 개의 복사본 핵심 히스톤의 H2A, H2B, H3, H4와 각각 구성되어있다. 반복적 DNA 분자를 따라 간격이 핵심 히스톤 octamers는 뉴 클레오 배열이라고합니다. 뉴 클레오 배열의 확장 구조는 구조 "문자열에 구슬"10 나노 섬유 또는로하고, 저 염분의 조건 2에서 체외에 존재하고있다. 10 ㎚ 섬유는 배열 내부 압축 및 / 또는 인터 - 배열 올리고머 (2)를 통해 고차 구조로 집광 할 수있다. 이러한 고차 구조, 염의 존재하에 유발 될 수있다또는 뉴 클레오 배열 3,4에 염색질 건축 단백질의 결합을 통해 영향을받을 수있다. 염색질 압축의 수준은 반비례 생체 5,6의 전사 율과 상관된다. 최근의 연구는 분화, 암 발생 및 기타 7,8 같은 공정에서 게놈의 조직 구조의 중요성을 강조한다. 염색질의 구조와 기능을 연구하는 뉴 클레오 배열의 사용은 널리되고있다. 여기에서 우리는 재조합 핵심 히스톤과 뉴 클레오 솜 위치의 DNA에서 뉴 클레오 배열의 조립하는 방법을 설명합니다.
뉴 클레오 솜 위치 시퀀스의 탠덤 반복으로 재조합 DNA를 사용하여 정기적으로 간격 뉴 클레오를 포함하는 배열을 재구성 할 수 있습니다. 더 인기있는 위치 시퀀스의 두 5S rRNA 유전자 서열과 "601"시퀀스 9,10입니다. (601) 순서는 SELEX 실험 및 MOR에서 파생 된전자 강하게 5S 순서 11 이상의 뉴 클레오을 배치합니다. 따라서, (601) 어레이의 링커 DNA 길이는 더 균일하다. Tandemly 반복 뉴 클레오 솜 위치 DNA는 겔 여과 4,12함으로써 얻어진다. 재조합 히스톤은 E.에서 정제된다 변성 조건 13 세 미만 대장균. 재조합 히스톤의 사용은 하나의 정중 뉴 클레오 배열 히스톤 조성물을 제어 할 수있다. 예를 들어, 코어 (15, 16)은 야생형 코어 히스톤 대체 할 수있는 특정 변이 (14) 또는 번역 후 변형을 담지 히스톤.
침강 속도 실험인가 원심력 17 세 미만 용액에 고분자의 침전 속도를 모니터링 할 수 있습니다. 이것은 샘플에서 거대 분자의 크기와 형태에 대한 정보를 산출한다. 침강 속도 실험 따라서 염색질 섬유 용액 상태의 변경 사항을 공부에 적합한 도구입니다크로 마틴 응축 18에 의한 구조. 중요한 것은 뉴 클레오 배열 재구성의 품질 관리 단계로서 침강 속도 실험을 사용하는 것이 우선 필요하다. DNA와 뉴 클레오는 적절한 몰비 결합하지 않는 경우, 배열 또는 과다 포화 핵심 히스톤있을 수 있습니다. 따라서, 침강 속도 실험으로부터 얻은 정보는 DNA가 적절히 뉴 클레오로 포화되는 것을 보장하기 위해 사용된다. 그것은 이전에 uncharacterized DNA 템플릿 작업 특히, 뉴 클레오와 DNA의 채도를 추정하는 다른 방법을 사용하는 것이 중요합니다. 그러므로, 우리는 원자 힘 현미경 (AFM)을 사용하여 뉴 클레오 어레이 분석 방법을 설명한다. AFM은 수있는 강력한 기술이다 같은 포화 레벨, 히스톤 변형의 존재의 효과 또는 MgCl2를 19, 20의 효과와 같은 매개 변수의 개수의 효과를 가시화. AFM은 응용하고있다ED는 시간 경과 영상 (21)를 사용하여 뉴 클레오 역학을 연구한다. 시험관들이 AFM 이미징 22 적당한 크기의 범위에 속하기 때문에 뉴 클레오 12 메르 배열 AFM 연구에 특히 의무가 있습니다 조립. 본 연구에서 우리는 품질 관리뿐만 아니라 ( "보는 것이 믿는 것") AUC의 데이터를 긍정하는 수단으로 뉴 클레오 배열의 AFM을 사용했습니다. 간단한 시각화 외에도, AFM은 추가적인 메트릭과 같은 샘플의 높이 프로파일의 측정을 허용한다.
프로토콜
1. Octamers 재조합 핵심 히스톤의 조립
이유 : 뉴 클레오 어레이 재구성의 첫 번째 단계는 동결 건조 된 재조합 코어 히스톤으로부터 네이티브 코어 히스톤 octamers을 제조하는 공정이다. 히스톤 단백질은 동일한 몰 양의 결합 버퍼를 재 접힘에 변성 버퍼 중 샘플을 투석에 의해 히스톤 octamers로 조립된다.
- 정화 (13)을 설명 된대로 (H2A, H2B, H3, H4) 재조합 핵심 히스톤을 냉동 건조.
- 전개 버퍼 (6 M 구 아니 염산, 20 MM 트리스 산도 7.5, 5 mM의 DTT)의 3 ㎖에 각 동결 건조의 핵심 히스톤의 약 5 밀리그램을 녹입니다. 각 나누어지는 적어도 한 시간 동안 분해 할 수 있습니다. 어떤 단백질이 용기의 측면에 남아 있는지 확인합니다.
- 참고로 전개 버퍼를 사용하여 276 nm에서 각 히스톤의 흡광도를 측정한다.
- 그들의 흡광 계수를 사용하여 각 히스톤의 몰 농도를 계산한다 (표 1 참조 Xenopus의 히스톤 흡광 계수의 경우). 단백질의 대부분이 용해되면 동결 건조 중량 및 추정에 히스톤의 흡광도 결정된 농도를 비교한다.
- 이 모든 히스톤에 대한 몰수의 제한 수있을 것입니다, 당신이 가장 적은 몰수이있는 히스톤 결정합니다.
- 동일한 몰 양으로 히스톤을 결합하고 1 ㎎ / ㎖의 최종 농도로 버퍼를 전개로 희석.
- 6-8 kDa의 MWCO 투석 튜브에 샘플을 놓습니다. 튜브를 밀봉하고 차가운 폴딩 버퍼의 2 L에 배치 (2 M의 NaCl, 10 MM 트리스 산도 7.5, 1 ㎜ EDTA, 5 MM의 β-머 캅토 에탄올).
- 교반하면서 4 ° C에서 18 시간 동안 샘플을 Dialyze. 확인 투석 튜브 자유롭게 적극적으로 회전 할 수있다, 또는 다른 히스톤이 침전하는 경우가 있습니다.
- 두 번 18 시간 동안 (완료 될 때까지 즉, 매 6 시간에 대해 폴딩 버퍼를 변경)의 투석 버퍼를 변경합니다. 침전물이 형성된 경우에도 폐기하지수율이 감소 하겠지만 샘플 일부 옥타 머를 복구 할 수 있습니다.
참고 : 다음 일에 대한 열 평형을 준비하는 단계 2.1-2.2를 참조하십시오.
2. 크기 배제 크로마토 그래피로 정제하여 히스톤 Octamers
이유 : 섹션 1과 체결 한 후, 샘플 히스톤 octamers뿐만 아니라 집계, H3/H4의 테트라, 그리고 H2A/H2B 이량 체와 같은 다른 히스톤 복합체를 포함합니다. 히스톤 octamers 멀리 크기 배제 크로마토 그래피 (SEC)를 사용하여 다른 착물로부터 정제한다.
- HiLoad FPLC 시스템에 60분의 16 Superdex200 60분의 16 (S200) 열을 연결합니다. 기포가 시스템에 입력하지 않았는지 확인합니다.
- 0.2 μm의 여과 물 S200 열을 청소합니다. 0.3 ㎖ / 분의 유속을 사용하여 0.5 ㎫의 배압 한도를 설정. 당신은 충분한 물이 있는지 확인하고, 열이 밤새 청소 할 수 있습니다.
- F의 S200의 열 및 샘플 루프를 평형PLC는 버퍼를 사용하여 리 폴딩. 0.2 μm의 여과 폴딩 버퍼의 1 L를 사용합니다. / 분으로 1 ㎖의 비율로 약 2 시간 동안 0.5 MPa의 최대 압력에서 칼럼을 통해 완충액을 폴딩 흐름.
- 리 폴딩 완충액을 1 ㎖로 원심 Vivaspin 농축기 (50 kDa의 MWCO)를 평형. 투석 튜브에서 샘플을 제거하고 침전물을 제거하기 위해 4 ° C에서 샘플을 원심 분리기. 집중에 샘플 상층 액을 피펫. 약 500 μL의 부피로 샘플을 집중한다.
- 새 컨테이너에 집중 옥타 머 샘플을 제거하고 1 ㎖는 버퍼를 재 접힘과 집중을 씻어. 500 μL에 린스를 집중하고, 옥타 머 샘플에 추가합니다. 이것은 집중에 남아있는 옥타 머를 구제하는 데 도움이됩니다.
- 4 ℃에서 10,000 rpm에서 5 분 동안 1.5 ML의 미세 원심 분리 튜브에 샘플을 스핀 상층 액을 수집하고 새로운 튜브로 전송할 수 있습니다. 이 FPL의 샘플을로드하기 전에 침전물을 제거하는 데 도움이C.
- S200 칼럼 상에 샘플을로드합니다. 하나의 정제에 1.5 ㎖의 전체 볼륨 또는 15 밀리그램의 옥타 머의 최대로드합니다.
- 갓 만든 리 폴딩 완충액을 사용하여 샘플을 용출. 1 ㎖ / 분의 유속 및 0.5 MPa의 배압 제한을 사용한다. 버퍼를 폴딩 두 열 볼륨 (400 ㎖)에 사용하고 2 ㎖ 분수를 수집합니다. 용출 동안 280 nm에서 흡광도를 모니터링합니다. 다른 종의 크기가 감소하는 순서로 용출됩니다. 히스톤 집계는 보통 약 84 ㎖ (그림 1)에서 약 45 ㎖, 옥타 약 65 ml로하고, 이량 체를 용출.
- 18~20% SDS-PAGE에서 샘플을 실행하여 그 봉우리에서 용출 분수를 분석합니다. 정제 octamers을 포함하는 분획을 네 가지 핵심 히스톤 단백질의 동일 몰 양의 (그림 1)이 있어야합니다.
- 정제 octamers를 포함하는 분수 수영장, Vivaspin 원심 필터를 15 ㎎ / ㎖를 ≤에 집중한다. 희석을두면, octamers는 끊을 수 있습니다 In을 H2A/H2B 이합체와 H3/H4의 사량.
- 280 nm에서 흡광도를 측정함으로써 옥타 머의 농도를 측정하고 표 1에서 흡광 계수를 사용하여 계산한다.
- 히스톤 octamers 4 ℃에서 접힘 버퍼에 짧은 기간 유지 될 수있다 장기적으로 저장되는 경우, octamers는 -20 ° C에서와 50 % 글리세롤 용액에 더 안정 될 것입니다. 글리세롤에 저장 Octamers 흡광도 측정 전에 신선한 폴딩 버퍼에 투석과 뉴 클레오 배열 reconstitutions에 사용되어야한다.
3. DNA에서 뉴 클레오 배열 및 정화 Octamers의 재구성
이유 : 뉴 클레오 배열에서 재구성 히스톤 octamers 및 템플릿 DNA가 특정의 몰 비율로 결합해야합니다. 2 M 염화나트륨의 DNA와 히스톤 octamers의 혼합물은 이온 강도가 감소하는 버퍼에 투석 단계입니다. 낮은 소금에 점차적 인 변화는 적절한 뉴 클레오 솜 형성을 보장합니다. N 얻기템플릿 DNA의 히스톤 옥타 머 포화 원하는 수준 ucleosomal 배열은 대규모 예비 재구성 다음에 작은 규모의 시험 reconstitutions가 필요합니다. 작은 규모의 reconstitutions에 의해 정의 된 적절한 조건은 예비 재구성을 안내하는 데 사용됩니다.
- 4,12 바와 같이 tandemly 반복 뉴 클레오 솜 위치 DNA를 정제 하였다. 간단히, Qiagen 사 GigaPrep 키트 또는 유사한 제품을 이용하여 플라스미드 DNA를 분리. 셋업 제한 효소 템플릿 DNA를 분리하고 작은 크기 (≤의 약 700 bp)의 플라스미드 DNA를 소화하기 위해 소화를. 템플릿 DNA는 멀리 크기 배제 크로마토 그래피 (SEC)를 사용하여 작은 플라스미드 DNA 잔재로부터 정제 할 수있다. 중력이 세파 크릴 S-1000의 구슬로 포장 약 115 cm의 높이, 열 공급은 601 207bp X 12mer의 DNA의 정화에 충분하다.
참고 : 플라스미드 DNA의 감소 비용으로 정화하지만, 증가 EFF를 위해ORT, 하나는 E.부터 플라스미드 DNA를 분리하기 위해, 페놀 / 클로로포름 DNA 정제하여 알칼리 용해를 사용 대장균 (23, 24). 플라스미드 DNA의 주형 DNA를 분리하기위한 전략 템플릿 DNA의 크기의 변화에 따라 달라질 수 있습니다. 삼성 전자의 경우는 제한 효소 템플릿 및 플라스미드 DNA 사이의 크기의 차이를 최대화하는 방식으로 소화를 설정하는 것이 중요합니다.
- 몰비 (R)은 reconstitutions에 사용할 판별. r은 반복의 DNA 몰에 옥타 머의 몰 비율이 동일하다. 의도가 포화 뉴 클레오 배열을 취득하는 경우 초기 작은 규모의 실험을 위해, 0.9, 1, 1.1의 R 값이 적합합니다.
- 테스트 할 각각의 몰비 약 18 μg의 DNA에 추가 옥타 머의 양을 계산함으로써 소규모의 샘플을 준비한다. DNA와 히스톤 octamers을 섞는다. 샘플의 NaCl의 최종 농도는 2 ≥ M을, 그리고 D의 최종 농도되어야NA는 약 0.3 ㎍ / ㎕를해야한다. 최종 샘플 버퍼 조건은 10 MM 트리스 산도 7.8, 1 mM의 EDTA를 포함해야합니다.
- 샘플은 현재 투석을위한 준비가되어 있습니다. 12K-14K MWCO 투석 튜브에 샘플을로드합니다. 다음과 같이 이온 강도가 감소하는 버퍼의 2L에 대해 샘플을 Dialyze.
10 MM 트리스 산도 7.8, 1 ㎜ EDTA : 모든 버퍼의 구성 요소. 버퍼 1 ~ 6 시간 동안 (1 M의 NaCl, 1 mM의 DTT 추가). 버퍼 2 하룻밤 (825 M의 NaCl, 1 mM의 DTT 추가). 버퍼 3 ~ 6 시간 동안 (2.5 밀리미터의 NaCl, 1 mM의 DTT 추가). 버퍼 4 하룻밤 (2.5 mM의 염화나트륨, 0.1 ㎜ PMSF 추가).
- 투석 튜브에서 샘플을 제거하고 섹션 4-6를 진행합니다. 소량의 샘플은 원하는 결과를 산출하는 경우, 대규모 섹션 3-6의 단계를 반복한다.
주 : 자원을 보존하기 위해, 소규모 샘플 하류 스크리닝 분석법을 위해 충분하기 위해 필요한 최소 체적을 가져야한다. E가하기 위해서는계속하지 0.3 ㎎ / ㎖ DNA에 여기에 나열된 침강 속도와 AFM 실험, 50 ㎕의 샘플에 대한 샘플이 적합합니다. 큰 규모의 크기는, 분취 샘플들은 의도 된 사용에 의존한다. 대규모 샘플은 작은 규모의 샘플과 동일한 방식으로 제조한다.
4. 재구성 된 뉴 클레오 배열의 침강 속도 분석
이유 : 용액에서 분자의 크기와 모양에 베크 XL-A / I 분석 초원 심 분리기 수익률 정보에 침강 속도 실험. 저염 조건에서 수행 침강 속도 실험을 재구성 배열이 뉴 클레오으로 포화되는 범위를 결정하는 데 사용됩니다.
- TEN 버퍼 (10 MM 트리스 산도 7.8, 1 ㎜ EDTA, 2.5 mM의 염화나트륨)를 사용하여 260 nm에서 0.5 정도 흡수에 샘플을 희석. 한쪽 샘플과 referen으로, 두 부문 중심으로 셀에 시료 400 μl를로드다른 25 CE (TEN 버퍼). (즉, 420 ㎕를 참조 솔루션을 추가) 샘플 메 니스 커스 (meniscus)보다 더 높은 기준 메 니스 커스 (meniscus)를 유지합니다.
- 로터에 세포를 넣고 적절히 세포와 로터 (25)의 하단에 해시 마크를 이용하여 셀을 정렬. 부드럽게 압축 공기를 이용하여 세포의 렌즈 먼지.
- , XL-A/XL-I 원심 분리기에 돌려 로터를 삽입하고, 연결하고 사용 설명서에 설명 된 바와 같이, 광학 장치를 고정합니다. 새로운 : 프로테옴 연구소 소프트웨어 선택 파일을 엽니 다.
- 3,000 rpm으로 25 ℃의 온도에서 단일 스캔 실행을 설정 옵션에서 선택 마지막 스캔 후 중지 및 실행 반경 보정 (로터가 AUC에 사용 된 마지막 회 인 경우 반경 보정이 필요하지 않습니다).
- 각 셀의 경우, 샘플의 이름을 파장 (260 나노 미터)을 선택, 흡광도를 선택하고 컴퓨터에서 파일을 저장할 위치를 선택합니다. 단일 스캔 실행을 시작합니다.
- 적절한 RA를 결정하는 하나의 스캔을 사용하여스캔 길이 (그림 2A)를 다이얼. 스캔 할 셀의 길이를 줄이면 실행 시간을 감소시킵니다. 그러나, 샘플 메 니스 커스 (meniscus)를 포함하고 매우 가깝거나 셀의 하단에 끝을 확장해야합니다.
참고 : 싱글 스캔이 측정 영역은 단지 샘플 메 니스 커스 (도 2A) 전에 시작하여 측정 줄어드는 것을 허용한다. AUC를 실행하는 동안 생성 된 검사의 대부분은 메 니스 커스 (meniscus)뿐만 아니라 안정적인 고원 (그림 2B) 지난 경계 분수가 있어야합니다. AUC 세포에 더러운 렌즈가 큰 스파이크 스캔을 생성 할 수있는 데이터의 분석에 영향을 미칠 수있다. 기능 UltraScan 소프트웨어는 분석 초 원심 데이터 (26)의 분석에 관한 정보를위한 좋은 자원이다 설명서가 포함되어 있습니다.
- XL-A/XL-I 제어 패널을 사용하여, 0의 속도와 키를 눌러 시동을 입력합니다. 이 진공을 모두 끌어 원심 분리기 챔버를 묻는 메시지가 표시됩니다아야 챔버의 온도는 평형. 실행을 시작하기 전에 온도 평형 수 있도록 1 시간을 기다립니다.
- 원하는 속도 및 스캔의 수에 대한 실행까지를 설정하는 소프트웨어를 사용합니다. 높은 속도는 해상도를 증가하지만, 샘플 셀의 바닥에 침전되기 전에 하나는 적어도 20 스캔 (바람직하게는 그 이상)를 수집한다. 그것은 실행의 진행 상황을 모니터링하고 누출 세포와 같은 잠재적 인 문제를 확인하기 위해 (옵션 메뉴에서 수행) 지난 몇 검사를 오버레이하는 것이 편리하다. 올바른 작동을 보장하기 위해 검사의 첫 번째 커플을 모니터링합니다.
5. 침강 속도 데이터를 편집 및 분석
이유 : 원시 침강 속도 데이터는 확산 보정 침강 계수 분포를 산출 프로그램에 의해 분석한다. 차례로이 정보는 관련 포화 레벨을 포함하는 재구성 된 샘플의 일부를 나타내고, 예를 들면 12 메르 DNA 템플릿을 사용하는 경우는 10, 11, 12 뉴 클레오 / DNA를 가지고 재구성 된 배열의 분획을 결정할 수있을 것이다.
- 데이터 26 편집 등의 UltraScan로 AUC 데이터 분석 소프트웨어를 사용합니다. 스캔 데이터의 편집은 각각의 셀에 대해 수행 및 후속 분석을위한 데이터 세트를 생성해야합니다. 스캔의 적절한 편집, 샘플 메 니스 커스를 정의하는 관심 영역에 데이터를 감소시키고 기준뿐만 아니라 고원 영역 (도 2)를 정의하는 것을 포함한다. 원하지 않는 스캔의이 시점에서 데이터 세트로부터 제거 될 수있다. 이 분석에서 불필요한 검사를 제거하는 것이 가능하기 때문에, 그것은 모든 스캔이이 시점에서 유지하는 것을 추천합니다.
- 우리는 강력하게 강화 된 반 Holde-Weischet 방법은 데이터 27를 분석하는 데 사용하는 것이 좋습니다. 이 분석 방법은 런의 과정을 통해 확산의 효과를 보정하고, 침강 계수의 적분 분포를 산출한다. 특히, 강화 된 반 Holde-Weischet 방법 (의 UltraScan에서 구현) 안정 고원 부족, 또는 분석 (28)에 메 니스 커스 (meniscus)을 삭제하지 않은 경계 분수를 포함하는 검사의 포함을 허용합니다.
- 조립 배열의 침강 계수의 분포를 결정합니다. 601-12 (207bp, 12 메르) 뉴 클레오 배열의 대표 결과는 그림 3에 나와 있습니다.
- 작은 규모의 샘플 중 하나가 침강 계수의 원하는 범위의 배열이 포함되는 경우, 제 3 항부터 증가 규모 과정을 반복합니다. 작은 규모의 샘플 중에 침강 계수의 적절한 분배가없는 경우 다음 섹션 3에서 시작하는 새로운 연구 범위와 작은 규모의 테스트를 반복합니다.
6. 원자 힘 현미경을 사용하여 뉴 클레오 배열을 시각화 (AFM)
이유 : AFM은 수 nucleosoma의 포화 수준의 시각화L 배열. 이 기술은 품질 관리 분석으로 AUC 및 제한 효소 소화에 의한 침강 속도를 보완한다.
- 전술 한 방법을 사용하여, 잘 포화 뉴 클레오 어레이 (도 5a)을 얻었다.
- 준비 APTES가 배치하여 운모 슬라이드를 처리 ~ 30 ㎕를 30 분 동안 0.22 μm의 여과 nanopure 물에 상용 (시그마 - 알드리치 (3 - 아미노 프로필) 트리에 톡시 실란의 A3648-100ML)의 1:1,000 희석.
- 30 분 후 여과 물 APTES를 씻어 부드럽게 질소 흐름을 건조.
- 슬라이드에 적절히 희석 뉴 클레오 배열 샘플 (~ 1.5 NG / μL)를 놓고 15 분 동안 실온에서 해당 품어.
- 필터링 된 샘플 버퍼와 오프 샘플링 이전 건조하고, AFM (이 경우 정신 병원 연구 MFP - 3D 원자 힘 현미경)의 무대에 장소 슬라이드 씻어.
- 512 스캔 라인을 2 × 2 μm의 스캔 이미징에 의해 시작합니다. SA의 수준을 확인하는 뉴 클레오 솜의 수를 계산turation (그림 5B).
- 이상적으로해야 잘 분리 된 뉴 클레오 배열과 슬라이드 이미지 영역.
- 높은 해상도 이미지의 경우 500 × 500 nm의 스캔 (그림 5C)를 얻을 수있다.
- 이미지는 평탄화 및 정신 병원에서 제공 MFP-3D 소프트웨어를 사용하여 분석 할 수있다.
- 각각의 이미지는 네 개의 사분면으로 나누어 뉴 클레오 (그림 5C, 오른쪽 패널 높이 추적에 대해받은 배열과 높이 프로파일을 그릴 수있는 잘 분리 배열과 여러 무료 손으로 라인을 명확하게보기를 얻기 위해 디지털 방식으로 확대 할 수 있습니다 및 (d)).
- 몇몇 그러한 이미지는 뉴 클레오 수백 포괄 어레이의 각 유형에 대해 분석한다. 높이 프로파일은, 기록 분류 및 MS Excel에서 그려집니다.
결과
프로토콜을 설명하기 위해 우리는 12 직렬의 601 위치 시퀀스 (601 207 × 12)의 207 bp의 반복으로 이루어진 재조합 Xenopus의 핵심 히스톤과 DNA에서 뉴 클레오 배열을 재구성. 우리는 먼저 동결 건조의 핵심 히스톤에서 네이티브 octamers을 조립 한 후 S200 열 (그림 1A)를 사용하여 FPLC에 의해 octamers을 정제. 큰 단지는 S200 열에서 이전 용출. 히스톤은 일반적으로 다음 순서로 용출 : 비 특이 히스톤 집계, 히스톤 옥타 머, H3/H4 량체 및 H2A/H2B 이합체 (그림 1A). S200 열에서 피크를 SDS-PAGE에 의해 분석 하였다. 정제 된 옥타 머 분획의 겔은 네 히스톤 단백질 (도 1b)의 몰량을 표시한다. Xenopus의 H2A와 H2B는 약 200 다에 따라 다릅니다 따라서 그들은 SDS 겔에 하나의 밴드로 표시 분자량이 있습니다. H3/H4의 테트라와 H2A/H2B 이량 체는 버전 후 용출 것이다, 그러나히스톤 옥타 머 피크의 Y 부근에 있습니다. 유지하는 분획을 선택하는 경우는 크로마토 그램의 피크 옥타 머의 단부를 향해 나오는 분획을 폐기 현명하다. 이 분수는 비 octameric 히스톤 복합체의 양이 증가 할 수 있습니다. 이 경우 64 ~ 67는 뉴 클레오 배열 재구성 (그림 1)를위한 풀링 및 저장된 분수.
의 UltraScan 내에서 강화 된 반 Holde-Weischet 분석에서 얻은 침전 계수의 확산 보정 배포를 보는 두 가지 방법이 있습니다. 빨간색 선은 침강 계수의 적분 분포를 나타냅니다. 그래프상의 임의의 주어진 포인트를 들어, y 축은 동일하거나 또는 x-축에 나타낸 값보다 적은 침강 계수를 갖는 샘플의 일부분을 나타낸다. 이종 샘플이 양의 기울기를 가진 곡선을 가지고 반면 따라서, 수직선은 동질 샘플 나타낸다. 녹색 선은 드입니다적분 분포 이로부터 파생 된. 피크 아래의 면적은 그 침강 계수를 갖는 샘플의 일부분에 비례한다. 완전히 포화 601 207 X 12 뉴 클레오 배열은 29S (29)의 침강 계수가있다. 도 3에 도시 된 샘플의 경우, 데이터는 어레이의 약 70 %가 포화하고 30 % 포화 위에있는 것을 나타낸다.
원자 힘 현미경은 체외에서 염색질 조직을 연구하는 인기있는 보완적인 접근 방식에 새로운 기술에서왔다. 여기에 우리가 재구성 한 후 템플릿 채도의 범위를 설정하는 분석 초 원심의 옆에 AFM을 사용했습니다. 그림 6에서 영상에 사용되는 ~ 27S 배열 (그림 4A)의 대부분은 AFM과 AUC 결과 사이 우수한 계약을 시연, 10-11 뉴 클레오 솜 (그림 4B)을 포함. 여기에서 얻어진 AFM 결과 따라서이 유효성을 확인접근과 뉴 클레오 배열의 특성에 대한 신뢰할 수있는 기술합니다.
| 히스톤 단백질 | Σ276, 펼쳐진 단백질 cm -1 M -1 | 분자량 다 |
| H2A | 4,350 | 13,960 |
| H2B | 7,250 | 13774 |
| H3 | 4,640 | 15,273 |
| H4 | 5,800 | 11,236 |
| 옥타 머 | 44,080 | 108486 |
표 1. 소멸 계수와는 Xenopus laevis의 핵심 히스톤과 renatured 히스톤 옥타 머의 분자량.
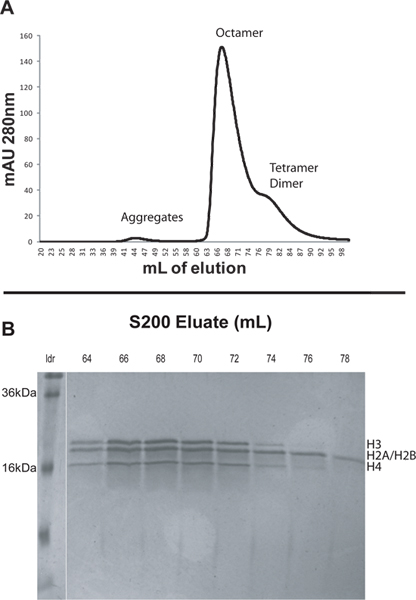
그림 1. 2 절에서 설명 된대로 S200 열에서 A. 용출 프로파일. 약 44 ㎖의에서 작은 피크가 큰 히스톤의 집계에 의한 것입니다. 67 ML의 저명한 피크는 히스톤 octamers입니다. 76-90 ML의 넓은 어깨는 H3/H4의 테트라와 H2A/H2B 이합체 S200 열에서 선택된 분수. B. 20 % SDS-PAGE가 포함되어 있습니다. 그들은 비 octameric 히스톤 복합체에 의해 오염 될 가능성이 가장 마찬가지로 옥타 머 피크 조기 분수를 수집해야합니다. 이 경우 64 ~ 67가 풀링 및 뉴 클레오 배열 재구성에 저장된 분수. 큰 그림을 보려면 여기를 클릭하십시오 .

그림 2. 라 3,000 rpm에서 수집 한 검사 A. ProteomeLab 소프트웨어 화면 캡처 (BELS) 추가. 샘플이 낮은 속도로 침전물을하지 않습니다. 단일 스캔은 AUC 세포 검사에 대한 측정 범위 (단계 4.6)의 UltraScan 프로그램 (추가 레이블) 26를 사용하여 얻은 침강 속도 검사의 일련의. B. 화면 캡처를 설정하는 데 사용됩니다. 검사의이 시리즈는 분석을위한 데이터 세트 (단계 5.1 참조)를 생성하기 위해 편집됩니다.
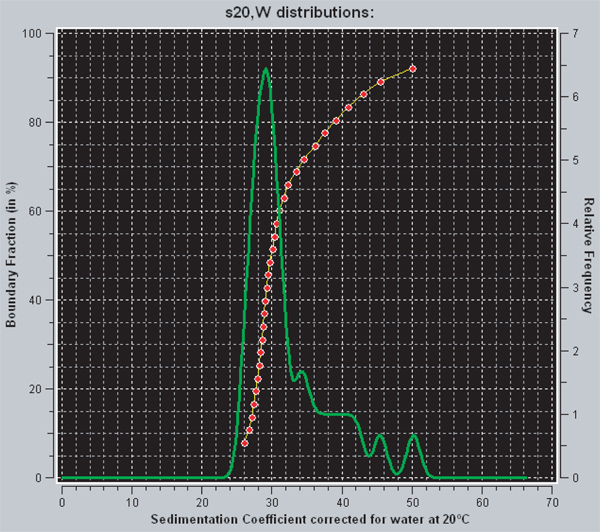
그림 3. UltraScanII에서 화면 캡처. 빨간색과 녹색 라인의 UltraScan에 침전 계수의 분포를 볼 수있는 두 가지 방법이 있습니다. 녹색 유도체 동안 적색 라인은, 침강 계수의 정수 분포이다. 해석에 관한 추가 정보는 결과 섹션에서 찾을 수 있습니다.
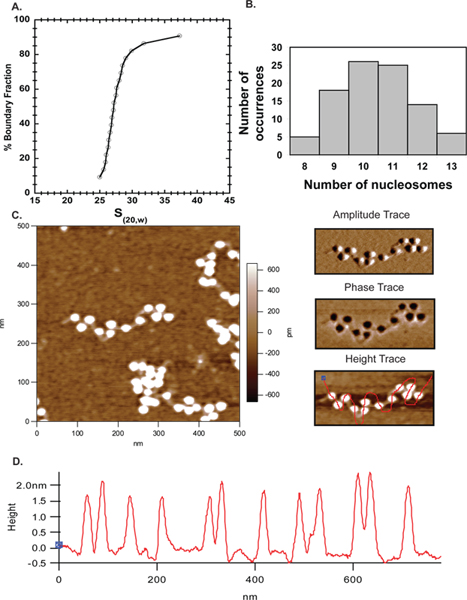
그림 4. AFM. 평균 침강 계수를 나타내는 마우스 옥타 머 조립 (601) 207 × 12 어레이에 대한 A. 침강 속도 프로파일은 27S입니다. B.이 동일 어레이는 AFM에 의해 촬영되었고, 여러 이미지에서 계산 된 뉴 클레오 솜의 수는 MS Excel에서 그려졌다. 줄거리는 배열의 대부분은. C. 500 × 500 링커 DNA 601 207 X 12 뉴 클레오 배열의 나노 스캔 명확하게 볼 수 AUC 데이터를 확증 10-11 뉴 클레오 있다고 지적했다. 오른쪽 상단 패널은 진폭 추적하고 중간 오른쪽 패널은 C에 나타낸 배열의 위상 추적이다. 오른쪽 하단 패널은 왼쪽에 있지만, 뉴 클레오 솜의 높이 프로파일을 결정하는 뉴 클레오을 그린 무료 손으로 라인과 같이 같은 높이 추적하다의. 위의 이미지에서 뉴 클레오 솜의 D. 높이 프로파일은 이전에 (40)을보고 모든 뉴 클레오는 1.5 ~ 2.5 nm의 높이 범위입니다 것을 보여줍니다.
토론
모델 뉴 클레오 어레이는 염색질 구조와 기능의 시험 관내 연구에 매우 유용한 도구이다. 예를 들어, 널리 용액 30-34에서 염색질 섬유 축합 메카니즘을 연구하는 데 사용하고, 그것이 가능 tetranucleosome (35)의 x-선 구조를 구하는되었습니다. 최근에 그들은 특정 핵심 히스톤 변형, 돌연변이 전사 후 수정 14-16,36의 구조적 효과를 해독에 유용 입증했다. 여기에서 우리는 뉴 클레오 솜 위치 DNA 재조합 핵심 히스톤에서 모델 뉴 클레오 배열의 어셈블리에 대한 일반적인 방법을 설명합니다.
정제 octamers 601 207 X 12 DNA에서 뉴 클레오 배열의 재구성은 순차적으로 2 M 2.5 밀리미터에서의 NaCl 농도를 낮추는 여러 가지 투석 단계를 포함하는, 간단합니다. 프로토콜의 가장 어려운 부분은 원하는 TEM을 산출 r 값을 사용하는 것이다판 포화 수준. 적절한 R-값은 우선 작은 규모에 경험적으로 결정 후 실험에 사용 뉴 클레오 배열을 생성하는 더 큰 규모로 반복된다. 우리의 경우, 우리는 주로 DNA 당 12 뉴 클레오 포화 (601) 207 × 12 DNA 템플릿을 얻기 위해 시도되었다. 완전 포화 601 207 X 12 뉴 클레오 배열의 침강 계수는 29 S입니다 ~ 27S 29에서 DNA 침전물 당 11 뉴 클레오 솜과 동일한 DNA 템플릿있다. 따라서, 분석 초 원심 분리기에서 침강 속도는 재구성 후 뉴 클레오 포화 레벨을 결정하는 매우 민감한 방법을 제공한다. 우리는 확산 보정 침강 계수 분포를 산출 강화 반 Holde-Weischet 방법을 사용하여 우리의 데이터를 분석했다. 균질하거나 이질적 샘플 재구성 후에 얼마나 하나를 알려주기 때문에이 정보는 필수적이다. 즉, 601 (207) 12 X DNA 템플릿, 그것을도 3은, 향상된 밴 1.1의 R에서 재구성 601 207 X 12 뉴 클레오 배열 Holde-Weischet 분석의 결과를 나타낸다 DNA 당 12 뉴 클레오, DNA 당 11 뉴 클레오 등을 가지고 시료의 분율을 나타내는이 대략 배열의 30 %는 포화에 있습니다. 샘플의 유틸리티는 일반적으로 포화 배열의 비율에 묶여 있지만, 일부 응용 프로그램은 매우 동질 및 / 또는 포화 배열을 요구할 수있다 안으로 배열이 사용되는 실험에 의존하고 있습니다. 다수의 방법은 뉴 클레오 배열의 균질성 및 채도를 향상시키기 위해 존재한다.
이 경우 과포화 어레이는 MgCl2를 (37)의 첨가시 선택적 침전에 의해 제거 될 수있다. 더 균질 한 배열로는 자당 구배 원심 분리, 분취 겔 전기 영동, 이온 교환 크로마토 그래피를 사용 10,13 시료를 정제함으로써 얻을 수있다. 수정 된 뉴cleosomal 배열 재구성 방법은 소금 투석 (38, 39)를 재구성하기 전에 샘플로 짧은 경쟁 DNA를 추가 호출합니다. 이것은 하나 이상의 DNA 템플릿을 포화하지 않고 히스톤 옥타 머의 과잉과 뉴 클레오 배열을 조립 할 수 있습니다. 뉴 클레오 배열 가능성 경쟁자 DNA 및 여분 히스톤의 제거를위한 정제 공정을 필요로 할 것이다 경쟁자 DNA를 사용하여 재구성.
비록 소규모의 파일럿 배열 어셈블리 후, 대규모로 재구성 된 뉴 클레오 배열 제대로 포화되지 않을 수있다. 샘플 템플릿 DNA와 히스톤 옥타 낭비를 방지하기 위해, 위에 또는 포화 배열 하에서 고정 할 수있다. 어레이 포화 위에있는 경우, 여분의 DNA 샘플에 첨가 될 수있다. 어레이 포화 미만인 경우, 여분 옥타 정정에 추가 될 수있다. 그러나, 이미 저 염분으로 투석 배열에 옥타 머를 추가하는 것은 해리 옥타 머 발생할 수 있습니다. 여분의 O를 추가하는 경우ctamer 또는 DNA는, 배열 벌크 샘플에 추가 옥타 또는 DNA를 추가하기 전에 다시 2 M의 NaCl에 배열을 dialyze합니다. 3.4 단계에 따라 낮은 소금 높은에서 고정 된 샘플 단계 투석을 반복합니다. 어레이를 정정하기 위해 추가 옥타 머의 양은 포화 어레이 (29) 아래에 대한 침강 계수들로부터 추정 될 수있다. 뉴 클레오 어레이의 샘플을 조정 한 후, 포화 레벨의 측정이 다시 이루어져야한다.
AFM은 뉴 클레오 배열을 특성화하기위한 강력한 방법이지만, 그것은 복잡하고 노동 집약적 인 과정입니다. 이것은 작은 규모의 reconstitutions를 선별 가난한 기술,하지만 대규모 샘플의 특성과 염색질 조직의 연구를위한 우수한 기술한다. 이전에 우리는 "하이퍼 응답"도 낮은의 MgCl2 농도로했다 macroH2A 삭제 구조에 의해 생성 된 "매크로"입자를 시각화하기 위해 AFM을 사용했습니다. LikewiSE는, 몬텔 등. (2009) H2A BBD의 변형 뉴 클레오 배열 야생 형식 배열에 비해 더 "열기"로 발생하는 것으로 나타났습니다. 따라서, AFM은 신뢰할 수있는 품질 제어를위한 기술뿐만 아니라, 일반적으로 염색질 섬유 구조의 연구이다.
공개
저자는 이익의 충돌이 없습니다.
감사의 말
이 작품은 NIH 보조금 GM45916 및 GM66834 JCH 및이 작품을 AK하는 국제 레트 증후군 재단의 화목에 의해 지원되었다는 NIH가 GM088409와 하워드 휴즈 의학 연구소에게 KL 공헌을 부여에 의해 지원되었다
자료
| Name | Company | Catalog Number | Comments |
| (3-Aminopropyl)triethoxysilane | Sigma-Aldrich | A3648-100ML | |
| 6-8 kDa MWCO Dialysis Tubing | Fisher | 21-152-5 | |
| HiLoad Superdex 200 16/60 Column | GE | 17-1069-01 | |
| Vivaspin 50 kDa MWCO Centrifugal Concentrator | Sartorius | VS2031 | |
| 12-14 kDa MWCO Dialysis Tubing | Fisher | 08-667A | |
| Illustra Sephacryl S-1000 Superfine | GE | 17-0476-01 | |
| XL-A/I Analytical Ultracentrifuge | Beckman-Coulter |
참고문헌
- Luger, K., Mader, A., Richmond, R., Crystal Sargent, D. structure of the nucleosome core particle at 2.8 A resolution. Nature. 7, (1997).
- Hansen, J. C. Conformational dynamics of the chromatin fiber in solution: determinants, mechanisms, and functions. Annual Review of Biophysics and Biomolecular Structure. 31, 361-392 (2002).
- McBryant, S., Adams, V., Hansen, J. Chromatin architectural proteins. Chromosome Research. 14 (1), 39-51 (2006).
- Hansen, J. C., Ausio, J., Stanik, V. H., van Holde, K. E. Homogeneous reconstituted oligonucleosomes, evidence for salt-dependent folding in the absence of histone H1. Biochemistry. 28 (23), 9129-9136 (1989).
- Szerlong, H. J., Prenni, J. E., Nyborg, J. K., Hansen, J. C. Activator-dependent p300 acetylation of chromatin in vitro: enhancement of transcription by disruption of repressive nucleosome-nucleosome interactions. The Journal of Biological Chemistry. 285 (42), 31954-31964 (2010).
- Cirillo, L. A., Lin, F. R., Cuesta, I., Friedman, D., Jarnik, M., Zaret, K. S. Opening of compacted chromatin by early developmental transcription factors HNF3 (FoxA) and GATA-4. Molecular Cell. 9 (2), 279-289 (2002).
- Nguyen, C., Gonzales, F. chromatin structure associated with methylation-induced gene silencing in cancer cells: correlation of accessibility, methylation, MeCP2 binding and acetylation. Nucleic Acids Research. 29 (22), 4598-4606 (2001).
- Cuesta, I., Zaret, K. S., Santisteban, P. The forkhead factor FoxE1 binds to the thyroperoxidase promoter during thyroid cell differentiation and modifies compacted chromatin structure. Molecular and Cellular Biology. 27 (20), 7302-7314 (2007).
- Simpson, R. T., Stafford, D. W. Structural features of a phased nucleosome core particle. Proceedings of the National Academy of Sciences of the U S A. 80 (1), 51-55 (1983).
- Lowary, P. T., Widom, J. New DNA sequence rules for high affinity binding to histone octamer and sequence-directed nucleosome positioning. Journal of Molecular Biology. 276 (1), 19-42 (1998).
- Lowary, P., Widlund, H., Cao, H. Sequence motifs and free energies of selected natural and non-natural nucleosome positioning DNA sequences. Journal of Molecular Biology. 288, 213-229 (1999).
- Gordon, F., Luger, K., Hansen, J. C. The Core Histone N-terminal Tail Domains Function Independently and Additively during Salt-dependent Oligomerization of Nucleosomal Arrays *. The Journal of Biological Chemistry. 280 (40), 33701-33706 (2005).
- Luger, K., Rechsteiner, T. J., Richmond, T. J. Expression and purification of recombinant histones and nucleosome reconstitution. Methods in Molecular Biology (Clifton, N.J.). 119 (4), 1-16 (1999).
- McBryant, S. J., Klonoski, J., et al. Determinants of histone H4 N-terminal domain function during nucleosomal array oligomerization: roles of amino acid sequence, domain length, and charge density. The Journal of Biological Chemistry. 284 (25), 16716-16722 (2009).
- Ma Shogren-Knaak, ., Fry, C. J., Peterson, C. L. A native peptide ligation strategy for deciphering nucleosomal histone modifications. The Journal of Biological Chemistry. 278 (18), 15744-158 (2003).
- Lu, X., Simon, M. The effect of H3K79 dimethylation and H4K20 trimethylation on nucleosome and chromatin structure. Nat Struct Mol Biol. 15 (10), 1122-1124 (2008).
- Ausio, J. Analytical Ultracentrifugation for the Analysis of Chromatin Structure. Biophysical Chemistry. 86 (2-3), 141-153 (2000).
- Hansen, J., Kreider, J., Demeler, B., Fletcher, T. Analytical ultracentrifugation and agarose gel electrophoresis as tools for studying chromatin folding in solution. Methods. 12 (1), 62-72 (1997).
- Montel, F., Menoni, H., et al. The dynamics of individual nucleosomes controls the chromatin condensation pathway: direct atomic force microscopy visualization of variant chromatin. Biophysical Journal. 97 (2), 544-5453 (2009).
- Muthurajan, U. M., McBryant, S. J., Lu, X., Hansen, J. C., Luger, K. The linker region of macroH2A promotes self-association of nucleosomal arrays. The Journal of Biological Chemistry. 286 (27), 23852-23864 (2011).
- Shlyakhtenko, L. S., Lushnikov, A. Y., Lyubchenko, Y. L. Dynamics of nucleosomes revealed by time-lapse atomic force microscopy. Biochemistry. 48 (33), 7842-7848 (2009).
- Lohr, D., Bash, R., Wang, H., Yodh, J., Lindsay, S. Using atomic force microscopy to study chromatin structure and nucleosome remodeling. Methods (San Diego, Calif). 41 (3), 333-341 (2007).
- Dyer, P. N., Edayathumangalam, R. S., et al. Reconstitution of nucleosome core particles from recombinant histones and DNA. Methods in Enzymology. 375, 23-44 (2004).
- Sambrook, J., Russell, D. . Molecular cloning: a laboratory manual. , (2001).
- Balbo, A., Zhao, H., Brown, P. H., Schuck, P. Assembly, loading, and alignment of an analytical ultracentrifuge sample cell. J. Vis. Exp. (33), e1530 (2009).
- Demeler, B. UltraScan: a comprehensive data analysis software package for analytical ultracentrifugation experiments. Modern Analytical Ultracentrifugation: Techniques. , 210-230 (2005).
- Holde, K. V., Weischet, W. Boundary analysis of sedimentation velocity experiments with monodisperse and paucidisperse solutes. Biopolymers. 17 (6), 1387-1403 (1978).
- Demeler, B., van Holde, K. E. Sedimentation velocity analysis of highly heterogeneous systems. Analytical Biochemistry. 335, 279-288 (2004).
- Hansen, J., Lohr, D. Assembly and structural properties of subsaturated chromatin arrays. Journal of Biological Chemistry. 8, 5840-5848 (1993).
- Routh, A., Sandin, S., Rhodes, D. Nucleosome repeat length and linker histone stoichiometry determine chromatin fiber structure. Proceedings of the National Academy of Sciences of the U S A. 105 (26), 8872-8877 (2008).
- Zhou, J., Fan, J. Y., Rangasamy, D., Tremethick, D. J. The nucleosome surface regulates chromatin compaction and couples it with transcriptional repression. Nature Structural & Molecular Biology. 14 (11), 1070-1076 (2007).
- Dorigo, B., Schalch, T., Kulangara, A., Duda, S., Schroeder, R. R., Richmond, T. J. Nucleosome arrays reveal the two-start organization of the chromatin fiber. Science (New York, N.Y.). 306 (5701), 1571-1573 (2004).
- Correll, S. J., Schubert, M. H., Grigoryev, S. a Short nucleosome repeats impose rotational modulations on chromatin fibre folding. The EMBO Journal. 31 (10), 2416-2426 (2012).
- Mcbryant, S. J., Krause, C., Woodcock, C. L., Hansen, J. C. The Silent Information Regulator 3 Protein , SIR3p , Binds to Chromatin Fibers and Assembles a Hypercondensed Chromatin Architecture in the Presence of Salt. Molecular and Cellular Biology. 28 (11), 3563-3572 (2008).
- Schalch, T., Duda, S., Sargent, D. F., Richmond, T. J. X-ray structure of a tetranucleosome and its implications for the chromatin fibre. Nature. 436 (7047), 138-1341 (2005).
- Fan, J. Y., Gordon, F., Luger, K., Hansen, J. C., Tremethick, D. J. The essential histone variant H2A.Z regulates the equilibrium between different chromatin conformational states. Nature Structural Biology. 9 (3), 172-176 (2002).
- Carruthers, L. M., Bednar, J., Woodcock, C. L., Hansen, J. C. Linker histones stabilize the intrinsic salt-dependent folding of nucleosomal arrays: mechanistic ramifications for higher-order chromatin folding. Biochemistry. 37 (42), 14776-14787 (1998).
- Huynh, V. A. T., Robinson, P. J. J., Rhodes, D. A Method for the In Vitro Reconstitution of a Defined "30 nm" Chromatin Fibre Containing Stoichiometric Amounts of the Linker Histone. Journal of Molecular Biology. 345 (5), 957-968 (2005).
- Dorigo, B., Schalch, T. Chromatin fiber folding: requirement for the histone H4 N-terminal tail. J. Mol. Biol. 2836 (03), 85-96 (2003).
- Qian, R. L., Liu, Z. X., et al. Visualization of chromatin folding patterns in chicken erythrocytes by atomic force microscopy (AFM. Cell Research. 7 (2), 143-150 (1997).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유