Method Article
Assemblea dei nucleosomal matrici da ricombinanti core istoni e nucleosomi posizionamento DNA
In questo articolo
Riepilogo
Un metodo viene presentato per la ricostituzione del modello nucleosomal array di istoni fondamentali ricombinanti e tandem ripetute di DNA posizionamento dei nucleosomi. Abbiamo anche descrivono come gli esperimenti di velocità di sedimentazione nel ultracentrifuga analitica e microscopia a forza atomica (AFM) vengono utilizzati per monitorare il grado di saturazione matrice nucleosomal dopo la ricostituzione.
Abstract
Octamers istoni core che sono ripetitivamente distanziati lungo una molecola di DNA sono chiamati matrici nucleosomal. Array nucleosomi sono ottenuti in uno dei due modi: la purificazione da fonti in vivo, o la ricostituzione in vitro da istoni fondamentali ricombinanti e tandem ripetute di DNA posizionamento dei nucleosomi. Il secondo metodo ha il vantaggio di consentire il montaggio di una più uniforme composizionale e matrice nucleosomal posizionamento preciso. Esperimenti velocità di sedimentazione del ultracentrifuga informazioni resa analitica circa le dimensioni e la forma delle macromolecole analizzando il tasso al quale migrano attraverso la soluzione sotto la forza centrifuga. Questa tecnica, insieme con microscopia a forza atomica, può essere utilizzato per il controllo di qualità, garantendo che la maggioranza dei modelli di DNA sono saturi di nucleosomi dopo ricostituzione. Qui si descrivono i protocolli necessari per ricostituire quantità milligrammo di lunghezza e compositivamente darray nucleosomal efined adatte per gli studi biochimici e biofisici della struttura e funzione della cromatina.
Introduzione
Genomi eucariotici non esistono DNA nudo, ma piuttosto vengono compattati e organizzati da proteine legate. Questi complessi di DNA e proteine sono noti come cromatina. L'unità di ripetizione di base della cromatina è il nucleosoma. Un nucleosoma è costituito da istoni ottamero e 146 paia di basi di DNA avvolto intorno al istoni ottamero circa 1,6 volte 1. Il ottamero istone è composto di due copie ciascuna delle istoni H2A nucleo, H2B, H3, e H4. Octamers istoni core che sono ripetitivamente distanziati lungo una molecola di DNA sono chiamati matrici nucleosomal. La struttura ampliata di matrici nucleosomal è stato definito come la fibra 10 nm o le "perle su una stringa" struttura ed è presente in vitro in condizioni di scarsa sale 2. La fibra 10 nm è in grado di condensare in strutture di ordine superiore attraverso intra-array compattazione e / o inter-array di oligomerizzazione 2. Queste strutture di ordine superiore possono essere indotte in presenza di sali,o possono essere influenzati tramite il legame di proteine architettoniche cromatina all'array nucleosomal 3,4. I livelli di compattazione della cromatina sono inversamente correlati con il tasso di trascrizione in vivo 5,6. Recenti ricerche evidenzia l'importanza della organizzazione strutturale di genomi in processi quali il differenziamento, lo sviluppo del cancro, e altri 7,8. L'uso di matrici nucleosomal per studiare la struttura e la funzione della cromatina è diffusa. Qui si descrive un metodo per l'assemblaggio di array nucleosomal da istoni fondamentali ricombinanti e nucleosoma posizionamento DNA.
Utilizzo di DNA ricombinante con ripetizioni in tandem di sequenze di posizionamento nucleosome consente la ricostituzione degli array che contengono nucleosomi intervalli regolari. Due delle sequenze di posizionamento più popolari sono la sequenza del gene rRNA 5S e la "601" sequenza 9,10. La sequenza 601 è stato derivato da esperimenti SELEX e more posizioni molto nucleosomi rispetto alla sequenza 5S 11. Di conseguenza, la lunghezza del DNA linker delle matrici 601 è più omogeneo. Ripetute in tandem nucleosome posizionamento DNA è ottenuta mediante gel filtrazione 4,12. Istoni ricombinanti sono purificati da E. coli in condizioni denaturanti 13. L'uso di istoni ricombinanti permette di controllare accuratamente la composizione istone delle matrici nucleosomal. Ad esempio, il nucleo istoni recante specifiche mutazioni 14 o modificazioni post-traslazionali 15,16 può essere sostituito istoni principali wild type.
Esperimenti di velocità di sedimentazione di monitorare il tasso di sedimentazione delle macromolecole in soluzione sotto una forza centrifuga applicata 17. Questo fornisce informazioni circa la dimensione e la forma di macromolecole in un campione. Esperimenti di velocità di sedimentazione sono quindi uno strumento adeguato per lo studio di soluzioni cambiamenti di stato in fibra di cromatinastruttura a causa di condensazione della cromatina 18. Soprattutto, è necessario prima utilizzare gli esperimenti di velocità di sedimentazione come un passo di controllo di qualità in nucleosomal matrice ricostituzione. Se il DNA e nucleosomi non sono combinati al corretto rapporto molare, le matrici possono essere sotto-o sovra-saturo di istoni core. Pertanto, le informazioni ottenute dagli esperimenti di velocità di sedimentazione viene utilizzato per garantire che il DNA sia correttamente saturo di nucleosomi. È importante utilizzare metodi alternativi per stimare la saturazione di DNA con nucleosomi, soprattutto se si lavora con uno stampo di DNA precedentemente uncharacterized. Pertanto, abbiamo anche descritto un metodo per l'analisi di matrici nucleosomal utilizzando microscopia a forza atomica (AFM). AFM è una tecnica potente che permette la visualizzazione degli effetti di una serie di parametri, quali il livello di saturazione, effetto della presenza di varianti dell'istone o gli effetti di MgCl 2 19,20. AFM è stato anche applied a studiare le dinamiche nucleosome utilizzando lasso di tempo di imaging 21. In vitro assemblati nucleosomal array di 12-mer sono particolarmente suscettibili agli studi AFM perché appartengono nell'intervallo giusta dimensione per imaging AFM 22. Nel presente studio abbiamo utilizzato AFM di array nucleosomal come controllo di qualità, nonché un mezzo di confermare i dati di AUC ("vedere per credere"). Oltre alla visualizzazione semplice, AFM permette la misurazione di profili altezza di campioni come metrica supplementare.
Protocollo
1. Assemblea dei ricombinanti fondamentali istoni in octamers
Motivazione: Il primo passo per nucleosomal matrice ricostituzione è quello di preparare native di base dell'istone octamers da liofilizzati istoni fondamentali ricombinanti. Istoni sono combinati in quantità molari uguali e assemblati in octamers istoni dai dialisi i campioni di un buffer di denaturazione in ripiegamento buffer.
- Purificare e liofilizzare istoni fondamentali ricombinanti (H2A, H2B, H3, H4), come descritto 13.
- Sciogliere circa 5 mg di ciascun nucleo istone liofilizzato in 3 ml di tampone di dispiegamento (6 M guanidina HCl, 20 mM Tris pH 7,5, 5 mM DTT). Consentire ad ogni aliquota da sciogliere per almeno un'ora. Assicurarsi che nessuna proteina rimane sulle pareti del contenitore.
- Misurare l'assorbanza di ciascun istone a 276 nm utilizzando tampone dispiegarsi come riferimento.
- Calcolare la molarità di ciascun istone usando i loro coefficienti di estinzione (Vedi Tabella 1 per Xenopus coefficienti di istone estinzione). Confrontare l'assorbanza determinata concentrazione dell'istone al peso secco liofilizzato, e stima se la maggior parte della proteina è disciolto.
- Determinare quale istone avete le talpe minor numero di, in quanto questo sarà il numero limite di moli per tutti gli istoni.
- Combinare gli istoni in quantità molari uguali e diluire con dispiegarsi tampone ad una concentrazione finale di 1 mg / ml.
- Mettere il campione in 6-8 tubi di dialisi kDa MWCO. Sigillare il tubo e riporre in 2 L di tampone ripiegamento fredda (2 M NaCl, 10 mM Tris pH 7.5, 1 mM EDTA, 5 mM β-mercaptoetanolo).
- Dializzare il campione per 18 ore a 4 ° C con agitazione. Assicurarsi che il tubo di dialisi può ruotare liberamente e con forza, altrimenti gli istoni può precipitare.
- Modificare il buffer di dialisi due volte nel periodo di 18 ore (cioè modificare il buffer di ripiegamento circa ogni 6 ore fino a cottura). Anche se si forma precipitato non scartarecampione, alcuni ottamero può essere recuperato anche se la resa sarà diminuito.
Nota: Vedere la fase 2,1-2,2 per preparare colonna di equilibrio per il giorno successivo.
2. Purificazione dell'istone octamers da Size Exclusion Chromatography
Motivazione: Dopo aver concluso con la sezione 1, i campioni conterranno octamers istoni così come altri complessi di istoni, quali inerti, tetrameri H3/H4, e dimeri H2A/H2B. Octamers istoni saranno purificati da questi complessi utilizzando cromatografia di esclusione dimensionale (SEC).
- Collegare un HiLoad 16/60 Superdex200 16/60 (S200) colonna a un sistema FPLC. Assicurarsi che le bolle d'aria non entrano nel sistema.
- Pulire la colonna S200 con 0,2 micron acqua filtrata. Utilizzare una portata di 0,3 ml / min e impostare un limite contropressione di 0,5 MPa. Assicuratevi di avere abbastanza acqua e permettere la colonna di pulire tutta la notte.
- Equilibrare la colonna e campionamento del loop S200 del FPLC utilizzando ripiegamento buffer. Utilizzare 1 L di 0,2 micron buffer di ripiegamento filtrato. Flusso ripiegamento tampone attraverso la colonna ad una velocità di 1 ml / min e una pressione massima di 0,5 MPa per circa 2 ore.
- Equilibrare un concentratore centrifugo Vivaspin (50 kDa MWCO) con 1 ml di tampone di ripiegatura. Rimuovere il campione dal tubo di dialisi e centrifugare il campione a 4 ° C per rimuovere eventuali precipitati. Pipettare il surnatante campione nel concentratore. Concentrare il campione ad un volume di circa 500 ml.
- Rimuovere il campione ottamero concentrato in un nuovo contenitore e risciacquare il concentratore di 1 ml di ripiegamento buffer. Concentrare il risciacquo fino a 500 microlitri, e aggiungerlo al campione ottamero. Questo aiuta a recuperare qualsiasi ottamero rimanente nel concentratore.
- Centrifugare il campione in una provetta per microcentrifuga da 1,5 ml per 5 min a 10.000 rpm a 4 ° C. Raccogliere il surnatante e trasferire in un nuovo tubo. Questo aiuta a rimuovere l'eventuale precipitato prima di caricare il campione sul FPLC.
- Caricare il campione attraverso la colonna S200. Caricare un massimo di 1,5 volume totale ml o 15 mg ottamero in una singola purificazione.
- Eluire l'esempio utilizzando buffer di ripiegamento appena fatto. Utilizzare una portata di 1 ml / min e un limite contropressione di 0,5 MPa. Utilizzare due volumi di colonna (400 ml) ripiegamento tampone e raccogliere 2 ml frazioni. Monitorare l'assorbanza a 280 nm durante l'eluizione. Le diverse specie saranno eluire in ordine decrescente. Aggregati istoni eluiscono solito a circa 45 ml, ottamero a circa 65 ml, e dimero di circa 84 ml (Figura 1).
- Analizzare le frazioni di eluizione da picchi di interesse eseguendo campioni su un 18-20% SDS-PAGE. Le frazioni contenenti octamers purificati dovrebbero avere la stessa quantità molari dei quattro proteine istoniche fondamentali (Figura 1).
- Si riuniscono le frazioni che contengono octamers purificati, e concentrare a ≤ 15 mg / ml con un filtro centrifugo Vivaspin. Se lasciato diluito, octamers possono dissociare idimeri nto H2A/H2B e tetrameri H3/H4.
- Determinare la concentrazione ottamero misurando l'assorbanza a 280 nm e calcolare utilizzando il coefficiente di estinzione da Tabella 1.
- Octamers istoni possono essere conservati a breve termine in tampone ripiegamento a 4 ° C. In caso di immagazzinaggio a lungo termine, i octamers saranno più stabili a -20 ° C e in una soluzione di glicerolo al 50%. Octamers conservati in glicerolo devono essere dializzati nel buffer di ripiegamento fresca prima misure di assorbanza e utilizzare in ricostituzioni di matrice nucleosomal.
3. Ricostituzione del nucleosomal matrici di DNA e purificata octamers
Motivazione: Ricostituzione di array nucleosomal richiede che octamers istoni e DNA stampo essere combinati in rapporti molari specifici. Miscele di DNA e istoni octamers a 2 M NaCl sono passo dializzato in buffer di diminuire forza ionica. Un cambiamento graduale di sale inferiore assicura la corretta formazione dei nucleosomi. Ottenere narray ucleosomal con il livello desiderato di saturazione istone ottamero del DNA stampo richiede piccole ricostruzioni prova scala seguite da una larga scala preparativa ricostituzione. Le condizioni appropriate definite dalle ricostruzioni piccola scala sono utilizzati per guidare la ricostituzione preparativa.
- Purificare tandem ripetute nucleosome posizionamento DNA come descritto 4,12. Brevemente, isolare il DNA plasmidico utilizzando il kit Qiagen GigaPrep o un prodotto simile. Impostare un enzima di restrizione digerire per liberare il DNA stampo e digerire il DNA plasmidico di dimensioni più piccole (≤ 700bp). Il DNA modello può poi essere purificato dal piccolo plasmide resti di DNA utilizzando la cromatografia di esclusione dimensionale (SEC). Una gravità alimentato colonna, con un'altezza di circa 115 centimetri imballato con Sephacryl perline S-1000 è sufficiente per la purificazione di 601 x 12mer 207bp DNA.
Note: Per la purificazione di DNA plasmidico a costo ridotto ma aumentato effort, si può usare lisi alcalina con fenolo / cloroformio purificazione DNA per isolare il DNA plasmidico da E. coli 23,24. Strategie per separare modello di DNA dal plasmide possono variare con i cambiamenti nel modello di dimensioni DNA. Per SEC è importante impostare l'enzima di restrizione digerire in modo da massimizzare la differenza di dimensioni tra il modello e il DNA plasmidico.
- Determinare quali rapporti molari (r) da utilizzare per ricostruzioni. R è uguale al rapporto tra le moli di ottamero di moli di ripetizione DNA. Per le prove iniziali di piccola scala, i valori r di 0.9, 1 e 1.1 sono appropriati se l'intento è quello di ottenere matrici nucleosomal saturi.
- Preparare campioni piccola scala calcolando la quantità di ottamero aggiungere a circa 18 mg di DNA per ogni rapporto molare da testare. Mescolare il DNA e octamers istoni. La concentrazione finale di NaCl nel campione deve essere ≥ 2 M, e la concentrazione finale di DNA dovrebbe essere intorno a 0,3 mg / mL. Le condizioni definitive tampone del campione devono comprendere anche 10 mM Tris pH 7.8, e 1 mM EDTA.
- I campioni sono ora pronti per dialisi. Caricare i campioni in tubi di dialisi 12k-14k MWCO. Dializzare campioni contro 2L di tampone decrescente di forza ionica come segue.
Componenti in tutti i buffer: 10 mM Tris pH 7,8, 1 mM EDTA. Buffer 1 (aggiungere 1 M NaCl, 1 mM DTT) per 5-6 ore. Buffer 2 (aggiungere 0,75 M NaCl, 1 mM DTT) durante la notte. Buffer 3 (aggiungere 2,5 mM NaCl, 1 mM DTT) per 5-6 ore. Buffer 4 (aggiungere 2,5 mM NaCl, 0,1 mM PMSF) durante la notte.
- Rimuovere i campioni dal tubo di dialisi e procedere alle sezioni 4-6. Se i campioni di piccole dimensioni producono i risultati desiderati, ripetere i passaggi nelle sezioni 3-6 su larga scala.
Nota: Al fine di preservare le risorse, i campioni su piccola scala devono avere il volume minimo necessario per bastare per test di screening a valle. Per avere enough campione per gli esperimenti di velocità di sedimentazione e AFM qui elencate, 50 campioni microlitri a 0.3 mg / ml di DNA sono appropriati. La dimensione di larga scala, campioni preparative dipendono dall'uso previsto. Un campione larga scala dovrebbe essere preparata nello stesso modo dei campioni su piccola scala.
4. La sedimentazione Velocity Analisi dei ricostituiti nucleosomal Array
Motivazioni: sedimentazione esperimenti velocità nelle informazioni rese ultracentrifuga analitica Beckman Xl-A / I sulla dimensione e la forma delle molecole in soluzione. Esperimenti di velocità di sedimentazione effettuati in condizioni di scarsa di sale vengono utilizzati per determinare la misura in cui le matrici ricostituiti sono saturi di nucleosomi.
- Diluire il campione ad una assorbanza circa 0,5 a 260 nm utilizzando TEN tampone (10 mM Tris pH 7,8, EDTA 1 mM, 2,5 mM di NaCl). Caricare 400 ml di campione in una cella con un centro due settore, con il campione su un lato e il riferimence (TEN buffer) dall'altro 25. Mantenere le menisco di riferimento superiori al menisco del campione (ovvero aggiungere 420 microlitri soluzione di riferimento).
- Caricare le cellule nel rotore, e allineare le celle correttamente utilizzando i marchi hash sul fondo delle celle e rotore 25. Spolverare delicatamente le lenti delle cellule utilizzando aria compressa.
- Accendere la centrifuga XL-A/XL-I, inserire il rotore, e collegare e fissare le ottiche come descritto nel manuale. Aprire il software Proteome Lab e selezionare il file: new.
- Impostare una singola esecuzione di scansione a 3.000 giri e una temperatura di 25 ° C. In Opzioni, selezionare fermata dopo l'ultima scansione e la calibrazione radiale run (calibrazione radiale non è necessario se il rotore è stato l'ultimo rotore utilizzato nella AUC).
- Per ogni cella, il nome dei campioni, selezionare una lunghezza d'onda (260 nm), selezionare assorbanza e scegliere un percorso di salvataggio del file sul vostro computer. Inizia la corsa singola scansione.
- Utilizzare la scansione singolo per determinare la ra appropriatacomporre lunghezza di scansione (Figura 2A). Diminuendo la lunghezza della cella da scansionare diminuisce il tempo di esecuzione. Tuttavia, assicurarsi di includere il menisco campione e prolungare l'estremità molto vicino, o nella parte inferiore della cella.
Nota: scansioni semplici permettono l'area di misura per essere accorciata misurazioni partire subito prima del menisco campione (Figura 2A). La maggior parte delle scansioni generati durante la corsa AUC dovrebbe avere frazioni contorno ultime menisco e plateau stabile (Figura 2B). Lenti sporche sulle cellule AUC possono generare scansioni con picchi di grandi dimensioni, questi possono influenzare l'analisi dei dati. Il software UltraScan include un manuale che è una grande risorsa per informazioni riguardanti l'analisi dei dati ultracentrifugazione analitica 26.
- Utilizzando il pannello di controllo XL-A/XL-I, immettere una velocità di 0 e premere start. Ciò richiederà la camera di centrifuga per tirare un vuoto e tuttiow la temperatura della camera di equilibrarsi. Attendere 1 ora per consentire il raggiungimento dell'equilibrio di temperatura prima di iniziare a correre.
- Utilizzare il software per impostare la corsa per la velocità desiderata e il numero di scansioni. Velocità più elevate aumenta la risoluzione, ma si deve raccogliere almeno 20 scansioni (preferibilmente più) prima che il campione è sedimentato sul fondo della cella. E 'conveniente per sovrapporre gli ultimi scansioni (compiuti nel menu opzioni) al fine di monitorare l'andamento della corsa e verificare la presenza di potenziali problemi come le cellule che perdono. Monitorare il primo paio di scansioni per garantire il corretto funzionamento.
5. Modifica e analisi sedimentazione Velocity dati
Motivazione: dati di velocità di sedimentazione Raw devono essere analizzati da un programma che produce una distribuzione coefficiente di sedimentazione di diffusione corretta. Questa informazione a sua volta indica la frazione di un campione ricostituito contenente un determinato livello di saturazione, ad esempio se si utilizza uno stampo di DNA 12-mer, sarà possibile determinare la frazione di matrici ricostituite che ha 10, 11 e 12 nucleosomi / DNA.
- Utilizzare il software di analisi dei dati AUC come UltraScan per modificare i dati di 26. Editing dei dati di scansione deve essere eseguita per ogni cella e crea un insieme di dati per le analisi successive. La corretta editing di scansioni prevede la definizione dei menisco campione, riducendo i dati ad una regione di interesse, e definendo partenza nonché regioni plateau (Figura 2). Scansioni indesiderati possono essere rimossi dal set di dati, a questo punto. Tuttavia, dal momento che è possibile rimuovere scansioni indesiderate durante l'analisi, si raccomanda che tutte le scansioni essere mantenuti a questo punto.
- Si consiglia vivamente che il furgone metodo Holde-Weischet rafforzata essere utilizzato per analizzare i dati di 27. Questo metodo di analisi corregge gli effetti della diffusione nel corso della corsa e cede la distribuzione integrale di coefficienti di sedimentazione. In particolare,, La maggiore van Holde-Weischet metodo (come attuato in UltraScan) consentirà l'inserimento delle scansioni che mancano altipiani stabili, o che contengono frazioni di contorno che non hanno eliminato il menisco nell'analisi 28.
- Determinare la distribuzione dei coefficienti di sedimentazione per le matrici assemblate. Risultati rappresentativi per 601-12 (207bp, 12-mer) array nucleosomi sono mostrati in Figura 3.
- Se uno dei campioni su piccola scala contiene array con la gamma desiderata dei coefficienti di sedimentazione, quindi ripetere il processo ad una maggiore scala che inizia con la sezione 3. Se nessuno dei campioni di piccole dimensioni ha una corretta distribuzione del coefficiente di sedimentazione ripetere le prove su piccola scala con una nuova gamma r dal capitolo 3.
6. Visualizzazione nucleosomal matrici utilizzando microscopia a forza atomica (AFM)
Motivazioni: AFM consente di visualizzare il livello di saturazione di nucleosomamatrici l. Questa tecnica è complementare velocità di sedimentazione per AUC e enzimi di restrizione come un test di controllo della qualità.
- Usando i metodi descritti sopra, ottenere una matrice nucleosoma ben saturo (Figura 5A).
- Preparare APTES trattati diapositive mica mettendo ~ 30 microlitri 1:1000 diluizione del commercio (Sigma-Aldrich (3-Aminopropyl) trietossisilano A3648-100 ml) a 0,22 micron acqua Nanopure filtrata per 30 min.
- Dopo 30 minuti risciacquare le APTES con acqua filtrata e asciugarli delicatamente in un flusso di azoto.
- Collocare opportunamente diluito campione matrice nucleosomi (~ 1,5 ng / mL) sulla slitta, e incubare coperto a temperatura ambiente per 15 min.
- Risciacquare campione fuori con tampone campione filtrato, asciutto come prima, e il luogo di diapositive sul palco di AFM (in questo caso una Asylum Research MFP-3D Atomic Force Microscope).
- Inizia l'imaging 2 x 2 micron scansioni con 512 linee di scansione. Contare il numero di nucleosomi per accertare il livello di saturation (Figura 5B).
- Idealmente si dovrebbe aree di immagine sul vetrino con gli array nucleosome ben separati.
- Per immagini ad alta risoluzione, una scansione nm 500 x 500 può essere ottenuto (Figura 5C).
- Le immagini possono essere appiattite e analizzati utilizzando il software MFP-3D fornita da Asylum.
- Ogni immagine può essere diviso in quattro quadranti e ingrandito in digitale per ottenere una visione più chiara di matrici ben separati e più linee a mano libera può essere tracciata attraverso le matrici e le isoipse sono ottenute ai nucleosomi (Figura 5C, a destra traccia pannello di altezza e 5D).
- Parecchie tali immagini devono essere analizzati per ciascun tipo di matrice comprende diverse centinaia di nucleosomi. Isoipse sono registrati, classificati e tracciati in MS Excel.
Risultati
Per illustrare il protocollo abbiamo ricostituito array nucleosomal da ricombinanti istoni fondamentali Xenopus e DNA, rappresentati da 12 ripetizioni tandem 207 bp della sequenza di posizionamento 601 (601 207 x 12). In primo luogo abbiamo assemblato octamers autoctoni istoni fondamentali liofilizzati e poi purificato il octamers da FPLC utilizzando una colonna S200 (Figura 1A). Complessi più grandi eluiscono prima dalla colonna S200. Istoni generalmente eluiscano in questo ordine: non specifici aggregati istoni, istoni ottamero, H3/H4 tetramero, e H2A/H2B dimero (Figura 1A). I picchi dalla colonna S200 sono stati analizzati mediante SDS-PAGE. I gel di frazioni ottamero purificati devono indicare quantità equimolari di quattro proteine istoniche (Figura 1B). Si noti che Xenopus H2A e H2B hanno pesi molecolari che differiscono soltanto di circa 200 Da e quindi appaiono come una singola banda su gel SDS. Tetrameri H3/H4 e dimeri H2A/H2B saranno eluire dopo, ma very vicino al picco istone ottamero. Quando si seleziona quali frazioni di mantenere è prudente scartare frazioni che compaiono verso la fine del picco ottamero nel cromatogramma. Queste frazioni possono avere una maggiore quantità di complessi istone non octameric. In questo caso le frazioni 64-67 sono stati raccolti e salvati per nucleosomal matrice ricostituzione (Figura 1).
All'interno UltraScan ci sono due modi diversi di intendere la distribuzione diffusione corretta dei coefficienti di sedimentazione che si ottiene da una maggiore analisi van Holde-Weischet. La linea rossa rappresenta la distribuzione integrale dei coefficienti di sedimentazione. Per un dato punto sul grafico, l'asse y indica la frazione del campione che ha un coefficiente di sedimentazione pari o inferiore al valore indicato l'asse x. Così, una linea verticale è indicativa di un campione omogeneo, mentre un campione eterogeneo avrà una curva con pendenza positiva. La linea verde è il detratti derivati della distribuzione integrale. L'area sotto il picco è proporzionale alla quota parte del campione che ha quel coefficiente di sedimentazione. Completamente saturi 601 207 x 12 array nucleosomal hanno un coefficiente di sedimentazione di 29S 29. Per l'esempio mostrato nella figura 3, i dati indicano che circa il 70% degli array sono saturi e 30% supera saturo.
Microscopia a forza atomica è passati da una tecnica emergente di un approccio complementare popolare per studiare l'organizzazione della cromatina in vitro. Qui abbiamo usato AFM fianco di ultracentrifugazione analitica per stabilire il grado di saturazione del modello dopo la ricostituzione. Nella figura 6 la maggioranza del ~ 27S array (Figura 4A) utilizzata per l'imaging conteneva 10-11 nucleosomi (Figura 4B), dimostrando eccellente accordo fra la AFM e risultati AUC. I risultati ottenuti AFM qui convalidare così questoavvicinare e renderlo una tecnica affidabile per la caratterizzazione di matrici nucleosomal.
| Istone proteine | Σ276, Proteine dispiegata cm -1 M -1 | Molecular Weight Da |
| H2A | 4.350 | 13.960 |
| H2B | 7.250 | 13.774 |
| H3 | 4.640 | 15.273 |
| H4 | 5.800 | 11.236 |
| Ottamero | 44.080 | 108.486 |
Tabella 1. Coefficienti di estinzione e pesi molecolari di Xenopus laevis istoni fondamentali e rinaturalizzato ottamero istoni.
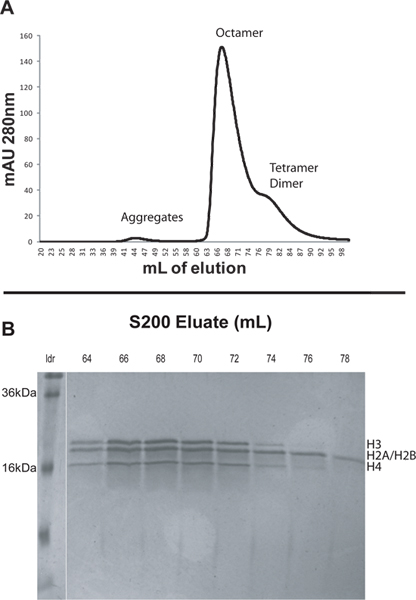
Figura 1. Profilo A. eluizione dalla colonna S200 come descritto nella sezione 2. L'piccolo picco a circa 44 ml è causa di grandi aggregati istoni. Il picco significativo a 67 ml è il octamers istoni. L'ampia spalla 76-90 ml contiene i tetrameri H3/H4 e H2A/H2B dimeri. B. 20% SDS-PAGE delle frazioni selezionate dalla colonna S200. Le prime frazioni dalla vetta ottamero devono essere raccolti in quanto sono meno probabilità di essere contaminati da complessi istone non octameric. In questo caso le frazioni 64-67 sono stati raccolti e salvati per nucleosomal matrice ricostituzione. Clicca qui per ingrandire la figura .

Figura 2. A. ProteomeLab schermo software di acquisizione di una singola scansione raccolti a 3.000 giri (con labels aggiunto). Notare che il campione non sedimento in questa bassa velocità. Il singolo di scansione viene utilizzato per impostare la gamma di misure per la scansione delle cellule AUC (fase 4.6). Cattura B. Schermo di una serie di sedimentazione scansioni di velocità ottenuto utilizzando il programma UltraScan (con le etichette aggiunti) 26. Questa serie di scansioni per generare un insieme di dati per l'analisi (vedere fase 5.1) viene modificato.
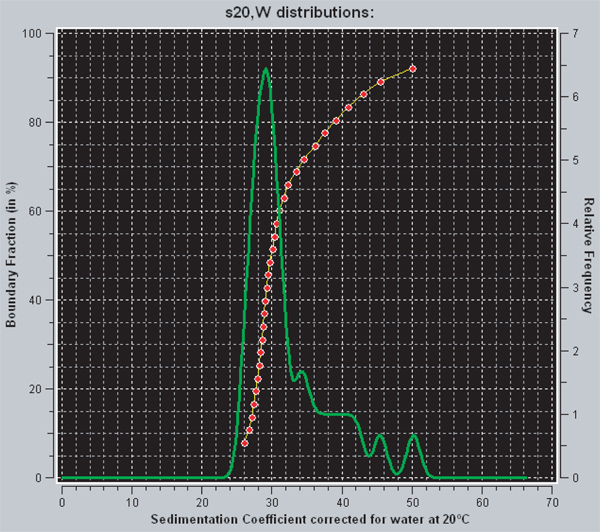
Figura 3. Un cattura schermo da UltraScanII. Le linee rosse e verdi sono due metodi diversi per la visualizzazione della distribuzione dei coefficienti di sedimentazione in UltraScan. La linea rossa è la distribuzione integrale di coefficienti di sedimentazione, mentre il verde è la derivata. Ulteriori informazioni sulla interpretazione può essere trovato nella sezione dei risultati.
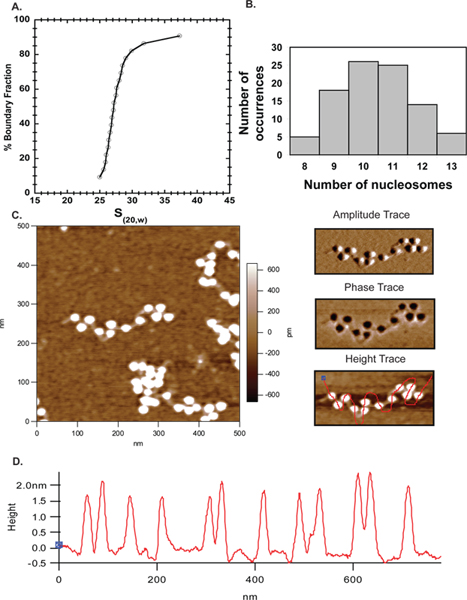
Figura 4. AFM. A. sedimentazione profilo di velocità di 601 207 x 12 matrici assemblate con il mouse ottamero che indicano il coefficiente medio di sedimentazione è 27S. B. Le stesse matrici in A sono stati ripresi dalla AFM e il numero di nucleosomi contati in diverse immagini sono state tracciate in MS Excel. La trama indicato che la maggioranza di array aveva 10-11 nucleosomi corroborare i dati di UAC. C. A 500 x 500 nm di scansione di 601 x 207 a 12 array nucleosome con il DNA linker chiaramente visibile. Il pannello in alto a destra è la traccia ampiezza e il pannello centrale di destra è la traccia fase per la matrice mostrata in C. Il pannello inferiore destra è la stessa traccia riportata in sinistra ma con una linea a mano libera che passa per i nucleosomi per determinare il profilo altezza del nucleosomas. D. profili altezza dei nucleosomi nell'immagine qui sopra dimostrano che tutti i nucleosomi nel raggio di 1,5-2,5 altezza nm come riportato in precedenza 40.
Discussione
Nucleosomal array modello sono uno strumento molto utile per lo studio in vitro della struttura e funzione della cromatina. Ad esempio, sono stati ampiamente utilizzati per studiare il meccanismo di fibra condensazione della cromatina in soluzione 30-34, e reso possibile ottenere una struttura a raggi X di un Tetranucleosome 35. Più di recente si sono dimostrate utili a decifrare gli effetti strutturali delle varianti specifiche dell'istone nucleo, mutanti e modificazioni post-14-16,36. Qui si descrive un metodo generale per l'assemblaggio del modello matrici nucleosomal da nucleosoma posizionamento DNA e istoni nucleo ricombinanti.
La ricostituzione degli array nucleosomal da octamers purificati e 601 207 x 12 DNA è semplice, che coinvolge diversi passaggi di dialisi che in sequenza abbassano la concentrazione di NaCl da 2 M a 2,5 mm. La parte più difficile del protocollo è quello di utilizzare un r-value che produce la temperatura desideratalivello di saturazione piatto. Il valore r appropriato viene determinata empiricamente su piccola scala e poi ripetuta su una scala più ampia per generare le matrici nucleosomal utilizzati per esperimenti. Nel nostro caso, abbiamo cercato di ottenere il 601 207 x 12 modelli di DNA per lo più saturi con 12 nucleosomi per DNA. Il coefficiente di sedimentazione di una saturazione completa 601 207 x 12 matrice nucleosomal è di 29 S, mentre lo stesso modello di DNA con solo 11 nucleosomi per sedimenti di DNA a ~ 27S 29. Così, velocità di sedimentazione in ultracentrifuga analitica fornisce un metodo molto sensibile per determinare il livello di saturazione nucleosoma dopo ricostituzione. Abbiamo analizzato i nostri dati utilizzando il metodo avanzato van Holde-Weischet, che produce una distribuzione coefficiente di sedimentazione di diffusione corretta. Questa informazione è essenziale perché racconta uno come omogenei o eterogenei il campione è dopo la ricostituzione. In altre parole, per un modello 601 207 x 12 DNA,indica la frazione del campione che ha 12 nucleosomi al DNA, 11 nucleosomi per DNA, ecc La Figura 3 mostra i risultati del furgone migliore analisi Holde-Weischet di 601 207 x 12 array nucleosomi ricostituiti durante un r di 1.1, in cui circa il 30% degli array sono più saturi. L'utilità del campione è di solito legata alla percentuale di array saturi, ma dipende esperimenti array verranno utilizzati in Alcune applicazioni possono richiedere matrici molto omogenei e / o saturi. Un certo numero di metodi esistenti per migliorare l'omogeneità e la saturazione di array nucleosomal.
In questo caso le matrici ipersaturi potrebbero essere rimossi mediante precipitazione selettiva in seguito all'aggiunta di MgCl 2 37. Array più omogenee possono anche essere ottenuti purificando l'esempio mediante centrifugazione su gradiente di saccarosio, gel elettroforesi preparativa, scambio ionico e cromatografia 10,13. A nu modificatocleosomal metodo array di ricostituzione prevede l'aggiunta di brevi DNA concorrente ai campioni prima della ricostituzione mediante dialisi sale 38,39. Questo permette di assemblare array nucleosomal con un eccesso di istone ottamero colpo sopra saturare il DNA stampo. Matrici nucleosomal ricostituito utilizzando concorrente DNA probabilmente richiederà una fase di purificazione per la rimozione del DNA competitore e istoni supplementare.
Anche dopo assembly matrice pilota su piccola scala, è possibile che le matrici nucleosomal ricostituite larga scala non saranno adeguatamente saturi. Per evitare sprechi di DNA stampo e istone ottamero nel campione, è possibile fissare sopra o sotto array saturi. Se gli array sono più saturi, il DNA può essere aggiunto al campione. Se gli array sono sotto saturi, ottamero extra può essere aggiunto per correggerlo. Tuttavia, l'aggiunta ottamero agli array già dializzati in bassa percentuale di sale può causare ottamero dissociazione. Se l'aggiunta o in piùctamer o DNA array, Dializzare le matrici di nuovo in 2 M NaCl prima di aggiungere ottamero extra o DNA per il campione globale. Ripetere il passaggio di dialisi per i campioni fissati da alto a basso contenuto di sale come da punto 3.4. La quantità di ottamero da aggiungere al fine di correggere gli array può essere stimata dai coefficienti di sedimentazione per meno di array saturi 29. Dopo aver regolato il campione di array nucleosomi, misurazioni del livello di saturazione devono essere effettuate nuovamente.
Mentre AFM è un metodo efficace per la caratterizzazione matrici nucleosomi, è complicato e un processo laborioso. Questo lo rende una scarsa tecnica per lo screening di piccole ricostruzioni scala, ma una tecnica eccellente per la caratterizzazione di campioni su larga scala e per lo studio di organizzazione della cromatina. In precedenza abbiamo utilizzato AFM per visualizzare particelle "macro" generati da una delezione costrutto macroH2A che doveva anche basse concentrazioni di MgCl2 "iper-reattivo". Likewise, Montel et al. (2009) hanno dimostrato che la variante H2A Bbd fa sì che le matrici nucleosome ad essere più "aperto" rispetto al tipo array selvatici. Pertanto, AFM è una tecnica affidabile per il controllo di qualità, nonché per lo studio della struttura della fibra cromatina in generale.
Divulgazioni
Gli autori non hanno conflitti di interesse.
Riconoscimenti
Questo lavoro è stato sostenuto da NIH sovvenzioni GM45916 e GM66834 di JCH e una borsa di studio dalla Sindrome di Rett Fondazione Internazionale per ak Questo lavoro è stato anche sostenuto da NIH concedere GM088409 e Howard Hughes Medical Institute contributi a KL
Materiali
| Name | Company | Catalog Number | Comments |
| (3-Aminopropyl)triethoxysilane | Sigma-Aldrich | A3648-100ML | |
| 6-8 kDa MWCO Dialysis Tubing | Fisher | 21-152-5 | |
| HiLoad Superdex 200 16/60 Column | GE | 17-1069-01 | |
| Vivaspin 50 kDa MWCO Centrifugal Concentrator | Sartorius | VS2031 | |
| 12-14 kDa MWCO Dialysis Tubing | Fisher | 08-667A | |
| Illustra Sephacryl S-1000 Superfine | GE | 17-0476-01 | |
| XL-A/I Analytical Ultracentrifuge | Beckman-Coulter |
Riferimenti
- Luger, K., Mader, A., Richmond, R., Crystal Sargent, D. structure of the nucleosome core particle at 2.8 A resolution. Nature. 7, (1997).
- Hansen, J. C. Conformational dynamics of the chromatin fiber in solution: determinants, mechanisms, and functions. Annual Review of Biophysics and Biomolecular Structure. 31, 361-392 (2002).
- McBryant, S., Adams, V., Hansen, J. Chromatin architectural proteins. Chromosome Research. 14 (1), 39-51 (2006).
- Hansen, J. C., Ausio, J., Stanik, V. H., van Holde, K. E. Homogeneous reconstituted oligonucleosomes, evidence for salt-dependent folding in the absence of histone H1. Biochemistry. 28 (23), 9129-9136 (1989).
- Szerlong, H. J., Prenni, J. E., Nyborg, J. K., Hansen, J. C. Activator-dependent p300 acetylation of chromatin in vitro: enhancement of transcription by disruption of repressive nucleosome-nucleosome interactions. The Journal of Biological Chemistry. 285 (42), 31954-31964 (2010).
- Cirillo, L. A., Lin, F. R., Cuesta, I., Friedman, D., Jarnik, M., Zaret, K. S. Opening of compacted chromatin by early developmental transcription factors HNF3 (FoxA) and GATA-4. Molecular Cell. 9 (2), 279-289 (2002).
- Nguyen, C., Gonzales, F. chromatin structure associated with methylation-induced gene silencing in cancer cells: correlation of accessibility, methylation, MeCP2 binding and acetylation. Nucleic Acids Research. 29 (22), 4598-4606 (2001).
- Cuesta, I., Zaret, K. S., Santisteban, P. The forkhead factor FoxE1 binds to the thyroperoxidase promoter during thyroid cell differentiation and modifies compacted chromatin structure. Molecular and Cellular Biology. 27 (20), 7302-7314 (2007).
- Simpson, R. T., Stafford, D. W. Structural features of a phased nucleosome core particle. Proceedings of the National Academy of Sciences of the U S A. 80 (1), 51-55 (1983).
- Lowary, P. T., Widom, J. New DNA sequence rules for high affinity binding to histone octamer and sequence-directed nucleosome positioning. Journal of Molecular Biology. 276 (1), 19-42 (1998).
- Lowary, P., Widlund, H., Cao, H. Sequence motifs and free energies of selected natural and non-natural nucleosome positioning DNA sequences. Journal of Molecular Biology. 288, 213-229 (1999).
- Gordon, F., Luger, K., Hansen, J. C. The Core Histone N-terminal Tail Domains Function Independently and Additively during Salt-dependent Oligomerization of Nucleosomal Arrays *. The Journal of Biological Chemistry. 280 (40), 33701-33706 (2005).
- Luger, K., Rechsteiner, T. J., Richmond, T. J. Expression and purification of recombinant histones and nucleosome reconstitution. Methods in Molecular Biology (Clifton, N.J.). 119 (4), 1-16 (1999).
- McBryant, S. J., Klonoski, J., et al. Determinants of histone H4 N-terminal domain function during nucleosomal array oligomerization: roles of amino acid sequence, domain length, and charge density. The Journal of Biological Chemistry. 284 (25), 16716-16722 (2009).
- Ma Shogren-Knaak, ., Fry, C. J., Peterson, C. L. A native peptide ligation strategy for deciphering nucleosomal histone modifications. The Journal of Biological Chemistry. 278 (18), 15744-158 (2003).
- Lu, X., Simon, M. The effect of H3K79 dimethylation and H4K20 trimethylation on nucleosome and chromatin structure. Nat Struct Mol Biol. 15 (10), 1122-1124 (2008).
- Ausio, J. Analytical Ultracentrifugation for the Analysis of Chromatin Structure. Biophysical Chemistry. 86 (2-3), 141-153 (2000).
- Hansen, J., Kreider, J., Demeler, B., Fletcher, T. Analytical ultracentrifugation and agarose gel electrophoresis as tools for studying chromatin folding in solution. Methods. 12 (1), 62-72 (1997).
- Montel, F., Menoni, H., et al. The dynamics of individual nucleosomes controls the chromatin condensation pathway: direct atomic force microscopy visualization of variant chromatin. Biophysical Journal. 97 (2), 544-5453 (2009).
- Muthurajan, U. M., McBryant, S. J., Lu, X., Hansen, J. C., Luger, K. The linker region of macroH2A promotes self-association of nucleosomal arrays. The Journal of Biological Chemistry. 286 (27), 23852-23864 (2011).
- Shlyakhtenko, L. S., Lushnikov, A. Y., Lyubchenko, Y. L. Dynamics of nucleosomes revealed by time-lapse atomic force microscopy. Biochemistry. 48 (33), 7842-7848 (2009).
- Lohr, D., Bash, R., Wang, H., Yodh, J., Lindsay, S. Using atomic force microscopy to study chromatin structure and nucleosome remodeling. Methods (San Diego, Calif). 41 (3), 333-341 (2007).
- Dyer, P. N., Edayathumangalam, R. S., et al. Reconstitution of nucleosome core particles from recombinant histones and DNA. Methods in Enzymology. 375, 23-44 (2004).
- Sambrook, J., Russell, D. . Molecular cloning: a laboratory manual. , (2001).
- Balbo, A., Zhao, H., Brown, P. H., Schuck, P. Assembly, loading, and alignment of an analytical ultracentrifuge sample cell. J. Vis. Exp. (33), e1530 (2009).
- Demeler, B. UltraScan: a comprehensive data analysis software package for analytical ultracentrifugation experiments. Modern Analytical Ultracentrifugation: Techniques. , 210-230 (2005).
- Holde, K. V., Weischet, W. Boundary analysis of sedimentation velocity experiments with monodisperse and paucidisperse solutes. Biopolymers. 17 (6), 1387-1403 (1978).
- Demeler, B., van Holde, K. E. Sedimentation velocity analysis of highly heterogeneous systems. Analytical Biochemistry. 335, 279-288 (2004).
- Hansen, J., Lohr, D. Assembly and structural properties of subsaturated chromatin arrays. Journal of Biological Chemistry. 8, 5840-5848 (1993).
- Routh, A., Sandin, S., Rhodes, D. Nucleosome repeat length and linker histone stoichiometry determine chromatin fiber structure. Proceedings of the National Academy of Sciences of the U S A. 105 (26), 8872-8877 (2008).
- Zhou, J., Fan, J. Y., Rangasamy, D., Tremethick, D. J. The nucleosome surface regulates chromatin compaction and couples it with transcriptional repression. Nature Structural & Molecular Biology. 14 (11), 1070-1076 (2007).
- Dorigo, B., Schalch, T., Kulangara, A., Duda, S., Schroeder, R. R., Richmond, T. J. Nucleosome arrays reveal the two-start organization of the chromatin fiber. Science (New York, N.Y.). 306 (5701), 1571-1573 (2004).
- Correll, S. J., Schubert, M. H., Grigoryev, S. a Short nucleosome repeats impose rotational modulations on chromatin fibre folding. The EMBO Journal. 31 (10), 2416-2426 (2012).
- Mcbryant, S. J., Krause, C., Woodcock, C. L., Hansen, J. C. The Silent Information Regulator 3 Protein , SIR3p , Binds to Chromatin Fibers and Assembles a Hypercondensed Chromatin Architecture in the Presence of Salt. Molecular and Cellular Biology. 28 (11), 3563-3572 (2008).
- Schalch, T., Duda, S., Sargent, D. F., Richmond, T. J. X-ray structure of a tetranucleosome and its implications for the chromatin fibre. Nature. 436 (7047), 138-1341 (2005).
- Fan, J. Y., Gordon, F., Luger, K., Hansen, J. C., Tremethick, D. J. The essential histone variant H2A.Z regulates the equilibrium between different chromatin conformational states. Nature Structural Biology. 9 (3), 172-176 (2002).
- Carruthers, L. M., Bednar, J., Woodcock, C. L., Hansen, J. C. Linker histones stabilize the intrinsic salt-dependent folding of nucleosomal arrays: mechanistic ramifications for higher-order chromatin folding. Biochemistry. 37 (42), 14776-14787 (1998).
- Huynh, V. A. T., Robinson, P. J. J., Rhodes, D. A Method for the In Vitro Reconstitution of a Defined "30 nm" Chromatin Fibre Containing Stoichiometric Amounts of the Linker Histone. Journal of Molecular Biology. 345 (5), 957-968 (2005).
- Dorigo, B., Schalch, T. Chromatin fiber folding: requirement for the histone H4 N-terminal tail. J. Mol. Biol. 2836 (03), 85-96 (2003).
- Qian, R. L., Liu, Z. X., et al. Visualization of chromatin folding patterns in chicken erythrocytes by atomic force microscopy (AFM. Cell Research. 7 (2), 143-150 (1997).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon