Method Article
核小体阵列从重组核心组蛋白和核小体定位的DNA组装
摘要
的方法,提出了用于从重组核心组蛋白模型核小体阵列的重构和串联重复的核小体定位的DNA。我们还描述沉降速度实验在分析型超速离心机,和原子力显微镜(AFM)是如何被用来监测核小体阵列饱和的重构后的程度。
摘要
被沿DNA分子重复间隔核心组蛋白八聚体被称为核小体阵列。核小体阵列中的两种方法之一获得:纯化来自体内来源的,或重组的来自重组核心组蛋白的体外和串联重复的核小体定位的DNA。后一种方法具有允许的更组分均匀的组装和精确定位的核小体阵列的优点。沉降速度实验中约大分子通过分析在它们通过离心力作用下溶液迁移率的大小和形状的分析型超速离心机产率的信息。该技术中,随着原子力显微镜,可以用于质量控制,确保多数的DNA模板被重构后饱和的核小体。在这里,我们描述了需要重建的长度毫克量和成分上D中的协议efined核小体阵列适用于染色质的结构和功能的生物化学和生物物理研究。
引言
真核基因组中不存在作为裸DNA,而是被压缩和被结合的蛋白质的组织。 DNA和蛋白质的这些复合物被称为染色质。染色质的基本重复单位是核小体。核小体是由组蛋白八聚体和146个碱基对的DNA周围的组蛋白八聚体包裹约1.6倍1的。组蛋白八聚体是由每个核心组蛋白H2A,H2B,H3和H4的两个副本。被沿DNA分子重复间隔核心组蛋白八聚体被称为核小体阵列。核小体阵列的扩展结构已经被称为10纳米纤维或“珠子串”结构,是目前在低盐条件下,2 体外 。在10纳米纤维能够通过阵列内部的压实和/或跨阵列齐聚2冷凝成高阶结构。这些高次结构的可诱导的盐的存在下,或者可以通过染色质的建筑蛋白的核小体阵列3,4的结合的影响。染色质压实水平呈负相关转录体内 5,6率。最近的研究突出在如分化,肿瘤发展和其他7,8流程的基因组的结构组织的重要性。用核小体阵列来研究染色质结构和功能已变得很普遍。这里,我们描述了从重组核心组蛋白和核小体定位核小体的DNA阵列的组件的方法。
使用重组DNA与核小体定位序列的串联重复允许对包含规则间隔的核小体阵列的重构。两个比较流行的定位序列是5S rRNA基因序列和“601”序列9,10。 601序列是由SELEX实验和铁道部导出Ë强烈持仓核比5S序列11。因此,601阵列的接头DNA长度更均匀。通过凝胶过滤得到的4,12串联重复的核小体定位的DNA。重组组蛋白是从大肠杆菌纯化变性条件下13 大肠杆菌 。使用重组组蛋白允许人们小心地控制在核小体阵列的组蛋白组合物。例如,核心组蛋白轴承特异性突变14或翻译后修饰15,16可以取代野生型核心组蛋白。
沉降速度实验监控解决方案大分子沉淀在施加离心力17的速度。这产生大约样品中大分子的大小和形状的信息。沉降速度实验,因而为研究染色质纤维溶液状态的变化适当的工具结构由于染色质浓缩18。重要的是,它是第一个必要使用沉降速度实验如在核小体阵列重建质量控制步骤。如果DNA的核小体和不结合在适当的摩尔比,该阵列可以是欠或过饱和的核心组蛋白。因此,从沉降速度实验所获得的信息用于确保该DNA被正确地饱和的核小体。使用替代方法来估算的DNA与核小体的饱和度是很重要的,特别是如果处理的是以前未知的DNA模板。因此,我们还描述了使用原子力显微镜(AFM)的核小体阵列的分析方法。 AFM是一个功能强大的技术,允许的许多参数,如饱和度的水平,组蛋白变体的存在的影响或MgCl 2的19,20的作用效果可视化。原子力显微镜也一直APPLI编辑使用时间推移成像21,研究核小体动力学。 体外核小体组装12聚体阵列是特别适合于原子力显微镜的研究,因为他们属于在正确的尺寸范围原子力显微镜成像22。在本研究中,我们使用了核小体阵列的原子力显微镜作为一种质量控制以及肯定从AUC的数据(“眼见为实”)的一种手段。除了简单的可视化,原子力显微镜允许样本作为一个附加度量值的高度轮廓测量。
研究方案
1。重组核心组蛋白八聚体进入组装
理由:在核小体阵列重建的第一步是准备从冻干重组核心组蛋白的原生核心组蛋白八聚体。组蛋白相结合,在等摩尔量和透析的样品出来变性缓冲到缓冲折叠组装成的组蛋白八聚体。
- 纯化和冻干的重组核心组蛋白(H2A,H2B,H3,H4)的描述13。
- 溶解约5毫克每冻干核心组蛋白在3ml去折叠缓冲液(6M盐酸胍,20毫摩尔Tris pH值7.5,5mM DTT的)的。允许每个等分试样溶解在至少一小时。确保没有蛋白仍保留在容器的侧面。
- 使用缓冲展开作为参考测量各组蛋白的吸光度在276纳米。
- 使用他们的消光系数计算出每组蛋白的摩尔浓度( 见表1 对非洲爪蟾组蛋白的消光系数)。如果大部分的蛋白质的溶解组蛋白的吸光度测定浓度进行比较冻干的干重,和估计。
- 确定哪些组蛋白你有最少的痣,因为这将是对所有的组蛋白的极限摩尔数。
- 结合组蛋白在等摩尔量和稀释用缓冲液展开,以1毫克/毫升的最终浓度。
- 将样品放入6-8 kDa的截留分子量透析管。密封管,并放入2升冷的重折叠缓冲液(2M氯化钠,的10mM Tris pH值7.5,1mM EDTA中,5mM的β-巯基乙醇)。
- 透析的样品18小时,在4℃搅拌。确保透析管可以自由大力旋转,否则可能组蛋白沉淀。
- 改变透析缓冲液两次在18小时内( 即改变复性缓冲大约每6小时至熟)。即使形成的沉淀不丢弃样品,一些八聚体可被回收,尽管产率会降低。
注:请参阅步骤2.1-2.2准备柱平衡第二天。
2。组蛋白八聚体通过尺寸排除色谱纯化
理由:第1结论后,样品将包含组蛋白八聚体以及其它组蛋白复合体,例如骨料,H3/H4四聚体,并且H2A/H2B二聚体。组蛋白八聚体将纯化远离使用尺寸排阻色谱法(SEC)的这些其它配合物。
- 连接的HiLoad 16/60 Superdex200 16/60(S200)列的FPLC系统。确保气泡不会进入系统。
- 用0.2微米的过滤水清洗S200列。用0.3毫升/分钟的流速,并设定为0.5MPa背压限制。确保你有足够的水,让列清理过夜。
- 平衡了F的S200柱和样品环PLC采用复性缓冲液。用1升的0.2微米过滤的重折叠缓冲液中。流过以1毫升/分钟的速率和0.5MPa的大约2小时的最大压力柱重折叠缓冲液中。
- 一个平衡的Vivaspin离心浓缩器(50 kDa的截留分子量)用1 ml复性缓冲液中。从透析管中取出样品并离心样品在4℃以除去任何沉淀物。吸取样品上清液进入选矿厂。样品浓缩至约500微升的体积。
- 取出浓八聚体样品到一个新的容器和冲洗选矿厂用1 ml复性缓冲液。浓缩清洗至500微升,并把它添加到八聚体的样品。这有助于挽救剩余的选矿厂任何八聚体。
- 在4℃下旋转样品在1.5ml微量离心管5分钟,以10,000 rpm收集上清液并转移到新管中。这有助于在FPL装样前,以消除任何沉淀物C.
- 样品装载到S200列。在一个单一的纯化加载最多1.5毫升总体积或15毫克八聚体。
- 使用新鲜的复性缓冲液洗脱样品。使用1毫升/分钟的流速和0.5MPa的背压限制。使用两个柱体积(400毫升)复性缓冲液和收集2毫升分数。洗脱过程中监测280nm处的吸光度。不同的物种将洗脱,以减少大小。组蛋白的聚集体,通常洗脱大约45毫升,八聚体在约65毫升,和二聚物在约84毫升( 图1)。
- 通过在18-20%SDS-PAGE上运行的样品分析来自所关注峰的洗脱级分。含有纯化的八聚体的级分应该具有四个核心组蛋白的等摩尔量( 图1)。
- 池包含八聚体纯化的分数,并集中到≤15毫克/毫升的用的Vivaspin离心过滤。如果不稀,八聚体可我撇清NTO H2A/H2B二聚体和H3/H4四聚体。
- 通过测量280nm处的吸光度测定的八聚体的浓度,并计算使用从表1中的消光系数。
- 组蛋白八聚体可以在4℃下保持短期复性缓冲液如果正在存储长期来看,八聚体将更加稳定,在-20℃和在50%的甘油溶液。存储在甘油八聚体应透析到新鲜的复性缓冲液的吸光度测量之前和核小体阵列reconstitutions使用。
3。从DNA核小体阵列和纯化八聚体的重构
理由:核小体阵列的重构要求的组蛋白八聚体和模板DNA在特定的摩尔比进行组合。在2 M氯化钠DNA和组蛋白八聚体的混合物步透析到降低离子强度的缓冲液。逐渐变化,以低盐,确保正确的核小体的形成。获得nucleosomal阵列的模板DNA的组蛋白八聚体饱和所需的水平,需要小规模试验reconstitutions其次是大规模制备重组。由小规模reconstitutions定义的适当的条件下用于指导制备重组。
- 净化所述4,12串联重复的核小体定位的DNA。简言之,隔离用Qiagen试剂盒GigaPrep或类似产品的质粒DNA。设置了一个限制,以便释放模板DNA和消化的质粒DNA,以更小的尺寸(≤700bp的)酶消化。模板DNA可以被纯化,远离使用尺寸排阻色谱法(SEC)的小质粒DNA残余。重力喂养列,约115厘米填充有Sephacryl S-1000珠的高度足以为601 207bp x 12聚体DNA纯化。
注:对于质粒DNA纯化以降低成本,但增加了EFFORT,可以用碱裂解用苯酚/氯仿纯化的DNA,以从大肠杆菌分离质粒DNA 大肠杆菌 23,24。策略用于分离质粒DNA为模板的DNA可以与变化的模板DNA的大小而有所不同。对于SEC它来设置限制在时装最大化的模板和质粒DNA之间的尺寸差异,酶消化是重要的。
- 确定哪些摩尔比(R)为使用为reconstitutions。r等于八聚体的摩尔量与DNA重复的摩尔数之比。对于最初的小规模试验中,0.9,1,和1.1的R值是合适的,如果目的是为了获得饱和的核小体阵列。
- 通过计算八聚体的量加至约的DNA 18微克每一摩尔比要测试的制备小规模的样品。混合DNA和组蛋白八聚体。盐的样品中的终浓度应≥2 M和D的最终浓度NA应该在0.3微克/微升。最终的样品缓冲液条件也应包括的10mM Tris pH值7.8,和1mM EDTA。
- 样品现在已经准备好透析。加载样品到12K-14K MWCO的透析管。透析的样品对减小离子强度如下的缓冲液2L。
10毫米的Tris pH值7.8,1 mM的EDTA:在所有的缓冲区组件。缓冲1(加1 M氯化钠,1 mM的DTT)为5-6小时。缓冲区2过夜(添加0.75 M氯化钠,1 mM的DTT)。缓冲区3(添加2.5 mM氯化钠,1mM的DTT)为5-6小时。缓冲液4℃过夜(添加2.5 mM氯化钠,0.1 mM的PMSF)。
- 从透析管中取出样品,并进行部分4-6。如果小规模的样本产生预期的效果,重复3-6节中的步骤上大的规模。
注:为了节约资源,小规模样品应该有足够的下游筛选试验所需的最低量。为了拥有电子nough样品为此处列出的沉降速度和AFM实验中,50微升样品在0.3毫克/毫升的DNA是合适的。大尺度的大小,制备的样品都依赖于他们的预期用途。甲大规模样品应以同样的方式作为小规模的样品制备。
4。再造核小体阵列的沉降速度分析
理由:在溶液中分子的大小和形状的贝克曼XL-A / I分析型超速离心机产量信息沉降速度实验。低盐条件下进行的沉降速度实验被用来确定到该重新构成的阵列是饱和与核小体的程度。
- 样品稀释到大约0.5在用TEN缓冲液(10mM的Tris pH值7.8,1毫摩尔EDTA,2.5 mM氯化钠)260 nm的吸光度。载入400μl的样品导入细胞用2扇区核心,具有在一侧的样品和参考价值。CE(TEN缓冲液)的其他25。保持参考半月板比样品液面较高的( 例如,添加420微升参比溶液)。
- 加载细胞进入转子,并适当地使用对细胞和转子25的底部的哈希标记对齐单元格。轻轻擦拭用压缩空气细胞的镜片。
- 打开XL-A/XL-I离心机,插入转子,并附加和说明书中描述的固定光学元件。打开蛋白质组实验室软件,然后选择文件:新。
- 设置一个单一的扫描运行在3,000 rpm和25℃的温度下在选项下,选择停止最后一次扫描后,并运行径向校准(径向校准是没有必要的,如果转子是在AUC最后使用的转子)。
- 对于每一个细胞,命名为样本,选择的波长(260纳米),选择吸收,并选择您的计算机上保存文件的位置。开始的单次扫描运行。
- 使用单次扫描,以确定相应的RA拨号扫描长度( 图2A)。减少将被扫描的单元格的长度减小了运行时间。但是,一定要包括样品半月板和扩展年底非常接近,或在池的底部。
注:单扫描允许测量区是由起始的测量只是示例弯月面( 图2A)之前缩短。最AUC的运行期间生成的扫描应该有边界馏分过去的弯液面以及稳定台地( 图2B)。对AUC细胞脏的镜头可以产生较大的尖峰扫描,这可能会影响数据的分析。该软件的UltraScan包括手册,该手册是关于超速离心分析数据26的分析信息的重要资源。
- 使用XL-A/XL-I控制面板,输入0的速度,然后按下开始。这将促使离心机腔抽成真空和所有OW的腔室的温度达到平衡。等待1小时,以允许温度平衡开始运行之前。
- 使用该软件来设置运行起来所需的速度和扫描次数。更高的速度提高分辨率,但应该至少收集20次扫描(最好是多个)之前,样品已经沉降到单元的底部。它可以方便地叠加在过去的几年扫描(在完成选项菜单),以监控运行的进程,并检查潜在的问题,如漏水细胞。监测的第一对夫妇的扫描,以确保正常运行。
5。编辑和分析沉降速度数据
理由:原沉降速度数据应该由程序产生一个扩散沉降修正系数分布进行分析。反过来这信息表示重构样品的馏分,它包含一个给定的饱和度, 例如 的,如果使用的是12 - 聚体DNA为模板,将有可能确定重构阵列具有10个,11个和12个核小体/ DNA的级分。
- 用AUC数据分析软件,如的UltraScan编辑数据26。扫描数据的编辑处理,必须对每个小区进行,并创建用于后续分析的数据集。适当的编辑扫描涉及定义的样本液面,减少了数据的感兴趣区域,并限定了基线以及平台区域( 图2)。无用的扫描,也可以从设置在这一点上的数据删除。然而,因为它可以在分析过程中除去不想要的扫描,建议所有的扫描保持在这一点上。
- 我们强烈建议增强面包车HOLDE-Weischet方法被用于分析数据27。此分析方法可以校正扩散在运行过程中的影响,并产生沉降系数的积分分布。特别是,增强面包车HOLDE-Weischet方法(如在实施的UltraScan)将允许包括一些缺乏稳定的高原,或包含边界部分已不清除半月板在分析28扫描。
- 确定沉降系数的组装阵列的分布。为601-12(207bp,12 -聚体)的核小体阵列代表性的结果示于图3。
- 如果小规模样品之一包含具有沉降系数的期望范围的阵列,然后以增加的比例开始第3重复上述过程。如果没有小规模的样本具有沉降系数的合理分配,然后用一个新的R系列开始于第3条重复了小规模试验。
6。可视化核小体阵列采用原子力显微镜(AFM)
原理:原子力显微镜允许nucleosoma的饱和水平的可视化升阵列。这种技术通过补充AUC和限制性内切酶消化沉降速度作为一种质量控制检测。
- 使用上述方法,获得良好的饱和核小体阵列( 图5A)。
- 准备APTES通过将处理过的云母滑动〜30μl的1:1000稀释的商业(Sigma-Aldrich公司(3 - 氨基丙基)三乙氧基硅烷A3648-100ML)的在0.22微米过滤的超纯水进行30分钟。
- 30分钟后,用过滤水冲洗APTES关闭并轻轻擦干他们在一个氮气流量。
- 放置在幻灯片上适当稀释的核小体阵列的样品(约1.5纳克/微升),孵育覆盖在室温下搅拌15分钟。
- 冲洗过滤样品缓冲液样了,像以前一样干,并在原子力显微镜(在这种情况下,一个庇护研究MFP的三维原子力显微镜)的阶段发生滑动。
- 首先,成像2×2μm的扫描,512扫描线。计数核小体的数量,以确定SA的电平turation( 图5B)。
- 理想情况下你应该与良好的分离核小体阵列幻灯片图像区域。
- 对于较高分辨率的图像,一个500×500纳米的扫描可以得到( 图5C)。
- 图像可以被夷为平地,利用庇护所提供的多功能三维软件进行分析。
- 每个图像可被划分为四个象限,并放大数字化以获得良好分离的阵列和多个空闲的手线的清晰的视野可以为核小体( 图5C,右面板高度跟踪获得通过阵列和高度轮廓绘制和图5D)。
- 几个这样的图片应该为每种类型的数组涵盖几百核小体进行分析。高度轮廓被记录,分类和绘制在MS Excel中。
结果
为了说明该协议,我们重组从重组非洲爪蟾核心组蛋白和DNA组成的12串联601定位序列(601 207×12)的207 bp的重复序列核小体阵列。我们首先组装从冻干的核心组蛋白八聚体原生,然后用S200柱( 图1A)纯化,八聚体,通过FPLC。更大的络合物较早洗脱从S200列。组蛋白一般洗脱顺序为:非特异性组蛋白聚集体,组蛋白八聚体,四聚体H3/H4和H2A/H2B二聚体( 图1A)。从S200柱的峰用SDS-PAGE进行分析。纯化的八聚体组分的凝胶应说明四个组蛋白( 图1B)等摩尔量。需要注意的是爪蟾 H2A和H2B有只相差约200 Da和因此它们出现在SDS凝胶上的单一条带的分子量。 H3/H4四聚体和二聚体H2A/H2B将洗脱后,但版本Ÿ接近组蛋白八聚体峰。当选择其中部分保持谨慎的做法是放弃对出现在色谱图中八聚体峰的结束部分。这些组分可具有非八聚体组蛋白复合物的量增加。在这种情况下,级分64-67合并,并且保存为核小体重构阵列( 图1)。
内的UltraScan有两种不同的查看从增强面包车HOLDE-Weischet分析中获得的沉降系数的扩散校正后的分配的方法。红线代表沉降系数的积分分布。为在图表上任意给定点中,y轴表示具有沉降系数等于或小于在X轴所指示的值的样本的分数。因此,垂直线指示的均匀样品,而多相样品将具有正斜率的曲线。绿线是德导数的积分分布。下一个峰的面积正比于具有该沉降系数样品的级分。完全饱和的601 207×12核小体阵列有29S 29的沉降系数。在图3所示的示例中,数据表明,该阵列的约70%是饱和的和30%的过饱和。
原子力显微镜已经从一个新兴技术,以流行的互补的方法来研究染色质组织体外 。这里,我们已经使用原子力显微镜一起分析超速离心法的重构后建立模板饱和的程度。 图6中的大部分〜27S阵列( 图4A)用于成像的含有10-11核小体( 图4B),表明了原子力显微镜和AUC结果之间非常吻合。这里获得的AFM结果从而验证了该接近并使其成为可靠的技术表征核小体阵列。
| 组蛋白 | Σ276,蛋白质折叠厘米-1 M -1 | 分子量大 |
| H2A | 4,350 | 13,960 |
| H2B | 7,250 | 13,774 |
| H3 | 4,640 | 15,273 |
| H4 | 5,800 | 11,236 |
| 八聚体 | 44,080 | 108486 |
表1中。消光系数和非洲爪蟾核心组蛋白和复性组蛋白八聚体的分子量。
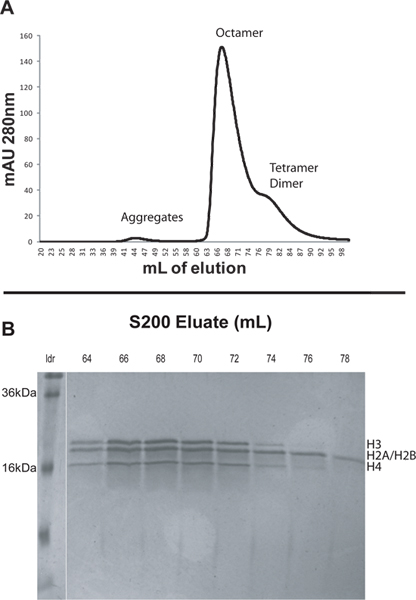
图1。从第2条所描述的S200列A洗脱曲线。在约44 ml用中小峰是由于大的组蛋白聚集体。在67毫升主峰是组蛋白八聚体。从76-90毫升的宽肩包含H3/H4四聚体和二聚体H2A/H2B。B. 20%SDS-PAGE电泳从S200柱选定馏分。从八聚体峰初馏分必须被收集,因为它们是最有可能被非八聚体组蛋白复合体被污染。在这种情况下,分数64-67分并保存为核小体阵列重建。 点击这里查看大图 。

图2。 A. ProteomeLab软件抓屏在3,000 rpm采集单次扫描(与LA贝尔所加)。需要注意的是样品不沉淀在这样低的速度。在单次扫描中用于设置测量范围的AUC细胞扫描(步骤4.6)中使用的UltraScan程序(与加标签)26获得的一系列沉降速率扫描。B.抓屏。该系列扫描编辑,生成分析数据集(见步骤5.1)。
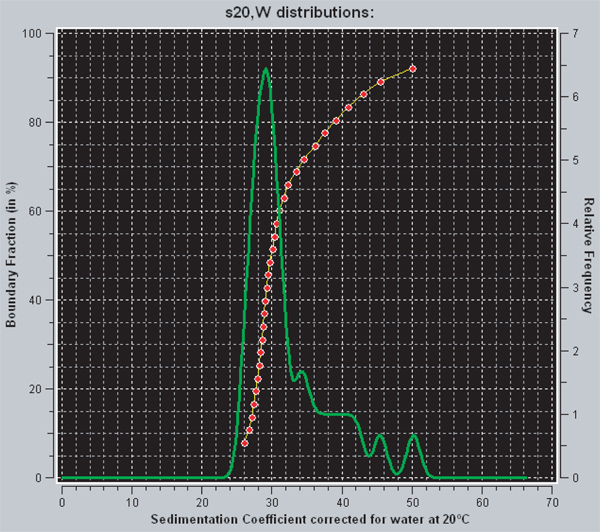
图3:从UltraScanII屏幕捕获,红色和绿色线是两种不同的方法用于查看的UltraScan沉降系数的分布。红线是沉降系数的积分分布,而绿色是衍生物。在解释额外的信息可以在结果一节中找到。
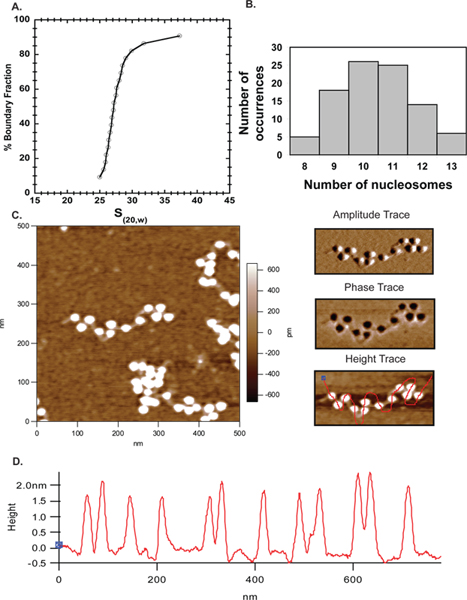
图4。AFM。为601 207×12阵列的组装用鼠标八聚物的平均沉降系数A.沉淀速度分布是27S。B.用同样的阵列,通过原子力显微镜成像和核小体在几个图像计数的数量绘制在MS Excel中。情节显示,大多数的阵列有10-11核确凿的AUC数据。C.一个500×500 601 207×12核阵列连接器的DNA纳米扫描清晰可见。右上方的面板是振幅跟踪和中间右侧面板是相位跟踪在C中所示的阵列。右下方的面板是在左边,但用另一只手线通过核小体绘制,以确定核小体的高度轮廓所示的相同高度跟踪秒。在上图中的核小体D.身高型材证明所有的核小体范围内1.5〜2.5 nm的高度上年度报告40。
讨论
核小体模型阵列是染色质的结构和功能的体外研究中一个非常有用的工具。例如,它们已被广泛用于研究的染色质纤维凝结的机理在溶液30-34,并且使得有可能获得一个tetranucleosome 35的X-射线结构。最近它们已被证明在解密的特定核心组蛋白变体,突变体和翻译后修饰14-16,36结构效应是有用的。在这里,我们描述了用于从核小体定位的DNA和重组核心组蛋白模型核小体阵列的组件的一般方法。
核小体阵列的八聚体纯化和601 207×12的DNA的重组是直接的,涉及多个透析步骤,按顺序从2米至2.5毫米降低NaCl浓度。该协议中最困难的部分是使用R-值,得到所需的统板饱和度。适当的r值是第一凭经验确定在小规模,然后重复在更大的规模,以产生用于实验的核小体阵列。在我们的例子中,我们试图获得大多饱和,每个DNA核小体12 601 207×12的DNA模板。完全饱和601 207×12核小体阵列的沉降系数为29 s,而每个DNA沉积物只有11核小体在27S〜29相同的DNA模板。因此,在分析型超速离心机沉降速率提供了一种用于确定重构后的核小体的饱和电平非常敏感的方法。我们使用增强的面包车HOLDE-Weischet方法,这将产生一个扩散校正的沉降系数分布分析我们的数据。这个信息非常重要,因为它告诉人们如何同质或异质的样品溶后。换句话说,对于一个601 207×12的DNA模板,它表明具有每DNA的核小体12,每DNA的核小体11 等 图3显示了增强面包车的重构以1.1一个r 601 207×12核小体阵列HOLDE-Weischet分析的结果,样品的组分,其中大致该阵列的30%以上的饱和。试样的实用程序通常依赖于饱和阵列的百分比,但依赖于阵列将被用来英寸某些应用可能需要非常均匀的和/或饱和的阵列的实验。为提高核小体阵列的均匀性和饱和度有许多方法存在的。
在这种情况下,过饱和的阵列可通过选择性沉淀在加入MgCl 2的37中被除去。更均匀的阵列,也可以通过使用蔗糖密度梯度离心法,制备型凝胶电泳,离子交换层析10,13纯化样品中获得。一种改进的NUcleosomal阵列重建方法调用,通过盐透析38,39增加短期竞争DNA的样本重建之前。这允许一个装配核小体阵列与过量的组蛋白八聚体的无DNA模板过饱和。核小体阵列采用竞争DNA可能会需要用于去除竞争DNA和组蛋白的额外的纯化步骤配制。
即使在小规模试点阵列组件,它有可能在重组大规模核小体阵列将无法正确饱和。为了避免浪费的模板DNA与组蛋白八聚体的样品中,有可能修复过的或饱和的阵列下。如果阵列是过度饱和的,额外的DNA可以被添加到样品中。如果数组下饱和,多余的八聚体可以被添加到纠正。然而,加入八聚体已经进入透析低盐阵列可导致八聚体的解离。如果增加额外的Ôctamer或DNA到阵列,增加额外的八聚体或DNA的大样前透析阵列回2 M氯化钠。从高分到低分盐,每步3.4重复步骤透析用于固定试样。八聚体,以便纠正阵列添加量可以从沉降系数饱和阵列29下估计。调节核小体阵列的样品之后,饱和电平的测量应再次进行。
虽然原子力显微镜是一种功能强大的方法表征核小体阵列,它是复杂的,劳动密集的过程。这使得一个贫穷的技术筛选小规模reconstitutions,但一个优秀的技术的大规模样本的特性和染色质组织的研究。此前我们已经用原子力显微镜可视化由macroH2A缺失构建,这是“超敏感”,甚至低浓度的氯化镁生成的“宏观”的粒子。 LikewiSE,蒙特尔等人(2009)显示,H2A BBD变异导致与野生型数组的数组核要更“开放”。因此,AFM是一种可靠的技术用于质量控制,以及对染色质纤维结构的一般研究。
披露声明
作者没有利益冲突。
致谢
这项工作是由美国国立卫生研究院资助GM45916和GM66834到JCH从Rett综合症国际基金会的奖学金,以AK这项工作的支持还得到美国国立卫生研究院授予GM088409和霍华德休斯医学研究所吉隆坡的贡献
材料
| Name | Company | Catalog Number | Comments |
| (3-Aminopropyl)triethoxysilane | Sigma-Aldrich | A3648-100ML | |
| 6-8 kDa MWCO Dialysis Tubing | Fisher | 21-152-5 | |
| HiLoad Superdex 200 16/60 Column | GE | 17-1069-01 | |
| Vivaspin 50 kDa MWCO Centrifugal Concentrator | Sartorius | VS2031 | |
| 12-14 kDa MWCO Dialysis Tubing | Fisher | 08-667A | |
| Illustra Sephacryl S-1000 Superfine | GE | 17-0476-01 | |
| XL-A/I Analytical Ultracentrifuge | Beckman-Coulter |
参考文献
- Luger, K., Mader, A., Richmond, R., Crystal Sargent, D. structure of the nucleosome core particle at 2.8 A resolution. Nature. 7, (1997).
- Hansen, J. C. Conformational dynamics of the chromatin fiber in solution: determinants, mechanisms, and functions. Annual Review of Biophysics and Biomolecular Structure. 31, 361-392 (2002).
- McBryant, S., Adams, V., Hansen, J. Chromatin architectural proteins. Chromosome Research. 14 (1), 39-51 (2006).
- Hansen, J. C., Ausio, J., Stanik, V. H., van Holde, K. E. Homogeneous reconstituted oligonucleosomes, evidence for salt-dependent folding in the absence of histone H1. Biochemistry. 28 (23), 9129-9136 (1989).
- Szerlong, H. J., Prenni, J. E., Nyborg, J. K., Hansen, J. C. Activator-dependent p300 acetylation of chromatin in vitro: enhancement of transcription by disruption of repressive nucleosome-nucleosome interactions. The Journal of Biological Chemistry. 285 (42), 31954-31964 (2010).
- Cirillo, L. A., Lin, F. R., Cuesta, I., Friedman, D., Jarnik, M., Zaret, K. S. Opening of compacted chromatin by early developmental transcription factors HNF3 (FoxA) and GATA-4. Molecular Cell. 9 (2), 279-289 (2002).
- Nguyen, C., Gonzales, F. chromatin structure associated with methylation-induced gene silencing in cancer cells: correlation of accessibility, methylation, MeCP2 binding and acetylation. Nucleic Acids Research. 29 (22), 4598-4606 (2001).
- Cuesta, I., Zaret, K. S., Santisteban, P. The forkhead factor FoxE1 binds to the thyroperoxidase promoter during thyroid cell differentiation and modifies compacted chromatin structure. Molecular and Cellular Biology. 27 (20), 7302-7314 (2007).
- Simpson, R. T., Stafford, D. W. Structural features of a phased nucleosome core particle. Proceedings of the National Academy of Sciences of the U S A. 80 (1), 51-55 (1983).
- Lowary, P. T., Widom, J. New DNA sequence rules for high affinity binding to histone octamer and sequence-directed nucleosome positioning. Journal of Molecular Biology. 276 (1), 19-42 (1998).
- Lowary, P., Widlund, H., Cao, H. Sequence motifs and free energies of selected natural and non-natural nucleosome positioning DNA sequences. Journal of Molecular Biology. 288, 213-229 (1999).
- Gordon, F., Luger, K., Hansen, J. C. The Core Histone N-terminal Tail Domains Function Independently and Additively during Salt-dependent Oligomerization of Nucleosomal Arrays *. The Journal of Biological Chemistry. 280 (40), 33701-33706 (2005).
- Luger, K., Rechsteiner, T. J., Richmond, T. J. Expression and purification of recombinant histones and nucleosome reconstitution. Methods in Molecular Biology (Clifton, N.J.). 119 (4), 1-16 (1999).
- McBryant, S. J., Klonoski, J., et al. Determinants of histone H4 N-terminal domain function during nucleosomal array oligomerization: roles of amino acid sequence, domain length, and charge density. The Journal of Biological Chemistry. 284 (25), 16716-16722 (2009).
- Ma Shogren-Knaak, ., Fry, C. J., Peterson, C. L. A native peptide ligation strategy for deciphering nucleosomal histone modifications. The Journal of Biological Chemistry. 278 (18), 15744-158 (2003).
- Lu, X., Simon, M. The effect of H3K79 dimethylation and H4K20 trimethylation on nucleosome and chromatin structure. Nat Struct Mol Biol. 15 (10), 1122-1124 (2008).
- Ausio, J. Analytical Ultracentrifugation for the Analysis of Chromatin Structure. Biophysical Chemistry. 86 (2-3), 141-153 (2000).
- Hansen, J., Kreider, J., Demeler, B., Fletcher, T. Analytical ultracentrifugation and agarose gel electrophoresis as tools for studying chromatin folding in solution. Methods. 12 (1), 62-72 (1997).
- Montel, F., Menoni, H., et al. The dynamics of individual nucleosomes controls the chromatin condensation pathway: direct atomic force microscopy visualization of variant chromatin. Biophysical Journal. 97 (2), 544-5453 (2009).
- Muthurajan, U. M., McBryant, S. J., Lu, X., Hansen, J. C., Luger, K. The linker region of macroH2A promotes self-association of nucleosomal arrays. The Journal of Biological Chemistry. 286 (27), 23852-23864 (2011).
- Shlyakhtenko, L. S., Lushnikov, A. Y., Lyubchenko, Y. L. Dynamics of nucleosomes revealed by time-lapse atomic force microscopy. Biochemistry. 48 (33), 7842-7848 (2009).
- Lohr, D., Bash, R., Wang, H., Yodh, J., Lindsay, S. Using atomic force microscopy to study chromatin structure and nucleosome remodeling. Methods (San Diego, Calif). 41 (3), 333-341 (2007).
- Dyer, P. N., Edayathumangalam, R. S., et al. Reconstitution of nucleosome core particles from recombinant histones and DNA. Methods in Enzymology. 375, 23-44 (2004).
- Sambrook, J., Russell, D. . Molecular cloning: a laboratory manual. , (2001).
- Balbo, A., Zhao, H., Brown, P. H., Schuck, P. Assembly, loading, and alignment of an analytical ultracentrifuge sample cell. J. Vis. Exp. (33), e1530 (2009).
- Demeler, B. UltraScan: a comprehensive data analysis software package for analytical ultracentrifugation experiments. Modern Analytical Ultracentrifugation: Techniques. , 210-230 (2005).
- Holde, K. V., Weischet, W. Boundary analysis of sedimentation velocity experiments with monodisperse and paucidisperse solutes. Biopolymers. 17 (6), 1387-1403 (1978).
- Demeler, B., van Holde, K. E. Sedimentation velocity analysis of highly heterogeneous systems. Analytical Biochemistry. 335, 279-288 (2004).
- Hansen, J., Lohr, D. Assembly and structural properties of subsaturated chromatin arrays. Journal of Biological Chemistry. 8, 5840-5848 (1993).
- Routh, A., Sandin, S., Rhodes, D. Nucleosome repeat length and linker histone stoichiometry determine chromatin fiber structure. Proceedings of the National Academy of Sciences of the U S A. 105 (26), 8872-8877 (2008).
- Zhou, J., Fan, J. Y., Rangasamy, D., Tremethick, D. J. The nucleosome surface regulates chromatin compaction and couples it with transcriptional repression. Nature Structural & Molecular Biology. 14 (11), 1070-1076 (2007).
- Dorigo, B., Schalch, T., Kulangara, A., Duda, S., Schroeder, R. R., Richmond, T. J. Nucleosome arrays reveal the two-start organization of the chromatin fiber. Science (New York, N.Y.). 306 (5701), 1571-1573 (2004).
- Correll, S. J., Schubert, M. H., Grigoryev, S. a Short nucleosome repeats impose rotational modulations on chromatin fibre folding. The EMBO Journal. 31 (10), 2416-2426 (2012).
- Mcbryant, S. J., Krause, C., Woodcock, C. L., Hansen, J. C. The Silent Information Regulator 3 Protein , SIR3p , Binds to Chromatin Fibers and Assembles a Hypercondensed Chromatin Architecture in the Presence of Salt. Molecular and Cellular Biology. 28 (11), 3563-3572 (2008).
- Schalch, T., Duda, S., Sargent, D. F., Richmond, T. J. X-ray structure of a tetranucleosome and its implications for the chromatin fibre. Nature. 436 (7047), 138-1341 (2005).
- Fan, J. Y., Gordon, F., Luger, K., Hansen, J. C., Tremethick, D. J. The essential histone variant H2A.Z regulates the equilibrium between different chromatin conformational states. Nature Structural Biology. 9 (3), 172-176 (2002).
- Carruthers, L. M., Bednar, J., Woodcock, C. L., Hansen, J. C. Linker histones stabilize the intrinsic salt-dependent folding of nucleosomal arrays: mechanistic ramifications for higher-order chromatin folding. Biochemistry. 37 (42), 14776-14787 (1998).
- Huynh, V. A. T., Robinson, P. J. J., Rhodes, D. A Method for the In Vitro Reconstitution of a Defined "30 nm" Chromatin Fibre Containing Stoichiometric Amounts of the Linker Histone. Journal of Molecular Biology. 345 (5), 957-968 (2005).
- Dorigo, B., Schalch, T. Chromatin fiber folding: requirement for the histone H4 N-terminal tail. J. Mol. Biol. 2836 (03), 85-96 (2003).
- Qian, R. L., Liu, Z. X., et al. Visualization of chromatin folding patterns in chicken erythrocytes by atomic force microscopy (AFM. Cell Research. 7 (2), 143-150 (1997).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。