Method Article
組換えコアヒストンおよびヌクレオソームポジショニングDNAからのヌクレオソーム配列の組み立て
要約
この方法は、組換えコアヒストンからモデルクレオソームアレイの再構成および縦列反復ヌクレオソームのポジショニングのDNAのために提示されている。また、分析用超遠心で沈降速度実験、および原子間力顕微鏡(AFM)は、再構成後のヌクレオソームアレイ飽和の程度をモニターするために使用される方法を記載している。
要約
繰り返しDNA分子に沿って配置されているコアヒストン八量はヌクレオソームアレイに呼ばれています。ヌクレオソームアレイは2つの方法のいずれかで得られる:in vivoでの情報源からの精製、または組換えコアヒストンからのin vitroで再構成および縦列反復ヌクレオソームのポジショニングのDNA。後者の方法は、以上の組成が均一のアセンブリーと正確に配置ヌクレオソームの配列を可能にするという利点があります。分析用超遠心で沈降速度実験は、それらが、遠心力下で溶液を通って移動する速度を解析することにより高分子のサイズおよび形状に関する情報を得た。この技術は、原子間力顕微鏡とともに、DNAテンプレートの大部分は再構成後にヌクレオソームで飽和されることを保証する品質管理のために使用することができる。ここでは、長さのミリグラム量を再構成するために必要と組成的にDのプロトコルを記述クロマチン構造および機能の生化学的および生物物理学的研究に適しefinedクレオソームアレイ。
概要
真核生物のゲノムは、裸のDNAとして存在していないのではなく、圧縮し、結合したタンパク質で編成されています。 DNAおよびタンパク質のこれらの複合体は、クロマチンとして知られている。クロマチンの基本繰り返し単位はヌクレオソームである。ヌクレオソームは、ヒストンオクタマーと約1.6倍の1オクタマーヒストンに巻きDNAの146塩基対で構成されています。ヒストン八量体は、2つのコピーのコアヒストンH2A、H2B、H3、およびH4の各々から構成されている。繰り返しDNA分子に沿って配置されているコアヒストン八量はヌクレオソームアレイに呼ばれています。ヌクレオソームアレイの拡張構造は、10nm繊維又は「ストリング上のビーズ」構造と呼ばれ、低塩条件2の下で、インビトロで存在されている。 10 nmの繊維は、アレイ内の圧縮および/ またはアレイ間オリゴマー2を介してより高次の構造へと凝縮することができる。これらの高次構造は、塩の存在下で誘導することができるまたはヌクレオソーム配列3,4にクロマチン建築のタンパク質の結合を介して影響を与えることができる。クロマチン圧縮のレベルは反比例ビボ 5,6 内の転写速度と相関している。最近の研究では、このような分化、癌の発生、その他7,8のようなプロセスにおけるゲノムの構造組織の重要性を強調しています。クロマチン構造及び機能を研究するためのヌクレオソームアレイの使用が普及している。ここでは、組換えコアヒストンとヌクレオソーム位置決めDNAからのヌクレオソームの配列の組立のための方法を説明します。
ヌクレオソーム位置決めシーケンスのタンデムリピートで組換えDNAを使用すると、規則的な間隔のヌクレオソームを含む配列の再構成を可能にします。より一般位置決め配列の二つは、5S rRNA遺伝子配列と「601」配列である9,10。 601配列を、SELEX実験やMORから派生したE強く5Sシーケンス11よりヌクレオソームを配置します。従って、アレイ601のリンカーDNAの長さはより均質である。縦列反復ヌクレオソーム位置決めDNAをゲル濾過4,12によって得られる。組換えヒストンは、E.から精製される変性条件13歳未満の大腸菌 。組換えヒストンの使用は1注意深くヌクレオソームアレイのヒストン組成を制御することができます。例えば、特定の変異14または翻訳後修飾を有するコアヒストン15,16は、野生型コアヒストンの代わりに用いることができる。
沈降速度実験は、遠心力17の下で、溶液中の高分子の沈降速度を監視します。これは、試料中の巨大分子の大きさや形状に関する情報が得られる。沈降速度実験従って、クロマチン繊維中の溶液の状態の変化を研究するための適切なツールであるクロマチン凝縮18による構造。重要なことは、ヌクレオソーム再構成アレイにおける品質管理工程として沈降速度実験を使用することがまず必要である。 DNAおよびヌクレオソームが適切なモル比で結合されない場合、アレイは過小または過剰飽和コアヒストンとすることができる。このように、沈降速度実験から得られた情報は、ヌクレオソームDNAが適切で飽和されていることを確認するために使用される。これは、以前に特徴づけられていないDNAテンプレートを扱う場合は特に、ヌクレオソームでDNAの飽和を推定するための代替方法を使用することが重要である。したがって、我々はまた、原子間力顕微鏡(AFM)を用いてヌクレオソームアレイの分析のための方法を記載している。 AFMは、例えば、飽和レベル、ヒストン変異体又は19,20のMgCl 2の影響の存在の効果などのパラメータの数の効果の可視化を可能にする強力な技術である。 AFMはまた、アプリをされているタイムラプスイメージング21を使用してヌクレオソームのダイナミクスを研究するエド。 インビトロでは 、彼らはAFMイメージング22に適した大きさの範囲内に属しているため、ヌクレオソーム12-merの配列は原子間力顕微鏡の研究に特に適している組み立て。本研究では、品質管理だけでなく、(「百聞は一見にしかず」)のAUCからデータを肯定する手段として、ヌクレオソームアレイのAFMを使用している。単純な可視化に加えて、AFMは、追加のメトリックとしての試料の高さプロファイルの測定を可能にする。
プロトコル
1。八量への組換えコアヒストンの組立
理由:ヌクレオソームのアレイ再構築の最初のステップは、凍結乾燥した組換えコアヒストンからネイティブコアヒストン八量を準備することです。ヒストンタンパク質は等モル量で混合し、リフォールディング緩衝液中に変性バッファからサンプルを透析することによってヒストン八量体に組み立てられる。
- 13に記載の組換えコアヒストン(H2A、H2B、H3、H4)を精製し、凍結乾燥する。
- アンフォールディングバッファー(6Mグアニジニウム塩酸、20mMのトリスpH7.5、5mMのDTT)3mlにそれぞれ凍結乾燥されたコアヒストンの約5ミリグラムを溶解する。各アリコートは、1時間以上溶解させる。全くタンパク質が容器の側面に残っていないことを確認してください。
- 参照として展開緩衝液を用いて、276 nmでの各ヒストンの吸光度を測定します。
- それらの吸光係数を用いて、各ヒストンのモル濃度を計算した( 表1を参照 アフリカツメガエルヒストン吸光係数のため)。タンパク質の大部分が溶解している場合は、凍結乾燥し、乾燥重量、および推定値にヒストンの吸光度決定された濃度を比較してください。
- これはすべてのヒストンのためのモル制限数であるように、あなたが最も少ないモル数を持っているヒストンを決定します。
- 等モル量のヒストンを結合および1 mg / mlの最終濃度になるようにバッファーを展開で希釈する。
- 6-8 kDaのMWCO透析チューブにサンプルを置きます。チューブを密封し、コールドリフォールディング緩衝液(2M NaCl、10mMのトリスpH7.5、1mMのEDTA、5mMのβ-メルカプトエタノール)2Lに配置します。
- 攪拌しながら4℃で18時間のためのサンプルを透析。透析チューブが自由に、激しく回転させることができ、そうでなければ、ヒストンが沈殿することを確認してください。
- 18時間の期間に2回透析緩衝液を変更(完了するまで、 すなわち 、すべての6時間約リフォールディング緩衝液に変更)。沈殿物が形成されていても破棄しない収率が低下するが、サンプルは、いくつかの八量体を回収することができる。
注:次の日のためにカラム平衡を準備するためにステップ2.1から2.2を参照してください。
2。サイズ排除クロマトグラフィーによるヒストン八量の精製
理由:セクション1で締結した後、サンプルはヒストン八量だけでなく、このような凝集、H3/H4四量体、およびH2A/H2B二量体などの他のヒストン複合体が含まれています。ヒストン八量体は、サイズ排除クロマトグラフィー(SEC)を用いて、これらの他の複合体から離れて精製される。
- FPLCシステムにハイロード16/60をSuperdex200 16/60(S200)のカラムを接続します。気泡がシステムに入っていないことを確認してください。
- 0.2μmのろ過された水で、S200列をきれいにしてください。 0.3ml /分の流速を使用し、0.5MPaでの背圧の上限を設定する。あなたは十分な水を持っていることを確認し、列は一晩をきれいにすることができます。
- FのS200カラムとサンプルループを平衡リフォールディング緩衝液を使用して、PLC。 0.2μmのフィルター再生バッファー1リットルを使用しています。 1ミリリットル/分と約2時間0.5メガパスカルの圧力の速度でカラムを介してバッファをリフォールディング流れ。
- リフォールディング緩衝液1mlでビバスピン遠心濃縮(50 kDaのMWCO)を平衡化させます。透析チューブからサンプルを取り出して、任意の沈殿物を除去するために4℃でサンプルを遠心分離する。コンセントレータへのサンプルの上清をピペットで。約500μlの容量にサンプルを濃縮。
- 新しいコンテナに集中八量サンプルを削除し、リフォールディング緩衝液1ミリリットルに濃縮装置をすすぐ。 500μlにリンスを下に集中し、八量サンプルに追加します。これは、コンセントレータに残っオクタマーをサルベージするのに役立ちます。
- 4℃にて10,000 rpmで5分間、1.5mlマイクロチューブ内の試料をスピン上清を回収し、新しいチューブに移す。これは、FPL上のサンプルをロードする前に任意の沈殿物を除去するのに役立ちますC.
- S200カラムにサンプルをロードします。シングル精製における最大1.5ミリリットルの全容量または15 mgの八量をロードします。
- 作りたてのリフォールディング緩衝液を使用してサンプルを溶出させる。 1ml /分の流速および0.5MPaでの背圧の上限を使用する。リフォールディング緩衝液2カラム容量(400ミリリットル)を使用し、2ミリリットルの分画を収集します。溶出中に280 nmの吸光度をモニターする。異なる種は、サイズが大きい順に溶出する。ヒストン凝集体は通常、約84ミリリットル( 図1)には、約45ミリリットル、オクタマー約65ミリリットルであり、二量体を溶出させる。
- 18から20まで%SDS-PAGEでサンプルを実行することで、関心のあるピークからの溶出画分を分析します。精製された八量を含む画分を4コアヒストンタンパク質の等モル量( 図1)を有するべきである。
- 精製された八量が含まれている画分をプールし、ビバスピン遠心濾過して15 mg / mlのを≤濃縮。希薄を放置すれば、八量私の関連付けを解除可能NTO H2A/H2B二量体およびH3/H4四量体。
- 280nmでの吸光度を測定することによって八量濃度を測定し、表1からの吸光係数を用いて計算する。
- ヒストン八量体を4℃でリフォールディングバッファー中で短期的に保つことができる長期間記憶されている場合、オクタマーを-20℃で50%グリセロール溶液中でより安定である。グリセロールに格納八量は吸光度測定の前に新鮮なリフォールディング緩衝液に透析し、ヌクレオソームのアレイ再構成の中で使用されるべきである。
3。 DNAからのヌクレオソーム配列と精製八量の再構成
理由:ヌクレオソームアレイの再構成は、ヒストン八量と鋳型DNAを特定のモル比で結合されている必要があります。 2MのNaCl中のDNAとヒストン八量体の混合物は、イオン強度を低下させるバッファに透析ステップである。低塩への段階的な変化は、適切なヌクレオソームの形成を確実にします。 Nの取得鋳型DNAのヒストンオクタマー飽和所望のレベルにucleosomalアレイは、大規模な取再構成に続いて小規模なテスト再構成の必要になります。小規模の再構成の適切な条件によって定義を分取再構成を誘導するために使用される。
- 4,12記載のように縦列反復ヌクレオソーム位置決めDNAを精製する。簡単に言うと、キアゲンGigaPrepキットや類似の製品を使用してプラスミドDNAを分離します。セットアップの制限酵素の鋳型DNAを解放し、小さいサイズ(≤の700bpの)へのプラスミドDNAを消化するためにダイジェストを。鋳型DNAは、次いでサイズ排除クロマトグラフィー(SEC)を用いてプラスミドDNA小さな残存物から精製することができる。セファクリルS-1000のビーズを充填した約115センチの高さで、列を与え重力は601 207bp X 12量体DNAの精製の ために十分である。
(注)減少したコストでプラスミドDNA精製のためではなく、EFFの増加ORT、一方はE.からプラスミドDNAを単離するためにフェノール/クロロホルムDNAの精製をアルカリ溶解を使用することができ大腸菌 23,24。プラスミドDNA鋳型からDNAを分離するための戦略は、鋳型DNAのサイズの変化によって変化し得る。 SECにとっては、制限酵素テンプレートプラスミドDNAの大きさの違いを最大化する方法でダイジェストを設定することが重要です。
- モル比(R)が再構成のために使用するかを決定します。Rは DNA反復のモル数に対する量体のモル比に等しい。意図は、飽和ヌクレオソームアレイを得ることである場合は、初期小規模試験のために、0.9、1および1.1のr値が適切である。
- テスト対象の各モル比で約18μgのDNAのに追加する八量の量を計算することにより、小規模なサンプルを準備します。 DNAやヒストン八量を混ぜる。試料中のNaClの最終濃度≥2 Mであるべきであり、Dの最終濃度NAは周り0.3μgの/μLである必要があります。最終サンプル緩衝液条件はまた、10mMのトリス、pH7.8、および1mM EDTAを含むべきである。
- サンプルは現在、透析のための準備が整いました。 12K-14K MWCO透析チューブにサンプルをロードします。次のようにイオン強度を低下させるバッファの2Lに対してサンプルを透析。
10 mMトリスpHは7.8、1 mMのEDTA:すべてのバッファでコンポーネント。バッファ1 5-6時間(1MのNaCl、1mMのDTTを加える)。バッファ2(0.75 M NaCl、1mMのDTTを追加)で一晩。バッファ3 5-6時間(2.5のNaCl、1mMのDTTを加える)。バッファ4晩(2.5のNaCl、0.1 mMのPMSFを加える)。
- 透析チューブからサンプルを取り出して、各項4-6に進んでください。小規模なサンプルが目的の結果が得られた場合は、大規模でのセクション3-6の手順を繰り返します。
注:リソースを節約するために、小規模なサンプルが下流のスクリーニングアッセイのために十分であるのに必要な最小体積を有するべきである。電子を有するためにここに記載されている沈降速度およびAFM実験のためのnoughサンプル、0.3 mg / mlのDNAを、50μlのサンプルが適切である。大規模の大きさは、調製用のサンプルは、その使用目的に依存している。大規模なサンプルは、小規模の試料と同様にして調製されるべきである。
4。再構成ヌクレオソーム配列の沈降速度解析
理由:溶液中の分子の大きさや形状にベックマンXL-A / I分析用超遠心利回り情報内沈降速度実験。低塩条件下で行わ沈降速度実験は、再構成されたアレイはヌクレオソームで飽和されている程度を決定するために使用される。
- TEN緩衝液(10mMトリス、pH7.8、1mMのEDTA、2.5mMのNaCl)を用いて260nmでの吸光度0.5の周りにサンプルを希釈する。片側のサンプルやreferenで、2部門の目玉で細胞内への試料400μLを読み込む他の25のCE(TENバッファー)。サンプル半月板よりも高い参照メニスカスを維持する( すなわち 420μL標準溶液を加える)。
- ローターに細胞をロードし、適切に細胞とロータ25の底にハッシュマークを用いて細胞を揃える。穏やかに圧縮空気を用いた細胞のレンズをほこり。
- 、XL-A/XL-I遠心機の電源を入れ、ローターを挿入し、添付して、マニュアルに記載されているように光学系を固定します。新しい:プロテオーム研究室ソフトウェアを選択し、ファイルを開きます。
- 3,000 rpmで、単一のスキャンを実行し、25℃の温度を設定しますオプション]で、[前回のスキャン後に停止し、実行ラジアル校正(ローターがAUCの最後に使用されたロータだった場合、ラジアルリブレーションは必要ありません)。
- 各セルに対して、サンプルに名前を付け、波長(260 nm)を選択し、吸光度を選択して、コンピュータ上のファイルを保存する場所を選択します。シングルスキャンの実行を開始します。
- 適切なRAを決定するために単一のスキャンを使用走査長( 図2A)をダイヤルします。スキャンされるセルの長さを小さくすると、実行時間が短縮されます。しかし、サンプルのメニスカスが含まれており、非常に近い、またはセルの下部に端を拡張するようにしてください。
注:単一のスキャンは、測定領域がちょうど試料のメニスカス( 図2A)の前に測定を開始することによって短縮することを可能にする。 AUCは、実行中に発生するスキャンの大半は、半月板だけでなく、安定した台地( 図2B)過去の境界画分を持っている必要があります。 AUCは、細胞上の汚れたレンズが大きなスパイクでスキャンを生成することができ、これらは、データの分析に影響を与えることがある。ウルトラスキャンソフトウェアは、分析超遠心データ26の分析に関する情報のための素晴らしいリソースですマニュアルが含まれています。
- XL-A/XL-Iコントロールパネルを使用して、0の速度と、プレスの開始を入力してください。これは真空とすべてをプルする遠心室プロンプトが表示されますわーチャンバーの温度が平衡する。ファイル名を指定して実行を開始する前に、温度平衡を可能にするために、1時間待ってください。
- 所望の速度とスキャン回数の実行を上に設定するためのソフトウェアを使用しています。より高速で、解像度を向上させるが、サンプルがセルの底に沈殿している前に、1は、少なくとも20のスキャン(好ましく)を収集する必要があります。これは、実行の進捗状況を監視し、漏れた細胞のような潜在的な問題をチェックするために、(オプションメニューで達成)の最後のいくつかのスキャンをオーバーレイすると便利です。正常な動作を保証するために、スキャンの最初のカップルを監視します。
5。編集と沈降速度データの分析
論理的根拠:プレーン沈降速度データは、補正された拡散沈降係数の分布が得られるプログラムによって解析されるべきである。今度はこの情報は、所与の飽和レベルを含有する再構成された試料の割合を示し、 例えば、 12-merのDNAテンプレートを使用する場合、それは10、11および12ヌクレオソーム/ DNAを有する再構成された配列の割合を決定することが可能となる。
- データ26を編集するようなウルトラスキャンなどのAUCデータ解析ソフトウェアを使用してください。スキャンデータの編集は、セルごとに行われ、その後の分析のためのデータセットを作成する必要があります。スキャンの適切な編集は、試料のメニスカスを画定する関心領域にデータを縮小し、ベースラインならびにプラトー領域( 図2)を規定することを含む。不要なスキャンも、この時点で、データセットから除去することができる。それは分析中に不要なスキャンを除去することが可能であるので、それはすべてのスキャンがこの時点で維持することが推奨される。
- 私たちは強く強化されたファン·ホルデ-Weischetメソッドは、データ27を分析するために使用されることをお勧めします。この分析方法は実験の過程にわたって拡散の影響を補正し、沈降係数の積分分布が得られる。特に、強化されたファン·ホルデ-Weischet法は、(ウルトラスキャンで実装されている)、安定した高原を欠いている、または分析28でメニスカスをクリアしていない境界画分を含んでいるのスキャンを含めることができるようになります。
- 組み立てアレイ用の沈降係数の分布を決定する。 601から12(207bp、12マー)ヌクレオソームアレイのための代表的な結果を図3に示す。
- 小規模のサンプルの1は沈降係数の所望の範囲を持つ配列が含まれている場合は、セクション3で始まる増加規模でプロセスを繰り返します。小規模サンプルのいずれも沈降係数の適切な分布を持っていない場合は、セクション3で始まる新しいRレンジで小規模なテストを繰り返します。
6。原子間力顕微鏡(AFM)を用いてヌクレオソーム配列を可視化する
理由:原子間力顕微鏡はnucleosomaの飽和レベルの可視化を可能にするL·アレイ。この技術は、品質管理アッセイとしてAUCおよび制限酵素消化による沈降速度を補完する。
- 上記の方法を用いて、ウェルを飽和ヌクレオソームアレイ( 図5A)を得た。
- 30分間、0.22μmのフィルターナノ純水に、商業の〜30μlの1:1,000希釈(シグマアルドリッチ(3 - アミノプロピル)トリエトキシシランA3648-100ML)を配置することにより、マイカスライドをあしらったAPTESを準備します。
- 30分後にろ過し、水でAPTESを洗い流し、ゆっくりと窒素フローでそれらを乾燥させます。
- スライド上で適切に希釈したヌクレオソーム配列のサンプル(〜1.5 ngの/μl)を配置し、15分間室温でカバーさインキュベートする。
- 、濾過された試料緩衝液でオフサンプルとして前に乾燥し、原子間力顕微鏡(この場合はアサイラムMFPの3次元原子間力顕微鏡)の舞台の上に置いてスライドすすいでください。
- 512本の走査線で2×2μmのスキャンを撮像して起動します。のSAのレベルを確認するためにヌクレオソームの数を数えるturation( 図5B)。
- 理想的にはよく分離ヌクレオソームアレイスライド上の画像領域をすべき。
- 高解像度画像は、500×500 nmのスキャンは、( 図5C)を得ることができる。
- 画像は平らにし、アサイラムが提供するMFPの3次元ソフトウエアを用いて分析することができる。
- 各画像は、4つの象限に分割され、ヌクレオソーム( 図5C、右のパネルの高さのトレースのために取得される配列や高度プロファイルを通して描くことができます十分に分離し、配列や複数のフリーハンドの線を明確に視野を得るために、デジタル的に拡大することができますおよび5D)。
- いくつかのこのような画像は、ヌクレオソームの数百を包含する配列の種類ごとに分析すべきである。高さプロファイルは、記録され分類され、MS Excelでプロットされている。
結果
プロトコルを説明するために、我々は601の位置決めシーケンス(601 207×12)の12タンデム207 bpの反復からなる組換えアフリカツメガエルコアヒストンとDNAからヌクレオソームの配列を再構成した。まず、凍結乾燥されたコアヒストンからネイティブ八量を組み立てた後、S200カラム( 図1A)を使用してFPLCによって八量を精製した。より大きな複合体は、S200カラムから先に溶出する。ヒストンは、一般的にこの順序で溶出:非特異的なヒストン凝集体、ヒストンオクタマー、H3/H4四量体、およびH2A/H2B二量体( 図1A)。 S200カラムからのピークをSDS-PAGEにより分析した。精製されたオクタマー画分のゲルは4ヒストンタンパク質( 図1B)の等モル量を示す必要があります。 アフリカツメガエル H2AとH2Bはわずか約200ダによって異なり、したがって、彼らはSDSゲル上の単一バンドとして現れる分子量を有していることに注意してください。 H3/H4四量体とH2A/H2B二量体は、バージョンの後に溶出しますが、ヒストンオクタマーピークに近いY。維持するためにどの分画を選択する際には、クロマトグラムのオクタマーピークの端に向かって表示される画分を廃棄することが賢明である。これらの画分は、非ヒストン八量体複合体の量が増加していてもよい。この場合、64から67は、ヌクレオソームの配列の再構成( 図1)のためにプールされ、保存された画分。
ウルトラスキャン内で強化されたファン·ホルデ-Weischet分析から得られた沈降係数の拡散補正後の分配を表示する2つの異なる方法が存在する。赤い線は、沈降係数の分布の積分を表す。グラフ上の任意の点については、y軸に等しいか又はx軸上に示される値未満の沈降係数を有するサンプルの割合を示す。異種のサンプルが正の傾きを有する曲線を持つことになりますしながら、このように、縦線は、均質なサンプルを示している。緑の線はデです一体型分布のrivative。ピーク下の面積は、その沈降係数を有するサンプルの割合に比例する。完全に飽和した601 207 X 12クレオソームアレイが29S 29の沈降係数を持っている。 図3に示すサンプルの場合、データはアレイの約70%が飽和であり、30%が過飽和であることを示している。
原子間力顕微鏡は、in vitroでクロマチン組織を研究するために人気の相補的なアプローチに新たな技術となっています。ここでは、再構成後のテンプレート彩度の範囲を確立するために、分析超遠心のと一緒に、AFMを使用している。 図6では、撮像に用いる〜27Sアレイ( 図4A)の大部分は、AFM及びAUC結果との間の優れた一致を示す、10-11ヌクレオソーム( 図4B)を含有した。ここで得られたAFMの結果は、このようにしてこれを検証するそれヌクレオソームアレイを特徴づけるための信頼性のある技術的なアプローチとなります。
| ヒストンタンパク質 | Σ276、非折り畳みタンパク質のCM -1 M -1 | 分子量ダ |
| H2A | 4350 | 13960 |
| H2B | 7,250 | 13774 |
| H3 | 4640 | 15273 |
| H4 | 5800 | 11236 |
| 8量体 | 44080 | 108486 |
表1。吸光係数およびアフリカツメガエルコアヒストンおよび復元させ、ヒストンオクタマーの分子量。
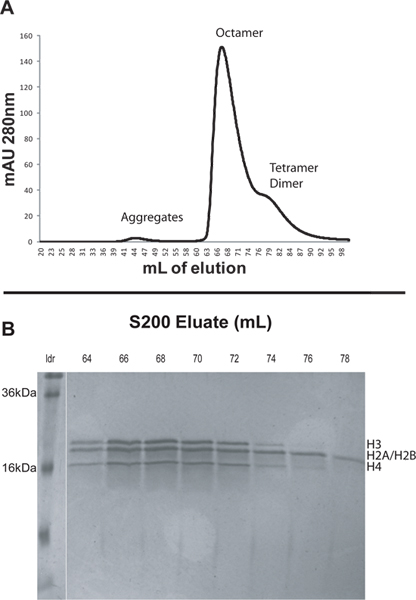
図1。セクション2で説明したように、S200列からAの溶出プロファイル。約44ミリリットルの小さなピークが大きいヒストン凝集によるものです。 67ミリリットルでの顕著なピークは、ヒストン八量です。 76〜90ミリリットルの広い肩はH3/H4四量体と二量体H2A/H2B S200欄から選択された画分の。B. 20%のSDS-PAGEが含まれています。彼らは非八量ヒストン複合体によって汚染され、少なくとも可能性が高いとして量体ピークから早期の画分を収集する必要があります。この場合、64〜67をプールし、ヌクレオソームのアレイの再構成のために保存した画分。 大きな画像を見るにはここをクリックしてください 。

図2。 LAで3,000 rpmで収集された単一のスキャンのA. ProteomeLabソフトウェア画面キャプチャ(ベル)を加えた。サンプルは、この低速で土砂をしないことに注意してください。シングルスキャンは、AUC細胞スキャン(ステップ4.6)のための測定値の範囲を設定するために使用されている。ウルトラスキャンプログラム(追加ラベル付き)26を使用して得られた沈降速度スキャンの一連のB.のスクリーンショット。スキャンこの一連の分析のためにデータ·セット(ステップ5.1参照)を生成するために編集される。
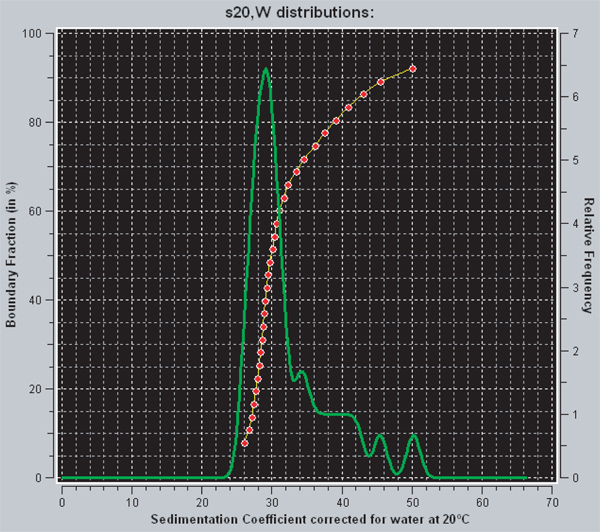
図3。UltraScanIIからのスクリーンキャプチャ。赤と緑の線はウルトラスキャンでの沈降係数の分布を表示するための2種類の方法があります。グリーンは、デリバティブている間赤い線は、沈降係数の積分分布である。解釈に関する追加情報は、結果セクションに記載されています。
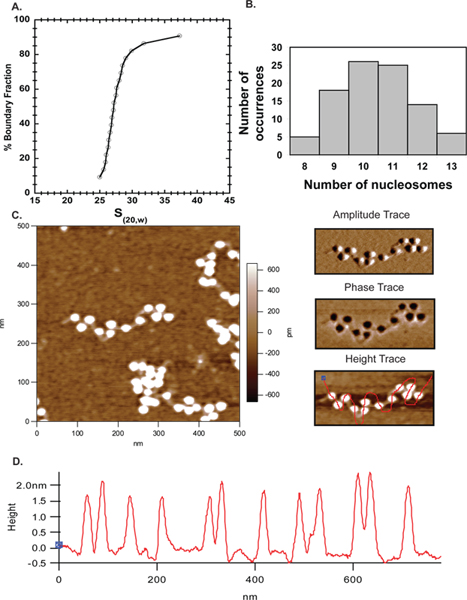
図4。原子間力顕微鏡。マウスのオクタマーと組み立て601 207×12アレイ用A.沈降速度プロファイルの平均沈降係数は27Sであることを示す。B. Aの同じ配列をAFMによって画像化され、いくつかの画像でカウントヌクレオソームの数は、MS Excelでプロットした。プロットは、配列の大半は、AUCのデータを裏付け10月11日ヌクレオソームを有することを示した。はっきり見えるリンカーDNAと601 207×12ヌクレオソームアレイのC. A 500×500 nmのスキャンを。右上のパネルには、振幅トレースで、中央の右側のパネルには、Cに示したアレイのための位相トレースである。右下のパネルには、左側にあるがヌクレオソームの高さプロファイルを決定するために、ヌクレオソームを通って引かフリーハンドラインで示されたのと同じ高さのトレースですS。上の画像にあるヌクレオソームのD.高さプロファイルは、以前に40を報告したように、すべてのヌクレオソームは、1.5〜2.5 nmの高さの中に及ぶことを示している。
ディスカッション
モデルヌクレオソームアレイは、クロマチン構造及び機能のin vitro研究のための非常に有用なツールである。例えば、それらは広く溶液30-34におけるクロマチン繊維縮合のメカニズムを研究するために使用され、tetranucleosome 35のX線構造を得ることが可能となされている。さらに最近では、それらは特定のコアヒストン変異体、突然変異体および翻訳後修飾14-16,36の構造的な影響を解読するのに有用であることが証明されている。ここでは、ヌクレオソーム位置決めDNAからモデルヌクレオソーム配列および組換えコアヒストンの組立のための一般的な方法を説明します。
精製された八量と601 207×12 DNAからのヌクレオソームの配列の再構成は順次、2M 2.5 mmのNaCl濃度を下げて、いくつかの透析工程を含む、簡単です。プロトコルの中で最も難しい部分は、目的のTEMをもたらすr値を使用することですプレート飽和レベル。適切なr値は、最初に小規模で実験的に決定した後、実験に使用されるヌクレオソームの配列を生成するために、より大きな規模で繰り返される。私たちのケースでは、主に、DNAあたり12ヌクレオソームで飽和601 207×12 DNAテンプレートを入手しようとしていた。完全に飽和した601 207×12ヌクレオソーム配列は〜27S 29におけるDNA堆積物当たりわずか11ヌクレオソームと同じDNAテンプレートが、29 Sでの沈降係数。したがって、分析用超遠心で沈降速度は、再構成後ヌクレオソーム飽和レベルを決定するための非常に感度の高い方法を提供する。我々は、拡散補正後の沈降係数の分布が得られる増強ファン·ホルデ-Weischet法を用いて、我々のデータを分析した。それは均質または不均質サンプルは、再構成後でどのように1に伝えますので、この情報は不可欠です。換言すれば、601 207×12 DNAテンプレートの、it図3は 、強化されたバン1.1のrにおいて再構成された601 207×12ヌクレオソームアレイのHolde-Weischet分析の結果を示すDNAあたり12ヌクレオソーム、DNAあたり11ヌクレオソーム等を有する試料の割合を示すもので略配列の30%が飽和を超えている。サンプルの有用性は、通常、飽和配列の割合に結びついたが、一部のアプリケーションでは、非常に均質な、および/または飽和の配列が必要な場合がありますインチのアレイが使用された実験に依存する。多くの方法は、ヌクレオソームアレイの均質性と彩度を向上させるために存在する。
この場合、過飽和アレイは37のMgCl 2を添加した際に選択的沈殿によって除去することができる。より均一なアレイは、ショ糖勾配遠心分離、分取ゲル電気泳動、イオン交換クロマトグラフィー10,13を用いて試料を精製することによって得ることができる。修正NUcleosomal配列の再構成法は、塩透析38,39を通じて再構成する前に、サンプルに短い競合DNAを加えることを求めている。これは1オーバーのDNAテンプレートを飽和させることなく、ヒストンオクタマー過剰のヌクレオソームの配列を組み立てることができます。ヌクレオソームアレイは、おそらく競合DNAとヒストンの余分な除去のための精製工程を必要とする競合DNAを用いて再構成した。
小さなスケールのパイロットアレイアセンブリ後には大規模に再構成されたヌクレオソームアレイが適切に飽和されないことが可能である。試料中の鋳型DNAおよびヒストン八量の浪費を避けるためには、上または飽和アレイの下に固定することができる。アレイは、飽和を超えている場合、余分なDNAを試料に添加することができる。アレイが飽和未満の場合、余分な量体はそれを修正するために添加することができる。しかし、すでに低塩に透析配列に八量を追加すると、解離オクタマー発生する可能性があります。余分なOを追加する場合ctamerまたはDNAは、配列バルクサンプルへの追加八量またはDNAを追加する前に戻る2MのNaClへの配列を透析する。ステップ3.4に従って高から低塩に固定したサンプルについて、手順透析。アレイを補正するために追加するオクタマーの量は飽和アレイ29の下で沈降係数から推定することができる。ヌクレオソームアレイのサンプルを調整した後、飽和レベルの測定が再度行われるべきである。
AFMは、ヌクレオソームアレイを特徴付けるための強力な方法であるが、複雑で労働集約的なプロセスである。これは、小規模な再構成のをスクリーニングするための技術が乏しいが、大規模なサンプルの特徴付けのためのクロマチン組織の研究のための優れた技術ができる。以前に私たちも、低いのMgCl 2濃度に対する「ハイパー応答性」だったmacroH2A欠失構築物によって生成された「マクロ」粒子を視覚化するために、AFMを使用している。 Likewiそれ自体、モンテルら (2009)H2A BBD変異体はヌクレオソーム配列は野生型配列と比較してより「オープン」であることが原因となることが示されている。そのため、AFMは、品質管理のためだけでなく、一般的なクロマチン繊維構造の研究のための信頼性の高い手法である。
開示事項
著者らは、利害の衝突を持っていません。
謝辞
この作品は、NIHの助成金GM45916およびGM66834 JCHし、この作品をAKための国際レット症候群財団のフェローシップでサポートされていたにもNIHがGM088409とハワードヒューズ医学研究所のKLへの貢献を助成金によって支えられて
資料
| Name | Company | Catalog Number | Comments |
| (3-Aminopropyl)triethoxysilane | Sigma-Aldrich | A3648-100ML | |
| 6-8 kDa MWCO Dialysis Tubing | Fisher | 21-152-5 | |
| HiLoad Superdex 200 16/60 Column | GE | 17-1069-01 | |
| Vivaspin 50 kDa MWCO Centrifugal Concentrator | Sartorius | VS2031 | |
| 12-14 kDa MWCO Dialysis Tubing | Fisher | 08-667A | |
| Illustra Sephacryl S-1000 Superfine | GE | 17-0476-01 | |
| XL-A/I Analytical Ultracentrifuge | Beckman-Coulter |
参考文献
- Luger, K., Mader, A., Richmond, R., Crystal Sargent, D. structure of the nucleosome core particle at 2.8 A resolution. Nature. 7, (1997).
- Hansen, J. C. Conformational dynamics of the chromatin fiber in solution: determinants, mechanisms, and functions. Annual Review of Biophysics and Biomolecular Structure. 31, 361-392 (2002).
- McBryant, S., Adams, V., Hansen, J. Chromatin architectural proteins. Chromosome Research. 14 (1), 39-51 (2006).
- Hansen, J. C., Ausio, J., Stanik, V. H., van Holde, K. E. Homogeneous reconstituted oligonucleosomes, evidence for salt-dependent folding in the absence of histone H1. Biochemistry. 28 (23), 9129-9136 (1989).
- Szerlong, H. J., Prenni, J. E., Nyborg, J. K., Hansen, J. C. Activator-dependent p300 acetylation of chromatin in vitro: enhancement of transcription by disruption of repressive nucleosome-nucleosome interactions. The Journal of Biological Chemistry. 285 (42), 31954-31964 (2010).
- Cirillo, L. A., Lin, F. R., Cuesta, I., Friedman, D., Jarnik, M., Zaret, K. S. Opening of compacted chromatin by early developmental transcription factors HNF3 (FoxA) and GATA-4. Molecular Cell. 9 (2), 279-289 (2002).
- Nguyen, C., Gonzales, F. chromatin structure associated with methylation-induced gene silencing in cancer cells: correlation of accessibility, methylation, MeCP2 binding and acetylation. Nucleic Acids Research. 29 (22), 4598-4606 (2001).
- Cuesta, I., Zaret, K. S., Santisteban, P. The forkhead factor FoxE1 binds to the thyroperoxidase promoter during thyroid cell differentiation and modifies compacted chromatin structure. Molecular and Cellular Biology. 27 (20), 7302-7314 (2007).
- Simpson, R. T., Stafford, D. W. Structural features of a phased nucleosome core particle. Proceedings of the National Academy of Sciences of the U S A. 80 (1), 51-55 (1983).
- Lowary, P. T., Widom, J. New DNA sequence rules for high affinity binding to histone octamer and sequence-directed nucleosome positioning. Journal of Molecular Biology. 276 (1), 19-42 (1998).
- Lowary, P., Widlund, H., Cao, H. Sequence motifs and free energies of selected natural and non-natural nucleosome positioning DNA sequences. Journal of Molecular Biology. 288, 213-229 (1999).
- Gordon, F., Luger, K., Hansen, J. C. The Core Histone N-terminal Tail Domains Function Independently and Additively during Salt-dependent Oligomerization of Nucleosomal Arrays *. The Journal of Biological Chemistry. 280 (40), 33701-33706 (2005).
- Luger, K., Rechsteiner, T. J., Richmond, T. J. Expression and purification of recombinant histones and nucleosome reconstitution. Methods in Molecular Biology (Clifton, N.J.). 119 (4), 1-16 (1999).
- McBryant, S. J., Klonoski, J., et al. Determinants of histone H4 N-terminal domain function during nucleosomal array oligomerization: roles of amino acid sequence, domain length, and charge density. The Journal of Biological Chemistry. 284 (25), 16716-16722 (2009).
- Ma Shogren-Knaak, ., Fry, C. J., Peterson, C. L. A native peptide ligation strategy for deciphering nucleosomal histone modifications. The Journal of Biological Chemistry. 278 (18), 15744-158 (2003).
- Lu, X., Simon, M. The effect of H3K79 dimethylation and H4K20 trimethylation on nucleosome and chromatin structure. Nat Struct Mol Biol. 15 (10), 1122-1124 (2008).
- Ausio, J. Analytical Ultracentrifugation for the Analysis of Chromatin Structure. Biophysical Chemistry. 86 (2-3), 141-153 (2000).
- Hansen, J., Kreider, J., Demeler, B., Fletcher, T. Analytical ultracentrifugation and agarose gel electrophoresis as tools for studying chromatin folding in solution. Methods. 12 (1), 62-72 (1997).
- Montel, F., Menoni, H., et al. The dynamics of individual nucleosomes controls the chromatin condensation pathway: direct atomic force microscopy visualization of variant chromatin. Biophysical Journal. 97 (2), 544-5453 (2009).
- Muthurajan, U. M., McBryant, S. J., Lu, X., Hansen, J. C., Luger, K. The linker region of macroH2A promotes self-association of nucleosomal arrays. The Journal of Biological Chemistry. 286 (27), 23852-23864 (2011).
- Shlyakhtenko, L. S., Lushnikov, A. Y., Lyubchenko, Y. L. Dynamics of nucleosomes revealed by time-lapse atomic force microscopy. Biochemistry. 48 (33), 7842-7848 (2009).
- Lohr, D., Bash, R., Wang, H., Yodh, J., Lindsay, S. Using atomic force microscopy to study chromatin structure and nucleosome remodeling. Methods (San Diego, Calif). 41 (3), 333-341 (2007).
- Dyer, P. N., Edayathumangalam, R. S., et al. Reconstitution of nucleosome core particles from recombinant histones and DNA. Methods in Enzymology. 375, 23-44 (2004).
- Sambrook, J., Russell, D. . Molecular cloning: a laboratory manual. , (2001).
- Balbo, A., Zhao, H., Brown, P. H., Schuck, P. Assembly, loading, and alignment of an analytical ultracentrifuge sample cell. J. Vis. Exp. (33), e1530 (2009).
- Demeler, B. UltraScan: a comprehensive data analysis software package for analytical ultracentrifugation experiments. Modern Analytical Ultracentrifugation: Techniques. , 210-230 (2005).
- Holde, K. V., Weischet, W. Boundary analysis of sedimentation velocity experiments with monodisperse and paucidisperse solutes. Biopolymers. 17 (6), 1387-1403 (1978).
- Demeler, B., van Holde, K. E. Sedimentation velocity analysis of highly heterogeneous systems. Analytical Biochemistry. 335, 279-288 (2004).
- Hansen, J., Lohr, D. Assembly and structural properties of subsaturated chromatin arrays. Journal of Biological Chemistry. 8, 5840-5848 (1993).
- Routh, A., Sandin, S., Rhodes, D. Nucleosome repeat length and linker histone stoichiometry determine chromatin fiber structure. Proceedings of the National Academy of Sciences of the U S A. 105 (26), 8872-8877 (2008).
- Zhou, J., Fan, J. Y., Rangasamy, D., Tremethick, D. J. The nucleosome surface regulates chromatin compaction and couples it with transcriptional repression. Nature Structural & Molecular Biology. 14 (11), 1070-1076 (2007).
- Dorigo, B., Schalch, T., Kulangara, A., Duda, S., Schroeder, R. R., Richmond, T. J. Nucleosome arrays reveal the two-start organization of the chromatin fiber. Science (New York, N.Y.). 306 (5701), 1571-1573 (2004).
- Correll, S. J., Schubert, M. H., Grigoryev, S. a Short nucleosome repeats impose rotational modulations on chromatin fibre folding. The EMBO Journal. 31 (10), 2416-2426 (2012).
- Mcbryant, S. J., Krause, C., Woodcock, C. L., Hansen, J. C. The Silent Information Regulator 3 Protein , SIR3p , Binds to Chromatin Fibers and Assembles a Hypercondensed Chromatin Architecture in the Presence of Salt. Molecular and Cellular Biology. 28 (11), 3563-3572 (2008).
- Schalch, T., Duda, S., Sargent, D. F., Richmond, T. J. X-ray structure of a tetranucleosome and its implications for the chromatin fibre. Nature. 436 (7047), 138-1341 (2005).
- Fan, J. Y., Gordon, F., Luger, K., Hansen, J. C., Tremethick, D. J. The essential histone variant H2A.Z regulates the equilibrium between different chromatin conformational states. Nature Structural Biology. 9 (3), 172-176 (2002).
- Carruthers, L. M., Bednar, J., Woodcock, C. L., Hansen, J. C. Linker histones stabilize the intrinsic salt-dependent folding of nucleosomal arrays: mechanistic ramifications for higher-order chromatin folding. Biochemistry. 37 (42), 14776-14787 (1998).
- Huynh, V. A. T., Robinson, P. J. J., Rhodes, D. A Method for the In Vitro Reconstitution of a Defined "30 nm" Chromatin Fibre Containing Stoichiometric Amounts of the Linker Histone. Journal of Molecular Biology. 345 (5), 957-968 (2005).
- Dorigo, B., Schalch, T. Chromatin fiber folding: requirement for the histone H4 N-terminal tail. J. Mol. Biol. 2836 (03), 85-96 (2003).
- Qian, R. L., Liu, Z. X., et al. Visualization of chromatin folding patterns in chicken erythrocytes by atomic force microscopy (AFM. Cell Research. 7 (2), 143-150 (1997).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved