Method Article
Versammlung der nukleosomalen Arrays von rekombinantem Core-Histone und DNA Nucleosome Positioning
In diesem Artikel
Zusammenfassung
Es wird ein Verfahren für die Wiederherstellung von Modell nukleosomalen Arrays aus rekombinanten Histone und hintereinander wiederholt Nukleosomen-Positionierung DNA vorgestellt. Wir beschreiben auch, wie Sedimentationsgeschwindigkeit Experimente in der analytischen Ultrazentrifuge, und Rasterkraftmikroskopie (AFM) werden verwendet, um das Ausmaß der nukleosomalen Array Sättigung nach Rekonstitution zu überwachen.
Zusammenfassung
Kern Histon Octamere, die wiederholt entlang eines DNA-Moleküls angeordnet sind nukleosomalen Arrays bezeichnet. Nukleosomalen Arrays werden in einem von zwei Wegen erreicht: Reinigung von in vivo-Quellen oder in vitro Rekonstitution von rekombinantem Core-Histone und tandemartig wiederholten DNA Nukleosom Positionierung. Das letztere Verfahren hat den Vorteil, dass für die Montage von einer in der Zusammensetzung einheitlich und genau positioniert nukleosomalen Array. Sedimentationsgeschwindigkeit Experimente in der analytischen Ultrazentrifuge Ausbeute Informationen über die Größe und Form von Makromolekülen durch die Analyse der Geschwindigkeit, mit der sie durch Lösung unter Zentrifugalkraft migrieren. Diese Technik, die zusammen mit der Rasterkraftmikroskopie kann zur Qualitätskontrolle verwendet werden, so dass der Großteil der DNA-Templates werden mit Nukleosomen nach Rekonstitution gesättigt. Hier beschreiben wir die erforderlichen Milligramm-Mengen der Länge und der Zusammensetzung zu rekonstituieren d Protokolleefined nukleosomalen Arrays für biochemische und biophysikalische Untersuchungen der Chromatin-Struktur und Funktion.
Einleitung
Eukaryotischen Genomen existieren nicht als nackte DNA, sondern verdichtet und durch gebundene Proteine organisiert. Diese Komplexe von DNA und Protein Chromatin. Die Grundstruktureinheit des Chromatins ist das Nukleosom. Ein Nukleosom umfasst Histonoktamer und 146 Basenpaare der DNA um die Histon-Oktamer gewickelt etwa 1,6 mal 1. Die Histonoktamer wird von jedem der Core-Histone H2A, H2B, H3, H4 und zwei Kopien zusammen. Kern Histon Octamere, die wiederholt entlang eines DNA-Moleküls angeordnet sind nukleosomalen Arrays bezeichnet. Die erweiterte Struktur nukleosomalen Arrays hat als die 10 nm-Faser oder den "Perlen auf einer Schnur"-Struktur bezeichnet worden ist und in vitro unter Niedrigsalzbedingungen 2 vorhanden. Die 10-nm-Faser ist in der Lage Kondensation in Strukturen höherer Ordnung durch intra-Array Verdichtung und / oder Inter-Array Oligomerisierung 2. Diese Strukturen höherer Ordnung in Gegenwart von Salzen induziert werden,oder durch die Bindung von Proteinen an Chromatin Architektur nukleosomalen Array 3,4 beeinflusst werden. Levels Verdichtung des Chromatins sind invers mit Transkriptionsrate in vivo 5,6 korreliert. Neue Forschung unterstreicht die Bedeutung der strukturellen Organisation der Genome in Prozesse wie Differenzierung, die Entwicklung von Krebs, und andere 7,8. Der Einsatz von Arrays nukleosomalen Chromatin Struktur und Funktion zu untersuchen hat sich weit verbreitet. Hier beschreiben wir ein Verfahren für die Montage von nukleosomalen Arrays aus rekombinanten Histone und Nukleosomen Positionierung DNA.
Mit Hilfe rekombinanter DNA mit Tandem-Wiederholungen von Nukleosomen-Positionierung Sequenzen ermöglicht die Wiederherstellung des Arrays, die regelmäßig angeordneten Nukleosomen enthalten. Zwei der populäreren Positionierung Sequenzen sind die 5S-rRNA-Gen-Sequenz und die "601"-Sequenz 9,10. Die 601-Sequenz wurde aus SELEX-Experimenten und mor abgeleitete-Positionen stark Nukleosomen als die 5S-Sequenz 11. Folglich ist die Linker-DNA Länge der Arrays 601 homogener. Tandemwiederholungen Nukleosom Positionierung DNA durch Gelfiltration 4,12 erhalten. Rekombinante Histone werden von E. gereinigt coli unter denaturierenden Bedingungen 13. Die Verwendung von rekombinanten Histone erlaubt es, sorgfältig zu steuern, die Histon-Zusammensetzung der nukleosomalen Arrays. Zum Beispiel, Kern-Histone tragen spezifische Mutationen 14 oder post-translationale Modifikationen 15,16 für Wildtyp-Core-Histone ersetzt werden.
Sedimentationsgeschwindigkeit Experimente überwachen die Geschwindigkeit der Sedimentation von Makromolekülen in Lösung unter einer angelegten Zentrifugalkraft 17. Dies liefert Informationen über die Größe und Form von Makromolekülen in einer Probe. Sedimentationsgeschwindigkeit Experimente sind daher ein geeignetes Instrument für die Untersuchung Lösung Zustandsänderungen in ChromatinfaserStruktur aufgrund Chromatin-Kondensation 18. Wichtig ist es zunächst erforderlich, Sedimentationsgeschwindigkeit Experimente als Qualitätskontrollschritt in nukleosomalen Array Rekonstitution zu verwenden. Wenn DNA und Nukleosomen sind nicht in der richtigen molaren Verhältnis kombiniert, können die Arrays unter-oder übergesättigten mit Core-Histone werden. Somit werden die Informationen aus der Sedimentationsgeschwindigkeit Experimente gewonnen wird verwendet, um sicherzustellen, dass die DNA richtig mit Nukleosomen gesättigt. Es ist wichtig, alternative Methoden anzuwenden, um die Sättigung der DNA mit Nukleosomen zu schätzen, vor allem, wenn die Arbeit mit einem zuvor nicht charakterisierten DNA-Vorlage. Deshalb beschreiben wir auch ein Verfahren zur Analyse von nukleosomalen Arrays mit Rasterkraftmikroskopie (AFM). AFM ist eine leistungsfähige Technik, die ermöglicht die Visualisierung der Wirkungen einer Anzahl von Parametern, wie der Pegel der Sättigung Wirkung der Anwesenheit von Histon-Varianten oder die Auswirkungen von MgCl 2 19,20. AFM hat auch Anwendungened, um Nukleosomen Dynamik Studie mit Zeitraffer-Imaging-21. In vitro zusammen nukleosomalen 12-mer-Arrays sind besonders geeignet, um AFM-Untersuchungen, weil sie in der richtigen Größenbereich für AFM 22 gehören. In der vorliegenden Studie haben wir von AFM nukleosomalen Arrays als Qualitätskontrolle als auch als Mittel zur Bekräftigung die Daten von AUC ("Sehen ist Glauben") eingesetzt. Neben einfachen Visualisierung ermöglicht AFM-Messung der Höhenprofile von Proben als eine zusätzliche Metrik.
Protokoll
1. Montage von rekombinantem Core-Histone in Octamere
Begründung: Der erste Schritt in nukleosomalen Array Rekonstitution ist es, nativen Kern Histon Octamere aus lyophilisierten rekombinanten Histone vorzubereiten. Histon-Proteine werden in äquimolaren Mengen kombiniert und durch Dialyse der Proben aus einem denaturierenden Puffer in Rückfaltungspuffer in Histon Octamere montiert.
- Entschlacken und lyophilisiert rekombinanten Histone (H2A, H2B, H3, H4) wie beschrieben 13.
- Auflösen etwa 5 mg jeder lyophilisierten Kern Histon in 3 ml Entfaltungspuffer (6 M Guanidinium-HCl, 20 mM Tris pH 7,5, 5 mM DTT). Es jedem Aliquot für mindestens eine Stunde auflösen. Sicherzustellen, dass kein Protein an den Seiten des Behälters verbleibt.
- Messen Sie die Absorption jeder Histon bei 276 nm mit Entfaltung Puffer als Referenz.
- Berechnen Sie die Molarität von jedem Histon mit ihren Extinktionskoeffizienten (siehe Tabelle 1 für Xenopus Histon Extinktionskoeffizienten). Vergleichen der Absorption bestimmten Konzentration des Histons an die lyophilisierte Trockenmasse, und Schätzung, wenn der Großteil des Proteins gelöst.
- Bestimmen Sie, welche Histon Sie die wenigsten Mol haben, da dies die Höchstzahl der Mole für alle Histone sein.
- Vereinige die Histone in gleichen molaren Mengen und Entfalten Puffer auf eine Endkonzentration von 1 mg / ml zu verdünnen.
- Legen Sie die Probe in 6-8 kDa MWCO Dialyseschlauch. Abdichten des Schlauchs aufnehmen und in 2 l kaltes Rückfaltungspuffer (2 M NaCl, 10 mM Tris pH 7,5, 1 mM EDTA, 5 mM β-Mercaptoethanol).
- Dialyse der Probe für 18 Stunden bei 4 ° C unter Rühren. Sicherstellen, dass die Dialyseschläuche frei und kräftig zu drehen, oder aber die Histone ausfallen.
- Ändern Sie zweimal in der 18 Stunden-Zeitraum (dh ändern Sie die Rückfaltungspuffer über jede 6 Stunden, bis getan) die Dialysepuffer. Auch wenn Niederschlag bildet nicht verwerfendie Probe kann einige Octamer gewonnen, wobei die Ausbeute wird verringert werden.
Hinweis: Siehe Schritt 2.1-2.2 Spaltenausgleich für den nächsten Tag vorzubereiten.
2. Reinigung von Histon Octamere durch Size Exclusion Chromatography
Begründung: Nach dem Abschluss mit § 1, werden Proben Histon Octamere sowie andere Histon-Komplexe wie Aggregate, H3/H4 Tetramere und Dimere H2A/H2B enthalten. Histon Octamere wird von diesen anderen Komplexen mit Größenausschluss-Chromatographie (SEC) gereinigt werden.
- Schließen Sie einen HiLoad 16/60 Superdex 200 16/60 (S200) Spalte zu einer FPLC-System. Stellen Sie sicher, dass keine Luftblasen in das System nicht.
- Reinigen Sie die Spalte S200 mit 0,2 um gefiltertes Wasser. Verwenden Sie eine Durchflussrate von 0,3 ml / min und setzen einen Gegendruck Grenze von 0,5 MPa. Stellen Sie sicher, Sie haben genug Wasser und lassen Sie die Spalte, über Nacht zu reinigen.
- Äquilibrieren Sie die S200-Säule und Probenschleife des FSPS über Rückfaltungspuffer. Verwenden 1 L von 0,2 um filtriert Rückfaltungspuffer. Rückfaltungspuffer fließen durch die Säule mit einer Geschwindigkeit von 1 ml / min und einem maximalen Druck von 0,5 MPa für ca. 2 Stunden.
- Äquilibrieren einer Vivaspin Zentrifugal-Konzentrator (MWCO 50 kDa) mit 1 ml Rückfaltungspuffer. Entfernen der Probe aus Dialyseschlauch und Zentrifugieren der Probe bei 4 ° C bis keine Niederschläge zu entfernen. Pipettieren des Probenüberstand in den Konzentrator. Konzentrieren sich die Probe bis auf ein Volumen von etwa 500 &mgr;.
- Entfernen Sie die konzentrierte Octamers Probe in einen neuen Container und spülen Sie den Konzentrator mit 1 ml Rückfaltungspuffer. Konzentrieren Sie die Spülung auf 500 ul und es an die Oktamer Probe hinzufügen. Dies hilft, das noch in der Konzentrator restlichen Octamers retten.
- Drehen die Probe in ein 1,5 ml Mikrozentrifugenröhrchen für 5 Minuten bei 10.000 UpM bei 4 ° C Sammeln Sie den Überstand und übertragen in ein neues Röhrchen. Dies hilft, jede Niederschlag vor dem Laden der Probe auf der FPL entfernenC.
- Laden der Probe auf die Säule S200. Laden Sie ein Maximum von 1,5 ml Volumen oder 15 mg Oktamer in einem einzigen Reinigungs.
- Eluiere das Beispiel mit frisch gemachten Rückfaltungspuffer. Verwenden einer Fließgeschwindigkeit von 1 ml / min und eines Rückdrucks Grenze von 0,5 MPa. Verwenden Sie zwei Säulenvolumen (400 ml) Rückfaltungspuffer und 2 ml Fraktionen. Überwachung der Absorption bei 280 nm während der Elution. Die verschiedenen Arten werden in der Reihenfolge der abnehmenden Größe zu eluieren. Histon-Aggregaten in der Regel eluieren bei etwa 45 ml, Oktamer bei etwa 65 ml, und Dimer zu etwa 84 ml (Abbildung 1).
- Analyse der Elutionsfraktionen von Peaks von Interesse indem Proben auf einem 18-20% SDS-PAGE. Fraktionen, die gereinigtes Octamere sollten gleichen molaren Mengen der vier Kern-Histon-Proteine (Abbildung 1).
- Pool der Fraktionen, die gereinigt Octamere enthalten und zu konzentrieren, um mit einer Vivaspin Zentrifugal-Filter ≤ 15 mg / ml. Wenn links verdünnter kann Octamere distanzieren into H2A/H2B Dimere und Tetramere H3/H4.
- Bestimmen die Octamer Konzentration durch Messen der Absorption bei 280 nm und die Berechnung unter Verwendung des Extinktionskoeffizienten von Tabelle 1.
- Histon Octamere kann kurzfristig in Rückfaltungspuffer bei 4 ° C gehalten werden, Wenn gespeichert wird langfristig die Octamere bei -20 ° C in einer 50% Glycerinlösung stabiler. Octamere in Glycerin gelagert werden, sollten vor der Absorptionsmessungen an die frische Rückfaltungspuffer dialysiert werden und die Verwendung in nukleosomalen Array Rekonstruktionen.
3. Rekonstitution der nukleosomalen Arrays aus DNA und gereinigtes Octamere
Begründung: Rekonstitution der nukleosomalen Arrays erfordert, dass Histon-Octamere-und Template-DNA an spezifischen molaren Verhältnissen kombiniert werden. Mischungen von DNA und Histon Octamere in 2 M NaCl sind Schritt in die Puffer abnehmender Ionenstärke dialysiert. Eine allmähliche Änderung niedrigeren Salzbildung gewährleistet eine ordnungsgemäße Nukleosomen. Beziehen nucleosomal Arrays mit dem gewünschten Grad der Sättigung Histonoktamer der Template-DNA erfordert kleinen Test Rekonstruktionen, gefolgt von einem großen Maßstab präparative Rekonstitution. Die durch die Klein Rekonstruktionen definierten geeigneten Bedingungen verwendet, um die Wiederherstellungs führen.
- Reinige hintereinander wiederholt, wie beschrieben, 4,12 Nukleosomen-Positionierung DNA. Kurz gesagt, die Plasmid-DNA zu isolieren, unter Verwendung eines Qiagen GigaPrep-Kit oder ein ähnliches Produkt. Set-up eines Restriktionsenzyms, um die Template-DNA freizusetzen und verdauen die Plasmid-DNA auf kleinere Größen (≤ 700 bp) zu verdauen. Die Matrizen-DNA kann dann von den kleinen Plasmid DNA Reste mit Größenausschluss-Chromatographie (SEC) gereinigt werden. Ein Schwerkraftsäule mit einer Höhe von etwa 115 cm mit Sephacryl S-1000 Perlen gepackt ist ausreichend für die Reinigung von 601 x 207bp 12mer DNA.
Hinweise: Für die Reinigung von Plasmid-DNA bei verringerten Kosten, sondern erhöht effort, kann man alkalische Lyse mit Phenol / Chloroform DNA-Reinigung, um Plasmid-DNA aus E. isolieren verwenden coli 23,24. Strategien zur Trennung von Template-DNA aus Plasmid-DNA kann mit Veränderungen in der DNA-Vorlage Größe variieren. Für SEC ist es wichtig, die Einrichtung der Restriktionsenzym in einer Weise, die den Unterschied in der Größe zwischen der Vorlage und Plasmid-DNA maximiert verdauen.
- Bestimmen, welche Molverhältnisse (r), die für Wiederherstellungen verwendet. R ist gleich dem Verhältnis der Mole von Oktamer auf Mole DNA wiederholen. Für einen ersten kleinen Versuche, r-Werte von 0,9, 1, 1,1 und angemessen, wenn die Absicht besteht, gesättigten nukleosomalen Arrays zu erhalten.
- Vorbereitung der kleinen Proben durch Berechnung des Betrags der Oktamer auf etwa 18 ug DNA pro Molverhältnis zu prüfenden hinzuzufügen. Mischen Sie die DNA und Histon Octamere. Die Endkonzentration von NaCl in der Probe sollte ≥ 2 m, und die Endkonzentration von DNA sollten rund 0,3 ug / ul sein. Die endgültige Probe Pufferbedingungen sollten auch 10 mM Tris pH 7,8, 1 mM EDTA.
- Die Proben sind nun bereit für die Dialyse. Laden Sie die Proben in 12k-14k MWCO Dialyseschlauch. Dialysiere die Proben gegen 2 l Puffer abnehmender Ionenstärke wie folgt.
Bauteile in den Puffer: 10 mM Tris pH 7,8, 1 mM EDTA. Puffer 1 (Add 1 M NaCl, 1 mM DTT) für 5-6 Std. Puffer 2 (Add 0,75 M NaCl, 1 mM DTT) über Nacht. Puffer 3 (Add 2,5 mM NaCl, 1 mM DTT) für 5-6 Std. Buffer 4 (Add 2,5 mM NaCl, 0,1 mM PMSF) über Nacht.
- Entfernen Sie die Proben aus dem Dialyseschlauch und fahren Sie Abschnitte 6.4. Wenn die kleinen Proben ergeben die gewünschten Ergebnisse, wiederholen Sie die Schritte in den Abschnitten 6.3 auf der großen Skala.
Hinweis: Um Ressourcen zu schonen, sollten kleinere Proben das Mindestvolumen notwendig, für nachgelagerte Screening-Tests genügen haben. Um e habennough Probe für die hier aufgeführten Sedimentationsgeschwindigkeit und AFM-Experimente wurden 50 ul-Proben mit 0,3 mg / ml DNA geeignet sind. Die Größe der in großem Maßstab, sind präparativen Proben abhängig von ihrem Verwendungszweck. Ein Groß Probe sollte in der gleichen Weise wie die kleinen Proben hergestellt werden.
4. Sedimentationsgeschwindigkeit Analyse der rekonstituierte nukleosomalen Arrays
Begründung: Sedimentationsgeschwindigkeit Experimente im Beckman XL-A / I analytischen Ultrazentrifuge Ausbeute Informationen über die Größe und Form der Moleküle in Lösung. Sedimentationsgeschwindigkeit Experimente unter Niedrigsalzbedingungen durchgeführt werden, verwendet, um das Ausmaß, in dem die rekonstituierten Arrays mit Nukleosomen gesättigten bestimmen.
- Die Probe zu verdünnen, um eine Absorption in der Umgebung von 0,5 bei 260 nm mit TEN-Puffer (10 mM Tris pH 7,8, 1 mM EDTA, 2,5 mM NaCl). Laden 400 ul Probe in eine Zelle mit einem Zweikernsektor, wobei die Probe auf der einen Seite und dem Refece (TEN-Puffer) 25 auf der anderen. Halten Sie die Referenz Meniskus höher als die Probe Meniskus (Hinzufügen von 420 ul Referenzlösung).
- Laden der Zellen in den Rotor, und richten die Zellen richtig mit den Hash-Zeichen auf dem Boden der Zellen und des Rotors 25. Vorsichtig reinigen Sie die Linsen der Zellen unter Verwendung von Druckluft.
- Schalten Sie die Zentrifuge XL-A/XL-I, legen Sie den Rotor und befestigen und sichern Sie die Optik wie in der Anleitung beschrieben. Öffnen Sie die Proteome Lab Software und wählen Sie Datei: Neu.
- Einrichten einer einzigen Scan laufen bei 3.000 Umdrehungen pro Minute und einer Temperatur von 25 ° C Unter Optionen wählen Sie Stop nach dem letzten Scan und Radiallauf Kalibrierung (radiale Kalibrierung ist nicht notwendig, wenn der Rotor war der letzte in der AUC verwendet Rotor).
- Für jede Zelle, die Bezeichnung der Proben, wählen Sie eine Wellenlänge (260 nm), wählen Absorption, und wählen Sie eine Datei speichern Speicherort auf Ihrem Computer. Beginnen Sie die einzelnen Scan laufen.
- Verwenden Sie die einzelnen Scan, um die entsprechende ra bestimmenWahlscanlänge (Abbildung 2A). Verringern der Länge der Zelle, die untersucht werden sollen verringert die Laufzeit. Jedoch sicher sein, um die Probe Meniskus sind und sich das Ende sehr nahe oder an der Unterseite der Zelle.
Anmerkung: Einzelscans kann der Messbereich durch Messungen begonnen, kurz bevor die Probenmeniskus (2A) verkürzt werden. Die meisten der während der AUC-Lauf erzeugt Scans sollten Grenze Fraktionen Vergangenheit des Meniskus sowie stabile Plateaus (2B) haben. Schmutzige Gläser auf den AUC-Zellen können Scans mit großen Spitzen erzeugen, können diese Analyse der Daten beeinflussen. Die Ultrascan-Software umfasst ein Handbuch, das eine große Ressource für Informationen über die Analyse von Daten analytische Ultrazentrifugation 26 ist.
- Mit der XL-A/XL-I Bedienfeld, geben Sie eine Geschwindigkeit von 0 und Start drücken. Dies wird die Zentrifugenkammer aufgefordert, um ein Vakuum zu ziehen und alleow die Temperatur der Kammer zu äquilibrieren. Warten Sie 1 Stunde, um für Temperaturausgleich vor Beginn einer Lauf zu ermöglichen.
- Verwenden Sie die Software, um den Lauf für die gewünschte Geschwindigkeit und Anzahl der Scans eingestellt. Höhere Geschwindigkeiten erhöhen Auflösung, aber man sollte mindestens 20 Scans (vorzugsweise mehr) zu sammeln, bevor die Probe auf dem Boden der Zelle sedimentiert. Es ist zweckmäßig, die letzten Scans (im Optionsmenü erreicht), um den Fortschritt der Lauf überwachen und auf mögliche Probleme wie undichte Zellen überlagern. Überwachen Sie die ersten paar Scans um den ordnungsgemäßen Betrieb zu gewährleisten.
5. Bearbeiten und Analysieren von Sedimentation Geschwindigkeitsdaten
Begründung: Rohmaterial Sedimentationsgeschwindigkeit Daten durch ein Programm, das eine Diffusionskorrigierten Sedimentationskoeffizientenverteilung ergibt analysiert werden. Diese Informationen wiederum gibt den Bruchteil eines rekonstituierten Probe, die einen bestimmten Sättigungspegel enthält, z. B. bei Verwendung eines 12-mer-DNA-Matrize wird es möglich sein, den Bruchteil des rekonstituierten Arrays 10, 11 und 12 Nukleosomen / DNA hat bestimmen.
- Verwenden Sie AUC Datenanalyse-Software wie Ultrascan, um die Daten 26 bearbeiten. Bearbeitung von Scan-Daten müssen für jede Zelle durchgeführt werden und erzeugt eine Datenmenge für eine nachfolgende Analyse. Ordnungsgemäßen Bearbeitung von Abtastungen beinhaltet die Definition der Probe Meniskus, wodurch die Daten in eine Region von Interesse, und Definieren Basis sowie Plateaubereiche (Fig. 2). Unerwünschte Scans können auch aus den Daten an dieser Stelle gesetzt entfernt werden. Jedoch, da es möglich ist, unerwünschte Scans während der Analyse zu entfernen, empfiehlt es sich, daß alle Abtastungen an dieser Stelle gehalten werden.
- Wir empfehlen dringend, dass die verbesserte van Holde-Weischet-Methode verwendet, um die Daten zu analysieren 27 werden. Dieses Analyseverfahren korrigiert für die Effekte der Diffusion im Laufe des Versuchs und liefert die integrale Verteilung der Sedimentationskoeffizienten. InsbesondereDie erhöhte van Holde-Weischet Verfahren (wie in Ultra implementiert) für die Aufnahme des Scans, die stabile Plateaus fehlt, oder die Randfraktionen, die den Meniskus in der Analyse 28 nicht gelöscht sind, enthalten kann.
- Bestimmung der Verteilung der Sedimentation Koeffizienten für die zusammengebauten Anordnungen. Repräsentative Ergebnisse für 601-12 (207bp, 12-mer) nukleosomalen Anordnungen in Fig. 3 gezeigt.
- Wenn eine der kleinen Proben enthält Arrays mit dem gewünschten Bereich von Sedimentationskoeffizienten, wiederholen Sie den Vorgang mit einer erhöhten Skala beginnend mit Abschnitt 3. Wenn keine der kleinen Proben hat eine angemessene Verteilung der Sedimentationskoeffizienten dann mit einem neuen R-Bereich ab Punkt 3 wiederholen Sie die kleinen Tests.
6. Visualisierung nukleosomalen Arrays Mit Atomic Force Microscopy (AFM)
Begründung: AFM ermöglicht die Visualisierung der Sättigungsgrad von nucleosomal-Arrays. Diese Technik ergänzt Sedimentationsgeschwindigkeit von AUC und Restriktionsverdau als Qualitätskontrolltest.
- Mit oben beschriebenen Methoden erhalten eine gut gesättigt Nukleosomen-Array (5A).
- Bereiten APTES behandelten Glimmer Folien, indem ~ 30 ul 1:1000 Verdünnung der kommerziellen (Sigma-Aldrich (3-Aminopropyl) triethoxysilan A3648-100 ml) in 0,22 um filtriert Nanopure-Wasser für 30 min.
- Nach 30 min spülen Sie die APTES mit gefiltertem Wasser und trocknen Sie sie sanft in einem Stickstoffstrom.
- Legen Sie entsprechend verdünnten Probe Nukleosomen Array (~ 1,5 ng / ul) auf der Folie, und bei Raumtemperatur 15 Minuten abgedeckt.
- Spülen Sie probieren mit gefilterten Probenpuffer, trocken wie zuvor, und statt Schlitten auf der Bühne des AFM (in diesem Fall ein Asylum Research MFP-3D Atomic Force Microscope).
- Starten Sie durch Abbildung 2 x 2 um Scans mit 512 Bildzeilen. Zähle die Anzahl der Nukleosomen auf Ebene der sa ermittelnturation (5B).
- Idealerweise sollten Sie Bildbereiche auf dem Objektträger mit gut getrennt Nukleosomen-Arrays.
- Für Bilder mit höherer Auflösung kann ein 500 x 500 nm-Scan erhalten werden (Abbildung 5C).
- Bilder können abgeflacht und mit der MFP-3D-Software zur Verfügung gestellt von Asylum analysiert.
- Jedes Bild kann in vier Quadranten unterteilt und in digital gezoomt, um eine bessere Sicht auf gut getrennten Arrays und mehrere Freihandlinien können durch die Arrays und Höhenprofile erstellt werden für die Nukleosomen (5C, rechte Tafel-Höhe Spur erhalten erhalten und 5D).
- Mehrere solcher Bilder für jedes Array-Typ, die mehrere Hunderte von Nukleosomen analysiert werden. Höhenprofile werden erfasst, kategorisiert und in MS Excel aufgetragen.
Ergebnisse
Um das Protokoll zu veranschaulichen wir rekonstituiert nukleosomalen Arrays aus rekombinanten Xenopus Histone und DNA, die aus 12 207 bp Tandem-Wiederholungen der Folge 601 Positionierung (601 207 x 12). Wir haben zuerst zusammengebaut nativen Octamere aus lyophilisierten Histone und reinigte die Octamere durch FPLC mit einem S200-Säule (Abbildung 1A) dann. Größere Komplexe eluieren früher aus der Säule S200. Histone eluieren in der Regel in dieser Reihenfolge: nicht-spezifische Histon-Aggregate, Histonoktamer, H3/H4 Tetramer und H2A/H2B Dimer (Abbildung 1A). Die Peaks von der Säule S200 wurden durch SDS-PAGE analysiert. Die Gele des gereinigten Fraktionen Octamer sollten angeben äquimolare Mengen der vier Histon-Proteine (Abbildung 1B). Beachten Sie, dass Xenopus H2A und H2B haben Molekulargewichte, die von nur etwa 200 Da unterscheiden und daher sind sie als eine einzelne Bande auf SDS-Gelen erscheint. H3/H4 Tetramere und Dimere H2A/H2B wird nach Elution, aber very in der Nähe des Histonoktamer Höhepunkt. Bei der Auswahl, welche Fraktionen zu halten, ist es ratsam, Fraktionen gegen Ende des Octamers Peak im Chromatogramm erscheinen zu verwerfen. Diese Fraktionen haben eine erhöhte Menge an nicht-Histon-Komplexen Octamer. In diesem Fall wird die Fraktionen 64-67 wurden vereinigt und zur Rekonstitution nukleosomalen Anordnung (Fig. 1) gespeichert.
In Ultra gibt es zwei unterschiedliche Arten von Anzeigen der Diffusionsverteilung korrigiert Sedimentations-Koeffizienten, die von der verbesserten van Holde Weischet-Analyse erhalten wird. Die rote Linie stellt die integrale Verteilung der Sedimentation Koeffizienten. Für jeden gegebenen Punkt auf der Kurve, zeigt die y-Achse den Anteil der Probe, die eine Sedimentation Koeffizient gleich oder kleiner als der auf der x-Achse angegebenen Wert hat. Somit ist eine vertikale Linie, welche aus einer homogenen Probe, während eine heterogene Probe wird eine Kurve mit positiver Steigung haben. Die grüne Linie ist der de-Derivat des integralen Verteilung. Die Fläche unter einem Peak ist proportional zu der Fraktion der Probe, die diese Sedimentationskoeffizienten hat. Vollständig gesättigte 601 207 x 12 nukleosomalen Arrays haben eine Sedimentationskoeffizient 29S 29. Bei der in Fig. 3 gezeigten Probe, die Daten zeigen, dass etwa 70% der Anordnungen sind gesättigte und 30% sind über gesättigt.
Rasterkraftmikroskopie hat sich von einer Zukunftstechnik zu einem beliebten komplementären Ansatz gegangen, um Chromatin-Organisation in vitro zu studieren. Hier haben wir neben der AFM analytische Ultrazentrifugation verwendet werden, um das Ausmaß der Vorlage Sättigung nach Rekonstitution zu etablieren. In Fig. 6 ist die Mehrheit der ~ 27S-Arrays (4A) für die Bildgebung verwendet wird, enthalten 10-11 Nukleosomen (4B) zeigt eine ausgezeichnete Übereinstimmung zwischen der AFM-und AUC-Ergebnisse. Die AFM-Ergebnisse bestätigen somit hier erhalten Sie diesenähern und machen es zu einem zuverlässigen Technik zur Charakterisierung nukleosomalen Arrays.
| Histon-Protein | Σ276, entfaltete Proteine cm -1 M -1 | Da Molekulargewicht |
| H2A | 4350 | 13.960 |
| H2B | 7250 | 13774 |
| H3 | 4640 | 15273 |
| H4 | 5800 | 11236 |
| Oktamer | 44080 | 108486 |
Tabelle 1. Extinktionskoeffizienten und Molekulargewichte von Xenopus laevis Histone und renaturiert Histonoktamer.
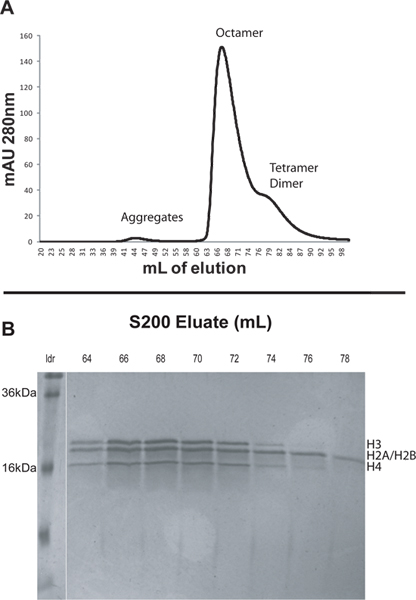
Abbildung 1. A. Elution Profil aus der S200-Säule, wie in Kapitel 2 beschrieben. Das kleine Peak bei etwa 44 ml ist aufgrund der großen Histon-Aggregate. Der prominente Peak bei 67 ml ist die Histon Octamere. Die breite Schulter 76-90 ml enthält die H3/H4 Tetramere und Dimere H2A/H2B. B. 20% SDS-PAGE ausgewählter Fraktionen aus der Säule S200. Frühen Fraktionen aus dem Octamer Peak gesammelt werden sollten, da sie am wenigsten von den nicht-Histon-Komplexen oktameren kontaminiert werden. In diesem Fall die Fraktionen 64-67 wurden vereinigt und zur Rekonstitution nukleosomalen Array gespeichert. Klicken Sie hier, um eine größere Abbildung anzuzeigen .

2. A. ProteomeLab Screen-Capture-Software von einem einzigen Scanvorgang bei 3000 Upm gesammelt (mit laBel hinzugefügt). Beachten Sie, dass die Probe nicht Sediment auf diesem niedrigen Geschwindigkeit. Die einzigen Scan wird verwendet, um den Bereich der Messungen für die AUC-Scan-Zelle (Schritt 4.6). B. Bildschirmerfassung aus einer Reihe von Sedimentationsgeschwindigkeit Scans mit Hilfe der Ultrascan-Programm (mit Etiketten hinzugefügt) 26 erhalten wird. Diese Serie von Scans bearbeitet wird, um einen Datensatz zur Analyse (siehe Schritt 5.1) zu erzeugen.
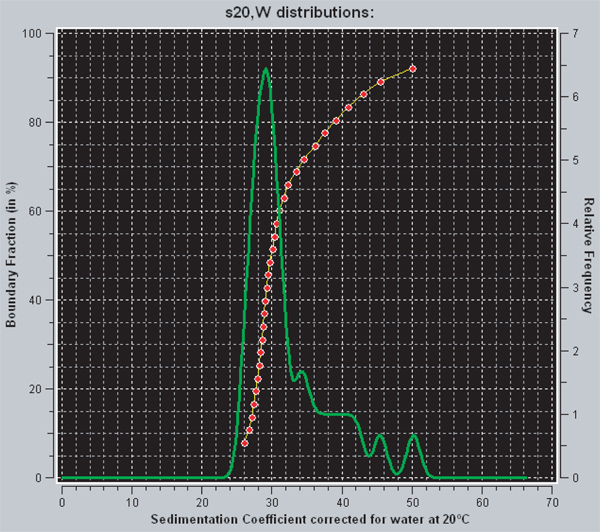
Abbildung 3. Ein Screen-Capture aus UltraScanII. Die roten und grünen Linien sind zwei verschiedene Methoden für die Anzeige der Verteilung der Sedimentationskoeffizienten in Ultrascan. Die rote Linie ist die integrale Verteilung der Sedimentationskoeffizienten, während die grüne ist die Ableitung. Zusätzliche Informationen über die Interpretation der Ergebnisse in Abschnitt.
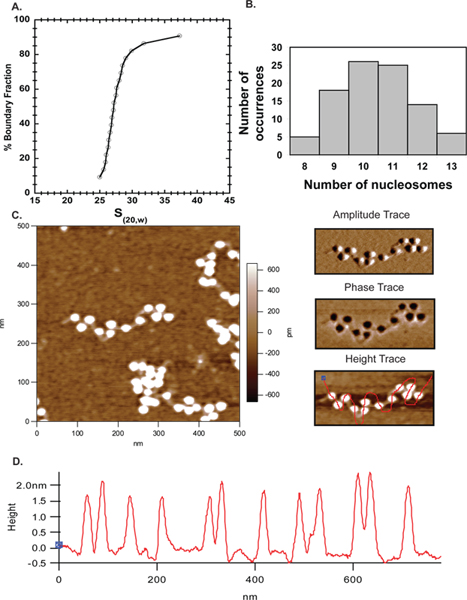
4. AFM. A. Sedimentation Geschwindigkeitsprofil 601 207 x 12 Arrays mit Maus Oktamer zusammengesetzt, die die Durchschnitts Sedimentationskoeffizient ist 27S. B. Die gleichen Anordnungen in A wurden durch AFM abgebildet und die Anzahl der Nukleosomen in mehreren Bildern gezählt wurden in MS Excel aufgetragen. Die Handlung angedeutet, dass eine Mehrheit von Arrays hatte 10-11 Nukleosomen erhärten die AUC-Daten. C. Ein 500 x 500 nm Scan von 601 207 x 12 Nukleosomen-Arrays mit dem Linker-DNA deutlich sichtbar. Der obere rechte Fenster ist die Amplitude und die mittlere Spur rechten Bereich ist die Phase, in der Spur für C gezeigt Array. Die untere rechte Fenster ist der auf der linken Seite, aber mit einer freien Hand Linie durch die Nukleosomen gezogen, um die Höhenprofil der Nukleosomen bestimmen gezeigt gleicher Höhe Spurs. D. Höhenprofile der Nukleosomen im Bild oben zeigen, dass alle Nukleosomen liegen innerhalb von 1,5 bis 2,5 nm Höhe wie zuvor berichtet 40.
Diskussion
Modell nukleosomalen Arrays sind ein sehr nützliches Werkzeug für die in vitro Untersuchung von Chromatin-Struktur und Funktion. Beispielsweise haben sie häufig verwendet, um den Mechanismus der Chromatinfaser Kondensation in Lösung 30-34 untersuchen, und machte es möglich, eine Röntgenstruktur eines tetranucleosome 35 erhalten. Vor kurzem haben sie nützlich bei der Entschlüsselung die strukturellen Auswirkungen der spezifischen Kern Histon-Varianten, Mutanten und posttranslationale Modifikationen 14-16,36 bewährt. Hier beschreiben wir eine allgemeine Methode für die Montage des Modells nukleosomalen-Arrays von Nukleosomen-Positionierung und rekombinante DNA-Core-Histone.
Die Rekonstitution nukleosomalen Arrays aus gereinigtem Octamere und 601 207 x 12 DNA ist einfach, die mehrere Dialyseschritte, die nacheinander die NaCl-Konzentration zu senken von 2 m bis 2,5 mm. Der schwierigste Teil des Protokolls ist es, ein r-Wert, der die gewünschte Tem ergibt verwendenPlatte Sättigungsgrad. Die entsprechenden r-Wert wird zunächst empirisch auf einem kleinen Maßstab bestimmt und dann in einem größeren Maßstab wiederholt, um die für Experimente verwendet nukleosomalen Arrays generieren. In unserem Fall haben wir versucht, 601 207 x 12 DNA-Vorlagen meist mit 12 Nukleosomen pro DNA gesättigt zu erhalten. Die Sedimentationskoeffizient eines vollständig gesättigten 601 207 x 12 nukleosomalen Array ist 29 S, während die gleiche DNA-Vorlage mit nur 11 Nukleosomen pro DNA-Sedimente bei ~ 29 27S. So Sedimentationsgeschwindigkeit in der analytischen Ultrazentrifuge bietet eine sehr empfindliche Methode für die Bestimmung der Nukleosomen-Sättigungsgrad nach Rekonstitution. Analysierten wir unsere Daten über den erweiterten van Holde Weischet-Methode, die eine diffusionskorrigierten Sedimentationskoeffizientenverteilung ergibt. Diese Information ist wichtig, weil es zeigt, wie ein homogener oder heterogener die Probe nach Rekonstitution. Mit anderen Worten, für eine 601 x 207 12-DNA-Matrize, eszeigt den Anteil der Probe, die 12 Nukleosomen pro DNA, 11 Nukleosomen pro DNA usw. hat Abbildung 3 zeigt die Ergebnisse der verbesserten van Holde Weischet-Analyse von 601 207 x 12 nukleosomalen Arrays in einem r von 1,1 rekonstituiert, wobei etwa 30% der Anordnungen sind über gesättigt. Die Nützlichkeit der Probe wird üblicherweise auf den Prozentsatz der gesättigten Arrays gebunden, sondern ist abhängig von den Experimenten die Anordnungen verwendet in. Einige Anwendungen können sehr homogene und / oder gesättigten Arrays erfordert. Eine Anzahl von Verfahren zur Verbesserung der Homogenität und der Sättigung nukleosomalen Arrays bestehen.
In diesem Fall könnten die übersättigte Arrays durch selektive Fällung durch die Zugabe von MgCl 2 37 entfernt werden. Homogenere Arrays können durch Reinigen der Probe unter Verwendung von Sucrose-Gradienten-Zentrifugation, präparative Gelelektrophorese und Ionenaustausch-Chromatographie 10,13 erzielt werden. Eine modifizierte nucleosomal Array Rekonstitution Methode ruft für das Hinzufügen von kurzen DNA-Konkurrent zu den Proben vor der Rekonstitution durch Salz Dialyse 38,39. Dies erlaubt es, nukleosomalen Arrays mit einem Überschuß an Histonoktamer ohne über Sättigen der DNA-Matrize zu montieren. Nukleosomalen Arrays rekonstituiert mit Kompetitor-DNA wird wahrscheinlich einen Reinigungsschritt zur Entfernung der Kompetitor-DNA und Histone zusätzlichen erfordern.
Auch nach kleinen Pilot Array Baugruppen ist es möglich, dass nukleosomalen Arrays in großem Maßstab nicht richtig wiederhergestellt gesättigt werden. Um zu vermeiden, verschwenden die Template-DNA und Histon-Oktamer in der Probe, ist es möglich, über oder unter gesättigten Arrays beheben. Wenn die Arrays über gesättigter kann zusätzliche DNA der Probe zugegeben werden. Sind die Arrays unter gesättigt ist, können zusätzliche Octamers hinzugefügt, um es zu korrigieren. Das Hinzufügen Octamers Arrays bereits in geringen Salz dialysiert können Octamers Dissoziation führen. Wenn das Hinzufügen zusätzlicher octamer oder DNA-Arrays, dialysiert die Arrays wieder in 2 M NaCl, bevor zusätzliche Octamers oder DNA an die Gesamtprobe. Wiederholen Sie Schritt Dialyse für feste Proben von hohen zu niedrigen Salz gemäß Schritt 3.4. Die Menge des Octamer, um die Arrays hinzufügen korrigieren kann von Sedimentationskoeffizienten für unter gesättigten Arrays 29 geschätzt werden. Nach dem Einstellen der Probe nukleosomalen Arrays, sollten Messungen der Sättigungsgrad wieder hergestellt werden.
Während AFM ist eine leistungsfähige Methode zur Charakterisierung nukleosomalen Arrays, ist es kompliziert und arbeitsintensiver Prozess. Dies macht es eine schlechte Technik zum Screening von kleinen Rekonstruktionen, aber eine hervorragende Technik zur Charakterisierung von großen Proben und für die Untersuchung von Chromatin-Organisation. Bisher haben wir AFM verwendet, um "Makro"-Partikel durch eine Streichung macroH2A Konstrukt, das "hyper-responsive", um auch niedrige MgCl2 Konzentrationen war erzeugt visualisieren. Ebenso genießense, Montel et al. (2009) haben gezeigt, dass H2A Bbd Variante bewirkt, dass die Nukleosomen-Arrays zu sein im Vergleich zu Wildtyp-Arrays mehr "offen". Daher ist AFM eine zuverlässige Technik für die Qualitätskontrolle, sowie für die Untersuchung von Chromatin Faserstruktur im allgemeinen.
Offenlegungen
Die Autoren haben keine Interessenkonflikte.
Danksagungen
Diese Arbeit wurde vom NIH Zuschüsse GM45916 und GM66834 zu JCH und ein Stipendium der Internationalen Rett Syndrome Foundation, diese Arbeit AK unterstützt wurde auch von NIH gewähren GM088409 und Howard Hughes Medical Institute Beiträge zur KL unterstützt
Materialien
| Name | Company | Catalog Number | Comments |
| (3-Aminopropyl)triethoxysilane | Sigma-Aldrich | A3648-100ML | |
| 6-8 kDa MWCO Dialysis Tubing | Fisher | 21-152-5 | |
| HiLoad Superdex 200 16/60 Column | GE | 17-1069-01 | |
| Vivaspin 50 kDa MWCO Centrifugal Concentrator | Sartorius | VS2031 | |
| 12-14 kDa MWCO Dialysis Tubing | Fisher | 08-667A | |
| Illustra Sephacryl S-1000 Superfine | GE | 17-0476-01 | |
| XL-A/I Analytical Ultracentrifuge | Beckman-Coulter |
Referenzen
- Luger, K., Mader, A., Richmond, R., Crystal Sargent, D. structure of the nucleosome core particle at 2.8 A resolution. Nature. 7, (1997).
- Hansen, J. C. Conformational dynamics of the chromatin fiber in solution: determinants, mechanisms, and functions. Annual Review of Biophysics and Biomolecular Structure. 31, 361-392 (2002).
- McBryant, S., Adams, V., Hansen, J. Chromatin architectural proteins. Chromosome Research. 14 (1), 39-51 (2006).
- Hansen, J. C., Ausio, J., Stanik, V. H., van Holde, K. E. Homogeneous reconstituted oligonucleosomes, evidence for salt-dependent folding in the absence of histone H1. Biochemistry. 28 (23), 9129-9136 (1989).
- Szerlong, H. J., Prenni, J. E., Nyborg, J. K., Hansen, J. C. Activator-dependent p300 acetylation of chromatin in vitro: enhancement of transcription by disruption of repressive nucleosome-nucleosome interactions. The Journal of Biological Chemistry. 285 (42), 31954-31964 (2010).
- Cirillo, L. A., Lin, F. R., Cuesta, I., Friedman, D., Jarnik, M., Zaret, K. S. Opening of compacted chromatin by early developmental transcription factors HNF3 (FoxA) and GATA-4. Molecular Cell. 9 (2), 279-289 (2002).
- Nguyen, C., Gonzales, F. chromatin structure associated with methylation-induced gene silencing in cancer cells: correlation of accessibility, methylation, MeCP2 binding and acetylation. Nucleic Acids Research. 29 (22), 4598-4606 (2001).
- Cuesta, I., Zaret, K. S., Santisteban, P. The forkhead factor FoxE1 binds to the thyroperoxidase promoter during thyroid cell differentiation and modifies compacted chromatin structure. Molecular and Cellular Biology. 27 (20), 7302-7314 (2007).
- Simpson, R. T., Stafford, D. W. Structural features of a phased nucleosome core particle. Proceedings of the National Academy of Sciences of the U S A. 80 (1), 51-55 (1983).
- Lowary, P. T., Widom, J. New DNA sequence rules for high affinity binding to histone octamer and sequence-directed nucleosome positioning. Journal of Molecular Biology. 276 (1), 19-42 (1998).
- Lowary, P., Widlund, H., Cao, H. Sequence motifs and free energies of selected natural and non-natural nucleosome positioning DNA sequences. Journal of Molecular Biology. 288, 213-229 (1999).
- Gordon, F., Luger, K., Hansen, J. C. The Core Histone N-terminal Tail Domains Function Independently and Additively during Salt-dependent Oligomerization of Nucleosomal Arrays *. The Journal of Biological Chemistry. 280 (40), 33701-33706 (2005).
- Luger, K., Rechsteiner, T. J., Richmond, T. J. Expression and purification of recombinant histones and nucleosome reconstitution. Methods in Molecular Biology (Clifton, N.J.). 119 (4), 1-16 (1999).
- McBryant, S. J., Klonoski, J., et al. Determinants of histone H4 N-terminal domain function during nucleosomal array oligomerization: roles of amino acid sequence, domain length, and charge density. The Journal of Biological Chemistry. 284 (25), 16716-16722 (2009).
- Ma Shogren-Knaak, ., Fry, C. J., Peterson, C. L. A native peptide ligation strategy for deciphering nucleosomal histone modifications. The Journal of Biological Chemistry. 278 (18), 15744-158 (2003).
- Lu, X., Simon, M. The effect of H3K79 dimethylation and H4K20 trimethylation on nucleosome and chromatin structure. Nat Struct Mol Biol. 15 (10), 1122-1124 (2008).
- Ausio, J. Analytical Ultracentrifugation for the Analysis of Chromatin Structure. Biophysical Chemistry. 86 (2-3), 141-153 (2000).
- Hansen, J., Kreider, J., Demeler, B., Fletcher, T. Analytical ultracentrifugation and agarose gel electrophoresis as tools for studying chromatin folding in solution. Methods. 12 (1), 62-72 (1997).
- Montel, F., Menoni, H., et al. The dynamics of individual nucleosomes controls the chromatin condensation pathway: direct atomic force microscopy visualization of variant chromatin. Biophysical Journal. 97 (2), 544-5453 (2009).
- Muthurajan, U. M., McBryant, S. J., Lu, X., Hansen, J. C., Luger, K. The linker region of macroH2A promotes self-association of nucleosomal arrays. The Journal of Biological Chemistry. 286 (27), 23852-23864 (2011).
- Shlyakhtenko, L. S., Lushnikov, A. Y., Lyubchenko, Y. L. Dynamics of nucleosomes revealed by time-lapse atomic force microscopy. Biochemistry. 48 (33), 7842-7848 (2009).
- Lohr, D., Bash, R., Wang, H., Yodh, J., Lindsay, S. Using atomic force microscopy to study chromatin structure and nucleosome remodeling. Methods (San Diego, Calif). 41 (3), 333-341 (2007).
- Dyer, P. N., Edayathumangalam, R. S., et al. Reconstitution of nucleosome core particles from recombinant histones and DNA. Methods in Enzymology. 375, 23-44 (2004).
- Sambrook, J., Russell, D. . Molecular cloning: a laboratory manual. , (2001).
- Balbo, A., Zhao, H., Brown, P. H., Schuck, P. Assembly, loading, and alignment of an analytical ultracentrifuge sample cell. J. Vis. Exp. (33), e1530 (2009).
- Demeler, B. UltraScan: a comprehensive data analysis software package for analytical ultracentrifugation experiments. Modern Analytical Ultracentrifugation: Techniques. , 210-230 (2005).
- Holde, K. V., Weischet, W. Boundary analysis of sedimentation velocity experiments with monodisperse and paucidisperse solutes. Biopolymers. 17 (6), 1387-1403 (1978).
- Demeler, B., van Holde, K. E. Sedimentation velocity analysis of highly heterogeneous systems. Analytical Biochemistry. 335, 279-288 (2004).
- Hansen, J., Lohr, D. Assembly and structural properties of subsaturated chromatin arrays. Journal of Biological Chemistry. 8, 5840-5848 (1993).
- Routh, A., Sandin, S., Rhodes, D. Nucleosome repeat length and linker histone stoichiometry determine chromatin fiber structure. Proceedings of the National Academy of Sciences of the U S A. 105 (26), 8872-8877 (2008).
- Zhou, J., Fan, J. Y., Rangasamy, D., Tremethick, D. J. The nucleosome surface regulates chromatin compaction and couples it with transcriptional repression. Nature Structural & Molecular Biology. 14 (11), 1070-1076 (2007).
- Dorigo, B., Schalch, T., Kulangara, A., Duda, S., Schroeder, R. R., Richmond, T. J. Nucleosome arrays reveal the two-start organization of the chromatin fiber. Science (New York, N.Y.). 306 (5701), 1571-1573 (2004).
- Correll, S. J., Schubert, M. H., Grigoryev, S. a Short nucleosome repeats impose rotational modulations on chromatin fibre folding. The EMBO Journal. 31 (10), 2416-2426 (2012).
- Mcbryant, S. J., Krause, C., Woodcock, C. L., Hansen, J. C. The Silent Information Regulator 3 Protein , SIR3p , Binds to Chromatin Fibers and Assembles a Hypercondensed Chromatin Architecture in the Presence of Salt. Molecular and Cellular Biology. 28 (11), 3563-3572 (2008).
- Schalch, T., Duda, S., Sargent, D. F., Richmond, T. J. X-ray structure of a tetranucleosome and its implications for the chromatin fibre. Nature. 436 (7047), 138-1341 (2005).
- Fan, J. Y., Gordon, F., Luger, K., Hansen, J. C., Tremethick, D. J. The essential histone variant H2A.Z regulates the equilibrium between different chromatin conformational states. Nature Structural Biology. 9 (3), 172-176 (2002).
- Carruthers, L. M., Bednar, J., Woodcock, C. L., Hansen, J. C. Linker histones stabilize the intrinsic salt-dependent folding of nucleosomal arrays: mechanistic ramifications for higher-order chromatin folding. Biochemistry. 37 (42), 14776-14787 (1998).
- Huynh, V. A. T., Robinson, P. J. J., Rhodes, D. A Method for the In Vitro Reconstitution of a Defined "30 nm" Chromatin Fibre Containing Stoichiometric Amounts of the Linker Histone. Journal of Molecular Biology. 345 (5), 957-968 (2005).
- Dorigo, B., Schalch, T. Chromatin fiber folding: requirement for the histone H4 N-terminal tail. J. Mol. Biol. 2836 (03), 85-96 (2003).
- Qian, R. L., Liu, Z. X., et al. Visualization of chromatin folding patterns in chicken erythrocytes by atomic force microscopy (AFM. Cell Research. 7 (2), 143-150 (1997).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten
Wir verwenden Cookies, um Ihre Erfahrung auf unserer Website zu verbessern.
Indem Sie unsere Website weiterhin nutzen oder auf „Weiter“ klicken, stimmen Sie zu, unsere Cookies zu akzeptieren.