Method Article
Un approccio efficiente alla transgenesi per la consegna genica nel cuore embrionale di topo
In questo articolo
Riepilogo
Questo protocollo presenta un quadro metodologico dettagliato per la transgenesi basata sull'elettroporazione di cellule cardiache in cuori di topo in via di sviluppo. Le risorse video fornite qui faciliteranno l'apprendimento di questa tecnica versatile.
Abstract
Il cuore dei mammiferi è un organo complesso formatosi durante lo sviluppo attraverso popolazioni molto diverse di cellule progenitrici. L'origine, i tempi di reclutamento e il destino di questi progenitori sono vitali per il corretto sviluppo di questo organo. I meccanismi molecolari che governano la morfogenesi del cuore sono essenziali per comprendere la patogenesi delle cardiopatie congenite e della rigenerazione cardiaca embrionale. Gli approcci classici per studiare questi meccanismi hanno impiegato la generazione di topi transgenici per valutare la funzione di geni specifici durante lo sviluppo cardiaco. Tuttavia, la transgenesi del topo è un processo complesso e dispendioso in termini di tempo che spesso non può essere eseguito per valutare il ruolo di geni specifici durante lo sviluppo del cuore. Per affrontare questo problema, abbiamo sviluppato un protocollo per l'elettroporazione e la coltura efficienti di cuori embrionali di topo, consentendo alla transgenesi transitoria di valutare rapidamente l'effetto del guadagno o della perdita di funzione dei geni coinvolti nello sviluppo cardiaco. Utilizzando questa metodologia, abbiamo sovraespresso con successo Meis1 nel cuore embrionale, con una preferenza per la trasfezione di cellule epicardiche, dimostrando le capacità della tecnica.
Introduzione
Il cuore è il primo organo che si forma durante lo sviluppo embrionale. Questo processo comporta la coordinazione spazio-temporale di varie popolazioni di cellule progenitrici provenienti da aree distinte dell'embrione. Tutto ciò avviene mentre il cuore in via di sviluppo continua a battere e a funzionare, sottolineando la notevole coordinazione necessaria per la sua formazione 1,2,3. Dato il ruolo cruciale del cuore, una stretta regolazione a livello cellulare e molecolare è essenziale per la sua corretta formazione 4,5. Identificare i meccanismi che controllano lo sviluppo del cuore è stato di grande interesse, in quanto sono cruciali per svelare le cardiopatie congenite, che colpiscono un numero considerevole di pazienti in tutto il mondo6. Inoltre, la comprensione dello sviluppo cardiaco è fondamentale per decifrare la rigenerazione cardiaca, poiché i cuori dei mammiferi postnatali conservano una capacità rigenerativa che viene persa o ostacolata in età adulta 7,8. Di conseguenza, l'analisi dei regolatori molecolari dello sviluppo cardiaco è fondamentale per far avanzare gli sforzi di ricerca sulle cardiopatie congenite e sulla rigenerazione cardiaca.
Nel perseguimento di questo obiettivo, c'è stata una crescente attenzione allo studio del ruolo dell'epicardio nello sviluppo e nella rigenerazione cardiaca9. L'epicardio è un sottile strato di tessuto mesoteliale che comprende lo strato più esterno del cuore dei mammiferi (Figura 1). Recenti studi hanno dimostrato l'importanza dell'epicardio durante il danno cardiaco, rivelando che questo tessuto è in grado di inviare segnali di proliferazione ai cardiomiociti nella zona interessata per mitigare il danno10,11. Nonostante l'importanza dell'epicardio, la conduzione di ulteriori indagini molecolari è stata messa alla prova dalla sua immensa eterogeneità. Gli esperimenti di RNAseq a singola cellula hanno rivelato l'eterogeneità dell'epicardio, che ospita più sottopopolazioni cellulari con firme trascrittomiche distinte 12,13,14,15,16. Pertanto, una strategia per lo screening dei potenziali regolatori dello sviluppo cardiaco dovrebbe tenere conto della diversità delle cellule progenitrici epicardiche.
In questo senso, l'adattabilità del modello murino alla modificazione genetica ha facilitato l'identificazione di numerosi geni cruciali per lo sviluppo del cuore, consentendo la generazione di linee mutanti con guadagno di funzione (GOF) o perdita di funzione (LOF) di geni specifici. Tuttavia, questi approcci implicano un notevole investimento di tempo e risorse sperimentali; pertanto, non sono pratici quando si valutano i ruoli di un gran numero di geni candidati. Inoltre, i geni dello sviluppo spesso esercitano funzioni pleiotropiche in diversi tessuti o sono necessari per lo sviluppo embrionale precoce, ostacolando l'interpretazione del loro contributo allo sviluppo in un processo specifico. Sebbene sia possibile indirizzare la funzione genica in strutture specifiche o in punti temporali dello sviluppo, ciò di solito richiede l'uso di costruzioni genetiche più complesse, che possono essere difficili da generare o sono generalmente non disponibili.
Per superare queste limitazioni, presentiamo una metodologia per elettroporare cuori embrionali di topo per la transgenesi transitoria (Figura 2). Abbinata alla coltura ex vivo e al cell sorting attivato da fluorescenza (FACS), questa strategia dimostra le sue capacità attraverso il GOF transitorio di Meis1, un gene ben caratterizzato implicato nello sviluppo e nella rigenerazione del cuore 17,18,19. In questo articolo, vengono esplorate anche altre potenziali applicazioni di questa metodologia, e vengono discussi i suoi vantaggi e limiti, nonché rispetto ai protocolli esistenti per la modulazione transitoria dell'espressione genica. Riteniamo che il quadro e gli esempi visivi presentati miglioreranno la comprensione della biologia dell'epicardio durante lo sviluppo e la malattia.
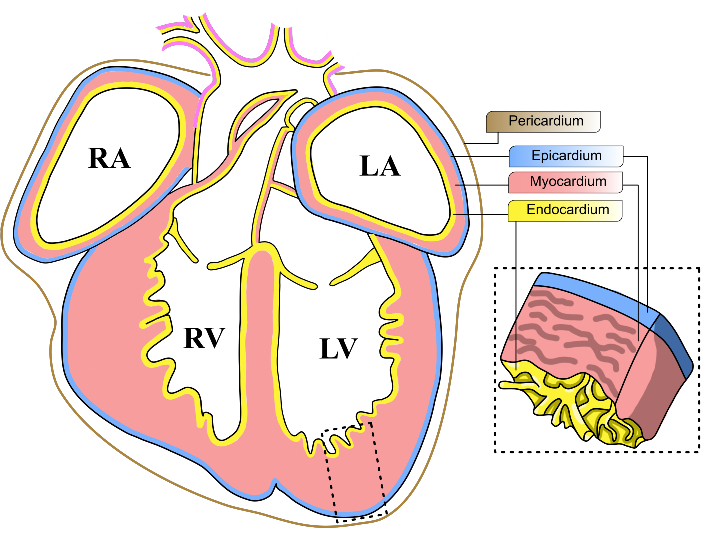
Figura 1: Strati cardiaci embrionali di topo. Diagramma schematico di una vista coronale di un cuore embrionale di topo E13-14. I tre principali strati cellulari del cuore sono rappresentati in giallo (endocardio), rosso (miocardio) e blu (epicardio). Il pericardio è rappresentato da una linea marrone. Le quattro camere del cuore sono abbreviate in LV, ventricolo sinistro; RV, ventricolo destro; LA, atrio sinistro; RA, atrio destro. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Panoramica schematica del protocollo di elettroporazione cardiaca. Fare clic qui per visualizzare una versione più grande di questa figura.
Protocollo
Tutte le procedure per gli animali sono state approvate dal Comitato Etico per la Sperimentazione Animale del CNIC e sono conformi alla legislazione vigente, tra cui la Direttiva UE 2010/63 e la Raccomandazione 2007/526/CE, come applicato dalla legge spagnola ai sensi del Real Decreto 53/2013. Per questo protocollo, sono stati impiegati topi CD-1 femmina wild-type di età compresa tra 15 e 21 settimane. I dettagli relativi agli animali, ai reagenti e alle attrezzature utilizzate sono elencati nella Tabella dei materiali.
1. Plasmide e preparazione degli strumenti
- Innanzitutto, preparare la miscela di elettroporazione aggiungendo il DNA plasmidico desiderato nel ghiaccio a una concentrazione finale di 1 μg/μL. Quindi, completare la miscela aggiungendo saccarosio alla concentrazione finale al 6% (disciolto in PBS), un colorante biocompatibile allo 0,01%, e completare con PBS fino ad un volume finale di 30 μL.
NOTA: Per campione vengono utilizzati circa 1-2 μl della miscela di elettroporazione. Scala le proporzioni della miscela di elettroporazione in base al numero di campioni nell'esperimento. Si ricorda che il DNA per l'elettroporazione deve essere purificato da colture batteriche, garantendo una stabilità ottimale e contaminanti minimi. Per maggiori dettagli sui plasmidi utilizzati per questo studio, fare riferimento alla sezione dei risultati rappresentativi. - Preparare il terreno di crescita integrando il Modified Eagle Medium (DMEM) di Dulbecco con il 10% di siero fetale bovino (FBS), il 2% di tampone Hepes e l'1% di penicillina/streptomicina. Conservare il terreno in un'incubatrice a 37 °C fino al momento dell'uso. Inoltre, scaldare 1x PBS a 37 °C, insieme al terreno di coltura.
- Preparare gli aghi allungando un capillare di vetro di 20 μm di diametro utilizzando un estrattore per pipette con le seguenti condizioni: calore = 550; tirare = 100; velocità = 250; tempo = 120. Il diametro finale dopo l'estrazione dovrebbe essere approssimativamente compreso tra 5-8 μm.
- Impostare i parametri dell'elettroporatore su cinque cicli da 50 ms a 40 V con intervalli di 900 ms. Preparare gli elettrodi, la pipetta orale e l'ago smussato (Figura 3A). Equipaggiare la pipetta con l'ago per caricare il DNA (Figura 3A).
2. Dissezione embrionale ed estrazione cardiaca
- Imposta un incrocio tra topi CD-1 wild-type. La mattina successiva, seleziona le femmine che presentano un tappo vaginale e annota questo momento come giorno embrionale E0.5. Eutanasia degli animali al giorno embrionale E12-12.5 mediante lussazione cervicale (seguendo protocolli istituzionalmente approvati).
- Estrarre con cura l'utero dal topo e metterlo in un tovagliolo di carta asciutto. Successivamente, tagliare l'utero con delle forbici sottili inserendo la punta delle forbici nel lato mesometrial e facendo scorrere lentamente il bordo della lama per tutta la lunghezza. Esponi le decidue e trasferiscile nel terreno di coltura. Per maggiori dettagli, si veda il lavoro di Behringer et al.20.
- Sezionare le decidue per estrarre gli embrioni, quindi posizionare gli embrioni in una capsula di Petri da 60 mm con terreno di coltura caldo. Usando una pinza fine, taglia con cura la coda e la testa dell'embrione in modo che rimanga solo il tronco del corpo. Lo stadio degli embrioni può essere verificato valutando le caratteristiche anatomiche, come descritto da Wong et al.21.
- Quindi, rimuovere con cautela quanto più tessuto possibile dal tronco dell'embrione senza danneggiare il cuore o le arterie polmonari e aortiche. Una volta che il cuore è stato esposto, posizionarlo immediatamente in un nuovo terreno di coltura caldo e tenerlo in un'incubatrice a 37 °C fino all'elettroporazione.
NOTA: Mantenere la temperatura del cuore è fondamentale per la sua sopravvivenza. Assicurarsi che il mezzo sia sempre caldo e sostituirlo ogni 5-10 minuti. D'altra parte, i campioni possono essere manipolati senza mantenere la sterilità, in quanto ciò non influirà sull'efficienza della procedura. Si prega di notare che il terreno di coltura contiene antibiotici.
3. Elettroporazione cardiaca e coltura ex vivo
- Caricare circa 10 μl di miscela plasmidica (preparata al punto 1.1) nell'ago aspirando con cautela con la pipetta orale.
- Preparare un singolo cuore per l'elettroporazione mettendolo in una capsula di Petri pulita da 60 mm con 1x PBS sterile riscaldato a 37 °C. Posizionare il piatto sotto il microscopio da dissezione (Figura 3A).
- Preparare gli elettrodi regolando la distanza tra i poli positivo e negativo a circa 4 mm (Figura 3B).
NOTA: 4 mm è la distanza di apertura in cui i cuori di mouse E12.5 si inseriscono negli elettrodi utilizzati in questo lavoro. Si prega di prendere in considerazione la possibilità di modificare questa distanza in base alle diverse dimensioni del campione o alle specifiche dell'elettrodo. - Perforare delicatamente lo strato più superficiale del cuore con l'ago nella pipetta orale. Pipettare con cautela 1-2 μL della miscela plasmidica e rimuovere l'ago. Possono essere eseguite iniezioni multiple per garantire una penetrazione ottimale, nonché per espandere il numero di celle elettroporate.
- Tenere gli elettrodi in posizione in modo che il cuore si trovi tra entrambi i poli (Figura 3C). Quindi, elettroporare il cuore con le condizioni sopra specificate.
NOTA: Per garantire la massima efficienza di elettroporazione, è possibile cambiare la polarità degli elettrodi e dare un altro impulso. - Trasferire il cuore elettroporoso in una nuova piastra a 12 pozzetti contenente 1 mL di terreno di coltura e posizionarlo a 37 °C, 5% CO2.
- Ripetere i passaggi da 3.1 a 3.6 fino a quando tutti i campioni dell'esperimento sono elettroporizzati. Infine, incubare tutti i cuori in un'incubatrice a 37 °C, 5% CO2, per 24 ore o fino all'analisi.
NOTA: Nelle nostre mani, i cuori potrebbero essere lasciati in coltura per un massimo di 48 ore senza apparenti segni di deterioramento (Filmato supplementare 1; Film supplementare 2; Figura supplementare 1).
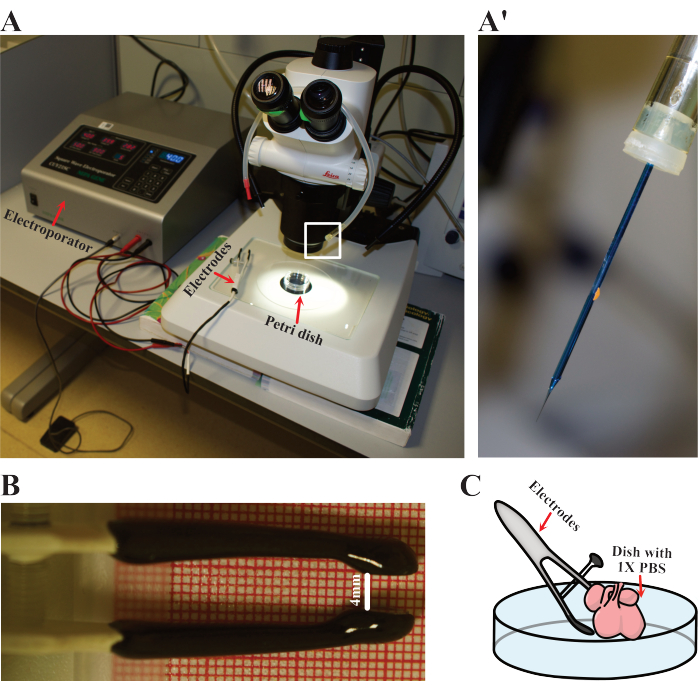
Figura 3: Configurazione dell'elettroporazione. (A) Configurazione utilizzata per l'elettroporazione. Le frecce rosse indicano l'elettroporatore, gli elettrodi e la capsula di Petri dove i cuori vengono elettroporati. (A') Dettaglio ravvicinato dell'ago per elettroporazione come evidenziato in (A) dal rettangolo bianco. (B) Dettaglio della distanza regolata degli elettrodi utilizzati nei cuori E12.5. (C) Rappresentazione schematica dell'elettroporazione. Clicca qui per visualizzare una versione più grande di questa figura.
4. Cell sorting e analisi immunoistochimica
- Per l'analisi di smistamento cellulare dei campioni, preparare un mezzo di digestione per dissociare il tessuto mescolando insieme 400 μL di liberasi (5 mg/ml), 300 μL di dispasi (0,1 g/mL), 4 μL di CaCl2 (0,4 μM), 50 μL di MgCl2 (5 μM) e completare fino a 10 mL di DMEM. Tenere in ghiaccio fino al momento dell'uso. Ogni cuore ha bisogno di circa 1 ml di terreno di digestione. Regolare i volumi in base al numero di campioni.
NOTA: Per l'immunoistochimica, fissare direttamente i cuori in 1,5 mL di PFA al 4% per 2 ore a RT o durante la notte a 4 °C. Il giorno successivo, lavare i cuori 3 volte con PBS e procedere alle procedure istologiche e di immunocolorazione a valle. Per un protocollo di immunoistochimica dettagliato, fare riferimento a Morris et al.22. - Trasferire ciascun cuore in coltura in una nuova provetta contenente 1 mL di terreno di digestione ghiacciato.
- Per facilitare la dissociazione, tritare il tessuto applicando delicatamente il piacere con una siringa 3-4 volte e incubare immediatamente in una piastra calda a 37 °C, 600 giri/min per 45 minuti.
- Filtrare la miscela di digestione attraverso un filtro cellulare da 70 μm in una nuova provetta e lasciarla svuotare per gravità. Quindi, recuperare il volume passante e filtrarlo nuovamente utilizzando un filtro cellulare da 40 μm in una nuova provetta da 50 mL.
- Aggiungere 500 μl di siero fetale bovino (FBS) e completare fino a 30 mL utilizzando DMEM freddo.
- Centrifugare la miscela a 240 x g per 10 minuti a temperatura ambiente.
- Scartare il surnatante e posizionare la provetta da 50 ml capovolta su un tovagliolo di carta per asciugarla completamente.
NOTA: Questo è un punto conveniente per mettere in pausa l'esperimento congelando il pellet della cella. In tal caso, risospendere le cellule con DMSO al 10% in FBS e conservarle a -80 °C. - Per la selezione cellulare, risospendere le cellule in 300 μL di tampone di smistamento (1% FBS, 1% penicillina/streptomicina in PBS) e aggiungere 0,3 μL di DAPI (1000x). Eseguire l'ordinamento delle celle.
Risultati
Per dimostrare l'efficacia di questa tecnica nell'esecuzione di esperimenti di guadagno di funzione (GOF) per regolatori dello sviluppo cardiaco rilevanti, un costrutto è stato elettroporatamente sovraesprimendo il fattore di trascrizione Meis1. Per raggiungere questo obiettivo, l'RNA è stato estratto dagli embrioni E9.5 ed è stata eseguita la trascrizione inversa per ottenere il DNA complementare (cDNA). Utilizzando il cDNA come modello, la sequenza codificante Meis1 è stata clonata (Tabella supplementare 1) in un plasmide di espressione pCAG (di seguito denominato pCAG::Meis1), mentre un plasmide costitutivo che esprime GFP (pCAG::GFP) è stato utilizzato come controllo. I cuori sono stati quindi elettroporati solo con pCAG::GFP o in combinazione con pCAG::Meis1.
Dopo 24 ore dopo l'elettroporazione (hpe), i cuori battevano e sembravano essere in buone condizioni (Figura 4A; Film supplementare 1; Filmato supplementare 2). Per valutare la vitalità delle cellule elettroporate, l'apoptosi è stata analizzata attraverso la colorazione immunoistochimica della caspasi 3. Sebbene siano state rilevate alcune cellule positive all'interno del cuore, né il miocardio né l'epicardio, dove si trova la maggior parte delle cellule elettroporate, sono risultati affetti da apoptosi (Figura 1C supplementare). Quando è stata valutata l'attività di fluorescenza, è stato osservato un segnale GFP a mosaico in quasi tutte e quattro le camere del cuore, indicando che questo protocollo è in grado di raggiungere la maggior parte delle strutture cardiache (Figura 4A). Per determinare quali tipi di cellule cardiache sono state elettroporizzate, i cuori elettroporati con pCAG::GFP sono stati immunocolorati contro i marcatori dell'epicardio, WT12, e del miocardio, MF2023, e quindi sottoposti a imaging utilizzando un microscopio confocale (Figura 4B). La maggior parte delle cellule GFP+ erano situate preferenzialmente nella regione esterna del cuore, il che corrispondeva in gran parte al segnale WT1, indicando che questo metodo è più efficiente nel raggiungere le cellule epicardiche (Figura 4B, punte di freccia bianche).
D'altra parte, i cuori elettroporati o di controllo (solo pCAG::GFP) sono stati dissociati e le cellule sono state sottoposte a smistamento cellulare attivato dalla fluorescenza (FACS) per isolare le cellule GFP+ dall'intero organo, come descritto nel passaggio 4 del protocollo. L'RNA è stato estratto dalle cellule selezionate ed è stata eseguita la PCR quantitativa a trascrizione inversa (RT-qPCR) per valutare i livelli di espressione di Meis1 nelle cellule trasfettate (Figura 4C, Tabella supplementare 1). L'espressione genica totale di Meis1 è stata normalizzata utilizzando i livelli di espressione del gene di pulizia del topo Eef224 e l'espressione genica relativa è stata calcolata utilizzando il metodo ΔΔCt25. Una significativa sovraregolazione dei livelli di RNA di Meis1 è stata osservata nei cuori elettroporati con il plasmide pCAG::Meis1 rispetto a quelli con solo il plasmide pCAG::GFP (Figura 4C). Pertanto, ciò dimostra il potenziale della tecnica di sovraesprimere i geni di interesse e affrontare l'esito molecolare.

Figura 4: Analisi molecolare di cuori elettroporati di E12.5 . (A) Fluorescenza GFP di un cuore elettroporato CAG::GFP 24 ore dopo l'elettroporazione. L'immagine inferiore mostra la fluorescenza GFP, mentre l'immagine superiore mostra una fusione di GFP e un campo luminoso dello stesso cuore. (B) Immunofluorescenza di sezioni istologiche cardiache elettroporate E12.5. MF20 marcare i cardiomiociti, mentre WT1 marcare le cellule epicardiche. Le immagini a destra corrispondono agli ingrandimenti del rettangolo bianco. Le punte di freccia bianche indicano le cellule GFP+ situate nell'epicardio, delimitate approssimativamente da una linea bianca tratteggiata. (C) Analisi RT-qPCR di cuori elettroporati con pCAG::Meis1 o pCAG::GFP (controllo). Ogni punto rappresenta una replica biologica ottenuta dall'unione di tre cuori. In totale, l'analisi qPCR è stata eseguita in triplicati con tre repliche biologiche distinte. L'analisi statistica è stata eseguita utilizzando il test t di Student (*P < 0,05). Barre della scala: 200 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
Figura supplementare 1: Efficienza dell'elettroporazione cardiaca e vitalità cellulare. (A) Espressione di GFP in due distinti cuori E12.5 elettroporati con il plasmide di controllo pCAG::GFP a 24 ore o 48 ore dopo l'elettroporazione (hpe). L'immagine del segnale GFP è stata sovrapposta a un'immagine in campo chiaro dello stesso cuore. (B) Quantificazione di cellule GFP+ in cuori da 24 hpe e 48 hpe elettroporati con pCAG::GFP di controllo. I punti indicano campioni distinti; Le linee continue indicano la media, mentre le barre di errore indicano la deviazione standard. Non è stata osservata alcuna differenza significativa tra le due condizioni ( t-test non parametrico; P > 0,9). (C) Immagine confocale di un cuore E12.5 elettroporato con pCAG::GFP immunocolorato contro la caspasi 3 scissa e MF20. La linea tratteggiata rappresenta lo strato miocardico del cuore. Le immagini a destra vengono ingrandite dal rettangolo bianco. Barre di scala: 100 μm. Clicca qui per scaricare questo file.
Tabella supplementare 1: Riassunto del primer e sequenze. Clicca qui per scaricare questo file.
Filmato supplementare 1: Cuore elettroporato pCAG::GFP dopo 24 ore. Filmato che cattura un cuore elettroporato con plasmide di controllo pCAG::GFP 24 ore dopo la procedura. Il cuore continua a battere e le cellule elettroporate esprimono segnali GFP. Clicca qui per scaricare questo filmato.
Filmato supplementare 2: Cuore elettroporoso pCAG::GFP dopo 48 ore. Filmato che mostra un cuore elettroporata con controllo pCAG::GFP, 48 h dopo l'elettroporazione. Clicca qui per scaricare questo filmato.
Discussione
Nel complesso, la metodologia qui descritta offre un quadro robusto per l'espressione di costrutti transgenici nell'epicardio in via di sviluppo (Figura 4B), come dimostrato dalla sovraespressione di Meis1 (Figura 4C). Con i costrutti appropriati, questo protocollo può essere utilizzato per valutare transitoriamente l'impatto del guadagno di funzione (GOF) o della perdita di funzione (LOF) di un gene specifico. La LOF può essere implementata nella tecnica trasfettando un plasmide che ha come bersaglio un gene candidato attraverso l'interferenza dell'RNA26. La valutazione rapida del gene GOF o LOF è particolarmente vantaggiosa, soprattutto considerando l'ampia eterogeneità trascrittomica delle cellule epicardiche12, che rende impraticabile la generazione di animali transgenici per valutare ogni gene candidato individualmente.
Poiché questo protocollo utilizza la coltura ex vivo, offre la possibilità di studiare il destino delle cellule epicardiche all'interno di una finestra temporale. Lo sviluppo dell'epicardio svolge un ruolo fondamentale nel modellare regioni distinte all'interno del cuore adulto 13,27,28; Pertanto, comprendere i suoi contributi allo sviluppo del cuore è fondamentale per sezionare le basi delle malformazioni cardiache congenite. Sebbene questo articolo non approfondisca questa applicazione, le linee guida delineate nel protocollo potrebbero potenzialmente essere utili per futuri studi di mappatura del destino dell'epicardio
Nonostante l'utilità pratica del metodo attuale, è necessario riconoscere alcuni limiti. Poiché questa metodologia si basa sulla transgenesi transitoria, l'espressione plasmidica può essere persa o diminuita nel tempo, limitando l'intervallo del protocollo per studiare una specifica finestra di sviluppo29. Tuttavia, in questo studio, la sovraespressione di GFP è stata mantenuta per tutta la durata delle colture post-elettroporazione (48 hpe). Inoltre, i potenziali limiti delle attuali tecnologie di coltura ex vivo ostacolano l'applicabilità del metodo per la valutazione delle fasi di sviluppo embrionale o postnatale tardive, soprattutto quando la coltura ex vivo dopo l'elettroporazione diventa impegnativa30. Sebbene esistano metodi più sofisticati di coltura embrionale ex vivo 31,32, possono richiedere l'uso di attrezzature meno accessibili ai ricercatori e potrebbero non garantire la sopravvivenza del campione.
Sebbene il protocollo qui presentato sia progettato per essere il più semplice possibile per migliorare la riproducibilità, include passaggi critici che sono strumentali per il successo. In primo luogo, la dissezione dell'embrione deve essere condotta con attenzione e rapidità per ridurre al minimo i danni cardiaci, che potrebbero compromettere la sopravvivenza dell'organo dopo l'elettroporazione. Si raccomanda vivamente una particolare attenzione durante questa fase, insieme all'uso di attrezzature di dissezione ottimali. Un altro aspetto critico del protocollo è l'esecuzione dell'elettroporazione stessa, poiché un'iniezione di plasmidi inadeguata può portare a risultati indesiderati. Pertanto, è consigliabile eseguire più iniezioni su un singolo cuore e iniettare il campione in modo piuttosto superficiale potrebbe essere utile per migliorare la sopravvivenza dell'organo. Infine, la dissociazione cellulare durante l'elaborazione cardiaca deve essere eseguita rigorosamente, rispettando i tempi previsti dal protocollo per massimizzare la vitalità cellulare.
In conclusione, l'identificazione dei geni chiave nella formazione del cuore è fondamentale per comprendere i meccanismi alla base delle malattie congenite cardiache e della rigenerazione. La metodologia qui stabilita consente uno screening rapido degli effetti del guadagno di funzione genica (GOF) e della perdita di funzione (LOF) nel cuore in via di sviluppo. Pur riconoscendo alcuni limiti, questo metodo mostra un notevole potenziale per l'estrapolazione a diversi stadi di sviluppo o per mappare il destino delle cellule epicardiche embrionali. Attraverso le linee guida visive fornite in questo articolo, miriamo a offrire una preziosa cassetta degli attrezzi per valutare l'immensa complessità e il potenziale dell'epicardio in via di sviluppo.
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Questo studio è stato supportato dalla sovvenzione RTI2018-097617-J-I00 dal Ministerio de Ciencia e Innovación spagnolo e da Acción 9 dall'Universidad de Jaén all'O.H.O. Grant PGC2018-096486-B-I00 dal Ministerio de Ciencia e Innovación spagnolo e dalla sovvenzione H2020-MSCA-ITN-2016-722427 dal programma Horizon 2020 dell'UE a M.T. JMG è stato supportato da una borsa di dottorato del Ministero della Scienza spagnolo e della Fundación Severo Ochoa (PRE2022-101884). Sia il CNIC che il CBMSO sono sostenuti dal Ministero della Scienza spagnolo, mentre il CNIC è sostenuto dalla Fondazione ProCNIC.
Materiali
| Name | Company | Catalog Number | Comments |
| #55 Forceps | Dumont | 11295-51 | |
| 12-well Clear Flat Bottom Multiwell Cell Culture Plate | BD Falcon | 353043 | |
| 35 mm vise table | Grandado | SKU 8798771617573 | |
| 40 µm Cell Strainer | Fischer Scientific | 08-771-1 | |
| 50 mL tubes | BD Falcon | 352070 | |
| 70 µm Cell Strainer | Corning | CLS431751 | |
| Anti-GFP Policlonal Antibody | Invitrogen | A10262 | 1:1000 dilution used |
| Anti-Myosin 4 (MF20) Monoclonal Antibody | Invitrogen | 14-6503-82 | 1:500 dilution used |
| CD1 Wild Type mice | Provided by Animalary Unit (CNIC) | ||
| Cleaved Caspase-3 (Asp175) Antibody | Cell Signalling Technologies | 9661 | 1:400 dilution used |
| DAPI | Cell Signalling Technologies | 4083 | 1:1000 dilution used |
| Dispase/collagenase | Roche | 10269638001 | |
| Distilled water | |||
| DMEM - Dulbecco's Modified Eagle Medium | Gibco | 10313021 | |
| Fetal Bovine Serum | Invitrogen | 10438-026 | |
| Heracell 150i CO2 Incubator | Thermo Scientific | 51032720 | |
| Leica Stereoscopic Microscope S8AP0 | Leica | 11524102 | |
| Liberase | Roche | 5401119001 | |
| Micropipette Puller Model P-97 | Sutter Instrument | SU-P-97 | |
| pCAG expression plasmid | Addgene | #89689 | |
| Penicillin-streptomycin | Invitrogen | 15070-063 | |
| Petri dishes 35 × 10 mm | BD Falcon | 351008 | |
| Petri dishes 60 × 15 mm | BD Falcon | 353002 | |
| Phenol Red | Merck | P3532 | |
| Pipette tips | Reused from old laboratory equipment | ||
| Rat Serum culture embryo, male rats SPRAGUE DAWLEY RjHan SD | Janvier Labs | 9979 | |
| Recombinant anti-Wilms Tumor Protein 1 (WT1) Antibody | Abcam | ab89901 | 1:300 dilution used |
| Square Wave Electroporator CUY21SC | Nepa Gene | CUY664-10X15 | |
| Sterile PBS | Provided and autoclaved by technical unit | ||
| Sucrose | Millipore | 84100 | |
| Tweezer electrodes with variable gap | Nepa Gene | CUY650P5 |
Riferimenti
- Tyser, R. C., et al. Calcium handling precedes cardiac differentiation to initiate the first heartbeat. eLife. 5, e17113(2016).
- Tyser, R. C. V., et al. Characterization of a common progenitor pool of the epicardium and myocardium. Science. 371 (6533), 2986(2021).
- Sendra, M., Domínguez, J., Torres, M., Ocaña, O. Dissecting the complexity of early heart progenitor cells. J Cardiovasc Dev Dis. 9 (1), 5(2021).
- Ivanovitch, K., Temiño, S., Torres, M. Live imaging of heart tube development in mouse reveals alternating phases of cardiac differentiation and morphogenesis. eLife. 6, e30668(2017).
- Ai, D., et al. Canonical Wnt signaling functions in second heart field to promote right ventricular growth. PNAS. 104 (22), 9319-9324 (2007).
- Zimmerman, M. S., et al. regional, and national burden of congenital heart disease, 1990-2017: A systematic analysis for the Global Burden of Disease Study 2017. Lancet Chil Adolesc Heal. 4 (3), 185-200 (2020).
- Xin, M., Olson, E. N., Bassel-Duby, R. Mending broken hearts: Cardiac development as a basis for adult heart regeneration and repair. Nat Rev Mol Cell Biol. 14 (8), 529-541 (2013).
- Porrello, E. R., et al. Transient regenerative potential of the neonatal mouse heart. Science. 331 (6020), 1078-1080 (2011).
- Cao, J., Poss, K. D. The epicardium as a hub for heart regeneration. Nat Rev Cardiol. 15 (10), 631-647 (2018).
- Zhou, B., et al. Adult mouse epicardium modulates myocardial injury by secreting paracrine factors. JCI. 121 (5), 1894-1904 (2011).
- Van Wijk, B., Gunst, Q. D., Moorman, A. F. M., Van Den Hoff, M. J. B. Cardiac regeneration from activated epicardium. PLOS One. 7 (9), e44692(2012).
- Hesse, J., et al. Single-cell transcriptomics defines heterogeneity of epicardial cells and fibroblasts within the infarcted murine heart. eLife. 10, e65921(2021).
- Streef, T. J., Smits, A. M. Epicardial contribution to the developing and injured heart: Exploring the Cellular composition of the epicardium. Front Cardiovasc Med. 8, 750243(2021).
- Sanchez-Fernandez, C., et al. Understanding epicardial cell heterogeneity during cardiogenesis and heart regeneration. J Cardiovasc Dev Dis. 10 (9), 376(2023).
- Quijada, P., et al. Coordination of endothelial cell positioning and fate specification by the epicardium. Nat Commun. 12 (1), 4155(2021).
- Mantri, M., et al. Spatiotemporal single-cell RNA sequencing of developing chicken hearts identifies interplay between cellular differentiation and morphogenesis. Nat Commun. 12 (1), 1771(2021).
- Paul, S., Zhang, X., He, J. Q. Homeobox gene Meis1 modulates cardiovascular regeneration. Semin Cell Dev Biol. 100, 52-61 (2020).
- Stankunas, K., et al. Pbx/Meis deficiencies demonstrate multigenetic origins of congenital heart disease. Circ Res. 103 (7), 702-709 (2008).
- Liu, Y., et al. Transcription factor Meis1 act as a new regulator of ischemic arrhythmias in mice. J Adv Res. 39, 275-289 (2022).
- Behringer, R. Manipulating the mouse embryo: A laboratory manual. , Cold Spring Harbor Lab Press. Cold Spring Harbor, New York. (2014).
- Wong, M. D., et al. 4D atlas of the mouse embryo for precise morphological staging. Development. 142 (20), 3583-3591 (2015).
- Morris, L., Klanke, C., Lang, S., Lim, F. Y., Crombleholme, T. TdTomato and EGFP identification in histological sections: Insight and alternatives. Biotech Histochem. 85 (6), 379-387 (2010).
- Schiaffino, S., Rossi, A. C., Smerdu, V., Leinwand, L. A., Reggiani, C. Developmental myosins: expression patterns and functional significance. Skelet. Muscle. 5 (1), 22(2015).
- Eissa, N., et al. Stability of reference genes for messenger RNA quantification by real-time pcr in mouse dextran sodium sulfate experimental colitis. PLOS One. 11 (5), e0156289(2016).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2−ΔΔCT method. Methods. 25 (4), 402-408 (2001).
- Lai, S. R., Andrews, L. G., Tollefsbol, T. O. RNA interference using a plasmid construct expressing short-hairpin RNA. Methods Mol Biol. 405, 31-37 (2007).
- Carmona, R., Barrena, S., López Gambero, A. J., Rojas, A., Muñoz-Chápuli, R. Epicardial cell lineages and the origin of the coronary endothelium. FASEB J. 34 (4), 5223-5239 (2020).
- Gittenberger-de Groot, A. C., Vrancken Peeters, M. P. F. M., Mentink, M. M. T., Gourdie, R. G., Poelmann, R. E. Epicardium-derived cells contribute a novel population to the myocardial wall and the atrioventricular cushions. Circ Res. 82 (10), 1043-1052 (1998).
- Chong, Z. X., Yeap, S. K., Ho, W. Y. Transfection types, methods and strategies: A technical review. Peer J. 9, e11165(2021).
- Kałużna, E., Nadel, A., Zimna, A., Rozwadowska, N., Kolanowski, T. Modeling the human heart ex vivo-Current possibilities and strive for future applications. JTERM. 16 (10), 853-874 (2022).
- Aguilera-Castrejon, A., et al. Ex utero mouse embryogenesis from pre-gastrulation to late organogenesis. Nature. 593 (7857), 119-124 (2021).
- Dyer, L. A., Patterson, C. A novel ex vivo culture method for the embryonic mouse heart. J Vis Exp. (75), e50359(2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon