Method Article
Un enfoque eficiente de transgénesis para la entrega de genes en el corazón embrionario de ratón
En este artículo
Resumen
Este protocolo presenta un marco metodológico detallado para la transgénesis de células cardíacas basada en electroporación en corazones de ratón en desarrollo. Los recursos de video que se proporcionan aquí facilitarán el aprendizaje de esta técnica versátil.
Resumen
El corazón de los mamíferos es un órgano complejo que se forma durante el desarrollo a través de poblaciones muy diversas de células progenitoras. El origen, el momento del reclutamiento y el destino de estos progenitores son vitales para el correcto desarrollo de este órgano. Los mecanismos moleculares que gobiernan la morfogénesis del corazón son esenciales para comprender la patogenia de las cardiopatías congénitas y la regeneración cardíaca embrionaria. Los enfoques clásicos para investigar estos mecanismos emplearon la generación de ratones transgénicos para evaluar la función de genes específicos durante el desarrollo cardíaco. Sin embargo, la transgénesis del ratón es un proceso complejo y lento que a menudo no se puede realizar para evaluar el papel de genes específicos durante el desarrollo del corazón. Para abordar esto, hemos desarrollado un protocolo para la electroporación eficiente y el cultivo de corazones embrionarios de ratón, lo que permite que la transgénesis transitoria evalúe rápidamente el efecto de la ganancia o pérdida de función de los genes involucrados en el desarrollo cardíaco. Utilizando esta metodología, sobreexpresamos con éxito Meis1 en el corazón embrionario, con preferencia por la transfección de células epicárdicas, demostrando las capacidades de la técnica.
Introducción
El corazón es el primer órgano que se forma durante el desarrollo embrionario. Este proceso implica la coordinación espacio-temporal de varias poblaciones de células progenitoras de distintas áreas del embrión. Todo esto ocurre mientras el corazón en desarrollo continúa latiendo y funcionando, enfatizando la notable coordinación requerida para su formación 1,2,3. Dado el papel crucial del corazón, una regulación estricta a nivel celular y molecular es esencial para su correcta formación 4,5. La identificación de los mecanismos que controlan el desarrollo del corazón ha sido de gran interés, ya que son cruciales para desentrañar los trastornos cardíacos congénitos, que afectan a un número sustancial de pacientes en todo el mundo6. Además, comprender el desarrollo del corazón es fundamental para descifrar la regeneración cardíaca, ya que el corazón de los mamíferos postnatales conserva una capacidad regenerativa que se pierde o se ve obstaculizada en la edad adulta 7,8. En consecuencia, la disección de los reguladores moleculares del desarrollo cardíaco es imprescindible para avanzar en los esfuerzos de investigación sobre las cardiopatías congénitas y la regeneración cardíaca.
En la búsqueda de este objetivo, ha habido un creciente enfoque en la investigación del papel del epicardio en el desarrollo y la regeneración cardíaca9. El epicardio es una capa delgada de tejido mesotelial que comprende la capa más externa del corazón de los mamíferos (Figura 1). Estudios recientes han demostrado la importancia del epicardio durante la lesión cardíaca, revelando que este tejido es capaz de enviar señales de proliferación a los cardiomiocitos en el área afectada para mitigar el daño 10,11. A pesar de la importancia del epicardio, la realización de nuevas investigaciones moleculares se ha visto dificultada por su inmensa heterogeneidad. Los experimentos de RNAseq de una sola célula han revelado la heterogeneidad del epicardio, que alberga múltiples subpoblaciones celulares con firmas transcriptómicas distintas 12,13,14,15,16. Por lo tanto, una estrategia para detectar posibles reguladores del desarrollo cardíaco debería acomodar la diversidad de células progenitoras epicárdicas.
En este sentido, la susceptibilidad del modelo murino a la modificación genética ha facilitado la identificación de numerosos genes cruciales para el desarrollo del corazón, permitiendo la generación de líneas mutantes con ganancia de función (GOF) o pérdida de función (LOF) de genes específicos. Sin embargo, estos enfoques implican una inversión considerable de tiempo y recursos experimentales; Por lo tanto, son poco prácticos a la hora de evaluar las funciones de un gran número de genes candidatos. Además, los genes del desarrollo a menudo ejercen funciones pleiotrópicas en diferentes tejidos o son necesarios para el desarrollo embrionario temprano, lo que dificulta la interpretación de su contribución al desarrollo en un proceso específico. Si bien es posible dirigirse a la función génica en estructuras específicas o puntos temporales del desarrollo, esto generalmente requiere el uso de construcciones genéticas más complejas, que pueden ser difíciles de generar o que generalmente no están disponibles.
Para superar estas limitaciones, presentamos una metodología para electroporatar corazones embrionarios de ratón para la transgénesis transitoria (Figura 2). Junto con el cultivo ex vivo y la clasificación celular activada por fluorescencia (FACS), esta estrategia demuestra sus capacidades a través del GOF transitorio de Meis1, un gen bien caracterizado implicado en el desarrollo y la regeneración del corazón 17,18,19. En este artículo también se exploran otras posibles aplicaciones de esta metodología, y se discuten sus ventajas y limitaciones, así como se comparan con los protocolos existentes para la modulación transitoria de la expresión génica. Creemos que el marco y los ejemplos visuales presentados mejorarán la comprensión de la biología del epicardio durante el desarrollo y la enfermedad.
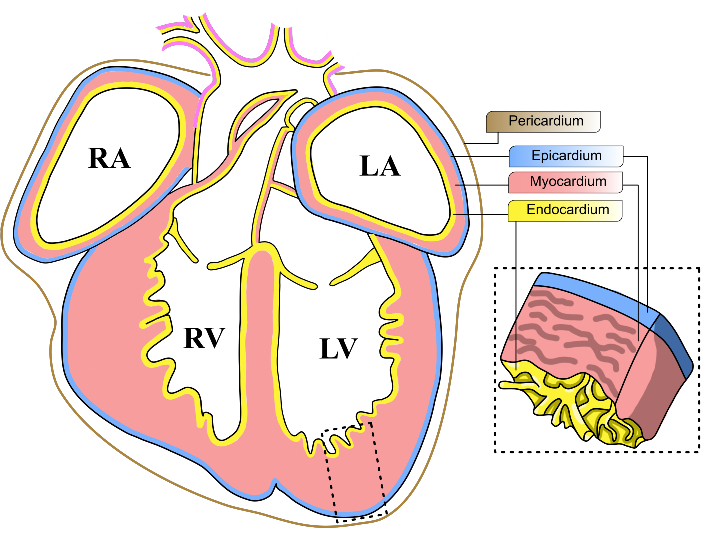
Figura 1: Capas de corazón embrionario de ratón. Diagrama esquemático de una vista coronal de un corazón embrionario de ratón E13-14. Las tres capas celulares principales del corazón están representadas en amarillo (endocardio), rojo (miocardio) y azul (epicardio). El pericardio está representado en una línea marrón. Las cuatro cavidades del corazón se abrevian como VI, ventrículo izquierdo; VD: ventrículo derecho; LA, aurícula izquierda; AR: aurícula derecha. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Resumen esquemático del protocolo de electroporación cardíaca. Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
Todos los procedimientos con animales fueron aprobados por el Comité de Ética de Experimentación Animal del CNIC y se ajustaron a la legislación vigente, incluida la Directiva de la UE 2010/63EU y la Recomendación 2007/526/CE, tal y como se aplica en la legislación española en virtud del Real Decreto 53/2013. Para este protocolo, se emplearon ratones hembra CD-1 de tipo salvaje de 15 a 21 semanas de edad. Los detalles sobre los animales, los reactivos y el equipo utilizado se enumeran en la Tabla de Materiales.
1. Preparación de plásmidos y herramientas
- En primer lugar, prepare la mezcla de electroporación añadiendo el ADN plasmídico deseado en hielo a una concentración final de 1 μg/μL. A continuación, completar la mezcla añadiendo sacarosa al 6% de concentración final (disuelta en PBS), un colorante biocompatible al 0,01%, y completar con PBS hasta un volumen final de 30 μL.
NOTA: Se utiliza aproximadamente 1-2 μL de la mezcla de electroporación por muestra. Escale las proporciones de la mezcla de electroporación de acuerdo con el número de muestras en el experimento. Recuerde que el ADN para la electroporación debe purificarse a partir de cultivos bacterianos, asegurando una estabilidad óptima y un mínimo de contaminantes. Para obtener más detalles sobre los plásmidos utilizados para este estudio, consulte la sección de resultados representativos. - Prepare el medio de crecimiento complementando el medio Eagle modificado de Dulbecco (DMEM) con un 10 % de suero fetal bovino (FBS), un 2 % de tampón Hepes y un 1 % de penicilina/estreptomicina. Mantenga el medio en una incubadora a 37 °C hasta su uso. Además, caliente 1x PBS a 37 °C, junto con el medio de crecimiento.
- Prepare las agujas estirando un capilar de vidrio de 20 μm de diámetro utilizando un extractor de pipetas con las siguientes condiciones: calor = 550; tirar = 100; velocidad = 250; tiempo = 120. El diámetro final después de la extracción debe estar aproximadamente entre 5-8 μm.
- Configure los parámetros del electroporador en cinco ciclos de 50 ms a 40 V con intervalos de 900 ms. Prepare los electrodos, la pipeta bucal y la aguja roma (Figura 3A). Equipe la pipeta con la aguja para cargar el ADN (Figura 3A).
2. Disección embrionaria y extracción del corazón
- Configura un cruce entre ratones CD-1 de tipo salvaje. A la mañana siguiente, seleccione las hembras que exhiben un tapón vaginal y anote esta hora como día embrionario E0.5. Eutanasia de los animales en el día embrionario E12-12.5 por luxación cervical (siguiendo protocolos aprobados institucionalmente).
- Extraiga con cuidado el útero del ratón y colóquelo en una toalla de papel seca. A continuación, corte el útero con unas tijeras finas insertando la punta de las tijeras en el lado del mesometrial y pasando lentamente el borde de la hoja por toda su longitud. Exponga las hojas caducas y transfiéralas al medio de crecimiento. Para más detalles, véase el trabajo de Behringer et al.20.
- Diseccionar las deciduae para extraer los embriones, luego colocar los embriones en una placa de Petri de 60 mm con medio de crecimiento tibio. Con unas pinzas finas, corte con cuidado la cola y la cabeza del embrión para que solo quede el tronco del cuerpo. El estadio de los embriones puede verificarse mediante la evaluación de las características anatómicas, tal como lo describen Wong et al.21.
- Luego, extraiga con precaución la mayor cantidad posible de tejido del tronco del embrión sin dañar el corazón ni las arterias pulmonar y aorta. Una vez que el corazón haya sido expuesto, colóquelo inmediatamente en un medio de crecimiento nuevo y cálido y manténgalo en una incubadora a 37 °C hasta la electroporación.
NOTA: Mantener la temperatura del corazón es crucial para su supervivencia. Asegúrese de que el medio esté siempre caliente y reemplácelo cada 5-10 minutos. Por otro lado, las muestras pueden manipularse sin mantener la esterilidad, ya que esto no afectará la eficiencia del procedimiento. Tenga en cuenta que el medio de crecimiento contiene antibióticos.
3. Electroporación cardíaca y cultivo ex vivo
- Cargue aproximadamente 10 μL de mezcla de plásmidos (preparada en el paso 1.1) en la aguja aspirando cuidadosamente con la pipeta bucal.
- Prepare un solo corazón para la electroporación colocándolo en una placa de Petri limpia de 60 mm con 1x PBS estéril calentado a 37 °C. Coloque el plato bajo el microscopio de disección (Figura 3A).
- Prepare los electrodos ajustando la distancia entre los polos positivo y negativo a aproximadamente 4 mm (Figura 3B).
NOTA: 4 mm es la distancia de apertura en la que encajan los corazones de ratón E12.5 en los electrodos utilizados en este trabajo. Considere cambiar esta distancia en función de los diferentes tamaños de muestra o especificaciones de electrodos. - Perforar suavemente la capa más superficial del corazón con la aguja en la pipeta bucal. Pipetear con cuidado 1-2 μL de la mezcla de plásmidos y retirar la aguja. Se pueden realizar múltiples inyecciones para asegurar una penetración óptima, así como para ampliar el número de células electroporadas.
- Sostenga los electrodos en su lugar de modo que el corazón quede ubicado entre ambos polos (Figura 3C). Luego, electropore el corazón con las condiciones especificadas anteriormente.
NOTA: Para garantizar la máxima eficiencia de electroporación, es posible cambiar la polaridad de los electrodos y dar otro pulso. - Transfiera el corazón electroporado a una nueva placa de 12 pocillos que contenga 1 mL de medio de crecimiento y colóquelo a 37 °C, 5% de CO2.
- Repita los pasos 3.1 a 3.6 hasta que todas las muestras del experimento estén electroporadas. Finalmente, incubar todos los corazones en una incubadora a 37 °C, 5% CO2, durante 24 h o hasta el análisis.
NOTA: En nuestras manos, los corazones podrían dejarse en cultivo hasta 48 h sin signos aparentes de deterioro (Vídeo Suplementario 1; Película Suplementaria 2; Figura complementaria 1).
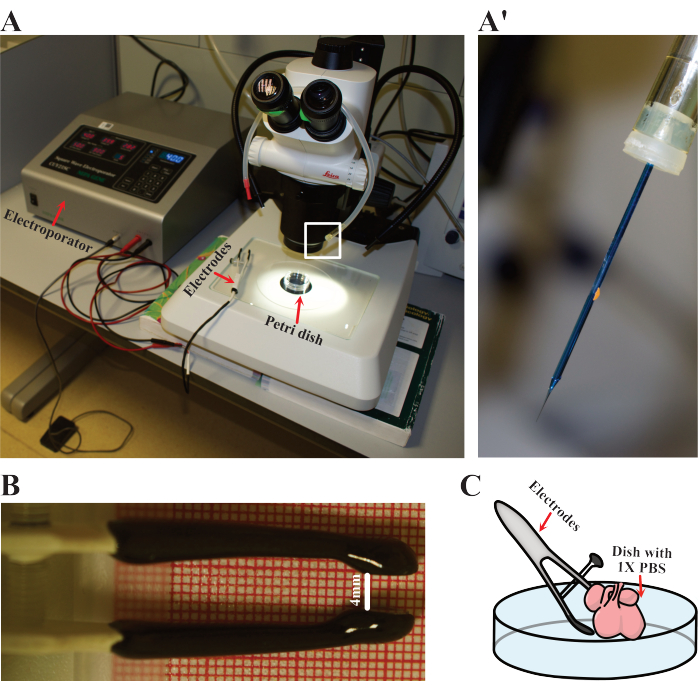
Figura 3: Configuración de la electroporación. (A) Configuración utilizada para la electroporación. Las flechas rojas indican el electroporador, los electrodos y la placa de Petri donde se electroporan los corazones. (A') Detalle cercano de la aguja de electroporación resaltado en (A) por el rectángulo blanco. (B) Detalle de la distancia ajustada de los electrodos utilizados en los corazones E12.5. (C) Representación esquemática de la electroporación. Haga clic aquí para ver una versión más grande de esta figura.
4. Clasificación celular y análisis inmunohistoquímico
- Para el análisis de clasificación celular de las muestras, prepare un medio de digestión para disociar el tejido mezclando 400 μL de liberasa (5 mg/ml), 300 μL de dispasa (0,1 g/mL), 4 μL de CaCl2 (0,4 μM), 50 μL de MgCl2 (5 μM) y complete hasta 10 mL de DMEM. Manténgalo en hielo hasta su uso. Cada corazón necesita aproximadamente 1 mL de medio de digestión. Ajuste los volúmenes de acuerdo con el número de muestras.
NOTA: Para inmunohistoquímica, fije directamente los corazones en 1,5 mL de PFA al 4% durante 2 h a RT o durante la noche a 4 °C. Al día siguiente, lave los corazones 3 veces con PBS y proceda a los procedimientos histológicos e inmunotinción posteriores. Para un protocolo detallado de inmunohistoquímica, consultar Morris et al.22. - Transfiera cada corazón en cultivo a un nuevo tubo que contenga 1 mL de medio de digestión helado.
- Para facilitar la disociación, picar el tejido aplicando suavemente el placer con una jeringa 3-4 veces e incubar inmediatamente en una placa caliente a 37 °C, 600 rpm durante 45 min.
- Filtre la mezcla de digestión a través de un colador de células de 70 μm en un nuevo tubo y déjelo vaciar por gravedad. A continuación, recupere el volumen de paso y vuelva a filtrarlo con un filtro de células de 40 μm en un nuevo tubo de 50 mL.
- Agregue 500 μL de suero fetal bovino (FBS) y complete hasta 30 mL con DMEM frío.
- Centrifugar la mezcla a 240 x g durante 10 min a temperatura ambiente.
- Deseche el sobrenadante y coloque el tubo de 50 ml boca abajo sobre una toalla de papel para que se seque por completo.
NOTA: Este es un punto conveniente para pausar el experimento congelando la pastilla de celda. Si es así, vuelva a suspender las células con DMSO al 10% en FBS y almacene a -80 °C. - Para la clasificación de células, vuelva a suspender las células en 300 μL de tampón de clasificación (1% de FBS, 1% de penicilina/estreptomicina en PBS) y agregue 0,3 μL de DAPI (1000x). Realizar la clasificación de celdas.
Resultados
Para demostrar la eficacia de esta técnica en la realización de experimentos de ganancia de función (GOF) para reguladores relevantes del desarrollo cardíaco, se electroporó un constructo que sobreexpresaba el factor de transcripción Meis1. Para ello, se extrajo ARN de embriones E9.5 y se realizó transcripción inversa para obtener ADN complementario (ADNc). Utilizando el ADNc como plantilla, la secuencia codificante de Meis1 se clonó (Tabla suplementaria 1) en un plásmido de expresión de pCAG (en lo sucesivo denominado pCAG::Meis1), mientras que un plásmido constitutivo que expresa GFP (pCAG::GFP) se utilizó como control. A continuación, los corazones se electroporaron solo con pCAG::GFP o en combinación con pCAG::Meis1.
Después de 24 h después de la electroporación (HPE), los corazones latían y parecían estar en buenas condiciones (Figura 4A; Película Suplementaria 1; Película complementaria 2). Para evaluar la viabilidad de las células electroporadas, se analizó la apoptosis mediante tinción inmunohistoquímica de caspasa 3. Aunque se detectaron algunas células positivas dentro del corazón, ni el miocardio ni el epicardio, donde se encuentran la mayoría de las células electroporadas, se encontraron afectados por la apoptosis (Figura 1C suplementaria). Cuando se evaluó la actividad fluorescente, se observó una señal de GFP en mosaico en casi las cuatro cámaras del corazón, lo que indica que este protocolo es capaz de llegar a la mayoría de las estructuras cardíacas (Figura 4A). Para determinar qué tipos de células cardíacas se electroporaron, los corazones electroporados con pCAG::GFP se inmunoteñieron con marcadores del epicardio, WT12, y miocardio, MF2023, y luego se obtuvieron imágenes con un microscopio confocal (Figura 4B). La mayoría de las células GFP+ se localizaron preferentemente en la región externa del corazón, que se correspondía en gran medida con la señal WT1, lo que indica que este método es más eficiente para llegar a las células epicárdicas (Figura 4B, puntas de flecha blancas).
Por otro lado, se disociaron los corazones electroporados por pCAG::Meis1 o de control (solo pCAG::GFP) y las células se sometieron a una clasificación celular activada por fluorescencia (FACS) para aislar las células GFP+ de todo el órgano, como se describe en el paso 4 del protocolo. Se extrajo ARN de las células seleccionadas y se realizó una PCR cuantitativa con transcripción inversa (RT-qPCR) para evaluar los niveles de expresión de Meis1 en las células transfectadas (Figura 4C, Tabla suplementaria 1). La expresión génica total de Meis1 se normalizó utilizando los niveles de expresión del gen de mantenimiento de ratón Eef224, y la expresión génica relativa se calculó utilizando el método ΔΔCt25. Se observó una regulación positiva significativa de los niveles de ARN de Meis1 en los corazones electroporados con el plásmido pCAG::Meis1 en comparación con aquellos con solo el plásmido pCAG::GFP (Figura 4C). Por lo tanto, esto demuestra el potencial de la técnica para sobreexpresar genes de interés y abordar el resultado molecular.

Figura 4: Análisis molecular de corazones electroporados de E12.5 . (A) Fluorescencia GFP de un corazón electroporado con CAG::GFP 24 h después de la electroporación. La imagen inferior muestra la fluorescencia de GFP, mientras que la imagen superior muestra una fusión de GFP y un campo brillante del mismo corazón. (B) Inmunofluorescencia de cortes histológicos cardíacos electroporados con E12.5. MF20 marca cardiomiocitos, mientras que WT1 marca células epicárdicas. Las imágenes de la derecha corresponden a ampliaciones del rectángulo blanco. Las puntas de flecha blancas indican células GFP+ ubicadas en el epicardio, delimitadas aproximadamente por una línea blanca punteada. (C) Análisis RT-qPCR de corazones electroporados con pCAG::Meis1 o pCAG::GFP (control). Cada punto representa una réplica biológica obtenida de la unión de tres corazones. En total, el análisis de qPCR se realizó por triplicado con tres réplicas biológicas distintas. El análisis estadístico se realizó mediante la prueba t de Student (*P < 0,05). Barras de escala: 200 μm. Haga clic aquí para ver una versión más grande de esta figura.
Figura complementaria 1: Eficiencia de la electroporación cardíaca y viabilidad celular. (A) Expresión de GFP en dos corazones E12.5 distintos electroporados con el plásmido de control pCAG::GFP a las 24 h o 48 h post-electroporación (hpe). La imagen de la señal GFP se superpuso a una imagen de campo claro del mismo corazón. (B) Cuantificación de células GFP+ en corazones de 24 hpe y 48 hpe electroporados con pCAG::GFP de control. Los puntos indican muestras distintas; Las líneas continuas indican la media, mientras que las barras de error indican la desviación estándar. No se observaron diferencias significativas entre ambas condiciones ( prueba t no paramétrica; P > 0,9). (C) Imagen confocal de un corazón E12.5 electroporado con pCAG::GFP inmunoteñido contra caspasa 3 y MF20 escindidos. La línea punteada representa la capa miocárdica del corazón. Las imágenes de la derecha se amplían desde el rectángulo blanco. Barras de escala: 100 μm. Haga clic aquí para descargar este archivo.
Tabla complementaria 1: Resumen del cebador y secuencias. Haga clic aquí para descargar este archivo.
Vídeo complementario 1: pCAG::GFP-corazón electroporado después de 24 h. Captura de vídeo de un corazón electroporado con plásmido de control pCAG::GFP 24 h después del procedimiento. El corazón sigue latiendo y las células electroporadas expresan señales de GFP. Haga clic aquí para descargar esta película.
Vídeo complementario 2: pCAG::GFP-corazón electroporado después de 48 h. Vídeo que muestra un corazón electroporado con control pCAG::GFP, 48 h después de la electroporación. Haga clic aquí para descargar esta película.
Discusión
En general, la metodología descrita aquí ofrece un marco robusto para expresar constructos transgénicos en el epicardio en desarrollo (Figura 4B), como lo demuestra la sobreexpresión de Meis1 (Figura 4C). Con los constructos apropiados, este protocolo se puede utilizar para evaluar transitoriamente el impacto de la ganancia de función (GOF) o la pérdida de función (LOF) de un gen específico. El LOF se puede implementar en la técnica mediante la transfectación de un plásmido dirigido a un gen candidato a través de la interferencia de ARN26. La evaluación rápida del gen GOF o LOF es particularmente ventajosa, especialmente considerando la gran heterogeneidad transcriptómica de las células epicárdicas12, lo que hace poco práctico generar animales transgénicos para evaluar cada gen candidato individualmente.
Debido a que este protocolo utiliza cultivo ex vivo, ofrece la posibilidad de investigar el destino de las células epicárdicas dentro de una ventana temporal. El epicardio en desarrollo desempeña un papel fundamental en la formación de distintas regiones dentro del corazón adulto 13,27,28; Por lo tanto, comprender sus contribuciones al desarrollo del corazón es fundamental para diseccionar las bases de las malformaciones cardíacas congénitas. Si bien este artículo no profundiza en esta aplicación, las pautas descritas en el protocolo podrían ser útiles para futuros estudios de mapeo del destino del epicardio
A pesar de la utilidad práctica del método actual, es necesario reconocer ciertas limitaciones. Dado que esta metodología se basa en la transgénesis transitoria, la expresión del plásmido puede perderse o disminuirse con el tiempo, lo que limita el alcance del protocolo para estudiar una ventana de desarrollo específica29. Sin embargo, en este estudio, la sobreexpresión de GFP se mantuvo durante toda la duración de los cultivos post-electroporación (48 hpe). Además, las limitaciones potenciales en las tecnologías actuales de cultivo ex vivo dificultan la aplicabilidad del método para evaluar las etapas de desarrollo embrionario tardío o postnatal, especialmente cuando el cultivo ex vivo después de la electroporación se vuelve desafiante30. A pesar de que existen métodos más sofisticados de cultivo de embriones ex vivo 31,32, pueden requerir el uso de equipos menos accesibles para los investigadores y pueden no garantizar la supervivencia de la muestra.
Si bien el protocolo presentado aquí está diseñado para ser lo más simple posible para mejorar la reproducibilidad, incluye pasos críticos que son fundamentales para el éxito. En primer lugar, la disección embrionaria debe realizarse con cuidado y rapidez para minimizar el daño cardíaco, que podría comprometer la supervivencia del órgano después de la electroporación. Se recomienda encarecidamente tener especial cuidado durante este paso, junto con el uso de un equipo de disección óptimo. Otro aspecto crítico del protocolo es realizar la electroporación en sí, ya que una inyección inadecuada de plásmidos puede dar lugar a resultados indeseables. Por lo tanto, es aconsejable realizar múltiples inyecciones en un solo corazón, y inyectar la muestra de una manera bastante superficial podría ser beneficioso para mejorar la supervivencia del órgano. Por último, la disociación celular durante el procesamiento cardíaco debe realizarse rigurosamente, cumpliendo con los tiempos previstos en el protocolo para maximizar la viabilidad celular.
En conclusión, la identificación de genes clave en la formación del corazón es fundamental para comprender los mecanismos subyacentes de las enfermedades congénitas cardíacas y la regeneración. La metodología establecida aquí permite la detección rápida de los efectos de ganancia de función (GOF) y pérdida de función (LOF) de genes en el corazón en desarrollo. Si bien reconoce ciertas limitaciones, este método exhibe un considerable potencial para la extrapolación a diferentes etapas del desarrollo o para mapear el destino de las células epicárdicas embrionarias. A través de las pautas visuales proporcionadas en este artículo, nuestro objetivo es ofrecer una valiosa caja de herramientas para evaluar la inmensa complejidad y el potencial del epicardio en desarrollo.
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Este estudio ha sido financiado por la subvención RTI2018-097617-J-I00 del Ministerio de Ciencia e Innovación y Acción 9 de la Universidad de Jaén a la O.H.O. La subvención PGC2018-096486-B-I00 del Ministerio de Ciencia e Innovación y la subvención H2020-MSCA-ITN-2016-722427 del programa Horizonte 2020 de la UE al M.T. JMG contó con el apoyo de una beca de doctorado del Ministerio de Ciencia de España y la Fundación Severo Ochoa (PRE2022-101884). Tanto el CNIC como el CBMSO cuentan con el apoyo del Ministerio de Ciencia español, y el CNIC cuenta con el apoyo de la Fundación ProCNIC.
Materiales
| Name | Company | Catalog Number | Comments |
| #55 Forceps | Dumont | 11295-51 | |
| 12-well Clear Flat Bottom Multiwell Cell Culture Plate | BD Falcon | 353043 | |
| 35 mm vise table | Grandado | SKU 8798771617573 | |
| 40 µm Cell Strainer | Fischer Scientific | 08-771-1 | |
| 50 mL tubes | BD Falcon | 352070 | |
| 70 µm Cell Strainer | Corning | CLS431751 | |
| Anti-GFP Policlonal Antibody | Invitrogen | A10262 | 1:1000 dilution used |
| Anti-Myosin 4 (MF20) Monoclonal Antibody | Invitrogen | 14-6503-82 | 1:500 dilution used |
| CD1 Wild Type mice | Provided by Animalary Unit (CNIC) | ||
| Cleaved Caspase-3 (Asp175) Antibody | Cell Signalling Technologies | 9661 | 1:400 dilution used |
| DAPI | Cell Signalling Technologies | 4083 | 1:1000 dilution used |
| Dispase/collagenase | Roche | 10269638001 | |
| Distilled water | |||
| DMEM - Dulbecco's Modified Eagle Medium | Gibco | 10313021 | |
| Fetal Bovine Serum | Invitrogen | 10438-026 | |
| Heracell 150i CO2 Incubator | Thermo Scientific | 51032720 | |
| Leica Stereoscopic Microscope S8AP0 | Leica | 11524102 | |
| Liberase | Roche | 5401119001 | |
| Micropipette Puller Model P-97 | Sutter Instrument | SU-P-97 | |
| pCAG expression plasmid | Addgene | #89689 | |
| Penicillin-streptomycin | Invitrogen | 15070-063 | |
| Petri dishes 35 × 10 mm | BD Falcon | 351008 | |
| Petri dishes 60 × 15 mm | BD Falcon | 353002 | |
| Phenol Red | Merck | P3532 | |
| Pipette tips | Reused from old laboratory equipment | ||
| Rat Serum culture embryo, male rats SPRAGUE DAWLEY RjHan SD | Janvier Labs | 9979 | |
| Recombinant anti-Wilms Tumor Protein 1 (WT1) Antibody | Abcam | ab89901 | 1:300 dilution used |
| Square Wave Electroporator CUY21SC | Nepa Gene | CUY664-10X15 | |
| Sterile PBS | Provided and autoclaved by technical unit | ||
| Sucrose | Millipore | 84100 | |
| Tweezer electrodes with variable gap | Nepa Gene | CUY650P5 |
Referencias
- Tyser, R. C., et al. Calcium handling precedes cardiac differentiation to initiate the first heartbeat. eLife. 5, e17113(2016).
- Tyser, R. C. V., et al. Characterization of a common progenitor pool of the epicardium and myocardium. Science. 371 (6533), 2986(2021).
- Sendra, M., Domínguez, J., Torres, M., Ocaña, O. Dissecting the complexity of early heart progenitor cells. J Cardiovasc Dev Dis. 9 (1), 5(2021).
- Ivanovitch, K., Temiño, S., Torres, M. Live imaging of heart tube development in mouse reveals alternating phases of cardiac differentiation and morphogenesis. eLife. 6, e30668(2017).
- Ai, D., et al. Canonical Wnt signaling functions in second heart field to promote right ventricular growth. PNAS. 104 (22), 9319-9324 (2007).
- Zimmerman, M. S., et al. regional, and national burden of congenital heart disease, 1990-2017: A systematic analysis for the Global Burden of Disease Study 2017. Lancet Chil Adolesc Heal. 4 (3), 185-200 (2020).
- Xin, M., Olson, E. N., Bassel-Duby, R. Mending broken hearts: Cardiac development as a basis for adult heart regeneration and repair. Nat Rev Mol Cell Biol. 14 (8), 529-541 (2013).
- Porrello, E. R., et al. Transient regenerative potential of the neonatal mouse heart. Science. 331 (6020), 1078-1080 (2011).
- Cao, J., Poss, K. D. The epicardium as a hub for heart regeneration. Nat Rev Cardiol. 15 (10), 631-647 (2018).
- Zhou, B., et al. Adult mouse epicardium modulates myocardial injury by secreting paracrine factors. JCI. 121 (5), 1894-1904 (2011).
- Van Wijk, B., Gunst, Q. D., Moorman, A. F. M., Van Den Hoff, M. J. B. Cardiac regeneration from activated epicardium. PLOS One. 7 (9), e44692(2012).
- Hesse, J., et al. Single-cell transcriptomics defines heterogeneity of epicardial cells and fibroblasts within the infarcted murine heart. eLife. 10, e65921(2021).
- Streef, T. J., Smits, A. M. Epicardial contribution to the developing and injured heart: Exploring the Cellular composition of the epicardium. Front Cardiovasc Med. 8, 750243(2021).
- Sanchez-Fernandez, C., et al. Understanding epicardial cell heterogeneity during cardiogenesis and heart regeneration. J Cardiovasc Dev Dis. 10 (9), 376(2023).
- Quijada, P., et al. Coordination of endothelial cell positioning and fate specification by the epicardium. Nat Commun. 12 (1), 4155(2021).
- Mantri, M., et al. Spatiotemporal single-cell RNA sequencing of developing chicken hearts identifies interplay between cellular differentiation and morphogenesis. Nat Commun. 12 (1), 1771(2021).
- Paul, S., Zhang, X., He, J. Q. Homeobox gene Meis1 modulates cardiovascular regeneration. Semin Cell Dev Biol. 100, 52-61 (2020).
- Stankunas, K., et al. Pbx/Meis deficiencies demonstrate multigenetic origins of congenital heart disease. Circ Res. 103 (7), 702-709 (2008).
- Liu, Y., et al. Transcription factor Meis1 act as a new regulator of ischemic arrhythmias in mice. J Adv Res. 39, 275-289 (2022).
- Behringer, R. Manipulating the mouse embryo: A laboratory manual. , Cold Spring Harbor Lab Press. Cold Spring Harbor, New York. (2014).
- Wong, M. D., et al. 4D atlas of the mouse embryo for precise morphological staging. Development. 142 (20), 3583-3591 (2015).
- Morris, L., Klanke, C., Lang, S., Lim, F. Y., Crombleholme, T. TdTomato and EGFP identification in histological sections: Insight and alternatives. Biotech Histochem. 85 (6), 379-387 (2010).
- Schiaffino, S., Rossi, A. C., Smerdu, V., Leinwand, L. A., Reggiani, C. Developmental myosins: expression patterns and functional significance. Skelet. Muscle. 5 (1), 22(2015).
- Eissa, N., et al. Stability of reference genes for messenger RNA quantification by real-time pcr in mouse dextran sodium sulfate experimental colitis. PLOS One. 11 (5), e0156289(2016).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2−ΔΔCT method. Methods. 25 (4), 402-408 (2001).
- Lai, S. R., Andrews, L. G., Tollefsbol, T. O. RNA interference using a plasmid construct expressing short-hairpin RNA. Methods Mol Biol. 405, 31-37 (2007).
- Carmona, R., Barrena, S., López Gambero, A. J., Rojas, A., Muñoz-Chápuli, R. Epicardial cell lineages and the origin of the coronary endothelium. FASEB J. 34 (4), 5223-5239 (2020).
- Gittenberger-de Groot, A. C., Vrancken Peeters, M. P. F. M., Mentink, M. M. T., Gourdie, R. G., Poelmann, R. E. Epicardium-derived cells contribute a novel population to the myocardial wall and the atrioventricular cushions. Circ Res. 82 (10), 1043-1052 (1998).
- Chong, Z. X., Yeap, S. K., Ho, W. Y. Transfection types, methods and strategies: A technical review. Peer J. 9, e11165(2021).
- Kałużna, E., Nadel, A., Zimna, A., Rozwadowska, N., Kolanowski, T. Modeling the human heart ex vivo-Current possibilities and strive for future applications. JTERM. 16 (10), 853-874 (2022).
- Aguilera-Castrejon, A., et al. Ex utero mouse embryogenesis from pre-gastrulation to late organogenesis. Nature. 593 (7857), 119-124 (2021).
- Dyer, L. A., Patterson, C. A novel ex vivo culture method for the embryonic mouse heart. J Vis Exp. (75), e50359(2013).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados