Method Article
Une approche de transgenèse efficace pour l’administration de gènes dans le cœur embryonnaire de souris
Dans cet article
Résumé
Ce protocole présente un cadre méthodologique détaillé pour la transgenèse des cellules cardiaques basée sur l’électroporation dans les cœurs de souris en développement. Les ressources vidéo fournies ici faciliteront l’apprentissage de cette technique polyvalente.
Résumé
Le cœur de mammifère est un organe complexe formé au cours du développement par des populations très diverses de cellules progénitrices. L’origine, le moment du recrutement et le destin de ces géniteurs sont essentiels au bon développement de cet organe. Les mécanismes moléculaires qui régissent la morphogenèse du cœur sont essentiels pour comprendre la pathogenèse des cardiopathies congénitales et de la régénération cardiaque embryonnaire. Les approches classiques pour étudier ces mécanismes utilisaient la génération de souris transgéniques pour évaluer la fonction de gènes spécifiques au cours du développement cardiaque. Cependant, la transgenèse chez la souris est un processus complexe et long qui ne peut souvent pas être effectué pour évaluer le rôle de gènes spécifiques au cours du développement cardiaque. Pour y remédier, nous avons développé un protocole d’électroporation et de culture efficaces de cœurs embryonnaires de souris, permettant la transgenèse transitoire pour évaluer rapidement l’effet du gain ou de la perte de fonction des gènes impliqués dans le développement cardiaque. En utilisant cette méthodologie, nous avons réussi à surexprimer Meis1 dans le cœur embryonnaire, avec une préférence pour la transfection des cellules épicardiques, démontrant ainsi les capacités de la technique.
Introduction
Le cœur est le premier organe formé au cours du développement embryonnaire. Ce processus implique la coordination spatio-temporelle de diverses populations de cellules progénitrices à partir de zones distinctes de l’embryon. Tout cela se produit pendant que le cœur en développement continue de battre et de fonctionner, mettant l’accent sur la coordination remarquable nécessaire à sa formation 1,2,3. Compte tenu du rôle crucial du cœur, une régulation stricte aux niveaux cellulaire et moléculaire est essentielle à sa bonne formation 4,5. L’identification des mécanismes qui contrôlent le développement cardiaque a été d’un grand intérêt, car ils sont cruciaux pour démêler les cardiopathies congénitales, qui touchent un nombre important de patients dans le mondeentier 6. De plus, la compréhension du développement cardiaque est essentielle pour déchiffrer la régénération cardiaque, car les cœurs postnatals des mammifères conservent une capacité de régénération qui est perdue ou entravée à l’âge adulte 7,8. Par conséquent, il est impératif de disséquer les régulateurs moléculaires du développement cardiaque pour faire progresser les efforts de recherche sur les cardiopathies congénitales et la régénération cardiaque.
Dans la poursuite de cet objectif, on s’est de plus en plus intéressé à l’étude du rôle de l’épicarde dans le développement et la régénération cardiaques9. L’épicarde est une fine couche de tissu mésothélial qui comprend la couche la plus externe du cœur de mammifère (Figure 1). Des études récentes ont montré l’importance de l’épicarde lors d’une lésion cardiaque, révélant que ce tissu est capable d’envoyer des signaux de prolifération aux cardiomyocytes de la zone touchée pour atténuer les dommages10,11. Malgré l’importance de l’épicarde, son immense hétérogénéité a été difficile à mener d’autres études moléculaires. Des expériences de RNAseq sur cellule unique ont révélé l’hétérogénéité de l’épicarde, abritant plusieurs sous-populations cellulaires avec des signatures transcriptomiques distinctes 12,13,14,15,16. Ainsi, une stratégie de dépistage des régulateurs potentiels du développement cardiaque devrait tenir compte de la diversité des cellules progénitrices épicardiques.
En ce sens, la facilité de modification génétique du modèle murin a facilité l’identification de nombreux gènes cruciaux pour le développement cardiaque, permettant la génération de lignées mutantes avec gain de fonction (GOF) ou perte de fonction (LOF) de gènes spécifiques. Cependant, ces approches impliquent un investissement considérable en temps et en moyens expérimentaux ; Par conséquent, ils ne sont pas pratiques lorsqu’il s’agit d’évaluer les rôles d’un grand nombre de gènes candidats. En outre, les gènes du développement exercent souvent des fonctions pléiotropiques dans différents tissus ou sont nécessaires au développement embryonnaire précoce, ce qui entrave l’interprétation de leur contribution au développement dans un processus spécifique. Bien qu’il soit possible de cibler la fonction des gènes à des structures spécifiques ou à des moments de développement, cela nécessite généralement l’utilisation de constructions génétiques plus complexes, qui peuvent être difficiles à générer ou qui sont généralement indisponibles.
Pour surmonter ces limitations, nous présentons une méthodologie permettant d’électropoler des cœurs embryonnaires de souris pour une transgenèse transitoire (Figure 2). Associée à la culture ex vivo et au tri cellulaire activé par fluorescence (FACS), cette stratégie démontre ses capacités grâce au GOF transitoire de Meis1, un gène bien caractérisé impliqué dans le développement et la régénération cardiaques 17,18,19. Dans cet article, d’autres applications potentielles de cette méthodologie sont également explorées, et ses avantages et limites sont discutés, ainsi que comparés aux protocoles existants pour moduler transitoirement l’expression des gènes. Nous pensons que le cadre et les exemples visuels présentés amélioreront la compréhension de la biologie de l’épicarde au cours du développement et de la maladie.
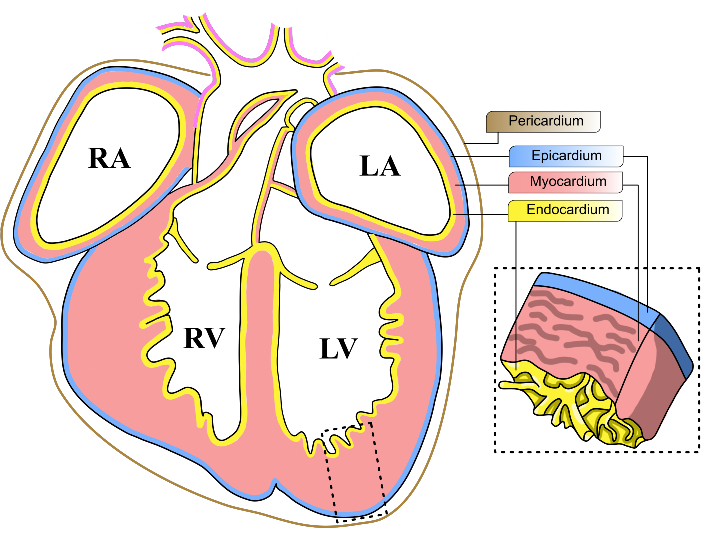
Figure 1 : Couches cardiaques embryonnaires de souris. Schéma de principe d’une vue coronale d’un cœur embryonnaire de souris E13-14. Les trois principales couches cellulaires du cœur sont représentées en jaune (endocarde), rouge (myocarde) et bleu (épicarde). Le péricarde est représenté par une ligne brune. Les quatre cavités du cœur sont abrégées en VG, ventricule gauche ; VD, ventricule droit ; LA, oreillette gauche ; RA, oreillette droite. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Vue d’ensemble schématique du protocole d’électroporation cardiaque. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocole
Toutes les procédures animales ont été approuvées par le Comité d’éthique de l’expérimentation animale du CNIC et conformes à la législation en vigueur, y compris la directive européenne 2010/63UE et la recommandation 2007/526/CE, telles qu’appliquées par la loi espagnole en vertu du décret royal 53/2013. Pour ce protocole, des souris CD-1 femelles de type sauvage âgées de 15 à 21 semaines ont été utilisées. Les détails concernant les animaux, les réactifs et l’équipement utilisés sont répertoriés dans la table des matériaux.
1. Préparation du plasmide et de l’outil
- Tout d’abord, préparez le mélange d’électroporation en ajoutant l’ADN plasmidique souhaité dans de la glace à une concentration finale de 1 μg/μL. Ensuite, complétez le mélange en ajoutant du saccharose à 6 % de concentration finale (dissous dans du PBS), un colorant biocompatible à 0,01 % et complétez avec du PBS jusqu’à un volume final de 30 μL.
REMARQUE : Environ 1 à 2 μL du mélange d’électroporation est utilisé par échantillon. Échelonnez les proportions du mélange d’électroporation en fonction du nombre d’échantillons dans l’expérience. N’oubliez pas que l’ADN pour l’électroporation doit être purifié à partir de cultures bactériennes, assurant une stabilité optimale et un minimum de contaminants. Pour plus de détails sur les plasmides utilisés pour cette étude, veuillez vous référer à la section des résultats représentatifs. - Préparez le milieu de croissance en complétant le milieu Eagle modifié (DMEM) de Dulbecco avec 10 % de sérum de veau fœtal (FBS), 2 % de tampon Hepes et 1 % de pénicilline/streptomycine. Maintenir le milieu dans un incubateur à 37 °C jusqu’à l’utilisation. De plus, réchauffez 1x PBS à 37 °C, avec le milieu de croissance.
- Préparez les aiguilles en étirant un capillaire en verre de 20 μm de diamètre à l’aide d’un extracteur de pipette dans les conditions suivantes : chaleur = 550 ; traction = 100 ; vitesse = 250 ; temps = 120. Le diamètre final après le tirage doit être d’environ 5 à 8 μm.
- Réglez les paramètres de l’électroporateur sur cinq cycles de 50 ms à 40 V avec des intervalles de 900 ms. Préparez les électrodes, la pipette à bouche et l’aiguille émoussée (Figure 3A). Équipez la pipette de l’aiguille pour charger l’ADN (Figure 3A).
2. Dissection d’embryons et extraction cardiaque
- Mettez en place un croisement entre des souris CD-1 de type sauvage. Le lendemain matin, sélectionnez les femelles qui présentent un bouchon vaginal et notez cette heure comme jour embryonnaire E0.5. Euthanasier les animaux au jour embryonnaire E12-12.5 par luxation cervicale (selon les protocoles approuvés par l’établissement).
- Extrayez soigneusement l’utérus de la souris et placez-le dans une serviette en papier sèche. Ensuite, coupez l’utérus à l’aide de ciseaux fins en insérant la pointe des ciseaux dans le côté mésométrial et en passant lentement le bord de la lame sur toute la longueur. Exposez les déciduae et transférez-les dans le milieu de croissance. Pour plus de détails, veuillez consulter les travaux de Behringer et al.20.
- Disséquez les déciduae pour extraire les embryons, puis placez les embryons dans une boîte de Pétri de 60 mm avec un milieu de croissance chaud. À l’aide de pinces fines, coupez soigneusement la queue et la tête de l’embryon de manière à ce qu’il ne reste que le tronc du corps. Le stade des embryons peut être vérifié en évaluant les caractéristiques anatomiques, comme décrit par Wong et al.21.
- Ensuite, retirez avec précaution autant de tissu que possible du tronc de l’embryon sans endommager le cœur ou les artères pulmonaires et aortales. Une fois le cœur exposé, placez-le immédiatement dans un nouveau milieu de croissance chaud et conservez-le dans un incubateur à 37 °C jusqu’à l’électroporation.
REMARQUE : Le maintien de la température cardiaque est crucial pour sa survie. Assurez-vous que le milieu est toujours chaud et remplacez-le toutes les 5 à 10 minutes. D’autre part, les échantillons peuvent être manipulés sans maintenir la stérilité, car cela n’affectera pas l’efficacité de la procédure. Veuillez noter que le milieu de croissance contient des antibiotiques.
3. Électroporation cardiaque et culture ex vivo
- Chargez environ 10 μL de mélange plasmidique (préparé à l’étape 1.1) dans l’aiguille en aspirant soigneusement avec la pipette à bouche.
- Préparez un seul cœur pour l’électroporation en le plaçant dans une boîte de Pétri propre de 60 mm avec 1x PBS stérile chauffé à 37 °C. Placez la parabole sous le microscope à dissection (Figure 3A).
- Préparez les électrodes en ajustant la distance entre les pôles positif et négatif à environ 4 mm (Figure 3B).
REMARQUE : 4 mm est la distance d’ouverture dans laquelle les cœurs de souris E12.5 s’insèrent dans les électrodes utilisées dans ce travail. Veuillez envisager de modifier cette distance en fonction de différentes tailles d’échantillons ou spécifications d’électrodes. - Percez doucement la couche la plus superficielle du cœur avec l’aiguille dans la pipette à bouche. Pipetez soigneusement 1 à 2 μL du mélange plasmidique et retirez l’aiguille. Des injections multiples peuvent être effectuées pour assurer une pénétration optimale, ainsi que pour augmenter le nombre de cellules électroporées.
- Maintenez les électrodes en place de manière à ce que le cœur soit situé entre les deux pôles (Figure 3C). Ensuite, électroporez le cœur dans les conditions spécifiées ci-dessus.
REMARQUE : Pour assurer une efficacité d’électroporation maximale, il est possible de changer la polarité des électrodes et de donner une autre impulsion. - Transférez le cœur électroporé dans une nouvelle plaque à 12 puits contenant 1 mL de milieu de croissance et placez-le à 37 °C, 5 % de CO2.
- Répétez les étapes 3.1 à 3.6 jusqu’à ce que tous les échantillons de l’expérience soient électroporés. Enfin, incubez tous les cœurs dans un incubateur à 37 °C, 5% CO2, pendant 24 h ou jusqu’à l’analyse.
REMARQUE : Dans nos mains, les cœurs peuvent être laissés en culture jusqu’à 48 h sans signes apparents de détérioration (film supplémentaire 1 ; Film supplémentaire 2 ; Figure supplémentaire 1).
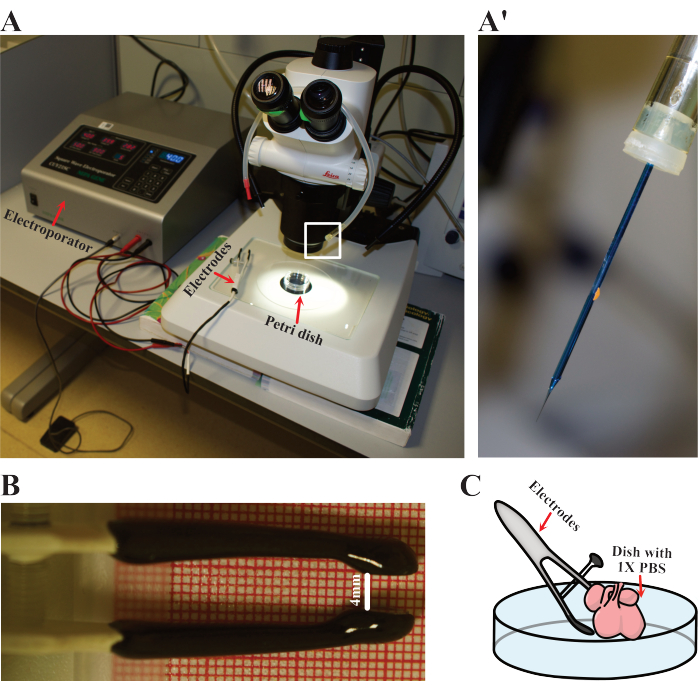
Figure 3 : Configuration de l’électroporation. (A) Configuration utilisée pour l’électroporation. Les flèches rouges indiquent l’électroporateur, les électrodes et la boîte de Pétri où les cœurs sont électroporés. (A') Détail rapproché de l’aiguille d’électroporation, comme le souligne (A) par le rectangle blanc. (B) Détail de la distance ajustée des électrodes utilisées dans les cœurs E12.5. (C) Représentation schématique de l’électroporation. Veuillez cliquer ici pour voir une version agrandie de cette figure.
4. Tri cellulaire et analyse immunohistochimique
- Pour l’analyse de tri cellulaire des échantillons, préparer un milieu de digestion pour dissocier les tissus en mélangeant 400 μL de libérase (5 mg/ml), 300 μL de dispase (0,1 g/mL), 4 μL de CaCl2(0,4 μM), 50 μL de MgCl2 (5 μM) et compléter jusqu’à 10 mL de DMEM. Garder sur la glace jusqu’à utilisation. Chaque cœur a besoin d’environ 1 ml de milieu de digestion. Ajustez les volumes en fonction du nombre d’échantillons.
REMARQUE : Pour l’immunohistochimie, fixer directement les cœurs dans 1,5 mL de PFA à 4 % pendant 2 h à l’heure de l’artérologie ou toute la nuit à 4 °C. Le lendemain, lavez les cœurs 3 fois avec du PBS et procédez aux procédures histologiques et d’immunocoloration en aval. Pour un protocole d’immunohistochimie détaillé, veuillez consulter Morris et coll.22. - Transférez chaque cœur en culture dans un nouveau tube contenant 1 mL de milieu de digestion glacé.
- Pour faciliter la dissociation, hachez le tissu en appliquant doucement le plaisir avec une seringue 3 à 4 fois et incubez immédiatement dans une plaque chauffante à 37 °C, 600 tr/min pendant 45 min.
- Filtrez le mélange de digestion à travers une passoire de 70 μm dans un nouveau tube et laissez-le se vider par gravité. Ensuite, récupérez le volume de passage et filtrez-le à nouveau à l’aide d’une crépine à cellules de 40 μm dans un nouveau tube de 50 mL.
- Ajouter 500 μL de sérum fœtal bovin (FBS) et compléter jusqu’à 30 mL avec du DMEM froid.
- Centrifuger le mélange à 240 x g pendant 10 min à température ambiante.
- Jetez le surnageant et placez le tube de 50 ml à l’envers sur une serviette en papier pour qu’il sèche complètement.
REMARQUE : Il s’agit d’un point pratique pour interrompre l’expérience en congelant la pastille de cellule. Si c’est le cas, remettre en suspension les cellules avec 10 % de DMSO dans le FBS et les stocker à -80 °C. - Pour le tri cellulaire, remettre les cellules en suspension dans 300 μL de tampon de tri (1 % FBS, 1 % pénicilline/streptomycine dans le PBS) et ajouter 0,3 μL de DAPI (1000x). Effectuez le tri des cellules.
Résultats
Pour démontrer l’efficacité de cette technique dans la réalisation d’expériences de gain de fonction (GOF) pour les régulateurs du développement cardiaque pertinents, une construction a été électroporée surexprimant le facteur de transcription Meis1. Pour ce faire, l’ARN a été extrait d’embryons E9.5, et une transcription inverse a été effectuée pour obtenir de l’ADN complémentaire (ADNc). En utilisant l’ADNc comme modèle, la séquence codante de Meis1 a été clonée (tableau supplémentaire 1) dans un plasmide d’expression pCAG (ci-après dénommé pCAG ::Meis1), tandis qu’un plasmide constitutif exprimant la GFP (pCAG ::GFP) a été utilisé comme témoin. Les cœurs ont ensuite été électroporés avec pCAG ::GFP uniquement ou en combinaison avec pCAG ::Meis1.
Après 24 h après l’électroporation (hpe), les cœurs battaient et semblaient être en bon état (figure 4A ; Film supplémentaire 1 ; Film supplémentaire 2). Pour évaluer la viabilité des cellules électroporées, l’apoptose a été analysée par coloration immunohistochimique de la caspase 3. Bien que certaines cellules positives aient été détectées dans le cœur, ni le myocarde ni l’épicarde, où se trouvent la majorité des cellules électroporées, ne se sont avérés être affectés par l’apoptose (Figure supplémentaire 1C). Lorsque l’activité de fluorescence a été évaluée, un signal GFP mosaïque a été observé dans presque toutes les quatre cavités du cœur, indiquant que ce protocole est capable d’atteindre la majorité des structures cardiaques (Figure 4A). Pour déterminer quels types de cellules cardiaques ont été électroporés, les cœurs électroporés par pCAG ::GFP ont été immunomarqués contre les marqueurs de l’épicarde, WT12, et du myocarde, MF2023, puis imagés à l’aide d’un microscope confocal (Figure 4B). La plupart des cellules GFP+ étaient préférentiellement situées dans la région externe du cœur, ce qui correspondait en grande partie au signal WT1, indiquant que cette méthode est la plus efficace pour atteindre les cellules épicardiques (Figure 4B, pointes de flèches blanches).
D’autre part, les cœurs électroporés ou témoins (pCAG ::GFP uniquement) ont été dissociés, et les cellules ont été soumises à un tri cellulaire activé par fluorescence (FACS) pour isoler les cellules GFP+ de l’organe entier, comme décrit à l’étape 4 du protocole. L’ARN a été extrait des cellules triées, et une PCR quantitative par transcription inverse (RT-qPCR) a été réalisée pour évaluer les niveaux d’expression de Meis1 dans les cellules transfectées (Figure 4C, Tableau supplémentaire 1). L’expression totale du gène Meis1 a été normalisée à l’aide des niveaux d’expression du gène d’entretien domestique de la souris Eef224, et l’expression relative des gènes a été calculée à l’aide de la méthode ΔΔCt25. Une régulation positive significative des niveaux d’ARN Meis1 a été observée dans les cœurs électroporés avec le plasmide pCAG ::Meis1 par rapport à ceux avec uniquement le plasmide pCAG ::GFP (Figure 4C). Ainsi, cela démontre le potentiel de la technique à surexprimer les gènes d’intérêt et à traiter le résultat moléculaire.

Figure 4 : Analyse moléculaire de cœurs électroporés E12.5. (A) Fluorescence GFP d’un cœur électroporé CAG ::GFP 24 h après l’électroporation. L’image du bas montre la fluorescence de la GFP, tandis que l’image du haut montre une fusion de la GFP et d’un champ lumineux du même cœur. (B) Immunofluorescence de coupes histologiques cardiaques électroporées E12.5. MF20 marque les cardiomyocytes, tandis que WT1 marque les cellules épicardiques. Les images de droite correspondent aux zooms du rectangle blanc. Les pointes de flèches blanches indiquent les cellules GFP+ situées dans l’épicarde, grossièrement délimitées par une ligne blanche pointillée. (C) Analyse RT-qPCR de cœurs électroporés avec pCAG ::Meis1 ou pCAG ::GFP (contrôle). Chaque point représente une réplique biologique obtenue à partir de la mise en commun de trois cœurs. Au total, l’analyse qPCR a été réalisée en trois exemplaires avec trois réplicats biologiques distincts. L’analyse statistique a été effectuée à l’aide du test t de Student (*P < 0,05). Barres d’échelle : 200 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Figure supplémentaire 1 : Efficacité de l’électroporation cardiaque et viabilité cellulaire. (A) Expression de la GFP dans deux cœurs E12.5 distincts électroporés avec le plasmide témoin pCAG ::GFP à 24 h ou 48 h après l’électroporation (hpe). L’image du signal GFP a été superposée à une image en champ clair du même cœur. (B) Quantification des cellules GFP+ dans des cœurs de 24 hpe et 48 hpe électroporés avec contrôle pCAG ::GFP. Les points indiquent des échantillons distincts ; Les lignes pleines indiquent la moyenne, tandis que les barres d’erreur indiquent l’écart-type. Aucune différence significative n’a été observée entre les deux conditions ( test t non paramétrique ; P > 0,9). (C) Image confocale d’un cœur E12.5 électroporé avec l’immunomarquage pCAG ::GFP contre la caspase clivée 3 et MF20. La ligne pointillée représente la couche myocardique du cœur. Les images de droite sont zoomées à partir du rectangle blanc. Barres d’échelle : 100 μm. Veuillez cliquer ici pour télécharger ce fichier.
Tableau supplémentaire 1 : Résumé et séquences de l’abécédaire. Veuillez cliquer ici pour télécharger ce fichier.
Vidéo supplémentaire 1 : Cœur électroporé pCAG ::GFP après 24 h. Capture vidéo d’un cœur électroporé avec le plasmide de contrôle pCAG ::GFP 24 h après l’intervention. Le cœur continue de battre et les cellules électroporées expriment des signaux GFP. Veuillez cliquer ici pour télécharger ce film.
Vidéo supplémentaire 2 : Cœur électroporé pCAG ::GFP après 48 h. Vidéo montrant un cœur électroporé avec contrôle pCAG ::GFP, 48 h après l’électroporation. Veuillez cliquer ici pour télécharger ce film.
Discussion
Dans l’ensemble, la méthodologie décrite ici offre un cadre robuste pour l’expression des constructions transgéniques dans l’épicarde en développement (Figure 4B), comme le démontre la surexpression de Meis1 (Figure 4C). Avec les constructions appropriées, ce protocole peut être utilisé pour évaluer transitoirement l’impact du gain de fonction (GOF) ou de la perte de fonction (LOF) d’un gène spécifique. Le LOF peut être implémenté dans la technique en transfectant un plasmide ciblant un gène candidat par interférence ARN26. L’évaluation rapide du gène GOF ou LOF est particulièrement avantageuse, surtout si l’on considère la grande hétérogénéité transcriptomique des cellules épicardiques12, ce qui rend peu pratique la génération d’animaux transgéniques pour évaluer chaque gène candidat individuellement.
Parce que ce protocole utilise la culture ex vivo, il offre la possibilité d’étudier le devenir des cellules épicardiques dans une fenêtre temporelle. L’épicarde en développement joue un rôle central dans la formation de régions distinctes du cœur adulte 13,27,28 ; Ainsi, la compréhension de ses contributions au développement cardiaque est essentielle pour disséquer la base des malformations cardiaques congénitales. Bien que cet article ne se penche pas sur cette application, les directives décrites dans le protocole pourraient potentiellement être utiles pour les futures études de cartographie du destin de l’épicarde
Malgré l’utilité pratique de la méthode actuelle, certaines limites doivent être reconnues. Étant donné que cette méthodologie repose sur la transgénèse transitoire, l’expression plasmidique peut être perdue ou diminuée au fil du temps, ce qui limite la portée du protocole pour étudier une fenêtre de développement spécifique29. Cependant, dans cette étude, la surexpression de la GFP s’est maintenue pendant toute la durée des cultures post-électroporation (48 hpe). De plus, les limites potentielles des technologies actuelles de culture ex vivo entravent l’applicabilité de la méthode pour évaluer les stades tardifs du développement embryonnaire ou postnatal, en particulier lorsque la culture ex vivo après électroporation devient difficile30. Bien qu’il existe des méthodes plus sophistiquées de culture embryonnaire ex vivo 31,32, elles peuvent nécessiter l’utilisation d’un équipement moins accessible aux chercheurs et ne pas garantir la survie de l’échantillon.
Bien que le protocole présenté ici soit conçu pour être aussi simple que possible afin d’améliorer la reproductibilité, il comprend des étapes essentielles qui sont essentielles au succès. Tout d’abord, la dissection d’embryons doit être effectuée avec soin et rapidité pour minimiser les dommages cardiaques, qui pourraient compromettre la survie de l’organe après l’électroporation. Une attention particulière lors de cette étape, ainsi que l’utilisation d’un équipement de dissection optimal, sont fortement recommandées. Un autre aspect critique du protocole est la réalisation de l’électroporation elle-même, car une injection inadéquate de plasmide peut entraîner des résultats indésirables. Par conséquent, il est conseillé d’effectuer plusieurs injections sur un seul cœur, et l’injection de l’échantillon de manière plutôt superficielle pourrait être bénéfique pour améliorer la survie des organes. Enfin, la dissociation cellulaire pendant le traitement cardiaque doit être rigoureusement effectuée, en respectant les temps prévus dans le protocole pour maximiser la viabilité cellulaire.
En conclusion, l’identification des gènes clés dans la formation du cœur est essentielle pour comprendre les mécanismes sous-jacents des maladies cardiaques congénitales et de la régénération. La méthodologie établie ici permet un dépistage rapide des effets de gain de fonction (GOF) et de perte de fonction (LOF) dans le cœur en développement. Tout en reconnaissant certaines limites, cette méthode présente un potentiel considérable d’extrapolation à différents stades de développement ou de cartographie du devenir des cellules épicardiques embryonnaires. À travers les directives visuelles fournies dans cet article, nous visons à offrir une boîte à outils précieuse pour évaluer l’immense complexité et le potentiel de l’épicarde en développement.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Cette étude a été financée par la subvention RTI2018-097617-J-I00 du Ministerio de Ciencia e Innovación espagnol et l’Acción 9 de l’Universidad de Jaén à l’O.H.O., la subvention PGC2018-096486-B-I00 du Ministerio de Ciencia e Innovación espagnol et la subvention H2020-MSCA-ITN-2016-722427 du programme Horizon 2020 de l’UE à M.T. JMG a été soutenue par une bourse de doctorat du ministère espagnol des Sciences et de la Fundación Severo Ochoa (PRE2022-101884). Le CNIC et le CBMSO sont tous deux soutenus par le ministère espagnol des Sciences, et le CNIC est soutenu par la Fondation ProCNIC.
matériels
| Name | Company | Catalog Number | Comments |
| #55 Forceps | Dumont | 11295-51 | |
| 12-well Clear Flat Bottom Multiwell Cell Culture Plate | BD Falcon | 353043 | |
| 35 mm vise table | Grandado | SKU 8798771617573 | |
| 40 µm Cell Strainer | Fischer Scientific | 08-771-1 | |
| 50 mL tubes | BD Falcon | 352070 | |
| 70 µm Cell Strainer | Corning | CLS431751 | |
| Anti-GFP Policlonal Antibody | Invitrogen | A10262 | 1:1000 dilution used |
| Anti-Myosin 4 (MF20) Monoclonal Antibody | Invitrogen | 14-6503-82 | 1:500 dilution used |
| CD1 Wild Type mice | Provided by Animalary Unit (CNIC) | ||
| Cleaved Caspase-3 (Asp175) Antibody | Cell Signalling Technologies | 9661 | 1:400 dilution used |
| DAPI | Cell Signalling Technologies | 4083 | 1:1000 dilution used |
| Dispase/collagenase | Roche | 10269638001 | |
| Distilled water | |||
| DMEM - Dulbecco's Modified Eagle Medium | Gibco | 10313021 | |
| Fetal Bovine Serum | Invitrogen | 10438-026 | |
| Heracell 150i CO2 Incubator | Thermo Scientific | 51032720 | |
| Leica Stereoscopic Microscope S8AP0 | Leica | 11524102 | |
| Liberase | Roche | 5401119001 | |
| Micropipette Puller Model P-97 | Sutter Instrument | SU-P-97 | |
| pCAG expression plasmid | Addgene | #89689 | |
| Penicillin-streptomycin | Invitrogen | 15070-063 | |
| Petri dishes 35 × 10 mm | BD Falcon | 351008 | |
| Petri dishes 60 × 15 mm | BD Falcon | 353002 | |
| Phenol Red | Merck | P3532 | |
| Pipette tips | Reused from old laboratory equipment | ||
| Rat Serum culture embryo, male rats SPRAGUE DAWLEY RjHan SD | Janvier Labs | 9979 | |
| Recombinant anti-Wilms Tumor Protein 1 (WT1) Antibody | Abcam | ab89901 | 1:300 dilution used |
| Square Wave Electroporator CUY21SC | Nepa Gene | CUY664-10X15 | |
| Sterile PBS | Provided and autoclaved by technical unit | ||
| Sucrose | Millipore | 84100 | |
| Tweezer electrodes with variable gap | Nepa Gene | CUY650P5 |
Références
- Tyser, R. C., et al. Calcium handling precedes cardiac differentiation to initiate the first heartbeat. eLife. 5, e17113(2016).
- Tyser, R. C. V., et al. Characterization of a common progenitor pool of the epicardium and myocardium. Science. 371 (6533), 2986(2021).
- Sendra, M., Domínguez, J., Torres, M., Ocaña, O. Dissecting the complexity of early heart progenitor cells. J Cardiovasc Dev Dis. 9 (1), 5(2021).
- Ivanovitch, K., Temiño, S., Torres, M. Live imaging of heart tube development in mouse reveals alternating phases of cardiac differentiation and morphogenesis. eLife. 6, e30668(2017).
- Ai, D., et al. Canonical Wnt signaling functions in second heart field to promote right ventricular growth. PNAS. 104 (22), 9319-9324 (2007).
- Zimmerman, M. S., et al. regional, and national burden of congenital heart disease, 1990-2017: A systematic analysis for the Global Burden of Disease Study 2017. Lancet Chil Adolesc Heal. 4 (3), 185-200 (2020).
- Xin, M., Olson, E. N., Bassel-Duby, R. Mending broken hearts: Cardiac development as a basis for adult heart regeneration and repair. Nat Rev Mol Cell Biol. 14 (8), 529-541 (2013).
- Porrello, E. R., et al. Transient regenerative potential of the neonatal mouse heart. Science. 331 (6020), 1078-1080 (2011).
- Cao, J., Poss, K. D. The epicardium as a hub for heart regeneration. Nat Rev Cardiol. 15 (10), 631-647 (2018).
- Zhou, B., et al. Adult mouse epicardium modulates myocardial injury by secreting paracrine factors. JCI. 121 (5), 1894-1904 (2011).
- Van Wijk, B., Gunst, Q. D., Moorman, A. F. M., Van Den Hoff, M. J. B. Cardiac regeneration from activated epicardium. PLOS One. 7 (9), e44692(2012).
- Hesse, J., et al. Single-cell transcriptomics defines heterogeneity of epicardial cells and fibroblasts within the infarcted murine heart. eLife. 10, e65921(2021).
- Streef, T. J., Smits, A. M. Epicardial contribution to the developing and injured heart: Exploring the Cellular composition of the epicardium. Front Cardiovasc Med. 8, 750243(2021).
- Sanchez-Fernandez, C., et al. Understanding epicardial cell heterogeneity during cardiogenesis and heart regeneration. J Cardiovasc Dev Dis. 10 (9), 376(2023).
- Quijada, P., et al. Coordination of endothelial cell positioning and fate specification by the epicardium. Nat Commun. 12 (1), 4155(2021).
- Mantri, M., et al. Spatiotemporal single-cell RNA sequencing of developing chicken hearts identifies interplay between cellular differentiation and morphogenesis. Nat Commun. 12 (1), 1771(2021).
- Paul, S., Zhang, X., He, J. Q. Homeobox gene Meis1 modulates cardiovascular regeneration. Semin Cell Dev Biol. 100, 52-61 (2020).
- Stankunas, K., et al. Pbx/Meis deficiencies demonstrate multigenetic origins of congenital heart disease. Circ Res. 103 (7), 702-709 (2008).
- Liu, Y., et al. Transcription factor Meis1 act as a new regulator of ischemic arrhythmias in mice. J Adv Res. 39, 275-289 (2022).
- Behringer, R. Manipulating the mouse embryo: A laboratory manual. , Cold Spring Harbor Lab Press. Cold Spring Harbor, New York. (2014).
- Wong, M. D., et al. 4D atlas of the mouse embryo for precise morphological staging. Development. 142 (20), 3583-3591 (2015).
- Morris, L., Klanke, C., Lang, S., Lim, F. Y., Crombleholme, T. TdTomato and EGFP identification in histological sections: Insight and alternatives. Biotech Histochem. 85 (6), 379-387 (2010).
- Schiaffino, S., Rossi, A. C., Smerdu, V., Leinwand, L. A., Reggiani, C. Developmental myosins: expression patterns and functional significance. Skelet. Muscle. 5 (1), 22(2015).
- Eissa, N., et al. Stability of reference genes for messenger RNA quantification by real-time pcr in mouse dextran sodium sulfate experimental colitis. PLOS One. 11 (5), e0156289(2016).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2−ΔΔCT method. Methods. 25 (4), 402-408 (2001).
- Lai, S. R., Andrews, L. G., Tollefsbol, T. O. RNA interference using a plasmid construct expressing short-hairpin RNA. Methods Mol Biol. 405, 31-37 (2007).
- Carmona, R., Barrena, S., López Gambero, A. J., Rojas, A., Muñoz-Chápuli, R. Epicardial cell lineages and the origin of the coronary endothelium. FASEB J. 34 (4), 5223-5239 (2020).
- Gittenberger-de Groot, A. C., Vrancken Peeters, M. P. F. M., Mentink, M. M. T., Gourdie, R. G., Poelmann, R. E. Epicardium-derived cells contribute a novel population to the myocardial wall and the atrioventricular cushions. Circ Res. 82 (10), 1043-1052 (1998).
- Chong, Z. X., Yeap, S. K., Ho, W. Y. Transfection types, methods and strategies: A technical review. Peer J. 9, e11165(2021).
- Kałużna, E., Nadel, A., Zimna, A., Rozwadowska, N., Kolanowski, T. Modeling the human heart ex vivo-Current possibilities and strive for future applications. JTERM. 16 (10), 853-874 (2022).
- Aguilera-Castrejon, A., et al. Ex utero mouse embryogenesis from pre-gastrulation to late organogenesis. Nature. 593 (7857), 119-124 (2021).
- Dyer, L. A., Patterson, C. A novel ex vivo culture method for the embryonic mouse heart. J Vis Exp. (75), e50359(2013).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon