Method Article
Utilizzo dello strumento Minigene E1A per studiare le modifiche allo splicing dell'mRNA
In questo articolo
Riepilogo
Questo protocollo presenta uno strumento rapido e utile per valutare il ruolo di una proteina con funzione insolita nella regolazione dello splicing alternativo dopo il trattamento chemioterapico.
Abstract
L'elaborazione dell'mRNA prevede più passaggi simultanei per preparare l'mRNA per la traduzione, come 5'capping, aggiunta poli-A e splicing. Oltre allo splicing costitutivo, lo splicing alternativo dell'mRNA consente l'espressione di proteine multifunzionali da un unico gene. Poiché gli studi interactome sono generalmente la prima analisi per proteine nuove o sconosciute, l'associazione della proteina esca con fattori di splicing è un'indicazione che può partecipare al processo di splicing dell'mRNA, ma determinare in quale contesto o quali geni sono regolati è un processo empirico. Un buon punto di partenza per valutare questa funzione è l'utilizzo dello strumento minigene classico. Qui presentiamo l'uso del minigene Adenovirale E1A per valutare i cambiamenti di splicing alternativi dopo diversi stimoli di stress cellulare. Abbiamo valutato lo splicing del minigene E1A nella proteina Nek4 stabilmente sovraespressiva di HEK293 dopo diversi trattamenti stressanti. Il protocollo include la trasfezione minigene E1A, il trattamento cellulare, l'estrazione dell'RNA e la sintesi cDNA, seguita dall'analisi PCR e gel e dalla quantificazione delle varianti spliced E1A. L'uso di questo metodo semplice e consolidato combinato con trattamenti specifici è un punto di partenza affidabile per far luce sui processi cellulari o su quali geni possono essere regolati dallo splicing dell'mRNA.
Introduzione
Lo splicing è tra i passaggi più importanti nell'elaborazione dell'mRNA eucariotico che avviene simultaneamente alla tappatura a 5 mRNA e alla poliadenylazione di 3'mRNA, che comprende la rimozione dell'introne seguita dalla giunzione dell'esonero. Il riconoscimento dei siti di giunzione (SS) da parte dello spliceosoma, un complesso di ribonucleoproteine contenente piccole ribonucleoproteine (snRNP U1, U2, U4 e U6), piccoli RRNA (snRNA) e diverse proteineregolatorie 1 è necessario per la giunzione.
Oltre alla rimozione dell'introne (splicing costitutivo), negli eucarioti, gli introni possono essere conservati e gli esoni possono essere esclusi, configurando il processo chiamato mRNA alternative splicing (AS). Lo splicing alternativo pre-mRNA espande la capacità di codifica dei genomi eucarioti permettendo la produzione di un numero grande e diversificato di proteine da un numero relativamente piccolo di geni. Si stima che il 95-100% degli mRNA umani che contengono più di un esone possano subire giunzione alternativa2,3. Questo è fondamentale per processi biologici come lo sviluppo neuronale, l'attivazione dell'apoptosi e la risposta allo stress cellulare4, fornendo all'organismo alternative per regolare il funzionamento cellulare utilizzando lo stesso repertorio di geni.
La macchina necessaria per lo splicing alternativo è la stessa utilizzata per lo splicing costitutivo e l'uso delle SS è il principale fattore determinante per l'occorrenza di giunzione alternativa. Lo splicing costitutivo è correlato all'uso di forti siti di giunzione, che di solito sono più simili ai motivi di consenso per il riconoscimento spliceosoma5.
Gli esoni alternativi sono tipicamente riconosciuti in modo meno efficiente rispetto agli esoni costitutivi una volta che i suoi elementi regolatori cis-, le sequenze nelle SS di 5 e 3 che fiancheggiano questi esoni, mostrano una capacità di legame inferiore allo spliceosoma. mRNA contiene anche regioni denominate esaltatori o silenziatori situati in esoni (esaltatori di splicing esonici (ESE) e silenziatori di giunzione esonici (ESS)) e introni (esaltatori di giunzione intronica (ISE) e silenziatori di giunzione intronica (ISSS)) che migliorano o reprimeno l'uso dell'esonero,rispettivamente 5. Queste sequenze sono riconosciute da elementi transre normativi o fattori di splicing (SF). Le SFs sono rappresentate principalmente da due famiglie di proteine, i fattori di splicing ricchi di serina/arginina (SRSF) che si legano alle ESE e alla famiglia delle ribonucleoproteine nucleari eterogenee (hnRNP) che si legano alle sequenze ESS5.
Lo splicing alternativo può essere modulato mediante fosforilazione/ defosforilazione dei trans- fattori che modificano i partner delle interazioni e localizzazione cellulare dei fattori di giunzione6,7,8. L'identificazione di nuovi regolatori dei fattori di splicing può fornire nuovi strumenti per regolare lo splicing e, di conseguenza, alcuni trattamenti contro il cancro.
9, in un profilo di espressione genica del microarray di mRNA, ha osservato cambiamenti consistenti nei livelli di componenti spliceosomiali in 101 linee cellulari e dopo diverse condizioni di stress (farmaci a base di platino, irradiazione gamma, inibitori della topoisomerasi, inibitori della tirosina chinasi e taxani). La relazione tra il modello di giunzione e l'efficacia della chemioterapia è già stata dimostrata nelle cellule tumorali polmonari, che sono resistenti alla chemioterapia, mostrando cambiamenti nel tasso di varianti della caspasi-910. Le cellule HEK293 trattate con pannello chemioterapici mostrano cambiamenti nello splicing con un aumento delle varianti pro-apoptotiche. Gabriel et al.11 ha osservato cambiamenti in almeno 700 eventi di splicing dopo il trattamento con cisplatino in diverse linee cellulari, sottolineando che le vie di splicing sono influenzate dal cisplatino. I modulatori di giunzione hanno già dimostrato attività antitumorale, dimostrando che lo splicing è importante per lo sviluppo tumorale e, principalmente, la risposta chemioterapica12. Pertanto, caratterizzare nuove proteine che regolano lo splicing dopo agenti stressanti cellulari, come la chemioterapica, è molto importante per scoprire nuove strategie di trattamento.
Gli indizi di regolazione alternativa dello splicing dagli studi interactome, particolarmente importanti per caratterizzare funzioni di proteine nuove o non simple, possono richiedere un approccio più generale e semplice per verificare il ruolo reale della proteina in AS. I minigeni sono strumenti importanti per l'analisi del ruolo generale di una proteina che influisce sulla regolazione dello splicing. Contengono segmenti di un gene di interesse contenente regioni genomiche alternativamente spliced e affiancate13. L'utilizzo di uno strumento minigene consente l'analisi dello splicing in vivo con diversi vantaggi come la lunghezza del minigene che è minore e quindi non è una limitazione alla reazione di amplificazione; lo stesso minigene può essere valutato in diverse linee cellulari; tutti i componenti cellulari, principalmente la loro regolazione della modifica post-traslazionale (fosforilazione e cambiamenti nei compartimenti cellulari) sono presenti e possono essereaffrontati 13,14. Inoltre, i cambiamenti nel modello di splicing alternativo possono essere osservati dopo lo stress cellulare e, l'uso di un sistema minigene, consentono di identificare il percorso modulato da diversi stimoli.
Esistono diversi sistemi minigeni già descritti che sono specifici per diversi tipi di eventi di splicing13,14, tuttavia, come saggio preliminare, il minigene E1A15 è un sistema di reporter alternativo molto consolidato per lo studio della selezione delle SS 5 in vivo. Da un solo gene, E1A, cinque mRNA sono prodotti da splicing alternativo basato sulla selezione di tre diversi siti di giunzione 5 ′ e di uno o un sito di giunzione 3 ′ maggiore ominore 16,17,18. L'espressione delle varianti E1A cambia in base al periodo di infezione adenovirale19,20.
Abbiamo dimostrato in precedenza che entrambe le isoforme Nek4 interagiscono con fattori di giunzione come SRSF1 e hnRNPA1 e mentre isoforma 2 cambia minigene E1A splicing alternativo, isoforma 1 non ha alcun effetto inquel 21. Poiché l'isoforma 1 è l'isoforma più abbondante e cambia la resistenza alla chemioterapia e la risposta ai danni al DNA, valutiamo se potrebbe cambiare il minigene E1A alternativo in una condizione di stress.
Il test minigene è un metodo semplice, a basso costo e rapido, poiché ha solo bisogno di estrazione di RNA, sintesi cDNA, amplificazione e analisi del gel di agarosio, e può essere uno strumento utile per valutare poiché un possibile effetto sullo splicing alternativo da parte di una proteina di interessi fino all'effetto di diversi trattamenti sul modello di splicing alternativo cellulare.
Protocollo
1. Cellule placcanti
NOTA: In questo protocollo descritto, le linee cellulari stabili HEK293, precedentemente generate per l'espressione induttibile stabile di Nek4, sono stateutilizzate 21, tuttavia, lo stesso protocollo è adatto a molte altre linee cellulari, come HEK29322, HeLa23,24,25,26, U-2 OS27, COS728, SH-SY5Y29. Il modello di espressione delle isoforme minigene E1A in condizioni basali varia tra queste cellule e deve essere caratterizzato per ogni condizione. Questo protocollo non è limitato alle linee cellulari stabili. L'approccio più comune nella valutazione della proteina candidata è la co-trasfezione transitoria di aumentare la sua quantità con una quantità fissa del minigene. Lo stesso protocollo è adatto per le celle ad eliminazione diretta.
- Celle a piastre considerando i controlli del veicolo, non trasfettato e della GFP.
- Cellule DI coltura HEK293 con espressione stabile del gene di interesse nel Mezzo aquila modificato (DMEM) di Dulbecco integrato con il 10% del siero bovino fetale (FBS), 4,5 g/L glucosio, 4 mM L-glutammina e mantenere con 100 μg/mL di igromicina B, su piastre trattate con coltura tissutale a 37 °C nel 5% di CO2 e un'atmosfera umidificata.
- Dividere le celle utilizzando lo 0,25% di tripside-EDTA. Piastra 3 x 105 cellule in piastre a 6 pozzi e incubarle per 24 ore a 37 °C nel 5% di CO2.
NOTA: Per la trasfezione, le cellule devono essere confluenti al 70-80% per ridurre la morte cellulare dopo la trasfezione.
2. Trasfezione cellulare
NOTA: Qui è stato utilizzato 1-2 μg di pMTE1A minigene plasmide, tuttavia la quantità di DNA, così come il tempo di espressione, deve essere mantenuta al minimo per evitare tossicità. Ad esempio, è stata osservata un'elevata tossicità nelle cellule HeLa dopo 30 ore di trasfezione con 1 μg di DNA pMTE1A. Per la trasfezione qui descritta, è stato utilizzato un reagente di trasfezione a base lipidica
- Controllare la confluenza cellulare 24 ore dopo la placcatura e trasfetto le celle stabili HEK293 solo quando il 70-80% confluente.
- Rimuovere attentamente il mezzo di coltura cellulare con una pipetta, invece di utilizzare un sistema di pompa per vuoto. Quindi aggiungere con cura 2 mL di mezzo DMEM completo senza antibiotici e rimettere la piastra nell'incubatrice.
- Preparare un tubo con il tampone di trasfezione (200 μL/porcile), aggiungere 2 μg di DNA, vortice di pMTE1A e quindi aggiungere 2 μL di reagente di trasfezione. Vortice di nuovo e incubare per 10 minuti a temperatura ambiente.
- Rimuovere le piastre dall'incubatore e aggiungere con cura la miscela di trasfezione dropwise.
- Alle cellule stabili HEK293 aggiungere la tetraciclina (0,5 μg/mL) per l'induzione dell'espressione Nek4 6 h dopo la trasfezione. Il cambiamento medio non è necessario.
NOTA: I volumi/importi descritti in questa sezione sono per un pozzo. Preparare la miscela per tutti i pozzi utilizzati nell'esperimento nello stesso tubo. - Preparare un pozzo per trasfetto con EGFP, o un altro fluoroforo che esprime plasmide per stimare l'efficienza di trasfezione. Risultati migliori possono essere osservati con un'efficienza di trasfezione di almeno il 40%, ma in precedenza sono state osservate buone prestazioni con minore efficienza di trasfezione.
- Quando si utilizza la cotrasfezione (proteina di interesse e minigene) mantenere un pozzo con cellule non trasfette per evitare di ottenere risultati dall'mRNA endogeno. Nel caso dell'E1A, le cellule HEK293 esprimono già il gene E1A30.
3. Preparare i farmaci
NOTA: Il tempo e la concentrazione del trattamento sono stati scelti in base ai risultati della letteratura, che indicanoe i cambiamenti nello splicing alternativo per alcuni geni.
- Eseguire una curva dose-risposta prima di iniziare il saggio per determinare la concentrazione minima all'induzione di giunzione alternativa senza alcun effetto sulla vitalità cellulare.
- Preparare una soluzione di calcio Paclitaxel a concentrazione di 5 mM in etanolo. La concentrazione finale è di 1 μM. Conservare a -20 °C. Utilizzare lo 0,02% di etanolo come controllo del veicolo.
- Preparare una soluzione di cisplatino diluindo in 0,9% NaCl a circa 0,5 mg/mL (1,66 mM). Proteggere da luce, vortice e incubare in un bagno termale, 37 °C per 30 min. La concentrazione finale è di 30 μM. Preparare fresco o conservare a 2-10 °C per un mese.
NOTA: Tutti i farmaci devono essere protetti dalla luce.
4. Trattamento e raccolta cellulare
NOTA: le cellule stabili HEK293 sono state raccolte 48 ore dopo la trasfezione e per questo sono state trattate 24 ore dopo la trasfezione perché il più alto livello di espressione Nek4 si ottiene entro 48 h. Tuttavia, in questo momento sono stati osservati alti livelli di espressione isoforma 13S (fino al 90%). Per diminuire la proporzione di isoforma 13S, provare a trattare e raccogliere le cellule 30 ore dopo il massimo della trasfezione.
- Utilizzare punte e tubi senza RNA/DNasi. Eseguire l'estrazione totale dell'RNA con reagente fenolo-cloroformio su raccomandazione del produttore.
- 24 ore dopo la trasfezione, verificare la morfologia cellulare e l'efficienza di trasfezione utilizzando un microscopio fluorescente.
- Rimuovere il mezzo di coltura cellulare, utilizzando preferenzialmente una pipetta anziché un sistema di pompaggio per vuoto. Quindi aggiungere il mezzo di coltura cellulare con la chemioterapica nella concentrazione finale precedentemente descritta.
- Incubare a 37 °C, 5% CO2 per 18 - 24 ore.
- Raccogliere l'RNA scartando il mezzo di coltura cellulare in un contenitore e aggiungendo 0,5 - 1 mL di reagente di estrazione dell'RNA direttamente al pozzo. Se i pozzi sono molto confluenti, utilizzare 1 mL di reagente di estrazione dell'RNA per migliorare la qualità dell'RNA.
- Omogeneizzare con la pipetta e trasferirlo in un tubo di centrifuga da 1,5 ml. A questo punto, il protocollo può essere messo in pausa memorizzando campioni a -80 °C o procedere immediatamente all'estrazione dell'RNA.
AVVERTENZA: Il reagente di estrazione dell'RNA a base di fenolo è tossico e tutte le procedure devono essere eseguite in una cappa chimica dei fumi e i residui vengono smaltiti correttamente.
5. Estrazione dell'RNA e sintesi del cDNA
- In una cappa aspirante scongelare i campioni e incubare per 5 minuti a temperatura ambiente. Aggiungere 0,1 -0,2 mL di cloroformio e agitare vigorosamente.
- Incubare per 3 minuti a temperatura ambiente.
- Centrifuga per 15 min a 12.000 x g e 4 °C.
- Raccogliere la fase acquosa superiore e trasferirlo in un nuovo tubo di centrifuga da 1,5 ml. Raccogliere circa il 60% del volume totale; tuttavia, non raccogliere il DNA o la fase organica (inferiore).
- Aggiungere 0,25 - 0,5 mL di isopropanolo e agitare per inversione 4 volte. Incubare per 10 minuti a temperatura ambiente.
- Centrifuga a 12.000 x g per 10 min, a 4 °C e scarta il supernatante.
- Lavare il pellet di RNA due volte con etanolo (75% in acqua trattata con dietilpirrocarbonato (DEPC). Centrifugare a 7.500 x g per 5 minuti e scartare l'etanolo.
- Rimuovere l'etanolo in eccesso invertendo il tubo su una carta assorbente e quindi lasciare il tubo aperto all'interno di una cappa aspirante per asciugare parzialmente il pellet per 5 -10 minuti.
- Rimospò il pellet di RNA in 15 μL di acqua trattata con DEPC.
- Quantificare l'RNA totale utilizzando l'assorbanza a 230 nm, 260 nm e 280 nm per verificare la qualità dell'RNA.
- Per verificare la qualità totale dell'RNA, eseguire un gel di agarosio dell'1% pretrattato con l'1,2% (v/v) di una soluzione di ipoclorito di sodio al 2,5% per 30 min31.
- Eseguire la sintesi di cDNA utilizzando 1-2 μg di RNA totale.
- Pipetta RNA, 1 μL di oligo-dT (50 μM), 1 μL di dNTP (10 mM) e compongono il volume a 12 μL con acqua priva di nucleasi. Incubare nel termociclo per 5 minuti a 65 °C.
- Rimuovere i campioni dal termociclo per raffreddarsi e preparare la miscela di reazione: 4 μL di tampone di transcriptasi inversa, 2 μL di ditiothreitol (DTT) e 1 μL di inibitore della ribonucleasi. Incubare a 37 °C per 2 min.
- Aggiungere 1 μL di trascrittasi inversa termo stabile. Incubare a 37 °C per 50 min e inattivare l'enzima a 70 °C per 15 min.
NOTA: Il cDNA può essere conservato a -20 °C per diverse settimane. Eseguire un controllo della transcriptasi non inversa (NRT) per la contaminazione genomica da discriminazione degli eventi di ritenzione di introno putativo.
6. pMTE1A minigene PCR
- Eseguire PCR con la composizione di reazione (1,5 mM di MgCl2, 0,3 mM di miscela dNTP, 0,5 μM di ogni primer, 2,5 U di una Taq Polimerasi ad avvio caldo e 150 ng di cDNA) con le seguenti condizioni:
94 °C per 2 min, 29 cicli di: 94 °C per 1 min, 50 °C per 2 min, 72 °C per 2 min, 72 °C per 10 min. - Caricare 20-25 μL del prodotto PCR in un gel di agarosio al 3% contenente macchia di acido nucleico ed eseguire a 100 V per circa 1 h.
7. Analisi del gel utilizzando un software di elaborazione e analisi delle immagini32
- Dopo la corsa, fotografare il gel (utilizzando un sistema di acquisizione dell'imaging gel) evitando qualsiasi saturazione della banda e quantificare le bande utilizzando un software di elaborazione delle immagini.
- Per la quantificazione si consideri che le bande a ~631 bp, ~493 bp e ~156 bp corrispondono rispettivamente agli isoformi 13S, 12S e 9S.
- Dal menu File del software, aprire il file di immagine ottenuto dal sistema di acquisizione dell'imaging. Converti in scala di grigi, regola luminosità e contrasto e rimuovi il rumore più esterno, se necessario.
- Disegnare un rettangolo intorno alla prima corsia con lo strumento Selezione rettangolo e selezionarlo tramite l'| Gel | Selezionare il comando Prima corsia oppure premendo la scelta rapida da tastiera.
- Usare il mouse per fare clic e tenere premuto nel mezzo del rettangolo sulla prima corsia e trascinarlo sulla corsia successiva. Vai ad Analizza | Gel | Selezionare Corsia successivaoppure premere il collegamento disponibile. Ripetere questo passaggio in tutte le corsie rimanenti.
- Dopo che tutte le corsie sono evidenziate e numerate, vai ad Analizza | Gel | Traccia corsie per disegnare un grafico di profilo di ogni corsia.
- Con lo strumento di selezione linea retta, disegna una linea attraverso la base di ogni picco corrispondente a ogni banda, tralascio il rumore di fondo. Dopo che tutte le cime, da ogni corsia, sono state chiuse, selezionare lo strumento Bacchetta e fare clic all'interno di ogni picco. Per ogni picco evidenziato, le misurazioni devono apparire nella finestra Risultati visualizzata.
- Sommare l'intensità di tutte le 3 bande per ogni campione e calcolare la percentuale per ogni isoforma relativa al totale.
- Tracciare le percentuali di ogni isoforma o le differenze nella percentuale rispetto ai campioni non trattati.
NOTA: Assicurarsi che la somma di tre varianti E1A sia uguale al 100%.
Risultati
Un test di siti di giunzione da 5' utilizzando il minigene E1A è stato eseguito per valutare i cambiamenti nel profilo di giunzione nelle cellule dopo l'esposizione della chemioterapia. Il ruolo di Nek4 - isoforma 1 nella regolazione AS nelle cellule stabili HEK293 dopo il trattamento con paclitaxel o cisplatino è stato valutato.
La regione Adenovirale E1A è responsabile della produzione di tre mRNA principali da un precursore dell'RNA a causa dell'uso di diversi donatori di giunzione. Condividono termini comuni da 5' e 3' ma differiscono per l'entità dei loro introni accisa. Gli MRNA Adenoviral E1A sono nominati in base ai loro coefficienti di sedimentazione, 13S, 12S e 9S. Durante la fase iniziale dell'infezione da adenovirus (circa 7 h), vengono prodotte proteine importanti per preparare la cellula infetta per la replicazione virale del DNA (13S - 723 aa e 12S - 586 aa) e, nella fase finale (circa 18 ore) oltre a quelle, viene prodotta una piccola proteina (9S -249 aa)20. Utilizzando un plasmide contenente il minigene di E1A, l'effetto sullo splicing alternativo può essere osservato nelle cellule dopo la trasfezione valutando la proporzione di mRNA da ogni isoforma prodotta: 13S: 631 bp, 12S: 493 bp e 9S 156 bp (Figure 1A e B).
L'espressione basale delle varianti di isoforme E1A dipende dalla linea cellulare e dal tempo dell'espressione E1A. È stato osservato che la linea cellulare stabile HEK293 (HEK293-Flag) o la ricombinazione HEK293 contenente il sito (HEK293-FRT - la linea cellulare originale) mostra un'espressione più alta di 13S rispetto alle cellule HeLa (HeLa-PLKO) che mostra livelli simili di isoforme 13S e 12S dopo 48 h di espressione E1A (Figura 1C e D).
L'alto livello di espressione 13S osservato nelle cellule stabili HEK293 è notevolmente diminuito in tempi più brevi di espressione E1A (circa 30 h). La proporzione (%) di 13S:12S:9S a 30 h e 48 h è rispettivamente di 60:33:7 e 80:15:5 (dati non rappresentati). Per questo motivo, è importante caratterizzare il profilo di giunzione e1A del minigene cellulare basale prima di iniziare gli esperimenti.
Le cellule esposte al cisplatino hanno mostrato uno spostamento nella selezione di giunzione 5 SS favorendo l'espressione 12S (un aumento di circa il 15% rispetto alle cellule non trattate). Questo effetto è stato osservato in HEK293 che esprime stabilmente il vettore vuoto flag così come l'isoforma 1 di Nek4. Quando si osservano cambiamenti importanti nella percentuale di espressione, un grafico con percentuali rappresenta chiaramente i risultati (Figura 2).
Quando si confrontano due condizioni (sovraespressione Flag e Nek4) che rispondono a un trattamento, di solito il modo migliore per rappresentare i dati è tracciare le differenze su un grafico, perché il livello basale di espressione può essere diverso e le percentuali non rifletteranno l'effetto reale del trattamento. Ciò può essere osservato nella figura 3. I cambiamenti nell'AS dopo il trattamento paclitaxel furono molto discreti, ma le direzioni dei cambiamenti erano opposte nelle cellule che esprimevano Flag e Nek4.
Nonostante piccoli cambiamenti dopo il trattamento, i risultati sono stati coerenti, indicando che il trattamento paclitaxel porta a una diminuzione dell'isoforma 13S, con un aumento di 12S e 9S nelle cellule che esprimono Flag, mentre, d'altra parte, nelle cellule che esprimono Nek4, si osserva l'effetto opposto.

Figura 1: Modello di giunzione Minigene E1A dipende dalla linea cellulare. A) Rappresentazione schematica dei siti di splicing minigene E1A. Le frecce indicano la regione di ricottura del primer per l'amplificazione degli isoformi minigene E1A. B)Isoforme generate da splicing alternativo del minigene E1A. C) HEK293 che esprime stabilmente il vettore vuoto flag (HEK293 -Flag), HeLa trasfetto con vettore PLKO (HeLa - PLKO) o, siti contenenti ricombinazione HEK293 (da ciò che HEK293 stabilmente esprimendo Flag o Nek4.1 sono stati generati - HEK293-FRT) sono stati transfettanti con pMTE1A plasmide. 48 ore dopo l'isolato dell'RNA totale di trasfezione e le isoforme E1A sono state separate in gel di agarosio(D). Grafico che confronta la percentuale di isoforma 13S, 12S e 9S nelle cellule HEK293-Flag e HeLa-PLKO in condizioni basali. Dati di tre esperimenti indipendenti. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Effetto del trattamento con cisplatino nel modello di giunzione minigene E1A. HEK293 che esprime stabilmente il vettore vuoto flag (Flag) o il tag Nek4.1 Flag fusa, sono stati transessuali con pMTE1A plasmide. Sei ore dopo che la tetraciclina di trasfezione è stata aggiunta all'induzione dell'espressione delle proteine. Da 24 h a 48 ore, il mezzo di coltura cellulare è stato sostituito per un mezzo contenente 30 μM di Cisplatino. Dopo 24 ore di incubazione, è stato estratto l'RNA totale e i prodotti della PCR sono stati separati al 3% di gel di agarosio. A)Sono raffigurate isoforme minigene predominanti E1A. I grafici B-D rappresentano la % di ogni isoforma rispetto alla somma di tre varianti (13S, 12S e 9S). In E, la differenza nella percentuale di espressione per ogni isoforma viene presentata rispetto al controllo del veicolo (medio). I grafici sono presentati come la media e il SEM di tre esperimenti indipendenti. * p< 0,05, ** p<0,01 in prova t non accoppiata. Clicca qui per visualizzare una versione più grande di questa figura.
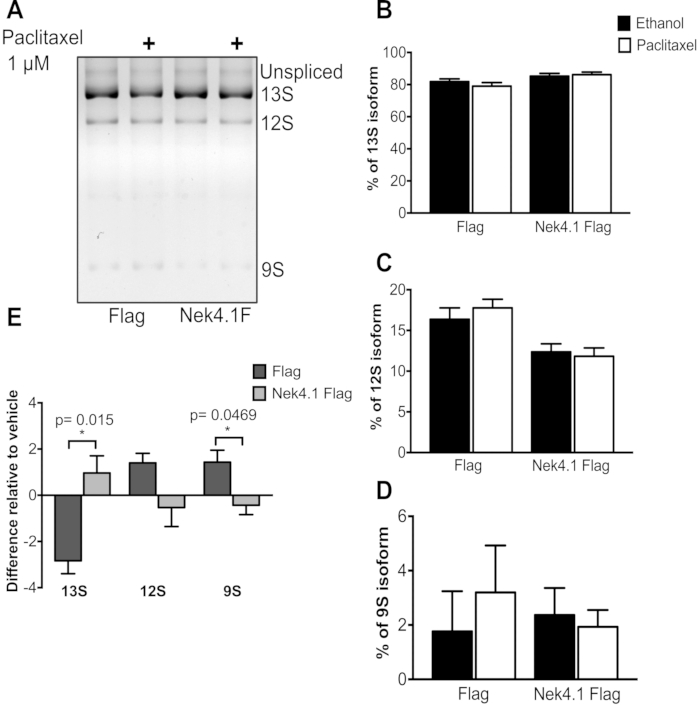
Figura 3: Effetto del trattamento paclitaxel nel modello di giunzione minigene E1A. HEK293 che esprime stabilmente il vettore vuoto flag (Flag) o la bandiera Nek4.1 fusa, sono stati trasfettanti con pMTE1A plasmide. Sei ore dopo che la tetraciclina di trasfezione è stata aggiunta all'induzione dell'espressione delle proteine. Da 24 h a 48 ore, il mezzo di coltura cellulare è stato sostituito da un mezzo contenente 1 μM di Paclitaxel o etanolo (0,02%) utilizzato come controllo del veicolo. Dopo 24 ore di incubazione, è stato estratto l'RNA totale e i prodotti della PCR sono stati separati al 3% di gel di agarosio. A)Sono raffigurate isoforme predominanti. I grafici B-D rappresentano la % di ogni isoforma rispetto alla somma di tre varianti (13S, 12S e 9S). In E, la differenza nella percentuale di espressione per ogni isoforma viene presentata rispetto al controllo del veicolo (etanolo). I grafici sono presentati come la media e il SEM di tre esperimenti indipendenti. * p< 0,05 in test t non accoppiato. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
I minigeni sono strumenti importanti per determinare gli effetti nello splicing alternativo globale in vivo. Il minigene adenovirale E1A è stato utilizzato con successo per decenni per valutare il ruolo delle proteine aumentando la quantità di queste nella cellula13,14. Qui, proponiamo l'uso minigene E1A per valutare lo splicing alternativo dopo l'esposizione chemioterapeutica. Fu utilizzata una linea cellulare stabile che esprimeva l'isoforma 1 di Nek4, evitando gli artefatti della sovraespressione causati dalla transitoria transitoria. L'isoforma 1 di Nek4 non ha mostrato effetto nel minigene E1A alternativo splicing in condizioni basali21, ma ha molti interattori correlati allo splicing, quindi, permettendoci di valutare l'effetto specifico del trattamento chemioterapico in E1A splicing alternativo in queste cellule.
Nonostante la sua bassa sensibilità, principalmente rispetto agli approcci radioattivi, il metodo qui descritto è semplice e non richiede reagenti speciali o condizioni di laboratorio. Tuttavia, è importante notare che il minigene E1A è un reporter globale delle selezioni 5 SS, anche se la selezione delle SS 3 può essere valutata con questo protocollo, il minigene reporter specifico deveessere utilizzato 14,33,34. Inoltre, i risultati possono essere influenzati dalla linea cellulare e devono essere attentamente valutati per evitare interpretazioni errate a causa del profilo di giunzione alternativo basale.
Di solito, grandi differenze nel modello di giunzione minigene E1A si osservano solo quando si cambia l'espressione dei fattori di giunzione. Altri cambiamenti sono meno evidenti a causa del gran numero di proteine che modulano l'attività di questi fattori. Per questo motivo, quando si iniziano gli studi per un candidato indiretto, dovrebbe essere preferito l'approccio classico, basato su quantità crescenti di questa proteina candidata. Quando si osserva un certo effetto, i trattamenti possono essere eseguiti per esplorare se la regolazione può essere specifica di un particolare stimolo cellulare.
Dopo un risultato positivo preliminare, è possibile eseguire la standardizzazione del tempo e della concentrazione del farmaco per ottimizzare l'esperimento.
Questo semplice protocollo è un saggio preliminare, un punto di partenza in grado di rispondere se la proteina di interesse mostra un effetto nello splicing alternativo e anche, quando qualche effetto nella regolazione dello splicing alternativo è già noto, può indirizzare gli studi verso il percorso più coerente in cui la proteina svolge un ruolo regolando lo splicing alternativo nella risposta chemioterapica.
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari concorrenti.
Riconoscimenti
Ringraziamo Fundação de Amparo a Pesquisa do Estado de São Paulo (FAPESP, attraverso Grant Temático 2017/03489-1 a JK e borsa di studio a FLB 2018/05350-3) e con il Conselho Nacional de Desenvolvimento Cientifico e Tecnológico (CNPq) per il finanziamento di questa ricerca. Vorremmo ringraziare il Dott. Adrian Krainer per aver fornito il plasmide pMTE1A e Zerler e colleghi per il loro lavoro nella clonazione E1A. Ringraziamo anche la Dott.ssa Patrícia Moriel, la Dott.ssa Wanda Pereira Almeida, il Prof.
Materiali
| Name | Company | Catalog Number | Comments |
| 100 pb DNA Ladder | Invitrogen | 15628-050 | |
| 6 wells plate | Sarstedt | 833920 | |
| Agarose | Sigma | A9539-250G | |
| Cisplatin | Sigma | P4394 | |
| DEPC water | ThermoFisher | AM9920 | |
| DMEM | ThermoFisher | 11965118 | |
| dNTP mix | ThermoFisher | 10297-018 | |
| Fetal Bovine Serum - FBS | ThermoFisher | 12657029 | |
| Fluorescent Microscope | Leica | DMIL LED FLUO | |
| Gel imaging acquisition system - ChemiDoc Gel Imagin System | Bio-Rad | ||
| GFP - pEGFPC3 | Clontech | ||
| HEK293 stable cells - HEK293 Flp-In | Generated from Flp-In™ T-REx™ 293 - Invitrogen and described in ref 21 | ||
| Hygromycin B | ThermoFisher | 10687010 | Used for Flp-In cells maintenemant |
| Image processing and analysis software - FIJI software | ref. 32 | ||
| Lipid- based transfection reagent - jetOPTIMUS Polyplus Reagent | Polyplus | 117-07 | |
| Oligo DT | ThermoFisher | 18418020 | |
| Paclitxel | Invitrogen | P3456 | |
| Plate Reader/ UV absorbance | Biotech | Epoch Biotek/ Take3 adapter | |
| pMTE1A plasmid | Provided by Dr. Adrian Krainer | ||
| pMTE1A F | Invitrogen | 5’ -ATTATCTGCCACGGAGGTGT-3 | |
| pMTE1A R | Invitrogen | 5’ -GGATAGCAGGCGCCATTTTA-3’ | |
| Refrigerated centrifuge | Eppendorf | F5810R | |
| Reverse Transcriptase - M-MLV | ThermoFisher | 28025013 | |
| Reverse transcriptase - Superscript IV | ThermoFisher | 18090050 | |
| Ribunuclease inhibitor RNAse OUT | ThermoFisher | 10777-019 | |
| RNA extraction phenol-chloroform based reagent - Trizol | ThermoFisher | 15596018 | |
| SybrSafe DNA gel stain | ThermoFisher | S33102 | |
| Taq Platinum | Thermo | 10966026 | |
| Tetracyclin | Sigma | T3383 | Used for Flag empty or Nek4- Flag expression induction |
| Thermocycler Bio-Rad | Bio-Rad | T100 | |
| Trypsin | Sigma | T4799 |
Riferimenti
- Ule, J., Blencowe, B. J. Alternative Splicing Regulatory Networks: Functions, Mechanisms, and Evolution. Molecular Cell. 76 (2), 329-345 (2019).
- Pan, Q., Shai, O., Lee, L. J., Frey, B. J., Blencowe, B. J. Deep surveying of alternative splicing complexity in the human transcriptome by high-throughput sequencing. Nature Genetics. 40 (12), 1413-1415 (2008).
- Nilsen, T. W., Graveley, B. R. Expansion of the eukaryotic proteome by alternative splicing. Nature. 463 (7280), 457-463 (2010).
- Stamm, S. Signals and their transduction pathways regulating alternative splicing: a new dimension of the human genome. Human Molecular Genetics. 11 (20), 2409-2416 (2002).
- Kornblihtt, A. R., et al. Alternative splicing: a pivotal step between eukaryotic transcription and translation. Nature Reviews Molecular Cell Biology. 14 (3), 153-165 (2013).
- Zhong, X. -. Y., Ding, J. -. H., Adams, J. A., Ghosh, G., Fu, X. -. D. Regulation of SR protein phosphorylation and alternative splicing by modulating kinetic interactions of SRPK1 with molecular chaperones. Genes & Development. 23 (4), 482-495 (2009).
- Misteli, T., Cáceres, J. F., Clement, J. Q., Krainer, A. R., Wilkinson, M. F., Spector, D. L. Serine Phosphorylation of SR Proteins Is Required for Their Recruitment to Sites of Transcription In Vivo. Journal of Cell Biology. 143 (2), 297-307 (1998).
- Kanopka, A., et al. Regulation of adenovirus alternative RNA splicing by dephosphorylation of SR proteins. Nature. 393 (6681), 185-187 (1998).
- Anufrieva, K. S., et al. Therapy-induced stress response is associated with downregulation of pre-mRNA splicing in cancer cells. Genome Medicine. 10 (1), 49 (2018).
- Shultz, J. C., et al. SRSF1 Regulates the Alternative Splicing of Caspase 9 Via A Novel Intronic Splicing Enhancer Affecting the Chemotherapeutic Sensitivity of Non-Small Cell Lung Cancer Cells. Molecular Cancer Research. 9 (7), 889-900 (2011).
- Gabriel, M., et al. Role of the splicing factor SRSF4 in cisplatin-induced modifications of pre-mRNA splicing and apoptosis. BMC Cancer. 15, (2015).
- Lee, S. C. -. W., Abdel-Wahab, O. Therapeutic targeting of splicing in cancer. Nature Medicine. 22 (9), 976-986 (2016).
- Cooper, T. A. Use of minigene systems to dissect alternative splicing elements. Methods. 37 (4), 331-340 (2005).
- Stoss, O., Stoilov, P., Hartmann, A. M., Nayler, O., Stamm, S. The in vivo minigene approach to analyze tissue-specific splicing. Brain Research Protocols. 4 (3), 383-394 (1999).
- Zerler, B., et al. Adenovirus E1A coding sequences that enable ras and pmt oncogenes to transform cultured primary cells. Molecular and Cellular Biology. 6 (3), 887-899 (1986).
- Gattoni, R., Schmitt, P., Stevenin, J. In vitro splicing of adenovirus E1A transcripts: characterization of novel reactions and of multiple branch points abnormally far from the 3' splice site. Nucleic Acids Research. 16 (6), 2389-2409 (1988).
- Stephens, C., Harlow, E. Differential splicing yields novel adenovirus 5 E1A mRNAs that encode 30 kd and 35 kd proteins. The EMBO journal. 6 (7), 2027-2035 (1987).
- Ulfendahl, P. J., et al. A novel adenovirus-2 E1A mRNA encoding a protein with transcription activation properties. The EMBO journal. 6 (7), 2037-2044 (1987).
- Berk, A. J., Sharp, P. A. Structure of the adenovirus 2 early mRNAs. Cell. 14 (3), 695-711 (1978).
- Svensson, C., Pettersson, U., Akusjärvi, G. Splicing of adenovirus 2 early region 1A mRNAs is non-sequential. Journal of Molecular Biology. 165 (3), 475-495 (1983).
- Basei, F. L., Meirelles, G. V., Righetto, G. L., dos Santos Migueleti, D. L., Smetana, J. H. C., Kobarg, J. New interaction partners for Nek4.1 and Nek4.2 isoforms: from the DNA damage response to RNA splicing. Proteome Science. 13 (1), 11 (2015).
- Zhou, Z., et al. The Akt-SRPK-SR Axis Constitutes a Major Pathway in Transducing EGF Signaling to Regulate Alternative Splicing in the Nucleus. Molecular Cell. 47 (3), 422-433 (2012).
- Caceres, J., Stamm, S., Helfman, D., Krainer, A. Regulation of alternative splicing in vivo by overexpression of antagonistic splicing factors. Science. 265 (5179), 1706-1709 (1994).
- Zhong, X. -. Y., Ding, J. -. H., Adams, J. A., Ghosh, G., Fu, X. -. D. Regulation of SR protein phosphorylation and alternative splicing by modulating kinetic interactions of SRPK1 with molecular chaperones. Genes & Development. 23 (4), 482-495 (2009).
- Naro, C., et al. The centrosomal kinase NEK2 is a novel splicing factor kinase involved in cell survival. Nucleic Acids Research. 42 (5), 3218-3227 (2014).
- Lu, C. -. C., Chen, T. -. H., Wu, J. -. R., Chen, H. -. H., Yu, H. -. Y., Tarn, W. -. Y. Phylogenetic and Molecular Characterization of the Splicing Factor RBM4. PLoS ONE. 8 (3), 59092 (2013).
- Jarnæss, E., et al. Splicing Factor Arginine/Serine-rich 17A (SFRS17A) Is an A-kinase Anchoring Protein That Targets Protein Kinase A to Splicing Factor Compartments. Journal of Biological Chemistry. 284 (50), 35154-35164 (2009).
- Bressan, G. C., et al. Functional association of human Ki-1/57 with pre-mRNA splicing events. FEBS Journal. 276 (14), 3770-3783 (2009).
- Vivarelli, S., et al. Paraquat Modulates Alternative Pre-mRNA Splicing by Modifying the Intracellular Distribution of SRPK2. PLoS ONE. 8 (4), 61980 (2013).
- Russell, W. C., Graham, F. L., Smiley, J., Nairn, R. Characteristics of a Human Cell Line Transformed by DNA from Human Adenovirus Type 5. Journal of General Virology. 36 (1), 59-72 (1977).
- Aranda, P. S., LaJoie, D. M., Jorcyk, C. L. Bleach gel: A simple agarose gel for analyzing RNA quality. Electrophoresis. 33 (2), 366-369 (2012).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Bai, Y. Control of 3' splice site choice in vivo by ASF/SF2 and hnRNP A1. Nucleic Acids Research. 27 (4), 1126-1134 (1999).
- Cote, G. J., Nguyen, N., Lips, C. J. M., Berget, S. M., Gagel, R. F. Validation of an in vitro RNA processing system for CT/CGRP precursor mRNA. Nucleic Acids Research. 19 (13), 3601-3606 (1991).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon