Method Article
Évaluation de la cytotoxicité à médiation cellulaire dépendante des anticorps dans les cellules cancéreuses à l’aide d’un essai biologique de prototoxicité à médiation cellulaire dépendante des anticorps
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ici, nous présentons un protocole pour un test de cytotoxicité à médiation cellulaire (ADCC) dépendant des anticorps à l’aide d’un kit de dosage biologique ADCC. Cette méthode offre un outil précieux pour élucider le mécanisme de l’ADCC et évaluer le potentiel thérapeutique des anticorps dans l’immunothérapie du cancer.
Résumé
La méthode de cytotoxicité à médiation cellulaire dépendante des anticorps (ADCC) représente un outil important pour évaluer l’efficacité des anticorps thérapeutiques dans l’immunothérapie du cancer. L’évaluation de l’activité de l’ADCC dans les cellules cancéreuses est essentielle pour le développement et l’optimisation des traitements à base d’anticorps. Ici, nous proposons une approche méthodologique de l’utilisation d’un kit d’essai biologique ADCC pour l’évaluation quantitative de la réaction ADCC en utilisant des cellules cancéreuses de la thyroïde comme cellules effectrices. Le protocole implique la co-culture de cellules effectrices avec des cellules cancéreuses cibles dans différents ratios en présence d’un anticorps thérapeutique. Le kit d’essai biologique ADCC utilisé dans cette expérience comprend des cellules effectrices génétiquement modifiées exprimant un gène rapporteur de la luciférase sous le contrôle d’éléments de réponse du facteur nucléaire des cellules T activées (NFAT). Lors de la liaison de l’antigène de surface sur les cellules cibles avec les anticorps et les cellules effectrices, les cellules effectrices libèrent de la luciférase, permettant la quantification de la cytotoxicité par mesure du signal de luminescence. Contrairement aux tests ADCC conventionnels, cette méthode a prouvé la liaison de l’antigène cible avec des anticorps et des cellules effectrices, ce qui peut produire des résultats fiables dans un court laps de temps.
Introduction
La cytotoxicité à médiation cellulaire dépendante des anticorps (ADCC) est un mécanisme important par lequel les anticorps exercent des effets de destruction cellulaire à médiation immunitaire 1,2,3. Les cellules immunitaires sont activées en se liant à l’anticorps thérapeutique, qui interagit avec les antigènes de surface des cellules cibles pour libérer des granzymes, la perforine, conduisant à la mort cellulaire cible. Ces cellules immunitaires comprennent les cellules tueuses naturelles (NK) et les neutrophiles 2,3,4,5,6,7. Le test ADCC est devenu un outil important pour évaluer l’efficacité de l’anticorps thérapeutique 8,9.
Dans le test ADCC conventionnel, les cellules mononucléées du sang périphérique (PBMC) ou les cellules tueuses naturelles sont utilisées comme cellules effectrices pour surveiller l’efficacité d’un anticorps thérapeutique en quantifiant le taux de mort cellulaire de la cible. Notre méthode utilise un kit d’essai biologique ADCC qui comprend des cellules effectrices génétiquement modifiées exprimant un gène rapporteur de la luciférase sous le contrôle d’éléments de réponse NFAT (Nuclear Factor of Activated T-cellules). Nous quantifions ensuite la liaison de l’antigène de surface sur les cellules cibles avec l’anticorps et les cellules effectrices. Cette méthode est basée sur la réaction ADCC se produisant dans un court laps de temps sans nécessiter de cellules PBMC humaines. Les étapes expérimentales comprennent la co-culture de cellules effectrices avec des cellules cibles en présence d’anticorps thérapeutiques.
Au cours de l’incubation, l’anticorps thérapeutique se lie à l’antigène cible à la surface des cellules cibles, ce qui conduit à la liaison des cellules effectrices et du fragment Fc d’un anticorps. Cela active l’élément de réponse NFAT et libère des signaux de luminescence pour l’évaluation quantitative de la réaction ADCC.
Avant d’effectuer l’expérience, l’expression de l’antigène cible dans les cellules cibles doit être confirmée par cytométrie en flux ou par western blot. Les cellules cibles sont cultivées et passées dans des plaques de 96 puits pendant 24 heures avant l’expérience. Différentes concentrations d’un anticorps thérapeutique sont ajoutées avec différents nombres de cellules effectrices pour obtenir le rapport effecteur/cellule cible calculé.
Les étapes clés de cette méthode comprennent (1) la préparation des cellules cibles et des cellules effectrices, (2) les rapports entre les cellules effectrices et les cellules cibles, (3) la préparation de différentes concentrations de l’anticorps et (4) la variation de la durée de l’incubation. Après l’incubation, les signaux de luminescence sont mesurés à l’aide d’un luminomètre, ce qui permet d’obtenir une lecture quantitative de l’activité de l’ADCC. Comparée à d’autres méthodes de mesure de l’ADCC, cette méthode est relativement simple à utiliser et les résultats sont précis.
L’essai biologique rapporteur ADCC indique la liaison de l’antigène cible, de l’anticorps thérapeutique et des cellules immunitaires dans l’activation de la voie ADCC. Cette liaison active la transcription génique par la voie NFAT dans les cellules effectrices conçues par les cellules Jurkat avec le récepteur FcγRIIIa exprimant de manière stable, la variante V158 (haute affinité). L’élément de réponse NFAT médie l’expression de la luciférase dans les cellules effectrices10,11. L’activité biologique de l’anticorps dans le mécanisme d’action (MOA) de l’ADCC est quantifiée par le signal de la luciférase produit par la voie NFAT. Le signal de la luciférase dans les cellules effectrices - cellules Jurket exprimant le récepteur FcγRIIIa - est quantifié à l’aide d’un lecteur de luminescence (Figure 1). Le rapport signal/bruit du test est élevé.
Protocole
1. Détection de l’expression de l’EGFR et du VEGFR dans les cellules cibles
REMARQUE : Utilisez le western blot pour détecter l’expression de l’antigène cible dans les cellules cibles.
- Préparation des échantillons
- Cultivez les cellules (lignées cellulaires de cancer de la thyroïde humain BHT-101 et SW-1736) dans des flacons T75 à l’aide d’un milieu du Roswell Park Memorial Institute (RPMI) complété par 10 % de sérum de veau fœtal (FBS), 1 % de L-glutamine et 1 % d’antibiotiques (pénicilline-streptomycine).
- Récoltez les cellules lorsqu’elles sont confluentes à 80 % en retirant le milieu de culture et en lavant les cellules avec du PBS. Ajouter 5 mL de PBS et gratter les cellules à l’aide d’un grattoir cellulaire pour retirer les cellules de la fiole de culture. Transférez les cellules dans un tube conique de 15 ml.
- Comptez les cellules à l’aide d’un compteur de cellules.
- Aliquote 5 × 106 de cellules BHT-101 et SW-1736 dans des tubes coniques de 15 ml.
- Faites tourner les cellules à 135 × g pendant 5 min à température ambiante. Retirez le PBS surnageant.
- Lyser la pastille cellulaire en ajoutant 100 μL de tampon de lyse RIPA contenant des inhibiteurs de la protéase-phosphatase pendant 5 min sur de la glace. Transférez le lysat dans un tube de 2 ml, centrifugez-le à 15 000 × g pendant 15 minutes et transférez le surnageant dans un nouveau tube de 2 ml.
- Mesurez la concentration en protéines du lysat cellulaire à l’aide d’un kit de dosage d’acide bicinchoninique (BCA).
- Aliquote 70 μg de protéines de lysat cellulaire de chaque échantillon, ajouter 2x tampon d’échantillon Laemmli complété par 10 % de 2-mercaptoéthanol et faire bouillir les échantillons à 100 °C pendant 5 min pour la dénaturation des échantillons.
- Électrophorèse sur gel et transfert membranaire
- Préparez un gel de polyacrylamide de dodécylsulfate de sodium (SDS) à 8 % en utilisant de l’eau distillée (4,7 ml), de l’acrylamide à 30 % (2,7 ml), un tampon tris à 1,5 m, un pH de 8,8 (2,5 ml), du dodécylsulfate de sodium à 10 % (0,1 ml), du persulfate d’ammonium à 10 % (100 μL) et TEMED (10 μL).
- Analyser 70 μg des échantillons dénaturés dans le gel de polyacrylamide SDS à 8 % à 80 V pendant 20 min et à 120 V pendant 100 min.
- Transférez la protéine sur le gel dans une membrane de fluorure de polyvinylidène (PVDF) à 100 V pendant 90 min.
- Après le transfert, bloquer la membrane avec du lait écrémé à 7 % dilué dans un tampon TBS-T (20 mM Tris pH 7,6, 140 mM NaCl, 0,2 % Tween-20) pendant 1 h.
- Incubation d’anticorps
- Incuber la membrane bloquée avec des anticorps monoclonaux anti-récepteur du facteur de croissance épidermique (EGFR) de souris et anti-récepteur du facteur de croissance endothélial vasculaire (VEGFR) de lapin (dilués à 1:1 000 dans du lait écrémé à 7 %) pendant la nuit à 4 °C.
- Après l’incubation, laver soigneusement la membrane avec le tampon Tris-buffered saline-Tween (TBS-T) (20 mM Tris pH 7,6, 140 mM NaCl, 0,2 % Tween-20) pendant 7 min et répéter 4 fois.
- Incuber la membrane avec l’anticorps secondaire conjugué à la HRP (dilué à 1:4 000 dans du lait écrémé à 7 % [TBS-T]) pendant 1 h.
- Après l’incubation, laver soigneusement la membrane avec un tampon TBS-T pendant 7 minutes et répéter 4 fois.
- Détection des protéines
- Incuber la membrane dans un substrat HRP chimiluminescent préparé en mélangeant 1 mL de solution de peroxyde de substrat HRP avec 1 mL de réactif Luminol de substrat HRP (les deux réactifs proviennent du kit de substrat chimiluminescent) pendant 2 min.
- Visualisez la membrane à l’aide d’un système d’imagerie.
2. Préparation des cellules cibles
- Cultivez les lymphocytes T cibles, les cellules BHT-101 et SW-1736 de manière exponentielle jusqu’à ce qu’ils confluent à 80 % dans un milieu RPMI complété par 10 % de FBS, 1 % de L-glutamine et 1 % d’antibiotiques (pénicilline-streptomycine) dans des flacons T-75.
- Après avoir retiré le support, lavez les cellules une fois avec du PBS. Incuber les cellules avec 1 ml de tampon de dissociation cellulaire non enzymatique pendant 5 min pour déloger les cellules adhérentes. Ajouter du PBS pour arrêter la réaction du tampon de dissociation cellulaire non enzymatique. Faites tourner les cellules à 135 × g pendant 5 minutes et ajoutez 5 ml de PBS.
REMARQUE : Ici, nous utilisons un tampon de dissociation cellulaire non enzymatique pour préserver l’intégrité de la protéine de surface de la membrane. - Comptez les cellules et ensemencez-les à 15 000 cellules/puits dans des microplaques de polystyrène blanc à 96 puits avec un fond transparent et plat.
3. Préparation de différentes concentrations de l’anticorps thérapeutique
- Pour suivre ce protocole, utilisez le Cetuximab (anticorps chimérique anti-EGFR) pour lier l’EGFR et utilisez le Bevacizumab (anticorps anti VEGF-A humanisé) comme contrôle négatif.
- Préparez le tampon d’essai biologique ADCC en ajoutant 1,4 mL de sérum à faible teneur en IgG dans 33,6 mL de RPMI-1640 (fourni dans le kit).
- Utilisez le tampon ADCC Bioassay pour la dilution des anticorps. Préparez 400 μL de chaque anticorps en trois concentrations différentes : 30 μg/mL, 3 μg/mL et 0,3 μg/mL (concentration 3x) pour obtenir la concentration finale de 10 μg/mL, 1 μg/mL et 0,1 μg/mL pour une couverture à large spectre afin de détecter la dose optimale.
REMARQUE : Les concentrations finales d’anticorps sont basées sur 25 μL de cellules cibles cultivées, 25 μL de cellules effectrices et 25 μL d’anticorps dans chaque puits d’une plaque de 96 puits.
4. Préparation des cellules effectrices
- Stockez les cellules effectrices à -80 °C avant utilisation.
- Préchauffez le tampon d’essai biologique ADCC dans un bain-marie à 37 °C pendant au moins 30 minutes avant de l’utiliser.
- Décongelez les cellules effectrices de l’ADCC Bioassay dans un entrepôt frigorifique à -80 °C en les plaçant dans un bain-marie à 37 °C (environ 2-3 min). Basculez doucement et inspectez visuellement le flacon, mais ne le retournez pas pendant le processus de décongélation.
- Transférez 630ul de cellules effectrices dans un tube de 15 ml contenant 3,6 ml de tampon de dosage ADCC. Bien mélanger en retournant doucement le tube 2x.
5. Incubation de cellules effectrices avec des anticorps et des cellules cibles
- Après une nuit d’incubation, prélever le milieu des cellules cibles (15 000 cellules cancéreuses par puits) et ajouter 25 μL de tampon d’essai biologique ADCC et 25 μL de cetuximab (antagoniste de l’EGFR) et de bevacizumab (antagoniste du VEGF) pour obtenir la concentration finale de 0,1 μg/mL, 1 μg/mL ou 10 μg/mL des cellules cibles dans chaque puits, conformément à la figure 2.
- Ajoutez 75 000 cellules effectrices dans 25 μL de tampon d’essai biologique ADCC par puits dans la microplaque de 96 puits contenant les cellules cibles. Les puits étiquetés sans anticorps monoclonaux agissent comme un contrôle sans anticorps (figure 2).
- Ajoutez le tampon d’essai biologique ADCC dans les puits étiquetés AB (tampon d’essai biologique ADCC) pour le contrôle à blanc.
- Incuber la plaque pendant 6 h.
6. Lecture quantitative de l’activité de l’ADCC
- Préparez le réactif de dosage de la luciférase 4 h avant la mesure en ajoutant le tampon de dosage de la luciférase au substrat de dosage de la luciférase (lyophilisé).
- Après 6 h d’incubation des cellules cibles, des anticorps et des cellules effectrices, ajouter 75 μL de réactif de dosage de la luciférase dans chaque puits et incuber pendant 30 min.
- Après la période d’incubation, mesurez le signal de luminescence dans chaque puits à l’aide d’un luminomètre, fournissant une lecture quantitative de l’activité de l’ADCC.
- Pour l’analyse des données, calculez l’induction du pli comme suit :
Induction du pli = RLU (induit par les anticorps - arrière-plan) / RLU (pas de contrôle des anticorps - arrière-plan).
où RLU est constitué d’unités de luminescence relatives ; Les anticorps induits sont les puits B3 à B8 et C3 à C8 ; Pas de contrôle des anticorps = B2 et C2 ; Contexte = RLU moyen du puits A2 à A5 (figure 2).
Résultats
L’expression de l’EGFR et du VEGFR dans les cellules cibles BHT-101 et SW-1736 a été détectée à l’aide du western blot. L’expression de l’EGFR a été détectée dans les cellules BHT-101 et SW-1736, mais pas dans l’expression du VEGFR (Figure 3).
À l’aide du kit d’essai biologique ADCC, nous avons détecté la réaction ADCC de l’anticorps anti-EGFR, cetuximab, en utilisant des lignées cellulaires positives à l’EGFR, BHT-101 et SW-1736, comme cellules cibles. Le bevacizumab, un inactivateur du VEGF, a été utilisé comme anticorps de contrôle négatif. Différentes concentrations d’anticorps et de cellules effectrices ont été incubées avec les cellules cibles. Le signal de luminescence a été détecté à l’aide d’un lecteur de plaques (Figure 2). Un repli plus élevé de l’induction de l’activité ADCC a été observé dans les groupes Cetuximab (anticorps anti-EGFR), mais aucune activité ADCC n’a été observée dans le Bevacizumab (antagoniste du VEGF) pour les deux lignées cellulaires. Cela a démontré l’effet ADCC ciblant les antigènes de la membrane extracellulaire EGFR pour les deux lignées cellulaires. En utilisant cette méthode, la réaction ADCC a été validée en présence de Cetuximab dans deux lignées cellulaires anaplasiques de cancer de la thyroïde (BHT101 et SW1736) (Figure 4).
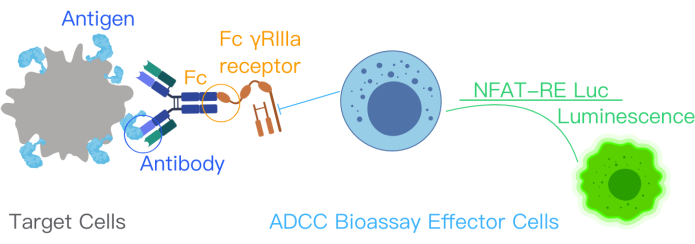
Figure 1 : Schéma de principe de la réaction de l’ADCC avec le kit d’essai biologique de l’ADCC. Les sites de liaison à l’antigène dans l’anticorps thérapeutique se lient aux antigènes de surface dans les cellules cibles. Cette liaison conduit à la liaison de la partie Fc de l’anticorps aux récepteurs FcƳRIIIa des cellules effectrices, qui ont été génétiquement modifiées avec NFAT-RE luc, produisant un signal de luminescence dans les cellules effectrices. Cette figure a été modifiée à partir du manuel du kit ADCC Reporter Bioassay Core avec la permission12. Abréviations : ADCC = Cytotoxicité à médiation cellulaire dépendante des anticorps ; Fc = Région cristallisable du fragment ; NFAT = facteur nucléaire de cellule T activée ; RE = Élément de réponse ; luc = Luciférase. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Lecture de la luminescence et schéma expérimental de la conception de plaques à 96 puits du système de co-culture ADCC. Les signaux de luminescence sont lus par un lecteur de luminescence. Rangée A :Les puits de la 1èreà la 9e contiennent un tampon d’essai biologique ADCC. Rangée B : 3à 8èmes puits contiennent le système de réaction ADCC de BHT101 (cellules cibles + anticorps + cellules effectrices). Le 2èmepuits est sans anticorps. Le 9èmepuits ne contient que le tampon d’essai biologique ADCC. Rangée C : 3à 8e puits contiennent le système de réaction ADCC de SW-1736 (cellule cible + anticorps + cellules effectrices). Le 2èmepuits est sans anticorps. Le 9èmepuits ne contient que le tampon d’essai biologique ADCC. Rangée D : 1st-9 th puits contiennent un tampon de dosage. Abréviations : ADCC = Cytotoxicité à médiation cellulaire dépendante des anticorps ; AB = tampon d’essai biologique ADCC ; VEGFR = récepteur du facteur de croissance de l’épithélium vasculaire ; EGFR = récepteur du facteur de croissance épidermique ; mAb = anticorps monoclonal. Veuillez cliquer ici pour voir une version agrandie de cette figure.
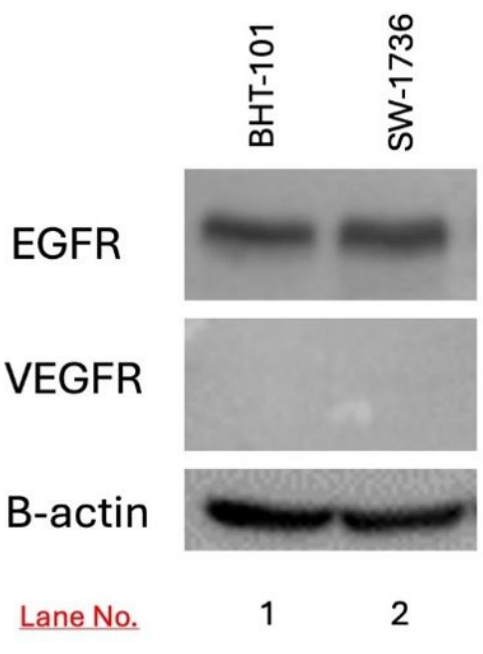
Figure 3 : Détection de l’expression de l’EGFR et du VEGFR dans le carcinome thyroïdien BHT-101 et SW-1736 à l’aide du western blot. L’expression de l’EGFR, mais pas du VEGFR, a été détectée dans les deux lignées cellulaires. L’actine B a été utilisée comme contrôle de charge. Abréviations : EGFR = récepteur du facteur de croissance épidermique ; VEGFR = facteur de croissance de l’épiderme vasculaire. Veuillez cliquer ici pour voir une version agrandie de cette figure.
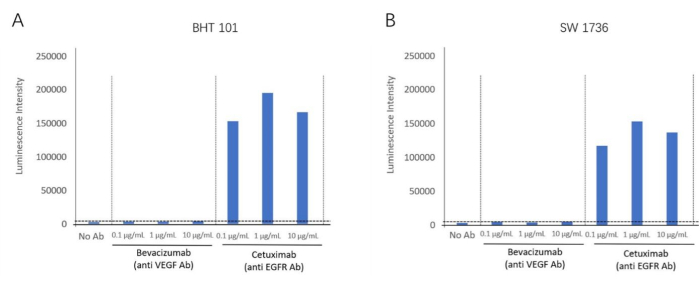
Figure 4 : Induction du repliement de l’activité de l’ADCC. (A) La lignée cellulaire du cancer de la thyroïde BHT-101 présente un effet ADCC en présence de l’anticorps anti-EGFR Cetuximab. (B) La lignée cellulaire du cancer de la thyroïde SW-1736 présente un effet ADCC en présence de l’anticorps anti-EGFR Cetuximab. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Ici, nous avons présenté la méthode ADCC Bioassay pour évaluer la réaction ADCC d’un anticorps thérapeutique. La méthode est simple et utilise un format simple « ajouter-mélanger-lire » pour la mesure.
Avant de réaliser l’expérience, l’expression de l’antigène cible dans les cellules cibles doit être confirmée soit par cytométrie en flux, soit par western blot. La cytométrie en flux sera un meilleur outil pour détecter l’antigène de surface. Cependant, l’utilisation de la cytométrie en flux peut stresser les cellules, provoquant l’apoptose et affectant la viabilité de la cellule et, par conséquent, l’analyse globale. De plus, il coûte plus cher que le western blot. Dans cette expérience, nous avons utilisé le western blot comme méthode plus rapide et plus rentable, car les antigènes ciblés sont des antigènes de surface cellulaire connus.
Des cellules Jurkat modifiées, qui expriment de manière stable le récepteur FcγRIIIa, la variante V158 (haute affinité) et un élément de réponse NFAT qui détermine l’expression de la luciférase des lucioles, ont été utilisées comme cellules effectrices. L’activation de la voie NFAT entraîne la production de luciférase, et son activité est quantifiée sous la forme d’une lecture de luminescence. Le signal représente également l’activité de l’ADCC.
Tout d’abord, les cellules cibles (T) sont incubées avec des cellules effectrices (E) en présence de quantités variables d’anticorps dans des plaques de 96 puits pendant 6 h à 37 °C dans un incubateur de CO2 humidifié. Le rapport effecteur/cible de 5:1 est utilisé pour optimiser le signal. Par exemple, 75 000 cellules effectrices ont été ajoutées à 15 000 cellules cibles. À la fin de l’incubation, le réactif d’essai de luciférase Bio-Glo est ajouté à chaque puits, incubé pendant 30 min, et la luminescence (RLU, unités de luciférase relatives) est mesurée à l’aide d’un lecteur de plaque de luminescence.
Les principales étapes critiques de ce protocole sont les suivantes : (1) La manipulation des cellules effectrices. Le flacon de cellules ne doit pas être inversé pendant le processus de décongélation, basculer doucement et doit être utilisé immédiatement après la décongélation pour éviter la mort cellulaire indésirable ou affecter les performances de détection biologique. (2). Il est important d’utiliser des microplaques de polystyrène blanc à 96 puits à fond transparent et plat pour les mesures de luminescence, car la luminescence est capturée à partir du bas du lecteur.
Dans cette étude, nous avons observé les réactions ADCC plus fortes et plus faibles de l’anticorps anti-EGFR, le cetuximab, en fonction de la quantité de cellules cibles et de la concentration d’anticorps. Quelques raisons de gérer l’étalonnage des réactions ADCC faibles : (1) Les lectures de l’essai biologique rapporteur ADCC proviennent de cellules effectrices (E), avec un nombre constant de 75 000 cellules par puits dans le système de réaction ADCC de cette étude. Par conséquent, l’optimisation de la qualité des cellules cibles (T) est un aspect qui peut être amélioré. Le rapport E :T dans cette étude était de 5:1, qui a pu être ajusté jusqu’à 20:1. (2) La concentration d’anticorps est également l’un des facteurs importants affectant la réaction de l’ADCC. Des ajustements peuvent être effectués par dilution en série des anticorps pour explorer la plage de concentration optimale, obtenant ainsi une réponse maximale dans le rapport ADCC. (3) Le temps d’incubation des anticorps, des cellules cibles et des cellules effectrices est également crucial pour les résultats expérimentaux. Dans cette étude, nous avons incubé pendant 6 h, qui peuvent être prolongés jusqu’à 24 h pour obtenir la réaction ADCC optimale. (4) De plus, la concentration de la solution tampon (faible IgG) pour le dosage de l’ADCC doit également être explorée. La concentration sérique optimale pour la réponse ADCC peut être atteinte dans la plage de 1 % à 10 %.
Cette méthode expérimentale est rapide et simple, permettant de la terminer en une journée grâce à un simple protocole « ajouter-mixer-lire ». Les résultats sont stables et se prêtent à des tests par lots. Cependant, ce kit de dosage biologique ne détecte pas la mort cellulaire comme les méthodes ADCC conventionnelles qui utilisent des cellules PBMC ou NK comme cellules effectrices12,13. Au lieu de cela, il utilise des cellules Jurkat exprimées par le récepteur FcγRIIIa comme cellules effectrices, ce qui coûtera plus cher car les cellules sont génétiquement modifiées. Par rapport aux expériences ADCCtraditionnelles13,14, il élimine la nécessité de donner du sang d’individus en bonne santé et évite le processus complexe d’extraction des cellules immunitaires, atténuant ainsi les variations individuelles qui pourraient avoir un impact sur les résultats. Les signaux de luminescence détectés proviennent de la liaison des cellules effectrices aux cellules cibles, et non de la mort réelle des cellules cibles. Par conséquent, il peut y avoir des différences par rapport aux méthodes de détection ADCC standard. De plus, le tampon de dissociation cellulaire, qui est plus coûteux que la trypsine, est utilisé pour détacher les cellules adhérentes afin de maintenir l’intégrité de la membrane.
L’analyse des gènes rapporteurs de l’ADCC démontre une précision et une stabilité excellentes, servant de méthode d’analyse d’efficacité pour la détection de masse des médicaments à base d’anticorps thérapeutiques. Il peut également servir de méthode d’analyse critique pour la caractérisation des molécules thérapeutiques et le développement de procédés15.
En résumé, l’ADCC est un mécanisme immunitaire important, et la détection quantitative de l’ADCC revêt une grande importance dans le domaine de l’immunothérapie. Cette méthode expérimentale offre un moyen efficace pour la mesure quantitative de l’ADCC15.
Déclarations de divulgation
Tous les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Nous sommes reconnaissants au Prof. Zeng (IMCB, A*STAR) de soutenir ce travail. Cette étude a été soutenue par la Fondation pour la jeunesse de la Fondation nationale des sciences naturelles de Chine (NSFC) (82202231) et le Projet des sciences et technologies médicales et de la santé de la province du Zhejiang, en Chine (2021KY110,2024KY824).
matériels
| Name | Company | Catalog Number | Comments |
| 0.5% Trypsin-EDTA | Gibco | 15400-054 | Dilute 10x in PBS to make 0.05% Trypsin |
| 1x Tris Buffer Saline (TBS) | 1st BASE | BUF-3030-1X1L | For membrane washing in western blotting |
| 1.5 M Tris Buffer, pH 8.8 | 1st BASE | BUF-1419-1L-pH8.8 | For SDS gel preparation |
| 2-Mercaptoethanol | Sigma Aldrich | M7522-100ML | For sample preparation of western blotting |
| 30% Acrylamide/Bis solution | Bio-Rad | #1610158 | For SDS gel preparation |
| 4x Laemmli Buffer | Bio-Rad | #1610747 | For sample preparation of western blotting |
| 96-well white polystyrene microplate with clear flat bottom | Corning Incorporated | 3610 | For ADCC assay |
| ADCC Bioassay Effector cells (0.65 mL) | Promega | G7011 | Includes in ADCC reporter bioassay core kit (Promega G7010), 1 x 1 vial |
| ADCC reporter bioassay core kit | Promega | G7010 | Mentioned as ADCC bioassay kit for ADCC assay in this experiment |
| Ammonium Persulfate | Sigma Aldrich | A3678-25G | For SDS gel preparation |
| Bevacizumab (Humanized Anti VEGF-antibody) | MVASI | - | Use as negative control antibody in ADCC asssay |
| BHT-101 | Leibniz Institute DSMZ | ACC279 | Human anaplastic papillary thyroid cancer cell line |
| Bio-Glo Luciferase Assay Buffer | Promega | G7941 | Includes in ADCC reporter bioassay core kit (Promega G7010), 1 x 10 mL |
| Bio-Glo Luciferase Assay Substrate (Lyophilized) | Promega | G7941 | Includes in ADCC reporter bioassay core kit (Promega G7010), 1 x 1 vial |
| Cell scraper | GenFollower | GD00235 | To remove cell from culture flask |
| Cetuximab (Chimeric anti-EGFR antibody) | ERBITUX | - | Use as therapeutic antibody in ADCC assay |
| Chemiluminescent HRP substrate | Merck Millipore | WBKLS0500 | For protein detection in western blotting |
| Distilled water | Gibco | 15230-162 | For SDS gel preparation |
| Fetal Bovine Serum (FBS) | Gibco | 10270-106 | Culture media supplement |
| iBright CL1500 imaging system | Thermo Scientific | 2462621100038 | For protein detection in western blotting |
| L-glutamine, 200 mM | Gibco | 25030-081 | Culture media supplement |
| Low IgG Serum | Promega | G7110 | Includes in ADCC reporter bioassay core kit (Promega G7010), 1 x 4 mL |
| Megafuge 8R | Thermo Scientific | 42876589 | Centrifuge |
| Mouse anti-EGFR monoclonal antibodies | BD Biosciences | 610016 | Primary antibody in western blotting |
| Mouse anti-VEGFR monoclonal antibodies | BD Biosciences | 571194 | Primary antibody in western blotting |
| non-enzymatic cell dissociation buffer | Sigma Aldrich | C5789-100ML | For cell harvesting from T75 flask |
| Penicillin-Streptomycin | PAN Biotech | P06-07100 | Antibacterial for culture media |
| Phosphate Buffered Saline (PBS), pH 7.2, Sterile filtered | 1st BASE | CUS-2048-1x1L | Use as washing solution for cells |
| Pierce BCA assay kit | Thermo Scientific | 23225 | To measure protein concentration |
| Protease and phosphatase inhibitor | Thermo Scientific | A32959 | For protein digestion in sample preparation for western blotting |
| PVDF membrane (Immobilin-P) | Merck Millipore | IPVH00010 | For protein transfer in western blotting |
| Rabbit anti-mouse IgG, Fcγ HRP-conjugated secondary antibody | Jackson ImmunoResearch | 315-035-046 | Secondary antibody in western blotting |
| Roswell Park Memorial Institute (RPMI) medium | Capricorn Scientific | RPMI-XA | Cell culture media |
| RPMI-1640 | Promega | G7080 | Includes in ADCC reporter bioassay core kit (Promega G7010), 1 x 36 mL |
| Skim milk powder | Merck Millipore | 70166-500G | For membrane blocking in western blotting |
| Sodium Dodecyl Sulfate | 1st BASE | BIO-2050-500g | For SDS gel preparation |
| SW-1736 | Cytion | 300453 | Human thyroid squamous cell cancer cell line |
| T75 culture flasks | SPL Lifesciences | 70075 | Cell culture flask |
| Tecan Multimode Reader model Spark 10M | Tecan | 1607000294 | for luminicence quantification |
| TEMED | Bio-Rad | #1610801 | For SDS gel preparation |
| Tween-20 | Promega | H5151 | For membrane washing in western blotting |
| Vi-cell XR cell viability analyzer | Beckman Coulter | AL15072 | Cell counter |
Références
- Zahavi, D., AlDeghaither, D., O'Connell, Enhancing antibody-dependent cell-mediated cytotoxicity: a strategy for improving antibody-based immunotherapy. Antib Ther. 1 (1), 7-12 (2018).
- Fenis, A., Demaria, O., Gauthier, L., Vivier, E. New immune cell engagers for cancer immunotherapy. Nat Rev Immunol. 24 (7), 471-486 (2024).
- Pinto, S., Pahl, J., Schottelius, A., Carter, P. J. Reimagining antibody-dependent cellular cytotoxicity in cancer: the potential of natural killer cell engagers. Trends Immunol. 43 (11), 932-946 (2022).
- Ochoa, M. C., et al. Antibody-dependent cell cytotoxicity: immunotherapy strategies enhancing effector NK cells. Immunol Cell Biol. 95 (4), 347-355 (2017).
- Wang, W., Erbe, A. K., Hank, J. A., Morris, Z. S. NK cell-mediated antibody-dependent cellular cytotoxicity in cancer immunotherapy. Front Immunol. 6, 368 (2015).
- Chung, S., et al. Characterization of in vitro antibody-dependent cell-mediated cytotoxicity activity of therapeutic antibodies - impact of effector cells. J Immunol Methods. 407, 63-75 (2014).
- Shimasaki, N., Jain, A., Campana, D. NK cells for cancer immunotherapy. Nat Rev Drug Discov. 19 (3), 200-218 (2020).
- Cheng, Z. J., et al. Development of a robust reporter-based ADCC assay with frozen, thaw-and-use cells to measure Fc effector function of therapeutic antibodies. J Immunol Methods. 414, 69-81 (2014).
- Parekh, B. S., et al. Development and validation of an antibody-dependent cell-mediated cytotoxicity-reporter gene assay. MAbs. 4 (3), 310-318 (2012).
- Hogarth, P. M., Pietersz, G. A. Fc receptor-targeted therapies for the treatment of inflammation, cancer and beyond. Nat Rev Drug Discov. 11 (4), 311-331 (2012).
- Chung, S., et al. Quantitative evaluation of fucose reducing effects in a humanized antibody on Fcgamma receptor binding and antibody-dependent cell-mediated cytotoxicity activities. MAbs. 4 (3), 326-340 (2012).
- . ADCC Reporter Bioassay Core Kit Technical Manual Available from: https://www.promega.sg/-/media/files/resources/protocols/technical-manuals/101/adcc-reporter-bioassay-core-kit-protocol.pdf?rev=bec36264c0b6470591ded081377d207d&sc_lang=en (2023)
- Miller, A. S., Tejada, M. L., Gazzano-Santoro, H. Methods for measuring antibody-dependent cell-mediated cytotoxicity in vitro. Methods Mol Biol. 1134, 59-65 (2014).
- Lo Nigro, C., et al. NK-mediated antibody-dependent cell-mediated cytotoxicity in solid tumors: biological evidence and clinical perspectives. Ann Transl Med. 7 (5), 105 (2019).
- Gómez Román, V. R., Murray, J. C., Weiner, L. M., Ackerman, M. E., Nimmerjahn, F. . Antibody Fc. , 1-27 (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon