Method Article
Malign Periferik Sinir Kılıfı Tümörünün Fare Modellerinde Terapötik Hedefleri Belirlemek için Genetik Profilleme ve Genom Ölçeğinde Bırakma Taraması
Bu Makalede
Özet
Genetiği değiştirilmiş fare modellerinde ortaya çıkan tümörlerde terapötik hedefleri ve karşılık gelen insan tümör tipini belirlemek ve karşılaştırmak için genomik analizler ve fonksiyonel genomik ekranlar kullanan türler arası karşılaştırmalı bir onkogenomik yaklaşım geliştirdik.
Özet
Malign Periferik Sinir Kılıfı Tümörleri (MPNST'ler) Schwann hücrelerinden veya öncüllerinden türetilir. Tümör yatkınlık sendromu nörofibromatozis tip 1 (NF1) olan hastalarda, MPNST'ler en sık görülen malignite ve önde gelen ölüm nedenidir. Bu nadir ve agresif yumuşak doku sarkomları, 5 yıllık hastalıksız sağkalım oranlarının %34-60 olduğu keskin bir gelecek sunmaktadır. MPNST'li bireyler için tedavi seçenekleri hayal kırıklığı yaratacak şekilde sınırlıdır ve şekil bozucu cerrahi en önde gelen tedavi seçeneğidir. Ras sinyalinin bir inhibitörü olan tipifarnib gibi bir zamanlar umut vaat eden birçok tedavi klinik olarak başarısız olmuştur. Benzer şekilde, epidermal büyüme faktörünü (EFGR) hedefleyen erlotinib ve vasküler endotelyal büyüme faktörü reseptörünü (VEGF), trombosit kaynaklı büyüme faktörü reseptörünü (PDGF) ve Raf'ı hedefleyen sorafenib ile yapılan faz II klinik çalışmalar da standart kemoterapi ile kombinasyon halinde hastalarda yanıt üretememiştir.
Son yıllarda, kanser hücre hatlarının genetik profillemesi ile birleştirilen fonksiyonel genomik tarama yöntemlerinin, temel sitoplazmik sinyal yolaklarının tanımlanması ve hedefe özgü tedavilerin geliştirilmesi için yararlı olduğu kanıtlanmıştır. Nadir tümör tipleri söz konusu olduğunda, türler arası karşılaştırmalı onkogenomik olarak bilinen bu yaklaşımın bir varyasyonu, yeni terapötik hedefleri belirlemek için giderek daha fazla kullanılmaktadır. Türler arası karşılaştırmalı onkogenomikte, genetik olarak tasarlanmış fare (GEM) modellerinde genetik profilleme ve fonksiyonel genomik gerçekleştirilir ve sonuçlar daha sonra mevcut olan nadir insan örneklerinde ve hücre hatlarında doğrulanır.
Bu makale, tüm ekzom dizileme (WES) kullanılarak insan ve fare MPNST hücrelerinde aday sürücü gen mutasyonlarının nasıl tanımlanacağını açıklamaktadır. Daha sonra, fare ve insan MPNST hücrelerindeki kritik sinyal yollarını tanımlamak ve karşılaştırmak ve bu yollardaki ilaçlanabilir hedefleri belirlemek için genom ölçeğinde shRNA taramalarının nasıl gerçekleştirileceğini açıklıyoruz. Bu metodolojiler, çeşitli insan kanseri türlerinde yeni terapötik hedeflerin belirlenmesinde etkili bir yaklaşım sağlar.
Giriş
Malign periferik sinir kılıfı tümörleri (MPNST'ler), tümör yatkınlık sendromu nörofibromatozis tip 1 (NF1) ile birlikte, sporadik olarak genel popülasyonda ve önceki radyoterapi bölgelerindeortaya çıkan oldukça agresif iğsi hücreli neoplazmlardır 1,2,3. NF1 hastaları, NF1 tümör baskılayıcı geninin vahşi tip bir kopyası ve fonksiyon kaybı mutasyonu olan ikinci bir NF1 aleli ile doğarlar. Bu haplo yetmezliği durumu, NF1 hastalarını, tümör oluşumunu tetikleyen vahşi tip NF1 genlerinde ikinci bir fonksiyon kaybı mutasyonuna duyarlı hale getirir. Bu "ikinci vuruş" NF1 mutasyonu Schwann hücre soyundaki bir hücrede meydana geldiğinde, ortaya çıkan tümör ya deride ortaya çıkan bir dermal nörofibrom ya da büyük sinirlerde veya sinir pleksuslarında gelişen bir pleksiform nörofibromdur. Dermal ve pleksiform nörofibromların patolojisi aynı olmasına rağmen, biyolojik davranışları oldukça farklıdır - hem dermal hem de pleksiform nörofibromlar iyi huylu olmasına rağmen, sadece pleksiform nörofibromlar dönüşüme uğrayabilir ve MPNST'lere yol açabilir. NF1 geni tarafından kodlanan Ras GTPaz aktive edici protein olan nörofibromin kaybına ek olarak, MPNST'ler, TP53 4,5,6,7, CDKN2A 8,9 ve PTEN 10 dahil olmak üzere diğer birçok tümör baskılayıcı genin mutasyonlarını taşır, policomb baskılayıcı kompleks 2 11,12 (PRC2 ; SUZ12 ve EED genleri) ve reseptör tirozin kinazların anormal ekspresyonu 1,2. NF1 ve yukarıda belirtilen diğer genlerin mutasyonları, sporadik ve radyasyona bağlı MPNST'lerdede mevcuttur 11,12.
MPNST'lerdeki genomik anormallikleri anlamamızdaki bu ilerlemeler, patogenezlerini anlamak için paha biçilmez olsa da, henüz MPNST'ler için etkili yeni tedavilerin geliştirilmesine yol açmamıştır. Yeni tedavilerin geliştirilmesini engelleyen önemli bir engel, MPNST'lerin nadir görülen kanserler olduğu gerçeğidir. Bu nedenle, Kanser Genom Atlası (TCGA) tarafından üstlenilenler gibi temel itici mutasyonları tanımlayan küresel analizler için gerekli olan çok sayıda hasta örneğini elde etmek zordur. Deneyimlerimize göre, mütevazı sayıda insan MPNST örneğinin bile biriktirilmesi yıllar alabilir. Bu tür sınırlamaların üstesinden gelmek için, diğer nadir kanser türlerini inceleyen birçok araştırmacı, temel sürücü gen mutasyonlarını tanımlamak, ilgilendikleri tümördeki temel sitoplazmik sinyal yollarını tanımlamak ve yeni terapötik hedefleri belirlemek için türler arası karşılaştırmalı onkogenomik kullanımına yönelmiştir. Tümör oluşumu için gerekli olan sinyal yolları, insanlar ve diğer omurgalı türleri arasında yüksek oranda korunduğundan, genom ölçekli shRNA ekranları gibi fonksiyonel genomik yaklaşımların uygulanması, bu yeni sürücü mutasyonları, sinyal yollarını ve terapötik hedefleri tanımlamanın etkili bir yolu olabilir 13,14,15,16,17,18,19, özellikle sınırlı sayıda mevcut olan nadir insan tümör tiplerini incelerken20.
Burada sunulan metodolojilerde, Schwann hücresine özgü büyüme faktörü nöregulin-1'in (NRG1) aşırı ekspresyonunun pleksiform nörofibromların patogenezini ve daha sonra MPNST'lere ilerlemesini teşvik ettiği genetik olarak tasarlanmış bir fare modeli (GEM) olan P 0-GGFβ3 farelerinden türetilen insan MPNST hücre hatlarında ve erken geçiş MPNST kültürlerinde genomik profilleme gerçekleştirmeye yönelik bu yaklaşımı açıklıyoruz 21, 22,23. Bu yaklaşımdaki ilk adım, P 0-GGFβ3 MPNST'lerde, insan MPNST hücre hatlarında ve cerrahi olarak rezeke edilen insan MPNST'lerinde aday sürücü genlerini tanımlamaktır. Bu mutasyonlardan etkilenen sinyal yollarını işlevsel olarak doğrulamak için, insan ve fare MPNST hücre hatlarında proliferasyon ve hayatta kalma için gerekli genleri tanımlamak için genom ölçeğinde shRNA ekranları kullanıyoruz. Proliferasyon ve hayatta kalma için gerekli genleri belirledikten sonra, İlaç Gen Etkileşimi Veritabanını kullanarak "hit" koleksiyonundaki ilaçlanabilir gen ürünlerini tanımlarız. Ayrıca, GEM modelinin ve insan MPNST'lerinin aynı genlere ve sinyal yollarına benzer bağımlılık gösterip göstermediğini belirlemek için insan ve fare MPNST hücrelerindeki "isabetleri" karşılaştırıyoruz. Proliferasyon ve hayatta kalma için gerekli genlerdeki örtüşmelerin ve etkilenen sinyal yolaklarının belirlenmesi, P 0-GGFβ3 fare modelini moleküler düzeyde doğrulamanın bir yolu olarak hizmet eder. Bu yaklaşım aynı zamanda, fare modelinin insan ekranlarının tamamlayıcısı olarak hizmet edebileceği yeni terapötik hedefleri belirlemek için insan ve fare ekranlarını birleştirmenin etkinliğini vurgulamaktadır. Bu türler arası yaklaşımın değeri, insan tümörlerinin ve hücre hatlarının elde edilmesinin zor olduğu nadir tümörlerde terapötik hedefler ararken özellikle belirgindir.
Protokol
Çalışmalara başlamadan önce, Kurumsal Hayvan Bakım ve Kullanım Komitesi (IACUC) ve Kurumsal Biyogüvenlik Komitesi (IBC) tarafından gözden geçirilen ve onaylanan viral vektörlerin işlenmesine yönelik hayvan prosedürlerini ve protokollerini yaptırın. Burada açıklanan prosedürler, Güney Carolina Tıp Üniversitesi'nin IACUC ve IBC Kurulları tarafından onaylanmıştır ve NIH Laboratuvar Hayvanlarının Bakımı ve Kullanımı Kılavuzu ve MUSC'nin kurumsal hayvan bakımı yönergelerine uygun olarak uygun şekilde eğitilmiş personel tarafından gerçekleştirilmiştir.
1. WES-Seq Analizleri ve Patojenik Varyantların Tanımlanması
- Piyasada bulunan standart adsorpsiyon silika jel bazlı yöntemler kullanarak 60 mm'lik bir tabak üzerinde büyütülen bir tümör örneğinden veya alt birleşen (%70) MPNST hücrelerinden genomik DNA'yı izole edin (ayrıntılı adımlar için üreticinin protokolüne bakın). Genel iş akışı Şekil 1'de şematize edilmiştir.
- Farklı genomik DNA miktarları veya konsantrasyonları belirtilmedikçe, dizileme çekirdeğine 50 ng/μL'de en az 10 μL genomik DNA gönderin.
- Çekirdek tesis, genomik DNA'yı sonikasyonla parçalar ve daha sonra tercih edilen yöntemi kullanarak saflaştırır. Ekzom yakalama ve kütüphane yapımı, tercih edilen ekzom dizileme kiti kullanılarak gerçekleştirilir ve amplifiye edilmiş yakalanan ekzoma indeks etiketleri eklenir.
- Tüm ekzom eşleştirilmiş uç dizilemesinin (WES; her uçtan 100 bp dizili) gerçekleştirilmesi için örnekleri dizileme çekirdeğine gönderin.
- Çekirdek tarafından oluşturulan FASTQ dosyaları araştırmacıya sağlanır. Analiz için yalnızca kalite ölçümlerini geçen FASTQ dosyalarını kullanın.
- Piyasada bulunan yazılım programlarını (örneğin, DNAStar19,20, Partek21 veya Varsome) kullanarak FASTQ dosyalarını hizalayın ve analiz edin. Varsayılan ayarları kullanarak FASTQ dosyalarını fare başvuru genomu GRCm38/mm10 ile hizalayın.
NOT: Örnek olarak DNAStar ve bir fare MPNST örneği kullanılarak, genel iş akışı Şekil 1 ve aşağıda kısaca açıklanmıştır.- DNAStar yazılımını açın ve SeqMan NGen iş akışını seçin.
- Varyant Analizi/Yeniden Sıralama'yı ve dizileme analizi türü NGS Tabanlı Amplicon'u, gen panelini veya ekzomu seçerek İş Akışı'nı seçin. Sonrakine tıkla.
- Genom Paketini İndir'i ve uygun referans genomu (ör. Mus_musculus-GRCm38-dbSNP146.zip veya Homo sapien-GRCh37.p13.zip) seçerek tercih edilen Referans Dizisini seçin. Çekirdek tesis bir yatak dosyası sağladıysa, bu yardımcı dosyayı yükleyin. Sonrakine tıkla.
- Uygun sıralayıcı teknolojisi olan Illumina'yı seçerek Giriş Dizileri'ni seçin ve sıralama okumalarının eşleştirilmiş uç olduğunu belirleyin. Ardından deneme kurulumunu, çoklu örneklemeyi seçin ve Ekle'yi seçerek sıralama FASTQ dosyalarını karşıya yükleyin. Birden fazla numune çalıştırıyorsanız, her bir çift uç setini benzersiz bir deney veya numune adı ile belirleyin Tümör, Hücre hattı veya A18, A202... Sonrakine tıkla.
- Varsa denetim veri kümesini ayarlayın. İleri'ye tıklayın ve Montaj Seçenekleri altında tekrar İleri'ye tıklayın. Analiz Seçenekleri altında, uygun Varyant Algılama modunu Diploid olarak tıklatın ve ardından İleri'ye tıklayın. Derleme Çıktısı altında, projenin dosya kaydetme konumunu adlandırın ve belirleyin; Sonrakine tıkla. Derlemeyi yerel bilgisayarda veya bulutta çalıştırın.
NOT: Elde edilen hizalama dosyaları, algılanan tek nükleotid polimorfizmlerinin (SNP'ler) varyant açıklamaları için ArrayStar iş akışında açılabilir. Bu iş akışı, gen kopya sayısı kazançlarını veya kayıplarını değil, SNP'leri algılar. Yüksek yoğunluklu SNP dizisi adı verilen ayrı bir analiz, kopya sayısı değişikliklerini algılar; Tüm ekzom dizilimi, kopya sayısı değişikliklerini güvenilir bir şekilde tespit etmez. Hizalama programıyla gerçekleştirilecek belirli adımlar için program kullanım kılavuzuna bakın. - Belirli verileri dahil etmek veya hariç tutmak için algılanan SNP'lerin varyant ek açıklamasına kullanıcı tanımlı filtreler uygulayın. İşlevsel olarak ilgili olası varyantların yoğunlaştırılmış bir listesini elde etmek için, bu hiyerarşideki varyant veri kümelerine aşağıdaki filtreleri uygulayın: kontrolde değil (normal bir kontrol örneği mevcutsa), popülasyon frekansı (gnomAD, ExAC, 1.000 genom frekansları), alel frekansı (≤0.001 veya% 0.1'i içerir), kapsama derinliği (<10 derinliği hariç), patojenite veya ClinVar sınıfı (dahil: patojenik, muhtemelen patojenik; hariç tut: belirsiz, muhtemelen iyi huylu, iyi huylu) ve istenirse SNP tipi ve kodlama etkisi (şunları içerir: eşanlamlı olmayan, yanlış anlamlı, saçma, çerçeve kaydırma, çerçeve içi, ekleme).
NOT: Bilinen ilgili bir kanser gen listesi, yalnızca belirli hastalıkla ilgili genleri çekmek için nihai listeye de uygulanabilir, bkz. adım 1.3.7. Bu filtreler, varyant gen listesini 20'den az gene kadar azaltabilir.- Sıralama hatası SNP'lerini filtrelemek için varyant alel frekansını kullanın. Homozigot ve heterozigot varyantlar, saf kirletici olmayan bir hücre popülasyonu için sırasıyla yaklaşık% 100 ve% 50'de temsil edilecektir (yerleşik bir tümör kaynaklı hücre hattı, tek bir izojenik tümör hücresi popülasyonunu temsil etmelidir). Bu durumda, varyantlar için %100-90 ve %50-40 alel frekanslarını filtre olarak uygulayın, böylece bunun altındaki varyantları kaldırın. Aynı veri kümesi için yüksek yoğunluklu SNP dizisi verileri de mevcutsa, homozigot veya heterozigot oranlardan daha düşük değişken alel frekanslarına sahip SNP'lere kopya sayısı kazancı veya kaybı uygulayın (yani, 2 kopya alel A ve 1 kopya alel B ile sonuçlanan kopya sayısı kazancı, %75, %25'lik uygun varyant alel frekansları yapar, sırasıyla).
- ArrayStar ile patojenik varyantları tanımladıktan sonra, varyant gen listesini bir csv, txt veya xls dosyası olarak dışa aktarın ve kaydedin ve örtüşen ve benzersiz mutasyona uğramış gen listelerini belirlemek için bu mutasyonları içeren genleri P 0-GGFβ3 tarafından oluşturulan tümörlerin kohortundakilerle karşılaştırın. Mutasyona uğramış genleri, insan muadilleriyle ilişkili bilinen mutasyona uğramış genlerle karşılaştırın (yani, Bushman Lab Kanser Gen listesi veya kullanıcı küratörlüğünde bir liste).
- İsteğe bağlı olarak, herhangi bir kullanıcı tanımlı filtre uygulamadan önce (1.3.6), açıklamalı dosyayı dışa aktarın ve VCF dosyası olarak kaydedin.
NOT: Bu VCF dosyası, ikincil bir analiz olarak karşılaştırma için diğer varyant efektör tahmin yazılımı Varsome veya VEP'e yüklenebilir. Filtrelenmiş varyant gen listeleri üzerinde biyolojik ve yolak analizi de yapılabilir.
- İsteğe bağlı olarak, herhangi bir kullanıcı tanımlı filtre uygulamadan önce (1.3.6), açıklamalı dosyayı dışa aktarın ve VCF dosyası olarak kaydedin.
- Protein sınıfı, yol, yol bileşeni ve gen ailesi ve ontoloji bilgilerini elde etmek için kullanıcı tarafından tercih edilen bir veritabanı aracılığıyla varyant gen listesinin fonksiyonel sınıflandırmasını gerçekleştirin.
NOT: Burada örnek olarak PANTHER (pantherdp.org) kullanılmıştır.- Gen kimliği ile filtrelenmiş gen listesini yükleyin.
- Organizmayı seçin (Mus musculus).
- Analizi seçin (Gen listesinde görüntülenen fonksiyonel sınıflandırma) ve filtrelenmiş varyant gen listelerinde biyolojik ve yol analizini gönder'e tıklayın.
2. Genom Ölçekli shRNA Ekranları
NOT: Düşük geçişli tümör kültürlerine sahip genom ölçekli fonksiyonel ekranlar için kullanılabilecek çeşitli shRNA ve CRISPR kütüphaneleri mevcuttur. Burada örnek olarak CELLECTA DECIPHER shRNA kütüphanelerinin kullanımını açıklıyoruz. CELLECTA DECIPHER lentiviral shRNA kütüphaneleri, havuzlanmış formatta RNAi genetik taramaları için optimize edilmiştir. Her transkript en az 5-6 benzersiz shRNA tarafından hedeflenir ve her lentiviral shRNA vektörü, PCR primer bölgeleri ile çevrili benzersiz bir genetik barkod içerir. Bu kütüphaneler, insan ve fare hastalıklarıyla ilgili genlerin çoğunu kapsar, ancak genomdaki tüm genleri kapsamaz. Cellecta insan kütüphanesi plazmid DNA havuzları üç modülde (İnsan Modülü I, II, III; 15.377 geni hedefler) bulunurken, fare kütüphanesi plazmid havuzları iki modülde (Fare Modülleri I ve II; 9.145 geni hedefler) mevcuttur. Bu kütüphaneler, proliferasyon ve/veya hayatta kalma için gerekli olan hedeflenmiş genlerin viral transdüksiyondan sonra farklı zaman noktalarında farklı şekilde eksprese edildiği "bırakma" testlerini gerçekleştirmek için kullanılır.
- Lentiviral Ambalaj
- 0. Gün: %10 fetal sığır serumu (FBS) içeren 30 mL/çanak antibiyotiksiz DMEM'de 10 milyon 293T hücre/15 cm'lik 10 tabak tabak.
- 1. Gün: Ertesi gün hücrelerin ~% 80 birleştiğini ve transfekte etmeye hazır olduğunu onaylayın. 50 mL'lik konik bir tüpte, aşağıdakileri şu sırayla karıştırın: 600 μL ambalaj plazmit karışımı (0.5 μg/μL), 60 μL plazmit barkod kitaplığı, 12 mL DMEM (serum veya antibiyotik yok) ve 600 μL transfeksiyon reaktifi. Oda sıcaklığında 15 dakika inkübe edin.
- 900 μL transfeksiyon reaktifi ve 12 mL DMEM'i ayrı bir 15 mL konik tüpe yerleştirin ve vorteksleme ile karıştırın. DNA karışımına 12.9 mL transfeksiyon reaktifi/DMEM karışımı ekleyin ve karıştırmak için hafifçe vurun. Daha fazla karıştırmadan oda sıcaklığında 15 dakika inkübe edin. Bu karışımın 2.5 mL'sini damla damlasına, 293T hücreden oluşan her 15 cm'lik bir tabağa ekleyin ve bir doku kültürü inkübatöründe gece boyunca inkübe edin.
- 2. Gün: Ertesi gün besiyerini antibiyotik içeren normal büyüme besiyeri ile değiştirin.
- 3. Gün: Ortamı toplayarak ve 0,2 μm'lik bir filtrasyon ünitesinden geçirerek ve 15 mL'lik konik tüplere alikosyon yaparak virüsü hasat edin; ayrıca viral titrede (48 saat virüs olarak kabul edilir) kullanılmak üzere kriyoviyallerde beş adet 1 mL filtrelenmiş virüs alikutu hazırlayın; Virüsü -80 °C'lik bir dondurucuda saklayın. Hücrelerin kurumasını önlemek için ortamı plakalar üzerinde birer birer 30 mL büyüme ortamı ile değiştirin.
- 4. Gün: Ortamı toplayarak ve 0,2 μm'lik bir filtreleme ünitesinden geçirerek virüsü hasat edin. Aliquot virüsü 15 mL konik tüplere. Bu 72 saat virüsü olarak kabul edilir; Virüsü -80 °C'lik bir dondurucuda saklayın.
- Titre Lentiviral Havuzlar
- 65 mL tümör hücresi büyüme ortamına 65 μL katyonik polimer (10 mg / mL) ekleyin. Polimer içeren ortamın 1 mL/oyuğunu on bir adet 6 oyuklu doku kültürü plakasına pipetleyin. Erken geçiş tümör hücrelerini tripsinize edin ve tercih edilen yöntemi kullanarak hücreleri sayın, böylece her kuyucuk 50.000 hücre/mL/kuyu alır. Dondurucudan 1 mL 48 saat lentivirüs alikotunu 37 ° C'lik bir su banyosunda çözdürün.
- Her viral modül için enfeksiyonu Tablo 1'de gösterildiği gibi hazırlayın.
- Tüm 6 oyuklu plakaları bir doku kültürü inkübatörüne yerleştirin. Ertesi gün, viral medyayı çıkarın ve taze büyüme medyası ile doldurun. 48 saatte, seçimi alan tüm kuyucuklara puromisin içeren ortam ekleyin. Kontrol hücreleri birleşmeden önce, tercih edilen yöntemi kullanarak hayatta kalan klonların hücre sayımlarını gerçekleştirin.
NOT: Transdüksiyon yapılmamış hücreleri öldürmek için gereken puromisin konsantrasyonu, bir "öldürme" eğrisinde bir dizi konsantrasyon test edilerek ampirik olarak önceden belirlenmelidir. Transdüksiyon yapılmamış kültürlerin ölümünü eşit şekilde indükleyen en düşük konsantrasyonu kullanın.
- Hedef Hücrelerin Lentiviral Enfeksiyonu
- MPNST hücrelerini tripsinize edin ve sayın. Modül başına toplam yirmi 15 cm'lik tabak için 15 cm'lik çanak başına 2,5 milyon hücre tabağı. Virüsü çözün ve 5 μg/mL katyonik polimer varlığında 0.5'lik bir MOI'de virüslü hücreleri transdüksiyon yapın (Şekil 2A).
- Ertesi sabah virüs içeren besiyerini çıkarın ve taze büyüme besiyeri ile değiştirin. Seçim işaretleyicisinin ekspresyonuna izin vermek için hücreleri 2 gün daha kültürleyin ve ardından dönüştürülmemiş hücreleri ortadan kaldırmak için kültürlere puromisin ekleyin.
- Puromisin ilavesinden üç gün sonra, hücreleri tripsinize edin ve hücre popülasyonunun yarısını bir masa üstü santrifüjde 5 dakika boyunca 200 × g'da santrifüjleyin. Peletlenmiş hücreleri gelecekteki genomik DNA hazırlığı için -80 °C dondurucuda saklayın; bu, referans zaman noktası veya zaman noktası 1'dir (T1).
- Hücre popülasyonunun diğer yarısını yeniden plakalayın ve yukarıdaki gibi hasat ve santrifüjlemeden önce yaklaşık 7 popülasyon ikiye katlayın. Bu hücre peleti, son zaman noktası (zaman noktası 2, T2) olarak hizmet edecek ve gelecekteki genomik DNA izolasyonu için -80 °C'de saklanacaktır (Şekil 2).
- Virüs Havuzları ile Dönüştürülen Hücrelerden Genomik DNA'nın İzole Edilmesi
- Hücre peletini -80 °C dondurucudan çözün ve peleti RNAse eklenmiş 10 mL yeniden süspansiyon tamponunda yeniden süspanse edin ve hemen iki adet 15 mL polimetilpenten tüpüne bölün (Şekil 2B).
- 5 mL'ye 500 μL %10 SDS ekleyin, karıştırın ve oda sıcaklığında 5 dakika inkübe edin. Daha sonra, sıcaklığı 4 ° C'de tuttuğunuzdan emin olarak, DNA'yı 30 s açık / 30 s kapalı 25 döngü boyunca sonikleştirmek için tüpleri bir DNA kesme cihazına yerleştirin.
- 5 mL fenol/kloroform, pH 8.0 (4 °C'de saklanır) ekleyin, kullanmadan önce fenol/kloroformu iyice karıştırdığınızdan emin olun. Fenol/kloroform ilavesinden sonra, maksimum ayarda 45-60 s kuvvetlice girdaplayarak iyice karıştırın. 60 dakika, -20 °C'de 7.200 × g'da santrifüjleyin.
NOT: Girdaptan sonra köpüklü/sütlü bir görünüm, tam süspansiyona işaret edecektir. - Berrak üst fazın 3 mL'sini 15 mL'lik taze bir tüpe aktarın ve 0.5 mL 3 M sodyum asetat ve 4 mL izopropanol ekleyin ve iyice karıştırın (Şekil 2C). 20 °C'de 7.200 × g'da 30 dakika santrifüjleyin.
- Santrifüjlemeyi takiben, süpernatanı atın ve ardından kalan sıvıyı dikkatlice pipetleyin. 0,5 mL %70 etanol ekleyin ve peleti yukarı ve aşağı pipetleyerek yerinden çıkarın. Yeniden süspanse edilmiş peleti 1,5 mL'lik bir santrifüj tüpüne aktarın ve aynı numunedeki her iki peleti tek bir 1,5 mL'lik santrifüj tüpünde birleştirin. 5 dakika boyunca tezgah üstü santrifüjde maksimum hızda santrifüjleyin.
- Süpernatanı atın ve kalan% 70 etanolü emmek için bir laboratuvar mendili kullanın. Peletleri 0,5 mL damıtılmış suda tekrar süspanse edin, peletin kurumasına izin vermediğinizden emin olun, çünkü bu yeniden süspansiyonu zorlaştıracaktır. DNA konsantrasyonunu ölçmeden önce numuneleri 4 °C'de saklayın.
- shRNA barkodlarını yükseltmek için iç içe geçmiş PCR
NOT: DNA'yı depodan alın ve buzun üzerine koyun. DNA konsantrasyonu 100 ng/μL'den düşükse, DNA'yı vakumla kurutun ve uygun hacimde damıtılmış su içinde yeniden süspanse edin. Bir seferde yalnızca bir modülün işlenmesini öneririz (zaman noktası 1 (T1) veya zaman noktası 2 (T2)). Sonunda bir araya getirilecek olan her zaman noktasının iki hazırlığını gerçekleştirin; Bu nedenle, yinelenen zaman noktalarını aynı gün işlememek önemlidir. Aşağıdaki protokol, genomik DNA'nın bir zaman noktası içindir.- 7 tüp kurun ve bunları açıklandığı gibi etiketleyin: Negatif kontrol, pozitif kontrol, genomik DNA için 4 tüp ve bir ana karışım için bir tüp (Şekil 3A). Negatif kontrol damıtılmış sudur; pozitif kontrol, lentivirüs oluşturmak için 293T transfeksiyonunda kullanılan plazmit DNA'dır.
- Önce Tablo 2'de gösterildiği gibi her tüpe su ekleyin.
- Tablo 3'te gösterildiği gibi bir Ana Karışım (MM) hazırlayın.
- Su içeren her tüpe 18 μL MM ekleyin. Ardından, 4 şablon tüpün her birine 25 μL genomik DNA ekleyin. Ardından, pozitif kontrol tüpüne 1 μL pozitif kontrol (10 ng/μL) ekleyin, diğer tüpleri pozitif kontrol DNA'sı ile kirletmemeye dikkat edin. Son olarak, her tüpe 2 μL polimeraz ekleyin, önce 4 numune tüpüne ve en son pozitif kontrol tüpüne ekleyin; PCR tüplerini karıştırın ve döndürün (Şekil 3).
- Tablo 4'te belirtilen koşullar altında PCR reaksiyonu gerçekleştirin.
- İlk PCR çalışırken, tüpleri Tablo 2'te belirtildiği gibi 5. PCR için hazırlayın. Yedi tüp ayarlayın: Negatif kontrol, pozitif kontrol, genomik DNA için 4 tüp ve MM için bir tüp (Şekil 3B).
- Su içeren her tüpe 22 μL MM ekleyin ve DNA ve polimerazı eklemeden önce ilk PCR turunun bitmesini bekleyin.
- PCR'nin ilk turu tamamlandıktan sonra, 4 numune tüpünü bir mikrosantrifüj tüpünde birleştirin ve karıştırın.
- Su içeren her numune tüpüne 25 μL ekleyin.
- Ardından, uygun şekilde etiketlenmiş tüplere 2 μL ilk PCR negatif kontrol ve 2 μL ilk PCR pozitif kontrol ekleyin.
- Her tüpe 2 μL polimeraz ekleyin, önce 4 numune tüpüne ve en son negatif kontrole ekleyin.
- Tablo 6'da belirtildiği gibi PCR reaksiyonu gerçekleştirin.
NOT: %3.5'lik bir agaroz jel üzerinde elektroforez yoluyla ilk PCR reaksiyonunun sonuçları analiz edildikten sonra, bol miktarda ürün elde edilirse, bu prosedürün bir sonraki tekrarı için döngü sayısı 9-10'a düşürülebilir; Aynı şekilde ürün verimi düşükse devirler 14'e çıkarılabilir. - İkinci PCR reaksiyonu tamamlandığında, 4 numuneyi bir mikrosantrifüj tüpünde ve 80 μL 6x yükleme boyasında birleştirin. Pozitif ve negatif kontroller için 20 μL numune kullanın.
- Tris-Borat-EDTA (TBE) tamponunda %3.5'lik bir agaroz jeli hazırlayın (Şekil 4A). Büyük numune hacmini karşılamak için, pozitif ve negatif kontroller için iki kuyu bıraktığınızdan emin olarak, birkaç oyuğu birbirine bantlayarak (hacme uygun bir tarak yoksa) jel tarağında büyük bir kuyucuk oluşturun. Jeli 1 saat boyunca 90 V'ta çalıştırın.
- Jeli görselleştirin ve yaklaşık 250 baz çiftinde (bp) bir bandı onaylayın.
- İlk Saflaştırma: Jel Ekstraksiyonu
- Temiz bir neşter veya tıraş bıçağı kullanarak 250 bp bandını kesin ve mümkün olduğunca fazla jeli kesin. Büyük bandı 4 parçaya bölün ve her parçayı temiz bir mikrosantrifüj tüpüne aktarın. Her jel diliminin ağırlığını ölçün ve kaydedin.
NOT: Her parça tüp başına yaklaşık 200 mg veya daha az olmalıdır. - 6 hacim çözündürme tamponu ekleyin (örneğin, jel parçası 200 mg ağırlığındaysa, 1.2 mL tampon ekleyin). Tüpleri 50 °C'lik bir su banyosuna yerleştirin ve jel dilimleri eriyene kadar her 10-15 dakikada bir döndürün. Ardından, her tüpe 1 hacim izopropanol ekleyin (örneğin, jel parçası 200 mg ağırlığındaysa 0.2 mL izopropanol).
- Saflaştırma için iki döndürme sütunu kullanarak, 4 tüpün hepsinden alınan numuneleri sütunlara yükleyin. Sütunlar yalnızca 750 μL tuttuğu için tüm numune hacmini işlemek için birden fazla döndürme gerçekleştirin. Sütunları geleneksel bir tezgah üstü mikrosantrifüjde 1 dakika boyunca 17.900 × g'da döndürün ve her seferinde akışı atın.
- Kolonları 750 μL yıkama tamponu ile yıkayın ve 17.900 × g'da 1 dakika santrifüjleyin. Yıkamanın ardından akışı boşaltın ve 17.900 × g'da 3 dakika santrifüj ederek sıkma kolonunu kurutun. DNA'yı kolon başına 50 μL damıtılmış su ile elute edin ve daha önce olduğu gibi 1 dakika santrifüjleyin ve numunenin toplam hacmi 100 μL için her iki tüpü birleştirin.
- Temiz bir neşter veya tıraş bıçağı kullanarak 250 bp bandını kesin ve mümkün olduğunca fazla jeli kesin. Büyük bandı 4 parçaya bölün ve her parçayı temiz bir mikrosantrifüj tüpüne aktarın. Her jel diliminin ağırlığını ölçün ve kaydedin.
- İkinci arıtma
- Bu sonraki saflaştırma adımı, küçük parçaları ortadan kaldırmayan Bağlayıcı Tampon 2'yi (PCR Saflaştırma Kitinden) kullanacaktır. 100 μL DNA'ya 400 μL tampon ekleyin ve bir spin kolonuna yükleyin (Şekil 4B). Numuneyi 17.900 × g'da 1 dakika santrifüjleyin.
- Daha sonra, membranı 650 μL yıkama tamponu ile yıkayın ve daha önce yapıldığı gibi sıkın. Döndürme kolonunu yukarıdaki gibi ek santrifüjleme ile 3 dakika kurutun.
- DNA'yı 30 μL damıtılmış su ile yükseltin ve 1 dakika boyunca maksimum hızda döndürün. Elüsyonun ardından, bir spektrofotometredeki konsantrasyonu kontrol edin; konsantrasyonun 10 ng/μL'den düşük veya 70-80 ng/μL'den yüksek olmadığından emin olun. DNA'yı -20 °C'lik bir dondurucuda saklayın.
- Güçlendirilmiş barkodların sıralanması
- Sıralama için, saflaştırılmış barkodları elüsyon tamponu (EB) kullanarak 0,75 ng/μL'ye seyreltin. Dizi çeşitliliği eklemek için, amplikonlar %30 (v/v) PhiX dahil olmak üzere 17 pM'de kümelenir. Üreticinin protokolüne göre otomatik küme oluşturma sisteminde tek uçlu (SE) kümeleme gerçekleştirin.
- Sıralama çekirdeğinin bir NextGen dizileyici üzerinde toplam 36 döngü tek uçlu dizileme gerçekleştirmesini sağlayın (Şekil 4C). 0,5 μM'de Illumina dizileme primerlerine özel astar GexSeqS ekleyin.
- Fastq dosyaları oluşturmak için başvurulan analiz yazılımını kullanın ve okuma uzunluklarını 18 nükleotidlere kırpmak için yazılım kullanarak bunları işleyin.
- Kırpılmış okumaları açmak için Barcode Analyzer ve Deconvoluter yazılımını kullanın. Referans zaman noktasındaki (T1) okuma sayılarının son zaman noktasına (T2) oranı olarak her shRNA için kat tükenme puanlarını hesaplayın.
- shRNA Tarama Sonuçlarının Analizi ve Aday Terapötik Hedeflerin Belirlenmesi
NOT: Cellecta shRNA kütüphanesinde, çoğu gen ya 5 (% 67) ya da 6 farklı shRNA (% 32) tarafından hedeflenir. Bununla birlikte, çoğu hücrede isabet etmesi gereken birkaç temizlik geni, çok sayıda shRNA tarafından hedeflenir ve negatifler için puan aralığını tanımlayan kontroller olarak hizmet eder.- Daha fazla sayıda shRNA tarafından hedeflenen genlere yönelik önyargıyı önlemek için, log tükenme puanlarını dönüştürür. Ampirik dağılımından her gen için 80. yüzdelik dilimi hesaplayarak kantil bir tahmin gerçekleştirin.
- Log katlama tükenme puanlarının boş bir dağılımını oluşturmak için, genlerin %>95'inin tükenmeyeceğini ve log-quantile puanlarının normal bir dağılıma sahip olacağını varsayalım. Boş dağılımın ortalamasını tahmin etmek için ampirik dağılımın medyanını kullanın. Bu boş dağılımı kullanarak, boş dağılımın 95. yüzdelik diliminden daha büyük olan log-kat tükenme puanlarına sahip tüm genleri 'isabet' olarak sınıflandırın.
NOT: Tüm isabetler, tükenme puanları kesme noktasının üzerinde olan en az iki shRNA'ya sahip olmalıdır (Şekil 5). - RNAi bırakma ekranı verilerinin kalitesini ve kesme noktasının geçerliliğini değerlendirmek için, karşılaştırma için COLT Kanser RNAi tarama girişimi24,25 tarafından 'temel temel' olarak tanımlanan bir dizi gen kullanın. Potansiyel terapötik hedeflerin ilk listesini oluşturmak için CEG'leri isabet listesinden çıkarın.
NOT: COLT setindeki genler, COLT tarafından taranan 72 kanser hücresi dizisinin %>50'sinde isabet olarak puan aldı. Temel esansiyel gen listesi 640 gen içerir. - Birden fazla MPNST hücre hattı tarafından yaygın veya tekdüze olarak gerekli olan potansiyel terapötik hedefleri belirlemek için, farklı MPNST hücre hatlarının veya erken geçiş kültürlerinin ekranlarında tanımlanan CEG olmayan isabetlerin Venn diyagramlarını oluşturun (Şekil 6A). MPNST hatlarının veya kültürlerinin tümünde veya çoğunda gerekli olan CEG olmayan isabetlere öncelik verin.
- Alternatif olarak, ürünleri tümör hücresi proliferasyonu ve/veya hayatta kalması için gerekli olan sinyal yollarının bileşenlerini kodlayan genleri tanımlamak için her hücre hattından veya erken geçiş kültüründen CEG olmayan isabet listesinde yol analizleri yapın. Ardından, CEG olmayanların yol analizinde tanımlanan sinyal yollarını, WES ile tanımlanan mutasyonlardan etkilendiği belirlenen sinyal yollarıyla karşılaştırın.
NOT: WES'te tanımlanan mutasyonlardan tutarlı bir şekilde etkilenen sinyal yollarını tanımlamayı ve bunları shRNA ekranlarında kritik olarak tanımlanan sinyal yollarıyla karşılaştırmayı özellikle yararlı buluyoruz.
- Alternatif olarak, ürünleri tümör hücresi proliferasyonu ve/veya hayatta kalması için gerekli olan sinyal yollarının bileşenlerini kodlayan genleri tanımlamak için her hücre hattından veya erken geçiş kültüründen CEG olmayan isabet listesinde yol analizleri yapın. Ardından, CEG olmayanların yol analizinde tanımlanan sinyal yollarını, WES ile tanımlanan mutasyonlardan etkilendiği belirlenen sinyal yollarıyla karşılaştırın.
- Terapötik ajanların halihazırda mevcut olduğu ilaçlanabilir hedefleri belirlemek için, İlaç Gen Etkileşimi Veritabanını (dgidb.org) kullanarak temel temel genlerin çıkarılmasından sonra kalan isabetlerin listesini tarayın.
NOT: Bu veri tabanı, genlerin bir listesinin aynı anda girilmesine ve taranmasına izin verir ve bu genleri hedeflemek için şu anda mevcut olan ilaçlar hakkında rehberlik sağlar. - Belirlenen ilaçlanabilir yüksek faizli hedefleri, ilk kütüphane ekranında kullanılanlardan farklı shRNA'larla gen ekspresyonlarını tek bir parti formatında (az önce açıklanan bir kütüphane-parti formatı değil) düşürerek doğrulayın. Gen ekspresyonunu yıkmak için iki ila üç farklı lentiviral shRNA kullanın. Enfeksiyondan üç ila dört gün sonra, bunun tümör hücresi proliferasyonu ve sağkalımı üzerindeki etkisini aşağıda açıklandığı gibi belirleyin.
3. Aday Terapötik Ajanlarla Zorlanan MPNST Hücrelerinde Hücre Sayıları ve Canlılığının Sitometre Testlerini Gerçekleştirin
- DMEM'deki MPNST hücrelerini% 80 birleşmeye kadar büyütün. Hücreleri oda sıcaklığında Hanks'in dengeli tuz çözeltisi (HBSS) ile durulayın.
NOT: Bu hücrelerin büyümesi yeniden kaplandıktan sonra bir süre gecikeceğinden, hücreleri birleşecek şekilde büyütmeyin. - Hücreleri enzimatik olmayan bir hücre ayrışma çözeltisi ile 30 s ila 1 dakika kaplayarak hücreleri substrattan ayırın. Ayrışma çözeltisinin 1 mL'si başına 5 mL DMEM ekleyin ve substrattan ayırmak için hücreleri hafifçe yukarı ve aşağı pipetleyin.
- Bir hemositometre kullanarak hücreleri sayın. Siyah duvarlı 96 oyuklu plakalarda oyuk başına 1.200 hücre yoğunluğunda plaka hücreleri; Test edilecek her ilaç seyreltmesi için en az üç kuyu açın ve bu deneylerin üç biyolojik kopyasını gerçekleştirin.
- Test edilecek ilacın başlangıç konsantrasyonlarını belirlemek için, diğer kanser hücresi tiplerine karşı hangi konsantrasyonların etkili olduğunu değerlendirmek için literatürü gözden geçirin. İlk deneylerde, diğer kanser türlerinde kullanılan ilaç konsantrasyonunun iki büyüklük sırası üzerinde ve iki büyüklük sırası altında bir aralığı test edin.
- Test edilecek ilacın dilüsyonlarını hazırlayın ve her bir dilüsyonu veya aracı en az üç kopya kuyucuğuna ekleyin.
- İlaçların eklenmesinden 1, 3, 5 ve 7 gün sonra doğrudan hücre sayılarını değerlendirin. Hoechst 33342'yi 5 μg/mL'lik bir nihai konsantrasyona ekleyin ve plakaları 37 °C'de 30 dakika inkübe edin. 100.000 ms pozlama süreleriyle toplam hücre sayısı için doğrudan hücre sayımı seçeneğini kullanarak yüksek verimli görüntüleme sitometresindeki plakaları okuyun.
- Yazılımı kullanarak okumaları analiz edin ve bunları bir elektronik tabloya aktarın ve istatistiksel analizler için uygun yazılımı kullanın.
- İlaçla tedavi edilen kuyucuklarda hücre sayılarında istatistiksel olarak anlamlı azalmalar gözlenirse, bu azalmanın kısmen hücre ölümünün indüksiyonundan kaynaklanıp kaynaklanmadığını belirlemek için bir "Canlı / Ölü" testi yapın.
- Adım 3.3'te açıklandığı gibi siyah duvarlı 96 oyuklu plakalardaki plaka hücreleri.
- İlaçları adım 3.5'te açıklandığı gibi hazırlayın ve ekleyin.
- İlacın eklenmesinden 1, 3, 5 ve 7 gün sonra hücre canlılığını ve ölümünü değerlendirin. Her bir oyukta 1 μM'lik bir nihai konsantrasyona kalsein ve 1 μM'lik bir nihai konsantrasyona propidyum iyodür ekleyin. Hücreleri 37 °C'de 15 dakika inkübe edin.
- Live+Dead yazılım seçeneğini kullanarak bir görüntüleme sitometresindeki hücreleri görüntüleyin. Yazılımı kullanarak okumaları analiz edin ve bunları bir elektronik tabloya aktarın ve istatistiksel analizler için uygun yazılımı kullanın.
Sonuçlar
Şekil 5 grafikleri, taranan her insan hücre hattında CEG olmayanlara (YANLIŞ olarak etiketlenmiş) kıyasla DOĞRU olarak etiketlenmiş temel temel genlerin (CEG'ler) tükenme puanlarını gösterir. Puanlar, genel puan dağılımının bir kutu grafiği temsili üzerinde çizilen bireysel genler için kat tükenme puanlarının log2'sini temsil eder. Her bir hücre hattındaki iki grup arasında tükenme puanlarının ortalamasında anlamlı bir fark olup olmadığını test etmek için öğrenci t-testi kullanıldı. Elde edilen p değerleri her panelde gösterilir. Ortalama kat tükenme puanlarının CEG'ler için CEG olmayanlara göre önemli ölçüde daha yüksek olduğunu unutmayın. Çekirdek Esansiyel Genler, tanım gereği, çoğu hücre tipinde proliferasyon ve / veya hayatta kalma için sürekli olarak gerekli olduğu için bu beklenen bir durumdur.
Şekil 6A, üç insan MPNST hücre hattı için "isabetlerin" bir Venn şemasını sunar. Tipik olarak, çok sayıda genin birden fazla hat arasında paylaşıldığını buluruz; bu isabetler, MPNST'lerin büyük bir alt kümesinin çoğalması ve/veya hayatta kalması için gerekli olması muhtemel proteinleri kodlayan genleri temsil ettikleri için yüksek bir önceliktir. Ayrıca, yalnızca bir hücre hattında isabet eden bir dizi gen olduğuna dikkat edin. Buna sıkça rastlıyoruz ve ekranların kalitesiz olduğunun bir göstergesi olarak algılanmamalı. Birden fazla çizgi arasında ortak isabet olan genler daha sonra, mevcut ajanlarla ilaçlanabilen proteinleri kodlayan bu alt kümedeki genleri tanımlamak için İlaç Gen Etkileşimi Veritabanı kullanılarak değerlendirilir. Daha sonra bunlardan birkaçını seçiyoruz ve karşılık gelen mRNA'nın ekspresyonunu shRNA'larla yıkarak bir başlangıç doğrulaması gerçekleştiriyoruz. Bazı shRNA'ların hedef dışı etkileri olacağından, her zaman aynı transkripti hedefleyen birden fazla shRNA'yı test ederiz. Şekil 6B, MPNST hücrelerini hedeflemeyen bir kontrol ve BCL6'yı hedefleyen çoklu shRNA'lar ile dönüştürdüğümüz temsili bir sonucu göstermektedir. Hücre sayıları daha sonra transdüksiyondan sonra değişen zamanlarda belirlendi. BCL6 shRNA'larının birçoğunun hücre sayılarını önemli ölçüde azalttığına dikkat edin; Eşlik eden immünoblotta gösterildiği gibi, hücre sayısındaki azalma derecesi, BCL6 yıkım derecesi ile ilişkilidir. Şekil 6C, erken geçişli bir P 0-GGFβ3 MPNST kültürü için temsili bir büyüme eğrisini göstermektedir.

Şekil 1: MPNST dokusunun veya erken geçiş MPNST hücrelerinin tüm ekzom dizilimini gerçekleştirmek için iş akışı. Şematik, tümör kaynaklı erken geçiş kültürlerinde bulunan varyant tespitinin genel iş akışını göstermektedir. DNA'yı erken geçiş kültürlerinden izole edin (1) ve kaliteli DNA'yı gönderme protokollerine (2) göre dizileme çekirdeğine gönderin. Dizileme çekirdeği, gönderilen DNA'nın kalitesini kontrol edecek ve gerekli tüm numune ve genom kütüphanesi hazırlıklarını gerçekleştirecektir. Çekirdek tesis, kullanıcılara kalite kontrol metriklerine sahip FASTQ sıralama dosyaları sağlayacaktır (3). Kullanıcılar, FASTQ dosyalarını kendi seçtikleri bir genom hizalama ve varyant arayan programına yükleyecektir. (4) Açıklamalı varyantlar, ilgili olmayan varyantları kaldırmak için kullanıcı tanımlı kriterlere göre filtrelenir. Gösterilen temsili veriler, rezeke edilen insan MPNST tümör örneğini tümörden türetilen bir hücre dizisi ile karşılaştırır26. (5) PANTHER ile fonksiyonel sınıflandırma analizi yapın. Kısaltma: MPNST = Malign Periferik Sinir Kılıfı Tümörü. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
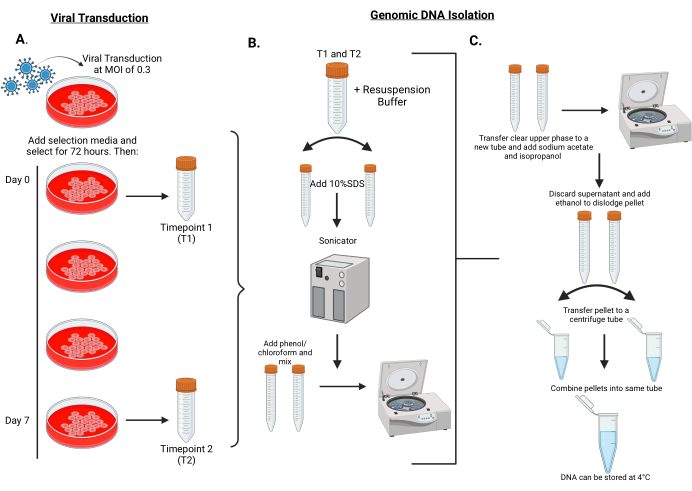
Şekil 2: ShRNA kütüphanelerinin MPNST hücrelerine viral transdüksiyonunu gerçekleştirmek ve genomik DNA'yı hücrelerden zaman noktası 1 ve zaman noktası 2'de izole etmek için iş akışı. (A) Hedef hücreler, barkodlu lentiviral partiküller ile 0.3'lük düşük bir MOI'de enfekte edilir ve 72 saat boyunca seçilir. Hücreler 5-7 popülasyon ikiye katlanması (yaklaşık 7 gün) için geçirilir. Gün 0 ve Gün 7'deki hücre peletleri, genomik DNA izolasyonu için -80 °C'de saklanır. Gün 0, Zaman noktası 1 (T1) ve Gün 7, Zaman noktası 2 (T2) olarak adlandırılır. (B) Genomik DNA izolasyonu, daha sonra iki adet 15 mL'lik tüpe bölünen resüspansiyon tamponunda hücre peletlerinin yeniden süspansiyonu ile başlar. Hücre lizizini kolaylaştırmak için, her tüpe% 10 SDS eklenir ve 30 s açık / 30 s kapalı 25 döngü için sonikleştirilir. Sonikasyonun ardından, fenol / kloroform her tüpe eklenir ve 45-60 s boyunca kuvvetlice girdaplanır. Tüpler daha sonra santrifüjlenir. (C) Berrak bir üst faz pipetlenir ve sodyum asetat/izopropanol ilavesiyle temiz bir tüpe eklenir ve iyice karıştırılır. Tüpler tekrar santrifüj edilir. Bu sefer, süpernatan atılır ve pelet% 70 etanol ilavesiyle yerinden çıkarılır. Yeniden süspanse edilmiş peletleri tek bir tüpte birleştirin ve bir tezgah üstü santrifüjde maksimum hızda döndürün. Süpernatanı atın ve peleti damıtılmış suda tekrar süspanse edin. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
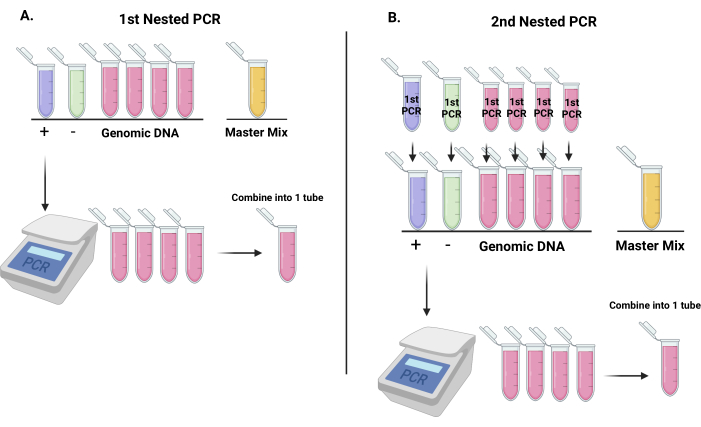
Şekil 3: Yeni nesil dizileme ile barkodların miktarının belirlenmesine hazırlık olarak lentiviral shRNA vektörlerinden barkod dizilerinin çoğaltılması için iş akışı. (A) İlk iç içe PCR reaksiyonu için tüplerin nasıl kurulacağının temsili (7 tüp: biri negatif kontrol için, biri pozitif kontrol için ve kalan 4 tüp genomik DNA için. Son tüp, ana karışım tüpü olarak işlev görecektir). İlk PCR reaksiyonunu takiben, genomik DNA tüplerini tek bir tüpte birleştirin ve karıştırın. (B) İlk iç içe geçmiş PCR reaksiyonundan elde edilen ürünler, ikinci iç içe geçmiş PCR reaksiyonu için şablon görevi görecektir. İkinci PCR'yi takiben, genomik DNA tüplerini tek bir tüpte birleştirin ve karıştırın. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
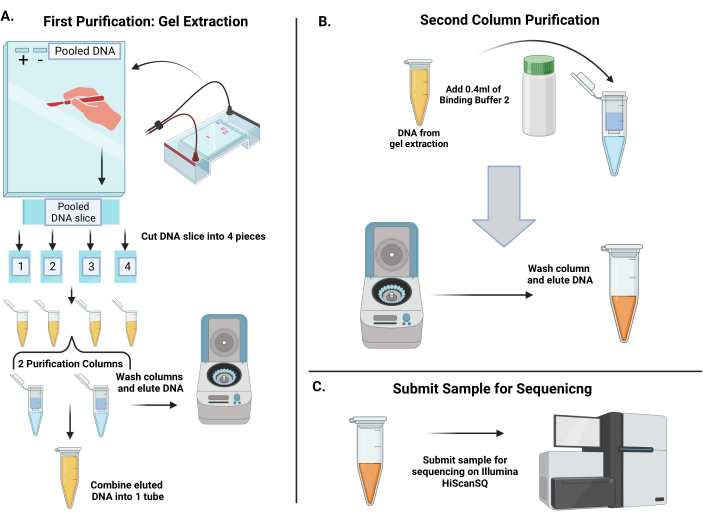
Şekil 4: Amplifiye edilmiş shRNA barkodlarını saflaştırmak ve barkodları zaman noktası 1 ve zaman noktası 2'deki temsillerini ölçmek için barkodları sıralamak için iş akışı . (A)% 3.5 agaroz jeli dökün. Havuzlanan DNA'nın hacmi bir kuyu sınırını aştığından, büyük bir kuyu oluşturmak için bir jel tarağın 4-6 dişini birbirine bantlayın. 6x yükleme boyası ile PCR ürünleri hazırlayın ve pozitif kontrol, negatif kontrol ve havuzlanmış DNA'yı jele yükleyin. Elektroforezi takiben, havuzlanmış DNA şeridinde yaklaşık 250 baz çiftinde büyük bir bant görünmelidir. Temiz bir neşter kullanarak tüm bandı kesin ve ardından 4 jel dilimi halinde kesin. Jel parçalarını çözündürün ve ardından DNA'yı elüte etmek için bunları iki döndürme sütununda birleştirin. Ayrıştırılmış DNA'yı tek bir tüpte birleştirin. (B) DNA, ikinci bir saflaştırma adımıyla saflaştırılır. Havuzlanmış DNA tüpüne Bağlayıcı Tampon 2 ekleyin ve ardından bir döndürme kolonuna pipetleyin. Membranı yıkayın ve ardından DNA'yı damıtılmış suda elüte edin. (C) Saflaştırılmış DNA'yı dizileme çekirdeğine gönderin. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
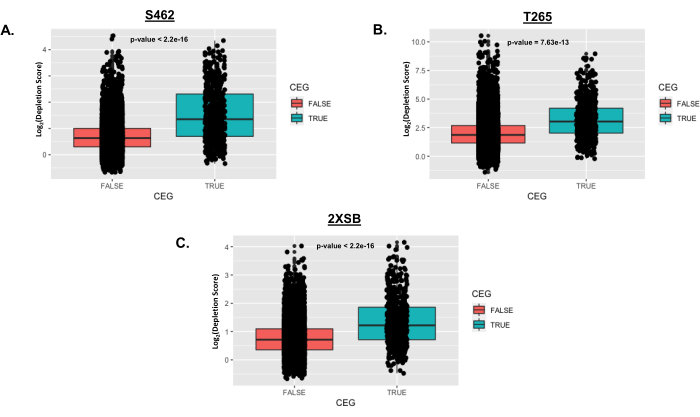
Şekil 5: Protokolde açıklandığı gibi analizden sonra Çekirdek Esansiyel Genlerin dağılımının temsili örnekleri. Bu örnekte, üç insan MPNST hücre hattı (S462, T265 ve 2XSB) hücresi Cellecta DECIPHER shRNA kütüphaneleri ile tarandı. Her insan MPNST hücre hattı için, Temel Temel Genler (CEG; Gerçek kutu grafiği)25 CEG'ler listesinde bulunmayan genlerinkine (Yanlış kutu grafiği). Tek tek veri noktaları, her kutu grafiğinin üzerine katmanlanır. P değerleri, CEG'lerin gen düzeyindeki tükenme skorlarını CEG olmayanlarla karşılaştıran standart bir t-testinden alınmıştır. Kısaltmalar: CEG = çekirdek esansiyel gen; MPNST = Malign Periferik Sinir Kılıfı Tümörü. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
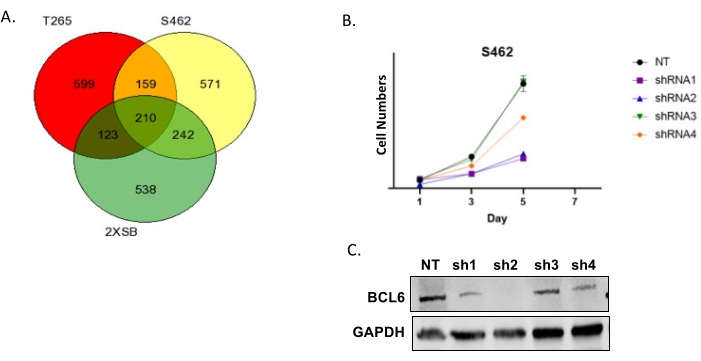
Şekil 6: Ekran sonuçlarının doğrulanması . (A) Üç insan MPNST hücre hattında üst üste binen isabetlerin temsili Venn şeması. (B) Dört farklı BCL6 shRNA'yı (shRNA1, shRNA2, shRNA3 ve shRNA4) eksprese eden hedeflemeyen (NT) bir lentiviral vektör ve lentivirüs ile transdüksiyona tabi tutulan S462 insan MPNST hücreleri. Hücreler lentivirüs ile transdüksiyona tabi tutuldu ve daha sonra 3 gün boyunca bir seçme ajanı (puromisin) ile muamele edildi. Hücre numaraları daha sonra önümüzdeki yedi gün boyunca değerlendirildi. (C) NT, shRNA1, shRNA2, shRNA3 ve shRNA4 lentivirüsü ile transdüksiyonu takiben BCL6 protein seviyelerini gösteren Western blot analizi. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Tablo 1: Lentiviral titre için plaka düzeni. Bu Tabloyu indirmek için lütfen buraya tıklayın.
Tablo 2: İlk PCR reaksiyonlarının ilk kurulumu. Bu Tabloyu indirmek için lütfen buraya tıklayın.
Tablo 3: İlk PCR reaksiyonu için ana karışımın hazırlanması. Bu Tabloyu indirmek için lütfen buraya tıklayın.
Tablo 4: Cellecta ilk PCR parametreleri. Bu Tabloyu indirmek için lütfen buraya tıklayın.
Tablo 5: İkinci PCR reaksiyonunun ilk kurulumu. Bu Tabloyu indirmek için lütfen buraya tıklayın.
Tablo 6: Cellecta ikinci PCR parametreleri. Bu Tabloyu indirmek için lütfen buraya tıklayın.
Tartışmalar
Burada sunulan ayrıntılı yöntemler periferik sinir sistemi neoplazisi ve MPNST patogenezini incelemek için geliştirilmiştir. Bu yöntemlerin etkili olduğunu bulmuş olsak da, burada tanımladığımız yöntemlerin bazı potansiyel sınırlamaları olduğu kabul edilmelidir. Aşağıda, bu sınırlamalardan bazılarını ve diğer model sistemlerde bunların üstesinden gelmek için potansiyel stratejileri tartışıyoruz.
Tüm ekzom diziliminin, P 0-GGFβ3 farelerinde ilgilenilen mutasyonları etkili bir şekilde tanımladığını bulduk. Bununla birlikte, tüm ekzom dizilemesinin kendisinin sınırlamaları olduğu kabul edilmelidir. İlk olarak, tüm ekzom dizilimi, füzyon gen ürünlerini tanımlamak için etkili bir yaklaşım değildir. Bunun nedeni, kromozomal kırılmaların çoğunluğunun ve müteakip füzyonun ağırlıklı olarak intergenik bölgeleri ve intronları içermesidir, çünkü bu bölgeler genomun çoğunluğunu temsil eder. Bunun yerine, eşleştirilmiş uçlu RNA-Seq'in füzyon genlerini çok daha etkili bir şekilde tanımladığını bulduk. Ayrıca, tüm ekzom dizilemesinin nispeten büyük kromozomal kayıp bölgelerini ne kadar etkili bir şekilde tanımladığı sorusu da vardır.
Bu tür kayıpları tanımlamak için çeşitli algoritmalar geliştirilmiş olsa da, "tüm ekzom dizilimi" teriminin kendisi yanıltıcıdır, çünkü ekzomun iyi çalışmalarda bile yakalanması genellikle ekzonik bölgelerin %5-10'unu kaçırır. Bu nedenle, tüm ekzom dizilemeyi rutin olarak dizi karşılaştırmalı genomik hibridizasyon (aCGH) gibi diğer yaklaşımlarla tamamlıyoruz. Kazanç ve kayıpları belirledikten sonra, bu aralıklardaki genleri inceliyoruz ve bunları insan muadilleriyle ilişkili bilinen sürücü mutasyonlarıyla karşılaştırıyoruz. Bununla birlikte, fare genomu insan genomundan daha kararlıdır27. Sonuç olarak, fare tümörleri tipik olarak insan neoplazmlarında görülene benzer kromotripsis göstermez. Fare tümörlerindeki patern bunun yerine çok daha basittir, güçlü seçici basınç altında meydana gelme eğiliminde olan nispeten az sayıda fokal delesyon ile tüm kromozom veya kromozom kazanımlarına veya kayıplarına yönelir22,23.
Genom ölçeğinde shRNA taramaları gerçekleştirirken karşılaştığımız bazı potansiyel tuzaklar var. Karşılaştığımız en yaygın sorunlardan biri, lentiviral vektörlerin hedef hücrelere nispeten zayıf transdüksiyonudur. Çoğu zaman sorunun, paketlenmiş lentiviral havuzların uygun olmayan titresinden kaynaklandığını görüyoruz. Erken geçişli fare tümör hücre kültürleri sınırlayıcı bir kaynak olduğundan, birçok araştırmacı bunun yerine lentivirüslerini daha kolay bulunabilen başka bir yerleşik hücre hattı kullanarak titremeye çalışacaktır. Bununla birlikte, bu yaklaşımla ilgili sorun, lentiviral transdüksiyonun etkinliğinin hücre tipinden hücre tipine önemli ölçüde değişebilmesidir. Bu nedenle, deneyde kullanılacak gerçek hücrelere lentivirüs uygulanmasını öneriyoruz. Nispeten düşük viral titrelerle ilgili sorunlarla da karşılaştık. Bu sorun çoğunlukla paketlenmiş virüs üretilirken 293T hücrelerinin zayıf transfeksiyonunu yansıtır.
Genom ölçeğinde shRNA taramaları gerçekleştirirken yanlış pozitif isabetler elde etmek mümkündür. Bu nedenle, bizi en çok ilgilendiren potansiyel olarak ilaçlanabilir hedefleri belirledikten sonra, shRNA taramalarımızın sonuçlarını her zaman doğrularız. Tipik olarak, yüksek faizli hedefleri doğrulamak için iki farklı yaklaşım kullanacağız. İlk olarak, ilk taramada kullanılanlardan farklı iki veya daha fazla shRNA kullanarak gen ekspresyonunu yıkıyoruz ve bunun tümör hücresi proliferasyonu ve hayatta kalması üzerindeki etkisini belirliyoruz. İkinci olarak, İlaç Gen Etkileşimi Veri Tabanında tanımlanan ilaç(lar)ı elde ediyoruz ve bunun tümör hücresi proliferasyonu ve sağkalımı üzerindeki etkisini belirliyoruz. Her iki yaklaşımı da birlikte kullanıyoruz çünkü shRNA'ların çalıştığı ve ilacın çalışmadığı durumlarla karşılaştık. Bu örneklerin en azından bazılarında, tüm ekzom dizisi veri setinin incelenmesi, hedeflenen proteinin, ilaç-protein etkileşimlerini potansiyel olarak etkileyen bir mutasyona sahip bir gen tarafından üretildiğini göstermiştir.
Yukarıda özetlenen yaklaşımlar, araştırmacıya nadir neoplazmlarda meydana gelen potansiyel sürücü mutasyonları tanımlamak, proliferasyon ve hayatta kalma için gerekli sinyal yollarını işlevsel olarak tanımlamak ve terapötik gelişim için hedeflere öncelik vermek için uygulanabilir bir araç sağlayacaktır. Diğer araştırmacıların bu yaklaşımları diğer insan kanserlerinde anahtar terapötik hedefleri belirlemek için yararlı bulacağını umuyoruz. Bununla birlikte, okuyucu, tümör patogenezinde yer alan genleri ve potansiyel terapötik hedefleri kodlayan genleri tanımlamak için kullanılabilecek başka fonksiyonel genomik yaklaşımlar olduğunun farkında olmalıdır. Örnek olarak, shRNA kütüphaneleri için tanımladığımıza benzer bir şekilde kullanılabilen CRISPR kütüphaneleri mevcuttur. Fonksiyonel taramalar, tümör oluşumunu destekleyen genleri tanımlamak için in vivo olarak da gerçekleştirilebilir. Bunun bir örneği olarak, Uyuyan Güzel transpozon tabanlı somatik mutajenez sistemi daha önce Schwann hücrelerini ve öncüllerini hedeflemek için kullanılmış ve MPNST patogenezinde yer alan birkaç yüz genin tanımlanmasına neden olmuştur28. Bu sistemler fonksiyonel genomiklere farklı şekillerde yaklaştıklarından, araştırmacının planlanan deneylerinin hedeflerini dikkatlice değerlendirmesini ve fonksiyonel genomik metodoloji seçimini bu hedeflere dayandırmasını öneririz.
Açıklamalar
Yazarların ifşa edecek herhangi bir çıkar çatışması yoktur.
Teşekkürler
Bu çalışma, Ulusal Nörolojik Hastalıklar ve İnme Enstitüsü'nden (R01 NS048353 ve R01 NS109655 SLC'ye hibelerle desteklenmiştir; R01 NS109655-03S1'den D.P.J.'ye), Ulusal Kanser Enstitüsü (R01 CA122804'den SLC'ye) ve Savunma Bakanlığı (X81XWH-09-1-0086 ve W81XWH-12-1-0164'ten SLC'ye).
Malzemeler
| Name | Company | Catalog Number | Comments |
| Bioruptor Sonication System | Diagenode | UCD-600 | |
| CASAVA 1.8.2 | |||
| Cbot | Illumina, San Diego, CA | N/A | |
| Celigo Image Cytometer | Nexcelom | N/A | |
| Cellecta Barcode Analyzer and Deconvoluter software | |||
| Citrisolve Hybrid | Decon Laboratories | 5989-27-5 | |
| Corning 96-well Black Microplate | Millipore Sigma | CLS3603 | |
| Diagenode Bioruptor 15ml conical tubes | Diagenode | C30010009 | |
| dNTP mix | Clontech | 639210 | |
| Eosin Y | Thermo Scientific | 7111 | |
| Elution buffer | Qiagen | 19086 | |
| Ethanol (200 Proof) | Decon Laboratories | 2716 | |
| Excel | Microsoft | ||
| FWDGEX 5’-CAAGCAGAAGACGGCATACGAGA-3’ | |||
| FWDHTS 5’-TTCTCTGGCAAGCAAAAGACGGCATA-3’ | |||
| GexSeqS (5’ AGAGGTTCAGAGTTCTACAGTCCGAA-3’ | HPLC purified | ||
| GraphPad Prism | Dotmatics | ||
| Harris Hematoxylin | Fisherbrand | 245-677 | |
| Illumina HiScanSQ | Illumina, San Diego, CA | N/A | |
| Paraformaldehyde (4%) | Thermo Scientific | J19943-K2 | |
| PLUS Transfection Reagent | Thermo Scientific | 11514015 | |
| Polybrene Transfection Reagent | Millipore Sigma | TR1003G | |
| PureLink Quick PCR Purification Kit | Invitrogen | K310001 | |
| Qiagen Buffer P1 | Qiagen | 19051 | |
| Qiagen Gel Extraction Kit | Qiagen | 28704 | |
| RevGEX 5’-AATGATACGGCGACCACCGAGA-3’ | |||
| RevHTS1 5’-TAGCCAACGCATCGCACAAGCCA-3’ | |||
| Titanium Taq polymerase | Clontech | 639210 | |
| Trimmomatic software | www.usadellab.org |
Referanslar
- Carroll, S. L. Molecular mechanisms promoting the pathogenesis of Schwann cell neoplasms. Acta Neuropathol. 123 (3), 321-348 (2012).
- Longo, J. F., Weber, S. M., Turner-Ivey, B. P., Carroll, S. L. Recent Advances in the Diagnosis and Pathogenesis of Neurofibromatosis Type 1 (NF1)-associated Peripheral Nervous System Neoplasms. Adv Anat Pathol. 25 (5), 353-368 (2018).
- Longo, J. F., Carroll, S. L. The RASopathies: Biology, genetics and therapeutic options. Adv Cancer Res. 153, 305-341 (2022).
- Birindelli, S., et al. Rb and TP53 pathway alterations in sporadic and NF1-related malignant peripheral nerve sheath tumors. Lab Invest. 81 (6), 833-844 (2001).
- Legius, E., et al. TP53 mutations are frequent in malignant NF1 tumors. Genes Chromosomes Cancer. 10 (4), 250-255 (1994).
- Menon, A. G., et al. Chromosome 17p deletions and p53 gene mutations associated with the formation of malignant neurofibrosarcomas in von Recklinghausen neurofibromatosis. Proc Natl Acad Sci U S A. 87 (14), 5435-5439 (1990).
- Upadhyaya, M., et al. Germline and somatic NF1 gene mutation spectrum in NF1-associated malignant peripheral nerve sheath tumors (MPNSTs). Hum Mutat. 29 (1), 74-82 (2008).
- Kourea, H. P., Orlow, I., Scheithauer, B. W., Cordon-Cardo, C., Woodruff, J. M. Deletions of the INK4A gene occur in malignant peripheral nerve sheath tumors but not in neurofibromas. Am J Pathol. 155 (6), 1855-1860 (1999).
- Nielsen, G. P., et al. Malignant transformation of neurofibromas in neurofibromatosis 1 is associated with CDKN2A/p16 inactivation. Am J Pathol. 155 (6), 1879-1884 (1999).
- Gregorian, C., et al. PTEN dosage is essential for neurofibroma development and malignant transformation. Proc Natl Acad Sci U S A. 106 (46), 19479-19484 (2009).
- Lee, W., et al. PRC2 is recurrently inactivated through EED or SUZ12 loss in malignant peripheral nerve sheath tumors. Nat Genet. 46 (11), 1227-1232 (2014).
- Zhang, M., et al. Somatic mutations of SUZ12 in malignant peripheral nerve sheath tumors. Nat Genet. 46 (11), 1170-1172 (2014).
- Varela, I., et al. Somatic structural rearrangements in genetically engineered mouse mammary tumors. Genome Biol. 11 (10), 100 (2010).
- Johnson, R. A., et al. Cross-species genomics matches driver mutations and cell compartments to model ependymoma. Nature. 466 (7306), 632-636 (2010).
- Kim, M., et al. Comparative oncogenomics identifies NEDD9 as a melanoma metastasis gene. Cell. 125 (7), 1269-1281 (2006).
- Zender, L., et al. Identification and validation of oncogenes in liver cancer using an integrative oncogenomic approach. Cell. 125 (7), 1253-1267 (2006).
- Uren, A. G., et al. Large-scale mutagenesis in p19(ARF)- and p53-deficient mice identifies cancer genes and their collaborative networks. Cell. 133 (4), 727-741 (2008).
- Starr, T. K., et al. A transposon-based genetic screen in mice identifies genes altered in colorectal cancer. Science. 323 (5922), 1747-1750 (2009).
- Dupuy, A. J., et al. A modified sleeping beauty transposon system that can be used to model a wide variety of human cancers in mice. Cancer Res. 69 (20), 8150-8156 (2009).
- Carroll, S. L. The Challenge of Cancer Genomics in Rare Nervous System Neoplasms: Malignant Peripheral Nerve Sheath Tumors as a Paradigm for Cross-Species Comparative Oncogenomics. Am J Pathol. 186 (3), 464-477 (2016).
- Huijbregts, R. P., Roth, K. A., Schmidt, R. E., Carroll, S. L. Hypertrophic neuropathies and malignant peripheral nerve sheath tumors in transgenic mice overexpressing glial growth factor beta3 in myelinating Schwann cells. J Neurosci. 23 (19), 7269-7280 (2003).
- Kazmi, S. J., et al. Transgenic mice overexpressing neuregulin-1 model neurofibroma-malignant peripheral nerve sheath tumor progression and implicate specific chromosomal copy number variations in tumorigenesis. Am J Pathol. 182 (3), 646-667 (2013).
- Brosius, S. N., et al. Neuregulin-1 overexpression and Trp53 haploinsufficiency cooperatively promote de novo malignant peripheral nerve sheath tumor pathogenesis. Acta Neuropathol. 127 (4), 573-591 (2014).
- Hart, T., Brown, K. R., Sircoulomb, F., Rottapel, R., Moffat, J. Measuring error rates in genomic perturbation screens: gold standards for human functional genomics. Mol Syst Biol. 10 (7), 733 (2014).
- Hart, T., et al. Evaluation and Design of Genome-Wide CRISPR/SpCas9 Knockout Screens. G3. 7 (8), 2719-2727 (2017).
- Longo, J. F., et al. Establishment and genomic characterization of a sporadic malignant peripheral nerve sheath tumor cell line. Sci Rep. 11 (1), 5690 (2021).
- Maser, R. S., et al. Chromosomally unstable mouse tumours have genomic alterations similar to diverse human cancers. Nature. 447 (7147), 966-971 (2007).
- Rahrmann, E. P., et al. Forward genetic screen for malignant peripheral nerve sheath tumor formation identifies new genes and pathways driving tumorigenesis. Nat Genet. 45 (7), 756-766 (2013).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır