Method Article
التنميط الجيني وفحص التسرب على نطاق الجينوم لتحديد الأهداف العلاجية في نماذج الفئران لورم غمد العصب المحيطي الخبيث
In This Article
Summary
لقد طورنا نهجا مقارنا لعلم الأورام الجيني عبر الأنواع باستخدام التحليلات الجينومية والشاشات الجينومية الوظيفية لتحديد ومقارنة الأهداف العلاجية في الأورام الناشئة في نماذج الفئران المعدلة وراثيا ونوع الورم البشري المقابل.
Abstract
أورام غمد الأعصاب المحيطية الخبيثة (MPNSTs) مشتقة من خلايا شوان أو سلائفها. في المرضى الذين يعانون من متلازمة حساسية الورم الورم العصبي الليفي من النوع 1 (NF1) ، تعد MPNSTs أكثر الأورام الخبيثة شيوعا والسبب الرئيسي للوفاة. توفر ساركوما الأنسجة الرخوة النادرة والعدوانية هذه مستقبلا صارخا ، مع معدلات بقاء خالية من الأمراض لمدة 5 سنوات تتراوح بين 34-60٪. خيارات العلاج للأفراد الذين يعانون من MPNSTs محدودة بشكل مخيب للآمال ، مع كون الجراحة المشوهة هي خيار العلاج الأول. العديد من العلاجات التي كانت واعدة في يوم من الأيام مثل tipifarnib ، وهو مثبط لإشارات Ras ، فشلت سريريا. وبالمثل ، فشلت التجارب السريرية للمرحلة الثانية مع erlotinib ، الذي يستهدف عامل نمو البشرة (EFGR) ، و sorafenib ، الذي يستهدف مستقبل عامل نمو بطانة الأوعية الدموية (VEGF) ، ومستقبلات عامل النمو المشتق من الصفائح الدموية (PDGF) ، و Raf ، بالاشتراك مع العلاج الكيميائي القياسي ، في إنتاج استجابة في المرضى.
في السنوات الأخيرة ، أثبتت طرق الفحص الجينومي الوظيفي جنبا إلى جنب مع التنميط الجيني لخطوط الخلايا السرطانية أنها مفيدة لتحديد مسارات الإشارات السيتوبلازمية الأساسية وتطوير علاجات محددة الهدف. في حالة أنواع الأورام النادرة ، يتم استخدام شكل مختلف من هذا النهج المعروف باسم علم الأورام الجيني المقارن عبر الأنواع بشكل متزايد لتحديد أهداف علاجية جديدة. في علم الأورام الجيني المقارن عبر الأنواع ، يتم إجراء التنميط الجيني وعلم الجينوم الوظيفي في نماذج الفئران المعدلة وراثيا (GEM) ثم يتم التحقق من صحة النتائج في العينات البشرية النادرة وخطوط الخلايا المتاحة.
تصف هذه الورقة كيفية تحديد طفرات الجينات الدافعة المرشحة في خلايا MPNST البشرية والفأر باستخدام تسلسل الإكسوم الكامل (WES). ثم نصف كيفية إجراء شاشات shRNA على نطاق الجينوم لتحديد ومقارنة مسارات الإشارات الحرجة في خلايا MPNST للفأر والإنسان وتحديد الأهداف القابلة للدواء في هذه المسارات. توفر هذه المنهجيات نهجا فعالا لتحديد أهداف علاجية جديدة في مجموعة متنوعة من أنواع السرطان البشري.
Introduction
أورام غمد الأعصاب المحيطية الخبيثة (MPNSTs) هي أورام خلايا المغزل شديدة العدوانية التي تنشأ بالاقتران مع متلازمة حساسية الورم العصبي الليفي من النوع 1 (NF1) ، بشكل متقطع في عموم السكان وفي مواقع العلاج الإشعاعي السابق1،2،3. يولد مرضى NF1 بنسخة من النوع البري من جين مثبط الورم NF1 وأليل NF1 ثان مع طفرة فقدان الوظيفة. هذه الحالة من عدم كفاية الفرداني تجعل مرضى NF1 عرضة لطفرة ثانية في فقدان الوظيفة في جين NF1 من النوع البري ، مما يؤدي إلى تكوين الأورام. عندما تحدث طفرة NF1 "الضربة الثانية" هذه في خلية في سلالة خلايا شوان ، يكون الورم الناتج إما ورم ليفي عصبي جلدي ينشأ في الجلد أو ورم ليفي عصبي ضفيري يتطور في الأعصاب الكبيرة أو الضفائر العصبية. على الرغم من أن أمراض الأورام الليفية العصبية الجلدية والضفيرية متطابقة ، إلا أن سلوكها البيولوجي مختلف تماما - على الرغم من أن كل من الأورام الليفية العصبية الجلدية والضفيرية حميدة ، إلا أن الأورام الليفية العصبية الضفيرية فقط هي التي يمكن أن تخضع للتحول وتؤدي إلى MPNSTs. بالإضافة إلى فقدان neurofibromin ، البروتين المنشط Ras GTPase المشفر بواسطة جين NF1 ، تحمل MPNSTs طفرات في العديد من الجينات الكابتة للورم الأخرى ، بما في ذلك TP534،5،6،7 و CDKN2A8،9 و PTEN 10 ، طفرات الجينات التي تشفر مكونات مجمع polycomb القمعي 2 11,12 (PRC2 ؛ ال SUZ12 وجينات EED) والتعبير الشاذ عن مستقبلات التيروزينكينازات 1،2. طفرات NF1 والجينات الأخرى المذكورة أعلاه موجودة أيضا في MPNSTs المتفرقة والناجمة عن الإشعاع11,12.
في حين أن هذه التطورات في فهمنا للتشوهات الجينومية في MPNSTs كانت لا تقدر بثمن لفهم التسبب في المرض ، إلا أنها لم تسفر بعد عن تطوير علاجات جديدة فعالة ل MPNSTs. أحد العوائق الرئيسية التي تعوق تطوير علاجات جديدة هو حقيقة أن MPNSTs هي سرطانات نادرة. ولهذا السبب، من الصعب الحصول على العدد الكبير من عينات المرضى المطلوبة للتحليلات العالمية التي تحدد طفرات المحرك الرئيسية مثل تلك التي يقوم بها أطلس جينوم السرطان (TCGA). في تجربتنا ، قد يستغرق تجميع حتى عدد متواضع من عينات MPNST البشرية سنوات. للتغلب على هذه القيود ، تحول العديد من الباحثين الذين يدرسون أنواعا أخرى من السرطان النادر إلى استخدام علم الأورام الجيني المقارن عبر الأنواع لتحديد الطفرات الجينية الأساسية للمحرك ، وتحديد مسارات الإشارات السيتوبلازمية الأساسية في الورم محل الاهتمام ، وتحديد أهداف علاجية جديدة. نظرا لأن مسارات الإشارات الضرورية لتكوين الأورام محفوظة بشكل كبير بين البشر وأنواع الفقاريات الأخرى ، فإن تطبيق مناهج الجينوم الوظيفي مثل شاشات shRNA على نطاق الجينوم يمكن أن يكون وسيلة فعالة لتحديد هذه الطفرات الدافعة الجديدة ، ومسارات الإشارات ، والأهداف العلاجية 13،14،15،16،17،18،19، خاصة عند دراسة أنواع الأورام البشرية النادرة المتوفرة في أعداد محدودة20.
في المنهجيات المقدمة هنا ، نصف هذا النهج لأداء التنميط الجينومي في خطوط خلايا MPNST البشرية ومزارع MPNST المبكرة المشتقة من الفئران P 0-GGFβ3 ، وهو نموذج فأر معدل وراثيا (GEM) حيث يعزز التعبير المفرط لخلية شوان لعامل النمو neuregulin-1 (NRG1) التسبب في الأورام الليفية العصبية الضفيرية وتطورها اللاحق إلى MPNSTs21 ، 22,23. تتمثل الخطوة الأولى في هذا النهج في تحديد جينات المحرك المرشحة في P 0-GGFβ3 MPNSTs ، وخطوط خلايا MPNST البشرية ، و MPNSTs البشرية التي تم استئصالها جراحيا. للتحقق وظيفيا من مسارات الإشارات المتأثرة بهذه الطفرات ، نستخدم بعد ذلك شاشات shRNA على نطاق الجينوم لتحديد الجينات المطلوبة للتكاثر والبقاء في خطوط خلايا MPNST البشرية والفئران. بعد تحديد الجينات المطلوبة للتكاثر والبقاء على قيد الحياة ، نحدد بعد ذلك منتجات الجينات القابلة للدواء ضمن مجموعة "الزيارات" باستخدام قاعدة بيانات التفاعل الجيني الدوائي. نقارن أيضا "الزيارات" في خلايا MPNST البشرية والفأر ، لتحديد ما إذا كان نموذج GEM و MPNSTs البشرية يظهران اعتمادا مشابها على نفس الجينات ومسارات الإشارة. يعمل تحديد التداخلات في الجينات المطلوبة للتكاثر والبقاء ومسارات الإشارات المتأثرة كوسيلة للتحقق من صحة نموذج الماوس P 0-GGFβ3 على المستوى الجزيئي. يؤكد هذا النهج أيضا على فعالية الجمع بين شاشات الإنسان والماوس لتحديد أهداف علاجية جديدة ، حيث يمكن أن يكون نموذج الماوس بمثابة مكمل للشاشات البشرية. تتضح قيمة هذا النهج عبر الأنواع بشكل خاص عند البحث عن أهداف علاجية في الأورام النادرة ، حيث يصعب الحصول على الأورام البشرية وخطوط الخلايا.
Protocol
قبل الشروع في الدراسات ، تمت مراجعة الإجراءات والبروتوكولات الحيوانية للتعامل مع النواقل الفيروسية والموافقة عليها من قبل اللجنة المؤسسية لرعاية واستخدام (IACUC) واللجنة المؤسسية للسلامة الأحيائية (IBC). تمت الموافقة على الإجراءات الموضحة هنا من قبل مجالس IACUC و IBC بجامعة ساوث كارولينا الطبية وتم تنفيذها من قبل موظفين مدربين تدريبا مناسبا وفقا لدليل المعاهد الوطنية للصحة لرعاية واستخدام المختبر وإرشادات رعاية المؤسسية الخاصة ب MUSC.
1. تحليلات WES-Seq وتحديد المتغيرات المسببة للأمراض
- عزل الحمض النووي الجينومي من عينة الورم أو خلايا MPNST الفرعية (70٪) المزروعة على طبق 60 مم ، باستخدام الطرق القياسية القائمة على هلام السيليكا الامتزاز المتاحة تجاريا (استشر بروتوكول الشركات المصنعة للحصول على خطوات مفصلة). يتم رسم سير العمل العام في الشكل 1.
- تقديم ما لا يقل عن 10 ميكرولتر من الحمض النووي الجينومي عند 50 نانوغرام / ميكرولتر إلى قلب التسلسل ما لم يتم تحديد كميات أو تركيزات مختلفة من الحمض النووي الجينومي.
- يقوم المرفق الأساسي بتفتيت الحمض النووي الجينومي عن طريق الصوتنة ثم تنقيته باستخدام الطريقة المفضلة. يتم تنفيذ التقاط الإكسوم وبناء المكتبة باستخدام مجموعة تسلسل الإكسوم المفضلة ويتم إضافة علامات الفهرس إلى الإكسوم المضخم الملتقط.
- أرسل العينات إلى قلب التسلسل لإجراء تسلسل كامل للإكسوم المزدوج (WES ؛ تسلسل 100 نقطة أساس من كل طرف).
- يتم توفير ملفات FASTQ التي تم إنشاؤها بواسطة النواة إلى المحقق. استخدم فقط ملفات FASTQ التي تجتاز مقاييس الجودة للتحليل.
- قم بمحاذاة وتحليل ملفات FASTQ باستخدام البرامج المتاحة تجاريا (مثل DNAStar19 أو 20 أو Partek21 أو Varsome). قم بمحاذاة ملفات FASTQ مع الجينوم المرجعي للماوس GRCm38 / mm10 باستخدام الإعدادات الافتراضية.
ملاحظة: باستخدام DNAStar وعينة MPNST الماوس كمثال، يتم رسم سير العمل العام في الشكل 1 وشرح بإيجاز أدناه.- افتح برنامج DNAStar واختر سير عمل SeqMan NGen.
- اختر سير العمل عن طريق تحديد تحليل المتغير/إعادة التسلسل ونوع تحليل التسلسل NGS المستند إلى Amplicon أو لوحة الجينات أو الإكسوم. انقر فوق التالي.
- اختر التسلسل المرجعي المفضل عن طريق تحديد تنزيل حزمة الجينوم والجينوم المرجعي المناسب (أي Mus_musculus-GRCm38-dbSNP146.zip أو Homo sapien-GRCh37.p13.zip). إذا قدم المرفق الأساسي ملف سرير ، فقم بتحميل هذا الملف الإضافي. انقر فوق التالي.
- اختر تسلسلات الإدخال عن طريق تحديد تقنية التسلسل المناسبة، Illumina، وقم بتعيين أن قراءات التسلسل مقترنة. بعد ذلك، حدد إعداد التجربة والعينات المتعددة وقم بتحميل ملفات FASTQ المتسلسلة عن طريق تحديد إضافة. في حالة تشغيل عينات متعددة ، قم بتعيين كل مجموعة نهاية مزدوجة بتجربة فريدة أو اسم عينة الورم أو خط الخلية أو A18 ، A202 ... انقر فوق التالي.
- قم بتعيين مجموعة بيانات عنصر التحكم إذا كان هناك واحد. انقر فوق التالي وانقر فوق التالي مرة أخرى ضمن خيارات التجميع. ضمن خيارات التحليل، انقر فوق وضع الكشف عن المتغير المناسب ك Diploid، ثم انقر فوق التالي. ضمن إخراج التجميع ، قم بتسمية وتعيين موقع حفظ الملف للمشروع ؛ انقر فوق التالي. قم بتشغيل التجميع على الكمبيوتر المحلي أو السحابة.
ملاحظة: يمكن فتح ملفات المحاذاة الناتجة في سير عمل ArrayStar للتعليق التوضيحي المتغير لتعدد أشكال النوكليوتيدات المفردة المكتشفة (SNPs). يكتشف سير العمل هذا SNPs ، وليس مكاسب أو خسائر رقم نسخة الجينات. يكتشف تحليل منفصل يسمى صفيف SNP عالي الكثافة تغييرات رقم النسخ ؛ لا يكتشف تسلسل الإكسوم الكامل تغييرات رقم النسخ بشكل موثوق. راجع دليل مستخدم البرنامج لمعرفة الخطوات المحددة التي يجب تنفيذها باستخدام برنامج المحاذاة. - قم بتطبيق عوامل التصفية المعرفة من قبل المستخدم على التعليق التوضيحي المتغير ل SNPs المكتشفة لتضمين بيانات معينة أو استبعادها. للحصول على قائمة مكثفة بالمتغيرات المحتملة ذات الصلة وظيفيا ، قم بتطبيق عوامل التصفية التالية على مجموعات بيانات المتغيرات في هذا التسلسل الهرمي: ليس تحت السيطرة (في حالة توفر عينة تحكم عادية) ، تردد السكان (gnomAD ، ExAC ، ترددات 1000 جينوم) ، تردد الأليل (بما في ذلك ≤0.001 أو 0.1٪) ، عمق التغطية (باستثناء <عمق 10) ، الإمراضية أو فئة ClinVar (تشمل: مسببة للأمراض ، من المحتمل أن تكون مسببة للأمراض ؛ استبعاد: غير مؤكدة ، من المحتمل أن تكون حميدة ، حميدة) ، وإذا رغبت في ذلك نوع SNP وتأثير الترميز (تشمل: غير مترادف ، وسوء المعنى ، وهراء ، وإزاحة الإطار ، وداخل الإطار ، والربط).
ملاحظة: يمكن أيضا تطبيق قائمة جينات السرطان المعروفة ذات الصلة على القائمة النهائية لسحب جينات محددة ذات صلة بالمرض فقط ، انظر الخطوة 1.3.7. يمكن لهذه المرشحات تقليل قائمة الجينات المتغيرة إلى أقل من 20 جينا.- استخدم تردد الأليل المتغير لتصفية خطأ التسلسل SNPs. سيتم تمثيل المتغيرات متماثلة الزيجوت وغير المتجانسة بنسبة 100٪ و 50٪ تقريبا على التوالي لمجموعة خلايا نقية غير ملوثة (يجب أن يمثل خط الخلية المشتق من الورم مجموعة واحدة من الخلايا السرطانية متساوية المنشأ). في هذه الحالة ، قم بتطبيق ترددات الأليل 100-90٪ و 50-40٪ للمتغيرات كمرشح ، وبالتالي إزالة أي متغيرات أقل من ذلك. إذا كانت بيانات صفيف SNP عالية الكثافة متاحة أيضا لنفس مجموعة البيانات ، فقم بتطبيق كسب أو فقدان رقم النسخ على SNPs ذات ترددات أليل متغيرة أقل من نسب متماثلة الزيجوت أو غير متجانسة الزيجوت (على سبيل المثال ، كسب رقم النسخ الناتج عن نسختين من الأليل A ونسخة 1 من الأليل B ستجعل ترددات الأليل المتغيرة المناسبة 75٪ ، 25٪ ، على التوالي).
- بعد تحديد المتغيرات المسببة للأمراض باستخدام ArrayStar ، قم بتصدير وحفظ قائمة الجينات المتغيرة كملف csv أو txt أو xls وقارن الجينات التي تحتوي على هذه الطفرات بتلك الموجودة في مجموعة الأورام التي تم إنشاؤها بواسطة P 0-GGFβ3 لتحديد قوائم الجينات الطافرة المتداخلة والفريدة. قارن الجينات الطافرة بالجينات الطافرة المعروفة المرتبطة بنظيراتها البشرية (مثل قائمة جينات السرطان في مختبر بوشمان أو قائمة برعاية المستخدم).
- اختياري ، قبل تطبيق أي عوامل تصفية معرفة من قبل المستخدم (1.3.6) ، قم بتصدير وحفظ الملف المشروح كملف VCF.
ملاحظة: يمكن تحميل هذا الملف VCF إلى برنامج تنبؤ مستجيب متغير آخر Varsome أو VEP للمقارنة كتحليل ثانوي. يمكن أيضا إجراء التحليل البيولوجي والمسار على قوائم الجينات المتغيرة المفلترة.
- اختياري ، قبل تطبيق أي عوامل تصفية معرفة من قبل المستخدم (1.3.6) ، قم بتصدير وحفظ الملف المشروح كملف VCF.
- إجراء تصنيف وظيفي لقائمة الجينات المتغيرة عبر قاعدة بيانات مفضلة لدى المستخدم للحصول على فئة البروتين والمسار ومكون المسار وعائلة الجينات ومعلومات الأنطولوجيا.
ملاحظة: يستخدم النمر (pantherdp.org) كمثال هنا.- تحميل قائمة الجينات التي تمت تصفيتها باستخدام معرف الجين.
- حدد الكائن الحي (Mus musculus).
- حدد التحليل (التصنيف الوظيفي المعروض في قائمة الجينات) وانقر فوق إرسال التحليل البيولوجي والمسار على قوائم الجينات المتغيرة التي تمت تصفيتها.
2. شاشات shRNA على نطاق الجينوم
ملاحظة: تتوفر العديد من مكتبات shRNA و CRISPR التي يمكن استخدامها للشاشات الوظيفية على نطاق الجينوم مع مزارع الأورام ذات المرور المنخفض. هنا ، نصف استخدام مكتبات CELLECTA DECIPHER shRNA كمثال. تم تحسين مكتبات CELLECTA Decipher shRNA عدسية الفيروسات لشاشات RNAi الجينية بتنسيق مجمع. يتم استهداف كل نسخة بواسطة ما لا يقل عن 5-6 shRNAs فريدة ويحتوي كل ناقل shRNA عدسي الفيروس على رمز شريطي جيني فريد محاط بمواقع أولية ل PCR. تغطي هذه المكتبات غالبية الجينات المرتبطة بأمراض الإنسان والفئران ولكنها لا تغطي جميع الجينات في الجينوم. تتوفر تجمعات الحمض النووي البلازميد لمكتبة Cellecta البشرية في ثلاث وحدات (الوحدة البشرية الأولى والثانية والثالثة ؛ تستهدف 15,377 جينا) بينما تتوفر تجمعات البلازميد في مكتبة الفئران في وحدتين (وحدات الماوس الأولى والثانية ؛ تستهدف 9,145 جينا). تستخدم هذه المكتبات لإجراء فحوصات "التسرب" التي يتم فيها التعبير عن الجينات المستهدفة المطلوبة للانتشار و / أو البقاء على قيد الحياة بشكل تفاضلي في نقاط زمنية مختلفة بعد نقل الفيروس.
- التغليف الفيروسي
- اليوم 0: طبق 10 أطباق من 10 ملايين خلية 293 تائية / طبق 15 سم في 30 مل / طبق DMEM خال من المضادات الحيوية يحتوي على 10٪ مصل بقري جنيني (FBS).
- اليوم 1: تأكد من أن الخلايا ~ 80٪ متقاربة في اليوم التالي وجاهزة للنقل. في أنبوب مخروطي سعة 50 مل ، امزج ما يلي بهذا الترتيب: 600 ميكرولتر من مزيج البلازميد للتغليف (0.5 ميكروغرام / ميكرولتر) ، 60 ميكرولتر من مكتبة الباركود البلازميد ، 12 مل من DMEM (بدون مصل أو مضادات حيوية) ، و 600 ميكرولتر من كاشف النقل. احتضان في درجة حرارة الغرفة لمدة 15 دقيقة.
- ضع 900 ميكرولتر من كاشف النقل و 12 مل DMEM في أنبوب مخروطي منفصل سعة 15 مل واخلطه بالدوامة. أضف 12.9 مل من مزيج كاشف النقل / DMEM إلى مزيج الحمض النووي وانقر للخلط. احتضان في درجة حرارة الغرفة لمدة 15 دقيقة دون مزيد من الخلط. أضف 2.5 مل من هذا المزيج ، قطرة ، إلى كل طبق 15 سم من 293 خلية تائية واحتضانها طوال الليل في حاضنة زراعة الأنسجة.
- اليوم 2: استبدل الوسائط في اليوم التالي بوسائط نمو منتظمة تحتوي على مضادات حيوية.
- اليوم 3: حصاد الفيروس عن طريق جمع الوسائط وتمريرها عبر وحدة ترشيح 0.2 ميكرومتر وتحويلها إلى أنابيب مخروطية سعة 15 مل ؛ أيضا إعداد خمسة 1 مل من حصص الفيروس المصفى في cryovials لاستخدامها في المعايرة الفيروسية (يعتبر فيروس 48 ساعة) ؛ قم بتخزين الفيروس في مجمد بدرجة حرارة -80 درجة مئوية. استبدل الوسائط ب 30 مل من وسائط النمو على الألواح واحدة تلو الأخرى لتجنب جفاف الخلايا.
- اليوم 4: حصاد الفيروس عن طريق جمع الوسائط وتمريرها عبر وحدة ترشيح 0.2 ميكرومتر. فيروس القسمة إلى أنابيب مخروطية سعة 15 مل. هذا يعتبر فيروس 72 ساعة. قم بتخزين الفيروس في مجمد بدرجة حرارة -80 درجة مئوية.
- برك عيار عدسي الفيروسات
- أضف 65 ميكرولتر من البوليمر الكاتيوني (10 مجم / مل) إلى 65 مل من وسائط نمو الخلايا السرطانية. ماصة 1 مل / بئر من الوسط المحتوي على البوليمر إلى إحدى عشرة لوحة زراعة أنسجة 6 آبار. التربسين في الخلايا السرطانية المارة مبكرا وعد الخلايا باستخدام الطريقة المفضلة بحيث يتلقى كل بئر 50000 خلية / مل / بئر. قم بإذابة 1 مل من قسامات فيروس lentivirus لمدة 48 ساعة من الفريزر في حمام مائي بدرجة حرارة 37 درجة مئوية.
- لكل وحدة فيروسية ، قم بإعداد العدوى كما هو موضح في الجدول 1.
- ضع جميع لوحات 6 آبار في حاضنة زراعة الأنسجة. في اليوم التالي ، قم بإزالة الوسائط الفيروسية وتجديدها بوسائط نمو جديدة. في 48 ساعة ، أضف الوسائط المحتوية على البوروميسين إلى جميع الآبار التي تتلقى الاختيار. قبل أن تتداخل خلايا التحكم ، قم بإجراء عدد الخلايا للنسخ المستنسخة الباقية باستخدام الطريقة المفضلة.
ملاحظة: يجب تحديد تركيز البوروميسين المطلوب لقتل الخلايا غير المنقولة مسبقا تجريبيا عن طريق اختبار مجموعة من التركيزات في منحنى "القتل". استخدم أقل تركيز يؤدي بشكل موحد إلى موت الثقافات غير المتحولة.
- عدوى فيروس العدسة للخلايا المستهدفة
- التربسين خلايا MPNST والعد. طبق 2.5 مليون خلية لكل طبق 15 سم ليصبح المجموع عشرين طبق 15 سم لكل وحدة. إذابة الفيروس وتحويل الخلايا مع الفيروس عند MOI من 0.5 في وجود 5 ميكروغرام / مل من البوليمر الموجبة (الشكل 2A).
- قم بإزالة الوسائط التي تحتوي على الفيروسات في صباح اليوم التالي واستبدلها بوسائط نمو جديدة. خلايا الثقافة لمدة 2 أيام إضافية للسماح بالتعبير عن علامة الاختيار ثم إضافة puromycin إلى الثقافات للقضاء على الخلايا غير المحولة.
- بعد ثلاثة أيام من إضافة البوروميسين ، يقوم التربسين بنصف الخلايا وأجهزة الطرد المركزي عند 200 × جم لمدة 5 دقائق في جهاز طرد مركزي منضدي. تخزين الخلايا المحببة في الثلاجة -80 درجة مئوية لإعداد الحمض النووي الجينومي في المستقبل ؛ هذه هي النقطة الزمنية المرجعية ، أو النقطة الزمنية 1 (T1).
- إعادة طلاء النصف الآخر من عدد الخلايا وتنميتها لمضاعفة عدد السكان تقريبا قبل الحصاد والطرد المركزي على النحو الوارد أعلاه. ستكون هذه الحبيبات الخلوية بمثابة النقطة الزمنية النهائية (النقطة الزمنية 2 ، T2) ويتم تخزينها عند -80 درجة مئوية لعزل الحمض النووي الجينومي في المستقبل (الشكل 2).
- عزل الحمض النووي الجينومي من الخلايا المنقولة ببرك الفيروسات
- قم بإذابة حبيبات الخلية من الفريزر -80 درجة مئوية وأعد تعليق الحبيبات في 10 مل من المخزن المؤقت لإعادة التعليق مع إضافة RNAse ، وانقسم على الفور إلى أنبوبين بولي ميثيل بنتين سعة 15 مل (الشكل 2 ب).
- أضف 500 ميكرولتر من 10٪ SDS لكل 5 مل ، واخلطها واحتضانها في درجة حرارة الغرفة لمدة 5 دقائق. بعد ذلك ، ضع الأنابيب في جهاز قص الحمض النووي لصوتنة الحمض النووي لمدة 25 دورة من 30 ثانية / 30 ثانية ، مع التأكد من الحفاظ على درجة الحرارة عند 4 درجات مئوية.
- أضف 5 مل من الفينول / الكلوروفورم ، درجة الحموضة 8.0 (مخزنة عند 4 درجات مئوية) ، مع التأكد من خلط الفينول / الكلوروفورم جيدا قبل الاستخدام. بعد إضافة الفينول / الكلوروفورم ، امزج جيدا عن طريق الدوامة بقوة على الإعداد الأقصى لمدة 45-60 ثانية. جهاز طرد مركزي لمدة 60 دقيقة ، -20 درجة مئوية عند 7200 × جم.
ملاحظة: المظهر الرغوي / اللبني بعد الدوامة سيشير إلى تعليق كامل. - انقل 3 مل من المرحلة العليا الصافية إلى أنبوب جديد سعة 15 مل وأضف 0.5 مل من أسيتات الصوديوم 3 أمتار و 4 مل من الأيزوبروبانول واخلطه جيدا (الشكل 2 ج). أجهزة الطرد المركزي لمدة 30 دقيقة عند 20 درجة مئوية عند 7200 × جم.
- بعد الطرد المركزي ، تخلص من المادة الطافية ثم قم بسحب السائل المتبقي بعناية. أضف 0.5 مل من الإيثانول بنسبة 70٪ وقم بإزاحة الحبيبات عن طريق السحب لأعلى ولأسفل. انقل الحبيبات المعاد تعليقها إلى أنبوب طرد مركزي سعة 1.5 مل وادمج كلتا الكريتين من نفس العينة في أنبوب طرد مركزي واحد سعة 1.5 مل. أجهزة الطرد المركزي بأقصى سرعة في جهاز طرد مركزي على الطاولة لمدة 5 دقائق.
- تخلص من المادة الطافية واستخدم مناديل معملية لامتصاص أي إيثانول متبقي بنسبة 70٪. أعد تعليق الحبيبات في 0.5 مل من الماء المقطر ، مع التأكد من عدم ترك الحبيبات تجف لأن هذا سيجعل إعادة التعليق صعبة. قم بتخزين العينات عند 4 درجات مئوية قبل قياس تركيز الحمض النووي.
- تفاعل البوليميراز المتسلسل المتداخل لتضخيم الرموز الشريطية shRNA
ملاحظة: استرجع الحمض النووي من التخزين وضعه على الجليد. إذا كان تركيز الحمض النووي أقل من 100 نانوغرام / ميكرولتر ، فقم بتجفيف الحمض النووي بالمكنسة الكهربائية وإعادة تعليقه في حجم مناسب من الماء المقطر. نوصي بمعالجة وحدة واحدة فقط في كل مرة (النقطة الزمنية 1 (T1) أو النقطة الزمنية 2 (T2)). قم بإجراء إعدادين لكل نقطة زمنية يتم تجميعها في النهاية ؛ وبالتالي ، من المهم عدم معالجة النقاط الزمنية المكررة في نفس اليوم. البروتوكول التالي هو نقطة زمنية واحدة من الحمض النووي الجينومي.- قم بإعداد 7 أنابيب وقم بتسميتها كما هو موضح: التحكم السلبي ، والتحكم الإيجابي ، و 4 أنابيب للحمض النووي الجينومي ، وواحد لمزيج رئيسي (الشكل 3 أ). السيطرة السلبية هي الماء المقطر. التحكم الإيجابي هو الحمض النووي البلازميد المستخدم في نقل 293T لتوليد فيروس lentivirus.
- أضف الماء إلى كل أنبوب أولا كما هو موضح في الجدول 2.
- قم بإعداد مزيج رئيسي (MM) كما هو موضح في الجدول 3.
- أضف 18 ميكرولتر من مم إلى كل أنبوب يحتوي على الماء. بعد ذلك ، أضف 25 ميكرولتر من الحمض النووي الجينومي إلى كل من أنابيب القوالب الأربعة. ثم أضف 1 ميكرولتر من التحكم الإيجابي (10 نانوغرام / ميكرولتر) إلى أنبوب التحكم الإيجابي ، مع الحرص على عدم تلويث الأنابيب الأخرى بالحمض النووي للتحكم الإيجابي. أخيرا ، أضف 2 ميكرولتر من البلمرة إلى كل أنبوب ، إضافة إلى أنابيب العينة 4 أولا وأنبوب التحكم الإيجابي أخيرا ؛ امزج وتدور أنابيب PCR (الشكل 3).
- إجراء تفاعل PCR في ظل الظروف المذكورة في الجدول 4.
- أثناء تشغيل تفاعل البوليميراز المتسلسل الأول ، قم بإعداد الأنابيب ل 2nd PCR كما هو موضح في الجدول 5. قم بإعداد سبعة أنابيب: التحكم السلبي ، والتحكم الإيجابي ، و 4 أنابيب للحمض النووي الجينومي ، وواحد ل MM (الشكل 3 ب).
- أضف 22 ميكرولتر من MM إلى كل أنبوب يحتوي على الماء وانتظر حتى تنتهي الجولة الأولى من تفاعل البوليميراز المتسلسل قبل إضافة الحمض النووي والبلمرة.
- بمجرد اكتمال الجولة الأولى من تفاعل البوليميراز المتسلسل ، ادمج 4 أنابيب عينة في أنبوب طرد مركزي واحد واخلطها.
- أضف 25 ميكرولتر لكل أنبوب عينة يحتوي على الماء.
- بعد ذلك ، أضف 2 ميكرولتر من أول تحكم سلبي في تفاعل البوليميراز المتسلسل و 2 ميكرولتر من أول تحكم إيجابي في تفاعل البوليميراز المتسلسل إلى الأنابيب ذات العلامات المناسبة.
- أضف 2 ميكرولتر من البلمرة إلى كل أنبوب ، إضافة إلى أنابيب العينة 4 أولا والتحكم السلبي أخيرا.
- إجراء تفاعل PCR كما هو مذكور في الجدول 6.
ملاحظة: عند تحليل النتائج من تفاعل PCR الأول عن طريق الكهربائي على هلام الأغاروز 3.5 ٪ ، إذا تم الحصول على وفرة من المنتج ، يمكن تقليل عدد الدورات إلى 9-10 للتكرار التالي لهذا الإجراء ؛ وبالمثل ، إذا كان عائد المنتج منخفضا ، فيمكن زيادة الدورات إلى 14. - عند اكتمال تفاعل تفاعل البوليميراز المتسلسل الثاني ، ادمج 4 عينات في أنبوب طرد مركزي واحد بالإضافة إلى 80 ميكرولتر من صبغة التحميل 6x. للضوابط الإيجابية والسلبية ، استخدم 20 ميكرولتر من العينة.
- قم بإعداد جل أغاروز بنسبة 3.5٪ في محلول Tris-Borate-EDTA (TBE) (الشكل 4 أ). لاستيعاب حجم العينة الكبير ، قم بإنشاء بئر واحد كبير في مشط الجل عن طريق لصق عدة آبار معا (في حالة عدم توفر مشط يناسب الحجم) ، مع التأكد من ترك بئرين متاحين للعناصر الموجبة والسلبية. قم بتشغيل الجل عند 90 فولت لمدة 1 ساعة.
- تصور الجل وقم بتأكيد النطاق عند حوالي 250 زوجا أساسيا (bp).
- التنقية الأولى: استخراج الجل
- باستخدام مشرط نظيف أو شفرة حلاقة ، قم باستئصال شريط 250 bp ، وقطع أكبر قدر ممكن من الجل. قسم الشريط الكبير إلى 4 قطع وانقل كل قطعة إلى أنبوب طرد مركزي نظيف. قم بقياس وتسجيل وزن كل شريحة هلام.
ملاحظة: يجب أن تكون كل قطعة حوالي 200 ملغ أو أقل لكل أنبوب. - أضف 6 مجلدات من محلول الذوبان (على سبيل المثال ، إذا كانت قطعة الجل تزن 200 مجم ، أضف 1.2 مل من المخزن المؤقت). ضع الأنابيب في حمام مائي بدرجة حرارة 50 درجة مئوية وقم بتدويرها كل 10-15 دقيقة حتى تذوب شرائح الهلام. ثم أضف 1 حجم من الأيزوبروبانول إلى كل أنبوب (على سبيل المثال ، 0.2 مل من الأيزوبروبانول إذا كانت قطعة الهلام تزن 200 مجم).
- باستخدام عمودين تدور للتنقية ، قم بتحميل العينات من جميع الأنابيب 4 في الأعمدة. قم بإجراء دورات متعددة لمعالجة كل حجم العينة لأن الأعمدة تحتوي على 750 ميكرولتر فقط. قم بتدوير الأعمدة عند 17900 × جم لمدة 1 دقيقة في جهاز طرد مركزي دقيق تقليدي على الطاولة وتجاهل التدفق في كل مرة.
- اغسل الأعمدة ب 750 ميكرولتر من المخزن المؤقت للغسيل وأجهزة الطرد المركزي عند 17900 × جم لمدة 1 دقيقة. بعد الغسيل ، تخلص من التدفق وجفف عمود الدوران عن طريق الطرد المركزي عند 17900 × جم لمدة 3 دقائق. قم بسحب الحمض النووي ب 50 ميكرولتر من الماء المقطر لكل عمود وأجهزة طرد مركزي كما كان سابقا لمدة 1 دقيقة وادمج كلا الأنبوبين لحجم إجمالي قدره 100 ميكرولتر من العينة.
- باستخدام مشرط نظيف أو شفرة حلاقة ، قم باستئصال شريط 250 bp ، وقطع أكبر قدر ممكن من الجل. قسم الشريط الكبير إلى 4 قطع وانقل كل قطعة إلى أنبوب طرد مركزي نظيف. قم بقياس وتسجيل وزن كل شريحة هلام.
- التنقية الثانية
- ستستخدم خطوة التنقية التالية هذه Binding Buffer 2 (من مجموعة تنقية PCR) ، والتي لا تقضي على الأجزاء الصغيرة. أضف 400 ميكرولتر من المخزن المؤقت إلى 100 ميكرولتر من الحمض النووي ، وقم بتحميله على عمود دوران واحد (الشكل 4 ب). أجهزة الطرد المركزي العينة في 17900 × غرام لمدة 1 دقيقة.
- بعد ذلك ، اغسل الغشاء ب 650 ميكرولتر من المخزن المؤقت للغسيل وقم بتدويره كما تم إجراؤه مسبقا. جفف عمود الدوران باستخدام جهاز طرد مركزي إضافي على النحو الوارد أعلاه لمدة 3 دقائق.
- قم بسحب الحمض النووي ب 30 ميكرولتر من الماء المقطر وقم بتدويره بأقصى سرعة لمدة 1 دقيقة. بعد الشطف ، تحقق من التركيز على مقياس الطيف الضوئي ؛ تأكد من أن التركيز لا يقل عن 10 نانوغرام / ميكرولتر أو أعلى من 70-80 نانوغرام / ميكرولتر. قم بتخزين الحمض النووي في مجمد -20 درجة مئوية.
- تسلسل الباركود المضخم
- للتسلسل ، قم بتخفيف الرموز الشريطية المنقاة إلى 0.75 نانوغرام / ميكرولتر باستخدام المخزن المؤقت للشطف (EB). لإضافة تنوع التسلسل ، يتم تجميع الأمبليكونات عند 17 pM ، بما في ذلك 30٪ (v / v) PhiX. قم بإجراء تجميع أحادي الطرف (SE) على نظام إنشاء مجموعة آلي وفقا لبروتوكول الشركة المصنعة.
- اطلب من نواة التسلسل إجراء ما مجموعه 36 دورة من التسلسل أحادي الطرف على جهاز تسلسل NextGen (الشكل 4C). أضف التمهيدي المخصص GexSeqS إلى بادئات تسلسل Illumina عند 0.5 ميكرومتر.
- استخدم برنامج التحليل المشار إليه لإنشاء ملفات Fastq ومعالجتها باستخدام برنامج لتقليص أطوال القراءة إلى 18 نيوكليوتيدات.
- استخدم محلل الباركود وبرنامج Deconvoluter لفك القراءات المشذبة. احسب درجات استنفاد الطيات لكل shRNA كنسبة من عدد القراءة في نقطة الوقت المرجعية (T1) مقابل النقطة الزمنية النهائية (T2).
- تحليل نتائج فحص shRNA وتحديد الأهداف العلاجية المرشحة
ملاحظة: في مكتبة Cellecta shRNA ، يتم استهداف معظم الجينات إما بواسطة 5 (67٪) أو 6 shRNAs مختلفة (32٪). ومع ذلك ، فإن العديد من جينات التدبير المنزلي ، والتي يجب أن تكون ناجحة في معظم الخلايا ، مستهدفة بأعداد كبيرة من shRNAs وتعمل كضوابط تحدد نطاق الدرجات للسلبيات.- لمنع التحيز نحو الجينات المستهدفة بأعداد أكبر من shRNAs ، قم بتحويل درجات النضوب. قم بإجراء تقدير كمي عن طريق حساب النسبة المئوية 80 لكل جين من توزيعه التجريبي.
- لإنشاء توزيع فارغ لدرجات استنفاد الطيات اللوغاريتمية ، افترض أن >95٪ من الجينات لن يتم استنفادها وأن درجاتها اللوغاريتمية الكمية سيكون لها توزيع طبيعي. استخدم وسيط التوزيع التجريبي لتقدير متوسط التوزيع الخالي. باستخدام هذا التوزيع الفارغ ، صنف جميع الجينات ذات درجات استنفاد الطيات اللوغاريتمية التي تكون أكبر من النسبة المئوية 95 للتوزيع الفارغ على أنها "ضربات".
ملاحظة: يجب أن تحتوي جميع الزيارات على اثنين على الأقل من shRNAs مع درجات استنفاد أعلى من نقطة القطع (الشكل 5). - لتقييم جودة بيانات شاشة تسرب RNAi وصحة نقطة القطع ، استخدم مجموعة من الجينات التي تم تعريفها على أنها "أساسية أساسية" من قبل مبادرة فحص COLT Cancer RNAi24,25 للمقارنة. قم بإزالة CEGs من قائمة الزيارات لإنتاج القائمة الأولية للأهداف العلاجية المحتملة.
ملاحظة: سجلت الجينات في مجموعة COLT كضربة في >50٪ من 72 خطا للخلايا السرطانية التي تم فحصها بواسطة COLT. تحتوي قائمة الجينات الأساسية الأساسية على 640 جينا. - لتحديد الأهداف العلاجية المحتملة المطلوبة بشكل شائع أو موحد من قبل خطوط خلايا MPNST المتعددة ، قم ببناء مخططات Venn للنتائج غير CEG المحددة في شاشات خطوط خلايا MPNST المختلفة أو ثقافات المرور المبكر (الشكل 6A). إعطاء الأولوية للنتائج غير CEG المطلوبة في جميع أو غالبية خطوط أو ثقافات MPNST.
- بدلا من ذلك ، قم بإجراء تحليلات المسار على قائمة ضرب غير CEG من كل خط خلية أو ثقافة مرور مبكرة لتحديد الجينات التي تشفر منتجاتها مكونات مسارات الإشارات المطلوبة لتكاثر الخلايا السرطانية و / أو البقاء على قيد الحياة. بعد ذلك ، قارن مسارات الإشارات التي تم تحديدها في تحليل المسار لغير CEGs بمسارات الإشارات التي تم تحديدها على أنها متأثرة بالطفرات المحددة مع WES.
ملاحظة: نجد أنه من المفيد بشكل خاص تحديد مسارات الإشارات التي تتأثر باستمرار بالطفرات المحددة في WES ومقارنتها بمسارات الإشارات التي تم تحديدها على أنها حرجة في شاشات shRNA.
- بدلا من ذلك ، قم بإجراء تحليلات المسار على قائمة ضرب غير CEG من كل خط خلية أو ثقافة مرور مبكرة لتحديد الجينات التي تشفر منتجاتها مكونات مسارات الإشارات المطلوبة لتكاثر الخلايا السرطانية و / أو البقاء على قيد الحياة. بعد ذلك ، قارن مسارات الإشارات التي تم تحديدها في تحليل المسار لغير CEGs بمسارات الإشارات التي تم تحديدها على أنها متأثرة بالطفرات المحددة مع WES.
- لتحديد الأهداف القابلة للدواء والتي تتوفر لها العوامل العلاجية بالفعل ، قم بفحص قائمة الزيارات المتبقية بعد إزالة الجينات الأساسية الأساسية باستخدام قاعدة بيانات التفاعل الجيني الدوائي (dgidb.org).
ملاحظة: تسمح قاعدة البيانات هذه بإدخال قائمة الجينات وفحصها في وقت واحد وتوفر إرشادات حول الأدوية المتاحة حاليا لاستهداف هذه الجينات. - تحقق من صحة الأهداف عالية الاهتمام القابلة للدواء المحددة أولا عن طريق هدم تعبيرها الجيني باستخدام shRNAs المتميزة عن تلك المستخدمة في شاشة المكتبة الأولية بتنسيق دفعة واحدة (وليس تنسيق مكتبة دفعة موصوف للتو). لهدم التعبير الجيني ، استخدم اثنين إلى ثلاثة من shRNAs الفيروسية العدسية المتميزة. بعد ثلاثة إلى أربعة أيام من الإصابة، حدد تأثير ذلك على تكاثر الخلايا السرطانية وبقائها على قيد الحياة كما هو موضح أدناه.
3. إجراء فحوصات مقياس الخلايا لأعداد الخلايا وصلاحيتها في خلايا MPNST التي تم تحديها باستخدام العوامل العلاجية المرشحة
- تنمو خلايا MPNST في DMEM إلى التقاء 80٪. شطف الخلايا بدرجة حرارة الغرفة محلول الملح المتوازن هانكس (HBSS).
ملاحظة: لا تنمو الخلايا إلى نقطة التقاء لأن نمو هذه الخلايا سوف يتأخر لفترة من الوقت بعد إعادة الطلاء. - افصل الخلايا عن الركيزة عن طريق تغطية الخلايا لمدة 30 ثانية إلى 1 دقيقة بمحلول تفكك الخلايا غير الأنزيمي. أضف 5 مل من DMEM لكل 1 مل من محلول التفكك وقم بسحب الخلايا برفق لأعلى ولأسفل للفصل عن الركيزة.
- عد الخلايا باستخدام مقياس الدم. خلايا الصفيحة بكثافة 1200 خلية لكل بئر في ألواح 96 بئرا ذات الجدران السوداء ؛ صفيحة ما لا يقل عن ثلاثة آبار لكل تخفيف دوائي سيتم اختباره وإجراء ثلاث نسخ بيولوجية مكررة من هذه التجارب.
- لتحديد التركيزات الأولية للدواء الذي سيتم اختباره ، راجع الأدبيات لتقييم التركيزات التي كانت فعالة ضد أنواع الخلايا السرطانية الأخرى. في التجارب الأولية ، اختبر نطاقا من أمرين من حيث الحجم أعلى وأمرين من الحجم أقل من تركيز الدواء المستخدم مع أنواع السرطان الأخرى.
- تحضير تخفيفات الدواء المراد اختباره وإضافة كل تخفيف أو مركبة إلى ثلاثة آبار مكررة على الأقل.
- تقييم أرقام الخلايا المباشرة 1 و 3 و 5 و 7 أيام بعد إضافة الأدوية. أضف Hoechst 33342 إلى تركيز نهائي قدره 5 ميكروغرام / مل واحتضان الألواح لمدة 30 دقيقة عند 37 درجة مئوية. اقرأ اللوحات على مقياس خلوي للتصوير عالي الإنتاجية باستخدام خيار عدد الخلايا المباشر لإجمالي عدد الخلايا مع أوقات تعرض تبلغ 100000 مللي ثانية.
- تحليل القراءات باستخدام البرنامج وتصديرها إلى جدول بيانات واستخدام البرامج المناسبة للتحليلات الإحصائية.
- إذا لوحظت انخفاضات ذات دلالة إحصائية في أعداد الخلايا في الآبار المعالجة بالعقاقير ، فقم بإجراء اختبار "حي / ميت" لتحديد ما إذا كان هذا الانخفاض يرجع جزئيا إلى تحريض موت الخلايا.
- خلايا اللوحة في 96 لوحة بئر ذات جدران سوداء كما هو موضح في الخطوة 3.3.
- تحضير الأدوية وإضافتها كما هو موضح في الخطوة 3.5.
- تقييم صلاحية الخلية وموتها بعد 1 و 3 و 5 و 7 أيام من إضافة الدواء. أضف الكالسيين AM إلى تركيز نهائي قدره 1 ميكرومتر ويوديد البروبيديوم إلى تركيز نهائي قدره 1 ميكرومتر في كل بئر. احتضان الخلايا لمدة 15 دقيقة عند 37 درجة مئوية.
- خلايا الصورة على مقياس خلوي للتصوير باستخدام خيار برنامج Live+Dead . تحليل القراءات باستخدام البرنامج وتصديرها إلى جدول بيانات واستخدام البرامج المناسبة للتحليلات الإحصائية.
النتائج
تعرض مخططات الشكل 5 درجات استنفاد الجينات الأساسية الأساسية (CEGs) المصنفة على أنها TRUE مقارنة بغير CEGs (المسمى FALSE) في كل خط خلية بشرية تم فحصه. تمثل النقاط log2 من درجات استنفاد الطيات للجينات الفردية ، والتي يتم رسمها على تمثيل boxplot لتوزيع النتيجة الإجمالي. تم استخدام اختبار t للطالب لاختبار وجود فرق كبير في متوسط درجات النضوب بين المجموعتين في كل خط خلوي. يشار إلى قيم p الناتجة في كل لوحة. لاحظ أن متوسط درجات استنفاد الطيات أعلى بكثير بالنسبة ل CEGs من غير CEGs. هذا متوقع لأن الجينات الأساسية الأساسية ، بحكم تعريفها ، مطلوبة باستمرار للتكاثر و / أو البقاء في معظم أنواع الخلايا.
يقدم الشكل 6 أ مخطط فن ل "الزيارات" لثلاثة خطوط خلايا MPNST بشرية. نجد عادة أن عددا كبيرا من الجينات مشتركة بين خطوط متعددة. هذه النتائج هي أولوية عالية لأنها تمثل الجينات المشفرة للبروتينات التي من المحتمل أن تكون ضرورية لانتشار و / أو بقاء مجموعة فرعية كبيرة من MPNSTs. لاحظ أيضا أن هناك عددا من الجينات التي يتم ضربها في خط خلية واحد فقط. نواجه هذا بشكل شائع ولا ينبغي اعتباره مؤشرا على أن الشاشات ذات جودة رديئة. ثم يتم تقييم الجينات الشائعة بين خطوط متعددة باستخدام قاعدة بيانات التفاعل الجيني الدوائي لتحديد الجينات داخل هذه المجموعة الفرعية التي تشفر البروتينات القابلة للدواء مع العوامل الموجودة. ثم نختار العديد منها ونجري تحققا أوليا عن طريق إسقاط تعبير mRNA المقابل باستخدام shRNAs. نظرا لأن بعض shRNAs سيكون لها تأثيرات خارج الهدف ، فإننا نختبر دائما العديد من shRNAs التي تستهدف نفس النص. يوضح الشكل 6B نتيجة تمثيلية قمنا فيها بتحويل خلايا MPNST مع عنصر تحكم غير مستهدف و shRNAs متعددة تستهدف BCL6. ثم تم تحديد أرقام الخلايا في أوقات مختلفة بعد النقل. لاحظ أن العديد من BCL6 shRNAs خفضت بشكل ملحوظ أرقام الخلايا. كما هو موضح في اللطخة المناعية المصاحبة ، ترتبط درجة الانخفاض في أعداد الخلايا بدرجة ضربة قاضية BCL6. يوضح الشكل 6C منحنى نمو تمثيلي لثقافة مرور مبكر P 0-GGFβ3 MPNST.

الشكل 1: سير العمل لإجراء تسلسل الإكسوم الكامل لأنسجة MPNST أو خلايا MPNST ذات المرور المبكر. يوضح التخطيطي سير العمل العام للكشف عن المتغيرات الموجودة في ثقافات المرور المبكر المشتقة من الورم. عزل الحمض النووي من ثقافات المرور المبكر (1) وتقديم الحمض النووي عالي الجودة إلى قلب التسلسل وفقا لبروتوكولات التقديم الخاصة بهم (2). سيتحقق جوهر التسلسل من جودة الحمض النووي المقدم ويقوم بجميع الاستعدادات اللازمة للعينات ومكتبة الجينوم. سيوفر المرفق الأساسي للمستخدمين ملفات تسلسل FASTQ بمقاييس مراقبة الجودة (3). سيقوم المستخدمون بتحميل ملفات FASTQ إلى محاذاة الجينوم وبرنامج المتصل المتغير الذي يختارونه. (4) تتم تصفية المتغيرات المشروحة وفقا لمعايير يحددها المستخدم لإزالة المتغيرات غير ذات الصلة. البيانات التمثيلية المعروضة تقارن عينة ورم MPNST البشرية المقطوعة مقابل خط الخلية المشتق من الورم26. (5) إجراء تحليل التصنيف الوظيفي مع النمر. اختصار: MPNST = ورم غمد العصب المحيطي الخبيث. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
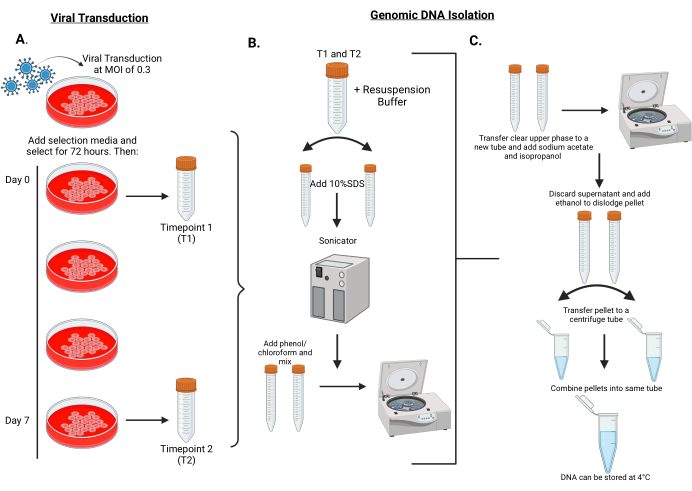
الشكل 2: سير العمل لإجراء النقل الفيروسي لمكتبات shRNA إلى خلايا MPNST وعزل الحمض النووي الجينومي من الخلايا عند النقطة الزمنية 1 والنقطة الزمنية 2. (أ) تصاب الخلايا المستهدفة عند MOI منخفض يبلغ 0.3 بجزيئات فيروسية عدسية مشفرة ومختارة لمدة 72 ساعة. يتم تمرير الخلايا لمدة 5-7 مضاعفة السكان (حوالي 7 أيام). يتم تخزين كريات الخلايا في اليوم 0 واليوم 7 عند -80 درجة مئوية لعزل الحمض النووي الجينومي. يشار إلى اليوم 0 بالنقطة الزمنية 1 (T1) ويشار إلى اليوم 7 بالنقطة الزمنية 2 (T2). (ب) يبدأ عزل الحمض النووي الجينومي بإعادة تعليق الكريات الخلوية في مخزن إعادة التعليق، ثم تنقسم إلى أنبوبين سعة 15 mL. لتسهيل تحلل الخلايا ، تتم إضافة 10٪ SDS إلى كل أنبوب وصوتنة لمدة 25 دورة من 30 ثانية على / 30 ثانية قبالة. بعد صوتنة ، يضاف الفينول / الكلوروفورم إلى كل أنبوب ودوامة بقوة لمدة 45-60 ثانية. ثم يتم طرد الأنابيب بالطرد المركزي. (ج) يتم سحب الطور العلوي الصافي وإضافته إلى أنبوب نظيف مع إضافة أسيتات الصوديوم / الأيزوبروبانول ويخلط جيدا. يتم طرد الأنابيب مرة أخرى. هذه المرة ، يتم التخلص من المادة الطافية ، ويتم إزاحة الحبيبات بإضافة 70٪ من الإيثانول. اجمع الكريات المعاد تعليقها في أنبوب واحد وقم بتدويرها بأقصى سرعة في جهاز طرد مركزي على الطاولة. تخلص من المادة الطافية وأعد تعليق الحبيبات في الماء المقطر. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
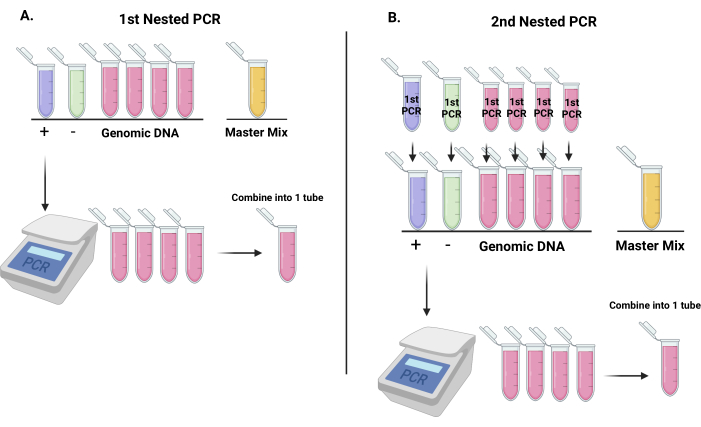
الشكل 3: سير العمل لتضخيم تسلسلات الباركود من نواقل shRNA الفيروسية عدسية استعدادا للقياس الكمي للرموز الشريطية مع تسلسل الجيل التالي. (أ) تمثيل كيفية إعداد الأنابيب لأول تفاعل تفاعل البوليميراز المتسلسل المتداخل (7 أنابيب: واحد للتحكم السلبي ، وواحد للتحكم الإيجابي ، والأنابيب الأربعة المتبقية للحمض النووي الجينومي. سيكون الأنبوب الأخير بمثابة أنبوب المزيج الرئيسي). بعد تفاعل تفاعل البوليميراز المتسلسل الأول ، ادمج أنابيب الحمض النووي الجينومية في أنبوب واحد واخلطها. (ب) ستكون نواتج تفاعل البوليميراز المتسلسل المتداخل الأول بمثابة قوالب لتفاعل تفاعل تفاعل البوليميراز المتسلسل المتداخل الثاني. بعد تفاعل البوليميراز المتسلسل الثاني ، ادمج أنابيب الحمض النووي الجينومية في أنبوب واحد واخلطها. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
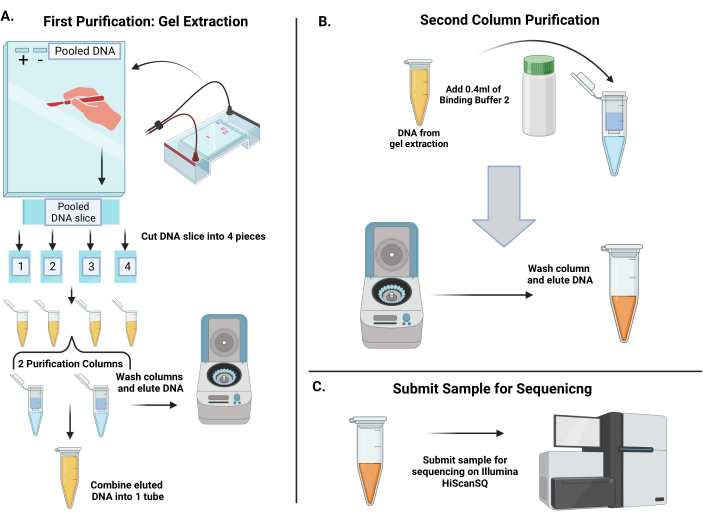
الشكل 4: سير العمل لتنقية الرموز الشريطية shRNA المضخمة وتسلسل الرموز الشريطية لتحديد تمثيلها عند النقطة الزمنية 1 والنقطة الزمنية 2 . (أ) صب 3.5٪ هلام أغاروز. نظرا لأن حجم الحمض النووي المجمع يتجاوز الحد الأقصى لبئر واحد ، فقم بربط 4-6 أسنان من مشط الجل معا لإنتاج بئر واحد كبير. قم بإعداد منتجات PCR بصبغة تحميل 6x وقم بتحميل التحكم الإيجابي والتحكم السلبي والحمض النووي المجمع في الجل. بعد الرحلان الكهربائي ، يجب أن يظهر شريط كبير عند حوالي 250 زوجا من القواعد في ممر الحمض النووي المجمع. باستخدام مشرط نظيف ، قم باستئصال الشريط بأكمله ، ثم قطعه إلى 4 شرائح هلام. إذابة قطع الهلام ثم دمجها في عمودين مغزلين لإذابة الحمض النووي. الجمع بين الحمض النووي المستخلصة في أنبوب واحد. (ب) ينقي الحمض النووي (DNA) من خلال خطوة تنقية ثانية. أضف Binding Buffer 2 إلى أنبوب الحمض النووي المجمع ، ثم ماصة على عمود الدوران. اغسل الغشاء ثم قم بإزالة الحمض النووي في الماء المقطر. ج: إرسال الحمض النووي المنقى إلى نواة التسلسل. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
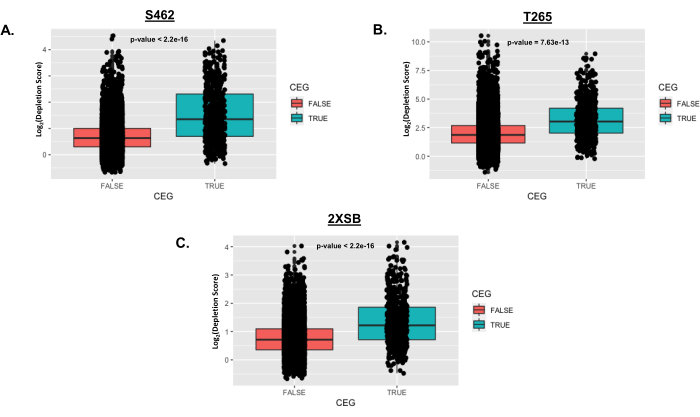
الشكل 5: أمثلة تمثيلية لتوزيع الجينات الأساسية الأساسية بعد التحليل كما هو موضح في البروتوكول. في هذا المثال ، تم فحص ثلاث خطوط خلايا MPNST بشرية (S462 و T265 و 2XSB) باستخدام مكتبات Cellecta DECIPHER shRNA. لكل خط خلية MPNST بشري ، تم إنشاء مخطط صندوقي لمقارنة درجات استنفاد مستوى الجينات للجينات في قائمة الجينات الأساسية الأساسية (CEG; مخطط مربع صحيح)25 إلى الجينات غير الموجودة في قائمة CEGs (مخطط مربع خطأ). يتم وضع نقاط البيانات الفردية فوق كل مخطط مربع. قيم P مأخوذة من اختبار t قياسي يقارن درجات استنفاد مستوى الجينات ل CEGs بغير CEGs. الاختصارات: CEG = الجين الأساسي الأساسي ؛ MPNST = ورم غمد العصب المحيطي الخبيث. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
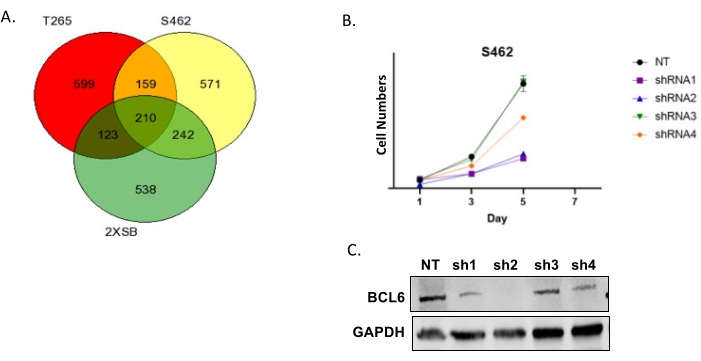
الشكل 6: التحقق من صحة نتائج الشاشة . (أ) مخطط فن التمثيلي للضربات المتداخلة في ثلاثة خطوط خلايا MPNST بشرية. (ب) خلايا MPNST البشرية S462 المنقولة بناقل فيروسي عدسي غير مستهدف (NT) وفيروس عدسي يعبر عن أربعة أنواع مختلفة من BCL6 shRNAs (shRNA1 و shRNA2 و shRNA3 و shRNA4). تم تحويل الخلايا بفيروس lentivirus ثم عولجت بعامل اختيار (puromycin) لمدة 3 أيام. ثم تم تقييم أرقام الخلايا على مدار الأيام السبعة التالية. (ج) تحليل اللطخة الغربية الذي يظهر مستويات البروتين في BCL6 بعد التحويل باستخدام NT و shRNA1 و shRNA2 و shRNA3 و shRNA4. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
الجدول 1: تخطيط لوحة لمعايرة الفيروسات العدسية. الرجاء الضغط هنا لتنزيل هذا الجدول.
الجدول 2: الإعداد الأولي لتفاعلات تفاعل البوليميراز المتسلسل الأولى. الرجاء الضغط هنا لتنزيل هذا الجدول.
الجدول 3: تحضير المزيج الرئيسي لأول تفاعل تفاعل البوليميراز المتسلسل. الرجاء الضغط هنا لتنزيل هذا الجدول.
الجدول 4: معلمات Cellecta الأولى PCR. الرجاء الضغط هنا لتنزيل هذا الجدول.
الجدول 5: الإعداد الأولي لتفاعل تفاعل البوليميراز المتسلسل الثاني. الرجاء الضغط هنا لتنزيل هذا الجدول.
الجدول 6: معلمات Cellecta PCR الثانية. الرجاء الضغط هنا لتنزيل هذا الجدول.
Discussion
تم تطوير الطرق التفصيلية المعروضة هنا لدراسة أورام الجهاز العصبي المحيطي والتسبب في MPNST. على الرغم من أننا وجدنا أن هذه الطرق فعالة ، إلا أنه يجب الاعتراف بوجود بعض القيود المحتملة على الطرق التي نصفها هنا. أدناه ، نناقش بعض هذه القيود والاستراتيجيات المحتملة للتغلب عليها في أنظمة نموذجية أخرى.
لقد وجدنا أن تسلسل الإكسوم الكامل يحدد بشكل فعال الطفرات ذات الأهمية في الفئران P 0-GGFβ3. ومع ذلك ، يجب الاعتراف بأن تسلسل الإكسوم بأكمله له حدود. أولا ، لا يعد تسلسل الإكسوم الكامل نهجا فعالا لتحديد منتجات جينات الاندماج. وذلك لأن معظم فواصل الكروموسومات والاندماج اللاحق تتضمن في الغالب المناطق بين الجينات والإنترونات لأن هذه المناطق تمثل غالبية الجينوم. لقد وجدنا بدلا من ذلك أن RNA-Seq مع قراءات النهاية المزدوجة تحدد بشكل أكثر فعالية جينات الاندماج. هناك أيضا سؤال حول مدى فعالية تسلسل الإكسوم الكامل في تحديد مناطق كبيرة نسبيا من فقدان الكروموسومات.
على الرغم من تطوير العديد من الخوارزميات لتحديد مثل هذه الخسائر ، فإن مصطلح "تسلسل الإكسوم الكامل" هو في حد ذاته مضلل لأن التقاط الإكسوم حتى في عمليات التشغيل الجيدة غالبا ما يفتقد ما يصل إلى 5-10٪ من المناطق الخارجية. لهذا السبب ، فإننا نكمل بشكل روتيني تسلسل الإكسوم الكامل بأساليب أخرى مثل التهجين الجينومي المقارن للصفيف (aCGH). بعد تحديد المكاسب والخسائر ، نفحص الجينات ضمن هذه الفترات ونقارنها بالطفرات الدافعة المعروفة المرتبطة بنظيراتها البشرية. ومع ذلك ، فإن جينوم الفأر أكثر استقرارا من الجينوم البشري27. وبالتالي ، لا تظهر أورام الفئران عادة كروموثريبسيس مماثلة لما يظهر في الأورام البشرية. بدلا من ذلك ، يكون النمط في أورام الفئران أبسط بكثير ، حيث يميل إلى مكاسب أو خسائر كروموسوم كامل أو كروموسوم مع عدد قليل نسبيا من عمليات الحذف البؤري التي تميل إلى الحدوث تحت ضغط انتقائي قوي22,23.
هناك بعض المزالق المحتملة التي واجهناها عند إجراء شاشات shRNA على نطاق الجينوم. واحدة من أكثر المشاكل شيوعا التي نواجهها هي النقل الضعيف نسبيا للنواقل الفيروسية العدسية إلى الخلايا المستهدفة. غالبا ما نجد أن المشكلة نشأت من المعايرة غير الصحيحة لبرك الفيروسات العدسية المعبأة. نظرا لأن مزارع الخلايا السرطانية للفأر في وقت مبكر هي مورد مقيد ، سيحاول العديد من الباحثين بدلا من ذلك معايرة فيروس العدس باستخدام خط خلوي آخر ثابت متاح بسهولة أكبر. ومع ذلك ، فإن المشكلة في هذا النهج هي أن كفاءة نقل الفيروسات العدسية يمكن أن تختلف اختلافا كبيرا من نوع الخلية إلى نوع الخلية. ولهذا السبب نوصي بمعايرة فيروس العدس على الخلايا الفعلية التي سيتم استخدامها في التجربة. لقد واجهنا أيضا مشاكل مع التتر الفيروسي المنخفض نسبيا. غالبا ما تعكس هذه المشكلة ضعف نقل الخلايا التائية 293 عند إنتاج الفيروس المعبأ.
من الممكن الحصول على نتائج إيجابية خاطئة عند إجراء شاشات shRNA على نطاق الجينوم. لهذا السبب ، بمجرد تحديد الأهداف المحتملة للتخدير التي تهمنا أكثر ، فإننا نتحقق دائما من صحة نتائج شاشات shRNA الخاصة بنا. عادة ، سنستخدم نهجين مختلفين للتحقق من صحة الأهداف ذات الفائدة العالية. أولا ، نهدم التعبير الجيني باستخدام اثنين أو أكثر من shRNAs متميزين عن تلك المستخدمة في الشاشة الأولية ونحدد تأثير ذلك على تكاثر الخلايا السرطانية وبقائها. ثانيا ، نحصل على الدواء (الأدوية) المحددة في قاعدة بيانات التفاعل الجيني للأدوية ونحدد تأثير ذلك على تكاثر الخلايا السرطانية وبقائها. نحن نستخدم كلا النهجين جنبا إلى جنب لأننا واجهنا ظروفا يعمل فيها shRNAs ولا يعمل الدواء. في بعض هذه الحالات على الأقل ، أظهر فحص مجموعة بيانات تسلسل الإكسوم بأكملها أن البروتين المستهدف يتم إنتاجه بواسطة جين يحتوي على طفرة يحتمل أن تؤثر على تفاعلات البروتين الدوائي.
ستوفر الأساليب الموضحة أعلاه للباحث وسيلة قابلة للتطبيق لتحديد الطفرات الدافعة المحتملة التي تحدث في الأورام النادرة ، وتحديد مسارات الإشارات المطلوبة للانتشار والبقاء وظيفيا ، وتحديد أولويات أهداف التطوير العلاجي. نأمل أن يجد الباحثون الآخرون هذه الأساليب مفيدة لتحديد الأهداف العلاجية الرئيسية في السرطانات البشرية الأخرى. ومع ذلك ، يجب أن يدرك القارئ أن هناك طرقا جينومية وظيفية أخرى يمكن استخدامها لتحديد الجينات المشاركة في التسبب في الورم والجينات التي تشفر الأهداف العلاجية المحتملة. على سبيل المثال ، تتوفر مكتبات CRISPR التي يمكن استخدامها بطريقة مماثلة لتلك التي نصفها لمكتبات shRNA. يمكن أيضا إجراء الشاشات الوظيفية في الجسم الحي لتحديد الجينات التي تعزز تكوين الأورام. وكمثال على ذلك ، تم استخدام نظام الطفرات الجسدية القائم على Sleeping Beauty transposon سابقا لاستهداف خلايا Schwann وسلائفها ، مما أدى إلى تحديد عدة مئات من الجينات المشاركة في التسبب في MPNST28. نظرا لأن هذه الأنظمة تتعامل مع علم الجينوم الوظيفي بطرق متميزة ، فإننا نوصي بأن ينظر الباحث بعناية في أهداف تجاربه المخطط لها وأن يبني اختياره لمنهجية الجينوم الوظيفي على تلك الأهداف.
Disclosures
ليس لدى المؤلفين أي تضارب في المصالح للكشف عنه.
Acknowledgements
تم دعم هذا العمل بمنح من المعهد الوطني للأمراض العصبية والسكتة الدماغية (R01 NS048353 و R01 NS109655 إلى S.L.C. ؛ R01 NS109655-03S1 إلى D.P.J.) ، والمعهد الوطني للسرطان (R01 CA122804 إلى S.L.C.) ، ووزارة الدفاع (X81XWH-09-1-0086 و W81XWH-12-1-0164 إلى S.L.C.)
Materials
| Name | Company | Catalog Number | Comments |
| Bioruptor Sonication System | Diagenode | UCD-600 | |
| CASAVA 1.8.2 | |||
| Cbot | Illumina, San Diego, CA | N/A | |
| Celigo Image Cytometer | Nexcelom | N/A | |
| Cellecta Barcode Analyzer and Deconvoluter software | |||
| Citrisolve Hybrid | Decon Laboratories | 5989-27-5 | |
| Corning 96-well Black Microplate | Millipore Sigma | CLS3603 | |
| Diagenode Bioruptor 15ml conical tubes | Diagenode | C30010009 | |
| dNTP mix | Clontech | 639210 | |
| Eosin Y | Thermo Scientific | 7111 | |
| Elution buffer | Qiagen | 19086 | |
| Ethanol (200 Proof) | Decon Laboratories | 2716 | |
| Excel | Microsoft | ||
| FWDGEX 5’-CAAGCAGAAGACGGCATACGAGA-3’ | |||
| FWDHTS 5’-TTCTCTGGCAAGCAAAAGACGGCATA-3’ | |||
| GexSeqS (5’ AGAGGTTCAGAGTTCTACAGTCCGAA-3’ | HPLC purified | ||
| GraphPad Prism | Dotmatics | ||
| Harris Hematoxylin | Fisherbrand | 245-677 | |
| Illumina HiScanSQ | Illumina, San Diego, CA | N/A | |
| Paraformaldehyde (4%) | Thermo Scientific | J19943-K2 | |
| PLUS Transfection Reagent | Thermo Scientific | 11514015 | |
| Polybrene Transfection Reagent | Millipore Sigma | TR1003G | |
| PureLink Quick PCR Purification Kit | Invitrogen | K310001 | |
| Qiagen Buffer P1 | Qiagen | 19051 | |
| Qiagen Gel Extraction Kit | Qiagen | 28704 | |
| RevGEX 5’-AATGATACGGCGACCACCGAGA-3’ | |||
| RevHTS1 5’-TAGCCAACGCATCGCACAAGCCA-3’ | |||
| Titanium Taq polymerase | Clontech | 639210 | |
| Trimmomatic software | www.usadellab.org |
References
- Carroll, S. L. Molecular mechanisms promoting the pathogenesis of Schwann cell neoplasms. Acta Neuropathol. 123 (3), 321-348 (2012).
- Longo, J. F., Weber, S. M., Turner-Ivey, B. P., Carroll, S. L. Recent Advances in the Diagnosis and Pathogenesis of Neurofibromatosis Type 1 (NF1)-associated Peripheral Nervous System Neoplasms. Adv Anat Pathol. 25 (5), 353-368 (2018).
- Longo, J. F., Carroll, S. L. The RASopathies: Biology, genetics and therapeutic options. Adv Cancer Res. 153, 305-341 (2022).
- Birindelli, S., et al. Rb and TP53 pathway alterations in sporadic and NF1-related malignant peripheral nerve sheath tumors. Lab Invest. 81 (6), 833-844 (2001).
- Legius, E., et al. TP53 mutations are frequent in malignant NF1 tumors. Genes Chromosomes Cancer. 10 (4), 250-255 (1994).
- Menon, A. G., et al. Chromosome 17p deletions and p53 gene mutations associated with the formation of malignant neurofibrosarcomas in von Recklinghausen neurofibromatosis. Proc Natl Acad Sci U S A. 87 (14), 5435-5439 (1990).
- Upadhyaya, M., et al. Germline and somatic NF1 gene mutation spectrum in NF1-associated malignant peripheral nerve sheath tumors (MPNSTs). Hum Mutat. 29 (1), 74-82 (2008).
- Kourea, H. P., Orlow, I., Scheithauer, B. W., Cordon-Cardo, C., Woodruff, J. M. Deletions of the INK4A gene occur in malignant peripheral nerve sheath tumors but not in neurofibromas. Am J Pathol. 155 (6), 1855-1860 (1999).
- Nielsen, G. P., et al. Malignant transformation of neurofibromas in neurofibromatosis 1 is associated with CDKN2A/p16 inactivation. Am J Pathol. 155 (6), 1879-1884 (1999).
- Gregorian, C., et al. PTEN dosage is essential for neurofibroma development and malignant transformation. Proc Natl Acad Sci U S A. 106 (46), 19479-19484 (2009).
- Lee, W., et al. PRC2 is recurrently inactivated through EED or SUZ12 loss in malignant peripheral nerve sheath tumors. Nat Genet. 46 (11), 1227-1232 (2014).
- Zhang, M., et al. Somatic mutations of SUZ12 in malignant peripheral nerve sheath tumors. Nat Genet. 46 (11), 1170-1172 (2014).
- Varela, I., et al. Somatic structural rearrangements in genetically engineered mouse mammary tumors. Genome Biol. 11 (10), 100(2010).
- Johnson, R. A., et al. Cross-species genomics matches driver mutations and cell compartments to model ependymoma. Nature. 466 (7306), 632-636 (2010).
- Kim, M., et al. Comparative oncogenomics identifies NEDD9 as a melanoma metastasis gene. Cell. 125 (7), 1269-1281 (2006).
- Zender, L., et al. Identification and validation of oncogenes in liver cancer using an integrative oncogenomic approach. Cell. 125 (7), 1253-1267 (2006).
- Uren, A. G., et al. Large-scale mutagenesis in p19(ARF)- and p53-deficient mice identifies cancer genes and their collaborative networks. Cell. 133 (4), 727-741 (2008).
- Starr, T. K., et al. A transposon-based genetic screen in mice identifies genes altered in colorectal cancer. Science. 323 (5922), 1747-1750 (2009).
- Dupuy, A. J., et al. A modified sleeping beauty transposon system that can be used to model a wide variety of human cancers in mice. Cancer Res. 69 (20), 8150-8156 (2009).
- Carroll, S. L. The Challenge of Cancer Genomics in Rare Nervous System Neoplasms: Malignant Peripheral Nerve Sheath Tumors as a Paradigm for Cross-Species Comparative Oncogenomics. Am J Pathol. 186 (3), 464-477 (2016).
- Huijbregts, R. P., Roth, K. A., Schmidt, R. E., Carroll, S. L. Hypertrophic neuropathies and malignant peripheral nerve sheath tumors in transgenic mice overexpressing glial growth factor beta3 in myelinating Schwann cells. J Neurosci. 23 (19), 7269-7280 (2003).
- Kazmi, S. J., et al. Transgenic mice overexpressing neuregulin-1 model neurofibroma-malignant peripheral nerve sheath tumor progression and implicate specific chromosomal copy number variations in tumorigenesis. Am J Pathol. 182 (3), 646-667 (2013).
- Brosius, S. N., et al. Neuregulin-1 overexpression and Trp53 haploinsufficiency cooperatively promote de novo malignant peripheral nerve sheath tumor pathogenesis. Acta Neuropathol. 127 (4), 573-591 (2014).
- Hart, T., Brown, K. R., Sircoulomb, F., Rottapel, R., Moffat, J. Measuring error rates in genomic perturbation screens: gold standards for human functional genomics. Mol Syst Biol. 10 (7), 733(2014).
- Hart, T., et al. Evaluation and Design of Genome-Wide CRISPR/SpCas9 Knockout Screens. G3. 7 (8), Bethesda. 2719-2727 (2017).
- Longo, J. F., et al. Establishment and genomic characterization of a sporadic malignant peripheral nerve sheath tumor cell line. Sci Rep. 11 (1), 5690(2021).
- Maser, R. S., et al. Chromosomally unstable mouse tumours have genomic alterations similar to diverse human cancers. Nature. 447 (7147), 966-971 (2007).
- Rahrmann, E. P., et al. Forward genetic screen for malignant peripheral nerve sheath tumor formation identifies new genes and pathways driving tumorigenesis. Nat Genet. 45 (7), 756-766 (2013).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved
We use cookies to enhance your experience on our website.
By continuing to use our website or clicking “Continue”, you are agreeing to accept our cookies.