Method Article
遗传分析和基因组规模的辍学筛选以确定恶性周围神经鞘瘤小鼠模型中的治疗靶点
摘要
我们开发了一种跨物种比较肿瘤基因组学方法,利用基因组分析和功能基因组筛选来识别和比较基因工程小鼠模型中产生的肿瘤和相应的人类肿瘤类型的治疗靶点。
摘要
恶性周围神经鞘瘤 (MPNST) 来源于雪旺细胞或其前体。在肿瘤易感综合征 1 型神经纤维瘤病 (NF1) 患者中,MPNST 是最常见的恶性肿瘤,也是导致死亡的主要原因。这些罕见且具有侵袭性的软组织肉瘤前景广阔,5 年无病生存率为 34-60%。MPNST患者的治疗选择非常有限,其中毁容手术是最重要的治疗选择。许多曾经很有前途的疗法,如Ras信号传导抑制剂tipifarnib,在临床上都失败了。同样,靶向表皮生长因子(EFGR)的厄洛替尼和靶向血管内皮生长因子受体(VEGF)、血小板衍生生长因子受体(PDGF)和Raf的索拉非尼联合标准化疗的II期临床试验也未能在患者中产生反应。
近年来,功能基因组筛选方法与癌细胞系的遗传分析相结合,已被证明可用于识别基本的细胞质信号通路和靶点特异性疗法的开发。在罕见肿瘤类型的情况下,这种方法的一种变体被称为跨物种比较肿瘤基因组学,越来越多地被用于确定新的治疗靶点。在跨物种比较肿瘤基因组学中,在基因工程小鼠(GEM)模型中进行遗传分析和功能基因组学,然后在可用的稀有人类标本和细胞系中验证结果。
本文介绍了如何使用全外显子组测序(WES)鉴定人和小鼠MPNST细胞中的候选驱动基因突变。然后,我们描述了如何进行基因组规模的shRNA筛选,以识别和比较小鼠和人类MPNST细胞中的关键信号通路,并确定这些通路中的可成药靶点。这些方法为识别各种人类癌症类型的新治疗靶点提供了一种有效的方法。
引言
恶性周围神经鞘瘤 (MPNST) 是高度侵袭性的梭形细胞肿瘤,与肿瘤易感综合征 1 型神经纤维瘤病 (NF1) 相关,散发于普通人群和既往放疗部位1,2,3。NF1 患者出生时具有 NF1 抑癌基因的野生型拷贝和具有功能丧失突变的第二个 NF1 等位基因。这种单倍体功能不全的状态使 NF1 患者易受其野生型 NF1 基因的第二次功能丧失突变的影响,从而触发肿瘤发生。当这种"第二次命中"NF1突变发生在雪旺细胞谱系的细胞中时,产生的肿瘤要么是皮肤中出现的真皮神经纤维瘤,要么是发生在大神经或神经丛中的丛状神经纤维瘤。虽然真皮和丛状神经纤维瘤的病理学相同,但它们的生物学行为却大不相同——虽然真皮和丛状神经纤维瘤都是良性的,但只有丛状神经纤维瘤才能发生转化并产生MPNST。除了神经纤维蛋白(由 NF1 基因编码的 Ras GTP 酶激活蛋白)的缺失外,MPNST 还携带多种其他抑癌基因的突变,包括 TP53 4,5,6,7、CDKN2A 8,9 和 PTEN 10,编码多梳抑制复合物 2 11,12 (PRC2; SUZ12 和 EED 基因)和受体酪氨酸激酶的异常表达 1,2。NF1 和上述其他基因的突变也存在于散发和辐射诱导的 MPNST中 11,12。
虽然我们对MPNSTs基因组异常的理解的这些进展对于理解其发病机制非常宝贵,但它们尚未导致MPNSTs的有效新疗法的开发。阻碍新疗法开发的一个主要障碍是MPNST是罕见的癌症。正因为如此,很难获得定义关键驱动突变的全球分析所需的大量患者样本,例如癌症基因组图谱 (TCGA) 进行的分析。根据我们的经验,即使积累少量的人类MPNST标本也可能需要数年时间。为了克服这些局限性,许多研究其他罕见癌症类型的研究人员已转向使用跨物种比较肿瘤基因组学来识别重要的驱动基因突变,定义其感兴趣的肿瘤中的基本细胞质信号通路,并确定新的治疗靶点。由于对肿瘤发生至关重要的信号通路在人类和其他脊椎动物物种之间高度保守,因此应用功能基因组学方法(如基因组规模的shRNA筛选)可以成为识别这些新的驱动突变、信号通路和治疗靶点的有效手段13,14,15,16,17,18,19,特别是在研究有限的人类肿瘤类型时,数量限制为20。
在这里介绍的方法中,我们描述了这种在人 MPNST 细胞系和源自 P 0-GGFβ3 小鼠的早期传代 MPNST 培养物中进行基因组分析的方法,这是一种基因工程小鼠模型 (GEM),其中生长因子神经调节蛋白-1 (NRG1) 的雪旺细胞特异性过表达促进丛状神经纤维瘤的发病机制及其随后进展为 MPNST21,22,23.该方法的第一步是鉴定 P 0-GGFβ3 MPNSTs、人 MPNST 细胞系和手术切除的人 MPNST 中的候选驱动基因。为了在功能上验证受这些突变影响的信号通路,我们然后使用基因组规模的shRNA筛选来鉴定人和小鼠MPNST细胞系中增殖和存活所需的基因。在确定增殖和存活所需的基因后,我们使用药物基因相互作用数据库在"命中"集合中识别可成药的基因产物。我们还比较了人和小鼠MPNST细胞中的"命中",以确定GEM模型和人类MPNST是否表现出对相同基因和信号通路的相似依赖性。鉴定增殖和存活所需的基因重叠以及受影响的信号通路可作为在分子水平上验证P 0-GGFβ3小鼠模型的一种手段。这种方法还强调了结合人类和小鼠筛选来识别新的治疗靶点的有效性,其中小鼠模型可以作为人类筛选的补充。在人类肿瘤和细胞系难以获得的罕见肿瘤中寻找治疗靶点时,这种跨物种方法的价值尤为明显。
研究方案
在研究开始之前,由机构动物护理和使用委员会 (IACUC) 和机构生物安全委员会 (IBC) 审查和批准处理病毒载体的动物程序和方案。此处描述的程序已获得南卡罗来纳医科大学的 IACUC 和 IBC 董事会的批准,并由经过适当培训的人员根据 NIH 实验动物护理和使用指南和 MUSC 的机构动物护理指南执行。
1. WES-Seq分析与致病变异鉴定
- 使用基于标准市售吸附硅胶的方法,从肿瘤样品或在 60 mm 培养皿上生长的亚汇合 (70%) MPNST 细胞中分离基因组 DNA(有关详细步骤,请参阅制造商的方案)。一般工作流程如图 1 所示。
- 将至少 10 μL 的 50 ng/μL 基因组 DNA 提交到测序核心,除非指定了不同数量或浓度的基因组 DNA。
- 核心设施通过超声处理片段化基因组DNA,然后使用首选方法纯化。使用优选的外显子组测序试剂盒进行外显子组捕获和文库构建,并将索引标签添加到扩增捕获的外显子组中。
- 将标本提交到测序核心,进行全外显子组双端测序(WES;从两端测序 100 bp)。
- 核心生成的 FASTQ 文件将提供给研究者。仅使用传递质量指标进行分析的 FASTQ 文件。
- 使用市售的软件程序(例如,DNAStar19,20,Partek21或Varsome)对齐和分析FASTQ文件。使用默认设置将FASTQ文件与小鼠参考基因组GRCm38 / mm10对齐。
注:以DNAStar和小鼠MPNST标本为例,一般工作流程如图所示 图1 并在下面简要解释。- 打开DNAStar软件,选择SeqMan NGen工作流程。
- 通过选择 变异分析/重测 序和测序分析类型( 基于 NGS 的扩增子、基因panel或外显子组)来选择工作流程。单击 "下一步"。
- 通过选择下载基因组包和适当的参考 基因组 (即 Mus_musculus-GRCm38-dbSNP146.zip 或 Homo sapien-GRCh37.p13.zip 来选择首选参考序列。如果核心设施提供了床位文件,请上传此辅助文件。单击 "下一步"。
- 通过选择适当的测序仪技术 Illumina 来选择输入序列,并指定 测序读数 是 成对端的。然后,选择实验设置、多样本,并通过选择 "添加"上传测序 FASTQ 文件。如果运行 多个样品,请使用唯一的实验或样品名称 Tumor、Cell line 或 A18、A202... 单击 "下一步"。
- 设置控制数据集(如果有)。单击"下一步",然后在"程序集选项"下再次单击"下一步"。在"分析选项"下,单击相应的"变异检测"模式作为"二倍体",然后单击"下一步"。在"程序集输出"下,命名并指定项目的文件保存位置;单击"下一步"。在本地计算机或云上运行程序集。
注:生成的比对文件可以在 ArrayStar 工作流程中打开,用于对检测到的单核苷酸多态性(SNP)进行变异注释。此工作流程检测的是 SNP,而不是基因拷贝数的增加或丢失。一种称为高密度 SNP 阵列的单独分析可检测拷贝数变化;全外显子组测序不能可靠地检测拷贝数变化。有关对准程序要执行的具体步骤,请参阅程序用户指南。 - 将用户定义的过滤器应用于检测到的 SNP 的变体注释,以包含或排除某些数据。要获得可能与功能相关的变异的精简列表,请将以下过滤器应用于此层次结构中的变异数据集:非对照(如果有正常 对照 样本)、 群体 频率(gnomAD、ExAC、1,000 个基因组频率)、 等位基因频率 (包括 ≤0.001 或 0.1%)、 覆盖深度 (不包括 <10 深度)、 致病性 或 ClinVar 等级 (包括:致病性、可能致病性;排除:不确定、可能是良性、良性),如果需要, SNP类型和编码影响 (包括:非同义词、错义、无意义、移码、框内、剪接)。
注意:已知的相关癌症基因列表也可以应用于最终列表,以仅提取特定的疾病相关基因,参见步骤 1.3.7。这些过滤器可以将变异基因列表减少到少于 20 个基因。- 使用变异等位基因频率过滤掉测序错误 SNP。 纯合子和杂合子变体将分别以大约 100% 和 50% 的比例表示纯非污染细胞群(已建立的肿瘤衍生细胞系应代表同基因肿瘤细胞的单个细胞群)。在这种情况下,对变异应用 100-90 % 和 50-40% 的等位基因频率 作为过滤器,从而去除低于该频率的任何变异。如果高密度 SNP 阵列数据也可用于同一数据集,则将拷贝数 增益 或 丢失 应用于变异等位基因频率 小于纯合或杂合比率的 SNP (即,拷贝数增加导致 2 个等位基因 A 拷贝和 1 个等位基因 B 拷贝将使适当的变异等位基因频率达到 75%、25%、 分别)。
- 使用ArrayStar鉴定致病变异后,将变异基因列表导出并保存为csv、txt或xls文件,并将包含这些突变的基因与P 0-GGFβ3产生的肿瘤队列中的基因进行比较,以确定重叠和独特的突变基因列表。将突变基因与与人类对应物相关的已知突变基因进行比较(即 Bushman Lab 癌症基因列表或用户策划的列表)。
- 可选,在应用任何用户定义的过滤器 (1.3.6) 之前,将注释文件导出并另存为 VCF 文件。
注:此 VCF 文件可以上传到其他变异效应器预测软件 Varsome 或 VEP 进行比较,作为二次分析进行比较。也可以对过滤后的变异基因列表进行生物学和通路分析。
- 可选,在应用任何用户定义的过滤器 (1.3.6) 之前,将注释文件导出并另存为 VCF 文件。
- 通过用户首选的数据库对变异基因列表进行功能分类,获取蛋白质类别、通路、通路组成以及基因家族和本体信息。
注意:此处以 PANTHER (pantherdp.org) 为例。- 上传带有基因 ID 的过滤基因列表。
- 选择生物体(Mus musculus)。
- 选择分析(在基因列表中查看功能分类),然后单击在过滤后的变异基因列表中 提交 生物学和通路分析。
2. 基因组规模shRNA筛选
注:有几种shRNA和CRISPR文库可用于低传代肿瘤培养的基因组规模功能筛选。在这里,我们以CELLECTA DECIPHER shRNA文库为例进行描述。CELLECTA DECIPHER 慢病毒 shRNA 文库针对 RNAi 基因筛选进行了优化,采用混合形式。每个转录本由至少 5-6 个独特的 shRNA 靶向,每个慢病毒 shRNA 载体都包含一个独特的遗传条形码,两侧是 PCR 引物位点。这些文库涵盖了大多数人类和小鼠疾病相关基因,但并未涵盖基因组中的所有基因。Cellecta 人文库质粒 DNA 库有三个模块(人模块 I、II、III;靶标 15,377 个基因),而小鼠文库质粒库有两个模块(小鼠模块 I 和 II;靶标 9,145 个基因)。这些文库用于进行"脱落"测定,其中增殖和/或存活所需的靶基因在病毒转导后的不同时间点差异表达。
- 慢病毒包装
- 第 0 天:将 10 个培养皿(1000 万个 293T 细胞/15 cm 培养皿)接种在含有 10% 胎牛血清 (FBS) 的 30 mL/培养皿中,不含抗生素的 DMEM。
- 第 1 天:确认细胞在第二天汇合度达到 ~80% 并准备转染。在 50 mL 锥形管中,按以下顺序混合以下内容:600 μL 包装质粒混合物 (0.5 μg/μL)、60 μL 质粒条形码库、12 mL DMEM(不含血清或抗生素)和 600 μL 转染试剂。在室温下孵育15分钟。
- 将 900 μL 转染试剂和 12 mL DMEM 放入单独的 15 mL 锥形管中,并通过涡旋混合。向 DNA 混合物中加入 12.9 mL 转染试剂/DMEM 混合物,轻弹混匀。在室温下孵育 15 分钟,无需进一步混合。将 2.5 mL 该混合物逐滴添加到每个 15 cm 的 293T 细胞培养皿中,并在组织培养箱中孵育过夜。
- 第 2 天:第二天用含有抗生素的常规生长培养基替换培养基。
- 第 3 天:通过收集培养基并将其通过 0.2 μm 过滤装置并分装到 15 mL 锥形管中来收获病毒;还要在冻存管中制备 5 份 1 mL 过滤病毒等分试样,用于病毒滴度测定(被认为是 48 小时病毒);将病毒储存在-80°C冰箱中。用 30 mL 生长培养基替换培养基,一次一个,以避免细胞变干。
- 第 4 天:通过收集培养基并将其通过 0.2 μm 过滤装置来收获病毒。将病毒分装到 15 mL 锥形管中。这被认为是 72 小时病毒;将病毒储存在-80°C冰箱中。
- 滴度慢病毒池
- 将 65 μL 阳离子聚合物 (10 mg/mL) 加入 65 mL 肿瘤细胞生长培养基中。将 1 mL/孔含聚合物的培养基移液到 11 个 6 孔组织培养板中。胰蛋白酶消化早期传代肿瘤细胞并使用优选方法对细胞进行计数,使每个孔接受 50,000 个细胞/mL/孔。在37°C水浴中从冰箱中解冻1mL等分试样的48小时慢病毒。
- 对于每个病毒模块,如 表1所示准备感染。
- 将所有 6 孔板放入组织培养箱中。第二天,去除病毒培养基并补充新鲜的生长培养基。在48小时,将含嘌呤霉素的培养基加入所有接受选择的孔中。在对照细胞汇合之前,使用首选方法对存活的克隆进行细胞计数。
注:杀死非转导细胞所需的嘌呤霉素浓度必须通过测试"杀伤"曲线中的浓度范围来凭经验预先确定。使用均匀诱导非转导培养物死亡的最低浓度。
- 靶细胞的慢病毒感染
- 胰蛋白酶消化MPNST细胞并计数。每个 15 cm 培养皿接种 250 万个细胞,每个模块总共 20 个 15 cm 培养皿。解冻病毒,并在5μg/ mL阳离子聚合物存在下以0.5的MOI转导细胞(图2A)。
- 第二天早上取出含病毒的培养基,并用新鲜的生长培养基替换。将细胞再培养 2 天以表达选择标记物,然后将嘌呤霉素添加到培养物中以消除非转导细胞。
- 加入嘌呤霉素三天后,胰蛋白酶消化细胞,并在台式离心机中以200×g离心5分钟。将沉淀的细胞储存在-80°C冰箱中,用于将来的基因组DNA制备;这是参考时间点或时间点 1 (T1)。
- 重新铺板另一半细胞群,并在收获和离心之前将其生长约7个细胞群倍增,如上所述。该细胞沉淀将作为最终时间点(时间点 2,T2),并储存在-80°C下用于将来的基因组DNA分离(图2)。
- 从病毒库转导的细胞中分离基因组DNA
- 从-80°C冰箱中解冻细胞沉淀,并将沉淀重悬于加入RNAse的10mL重悬缓冲液中,并立即分裂成两个15mL聚甲基戊烯管(图2B)。
- 每 5 mL 加入 500 μL 10% SDS,混合并在室温下孵育 5 分钟。接下来,将试管放入DNA剪切装置中,对DNA进行25个周期的超声处理,每次30秒开/30秒关,确保温度保持在4°C。
- 加入5mL苯酚/氯仿,pH 8.0(储存在4°C),确保在使用前充分混合苯酚/氯仿。加入苯酚/氯仿后,在最大设置下剧烈涡旋45-60秒,充分混合。离心60分钟,-20°C,7,200× g。
注意: 涡旋后出现泡沫/乳白色外观将表示完全重悬。 - 将 3 mL 的透明上相转移到新鲜的 15 mL 管中,加入 0.5 mL 的 3 M 乙酸钠和 4 mL 异丙醇并充分混合(图 2C)。在20°C下以7,200× g离心30分钟。
- 离心后,弃去上清液,然后小心地移出剩余的液体。加入 0.5 mL 70% 乙醇,上下移液去除沉淀。将重新悬浮的沉淀转移到 1.5 mL 离心管中,并将来自同一样品的两种沉淀合并到单个 1.5 mL 离心管中。在台式离心机中以最大速度离心5分钟。
- 弃去上清液,用实验室擦拭吸收任何残留的70%乙醇。将沉淀重新悬浮在 0.5 mL 蒸馏水中,确保不要让沉淀变干,因为这会使重悬变得困难。在测量DNA浓度之前将样品储存在4°C。
- 巢式PCR扩增shRNA条形码
注意:从储存中取出 DNA 并置于冰上。如果 DNA 浓度低于 100 ng/μL,则快速真空干燥 DNA 并重悬于适当体积的蒸馏水中。我们建议一次只处理一个模块(时间点 1 (T1) 或时间点 2 (T2))。对每个时间点进行两次准备,最后进行合并;因此,重要的是不要在同一天处理复制时间点。以下方案适用于基因组DNA的一个时间点。- 设置7个试管并按描述标记它们:阴性对照,阳性对照,4个用于基因组DNA的试管,以及一个用于预混液的试管(图3A)。阴性对照为蒸馏水;阳性对照是用于 293T 转染生成慢病毒的质粒 DNA。
- 如 表2所示,首先向每个管子加水。
- 如 表3所示制备预混液(MM)。
- 向每个装有水的试管中加入 18 μL MM。接下来,向 4 个模板管中的每一个中加入 25 μL 基因组 DNA。然后,向阳性对照管中加入 1 μL 阳性对照 (10 ng/μL),注意不要用阳性对照 DNA 污染其他管。最后,向每个试管中加入 2 μL 聚合酶,先加入 4 个样品管,最后加入阳性对照管;混合并旋转PCR管(图3)。
- 在 表4所述条件下进行PCR反应。
- 在运行第一次PCR时,如表5所示,为第二次PCR准备试管。设置七个试管:阴性对照,阳性对照,4个基因组DNA管和一个MM管(图3B)。
- 向每个装有水的试管中加入 22 μL MM,等待第一轮 PCR 完成,然后再加入 DNA 和聚合酶。
- 第一轮PCR完成后,将4个样品管合并到一个微量离心管中并混合。
- 向每个装有水的样品管中加入 25 μL。
- 接下来,将 2 μL 的第一个 PCR 阴性对照和 2 μL 的第一个 PCR 阳性对照加入适当标记的试管中。
- 向每个试管中加入 2 μL 聚合酶,先加入 4 个样品管,最后加入阴性对照。
- 如 表6所述进行PCR反应。
注:在3.5%琼脂糖凝胶上通过电泳分析第一次PCR反应的结果后,如果获得丰度的产物,则下一次重复该过程的循环次数可以减少到9-10次;同样,如果产品产量较低,则可以将循环次数增加到 14 次。 - 第二次PCR反应完成后,将4个样品合并到一个微量离心管中,加80μL的6x上样染料。对于阳性和阴性对照,使用 20 μL 样品。
- 在Tris-硼酸盐-EDTA(TBE)缓冲液中制备3.5%琼脂糖凝胶(图4A)。为了容纳大样品体积,通过将几个孔粘在一起(如果没有适合该体积的梳子),在凝胶梳中创建一个大孔,确保为阳性和阴性对照留出两个孔。将凝胶在90V下运行1小时。
- 观察凝胶并确认大约 250 个碱基对 (bp) 的条带。
- 第一次纯化:凝胶提取
- 使用干净的手术刀或剃须刀片切除 250 bp 条带,切除尽可能多的凝胶。将大条带切成 4 块,并将每块转移到干净的微量离心管中。测量并记录每个凝胶切片的重量。
注意:每管应约为 200 毫克或更少。 - 加入 6 体积的增溶缓冲液(例如,如果凝胶片重 200 mg,则加入 1.2 mL 缓冲液)。将试管置于50°C水浴中,每10-15分钟旋转一次,直到凝胶切片溶解。然后,向每个试管中加入 1 体积的异丙醇(例如,如果凝胶片重 200 mg,则加入 0.2 mL 异丙醇)。
- 使用两个离心柱进行纯化,将所有 4 个试管中的样品加载到柱中。进行多次离心以处理所有样品体积,因为色谱柱仅可容纳 750 μL。 在传统的台式微量离心机中以 17,900 × g 旋转色谱柱 1 分钟,每次丢弃流出物。
- 用 750 μL 洗涤缓冲液洗涤色谱柱,并以 17,900 × g 离心 1 分钟。洗涤后,丢弃流出物并通过以17,900 × g 离心3分钟来干燥离心柱。每列用 50 μL 蒸馏水洗脱 DNA,并像以前一样离心 1 分钟,并将两个管合并,使总体积的样品为 100 μL。
- 使用干净的手术刀或剃须刀片切除 250 bp 条带,切除尽可能多的凝胶。将大条带切成 4 块,并将每块转移到干净的微量离心管中。测量并记录每个凝胶切片的重量。
- 二次净化
- 下一个纯化步骤将使用结合缓冲液 2(来自 PCR 纯化试剂盒),它不能消除小片段。将 400 μL 缓冲液加入 100 μL DNA 中,并将其加载到一个离心柱上(图 4B)。将样品以17,900× g 离心1分钟。
- 接下来,用 650 μL 洗涤缓冲液洗涤膜并按照先前的方法旋转。如上所述,用额外的离心干燥离心柱3分钟。
- 用 30 μL 蒸馏水洗脱 DNA,并以最大速度旋转 1 分钟。洗脱后,在分光光度计上检查浓度;确保浓度不低于 10 ng/μL 或高于 70-80 ng/μL。 将DNA储存在-20°C冰箱中。
- 扩增条码的测序
- 对于测序,使用洗脱缓冲液 (EB) 将纯化的条形码稀释至 0.75 ng/μL。为了增加序列多样性,扩增子以 17 pM 聚集,包括 30% (v/v) PhiX。根据制造商的协议,在自动集群生成系统上执行单端 (SE) 集群。
- 让测序核心在NextGen测序仪上执行总共36个周期的单端测序(图4C)。将定制引物GexSeqS以0.5μM的浓度添加到Illumina测序引物中。
- 使用参考的分析软件生成 Fastq 文件,并使用软件对其进行处理,以将读取长度修剪为 18 个核苷酸。
- 使用条形码分析仪和反卷积软件对修剪的读取进行去卷积。计算每个shRNA的倍数耗竭分数,作为参考时间点(T1)与最终时间点(T2)的读取计数之比。
- shRNA筛选结果分析及候选治疗靶点鉴定
注:在 Cellecta shRNA 文库中,大多数基因由 5 个 (67%) 或 6 个不同的 shRNA (32%) 靶向。然而,在大多数细胞中应该被命中的几个管家基因被大量shRNA靶向,并作为定义阴性分数范围的对照。- 为了防止偏向于大量shRNA靶向的基因,对数转换耗竭分数。通过从其经验分布中计算每个基因的第 80 个百分位数来执行分位数估计。
- 要生成对数倍数耗竭分数的零分布,假设 >95% 的基因不会被耗竭,并且它们的对数分位数分数将具有正态分布。使用经验分布的中位数来估计原分布的均值。使用此零分布,将对数倍数耗竭分数大于零分布的第 95 个百分位数的所有基因分类为"命中"。
注意:所有命中应至少具有两个耗竭分数高于临界点的shRNA(图5)。 - 为了评估 RNAi 脱落筛选数据的质量和临界点的有效性,请使用一组被 COLT 癌症 RNAi 筛查倡议24,25 定义为"核心必需"的基因进行比较。从命中列表中删除 CEG,以生成潜在治疗靶点的初始列表。
注:在COLT筛选的72个癌细胞系中,COLT集中的基因在>50%中得分为命中。核心必需基因列表包含 640 个基因。 - 为了确定多个MPNST细胞系通常或一致需要的潜在治疗靶点,构建在不同MPNST细胞系或早期传代培养物的筛选中鉴定的非CEG命中的维恩图(图6A)。优先考虑所有或大多数 MPNST 品系或文化中所需的非 CEG 命中。
- 或者,对每个细胞系或早期传代培养物的非 CEG 命中列表进行通路分析,以鉴定其产物编码肿瘤细胞增殖和/或存活所需的信号通路组分的基因。然后,将非 CEG 通路分析中鉴定的信号通路与鉴定为受 WES 鉴定突变影响的信号通路进行比较。
注:我们发现识别始终受WES中鉴定的突变影响的信号通路,并将其与shRNA筛选中确定为关键的信号通路进行比较特别有用。
- 或者,对每个细胞系或早期传代培养物的非 CEG 命中列表进行通路分析,以鉴定其产物编码肿瘤细胞增殖和/或存活所需的信号通路组分的基因。然后,将非 CEG 通路分析中鉴定的信号通路与鉴定为受 WES 鉴定突变影响的信号通路进行比较。
- 为了确定已经可用的治疗药物的可成药靶点,使用药物基因相互作用数据库 (dgidb.org) 筛选去除核心必需基因后剩余的命中列表。
注:该数据库允许同时输入和筛选基因列表,并提供有关目前可用于靶向这些基因的药物的指导。 - 首先,通过用与初始文库筛选中使用的shRNA不同的shRNA敲低其基因表达,以单批次形式(而不是刚才描述的文库批次格式)来验证已鉴定的可成药的高兴趣靶标。要敲低基因表达,请使用两到三个不同的慢病毒 shRNA。感染后三到四天,确定其对肿瘤细胞增殖和存活的影响,如下所述。
3. 对候选治疗剂攻击的 MPNST 细胞的细胞数量和活力进行细胞计数器检测
- 在DMEM中培养MPNST细胞至80%汇合度。用室温汉克斯平衡盐溶液(HBSS)冲洗细胞。
注意:不要将细胞培养到汇合,因为这些细胞的生长在重新电镀后会滞后一段时间。 - 通过用非酶细胞解离溶液覆盖细胞30秒至1分钟,将细胞从基质上分离。每 1 mL 解离溶液加入 5 mL DMEM,轻轻上下移取细胞以从底物上分离。
- 使用血细胞计数器计数细胞。在黑壁 96 孔板中以每孔 1,200 个细胞的密度铺板细胞;为将要测试的每种药物稀释度铺板至少三个孔,并对这些实验进行三次生物学重复。
- 为了确定将要测试的药物的初始浓度,请查看文献以评估哪些浓度对其他癌细胞类型有效。在最初的实验中,测试比其他癌症类型使用的药物浓度高两个数量级和低于两个数量级的范围。
- 准备待测药物的稀释液,并将每种稀释液或载体添加到至少三个重复孔中。
- 评估加药后 1、3、5 和 7 天的直接细胞数。加入Hoechst 33342至终浓度为5μg/ mL,并在37°C下孵育板30分钟。 使用 直接细胞计数总细胞数 选项在高通量成像细胞仪上读取孔板, 曝光时间为 100,000 毫秒。
- 使用软件分析读数并将其导出到电子表格中,并使用适当的软件进行统计分析。
- 如果在药物处理的孔中观察到细胞数量在统计学上显着减少,则进行"活/死"测定以确定这种减少是否部分归因于细胞死亡的诱导。
- 如步骤3.3所述,在黑壁96孔板中铺板细胞。
- 按照步骤3.5中的说明准备和添加药物。
- 在加药后 1、3、5 和 7 天评估细胞活力和死亡。在每个孔中加入钙黄绿素AM至终浓度为1μM,加入碘化丙啶至终浓度为1μM。将细胞在37°C孵育15分钟。
- 使用 Live+Dead 软件选项在成像细胞仪上对细胞进行成像。使用软件分析读数并将其导出到电子表格中,并使用适当的软件进行统计分析。
结果
图 5 图显示了在筛选的每个人细胞系中标记为 TRUE 的核心必需基因 (CEG) 与非 CEG(标记为 FALSE)的耗竭评分。点表示单个基因的倍数耗竭分数的 log2,这些分数绘制在总体分数分布的箱线图表示上。采用Student's t检验检验各细胞系两组平均耗竭评分差异有统计学意义。生成的 p 值显示在每个面板中。请注意,CEG 的平均倍数耗竭评分明显高于非 CEG。这是意料之中的,因为根据定义,核心必需基因是大多数细胞类型增殖和/或存活所必需的。
图6A显示了三种人MPNST细胞系的"命中"维恩图。我们通常会发现大量基因在多个品系之间共享;这些命中是高优先级的,因为它们代表了编码蛋白质的基因,这些蛋白质可能对大量MPNST的增殖和/或存活至关重要。 另请注意,有许多基因仅在一个细胞系中被命中。我们经常遇到这种情况,不应将其视为屏幕质量差的迹象。然后使用药物基因相互作用数据库评估多系之间常见命中的基因,以识别该子集中编码可用现有药物成药的蛋白质的基因。然后,我们选择其中的几个,并通过用shRNA敲低相应mRNA的表达来执行初始验证。由于一些shRNA会产生脱靶效应,因此我们总是测试靶向同一转录本的多个shRNA。图 6B 显示了一个代表性结果,其中我们用非靶向对照和靶向 BCL6 的多个 shRNA 转导了 MPNST 细胞。然后在转导后的不同时间测定细胞数量。请注意,一些 BCL6 shRNA 显着减少了细胞数量;如随附的免疫印迹所示,细胞数量的减少程度与BCL6敲低的程度相关。图6C显示了早期传代P 0-GGFβ3 MPNST培养物的代表性生长曲线。

图 1:对 MPNST 组织或早期传代 MPNST 细胞进行全外显子组测序的工作流程。示意图说明了肿瘤来源的早期传代培养物中存在的变异检测的一般工作流程。从早期传代培养物中分离 DNA (1),并根据其提交方案将高质量 DNA 提交到测序核心 (2)。测序核心将检查提交的DNA的质量,并执行所有必要的样本和基因组文库制备。核心设施将为用户提供具有质量控制指标的 FASTQ 测序文件 (3)。用户会将 FASTQ 文件上传到他们选择的基因组比对和变异调用程序。(4) 根据用户定义的标准过滤带注释的变体,以删除不相关的变体。所示的代表性数据比较了切除的人 MPNST 肿瘤样本与源自肿瘤的细胞系26。(5)使用PANTHER进行功能分类分析。缩写:MPNST = 恶性周围神经鞘瘤。请点击这里查看此图的较大版本.
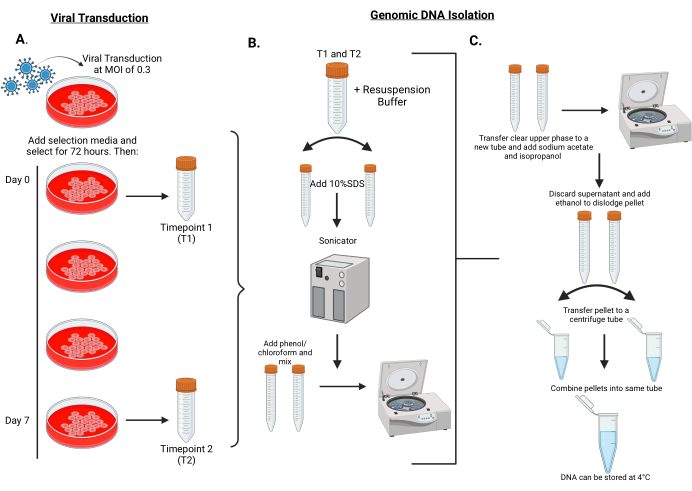
图 2:在时间点 1 和时间点 2 将 shRNA 文库转导到 MPNST 细胞中并从细胞中分离基因组 DNA 的工作流程 。 (A)用带条形码的慢病毒颗粒以0.3的低MOI感染靶细胞,并选择72小时。将细胞传代5-7个群体倍增(约7天)。将第0天和第7天的细胞沉淀储存在-80°C下用于基因组DNA分离。第 0 天称为时间点 1 (T1),第 7 天称为时间点 2 (T2)。(B) 基因组 DNA 分离首先将细胞沉淀重悬于重悬缓冲液中,然后将其分成两个 15 mL 试管。为了促进细胞裂解,将10%SDS加入每个试管中,并超声处理25个周期,每次30秒开/30秒关。超声处理后,将苯酚/氯仿加入每个管中,并剧烈涡旋45-60秒。然后离心管。(C) 移出透明的上相,加入醋酸钠/异丙醇并充分混合到干净的试管中。再次离心管。这一次,弃去上清液,加入70%乙醇除去沉淀。将重悬的颗粒合并到一个管中,并在台式离心机中以最大速度旋转。弃去上清液,将沉淀重悬于蒸馏水中。 请点击这里查看此图的较大版本.
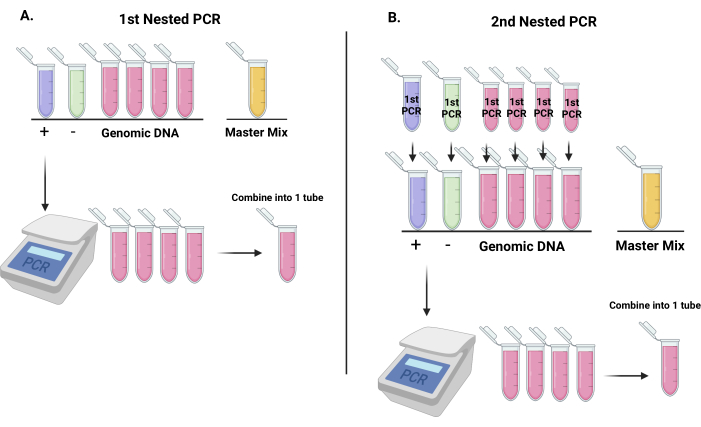
图 3:从慢病毒 shRNA 载体中扩增条形码序列的工作流程,为使用下一代测序对条形码进行定量做准备。 (A) 如何为第一个巢式 PCR 反应设置试管的表示(7 个试管:一个用于阴性对照,一个用于阳性对照,其余 4 个试管用于基因组 DNA。最后一根管将用作预混管)。在第一次PCR反应后,将基因组DNA管合并到一个管中并混合。(B) 第一次巢式PCR反应的产物将作为第二次巢式PCR反应的模板。在第二次PCR之后,将基因组DNA管合并到一个管中并混合。请点击这里查看此图的较大版本.
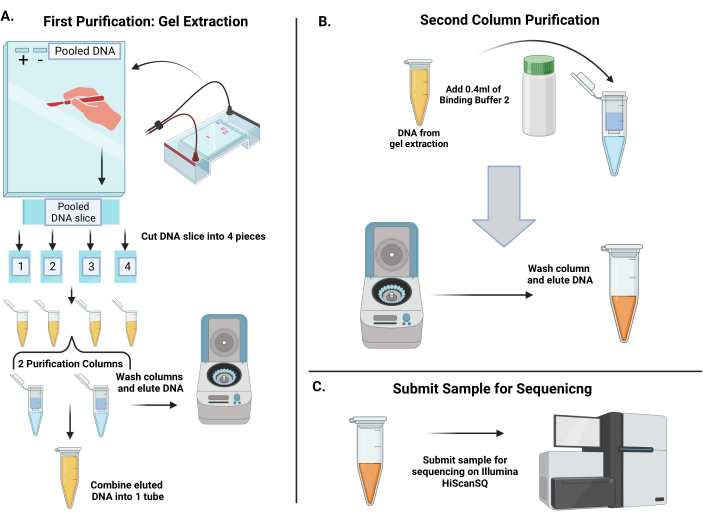
图 4:纯化扩增的 shRNA 条形码和测序条形码以量化其在时间点 1 和时间点 2 的表示的工作流程 。 (A) 倒入 3.5% 琼脂糖凝胶。由于合并的 DNA 的体积超过了一个孔的极限,因此将凝胶的 4-6 个齿梳在一起以产生一个大孔。用 6x 上样染料制备 PCR 产物,并将阳性对照、阴性对照和混合 DNA 上样到凝胶中。电泳后,合并的DNA泳道中应出现约250个碱基对的大条带。使用干净的手术刀,切除整个带子,然后切成 4 片凝胶片。溶解凝胶片段,然后将它们合并到两个离心柱中以洗脱DNA。将洗脱的DNA合并到一个试管中。(B) 通过第二个纯化步骤纯化 DNA。将结合缓冲液 2 加入混合 DNA 管中,然后移液到离心柱上。洗涤膜,然后在蒸馏水中洗脱DNA。(C) 将纯化的 DNA 提交到测序核心。 请点击这里查看此图的较大版本.
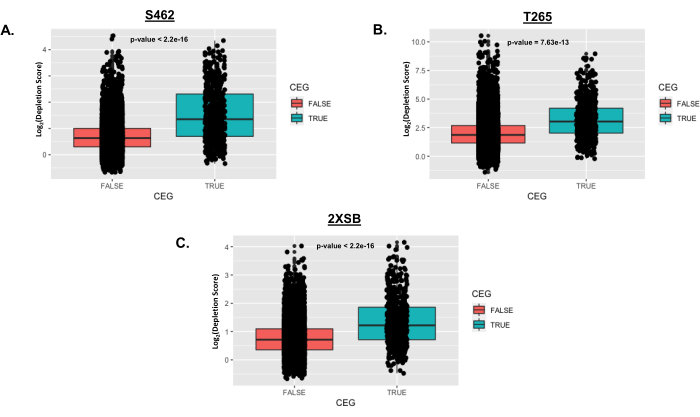
图 5:方案中描述的分析后核心必需基因分布的代表性示例。 在本例中,使用 Cellecta DECIPHER shRNA 文库筛选了三种人 MPNST 细胞系(S462、T265 和 2XSB)细胞。对于每个人类 MPNST 细胞系,创建一个箱线图来比较核心必需基因列表中基因的基因水平耗竭评分 (CEG;真箱线图)25 到在 CEG 列表中未找到的基因(假箱线图)。单个数据点分层在每个箱形图的顶部。P 值来自标准 t 检验,比较 CEG 与非 CEG 的基因水平耗竭评分。缩写:CEG=核心必需基因;MPNST = 恶性周围神经鞘瘤。 请点击这里查看此图的较大版本.
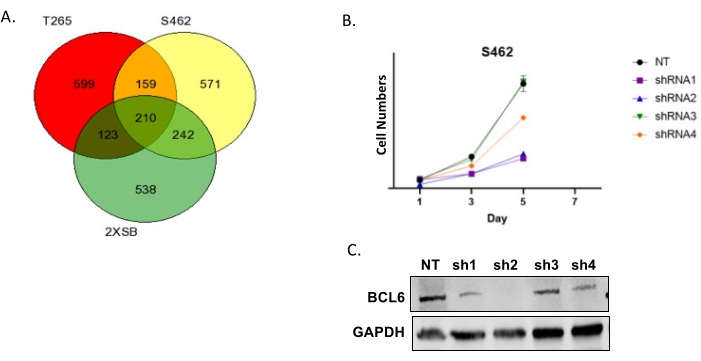
图 6:屏幕结果的验证。 (A) 三种人 MPNST 细胞系中重叠命中的代表性维恩图。(B) 用非靶向 (NT) 慢病毒载体和表达四种不同 BCL6 shRNA(shRNA1、shRNA2、shRNA3 和 shRNA4)的慢病毒转导的 S462 人 MPNST 细胞。用慢病毒转导细胞,然后用选择剂(嘌呤霉素)处理 3 天。然后在接下来的七天内评估细胞数量。(C) 蛋白质印迹分析显示 NT、shRNA1、shRNA2、shRNA3 和 shRNA4 慢病毒转导后 BCL6 的蛋白水平。请点击这里查看此图的较大版本.
表 1:慢病毒滴度测定的板布局。请按此下载此表格。
表2:首次PCR反应的初始设置。请按此下载此表格。
表3:用于首次PCR反应的预混液的制备。请按此下载此表格。
表4:Cellecta首次PCR参数。请按此下载此表格。
表5:第二次PCR反应的初始设置。请按此下载此表格。
表6:Cellecta第二次PCR参数。请按此下载此表格。
讨论
本文介绍的详细方法旨在研究周围神经系统肿瘤和MPNST发病机制。尽管我们发现这些方法是有效的,但应该认识到,我们在这里描述的方法存在一些潜在的局限性。下面,我们将讨论其中的一些局限性以及在其他模型系统中克服这些局限性的潜在策略。
我们发现全外显子组测序可有效识别 P 0-GGFβ3 小鼠中感兴趣的突变。然而,应该认识到,整个外显子组测序本身也有局限性。首先,全外显子组测序不是鉴定融合基因产物的有效方法。这是因为大多数染色体断裂和随后的融合主要涉及基因间区域和内含子,因为这些区域代表了基因组的大部分。相反,我们发现具有双端读长的RNA-Seq可以更有效地识别融合基因。还有一个问题是,全外显子组测序如何有效地识别相对较大的染色体丢失区域。
尽管已经开发了几种算法来识别此类损失,但术语"全外显子组测序"本身具有误导性,因为即使在良好的运行中,外显子组的捕获也经常会遗漏多达 5-10% 的外显子区域。因此,我们通常用其他方法(如阵列比较基因组杂交(aCGH))来补充全外显子组测序。在确定收益和损失后,我们检查这些区间内的基因,并将它们与与人类对应物相关的已知驱动突变进行比较。然而,小鼠基因组比人类基因组更稳定27。因此,小鼠肿瘤通常不会表现出类似于人类肿瘤的染色体。相反,小鼠肿瘤的模式要简单得多,倾向于整个染色体或染色体的获得或损失,而局灶性缺失相对较少,这些缺失往往在强大的选择压力下发生22,23。
在进行基因组规模的shRNA筛选时,我们遇到了一些潜在的陷阱。我们遇到的最常见的问题之一是慢病毒载体转导到靶细胞中相对较差。我们最常发现,问题源于包装的慢病毒池滴度不当。由于早期传代小鼠肿瘤细胞培养物是一种有限的资源,因此许多研究人员将尝试使用另一种更容易获得的已建立细胞系来滴定他们的慢病毒。然而,这种方法的问题在于,慢病毒转导的效率可能因细胞类型而异。正是出于这个原因,我们建议在实验中使用的实际细胞上滴度慢病毒。我们还遇到了病毒滴度相对较低的问题。该问题通常反映出在生产包装病毒时 293T 细胞的转染不良。
在进行基因组规模的shRNA筛选时,可能会获得假阳性命中。正因为如此,一旦我们确定了我们最感兴趣的潜在成药靶点,我们总是会验证我们的shRNA筛选结果。通常,我们将使用两种不同的方法来验证高兴趣目标。首先,我们使用两种或多种与初始筛选中使用的shRNA不同的shRNA敲低基因表达,并确定这对肿瘤细胞增殖和存活的影响。其次,我们获得药物基因相互作用数据库中鉴定的药物,并确定其对肿瘤细胞增殖和存活的影响。我们同时使用这两种方法,因为我们遇到过shRNA起作用而药物不起作用的情况。至少在其中一些情况下,对整个外显子组序列数据集的检查表明,靶蛋白是由具有可能影响药物-蛋白质相互作用的突变的基因产生的。
上述方法将为研究者提供一种适用的方法,用于识别罕见肿瘤中发生的潜在驱动突变,在功能上识别增殖和存活所需的信号通路,并确定治疗开发的靶点。我们希望其他研究人员会发现这些方法有助于确定其他人类癌症的关键治疗靶点。不过,读者应该意识到,还有其他功能基因组方法可用于识别参与肿瘤发病机制的基因和编码潜在治疗靶点的基因。例如,CRISPR文库的使用方式类似于我们描述的shRNA文库。功能筛选也可以 在体内 进行,以鉴定促进肿瘤发生的基因。例如,基于睡美人转座子的体细胞诱变系统先前已被用于靶向雪旺细胞及其前体,从而鉴定出数百个参与 MPNST 发病机制的基因28。由于这些系统以不同的方式处理功能基因组学,我们建议研究者仔细考虑其计划实验的目标,并根据这些目标选择功能基因组学方法。
披露声明
作者没有要披露的利益冲突。
致谢
这项工作得到了美国国家神经疾病和中风研究所(R01 NS048353和R01 NS109655 to SLC;R01 NS109655-03S1 至 D.P.J.)、美国国家癌症研究所(R01 CA122804 至 S.L.C.)和国防部(X81XWH-09-1-0086 和 W81XWH-12-1-0164 至 S.L.C.)。
材料
| Name | Company | Catalog Number | Comments |
| Bioruptor Sonication System | Diagenode | UCD-600 | |
| CASAVA 1.8.2 | |||
| Cbot | Illumina, San Diego, CA | N/A | |
| Celigo Image Cytometer | Nexcelom | N/A | |
| Cellecta Barcode Analyzer and Deconvoluter software | |||
| Citrisolve Hybrid | Decon Laboratories | 5989-27-5 | |
| Corning 96-well Black Microplate | Millipore Sigma | CLS3603 | |
| Diagenode Bioruptor 15ml conical tubes | Diagenode | C30010009 | |
| dNTP mix | Clontech | 639210 | |
| Eosin Y | Thermo Scientific | 7111 | |
| Elution buffer | Qiagen | 19086 | |
| Ethanol (200 Proof) | Decon Laboratories | 2716 | |
| Excel | Microsoft | ||
| FWDGEX 5’-CAAGCAGAAGACGGCATACGAGA-3’ | |||
| FWDHTS 5’-TTCTCTGGCAAGCAAAAGACGGCATA-3’ | |||
| GexSeqS (5’ AGAGGTTCAGAGTTCTACAGTCCGAA-3’ | HPLC purified | ||
| GraphPad Prism | Dotmatics | ||
| Harris Hematoxylin | Fisherbrand | 245-677 | |
| Illumina HiScanSQ | Illumina, San Diego, CA | N/A | |
| Paraformaldehyde (4%) | Thermo Scientific | J19943-K2 | |
| PLUS Transfection Reagent | Thermo Scientific | 11514015 | |
| Polybrene Transfection Reagent | Millipore Sigma | TR1003G | |
| PureLink Quick PCR Purification Kit | Invitrogen | K310001 | |
| Qiagen Buffer P1 | Qiagen | 19051 | |
| Qiagen Gel Extraction Kit | Qiagen | 28704 | |
| RevGEX 5’-AATGATACGGCGACCACCGAGA-3’ | |||
| RevHTS1 5’-TAGCCAACGCATCGCACAAGCCA-3’ | |||
| Titanium Taq polymerase | Clontech | 639210 | |
| Trimmomatic software | www.usadellab.org |
参考文献
- Carroll, S. L. Molecular mechanisms promoting the pathogenesis of Schwann cell neoplasms. Acta Neuropathol. 123 (3), 321-348 (2012).
- Longo, J. F., Weber, S. M., Turner-Ivey, B. P., Carroll, S. L. Recent Advances in the Diagnosis and Pathogenesis of Neurofibromatosis Type 1 (NF1)-associated Peripheral Nervous System Neoplasms. Adv Anat Pathol. 25 (5), 353-368 (2018).
- Longo, J. F., Carroll, S. L. The RASopathies: Biology, genetics and therapeutic options. Adv Cancer Res. 153, 305-341 (2022).
- Birindelli, S., et al. Rb and TP53 pathway alterations in sporadic and NF1-related malignant peripheral nerve sheath tumors. Lab Invest. 81 (6), 833-844 (2001).
- Legius, E., et al. TP53 mutations are frequent in malignant NF1 tumors. Genes Chromosomes Cancer. 10 (4), 250-255 (1994).
- Menon, A. G., et al. Chromosome 17p deletions and p53 gene mutations associated with the formation of malignant neurofibrosarcomas in von Recklinghausen neurofibromatosis. Proc Natl Acad Sci U S A. 87 (14), 5435-5439 (1990).
- Upadhyaya, M., et al. Germline and somatic NF1 gene mutation spectrum in NF1-associated malignant peripheral nerve sheath tumors (MPNSTs). Hum Mutat. 29 (1), 74-82 (2008).
- Kourea, H. P., Orlow, I., Scheithauer, B. W., Cordon-Cardo, C., Woodruff, J. M. Deletions of the INK4A gene occur in malignant peripheral nerve sheath tumors but not in neurofibromas. Am J Pathol. 155 (6), 1855-1860 (1999).
- Nielsen, G. P., et al. Malignant transformation of neurofibromas in neurofibromatosis 1 is associated with CDKN2A/p16 inactivation. Am J Pathol. 155 (6), 1879-1884 (1999).
- Gregorian, C., et al. PTEN dosage is essential for neurofibroma development and malignant transformation. Proc Natl Acad Sci U S A. 106 (46), 19479-19484 (2009).
- Lee, W., et al. PRC2 is recurrently inactivated through EED or SUZ12 loss in malignant peripheral nerve sheath tumors. Nat Genet. 46 (11), 1227-1232 (2014).
- Zhang, M., et al. Somatic mutations of SUZ12 in malignant peripheral nerve sheath tumors. Nat Genet. 46 (11), 1170-1172 (2014).
- Varela, I., et al. Somatic structural rearrangements in genetically engineered mouse mammary tumors. Genome Biol. 11 (10), 100 (2010).
- Johnson, R. A., et al. Cross-species genomics matches driver mutations and cell compartments to model ependymoma. Nature. 466 (7306), 632-636 (2010).
- Kim, M., et al. Comparative oncogenomics identifies NEDD9 as a melanoma metastasis gene. Cell. 125 (7), 1269-1281 (2006).
- Zender, L., et al. Identification and validation of oncogenes in liver cancer using an integrative oncogenomic approach. Cell. 125 (7), 1253-1267 (2006).
- Uren, A. G., et al. Large-scale mutagenesis in p19(ARF)- and p53-deficient mice identifies cancer genes and their collaborative networks. Cell. 133 (4), 727-741 (2008).
- Starr, T. K., et al. A transposon-based genetic screen in mice identifies genes altered in colorectal cancer. Science. 323 (5922), 1747-1750 (2009).
- Dupuy, A. J., et al. A modified sleeping beauty transposon system that can be used to model a wide variety of human cancers in mice. Cancer Res. 69 (20), 8150-8156 (2009).
- Carroll, S. L. The Challenge of Cancer Genomics in Rare Nervous System Neoplasms: Malignant Peripheral Nerve Sheath Tumors as a Paradigm for Cross-Species Comparative Oncogenomics. Am J Pathol. 186 (3), 464-477 (2016).
- Huijbregts, R. P., Roth, K. A., Schmidt, R. E., Carroll, S. L. Hypertrophic neuropathies and malignant peripheral nerve sheath tumors in transgenic mice overexpressing glial growth factor beta3 in myelinating Schwann cells. J Neurosci. 23 (19), 7269-7280 (2003).
- Kazmi, S. J., et al. Transgenic mice overexpressing neuregulin-1 model neurofibroma-malignant peripheral nerve sheath tumor progression and implicate specific chromosomal copy number variations in tumorigenesis. Am J Pathol. 182 (3), 646-667 (2013).
- Brosius, S. N., et al. Neuregulin-1 overexpression and Trp53 haploinsufficiency cooperatively promote de novo malignant peripheral nerve sheath tumor pathogenesis. Acta Neuropathol. 127 (4), 573-591 (2014).
- Hart, T., Brown, K. R., Sircoulomb, F., Rottapel, R., Moffat, J. Measuring error rates in genomic perturbation screens: gold standards for human functional genomics. Mol Syst Biol. 10 (7), 733 (2014).
- Hart, T., et al. Evaluation and Design of Genome-Wide CRISPR/SpCas9 Knockout Screens. G3. 7 (8), 2719-2727 (2017).
- Longo, J. F., et al. Establishment and genomic characterization of a sporadic malignant peripheral nerve sheath tumor cell line. Sci Rep. 11 (1), 5690 (2021).
- Maser, R. S., et al. Chromosomally unstable mouse tumours have genomic alterations similar to diverse human cancers. Nature. 447 (7147), 966-971 (2007).
- Rahrmann, E. P., et al. Forward genetic screen for malignant peripheral nerve sheath tumor formation identifies new genes and pathways driving tumorigenesis. Nat Genet. 45 (7), 756-766 (2013).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。