Method Article
Profilage génétique et criblage de décrochage à l’échelle du génome pour identifier des cibles thérapeutiques dans des modèles murins de tumeur maligne de la gaine nerveuse périphérique
Dans cet article
Résumé
Nous avons développé une approche d’oncogénomique comparative inter-espèces utilisant des analyses génomiques et des criblages génomiques fonctionnels pour identifier et comparer des cibles thérapeutiques dans les tumeurs apparaissant dans des modèles murins génétiquement modifiés et le type de tumeur humaine correspondant.
Résumé
Les tumeurs malignes de la gaine des nerfs périphériques (MPNST) sont dérivées de cellules de Schwann ou de leurs précurseurs. Chez les patients atteints du syndrome de susceptibilité tumorale neurofibromatose de type 1 (NF1), les MPNST sont la tumeur maligne la plus fréquente et la principale cause de décès. Ces sarcomes des tissus mous, rares et agressifs, offrent un avenir radieux, avec des taux de survie sans maladie à 5 ans de 34 à 60 %. Les options de traitement pour les personnes atteintes de MPNST sont décevantes et limitées, la chirurgie défigurante étant la principale option de traitement. De nombreuses thérapies autrefois prometteuses telles que le tipifarnib, un inhibiteur de la signalisation Ras, ont échoué cliniquement. De même, les essais cliniques de phase II avec l’erlotinib, qui cible le facteur de croissance épidermique (EFGR), et le sorafénib, qui cible le récepteur du facteur de croissance de l’endothélium vasculaire (VEGF), le récepteur du facteur de croissance dérivé des plaquettes (PDGF), et le Raf, en association avec la chimiothérapie standard, n’ont pas non plus réussi à produire de réponse chez les patients.
Au cours des dernières années, les méthodes de criblage génomique fonctionnel combinées au profilage génétique des lignées cellulaires cancéreuses se sont avérées utiles pour identifier les voies de signalisation cytoplasmique essentielles et le développement de thérapies spécifiques à la cible. Dans le cas des types de tumeurs rares, une variante de cette approche connue sous le nom d’oncogénomique comparative inter-espèces est de plus en plus utilisée pour identifier de nouvelles cibles thérapeutiques. Dans le cadre de l’oncogénomique comparative inter-espèces, le profilage génétique et la génomique fonctionnelle sont effectués dans des modèles de souris génétiquement modifiées (GEM) et les résultats sont ensuite validés dans les rares spécimens humains et lignées cellulaires disponibles.
Cet article décrit comment identifier les mutations des gènes moteurs candidats dans les cellules MPNST humaines et murines à l’aide du séquençage de l’exome entier (WES). Nous décrivons ensuite comment effectuer des criblages d’ARNsh à l’échelle du génome pour identifier et comparer les voies de signalisation critiques dans les cellules MPNST de souris et humaines et identifier des cibles médicamenteuses dans ces voies. Ces méthodologies fournissent une approche efficace pour identifier de nouvelles cibles thérapeutiques dans une variété de types de cancer humain.
Introduction
Les tumeurs malignes de la gaine des nerfs périphériques (MPNST) sont des néoplasmes fusiformes très agressifs qui surviennent en association avec le syndrome de susceptibilité tumorale neurofibromatose de type 1 (NF1), sporadiquement dans la population générale et aux sites de radiothérapie antérieure 1,2,3. Les patients atteints de NF1 naissent avec une copie de type sauvage du gène suppresseur de tumeur NF1 et un deuxième allèle NF1 avec une mutation de perte de fonction. Cet état d’haploinsuffisance rend les patients atteints de NF1 sensibles à une deuxième mutation de perte de fonction dans leur gène NF1 de type sauvage, qui déclenche la tumorigenèse. Lorsque cette mutation NF1 « second coup » se produit dans une cellule de la lignée cellulaire de Schwann, la tumeur qui en résulte est soit un neurofibrome dermique apparaissant dans la peau, soit un neurofibrome plexiforme qui se développe dans les gros nerfs ou les plexus nerveux. Bien que la pathologie des neurofibromes dermiques et plexiformes soit identique, leur comportement biologique est très différent - bien que les neurofibromes dermiques et plexiformes soient bénins, seuls les neurofibromes plexiformes peuvent subir une transformation et donner lieu à des MPNST. En plus de la perte de la neurofibromine, la protéine activatrice de la GTPase Ras codée par le gène NF1, les MPNST portent des mutations de plusieurs autres gènes suppresseurs de tumeurs, notamment TP53 4,5,6,7, CDKN2A8,9 et PTEN 10, des mutations de gènes codant pour des composants du complexe répressif polycomb 2 11,12 ( PRC2 ; le SUZ12 et les gènes EED) et l’expression aberrante des récepteurs tyrosine kinases 1,2. Des mutations de NF1 et des autres gènes mentionnés ci-dessus sont également présentes dans les MPNST sporadiques et radio-induites11,12.
Bien que ces progrès dans notre compréhension des anomalies génomiques dans les MPNST aient été inestimables pour comprendre leur pathogenèse, ils n’ont pas encore abouti à la mise au point de nouvelles thérapies efficaces pour les MPNST. L’un des principaux obstacles à la mise au point de nouveaux traitements est le fait que les MPNST sont des cancers rares. Pour cette raison, il est difficile d’obtenir le grand nombre d’échantillons de patients nécessaires pour les analyses globales définissant les mutations clés telles que celles entreprises par l’Atlas du génome du cancer (TCGA). D’après notre expérience, l’accumulation d’un nombre même modeste de spécimens humains de MPNST peut prendre des années. Pour surmonter ces limitations, de nombreux chercheurs qui étudient d’autres types de cancers rares se sont tournés vers l’utilisation de l’oncogénomique comparative inter-espèces pour identifier les mutations génétiques essentielles des gènes, définir les voies de signalisation cytoplasmique essentielles dans leur tumeur d’intérêt et identifier de nouvelles cibles thérapeutiques. Étant donné que les voies de signalisation essentielles à la tumorigenèse sont hautement conservées entre les humains et les autres espèces de vertébrés, l’application d’approches de génomique fonctionnelle telles que les criblages d’ARNsh à l’échelle du génome peut être un moyen efficace d’identifier ces nouvelles mutations pilotes, voies de signalisation et cibles thérapeutiques 13,14,15,16,17,18,19 , en particulier lors de l’étude de types de tumeurs humaines rares qui sont disponibles en nombre limité20.
Dans les méthodologies présentées ici, nous décrivons cette approche pour effectuer le profilage génomique dans les lignées cellulaires MPNST humaines et les cultures MPNST à passage précoce dérivées de souris P 0-GGFβ3, un modèle murin génétiquement modifié (GEM) dans lequel la surexpression spécifique des cellules de Schwann du facteur de croissance neuréguline-1 (NRG1) favorise la pathogenèse des neurofibromes plexiformes et leur progression ultérieure vers les MPNSTs21, 22 et 23. La première étape de cette approche consiste à identifier les gènes pilotes candidats dans les MPNST P 0-GGFβ3, les lignées cellulaires humaines MPNST et les MPNST humains réséqués chirurgicalement. Pour valider fonctionnellement les voies de signalisation affectées par ces mutations, nous utilisons ensuite des criblages d’ARNsh à l’échelle du génome pour identifier les gènes nécessaires à la prolifération et à la survie dans les lignées cellulaires MPNST humaines et murines. Après avoir identifié les gènes nécessaires à la prolifération et à la survie, nous identifions les produits géniques médicamenteux dans la collection de « résultats » à l’aide de la base de données sur les interactions géniques médicamenteuses. Nous comparons également les « hits » dans les cellules MPNST humaines et murines, afin de déterminer si le modèle GEM et les MPNST humains présentent une dépendance similaire aux mêmes gènes et voies de signalisation. L’identification des chevauchements dans les gènes nécessaires à la prolifération et à la survie et dans les voies de signalisation affectées permet de valider le modèle murin P 0-GGFβ3 au niveau moléculaire. Cette approche met également l’accent sur l’efficacité de la combinaison des cribles humains et murins pour identifier de nouvelles cibles thérapeutiques, où le modèle murin peut servir de complément aux cribles humains. La valeur de cette approche inter-espèces est particulièrement évidente lorsqu’il s’agit de rechercher des cibles thérapeutiques dans les tumeurs rares, où les tumeurs humaines et les lignées cellulaires sont difficiles à obtenir.
Protocole
Avant le début des études, faire examiner et approuver les procédures et les protocoles de manipulation des vecteurs viraux sur les animaux par le Comité institutionnel de protection et d’utilisation des animaux (IACUC) et le Comité de biosécurité institutionnel (IBC). Les procédures décrites ici ont été approuvées par les conseils d’administration de l’IACUC et de l’IBC de l’Université médicale de Caroline du Sud et ont été effectuées par un personnel correctement formé conformément au Guide des NIH pour les soins et l’utilisation des animaux de laboratoire et aux directives institutionnelles de MUSC en matière de soins aux animaux.
1. Analyses WES-Seq et identification des variants pathogènes
- Isolez l’ADN génomique d’un échantillon de tumeur ou de cellules MPNST sous-confluentes (70 %) cultivées sur une boîte de 60 mm, en utilisant des méthodes standard d’adsorption à base de gel de silice disponibles dans le commerce (consultez le protocole des fabricants pour les étapes détaillées). Le flux de travail général est illustré à la figure 1.
- Soumettre au moins 10 μL d’ADN génomique à 50 ng/μL au carottage de séquençage, à moins que des quantités ou des concentrations différentes d’ADN génomique ne soient spécifiées.
- La plateforme fragmente l’ADN génomique par sonication, puis le purifie à l’aide de la méthode préférée. La capture de l’exome et la construction de la bibliothèque sont effectuées à l’aide du kit de séquençage de l’exome préféré et des étiquettes d’index sont ajoutées à l’exome capturé amplifié.
- Soumettre des échantillons à la carotte de séquençage pour qu’un séquençage de l’exome entier par paires (WES ; 100 pb séquencés à partir de chaque extrémité) soit effectué.
- Les fichiers FASTQ générés par le noyau sont fournis à l’enquêteur. Utilisez uniquement des fichiers FASTQ qui transmettent des mesures de qualité pour l’analyse.
- Alignez et analysez les fichiers FASTQ à l’aide de logiciels disponibles dans le commerce (par exemple, DNAStar19,20, Partek21 ou Varsome). Alignez les fichiers FASTQ sur le génome de référence de la souris GRCm38/mm10 en utilisant les paramètres par défaut.
REMARQUE : À l’aide de DNAStar et d’un spécimen MPNST de souris à titre d’exemple, le flux de travail général est schématisé dans Graphique 1 et brièvement expliqué ci-dessous.- Ouvrez le logiciel DNAStar et choisissez le flux de travail SeqMan NGen.
- Choisissez Workflow en sélectionnant Variant Analysis/Resequencing (Analyse/Reséquençage) des variants et le type d’analyse de séquençage (NGS-Based Amplicon), panel de gènes ou exome (Amplicon basé sur NGS), panel de gènes ou exome. Cliquez sur Suivant.
- Choisissez la séquence de référence préférée en sélectionnant Télécharger le paquet de génome et le génome de référence approprié ( c’est-à-dire Mus_musculus-GRCm38-dbSNP146.zip ou Homo sapien-GRCh37.p13.zip). Si l’établissement principal a fourni un fichier de lit, téléchargez ce fichier auxiliaire. Cliquez sur Suivant.
- Choisissez Séquences d’entrée en sélectionnant la technologie de séquenceur appropriée, Illumina, et indiquez que les lectures de séquençage sont appariées. Ensuite, sélectionnez la configuration de l’expérience, le multi-échantillon, puis téléchargez les fichiers FASTQ de séquençage en sélectionnant Ajouter. Si vous exécutez plusieurs échantillons, désignez chaque ensemble d’extrémités appariées avec une expérience ou un nom d’échantillon unique Tumeur, Lignée cellulaire ou A18, A202... Cliquez sur Suivant.
- Définissez le jeu de données de contrôle, le cas échéant. Cliquez sur Suivant, puis sur Suivant sous Options d’assemblage. Sous Options d’analyse, cliquez sur le mode de détection de variante Diploïde approprié, puis cliquez sur Suivant. Sous Sortie de l’assemblage, nommez et désignez l’emplacement d’enregistrement du fichier du projet ; cliquez sur Suivant. Exécutez l’assembly sur l’ordinateur local ou dans le cloud.
REMARQUE : Les fichiers d’alignement résultants peuvent être ouverts dans le flux de travail ArrayStar pour l’annotation des variantes des polymorphismes mononucléotidiques (SNP) détectés. Ce flux de travail détecte les SNP, et non les gains ou les pertes de nombre de copies de gènes. Une analyse distincte appelée réseau SNP haute densité détecte les changements de nombre de copies ; Le séquençage de l’exome entier ne détecte pas de manière fiable les changements du nombre de copies. Consultez le guide de l’utilisateur du programme pour connaître les étapes spécifiques à effectuer avec le programme d’alignement. - Appliquez des filtres définis par l’utilisateur à l’annotation de variante des SNP détectés afin d’inclure ou d’exclure certaines données. Pour obtenir une liste condensée des variants susceptibles d’être pertinents sur le plan fonctionnel, appliquez les filtres suivants aux ensembles de données de variants de cette hiérarchie : non sous contrôle (si un échantillon témoin normal est disponible), fréquence de la population (gnomAD, ExAC, fréquences de 1 000 génomes), fréquence des allèles (inclure ≤0,001 ou 0,1 %), profondeur de couverture (exclure pathogénicité ou classe ClinVar (inclure : pathogène, probablement pathogène ; exclure : incertain, probablement bénin, bénin), et si désiré le type de SNP et l’impact du codage (inclure : non synonyme, faux-sens, non-sens, décalage de trame, in-frame, épissage).
REMARQUE : Une liste de gènes cancéreux pertinents connus peut également être appliquée à la liste finale pour n’extraire que des gènes spécifiques pertinents pour la maladie, voir l’étape 1.3.7. Ces filtres peuvent réduire la liste des gènes variants à moins de 20 gènes.- Utilisez la fréquence des allèles variants pour filtrer les SNP d’erreur de séquençage. Les variants homozygotes et hétérozygotes seront représentés approximativement à 100 % et 50 %, respectivement, pour une population de cellules pures non contaminantes (une lignée cellulaire dérivée d’une tumeur établie doit représenter une seule population de cellules tumorales isogéniques). Dans ce cas, appliquez des fréquences alléliques de 100 à 90 % et de 50 à 40 % pour les variants comme filtre, supprimant ainsi toutes les variantes inférieures à ce seuil. Si des données de puces SNP à haute densité sont également disponibles pour le même ensemble de données, appliquez le gain ou la perte de nombre de copies aux SNP dont la fréquence des allèles variants est inférieure aux rapports homozygotes ou hétérozygotes (c’est-à-dire que le gain du nombre de copies résultant en 2 copies de l’allèle A et 1 copie de l’allèle B donnerait des fréquences d’allèles variantes appropriées de 75 %, 25 %, respectivement).
- Après avoir identifié les variants pathogènes avec ArrayStar, exportez et enregistrez la liste des gènes variants sous forme de fichier csv, txt ou xls et comparez les gènes contenant ces mutations à ceux de la cohorte de tumeurs générées par P 0-GGFβ3 pour déterminer les listes de gènes mutés uniques et qui se chevauchent. Comparez les gènes mutés aux gènes mutés connus associés à leurs homologues humains (c.-à-d. la liste des gènes du cancer du laboratoire Bushman ou une liste organisée par l’utilisateur).
- Si vous le souhaitez, avant d’appliquer des filtres définis par l’utilisateur (1.3.6), exportez et enregistrez le fichier annoté en tant que fichier VCF.
REMARQUE : Ce fichier VCF peut être téléchargé vers un autre logiciel de prédiction d’effecteur de variant, Varsome ou VEP, à des fins de comparaison en tant qu’analyse secondaire. L’analyse biologique et l’analyse des voies peuvent également être effectuées sur les listes de gènes variants filtrés.
- Si vous le souhaitez, avant d’appliquer des filtres définis par l’utilisateur (1.3.6), exportez et enregistrez le fichier annoté en tant que fichier VCF.
- Effectuez la classification fonctionnelle de la liste des gènes variants via une base de données préférée de l’utilisateur pour obtenir des informations sur la classe de protéines, la voie, le composant de la voie, la famille de gènes et l’ontologie.
NOTE : PANTHER (pantherdp.org) est utilisé comme exemple ici.- Téléchargez la liste des gènes filtrés avec l’ID du gène.
- Sélectionnez l’organisme (Mus musculus).
- Sélectionnez l’analyse (Classification fonctionnelle affichée dans la liste des gènes) et cliquez sur Soumettre l’analyse biologique et l’analyse des voies sur les listes de gènes variants filtrés.
2. Criblages d’ARNsh à l’échelle du génome
REMARQUE : Plusieurs banques shRNA et CRISPR sont disponibles et peuvent être utilisées pour les criblages fonctionnels à l’échelle du génome avec des cultures tumorales à faible passage. Nous décrivons ici l’utilisation des banques d’ARNh CELLECTA DECIPHER à titre d’exemple. Les banques d’ARNh lentiviraux de CELLECTA DECIPHER sont optimisées pour les criblages génétiques d’ARNi en format groupé. Chaque transcrit est ciblé par au moins 5 à 6 shRNA uniques et chaque vecteur shRNA lentiviral contient un code-barres génétique unique flanqué de sites d’amorce PCR. Ces banques couvrent la majorité des gènes liés aux maladies humaines et de souris, mais ne couvrent pas tous les gènes du génome. Les pools d’ADN plasmidique de la bibliothèque humaine de Cellecta sont disponibles en trois modules (Human Module I, II, III ; cible 15 377 gènes) tandis que les pools de plasmides de la bibliothèque de souris sont disponibles en deux modules (Mouse Modules I et II ; cible 9 145 gènes). Ces banques sont utilisées pour effectuer des tests de « décrochage » dans lesquels les gènes ciblés nécessaires à la prolifération et/ou à la survie sont exprimés de manière différentielle à différents moments après la transduction virale.
- Emballage lentiviral
- Jour 0 : Assiette de 10 millions de cellules 293T/boîte de 15 cm dans 30 mL/plat de DMEM sans antibiotiques contenant 10 % de sérum de veau fœtal (FBS).
- Jour 1 : Confirmez que les cellules sont confluentes à ~80 % le lendemain et prêtes à être transfectées. Dans un tube conique de 50 mL, mélanger dans cet ordre : 600 μL de mélange plasmidique d’emballage (0,5 μg/μL), 60 μL de bibliothèque de codes-barres plasmidiques, 12 mL de DMEM (sans sérum ni antibiotiques) et 600 μL de réactif de transfection. Incuber à température ambiante pendant 15 min.
- Placer 900 μL de réactif de transfection et 12 mL de DMEM dans un tube conique séparé de 15 mL et mélanger par vortex. Ajouter 12,9 mL du mélange réactif de transfection/DMEM au mélange d’ADN et feuilleter pour mélanger. Incuber à température ambiante pendant 15 minutes sans mélanger davantage. Ajouter 2,5 mL de ce mélange, goutte à goutte, à chaque boîte de 15 cm de cellules 293T et incuber toute la nuit dans un incubateur de culture tissulaire.
- Jour 2 : Remplacez le milieu le lendemain par un milieu de culture ordinaire contenant des antibiotiques.
- Jour 3 : Récolter le virus en recueillant le milieu et en le faisant passer à travers une unité de filtration de 0,2 μm et en l’aliquoteant dans des tubes coniques de 15 mL ; préparer également cinq aliquotes de 1 mL de virus filtré dans des flacons cryogéniques pour le titrage viral (considéré comme un virus de 48 h) ; conserver le virus dans un congélateur à -80 °C. Remplacez les milieux par 30 mL de milieu de culture sur les plaques une à la fois pour éviter le dessèchement des cellules.
- Jour 4 : Récoltez le virus en collectant le milieu et en le faisant passer dans une unité de filtration de 0,2 μm. Aliquote dans des tubes coniques de 15 mL. C’est ce qu’on appelle le virus 72 h ; conserver le virus dans un congélateur à -80 °C.
- Piscines lentivirales à titre
- Ajouter 65 μL de polymère cationique (10 mg/mL) à 65 mL de milieu de croissance des cellules tumorales. Pipeter 1 mL/puits du milieu contenant du polymère dans onze plaques de culture tissulaire à 6 puits. Trypsiniser les cellules tumorales à passage précoce et compter les cellules en utilisant la méthode préférée afin que chaque puits reçoive 50 000 cellules/mL/puits. Décongeler 1 mL d’aliquotes de lentivirus 48 h du congélateur au bain-marie à 37 °C.
- Pour chaque module viral, préparez l’infection comme indiqué dans le tableau 1.
- Placez toutes les plaques à 6 puits dans un incubateur de culture tissulaire. Le lendemain, retirez les milieux viraux et reconstituez-les avec de nouveaux milieux de culture. À 48 h, ajouter un milieu contenant de la puromycine à tous les puits recevant la sélection. Avant que les cellules témoins ne soient confluentes, effectuez le comptage cellulaire des clones survivants en utilisant la méthode préférée.
REMARQUE : La concentration de puromycine nécessaire pour tuer les cellules non transduites doit être prédéterminée empiriquement en testant une gamme de concentrations dans une courbe de « destruction ». Utiliser la concentration la plus faible qui induit uniformément la mort des cultures non transduites.
- Infection lentivirale des cellules cibles
- Trypsiniser les cellules MPNST et les compter. Assiette : 2,5 millions de cellules par plat de 15 cm pour un total de vingt plats de 15 cm par module. Décongeler le virus et transduire les cellules avec le virus à un moment d’inertie de 0,5 en présence de 5 μg/mL de polymère cationique (figure 2A).
- Retirez les milieux contenant des virus le lendemain matin et remplacez-les par des milieux de culture neufs. Cultivez les cellules pendant 2 jours supplémentaires pour permettre l’expression du marqueur de sélection, puis ajoutez de la puromycine aux cultures pour éliminer les cellules non transduites.
- Trois jours après l’ajout de puromycine, trypsiniser les cellules et centrifuger la moitié de la population cellulaire à 200 × g pendant 5 min dans une centrifugeuse de table. Conservez les cellules granulées dans le congélateur à -80 °C pour la préparation future de l’ADN génomique ; il s’agit du point temporel de référence, ou point temporel 1 (T1).
- Replaquez l’autre moitié de la population cellulaire et cultivez-la pour environ 7 doublements de population avant de récolter et de centrifuger comme ci-dessus. Cette pastille cellulaire servira de point de temps final (point de temps 2, T2) et sera stockée à -80 °C pour l’isolement futur de l’ADN génomique (Figure 2).
- Isolement de l’ADN génomique à partir de cellules transduites avec des pools de virus
- Décongeler la pastille cellulaire du congélateur à -80 °C et la remettre en suspension dans 10 mL de tampon de remise en suspension additionné d’ARNase, puis la diviser immédiatement en deux tubes de polyméthylpentène de 15 mL (figure 2B).
- Ajouter 500 μL de SDS à 10 % par 5 mL, mélanger et incuber à température ambiante pendant 5 minutes. Ensuite, placez des tubes dans un dispositif de cisaillement de l’ADN pour soniser l’ADN pendant 25 cycles de 30 s marche/30 s d’arrêt, en veillant à maintenir la température à 4 °C.
- Ajouter 5 mL de phénol/chloroforme, pH 8,0 (conservé à 4 °C), en veillant à bien mélanger le phénol/chloroforme avant utilisation. Après l’ajout de phénol/chloroforme, bien mélanger en tourbillonnant vigoureusement au réglage maximum pendant 45 à 60 s. Centrifuger pendant 60 min, -20 °C à 7 200 × g.
REMARQUE : Un aspect mousseux/laiteux après le vortex signalera une remise en suspension complète. - Transférer 3 mL de la phase supérieure transparente dans un tube neuf de 15 mL et ajouter 0,5 mL d’acétate de sodium 3 M et 4 mL d’isopropanol et bien mélanger (figure 2C). Centrifuger pendant 30 min à 20 °C à 7 200 × g.
- Après la centrifugation, jeter le surnageant, puis pipeter soigneusement le liquide restant. Ajouter 0,5 mL d’éthanol à 70 % et déloger la pastille en pipetant de haut en bas. Transférez la pastille remise en suspension dans un tube à centrifuger de 1,5 mL et combinez les deux pastilles du même échantillon dans un seul tube à centrifuger de 1,5 mL. Centrifuger à vitesse maximale dans une centrifugeuse de paillasse pendant 5 min.
- Jetez le surnageant et utilisez une lingette de laboratoire pour absorber tout résidu d’éthanol à 70 %. Remettre la pastille en suspension dans 0,5 mL d’eau distillée, en veillant à ne pas laisser la pastille sécher car cela rendrait la remise en suspension difficile. Conservez les échantillons à 4 °C avant de mesurer la concentration d’ADN.
- PCR imbriquée pour amplifier les codes-barres shRNA
REMARQUE : Récupérez l’ADN de l’entreposage et placez-le sur de la glace. Si la concentration d’ADN est inférieure à 100 ng/μL, séchez rapidement l’ADN sous vide et mettez-le en suspension dans un volume approprié d’eau distillée. Nous vous recommandons de ne traiter qu’un seul module à la fois (point de temps 1 (T1) ou point de temps 2 (T2)). Effectuez deux préparations de chaque point temporel qui doivent être regroupées à la fin ; Par conséquent, il est important de ne pas traiter les points temporels répliqués le même jour. Le protocole suivant concerne un point de temps de l’ADN génomique.- Installez 7 tubes et étiquetez-les comme décrit : témoin négatif, témoin positif, 4 tubes pour l’ADN génomique et un pour un mélange maître (Figure 3A). Le témoin négatif est l’eau distillée ; le contrôle positif est l’ADN plasmidique utilisé dans la transfection 293T pour générer le lentivirus.
- Ajoutez d’abord de l’eau dans chaque tube comme indiqué dans le tableau 2.
- Préparez un Master Mix (MM) comme indiqué dans le tableau 3.
- Ajouter 18 μL de MM dans chaque tube contenant de l’eau. Ensuite, ajoutez 25 μL d’ADN génomique à chacun des 4 tubes modèles. Ensuite, ajoutez 1 μL de témoin positif (10 ng/μL) dans le tube témoin positif, en prenant soin de ne pas contaminer d’autres tubes avec de l’ADN témoin positif. Enfin, ajoutez 2 μL de polymérase dans chaque tube, en ajoutant d’abord aux 4 tubes d’échantillon et en dernier au tube témoin positif ; mélanger et faire tourner les tubes PCR (Figure 3).
- Effectuer une réaction PCR dans les conditions indiquées dans le tableau 4.
- Pendant que la première PCR est en cours, préparez les tubes pour la 2e PCR comme indiqué dans le tableau 5. Mettez en place sept tubes : témoin négatif, témoin positif, 4 tubes pour l’ADN génomique et un pour le MM (Figure 3B).
- Ajouter 22 μL de MM dans chaque tube contenant de l’eau et attendre la fin de la première série de PCR avant d’ajouter l’ADN et la polymérase.
- Une fois le premier cycle de PCR terminé, combinez 4 tubes d’échantillons dans un tube de microcentrifugation et mélangez.
- Ajouter 25 μL dans chaque tube d’échantillon contenant de l’eau.
- Ensuite, ajoutez 2 μL du premier témoin négatif à la PCR et 2 μL du premier témoin positif à la PCR dans les tubes correctement étiquetés.
- Ajouter 2 μL de polymérase dans chaque tube, en ajoutant d’abord aux 4 tubes d’échantillon et en dernier le témoin négatif.
- Effectuer une réaction PCR comme indiqué dans le tableau 6.
REMARQUE : Après avoir analysé les résultats de la première réaction PCR par électrophorèse sur un gel d’agarose à 3,5 %, si une abondance de produit est obtenue, le nombre de cycles peut être réduit à 9-10 pour la prochaine répétition de cette procédure ; De même, si le rendement du produit est faible, les cycles peuvent être augmentés à 14. - Lorsque la deuxième réaction PCR est terminée, combinez 4 échantillons dans un tube de microcentrifugation plus 80 μL de colorant de charge 6x. Pour les témoins positifs et négatifs, utiliser 20 μL de l’échantillon.
- Préparer un gel d’agarose à 3,5 % dans un tampon Tris-Borate-EDTA (TBE) (Figure 4A). Pour s’adapter au grand volume d’échantillon, créez un grand puits dans le peigne à gel en collant plusieurs puits ensemble (si un peigne adapté au volume n’est pas disponible), en vous assurant de laisser deux puits disponibles pour les témoins positifs et négatifs. Faites fonctionner le gel à 90 V pendant 1 h.
- Visualisez le gel et confirmez une bande à environ 250 paires de bases (pb).
- Première purification : Extraction sur gel
- À l’aide d’un scalpel ou d’une lame de rasoir propre, exciser la bande de 250 pb, en coupant autant de gel que possible. Divisez la grande bande en 4 morceaux et transférez chaque morceau dans un tube de microcentrifugation propre. Mesurez et notez le poids de chaque tranche de gel.
REMARQUE : Chaque morceau doit être d’environ 200 mg ou moins par tube. - Ajouter 6 volumes de tampon de solubilisation (p. ex., si le morceau de gel pèse 200 mg, ajouter 1,2 mL de tampon). Placez les tubes dans un bain-marie à 50 °C et tournez-les toutes les 10-15 minutes jusqu’à ce que les tranches de gel soient dissoutes. Ensuite, ajoutez 1 volume d’isopropanol dans chaque tube (par exemple, 0,2 mL d’isopropanol si le morceau de gel pèse 200 mg).
- À l’aide de deux colonnes de centrifugation pour la purification, chargez les échantillons des 4 tubes dans les colonnes. Effectuez plusieurs essorages pour traiter tout le volume de l’échantillon, car les colonnes ne contiennent que 750 μL. Faites tourner les colonnes à 17 900 × g pendant 1 min dans une microcentrifugeuse de paillasse conventionnelle et jetez l’écoulement à chaque fois.
- Laver les colonnes avec 750 μL de tampon de lavage et centrifuger à 17 900 × g pendant 1 min. Après le lavage, jeter l’écoulement et sécher la colonne d’essorage par centrifugation à 17 900 × g pendant 3 min. Éluer l’ADN avec 50 μL d’eau distillée par colonne et centrifuger comme précédemment pendant 1 min et combiner les deux tubes pour un volume total de 100 μL de l’échantillon.
- À l’aide d’un scalpel ou d’une lame de rasoir propre, exciser la bande de 250 pb, en coupant autant de gel que possible. Divisez la grande bande en 4 morceaux et transférez chaque morceau dans un tube de microcentrifugation propre. Mesurez et notez le poids de chaque tranche de gel.
- Deuxième purification
- Cette prochaine étape de purification utilisera le tampon de liaison 2 (du kit de purification PCR), qui n’élimine pas les petits fragments. Ajoutez 400 μL de tampon aux 100 μL d’ADN et chargez-le sur une colonne de spin (Figure 4B). Centrifuger l’échantillon à 17 900 × g pendant 1 min.
- Ensuite, lavez la membrane avec 650 μL de tampon de lavage et essorez-la comme précédemment. Séchez la colonne d’essorage avec une centrifugation supplémentaire comme ci-dessus pendant 3 min.
- Éluez l’ADN avec 30 μL d’eau distillée et essorez à vitesse maximale pendant 1 min. Après l’élution, vérifier la concentration à l’aide d’un spectrophotomètre ; s’assurer que la concentration n’est pas inférieure à 10 ng/μL ni supérieure à 70-80 ng/μL. Conservez l’ADN dans un congélateur à -20 °C.
- Séquençage de codes-barres amplifiés
- Pour le séquençage, diluer les codes-barres purifiés à 0,75 ng/μL à l’aide d’un tampon d’élution (EB). Pour ajouter de la diversité de séquences, les amplicons sont regroupés à 17 pM, dont 30 % (v/v) de PhiX. Effectuez un clustering à extrémité unique (SE) sur un système de génération de cluster automatisé selon le protocole du fabricant.
- Demandez au cœur de séquençage d’effectuer un total de 36 cycles de séquençage à une extrémité sur un séquenceur NextGen (Figure 4C). Ajoutez l’amorce personnalisée GexSeqS aux amorces de séquençage Illumina à 0,5 μM.
- Utilisez le logiciel d’analyse référencé pour générer des fichiers Fastq et traitez-les à l’aide d’un logiciel pour réduire la longueur de lecture à 18 nucléotides.
- Utilisez le logiciel Barcode Analyzer et Deconvoluter pour déconvoluer les lectures tronquées. Calculez les scores d’épuisement des plis pour chaque shRNA comme le rapport entre le nombre de lectures au point de temps de référence (T1) et le point de temps final (T2).
- Analyse des résultats de criblage de shRNA et identification de cibles thérapeutiques candidates
REMARQUE : Dans la bibliothèque d’ARNh de Cellica, la plupart des gènes sont ciblés par 5 (67 %) ou 6 shRNA différents (32 %). Cependant, plusieurs gènes d’entretien, qui devraient être atteints dans la plupart des cellules, sont ciblés par un grand nombre de shRNA et servent de contrôles qui définissent la gamme de scores pour les négatifs.- Pour éviter les biais en faveur des gènes ciblés par un plus grand nombre d’ARNsh, transformez logarithmique les scores d’appauvrissement. Effectuez une estimation quantile en calculant le 80e percentile de chaque gène à partir de sa distribution empirique.
- Pour générer une distribution nulle des scores d’appauvrissement logarithmique, supposons que >95 % des gènes ne seront pas appauvris et que leurs scores log-quantile auront une distribution normale. Utilisez la médiane de la distribution empirique pour estimer la moyenne de la distribution nulle. À l’aide de cette distribution nulle, classez tous les gènes dont les scores d’appauvrissement logarithmique sont supérieurs au 95e percentile de la distribution nulle comme des « résultats ».
REMARQUE : Tous les résultats doivent avoir au moins deux shRNA avec des scores d’appauvrissement supérieurs au seuil (Figure 5). - Pour évaluer la qualité des données de dépistage de l’ARNi et la validité du seuil de coupure, utilisez un ensemble de gènes qui ont été définis comme « essentiels » par l’initiative de dépistage de l’ARNi du cancer COLT24,25 à des fins de comparaison. Retirer les CEG de la liste des résultats pour produire la liste initiale des cibles thérapeutiques potentielles.
REMARQUE : Les gènes de l’ensemble COLT ont été marqués comme un succès dans >50 % des 72 lignées cellulaires cancéreuses qui ont été criblées par COLT. La liste des gènes essentiels de base contient 640 gènes. - Pour identifier les cibles thérapeutiques potentielles qui sont couramment ou uniformément requises par plusieurs lignées cellulaires MPNST, construire des diagrammes de Venn des hits non-CEG identifiés dans les criblages de différentes lignées cellulaires MPNST ou les cultures à passage précoce (Figure 6A). Donner la priorité aux hits non-CEG qui sont requis dans toutes ou la majorité des lignées ou cultures MPNST.
- Vous pouvez également effectuer des analyses de voies sur la liste de résultats non CEG de chaque lignée cellulaire ou culture à passage précoce pour identifier les gènes dont les produits codent pour des composants des voies de signalisation nécessaires à la prolifération et/ou à la survie des cellules tumorales. Ensuite, comparez les voies de signalisation identifiées dans l’analyse des voies des non-CEG aux voies de signalisation qui ont été identifiées comme affectées par des mutations identifiées avec WES.
NOTE : Nous trouvons qu’il est particulièrement utile d’identifier les voies de signalisation qui sont systématiquement affectées par les mutations identifiées dans WES et de les comparer aux voies de signalisation identifiées comme critiques dans les cribles d’ARNsh.
- Vous pouvez également effectuer des analyses de voies sur la liste de résultats non CEG de chaque lignée cellulaire ou culture à passage précoce pour identifier les gènes dont les produits codent pour des composants des voies de signalisation nécessaires à la prolifération et/ou à la survie des cellules tumorales. Ensuite, comparez les voies de signalisation identifiées dans l’analyse des voies des non-CEG aux voies de signalisation qui ont été identifiées comme affectées par des mutations identifiées avec WES.
- Pour identifier les cibles médicamenteuses pour lesquelles des agents thérapeutiques sont déjà disponibles, passer au crible la liste des résultats restants après l’ablation des gènes essentiels de base à l’aide de la base de données sur les interactions géniques médicamenteuses (dgidb.org).
REMARQUE : Cette base de données permet d’entrer et de dépister simultanément une liste de gènes et fournit des conseils sur les médicaments actuellement disponibles pour cibler ces gènes. - Validez d’abord les cibles à haut intérêt médicamenteux identifiées en inhibant leur expression génique avec des shRNA distincts de ceux utilisés dans l’écran initial de la bibliothèque dans un format de lot unique (et non un format de lot de bibliothèque que nous venons de décrire). Pour inhiber l’expression des gènes, utilisez deux à trois shRNA lentiviraux distincts. Trois à quatre jours après l’infection, déterminez l’effet que cela a sur la prolifération et la survie des cellules tumorales comme décrit ci-dessous.
3. Effectuer des tests cytométriques du nombre de cellules et de la viabilité dans les cellules MPNST mises à l’épreuve avec des agents thérapeutiques candidats
- Cultivez les cellules MPNST dans le DMEM jusqu’à 80 % de confluence. Rincez les cellules avec la solution saline équilibrée de Hanks (HBSS) à température ambiante.
REMARQUE : Ne cultivez pas de cellules jusqu’à la confluence car la croissance de ces cellules sera retardée pendant un certain temps après le replacage. - Détacher les cellules du substrat en les recouvrant pendant 30 s à 1 min avec une solution de dissociation cellulaire non enzymatique. Ajouter 5 mL de DMEM pour 1 mL de solution de dissociation et pipeter doucement les cellules de haut en bas pour qu’elles se détachent du substrat.
- Comptez les cellules à l’aide d’un hémocytomètre. Cellules à plaques d’une densité de 1 200 cellules par puits dans des plaques à paroi noire de 96 puits ; Plaquer au moins trois puits pour chaque dilution de médicament qui sera testée et effectuer trois répétitions biologiques de ces expériences.
- Pour déterminer les concentrations initiales du médicament qui sera testé, examinez la littérature afin d’évaluer quelles concentrations ont été efficaces contre d’autres types de cellules cancéreuses. Dans les expériences initiales, testez une plage de deux ordres de grandeur au-dessus et de deux ordres de grandeur en dessous de la concentration de médicament utilisée avec d’autres types de cancer.
- Préparez les dilutions du médicament à tester et ajoutez chaque dilution ou véhicule à au moins trois puits de répétition.
- Évaluez le nombre direct de cellules 1, 3, 5 et 7 jours après l’ajout de médicaments. Ajouter Hoechst 33342 jusqu’à une concentration finale de 5 μg/mL et incuber les plaques pendant 30 min à 37 °C. Lire les plaques sur un cytomètre d’imagerie à haut débit à l’aide de l’option de numération cellulaire directe pour le nombre total de cellules avec des temps d’exposition de 100 000 ms.
- Analysez les lectures à l’aide du logiciel et exportez-les dans une feuille de calcul et utilisez un logiciel approprié pour les analyses statistiques.
- Si des réductions statistiquement significatives du nombre de cellules sont observées dans les puits traités par des médicaments, effectuer un test « vivant/mort » pour déterminer si cette réduction est due, en partie, à l’induction de la mort cellulaire.
- Cellules de plaque dans des plaques à 96 puits à paroi noire, comme décrit à l’étape 3.3.
- Préparez et ajoutez les médicaments comme décrit à l’étape 3.5.
- Évaluez la viabilité cellulaire et la mort 1, 3, 5 et 7 jours après l’ajout du médicament. Ajouter la calcéine AM à une concentration finale de 1 μM et l’iodure de propidium à une concentration finale de 1 μM dans chaque puits. Incuber les cellules pendant 15 min à 37 °C.
- Imagez des cellules sur un cytomètre d’imagerie à l’aide de l’option logicielle Live+Dead . Analysez les lectures à l’aide du logiciel et exportez-les dans une feuille de calcul et utilisez un logiciel approprié pour les analyses statistiques.
Résultats
Les graphiques de la figure 5 montrent les scores d’appauvrissement des gènes essentiels de base (CEG) étiquetés comme VRAI par rapport aux non-CEG (étiquetés comme FAUX) dans chaque lignée cellulaire humaine qui a été criblée. Les points représentent log2 des scores d’épuisement des plis pour les gènes individuels, qui sont tracés sur une représentation en boîte à moustaches de la distribution globale des scores. Le test t de Student a été utilisé pour tester une différence significative dans la moyenne des scores d’épuisement entre les deux groupes dans chaque lignée cellulaire. Les valeurs p résultantes sont indiquées dans chaque panneau. Il est à noter que les scores moyens d’épuisement des plis sont significativement plus élevés pour les CEG que pour les non-CEG. On s’y attend, car les gènes essentiels centraux sont, par définition, systématiquement nécessaires à la prolifération et/ou à la survie dans la plupart des types de cellules.
La figure 6A présente un diagramme de Venn des « correspondances » pour trois lignées cellulaires MPNST humaines. Nous constatons généralement qu’un grand nombre de gènes sont partagés entre plusieurs lignées ; ces résultats sont hautement prioritaires car ils représentent des gènes codant pour des protéines susceptibles d’être essentielles à la prolifération et/ou à la survie d’un grand sous-ensemble de MPNST. Notez également qu’il existe un certain nombre de gènes qui sont des résultats dans une seule lignée cellulaire. Nous rencontrons cela fréquemment et cela ne doit pas être considéré comme une indication que les écrans sont de mauvaise qualité. Les gènes qui sont communs entre plusieurs lignées sont ensuite évalués à l’aide de la base de données sur les interactions géniques médicamenteuses afin d’identifier les gènes de ce sous-ensemble qui codent pour des protéines pouvant être médicamentées avec des agents existants. Nous sélectionnons ensuite plusieurs d’entre eux et effectuons une première validation en inhibant l’expression de l’ARNm correspondant avec des shRNAs. Étant donné que certains shRNAs auront des effets hors cible, nous testons toujours plusieurs shRNAs ciblant le même transcrit. La figure 6B montre un résultat représentatif dans lequel nous avons transduit des cellules MPNST avec un témoin non ciblant et plusieurs shRNAs ciblant BCL6. Le nombre de cellules a ensuite été déterminé à différents moments après la transduction. Il est à noter que plusieurs des shRNAs de BCL6 ont considérablement réduit le nombre de cellules ; comme le montre l’immunoblot qui l’accompagne, le degré de diminution du nombre de cellules est corrélé au degré d’inactivation de BCL6. La figure 6C montre une courbe de croissance représentative d’une culture MPNST P 0-GGFβ3 à passage précoce.

Figure 1 : Flux de travail pour effectuer le séquençage de l’exome entier du tissu MPNST ou des cellules MPNST à passage précoce. Le schéma illustre le flux de travail général de détection des variants présents dans les cultures à passage précoce dérivées de tumeurs. Isolez l’ADN des cultures à passage précoce (1) et soumettez l’ADN de qualité au noyau de séquençage selon leurs protocoles de soumission (2). Le noyau de séquençage vérifiera la qualité de l’ADN soumis et effectuera toutes les préparations nécessaires pour les échantillons et la bibliothèque de génomes. L’installation centrale fournira aux utilisateurs des fichiers de séquençage FASTQ avec des mesures de contrôle de la qualité (3). Les utilisateurs téléchargeront les fichiers FASTQ dans un programme d’alignement du génome et d’appel de variantes de leur choix. (4) Les variantes annotées sont filtrées selon des critères définis par l’utilisateur afin de supprimer les variantes non pertinentes. Les données représentatives présentées comparent un échantillon de tumeur MPNST humaine réséquée à une lignée cellulaire dérivée de la tumeur26. (5) Effectuer une analyse de classification fonctionnelle avec PANTHER. Abréviation : MPNST = Tumeur maligne de la gaine des nerfs périphériques. Veuillez cliquer ici pour voir une version agrandie de cette figure.
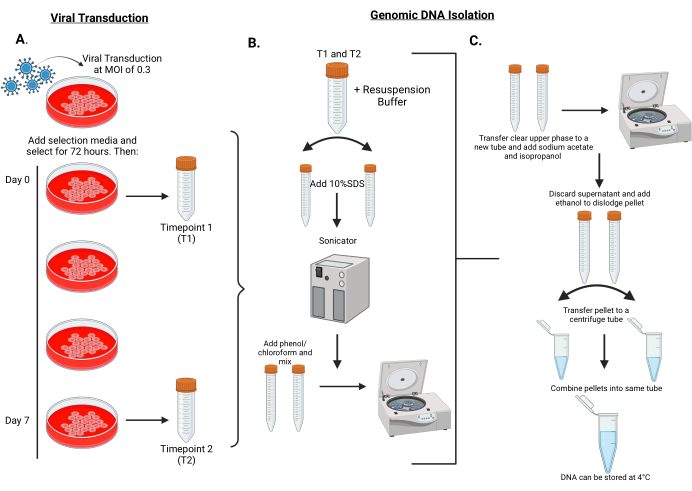
Figure 2 : Flux de travail pour effectuer la transduction virale des banques d’ARNsh dans les cellules MPNST et isoler l’ADN génomique des cellules aux points temporels 1 et 2. (A) Les cellules cibles sont infectées à un faible moment d’inertie de 0,3 avec des particules lentivirales à code-barres et sélectionnées pendant 72 h. Les cellules sont traversées pendant 5 à 7 doublements de population (environ 7 jours). Les pastilles cellulaires du jour 0 et du jour 7 sont stockées à -80 °C pour l’isolement de l’ADN génomique. Le jour 0 est appelé point de temps 1 (T1) et le jour 7 est appelé point de temps 2 (T2). (B) L’isolement de l’ADN génomique commence par la remise en suspension de pastilles cellulaires dans un tampon de remise en suspension qui sont ensuite divisées en deux tubes de 15 mL. Pour faciliter la lyse cellulaire, 10 % de SDS sont ajoutés à chaque tube et sonisés pendant 25 cycles de 30 s marche/30 s d’arrêt. Après la sonication, du phénol/chloroforme est ajouté à chaque tube et tourbillonné vigoureusement pendant 45 à 60 s. Les tubes sont ensuite centrifugés. (C) Une phase supérieure transparente est pipetée et ajoutée à un tube propre avec l’ajout d’acétate de sodium / isopropanol et bien mélangée. Les tubes sont à nouveau centrifugés. Cette fois, le surnageant est éliminé et la pastille est délogée avec l’ajout de 70% d’éthanol. Combinez les pastilles remises en suspension dans un tube et essorez-les à vitesse maximale dans une centrifugeuse de paillasse. Jetez le surnageant et remettez la pastille en suspension dans de l’eau distillée. Veuillez cliquer ici pour voir une version agrandie de cette figure.
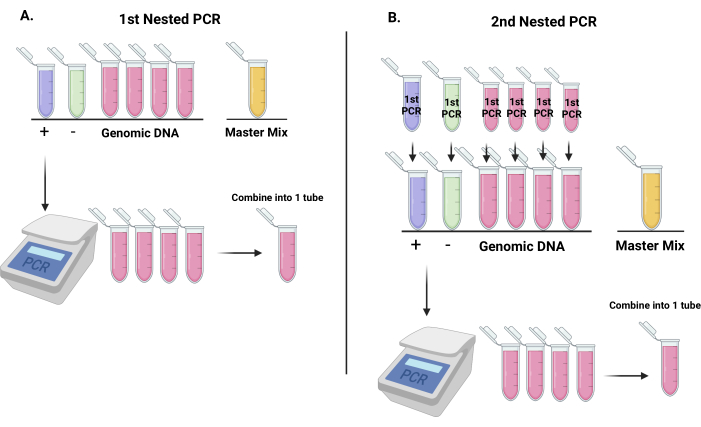
Figure 3 : Flux de travail pour l’amplification de séquences de codes-barres à partir de vecteurs d’ARNsh lentiviraux en vue de la quantification des codes-barres avec le séquençage de nouvelle génération. (A) Représentation de la façon de mettre en place des tubes pour la première réaction de PCR imbriquée (7 tubes : un pour le contrôle négatif, un pour le contrôle positif et les 4 tubes restants pour l’ADN génomique. Le dernier tube servira de tube de mélange principal). Après la première réaction de PCR, combinez les tubes d’ADN génomique en un seul tube et mélangez. (B) Les produits de la première réaction de PCR imbriquée serviront de modèles pour la deuxième réaction de PCR imbriquée. Après la deuxième PCR, combinez les tubes d’ADN génomique en un seul tube et mélangez. Veuillez cliquer ici pour voir une version agrandie de cette figure.
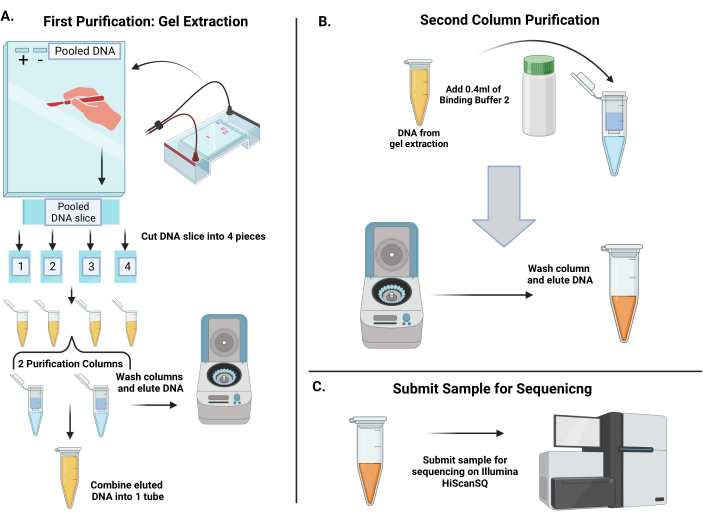
Figure 4 : Flux de travail pour purifier les codes-barres de l’ARNsh amplifié et séquencer les codes-barres pour quantifier leur représentation aux points de temps 1 et 2. (A) Versez un gel d’agarose à 3,5 %. Étant donné que le volume de l’ADN mis en commun dépasse la limite d’un puits, collez 4 à 6 dents d’un peigne en gel ensemble pour produire un grand puits. Préparez les produits PCR avec un colorant de charge 6x et chargez le contrôle positif, le contrôle négatif et l’ADN mis en commun dans le gel. Après l’électrophorèse, une grande bande d’environ 250 paires de bases devrait apparaître dans la voie de l’ADN mis en commun. À l’aide d’un scalpel propre, exciser toute la bande, puis la couper en 4 tranches de gel. Solubiliser les morceaux de gel, puis les combiner en deux colonnes de spin pour éluer l’ADN. Combinez l’ADN élué dans un tube. (B) L’ADN est purifié par une deuxième étape de purification. Ajoutez le tampon de liaison 2 dans le tube d’ADN mis en commun, puis pipetez sur une colonne de spin. Lavez la membrane, puis éluez l’ADN dans de l’eau distillée. (C) Soumettre l’ADN purifié au noyau de séquençage. Veuillez cliquer ici pour voir une version agrandie de cette figure.
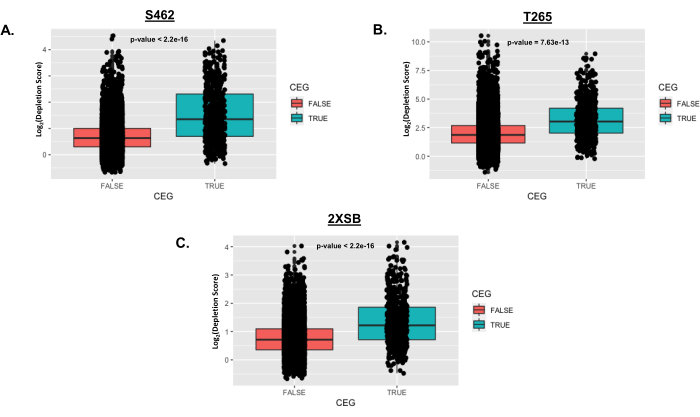
Figure 5 : Exemples représentatifs de la distribution des gènes essentiels de base après analyse telle que décrite dans le protocole. Dans cet exemple, trois lignées cellulaires MPNST humaines (S462, T265 et 2XSB) ont été criblées à l’aide de banques d’ARNh DECIPHER de Cellecta. Pour chaque lignée cellulaire MPNST humaine, une boîte à moustaches a été créée pour comparer les scores d’appauvrissement au niveau des gènes pour les gènes de la liste des gènes essentiels de base (CEG ; Boîte à moustaches vraie)25 à celle des gènes qui ne se trouvent pas dans la liste des CEG (boîte à moustaches fausse). Les points de données individuels sont superposés au-dessus de chaque boîte à moustaches. Les valeurs P proviennent d’un test t standard comparant les scores d’appauvrissement au niveau des gènes des CEG à ceux des non-CEG. Abréviations : CEG = gène essentiel de base ; MPNST = Tumeur maligne de la gaine des nerfs périphériques. Veuillez cliquer ici pour voir une version agrandie de cette figure.
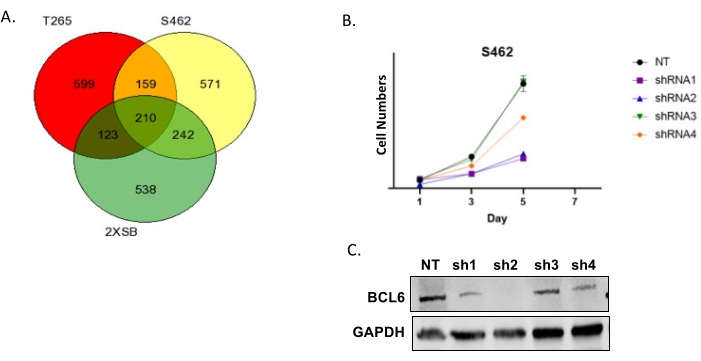
Figure 6 : Validation des résultats du criblage. (A) Diagramme de Venn représentatif des résultats qui se chevauchent dans trois lignées cellulaires MPNST humaines. (B) Cellules MPNST humaines S462 transduites avec un vecteur lentiviral non ciblé (NT) et un lentivirus exprimant quatre shRNAs BCL6 différents (shRNA1, shRNA2, shRNA3 et shRNA4). Les cellules ont été transduites avec du lentivirus, puis traitées avec un agent de sélection (puromycine) pendant 3 jours. Le nombre de cellules a ensuite été évalué au cours des sept jours suivants. (C) Analyse par transfert Western montrant les niveaux de protéines de BCL6 après transduction avec les lentivirus NT, shRNA1, shRNA2, shRNA3 et shRNA4. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tableau 1 : Disposition des plaques pour le titrage lentiviral. Veuillez cliquer ici pour télécharger ce tableau.
Tableau 2 : Mise en place initiale des premières réactions PCR. Veuillez cliquer ici pour télécharger ce tableau.
Tableau 3 : Préparation du mélange maître pour la première réaction de PCR. Veuillez cliquer ici pour télécharger ce tableau.
Tableau 4 : Paramètres de la première PCR de Cellica. Veuillez cliquer ici pour télécharger ce tableau.
Tableau 5 : Configuration initiale de la deuxième réaction PCR. Veuillez cliquer ici pour télécharger ce tableau.
Tableau 6 : Paramètres de la deuxième PCR de Cellica. Veuillez cliquer ici pour télécharger ce tableau.
Discussion
Les méthodes détaillées présentées ici ont été développées pour étudier la néoplasie du système nerveux périphérique et la pathogenèse MPNST. Bien que nous ayons trouvé ces méthodes efficaces, il faut reconnaître qu’il existe certaines limites potentielles aux méthodes que nous décrivons ici. Ci-dessous, nous discutons de certaines de ces limitations et des stratégies potentielles pour les surmonter dans d’autres systèmes de modèles.
Nous avons constaté que le séquençage de l’exome entier identifie efficacement les mutations d’intérêt chez les souris P 0-GGFβ3. Il faut cependant reconnaître que le séquençage de l’exome lui-même a des limites. Premièrement, le séquençage de l’exome entier n’est pas une approche efficace pour identifier les produits génétiques de fusion. En effet, la majorité des cassures chromosomiques et de la fusion subséquente impliquent principalement des régions et des introns intergéniques, car ces régions représentent la majorité du génome. Au lieu de cela, nous avons constaté que le séquençage de l’ARN avec des lectures d’extrémité appariées identifie beaucoup plus efficacement les gènes de fusion. Il y a aussi la question de l’efficacité avec laquelle le séquençage de l’exome entier identifie des régions relativement grandes de perte chromosomique.
Bien que plusieurs algorithmes aient été développés pour identifier de telles pertes, le terme « séquençage de l’exome entier » est lui-même trompeur car la capture de l’exome, même dans les bonnes séries, manque souvent jusqu’à 5 à 10 % des régions exoniques. Pour cette raison, nous complétons régulièrement le séquençage de l’exome entier par d’autres approches telles que l’hybridation génomique comparative sur puce (aCGH). Après avoir identifié les gains et les pertes, nous examinons les gènes au sein de ces intervalles et les comparons aux mutations pilotes connues qui sont associées à leurs homologues humains. Cependant, le génome de la souris est plus stable que le génome humain27. Par conséquent, les tumeurs de souris ne présentent généralement pas de chromothripsis analogue à ce que l’on observe dans les néoplasmes humains. Le schéma dans les tumeurs de souris est au contraire beaucoup plus simple, tendant vers des gains ou des pertes de chromosomes entiers avec relativement peu de délétions focales qui ont tendance à se produire sous une forte pression sélective22,23.
Il y a quelques pièges potentiels que nous avons rencontrés lors de la réalisation de criblages d’ARNsh à l’échelle du génome. L’un des problèmes les plus courants que nous rencontrons est la transduction relativement faible des vecteurs lentiviraux dans les cellules cibles. Nous constatons le plus souvent que le problème est dû à un titre incorrect des pools de lentiviraux emballés. Étant donné que les cultures de cellules tumorales de souris à passage précoce sont une ressource limitative, de nombreux chercheurs tenteront plutôt de titrer leur lentivirus en utilisant une autre lignée cellulaire établie qui est plus facilement disponible. Le problème avec cette approche, cependant, est que l’efficacité de la transduction lentivirale peut varier considérablement d’un type de cellule à l’autre. C’est pour cette raison que nous recommandons de titrer le lentivirus sur les cellules réelles qui seront utilisées dans l’expérience. Nous avons également rencontré des problèmes avec des titres viraux relativement faibles. Ce problème reflète le plus souvent une mauvaise transfection des cellules 293T lors de la production du virus emballé.
Il est possible d’obtenir des résultats faussement positifs lors de l’exécution de criblages d’ARNsh à l’échelle du génome. De ce fait, une fois que nous avons identifié les cibles potentiellement médicamenteuses qui nous intéressent le plus, nous validons toujours les résultats de nos criblages d’ARNsh. En règle générale, nous utiliserons deux approches différentes pour valider les cibles à taux d’intérêt élevé. Tout d’abord, nous inhibons l’expression des gènes à l’aide de deux ou plusieurs shRNA distincts de ceux utilisés dans le criblage initial et déterminons l’effet que cela a sur la prolifération et la survie des cellules tumorales. Deuxièmement, nous obtenons le(s) médicament(s) identifié(s) dans la base de données sur les interactions géniques médicamenteuses et déterminons l’effet que cela a sur la prolifération et la survie des cellules tumorales. Nous utilisons les deux approches en tandem parce que nous avons rencontré des circonstances dans lesquelles les shRNAs fonctionnent et le médicament ne fonctionne pas. Dans au moins certains de ces cas, l’examen de l’ensemble de la séquence de l’exome a montré que la protéine ciblée est produite par un gène qui présente une mutation susceptible d’affecter les interactions médicament-protéine.
Les approches décrites ci-dessus fourniront à l’investigateur un moyen applicable d’identifier les mutations pilotes potentielles qui se produisent dans les néoplasmes rares, d’identifier fonctionnellement les voies de signalisation nécessaires à la prolifération et à la survie, et de hiérarchiser les cibles pour le développement thérapeutique. Nous espérons que d’autres chercheurs trouveront ces approches utiles pour identifier des cibles thérapeutiques clés dans d’autres cancers humains. Le lecteur doit cependant savoir qu’il existe d’autres approches génomiques fonctionnelles qui peuvent être utilisées pour identifier les gènes impliqués dans la pathogenèse tumorale et les gènes codant pour des cibles thérapeutiques potentielles. À titre d’exemple, il existe des banques CRISPR qui peuvent être utilisées d’une manière analogue à celle que nous décrivons pour les banques shRNA. Des criblages fonctionnels peuvent également être réalisés in vivo pour identifier les gènes favorisant la tumorigenèse. À titre d’exemple, le système de mutagénèse somatique basé sur les transposons de La Belle au bois dormant a déjà été utilisé pour cibler les cellules de Schwann et leurs précurseurs, ce qui a permis d’identifier plusieurs centaines de gènes impliqués dans la pathogenèse du MPNST28. Étant donné que ces systèmes abordent la génomique fonctionnelle de manière distincte, nous recommandons au chercheur d’examiner attentivement les objectifs de ses expériences prévues et de fonder son choix d’une méthodologie de génomique fonctionnelle sur ces objectifs.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Ces travaux ont été financés par des subventions de l’Institut national des maladies neurologiques et des accidents vasculaires cérébraux (R01 NS048353 et R01 NS109655 à S.L.C. ; R01 NS109655-03S1 à D.P.J.), l’Institut national du cancer (R01 CA122804 à S.L.C.) et le ministère de la Défense (X81XWH-09-1-0086 et W81XWH-12-1-0164 à S.L.C.).
matériels
| Name | Company | Catalog Number | Comments |
| Bioruptor Sonication System | Diagenode | UCD-600 | |
| CASAVA 1.8.2 | |||
| Cbot | Illumina, San Diego, CA | N/A | |
| Celigo Image Cytometer | Nexcelom | N/A | |
| Cellecta Barcode Analyzer and Deconvoluter software | |||
| Citrisolve Hybrid | Decon Laboratories | 5989-27-5 | |
| Corning 96-well Black Microplate | Millipore Sigma | CLS3603 | |
| Diagenode Bioruptor 15ml conical tubes | Diagenode | C30010009 | |
| dNTP mix | Clontech | 639210 | |
| Eosin Y | Thermo Scientific | 7111 | |
| Elution buffer | Qiagen | 19086 | |
| Ethanol (200 Proof) | Decon Laboratories | 2716 | |
| Excel | Microsoft | ||
| FWDGEX 5’-CAAGCAGAAGACGGCATACGAGA-3’ | |||
| FWDHTS 5’-TTCTCTGGCAAGCAAAAGACGGCATA-3’ | |||
| GexSeqS (5’ AGAGGTTCAGAGTTCTACAGTCCGAA-3’ | HPLC purified | ||
| GraphPad Prism | Dotmatics | ||
| Harris Hematoxylin | Fisherbrand | 245-677 | |
| Illumina HiScanSQ | Illumina, San Diego, CA | N/A | |
| Paraformaldehyde (4%) | Thermo Scientific | J19943-K2 | |
| PLUS Transfection Reagent | Thermo Scientific | 11514015 | |
| Polybrene Transfection Reagent | Millipore Sigma | TR1003G | |
| PureLink Quick PCR Purification Kit | Invitrogen | K310001 | |
| Qiagen Buffer P1 | Qiagen | 19051 | |
| Qiagen Gel Extraction Kit | Qiagen | 28704 | |
| RevGEX 5’-AATGATACGGCGACCACCGAGA-3’ | |||
| RevHTS1 5’-TAGCCAACGCATCGCACAAGCCA-3’ | |||
| Titanium Taq polymerase | Clontech | 639210 | |
| Trimmomatic software | www.usadellab.org |
Références
- Carroll, S. L. Molecular mechanisms promoting the pathogenesis of Schwann cell neoplasms. Acta Neuropathol. 123 (3), 321-348 (2012).
- Longo, J. F., Weber, S. M., Turner-Ivey, B. P., Carroll, S. L. Recent Advances in the Diagnosis and Pathogenesis of Neurofibromatosis Type 1 (NF1)-associated Peripheral Nervous System Neoplasms. Adv Anat Pathol. 25 (5), 353-368 (2018).
- Longo, J. F., Carroll, S. L. The RASopathies: Biology, genetics and therapeutic options. Adv Cancer Res. 153, 305-341 (2022).
- Birindelli, S., et al. Rb and TP53 pathway alterations in sporadic and NF1-related malignant peripheral nerve sheath tumors. Lab Invest. 81 (6), 833-844 (2001).
- Legius, E., et al. TP53 mutations are frequent in malignant NF1 tumors. Genes Chromosomes Cancer. 10 (4), 250-255 (1994).
- Menon, A. G., et al. Chromosome 17p deletions and p53 gene mutations associated with the formation of malignant neurofibrosarcomas in von Recklinghausen neurofibromatosis. Proc Natl Acad Sci U S A. 87 (14), 5435-5439 (1990).
- Upadhyaya, M., et al. Germline and somatic NF1 gene mutation spectrum in NF1-associated malignant peripheral nerve sheath tumors (MPNSTs). Hum Mutat. 29 (1), 74-82 (2008).
- Kourea, H. P., Orlow, I., Scheithauer, B. W., Cordon-Cardo, C., Woodruff, J. M. Deletions of the INK4A gene occur in malignant peripheral nerve sheath tumors but not in neurofibromas. Am J Pathol. 155 (6), 1855-1860 (1999).
- Nielsen, G. P., et al. Malignant transformation of neurofibromas in neurofibromatosis 1 is associated with CDKN2A/p16 inactivation. Am J Pathol. 155 (6), 1879-1884 (1999).
- Gregorian, C., et al. PTEN dosage is essential for neurofibroma development and malignant transformation. Proc Natl Acad Sci U S A. 106 (46), 19479-19484 (2009).
- Lee, W., et al. PRC2 is recurrently inactivated through EED or SUZ12 loss in malignant peripheral nerve sheath tumors. Nat Genet. 46 (11), 1227-1232 (2014).
- Zhang, M., et al. Somatic mutations of SUZ12 in malignant peripheral nerve sheath tumors. Nat Genet. 46 (11), 1170-1172 (2014).
- Varela, I., et al. Somatic structural rearrangements in genetically engineered mouse mammary tumors. Genome Biol. 11 (10), 100 (2010).
- Johnson, R. A., et al. Cross-species genomics matches driver mutations and cell compartments to model ependymoma. Nature. 466 (7306), 632-636 (2010).
- Kim, M., et al. Comparative oncogenomics identifies NEDD9 as a melanoma metastasis gene. Cell. 125 (7), 1269-1281 (2006).
- Zender, L., et al. Identification and validation of oncogenes in liver cancer using an integrative oncogenomic approach. Cell. 125 (7), 1253-1267 (2006).
- Uren, A. G., et al. Large-scale mutagenesis in p19(ARF)- and p53-deficient mice identifies cancer genes and their collaborative networks. Cell. 133 (4), 727-741 (2008).
- Starr, T. K., et al. A transposon-based genetic screen in mice identifies genes altered in colorectal cancer. Science. 323 (5922), 1747-1750 (2009).
- Dupuy, A. J., et al. A modified sleeping beauty transposon system that can be used to model a wide variety of human cancers in mice. Cancer Res. 69 (20), 8150-8156 (2009).
- Carroll, S. L. The Challenge of Cancer Genomics in Rare Nervous System Neoplasms: Malignant Peripheral Nerve Sheath Tumors as a Paradigm for Cross-Species Comparative Oncogenomics. Am J Pathol. 186 (3), 464-477 (2016).
- Huijbregts, R. P., Roth, K. A., Schmidt, R. E., Carroll, S. L. Hypertrophic neuropathies and malignant peripheral nerve sheath tumors in transgenic mice overexpressing glial growth factor beta3 in myelinating Schwann cells. J Neurosci. 23 (19), 7269-7280 (2003).
- Kazmi, S. J., et al. Transgenic mice overexpressing neuregulin-1 model neurofibroma-malignant peripheral nerve sheath tumor progression and implicate specific chromosomal copy number variations in tumorigenesis. Am J Pathol. 182 (3), 646-667 (2013).
- Brosius, S. N., et al. Neuregulin-1 overexpression and Trp53 haploinsufficiency cooperatively promote de novo malignant peripheral nerve sheath tumor pathogenesis. Acta Neuropathol. 127 (4), 573-591 (2014).
- Hart, T., Brown, K. R., Sircoulomb, F., Rottapel, R., Moffat, J. Measuring error rates in genomic perturbation screens: gold standards for human functional genomics. Mol Syst Biol. 10 (7), 733 (2014).
- Hart, T., et al. Evaluation and Design of Genome-Wide CRISPR/SpCas9 Knockout Screens. G3. 7 (8), 2719-2727 (2017).
- Longo, J. F., et al. Establishment and genomic characterization of a sporadic malignant peripheral nerve sheath tumor cell line. Sci Rep. 11 (1), 5690 (2021).
- Maser, R. S., et al. Chromosomally unstable mouse tumours have genomic alterations similar to diverse human cancers. Nature. 447 (7147), 966-971 (2007).
- Rahrmann, E. P., et al. Forward genetic screen for malignant peripheral nerve sheath tumor formation identifies new genes and pathways driving tumorigenesis. Nat Genet. 45 (7), 756-766 (2013).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon
Nous utilisons des cookies afin d'améliorer votre expérience sur notre site web.
En continuant à utiliser notre site ou en cliquant sur le bouton ''continuer'', vous acceptez l'utilisation de cookies.