Method Article
Genetisches Profiling und Dropout-Screening im Genommaßstab zur Identifizierung therapeutischer Ziele in Mausmodellen für bösartige periphere Nervenscheidentumoren
In diesem Artikel
Zusammenfassung
Wir haben einen speziesübergreifenden vergleichenden Onkogenomik-Ansatz entwickelt, der genomische Analysen und funktionelle genomische Screenings verwendet, um therapeutische Ziele in Tumoren, die in gentechnisch veränderten Mausmodellen auftreten, und den entsprechenden menschlichen Tumortyp zu identifizieren und zu vergleichen.
Zusammenfassung
Maligne periphere Nervenscheidentumoren (MPNSTs) stammen von Schwann-Zellen oder ihren Vorläufern ab. Bei Patienten mit dem Tumor-Suszeptibilitäts-Syndrom Neurofibromatose Typ 1 (NF1) sind MPNSTs die häufigste Malignität und die häufigste Todesursache. Diese seltenen und aggressiven Weichteilsarkome bieten mit einer krankheitsfreien 5-Jahres-Überlebensrate von 34-60% eine düstere Zukunft. Die Behandlungsmöglichkeiten für Menschen mit MPNST sind enttäuschend begrenzt, wobei eine entstellende Operation die wichtigste Behandlungsoption ist. Viele einst vielversprechende Therapien wie Tipifarnib, ein Inhibitor des Ras-Signalwegs, haben klinisch versagt. Auch klinische Phase-II-Studien mit Erlotinib, das auf den epidermalen Wachstumsfaktor (EFGR) abzielt, und Sorafenib, das auf den vaskulären endothelialen Wachstumsfaktorrezeptor (VEGF), den Thrombozyten-Wachstumsfaktor-Rezeptor (PDGF) abzielt, und Raf in Kombination mit einer Standard-Chemotherapie haben bei den Patienten kein Ansprechen hervorgerufen.
In den letzten Jahren haben sich funktionelle genomische Screening-Methoden in Kombination mit der genetischen Profilierung von Krebszelllinien als nützlich erwiesen, um essentielle zytoplasmatische Signalwege zu identifizieren und zielspezifische Therapien zu entwickeln. Bei seltenen Tumorarten wird zunehmend eine Variante dieses Ansatzes, die sogenannte speziesübergreifende vergleichende Onkogenomik, eingesetzt, um neue therapeutische Ziele zu identifizieren. In der artübergreifenden vergleichenden Onkogenomik werden genetische Profilierung und funktionelle Genomik in gentechnisch veränderten Mausmodellen (GEM) durchgeführt und die Ergebnisse dann in den seltenen menschlichen Proben und Zelllinien validiert, die zur Verfügung stehen.
In diesem Artikel wird beschrieben, wie Kandidaten-Treibergenmutationen in menschlichen und Maus-MPNST-Zellen mittels Whole-Exom-Sequenzierung (WES) identifiziert werden können. Anschließend beschreiben wir, wie shRNA-Screenings auf Genomebene durchgeführt werden können, um kritische Signalwege in MPNST-Zellen von Mäusen und Menschen zu identifizieren und zu vergleichen und medikamentöse Ziele in diesen Signalwegen zu identifizieren. Diese Methoden bieten einen effektiven Ansatz zur Identifizierung neuer therapeutischer Ziele bei einer Vielzahl von Krebsarten beim Menschen.
Einleitung
Maligne periphere Nervenscheidentumoren (MPNSTs) sind hochaggressive Spindelzellneoplasien, die in Verbindung mit dem Tumor-Suszeptibilitätssyndrom Neurofibromatose Typ 1 (NF1) sporadisch in der Allgemeinbevölkerung und an Stellen früherer Strahlentherapie auftreten 1,2,3. NF1-Patienten werden mit einer Wildtyp-Kopie des NF1-Tumorsuppressorgens und einem zweiten NF1-Allel mit einer Loss-of-Function-Mutation geboren. Dieser Zustand der Haploinsuffizienz macht NF1-Patienten anfällig für eine zweite Loss-of-Function-Mutation in ihrem Wildtyp-NF1-Gen, die die Tumorgenese auslöst. Wenn diese "Second Hit"-NF1-Mutation in einer Zelle der Schwann-Zelllinie auftritt, ist der resultierende Tumor entweder ein dermales Neurofibrom, das in der Haut entsteht, oder ein plexiformes Neurofibrom, das sich in großen Nerven oder Nervengeflechten entwickelt. Obwohl die Pathologie dermaler und plexiformer Neurofibrome identisch ist, ist ihr biologisches Verhalten sehr unterschiedlich - obwohl sowohl dermale als auch plexiforme Neurofibrome gutartig sind, können nur plexiforme Neurofibrome eine Transformation durchlaufen und MPNSTs hervorrufen. Neben dem Verlust von Neurofibromin, dem Ras-GTPase-aktivierenden Protein, das vom NF1-Gen kodiert wird, tragen MPNSTs Mutationen mehrerer anderer Tumorsuppressorgene, darunter TP53 4,5,6,7, CDKN2A 8,9 und PTEN 10, Mutationen von Genen, die für Komponenten des Polycomb-Repressionskomplexes 2 11,12 (PRC2; die SUZ12- und EED-Gene) und aberrante Expression von Rezeptor-Tyrosinkinasen 1,2. Mutationen von NF1 und den anderen oben genannten Genen sind auch in sporadischen und strahleninduzierten MPNSTs vorhanden11,12.
Obwohl diese Fortschritte in unserem Verständnis der genomischen Anomalien in MPNSTs von unschätzbarem Wert für das Verständnis ihrer Pathogenese waren, haben sie noch nicht zur Entwicklung wirksamer neuer Therapien für MPNSTs geführt. Ein großes Hindernis für die Entwicklung neuer Therapien ist die Tatsache, dass MPNSTs seltene Krebsarten sind. Aus diesem Grund ist es schwierig, die große Anzahl von Patientenproben zu erhalten, die für globale Analysen zur Definition wichtiger Treibermutationen erforderlich sind, wie sie vom Cancer Genome Atlas (TCGA) durchgeführt werden. Unserer Erfahrung nach kann es Jahre dauern, selbst eine bescheidene Anzahl menschlicher MPNST-Exemplare zu sammeln. Um solche Einschränkungen zu überwinden, haben sich viele Forscher, die andere seltene Krebsarten untersuchen, der Verwendung der vergleichenden Onkogenomik zwischen verschiedenen Spezies zugewandt, um wesentliche Treibergenmutationen zu identifizieren, die wesentlichen zytoplasmatischen Signalwege in ihrem interessierenden Tumor zu definieren und neue therapeutische Ziele zu identifizieren. Da die Signalwege, die für die Tumorentstehung essentiell sind, zwischen Menschen und anderen Wirbeltierarten hochkonserviert sind, kann die Anwendung funktioneller Genomik-Ansätze wie shRNA-Screenings auf Genomebene ein wirksames Mittel sein, um diese neuen Treibermutationen, Signalwege und therapeutischen Ziele zu identifizieren 13,14,15,16,17,18,19 , insbesondere bei der Untersuchung seltener menschlicher Tumorarten, die in begrenzter Anzahl verfügbar sind20.
In den hier vorgestellten Methoden beschreiben wir diesen Ansatz zur Durchführung von genomischen Profilen in humanen MPNST-Zelllinien und frühen MPNST-Kulturen, die von P 0-GGFβ3-Mäusen stammen, einem gentechnisch veränderten Mausmodell (GEM), in dem die Schwann-Zell-spezifische Überexpression des Wachstumsfaktors Neuregulin-1 (NRG1) die Pathogenese plexiformer Neurofibrome und deren anschließende Progression zu MPNSTs fördert21, 22,23. Der erste Schritt in diesem Ansatz besteht darin, Kandidatentreibergene in P 0-GGFβ3-MPNSTs, humanen MPNST-Zelllinien und chirurgisch resezierten humanen MPNSTs zu identifizieren. Um die Signalwege, die von diesen Mutationen betroffen sind, funktionell zu validieren, verwenden wir dann shRNA-Screenings auf Genomebene, um die Gene zu identifizieren, die für die Proliferation und das Überleben in menschlichen und Maus-MPNST-Zelllinien erforderlich sind. Nachdem wir die Gene identifiziert haben, die für die Proliferation und das Überleben erforderlich sind, identifizieren wir die medikamentösen Genprodukte innerhalb der Sammlung von "Treffern" mit Hilfe der Drug Gene Interaction Database. Wir vergleichen auch die "Treffer" in menschlichen und Maus-MPNST-Zellen, um festzustellen, ob das GEM-Modell und die menschlichen MPNSTs eine ähnliche Abhängigkeit von denselben Genen und Signalwegen aufweisen. Die Identifizierung von Überlappungen in den Genen, die für die Proliferation und das Überleben erforderlich sind, und den betroffenen Signalwegen dient als Mittel zur Validierung des P 0-GGFβ3-Mausmodells auf molekularer Ebene. Dieser Ansatz unterstreicht auch die Wirksamkeit der Kombination von Human- und Mausbildschirmen, um neue therapeutische Ziele zu identifizieren, wobei das Mausmodell als Ergänzung zu den menschlichen Bildschirmen dienen kann. Der Wert dieses speziesübergreifenden Ansatzes zeigt sich besonders bei der Suche nach therapeutischen Zielen in seltenen Tumoren, bei denen menschliche Tumore und Zelllinien schwer zu gewinnen sind.
Protokoll
Lassen Sie vor Beginn der Studien Tierverfahren und -protokolle für den Umgang mit viralen Vektoren vom Institutional Animal Care and Use Committee (IACUC) und dem Institutional Biosafety Committee (IBC) überprüfen und genehmigen. Die hier beschriebenen Verfahren wurden von den IACUC- und IBC-Gremien der Medical University of South Carolina genehmigt und von entsprechend geschultem Personal in Übereinstimmung mit dem NIH-Leitfaden für die Pflege und Verwendung von Labortieren und den institutionellen Tierpflegerichtlinien des MUSC durchgeführt.
1. WES-Seq-Analysen und Identifizierung pathogener Varianten
- Isolierung genomischer DNA aus einer Tumorprobe oder subkonfluenten (70 %) MPNST-Zellen, die auf einer 60-mm-Schale gezüchtet wurden, unter Verwendung handelsüblicher Adsorptionsmethoden auf Basis von Kieselgel (detaillierte Schritte finden Sie im Protokoll des Herstellers). Der allgemeine Arbeitsablauf ist in Abbildung 1 dargestellt.
- Mindestens 10 μl genomische DNA mit 50 ng/μl in den Sequenzierungskern geben, es sei denn, es sind andere Mengen oder Konzentrationen genomischer DNA angegeben.
- Die Core Facility fragmentiert die genomische DNA durch Beschallung und reinigt sie dann mit der bevorzugten Methode. Die Exomerfassung und der Aufbau der Bibliothek werden mit dem bevorzugten Exom-Sequenzierungskit durchgeführt, und dem amplifizierten erfassten Exom werden Index-Tags hinzugefügt.
- Schicken Sie Proben an den Sequenzierungskern, um eine Sequenzierung des gesamten Exompaares (WES; 100 bp sequenziert von jedem Ende) durchführen zu lassen.
- FASTQ-Dateien, die vom Kern generiert werden, werden dem Prüfer zur Verfügung gestellt. Verwenden Sie nur FASTQ-Dateien, die Qualitätsmetriken für die Analyse bestehen.
- Richten Sie die FASTQ-Dateien aus und analysieren Sie sie mit handelsüblichen Softwareprogrammen (z. B. DNAStar19,20, Partek21 oder Varsome). Richten Sie die FASTQ-Dateien mit den Standardeinstellungen am Maus-Referenzgenom GRCm38/mm10 aus.
HINWEIS: Am Beispiel von DNAStar und einer MPNST-Mausprobe wird der allgemeine Arbeitsablauf in Abbildung 1 und im Folgenden kurz erläutert.- Öffnen Sie die DNAStar-Software und wählen Sie den SeqMan NGen-Workflow.
- Wählen Sie Workflow aus, indem Sie Variantenanalyse/Resequenzierung und die Art der Sequenzierungsanalyse NGS-basiertes Amplicon, Genpanel oder Exom auswählen. Klicken Sie auf Weiter.
- Wählen Sie die bevorzugte Referenzsequenz aus, indem Sie Genompaket herunterladen und das entsprechende Referenzgenom auswählen (z. B. Mus_musculus-GRCm38-dbSNP146.zip oder Homo sapien-GRCh37.p13.zip). Wenn die Kerneinrichtung eine Bettdatei bereitgestellt hat, laden Sie diese Hilfsdatei hoch. Klicken Sie auf Weiter.
- Wählen Sie Eingabesequenzen, indem Sie die entsprechende Sequenzer-Technologie, Illumina, auswählen, und legen Sie fest, dass die Sequenzierungslesevorgänge paarweise ausgeführt werden. Wählen Sie dann das Experiment-Setup und die Mehrfachstichprobe aus, und laden Sie die FASTQ-Sequenzierungsdateien hoch, indem Sie Hinzufügen auswählen. Wenn Sie mehrere Proben durchführen, kennzeichnen Sie jeden Satz mit einem eindeutigen Experiment oder Probennamen Tumor, Zelllinie oder A18, A202... Klicken Sie auf Weiter.
- Legen Sie das Kontrolldataset fest, falls vorhanden. Klicken Sie auf Weiter, und klicken Sie unter Baugruppenoptionen erneut auf Weiter. Klicken Sie unter Analyseoptionen auf den entsprechenden Variantenerkennungsmodus als Diploid, und klicken Sie dann auf Weiter. Geben Sie unter Baugruppenausgabe den Dateispeicherort des Projekts an, und legen Sie ihn fest. Klicken Sie auf Weiter. Führen Sie die Assembly auf dem lokalen Computer oder in der Cloud aus.
HINWEIS: Die resultierenden Ausrichtungsdateien können im ArrayStar-Workflow für die Variantenannotation von erkannten Einzelnukleotidpolymorphismen (SNPs) geöffnet werden. Dieser Workflow erkennt SNPs, nicht Genkopienzahlgewinne oder -verluste. Eine separate Analyse, die als SNP-Array mit hoher Dichte bezeichnet wird, erkennt Änderungen der Kopienzahl. Die Sequenzierung des gesamten Exoms erkennt Änderungen der Kopienzahl nicht zuverlässig. Im Benutzerhandbuch des Programms finden Sie Informationen zu den spezifischen Schritten, die mit dem Ausrichtungsprogramm ausgeführt werden müssen. - Wenden Sie benutzerdefinierte Filter auf die Variantenannotation erkannter SNPs an, um bestimmte Daten ein- oder auszuschließen. Um eine komprimierte Liste wahrscheinlich funktionell relevanter Varianten zu erhalten, wenden Sie die folgenden Filter auf die Variantendatensätze in dieser Hierarchie an: nicht in der Kontrolle (wenn eine normale Kontrollstichprobe verfügbar ist), Populationshäufigkeit (gnomAD, ExAC, 1.000 Genomhäufigkeiten), Allelhäufigkeit (einschließlich ≤0,001 oder 0,1 %), Abdeckungstiefe (<10 Tiefe ausschließen), Pathogenität oder ClinVar-Klasse (einschließlich: pathogen, wahrscheinlich pathogen; ausschließen: unsicher, wahrscheinlich gutartig, gutartig) und, falls gewünscht, SNP-Typ und Codierungsauswirkung (einschließlich: nicht-synonym, missense, nonsense, frameshift, in-frame, spleißen).
HINWEIS: Eine bekannte relevante Krebsgenliste kann auch auf die endgültige Liste angewendet werden, um nur bestimmte krankheitsrelevante Gene abzurufen, siehe Schritt 1.3.7. Diese Filter können die Liste der Genvarianten auf weniger als 20 Gene reduzieren.- Verwenden Sie die Varianten-Allelfrequenz, um Sequenzierungsfehler-SNPs herauszufiltern. Homozygote und heterozygote Varianten werden zu etwa 100 % bzw. 50 % für eine reine, nicht-kontaminierende Zellpopulation repräsentiert (eine etablierte Tumorzelllinie sollte eine einzige Population isogener Tumorzellen darstellen). Wenden Sie in diesem Fall 100-90 % und 50-40 % Allelfrequenzen für Varianten als Filter an und entfernen Sie so alle darunter liegenden Varianten. Wenn SNP-Array-Daten mit hoher Dichte auch für denselben Datensatz verfügbar sind, wenden Sie den Kopienzahlgewinn oder -verlust auf SNPs mit unterschiedlichen Allelfrequenzen an, die kleiner als homozygote oder heterozygote Verhältnisse sind (d. h. ein Kopienzahlgewinn, der zu 2 Kopien von Allel A und 1 Kopie von Allel B führt, würde geeignete Allelfrequenzen von 75 %, 25 % ergeben, bzw. ).
- Nachdem Sie pathogene Varianten mit ArrayStar identifiziert haben, exportieren und speichern Sie die Varianten-Genliste als csv-, txt- oder xls-Datei und vergleichen Sie die Gene, die diese Mutationen enthalten, mit denen in der Kohorte von P 0-GGFβ3-generierten Tumoren, um überlappende und einzigartige mutierte Genlisten zu bestimmen. Vergleichen Sie die mutierten Gene mit bekannten mutierten Genen, die mit ihren menschlichen Gegenstücken assoziiert sind (z. B. die Krebsgenliste des Bushman Lab oder eine vom Benutzer kuratierte Liste).
- Optional: Bevor Sie benutzerdefinierte Filter (1.3.6) anwenden, exportieren und speichern Sie die mit Anmerkungen versehene Datei als VCF-Datei.
HINWEIS: Diese VCF-Datei kann zum Vergleich als Sekundäranalyse in eine andere Effektor-Prädiktor-Software Varsome oder VEP hochgeladen werden. Biologische und Signalweganalysen können auch an den gefilterten Genlisten durchgeführt werden.
- Optional: Bevor Sie benutzerdefinierte Filter (1.3.6) anwenden, exportieren und speichern Sie die mit Anmerkungen versehene Datei als VCF-Datei.
- Führen Sie eine funktionelle Klassifizierung der Genliste über eine vom Benutzer bevorzugte Datenbank durch, um Informationen zu Proteinklassen, Signalwegen, Signalwegkomponenten sowie Genfamilien und Ontologien zu erhalten.
HINWEIS: PANTHER (pantherdp.org) wird hier als Beispiel verwendet.- Laden Sie eine gefilterte Genliste mit Gen-ID hoch.
- Wählen Sie den Organismus (Mus musculus) aus.
- Wählen Sie Analyse (Funktionelle Klassifikation in der Genliste angezeigt) und klicken Sie auf Biologische und Signalweganalyse für gefilterte Genlisten der Varianten.
2. shRNA-Screenings im Genommaßstab
HINWEIS: Es stehen mehrere shRNA- und CRISPR-Bibliotheken zur Verfügung, die für funktionelle Screenings im Genommaßstab mit Low-Passage-Tumorkulturen verwendet werden können. Hier beschreiben wir beispielhaft die Verwendung von CELLECTA DECIPHER shRNA-Bibliotheken. CELLECTA DECIPHER lentivirale shRNA-Bibliotheken sind für RNAi-Genscreenings im gepoolten Format optimiert. Jedes Transkript wird von mindestens 5-6 einzigartigen shRNAs anvisiert, und jeder lentivirale shRNA-Vektor enthält einen eindeutigen genetischen Barcode, der von PCR-Primerstellen flankiert wird. Diese Bibliotheken decken die Mehrheit der krankheitsrelevanten Gene von Mensch und Maus ab, aber nicht alle Gene im Genom. Die Plasmid-DNA-Pools der Cellecta-Bibliothek sind in drei Modulen verfügbar (Humanmodul I, II, III; zielt auf 15.377 Gene ab), während die Plasmidpools der Mausbibliothek in zwei Modulen (Mausmodule I und II; auf 9.145 Gene) verfügbar sind. Diese Bibliotheken werden verwendet, um "Drop-out"-Assays durchzuführen, bei denen Zielgene, die für die Proliferation und/oder das Überleben erforderlich sind, zu verschiedenen Zeitpunkten nach der viralen Transduktion unterschiedlich exprimiert werden.
- Lentivirale Verpackung
- Tag 0: 10 Schalen mit 10 Millionen 293T-Zellen/15-cm-Schale in 30 ml/Schale antibiotikafreies DMEM mit 10 % fötalem Kälberserum (FBS) anrichten.
- Tag 1: Vergewissern Sie sich, dass die Zellen am nächsten Tag zu ~80% konfluieren und bereit für die Transfektion sind. Mischen Sie in einem konischen 50-ml-Röhrchen Folgendes in dieser Reihenfolge: 600 μl Verpackungsplasmidmischung (0,5 μg/μl), 60 μl Plasmid-Barcode-Bibliothek, 12 ml DMEM (kein Serum oder Antibiotika) und 600 μl Transfektionsreagenz. 15 Minuten bei Raumtemperatur inkubieren.
- 900 μl Transfektionsreagenz und 12 ml DMEM in ein separates konisches 15-ml-Röhrchen geben und durch Vortexen mischen. 12,9 ml des Transfektionsreagenz/DMEM-Gemischs zur DNA-Mischung geben und zum Mischen schnippen. 15 Minuten bei Raumtemperatur ohne weiteres Mischen inkubieren. Geben Sie 2,5 ml dieser Mischung tropfenweise in jede 15-cm-Schale mit 293T-Zellen und inkubieren Sie über Nacht in einem Gewebekultur-Inkubator.
- Tag 2: Ersetzen Sie die Medien am nächsten Tag durch normale Wachstumsmedien, die Antibiotika enthalten.
- Tag 3: Ernten Sie das Virus, indem Sie das Medium sammeln und durch eine 0,2-μm-Filtrationseinheit leiten und in konische 15-ml-Röhrchen aliquotieren. außerdem werden fünf 1-ml-Aliquots des gefilterten Virus in Kryoröhrchen für die Verwendung bei der Virustitrierung (als 48-Stunden-Virus betrachtet) hergestellt; Lagern Sie das Virus in einem Gefrierschrank bei -80 °C. Ersetzen Sie das Medium nacheinander durch 30 ml Nährmedium auf den Platten, um ein Austrocknen der Zellen zu vermeiden.
- Tag 4: Ernten Sie Viren, indem Sie das Medium sammeln und durch eine 0,2-μm-Filtrationseinheit leiten. Aliquotes Virus in konische 15-ml-Röhrchen. Dies gilt als 72-Stunden-Virus; Lagern Sie das Virus in einem Gefrierschrank bei -80 °C.
- Lentivirale Titer-Pools
- 65 μl kationisches Polymer (10 mg/ml) zu 65 ml Tumorzellwachstumsmedium geben. Pipettieren Sie 1 ml/Well des polymerhaltigen Mediums in elf 6-Well-Gewebekulturplatten. Trypsinisieren Sie Tumorzellen in der frühen Passage und zählen Sie die Zellen mit der bevorzugten Methode, so dass jede Vertiefung 50.000 Zellen/ml/Well erhält. 1 ml Aliquots von 48 h Lentiviren aus dem Gefrierschrank in einem 37 °C warmen Wasserbad auftauen.
- Bereiten Sie für jedes Virusmodul die Infektion vor, wie in Tabelle 1 dargestellt.
- Legen Sie alle 6-Well-Platten in einen Gewebekultur-Inkubator. Entfernen Sie am nächsten Tag virale Medien und füllen Sie sie mit frischem Wachstumsmedium auf. Nach 48 Stunden wird Puromycin-haltiges Medium in alle Vertiefungen gegeben, die ausgewählt werden. Bevor die Kontrollzellen konfluieren, führen Sie die Zellzählung der überlebenden Klone mit der bevorzugten Methode durch.
HINWEIS: Die Konzentration von Puromycin, die erforderlich ist, um nicht transduzierte Zellen abzutöten, muss empirisch vorbestimmt werden, indem ein Konzentrationsbereich in einer "Kill"-Kurve getestet wird. Verwenden Sie die niedrigste Konzentration, die gleichmäßig den Tod von nicht transduzierten Kulturen induziert.
- Lentivirale Infektion von Zielzellen
- MPNST-Zellen trypsinisieren und zählen. Platte 2,5 Millionen Zellen pro 15-cm-Schale, insgesamt zwanzig 15-cm-Schalen pro Modul. Das Virus wird aufgetaut und die Zellen mit dem Virus bei einem MOI von 0,5 in Gegenwart von 5 μg/ml kationischem Polymer transduziert (Abbildung 2A).
- Entfernen Sie virenhaltige Medien am nächsten Morgen und ersetzen Sie sie durch frische Nährmedien. Kultivieren Sie die Zellen für weitere 2 Tage, um die Expression des Selektionsmarkers zu ermöglichen, und fügen Sie dann Puromycin zu den Kulturen hinzu, um nicht transduzierte Zellen zu eliminieren.
- Drei Tage nach der Zugabe von Puromycin werden die Zellen trypsinisiert und die Hälfte der Zellpopulation bei 200 × g für 5 Minuten in einer Tischzentrifuge zentrifugiert. Lagern Sie die pelletierten Zellen im Gefrierschrank bei -80 °C für die zukünftige genomische DNA-Vorbereitung. Dies ist der Referenzzeitpunkt oder Zeitpunkt 1 (T1).
- Die andere Hälfte der Zellpopulation wird neu beschichtet und für etwa 7 Populationsverdopplungen gezüchtet, bevor sie wie oben geerntet und zentrifugiert wird. Dieses Zellpellet dient als letzter Zeitpunkt (Zeitpunkt 2, T2) und wird bei -80 °C für die zukünftige genomische DNA-Isolierung gelagert (Abbildung 2).
- Isolierung genomischer DNA aus Zellen, die mit Viruspools transduziert wurden
- Das Zellpellet wird aus dem Gefrierschrank bei -80 °C aufgetaut und in 10 ml Resuspensionspuffer mit zugesetzter RNAse erneut suspendiert und sofort in zwei 15-ml-Polymethylpentenröhrchen aufgeteilt (Abbildung 2B).
- 500 μl 10 % SDS pro 5 ml hinzufügen, mischen und 5 Minuten lang bei Raumtemperatur inkubieren. Als Nächstes legen Sie die Röhrchen in ein DNA-Schergerät, um die DNA für 25 Zyklen von 30 s an und 30 s aus zu beschallen und dabei darauf zu achten, dass die Temperatur bei 4 °C bleibt.
- Fügen Sie 5 ml Phenol/Chloroform mit einem pH-Wert von 8,0 (bei 4 °C gelagert) hinzu und mischen Sie das Phenol/Chloroform vor Gebrauch gut. Nach der Zugabe von Phenol/Chloroform gut mischen, indem man bei maximaler Einstellung 45-60 s kräftig wirbelt. 60 min, -20 °C bei 7.200 × g zentrifugieren.
HINWEIS: Ein schaumiges/milchiges Aussehen nach dem Wirbeln signalisiert eine vollständige Resuspension. - 3 ml der klaren oberen Phase in ein frisches 15-ml-Röhrchen überführen und 0,5 ml 3 M Natriumacetat und 4 ml Isopropanol hinzufügen und gut mischen (Abbildung 2C). 30 min bei 20 °C bei 7.200 × g zentrifugieren.
- Nach dem Zentrifugieren den Überstand verwerfen und dann die restliche Flüssigkeit vorsichtig abpipettieren. Fügen Sie 0,5 ml 70%iges Ethanol hinzu und lösen Sie das Pellet durch Auf- und Abpipettieren. Füllen Sie das resuspendierte Pellet in ein 1,5-ml-Zentrifugenröhrchen und kombinieren Sie beide Pellets aus derselben Probe in einem einzigen 1,5-ml-Zentrifugenröhrchen. Zentrifugieren Sie bei maximaler Geschwindigkeit in einer Tischzentrifuge für 5 Minuten.
- Entsorgen Sie den Überstand und verwenden Sie ein Labortuch, um alle verbleibenden 70 % Ethanol zu absorbieren. Suspendieren Sie das Pellet erneut in 0,5 ml destilliertem Wasser und achten Sie darauf, das Pellet nicht austrocknen zu lassen, da dies die Resuspension erschwert. Lagerung der Proben bei 4 °C, bevor die DNA-Konzentration gemessen wird.
- Verschachtelte PCR zur Amplifikation von shRNA-Barcodes
HINWEIS: Entnehmen Sie die DNA aus dem Lager und legen Sie sie auf Eis. Wenn die DNA-Konzentration niedriger als 100 ng/μl ist, wird die DNA im Eiltempo vakuumgetrocknet und in einem geeigneten Volumen destilliertem Wasser resuspendiert. Wir empfehlen, jeweils nur ein Modul zu bearbeiten (Zeitpunkt 1 (T1) oder Zeitpunkt 2 (T2)). Führen Sie zwei Vorbereitungen für jeden Zeitpunkt durch, die am Ende zusammengefasst werden sollen. Daher ist es wichtig, Replikationszeitpunkte nicht am selben Tag zu verarbeiten. Das folgende Protokoll bezieht sich auf einen Zeitpunkt der genomischen DNA.- Stellen Sie 7 Röhrchen auf und beschriften Sie sie wie beschrieben: Negativkontrolle, Positivkontrolle, 4 Röhrchen für genomische DNA und eines für einen Mastermix (Abbildung 3A). Die Negativkontrolle ist destilliertes Wasser; Die Positivkontrolle ist die Plasmid-DNA, die bei der 293T-Transfektion zur Generierung des Lentivirus verwendet wird.
- Geben Sie zuerst Wasser in jedes Röhrchen, wie in Tabelle 2 angegeben.
- Bereiten Sie einen Mastermix (MM) vor, wie in Tabelle 3 gezeigt.
- Geben Sie 18 μl MM in jedes Röhrchen mit Wasser. Als nächstes fügen Sie 25 μl genomische DNA zu jedem der 4 Template-Röhrchen hinzu. Geben Sie dann 1 μl Positivkontrolle (10 ng/μl) in das Positivkontrollröhrchen, wobei darauf zu achten ist, dass andere Röhrchen nicht mit Positivkontroll-DNA kontaminiert werden. Zum Schluss fügen Sie 2 μl Polymerase in jedes Röhrchen hinzu und geben Sie zuerst in die 4 Probenröhrchen und zuletzt in das Positivkontrollröhrchen. mischen und schleudern Sie die PCR-Röhrchen herunter (Abbildung 3).
- Die PCR-Reaktion ist unter den in Tabelle 4 angegebenen Bedingungen durchzuführen.
- Während die erste PCR läuft, bereiten Sie die Röhrchen für die 2. PCR vor, wie in Tabelle 5 angegeben. Richten Sie sieben Röhrchen ein: Negativkontrolle, Positivkontrolle, 4 Röhrchen für genomische DNA und eines für MM (Abbildung 3B).
- Geben Sie 22 μl MM in jedes Röhrchen mit Wasser und warten Sie, bis die erste PCR-Runde abgeschlossen ist, bevor Sie die DNA und Polymerase hinzufügen.
- Sobald die erste PCR-Runde abgeschlossen ist, kombinieren Sie 4 Probenröhrchen zu einem Mikrozentrifugenröhrchen und mischen Sie.
- Geben Sie 25 μl in jedes Probenröhrchen mit Wasser.
- Als nächstes werden 2 μl der ersten PCR-Negativkontrolle und 2 μl der ersten PCR-Positivkontrolle in entsprechend markierte Röhrchen gegeben.
- Geben Sie 2 μl Polymerase in jedes Röhrchen und geben Sie zuerst die 4 Probenröhrchen und zuletzt die Negativkontrolle.
- Führen Sie die PCR-Reaktion wie in Tabelle 6 angegeben durch.
HINWEIS: Nach der Analyse der Ergebnisse der ersten PCR-Reaktion durch Elektrophorese an einem 3,5%igen Agarose-Gel kann die Anzahl der Zyklen für die nächste Wiederholung dieses Verfahrens auf 9-10 reduziert werden, wenn eine Fülle von Produkten erhalten wird. Wenn die Produktausbeute gering ist, können die Zyklen auf 14 erhöht werden. - Wenn die zweite PCR-Reaktion abgeschlossen ist, mischen Sie 4 Proben in ein Mikrozentrifugenröhrchen plus 80 μl 6-fachen Beladungsfarbstoff. Für Positiv- und Negativkontrollen verwenden Sie 20 μl der Probe.
- Bereiten Sie ein 3,5%iges Agarose-Gel in Trisborat-EDTA (FSME)-Puffer vor (Abbildung 4A). Um das große Probenvolumen unterzubringen, erstellen Sie eine große Vertiefung im Gelkamm, indem Sie mehrere Vertiefungen zusammenkleben (wenn kein Kamm für das Volumen verfügbar ist), und stellen Sie sicher, dass zwei Vertiefungen für die Positiv- und Negativkontrollen zur Verfügung stehen. Lassen Sie das Gel 1 Stunde lang bei 90 V laufen.
- Visualisieren Sie das Gel und bestätigen Sie eine Bande bei ca. 250 Basenpaaren (bp).
- Erste Reinigung: Gel-Extraktion
- Schneiden Sie mit einem sauberen Skalpell oder einer Rasierklinge das 250-bp-Band heraus und schneiden Sie so viel Gel wie möglich ab. Teilen Sie das große Band in 4 Stücke und geben Sie jedes Stück in ein sauberes Mikrozentrifugenröhrchen. Messen und notieren Sie das Gewicht jeder Gelscheibe.
HINWEIS: Jedes Stück sollte etwa 200 mg oder weniger pro Röhrchen enthalten. - Fügen Sie 6 Volumina Solubilisierungspuffer hinzu (z. B. wenn das Gelstück 200 mg wiegt, fügen Sie 1,2 ml Puffer hinzu). Die Röhrchen in ein 50 °C warmes Wasserbad legen und alle 10-15 min drehen, bis sich die Gelscheiben aufgelöst haben. Geben Sie dann 1 Volumen Isopropanol in jedes Röhrchen (z. B. 0,2 ml Isopropanol, wenn das Gelstück 200 mg wiegt).
- Verwenden Sie zwei Spin-Säulen für die Aufreinigung und laden Sie die Proben aus allen 4 Röhrchen in die Säulen. Führen Sie mehrere Drehungen durch, um das gesamte Probenvolumen zu verarbeiten, da die Säulen nur 750 μl fassen. Schleudern Sie die Säulen bei 17.900 × g für 1 Minute in einer herkömmlichen Tischmikrozentrifuge und verwerfen Sie den Durchfluss jedes Mal.
- Waschen Sie die Säulen mit 750 μl Waschpuffer und zentrifugieren Sie 1 Minute lang bei 17.900 × g . Nach dem Waschen wird der Durchfluss verworfen und die Schleudersäule durch Zentrifugieren bei 17.900 × g für 3 Minuten getrocknet. Elutieren Sie die DNA mit 50 μL destilliertem Wasser pro Säule und zentrifugieren Sie wie zuvor für 1 min und kombinieren Sie beide Röhrchen für ein Gesamtvolumen von 100 μL der Probe.
- Schneiden Sie mit einem sauberen Skalpell oder einer Rasierklinge das 250-bp-Band heraus und schneiden Sie so viel Gel wie möglich ab. Teilen Sie das große Band in 4 Stücke und geben Sie jedes Stück in ein sauberes Mikrozentrifugenröhrchen. Messen und notieren Sie das Gewicht jeder Gelscheibe.
- Zweite Reinigung
- In diesem nächsten Aufreinigungsschritt wird Bindungspuffer 2 (aus dem PCR-Aufreinigungskit) verwendet, der kleine Fragmente nicht eliminiert. Zu den 100 μl DNA werden 400 μl Puffer hinzugefügt und auf eine Spin-Säule geladen (Abbildung 4B). Die Probe wird 1 Minute lang bei 17.900 × g zentrifugiert.
- Als nächstes wird die Membran mit 650 μl Waschpuffer gewaschen und wie zuvor geschleudert. Trocknen Sie die Schleudersäule mit zusätzlicher Zentrifugation wie oben für 3 min.
- Die DNA wird mit 30 μl destilliertem Wasser eluiert und 1 Minute lang bei maximaler Geschwindigkeit geschleudert. Nach der Elution ist die Konzentration mit einem Spektralphotometer zu überprüfen. Stellen Sie sicher, dass die Konzentration nicht niedriger als 10 ng/μl und nicht höher als 70-80 ng/μl ist. Lagern Sie die DNA in einem Gefrierschrank bei -20 °C.
- Sequenzierung von amplifizierten Barcodes
- Für die Sequenzierung werden gereinigte Barcodes mit Elutionspuffer (EB) auf 0,75 ng/μl verdünnt. Um die Sequenzvielfalt zu erhöhen, werden die Amplikons bei 17 pM geclustert, einschließlich 30 % (v/v) PhiX. Führen Sie Single-End-Clustering (SE) auf einem automatisierten Cluster-Generierungssystem gemäß dem Protokoll des Herstellers durch.
- Lassen Sie den Sequenzierungskern insgesamt 36 Single-End-Sequenzierungszyklen auf einem NextGen-Sequenzer durchführen (Abbildung 4C). Fügen Sie den Illumina-Sequenzierungsprimern bei 0,5 μM einen benutzerdefinierten Primer GexSeqS hinzu.
- Verwenden Sie die referenzierte Analysesoftware, um Fastq-Dateien zu generieren und sie mit einer Software zu verarbeiten, um die Leselängen auf 18 Nukleotide zu kürzen.
- Verwenden Sie Barcode Analyzer und Deconvoluter-Software, um getrimmte Lesevorgänge zu dekonvolutieren. Berechnen Sie die Fold-Depletion-Werte für jede shRNA als Verhältnis der Anzahl der Lesevorgänge zum Referenzzeitpunkt (T1) zum letzten Zeitpunkt (T2).
- Analyse der shRNA-Screening-Ergebnisse und Identifizierung potenzieller therapeutischer Ziele
HINWEIS: In der shRNA-Bibliothek von Cellecta werden die meisten Gene entweder von 5 (67 %) oder 6 verschiedenen shRNAs (32 %) angegriffen. Mehrere Housekeeping-Gene, die in den meisten Zellen Treffer sein sollten, werden jedoch von einer großen Anzahl von shRNAs angegriffen und dienen als Kontrollen, die den Bereich der Scores für negative Werte definieren.- Um eine Verzerrung gegenüber Genen zu verhindern, auf die eine größere Anzahl von shRNAs abzielt, sollten die Depletionswerte logarithmisch transformiert werden. Führen Sie eine Quantilschätzung durch, indem Sie das 80. Perzentil für jedes Gen aus seiner empirischen Verteilung berechnen.
- Um eine Nullverteilung der logarithmischen Fold-Depletion-Werte zu generieren, nehmen Sie an, dass >95 % der Gene nicht erschöpft werden und dass ihre logarithmischen Quantil-Werte eine Normalverteilung aufweisen. Verwenden Sie den Median der empirischen Verteilung, um den Mittelwert der Nullverteilung zu schätzen. Klassifizieren Sie anhand dieser Nullverteilung alle Gene mit logarithmisch-fachen Verarmungswerten, die größer als das 95. Perzentil der Nullverteilung sind, als "Treffer".
HINWEIS: Alle Treffer sollten mindestens zwei shRNAs mit Verarmungswerten über dem Grenzwert aufweisen (Abbildung 5). - Um die Qualität der RNAi-Dropout-Screening-Daten und die Validität des Cut-Points zu bewerten, wird eine Reihe von Genen verwendet, die von der COLT Cancer RNAi Screening Initiative24,25 als "Core Essential" definiert wurden. Entfernen Sie CEGs aus der Trefferliste, um die anfängliche Liste potenzieller therapeutischer Ziele zu erstellen.
HINWEIS: Gene im COLT-Set wurden in >50 % der 72 Krebszelllinien, die von COLT gescreent wurden, als Treffer gewertet. Die Liste der essentiellen Gene enthält 640 Gene. - Um potenzielle therapeutische Ziele zu identifizieren, die häufig oder einheitlich von mehreren MPNST-Zelllinien benötigt werden, erstellen Sie Venn-Diagramme der Nicht-CEG-Treffer, die in Screenings verschiedener MPNST-Zelllinien oder früher Passagekulturen identifiziert wurden (Abbildung 6A). Priorisieren Sie Nicht-CEG-Treffer, die in allen oder den meisten MPNST-Linien oder -Kulturen erforderlich sind.
- Alternativ können Sie Signalweganalysen auf der Nicht-CEG-Trefferliste jeder Zelllinie oder Frühpassagekultur durchführen, um Gene zu identifizieren, deren Produkte Komponenten von Signalwegen kodieren, die für die Proliferation und/oder das Überleben von Tumorzellen erforderlich sind. Vergleichen Sie dann die Signalwege, die in der Signalweganalyse von Nicht-CEGs identifiziert wurden, mit den Signalwegen, die von Mutationen betroffen sind, die mit WES identifiziert wurden.
HINWEIS: Wir finden es besonders nützlich, Signalwege zu identifizieren, die konsistent von Mutationen betroffen sind, die in WES identifiziert wurden, und sie mit den Signalwegen zu vergleichen, die in shRNA-Screenings als kritisch identifiziert wurden.
- Alternativ können Sie Signalweganalysen auf der Nicht-CEG-Trefferliste jeder Zelllinie oder Frühpassagekultur durchführen, um Gene zu identifizieren, deren Produkte Komponenten von Signalwegen kodieren, die für die Proliferation und/oder das Überleben von Tumorzellen erforderlich sind. Vergleichen Sie dann die Signalwege, die in der Signalweganalyse von Nicht-CEGs identifiziert wurden, mit den Signalwegen, die von Mutationen betroffen sind, die mit WES identifiziert wurden.
- Um medikamentöse Ziele zu identifizieren, für die bereits Therapeutika verfügbar sind, überprüfen Sie die Liste der Treffer, die nach der Entfernung der essentiellen Kerngene verbleiben, mithilfe der Drug Gene Interaction Database (dgidb.org).
HINWEIS: Diese Datenbank ermöglicht die gleichzeitige Eingabe und Überprüfung von Genen und bietet Hinweise zu Medikamenten, die derzeit für diese Gene verfügbar sind. - Validieren Sie zunächst die identifizierten medikamentösen High-Interest-Targets, indem Sie ihre Genexpression mit shRNAs reduzieren, die sich von denen unterscheiden, die im anfänglichen Bibliotheksbildschirm in einem einzigen Batch-Format (nicht in einem gerade beschriebenen Bibliotheks-Batch-Format) verwendet werden. Um die Genexpression zu unterbinden, verwenden Sie zwei bis drei verschiedene lentivirale shRNAs. Bestimmen Sie drei bis vier Tage nach der Infektion die Auswirkungen, die dies auf die Proliferation und das Überleben der Tumorzellen hat, wie unten beschrieben.
3. Durchführung von Zytometer-Assays der Zellzahl und Viabilität in MPNST-Zellen, die mit potenziellen Therapeutika konfrontiert sind
- Züchten Sie MPNST-Zellen in DMEM auf 80 % Konfluenz. Spülen Sie die Zellen mit Hanks' ausgewogener Salzlösung (HBSS) bei Raumtemperatur.
HINWEIS: Züchten Sie Zellen nicht bis zur Konfluenz, da das Wachstum dieser Zellen nach der Neubeschichtung eine Zeit lang verzögert wird. - Lösen Sie die Zellen vom Substrat, indem Sie die Zellen 30 s bis 1 min lang mit einer nicht-enzymatischen Zelldissoziationslösung abdecken. Geben Sie 5 ml DMEM pro 1 ml der Dissoziationslösung hinzu und pipetieren Sie die Zellen vorsichtig auf und ab, um sich vom Substrat zu lösen.
- Zählen Sie die Zellen mit einem Hämozytometer. Plattenzellen mit einer Dichte von 1.200 Zellen pro Well in schwarzwandigen 96-Well-Platten; Bestücken Sie mindestens drei Vertiefungen für jede Arzneimittelverdünnung, die getestet werden soll, und führen Sie drei biologische Wiederholungen dieser Experimente durch.
- Um die anfänglichen Konzentrationen des zu testenden Arzneimittels zu bestimmen, überprüfen Sie die Literatur, um zu beurteilen, welche Konzentrationen gegen andere Krebszelltypen wirksam waren. Testen Sie in ersten Experimenten einen Bereich von zwei Größenordnungen über und zwei Größenordnungen unter der Arzneimittelkonzentration, die bei anderen Krebsarten verwendet wird.
- Bereiten Sie Verdünnungen des zu testenden Arzneimittels vor und geben Sie jede Verdünnung oder jedes Vehikel in mindestens drei Wiederholungsvertiefungen.
- Beurteilen Sie die Anzahl der direkten Zellen 1, 3, 5 und 7 Tage nach der Zugabe von Arzneimitteln. Geben Sie Hoechst 33342 zu einer Endkonzentration von 5 μg/ml und inkubieren Sie die Platten 30 Minuten lang bei 37 °C. Lesen Sie Platten auf einem Hochdurchsatz-Bildgebungszytometer mit der Option "Direkte Zellzahl für die Gesamtzellzahl" mit 100.000 ms Belichtungszeiten.
- Analysieren Sie die Messwerte mit der Software und exportieren Sie sie in eine Tabellenkalkulation und verwenden Sie entsprechende Software für statistische Analysen.
- Wenn statistisch signifikante Reduktionen der Zellzahl in medikamentös behandelten Wells beobachtet werden, führen Sie einen "Lebend/Tot"-Assay durch, um festzustellen, ob diese Verringerung teilweise auf die Induktion des Zelltods zurückzuführen ist.
- Plattenzellen in schwarzwandigen 96-Well-Platten, wie in Schritt 3.3 beschrieben.
- Bereiten Sie Arzneimittel vor und fügen Sie sie hinzu, wie in Schritt 3.5 beschrieben.
- Beurteilen Sie die Lebensfähigkeit und den Tod der Zellen 1, 3, 5 und 7 Tage nach der Zugabe des Arzneimittels. Calcein AM bis zu einer Endkonzentration von 1 μM und Propidiumiodid bis zu einer Endkonzentration von 1 μM in jede Vertiefung geben. Die Zellen werden 15 Minuten lang bei 37 °C inkubiert.
- Bilde Zellen auf einem bildgebenden Zytometer mit der Live +Dead-Softwareoption . Analysieren Sie die Messwerte mit der Software und exportieren Sie sie in eine Tabellenkalkulation und verwenden Sie entsprechende Software für statistische Analysen.
Ergebnisse
Abbildung 5 zeigt die Depletionswerte von essentiellen Kerngenen (CEGs), die als WAHR markiert sind, im Vergleich zu Nicht-CEGs (als FALSCH markiert) in jeder gescreenten menschlichen Zelllinie. Die Punkte stellen log2 der Fold-Depletion-Scores für einzelne Gene dar, die über einer Boxplot-Darstellung der Gesamtscore-Verteilung dargestellt werden. Der Student's t-Test wurde verwendet, um einen signifikanten Unterschied im Mittelwert der Depletionswerte zwischen den beiden Gruppen in jeder Zelllinie zu testen. Die resultierenden p-Werte werden in jedem Feld angezeigt. Beachten Sie, dass die durchschnittlichen Fold-Depletion-Werte für die CEGs signifikant höher sind als für die Nicht-CEGs. Dies ist zu erwarten, da essentielle Kerngene definitionsgemäß für die Proliferation und/oder das Überleben in den meisten Zelltypen erforderlich sind.
Abbildung 6A zeigt ein Venn-Diagramm der "Treffer" für drei humane MPNST-Zelllinien. Wir stellen in der Regel fest, dass eine große Anzahl von Genen von mehreren Linien geteilt wird. Diese Treffer haben eine hohe Priorität, da sie Gene darstellen, die für Proteine kodieren, die wahrscheinlich für die Proliferation und/oder das Überleben einer großen Untergruppe von MPNSTs unerlässlich sind. Beachten Sie auch, dass es eine Reihe von Genen gibt, die nur in einer Zelllinie getroffen werden. Dies ist häufig der Fall und sollte nicht als Hinweis darauf gewertet werden, dass die Bildschirme von schlechter Qualität sind. Die Gene, die gemeinsame Treffer zwischen mehreren Linien sind, werden dann mit Hilfe der Drug Gene Interaction Database bewertet, um Gene innerhalb dieser Untergruppe zu identifizieren, die Proteine kodieren, die mit bestehenden Wirkstoffen medikamentös behandelt werden können. Wir wählen dann mehrere davon aus und führen eine erste Validierung durch, indem wir die Expression der entsprechenden mRNA mit shRNAs ausschalten. Da einige shRNAs Off-Target-Effekte haben, testen wir immer mehrere shRNAs, die auf dasselbe Transkript abzielen. Abbildung 6B zeigt ein repräsentatives Ergebnis, in dem wir MPNST-Zellen mit einer nicht-zielgerichteten Kontrolle und mehreren shRNAs, die auf BCL6 abzielen, transduziert haben. Die Zellzahlen wurden dann zu unterschiedlichen Zeitpunkten nach der Transduktion bestimmt. Beachten Sie, dass mehrere der BCL6-shRNAs die Zellzahl deutlich reduzierten; Wie im beiliegenden Immunoblot gezeigt, korreliert der Grad der Abnahme der Zellzahl mit dem Grad des BCL6-Knockdowns. Abbildung 6C zeigt eine repräsentative Wachstumskurve für eine frühe Passage P 0-GGFβ3 MPNST-Kultur.

Abbildung 1: Arbeitsablauf für die Durchführung der Sequenzierung des gesamten Exoms von MPNST-Gewebe oder MPNST-Zellen in der frühen Passage. Das Schema veranschaulicht den allgemeinen Ablauf des Variantennachweises in tumorabgeleiteten Frühpassagekulturen. Isolierung von DNA aus frühen Passagekulturen (1) und Übermittlung von Qualitäts-DNA an den Sequenzierungskern gemäß ihren Einreichungsprotokollen (2). Der Sequenzierungskern prüft die Qualität der eingereichten DNA und führt alle notwendigen Proben- und Genombibliotheksvorbereitungen durch. Die Core Facility wird den Nutzern FASTQ-Sequenzierungsdateien mit Qualitätskontrollmetriken zur Verfügung stellen (3). Die Benutzer laden die FASTQ-Dateien in ein Genom-Alignment- und Variant-Caller-Programm ihrer Wahl hoch. (4) Annotierte Varianten werden nach benutzerdefinierten Kriterien gefiltert, um nicht relevante Varianten zu entfernen. Repräsentative Daten vergleichen resezierte menschliche MPNST-Tumorprobe mit einer Zelllinie, die aus dem Tumor gewonnen wurde26. (5) Führen Sie eine funktionale Klassifikationsanalyse mit PANTHER durch. Abkürzung: MPNST = Malignant Peripheral Nerve Sheath Tumor. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
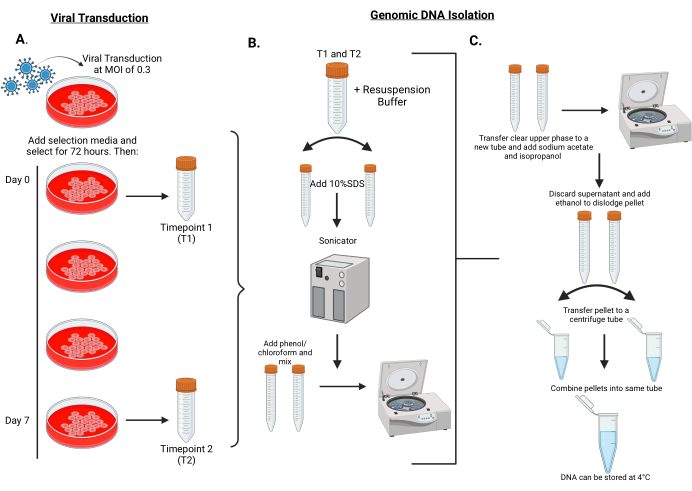
Abbildung 2: Arbeitsablauf für die Durchführung der viralen Transduktion der shRNA-Bibliotheken in MPNST-Zellen und die Isolierung genomischer DNA aus den Zellen zum Zeitpunkt 1 und 2. (A) Die Zielzellen werden mit einem niedrigen MOI von 0,3 mit barcodedierten lentiviralen Partikeln infiziert und für 72 Stunden selektiert. Die Zellen werden für 5-7 Populationsverdopplungen (ca. 7 Tage) durchgelassen. Die Zellpellets an Tag 0 und Tag 7 werden zur genomischen DNA-Isolierung bei -80 °C gelagert. Tag 0 wird als Zeitpunkt 1 (T1) und Tag 7 als Zeitpunkt 2 (T2) bezeichnet. (B) Die genomische DNA-Isolierung beginnt mit der Resuspension von Zellpellets in Resuspensionspuffer, die dann in zwei 15-ml-Röhrchen aufgeteilt werden. Um die Zelllyse zu erleichtern, wird jedem Röhrchen 10 % SDS zugesetzt und 25 Zyklen von 30 s an und 30 s aus beschallt. Nach der Beschallung wird Phenol/Chloroform in jedes Röhrchen gegeben und 45-60 s lang kräftig vortexed. Anschließend werden die Röhrchen zentrifugiert. (C) Eine klare obere Phase wird abgepipettiert und unter Zugabe von Natriumacetat/Isopropanol in ein sauberes Röhrchen gegeben und gut vermischt. Die Röhrchen werden erneut zentrifugiert. Diesmal wird der Überstand verworfen und das Pellet unter Zugabe von 70% Ethanol gelöst. Kombinieren Sie die resuspendierten Pellets in einem Röhrchen und schleudern Sie sie bei maximaler Geschwindigkeit in einer Tischzentrifuge. Verwerfen Sie den Überstand und suspendieren Sie das Pellet wieder in destilliertem Wasser. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
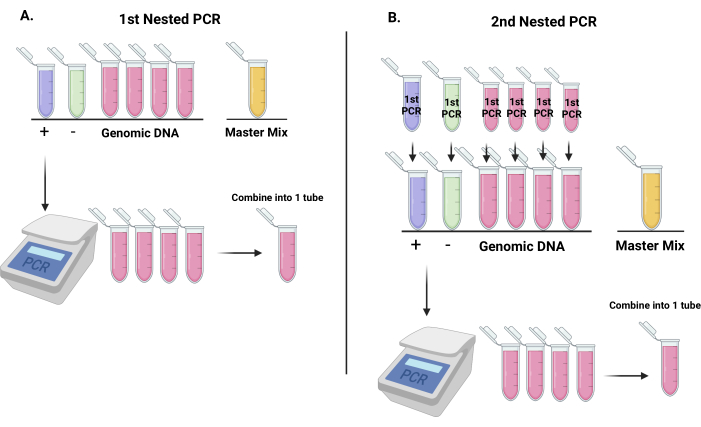
Abbildung 3: Arbeitsablauf zur Amplifikation von Barcode-Sequenzen aus lentiviralen shRNA-Vektoren in Vorbereitung auf die Quantifizierung von Barcodes mit Next-Generation-Sequencing. (A) Darstellung der Einrichtung von Röhrchen für die erste verschachtelte PCR-Reaktion (7 Röhrchen: eines für die Negativkontrolle, eines für die Positivkontrolle und die restlichen 4 Röhrchen für genomische DNA. Das letzte Röhrchen dient als Mastermix-Röhrchen). Nach der ersten PCR-Reaktion werden die genomischen DNA-Röhrchen zu einem Röhrchen zusammengefügt und vermischt. (B) Die Produkte aus der ersten verschachtelten PCR-Reaktion dienen als Matrizen für die zweite verschachtelte PCR-Reaktion. Nach der zweiten PCR werden genomische DNA-Röhrchen zu einem Röhrchen kombiniert und vermischt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
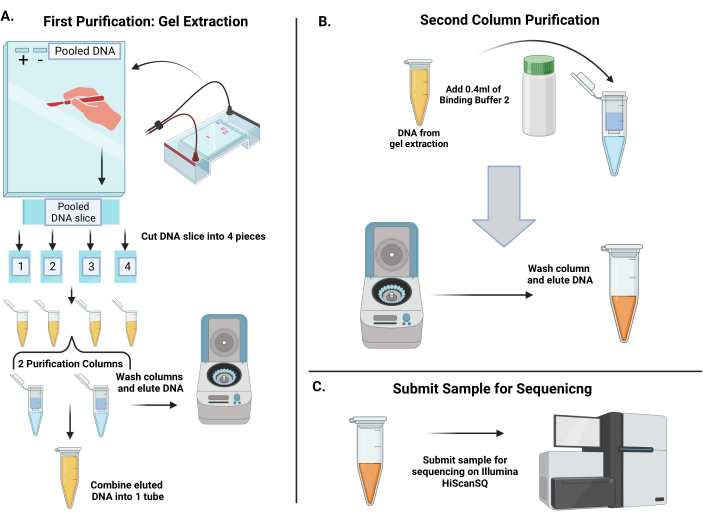
Abbildung 4: Arbeitsablauf für die Aufreinigung der amplifizierten shRNA-Barcodes und die Sequenzierung von Barcodes zur Quantifizierung ihrer Darstellung zum Zeitpunkt 1 und 2. (A) Gießen Sie ein 3,5%iges Agarose-Gel ein. Da das Volumen der gepoolten DNA die Grenze für eine Vertiefung überschreitet, kleben Sie 4-6 Zähne eines Gelkamms zusammen, um eine große Vertiefung zu erhalten. Bereiten Sie PCR-Produkte mit 6-fachem Beladungsfarbstoff vor und laden Sie die Positivkontrolle, die Negativkontrolle und die gepoolte DNA in das Gel. Nach der Elektrophorese sollte eine große Bande mit etwa 250 Basenpaaren in der gepoolten DNA-Spur erscheinen. Mit einem sauberen Skalpell das gesamte Band herausschneiden und dann in 4 Gelscheiben schneiden. Solubilisieren Sie die Gelstücke und kombinieren Sie sie dann zu zwei Spinsäulen, um DNA zu eluieren. Kombinieren Sie die eluierte DNA in einem Röhrchen. (B) Die DNA wird durch einen zweiten Reinigungsschritt gereinigt. Geben Sie Bindungspuffer 2 in das Röhrchen mit gepoolter DNA und pipettieren Sie es dann auf eine Spin-Säule. Waschen Sie die Membran und eluieren Sie dann die DNA in destilliertem Wasser. (C) Die gereinigte DNA wird dem Sequenzierungskern zugeführt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
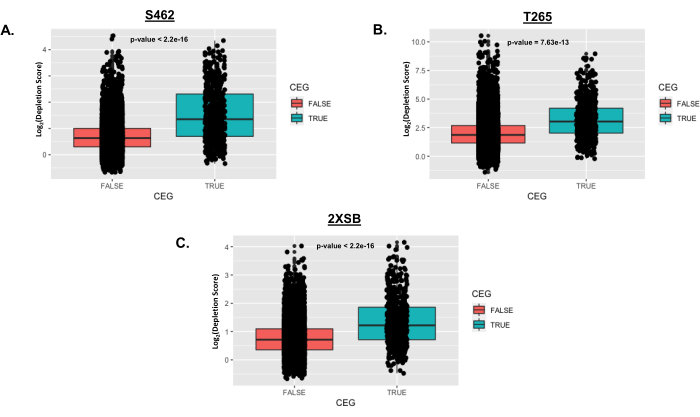
Abbildung 5: Repräsentative Beispiele für die Verteilung der essentiellen Kerngene nach der Analyse, wie im Protokoll beschrieben. In diesem Beispiel wurden drei humane MPNST-Zelllinien (S462, T265 und 2XSB) mit Cellecta DECIPHER shRNA-Bibliotheken gescreent. Für jede menschliche MPNST-Zelllinie wurde ein Boxplot erstellt, um die Depletionswerte auf Genebene für Gene in der Liste der Core Essential Genes (CEG; True Box Plot)25 zu dem von Genen, die nicht in der Liste der CEGs gefunden werden (False Box Plot). Einzelne Datenpunkte werden über jeden Boxplot gelegt. Die P-Werte stammen aus einem Standard-t-Test, der die Depletionswerte von CEGs auf Genebene mit denen von Nicht-CEGs vergleicht. Abkürzungen: CEG = Core Essential Gen; MPNST = Bösartiger peripherer Nervenscheidentumor. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
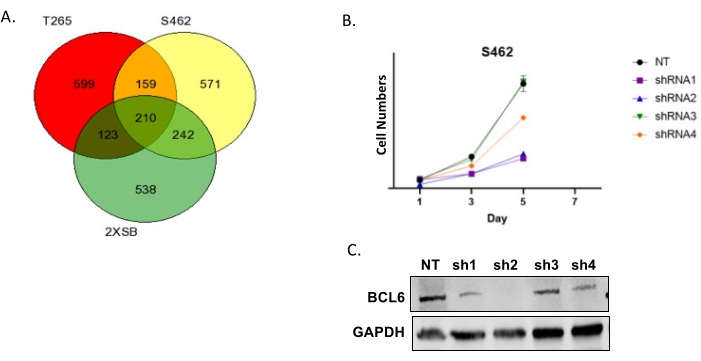
Abbildung 6: Validierung der Screening-Ergebnisse . (A) Repräsentatives Venn-Diagramm überlappender Treffer in drei humanen MPNST-Zelllinien. (B) S462 humane MPNST-Zellen, transduziert mit einem nicht-zielgerichteten (NT) lentiviralen Vektor und Lentivirus, die vier verschiedene BCL6-shRNAs (shRNA1, shRNA2, shRNA3 und shRNA4) exprimieren. Die Zellen wurden mit Lentivirus transduziert und dann 3 Tage lang mit einem Selektionsmittel (Puromycin) behandelt. Die Zellzahlen wurden dann in den nächsten sieben Tagen ausgewertet. (C) Western-Blot-Analyse, die Proteinspiegel von BCL6 nach Transduktion mit NT,shRNA1-, shRNA2-, shRNA3- und shRNA4-Lentivirus zeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Tabelle 1: Plattenlayout für die lentivirale Titerierung. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 2: Initiale Einrichtung der ersten PCR-Reaktionen. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 3: Herstellung des Mastermixes für die erste PCR-Reaktion. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 4: Erste PCR-Parameter von Cellecta. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 5: Anfänglicher Aufbau der zweiten PCR-Reaktion. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 6: Cellecta zweite PCR-Parameter. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Diskussion
Die hier vorgestellten detaillierten Methoden wurden entwickelt, um die Neoplasie des peripheren Nervensystems und die MPNST-Pathogenese zu untersuchen. Obwohl wir festgestellt haben, dass diese Methoden effektiv sind, sollten wir uns darüber im Klaren sein, dass es einige potenzielle Einschränkungen für die hier beschriebenen Methoden gibt. Im Folgenden diskutieren wir einige dieser Einschränkungen und mögliche Strategien, um sie in anderen Modellsystemen zu überwinden.
Wir haben herausgefunden, dass die Sequenzierung des gesamten Exoms Mutationen von Interesse in P 0-GGFβ3-Mäusen effektiv identifiziert. Es sollte jedoch anerkannt werden, dass die gesamte Exom-Sequenzierung selbst Grenzen hat. Erstens ist die Sequenzierung des gesamten Exoms kein effektiver Ansatz zur Identifizierung von Fusionsgenprodukten. Dies liegt daran, dass die Mehrzahl der Chromosomenbrüche und der anschließenden Fusion überwiegend intergene Regionen und Introns betreffen, da diese Regionen den Großteil des Genoms ausmachen. Stattdessen haben wir herausgefunden, dass RNA-Seq mit Paired-End-Reads Fusionsgene viel effektiver identifiziert. Es stellt sich auch die Frage, wie effektiv die Sequenzierung des gesamten Exoms relativ große Regionen des Chromosomenverlusts identifiziert.
Obwohl mehrere Algorithmen entwickelt wurden, um solche Verluste zu identifizieren, ist der Begriff "Whole Exom Sequencing" selbst irreführend, da die Erfassung des Exoms selbst bei guten Läufen oft bis zu 5-10% der exonischen Regionen verfehlt. Aus diesem Grund ergänzen wir die Sequenzierung des gesamten Exoms routinemäßig mit anderen Ansätzen wie der Array-vergleichenden genomischen Hybridisierung (aCGH). Nachdem wir Gewinne und Verluste identifiziert haben, untersuchen wir die Gene innerhalb dieser Intervalle und vergleichen sie mit den bekannten Treibermutationen, die mit ihren menschlichen Gegenstücken assoziiert sind. Das Genom der Maus ist jedoch stabiler als das menschliche Genom27. Folglich zeigen Maustumoren in der Regel keine Chromothripsis analog zu dem, was bei menschlichen Neoplasien beobachtet wird. Das Muster bei Maustumoren ist stattdessen viel einfacher und tendiert zu Gewinnen oder Verlusten des gesamten Chromosoms oder der Chromosomen mit relativ wenigen fokalen Deletionen, die tendenziell unter starkem Selektionsdruck auftreten22,23.
Es gibt einige potenzielle Fallstricke, auf die wir bei der Durchführung von shRNA-Screenings auf Genomebene gestoßen sind. Eines der häufigsten Probleme, auf die wir stoßen, ist die relativ schlechte Transduktion der lentiviralen Vektoren in die Zielzellen. Wir stellen am häufigsten fest, dass das Problem durch eine unsachgemäße Titrierung der verpackten lentiviralen Pools entstanden ist. Da die frühe Passage von Maustumorzellkulturen eine begrenzte Ressource darstellt, werden viele Forscher stattdessen versuchen, ihr Lentivirus mit einer anderen etablierten Zelllinie zu titrieren, die leichter verfügbar ist. Das Problem bei diesem Ansatz ist jedoch, dass die Effizienz der lentiviralen Transduktion von Zelltyp zu Zelltyp sehr unterschiedlich sein kann. Aus diesem Grund empfehlen wir, das Lentivirus auf die tatsächlichen Zellen zu übertragen, die im Experiment verwendet werden. Wir sind auch auf Probleme mit relativ niedrigen Virustitern gestoßen. Dieses Problem ist in den meisten Fällen auf eine schlechte Transfektion von 293T-Zellen bei der Produktion des verpackten Virus zurückzuführen.
Es ist möglich, falsch positive Treffer zu erhalten, wenn shRNA-Screenings auf Genomebene durchgeführt werden. Aus diesem Grund validieren wir, sobald wir die potenziell medikamentösen Ziele identifiziert haben, die für uns am interessantesten sind, immer die Ergebnisse unserer shRNA-Screenings. In der Regel verwenden wir zwei verschiedene Ansätze, um hochinteressante Ziele zu validieren. Zunächst unterbinden wir die Genexpression mit zwei oder mehr shRNAs, die sich von denen unterscheiden, die im ersten Screening verwendet wurden, und bestimmen die Auswirkungen, die dies auf die Proliferation und das Überleben von Tumorzellen hat. Zweitens erhalten wir das/die in der Drug Gene Interaction Database identifizierte(n) Medikament(e) und bestimmen die Auswirkungen, die dies auf die Proliferation und das Überleben von Tumorzellen hat. Wir verwenden beide Ansätze gleichzeitig, weil wir auf Umstände gestoßen sind, in denen die shRNAs funktionieren und das Medikament nicht. In zumindest einigen dieser Fälle hat die Untersuchung des gesamten Exom-Sequenzdatensatzes gezeigt, dass das Zielprotein von einem Gen produziert wird, das eine Mutation aufweist, die möglicherweise die Wechselwirkungen zwischen Arzneimitteln und Proteinen beeinflusst.
Die oben skizzierten Ansätze werden dem Forscher ein anwendbares Mittel an die Hand geben, um potenzielle Treibermutationen zu identifizieren, die bei seltenen Neoplasien auftreten, die für die Proliferation und das Überleben erforderlichen Signalwege funktionell zu identifizieren und Ziele für die therapeutische Entwicklung zu priorisieren. Wir hoffen, dass andere Forscher diese Ansätze nützlich finden werden, um wichtige therapeutische Ziele bei anderen menschlichen Krebsarten zu identifizieren. Der Leser sollte sich jedoch darüber im Klaren sein, dass es andere funktionelle genomische Ansätze gibt, mit denen Gene, die an der Tumorpathogenese beteiligt sind, und Gene, die für potenzielle therapeutische Ziele kodieren, identifiziert werden können. Als Beispiel stehen CRISPR-Bibliotheken zur Verfügung, die analog zu shRNA-Bibliotheken verwendet werden können. Funktionelle Screenings können auch in vivo durchgeführt werden, um Gene zu identifizieren, die die Tumorgenese fördern. Als Beispiel dafür wurde das auf Dornröschen-Transposon basierende somatische Mutagenesesystem bereits verwendet, um Schwann-Zellen und ihre Vorläufer anzugreifen, was zur Identifizierung von mehreren hundert Genen führte, die an der MPNST-Pathogenese beteiligt sind28. Da diese Systeme die funktionelle Genomik auf unterschiedliche Weise angehen, empfehlen wir dem Forscher, die Ziele seiner geplanten Experimente sorgfältig abzuwägen und seine Auswahl einer funktionellen Genomik-Methodik auf diese Ziele zu stützen.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Diese Arbeit wurde durch Zuschüsse des National Institute of Neurological Diseases and Stroke (R01 NS048353 und R01 NS109655 an S.L.C. unterstützt; R01 NS109655-03S1 an D.P.J.), das National Cancer Institute (R01 CA122804 an S.L.C.) und das Verteidigungsministerium (X81XWH-09-1-0086 und W81XWH-12-1-0164 an S.L.C.).
Materialien
| Name | Company | Catalog Number | Comments |
| Bioruptor Sonication System | Diagenode | UCD-600 | |
| CASAVA 1.8.2 | |||
| Cbot | Illumina, San Diego, CA | N/A | |
| Celigo Image Cytometer | Nexcelom | N/A | |
| Cellecta Barcode Analyzer and Deconvoluter software | |||
| Citrisolve Hybrid | Decon Laboratories | 5989-27-5 | |
| Corning 96-well Black Microplate | Millipore Sigma | CLS3603 | |
| Diagenode Bioruptor 15ml conical tubes | Diagenode | C30010009 | |
| dNTP mix | Clontech | 639210 | |
| Eosin Y | Thermo Scientific | 7111 | |
| Elution buffer | Qiagen | 19086 | |
| Ethanol (200 Proof) | Decon Laboratories | 2716 | |
| Excel | Microsoft | ||
| FWDGEX 5’-CAAGCAGAAGACGGCATACGAGA-3’ | |||
| FWDHTS 5’-TTCTCTGGCAAGCAAAAGACGGCATA-3’ | |||
| GexSeqS (5’ AGAGGTTCAGAGTTCTACAGTCCGAA-3’ | HPLC purified | ||
| GraphPad Prism | Dotmatics | ||
| Harris Hematoxylin | Fisherbrand | 245-677 | |
| Illumina HiScanSQ | Illumina, San Diego, CA | N/A | |
| Paraformaldehyde (4%) | Thermo Scientific | J19943-K2 | |
| PLUS Transfection Reagent | Thermo Scientific | 11514015 | |
| Polybrene Transfection Reagent | Millipore Sigma | TR1003G | |
| PureLink Quick PCR Purification Kit | Invitrogen | K310001 | |
| Qiagen Buffer P1 | Qiagen | 19051 | |
| Qiagen Gel Extraction Kit | Qiagen | 28704 | |
| RevGEX 5’-AATGATACGGCGACCACCGAGA-3’ | |||
| RevHTS1 5’-TAGCCAACGCATCGCACAAGCCA-3’ | |||
| Titanium Taq polymerase | Clontech | 639210 | |
| Trimmomatic software | www.usadellab.org |
Referenzen
- Carroll, S. L. Molecular mechanisms promoting the pathogenesis of Schwann cell neoplasms. Acta Neuropathol. 123 (3), 321-348 (2012).
- Longo, J. F., Weber, S. M., Turner-Ivey, B. P., Carroll, S. L. Recent Advances in the Diagnosis and Pathogenesis of Neurofibromatosis Type 1 (NF1)-associated Peripheral Nervous System Neoplasms. Adv Anat Pathol. 25 (5), 353-368 (2018).
- Longo, J. F., Carroll, S. L. The RASopathies: Biology, genetics and therapeutic options. Adv Cancer Res. 153, 305-341 (2022).
- Birindelli, S., et al. Rb and TP53 pathway alterations in sporadic and NF1-related malignant peripheral nerve sheath tumors. Lab Invest. 81 (6), 833-844 (2001).
- Legius, E., et al. TP53 mutations are frequent in malignant NF1 tumors. Genes Chromosomes Cancer. 10 (4), 250-255 (1994).
- Menon, A. G., et al. Chromosome 17p deletions and p53 gene mutations associated with the formation of malignant neurofibrosarcomas in von Recklinghausen neurofibromatosis. Proc Natl Acad Sci U S A. 87 (14), 5435-5439 (1990).
- Upadhyaya, M., et al. Germline and somatic NF1 gene mutation spectrum in NF1-associated malignant peripheral nerve sheath tumors (MPNSTs). Hum Mutat. 29 (1), 74-82 (2008).
- Kourea, H. P., Orlow, I., Scheithauer, B. W., Cordon-Cardo, C., Woodruff, J. M. Deletions of the INK4A gene occur in malignant peripheral nerve sheath tumors but not in neurofibromas. Am J Pathol. 155 (6), 1855-1860 (1999).
- Nielsen, G. P., et al. Malignant transformation of neurofibromas in neurofibromatosis 1 is associated with CDKN2A/p16 inactivation. Am J Pathol. 155 (6), 1879-1884 (1999).
- Gregorian, C., et al. PTEN dosage is essential for neurofibroma development and malignant transformation. Proc Natl Acad Sci U S A. 106 (46), 19479-19484 (2009).
- Lee, W., et al. PRC2 is recurrently inactivated through EED or SUZ12 loss in malignant peripheral nerve sheath tumors. Nat Genet. 46 (11), 1227-1232 (2014).
- Zhang, M., et al. Somatic mutations of SUZ12 in malignant peripheral nerve sheath tumors. Nat Genet. 46 (11), 1170-1172 (2014).
- Varela, I., et al. Somatic structural rearrangements in genetically engineered mouse mammary tumors. Genome Biol. 11 (10), 100 (2010).
- Johnson, R. A., et al. Cross-species genomics matches driver mutations and cell compartments to model ependymoma. Nature. 466 (7306), 632-636 (2010).
- Kim, M., et al. Comparative oncogenomics identifies NEDD9 as a melanoma metastasis gene. Cell. 125 (7), 1269-1281 (2006).
- Zender, L., et al. Identification and validation of oncogenes in liver cancer using an integrative oncogenomic approach. Cell. 125 (7), 1253-1267 (2006).
- Uren, A. G., et al. Large-scale mutagenesis in p19(ARF)- and p53-deficient mice identifies cancer genes and their collaborative networks. Cell. 133 (4), 727-741 (2008).
- Starr, T. K., et al. A transposon-based genetic screen in mice identifies genes altered in colorectal cancer. Science. 323 (5922), 1747-1750 (2009).
- Dupuy, A. J., et al. A modified sleeping beauty transposon system that can be used to model a wide variety of human cancers in mice. Cancer Res. 69 (20), 8150-8156 (2009).
- Carroll, S. L. The Challenge of Cancer Genomics in Rare Nervous System Neoplasms: Malignant Peripheral Nerve Sheath Tumors as a Paradigm for Cross-Species Comparative Oncogenomics. Am J Pathol. 186 (3), 464-477 (2016).
- Huijbregts, R. P., Roth, K. A., Schmidt, R. E., Carroll, S. L. Hypertrophic neuropathies and malignant peripheral nerve sheath tumors in transgenic mice overexpressing glial growth factor beta3 in myelinating Schwann cells. J Neurosci. 23 (19), 7269-7280 (2003).
- Kazmi, S. J., et al. Transgenic mice overexpressing neuregulin-1 model neurofibroma-malignant peripheral nerve sheath tumor progression and implicate specific chromosomal copy number variations in tumorigenesis. Am J Pathol. 182 (3), 646-667 (2013).
- Brosius, S. N., et al. Neuregulin-1 overexpression and Trp53 haploinsufficiency cooperatively promote de novo malignant peripheral nerve sheath tumor pathogenesis. Acta Neuropathol. 127 (4), 573-591 (2014).
- Hart, T., Brown, K. R., Sircoulomb, F., Rottapel, R., Moffat, J. Measuring error rates in genomic perturbation screens: gold standards for human functional genomics. Mol Syst Biol. 10 (7), 733 (2014).
- Hart, T., et al. Evaluation and Design of Genome-Wide CRISPR/SpCas9 Knockout Screens. G3. 7 (8), 2719-2727 (2017).
- Longo, J. F., et al. Establishment and genomic characterization of a sporadic malignant peripheral nerve sheath tumor cell line. Sci Rep. 11 (1), 5690 (2021).
- Maser, R. S., et al. Chromosomally unstable mouse tumours have genomic alterations similar to diverse human cancers. Nature. 447 (7147), 966-971 (2007).
- Rahrmann, E. P., et al. Forward genetic screen for malignant peripheral nerve sheath tumor formation identifies new genes and pathways driving tumorigenesis. Nat Genet. 45 (7), 756-766 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten
Wir verwenden Cookies, um Ihre Erfahrung auf unserer Website zu verbessern.
Indem Sie unsere Website weiterhin nutzen oder auf „Weiter“ klicken, stimmen Sie zu, unsere Cookies zu akzeptieren.