Method Article
悪性末梢神経鞘腫瘍のマウスモデルにおける治療標的を特定するための遺伝子プロファイリングおよびゲノムスケールドロップアウトスクリーニング
要約
私たちは、遺伝子改変マウスモデルで発生した腫瘍の治療標的と対応するヒト腫瘍の種類を同定および比較するために、ゲノム解析と機能的ゲノムスクリーニングを利用した種間比較腫瘍ゲノミクスアプローチを開発しました。
要約
悪性末梢神経鞘腫瘍(MPNST)は、シュワン細胞またはその前駆細胞に由来する。腫瘍感受性症候群神経線維腫症1型(NF1)の患者では、MPNSTは最も一般的な悪性腫瘍であり、主要な死因です。これらのまれで侵攻性の軟部肉腫は、5年無病生存率が34〜60%であり、厳しい未来を提供します。MPNST患者の治療選択肢は残念なことに限られており、外観を損なう手術が最も重要な治療選択肢です。Rasシグナル伝達の阻害剤であるチピファルニブなど、かつては有望視されていた多くの治療法が臨床的に失敗しています。同様に、上皮成長因子(EFGR)を標的とするエルロチニブ、血管内皮増殖因子受容体(VEGF)、血小板由来成長因子受容体(PDGF)、およびRafを標的とするソラフェニブを標準化学療法と併用した第II相臨床試験でも、患者に反応を示すことができませんでした。
近年、がん細胞株の遺伝子プロファイリングと組み合わせた機能的ゲノムスクリーニング法は、必須の細胞質シグナル伝達経路の同定や標的特異的治療法の開発に有用であることが証明されています。稀な腫瘍型の場合、異種間比較腫瘍ゲノミクスとして知られるこのアプローチのバリエーションが、新規治療標的の同定にますます用いられている。異種間比較腫瘍ゲノミクスでは、遺伝子プロファイリングと機能ゲノミクスが遺伝子改変マウス(GEM)モデルで実施され、その結果が入手可能な希少なヒト検体および細胞株で検証されます。
この論文では、全エクソームシーケンシング(WES)を使用して、ヒトおよびマウスのMPNST細胞における候補ドライバー遺伝子変異を同定する方法について説明します。次に、ゲノムスケールのshRNAスクリーニングを実施して、マウスおよびヒトMPNST細胞の重要なシグナル伝達経路を同定および比較し、これらの経路における創薬可能な標的を同定する方法について説明します。これらの方法論は、さまざまな種類のヒトがんにおける新しい治療標的を特定するための効果的なアプローチを提供します。
概要
悪性末梢神経鞘腫瘍(MPNST)は、腫瘍感受性症候群神経線維腫症1型(NF1)に関連して発生する非常に攻撃的な紡錘細胞腫瘍であり、一般集団および以前の放射線療法の部位で散発的に発生します1,2,3。NF1患者は、NF1腫瘍抑制遺伝子の野生型コピーと、機能喪失変異を有する第2のNF1対立遺伝子を持って生まれる。このハプロ不全の状態により、NF1患者は野生型NF1遺伝子の2回目の機能喪失変異の影響を受けやすくなり、腫瘍形成を引き起こします。この「セカンドヒット」NF1変異がシュワン細胞系統の細胞に発生すると、結果として生じる腫瘍は、皮膚に発生する真皮神経線維腫か、大神経または神経叢に発生する網状神経線維腫のいずれかである。皮膚神経線維腫と網状神経線維腫の病理は同じですが、それらの生物学的挙動はまったく異なります-真皮神経線維腫と網状神経線維腫はどちらも良性ですが、網状神経線維腫のみが形質転換を受け、MPNSTを引き起こす可能性があります。NF1遺伝子によってコードされるRas GTPase活性化タンパク質であるニューロフィブロミンの喪失に加えて、MPNSTは、TP53 4,5,6,7、CDKN2A 8,9、およびPTEN 10、ポリコーム抑制複合体の構成要素をコードする遺伝子の変異を含む、他の複数の腫瘍抑制遺伝子の変異を運びます2 11,12 (PRC2; SUZ12およびEED遺伝子)および受容体チロシンキナーゼの異常な発現1,2。NF1および上記の他の遺伝子の変異は、散発性および放射線誘発性MPNSTにも存在する11,12。
MPNSTのゲノム異常の理解におけるこれらの進歩は、その病因を理解する上で非常に貴重ですが、MPNSTの効果的な新しい治療法の開発にはまだ至っていません。新しい治療法の開発を妨げる大きな障壁は、MPNSTがまれながんであるという事実です。このため、The Cancer Genome Atlas(TCGA)で実施されているような主要なドライバー変異を定義する全体的な解析に必要な多数の患者サンプルを入手することは困難です。私たちの経験では、適度な数のヒトMPNST標本を蓄積するだけでも何年もかかることがあります。このような限界を克服するために、他の希少がん種を研究する多くの研究者は、種間比較腫瘍ゲノミクスを使用して、必須のドライバー遺伝子変異を同定し、目的の腫瘍に必須の細胞質シグナル伝達経路を定義し、新しい治療標的を特定しています。腫瘍形成に不可欠なシグナル伝達経路は、ヒトと他の脊椎動物種の間で高度に保存されているため、ゲノムスケールのshRNAスクリーニングなどの機能ゲノミクスアプローチを適用することは、これらの新しいドライバー変異、シグナル伝達経路、および治療標的を特定するための効果的な手段となり得ます13,14,15,16,17,18,19、特に限られた数で入手可能なまれなヒト腫瘍タイプを研究する場合20.
ここで紹介する方法論では、成長因子ニューレグリン-1(NRG1)のシュワン細胞特異的な過剰発現が叢状神経線維腫の病因とその後のMPNSTへの進行を促進する遺伝子改変マウスモデル(GEM)であるP0-GGFβ3マウスに由来するヒトMPNST細胞株および早期継代MPNST培養においてゲノムプロファイリングを実施するためのこのアプローチについて説明します21。22,23。このアプローチの最初のステップは、P 0-GGFβ3 MPNST、ヒトMPNST細胞株、および外科的に切除されたヒトMPNSTの候補ドライバー遺伝子を同定することです。次に、これらの変異の影響を受けるシグナル伝達経路を機能的に検証するために、ゲノムスケールのshRNAスクリーニングを使用して、ヒトおよびマウスのMPNST細胞株における増殖と生存に必要な遺伝子を同定します。増殖と生存に必要な遺伝子を同定した後、Drug Gene Interaction Databaseを用いて「ヒット」の集合体の中から創薬可能な遺伝子産物を同定します。また、ヒトとマウスのMPNST細胞の「ヒット」を比較して、GEMモデルとヒトMPNSTが同じ遺伝子とシグナル伝達経路に同様の依存性を示すかどうかを判断します。増殖と生存に必要な遺伝子の重複と、影響を受けるシグナル伝達経路を同定することは、P 0-GGFβ3マウスモデルを分子レベルで検証する手段として役立ちます。また、このアプローチでは、ヒトとマウスのスクリーニングを組み合わせて新しい治療標的を同定することの有効性も強調されており、マウスモデルはヒトのスクリーニングを補完する役割を果たすことができます。この異種間アプローチの価値は、ヒト腫瘍や細胞株の入手が困難な希少腫瘍の治療標的を探す場合に特に顕著です。
プロトコル
研究の開始前に、ウイルスベクターを取り扱うための動物の手順とプロトコルを、施設の動物のケアと使用委員会(IACUC)および施設のバイオセーフティ委員会(IBC)によってレビューおよび承認してもらいます。ここに記載されている手順は、サウスカロライナ医科大学のIACUCおよびIBC理事会によって承認され、実験動物のケアと使用に関するNIHガイドおよびMUSCの施設動物ケアガイドラインに従って、適切な訓練を受けた担当者によって実施されました。
1. WES-Seq解析と病原性多様体の同定
- 標準的な市販の吸着シリカゲルベースの方法を使用して、60 mmの皿上で増殖した腫瘍サンプルまたはサブコンフルエント(70%)MPNST細胞からゲノムDNAを単離します(詳細な手順については、メーカーのプロトコルを参照してください)。一般的なワークフローを 図 1 に示します。
- ゲノムDNAの量または濃度が異なる場合を除き、少なくとも10 μLのゲノムDNAを50 ng/μLでシーケンシングコアに提出してください。
- コア施設は、超音波処理によってゲノムDNAを断片化し、次いで好ましい方法を用いてそれを精製する。エクソーム捕捉およびライブラリ構築は、好ましいエクソーム配列決定キットを用いて行われ、インデックスタグは、増幅された捕捉エクソームに付加される。
- 検体をシーケンシングコアに提出し、全エクソームペアエンドシーケンシング(WES;両末端から100 bpシーケンシング)を実施します。
- コアによって生成されたFASTQファイルは、調査員に提供されます。分析には、品質指標に合格する FASTQ ファイルのみを使用してください。
- 市販のソフトウェアプログラム(DNAStar19、20、Partek21、Varsomeなど)を使用してFASTQファイルをアライメントおよび解析します。デフォルト設定を使用して、FASTQファイルをマウスリファレンスゲノムGRCm38/mm10にアライメントします。
注:DNAStarとマウスMPNST標本を例にとると、一般的なワークフローは 図 1 以下で簡単に説明します。- DNAStar ソフトウェアを開き、SeqMan NGen ワークフローを選択します。
- Variant Analysis/Resequencingを選択し、シーケンシング解析のタイプとしてNGSベースのアンプリコン、遺伝子パネル、エクソームのいずれかを選択して、ワークフローを選択します。「次へ」をクリックします。
- Download Genome Packageと適切な参照ゲノム(例:Mus_musculus-GRCm38-dbSNP146.zipまたはHomo sapien-GRCh37.p13.zip)を選択して、希望する参照配列を選択します。コアファシリティからベッドファイルの提供があった場合は、この補助ファイルをアップロードします。「次へ」をクリックします。
- 適切なシーケンサーテクノロジーである Illuminaを選択して[Input Sequences]を選択し、 シーケンシングリード が ペアエンドであることを指定します。次に、実験のセットアップ、マルチサンプルを選択し、 追加を選択してシーケンスFASTQファイルをアップロードします。複数のサンプルを実行する場合は、各ペアエンドセットに一意の実験名または サンプル名( 腫瘍、細胞株 、または A18、A202... 「 次へ」をクリックします。
- コントロールデータセットがある場合は、それを設定します。「次へ」(Next) をクリックし、「アセンブリオプション」(Assembly Options) でもう一度「次へ」(Next) をクリックします。[解析オプション]で、適切な[バリアント検出モード]で[Diploid]をクリックし、[次へ]をクリックします。[アセンブリ出力]で、プロジェクトのファイルの保存場所に名前を付けて指定します。「次へ」をクリックします。ローカル コンピューターまたはクラウドでアセンブリを実行します。
注:得られたアライメントファイルは、検出された一塩基多型(SNP)のバリアントアノテーションのために ArrayStar ワークフローで開くことができます。このワークフローでは、遺伝子コピー数の増加や減少ではなく、SNPを検出します。高密度SNPアレイと呼ばれる別の分析では、コピー数の変化を検出します。全エクソームシーケンシングでは、コピー数の変化を確実に検出することはできません。アライメントプログラムで実行する具体的な手順については、プログラムのユーザーガイドを参照してください。 - 検出されたSNPのバリアントアノテーションにユーザー定義のフィルターを適用して、特定のデータを含めたり除外したりします。機能的に関連性がある可能性のあるバリアントの要約リストを取得するには、この階層内のバリアントデータセットに次のフィルターを適用します: 制御なし (正常な対照サンプルが利用可能な場合)、 集団 頻度 (gnomAD、ExAC、1,000 ゲノム頻度)、 対立遺伝子頻度 (≤0.001 または 0.1% を含む)、カバレッジ深度 (<10 深度 を除く)、 病原性 または ClinVar クラス (含む:病原性、病原性の可能性が高い;除外:不確実、良性の可能性が高い、良性)、および必要に応じて SNPの種類とコーディングの影響 (非同義、ミスセンス、ナンセンス、フレームシフト、インフレーム、スプライシングを含む)。
注:既知の関連するがん遺伝子リストを最終リストに適用して、特定の疾患関連遺伝子のみをプルすることもできます(ステップ1.3.7を参照)。これらのフィルターは、バリアント遺伝子リストを20遺伝子未満に減らすことができます。- 変異アレル頻度を使用して、シーケンシングエラーSNPを除外します。 ホモ接合体およびヘテロ接合体バリアントは、純粋な非汚染細胞集団に対して、それぞれ約100%および50%で表されます(確立された腫瘍由来細胞株は、同原性腫瘍細胞の単一集団を表す必要があります)。この場合、バリアントに100〜90%および50〜40%の対立遺伝子頻度をフィルターとして適用し、それ以下のバリアントを削除します。同じデータセットに対して高密度SNPアレイデータも利用可能な場合は、ホモ接合体またはヘテロ接合体の比率よりも低いバリアント対立遺伝子頻度を有するSNPにコピー数ゲインまたはコピー損失を適用する(すなわち、対立遺伝子Aの2コピーおよび対立遺伝子Bの1コピーをもたらすコピー数ゲインは、75%、25%の適切なバリアント対立遺伝子頻度を作るであろう、 それぞれ)。
- ArrayStarで病原性多様体を同定した後、変異遺伝子リストをcsv、txt、またはxlsファイルとしてエクスポートして保存し、これらの変異を含む遺伝子をP 0-GGFβ3で生成された腫瘍のコホートの遺伝子と比較して、重複する固有の変異遺伝子リストを決定します。変異した遺伝子を、ヒトの対応する遺伝子に関連する既知の変異遺伝子と比較します(つまり、Bushman Lab Cancer Geneリストまたはユーザーがキュレーションしたリスト)。
- オプションで、ユーザー定義フィルター (1.3.6) を適用する前に、注釈付きファイルを VCF ファイルとしてエクスポートして保存します。
注:このVCFファイルは、他のバリアントエフェクタ予測ソフトウェアVarsomeまたはVEPにアップロードして、二次解析として比較することができます。生物学的および経路解析は、フィルタリングされたバリアント遺伝子リストに対しても実行できます。
- オプションで、ユーザー定義フィルター (1.3.6) を適用する前に、注釈付きファイルを VCF ファイルとしてエクスポートして保存します。
- ユーザーが好むデータベースを介してバリアント遺伝子リストの機能分類を実行し、タンパク質クラス、パスウェイ、パスウェイコンポーネント、および遺伝子ファミリーおよびオントロジー情報を取得します。
注:ここでは、PANTHER(pantherdp.org)を例として使用しています。- フィルターをかけた遺伝子リストを遺伝子IDでアップロードします。
- 生物(Mus musculus)を選択します。
- analysis (Functional classification displayed in gene list)を選択し、 Submit biological and pathway analysis on filtered variant gene listsをクリックします。
2. ゲノムスケールshRNAスクリーニング
注:低継代腫瘍培養のゲノムスケール機能スクリーニングに使用できるshRNAおよびCRISPRライブラリーがいくつか用意されています。ここでは、CELLECTA DECIPHER shRNAライブラリーの使用を例に説明します。CELLECTA DECIPHERレンチウイルスshRNAライブラリーは、プール形式のRNAi遺伝子スクリーニング用に最適化されています。各転写産物は少なくとも5〜6個の固有のshRNAを標的としており、各レンチウイルスshRNAベクターには、PCRプライマー部位に挟まれた固有の遺伝子バーコードが含まれています。これらのライブラリは、ヒトおよびマウスの疾患関連遺伝子の大部分をカバーしていますが、ゲノム内のすべての遺伝子をカバーしているわけではありません。CellectaのヒトライブラリープラスミドDNAプールは、3つのモジュール(ヒトモジュールI、II、III、ターゲット15,377遺伝子)で、マウスライブラリープラスミドプールは、2つのモジュール(マウスモジュールIおよびII、ターゲット9,145遺伝子)で利用できます。これらのライブラリーは、ウイルス形質導入後の異なる時点で、増殖および/または生存に必要な標的遺伝子を異なる時点で異なる形で発現させる「ドロップアウト」アッセイを実行するために使用されます。
- レンチウイルス包装
- 0日目:1000万個の293T細胞/15cmディッシュを、10%ウシ胎児血清(FBS)を含む抗生物質不使用のDMEM30 mL/ディッシュに10皿播種します。
- 1日目:翌日に細胞が~80%コンフルエントになり、トランスフェクションの準備が整っていることを確認します。50 mL のコニカルチューブに、600 μL の包装用プラスミドミックス(0.5 μg/μL)、60 μL のプラスミドバーコードライブラリー、12 mL の DMEM(血清または抗生物質を含まない)、600 μL のトランスフェクション試薬の順に混合します。室温で15分間インキュベートします。
- トランスフェクション試薬 900 μL と DMEM 12 mL を別の 15 mL コニカルチューブに入れ、ボルテックスで混合します。12.9 mLのトランスフェクション試薬/DMEMミックスをDNAミックスに加え、フリックして混合します。さらに混合せずに室温で15分間インキュベートします。この混合物2.5 mLを293T細胞の15 cmの各皿に滴下し、組織培養インキュベーターで一晩インキュベートします。
- 2日目:翌日、培地を抗生物質を含む通常の増殖培地と交換します。
- 3日目:培地を回収し、0.2 μmのろ過ユニットに通し、15 mLのコニカルチューブに分注してウイルスを回収します。また、ウイルス力価測定に使用するために、クライオバイアルでろ過したウイルスの1 mLアリコートを5つ調製します(48時間ウイルスと見なされます)。ウイルスは-80°Cの冷凍庫で保管してください。細胞が乾燥しないように、培地をプレート上で一度に1つずつ30 mLの増殖培地と交換します。
- 4日目:培地を採取し、0.2μmのろ過ユニットに通してウイルスを採取します。ウイルスを15 mLのコニカルチューブに分注します。これは72時間ウイルスと見なされます。ウイルスは-80°Cの冷凍庫で保管してください。
- 力価レンチウイルスプール
- 65 mLの腫瘍細胞増殖培地に65 μLのカチオン性ポリマー(10 mg/mL)を加えます。1 mL/ウェルのポリマー含有培地を11枚の6ウェル組織培養プレートにピペットで移します。早期継代腫瘍細胞をトリプシン化し、各ウェルが50,000細胞/mL/ウェルを受け取るように、好ましい方法を使用して細胞をカウントします。48時間レンチウイルス1 mLアリコートを冷凍庫から37°Cのウォーターバスで解凍します。
- 各ウイルスモジュールについて、 表1に示すように感染を準備します。
- すべての6ウェルプレートを組織培養インキュベーターに入れます。翌日、ウイルス培地を取り除き、新しい増殖培地を補充します。48時間後に、選択を受けるすべてのウェルにピューロマイシン含有培地を添加します。コントロール細胞がコンフルエントになる前に、希望する方法を使用して、生き残ったクローンの細胞カウントを行います。
注:形質導入されていない細胞を殺すために必要なピューロマイシンの濃度は、「死滅」曲線で濃度の範囲をテストすることにより、経験的に事前に決定する必要があります。形質導入されていない培養物の死滅を均一に誘導する最低濃度を使用してください。
- 標的細胞のレンチウイルス感染
- MPNST細胞をトリプシン化してカウントします。15 cmディッシュあたり250万個の細胞をプレートし、モジュールあたり合計20個の15 cmディッシュを播種します。ウイルスを融解し、5 μg/mLのカチオン性ポリマーの存在下でMOI0.5でウイルスを細胞に形質導入します(図2A)。
- 翌朝、ウイルスを含む培地を取り除き、新しい増殖培地と交換します。細胞をさらに2日間培養して選択マーカーを発現させ、ピューロマイシンを培養物に添加して非形質導入細胞を排除します。
- ピューロマイシンを添加した3日後、細胞をトリプシン化し、卓上遠心分離機で細胞集団の半分を200 × gで5分間遠心分離します。ペレット化した細胞を-80°Cの冷凍庫で保存し、将来のゲノムDNA調製に役立てます。これは、基準時点、または時点 1 (T1) です。
- 細胞集団の残りの半分を再播種し、上記のように回収および遠心分離する前に、約7つの集団倍増のために増殖させます。この細胞ペレットは、最終時点(時点2、 T2)として機能し、将来のゲノムDNA単離のために-80°Cで保存されます(図2)。
- ウイルスプールで形質導入した細胞からのゲノムDNAの単離
- 細胞ペレットを-80°Cの冷凍庫から解凍し、RNAseを添加した10 mLの再懸濁バッファーにペレットを再懸濁し、すぐに2本の15 mLポリメチルペンテンチューブに分割します(図2B)。
- 5 mLあたり500 μLの10% SDSを添加し、混合し、室温で5分間インキュベートします。次に、チューブをDNA剪断装置に入れて、温度を4°Cに保ちながら、30秒オン/30秒オフの25サイクルでDNAを超音波処理します。
- 使用前にフェノール/クロロホルムをよく混合し、pH 8.0(4°Cで保存)のフェノール/クロロホルム5 mLを加えます。フェノール/クロロホルムを添加した後、最大設定で45〜60秒間激しくボルテックスしてよく混合します。7,200 × gで-20°Cで60分間遠心分離します。
注:ボルテックス後の泡状/乳白色の外観は、完全な再懸濁の合図です。 - 透明な上相 3 mL を新しい 15 mL チューブに移し、0.5 mL の 3 M 酢酸ナトリウムと 4 mL のイソプロパノールを加えてよく混合します(図 2C)。7,200 × g、20°Cで30分間遠心分離します。
- 遠心分離後、上清を廃棄し、残りの液体を慎重にピペットで取り除きます。0.5 mLの70%エタノールを加え、ピペッティングで上下させてペレットを取り除きます。再懸濁したペレットを1.5 mLの遠心チューブに移し、同じサンプルから採取した両方のペレットを1本の1.5 mL遠心チューブに混ぜ合わせます。卓上型遠心分離機で最高速度で5分間遠心分離します。
- 上清を廃棄し、ラボ用ワイプを使用して、残留した70%エタノールを吸収します。ペレットを0.5 mLの蒸留水に再懸濁し、再懸濁が困難になるため、ペレットを乾燥させないように注意してください。DNA濃度を測定する前に、サンプルを4°Cで保存してください。
- shRNAバーコードを増幅するためのネストPCR
注:ストレージからDNAを取り出し、氷の上に置きます。DNA濃度が100 ng/μL未満の場合は、DNAをスピードバキュームドライし、適切な量の蒸留水に再懸濁します。一度に処理するモジュールは 1 つだけにすることをお勧めします (時点 1 (T1) または時点 2 (T2))。最後にプールされる各時点の 2 つの準備を実行します。したがって、レプリケート時点を同じ日に処理しないことが重要です。次のプロトコルはgenomic DNAの1つの時点のためである。- 7本のチューブをセットアップし、説明されているようにラベルを付けます:ネガティブコントロール、ポジティブコントロール、ゲノムDNA用のチューブ4本、マスターミックス用のチューブ1本(図3A)。ネガティブコントロールは蒸留水です。ポジティブコントロールは、レンチウイルスを産生するための293Tトランスフェクションで使用されたプラスミドDNAです。
- 表2に示すように、最初に各チューブに水を追加します。
- 表3に示すようにマスターミックス(MM)を調製します。
- 水を入れた各チューブに18μLのMMを加えます。次に、25 μLのゲノムDNAを4本のテンプレートチューブのそれぞれに加えます。次に、ポジティブコントロールチューブに1 μL(10 ng/μL)を加え、他のチューブをポジティブコントロールDNAで汚染しないように注意します。最後に、各チューブに2 μLのポリメラーゼを添加し、最初に4本のサンプルチューブに、最後にポジティブコントロールチューブに添加します。PCRチューブを混合し、スピンダウンします(図3)。
- 表4に記載の条件でPCR反応を行う。
- 最初のPCRが行われている間に、表5に示すように、2回目のPCR用のチューブを準備します。ネガティブコントロール、ポジティブコントロール、ゲノムDNA用チューブ4本、MM用チューブ1本の計7本をセットアップします(図3B)。
- 水を含む各チューブに22 μLのMMを加え、PCRの最初のラウンドが終了するのを待ってから、DNAとポリメラーゼを添加します。
- 最初のPCRが完了したら、4本のサンプルチューブを1本の微量遠心チューブに混ぜ合わせます。
- 水を入れた各サンプルチューブに25 μLを加えます。
- 次に、1 μLの1回目のPCRネガティブコントロールと2 μLの1回目のPCRポジティブコントロールを、適切にラベル付けしたチューブに加えます。
- 各チューブに2 μLのポリメラーゼを加え、最初に4本のサンプルチューブに添加し、最後にネガティブコントロールを加えます。
- 表6に示すPCR反応を行う。
注:3.5%アガロースゲルの電気泳動による最初のPCR反応の結果を分析した際、豊富な生成物が得られた場合、この手順の次の繰り返しのためにサイクル数を9〜10に減らすことができます。同様に、製品の歩留まりが低い場合は、サイクルを14に増やすことができます。 - 2回目のPCR反応が完了したら、4つのサンプルを1本の微量遠心チューブに混ぜ合わせ、さらに80 μLの6倍ローディング色素を入れます。ポジティブコントロールとネガティブコントロールには、20 μLのサンプルを使用します。
- Tris-Borate-EDTA(TBE)バッファーで3.5%アガロースゲルを調製します(図4A)。大量のサンプルに対応するために、複数のウェルをテープでつなぎ合わせてゲルコームに 1 つの大きなウェルを作成し(容量に合うコームが利用できない場合)、ポジティブコントロールとネガティブコントロール用に 2 つのウェルを使用可能にしておきます。ゲルを90Vで1時間泳動します。
- ゲルを可視化し、約 250 塩基対 (bp) のバンドを確認します。
- 最初の精製:ゲル抽出
- 清潔なメスまたはカミソリの刃を使用して、250 bpバンドを切除し、できるだけ多くのゲルを切り取ります。大きなバンドを4つに分割し、各ピースを清潔な微量遠心チューブに移します。各ゲルスライスの重量を測定して記録します。
注:各ピースは、チューブあたり約200 mg以下である必要があります。 - 6 容量の可溶化バッファーを添加します (例: ゲル片の重量が 200 mg の場合は、1.2 mL のバッファーを添加します)。チューブを50°Cのウォーターバスに入れ、ゲルスライスが溶解するまで10〜15分ごとに回転させます。次に、各チューブに1容量のイソプロパノールを加えます(例:ゲル片の重量が200 mgの場合は0.2 mLのイソプロパノール)。
- 精製に 2 つのスピンカラムを使用して、4 本のチューブすべてからサンプルをカラムにロードします。カラムの保持量がわずか750 μLであるため、複数のスピンを実行してすべてのサンプル量を処理します。 従来の卓上型微量遠心分離機でカラムを 17,900 × g で 1 分間スピンし、その都度フロースルーを廃棄します。
- カラムを 750 μL の洗浄バッファーで洗浄し、17,900 × g で 1 分間遠心分離します。洗浄後、フロースルーを廃棄し、17,900 × g で 3 分間遠心分離してスピンカラムを乾燥させます。1カラムあたり50 μLの蒸留水でDNAを溶出し、前と同様に1分間遠心分離し、両方のチューブを組み合わせて合計100 μLのサンプルにします。
- 清潔なメスまたはカミソリの刃を使用して、250 bpバンドを切除し、できるだけ多くのゲルを切り取ります。大きなバンドを4つに分割し、各ピースを清潔な微量遠心チューブに移します。各ゲルスライスの重量を測定して記録します。
- 2回目の精製
- この次の精製ステップでは、結合バッファー 2 (PCR 精製キットに付属) を使用しますが、これは小さなフラグメントを除去しません。100 μL の DNA に 400 μL のバッファーを添加し、1 つのスピンカラムにロードします(図 4B)。サンプルを17,900 × g で1分間遠心分離します。
- 次に、メンブレンを650μLの洗浄バッファーで洗浄し、先ほど行ったようにスピンします。上記のように遠心分離を追加してスピンカラムを3分間乾燥させます。
- 30 μLの蒸留水でDNAを溶出し、最高速度で1分間スピンします。溶出後、分光光度計で濃度を確認します。濃度が10 ng / μL以上または70〜80 ng / μLを超えないようにしてください。 DNAを-20°Cの冷凍庫で保存します。
- 増幅バーコードのシーケンシング
- シーケンシングでは、溶出バッファー(EB)を使用して精製バーコードを 0.75 ng/μL に希釈します。配列の多様性を高めるために、30%(v/v)の PhiX を含むアンプリコンを 17 pM でクラスター化します。製造元のプロトコルに従って、自動クラスター生成システムでシングルエンド (SE) クラスタリングを実行します。
- シーケンシングコアで、NextGenシーケンサーで合計36サイクルのシングルエンドシーケンシングを実行します(図4C)。カスタムプライマー GexSeqS を Illumina シーケンシングプライマーに 0.5 μM で添加します。
- 参照した解析ソフトウェアを使用してFastqファイルを生成し、リード長を18ヌクレオチドにトリミングするソフトウェアを使用して処理します。
- バーコードアナライザーとデコンボリューションソフトウェアを使用して、トリミングされた読み取りをデコンボリューションします。各shRNAのフォールド枯渇スコアを、基準時点(T1)と最終時点(T2)におけるリードカウントの比として計算します。
- shRNAスクリーニング結果の解析と治療標的候補の同定
注:Cellecta shRNAライブラリーでは、ほとんどの遺伝子が5種類(67%)または6種類(32%)のshRNAを標的としています。しかし、ほとんどの細胞でヒットするはずのいくつかのハウスキーピング遺伝子は、多数のshRNAの標的となり、陰性のスコアの範囲を定義するコントロールとして機能します。- より多くのshRNAが標的とする遺伝子への偏りを防ぐために、枯渇スコアを対数変換します。経験分布から各遺伝子の 80 パーセンタイルを計算することにより、分位数推定を実行します。
- 対数フォールド枯渇スコアの帰無分布を生成するには、遺伝子の>95%が枯渇せず、それらの対数分位スコアが正規分布を持つと仮定します。経験分布の中央値を使用して、帰無分布の平均を推定します。この帰無分布を使用して、帰無分布の95パーセンタイルよりも大きい対数分割枯渇スコアを持つすべての遺伝子を「ヒット」として分類します。
注:すべてのヒットには、枯渇スコアがカットポイントを超えるshRNAが少なくとも2つ必要です(図5)。 - RNAiドロップアウトスクリーニングデータの品質とカットポイントの妥当性を評価するには、COLT Cancer RNAiスクリーニングイニシアチブ24,25によって「コア必須」と定義された一連の遺伝子を使用して比較します。ヒットのリストから CEG を削除して、潜在的な治療標的の初期リストを作成します。
注:COLTセットの遺伝子は、COLTによってスクリーニングされた72のがん細胞株の>50%でヒットとしてスコアリングされました。コア必須遺伝子リストには、640の遺伝子が含まれています。 - 複数のMPNST細胞株に共通または一様に必要とされる潜在的な治療標的を特定するには、異なるMPNST細胞株または初期継代培養のスクリーニングで同定された非CEGヒットのベン図を作成します(図6A)。MPNST 回線またはカルチャのすべてまたは大部分で必要な非 CEG ヒットを優先します。
- あるいは、各細胞株または早期継代培養の非CEGヒットリストでパスウェイ解析を行い、腫瘍細胞の増殖および/または生存に必要なシグナル伝達経路の構成要素をコードする遺伝子を同定します。次に、非CEGの経路解析で同定されたシグナル伝達経路を、WESで同定された変異の影響を受けていると同定されたシグナル伝達経路と比較します。
注:WESで同定された変異によって一貫して影響を受けるシグナル伝達経路を同定し、shRNAスクリーニングで重要と同定されたシグナル伝達経路と比較することは特に有用であると考えています。
- あるいは、各細胞株または早期継代培養の非CEGヒットリストでパスウェイ解析を行い、腫瘍細胞の増殖および/または生存に必要なシグナル伝達経路の構成要素をコードする遺伝子を同定します。次に、非CEGの経路解析で同定されたシグナル伝達経路を、WESで同定された変異の影響を受けていると同定されたシグナル伝達経路と比較します。
- 治療薬がすでに利用可能な創薬可能な標的を特定するには、Drug Gene Interaction Database(dgidb.org)を使用して、コア必須遺伝子の除去後に残ったヒットのリストをスクリーニングします。
注:このデータベースでは、遺伝子のリストを同時に入力してスクリーニングすることができ、これらの遺伝子を標的とするために現在利用可能な薬剤に関するガイダンスを提供します。 - まず、最初のライブラリスクリーニングで使用したものとは異なるshRNAで遺伝子発現をノックダウンすることにより、同定された創薬可能な高目的ターゲットをシングルバッチフォーマット(前述のライブラリバッチフォーマットではありません)でバリデーションします。遺伝子発現をノックダウンするには、2〜3種類の異なるレンチウイルスshRNAを使用します。感染後3〜4日で、以下に説明するように、これが腫瘍細胞の増殖と生存に及ぼす影響を決定します。
3. 治療薬候補を試用したMPNST細胞の細胞数と生存率のサイトメーターアッセイの実施
- DMEMでMPNST細胞を80%のコンフルエントに増殖させます。細胞を室温のハンクス平衡塩溶液(HBSS)で洗い流します。
注:これらの細胞の増殖は再プレーティング後しばらく遅れるため、細胞をコンフルエントに増殖させないでください。 - 非酵素的細胞解離溶液で30秒〜1分間細胞を覆って、細胞を基質から剥離します。解離溶液1 mLあたり5 mLのDMEMを添加し、細胞を静かに上下にピペットして基質から剥離します。
- 血球計算盤を使用して細胞をカウントします。ウェルあたり 1,200 細胞の密度で細胞を黒壁の 96 ウェルプレートに播種します。試験する薬物希釈液ごとに少なくとも3つのウェルを播種し、これらの実験の生物学的複製を3回実施します。
- 試験される薬剤の初期濃度を決定するには、文献をレビューして、他の種類のがん細胞に対して有効な濃度を評価します。最初の実験では、他の種類のがんで使用される薬物濃度よりも2桁上、2桁下の範囲でテストします。
- 試験する薬物の希釈液を調製し、各希釈液またはビヒクルを少なくとも3つの複製ウェルに添加します。
- 薬物添加後1、3、5、および7日で直接細胞数を評価します。Hoechst 33342を最終濃度5 μg/mLになるように添加し、プレートを37°Cで30分間インキュベートします。 100,000 msの露光時間で、総細胞数に対する直接細胞数オプションを使用して、ハイスループットイメージングサイトメーターでプレートを読み取ります。
- ソフトウェアを使用して読み取り値を解析し、スプレッドシートにエクスポートし、統計分析に適切なソフトウェアを使用します。
- 薬物処理ウェルで細胞数の統計的に有意な減少が観察された場合は、「生死」アッセイを実施して、この減少が細胞死の誘導に部分的に起因しているかどうかを判断します。
- ステップ3.3で説明した黒壁の96ウェルプレートに細胞をプレートします。
- ステップ3.5の説明に従って薬を準備して追加します。
- 薬物添加後1、3、5、および7日後の細胞生存率と死を評価します。各ウェルにカルセインAMを最終濃度1 μMに添加し、ヨウ化プロピジウムを最終濃度1 μMに添加します。細胞を37°Cで15分間インキュベートします。
- Live+Deadソフトウェアオプションを使用したイメージングサイトメーターで細胞をイメージングします。ソフトウェアを使用して読み取り値を解析し、スプレッドシートにエクスポートし、統計分析に適切なソフトウェアを使用します。
結果
図5のプロットは、スクリーニングした各ヒト細胞株において、TRUEとラベル付けされたコア必須遺伝子(CEG)の枯渇スコアを、非CEG(FALSEとラベル付け)と比較したものです。ポイントは、個々の遺伝子の倍数枯渇スコアのlog2を表し、全体的なスコア分布の箱ひげ図表現にプロットされます。スチューデントの t検定は、各細胞株の2つのグループ間の枯渇スコアの平均の有意差を検定するために使用されました。結果の p値は、各パネルに示されます。平均フォールド枯渇スコアは、CEGの方が非CEGよりも有意に高いことに注意してください。これは、コア必須遺伝子が、定義上、ほとんどの細胞タイプで増殖および/または生存に一貫して必要であるため、予想されます。
図6Aは、3つのヒトMPNST細胞株の「ヒット」のベン図を示しています。通常、多数の遺伝子が複数の系統間で共有されていることがわかります。これらのヒットは、MPNSTの大きなサブセットの増殖および/または生存に不可欠である可能性が高いタンパク質をコードする遺伝子を表すため、優先度が高い。 また、1つの細胞株のみでヒットする遺伝子が多数あることにも注意してください。これは一般的に発生し、画面の品質が低いことを示していると見なすべきではありません。次に、複数の系統間で共通してヒットする遺伝子をDrug Gene Interaction Databaseを用いて評価し、既存の薬剤で創薬可能なタンパク質をコードする遺伝子をこのサブセット内で同定します。次に、これらの中からいくつかを選択し、対応するmRNAの発現をshRNAでノックダウンすることにより、初期バリデーションを行います。一部のshRNAはオフターゲット効果を持つため、常に同じ転写産物を標的とする複数のshRNAを試験します。図6Bは、ノンターゲティングコントロールとBCL6を標的とする複数のshRNAを用いてMPNST細胞を形質導入した代表的な結果を示しています。次に、形質導入後のさまざまな時期に細胞数を決定しました。BCL6 shRNAのいくつかは、細胞数を著しく減少させました。付随するイムノブロットに示されているように、細胞数の減少の程度はBCL6ノックダウンの程度と相関しています。図6Cは、早期継代P0−GGFβ3 MPNST培養の代表的な増殖曲線を示す。

図1:MPNST組織または早期継代MPNST細胞の全エクソームシーケンシングを実施するためのワークフロー。概略図は、腫瘍由来の早期継代培養物に存在するバリアント検出の一般的なワークフローを示しています。初期の継代培養から DNA を単離し(1)、登録プロトコル(2)に従って高品質の DNA をシーケンシングコアに提出します。シーケンシングコアは、提出されたDNAの品質をチェックし、必要なすべてのサンプルおよびゲノムライブラリの準備を行います。コアファシリティは、品質管理指標を含むFASTQシーケンシングファイルをユーザーに提供します(3)。ユーザーは、FASTQファイルを任意のゲノムアライメントおよびバリアント呼び出し元プログラムにアップロードします。(4) 注釈付きバリアントは、ユーザー定義の基準でフィルタリングされ、関連性のないバリアントが削除されます。示されている代表的なデータは、切除されたヒトMPNST腫瘍サンプルと腫瘍由来の細胞株を比較したものである26。(5) PANTHERによる機能分類解析を行う。略語: MPNST = 悪性末梢神経鞘腫瘍。この図の拡大版をご覧になるには、ここをクリックしてください。
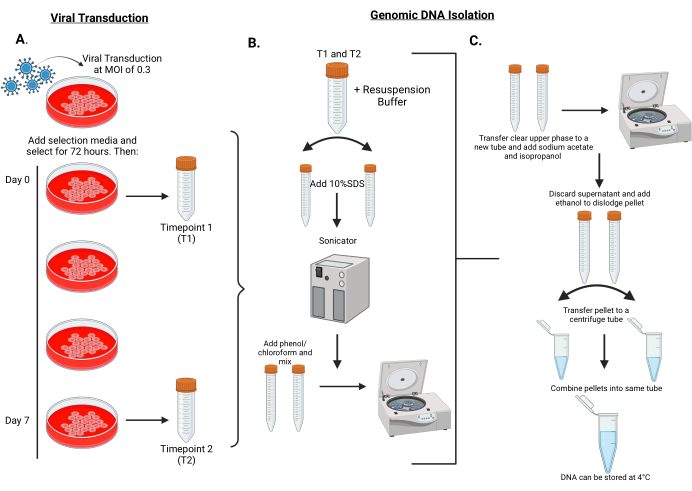
図2:shRNAライブラリーのMPNST細胞へのウイルス形質導入を行い、時点1および時点2の細胞からゲノムDNAを単離するためのワークフロー。 (A)標的細胞を0.3の低MOIでバーコードレンチウイルス粒子に感染させ、72時間選択します。細胞は5〜7集団倍増(約7日)継代されます。0日目と7日目の細胞ペレットは、ゲノムDNA単離のために-80°Cで保存されます。0 日目は時点 1 (T1) と呼ばれ、7 日目は時点 2 (T2) と呼ばれます。(B)ゲノムDNAの単離は、細胞ペレットを再懸濁バッファーに再懸濁することから始まり、その後、2本の15 mLチューブに分割されます。細胞溶解を促進するために、10%SDSを各チューブに添加し、30秒オン/30秒オフの25サイクルで超音波処理します。超音波処理後、フェノール/クロロホルムを各チューブに添加し、45〜60秒間激しくボルテックスする。その後、チューブを遠心分離します。(C)透明な上相をピペッティングし、酢酸ナトリウム/イソプロパノールを添加したクリーンチューブに添加し、よく混合します。チューブは再び遠心分離されます。このとき、上澄み液を捨て、70%エタノールを加えてペレットを除去する。再懸濁したペレットを1本のチューブに混ぜ合わせ、卓上遠心分離機で最高速度で回転させます。上清を捨て、ペレットを蒸留水に再懸濁します。この図の拡大版をご覧になるには、ここをクリックしてください。
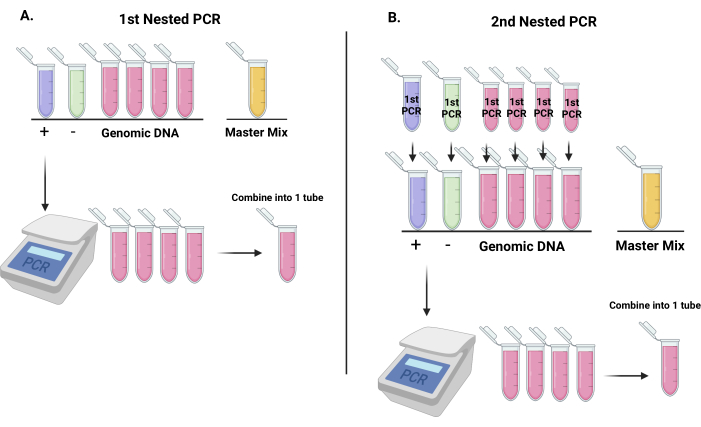
図3:次世代シーケンシングによるバーコードの定量化に備え、レンチウイルスshRNAベクターからバーコード配列を増幅するワークフロー 。 (A)最初のネストPCR反応用のチューブのセットアップ方法の表現(7本のチューブ:1本はネガティブコントロール用、もう1本はポジティブコントロール用、残りの4本はゲノムDNA用。最後のチューブはマスターミックスチューブとして機能します)。最初のPCR反応に続いて、ゲノムDNAチューブを1本のチューブに結合し、混合します。(B)最初のネストされたPCR反応からの生成物は、2番目のネストされたPCR反応のテンプレートとして機能します。2回目のPCRに続いて、ゲノムDNAチューブを1本のチューブに結合し、混合します。 この図の拡大版をご覧になるには、ここをクリックしてください。
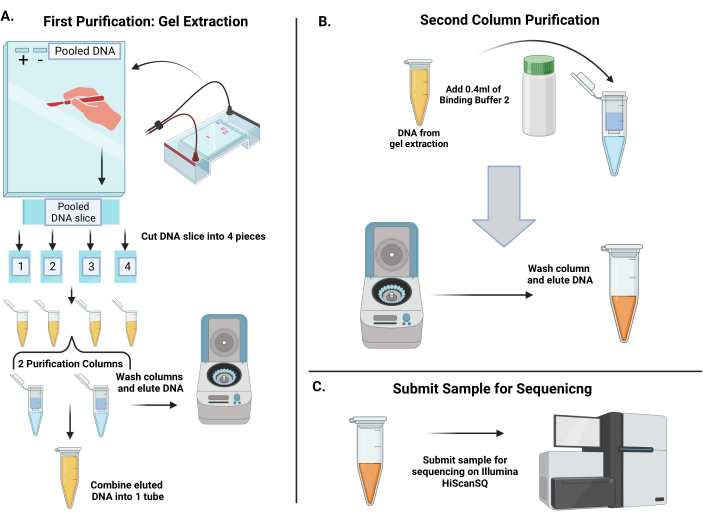
図4:増幅したshRNAバーコードを精製し、バーコードをシーケンシングして、時点1と時点2での発現を定量化するワークフロー 。 (A)3.5%アガロースゲルを流し込みます。プールされたDNAの量は1つのウェルの制限を超えるため、ゲルコームの4〜6本の歯をテープでつなぎ合わせて、1つの大きなウェルを作成します。6倍のローディング色素でPCR産物を調製し、ポジティブコントロール、ネガティブコントロール、およびプールされたDNAをゲルにロードします。電気泳動後、約250塩基対の大きなバンドがプールされたDNAレーンに現れます。清潔なメスを使用して、バンド全体を切除し、4つのゲルスライスにカットします。ゲル片を可溶化し、2つのスピンカラムに結合してDNAを溶出します。溶出したDNAを1本のチューブに結合します。(B)DNAは、第2の精製ステップを経て精製される。結合バッファー 2 をプールした DNA のチューブに添加し、スピンカラムにピペットで移します。メンブレンを洗浄し、蒸留水でDNAを溶出します。(C)精製したDNAをシーケンシングコアに提出する。 この図の拡大版をご覧になるには、ここをクリックしてください。
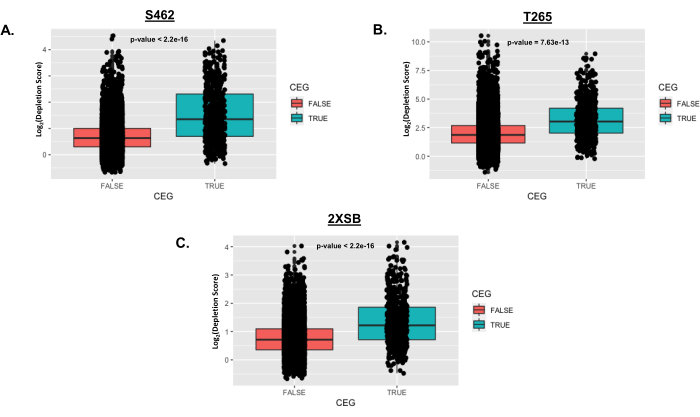
図5:プロトコルに記載されている解析後のCore Essential Genesの分布の代表例。 この例では、3つのヒトMPNST細胞株(S462、T265、および2XSB)細胞をCellecta DECIPHER shRNAライブラリーでスクリーニングしました。各ヒトMPNST細胞株について、Core Essential Genes(CEG;真の箱ひげ図)25 をCEGのリストにない遺伝子の箱ひげ図(偽箱ひげ図)と比較します。個々のデータポイントは、各箱ひげ図の上に重ねられます。p値は、CEGと非CEGの遺伝子レベルの枯渇スコアを比較する標準的な t検定からのものです。略語:CEG =コア必須遺伝子;MPNST = 悪性末梢神経鞘腫瘍。 この図の拡大版をご覧になるには、ここをクリックしてください。
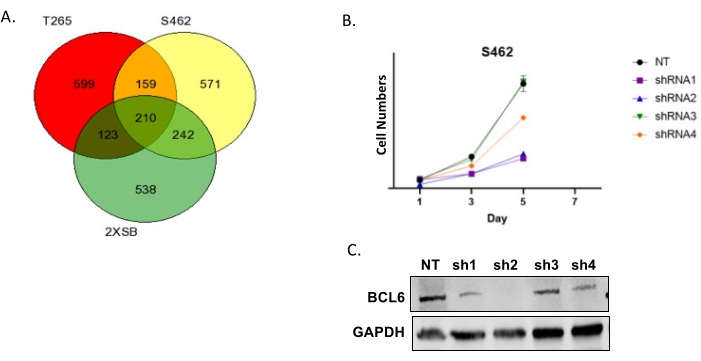
図6:スクリーニング結果の検証 。 (A)3つのヒトMPNST細胞株におけるオーバーラップヒットの代表的なベン図。(B)4つの異なる BCL6 shRNA(shRNA1、shRNA2、shRNA3、shRNA4)を発現するノンターゲティング(NT)レンチウイルスベクターとレンチウイルスを用いて形質導入したS462ヒトMPNST細胞。細胞をレンチウイルスで形質導入し、次いで選抜剤(ピューロマイシン)で3日間処理した。その後、細胞数を7日間にわたって評価した。(C)NT、shRNA1、shRNA2、shRNA3、shRNA4レンチウイルスによる形質導入後のBCL6のタンパク質レベルを示すウェスタンブロット解析。 この図の拡大版をご覧になるには、ここをクリックしてください。
表1:レンチウイルス力価測定のプレートレイアウト。この表をダウンロードするには、ここをクリックしてください。
表2:最初のPCR反応の初期設定。この表をダウンロードするには、ここをクリックしてください。
表3:最初のPCR反応のためのマスターミックスの調製。この表をダウンロードするには、ここをクリックしてください。
表4:Cellectaの最初のPCRパラメータ。この表をダウンロードするには、ここをクリックしてください。
表5:2回目のPCR反応の初期設定。この表をダウンロードするには、ここをクリックしてください。
表6:CellectaのセカンドPCRパラメータ。この表をダウンロードするには、ここをクリックしてください。
ディスカッション
ここで紹介する詳細な方法は、末梢神経系腫瘍とMPNSTの病因を研究するために開発されました。これらの方法は効果的であることがわかりましたが、ここで説明する方法にはいくつかの潜在的な制限があることを認識する必要があります。以下では、これらの制限のいくつかと、他のモデルシステムでそれらを克服するための潜在的な戦略について説明します。
我々は、全エクソームシーケンシングがP 0-GGFβ3マウスの目的の変異を効果的に同定することを見出しました。ただし、全エクソームシーケンシング自体には限界があることを認識する必要があります。第一に、全エクソームシーケンシングは、融合遺伝子産物を同定するための効果的なアプローチではありません。これは、染色体切断とその後の融合の大部分が、ゲノムの大部分を占める遺伝子間領域とイントロンが主に関与しているためです。その代わりに、ペアエンドリードのRNA-Seqの方が、融合遺伝子をより効果的に同定できることを発見しました。また、全エクソームシーケンシングが染色体喪失の比較的大きな領域をどの程度効果的に同定するかという問題もあります。
このような損失を特定するためにいくつかのアルゴリズムが開発されているが、「全エクソームシーケンシング」という用語自体が誤解を招く原因となるのは、エクソームの捕捉が良好なランであっても、エクソン領域の最大5〜10%を見逃すことが多いからである。このため、私たちは日常的に全エクソームシーケンシングをアレイ比較ゲノムハイブリダイゼーション(aCGH)などの他のアプローチで補完しています。利益と損失を特定した後、これらの間隔内の遺伝子を調べ、それらをヒトの対応する遺伝子に関連する既知のドライバー変異と比較します。しかし、マウスのゲノムはヒトゲノムよりも安定している27。その結果、マウス腫瘍は通常、ヒト新生物にみられるものに類似した染色体を示さない。マウス腫瘍のパターンは、その代わりはるかに単純であり、染色体全体または染色体の増減に向かう傾向があり、強い選択圧下で発生する傾向がある限局性欠失が比較的少ない22,23。
ゲノムスケールのshRNAスクリーニングを行う際に遭遇した潜在的な落とし穴がいくつかあります。私たちが遭遇する最も一般的な問題の1つは、レンチウイルスベクターの標的細胞への形質導入が比較的不十分であることです。ほとんどの場合、問題はパッケージ化されたレンチウイルスプールの不適切な力価から発生したことがわかります。早期継代マウス腫瘍細胞培養は限られたリソースであるため、多くの研究者は代わりに、より容易に入手できる別の確立された細胞株を使用してレンチウイルスの力価を測ろうとします。しかし、このアプローチの問題点は、レンチウイルス形質導入の効率が細胞の種類によって大きく異なる可能性があることです。このため、実験で使用する実際の細胞でレンチウイルスに力価を投与することをお勧めします。また、ウイルス力価が比較的低いという問題にも直面しています。この問題は、ほとんどの場合、パッケージ化されたウイルスを産生する際の293T細胞のトランスフェクションが不十分であることを反映しています。
ゲノムスケールのshRNAスクリーニングを行うと、偽陽性ヒットが得られる可能性があります。このため、最も関心のある創薬可能な標的を特定したら、常にshRNAスクリーニングの結果を検証しています。通常、関心の高いターゲットを検証するために、2つの異なるアプローチを使用します。まず、初期スクリーニングで用いたものとは異なる2種類以上のshRNAを用いて遺伝子発現をノックダウンし、これが腫瘍細胞の増殖と生存に及ぼす影響を明らかにします。次に、Drug Gene Interaction Databaseで同定された薬剤を入手し、これが腫瘍細胞の増殖と生存に及ぼす影響を明らかにします。この2つのアプローチを併用しているのは、shRNAが効き、薬剤が効かない状況に遭遇したからです。これらの事例の少なくとも一部では、エクソーム配列データセット全体の検査により、標的タンパク質は、薬物間タンパク質相互作用に影響を与える可能性のある変異を有する遺伝子によって産生されることが示されています。
上記で概説したアプローチは、研究者に、まれな新生物で発生する潜在的なドライバー変異を特定し、増殖と生存に必要なシグナル伝達経路を機能的に特定し、治療開発の標的に優先順位を付けるための適用可能な手段を提供します。他の研究者が、これらのアプローチが他のヒトがんの主要な治療標的を特定するのに役立つことを願っています。しかし、読者は、腫瘍の病因に関与する遺伝子や潜在的な治療標的をコードする遺伝子を特定するために使用できる他の機能ゲノムアプローチがあることに注意する必要があります。一例として、shRNAライブラリーについて説明したのと同様の方法で使用できるCRISPRライブラリーが利用可能です。機能スクリーニングは、腫瘍形成を促進する遺伝子を同定するためにin vivo で実施することもできる。この一例として、眠れる森の美女のトランスポゾンベースの体細胞突然変異誘発系は、以前にシュワン細胞とその前駆体を標的とするために使用され、その結果、MPNSTの病因に関与する数百の遺伝子が同定されました28。これらのシステムは機能ゲノミクスに異なる方法でアプローチするため、研究者は計画された実験の目標を慎重に検討し、それらの目標に基づいて機能ゲノミクス方法論を選択することをお勧めします。
開示事項
著者には開示すべき利益相反はありません。
謝辞
この研究は、国立神経疾患・脳卒中研究所(R01 NS048353およびR01 NS109655 to S.L.C.;R01 NS109655-03S1 から D.P.J.)、国立がん研究所 (R01 CA122804 から S.L.C.)、および国防総省 (X81XWH-09-1-0086 および W81XWH-12-1-0164 から S.L.C. へ)。
資料
| Name | Company | Catalog Number | Comments |
| Bioruptor Sonication System | Diagenode | UCD-600 | |
| CASAVA 1.8.2 | |||
| Cbot | Illumina, San Diego, CA | N/A | |
| Celigo Image Cytometer | Nexcelom | N/A | |
| Cellecta Barcode Analyzer and Deconvoluter software | |||
| Citrisolve Hybrid | Decon Laboratories | 5989-27-5 | |
| Corning 96-well Black Microplate | Millipore Sigma | CLS3603 | |
| Diagenode Bioruptor 15ml conical tubes | Diagenode | C30010009 | |
| dNTP mix | Clontech | 639210 | |
| Eosin Y | Thermo Scientific | 7111 | |
| Elution buffer | Qiagen | 19086 | |
| Ethanol (200 Proof) | Decon Laboratories | 2716 | |
| Excel | Microsoft | ||
| FWDGEX 5’-CAAGCAGAAGACGGCATACGAGA-3’ | |||
| FWDHTS 5’-TTCTCTGGCAAGCAAAAGACGGCATA-3’ | |||
| GexSeqS (5’ AGAGGTTCAGAGTTCTACAGTCCGAA-3’ | HPLC purified | ||
| GraphPad Prism | Dotmatics | ||
| Harris Hematoxylin | Fisherbrand | 245-677 | |
| Illumina HiScanSQ | Illumina, San Diego, CA | N/A | |
| Paraformaldehyde (4%) | Thermo Scientific | J19943-K2 | |
| PLUS Transfection Reagent | Thermo Scientific | 11514015 | |
| Polybrene Transfection Reagent | Millipore Sigma | TR1003G | |
| PureLink Quick PCR Purification Kit | Invitrogen | K310001 | |
| Qiagen Buffer P1 | Qiagen | 19051 | |
| Qiagen Gel Extraction Kit | Qiagen | 28704 | |
| RevGEX 5’-AATGATACGGCGACCACCGAGA-3’ | |||
| RevHTS1 5’-TAGCCAACGCATCGCACAAGCCA-3’ | |||
| Titanium Taq polymerase | Clontech | 639210 | |
| Trimmomatic software | www.usadellab.org |
参考文献
- Carroll, S. L. Molecular mechanisms promoting the pathogenesis of Schwann cell neoplasms. Acta Neuropathol. 123 (3), 321-348 (2012).
- Longo, J. F., Weber, S. M., Turner-Ivey, B. P., Carroll, S. L. Recent Advances in the Diagnosis and Pathogenesis of Neurofibromatosis Type 1 (NF1)-associated Peripheral Nervous System Neoplasms. Adv Anat Pathol. 25 (5), 353-368 (2018).
- Longo, J. F., Carroll, S. L. The RASopathies: Biology, genetics and therapeutic options. Adv Cancer Res. 153, 305-341 (2022).
- Birindelli, S., et al. Rb and TP53 pathway alterations in sporadic and NF1-related malignant peripheral nerve sheath tumors. Lab Invest. 81 (6), 833-844 (2001).
- Legius, E., et al. TP53 mutations are frequent in malignant NF1 tumors. Genes Chromosomes Cancer. 10 (4), 250-255 (1994).
- Menon, A. G., et al. Chromosome 17p deletions and p53 gene mutations associated with the formation of malignant neurofibrosarcomas in von Recklinghausen neurofibromatosis. Proc Natl Acad Sci U S A. 87 (14), 5435-5439 (1990).
- Upadhyaya, M., et al. Germline and somatic NF1 gene mutation spectrum in NF1-associated malignant peripheral nerve sheath tumors (MPNSTs). Hum Mutat. 29 (1), 74-82 (2008).
- Kourea, H. P., Orlow, I., Scheithauer, B. W., Cordon-Cardo, C., Woodruff, J. M. Deletions of the INK4A gene occur in malignant peripheral nerve sheath tumors but not in neurofibromas. Am J Pathol. 155 (6), 1855-1860 (1999).
- Nielsen, G. P., et al. Malignant transformation of neurofibromas in neurofibromatosis 1 is associated with CDKN2A/p16 inactivation. Am J Pathol. 155 (6), 1879-1884 (1999).
- Gregorian, C., et al. PTEN dosage is essential for neurofibroma development and malignant transformation. Proc Natl Acad Sci U S A. 106 (46), 19479-19484 (2009).
- Lee, W., et al. PRC2 is recurrently inactivated through EED or SUZ12 loss in malignant peripheral nerve sheath tumors. Nat Genet. 46 (11), 1227-1232 (2014).
- Zhang, M., et al. Somatic mutations of SUZ12 in malignant peripheral nerve sheath tumors. Nat Genet. 46 (11), 1170-1172 (2014).
- Varela, I., et al. Somatic structural rearrangements in genetically engineered mouse mammary tumors. Genome Biol. 11 (10), 100(2010).
- Johnson, R. A., et al. Cross-species genomics matches driver mutations and cell compartments to model ependymoma. Nature. 466 (7306), 632-636 (2010).
- Kim, M., et al. Comparative oncogenomics identifies NEDD9 as a melanoma metastasis gene. Cell. 125 (7), 1269-1281 (2006).
- Zender, L., et al. Identification and validation of oncogenes in liver cancer using an integrative oncogenomic approach. Cell. 125 (7), 1253-1267 (2006).
- Uren, A. G., et al. Large-scale mutagenesis in p19(ARF)- and p53-deficient mice identifies cancer genes and their collaborative networks. Cell. 133 (4), 727-741 (2008).
- Starr, T. K., et al. A transposon-based genetic screen in mice identifies genes altered in colorectal cancer. Science. 323 (5922), 1747-1750 (2009).
- Dupuy, A. J., et al. A modified sleeping beauty transposon system that can be used to model a wide variety of human cancers in mice. Cancer Res. 69 (20), 8150-8156 (2009).
- Carroll, S. L. The Challenge of Cancer Genomics in Rare Nervous System Neoplasms: Malignant Peripheral Nerve Sheath Tumors as a Paradigm for Cross-Species Comparative Oncogenomics. Am J Pathol. 186 (3), 464-477 (2016).
- Huijbregts, R. P., Roth, K. A., Schmidt, R. E., Carroll, S. L. Hypertrophic neuropathies and malignant peripheral nerve sheath tumors in transgenic mice overexpressing glial growth factor beta3 in myelinating Schwann cells. J Neurosci. 23 (19), 7269-7280 (2003).
- Kazmi, S. J., et al. Transgenic mice overexpressing neuregulin-1 model neurofibroma-malignant peripheral nerve sheath tumor progression and implicate specific chromosomal copy number variations in tumorigenesis. Am J Pathol. 182 (3), 646-667 (2013).
- Brosius, S. N., et al. Neuregulin-1 overexpression and Trp53 haploinsufficiency cooperatively promote de novo malignant peripheral nerve sheath tumor pathogenesis. Acta Neuropathol. 127 (4), 573-591 (2014).
- Hart, T., Brown, K. R., Sircoulomb, F., Rottapel, R., Moffat, J. Measuring error rates in genomic perturbation screens: gold standards for human functional genomics. Mol Syst Biol. 10 (7), 733(2014).
- Hart, T., et al. Evaluation and Design of Genome-Wide CRISPR/SpCas9 Knockout Screens. G3. 7 (8), Bethesda. 2719-2727 (2017).
- Longo, J. F., et al. Establishment and genomic characterization of a sporadic malignant peripheral nerve sheath tumor cell line. Sci Rep. 11 (1), 5690(2021).
- Maser, R. S., et al. Chromosomally unstable mouse tumours have genomic alterations similar to diverse human cancers. Nature. 447 (7147), 966-971 (2007).
- Rahrmann, E. P., et al. Forward genetic screen for malignant peripheral nerve sheath tumor formation identifies new genes and pathways driving tumorigenesis. Nat Genet. 45 (7), 756-766 (2013).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved
当社はcookieを使用しています。
「続行」をクリックすることで、当社のcookieへの同意となります。