Method Article
Profilazione genetica e screening dell'abbandono su scala genomica per identificare bersagli terapeutici in modelli murini di tumore maligno della guaina nervosa periferica
In questo articolo
Riepilogo
Abbiamo sviluppato un approccio oncogenomico comparativo cross-species utilizzando analisi genomiche e screening genomici funzionali per identificare e confrontare bersagli terapeutici nei tumori che insorgono in modelli murini geneticamente modificati e il corrispondente tipo di tumore umano.
Abstract
I tumori maligni della guaina dei nervi periferici (MPNST) derivano dalle cellule di Schwann o dai loro precursori. Nei pazienti con sindrome da suscettibilità tumorale neurofibromatosi di tipo 1 (NF1), le MPNST sono la neoplasia maligna più comune e la principale causa di morte. Questi sarcomi dei tessuti molli, rari e aggressivi, offrono un futuro difficile, con tassi di sopravvivenza libera da malattia a 5 anni del 34-60%. Le opzioni terapeutiche per gli individui affetti da MPNST sono deludentemente limitate, con la chirurgia deturpante che è l'opzione di trattamento principale. Molte terapie un tempo promettenti come il tipifarnib, un inibitore della segnalazione Ras, hanno fallito clinicamente. Allo stesso modo, anche gli studi clinici di fase II con erlotinib, che ha come bersaglio il fattore di crescita epidermico (EFGR), e sorafenib, che ha come bersaglio il recettore del fattore di crescita dell'endotelio vascolare (VEGF), il recettore del fattore di crescita derivato dalle piastrine (PDGF) e Raf, in combinazione con la chemioterapia standard, non sono riusciti a produrre una risposta nei pazienti.
Negli ultimi anni, i metodi di screening genomico funzionale combinati con la profilazione genetica delle linee cellulari tumorali si sono dimostrati utili per identificare le vie di segnalazione citoplasmatiche essenziali e lo sviluppo di terapie specifiche per il bersaglio. Nel caso di tipi di tumori rari, una variante di questo approccio, nota come oncogenomica comparativa cross-species, viene sempre più utilizzata per identificare nuovi bersagli terapeutici. Nell'oncogenomica comparativa interspecie, la profilazione genetica e la genomica funzionale vengono eseguite in modelli murini geneticamente modificati (GEM) e i risultati vengono quindi convalidati nei rari campioni umani e nelle linee cellulari disponibili.
Questo articolo descrive come identificare le mutazioni del gene driver candidato nelle cellule MPNST umane e murine utilizzando il sequenziamento dell'intero esoma (WES). Descriviamo quindi come eseguire screening shRNA su scala genomica per identificare e confrontare le vie di segnalazione critiche nelle cellule MPNST di topo e umane e identificare bersagli farmacologici in queste vie. Queste metodologie forniscono un approccio efficace per identificare nuovi bersagli terapeutici in una varietà di tipi di cancro umano.
Introduzione
I tumori maligni della guaina dei nervi periferici (MPNST) sono neoplasie a cellule fusiformi altamente aggressive che insorgono in associazione con la sindrome da suscettibilità tumorale neurofibromatosi di tipo 1 (NF1), sporadicamente nella popolazione generale e nei siti di precedente radioterapia 1,2,3. I pazienti affetti da NF1 nascono con una copia wild-type del gene oncosoppressore NF1 e un secondo allele NF1 con una mutazione con perdita di funzione. Questo stato di aploinsufficienza rende i pazienti affetti da NF1 suscettibili a una seconda mutazione con perdita di funzione nel loro gene NF1 wild-type, che innesca la tumorigenesi. Quando questa mutazione NF1 "di secondo colpo" si verifica in una cellula della linea cellulare di Schwann, il tumore risultante è un neurofibroma dermico che insorge nella pelle o un neurofibroma plessiforme che si sviluppa in grandi nervi o plessi nervosi. Sebbene la patologia dei neurofibromi dermici e plessiformi sia identica, il loro comportamento biologico è molto diverso: sebbene sia i neurofibromi dermici che quelli plessiformi siano benigni, solo i neurofibromi plessiformi possono subire una trasformazione e dare origine a MPNST. Oltre alla perdita di neurofibromina, la proteina attivante la Ras GTPasi codificata dal gene NF1, le MPNST sono portatrici di mutazioni di molti altri geni oncosoppressori, tra cui TP53 4,5,6,7, CDKN2A 8,9 e PTEN 10, mutazioni di geni che codificano componenti del complesso repressivo polycomb 2 11,12 (PRC2 ; geni SUZ12 e EED) e l'espressione aberrante dei recettori tirosin-chinasici 1,2. Mutazioni di NF1 e degli altri geni sopra menzionati sono presenti anche nelle MPNST sporadiche e indotte da radiazioni11,12.
Sebbene questi progressi nella nostra comprensione delle anomalie genomiche nelle MPNST siano stati preziosi per la comprensione della loro patogenesi, non hanno ancora portato allo sviluppo di nuove terapie efficaci per le MPNST. Uno dei principali ostacoli che ostacolano lo sviluppo di nuovi trattamenti è il fatto che gli MPNST sono tumori rari. Per questo motivo, è difficile ottenere il gran numero di campioni di pazienti necessari per le analisi globali che definiscono le mutazioni chiave come quelle intraprese dal Cancer Genome Atlas (TCGA). In base alla nostra esperienza, l'accumulo anche di un numero modesto di campioni umani di MPNST può richiedere anni. Per superare tali limitazioni, molti ricercatori che studiano altri tipi di cancro rari si sono rivolti all'uso dell'oncogenomica comparativa interspecie per identificare le mutazioni geniche driver essenziali, definire le vie di segnalazione citoplasmatiche essenziali nel loro tumore di interesse e identificare nuovi bersagli terapeutici. Poiché le vie di segnalazione essenziali per la tumorigenesi sono altamente conservate tra gli esseri umani e altre specie di vertebrati, l'applicazione di approcci di genomica funzionale come gli screening shRNA su scala genomica può essere un mezzo efficace per identificare queste nuove mutazioni driver, vie di segnalazione e bersagli terapeutici 13,14,15,16,17,18,19 , in particolare quando si studiano tipi di tumori umani rari che sono disponibili in numero limite20.
Nelle metodologie qui presentate, descriviamo questo approccio per eseguire la profilazione genomica in linee cellulari umane di MPNST e colture di MPNST di passaggio precoce derivate da topi P 0-GGFβ3, un modello murino geneticamente modificato (GEM) in cui la sovraespressione specifica delle cellule di Schwann del fattore di crescita neuregulina-1 (NRG1) promuove la patogenesi dei neurofibromi plessiformi e la loro successiva progressione verso MPNSTs21, 22,23. Il primo passo in questo approccio consiste nell'identificare i geni driver candidati nelle MPNST P 0-GGFβ3, nelle linee cellulari umane MPNST e nelle MPNST umane resecate chirurgicamente. Per convalidare funzionalmente le vie di segnalazione influenzate da queste mutazioni, utilizziamo quindi screening shRNA su scala genomica per identificare i geni necessari per la proliferazione e la sopravvivenza in linee cellulari MPNST umane e murine. Dopo aver identificato i geni necessari per la proliferazione e la sopravvivenza, identifichiamo i prodotti genici farmacologici all'interno della raccolta di "hit" utilizzando il Drug Gene Interaction Database. Confrontiamo anche gli "hit" nelle cellule MPNST umane e murine, per determinare se il modello GEM e le MPNST umane dimostrano una dipendenza simile dagli stessi geni e vie di segnalazione. L'identificazione delle sovrapposizioni nei geni necessari per la proliferazione e la sopravvivenza e le vie di segnalazione interessate serve come mezzo per convalidare il modello murino P 0-GGFβ3 a livello molecolare. Questo approccio sottolinea anche l'efficacia della combinazione di schermi umani e murini per identificare nuovi bersagli terapeutici, in cui il modello murino può fungere da complemento agli schermi umani. Il valore di questo approccio interspecie è particolarmente evidente quando si cercano bersagli terapeutici nei tumori rari, dove i tumori umani e le linee cellulari sono difficili da ottenere.
Protocollo
Prima dell'inizio degli studi, le procedure e i protocolli per la manipolazione dei vettori virali devono essere esaminati e approvati dal Comitato istituzionale per la cura e l'uso degli animali (IACUC) e dal Comitato istituzionale per la biosicurezza (IBC). Le procedure qui descritte sono state approvate dai consigli IACUC e IBC dell'Università di Medicina della Carolina del Sud e sono state eseguite da personale adeguatamente addestrato in conformità con la Guida NIH per la cura e l'uso degli animali da laboratorio e le linee guida istituzionali per la cura degli animali del MUSC.
1. Analisi WES-SEQ e identificazione di varianti patogene
- Isolare il DNA genomico da un campione tumorale o da cellule MPNST sub-confluenti (70%) coltivate su un piatto da 60 mm, utilizzando metodi standard disponibili in commercio a base di gel di silice (consultare il protocollo dei produttori per i passaggi dettagliati). Il flusso di lavoro generale è illustrato nella Figura 1.
- Inviare almeno 10 μL di DNA genomico a 50 ng/μL al nucleo di sequenziamento, a meno che non siano specificate quantità o concentrazioni diverse di DNA genomico.
- La struttura centrale frammenta il DNA genomico mediante sonicazione e quindi lo purifica utilizzando il metodo preferito. La cattura dell'esoma e la costruzione della libreria vengono eseguite utilizzando il kit di sequenziamento dell'esoma preferito e i tag indice vengono aggiunti all'esoma catturato amplificato.
- Inviare i campioni al nucleo di sequenziamento per eseguire il sequenziamento dell'intero esoma a due estremità (WES; sequenziato a 100 bp da ciascuna estremità).
- I file FASTQ generati dal core vengono forniti allo sperimentatore. Utilizzare solo file FASTQ che superano le metriche di qualità per l'analisi.
- Allineare e analizzare i file FASTQ utilizzando programmi software disponibili in commercio (ad esempio, DNAStar19,20, Partek21 o Varsome). Allineare i file FASTQ al genoma di riferimento del topo GRCm38/mm10 utilizzando le impostazioni predefinite.
NOTA: Utilizzando DNAStar e un campione MPNST di topo come esempio, il flusso di lavoro generale è schematizzato in Figura 1 e brevemente spiegato di seguito.- Apri il software DNAStar e scegli il flusso di lavoro SeqMan NGen.
- Scegliere Flusso di lavoro selezionando Variant Analysis/Resequencing (Analisi delle varianti/Risequenziamento ) e il tipo di analisi di sequenziamento : Amplicone basato su NGS, pannello genico o esoma. Fare clic su Next (Avanti).
- Scegli la sequenza di riferimento preferita selezionando Scarica pacchetto genoma e il genoma di riferimento appropriato (ad esempio Mus_musculus-GRCm38-dbSNP146.zip o Homo sapien-GRCh37.p13.zip). Se la struttura principale ha fornito un file di letto, caricare questo file ausiliario. Fare clic su Next (Avanti).
- Scegliere Sequenze di input selezionando la tecnologia di sequenziamento appropriata, Illumina, e indicare che le letture di sequenziamento sono accoppiate-end. Selezionare quindi l'impostazione dell'esperimento, il multicampione e caricare i file FASTQ di sequenziazione selezionando Aggiungi. Se si eseguono più campioni, designare ogni set di estremità accoppiate con un esperimento univoco o un nome di campione Tumore, Linea cellulare o A18, A202... Fare clic su Next (Avanti).
- Impostare il set di dati del controllo , se presente. Fate clic su Succ (Next ), quindi di nuovo su Succ (Next ) in Opzioni assieme (Assembly Options). In Opzioni di analisi, fare clic sulla modalità di rilevamento delle varianti appropriata come Diploide, quindi fare clic su Avanti. In Output assieme, assegnare un nome e designare il percorso di salvataggio dei file del progetto; fare clic su Avanti. Eseguire l'assembly nel computer locale o nel cloud.
NOTA: I file di allineamento risultanti possono essere aperti nel flusso di lavoro ArrayStar per l'annotazione delle varianti dei polimorfismi a singolo nucleotide (SNP) rilevati. Questo flusso di lavoro rileva gli SNP, non i guadagni o le perdite del numero di copie del gene. Un'analisi separata, chiamata array SNP ad alta densità, rileva le variazioni del numero di copie; Il sequenziamento dell'intero esoma non rileva in modo affidabile le variazioni del numero di copie. Consultare la guida per l'utente del programma per i passaggi specifici da eseguire con il programma di allineamento. - Applica filtri definiti dall'utente all'annotazione delle varianti degli SNP rilevati per includere o escludere determinati dati. Per ottenere un elenco condensato di varianti probabilmente rilevanti dal punto di vista funzionale, applicare i seguenti filtri ai set di dati delle varianti in questa gerarchia: non nel controllo (se è disponibile un campione di controllo normale), frequenza della popolazione (gnomAD, ExAC, frequenze di 1.000 genomi), frequenza allelica (includere ≤0,001 o 0,1%), profondità di copertura (escludere patogenicità o classe ClinVar (includere: patogeno, probabilmente patogeno; escludere: incerto, probabilmente benigno, benigno) e, se desiderato, il tipo di SNP e l'impatto della codifica (includere: non sinonimo, missenso, assurdità, frameshift, in-frame, splicing).
NOTA: Un elenco di geni tumorali rilevanti noti può anche essere applicato all'elenco finale per estrarre solo geni specifici rilevanti per la malattia, vedere il passaggio 1.3.7. Questi filtri possono ridurre l'elenco dei geni varianti a meno di 20 geni.- Utilizzare la frequenza allelica variante per filtrare gli SNP degli errori di sequenziamento. Le varianti omozigoti ed eterozigoti saranno rappresentate approssimativamente al 100% e al 50%, rispettivamente per una popolazione cellulare pura non contaminante (una linea cellulare derivata dal tumore dovrebbe rappresentare una singola popolazione di cellule tumorali isogeniche). In questo caso, applicare come filtro le frequenze alleliche del 100-90% e del 50-40% per le varianti, rimuovendo così tutte le varianti al di sotto di tale valore. Se per lo stesso set di dati sono disponibili anche dati di array SNP ad alta densità, applicare il guadagno o la perdita del numero di copie agli SNP con frequenze alleliche varianti inferiori ai rapporti omozigoti o eterozigoti (ad esempio, il guadagno del numero di copie risultante in 2 copie dell'allele A e 1 copia dell'allele B renderebbe appropriate le frequenze alleliche varianti del 75%, 25%, rispettivamente).
- Dopo aver identificato le varianti patogene con ArrayStar, esporta e salva l'elenco dei geni delle varianti come file csv, txt o xls e confronta i geni contenenti queste mutazioni con quelli della coorte di tumori generati da P 0-GGFβ3 per determinare elenchi di geni mutati sovrapposti e unici. Confrontare i geni mutati con i geni mutati noti associati alle loro controparti umane (ad esempio l'elenco dei geni del cancro del laboratorio Bushman o un elenco curato dall'utente).
- Facoltativo, prima di applicare qualsiasi filtro definito dall'utente (1.3.6), esportare e salvare il file annotato come file VCF.
NOTA: Questo file VCF può essere caricato in un altro software predittivo di varianti Varsome o VEP per il confronto come analisi secondaria. L'analisi biologica e di pathway può essere eseguita anche sulle liste di geni varianti filtrate.
- Facoltativo, prima di applicare qualsiasi filtro definito dall'utente (1.3.6), esportare e salvare il file annotato come file VCF.
- Esegui la classificazione funzionale dell'elenco dei geni varianti tramite un database preferito dall'utente per ottenere informazioni sulla classe delle proteine, sul percorso, sul componente del percorso, sulla famiglia genica e sull'ontologia.
NOTA: PANTHER (pantherdp.org) viene utilizzato come esempio qui.- Carica l'elenco dei geni filtrati con l'ID del gene.
- Selezionare l'organismo (Mus musculus).
- Selezionare l'analisi (classificazione funzionale visualizzata nell'elenco dei geni) e fare clic su Invia analisi biologica e del percorso negli elenchi di geni varianti filtrati.
2. Screening shRNA su scala genomica
NOTA: Sono disponibili diverse librerie shRNA e CRISPR che possono essere utilizzate per screening funzionali su scala genomica con colture tumorali a basso passaggio. Qui, descriviamo l'uso delle librerie shRNA CELLECTA DECIPHER come esempio. Le librerie lentivirali shRNA di CELLECTA DECIPHER sono ottimizzate per gli screening genetici RNAi in formato pooled. Ogni trascritto è bersagliato da almeno 5-6 shRNA unici e ogni vettore lentivirale di shRNA contiene un codice a barre genetico univoco affiancato da siti primer PCR. Queste librerie coprono la maggior parte dei geni rilevanti per le malattie umane e murine, ma non coprono tutti i geni del genoma. I pool di DNA plasmidico della libreria umana Cellecta sono disponibili in tre moduli (Human Module I, II, III; si rivolge a 15.377 geni) mentre i pool di plasmidi della libreria murina sono disponibili in due moduli (Mouse Modules I e II; target 9.145 geni). Queste librerie sono utilizzate per eseguire saggi di "drop-out" in cui i geni mirati necessari per la proliferazione e/o la sopravvivenza sono espressi in modo differenziato in diversi punti temporali dopo la trasduzione virale.
- Confezionamento lentivirale
- Giorno 0: Piastra 10 piastre da 10 milioni di cellule 293T/15 cm in DMEM da 30 ml/piatto privo di antibiotici contenente il 10% di siero fetale bovino (FBS).
- Giorno 1: Verificare che le cellule siano confluenti al ~80% il giorno successivo e pronte per la trasfezione. In una provetta conica da 50 mL, miscelare quanto segue in questo ordine: 600 μL di miscela plasmidica di confezionamento (0,5 μg/μL), 60 μL di libreria di codici a barre plasmidici, 12 mL di DMEM (senza siero o antibiotici) e 600 μL di reagente di trasfezione. Incubare a temperatura ambiente per 15 min.
- Mettere 900 μL di reagente di trasfezione e 12 mL di DMEM in una provetta conica separata da 15 mL e miscelare mediante vortex. Aggiungere 12,9 mL della miscela di reagente di trasfezione/DMEM alla miscela di DNA e mescolare per mescolare. Incubare a temperatura ambiente per 15 minuti senza mescolare ulteriormente. Aggiungere 2,5 mL di questa miscela, goccia a goccia, a ciascun piatto da 15 cm di cellule 293T e incubare per una notte in un incubatore per colture tissutali.
- Giorno 2: Sostituire i terreni il giorno seguente con terreni di coltura regolari contenenti antibiotici.
- Giorno 3: Raccogliere il virus raccogliendo il terreno e facendolo passare attraverso un'unità di filtrazione da 0,2 μm e aliquotandolo in provette coniche da 15 mL; preparare anche cinque aliquote da 1 mL di virus filtrato in crioviali da utilizzare nel titolo virale (considerato virus 48 h); conservare il virus in un congelatore a -80 °C. Sostituire il terreno con 30 ml di terreno di coltura sulle piastre uno alla volta per evitare che le cellule si secchino.
- Giorno 4: Raccogliere il virus raccogliendo il terreno e facendolo passare attraverso un'unità di filtrazione da 0,2 μm. Aliquote del virus in provette coniche da 15 ml. Questo è considerato virus 72 h; conservare il virus in un congelatore a -80 °C.
- Pool lentivirali titolati
- Aggiungere 65 μL di polimero cationico (10 mg/mL) a 65 mL di terreno di crescita delle cellule tumorali. Pipettare 1 mL/pozzetto del terreno contenente polimero in undici piastre di coltura tissutale a 6 pozzetti. Tripsinizzare le cellule tumorali di passaggio precoce e contare le cellule utilizzando il metodo preferito in modo che ogni pozzetto riceva 50.000 cellule/mL/pozzetto. Scongelare 1 mL di aliquote di lentivirus 48 h dal congelatore a bagnomaria a 37 °C.
- Per ogni modulo virale, preparare l'infezione come mostrato nella Tabella 1.
- Collocare tutte le piastre a 6 pozzetti in un incubatore per colture tissutali. Il giorno seguente, rimuovere i substrati virali e rifornirli con nuovi substrati di crescita. A 48 ore, aggiungere il terreno contenente puromicina a tutti i pozzetti che ricevono la selezione. Prima che le cellule di controllo siano confluenti, eseguire la conta cellulare dei cloni sopravvissuti utilizzando il metodo preferito.
NOTA: La concentrazione di puromicina necessaria per uccidere le cellule non trasdotte deve essere predeterminata empiricamente testando un intervallo di concentrazioni in una curva di "uccisione". Utilizzare la concentrazione più bassa che induce uniformemente la morte delle colture non trasdotte.
- Infezione lentivirale delle cellule bersaglio
- Tripsinizzare le cellule MPNST e contarle. Piastra 2,5 milioni di cellule per piatto da 15 cm per un totale di venti piatti da 15 cm per modulo. Scongelare il virus e trasdurre le cellule con il virus a un MOI di 0,5 in presenza di 5 μg/mL di polimero cationico (Figura 2A).
- Rimuovere il terreno contenente virus la mattina seguente e sostituirlo con un terreno di coltura fresco. Coltivare le cellule per altri 2 giorni per consentire l'espressione del marcatore di selezione e quindi aggiungere puromicina alle colture per eliminare le cellule non trasdotte.
- Tre giorni dopo l'aggiunta di puromicina, tripsinizzare le cellule e centrifugare metà della popolazione cellulare a 200 × g per 5 minuti in una centrifuga da tavolo. Conservare le cellule pellettate nel congelatore a -80 °C per la futura preparazione del DNA genomico; questo è il punto temporale di riferimento, o punto temporale 1 (T1).
- Riplaccare l'altra metà della popolazione cellulare e farla crescere per circa 7 raddoppi di popolazione prima di raccogliere e centrifugare come sopra. Questo pellet cellulare servirà come punto temporale finale (punto temporale 2, T2) e viene conservato a -80 °C per il futuro isolamento del DNA genomico (Figura 2).
- Isolare il DNA genomico da cellule trasdotte con pool virali
- Scongelare il pellet cellulare dal congelatore a -80 °C e risospendere il pellet in 10 mL di tampone di risospensione con l'aggiunta di RNAsi e dividere immediatamente in due provette di polimetilpentene da 15 mL (Figura 2B).
- Aggiungere 500 μL di SDS al 10% per 5 mL, mescolare e incubare a temperatura ambiente per 5 minuti. Successivamente, posizionare le provette in un dispositivo di taglio del DNA per sonicare il DNA per 25 cicli di 30 s acceso/30 s spento, assicurandosi di mantenere la temperatura a 4 °C.
- Aggiungere 5 mL di fenolo/cloroformio, pH 8,0 (conservato a 4 °C), avendo cura di mescolare bene il fenolo/cloroformio prima dell'uso. Dopo l'aggiunta di fenolo/cloroformio, mescolare bene agitando energicamente alla massima temperatura per 45-60 s. Centrifugare per 60 min, -20 °C a 7.200 × g.
NOTA: Un aspetto schiumoso/lattiginoso dopo il vortice segnalerà la completa risospensione. - Trasferire 3 mL della fase superiore trasparente in una provetta fresca da 15 mL e aggiungere 0,5 mL di acetato di sodio 3 M e 4 mL di isopropanolo e mescolare bene (Figura 2C). Centrifugare per 30 min a 20 °C a 7.200 × g.
- Dopo la centrifugazione, scartare il surnatante e quindi pipettare con cura il liquido rimanente. Aggiungere 0,5 mL di etanolo al 70% e rimuovere il pellet pipettando su e giù. Trasferire il pellet risospeso in una provetta da centrifuga da 1,5 mL e combinare entrambi i pellet dello stesso campione in un'unica provetta da centrifuga da 1,5 mL. Centrifugare alla massima velocità in una centrifuga da banco per 5 min.
- Scartare il surnatante e utilizzare una salvietta da laboratorio per assorbire l'etanolo residuo al 70%. Risospendere il pellet in 0,5 mL di acqua distillata, facendo attenzione a non lasciare che il pellet si asciughi in quanto ciò renderebbe difficile la risospensione. Conservare i campioni a 4 °C prima di misurare la concentrazione di DNA.
- PCR nidificata per amplificare i codici a barre shRNA
NOTA: Recuperare il DNA dalla conservazione e metterlo sul ghiaccio. Se la concentrazione di DNA è inferiore a 100 ng/μL, asciugare rapidamente il DNA sotto vuoto e risospenderlo in un volume appropriato di acqua distillata. Si consiglia di elaborare un solo modulo alla volta (punto temporale 1 (T1) o punto temporale 2 (T2)). Eseguire due preparazioni di ogni punto temporale che devono essere raggruppate alla fine; Pertanto, è importante non elaborare i punti temporali replicati nello stesso giorno. Il seguente protocollo è per un punto temporale del DNA genomico.- Impostare 7 provette ed etichettarle come descritto: controllo negativo, controllo positivo, 4 provette per il DNA genomico e una per una miscela master (Figura 3A). Il controllo negativo è l'acqua distillata; il controllo positivo è il DNA plasmidico utilizzato nella trasfezione 293T per generare lentivirus.
- Aggiungere prima l'acqua in ogni tubo come indicato nella Tabella 2.
- Preparare un Master Mix (MM) come mostrato nella Tabella 3.
- Aggiungere 18 μL di MM a ciascuna provetta contenente acqua. Successivamente, aggiungere 25 μL di DNA genomico a ciascuna delle 4 provette stampo. Quindi, aggiungere 1 μL di controllo positivo (10 ng/μL) alla provetta di controllo positivo, facendo attenzione a non contaminare altre provette con DNA di controllo positivo. Infine, aggiungere 2 μL di polimerasi a ciascuna provetta, aggiungendo prima alle 4 provette del campione e infine alla provetta di controllo positivo; mescolare e centrifugare le provette PCR (Figura 3).
- Eseguire la reazione PCR nelle condizioni indicate nella Tabella 4.
- Mentre la prima PCR è in esecuzione, preparare le provette per la2a PCR come indicato nella Tabella 5. Impostare sette provette: controllo negativo, controllo positivo, 4 provette per il DNA genomico e una per il MM (Figura 3B).
- Aggiungere 22 μL di MM a ciascuna provetta contenente acqua e attendere il completamento del primo ciclo di PCR prima di aggiungere il DNA e la polimerasi.
- Una volta completato il primo ciclo di PCR, unire 4 provette in una provetta per microcentrifuga e mescolare.
- Aggiungere 25 μL a ciascuna provetta contenente acqua.
- Successivamente, aggiungere 2 μL del primo controllo negativo della PCR e 2 μL del primo controllo positivo della PCR alle provette opportunamente etichettate.
- Aggiungere 2 μL di polimerasi a ciascuna provetta, aggiungendo prima alle 4 provette del campione e infine al controllo negativo.
- Eseguire la reazione PCR come indicato nella Tabella 6.
NOTA: Dopo aver analizzato i risultati della prima reazione di PCR mediante elettroforesi su un gel di agarosio al 3,5%, se si ottiene un'abbondanza di prodotto, il numero di cicli può essere ridotto a 9-10 per la successiva ripetizione di questa procedura; Allo stesso modo, se la resa del prodotto è bassa, i cicli possono essere aumentati a 14. - Al termine della seconda reazione PCR, combinare 4 campioni in una provetta per microcentrifuga più 80 μL di colorante di caricamento 6x. Per i controlli positivi e negativi, utilizzare 20 μL del campione.
- Preparare un gel di agarosio al 3,5% in tampone Tris-Borato-EDTA (TBE) (Figura 4A). Per accogliere l'ampio volume del campione, creare un pozzetto grande nel pettine gel fissando diversi pozzetti insieme (se non è disponibile un pettine adatto al volume), assicurandosi di lasciare disponibili due pozzetti per i controlli positivo e negativo. Far funzionare il gel a 90 V per 1 ora.
- Visualizzare il gel e confermare la presenza di una banda a circa 250 paia di basi (bp).
- Prima purificazione: estrazione del gel
- Usando un bisturi pulito o una lametta da barba, asportare la fascia da 250 bp, tagliando quanto più gel possibile. Sezionare la banda grande in 4 pezzi e trasferire ogni pezzo in una provetta per microcentrifuga pulita. Misura e registra il peso di ogni fetta di gel.
NOTA: Ogni pezzo deve essere di circa 200 mg o meno per provetta. - Aggiungere 6 volumi di tampone di solubilizzazione (ad esempio, se il pezzo di gel pesa 200 mg, aggiungere 1,2 mL di tampone). Mettere i tubi a bagnomaria a 50 °C e ruotare ogni 10-15 minuti fino a quando le fette di gel non si sono sciolte. Quindi, aggiungere 1 volume di isopropanolo a ciascuna provetta (ad esempio, 0,2 ml di isopropanolo se il pezzo di gel pesa 200 mg).
- Utilizzando due colonne rotanti per la purificazione, caricare i campioni di tutte e 4 le provette nelle colonne. Eseguire più rotazioni per elaborare tutto il volume del campione poiché le colonne contengono solo 750 μL. Ruotare le colonne a 17.900 × g per 1 minuto in una microcentrifuga da banco convenzionale ed eliminare il flusso ogni volta.
- Lavare le colonne con 750 μL di tampone di lavaggio e centrifugare a 17.900 × g per 1 min. Dopo il lavaggio, eliminare il flusso e asciugare la colonna centrifuga centrifugando a 17.900 × g per 3 min. Eluire il DNA con 50 μL di acqua distillata per colonna e centrifugare come in precedenza per 1 minuto e unire entrambe le provette per un volume totale di 100 μL del campione.
- Usando un bisturi pulito o una lametta da barba, asportare la fascia da 250 bp, tagliando quanto più gel possibile. Sezionare la banda grande in 4 pezzi e trasferire ogni pezzo in una provetta per microcentrifuga pulita. Misura e registra il peso di ogni fetta di gel.
- Seconda purificazione
- Questa fase successiva di purificazione utilizzerà il tampone legante 2 (dal kit di purificazione PCR), che non elimina i piccoli frammenti. Aggiungere 400 μL di tampone ai 100 μL di DNA e caricarlo su una colonna di spin (Figura 4B). Centrifugare il campione a 17.900 × g per 1 min.
- Successivamente, lavare la membrana con 650 μL di tampone di lavaggio e centrifugare come precedentemente eseguito. Asciugare la colonna di centrifugazione con centrifugazione aggiuntiva come sopra per 3 minuti.
- Eluire il DNA con 30 μL di acqua distillata e centrifugare alla massima velocità per 1 min. Dopo l'eluizione, controllare la concentrazione su uno spettrofotometro; assicurarsi che la concentrazione non sia inferiore a 10 ng/μL o superiore a 70-80 ng/μL. Conservare il DNA in un congelatore a -20 °C.
- Sequenziamento di codici a barre amplificati
- Per il sequenziamento, diluire i codici a barre purificati a 0,75 ng/μL utilizzando un tampone di eluizione (EB). Per aggiungere diversità di sequenza, gli ampliconi sono raggruppati a 17 pM, incluso il 30% (v/v) di PhiX. Eseguire il clustering single-end (SE) su un sistema di generazione cluster automatizzato in base al protocollo del produttore.
- Fare in modo che il core di sequenziamento esegua un totale di 36 cicli di sequenziamento single-end su un sequenziatore NextGen (Figura 4C). Aggiungere il primer personalizzato GexSeqS ai primer di sequenziamento Illumina a 0,5 μM.
- Utilizzare il software di analisi di riferimento per generare file Fastq ed elaborarli utilizzando il software per ridurre le lunghezze di lettura a 18 nucleotidi.
- Utilizza il software Barcode Analyzer e Deconvoluter per decontorcere le letture tagliate. Calcola i punteggi di deplezione della piega per ogni shRNA come rapporto tra i conteggi delle letture al punto temporale di riferimento (T1) rispetto al punto temporale finale (T2).
- Analisi dei risultati dello screening shRNA e identificazione di bersagli terapeutici candidati
NOTA: Nella libreria shRNA di Cellecta, la maggior parte dei geni è bersagliata da 5 (67%) o 6 diversi shRNA (32%). Tuttavia, diversi geni housekeeping, che dovrebbero essere colpiti nella maggior parte delle cellule, sono bersagliati da un gran numero di shRNA e fungono da controlli che definiscono l'intervallo di punteggi per i negativi.- Per prevenire pregiudizi verso i geni presi di mira da un numero maggiore di shRNA, i punteggi di deplezione sono stati trasformati logaritmicamente. Eseguire una stima quantilica calcolando l'80° percentile per ciascun gene dalla sua distribuzione empirica.
- Per generare una distribuzione nulla dei punteggi di deplezione della piega logaritmica, si supponga che il >95% dei geni non sarà esaurito e che i loro punteggi dei quantili logaritmici avranno una distribuzione normale. Usa la mediana della distribuzione empirica per stimare la media della distribuzione nulla. Utilizzando questa distribuzione nulla, classificare tutti i geni con punteggi di deplezione log-fold che sono più grandi del 95° percentile della distribuzione nulla come "hit".
NOTA: Tutti gli hit devono avere almeno due shRNA con punteggi di deplezione superiori al punto di taglio (Figura 5). - Per valutare la qualità dei dati dello screening dell'RNAi dropout e la validità del cut-point, utilizzare una serie di geni che sono stati definiti come "core essential" dall'iniziativa di screening RNAi COLT Cancer RNAi24,25 per il confronto. Rimuovere i CEG dall'elenco dei riscontri per produrre l'elenco iniziale dei potenziali bersagli terapeutici.
NOTA: I geni nel set COLT hanno ottenuto un punteggio positivo nel >50% delle 72 linee cellulari tumorali che sono state sottoposte a screening da COLT. L'elenco dei geni essenziali contiene 640 geni. - Per identificare potenziali bersagli terapeutici che sono comunemente o uniformemente richiesti da più linee cellulari MPNST, costruire diagrammi di Venn dei risultati non CEG identificati negli screening di diverse linee cellulari MPNST o colture di passaggio precoce (Figura 6A). Assegnare la priorità ai riscontri non CEG necessari in tutte o nella maggior parte delle linee o delle impostazioni cultura MPNST.
- In alternativa, eseguire analisi del percorso sulla lista di risultati non CEG da ciascuna linea cellulare o coltura di passaggio precoce per identificare i geni i cui prodotti codificano componenti delle vie di segnalazione necessarie per la proliferazione e/o la sopravvivenza delle cellule tumorali. Quindi, confrontare le vie di segnalazione identificate nell'analisi della via dei non-CEG con le vie di segnalazione che sono state identificate come influenzate da mutazioni identificate con WES.
NOTA: Troviamo particolarmente utile identificare le vie di segnalazione che sono costantemente influenzate dalle mutazioni identificate in WES e confrontarle con le vie di segnalazione identificate come critiche negli screening shRNA.
- In alternativa, eseguire analisi del percorso sulla lista di risultati non CEG da ciascuna linea cellulare o coltura di passaggio precoce per identificare i geni i cui prodotti codificano componenti delle vie di segnalazione necessarie per la proliferazione e/o la sopravvivenza delle cellule tumorali. Quindi, confrontare le vie di segnalazione identificate nell'analisi della via dei non-CEG con le vie di segnalazione che sono state identificate come influenzate da mutazioni identificate con WES.
- Per identificare i bersagli farmacologici per i quali sono già disponibili agenti terapeutici, esaminare l'elenco dei risultati rimanenti dopo la rimozione dei geni essenziali di base utilizzando il Drug Gene Interaction Database (dgidb.org).
NOTA: Questo database consente di inserire e sottoporre a screening un elenco di geni contemporaneamente e fornisce indicazioni sui farmaci attualmente disponibili per colpire questi geni. - Convalidare prima i bersagli ad alto interesse farmacologico identificati abbattendo la loro espressione genica con shRNA distinti da quelli utilizzati nella schermata iniziale della libreria in un singolo formato batch (non un formato libreria-batch appena descritto). Per abbattere l'espressione genica, utilizzare due o tre shRNA lentivirali distinti. Tre o quattro giorni dopo l'infezione, determinare l'effetto che questo ha sulla proliferazione e sulla sopravvivenza delle cellule tumorali come descritto di seguito.
3. Eseguire saggi citometrici del numero di cellule e della vitalità nelle cellule MPNST sfidate con agenti terapeutici candidati
- Far crescere le cellule MPNST in DMEM fino all'80% di confluenza. Risciacquare le celle con la soluzione salina bilanciata di Hanks (HBSS) a temperatura ambiente.
NOTA: Non far crescere le cellule fino alla confluenza poiché la crescita di queste cellule sarà ritardata per un po' di tempo dopo la riplaccatura. - Staccare le cellule dal substrato coprendo le cellule per 30 secondi a 1 minuto con una soluzione di dissociazione cellulare non enzimatica. Aggiungere 5 mL di DMEM per 1 mL di soluzione di dissociazione e pipettare delicatamente le cellule su e giù per staccarle dal substrato.
- Conta le cellule usando un emocitometro. Piastre con una densità di 1.200 cellule per pozzetto in piastre a 96 pozzetti con pareti nere; Piastra almeno tre pozzetti per ogni diluizione del farmaco che verrà testata ed eseguire tre repliche biologiche di questi esperimenti.
- Per determinare le concentrazioni iniziali del farmaco che verrà testato, esaminare la letteratura per valutare quali concentrazioni sono state efficaci contro altri tipi di cellule tumorali. Negli esperimenti iniziali, testare un intervallo di due ordini di grandezza al di sopra e due ordini di grandezza al di sotto della concentrazione del farmaco utilizzato con altri tipi di cancro.
- Preparare le diluizioni del farmaco da testare e aggiungere ogni diluizione o veicolo ad almeno tre pozzetti replicati.
- Valutare il numero di cellule dirette 1, 3, 5 e 7 giorni dopo l'aggiunta dei farmaci. Aggiungere Hoechst 33342 a una concentrazione finale di 5 μg/mL e incubare le piastre per 30 minuti a 37 °C. Lettura delle piastre su un citometro per imaging ad alto rendimento utilizzando l'opzione di conteggio diretto delle cellule per il numero totale di cellule con tempi di esposizione di 100.000 ms.
- Analizza le letture utilizzando il software ed esportale in un foglio di calcolo e utilizza un software appropriato per le analisi statistiche.
- Se si osservano riduzioni statisticamente significative del numero di cellule nei pozzetti trattati con il farmaco, eseguire un test "Vivo/Morto" per determinare se questa riduzione è dovuta, in parte, all'induzione della morte cellulare.
- Celle a piastre in piastre a 96 pozzetti con pareti nere, come descritto al punto 3.3.
- Preparare e aggiungere i farmaci come descritto al punto 3.5.
- Valutare la vitalità cellulare e la morte 1, 3, 5 e 7 giorni dopo l'aggiunta del farmaco. Aggiungere calceina AM a una concentrazione finale di 1 μM e ioduro di propidio a una concentrazione finale di 1 μM in ciascun pozzetto. Incubare le cellule per 15 minuti a 37 °C.
- Immagini di cellule su un citometro per imaging utilizzando l'opzione software Live+Dead . Analizza le letture utilizzando il software ed esportale in un foglio di calcolo e utilizza un software appropriato per le analisi statistiche.
Risultati
I grafici della Figura 5 mostrano i punteggi di deplezione dei geni essenziali fondamentali (GEM) etichettati come TRUE rispetto ai non-CEG (etichettati come FALSE) in ciascuna linea cellulare umana sottoposta a screening. I punti rappresentano log2 dei punteggi di deplezione della piega per i singoli geni, che sono tracciati su una rappresentazione boxplot della distribuzione complessiva del punteggio. Il test t di Student è stato utilizzato per verificare una differenza significativa nella media dei punteggi di deplezione tra i due gruppi in ciascuna linea cellulare. I valori p risultanti sono indicati in ogni pannello. Si noti che i punteggi medi di deplezione della piega sono significativamente più alti per i CEG rispetto ai non CET. Ciò è previsto in quanto i geni essenziali del nucleo sono, per definizione, costantemente necessari per la proliferazione e/o la sopravvivenza nella maggior parte dei tipi di cellule.
La Figura 6A presenta un diagramma di Venn degli "hit" per tre linee cellulari umane MPNST. In genere troviamo che un gran numero di geni è condiviso tra più linee; questi hit sono una priorità elevata in quanto rappresentano geni che codificano proteine che sono probabilmente essenziali per la proliferazione e/o la sopravvivenza di un ampio sottogruppo di MPNST. Si noti inoltre che ci sono un certo numero di geni che sono hit in una sola linea cellulare. Lo incontriamo comunemente e non dovrebbe essere preso come un'indicazione che gli schermi siano di scarsa qualità. I geni che sono hit comuni tra più linee vengono quindi valutati utilizzando il Drug Gene Interaction Database per identificare i geni all'interno di questo sottoinsieme che codificano proteine che sono farmacologiche con agenti esistenti. Ne selezioniamo quindi diversi ed eseguiamo una convalida iniziale abbattendo l'espressione dell'mRNA corrispondente con shRNA. Poiché alcuni shRNA avranno effetti off-target, testiamo sempre più shRNA che mirano allo stesso trascritto. La Figura 6B mostra un risultato rappresentativo in cui abbiamo trasdotto cellule MPNST con un controllo non mirato e shRNA multipli mirati a BCL6. Il numero di cellule è stato quindi determinato in momenti variabili dopo la trasduzione. Si noti che molti degli shRNA BCL6 hanno ridotto notevolmente il numero di cellule; come mostrato nell'immunoblot di accompagnamento, il grado di diminuzione del numero di cellule è correlato al grado di knockdown di BCL6. La Figura 6C mostra una curva di crescita rappresentativa per una coltura MPNST P 0-GGFβ3 a passaggio precoce.

Figura 1: Flusso di lavoro per l'esecuzione del sequenziamento dell'intero esoma del tessuto MPNST o delle cellule MPNST di passaggio precoce. Lo schema illustra il flusso di lavoro generale del rilevamento delle varianti presenti nelle colture di passaggio precoce derivate dal tumore. Isolare il DNA da colture di passaggio precoce (1) e inviare DNA di qualità al nucleo di sequenziamento secondo i loro protocolli di sottomissione (2). Il nucleo di sequenziamento controllerà la qualità del DNA presentato ed eseguirà tutte le preparazioni necessarie per il campione e la libreria del genoma. La struttura principale fornirà agli utenti file di sequenziamento FASTQ con metriche di controllo della qualità (3). Gli utenti caricheranno i file FASTQ in un programma di allineamento del genoma e di chiamata delle varianti di loro scelta. (4) Le varianti annotate vengono filtrate in base a criteri definiti dall'utente per rimuovere le varianti non pertinenti. I dati rappresentativi mostrati confrontano un campione di tumore umano MPNST resecato rispetto a una linea cellulare derivata dal tumore26. (5) Eseguire l'analisi di classificazione funzionale con PANTHER. Abbreviazione: MPNST = Tumore maligno della guaina dei nervi periferici. Fare clic qui per visualizzare una versione più grande di questa figura.
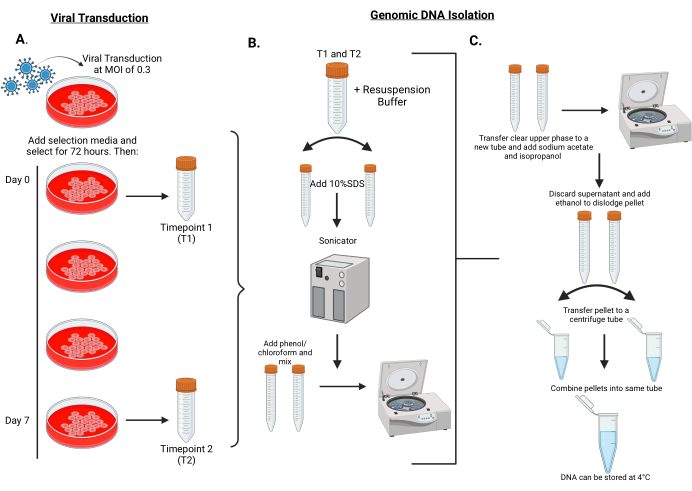
Figura 2: Flusso di lavoro per l'esecuzione della trasduzione virale delle librerie shRNA in cellule MPNST e l'isolamento del DNA genomico dalle cellule al punto temporale 1 e al punto temporale 2. (A) Le cellule bersaglio vengono infettate a un basso MOI di 0,3 con particelle lentivirali codificate a barre e selezionate per 72 ore. Le cellule vengono fatte passare per 5-7 raddoppi di popolazione (circa 7 giorni). I pellet cellulari al giorno 0 e al giorno 7 vengono conservati a -80 °C per l'isolamento del DNA genomico. Il giorno 0 è indicato come punto temporale 1 (T1) e il giorno 7 è indicato come punto temporale 2 (T2). (B) L'isolamento del DNA genomico inizia con la risospensione dei pellet cellulari in tampone di risospensione che vengono poi divisi in due provette da 15 mL. Per facilitare la lisi cellulare, il 10% di SDS viene aggiunto a ciascuna provetta e sonicata per 25 cicli di 30 s acceso/30 s spento. Dopo la sonicazione, il fenolo/cloroformio viene aggiunto a ciascuna provetta e fatto vorticare vigorosamente per 45-60 s. Le provette vengono quindi centrifugate. (C) Una fase superiore trasparente viene pipettata e aggiunta a una provetta pulita con l'aggiunta di acetato di sodio/isopropanolo e mescolata bene. Le provette vengono centrifugate di nuovo. Questa volta, il surnatante viene scartato e il pellet viene rimosso con l'aggiunta di etanolo al 70%. Unire i pellet risospesi in un unico tubo e centrifugare alla massima velocità in una centrifuga da banco. Scartare il surnatante e risospendere il pellet in acqua distillata. Fare clic qui per visualizzare una versione più grande di questa figura.
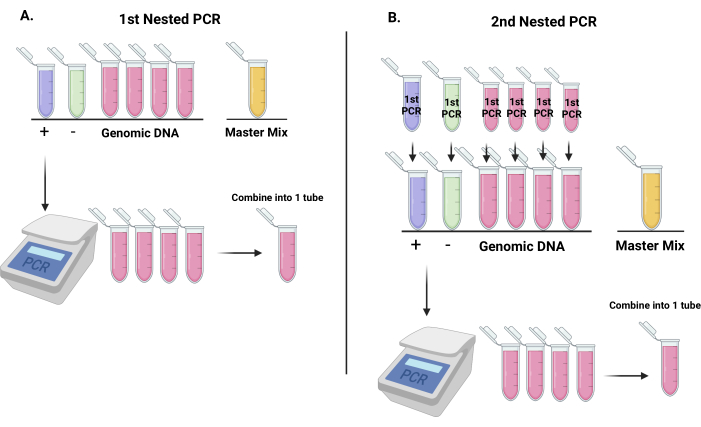
Figura 3: Flusso di lavoro per l'amplificazione di sequenze di codici a barre da vettori shRNA lentivirali in preparazione alla quantificazione dei codici a barre con sequenziamento di nuova generazione . (A) Rappresentazione di come impostare le provette per la prima reazione di PCR annidata (7 provette: una per il controllo negativo, una per il controllo positivo e le restanti 4 provette per il DNA genomico. L'ultimo tubo fungerà da tubo di miscelazione principale). Dopo la prima reazione di PCR, unire le provette di DNA genomico in un'unica provetta e mescolare. (B) I prodotti della prima reazione di PCR annidata serviranno come modelli per la seconda reazione di PCR annidata. Dopo la seconda PCR, combinare le provette di DNA genomico in un'unica provetta e mescolare. Fare clic qui per visualizzare una versione più grande di questa figura.
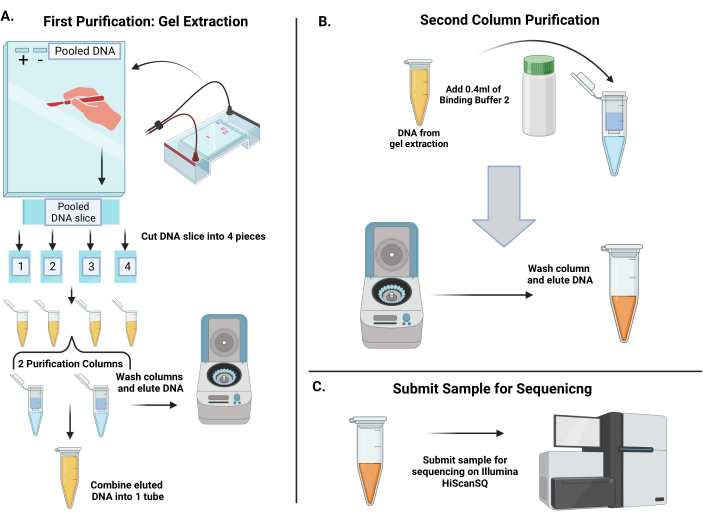
Figura 4: Flusso di lavoro per la purificazione dei codici a barre shRNA amplificati e il sequenziamento dei codici a barre per quantificare la loro rappresentazione al punto temporale 1 e al punto temporale 2. (A) Versare un gel di agarosio al 3,5%. Poiché il volume del DNA accumulato supera il limite per un pozzetto, fissare insieme 4-6 denti di un pettine di gel per produrre un pozzetto grande. Preparare i prodotti PCR con il colorante di caricamento 6x e caricare il controllo positivo, il controllo negativo e il DNA aggregato nel gel. Dopo l'elettroforesi, una grande banda a circa 250 coppie di basi dovrebbe apparire nella corsia del DNA aggregato. Usando un bisturi pulito, asportare l'intera fascia, quindi tagliare in 4 fette di gel. Solubilizzare i pezzi di gel e poi combinarli in due colonne di spin per eluire il DNA. Combina il DNA eluito in un'unica provetta. (B) Il DNA viene purificato attraverso una seconda fase di purificazione. Aggiungere il tampone di legame 2 alla provetta di DNA in pool, quindi pipettare su una colonna di rotazione. Lavare la membrana e poi eluire il DNA in acqua distillata. (C) Inviare il DNA purificato al nucleo di sequenziamento. Fare clic qui per visualizzare una versione più grande di questa figura.
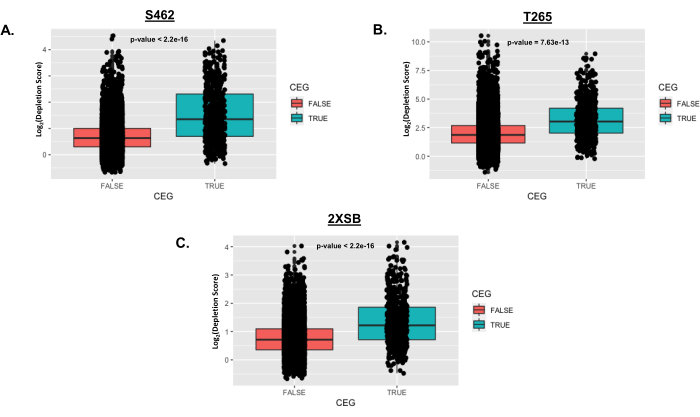
Figura 5: Esempi rappresentativi della distribuzione dei Core Essential Genes dopo l'analisi come descritto nel protocollo. In questo esempio, tre linee cellulari umane MPNST (S462, T265 e 2XSB) sono state sottoposte a screening con le librerie shRNA Cellecta DECIPHER. Per ogni linea cellulare umana MPNST, è stato creato un boxplot per confrontare i punteggi di deplezione a livello genico per i geni nell'elenco dei Core Essential Genes (CEG; True box plot)25 a quello dei geni che non si trovano nell'elenco dei CEG (False box plot). I singoli punti dati sono sovrapposti a ciascun box plot. I valori P provengono da un test t standard che confronta i punteggi di deplezione a livello genico dei CEG con quelli dei non-EG. Abbreviazioni: CEG = core essential gene; MPNST = Tumore maligno della guaina dei nervi periferici. Fare clic qui per visualizzare una versione più grande di questa figura.
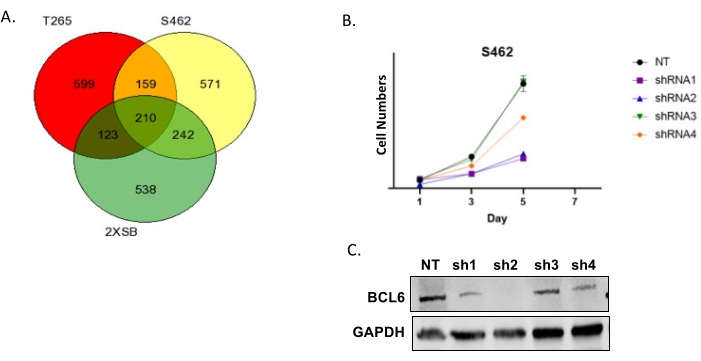
Figura 6: Convalida dei risultati dello screening . (A) Diagramma di Venn rappresentativo di hit sovrapposte in tre linee cellulari umane MPNST. (B) Cellule umane MPNST S462 trasdotte con un vettore lentivirale non bersaglio (NT) e lentivirus che esprimono quattro diversi shRNA BCL6 (shRNA1, shRNA2, shRNA3 e shRNA4). Le cellule sono state trasdotte con lentivirus e poi trattate con un agente di selezione (puromicina) per 3 giorni. Il numero di cellule è stato poi valutato nei sette giorni successivi. (C) Analisi Western blot che mostra i livelli proteici di BCL6 dopo trasduzione con lentivirus NT, shRNA1, shRNA2, shRNA3 e shRNA4. Fare clic qui per visualizzare una versione più grande di questa figura.
Tabella 1: Disposizione delle piastre per la titolazione lentivirale. Clicca qui per scaricare questa tabella.
Tabella 2: Impostazione iniziale delle prime reazioni di PCR. Clicca qui per scaricare questa tabella.
Tabella 3: Preparazione della miscela master per la prima reazione di PCR. Clicca qui per scaricare questa tabella.
Tabella 4: Primi parametri della PCR di Cellecta. Clicca qui per scaricare questa tabella.
Tabella 5: Configurazione iniziale della seconda reazione di PCR. Clicca qui per scaricare questa tabella.
Tabella 6: Parametri della seconda PCR di Cellecta. Clicca qui per scaricare questa tabella.
Discussione
I metodi dettagliati qui presentati sono stati sviluppati per studiare la neoplasia del sistema nervoso periferico e la patogenesi della MPNST. Sebbene abbiamo riscontrato l'efficacia di questi metodi, è necessario riconoscere che esistono alcune potenziali limitazioni ai metodi descritti in questa sede. Di seguito, discutiamo alcune di queste limitazioni e le potenziali strategie per superarle in altri sistemi modello.
Abbiamo scoperto che il sequenziamento dell'intero esoma identifica efficacemente le mutazioni di interesse nei topi P 0-GGFβ3. Va riconosciuto, tuttavia, che il sequenziamento dell'intero esoma ha dei limiti. In primo luogo, il sequenziamento dell'intero esoma non è un approccio efficace per identificare i prodotti genici di fusione. Questo perché la maggior parte delle rotture cromosomiche e della successiva fusione coinvolgono prevalentemente regioni intergeniche e introni, poiché tali regioni rappresentano la maggior parte del genoma. Abbiamo invece scoperto che l'RNA-Seq con letture paired-end identifica in modo molto più efficace i geni di fusione. C'è anche la questione dell'efficacia con cui il sequenziamento dell'intero esoma identifica regioni relativamente grandi di perdita cromosomica.
Sebbene siano stati sviluppati diversi algoritmi per identificare tali perdite, il termine "sequenziamento dell'intero esoma" è di per sé fuorviante perché la cattura dell'esoma, anche in buone esecuzioni, spesso perde fino al 5-10% delle regioni esoniche. Per questo motivo, integriamo abitualmente il sequenziamento dell'intero esoma con altri approcci come l'ibridazione genomica comparativa di array (aCGH). Dopo aver identificato i guadagni e le perdite, esaminiamo i geni all'interno di questi intervalli e li confrontiamo con le mutazioni driver note associate alle loro controparti umane. Tuttavia, il genoma del topo è più stabile del genoma umano27. Di conseguenza, i tumori del topo in genere non mostrano cromotripsi analoga a quella osservata nelle neoplasie umane. Il pattern nei tumori murini è invece molto più semplice, tendente verso guadagni o perdite di cromosomi interi o cromosomici con relativamente poche delezioni focali che tendono a verificarsi sotto una forte pressione selettiva22,23.
Ci sono alcune potenziali insidie che abbiamo incontrato durante l'esecuzione di screening shRNA su scala genomica. Uno dei problemi più comuni che incontriamo è la trasduzione relativamente scarsa dei vettori lentivirali nelle cellule bersaglio. Il più delle volte scopriamo che il problema è sorto da una titolazione impropria dei pool lentivirali confezionati. Poiché le colture di cellule tumorali di topo a passaggio precoce sono una risorsa limitante, molti ricercatori tenteranno invece di titolare il loro lentivirus utilizzando un'altra linea cellulare consolidata che è più facilmente disponibile. Il problema con questo approccio, tuttavia, è che l'efficienza della trasduzione lentivirale può variare considerevolmente da tipo di cellula a tipo di cellula. È per questo motivo che si consiglia di titolare il lentivirus sulle cellule reali che verranno utilizzate nell'esperimento. Abbiamo anche riscontrato problemi con titoli virali relativamente bassi. Questo problema riflette il più delle volte una scarsa trasfezione delle cellule 293T durante la produzione del virus impacchettato.
È possibile ottenere risultati falsi positivi durante l'esecuzione di screening shRNA su scala genomica. Per questo motivo, una volta identificati i bersagli potenzialmente farmacologici che sono di maggiore interesse per noi, convalidiamo sempre i risultati dei nostri screening shRNA. In genere, utilizzeremo due approcci diversi per convalidare gli obiettivi ad alto interesse. In primo luogo, abbattiamo l'espressione genica utilizzando due o più shRNA distinti da quelli utilizzati nello screening iniziale e determiniamo l'effetto che questo ha sulla proliferazione e sulla sopravvivenza delle cellule tumorali. In secondo luogo, otteniamo i farmaci identificati nel Drug Gene Interaction Database e determiniamo l'effetto che questo ha sulla proliferazione e sulla sopravvivenza delle cellule tumorali. Usiamo entrambi gli approcci in tandem perché abbiamo incontrato circostanze in cui gli shRNA funzionano e il farmaco no. Almeno in alcuni di questi casi, l'esame dell'intero set di dati della sequenza dell'esoma ha dimostrato che la proteina bersaglio è prodotta da un gene che ha una mutazione che potenzialmente influenza le interazioni farmaco-proteina.
Gli approcci sopra descritti forniranno allo sperimentatore un mezzo applicabile per identificare le potenziali mutazioni driver che si verificano nelle neoplasie rare, identificare funzionalmente le vie di segnalazione necessarie per la proliferazione e la sopravvivenza e dare priorità ai bersagli per lo sviluppo terapeutico. Ci auguriamo che altri ricercatori trovino questi approcci utili per identificare i principali bersagli terapeutici in altri tumori umani. Il lettore dovrebbe essere consapevole, tuttavia, che ci sono altri approcci genomici funzionali che possono essere utilizzati per identificare i geni coinvolti nella patogenesi del tumore e i geni che codificano potenziali bersagli terapeutici. Ad esempio, sono disponibili librerie CRISPR che possono essere utilizzate in modo analogo a quello che descriviamo per le librerie shRNA. Gli screening funzionali possono anche essere eseguiti in vivo per identificare i geni che promuovono la tumorigenesi. Ad esempio, il sistema di mutagenesi somatica basato su trasposoni Sleeping Beauty è stato precedentemente utilizzato per colpire le cellule di Schwann e i loro precursori, con conseguente identificazione di diverse centinaia di geni coinvolti nella patogenesi della MPNST28. Poiché questi sistemi si avvicinano alla genomica funzionale in modi diversi, raccomandiamo che il ricercatore consideri attentamente gli obiettivi degli esperimenti pianificati e basi la selezione di una metodologia di genomica funzionale su tali obiettivi.
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto da sovvenzioni del National Institute of Neurological Diseases and Stroke (R01 NS048353 e R01 NS109655 a S.L.C.; R01 NS109655-03S1 a D.P.J.), il National Cancer Institute (R01 CA122804 a S.L.C.) e il Dipartimento della Difesa (X81XWH-09-1-0086 e W81XWH-12-1-0164 a S.L.C.).
Materiali
| Name | Company | Catalog Number | Comments |
| Bioruptor Sonication System | Diagenode | UCD-600 | |
| CASAVA 1.8.2 | |||
| Cbot | Illumina, San Diego, CA | N/A | |
| Celigo Image Cytometer | Nexcelom | N/A | |
| Cellecta Barcode Analyzer and Deconvoluter software | |||
| Citrisolve Hybrid | Decon Laboratories | 5989-27-5 | |
| Corning 96-well Black Microplate | Millipore Sigma | CLS3603 | |
| Diagenode Bioruptor 15ml conical tubes | Diagenode | C30010009 | |
| dNTP mix | Clontech | 639210 | |
| Eosin Y | Thermo Scientific | 7111 | |
| Elution buffer | Qiagen | 19086 | |
| Ethanol (200 Proof) | Decon Laboratories | 2716 | |
| Excel | Microsoft | ||
| FWDGEX 5’-CAAGCAGAAGACGGCATACGAGA-3’ | |||
| FWDHTS 5’-TTCTCTGGCAAGCAAAAGACGGCATA-3’ | |||
| GexSeqS (5’ AGAGGTTCAGAGTTCTACAGTCCGAA-3’ | HPLC purified | ||
| GraphPad Prism | Dotmatics | ||
| Harris Hematoxylin | Fisherbrand | 245-677 | |
| Illumina HiScanSQ | Illumina, San Diego, CA | N/A | |
| Paraformaldehyde (4%) | Thermo Scientific | J19943-K2 | |
| PLUS Transfection Reagent | Thermo Scientific | 11514015 | |
| Polybrene Transfection Reagent | Millipore Sigma | TR1003G | |
| PureLink Quick PCR Purification Kit | Invitrogen | K310001 | |
| Qiagen Buffer P1 | Qiagen | 19051 | |
| Qiagen Gel Extraction Kit | Qiagen | 28704 | |
| RevGEX 5’-AATGATACGGCGACCACCGAGA-3’ | |||
| RevHTS1 5’-TAGCCAACGCATCGCACAAGCCA-3’ | |||
| Titanium Taq polymerase | Clontech | 639210 | |
| Trimmomatic software | www.usadellab.org |
Riferimenti
- Carroll, S. L. Molecular mechanisms promoting the pathogenesis of Schwann cell neoplasms. Acta Neuropathol. 123 (3), 321-348 (2012).
- Longo, J. F., Weber, S. M., Turner-Ivey, B. P., Carroll, S. L. Recent Advances in the Diagnosis and Pathogenesis of Neurofibromatosis Type 1 (NF1)-associated Peripheral Nervous System Neoplasms. Adv Anat Pathol. 25 (5), 353-368 (2018).
- Longo, J. F., Carroll, S. L. The RASopathies: Biology, genetics and therapeutic options. Adv Cancer Res. 153, 305-341 (2022).
- Birindelli, S., et al. Rb and TP53 pathway alterations in sporadic and NF1-related malignant peripheral nerve sheath tumors. Lab Invest. 81 (6), 833-844 (2001).
- Legius, E., et al. TP53 mutations are frequent in malignant NF1 tumors. Genes Chromosomes Cancer. 10 (4), 250-255 (1994).
- Menon, A. G., et al. Chromosome 17p deletions and p53 gene mutations associated with the formation of malignant neurofibrosarcomas in von Recklinghausen neurofibromatosis. Proc Natl Acad Sci U S A. 87 (14), 5435-5439 (1990).
- Upadhyaya, M., et al. Germline and somatic NF1 gene mutation spectrum in NF1-associated malignant peripheral nerve sheath tumors (MPNSTs). Hum Mutat. 29 (1), 74-82 (2008).
- Kourea, H. P., Orlow, I., Scheithauer, B. W., Cordon-Cardo, C., Woodruff, J. M. Deletions of the INK4A gene occur in malignant peripheral nerve sheath tumors but not in neurofibromas. Am J Pathol. 155 (6), 1855-1860 (1999).
- Nielsen, G. P., et al. Malignant transformation of neurofibromas in neurofibromatosis 1 is associated with CDKN2A/p16 inactivation. Am J Pathol. 155 (6), 1879-1884 (1999).
- Gregorian, C., et al. PTEN dosage is essential for neurofibroma development and malignant transformation. Proc Natl Acad Sci U S A. 106 (46), 19479-19484 (2009).
- Lee, W., et al. PRC2 is recurrently inactivated through EED or SUZ12 loss in malignant peripheral nerve sheath tumors. Nat Genet. 46 (11), 1227-1232 (2014).
- Zhang, M., et al. Somatic mutations of SUZ12 in malignant peripheral nerve sheath tumors. Nat Genet. 46 (11), 1170-1172 (2014).
- Varela, I., et al. Somatic structural rearrangements in genetically engineered mouse mammary tumors. Genome Biol. 11 (10), 100 (2010).
- Johnson, R. A., et al. Cross-species genomics matches driver mutations and cell compartments to model ependymoma. Nature. 466 (7306), 632-636 (2010).
- Kim, M., et al. Comparative oncogenomics identifies NEDD9 as a melanoma metastasis gene. Cell. 125 (7), 1269-1281 (2006).
- Zender, L., et al. Identification and validation of oncogenes in liver cancer using an integrative oncogenomic approach. Cell. 125 (7), 1253-1267 (2006).
- Uren, A. G., et al. Large-scale mutagenesis in p19(ARF)- and p53-deficient mice identifies cancer genes and their collaborative networks. Cell. 133 (4), 727-741 (2008).
- Starr, T. K., et al. A transposon-based genetic screen in mice identifies genes altered in colorectal cancer. Science. 323 (5922), 1747-1750 (2009).
- Dupuy, A. J., et al. A modified sleeping beauty transposon system that can be used to model a wide variety of human cancers in mice. Cancer Res. 69 (20), 8150-8156 (2009).
- Carroll, S. L. The Challenge of Cancer Genomics in Rare Nervous System Neoplasms: Malignant Peripheral Nerve Sheath Tumors as a Paradigm for Cross-Species Comparative Oncogenomics. Am J Pathol. 186 (3), 464-477 (2016).
- Huijbregts, R. P., Roth, K. A., Schmidt, R. E., Carroll, S. L. Hypertrophic neuropathies and malignant peripheral nerve sheath tumors in transgenic mice overexpressing glial growth factor beta3 in myelinating Schwann cells. J Neurosci. 23 (19), 7269-7280 (2003).
- Kazmi, S. J., et al. Transgenic mice overexpressing neuregulin-1 model neurofibroma-malignant peripheral nerve sheath tumor progression and implicate specific chromosomal copy number variations in tumorigenesis. Am J Pathol. 182 (3), 646-667 (2013).
- Brosius, S. N., et al. Neuregulin-1 overexpression and Trp53 haploinsufficiency cooperatively promote de novo malignant peripheral nerve sheath tumor pathogenesis. Acta Neuropathol. 127 (4), 573-591 (2014).
- Hart, T., Brown, K. R., Sircoulomb, F., Rottapel, R., Moffat, J. Measuring error rates in genomic perturbation screens: gold standards for human functional genomics. Mol Syst Biol. 10 (7), 733 (2014).
- Hart, T., et al. Evaluation and Design of Genome-Wide CRISPR/SpCas9 Knockout Screens. G3. 7 (8), 2719-2727 (2017).
- Longo, J. F., et al. Establishment and genomic characterization of a sporadic malignant peripheral nerve sheath tumor cell line. Sci Rep. 11 (1), 5690 (2021).
- Maser, R. S., et al. Chromosomally unstable mouse tumours have genomic alterations similar to diverse human cancers. Nature. 447 (7147), 966-971 (2007).
- Rahrmann, E. P., et al. Forward genetic screen for malignant peripheral nerve sheath tumor formation identifies new genes and pathways driving tumorigenesis. Nat Genet. 45 (7), 756-766 (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon
Utilizziamo i cookies per migliorare la tua esperienza sul nostro sito web.
Continuando a utilizzare il nostro sito web o cliccando “Continua”, accetti l'utilizzo dei cookies.