Method Article
Zebra Balığı Kalplerinde CRISPR-Cas9 Aracılı Hassas Knock-In Düzenlemeleri
Bu Makalede
Özet
Bu protokol, CRISPR-Cas9 teknolojisini kullanarak zebra balığı embriyolarında hassas knock-in düzenlemelerini kolaylaştırmak için bir yaklaşımı açıklamaktadır. Uzun QT Sendromu ile ilişkili bir gen varyantını modellemek için bu tekniklerin uygulanabilirliğini göstermek için bir fenotipleme boru hattı sunulmuştur.
Özet
Hayvan modellerinde düzenli olarak kümelenmiş aralıklı kısa palindromik tekrarlar (CRISPR), fizyolojik olayların incelenmesi için kesin genetik manipülasyon sağlar. Zebra balığı, tüm organ ve organizma düzeyinde kalıtsal hastalık, gelişim ve toksikoloji ile ilgili sayısız soruyu incelemek için etkili bir genetik model olarak kullanılmıştır. İyi açıklamalı ve haritalanmış zebra balığı genomu nedeniyle, gen düzenleme için çok sayıda araç geliştirilmiştir. Bununla birlikte, CRISPR kullanarak hassas knock-in düzenlemeleri üretmenin etkinliği ve tespit etme kolaylığı sınırlayıcı bir faktördür. Burada açıklanan, kardiyak repolarizasyondan sorumlu ve elektriksel bozukluk Uzun QT Sendromu (LQTS) ile ilişkili bir gendeki hassas düzenlemelerin basit tespiti ile CRISPR-Cas9 tabanlı bir knock-in yaklaşımıdır. Bu iki tek kılavuzlu RNA (sgRNA) yaklaşımı, hedef diziyi heyecanlandırır ve değiştirir ve genetik olarak kodlanmış bir muhabir genini birbirine bağlar. Bu yaklaşımın yararı, vahşi tip ve gen düzenlenmiş zebra balığı larvalarında kardiyak elektriksel fonksiyonun invaziv olmayan fenotipik ölçümlerini tanımlayarak gösterilmiştir. Bu yaklaşım, bütün bir organizmada hastalıkla ilişkili varyantların verimli bir şekilde çalışılmasını sağlar. Ayrıca, bu strateji, muhabir genleri, ortologlar veya gen editörleri gibi tercih edilen eksojen dizilerin eklenmesi için olanaklar sunar.
Giriş
Hayvan modellerinde CRISPR tabanlı gen düzenleme stratejileri, genetik olarak kalıtsal hastalık, gelişim ve toksikolojinin tüm organizma düzeyindeincelenmesini sağlar 1,2,3. Zebra balığı, insanlara birçok fizyolojik açıdan murin veya insan kaynaklı hücre modellerinden daha yakın olan güçlü bir model sağlar4. Zebra balıklarında hem ileri5 hem de ters genetik tarama6 için kapsamlı bir dizi genetik araç ve strateji kullanılmıştır. Zebra balığındaki kapsamlı genetik haritalama ve ek açıklama, hedeflenen gen nakavtlarını (KO'lar) ve hassas vuruşları (KI'lar)7 tasarlamak için birincil teknik olarak gen düzenleme yaklaşımlarını kolaylaştırmıştır.
Buna rağmen, zebra balıklarında hassas KI düzenlemeleri oluşturmak, düşük verimlilik ve doğru algılamanın zorluğu ile sınırlıdır. Transkripsiyon faktörü benzeri efektör nükleazlar (TALEN'ler) KI'lar8 için başarıyla kullanılmış ve optimize edilmiş olmasına rağmen, CRISPR daha basit sgRNA hedeflemesi ile gelişmiş bir gen düzenleme stratejisi sağlar. Çok sayıda çalışma, zebra balığı 9,10,11,12,13,14,15,16,17,18,19,20'de hassas KI'lar oluşturmak için CRISPR'yi kullanmıştır, ancak CRISPR aracılı homolojiye yönelik onarım (HDR) yoluyla oluşturulan bu düzenlemeler düşük içsel başarı ile verimsiz olma eğilimindedir. birincil ekran olarak genotipleme gerektiren oranlar 9,10,14,21. Bu, zebra balıklarında verimli bir KI CRISPR sistemine ve hassas düzenlemeleri tespit etmek için güvenilir, yüksek verimli bir sisteme duyulan ihtiyacı göstermektedir.
Bu çalışmanın amacı, zebra balığı kalplerinde başarılı düzenlemelerin basit ve yüksek verimli tespiti ile hassas bir kardiyak gen KI oluşturmak için bir platform tanımlamaktı. TALEN yaklaşımı8'e dayanan bir CRISPR-Cas9 tabanlı iki sgRNA ekzon replasman yaklaşımı tanımlanmıştır. Bu yaklaşım, iki sgRNA kılavuzu kullanılarak hedef dizinin eksizyonunu ve ilgilenilen KI'nın yanı sıra genetik olarak kodlanmış bir intronic muhabir genini içeren eksojen bir şablon dizisi ile değiştirilmesini içerir (Şekil 1). Genetik olarak kodlanmış bir floresan muhabirin hedef gen intronik dizisine entegrasyonu, pozitif düzenlemelerin etkili bir şekilde tespit edilmesini sağlar. Daha sonra, zebra balığı larvalarında, bireyleri ani kardiyak ölüme yatkın kılan bir kardiyak elektriksel bozukluk olan kalıtsal LQTS ile ilişkili gen varyantlarının non-invaziv karakterizasyonu için kardiyak elektriksel fonksiyonu değerlendirmek için bir fenotipleme platformu tanımlanmıştır.
Bu yaklaşımlar, kalıtsal hastalıkları modellemek ve gen ekspresyon kalıplarının haritalanması ve gelişimsel düzenleme gibi biyolojik ve fizyolojik soruları ele almak için zebra balığı KI gen düzenlemelerine erişimi ve bunların kullanımını artıracaktır. Zebra balığı kalpleri, insan kardiyak elektrofizyolojik özelliklerine murin modellerinden daha iyi paralel olduğundan, kalp hastalığı modellemesiiçin genetik olarak izlenebilir bir sistem olarak özellikle çekici olabilirler 7,22,23.
Protokol
Zebra balığı kullanan çalışmalar, Simon Fraser Üniversitesi Hayvan Bakım Komitesi ve Kanada Hayvan Bakımı Konseyi'nin politika ve prosedürleri ile uyumlu olarak yürütülmüş ve 1264K-18 numaralı protokol kapsamında tamamlanmıştır.
1. Hassas düzenlemeler için CRISPR bileşenlerinin tasarımı
- KI hedef bölgesini içeren diziyi çıkarmak için kullanılacak iki sgRNA kılavuzunu tasarlamak için, önce ilgilenilen gen için zebra balığı ortologunu tanımlayın.
NOT: Şekil 2 , iki sgRNA CRISPR-Cas9 yaklaşımını kullanarak hassas düzenlemeler tasarlama adımlarına özet bir genel bakış sunmaktadır. - Daha sonra, Danio rerio'nun bir tür olarak seçilmesini ve kullanılacak Cas enzimini içeren CRISPOR24 gibi bir tasarım yazılımı aracı kullanın.
NOT: Bu çalışmanın ilgi çekici geni zkcnh6a idi (Ensembl Transcript ID: ENSDART00000090809.6; UniProt Protein ID: B3DJX4) ve hedef mutasyon amino asit, R56Q idi.- İki-sgRNA yaklaşımı için, hedef ekzondan önce gelen bir sgRNA konumu ve hemen aşağı akış intronunda bulunan ikinci bir sgRNA seçin.
- Seçilen sgRNA'ların yüksek özgüllüğe ve düşük tahmin edilen hedef dışı bağlanmaya sahip olduğundan emin olun. Minimum hedef dışı bağlamaya sahip kılavuzları belirlemek için CRISPOR sıralamalarını kullanın. Potansiyel hedef dışı hedeflerin tohum dizisinde uyumsuzluk olmayan kılavuzları dikkate almayın.
- Adım 6.1.3'te PCR tabanlı Sanger dizileme genotiplemesi için en olası potansiyel hedef dışı bölgeleri belirleyin (CRISPOR puanlarına dayanarak, ilk üç ekzon potansiyel hedef dışı bölgeyi seçin).
- İki sgRNA seçildikten sonra, her biri için ters tamamlayıcıyı elde edin, böylece kullanılacak dört oligonükleotid vardır: mutasyondan önce gelen iki tamamlayıcı oligo ve aşağı yönde olan iki tamamlayıcı oligo.
- Dört oligonun her birinde, tercih edilen bir kılavuz plazmidine dahil edilmek üzere uyumlu kısıtlama bölgeleri ekleyin; DR274 plazmidine entegrasyon için, çıkıntı oluşturmak üzere 5' BsaI kısıtlama bölgesini kullanın. Bsa1 tanıma sahasının CRISPOR'dan seçilen kılavuzun 5' ucunda ve Bsa1 tanıma sahasının DR274 plazmidindeki kılavuzların doğru yönlendirilmesini sağlamak için tamamlayıcı tellerin 5' ucunda tasarlandığından emin olun (bkz. Şekil 3).
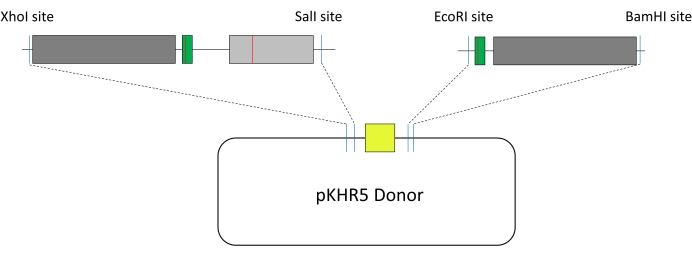
- Zebra balıklarında HDR için kullanılan eksojen şablonu tasarlamak için (Şekil 1), pKHR5 plazmidinde bulunan mVenus YFP muhabir genini çevreleyen iki dizi parçası seçin.
NOT: Amaçlanan değişiklik/düzenleme, yukarı akış veya aşağı akış parçasına dahil edilebilir.- Ensembl'i kullanarak, şablonu oluşturmak için kullanılacak yaklaşık 2 kb yan dizisi (homoloji kolu) dahil olmak üzere hedef bölgeyi ilgilenilen gen dizisi içinde bulun.
NOT: Homoloji kolları, hedef bölgenin yukarı ve aşağı akış olmak üzere simetrik veya asimetrik 25,26 ve her biri yaklaşık 1 kb olabilir. - Şablonu, mVenüs YFP muhabir geninin her iki tarafına yerleştirilecek iki bölüme ayırın (bkz. Şekil 4). Kodlama dizisinin kesintiye uğramaması için bölünmüş sitenin bir intron içinde olduğundan emin olun.
NOT: Gen iyi karakterize edilmişse, ekleme bölgeleri veya düzenleyici bölgeler gibi introndaki işlevsel rolleri kontrol edin. 5' veya 3' uçlarına yakın bölgeler mRNA eklemesinde daha sık rol oynar. - Cas enziminin yeniden kesilmesini önlemek için i) kılavuz protospacer bitişik motifindeki (PAM) veya tohum dizisindeki sessiz mutasyonları (Cas enziminin hedefleyebileceği alternatif PAM bölgelerinin farkında olun) içeren modifikasyonları şablon dizisine dahil edin; ii) faizin değiştirilmesi; iii) mVenüs YFP muhabir genini içeren pKHR5 plazmidine klonlamayı kolaylaştırmak için kısıtlama endonükleaz bölgelerinin oluşturulması (bkz. Şekil 3).
NOT: Bu çalışmada, ilk şablon segmenti R56Q mutasyon bölgesinin yukarı akışını ve SalI aşağı akışını içerirken, ikinci şablon segmenti kılavuz hedef dizisinin yukarı akışının EcoRI'sini ve BamHI aşağı akışını içeriyordu. Seçilen kısıtlama bölgelerinden herhangi biri şablon dizisinde mevcutsa, bunları susturmak için mutasyonlar gerekli olacaktır veya Gibson Assembly gibi alternatif yaklaşımlar kullanılabilir.
- Ensembl'i kullanarak, şablonu oluşturmak için kullanılacak yaklaşık 2 kb yan dizisi (homoloji kolu) dahil olmak üzere hedef bölgeyi ilgilenilen gen dizisi içinde bulun.
2. Embriyo mikroenjeksiyonu için CRISPR bileşenlerinin hazırlanması
- Cas9'u mikroenjeksiyonlardan 1 hafta önce mikroenjeksiyon için hazırlayın. Cas9 proteini kullanın veya in vitro transkripsiyon yoluyla Cas9 mRNA'yı hazırlayın.
NOT: Bu çalışmada, Cas9 mRNA kullanılmıştır, çünkü verimlilikler daha yüksek olma eğilimindedir.- Ticari olarak temin edilebilen XL1 Blue bakteriyel agar bıçağının (Cas9 plazmid içeren) bakteriyel kültürlerini ampisilin gibi uygun bir antibiyotik kullanarak yükseltin. −80 ° C'de uzun süreli depolama için yedek bir gliserol stoğu oluşturmak için 675 μL sıvı kültür (325 μL gliserol ile) kullanın.
- Miniprep kiti ile birlikte verilen protokole göre bir miniprep saflaştırması için sıvı kültürün geri kalanını kullanın. Son saflaştırılmış DNA'yı, sağlanan elüsyon tamponunun 50 μL'sinde yeniden askıya alın. Verimi ve saflığı incelemek için ürünü bir spektrofotometre ile ölçün.
- Uygun bir kısıtlama enzimi kullanarak ve ilgili enzim için listelenen uygun tampon ve kuluçka süresini kullanarak restriksiyon sindirimi yoluyla saflaştırılmış DNA'nın 2 μg'sini doğrusallaştırın.
- Doğrusallaştırılmış plazmidi bir PCR Saflaştırma Kiti kullanarak saflaştırın ve sağlanan elüsyon tamponunun 30 μL'sinde yeniden askıya alın.
- Ürünü ölçtükten sonra, bunu ilgilenilen destekçi için uygun transkripsiyon kitini kullanarak in vitro transkripsiyon için bir şablon olarak kullanın. Sağlanan protokolü izleyin ve lityum klorür çökeltme27 ile saflaştırın. Saflaştırılmış RNA'yı 10 μL nükleaz içermeyenH2O içinde yeniden askıya alın ve mikroenjeksiyon karışımında kullanılmak üzere -20 ° C'de saklamadan önce miktarını artırın.
- İki sgRNA kılavuzunu hazırlayın.
- Ticari olarak temin edilebilen XL1 Blue bakteriyel agar bıçağından bakteri kültürlerini yükselterek sgRNA plazmidini bir iskele ile hazırlayın (ayrıntılar için Malzeme Tablosuna bakınız), yukarıdaki MLM3613 ile aynı şekilde (adım 2.1.1), ampisilin yerine kanamisin kullanın.
- Yukarıda tasarlanan sgRNA kılavuzları için iki çift tamamlayıcı tek sarmallı oligonükleotidin (ssODN'ler) ilk önce ssODN'leri 1x tavlama tamponunda 100 μM'lik bir konsantrasyona resüpres ederek tavlayın.
- İki sgRNA'nın her biri için ayrı reaksiyonlarda, bir termal döngüleyici kullanarak tamamlayıcı ssODN çiftini tavlayın. Her bir tamamlayıcı ssODN çiftinin 2 μg'sini 50 μL tavlama tamponu ile karıştırın ve 2 dakika boyunca 95 ° C'de inkübe edin, ardından 45 dakika boyunca 25 ° C'ye soğutun.
- Bir gRNA iskelesi içeren ticari olarak temin edilebilen bir plazmidi sindirin. 1 μL BsaI, 2 μL uygun tampon veddH 2O ila 20 μL kullanarak 1 saat boyunca 37 °C'de 2 μL DR274 plazmidini sindirin. Jel elektroforez28 kullanarak doğrusallaştırmayı onaylayın (isteğe bağlı: PCR Saflaştırma Kiti kullanarak saflaştırın).
- İki ayrı ligasyon reaksiyonunda (her sgRNA için bir tane), tavlanmış ssODN'leri doğrusallaştırılmış DR274 plazmid ile yaslanın. Bir molar insert: vektör oranı 3: 1 kullanın, çevrimiçi bir ligasyon hesaplayıcısı aracılığıyla uygun kütleyi hesaplayın. Kesici uç ve vektörün gerekli kütlesini 1 μL T4 DNA ligaz, 2 μL ligasyon tamponu ve ddH 2 O ila12μL ile karıştırın ve 12 saat boyunca oda sıcaklığında inkübe edin.
- Standart yaklaşımları kullanarak bağlı ürünün 2 μL'sini uygun yetkin hücrelere (10β hücreler gibi) dönüştürün ve ardından ticari olarak temin edilebilen bir Miniprep Kiti kullanarak ürünü yükseltin ve saflaştırın. İsteğe bağlı: Bu ürünün gliserol stoğunu oluşturun.
- İki sgRNA'yı, uzay dizisinin sonuna mümkün olduğunca yakın olan 3' aşağı akış kısıtlama bölgesini kullanarak her kılavuzun 2 μg'sini doğrusallaştırarak transkripte edin. DR274 plazmidi için, HindIII ile doğrusallaştırın ve daha sonra bir PCR Saflaştırma Kiti kullanarak RNA şablonunu saflaştırın, elüsyon tamponunun 30 μL'sinde yeniden süspansiyon yapın.
- Bir RNA transkripsiyon kiti kullanarak iki kılavuzu transkripte edin. Üreticinin protokolünü takip edin ve lityum klorür çökeltme27 ile saflaştırın. İki saflaştırılmış sgRNA kılavuzunu 10 μL nükleaz içermeyenH2O içinde yeniden askıya alın, mikroenjeksiyon karışımında kullanılmak üzere -20 ° C'de nicelleştirin ve saklayın.
NOT: RNA transkripsiyon kiti 5' kapak veya poli-A kuyruğu içeremez.
- Çift sarmallı, dışsal HDR muhabir şablonunu hazırlayın.
NOT: Şablon, mVenus YFP muhabir geninin biri yukarı ve diğeri aşağı akış olmak üzere iki parça halinde sentezlenir. Bu iki segment, ticari bir sağlayıcı aracılığıyla sipariş edilen ve daha sonra sırasıyla pKHR5 (mVenus YFP içeren) plazmidine bağlanan sentetik yapılardır.- pKHR5 plazmidini, ticari olarak temin edilebilen DH5α bakteri suşundan bakteri kültürlerini yükselterek hazırlayın (ayrıntılar için Malzeme Tablosu'na bakın) yukarıdaki MLM3613 ile aynı şekilde (adım 2.1.1).
NOT: pKHR5 plazmidi mVenüs YFP muhabir gen dizisini içerir. - Yukarıda tasarlanan iki şablon segmentini TE tamponunda 100 μM'ye kadar yeniden askıya alın ve ardından 10β hücrelere dönüştürün.
- İlk şablon segmentini (mVenüs YFP muhabir geninin bir yukarı akışı) ve pKHR5 plazmidini adım 1.3.3'te seçilen kısıtlama enzimlerini kullanarak sindirin.
NOT: pKHR5'in sıralı özeti, MCS'de seçilen kısıtlama bölgelerinin yakınlığı nedeniyle gereklidir. - Yukarı akış şablon segmenti için pKHR5 plazmidini hazırlamak için, SalI ile 4 μg pKHR5'i (adım 2.2.4'e göre) sindirin ve ardından bir PCR saflaştırma kiti kullanarak saflaştırın. 30 μL ddH2O'da yeniden askıya alın ve bunu XhoI ile ikinci sindirim reaksiyonu için bir şablon olarak kullanın. PCR Saflaştırma Kitini kullanarak ürünü saflaştırın.
- Şablon segmentinin 2 μg'ını (adım 2.3.2), 1 μL XhoI, 1 μL SalI, 2 μL uygun tamponu ve ddH 2 O ila20μL'yi 37 °C'de 1 saat boyunca sindirerek ilk şablon segmentini hazırlayın ve ürünü jel saflaştırın.
- Adım 2.2.5'te açıklanan reaksiyon koşullarını kullanarak yukarı akış şablon segmentini adım 2.3.5'ten hazırlanan pKHR5'e bağlayın. Bağlı ürünü yetkin 10β hücrelere dönüştürün, bir miniprep kullanarak yükseltin ve saflaştırın (isteğe bağlı: bu ürünün gliserol stoğunu oluşturun).
- Adım 2.3.6'daki (mVenus YFP muhabir geninin yukarı akışındaki pKHR5 plazmidine bağlanmış ilk şablon segmentini içeren) bağlı ürünü kullanın ve ikinci (mVenus muhabirinin aşağı akışı) şablon segmentine hazırlanmak için sindirim. Bağlı ürünün 4 μg'sini (adım 2.2.4'te olduğu gibi adım 2.3.6'dan itibaren) BamHI ile sindirin ve ardından bir PCR saflaştırma kiti kullanarak saflaştırın. 30 μL ddH2O'da yeniden askıya alın ve bunu EcoRI ile ikinci özet reaksiyonu için şablon olarak kullanın. PCR Saflaştırma Kitini kullanarak ürünü saflaştırın.
- İkinci şablon segmentini, şablon segmentinin 2 μg'sini (adım 2.3.2), 1 μL BamHI, 1 μL EcoRI, 2 μL uygun tamponu ve ddH 2 O ila20μL'yi 37 °C'de 1 saat boyunca sindirerek hazırlayın ve ürünü jel saflaştırın.
- Adım 2.2.5'te açıklanan reaksiyon koşullarını kullanarak aşağı akış şablon segmentini hazırlanan pKHR5'e (adım 2.3.7'den itibaren) bağlayın. Bağlı ürünü yetkin hücrelere dönüştürün, bir miniprep kiti kullanarak yükseltin ve saflaştırın. pKHR5 içindeki mVenus YFP muhabir geninin her iki tarafına bağlı her iki şablon segmentini de içeren bu nihai ürünün gliserol stoğunu oluşturun.
- pKHR5 plazmidini, ticari olarak temin edilebilen DH5α bakteri suşundan bakteri kültürlerini yükselterek hazırlayın (ayrıntılar için Malzeme Tablosu'na bakın) yukarıdaki MLM3613 ile aynı şekilde (adım 2.1.1).
- Cas9 mRNA, iki sgRNA ve eksojen HDR muhabir şablonunu kullanarak mikroenjeksiyon karışımını hazırlayın.
- 200 ng/μL Cas9 mRNA, her sgRNA'nın 100 ng/μL ve 200 ng/μL eksojen HDR muhabir şablonunu 1x enjeksiyon tamponunda 20 μL'lik son hacme karıştırın.
- Mikroenjeksiyon karışımını -20 ° C'de saklayın ve kullanılmayan karışımı üç donma-çözülme döngüsünden sonra atın.
NOT: Her embriyonun yumurta sarısı kesesine mikroenjeksiyon için bu mikroenjeksiyon karışımının 4 nL'sini kullanın.
3. Zebra balığı yetiştiriciliği ve embriyo mikroenjeksiyonu
NOT: Zebra balığı yetiştiriciliği ve tek hücreli embriyoların mikroenjeksiyonu için protokoller daha önce tanımlanmıştır 29,30,31.
- Üreme için, AB suşunun zebra balıklarını kullanın ve bunlar 6-12 aylıktır. Embriyoları tek hücreli aşamada döllenme sonrası yaklaşık 40 dakikada enjekte edin (bkz. Şekil 5).
NOT: Enjekte edilen embriyoların biyolojik cinsiyeti bilinmiyordu; cinsel dimorfizm, yaklaşık 3 aylık32 yaşına kadar belirgin değildir.
4. CRISPR-Cas9 tarafından düzenlenmiş larva zebra balıklarının muhabir gen taraması
- Başarılı HDR düzenlemelerini taramak için CRISPR-Cas9 bileşenlerinin mikroenjeksiyonunu takiben zebra balığı larvalarında YFP entegrasyonunu görselleştirin.
- 25 mm'lik bir Petri kabında, 24 zebra balığı larvasını döllenmeden 3 gün sonra (dpf) % 0.3 trikain metan sülfonat (MS-222, HEPES ve sodyum hidroksit ile pH 7.0-7.4'e tamponlanmış) içinde, kendi kendini düzeltme reflekslerini (tipik olarak 1-2 dakika) kaybedene kadar anestezi uygulayın. Anestezi uygulandıktan sonra, her larvayı 24 kuyucuklu bir plakanın ayrı bir kuyucuğuna aktarın.
- GFP / YFP'yi tespit edebilen bir mikroskop kullanarak, her bir larvanın gözünde muhabir gen floresansını tarayın.
- Her larvanın görüntülerini yakalayın ve muhabir gen ekspresyonunun varlığını veya yokluğunu belgeleyin.
5. CRISPR-Cas9 tarafından düzenlenmiş larva zebra balıklarının fenotiplenmesi
- Muhabir gen taramasını takiben, her larva üzerinde kardiyak fenotipleme (kalp atış hızı, perikardiyal boyutlar, EKG) uygulayın. Fenotip, eşit sayıda muhabir gen-pozitif ve -negatif larvadır.
- Larvalar uyuşturulurken kalp atış hızını ve perikardiyal boyutları ölçmek için bir CCD kamera (örneğin, blackfly USB3) ve video ve görüntü kayıt yazılımı (örneğin, Micromanager for ImageJ) kullanın.
- Kalp atış hızını ölçmek için, Micromanager'ı kullanarak, kalbi yakalamak ve diğer yapıları dışlamak için bir ilgi alanı (ROI) oluşturun.
- Videoyu görüntü dizisi olarak ImageJ'ye içe aktarın ve görüntü sayısı altına doğru kare sayısının girildiğinden emin olun.
- Dosya açıldıktan sonra, kalpte bir YG çizmek için dikdörtgen seçim aracını kullanın, ancak diğer hareketli öğeleri hariç tutun ve YG'yi YG yöneticisine kaydedin (analiz | araçları | Yatırım getirisi yöneticisi).
- Yüklemek | eklentileri tıklayın ve kodu varsayılan klasöre yüklemek için kalp atış hızı algoritmasını seçin, ardından eklentiler sekmesinin altındaki eklentiyi seçin. Açılır pencereden dakika başına vuruşları (bpm) kaydedin.
NOT: Görüntü algılama algoritmaları, ventriküler sistolik kasılma ile ilişkili piksel yoğunluğu değişikliklerini tek tek ölçerek kalp atış hızını algılamak için özel olarak yazılmıştır. Kod https://github.com/dpoburko/zFish_HR bulunabilir.
- Perikardiyal kese ve gözlerden birinin etrafında YG'leri serbest çizmek için ImageJ gibi serbest bir araç kullanarak perikardiyal boyutları ölçün. Görüntüyü ImageJ'de açın ve poligon seçim aracını kullanarak önce perikardiyal kesenin etrafına bir YG çizin, bunu adım 5.1.2'deki gibi YG yöneticisine kaydedin ve göz için tekrarlayın. YG yönetimir'de bu iki YG'yi seçin ve ardından ölç'ü tıklayın. Daha sonra her larvadaki göz bölgesine normalleştirilmiş perikardiyal kesenin alanını hesaplamak için her birinin alanını kaydedin.
- Kalp atış hızı ve perikardiyal ölçümleri takiben, bireysel larvalardan EKG'yi kaydedin.
NOT: Zebra balığı EKG'sini kaydetme protokolleri daha önce33,34,35,36 olarak tanımlanmıştır.
6. CRISPR-Cas9 tarafından düzenlenmiş larva zebra balıklarının genotiplendirilmesi
- Fenotipik analizleri takiben, doğru ve kesin HDR gen düzenlemesini doğrulamak için hedef üzerinde ve potansiyel olarak hedef dışı genotipleme yapın.
- HOTShot yöntemi37'yi kullanarak gDNA'yı izole etmek için% 0.3 MS-222 ve kuyruk klipsi içinde her 3 dpf larvayı anestezi altına alın. Eksize edilen her kuyruk klipsini 20 dakika boyunca 95 °C'de 25 mM NaOH'nin 15 μL'sinde inkübe edin. Ardından, 1,5 μL Tris-HCl ile nötralize edin ve 30 s için 13.800 x g'de santrifüj yapın. Ekstrakte edilmiş gDNA içeren süpernatanı koruyun.
- Larvaları E3 ortamında kurtarın ve daha fazla gelişme veya çalışma amaçlanıyorsa bunları konut sistemine geri döndürün.
- Çıkarılan gDNA'yı şablon olarak kullanarak, hedef içi ve potansiyel hedef dışı bölgelerin PCR tabanlı Sanger dizilimini gerçekleştirin.
NOT: İsteğe bağlı: İç içe geçmiş bir PCR yaklaşımı bazı gen bölgeleri için yararlı olabilir. - Hedef üzerindeki primer tasarımının mutasyon bölgesini ve en yakın sgRNA bağlanma bölgesini yakaladığından emin olun. İlgilenilen genle entegrasyonu doğrulamak için yerleştirilen homoloji kolundan ve hedef genden geçişi tespit etmek için ayrı bir dizileme astarı tasarlayın. Adım 1.2.3'te tanımlanan ilk üç potansiyel hedef dışı alanı sıralamak için astarlar tasarlayın.
NOT: Kılavuz tasarım yazılım programları genellikle astarların kullanılmasını önerir, ancak en iyi sonuçları elde etmek için özelleştirme gerekebilir. - Her zebra balığı için tanımlanabilir hedef içi ve dışı genotipleme, kalp atış hızı, perikardiyal boyutlar, EKG fenotiplemesi ve muhabir gen verilerini derleyin.
Sonuçlar
Bu iki sgRNA ekzon replasmanı CRISPR yaklaşımının başarılı kullanımı, zebra balığındaki zkcnh6a geninde LQTS ile ilişkili varyant R56Q'yu tasarlamak için hassas bir düzenlemenin tanıtılması ve basit bir şekilde tespit edilmesiyle vurgulanmaktadır. Şekil 6 , yukarıda tarif edildiği gibi CRISPR bileşenleri ile tek hücreli embriyo aşamasında enjekte edilen temsili bir 3 dpf larvasını göstermektedir. Şekil 6A, YFP mVenus muhabirinin göz merceğindeki gen ekspresyonunun varlığını, başarılı şablon entegrasyonunun olumlu bir muhabiri olarak göstermektedir. Şekil 6B, C, sırasıyla vahşi tip ve muhabir gen pozitif balıkların kuyruk klipsi örneklerinden izole edilen genomik DNA'dan elde edilen Sanger dizileme kromatogramlarını göstermektedir. Muhabir gen pozitif balıkların, R56Q varyantını zkcnh6a'ya tanıtan kesin düzenlemeye, G'den A'ya sahip oldukları bulundu. Genotipleme, YFP muhabiri gen ekspresyonu ile hassas R56Q gen düzenlemesinin varlığı arasında% 100 bir korelasyon gösterdi ve bu floresan tarama aracını doğruladı.
Genle düzenlenmiş zebra balığı larvalarının fenotiplenmesi 3 dpf'de gerçekleştirildi. Şekil 7, vahşi tip ve R56Q geni düzenlenmiş larvalardan temsili sonuçları göstermektedir. Kalp atış hızı, yukarıda açıklandığı gibi video çekimi ile algılandı. Perikardiyal boyutların göz alanı oranı olarak ölçülmesine bir örnek gösterilmiştir (Şekil 7A). Şekil 7B, normalize perikardiyal boyutlara karşı kalp atış hızını çizerek, zebra balığı8,38,39,40'ta kardiyak repolarizasyon bozuklukları ile ilişkili olan artan perikardiyal ödem ile bradikardi eğilimini vurgulamaktadır. Şekil 7C, 3 dpf larvadan EKG kayıtlarının temsili bir örneğini göstermektedir. Standart aralıklar (QT, QRS) ortalama EKG sinyallerinden ölçüldü.

Şekil 1: HDR şablonunun zebra balığı genomuna entegrasyonu. Koyu gri, homoloji kolları; Cas9'un yeniden kesilmesini önlemek için sessiz mutasyona sahip yeşil, sgRNA kılavuz hedefleri; açık gri, hedef ekzon; kırmızı çizgi, nokta mutasyonu; α-kristalin promotörü altında sarı, mVenüs YFP muhabir geni; kesikli çizgiler homolojiyi gösterir. Burada, hedeflenen kesin düzenleme, zkcnh6a geninin ekzon 2'sinde R56Q idi. Kısaltmalar: HDR = homolojiye yönelik onarım; sgRNA = tek kılavuzlu RNA; YFP = sarı floresan protein; DSB = çift sarmallı kopma; WT = vahşi tür. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.

Şekil 2: İki sgRNA CRISPR-Cas9 yaklaşımını kullanarak zebra balığı genlerinde hassas düzenlemeler yapma adımlarının özeti (ilgili protokol adım numaraları parantez içinde belirtilmiştir). Kısaltmalar: sgRNA = tek kılavuzlu RNA; YFP = sarı floresan protein; gDNA = genomik DNA; EKG = elektrokardiyogram; dpf = döllenmeden sonraki günler; MS-222 = trikain metan sülfonat. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
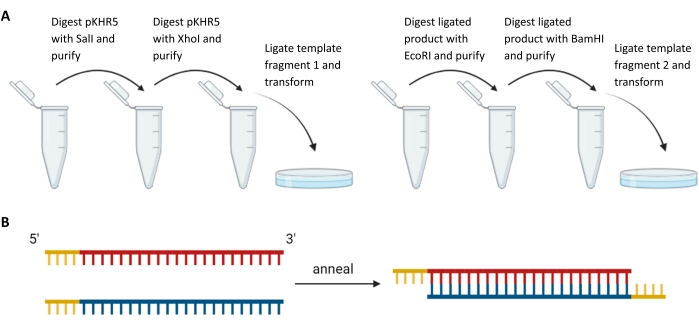
Şekil 3: Eksojen şablon fragmanlarının ve sgRNA kılavuzlarının hazırlanması . (A) pKHR5'te mVenus YFP muhabir gen dizisinin yukarı ve aşağı akış şablon parçalarının sıralı sindirimi ve ligasyonu. (B) Tamamlayıcı sgRNA çiftlerinin DR274'e bağlanma için kısıtlama çıkıntısı ile tavlanması. Kısaltmalar: sgRNA = tek kılavuzlu RNA; YFP = sarı floresan proteini. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
Şekil 4: HDR şablonunun yapımı. Koyu gri, homoloji kolları; Cas9'un yeniden kesilmesini önlemek için sessiz mutasyona sahip yeşil, sgRNA kılavuz hedefleri; açık gri, hedef ekzon; kırmızı çizgi, nokta mutasyonu; α-kristalin promotörü altında sarı, mVenüs YFP muhabir geni; koyu mavi çizgi, kısıtlama siteleri eklendi. İki şablon parçası pKHR5 plazmid donörüne entegre edilmiştir. Kısaltmalar: HDR = homolojiye yönelik onarım; sgRNA = tek kılavuzlu RNA. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.

Şekil 5: Tek hücreli zebra balığı embriyolarının CRISPR-Cas9 bileşenleri ile mikroenjeksiyonu. Ölçek çubuğu = 0,5 mm. Kısaltmalar: HDR = homolojiye yönelik onarım; sgRNA = tek kılavuzlu RNA. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.

Şekil 6: mVenüs YFP muhabir gen floresansının kolay tespiti, hedef gene pozitif HDR eksojen şablon entegrasyonunu gösterir. (A) Düzenlenmiş bir zebra balığı larvasında zebra balığı gözünde (ok) mVenüs YFP ifadesi örneği. (B) Başarılı düzenlemeler, kromatogramların sıralanmasıyla onaylanır (sol, WT; sağ, R56Q düzenleme). Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.

Şekil 7: Zkcnh6a hedef genindeki hassas R56Q düzenlemesini takiben 3 dpf zebra balığında kardiyak sonuçların fenotipik analizi . (A) ImageJ'deki çokgen aracını kullanarak göz boyutuna göre perikardiyal boyutların görüntü tespiti. Perikardiyal kesenin sınırları, yarı saydamlık ve pigmentasyondaki değişikliklere dayanarak kullanıcı tarafından tek bir kayıt çerçevesinden işaretlendi. Normal perikardiyal boyutlara ve perikardiyal efüzyona örnekler gösterilmiştir. Ölçek çubuğu = 0,5 mm. (B) Perikard boyutları (göz boyutuna göre) ile kalp atış hızı arasındaki korelasyon, R2 = 0,33. (C) 3 dpf zebra balığı larva kalbinden (solda) ve ortalama komplekslerden (sağda) EKG kaydı örneği. Kalp atış hızı, 131 bpm; kalp atış hızı düzeltilmiş QTc aralığı, 460 ms. Kısaltmalar: dpf = döllenmeden sonraki günler; EKG = elektrokardiyogram. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
Tartışmalar
CRISPR-Cas9 kullanarak hassas gen düzenlemelerinin mühendisliği, HDR mekanizmalarının düşük verimlilikleri ve verimli tespitleri nedeniyle zorlanmaktadır. Burada, zebra balığında pozitif düzenlemelerin doğrudan görsel olarak algılanmasıyla hassas düzenlemeler üreten CRISPR-Cas9 tabanlı iki sgRNA ekzon değiştirme yaklaşımı açıklanmaktadır. Bu yaklaşımın etkinliği, zkcnh6a geninde kesin düzenlemeler üreterek gösterilmiştir. Bu yazıda, gen düzenlenmiş zebra balığı larvalarında kardiyak fonksiyonun, kalp atış hızı, perikardiyal boyutlar ve EKG morfolojisinin non-invaziv fenotipik ölçümleri kullanılarak nasıl değerlendirilebileceği gösterilmektedir. Bir gen düzenlemesinin tanıtılmasından fenotipik değerlendirmeye kadar olan bu yaklaşım, yaklaşık 1 hafta içinde baştan sona tamamlanabilir.
Yukarıdaki düzenleme ve fenotipleme yaklaşımının faydaları, CRISPR modifikasyon tasarımının kolaylığı, çoklu fizyolojik sistemlerde geniş uygulanabilirlik, büyük genler veya gen parçaları ekleme yeteneği ve varyant etkilerini gelişim ve nesiller boyunca uzunlamasına izleme yeteneğidir. Bu yaklaşımdaki hassas düzenlemelerin başarısı, zebra balığı14'teki düzenlemelerin verimliliğini arttırdığı gösterilen büyük şablon boyutunun (muhabir gen eki ve uzun homoloji kolları nedeniyle) ve zebra balığı TALEN kaynaklı düzenlemelerde etkili bir şekilde kullanılan iki sgRNA kılavuzları stratejisinin kombinasyonu ile ilgiliolabilir.
Tarif edilen yaklaşımın özel bir gücü, büyük genler veya gen parçaları ekleme yeteneğidir. Bu, örneğin, insan ortologları41'i eklemek için yararlı olabilir, bu da daha klinik olarak çevrilebilir karakterizasyon ve ortologlar arasında karşılaştırma sağlar. Alternatif olarak, Cas enzimlerini kodlayan genler de eklenebilir, bu da in vivo CRISPR düzenleme mekanizmalarına sahip bir zebra balığı serisine izin vererek indüklenebilir bir sistem sağlar. Benzer şekilde, ana düzenleme gibi alternatif CRISPR mekanizmaları entegre edilebilir ve hassas ve verimli bir şekilde kolayca düzenlenen bir zebra balığı serisiyle sonuçlanabilir.
Bu yaklaşımın avantajlarına rağmen, bazı sınırlamalar vardır. İlk olarak, sadece tek bir gen ve lokus modifiye edilmiştir ve bu yaklaşımın ne kadar geniş çapta uygulanabilir olduğunu değerlendirmek için diğer bölgelerde veya diğer genlerde daha fazla test yapılması gerekmektedir. Gerekli uzun homoloji kolları nedeniyle, şablon tasarım maliyetleri daha yüksektir; ancak, bu etkili tarama ile dengelenebilir. Diğer bir sınırlama ise tarama yaklaşımının floresan algılama yeteneği gerektirmesidir. Bununla birlikte, optik gereksinimler nispeten düşüktür ve özel olarak üretilebilir veya makul derecede düşük bir maliyetle ticari olarak satın alınabilir. İki sgRNA yaklaşımı kullanmak, potansiyel hedef dışı olayların sayısını artırır; Bununla birlikte, bu, iki sgRNA kılavuzunun her ikisinin de muhabir gen ekspresyonunu sağlamak için şablonun dahil edilmesini kolaylaştıracak şekilde tavlanacağı olasılığının düşük olması ile hafifletilmiştir. Son olarak, Cas9 mRNA'nın kullanılması, Cas9 daha sonraki gelişim aşamalarına kadar aktif olmadığından mozaikliklere yol açabilir. Bu, belirli doku tiplerini sıralayarak açıklanabilir; Bununla birlikte, zebra balığı larvalarının büyüklüğü göz önüne alındığında, bu teknik olarak zordur.
Özetle, zebra balığındaki bu CRISPR-Cas9 iki-sgRNA hassas düzenleme yaklaşımı, pozitif düzenlemelerin basit görsel olarak algılanmasını sağlar ve herhangi bir lokusta ilgilenilen büyük genleri içerecek şekilde uyarlanabilir. Fenotipik önlemlerle birleştirildiğinde, bu, klinik olarak ilgili kardiyak varyantları incelemek için güvenilir ve yüksek verimli bir platform sağlar.
Açıklamalar
Yazarların açıklayacağı bir çıkar çatışması yoktur.
Teşekkürler
Bu araştırma, Kanada Sağlık Araştırma Enstitüleri Proje hibesi (T.W.C.) ve Kanada Keşif hibeleri Doğa Bilimleri ve Mühendislik Araştırma Konseyi (T.W.C.) tarafından desteklenmiştir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Program | |||
| CRISPOR | TEFOR Infrastructure | ||
| ENSEMBL | European Bioinformatics Institute | ||
| ImageJ | National Institutes of Health (NIH) | ||
| Micro-Manager | Open Source (Github) | ||
| NEBiocalculator | New England Biolabs (NEB) | ||
| EQUIPMENT | |||
| 24-well Plate | VWR | ||
| 25 mm Petri Dish | VWR | ||
| Blackfly USB3 Camera | Teledyne FLIR | ||
| C1000 Thermal Cycler | Bio-Rad | ||
| Centrifuge 5415C | Eppendorf | ||
| EZNA Gel Extraction Kit | Omega Biotek | ||
| MAXIscript T7 Transcription Kit | Invitrogen | ||
| MaxQ 5000 Incubator | Barnstead Lab Line | ||
| Miniprep Kit | Qiagen | ||
| mMessage mMachine T7 Ultra Transcription Kit | Invitrogen | ||
| ND1000 Spectrophotometer | Nanodrop | ||
| PCR Purification Kit | Qiagen | ||
| PLI 100A Picoinjector | Harvard Apparatus | ||
| PowerPac Basic Power Supply | Bio-Rad | ||
| Stemi 305 Steroscope | Zeiss | ||
| Wide Mini Sub Cell GT Electrophoresis System | Bio-Rad | ||
| ZebTec Zebrafish Housing System | Tecniplast | ||
| SERVICES | |||
| Gene Synthesis | Genewiz | ||
| Sanger Sequencing | Genewiz | ||
| REAGENTS | |||
| 10β Competent Cells | NEB | ||
| 10X PCR Buffer | Qiagen | ||
| 100 mM Nucleotide Mixture | ABM | ||
| Ampicillin | Sigma | ||
| BamHI Endonuclease w/ buffer | NEB | ||
| BsaI Endonuclease w/ buffer | NEB | ||
| DR274 Plasmid (XL1 Blue bacterial agar stab) | Addgene | ||
| EcoRI Endonuclease w/ buffer | NEB | ||
| Glycerol | |||
| HEPES | Sigma | ||
| HindIII Endonuclease w/ buffer | NEB | ||
| Kanamycin | Sigma | ||
| Methylene Blue | Sigma | ||
| MLM3613 Plasmid (XL1 Blue bacterial agar stab) | Addgene | ||
| MS-222 (Tricaine) | Sigma | ||
| pKHR5 Plasmid (DH5α bacterial agar stab) | Addgene | ||
| PmeI Endonuclease w/ buffer | NEB | ||
| SalI Endonuclease w/ buffer | NEB | ||
| Sodium Hydroxide | Sigma | ||
| T4 Ligase w/ buffer | Sigma | ||
| Taq Polymerase | Qiagen | ||
| TE Buffer | Sigma | ||
| Tris Hydrochloride | Sigma | ||
| XhoI Endonuclease w/ buffer | NEB | ||
| RECIPES | |||
| Solution | Component | Supplier | |
| Annealing Buffer (pH 7.5-8.0) | 10 mM Tris | Sigma | |
| 50 mM NaCl | Sigma | ||
| 1 mM EDTA | Sigma | ||
| E3 Media (pH 7.2) | 5 mM NaCl | Sigma | |
| 0.17 mM KCl | Sigma | ||
| 0.33 mM CaCl2 | Sigma | ||
| 0.33 mM MgSO4 | Sigma | ||
| Injection Buffer (pH 7.5) | 20 mM HEPES | Sigma | |
| 150 mM KCl | Sigma |
Referanslar
- Zarei, A., Razban, V., Hosseini, S. E., Tabei, S. M. B. Creating cell and animal models of human disease by genome editing using CRISPR/Cas9. The Journal of Gene Medicine. 21 (4), 3082(2019).
- Lee, H., Yoon, D. E., Kim, K. Genome editing methods in animal models. Animal Cells and Systems. 24 (1), 8-16 (2020).
- Li, Q., et al. Applications of genome editing technology in animal disease modeling and gene therapy. Computational and Structural Biotechnology Journal. 17, 689-698 (2019).
- Gut, P., Reischauer, S., Stainier, D. Y. R., Arnaout, R. Little fish, big data: Zebrafish as a model for cardiovascular and metabolic disease. Physiological Reviews. 97 (3), 889-938 (2017).
- Kegel, L., et al. Forward genetic screen using zebrafish to identify new genes involved in myelination. Oligodendrocytes: Methods and Protocols. 1936, 185-209 (2019).
- Shah, A. N., Davey, C. F., Whitebirch, A. C., Miller, A. C., Moens, C. B. Rapid reverse genetic screening using CRISPR in zebrafish. Nature Methods. 12 (6), 535-540 (2015).
- González-Rosa, J. M. Zebrafish models of cardiac disease: From fortuitous mutants to precision medicine. Circulation Research. 130 (12), 1803-1826 (2022).
- Hoshijima, K., Jurynec, M. J., Grunwald, D. J. Precise editing of the Zebrafish genome made simple and efficient. Developmental Cell. 36 (6), 654-667 (2016).
- Albadri, S., Del Bene, F., Revenu, C. Genome editing using CRISPR/Cas9-based knock-in approaches in zebrafish. Methods. 121-122, 77-85 (2017).
- Armstrong, G. A. B., et al. Homology directed knockin of point mutations in the zebrafish tardbp and fus genes in ALS using the CRISPR/Cas9 system. PLoS One. 11 (3), 0150188(2016).
- Bai, H., et al. CRISPR/Cas9-mediated precise genome modification by a long ssDNA template in zebrafish. BMC Genomics. 21 (1), 67(2020).
- de Vrieze, E., et al. Efficient generation of knock-in zebrafish models for inherited disorders using CRISPR-Cas9 ribonucleoprotein complexes. International Journal of Molecular Sciences. 22 (17), 9429(2021).
- Eschstruth, A., Schneider-Maunoury, S., Giudicelli, F. Creation of zebrafish knock-in reporter lines in the nefma gene by Cas9-mediated homologous recombination. Genesis. 58 (1), 23340(2020).
- Irion, U., Krauss, J., Nüsslein-Volhard, C. Precise and efficient genome editing in zebrafish using the CRISPR/Cas9 system. Development. 141 (24), 4827-4830 (2014).
- Kimura, Y., Hisano, Y., Kawahara, A., Higashijima, S. Efficient generation of knock-in transgenic zebrafish carrying reporter/driver genes by CRISPR/Cas9-mediated genome engineering. Scientific Reports. 4 (1), 6545(2014).
- Levic, D. S., Yamaguchi, N., Wang, S., Knaut, H., Bagnat, M. Knock-in tagging in zebrafish facilitated by insertion into non-coding regions. Development. 148 (19), (2021).
- Prykhozhij, S. V., et al. Optimized knock-in of point mutations in zebrafish using CRISPR/Cas9. Nucleic Acids Research. 46 (17), 102(2018).
- Wierson, W. A., et al. Efficient targeted integration directed by short homology in zebrafish and mammalian cells. eLife. 9, 53968(2020).
- Boel, A., et al. CRISPR/Cas9-mediated homology-directed repair by ssODNs in zebrafish induces complex mutational patterns resulting from genomic integration of repair-template fragments. Disease Models & Mechanisms. 11 (10), (2018).
- Tessadori, F., et al. Effective CRISPR/Cas9-based nucleotide editing in zebrafish to model human genetic cardiovascular disorders. Disease Models & Mechanisms. 11 (10), (2018).
- Zhang, Y., Zhang, Z., Ge, W. An efficient platform for generating somatic point mutations with germline transmission in the zebrafish by CRISPR/Cas9-mediated gene editing. The Journal of Biological Chemistry. 293 (17), 6611-6622 (2018).
- Vornanen, M., Hassinen, M. Zebrafish heart as a model for human cardiac electrophysiology. Channels. 10 (2), 101-110 (2016).
- Nemtsas, P., Wettwer, E., Christ, T., Weidinger, G., Ravens, U. Adult zebrafish heart as a model for human heart? An electrophysiological study. Journal of Molecular and Cellular Cardiology. 48 (1), 161-171 (2010).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biology. 17 (1), 148(2016).
- Liang, X., Potter, J., Kumar, S., Ravinder, N., Chesnut, J. D. Enhanced CRISPR/Cas9-mediated precise genome editing by improved design and delivery of gRNA, Cas9 nuclease, and donor DNA. Journal of Biotechnology. 241, 136-146 (2017).
- Richardson, C. D., Ray, G. J., DeWitt, M. A., Curie, G. L., Corn, J. E. Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nature Biotechnology. 34 (3), 339-344 (2016).
- Sambrook, J., Fritsch, E. F., Maniatis, T. Precipitation of Large RNAs with Lithium Chloride. Molecular Cloning: A Laboratory Manual, Book 3. E.15. , Cold Spring Harbor Laboratory. Long Island, NY. (1989).
- Sambrook, J., Fritsch, E. F., Maniatis, T. Agarose Gel Electrophoresis. Molecular Cloning: A Laboratory Manual, Book 1. , Cold Spring Harbor Laboratory. Long Island, NY. 3-20 (1989).
- Sorlien, E. L., Witucki, M. A., Ogas, J. Efficient production and identification of CRISPR/Cas9-generated gene knockouts in the model system Danio rerio. Journal of Visualized Experiments. (138), e56969(2018).
- Avdesh, A., et al. Regular care and maintenance of a zebrafish (Danio rerio) laboratory: An introduction. Journal of Visualized Experiments. (69), e4196(2012).
- Rosen, J. N., Sweeney, M. F., Mably, J. D. Microinjection of zebrafish embryos to analyze gene function. Journal of Visualized Experiments. (25), e1115(2009).
- Kossack, M. E., Draper, B. W. Genetic regulation of sex determination and maintenance in zebrafish (Danio rerio). Current Topics in Developmental Biology. 134, 119-149 (2019).
- Tanaka, Y., et al. Functional analysis of KCNH2 gene mutations of type 2 long QT syndrome in larval zebrafish using microscopy and electrocardiography. Heart and Vessels. 34 (1), 159-166 (2019).
- Dhillon, S. S., et al. Optimisation of embryonic and larval ECG measurement in zebrafish for quantifying the effect of QT prolonging drugs. PLoS One. 8 (4), 60552(2013).
- Yu, F., et al. Evolving cardiac conduction phenotypes in developing zebrafish larvae: Implications to drug sensitivity. Zebrafish. 7 (4), 325-331 (2010).
- Hurst, R. M. Development and optimization of tools for embryonic electrocardiograph recording for heart dysfunction in zebrafish. , University of Birmingham. PhD thesis (2018).
- Meeker, N. D., Hutchinson, S. A., Ho, L., Trede, N. S. Method for isolation of PCR-ready genomic DNA from zebrafish tissues. BioTechniques. 43 (5), 610-614 (2007).
- Arnaout, R., et al. Zebrafish model for human long QT syndrome. Proceedings of the National Academy of Sciences of the United States of America. 104 (27), 11316-11321 (2007).
- Milan, D. J., Peterson, T. A., Ruskin, J. N., Peterson, R. T., MacRae, C. A. Drugs that induce repolarization abnormalities cause bradycardia in zebrafish. Circulation. 107 (10), 1355-1358 (2003).
- Langheinrich, U., Vacun, G., Wagner, T. Zebrafish embryos express an orthologue of HERG and are sensitive toward a range of QT-prolonging drugs inducing severe arrhythmia. Toxicology and Applied Pharmacology. 193 (3), 370-382 (2003).
- MacRae, C. A. Cardiac arrhythmia: In vivo screening in the zebrafish to overcome complexity in drug discovery. Expert Opinion on Drug Discovery. 5 (7), 619-632 (2010).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır