Method Article
تعديلات دقيقة بوساطة CRISPR-Cas9 في قلوب الزرد
In This Article
Summary
يصف هذا البروتوكول نهجا لتسهيل التعديلات الدقيقة في أجنة الزرد باستخدام تقنية CRISPR-Cas9. يتم تقديم خط أنابيب التنميط الظاهري لإثبات قابلية تطبيق هذه التقنيات لنمذجة متغير جيني مرتبط بمتلازمة فترة QT الطويلة.
Abstract
تتيح التكرارات العنقودية القصيرة المتباعدة بانتظام (CRISPR) في النماذج الحيوانية التلاعب الجيني الدقيق لدراسة الظواهر الفسيولوجية. تم استخدام الزرد كنموذج وراثي فعال لدراسة العديد من الأسئلة المتعلقة بالأمراض الوراثية والتطور وعلم السموم على مستوى الأعضاء والكائنات الحية بأكملها. نظرا لجينوم سمك الزرد المشروح جيدا والمعين ، تم تطوير العديد من الأدوات لتحرير الجينات. ومع ذلك ، فإن فعالية توليد وسهولة اكتشاف التعديلات الدقيقة باستخدام كريسبر هي عامل مقيد. يوصف هنا نهج قائم على CRISPR-Cas9 مع الكشف البسيط عن التعديلات الدقيقة في الجين المسؤول عن إعادة استقطاب القلب والمرتبط بالاضطراب الكهربائي ، متلازمة فترة QT الطويلة (LQTS). هذا النهج ثنائي الدليل RNA (sgRNA) يستأصل ويحل محل التسلسل المستهدف ويربط جين مراسل مشفر وراثيا. يتم توضيح فائدة هذا النهج من خلال وصف قياسات النمط الظاهري غير الغازية للوظيفة الكهربائية للقلب في يرقات الزرد من النوع البري والمحرر جينيا. يتيح هذا النهج الدراسة الفعالة للمتغيرات المرتبطة بالمرض في كائن حي بأكمله. علاوة على ذلك ، توفر هذه الاستراتيجية إمكانيات لإدخال تسلسلات خارجية من الاختيار ، مثل جينات المراسل ، أو تقويم العظام ، أو محرري الجينات.
Introduction
تتيح استراتيجيات تحرير الجينات القائمة على كريسبر في النماذج الحيوانية دراسة الأمراض الوراثية وراثيا وتطورها وعلم السموم على مستوى الكائن الحي بأكمله1،2،3. يوفر الزرد نموذجا قويا أقرب في العديد من الجوانب الفسيولوجية للبشر من الفئران أو نماذج الخلايا المشتقة من الإنسان4. تم استخدام مجموعة واسعة من الأدوات والاستراتيجيات الجينية في الزرد لكل من الفحص الجيني الأمامي5 والعكسي6. سهلت الخرائط الجينية الشاملة والتعليقات التوضيحية في أسماك الزرد نهج تحرير الجينات كتقنية أولية لهندسة الضربات القاضية الجينية المستهدفة (KOs) والضربات الدقيقة (KIs)7.
على الرغم من ذلك ، فإن إنشاء تعديلات KI دقيقة في الزرد محدود بسبب الكفاءة المنخفضة وصعوبة الكشف الدقيق. على الرغم من أن نوكلياز المستجيب الشبيه بعامل النسخ (TALENs) قد تم استخدامه بنجاح وتحسينه ل KIs8 ، إلا أن CRISPR توفر استراتيجية محسنة لتحرير الجينات مع استهداف sgRNA أبسط. استخدمت العديد من الدراسات كريسبر لتوليد KIs دقيقة في أسماك الزرد9،10،11،12،13،14،15،16،17،18،19،20 ، على الرغم من أن هذه التعديلات التي تم إنشاؤها من خلال الإصلاح الموجه بالتماثل بوساطة كريسبر (HDR) تميل إلى أن تكون غير فعالة مع نجاح جوهري منخفض المعدلات التي تتطلب التنميط الجيني كشاشة أساسية9،10،14،21. يوضح هذا الحاجة إلى نظام KI CRISPR فعال في الزرد ، بالإضافة إلى نظام موثوق عالي الإنتاجية للكشف عن التعديلات الدقيقة.
كان الهدف من هذه الدراسة هو وصف منصة لتوليد جين قلبي دقيق KI في قلوب الزرد مع اكتشاف بسيط وعالي الإنتاجية للتعديلات الناجحة. تم وصف نهج استبدال إكسون ثنائي sgRNA قائم على CRISPR-Cas9 ، والذي يعتمد على نهج TALEN8. يتضمن هذا النهج استئصال التسلسل المستهدف باستخدام دليلين من sgRNA واستبداله بتسلسل قالب خارجي يحتوي على KI محل الاهتمام بالإضافة إلى جين مراسل intronic مشفر وراثيا (الشكل 1). يتيح دمج مراسل الفلورسنت المشفر وراثيا ضمن تسلسل الجين المستهدف الكشف الفعال عن التعديلات الإيجابية. ثم يتم وصف منصة التنميط الظاهري لتقييم الوظيفة الكهربائية للقلب في يرقات الزرد للتوصيف غير الجراحي للمتغيرات الجينية المرتبطة ب LQTS الموروثة ، وهو اضطراب كهربائي في القلب يهيئ الأفراد للموت القلبي المفاجئ.
ستعزز هذه الأساليب الوصول إلى تعديلات جينات KI لسمك الزرد واستخدامها لنمذجة الأمراض الوراثية ومعالجة الأسئلة البيولوجية والفسيولوجية ، مثل رسم خرائط أنماط التعبير الجيني ، والتنظيم التنموي. نظرا لأن قلوب الزرد توازي الخصائص الكهربية للقلب البشري بشكل أفضل من نماذج الفئران ، فقد تكون جذابة بشكل خاص كنظام قابل للتتبع وراثيا لنمذجة أمراض القلب7،22،23.
Protocol
أجريت الدراسات باستخدام الزرد بالاتفاق مع سياسات وإجراءات لجنة رعاية الحيوان بجامعة سيمون فريزر والمجلس الكندي لرعاية الحيوان وتم الانتهاء منها بموجب البروتوكول # 1264K-18.
1. تصميم مكونات كريسبر لإجراء تعديلات دقيقة
- لتصميم أدلة sgRNA ثنائية التي سيتم استخدامها لاستئصال التسلسل الذي يحتوي على موقع هدف KI ، حدد أولا أورثولوج الزرد للجين محل الاهتمام.
ملاحظة: يقدم الشكل 2 نظرة عامة موجزة عن خطوات هندسة عمليات التحرير الدقيقة باستخدام نهج sgRNA CRISPR-Cas9. - بعد ذلك ، استخدم أداة برمجية للتصميم ، مثل CRISPOR24 ، والتي تتضمن اختيار Danio rerio كنوع وإنزيم Cas الذي سيتم استخدامه.
ملاحظة: كان الجين محل الاهتمام لهذه الدراسة هو zkcnh6a (معرف نسخة المجموعة: ENSDART00000090809.6; UniProt Protein ID: B3DJX4) ، وكانت الطفرة المستهدفة هي الأحماض الأمينية ، R56Q.- بالنسبة لنهج sgRNA الثنائي ، اختر موقعا واحدا ل sgRNA يسبق exon المستهدف و sgRNA ثانيا يقع داخل المصب المباشر intron.
- تأكد من أن sgRNAs المحددة لها خصوصية عالية وربط منخفض متوقع خارج الهدف. استخدم تصنيفات كريسبر لتحديد الأدلة ذات الحد الأدنى من الربط خارج الهدف. لا تفكر في الأدلة التي لا تحتوي على عدم تطابق في تسلسل البذور للأهداف المحتملة خارج الأهداف.
- حدد المواقع المحتملة الأكثر احتمالا خارج الهدف (بناء على درجات CRISPOR ، حدد أفضل ثلاثة مواقع محتملة خارج الهدف من Exon) للتنميط الجيني لتسلسل Sanger المستند إلى تفاعل البوليميراز المتسلسل في الخطوة 6.1.3.
- بمجرد اختيار اثنين من sgRNAs ، احصل على المكمل العكسي لكل منهما ، بحيث يكون هناك أربعة قليل النيوكليوتيدات التي سيتم استخدامها: اثنين من oligos التكميلية التي تسبق الطفرة واثنين من oligos التكميلية التي هي في اتجاه مجرى النهر.
- في كل من oligos الأربعة ، أضف مواقع تقييد متوافقة لدمجها في بلازميد دليل من اختيارك ؛ للتكامل في بلازميد DR274 ، استخدم موقع تقييد BsaI 5 'لإنشاء نتوء. تأكد من تصميم موقع التعرف على Bsa1 في الطرف 5 'من الدليل المختار من CRISPOR وأن موقع التعرف Bsa1 مصمم في الطرف 5 'من الخيوط التكميلية لضمان التوجيه الصحيح للأدلة في بلازميد DR274 (انظر الشكل 3).
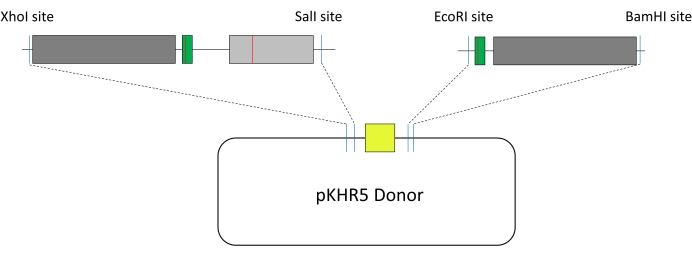
- لتصميم القالب الخارجي المستخدم ل HDR في الزرد (الشكل 1) ، اختر جزأين متتاليين يحيطان بجين مراسل mVenus YFP الموجود في بلازميد pKHR5.
ملاحظة: يمكن تضمين التعديل / التحرير المقصود في جزء المنبع أو المصب.- باستخدام Ensembl ، حدد موقع الموقع المستهدف داخل تسلسل الجينات محل الاهتمام ، بما في ذلك تسلسل المرافقة 2 كيلو بايت تقريبا (ذراع التماثل) ، والذي سيتم استخدامه لصنع القالب.
ملاحظة: قد تكون أذرع التماثل متماثلة أو غير متماثلة25,26 وحوالي 1 كيلو بايت لكل منهما ، في اتجاه المنبع والمصب للموقع المستهدف. - قسم القالب إلى جزأين سيتم إدخالهما على جانبي جين مراسل mVenus YFP (انظر الشكل 4). تأكد من أن موقع الانقسام في intron بحيث لا يتم مقاطعة تسلسل الترميز.
ملاحظة: إذا كان الجين مميزا بشكل جيد ، فتحقق من الأدوار الوظيفية في الإنترون ، مثل مواقع الربط أو المناطق التنظيمية. غالبا ما تشارك المناطق القريبة من نهايات 5 أو 3 في ربط mRNA. - دمج التعديلات في تسلسل القالب التي تشمل i) الطفرات الصامتة في الشكل المجاور للمسافة الأولية (PAM) أو تسلسل البذور (كن على دراية بمواقع PAM البديلة التي قد يستهدفها إنزيم Cas) لمنع إعادة قطع إنزيم Cas ؛ ب) تعديل الفائدة ؛ ج) إنشاء مواقع تقييد نوكلياز لتسهيل الاستنساخ في بلازميد pKHR5 ، الذي يحتوي على جين مراسل mVenus YFP (انظر الشكل 3).
ملاحظة: في هذه الدراسة ، احتوى مقطع القالب الأول على XhoI في المنبع لموقع طفرة R56Q و SalI في اتجاه مجرى النهر ، بينما احتوى الجزء الثاني من القالب على EcoRI في المنبع من تسلسل الهدف الإرشادي و BamHI في اتجاه المصب. إذا كان أي من مواقع التقييد المحددة موجودا ضمن تسلسل القالب ، فستكون هناك حاجة إلى طفرات لإسكاتها ، أو يمكن استخدام طرق بديلة مثل Gibson Assembly.
- باستخدام Ensembl ، حدد موقع الموقع المستهدف داخل تسلسل الجينات محل الاهتمام ، بما في ذلك تسلسل المرافقة 2 كيلو بايت تقريبا (ذراع التماثل) ، والذي سيتم استخدامه لصنع القالب.
2. تحضير مكونات كريسبر للحقن المجهري للجنين
- تحضير Cas9 للحقن المجهري 1 أسبوع قبل الحقن المجهري. استخدم بروتين Cas9 أو قم بإعداد Cas9 mRNA عن طريق النسخ في المختبر .
ملاحظة: في هذه الدراسة ، تم استخدام Cas9 mRNA ، لأن الكفاءة تميل إلى أن تكون أعلى.- تضخيم الثقافات البكتيرية لطعنة أجار بكتيرية XL1 Blue المتاحة تجاريا (تحتوي على بلازميد Cas9) باستخدام مضاد حيوي مناسب مثل الأمبيسلين. استخدم 675 ميكرولتر من المزرعة السائلة (مع 325 ميكرولتر من الجلسرين) لإنشاء مخزون احتياطي من الجلسرين للتخزين طويل الأجل عند -80 درجة مئوية.
- استخدم ما تبقى من المزرعة السائلة لتنقية miniprep ، وفقا للبروتوكول المقدم مع مجموعة miniprep. أعد تعليق الحمض النووي المنقى النهائي في 50 ميكرولتر من مخزن الشطف المقدم. تحديد كمية المنتج عبر مقياس الطيف الضوئي لفحص العائد والنقاء.
- خطي 2 ميكروغرام من الحمض النووي المنقى عن طريق هضم التقييد باستخدام إنزيم تقييد مناسب واستخدام المخزن المؤقت المناسب ووقت الحضانة كما هو مدرج للإنزيم محل الاهتمام.
- قم بتنقية البلازميد الخطي باستخدام مجموعة تنقية PCR ، وقم بتعليقه في 30 ميكرولتر من المخزن المؤقت للشطف المقدم.
- بعد تحديد المنتج ، استخدم هذا كقالب للنسخ في المختبر باستخدام مجموعة النسخ المناسبة للمروج محل الاهتمام. اتبع البروتوكول المقدم وقم بالتنقية عن طريق ترسيب كلوريد الليثيوم27. أعد تعليق الحمض النووي الريبي المنقى في 10 ميكرولتر من H2O الخالي من النيوكلياز وقم بتحديده قبل تخزينه عند -20 درجة مئوية لاستخدامه في مزيج الحقن المجهري.
- قم بإعداد دليلي sgRNA.
- قم بإعداد بلازميد sgRNA بسقالة عن طريق تضخيم الثقافات البكتيرية من طعنة أجار البكتيرية XL1 Blue المتاحة تجاريا (انظر جدول المواد للحصول على التفاصيل) بنفس طريقة MLM3613 أعلاه (الخطوة 2.1.1) ، باستثناء استخدام كاناميسين بدلا من الأمبيسلين.
- قم بتلدين زوجين من قليل النيوكليوتيدات التكميلية أحادية الشريط (ssODNs) لأدلة sgRNA المصممة أعلاه عن طريق إعادة تعليق ssODNs أولا في مخزن مؤقت للتلدين 1x إلى تركيز 100 ميكرومتر.
- في تفاعلات منفصلة لكل من sgRNAs ، قم بتلدين زوج ssODNs التكميلية باستخدام دورة حرارية. امزج 2 ميكروغرام من كل زوج ssODN مكمل مع 50 ميكرولتر من مخزن التلدين المؤقت واحتضانه عند 95 درجة مئوية لمدة دقيقتين ، ثم تبرد إلى 25 درجة مئوية خلال 45 دقيقة.
- هضم البلازميد المتاح تجاريا الذي يحتوي على سقالة gRNA. اهضم 2 ميكروغرام من بلازميد DR274 باستخدام 1 ميكرولتر من BsaI ، و 2 ميكرولتر من المخزن المؤقت المناسب ، و ddH2O إلى 20 ميكرولتر عند 37 درجة مئوية لمدة 1 ساعة. تأكيد الخطية (اختياري: تنقية باستخدام مجموعة تنقية PCR) باستخدام هلام الكهربائي28.
- في تفاعلي ربط منفصلين (واحد لكل sgRNA) ، قم بربط ssODNs الملدنة باستخدام بلازميد DR274 الخطي. استخدم نسبة الإدراج المولي: المتجه 3: 1 ، وحساب الكتلة المناسبة من خلال آلة حاسبة للربط عبر الإنترنت. امزج الكتلة المطلوبة من الإدخال والمتجه مع 1 ميكرولتر من T4 DNA ligase ، و 2 ميكرولتر من مخزن الربط ، و ddH2O إلى 12 ميكرولتر واحتضانها في درجة حرارة الغرفة لمدة 12 ساعة.
- قم بتحويل 2 ميكرولتر من المنتج المربوط إلى خلايا مختصة مناسبة (مثل خلايا 10β) باستخدام الأساليب القياسية ، ثم قم بتضخيم المنتج وتنقيته باستخدام مجموعة Miniprep المتاحة تجاريا. اختياري: قم بإنشاء مخزون الجلسرين لهذا المنتج.
- قم بنسخ sgRNAs عن طريق كتابة 2 ميكروغرام من كل دليل باستخدام موقع تقييد المصب 3 'القريب من نهاية تسلسل الفضاء قدر الإمكان. بالنسبة لبلازميد DR274 ، قم بالكتابة الخطية باستخدام HindIII ، ثم قم بتنقية قالب RNA باستخدام مجموعة تنقية PCR ، مع إعادة التعليق في 30 ميكرولتر من المخزن المؤقت للشطف.
- قم بنسخ الدليلين باستخدام مجموعة نسخ الحمض النووي الريبي. اتبع بروتوكول الشركة المصنعة وقم بالتنقية عن طريق ترسيب كلوريد الليثيوم27. أعد تعليق دليلي sgRNA المنقى في 10 ميكرولتر من H2Oالخالي من النيوكلياز ، وقم بتحديده وتخزينه عند -20 درجة مئوية للاستخدام في مزيج الحقن المجهري.
ملاحظة: لا يمكن أن تتضمن مجموعة نسخ الحمض النووي الريبي غطاء 5 بوصات أو ذيل بولي-أ.
- قم بإعداد قالب مراسل HDR الخارجي المزدوج الذي تقطعت به السبل.
ملاحظة: يتم تصنيع القالب في جزأين ، أحدهما في المنبع والآخر في اتجاه مجرى جين مراسل mVenus YFP. هذان الجزءان عبارة عن تركيبات اصطناعية يتم طلبها من خلال مزود تجاري ثم يتم ربطها في بلازميد pKHR5 (الذي يحتوي على mVenus YFP) بالتتابع.- قم بإعداد بلازميد pKHR5 عن طريق تضخيم الثقافات البكتيرية من السلالة البكتيرية DH5α المتاحة تجاريا (انظر جدول المواد للحصول على التفاصيل) بنفس طريقة MLM3613 أعلاه (الخطوة 2.1.1).
ملاحظة: يحتوي بلازميد pKHR5 على تسلسل جين مراسل mVenus YFP. - أعد تعليق مقطعي القالب المصممان أعلاه إلى 100 ميكرومتر في المخزن المؤقت TE ثم قم بالتحويل إلى خلايا 10β.
- اهضم مقطع القالب الأول (الجزء العلوي من جين مراسل mVenus YFP) وبلازميد pKHR5 باستخدام إنزيمات التقييد المحددة في الخطوة 1.3.3.
ملاحظة: يعد الملخص المتسلسل ل pKHR5 ضروريا نظرا لقرب مواقع التقييد المحددة في MCS. - لتحضير بلازميد pKHR5 لمقطع قالب المنبع ، اهضم 4 ميكروغرام من pKHR5 (وفقا للخطوة 2.2.4) باستخدام SalI ثم قم بالتنقية باستخدام مجموعة تنقية PCR. أعد التعليق في 30 ميكرولتر من ddH2O واستخدم هذا كقالب لتفاعل الهضم الثاني مع XhoI. تنقية المنتج باستخدام مجموعة تنقية PCR.
- قم بإعداد مقطع القالب الأول عن طريق هضم 2 ميكروغرام من مقطع القالب (الخطوة 2.3.2) ، و 1 ميكرولتر من XhoI ، و 1 ميكرولتر من SalI ، و 2 ميكرولتر من المخزن المؤقت المناسب ، و ddH2O إلى 20 ميكرولتر لمدة 1 ساعة عند 37 درجة مئوية وتنقية المنتج الهلام.
- قم بربط مقطع قالب المنبع من الخطوة 2.3.5 إلى pKHR5 المحضر باستخدام ظروف التفاعل الموضحة في الخطوة 2.2.5. قم بتحويل المنتج المربوط إلى خلايا 10β مختصة ، وقم بتضخيمه وتنقيته باستخدام miniprep (اختياري: إنشاء مخزون الجلسرين لهذا المنتج).
- استخدم المنتج المربوط من الخطوة 2.3.6 (التي تحتوي على مقطع القالب الأول المرتبط ببلازميد pKHR5 في المنبع لجين مراسل mVenus YFP) وقم بهضمه للتحضير لمقطع القالب الثاني (المصب لمراسل mVenus). اهضم 4 ميكروغرام من المنتج المربوط (من الخطوة 2.3.6 ، كما في الخطوة 2.2.4) باستخدام BamHI ، ثم قم بالتنقية باستخدام مجموعة تنقية PCR. أعد التعليق في 30 ميكرولتر من ddH2O واستخدم هذا كقالب لتفاعل الهضم الثاني مع EcoRI. تنقية المنتج باستخدام مجموعة تنقية PCR.
- قم بإعداد مقطع القالب الثاني عن طريق هضم 2 ميكروغرام من مقطع القالب (الخطوة 2.3.2) ، و 1 ميكرولتر من BamHI ، و 1 ميكرولتر من EcoRI ، و 2 ميكرولتر من المخزن المؤقت المناسب ، و ddH2O إلى 20 ميكرولتر لمدة 1 ساعة عند 37 درجة مئوية وتنقية المنتج الهلام.
- قم بربط مقطع قالب المصب في pKHR5 المعد (من الخطوة 2.3.7) باستخدام ظروف التفاعل الموضحة في الخطوة 2.2.5. قم بتحويل المنتج المربوط إلى خلايا مختصة ، وقم بتضخيمه وتنقيته باستخدام مجموعة miniprep. قم بإنشاء مخزون الجلسرين لهذا المنتج النهائي ، والذي يحتوي على كلا قطعتي القالب المربوطين على جانبي جين مراسل mVenus YFP داخل pKHR5.
- قم بإعداد بلازميد pKHR5 عن طريق تضخيم الثقافات البكتيرية من السلالة البكتيرية DH5α المتاحة تجاريا (انظر جدول المواد للحصول على التفاصيل) بنفس طريقة MLM3613 أعلاه (الخطوة 2.1.1).
- قم بإعداد مزيج الحقن المجهري باستخدام Cas9 mRNA ، واثنين من sgRNAs ، وقالب مراسل HDR الخارجي.
- امزج 200 نانوغرام / ميكرولتر من Cas9 mRNA ، و 100 نانوغرام / ميكرولتر من كل sgRNA ، و 200 نانوغرام / ميكرولتر من قالب مراسل HDR خارجي المنشأ في 1x مخزن حقن مؤقت إلى حجم نهائي يبلغ 20 ميكرولتر.
- قم بتخزين مزيج الحقن الدقيق عند -20 درجة مئوية وتخلص من المزيج غير المستخدم بعد ثلاث دورات تجميد وذوبان.
ملاحظة: استخدم 4 نانولتر من مزيج الحقن الدقيق هذا للحقن المجهري في كيس الصفار لكل جنين.
3. تربية الزرد والحقن المجهري للجنين
ملاحظة: تم وصف بروتوكولات تربية الزرد والحقن الدقيق للأجنة أحادية الخلية سابقا29،30،31.
- للتكاثر ، استخدم الزرد من سلالة AB والتي تتراوح أعمارها بين 6 و 12 شهرا. حقن الأجنة في مرحلة الخلية الواحدة بعد 40 دقيقة تقريبا من الإخصاب (انظر الشكل 5).
ملاحظة: لم يكن الجنس البيولوجي للأجنة المحقونة معروفا. إزدواج الشكل الجنسي غير واضح حتى حوالي 3 أشهر من سن32.
4. الفحص الجيني للمراسل لسمك الزرد اليرقات المحرر بواسطة CRISPR-Cas9
- تصور تكامل YFP في يرقات الزرد بعد الحقن الدقيق لمكونات CRISPR-Cas9 لفحص تعديلات HDR الناجحة.
- في طبق بتري 25 مم ، قم بتخدير 24 يرقة من يرقات الزرد في 3 أيام بعد الإخصاب (dpf) في 0.3٪ سلفونات الميثان التريكائين (MS-222 ، مخزنة إلى درجة الحموضة 7.0-7.4 مع HEPES وهيدروكسيد الصوديوم) حتى تفقد منعكسها الذاتي (عادة 1-2 دقيقة). بمجرد التخدير ، انقل كل يرقة إلى بئر فردي من صفيحة 24 بئرا.
- باستخدام مجهر قادر على اكتشاف GFP / YFP ، قم بفحص مضان جين المراسل في عيون كل يرقة على حدة.
- التقاط صور لكل يرقة وتوثيق وجود أو عدم وجود التعبير الجيني للمراسل.
5. التنميط الظاهري لسمك الزرد اليرقات المحرر بواسطة CRISPR-Cas9
- بعد الفحص الجيني للمراسل ، قم بإجراء التنميط الظاهري للقلب (معدل ضربات القلب ، أبعاد التامور ، تخطيط القلب) على كل يرقة. النمط الظاهري عدد متساو من يرقات المراسل الإيجابية للجين والسالبة.
- استخدم كاميرا CCD (على سبيل المثال ، Blackfly USB3) وبرنامج تسجيل الفيديو والصور (على سبيل المثال ، Micromanager for ImageJ) لقياس معدل ضربات القلب وأبعاد التامور أثناء تخدير اليرقات.
- لقياس معدل ضربات القلب ، باستخدام Micromanager ، قم بإنشاء منطقة اهتمام (ROI) لالتقاط القلب واستبعاد الهياكل الأخرى.
- قم باستيراد الفيديو إلى ImageJ كتسلسل صورة ، وتأكد من إدخال العدد الصحيح من الإطارات تحت عدد الصور.
- بعد فتح الملف ، استخدم أداة تحديد المستطيل لرسم عائد استثمار داخل القلب ولكن مع استبعاد العناصر المتحركة الأخرى ، وحفظ عائد الاستثمار في مدير عائد الاستثمار (تحليل أدوات | | مدير عائد الاستثمار).
- انقر فوق المكونات الإضافية | التثبيت ، وحدد خوارزمية معدل ضربات القلب لتثبيت الرمز في المجلد الافتراضي ، ثم حدد المكون الإضافي في أسفل علامة تبويب المكونات الإضافية . سجل النبضات في الدقيقة (نبضة في الدقيقة) من النافذة المنبثقة.
ملاحظة: تمت كتابة خوارزميات الكشف عن الصور خصيصا للكشف عن معدل ضربات القلب عن طريق قياس تغيرات كثافة البكسل الفردية المرتبطة بالانقباضي البطيني. يمكن العثور على الرمز في https://github.com/dpoburko/zFish_HR.
- قم بقياس أبعاد التامور باستخدام أداة مجانية مثل ImageJ لرسم عائد الاستثمار الحر حول كيس التامور وإحدى العينين. افتح الصورة في ImageJ واستخدم أداة تحديد المضلع لرسم عائد استثمار أولا حول كيس التامور، وحفظه في مدير عائد الاستثمار كما في الخطوة 5.1.2، وكرر للعين. حدد هذين عائدي الاستثمار في عائد الاستثمار إدارةr، ثم انقر على قياس. سجل المنطقة لكل منها لحساب منطقة كيس التامور الطبيعي في وقت لاحق إلى منطقة العين في كل يرقة.
- بعد معدل ضربات القلب وقياسات التامور ، سجل تخطيط القلب من اليرقات الفردية.
ملاحظة: تم وصف بروتوكولات تسجيل ECG لسمك الزرد سابقا33،34،35،36.
6. التنميط الجيني لسمك الزرد اليرقات المحرر بواسطة CRISPR-Cas9
- بعد تحليلات النمط الظاهري ، قم بإجراء التنميط الجيني المحتمل خارج الهدف لتأكيد التحرير الجيني الدقيق والدقيق ل HDR.
- تخدير كل يرقة 3 dpf في 0.3٪ MS-222 ومشبك الذيل لعزل gDNA باستخدام طريقة HOTShot37. احتضان كل مشبك ذيل مستأصل في 15 ميكرولتر من 25 mM NaOH عند 95 درجة مئوية لمدة 20 دقيقة. بعد ذلك ، قم بتحييد 1.5 ميكرولتر من Tris-HCl وأجهزة الطرد المركزي عند 13800 × جم لمدة 30 ثانية. احتفظ بالمادة الطافية التي تحتوي على gDNA المستخرج.
- استعادة اليرقات في وسائط E3 وإعادتها إلى نظام الإسكان إذا كان هناك مزيد من التطوير أو الدراسة.
- باستخدام gDNA المستخرج كقالب ، قم بإجراء تسلسل Sanger المستند إلى PCR للمواقع المستهدفة والمحتملة خارج الهدف.
ملاحظة: اختياري: قد يكون نهج تفاعل البوليميراز المتسلسل المتداخل مفيدا لبعض مناطق الجينات. - تأكد من أن تصميم التمهيدي المستهدف يلتقط موقع الطفرة وأقرب موقع ربط sgRNA. صمم برايمر تسلسل منفصل للكشف عن الانتقال من ذراع التماثل المدرج والجين المستهدف لتأكيد الاندماج في الجين محل الاهتمام. تصميم البادئات لتسلسل أفضل ثلاثة مواقع محتملة خارج الهدف تم تحديدها في الخطوة 1.2.3.
ملاحظة: غالبا ما تقترح برامج تصميم الدليل مواد أولية لاستخدامها ، ولكن قد يكون التخصيص ضروريا لتحقيق أفضل النتائج. - تجميع التنميط الجيني داخل وخارج الهدف ، ومعدل ضربات القلب ، وأبعاد التامور ، والتنميط الظاهري لتخطيط القلب ، وبيانات الجينات الصحفية التي يمكن تحديدها لكل سمكة زرد.
النتائج
يتم تسليط الضوء على الاستخدام الناجح لنهج كريسون البديل ثنائي الحمض النووي الريبي من خلال الإدخال والكشف البسيط عن تحرير دقيق لهندسة المتغير المرتبط ب LQTS ، R56Q ، في جين zkcnh6a في الزرد. يوضح الشكل 6 3 يرقات تمثيلية من ألياف الحافظة المحقونة في مرحلة الجنين أحادي الخلية بمكونات كريسبر كما هو موضح أعلاه. يوضح الشكل 6A وجود التعبير الجيني لمراسل YFP mVenus في عدسة العين كمراسل إيجابي لتكامل القالب الناجح. يوضح الشكل 6B ، C تسلسل كروماتوجرام سانجر الذي تم الحصول عليه من الحمض النووي الجينومي المعزول من عينات مقطع الذيل للأسماك من النوع البري والأسماك الإيجابية للجين ، على التوالي. تم العثور على الأسماك الإيجابية للجين المراسل لديها التعديل الدقيق ، من G إلى A ، والذي يقدم متغير R56Q في zkcnh6a. أظهر التنميط الجيني وجود علاقة بنسبة 100٪ بين التعبير الجيني لمراسل YFP ووجود تحرير جين R56Q الدقيق ، مما يؤكد صحة أداة فحص التألق هذه.
تم إجراء التنميط الظاهري ليرقات الزرد المعدلة جينيا عند 3 dpf. يوضح الشكل 7 نتائج تمثيلية من اليرقات من النوع البري و R56Q المعدلة جينيا. تم الكشف عن معدل ضربات القلب عن طريق التقاط الفيديو كما هو موضح أعلاه. يظهر مثال على قياس أبعاد التامور كنسبة من مساحة العين (الشكل 7 أ). يرسم الشكل 7B معدل ضربات القلب مقابل أبعاد التامور الطبيعية ، مع تسليط الضوء على اتجاه بطء القلب مع زيادة وذمة التامور ، والتي ترتبط باضطرابات إعادة استقطاب القلب في الزرد8،38،39،40. يوضح الشكل 7C مثالا تمثيليا لتسجيلات تخطيط القلب من 3 يرقات dpf. تم قياس الفواصل الزمنية القياسية (QT ، QRS) من متوسط إشارات تخطيط القلب.

الشكل 1: دمج قالب HDR في جينوم الزرد. رمادي غامق ، أذرع التماثل ؛ الأخضر ، أهداف دليل sgRNA مع طفرة صامتة لمنع إعادة قطع Cas9 ؛ رمادي فاتح ، إكسون الهدف من الاهتمام ؛ الخط الأحمر ، طفرة النقطة ؛ الجين الأصفر ، mVenus YFP المراسل تحت مروج بلوري α ؛ تشير الخطوط المتقطعة إلى التماثل. هنا ، كان التعديل الدقيق المستهدف هو R56Q في exon 2 من جين zkcnh6a . الاختصارات: HDR = إصلاح موجه بالتماثل ؛ sgRNA = الحمض النووي الريبي أحادي الدليل ؛ YFP = بروتين الفلورسنت الأصفر ؛ DSB = كسر مزدوج تقطعت بهم السبل ؛ WT = النوع البري. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.

الشكل 2: ملخص خطوات هندسة التعديلات الدقيقة في جينات الزرد باستخدام نهج sgRNA CRISPR-Cas9 (يشار إلى أرقام خطوات البروتوكول ذات الصلة بين قوسين). الاختصارات: sgRNA = الحمض النووي الريبي أحادي الدليل ؛ YFP = بروتين الفلورسنت الأصفر ؛ gDNA = الحمض النووي الجينومي ؛ تخطيط القلب = مخطط كهربية القلب. DPF = أيام ما بعد الإخصاب ؛ MS-222 = سلفونات الميثان تريكايين. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
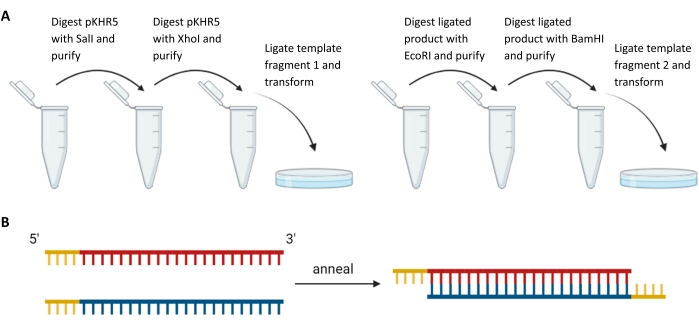
الشكل 3: تحضير أجزاء القالب الخارجية وأدلة sgRNA . (أ) الهضم المتسلسل وربط شظايا القالب في المنبع والمصب لتسلسل جين مراسل mVenus YFP في pKHR5. (ب) تلدين أزواج sgRNA التكميلية مع تراكم التقييد للربط في DR274. الاختصارات: sgRNA = الحمض النووي الريبي أحادي الدليل ؛ YFP = بروتين الفلورسنت الأصفر. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
الشكل 4: بناء قالب HDR. رمادي غامق ، أذرع التماثل ؛ الأخضر ، أهداف دليل sgRNA مع طفرة صامتة لمنع إعادة قطع Cas9 ؛ رمادي فاتح ، إكسون الهدف من الاهتمام ؛ الخط الأحمر ، طفرة النقطة ؛ الجين الأصفر ، mVenus YFP المراسل تحت مروج بلوري α ؛ خط أزرق داكن ، تمت إضافة مواقع التقييد. تم دمج جزأين من القالب في متبرع البلازميد pKHR5. الاختصارات: HDR = إصلاح موجه بالتماثل ؛ sgRNA = الحمض النووي الريبي أحادي الدليل. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.

الشكل 5: الحقن المجهري لأجنة الزرد وحيدة الخلية بمكونات كريسبر-كاس9. شريط المقياس = 0.5 مم. الاختصارات: HDR = إصلاح موجه بالتماثل ؛ sgRNA = الحمض النووي الريبي أحادي الدليل. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.

الشكل 6: يشير الكشف السهل عن مضان جين مراسل mVenus YFP إلى تكامل قالب HDR الخارجي الإيجابي في الجين المستهدف. (أ) مثال على تعبير mVenus YFP في عين الزرد (السهم) في يرقة الزرد المعدلة. (ب) يتم تأكيد التعديلات الناجحة من خلال تسلسل الكروماتوجرام (يسار ، WT ؛ يمين ، تحرير R56Q). الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.

الشكل 7: تحليل النمط الظاهري للعواقب القلبية في 3 أسماك الزرد dpf بعد تحرير R56Q الدقيق في الجين المستهدف zkcnh6a. (أ) الكشف عن صورة أبعاد التامور بالنسبة لحجم العين باستخدام أداة المضلع في ImageJ. تم تمييز حدود كيس التامور من قبل المستخدم من إطار تسجيل واحد بناء على التغيرات في الشفافية والتصبغ. يتم عرض أمثلة على أبعاد التامور الطبيعية وانصباب التامور. شريط المقياس = 0.5 مم. (ب) الارتباط بين أبعاد التامور (بالنسبة إلى بعد العين) ومعدل ضربات القلب ، R2 = 0.33. (ج) مثال على تسجيل تخطيط القلب من قلب يرقة الزرد 3 dpf (يسار) ومتوسط المجمعات (يمين). معدل ضربات القلب ، 131 نبضة في الدقيقة ؛ الفاصل الزمني QTc المصحح لمعدل ضربات القلب ، 460 مللي ثانية. الاختصارات: dpf = أيام ما بعد الإخصاب ؛ تخطيط القلب = مخطط كهربية القلب. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
Discussion
تواجه هندسة التعديلات الجينية الدقيقة باستخدام CRISPR-Cas9 تحديا بسبب الكفاءة المنخفضة لآليات HDR واكتشافها الفعال. هنا ، يتم وصف نهج استبدال إكسون ثنائي sgRNA قائم على CRISPR-Cas9 ينتج تعديلات دقيقة في الزرد مع اكتشاف مرئي مباشر للتعديلات الإيجابية. يتم إثبات فعالية هذا النهج من خلال توليد تعديلات دقيقة في جين zkcnh6a . توضح هذه الورقة كيف يمكن تقييم وظيفة القلب في يرقات الزرد المعدلة جينيا باستخدام مقاييس النمط الظاهري غير الغازية لمعدل ضربات القلب وأبعاد التامور ومورفولوجيا تخطيط القلب. يمكن إكمال هذا النهج ، من إدخال تعديل الجينات إلى تقييم النمط الظاهري ، من البداية إلى النهاية في غضون أسبوع 1 تقريبا.
تتمثل فوائد نهج التحرير والتنميط الظاهري أعلاه في سهولة تصميم تعديل كريسبر ، والتطبيق الواسع في أنظمة فسيولوجية متعددة ، والقدرة على إدخال جينات كبيرة أو شظايا جينية ، والقدرة على تتبع التأثيرات المتغيرة طوليا من خلال التطور والأجيال. قد يكون نجاح التعديلات الدقيقة في هذا النهج مرتبطا بمزيج من حجم القالب الكبير (بسبب إدخال جين المراسل وأذرع التماثل الطويلة) ، والتي ثبت أنها تزيد من كفاءة التعديلات في الزرد14 ، واستراتيجية دليلي sgRNA ، والتي تم استخدامها بشكل فعال في التعديلات التي يسببها الزرد TALEN8.
تتمثل إحدى نقاط القوة الخاصة للنهج الموصوف في القدرة على إدخال جينات كبيرة أو شظايا جينية. قد يكون هذا مفيدا ، على سبيل المثال ، لإدراج orthologsالبشرية 41 ، مما يسمح بتوصيف ومقارنة أكثر قابلية للترجمة سريريا بين orthologs. بدلا من ذلك ، يمكن أيضا إدخال الجينات التي تشفر إنزيمات Cas ، مما يسمح بخط من الزرد مع آليات تحرير CRISPR في الجسم الحي ، مما يوفر نظاما قابلا للتحريض. وبالمثل ، يمكن دمج آليات كريسبر البديلة ، مثل التحرير الأولي ، وينتج عنها خط من الزرد يتم تحريره بسهولة بدقة وكفاءة.
على الرغم من مزايا هذا النهج ، هناك بعض القيود. أولا، لم يتم تعديل سوى جين واحد وموضع واحد، ومن الضروري إجراء المزيد من الاختبارات في مواقع أخرى أو في جينات أخرى لتقييم مدى قابلية تطبيق هذا النهج على نطاق واسع. نظرا لأذرع التماثل الطويلة المطلوبة ، تكون تكاليف تصميم القالب أعلى ؛ ومع ذلك ، قد يتم تعويض ذلك عن طريق الفحص الفعال. قيد آخر هو أن نهج الفحص يتطلب القدرة على الكشف عن مضان. ومع ذلك ، فإن المتطلبات البصرية منخفضة نسبيا ويمكن بناؤها حسب الطلب أو شراؤها تجاريا بتكلفة منخفضة بشكل معقول. يؤدي استخدام نهج sgRNA الثنائي إلى زيادة عدد الأحداث المحتملة خارج الهدف ؛ ومع ذلك ، من المحتمل أن يتم تخفيف ذلك من خلال الاحتمال الأقل بأن دليلي sgRNA سوف يتصلدان بطريقة تسهل دمج القالب لإنتاج التعبير الجيني للمراسل. أخيرا ، قد يؤدي استخدام Cas9 mRNA إلى ظهور فسيفساء لأن Cas9 لا ينشط حتى مراحل النمو اللاحقة. يمكن تفسير ذلك عن طريق تسلسل أنواع معينة من الأنسجة. ومع ذلك ، نظرا لحجم يرقات الزرد ، فإن هذا يمثل تحديا تقنيا.
باختصار ، يتيح نهج التحرير الدقيق CRISPR-Cas9 ثنائي sgRNA في الزرد الكشف البصري البسيط عن التعديلات الإيجابية ويمكن تكييفه لدمج الجينات الكبيرة ذات الأهمية في أي مكان. إلى جانب مقاييس النمط الظاهري ، يسمح هذا بمنصة موثوقة وعالية الإنتاجية لدراسة المتغيرات القلبية ذات الصلة سريريا.
Disclosures
ليس لدى المؤلفين أي تضارب في المصالح للكشف عنه.
Acknowledgements
تم دعم هذا البحث من خلال منحة مشروع المعاهد الكندية للبحوث الصحية (T.W.C.) ومنح مجلس أبحاث العلوم الطبيعية والهندسة في كندا ديسكفري (T.W.C.).
Materials
| Name | Company | Catalog Number | Comments |
| Program | |||
| CRISPOR | TEFOR Infrastructure | ||
| ENSEMBL | European Bioinformatics Institute | ||
| ImageJ | National Institutes of Health (NIH) | ||
| Micro-Manager | Open Source (Github) | ||
| NEBiocalculator | New England Biolabs (NEB) | ||
| EQUIPMENT | |||
| 24-well Plate | VWR | ||
| 25 mm Petri Dish | VWR | ||
| Blackfly USB3 Camera | Teledyne FLIR | ||
| C1000 Thermal Cycler | Bio-Rad | ||
| Centrifuge 5415C | Eppendorf | ||
| EZNA Gel Extraction Kit | Omega Biotek | ||
| MAXIscript T7 Transcription Kit | Invitrogen | ||
| MaxQ 5000 Incubator | Barnstead Lab Line | ||
| Miniprep Kit | Qiagen | ||
| mMessage mMachine T7 Ultra Transcription Kit | Invitrogen | ||
| ND1000 Spectrophotometer | Nanodrop | ||
| PCR Purification Kit | Qiagen | ||
| PLI 100A Picoinjector | Harvard Apparatus | ||
| PowerPac Basic Power Supply | Bio-Rad | ||
| Stemi 305 Steroscope | Zeiss | ||
| Wide Mini Sub Cell GT Electrophoresis System | Bio-Rad | ||
| ZebTec Zebrafish Housing System | Tecniplast | ||
| SERVICES | |||
| Gene Synthesis | Genewiz | ||
| Sanger Sequencing | Genewiz | ||
| REAGENTS | |||
| 10β Competent Cells | NEB | ||
| 10X PCR Buffer | Qiagen | ||
| 100 mM Nucleotide Mixture | ABM | ||
| Ampicillin | Sigma | ||
| BamHI Endonuclease w/ buffer | NEB | ||
| BsaI Endonuclease w/ buffer | NEB | ||
| DR274 Plasmid (XL1 Blue bacterial agar stab) | Addgene | ||
| EcoRI Endonuclease w/ buffer | NEB | ||
| Glycerol | |||
| HEPES | Sigma | ||
| HindIII Endonuclease w/ buffer | NEB | ||
| Kanamycin | Sigma | ||
| Methylene Blue | Sigma | ||
| MLM3613 Plasmid (XL1 Blue bacterial agar stab) | Addgene | ||
| MS-222 (Tricaine) | Sigma | ||
| pKHR5 Plasmid (DH5α bacterial agar stab) | Addgene | ||
| PmeI Endonuclease w/ buffer | NEB | ||
| SalI Endonuclease w/ buffer | NEB | ||
| Sodium Hydroxide | Sigma | ||
| T4 Ligase w/ buffer | Sigma | ||
| Taq Polymerase | Qiagen | ||
| TE Buffer | Sigma | ||
| Tris Hydrochloride | Sigma | ||
| XhoI Endonuclease w/ buffer | NEB | ||
| RECIPES | |||
| Solution | Component | Supplier | |
| Annealing Buffer (pH 7.5-8.0) | 10 mM Tris | Sigma | |
| 50 mM NaCl | Sigma | ||
| 1 mM EDTA | Sigma | ||
| E3 Media (pH 7.2) | 5 mM NaCl | Sigma | |
| 0.17 mM KCl | Sigma | ||
| 0.33 mM CaCl2 | Sigma | ||
| 0.33 mM MgSO4 | Sigma | ||
| Injection Buffer (pH 7.5) | 20 mM HEPES | Sigma | |
| 150 mM KCl | Sigma |
References
- Zarei, A., Razban, V., Hosseini, S. E., Tabei, S. M. B. Creating cell and animal models of human disease by genome editing using CRISPR/Cas9. The Journal of Gene Medicine. 21 (4), 3082(2019).
- Lee, H., Yoon, D. E., Kim, K. Genome editing methods in animal models. Animal Cells and Systems. 24 (1), 8-16 (2020).
- Li, Q., et al. Applications of genome editing technology in animal disease modeling and gene therapy. Computational and Structural Biotechnology Journal. 17, 689-698 (2019).
- Gut, P., Reischauer, S., Stainier, D. Y. R., Arnaout, R. Little fish, big data: Zebrafish as a model for cardiovascular and metabolic disease. Physiological Reviews. 97 (3), 889-938 (2017).
- Kegel, L., et al. Forward genetic screen using zebrafish to identify new genes involved in myelination. Oligodendrocytes: Methods and Protocols. 1936, 185-209 (2019).
- Shah, A. N., Davey, C. F., Whitebirch, A. C., Miller, A. C., Moens, C. B. Rapid reverse genetic screening using CRISPR in zebrafish. Nature Methods. 12 (6), 535-540 (2015).
- González-Rosa, J. M. Zebrafish models of cardiac disease: From fortuitous mutants to precision medicine. Circulation Research. 130 (12), 1803-1826 (2022).
- Hoshijima, K., Jurynec, M. J., Grunwald, D. J. Precise editing of the Zebrafish genome made simple and efficient. Developmental Cell. 36 (6), 654-667 (2016).
- Albadri, S., Del Bene, F., Revenu, C. Genome editing using CRISPR/Cas9-based knock-in approaches in zebrafish. Methods. 121-122, 77-85 (2017).
- Armstrong, G. A. B., et al. Homology directed knockin of point mutations in the zebrafish tardbp and fus genes in ALS using the CRISPR/Cas9 system. PLoS One. 11 (3), 0150188(2016).
- Bai, H., et al. CRISPR/Cas9-mediated precise genome modification by a long ssDNA template in zebrafish. BMC Genomics. 21 (1), 67(2020).
- de Vrieze, E., et al. Efficient generation of knock-in zebrafish models for inherited disorders using CRISPR-Cas9 ribonucleoprotein complexes. International Journal of Molecular Sciences. 22 (17), 9429(2021).
- Eschstruth, A., Schneider-Maunoury, S., Giudicelli, F. Creation of zebrafish knock-in reporter lines in the nefma gene by Cas9-mediated homologous recombination. Genesis. 58 (1), 23340(2020).
- Irion, U., Krauss, J., Nüsslein-Volhard, C. Precise and efficient genome editing in zebrafish using the CRISPR/Cas9 system. Development. 141 (24), 4827-4830 (2014).
- Kimura, Y., Hisano, Y., Kawahara, A., Higashijima, S. Efficient generation of knock-in transgenic zebrafish carrying reporter/driver genes by CRISPR/Cas9-mediated genome engineering. Scientific Reports. 4 (1), 6545(2014).
- Levic, D. S., Yamaguchi, N., Wang, S., Knaut, H., Bagnat, M. Knock-in tagging in zebrafish facilitated by insertion into non-coding regions. Development. 148 (19), (2021).
- Prykhozhij, S. V., et al. Optimized knock-in of point mutations in zebrafish using CRISPR/Cas9. Nucleic Acids Research. 46 (17), 102(2018).
- Wierson, W. A., et al. Efficient targeted integration directed by short homology in zebrafish and mammalian cells. eLife. 9, 53968(2020).
- Boel, A., et al. CRISPR/Cas9-mediated homology-directed repair by ssODNs in zebrafish induces complex mutational patterns resulting from genomic integration of repair-template fragments. Disease Models & Mechanisms. 11 (10), (2018).
- Tessadori, F., et al. Effective CRISPR/Cas9-based nucleotide editing in zebrafish to model human genetic cardiovascular disorders. Disease Models & Mechanisms. 11 (10), (2018).
- Zhang, Y., Zhang, Z., Ge, W. An efficient platform for generating somatic point mutations with germline transmission in the zebrafish by CRISPR/Cas9-mediated gene editing. The Journal of Biological Chemistry. 293 (17), 6611-6622 (2018).
- Vornanen, M., Hassinen, M. Zebrafish heart as a model for human cardiac electrophysiology. Channels. 10 (2), 101-110 (2016).
- Nemtsas, P., Wettwer, E., Christ, T., Weidinger, G., Ravens, U. Adult zebrafish heart as a model for human heart? An electrophysiological study. Journal of Molecular and Cellular Cardiology. 48 (1), 161-171 (2010).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biology. 17 (1), 148(2016).
- Liang, X., Potter, J., Kumar, S., Ravinder, N., Chesnut, J. D. Enhanced CRISPR/Cas9-mediated precise genome editing by improved design and delivery of gRNA, Cas9 nuclease, and donor DNA. Journal of Biotechnology. 241, 136-146 (2017).
- Richardson, C. D., Ray, G. J., DeWitt, M. A., Curie, G. L., Corn, J. E. Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nature Biotechnology. 34 (3), 339-344 (2016).
- Sambrook, J., Fritsch, E. F., Maniatis, T. Precipitation of Large RNAs with Lithium Chloride. Molecular Cloning: A Laboratory Manual, Book 3. E.15. , Cold Spring Harbor Laboratory. Long Island, NY. (1989).
- Sambrook, J., Fritsch, E. F., Maniatis, T. Agarose Gel Electrophoresis. Molecular Cloning: A Laboratory Manual, Book 1. , Cold Spring Harbor Laboratory. Long Island, NY. 3-20 (1989).
- Sorlien, E. L., Witucki, M. A., Ogas, J. Efficient production and identification of CRISPR/Cas9-generated gene knockouts in the model system Danio rerio. Journal of Visualized Experiments. (138), e56969(2018).
- Avdesh, A., et al. Regular care and maintenance of a zebrafish (Danio rerio) laboratory: An introduction. Journal of Visualized Experiments. (69), e4196(2012).
- Rosen, J. N., Sweeney, M. F., Mably, J. D. Microinjection of zebrafish embryos to analyze gene function. Journal of Visualized Experiments. (25), e1115(2009).
- Kossack, M. E., Draper, B. W. Genetic regulation of sex determination and maintenance in zebrafish (Danio rerio). Current Topics in Developmental Biology. 134, 119-149 (2019).
- Tanaka, Y., et al. Functional analysis of KCNH2 gene mutations of type 2 long QT syndrome in larval zebrafish using microscopy and electrocardiography. Heart and Vessels. 34 (1), 159-166 (2019).
- Dhillon, S. S., et al. Optimisation of embryonic and larval ECG measurement in zebrafish for quantifying the effect of QT prolonging drugs. PLoS One. 8 (4), 60552(2013).
- Yu, F., et al. Evolving cardiac conduction phenotypes in developing zebrafish larvae: Implications to drug sensitivity. Zebrafish. 7 (4), 325-331 (2010).
- Hurst, R. M. Development and optimization of tools for embryonic electrocardiograph recording for heart dysfunction in zebrafish. , University of Birmingham. PhD thesis (2018).
- Meeker, N. D., Hutchinson, S. A., Ho, L., Trede, N. S. Method for isolation of PCR-ready genomic DNA from zebrafish tissues. BioTechniques. 43 (5), 610-614 (2007).
- Arnaout, R., et al. Zebrafish model for human long QT syndrome. Proceedings of the National Academy of Sciences of the United States of America. 104 (27), 11316-11321 (2007).
- Milan, D. J., Peterson, T. A., Ruskin, J. N., Peterson, R. T., MacRae, C. A. Drugs that induce repolarization abnormalities cause bradycardia in zebrafish. Circulation. 107 (10), 1355-1358 (2003).
- Langheinrich, U., Vacun, G., Wagner, T. Zebrafish embryos express an orthologue of HERG and are sensitive toward a range of QT-prolonging drugs inducing severe arrhythmia. Toxicology and Applied Pharmacology. 193 (3), 370-382 (2003).
- MacRae, C. A. Cardiac arrhythmia: In vivo screening in the zebrafish to overcome complexity in drug discovery. Expert Opinion on Drug Discovery. 5 (7), 619-632 (2010).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved