Method Article
CRISPR-Cas9 매개 제브라피쉬 하트의 정확한 녹인 편집
요약
이 프로토콜은 CRISPR-Cas9 기술을 사용하여 제브라피쉬 배아에서 정밀한 녹인 편집을 용이하게 하는 접근 방식을 설명합니다. 표현형 파이프라인은 Long QT 증후군 관련 유전자 변이체를 모델링하기 위한 이러한 기술의 적용 가능성을 입증하기 위해 제시됩니다.
초록
동물 모델에서 규칙적으로 간격을 둔 짧은 회문 반복(CRISPR)이 클러스터링되어 생리적 현상 연구를 위한 정확한 유전자 조작이 가능합니다. Zebrafish는 전체 장기 및 유기체 수준에서 유전성 질병, 발달 및 독성학과 관련된 수많은 질문을 연구하는 효과적인 유전 모델로 사용되었습니다. 주석이 잘 달리고 매핑된 제브라피쉬 게놈으로 인해 유전자 편집을 위한 수많은 도구가 개발되었습니다. 그러나 CRISPR을 사용하여 정확한 녹인 편집을 생성하는 효율성과 용이함은 제한 요소입니다. 여기에 설명된 것은 심장 재분극을 담당하고 전기 장애인 LQTS(Long QT 증후군)와 관련된 유전자에서 정밀한 편집을 간단하게 감지하는 CRISPR-Cas9 기반 녹인 접근 방식입니다. 이 2-단일 가이드 RNA(sgRNA) 접근법은 표적 서열을 절제 및 대체하고 유전적으로 암호화된 리포터 유전자를 연결합니다. 이 접근법의 유용성은 야생형 및 유전자 편집 제브라 피쉬 유충에서 심장 전기 기능의 비 침습적 표현형 측정을 설명함으로써 입증됩니다. 이 접근법은 전체 유기체에서 질병 관련 변이체의 효율적인 연구를 가능하게합니다. 또한, 이 전략은 리포터 유전자, 오르토로그 또는 유전자 편집자와 같은 외인성 선택 서열의 삽입 가능성을 제공합니다.
서문
동물 모델에서 CRISPR 기반 유전자 편집 전략을 사용하면 전체 유기체 수준 1,2,3에서 유전적으로 유전되는 질병, 발달 및 독성학을 연구할 수 있습니다. 제브라피쉬는 쥐 또는 인간 유래세포 모델보다 다양한 생리학적 측면에서 인간에게 더 가까운 강력한 모델을 제공합니다4. 광범위한 유전 도구와 전략이 제브라 피쉬에서 전진5 및 역 유전자 스크리닝6 모두에 사용되었습니다. 제브라피쉬의 포괄적인 유전자 매핑 및 주석은 표적 유전자 녹아웃(KO) 및 정밀 녹인(KI)을 엔지니어링하는 주요 기술로서 유전자 편집 접근 방식을 촉진했습니다7.
그럼에도 불구하고 제브라 피쉬에서 정확한 KI 편집을 생성하는 것은 낮은 효율성과 정확한 탐지의 어려움으로 인해 제한됩니다. 전사 인자 유사 이펙터 뉴클레아제(TALENs)가 KI8에 성공적으로 사용되고 최적화되었지만, CRISPR은 더 간단한 sgRNA 표적화로 개선된 유전자 편집 전략을 제공합니다. 수많은 연구에서 CRISPR을 사용하여 제브라피쉬 9,10,11,12,13,14,15,16,17,18,19,20에서 정확한 KI를 생성했지만, CRISPR 매개 상동성 지향 복구(HDR)를 통해 생성된 이러한 편집은 낮은 내재적 성공으로 비효율적인 경향이 있습니다. 유전형 분석을 기본 화면으로 요구하는 비율 9,10,14,21. 이는 제브라피쉬의 효율적인 KI CRISPR 시스템과 정밀한 편집을 감지하기 위한 신뢰할 수 있는 고처리량 시스템의 필요성을 보여줍니다.
이 연구의 목표는 성공적인 편집의 간단하고 높은 처리량 검출을 통해 제브라 피쉬 심장에서 정확한 심장 유전자 KI를 생성하는 플랫폼을 설명하는 것이 었습니다. CRISPR-Cas9 기반 2-sgRNA 엑손 치환 접근법이 설명되며, 이는 TALEN 접근법8에 기초한다. 이 접근법은 2-sgRNA 가이드를 사용하여 표적 서열을 절제하고 관심 있는 KI와 유전적으로 암호화된 인트론 리포터 유전자를 포함하는 외인성 주형 서열로 대체하는 것을 포함합니다(그림 1). 표적 유전자 인트론 서열 내에 유전적으로 암호화된 형광 리포터를 통합하면 양성 편집을 효율적으로 검출할 수 있습니다. 그런 다음 개인을 갑작스런 심장 사망에 걸리게하는 심장 전기 장애 인 유전 된 LQTS와 관련된 유전자 변이체의 비 침습적 특성화를 위해 제브라 피쉬 유충의 심장 전기 기능을 평가하기위한 표현형 플랫폼을 설명합니다.
이러한 접근법은 유전 질환을 모델링하고 유전자 발현 패턴 매핑 및 발달 조절과 같은 생물학적 및 생리학적 문제를 해결하기 위해 제브라피쉬 KI 유전자 편집에 대한 접근 및 사용을 향상시킬 것입니다. 제브라 피쉬 심장은 쥐 모델보다 인간 심장 전기 생리 학적 특성과 더 잘 평행하기 때문에 심장 질환 모델링을위한 유 전적으로 다루기 쉬운 시스템으로서 특히 매력적 일 수 있습니다 7,22,23.
프로토콜
제브라 피쉬를 사용한 연구는 Simon Fraser University 동물 관리위원회와 캐나다 동물 관리위원회의 정책 및 절차에 따라 수행되었으며 프로토콜 # 1264K-18에 따라 완료되었습니다.
1. 정밀한 편집을 위한 CRISPR 부품 설계
- KI 표적 부위를 포함하는 서열을 절제하는 데 사용될 2-sgRNA 가이드를 설계하려면 먼저 관심 유전자에 대한 제브라피쉬 오르토로그를 식별합니다.
참고: 그림 2 는 two-sgRNA CRISPR-Cas9 접근법을 사용하여 정밀한 편집을 엔지니어링하는 단계에 대한 요약 개요를 제공합니다. - 다음으로, CRISPOR24와 같은 설계 소프트웨어 도구를 사용하는데, 여기에는 사용할 Cas 효소의 종으로서의 Danio rerio 에 대한 선택이 포함됩니다.
참고: 이 연구의 관심 유전자는 zkcnh6a (Ensembl 전사체 ID: ENSDART00000090809.6; UniProt 단백질 ID: B3DJX4), 표적 돌연변이는 아미노산, R56Q였다.- 2-sgRNA 접근법의 경우 표적 엑손 앞에 있는 하나의 sgRNA 위치와 바로 다운스트림 인트론 내에 위치한 두 번째 sgRNA를 선택합니다.
- 선택된 sgRNA가 높은 특이성과 낮은 예측된 오프-타겟 결합을 갖도록 한다. CRISPOR 순위를 사용하여 최소한의 오프 타겟 바인딩으로 가이드를 식별합니다. 잠재적인 오프 타겟의 시드 시퀀스에 불일치가 없는 가이드를 고려하지 마십시오.
- 6.1.3단계에서 PCR 기반 Sanger 시퀀싱 유전형 분석에 대해 가장 가능성이 높은 잠재적 표적 외 부위(CRISPOR 점수에 따라 상위 3개의 엑손 잠재적 표적 외 부위 선택)를 식별합니다.
- 2개의 sgRNA가 선택되면 각각에 대한 역방향 보체를 얻어 4개의 올리고뉴클레오티드(돌연변이 앞에 오는 2개의 상보성 올리고와 다운스트림에 있는 2개의 상보성 올리고)가 사용되도록 합니다.
- 4개의 올리고 각각에, 선택한 가이드 플라스미드에 혼입시키기 위해 상용성있는 제한 부위를 추가하고; DR274 플라스미드에 통합하려면 5' BsaI 제한 부위를 사용하여 오버행을 생성합니다. Bsa1 인식 부위가 CRISPOR에서 선택한 가이드의 5' 끝에서 엔지니어링되고 Bsa1 인식 부위가 DR5 플라스미드에서 가이드의 올바른 방향을 보장하기 위해 상보 가닥의 274' 끝에서 설계되었는지 확인합니다( 그림 3 참조).
- 제브라피쉬에서 HDR에 사용되는 외인성 주형을 설계하려면(그림 1), pKHR5 플라스미드에 수용된 mVenus YFP 리포터 유전자의 측면에 있는 두 개의 서열 단편을 선택합니다.
참고: 의도한 수정/편집은 업스트림 또는 다운스트림 조각에 포함될 수 있습니다.- Ensembl을 사용하여, 대략 2 kb 플랭킹 서열(상동성 암)을 포함하는 관심 유전자 서열 내에서 표적 부위를 위치시키고, 이는 주형을 만드는 데 사용될 것이다.
참고: 상동성 암은 대칭 또는 비대칭25,26 및 대략 1kb, 표적 부위의 상류 및 하류일 수 있다. - 주형을 mVenus YFP 리포터 유전자의 양쪽에 삽입될 두 개의 세그먼트로 나눕니다( 그림 4 참조). 코딩 시퀀스가 중단되지 않도록 분할 사이트가 인트론에 있는지 확인하십시오.
참고: 유전자가 잘 특성화되면 접합 부위 또는 조절 영역과 같은 인트론의 기능적 역할을 확인하십시오. 5' 또는 3' 말단에 가까운 영역은 mRNA 스플라이싱에 더 자주 관여합니다. - Cas 효소 재절단을 방지하기 위해 i) 가이드 프로토스페이서 인접 모티프 (PAM) 또는 시드 서열 (Cas 효소가 표적화할 수 있는 대체 PAM 부위를 알고 있어야 함) 내의 침묵 돌연변이를 포함하는 변형을 주형 서열에 통합하고; ii) 이해관계의 수정; iii) mVenus YFP 리포터 유전자를 함유하는 pKHR5 플라스미드 내로의 클로닝을 용이하게 하기 위한 제한 엔도뉴클레아제 부위의 생성( 도 3 참조).
참고: 이 연구에서 첫 번째 템플릿 세그먼트에는 R56Q 돌연변이 부위의 XhoI 업스트림과 SalI 다운스트림이 포함되었으며, 두 번째 템플릿 세그먼트에는 가이드 표적 서열의 상류에 EcoRI 및 BamHI 다운스트림이 있었습니다. 선택된 제한 부위 중 임의의 것이 템플릿 서열 내에 존재하는 경우, 이를 침묵시키기 위한 돌연변이가 필요하거나 Gibson Assembly와 같은 대체 접근법이 사용될 수 있다.
- Ensembl을 사용하여, 대략 2 kb 플랭킹 서열(상동성 암)을 포함하는 관심 유전자 서열 내에서 표적 부위를 위치시키고, 이는 주형을 만드는 데 사용될 것이다.
2. 배아 미세 주입을 위한 CRISPR 성분의 제조
- 미세주사 1주일 전에 미세주입을 위해 Cas9를 준비한다. Cas9 단백질을 사용하거나 시험관 내 전사를 통해 Cas9 mRNA를 준비합니다.
참고: 이 연구에서는 효율성이 더 높은 경향이 있기 때문에 Cas9 mRNA가 사용되었습니다.- 암피실린과 같은 적절한 항생제를 사용하여 시판되는 XL1 Blue 박테리아 한천 찌르기(Cas9 플라스미드 함유)의 박테리아 배양을 증폭합니다. 675μL의 액체 배양(325μL의 글리세롤 포함)을 사용하여 -80°C에서 장기 보관을 위한 백업 글리세롤 스톡을 생성합니다.
- 미니프렙 키트와 함께 제공된 프로토콜에 따라 미니프렙 정제를 위해 나머지 액체 배양액을 사용하십시오. 제공된 용리 완충액의 50 μL에 최종 정제된 DNA를 재현탁시킨다. 분광 광도계를 통해 생성물을 정량화하여 수율과 순도를 검사합니다.
- 적절한 제한 효소를 사용하고 관심 효소에 대해 나열된 적절한 완충액과 배양 시간을 사용하여 제한 분해를 통해 정제된 DNA 2μg을 선형화합니다.
- PCR 정제 키트를 사용하여 선형화된 플라스미드를 정제하고, 제공된 용리 완충액 30μL에 재현탁시킨다.
- 생성물을 정량화한 후, 이것을 관심 프로모터에 대한 적절한 전사 키트를 사용하여 시험관내 전사를 위한 주형으로 사용한다. 제공된 프로토콜을 따르고 염화리튬 침전을 통해 정제하십시오27. 정제된 RNA를 10μL의 뉴클레아제가 없는H2O에 재현탁시키고 미세주입 혼합물에 사용하기 위해 -20°C에서 저장하기 전에 정량화합니다.
- 두 개의 sgRNA 가이드를 준비합니다.
- 암피실린 대신 카나마이신을 사용하는 것을 제외하고는, 상기 MLM3613과 동일한 방법(단계 2.1.1)으로 시판되는 XL1 Blue 박테리아 한천 찌르기(자세한 내용은 재료 표 참조)로부터 박테리아 배양물을 증폭하여 스캐폴드로 sgRNA 플라스미드를 제조한다.
- 상기 설계된 sgRNA 가이드를 위한 두 쌍의 상보적인 단일-가닥 올리고뉴클레오티드(ssODNs)를 먼저 1x 어닐링 완충액에 100μM의 농도로 재현탁시킴으로써 어닐링한다.
- 2개의 sgRNA 각각에 대한 별개의 반응에서, 열 순환기를 사용하여 상보적인 ssODN의 쌍을 어닐링한다. 각 상보성 ssODN 쌍 2μg을 어닐링 완충액 50μL와 혼합하고 95°C에서 2분 동안 배양한 다음 45분에 걸쳐 25°C로 냉각합니다.
- gRNA 스캐폴드를 함유하는 시판되는 플라스미드를 다이렉트한다. 1 μL의 BsaI, 2 μL의 적절한 완충액, 및 ddH2O를 사용하여 2 μg의DR274플라스미드를 37°C에서 20 μL로 1시간 동안 소화시켰다. 겔 전기영동28을 사용하여 선형화 확인(옵션: PCR 정제 키트를 사용하여 정제).
- 두 개의 개별 결찰 반응(각 sgRNA에 대해 하나씩)에서 어닐링된 ssODN을 선형화된 DR274 플라스미드로 결찰합니다. 몰 삽입:벡터 비율을 3:1로 사용하여 온라인 결찰 계산기를 통해 적절한 질량을 계산합니다. 삽입물 및 벡터의 필요한 질량을 1μL의 T4 DNA 리가제, 2μL의 결찰 완충액 및 ddH2O를12μL로 혼합하고 실온에서 12시간 동안 배양합니다.
- 표준 접근법을 사용하여 결찰된 제품 2μL를 적절한 적격 세포(예: 10β 세포)로 형질전환한 다음 시판되는 Miniprep 키트를 사용하여 제품을 증폭 및 정제합니다. 선택 사항 :이 제품의 글리세롤 스톡을 만듭니다.
- 공간 서열의 끝에 가능한 한 가까운 3' 다운스트림 제한 부위를 사용하여 각 가이드의 2μg을 선형화하여 두 sgRNA를 전사합니다. DR274 플라스미드의 경우 HindIII로 선형화한 다음 PCR 정제 키트를 사용하여 RNA 주형을 정제하고 용리 완충액 30μL에 재현탁합니다.
- RNA 전사 키트를 사용하여 두 가이드를 전사합니다. 제조업체의 프로토콜을 따르고 염화리튬 침전을 통해 정제하십시오27. 2개의 정제된 sgRNA 가이드를 10μL의 뉴클레아제가 없는H2O에 재현탁시키고, 정량하고, 미세주입 혼합물에 사용하기 위해 -20°C에서 보관한다.
참고: RNA 전사 키트는 5' 캡 또는 폴리-A 테일을 통합할 수 없습니다.
- 이중 가닥, 외인성 HDR 리포터 템플릿을 준비합니다.
참고: 주형은 mVenus YFP 리포터 유전자의 업스트림 및 다운스트림의 두 부분으로 합성됩니다. 이들 2개의 세그먼트는 상업적 제공자를 통해 주문된 후 순차적으로 pKHR5(mVenus YFP 함유) 플라스미드로 라이게이션된 합성 작제물이다.- 상기 MLM3613과 동일한 방법으로 시판되는 DH5α 박테리아 균주(자세한 내용은 재료 표 참조)로부터 박테리아 배양물을 증폭하여 pKHR5 플라스미드를 제조한다(단계 2.1.1).
참고: pKHR5 플라스미드는 mVenus YFP 리포터 유전자 서열을 포함합니다. - 위에서 설계된 두 개의 템플릿 세그먼트를 TE 버퍼에서 100μM로 재현탁한 다음 10β 세포로 변환합니다.
- 제1 주형 세그먼트 (mVenus YFP 리포터 유전자의 상류) 및 pKHR5 플라스미드를 단계 1.3.3에서 선택된 제한 효소를 사용하여 분해한다.
참고: pKHR5의 순차적 분해는 MCS에서 선택된 제한 부위가 매우 가깝기 때문에 필요합니다. - 업스트림 주형 세그먼트에 대한 pKHR5 플라스미드를 준비하기 위해, SalI로 4μg의 pKHR5(단계 2.2.4에 따라)를 분해한 다음, PCR 정제 키트를 사용하여 정제한다. 30 μL의 ddH2O에 재현탁하고 이것을 XhoI와의 두 번째 소화 반응을위한 템플릿으로 사용하십시오. PCR 정제 키트를 사용하여 제품을 정제합니다.
- 20 μL의 주형 세그먼트(단계 2.3.2), 1 μL의 XhoI, 1 μL의 SalI, 2 μL의 적절한 완충액, 및ddH2O를 20 μL로 37°C에서 1시간 동안 분해하여 제1 주형 세그먼트를 제조하고, 생성물을 겔-정제한다.
- 단계 2.3.5의 상류 템플릿 세그먼트를 단계 2.2.5에 설명된 반응 조건을 사용하여 제조된 pKHR5로 라이게이트한다. 결찰된 제품을 유능한 10β 세포로 변환하고, 증폭하고, miniprep을 사용하여 정제합니다(옵션: 이 제품의 글리세롤 스톡 생성).
- 단계 2.3.6(mVenus YFP 리포터 유전자의 상류에 pKHR5 플라스미드로 라이게이션된 첫 번째 주형 세그먼트를 포함함)의 결찰된 생성물을 사용하고 제2(mVenus 리포터의 하류) 주형 세그먼트를 준비하기 위해 다이제스트한다. BamHI로 4μg의 라이게이션된 생성물(단계 2.2.4에서와 같이 단계 2.3.6에서)을 분해한 다음 PCR 정제 키트를 사용하여 정제합니다. 30 μL의ddH2O에 재현탁하고 이것을 EcoRI를 사용한 두 번째 분해 반응을 위한 템플릿으로 사용합니다. PCR 정제 키트를 사용하여 제품을 정제합니다.
- 2μg의 템플릿 세그먼트(단계 2.3.2), 1μL의 BamHI, 1μL의 EcoRI, 2μL의 적절한 완충액, 및ddH2O를 20μL로 37°C에서 1시간 동안 분해하여 두 번째 템플릿 세그먼트를 준비하고 생성물을 겔-정제한다.
- 단계 2.2.5에 기재된 반응 조건을 사용하여 준비된 pKHR5(단계 2.3.7로부터) 내로 다운스트림 템플릿 세그먼트를 라이게이션한다. 결찰된 제품을 유능한 세포로 변환하고, 증폭하고, 미니프렙 키트를 사용하여 정제합니다. pKHR5 내에서 mVenus YFP 리포터 유전자의 양쪽에 결찰된 두 주형 세그먼트를 모두 포함하는 이 최종 생성물의 글리세롤 스톡을 생성한다.
- 상기 MLM3613과 동일한 방법으로 시판되는 DH5α 박테리아 균주(자세한 내용은 재료 표 참조)로부터 박테리아 배양물을 증폭하여 pKHR5 플라스미드를 제조한다(단계 2.1.1).
- Cas9 mRNA, 2개의 sgRNA 및 외인성 HDR 리포터 템플릿을 사용하여 미세주입 혼합물을 준비합니다.
- 200 ng/μL의 Cas9 mRNA, 100 ng/μL의 각 sgRNA 및 200 ng/μL의 외인성 HDR 리포터 템플릿을 1x 주입 완충액에 혼합하여 최종 부피 20μL까지 혼합합니다.
- 미세 주입 혼합물을 -20 ° C에서 보관하고 세 번의 동결-해동 사이클 후에 사용하지 않은 혼합물을 폐기하십시오.
알림: 이 미세 주입 혼합물 4nL를 사용하여 각 배아의 난황낭에 미세 주입하십시오.
3. 제브라 피쉬와 배아 미세 주사의 번식
참고 : 제브라 피쉬 번식 및 단일 세포 배아의 미세 주입을위한 프로토콜은 이전에 설명되었습니다 29,30,31.
- 번식을 위해서는 6-12 개월 된 AB 균주의 제브라 피쉬를 사용하십시오. 수정 후 약 40 분에 단일 세포 단계에서 배아를 주입합니다 ( 그림 5 참조).
참고: 주입된 배아의 생물학적 성별은 알려지지 않았습니다. 성적 이형 태성은 약 3 개월이 될 때까지 분명하지않습니다.
4. CRISPR-Cas9 편집 유충 제브라피쉬의 리포터 유전자 스크리닝
- 성공적인 HDR 편집을 위해 CRISPR-Cas9 구성 요소를 미세 주입한 후 제브라피쉬 유충의 YFP 통합을 시각화합니다.
- 25mm 페트리 접시에 24마리의 제브라피쉬 유충을 수정 후 3일에 0.3% 트리카인 메탄 설포네이트(MS-222, hepes 및 수산화나트륨으로 pH 7.0-7.4로 완충됨)로 마취하여 자가 교정 반사(일반적으로 1-2분)를 잃을 때까지 마취합니다. 마취되면 각 유충을 24 웰 플레이트의 개별 우물로 옮깁니다.
- GFP/YFP를 검출할 수 있는 현미경을 사용하여 각 유충의 눈에서 리포터 유전자 형광을 스크리닝합니다.
- 각 유충의 이미지를 캡처하고 리포터 유전자 발현의 유무를 문서화합니다.
5. CRISPR-Cas9 편집 유충 제브라피쉬의 표현형 분석
- 리포터 유전자 스크리닝 후 각 유충에 대해 심장 표현형 (심박수, 심낭 치수, ECG)을 수행합니다. 표현형은 동일한 수의 리포터 유전자 양성 및 음성 유충입니다.
- CCD 카메라(예: 블랙플라이 USB3)와 비디오 및 이미지 녹화 소프트웨어(예: ImageJ용 마이크로매니저)를 사용하여 유충이 마취되는 동안 심박수와 심낭 치수를 측정합니다.
- 심박수를 측정하려면 Micromanager를 사용하여 관심 영역(ROI)을 만들어 심장을 캡처하고 다른 구조를 제외합니다.
- 비디오를 이미지 시퀀스로 ImageJ로 가져오고 이미지 수에 올바른 프레임 수가 입력되었는지 확인합니다.
- 파일이 열리면 사각형 선택 도구를 사용하여 다른 움직이는 요소를 제외한 마음 속에 ROI를 그리고 ROI 관리자에 ROI를 저장합니다(| 도구 분석 | ROI 관리자).
- |설치할 플러그인을 클릭하고 심박수 알고리즘을 선택하여 기본 폴더에 코드를 설치한 다음 플러그인 탭 하단에서 플러그인을 선택합니다. 팝업 창에서 분당 비트 수(bpm)를 기록합니다.
알림: 이미지 감지 알고리즘은 심실 수축기 수축과 관련된 개별 픽셀 밀도 변화를 측정하여 심박수를 감지하도록 맞춤 작성되었습니다. 코드는 https://github.com/dpoburko/zFish_HR 에서 찾을 수 있습니다.
- ImageJ와 같은 무료 도구를 사용하여 심낭 치수를 측정하여 심낭과 눈 중 하나 주변의 ROI를 자유롭게 그릴 수 있습니다. ImageJ에서 이미지를 열고 다각형 선택 도구를 사용하여 먼저 심낭 주위에 ROI를 그린 다음 5.1.2단계와 같이 ROI 관리자에 저장하고 눈에 대해 반복합니다. ROI 관리r에서 이 두 ROI를 선택한 다음 측정을 클릭합니다. 나중에 각 유충의 눈 영역으로 정규화 된 심낭의 면적을 계산하기 위해 각각의 면적을 기록하십시오.
- 심박수 및 심낭 측정 후 개별 유충의 ECG를 기록합니다.
참고: 제브라피쉬 ECG를 기록하기 위한 프로토콜은 이전에33,34,35,36에 설명되어 있습니다.
6. CRISPR-Cas9 편집 유충 제브라피쉬의 유전형 분석
- 표현형 분석 후 온-타겟 및 잠재적 오프 타겟 유전형 분석을 수행하여 정확하고 정밀한 HDR 유전자 편집을 확인합니다.
- HOTShot 방법37을 사용하여 gDNA를 분리하기 위해 0.3% MS-222 및 테일 클립에 각각 3개의 dpf 유충을 마취합니다. 절제된 각 테일 클립을 95°C에서 20분 동안 25mM NaOH의 15μL에서 배양합니다. 그런 다음 1.5μL의 Tris-HCl로 중화하고 13,800 x g 에서 30초 동안 원심분리합니다. 추출된 gDNA를 포함하는 상청액을 보유한다.
- E3 배지에서 유충을 회수하고 추가 개발 또는 연구가 의도 된 경우 주택 시스템으로 되돌립니다.
- 추출된 gDNA를 주형으로 사용하여 표적 및 잠재적 표적 외 부위의 PCR 기반 Sanger 시퀀싱을 수행합니다.
참고: 선택 사항: 중첩된 PCR 접근법은 일부 유전자 영역에 유용할 수 있습니다. - 표적 프라이머 설계가 돌연변이 부위와 가장 가까운 sgRNA 결합 부위를 포착하는지 확인합니다. 관심있는 유전자로의 통합을 확인하기 위해 삽입된 상동성 암과 표적 유전자로부터의 전이를 검출하기 위해 별도의 시퀀싱 프라이머를 설계한다. 단계 1.2.3에서 확인된 상위 3개의 잠재적인 오프-타겟 부위를 서열화하도록 프라이머를 설계한다.
참고: 가이드 디자인 소프트웨어 프로그램은 종종 사용할 프라이머를 제안하지만 최적의 결과를 얻으려면 사용자 정의가 필요할 수 있습니다. - 각 제브라피쉬에 대해 식별 가능한 온-오프-타겟 유전형 분석, 심박수, 심낭 치수, ECG 표현형 및 리포터 유전자 데이터를 컴파일합니다.
결과
이 2-sgRNA 엑손 대체 CRISPR 접근법의 성공적인 사용은 제브라피쉬의 zkcnh6a 유전자에서 LQTS 관련 변이체 R56Q를 엔지니어링하기 위한 정밀 편집의 도입 및 간단한 검출로 강조됩니다. 도 6 은 상기와 같이 CRISPR 성분으로 단세포 배아 단계에서 주입된 대표적인 3 dpf 유충을 나타낸다. 도 6A 는 성공적인 주형 통합의 양성 리포터로서 눈 렌즈에서 YFP mVenus 리포터 유전자 발현의 존재를 보여준다. 그림 6B, C 는 각각 야생형 및 리포터 유전자 양성 어류의 꼬리 클립 샘플에서 분리 된 게놈 DNA에서 얻은 Sanger 시퀀싱 크로마토 그램을 보여줍니다. 리포터 유전자 양성 물고기는 Zkcnh6a에 R56Q 변이체를 도입하는 정확한 편집 G to A를 갖는 것으로 밝혀졌습니다. 유전형 분석은 YFP 리포터 유전자 발현과 정확한 R56Q 유전자 편집의 존재 사이에 100% 상관관계를 보여주어 이 형광 스크리닝 도구를 검증했습니다.
유전자 편집 제브라피쉬 유충의 표현형은 3dpf에서 수행되었습니다. 도 7은 야생형 및 R56Q 유전자 편집 유충으로부터의 대표적인 결과를 나타낸다. 심박수는 전술한 바와 같이 비디오 캡처에 의해 검출되었다. 눈 면적의 비로서 심낭 치수를 측정한 예가 도시되어 있다(도 7A). 그림 7B는 정상화 된 심낭 치수에 대한 심박수를 표시하여 제브라 피쉬 8,38,39,40의 심장 재분극 장애와 관련된 심낭 부종이 증가하는 서맥의 추세를 강조합니다. 도 7C는 3개의 dpf 유충으로부터의 ECG 기록의 대표적인 예를 나타낸다. 표준 간격(QT, QRS)은 평균화된 ECG 신호로부터 측정하였다.

그림 1: HDR 템플릿을 제브라피쉬 게놈에 통합. 다크 그레이, 상동성 암; 녹색, Cas9 재절단을 방지하기 위해 조용한 돌연변이가 있는 sgRNA 가이드 표적; 밝은 회색, 관심있는 엑손 대상; 빨간 선, 점 돌연변이; 노란색, α-결정질 프로모터 하의 mVenus YFP 리포터 유전자; 점선은 상 동성을 나타냅니다. 여기서, 표적화된 정밀 편집은 zkcnh6a 유전자의 엑손 2에서 R56Q였다. 약어 : HDR = 상 동성 지향 수리; sgRNA = 단일 가이드 RNA; YFP = 황색 형광 단백질; DSB = 이중 가닥 파손; WT = 야생형. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 2: two-sgRNA CRISPR-Cas9 접근법을 사용하여 제브라피쉬 유전자의 정밀한 편집을 엔지니어링하는 단계 요약(관련 프로토콜 단계 번호는 괄호 안에 표시됨). 약어 : sgRNA = 단일 가이드 RNA; YFP = 황색 형광 단백질; gDNA = 게놈 DNA; 심전도 = 심전도; DPF = 수정 후 일수; MS-222 = 트리카인 메탄설포네이트. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
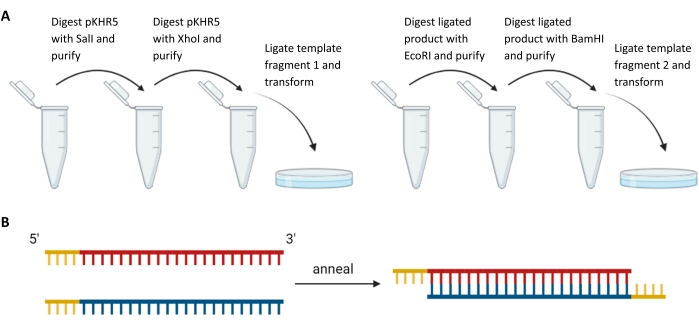
그림 3: 외인성 주형 단편 및 sgRNA 가이드의 준비 . (A) pKHR5에서 mVenus YFP 리포터 유전자 서열의 상류 및 하류의 주형 단편의 순차적 소화 및 결찰. (B) DR274로의 결찰을 위한 제한 오버행이 있는 상보적인 sgRNA 쌍의 어닐링. 약어 : sgRNA = 단일 가이드 RNA; YFP = 황색 형광 단백질. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
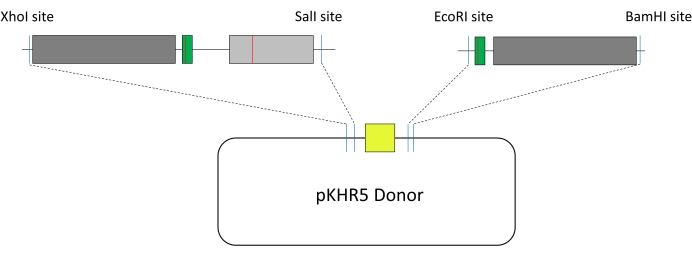
그림 4: HDR 템플릿의 구성. 다크 그레이, 상동성 암; 녹색, Cas9 재절단을 방지하기 위해 조용한 돌연변이가 있는 sgRNA 가이드 표적; 밝은 회색, 관심있는 엑손 대상; 빨간 선, 점 돌연변이; 노란색, α-결정질 프로모터 하의 mVenus YFP 리포터 유전자; 진한 파란색 선, 제한 사이트 추가. 2개의 주형 단편은 pKHR5 플라스미드 공여체에 통합된다. 약어 : HDR = 상 동성 지향 수리; sgRNA = 단일 가이드 RNA. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 5: CRISPR-Cas9 성분이 포함된 단세포 제브라피쉬 배아의 미세 주입. 스케일 바 = 0.5mm. 약어 : HDR = 상 동성 지향 수리; sgRNA = 단일 가이드 RNA. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 6: mVenus YFP 리포터 유전자의 손쉬운 검출 형광은 표적 유전자로의 양성 HDR 외인성 주형 통합을 나타냅니다. (A) 편집된 제브라피쉬 유충의 제브라피쉬 눈(화살표)에서 mVenus YFP 발현의 예. (B) 성공적인 편집은 크로마토 그램 (왼쪽, WT, 오른쪽, R56Q 편집)을 시퀀싱하여 확인됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 7: zkcnh6a 표적 유전자에서 정확한 R56Q 편집 후 3 dpf 제브라피쉬의 심장 결과에 대한 표현형 분석 . (A) ImageJ의 다각형 도구를 사용하여 눈 크기에 대한 심낭 치수의 이미지 감지. 심낭의 경계는 반투명도 및 색소침착의 변화에 기초하여 단일 기록 프레임으로부터 사용자에 의해 표시되었다. 정상적인 심낭 치수 및 심낭 삼출의 예가 표시됩니다. 스케일 바 = 0.5 mm. (B) 심낭 치수 (눈 치수에 비해)와 심박수 간의 상관 관계, R2 = 0.33. (C) 3 dpf 제브라피쉬 유충 심장(왼쪽)과 평균 복합체(오른쪽)로부터의 ECG 기록의 예. 심박수, 131 bpm; 심박수 보정 QTc 간격, 460ms. 약어 : dpf = 수정 후 일수; 심전도 = 심전도. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
CRISPR-Cas9를 사용한 정밀한 유전자 편집 엔지니어링은 HDR 메커니즘의 낮은 효율성과 효율적인 검출로 인해 어려움을 겪고 있습니다. 여기에서는 양성 편집을 직접 시각적으로 감지하여 제브라피쉬에서 정밀한 편집을 생성하는 CRISPR-Cas9 기반 2-sgRNA 엑손 대체 접근법에 대해 설명합니다. 이 접근법의 효능은 zkcnh6a 유전자에서 정확한 편집을 생성함으로써 입증됩니다. 이 논문은 유전자 편집 제브라 피쉬 유충의 심장 기능이 심박수, 심낭 치수 및 ECG 형태의 비 침습적 표현형 측정을 사용하여 어떻게 평가 될 수 있는지 보여줍니다. 유전자 편집 도입에서 표현형 평가에 이르기까지이 접근법은 약 1 주일 이내에 처음부터 끝까지 완료 될 수 있습니다.
위의 편집 및 표현형 접근법의 이점은 CRISPR 변형 설계의 용이성, 여러 생리학적 시스템에서의 광범위한 적용 가능성, 큰 유전자 또는 유전자 단편을 삽입할 수 있는 능력, 개발 및 세대를 통해 종단적으로 변이 효과를 추적하는 능력입니다. 이 접근법에서 정밀한 편집의 성공은 제브라피쉬14에서 편집의 효율성을 증가시키는 것으로 밝혀진 큰 주형 크기(리포터 유전자 삽입물과 긴 상동성 팔로 인해)와 제브라피쉬 탈렌 유도 편집8에 효과적으로 사용된 2-sgRNA-가이드 전략의 조합과 관련이 있을 수 있다.
설명된 접근법의 한 가지 특별한 강점은 큰 유전자 또는 유전자 단편을 삽입하는 능력이다. 이는 예를 들어, 인간 orthologs41을 삽입하는 데 유용할 수 있으며, 이는 ortholog 간의 보다 임상적으로 번역가능한 특성화 및 비교를 허용한다. 대안적으로, Cas 효소를 암호화하는 유전자가 또한 삽입될 수 있어서, 생체 내 CRISPR 편집 메커니즘을 갖는 제브라피쉬 계통을 허용하여, 유도성 시스템을 제공할 수 있다. 마찬가지로, 프라임 편집과 같은 대체 CRISPR 메커니즘이 통합되어 정확하고 효율적으로 쉽게 편집되는 제브라피쉬 라인을 만들 수 있습니다.
이 방법의 장점에도 불구하고 몇 가지 제한 사항이 있습니다. 첫째, 단일 유전자와 유전자좌 만 변형되었으며,이 접근법이 얼마나 광범위하게 적용 가능한지 평가하기 위해서는 다른 부위 또는 다른 유전자에서 추가 테스트가 필요합니다. 긴 상동성 암이 필요하기 때문에 템플릿 디자인 비용이 더 높습니다. 그러나 이는 효율적인 스크리닝으로 상쇄될 수 있습니다. 또 다른 한계는 스크리닝 접근법이 형광 검출 능력을 요구한다는 것입니다. 그러나 광학 요구 사항은 상대적으로 낮으며 합리적으로 저렴한 비용으로 맞춤 제작하거나 상업적으로 구입할 수 있습니다. 2-sgRNA 접근법을 사용하면 잠재적인 오프 타겟 이벤트의 수가 증가합니다. 그러나, 이는 2개의 sgRNA 가이드가 리포터 유전자 발현을 산출하기 위해 주형의 혼입을 용이하게 하는 방식으로 둘 다 어닐링될 가능성이 낮기 때문에 완화될 가능성이 높다. 마지막으로, Cas9 mRNA를 사용하면 Cas9가 후기 발달 단계까지 활성화되지 않기 때문에 모자이크가 발생할 수 있습니다. 이것은 특정 조직 유형을 시퀀싱하여 설명 할 수 있습니다. 그러나 제브라 피쉬 유충의 크기를 감안할 때 이것은 기술적으로 어렵습니다.
요약하면, 제브라피쉬에서 이 CRISPR-Cas9 2-sgRNA 정밀 편집 접근 방식은 양성 편집의 간단한 시각적 감지를 가능하게 하고 모든 유전자좌에서 관심 있는 큰 유전자를 통합하도록 조정될 수 있습니다. 표현형 측정과 결합하면 임상적으로 관련된 심장 변이체를 연구하기 위한 신뢰할 수 있고 처리량이 높은 플랫폼이 가능합니다.
공개
저자는 공개 할 이해 상충이 없습니다.
감사의 말
이 연구는 캐나다 보건원 연구 프로젝트 보조금 (TWC)과 캐나다 디스커버리 보조금의 자연 과학 및 공학 연구위원회 (TWC)의 지원을 받았습니다.
자료
| Name | Company | Catalog Number | Comments |
| Program | |||
| CRISPOR | TEFOR Infrastructure | ||
| ENSEMBL | European Bioinformatics Institute | ||
| ImageJ | National Institutes of Health (NIH) | ||
| Micro-Manager | Open Source (Github) | ||
| NEBiocalculator | New England Biolabs (NEB) | ||
| EQUIPMENT | |||
| 24-well Plate | VWR | ||
| 25 mm Petri Dish | VWR | ||
| Blackfly USB3 Camera | Teledyne FLIR | ||
| C1000 Thermal Cycler | Bio-Rad | ||
| Centrifuge 5415C | Eppendorf | ||
| EZNA Gel Extraction Kit | Omega Biotek | ||
| MAXIscript T7 Transcription Kit | Invitrogen | ||
| MaxQ 5000 Incubator | Barnstead Lab Line | ||
| Miniprep Kit | Qiagen | ||
| mMessage mMachine T7 Ultra Transcription Kit | Invitrogen | ||
| ND1000 Spectrophotometer | Nanodrop | ||
| PCR Purification Kit | Qiagen | ||
| PLI 100A Picoinjector | Harvard Apparatus | ||
| PowerPac Basic Power Supply | Bio-Rad | ||
| Stemi 305 Steroscope | Zeiss | ||
| Wide Mini Sub Cell GT Electrophoresis System | Bio-Rad | ||
| ZebTec Zebrafish Housing System | Tecniplast | ||
| SERVICES | |||
| Gene Synthesis | Genewiz | ||
| Sanger Sequencing | Genewiz | ||
| REAGENTS | |||
| 10β Competent Cells | NEB | ||
| 10X PCR Buffer | Qiagen | ||
| 100 mM Nucleotide Mixture | ABM | ||
| Ampicillin | Sigma | ||
| BamHI Endonuclease w/ buffer | NEB | ||
| BsaI Endonuclease w/ buffer | NEB | ||
| DR274 Plasmid (XL1 Blue bacterial agar stab) | Addgene | ||
| EcoRI Endonuclease w/ buffer | NEB | ||
| Glycerol | |||
| HEPES | Sigma | ||
| HindIII Endonuclease w/ buffer | NEB | ||
| Kanamycin | Sigma | ||
| Methylene Blue | Sigma | ||
| MLM3613 Plasmid (XL1 Blue bacterial agar stab) | Addgene | ||
| MS-222 (Tricaine) | Sigma | ||
| pKHR5 Plasmid (DH5α bacterial agar stab) | Addgene | ||
| PmeI Endonuclease w/ buffer | NEB | ||
| SalI Endonuclease w/ buffer | NEB | ||
| Sodium Hydroxide | Sigma | ||
| T4 Ligase w/ buffer | Sigma | ||
| Taq Polymerase | Qiagen | ||
| TE Buffer | Sigma | ||
| Tris Hydrochloride | Sigma | ||
| XhoI Endonuclease w/ buffer | NEB | ||
| RECIPES | |||
| Solution | Component | Supplier | |
| Annealing Buffer (pH 7.5-8.0) | 10 mM Tris | Sigma | |
| 50 mM NaCl | Sigma | ||
| 1 mM EDTA | Sigma | ||
| E3 Media (pH 7.2) | 5 mM NaCl | Sigma | |
| 0.17 mM KCl | Sigma | ||
| 0.33 mM CaCl2 | Sigma | ||
| 0.33 mM MgSO4 | Sigma | ||
| Injection Buffer (pH 7.5) | 20 mM HEPES | Sigma | |
| 150 mM KCl | Sigma |
참고문헌
- Zarei, A., Razban, V., Hosseini, S. E., Tabei, S. M. B. Creating cell and animal models of human disease by genome editing using CRISPR/Cas9. The Journal of Gene Medicine. 21 (4), 3082(2019).
- Lee, H., Yoon, D. E., Kim, K. Genome editing methods in animal models. Animal Cells and Systems. 24 (1), 8-16 (2020).
- Li, Q., et al. Applications of genome editing technology in animal disease modeling and gene therapy. Computational and Structural Biotechnology Journal. 17, 689-698 (2019).
- Gut, P., Reischauer, S., Stainier, D. Y. R., Arnaout, R. Little fish, big data: Zebrafish as a model for cardiovascular and metabolic disease. Physiological Reviews. 97 (3), 889-938 (2017).
- Kegel, L., et al. Forward genetic screen using zebrafish to identify new genes involved in myelination. Oligodendrocytes: Methods and Protocols. 1936, 185-209 (2019).
- Shah, A. N., Davey, C. F., Whitebirch, A. C., Miller, A. C., Moens, C. B. Rapid reverse genetic screening using CRISPR in zebrafish. Nature Methods. 12 (6), 535-540 (2015).
- González-Rosa, J. M. Zebrafish models of cardiac disease: From fortuitous mutants to precision medicine. Circulation Research. 130 (12), 1803-1826 (2022).
- Hoshijima, K., Jurynec, M. J., Grunwald, D. J. Precise editing of the Zebrafish genome made simple and efficient. Developmental Cell. 36 (6), 654-667 (2016).
- Albadri, S., Del Bene, F., Revenu, C. Genome editing using CRISPR/Cas9-based knock-in approaches in zebrafish. Methods. 121-122, 77-85 (2017).
- Armstrong, G. A. B., et al. Homology directed knockin of point mutations in the zebrafish tardbp and fus genes in ALS using the CRISPR/Cas9 system. PLoS One. 11 (3), 0150188(2016).
- Bai, H., et al. CRISPR/Cas9-mediated precise genome modification by a long ssDNA template in zebrafish. BMC Genomics. 21 (1), 67(2020).
- de Vrieze, E., et al. Efficient generation of knock-in zebrafish models for inherited disorders using CRISPR-Cas9 ribonucleoprotein complexes. International Journal of Molecular Sciences. 22 (17), 9429(2021).
- Eschstruth, A., Schneider-Maunoury, S., Giudicelli, F. Creation of zebrafish knock-in reporter lines in the nefma gene by Cas9-mediated homologous recombination. Genesis. 58 (1), 23340(2020).
- Irion, U., Krauss, J., Nüsslein-Volhard, C. Precise and efficient genome editing in zebrafish using the CRISPR/Cas9 system. Development. 141 (24), 4827-4830 (2014).
- Kimura, Y., Hisano, Y., Kawahara, A., Higashijima, S. Efficient generation of knock-in transgenic zebrafish carrying reporter/driver genes by CRISPR/Cas9-mediated genome engineering. Scientific Reports. 4 (1), 6545(2014).
- Levic, D. S., Yamaguchi, N., Wang, S., Knaut, H., Bagnat, M. Knock-in tagging in zebrafish facilitated by insertion into non-coding regions. Development. 148 (19), (2021).
- Prykhozhij, S. V., et al. Optimized knock-in of point mutations in zebrafish using CRISPR/Cas9. Nucleic Acids Research. 46 (17), 102(2018).
- Wierson, W. A., et al. Efficient targeted integration directed by short homology in zebrafish and mammalian cells. eLife. 9, 53968(2020).
- Boel, A., et al. CRISPR/Cas9-mediated homology-directed repair by ssODNs in zebrafish induces complex mutational patterns resulting from genomic integration of repair-template fragments. Disease Models & Mechanisms. 11 (10), (2018).
- Tessadori, F., et al. Effective CRISPR/Cas9-based nucleotide editing in zebrafish to model human genetic cardiovascular disorders. Disease Models & Mechanisms. 11 (10), (2018).
- Zhang, Y., Zhang, Z., Ge, W. An efficient platform for generating somatic point mutations with germline transmission in the zebrafish by CRISPR/Cas9-mediated gene editing. The Journal of Biological Chemistry. 293 (17), 6611-6622 (2018).
- Vornanen, M., Hassinen, M. Zebrafish heart as a model for human cardiac electrophysiology. Channels. 10 (2), 101-110 (2016).
- Nemtsas, P., Wettwer, E., Christ, T., Weidinger, G., Ravens, U. Adult zebrafish heart as a model for human heart? An electrophysiological study. Journal of Molecular and Cellular Cardiology. 48 (1), 161-171 (2010).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biology. 17 (1), 148(2016).
- Liang, X., Potter, J., Kumar, S., Ravinder, N., Chesnut, J. D. Enhanced CRISPR/Cas9-mediated precise genome editing by improved design and delivery of gRNA, Cas9 nuclease, and donor DNA. Journal of Biotechnology. 241, 136-146 (2017).
- Richardson, C. D., Ray, G. J., DeWitt, M. A., Curie, G. L., Corn, J. E. Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nature Biotechnology. 34 (3), 339-344 (2016).
- Sambrook, J., Fritsch, E. F., Maniatis, T. Precipitation of Large RNAs with Lithium Chloride. Molecular Cloning: A Laboratory Manual, Book 3. E.15. , Cold Spring Harbor Laboratory. Long Island, NY. (1989).
- Sambrook, J., Fritsch, E. F., Maniatis, T. Agarose Gel Electrophoresis. Molecular Cloning: A Laboratory Manual, Book 1. , Cold Spring Harbor Laboratory. Long Island, NY. 3-20 (1989).
- Sorlien, E. L., Witucki, M. A., Ogas, J. Efficient production and identification of CRISPR/Cas9-generated gene knockouts in the model system Danio rerio. Journal of Visualized Experiments. (138), e56969(2018).
- Avdesh, A., et al. Regular care and maintenance of a zebrafish (Danio rerio) laboratory: An introduction. Journal of Visualized Experiments. (69), e4196(2012).
- Rosen, J. N., Sweeney, M. F., Mably, J. D. Microinjection of zebrafish embryos to analyze gene function. Journal of Visualized Experiments. (25), e1115(2009).
- Kossack, M. E., Draper, B. W. Genetic regulation of sex determination and maintenance in zebrafish (Danio rerio). Current Topics in Developmental Biology. 134, 119-149 (2019).
- Tanaka, Y., et al. Functional analysis of KCNH2 gene mutations of type 2 long QT syndrome in larval zebrafish using microscopy and electrocardiography. Heart and Vessels. 34 (1), 159-166 (2019).
- Dhillon, S. S., et al. Optimisation of embryonic and larval ECG measurement in zebrafish for quantifying the effect of QT prolonging drugs. PLoS One. 8 (4), 60552(2013).
- Yu, F., et al. Evolving cardiac conduction phenotypes in developing zebrafish larvae: Implications to drug sensitivity. Zebrafish. 7 (4), 325-331 (2010).
- Hurst, R. M. Development and optimization of tools for embryonic electrocardiograph recording for heart dysfunction in zebrafish. , University of Birmingham. PhD thesis (2018).
- Meeker, N. D., Hutchinson, S. A., Ho, L., Trede, N. S. Method for isolation of PCR-ready genomic DNA from zebrafish tissues. BioTechniques. 43 (5), 610-614 (2007).
- Arnaout, R., et al. Zebrafish model for human long QT syndrome. Proceedings of the National Academy of Sciences of the United States of America. 104 (27), 11316-11321 (2007).
- Milan, D. J., Peterson, T. A., Ruskin, J. N., Peterson, R. T., MacRae, C. A. Drugs that induce repolarization abnormalities cause bradycardia in zebrafish. Circulation. 107 (10), 1355-1358 (2003).
- Langheinrich, U., Vacun, G., Wagner, T. Zebrafish embryos express an orthologue of HERG and are sensitive toward a range of QT-prolonging drugs inducing severe arrhythmia. Toxicology and Applied Pharmacology. 193 (3), 370-382 (2003).
- MacRae, C. A. Cardiac arrhythmia: In vivo screening in the zebrafish to overcome complexity in drug discovery. Expert Opinion on Drug Discovery. 5 (7), 619-632 (2010).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유