Method Article
Edições de knock-in precisas mediadas por CRISPR-Cas9 em corações de peixe-zebra
Neste Artigo
Resumo
Este protocolo descreve uma abordagem para facilitar edições precisas em embriões de peixe-zebra usando a tecnologia CRISPR-Cas9. Um pipeline de fenotipagem é apresentado para demonstrar a aplicabilidade dessas técnicas para modelar uma variante gênica associada à Síndrome do QT Longo.
Resumo
Repetições palindrômicas curtas agrupadas regularmente interespaçadas (CRISPR) em modelos animais permitem manipulação genética precisa para o estudo de fenômenos fisiológicos. O peixe-zebra tem sido usado como um modelo genético eficaz para estudar inúmeras questões relacionadas a doenças hereditárias, desenvolvimento e toxicologia no nível de todo o órgão e organismo. Devido ao genoma do peixe-zebra bem anotado e mapeado, inúmeras ferramentas para edição de genes foram desenvolvidas. No entanto, a eficácia de gerar e a facilidade de detectar edições precisas usando CRISPR é um fator limitante. Descrita aqui está uma abordagem knock-in baseada em CRISPR-Cas9 com a simples detecção de edições precisas em um gene responsável pela repolarização cardíaca e associado ao distúrbio elétrico, Síndrome do QT Longo (SQTL). Esta abordagem de RNA de guia único (sgRNA) extirpa e substitui a sequência alvo e liga um gene repórter geneticamente codificado. A utilidade dessa abordagem é demonstrada pela descrição de medidas fenotípicas não invasivas da função elétrica cardíaca em larvas de peixe-zebra do tipo selvagem e editadas por genes. Esta abordagem permite o estudo eficiente de variantes associadas à doença em todo um organismo. Além disso, essa estratégia oferece possibilidades para a inserção de sequências exógenas de escolha, como genes repórteres, ortólogos ou editores de genes.
Introdução
As estratégias de edição de genes baseadas em CRISPR em modelos animais permitem o estudo de doenças, desenvolvimento e toxicologia geneticamente hereditárias no nível de todo o organismo 1,2,3. O peixe-zebra fornece um modelo poderoso que está mais próximo em numerosos aspectos fisiológicos dos seres humanos do que os modelos celulares murinos ou derivados de humanos4. Uma extensa gama de ferramentas e estratégias genéticas tem sido usada no peixe-zebra tanto para triagem genética direta5 quanto reversa6. O mapeamento genético abrangente e a anotação em peixes-zebra facilitaram abordagens de edição de genes como uma técnica primária para projetar knockouts de genes direcionados (KOs) e knock-ins precisos (KIs)7.
Apesar disso, a geração de edições precisas de KI no peixe-zebra é limitada pela baixa eficiência e pela dificuldade de detecção precisa. Embora as nucleases efetoras semelhantes a fatores de transcrição (TALENs) tenham sido usadas e otimizadas com sucesso para KIs8, o CRISPR fornece uma estratégia aprimorada de edição de genes com direcionamento de sgRNA mais simples. Numerosos estudos têm utilizado a CRISPR para gerar KIs precisas em peixes-zebra 9,10,11,12,13,14,15,16,17,18,19,20, embora essas edições geradas através do reparo dirigido por homologia (HDR) mediado por CRISPR tendam a ser ineficientes com baixo sucesso intrínseco taxas que requerem genotipagem como tela primária 9,10,14,21. Isso demonstra a necessidade de um sistema KI CRISPR eficiente em peixe-zebra, bem como um sistema confiável de alto rendimento para detectar edições precisas.
O objetivo deste estudo foi descrever uma plataforma para gerar um gene KI cardíaco preciso em corações de peixe-zebra com detecção simples e de alto rendimento de edições bem-sucedidas. Uma abordagem de substituição de éxons de dois sgRNA baseada em CRISPR-Cas9 é descrita, que é baseada em uma abordagem TALEN8. Essa abordagem envolve a excisão da sequência alvo usando guias de dois sgRNA e a substituição por uma sequência de modelo exógena que contém o KI de interesse, bem como um gene repórter intrônico geneticamente codificado (Figura 1). A integração de um repórter fluorescente geneticamente codificado dentro da sequência intrônica do gene alvo permite a detecção eficiente de edições positivas. Uma plataforma de fenotipagem é então descrita para avaliar a função elétrica cardíaca em larvas de peixe-zebra para caracterização não invasiva das variantes genéticas associadas à SQTL hereditária, um distúrbio elétrico cardíaco que predispõe os indivíduos à morte súbita cardíaca.
Essas abordagens aumentarão o acesso e o uso de edições do gene KI do peixe-zebra para modelar doenças hereditárias e abordar questões biológicas e fisiológicas, como o mapeamento de padrões de expressão gênica e a regulação do desenvolvimento. Como os corações de peixe-zebra são mais paralelos às características eletrofisiológicas cardíacas humanas do que os modelos murinos, eles podem ser particularmente atraentes como um sistema geneticamente tratável para modelagem de doenças cardíacas 7,22,23.
Protocolo
Estudos usando peixe-zebra foram conduzidos de acordo com as políticas e procedimentos do Comitê de Cuidados com Animais da Universidade Simon Fraser e do Conselho Canadense de Cuidados com Animais e foram concluídos sob o protocolo # 1264K-18.
1. Projeto de componentes CRISPR para edições precisas
- Para projetar os guias de dois sgRNA que serão usados para extirpar a sequência contendo o local alvo do KI, primeiro identifique o ortolog do peixe-zebra para o gene de interesse.
NOTA: A Figura 2 fornece uma visão geral resumida das etapas para projetar edições precisas usando a abordagem CRISPR-Cas9 de dois sgRNAs. - Em seguida, use uma ferramenta de software de design, como o CRISPOR24, que inclui a seleção de Danio rerio como espécie e da enzima Cas a ser usada.
NOTA: O gene de interesse para este estudo foi zkcnh6a (Ensembl Transcript ID: ENSDART00000090809.6; UniProt Protein ID: B3DJX4), e a mutação alvo foi o aminoácido, R56Q.- Para a abordagem de dois sgRNA, escolha um local de sgRNA que precede o éxon alvo e um segundo sgRNA que está localizado dentro do intron a jusante imediato.
- Certifique-se de que os sgRNAs selecionados tenham alta especificidade e baixa ligação fora do alvo prevista. Use classificações CRISPOR para identificar guias com o mínimo de vinculação fora do alvo. Não considere guias sem incompatibilidades na sequência de sementes de possíveis alvos fora dos alvos.
- Identifique os locais potenciais fora do alvo mais prováveis (com base nas pontuações do CRISPOR, selecione os três principais locais potenciais fora do alvo) para genotipagem de sequenciamento de Sanger baseada em PCR na etapa 6.1.3.
- Uma vez selecionados os dois sgRNAs, obtenha-se o complemento reverso para cada um, de modo que haja quatro oligonucleotídeos a serem usados: dois oligos complementares que precedem a mutação e dois oligos complementares que estão a jusante.
- Em cada um dos quatro oligos, adicionar locais de restrição compatíveis para incorporação em um plasmídeo guia de escolha; para integração no plasmídeo DR274, use um site de restrição BsaI de 5' para criar uma saliência. Certifique-se de que o local de reconhecimento Bsa1 é projetado na extremidade 5' da guia selecionada do CRISPOR e o local de reconhecimento Bsa1 é projetado na extremidade 5' das cadeias complementares para garantir a orientação correta das guias no plasmídeo DR274 (ver Figura 3).
- Para projetar o modelo exógeno usado para HDR em peixe-zebra (Figura 1), escolha dois fragmentos de sequência que irão flanquear o gene repórter mVenus YFP alojado no plasmídeo pKHR5.
NOTA: A modificação/edição pretendida pode ser incluída no fragmento upstream ou downstream.- Usando o Ensembl, localize o local alvo dentro da sequência gênica de interesse, incluindo aproximadamente a sequência de flanco de 2 kb (braço de homologia), que será usada para fazer o modelo.
NOTA: Os braços de homologia podem ser simétricos ou assimétricos25,26 e aproximadamente 1 kb cada, a montante e a jusante do local de destino. - Divida o modelo em dois segmentos que serão inseridos em ambos os lados do gene repórter mVenus YFP (consulte a Figura 4). Certifique-se de que o site dividido esteja em um íntron para que a sequência de codificação não seja interrompida.
NOTA: Se o gene estiver bem caracterizado, verifique se há papéis funcionais no intron, como locais de splicing ou regiões reguladoras. Regiões próximas às extremidades de 5' ou 3' estão mais frequentemente envolvidas no splicing de mRNA. - Incorporar modificações na sequência do modelo que incluam i) mutações silenciosas no motivo adjacente do protoespaçador guia (PAM) ou na sequência de sementes (estar ciente dos locais PAM alternativos que a enzima Cas pode atingir) para evitar o recorte da enzima Cas; ii) a modificação de interesses; iii) a criação de sítios de endonuclease de restrição para facilitar a clonagem no plasmídeo pKHR5, que contém o gene reportador mVenus YFP (ver Figura 3).
NOTA: Neste estudo, o primeiro segmento de modelo continha XhoI a montante do local de mutação R56Q e SalI a jusante, enquanto o segundo segmento de modelo tinha EcoRI a montante da sequência de destino de guia e BamHI a jusante. Se algum dos locais de restrição selecionados estiver presente na sequência do modelo, mutações para silenciá-los serão necessárias, ou abordagens alternativas, como a Gibson Assembly, poderão ser usadas.
- Usando o Ensembl, localize o local alvo dentro da sequência gênica de interesse, incluindo aproximadamente a sequência de flanco de 2 kb (braço de homologia), que será usada para fazer o modelo.
2. Preparação de componentes CRISPR para microinjeção embrionária
- Prepare o Cas9 para microinjeção 1 semana antes das microinjeções. Use a proteína Cas9 ou prepare o mRNA Cas9 via transcrição in vitro .
NOTA: Neste estudo, utilizou-se o mRNA Cas9, uma vez que as eficiências tenderam a ser maiores.- Amplificar culturas bacterianas de facadas de ágar bacteriano XL1 Blue comercialmente disponíveis (contendo plasmídeo Cas9) usando um antibiótico apropriado, como a ampicilina. Use 675 μL de cultura líquida (com 325 μL de glicerol) para criar um estoque de glicerol de backup para armazenamento a longo prazo a -80 °C.
- Use o restante da cultura líquida para uma purificação de miniprep, de acordo com o protocolo fornecido com o kit de miniprep. Ressuspender o ADN purificado final em 50 μL do tampão de eluição fornecido. Quantificar o produto através de um espectrofotômetro para examinar o rendimento e a pureza.
- Linearizar 2 μg do DNA purificado através de digestão de restrição usando uma enzima de restrição apropriada e usando o tampão apropriado e o tempo de incubação, conforme listado para a enzima de interesse.
- Purificar o plasmídeo linearizado utilizando um Kit de Purificação por PCR, ressuspendendo-o em 30 μL do tampão de eluição fornecido.
- Depois de quantificar o produto, use-o como um modelo para transcrição in vitro usando o kit de transcrição apropriado para o promotor de interesse. Seguir o protocolo fornecido e purificar via precipitação de cloreto de lítio27. Ressuspender o ARN purificado em 10 μL de H2O livre de nuclease e quantificá-lo antes de o armazenar a -20 °C para utilização na mistura de microinjeção.
- Prepare os dois guias de sgRNA.
- Prepare o plasmídeo de sgRNA com um andaime amplificando as culturas bacterianas da facada de ágar bacteriano XL1 Blue disponível comercialmente (consulte a Tabela de Materiais para obter detalhes) da mesma forma que o MLM3613 acima (etapa 2.1.1), exceto use canamicina em vez de ampicilina.
- Recozir os dois pares de oligonucleotídeos complementares de fita simples (ssODNs) para as guias de sgRNA projetadas acima, primeiro ressuspendendo os ssODNs em 1x tampão de recozimento para uma concentração de 100 μM.
- Em reações separadas para cada um dos dois sgRNAs, recoza o par de ssODNs complementares usando um termociclador. Misturar 2 μg de cada par de sssODN complementar com 50 μL de tampão de recozimento e incubar a 95 °C durante 2 min, arrefecer depois a 25 °C durante 45 min.
- Digerir um plasmídeo comercialmente disponível que contenha um andaime de gRNA. Digerir 2 μg do plasmídeo DR274 usando 1 μL de BsaI, 2 μL de tampão apropriado e ddH2O a 20 μL a 37 °C por 1 h. Confirme a linearização (opcional: purificar usando um Kit de Purificação por PCR) usando eletroforese em gel28.
- Em duas reações de ligadura separadas (uma para cada sgRNA), ligue os ssODNs recozidos com o plasmídeo DR274 linearizado. Use uma relação molar insert:vector de 3:1, calculando a massa apropriada através de uma calculadora de ligadura online. Misturar a massa necessária da pastilha e do vector com 1 μL de DNA ligase T4, 2 μL de tampão de ligadura e ddH2O a 12 μL e incubar à temperatura ambiente durante 12 h.
- Transformar 2 μL do produto ligado em células competentes apropriadas (como células de 10β) usando abordagens padrão e, em seguida, amplificar e purificar o produto usando um Kit Miniprep comercialmente disponível. Opcional: criar um estoque de glicerol deste produto.
- Transcreva os dois sgRNAs linearizando 2 μg de cada guia usando um local de restrição a jusante de 3' que esteja o mais próximo possível do final da sequência espacial. Para o plasmídeo DR274, linearize com HindIII e, em seguida, purifique o molde de RNA usando um Kit de Purificação de PCR, ressuspendendo em 30 μL do tampão de eluição.
- Transcreva os dois guias usando um kit de transcrição de RNA. Siga o protocolo do fabricante e purifique via precipitação de cloreto de lítio27. Ressuscite as duas guias de sgRNA purificadas em 10 μL de H2O livre de nuclease, quantifique e armazene a -20 °C para uso na mistura de microinjeção.
NOTA: O kit de transcrição de RNA não pode incorporar uma tampa de 5' ou cauda poli-A.
- Prepare o modelo de repórter HDR exógeno de cadeia dupla.
NOTA: O modelo é sintetizado em duas partes, uma a montante e outra a jusante do gene repórter mVenus YFP. Esses dois segmentos são construções sintéticas ordenadas através de um provedor comercial e, em seguida, ligadas ao plasmídeo pKHR5 (que contém mVenus YFP) sequencialmente.- Preparar o plasmídeo pKHR5 amplificando as culturas bacterianas da estirpe bacteriana DH5α comercialmente disponível (ver Tabela de Materiais para mais pormenores) da mesma forma que a MLM3613 acima (passo 2.1.1).
NOTA: O plasmídeo pKHR5 contém a sequência do gene reportador mVenus YFP. - Ressuspenda os dois segmentos de modelo projetados acima para 100 μM no buffer TE e, em seguida, transforme-se em células de 10β.
- Digerir o primeiro segmento modelo (o a montante do gene reportador mVenus YFP) e o plasmídeo pKHR5 utilizando as enzimas de restrição selecionadas no passo 1.3.3.
NOTA: O resumo sequencial do pKHR5 é necessário devido à proximidade dos locais de restrição selecionados no MCS. - Para preparar o plasmídeo pKHR5 para o segmento de modelo a montante, digerir 4 μg de pKHR5 (de acordo com o passo 2.2.4) com SalI e, em seguida, purificar utilizando um kit de purificação por PCR. Ressuscite em 30 μL de ddH2O e use-o como um modelo para a segunda reação de digestão com XhoI. Purifice o produto usando o Kit de Purificação por PCR.
- Preparar o primeiro segmento de modelo digerindo 2 μg do segmento de modelo (passo 2.3.2), 1 μL de XhoI, 1 μL de SalI, 2 μL de tampão apropriado e ddH2O a 20 μL durante 1 h a 37 °C e purificar o produto em gel.
- Ligue o segmento do modelo a montante da etapa 2.3.5 para o pKHR5 preparado usando as condições de reação descritas na etapa 2.2.5. Transforme o produto ligado em células 10β competentes, amplifice e purifique usando um miniprep (opcional: crie um estoque de glicerol deste produto).
- Use o produto ligado da etapa 2.3.6 (que contém o primeiro segmento de modelo ligado ao plasmídeo pKHR5 a montante do gene repórter mVenus YFP) e digerir para se preparar para o segundo segmento de modelo (a jusante do repórter mVenus). Digerir 4 μg do produto ligado (a partir do passo 2.3.6, como no passo 2.2.4) com BamHI e, em seguida, purificar utilizando um kit de purificação por PCR. Ressuscite em 30 μL de ddH2O e use-o como modelo para a segunda reação digestiva com EcoRI. Purifice o produto usando o Kit de Purificação por PCR.
- Preparar o segundo segmento de modelo digerindo 2 μg do segmento de modelo (etapa 2.3.2), 1 μL de BamHI, 1 μL de EcoRI, 2 μL de tampão apropriado e ddH2O a 20 μL por 1 h a 37 °C e purificar o produto em gel.
- Ligue o segmento do modelo a jusante para o pKHR5 preparado (a partir do passo 2.3.7) utilizando as condições de reacção descritas no passo 2.2.5. Transforme o produto ligado em células competentes, amplie e purifique usando um kit miniprep. Crie um estoque de glicerol deste produto final, que contém ambos os segmentos de modelo ligados de ambos os lados do gene repórter mVenus YFP dentro do pKHR5.
- Preparar o plasmídeo pKHR5 amplificando as culturas bacterianas da estirpe bacteriana DH5α comercialmente disponível (ver Tabela de Materiais para mais pormenores) da mesma forma que a MLM3613 acima (passo 2.1.1).
- Prepare a mistura de microinjeção usando o mRNA Cas9, dois sgRNAs e o modelo de repórter HDR exógeno.
- Misture 200 ng/μL de mRNA Cas9, 100 ng/μL de cada sgRNA e 200 ng/μL de modelo de repórter HDR exógeno em buffer de injeção 1x para um volume final de 20 μL.
- Conservar a mistura de microinjeção a -20 °C e eliminar a mistura não utilizada após três ciclos de congelamento-descongelamento.
NOTA: Use 4 nL desta mistura de microinjeção para microinjeção no saco vitelino de cada embrião.
3. Criação de peixe-zebra e microinjeção de embriões
NOTA: Protocolos para a criação de peixes-zebra e a microinjeção de embriões unicelulares foram descritos anteriormente 29,30,31.
- Para a reprodução, use peixe-zebra da cepa AB e que são 6-12 meses de idade. Injetar os embriões na fase unicelular aproximadamente 40 minutos após a fertilização (ver Figura 5).
NOTA: O sexo biológico dos embriões injetados não era conhecido; o dimorfismo sexual não é evidente até aproximadamente 3 meses de idade32.
4. Triagem genética do repórter de peixe-zebra larval editado por CRISPR-Cas9
- Visualize a integração do YFP em larvas de peixe-zebra após a microinjeção de componentes CRISPR-Cas9 para rastrear edições HDR bem-sucedidas.
- Em uma placa de Petri de 25 mm, anestesiar 24 larvas de peixe-zebra aos 3 dias após a fertilização (dpf) em sulfonato de metano tricadino a 0,3% (MS-222, tamponada para pH 7,0-7,4 com HEPES e hidróxido de sódio) até que elas percam seu reflexo de auto-endireitamento (tipicamente 1-2 min). Uma vez anestesiada, transfira cada larva para um poço individual de uma placa de 24 poços.
- Usando um microscópio capaz de detectar GFP / YFP, rastreie a fluorescência do gene repórter nos olhos de cada larva individual.
- Capture imagens de cada larva e documente a presença ou ausência de expressão gênica do repórter.
5. Fenotipagem de larvas de peixe-zebra editado por CRISPR-Cas9
- Após a triagem do gene do repórter, realize fenotipagem cardíaca (frequência cardíaca, dimensões pericárdicas, ECG) em cada larva. Fenótipo um número igual de larvas geneticamente positivas e negativas do repórter.
- Use uma câmera CCD (por exemplo, blackfly USB3) e um software de gravação de vídeo e imagem (por exemplo, Micromanager for ImageJ) para medir a frequência cardíaca e as dimensões pericárdicas enquanto as larvas estão anestesiadas.
- Para medir a frequência cardíaca, usando o Micromanager, crie uma região de interesse (ROI) de modo a capturar o coração e excluir outras estruturas.
- Importe o vídeo para o ImageJ como uma sequência de imagens e verifique se o número correto de quadros é inserido em número de imagens.
- Depois que o arquivo estiver aberto, use a ferramenta de seleção de retângulo para desenhar um ROI dentro do coração, mas excluindo outros elementos móveis, e salve o ROI no gerenciador de ROI (analisar | ferramentas | Gerente de ROI).
- Clique em plug-ins | instalar e selecione o algoritmo de frequência cardíaca para instalar o código na pasta padrão e, em seguida, selecione o plug-in na parte inferior da guia plug-ins. Grave as batidas por minuto (bpm) da janela pop-up.
NOTA: Os algoritmos de detecção de imagem foram escritos sob medida para detectar a frequência cardíaca medindo as alterações individuais da densidade de pixels associadas à contração sistólica ventricular. O código pode ser encontrado em https://github.com/dpoburko/zFish_HR.
- Meça as dimensões pericárdicas usando uma ferramenta livre, como o ImageJ, para desenhar ROIs ao redor do saco pericárdico e de um dos olhos. Abra a imagem no ImageJ e use a ferramenta de seleção de polígonos para desenhar um ROI primeiro ao redor do saco pericárdico, salvando-o no gerenciador de ROI como na etapa 5.1.2 e repita para o olho. Selecione esses dois ROIs no gerenciador de ROI re clique em medir. Registre a área de cada um para calcular posteriormente a área do saco pericárdico normalizado para a área dos olhos em cada larva.
- Após as medidas de frequência cardíaca e pericárdica, registre o ECG de larvas individuais.
NOTA: Protocolos para registro de ECG de peixe-zebra foram descritos anteriormente 33,34,35,36.
6. Genotipagem de larvas de peixe-zebra editado por CRISPR-Cas9
- Após análises fenotípicas, realize genotipagem dentro e fora do alvo para confirmar a edição precisa e precisa do gene HDR.
- Anestesiar cada larva de 3 dpf em MS-222 a 0,3% e clipe de cauda para isolar o gDNA usando o método HOTShot37. Incubar cada clipe de cauda excisado em 15 μL de NaOH de 25 mM a 95 °C durante 20 min. Em seguida, neutralize com 1,5 μL de Tris-HCl e centrifugar a 13.800 x g por 30 s. Reter o sobrenadante, que contém gDNA extraído.
- Recupere as larvas em meios E3 e devolva-as ao sistema habitacional se for pretendido um maior desenvolvimento ou estudo.
- Usando o gDNA extraído como um modelo, execute o sequenciamento Sanger baseado em PCR de locais no alvo e potenciais fora do alvo.
NOTA: Opcional: uma abordagem de PCR aninhada pode ser benéfica para algumas regiões genéticas. - Certifique-se de que o projeto do primer no alvo capture o local da mutação e o local de ligação do sgRNA mais próximo. Projete um primer de sequenciamento separado para detectar a transição do braço de homologia inserido e do gene alvo para confirmar a integração no gene de interesse. Projetar primers para sequenciar os três principais locais potenciais fora do alvo identificados na etapa 1.2.3.
NOTA: Os programas de software de design de guia geralmente sugerem primers para usar, mas a personalização pode ser necessária para alcançar os melhores resultados. - Compile genotipagem dentro e fora do alvo, frequência cardíaca, dimensões pericárdicas, fenotipagem de ECG e dados do gene repórter identificáveis para cada peixe-zebra.
Resultados
O uso bem-sucedido desta abordagem CRISPR de substituição de éxons de dois sgRNA é destacado pela introdução e detecção simples de uma edição precisa para projetar a variante associada ao LQTS, R56Q, no gene zkcnh6a em peixe-zebra. A Figura 6 mostra larvas representativas de 3 dpf injetadas no estágio embrionário unicelular com componentes CRISPR, conforme descrito acima. A Figura 6A mostra a presença da expressão gênica do repórter mVenus YFP na lente ocular como um repórter positivo da integração bem-sucedida do modelo. A Figura 6B,C mostra cromatogramas de sequenciamento de Sanger obtidos a partir de DNA genômico isolado de amostras de clipe de cauda de peixes do tipo selvagem e do gene repórter positivos, respectivamente. Descobriu-se que os peixes geneticamente positivos do repórter tinham a edição precisa, G a A, que introduz a variante R56Q no zkcnh6a. A genotipagem mostrou uma correlação de 100% entre a expressão gênica do repórter YFP e a presença da edição precisa do gene R56Q, validando essa ferramenta de triagem de fluorescência.
A fenotipagem de larvas de peixe-zebra editadas por genes foi realizada a 3 dpf. A Figura 7 mostra resultados representativos de larvas do tipo selvagem e editadas pelo gene R56Q. A frequência cardíaca foi detectada por captura de vídeo, conforme descrito acima. Um exemplo da medida das dimensões pericárdicas como proporção da área ocular é mostrado (Figura 7A). A Figura 7B apresenta gráficos da frequência cardíaca frente às dimensões pericárdicas normalizadas, destacando uma tendência de bradicardia com edema pericárdico crescente, que está associado a distúrbios de repolarização cardíaca em peixes-zebra 8,38,39,40. A Figura 7C mostra um exemplo representativo de registros de ECG de larvas de 3 dpf. Os intervalos padrão (QT, QRS) foram medidos a partir da média dos sinais de ECG.

Figura 1: Integração do modelo HDR no genoma do peixe-zebra. Cinzento escuro, braços de homologia; alvos verdes de guia de sgRNA com mutação silenciosa para evitar o recorte de Cas9; cinza claro, éxon alvo de interesse; linha vermelha, mutação pontual; amarelo, gene repórter mVenus YFP sob um promotor de α-cristalina; linhas tracejadas indicam homologia. Aqui, a edição precisa alvo foi R56Q no éxon 2 do gene zkcnh6a . Abreviaturas: HDR = reparo dirigido por homologia; sgRNA = RNA de guia única; YFP = proteína fluorescente amarela; DSB = quebra de fita dupla; WT = tipo selvagem. Por favor, clique aqui para ver uma versão maior desta figura.

Figura 2: Resumo das etapas para projetar edições precisas em genes de peixe-zebra usando a abordagem CRISPR-Cas9 de dois sgRNA (os números de passo do protocolo relacionados são indicados entre parênteses). Abreviaturas: sgRNA = RNA de guia único; YFP = proteína fluorescente amarela; gDNA = DNA genômico; ECG = eletrocardiograma; dpf = dias pós-adubação; MS-222 = sulfonato de metano de tricaína. Por favor, clique aqui para ver uma versão maior desta figura.
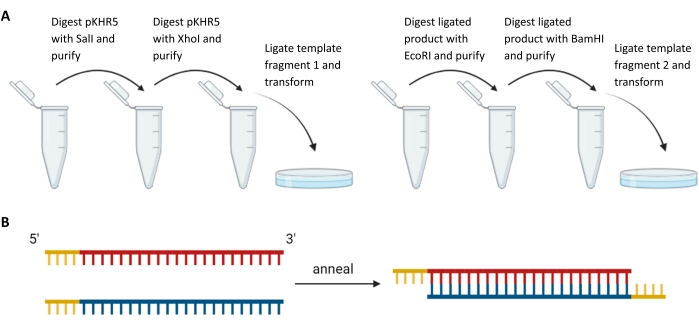
Figura 3: Preparação de fragmentos de molde exógenos e guias de sgRNA. (A) Digestão sequencial e ligadura de fragmentos de molde a montante e a jusante da sequência do gene reportador mVenus YFP em pKHR5. (B) Recozimento de pares complementares de sgRNA com saliência de restrição para ligadura em DR274. Abreviaturas: sgRNA = RNA de guia único; YFP = proteína fluorescente amarela. Por favor, clique aqui para ver uma versão maior desta figura.
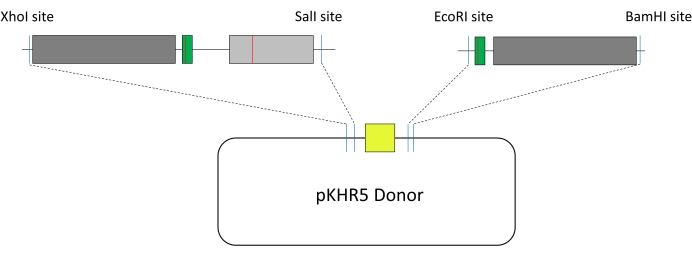
Figura 4: Construção do modelo HDR. Cinzento escuro, braços de homologia; alvos verdes de guia de sgRNA com mutação silenciosa para evitar o recorte de Cas9; cinza claro, éxon alvo de interesse; linha vermelha, mutação pontual; amarelo, gene repórter mVenus YFP sob um promotor de α-cristalina; linha azul escura, adicionado sites de restrição. Os dois fragmentos de modelo são integrados no doador de plasmídeo pKHR5. Abreviaturas: HDR = reparo dirigido por homologia; sgRNA = RNA de guia única. Por favor, clique aqui para ver uma versão maior desta figura.

Figura 5: Microinjeção de embriões de peixe-zebra unicelular com componentes CRISPR-Cas9. Barra de escala = 0,5 mm. Abreviaturas: HDR = reparo dirigido por homologia; sgRNA = RNA de guia única. Por favor, clique aqui para ver uma versão maior desta figura.

Figura 6: A fácil detecção da fluorescência do gene repórter mVenus YFP indica integração positiva do modelo exógeno HDR no gene alvo. (A) Exemplo de expressão de mVenus YFP em um olho de peixe-zebra (seta) em uma larva de peixe-zebra editada. (B) As edições bem-sucedidas são confirmadas por cromatogramas de sequenciamento (esquerda, WT; direita, edição R56Q). Por favor, clique aqui para ver uma versão maior desta figura.

Figura 7: Análise fenotípica das consequências cardíacas em peixes-zebra de 3 dpf após a edição precisa do R56Q no gene alvo zkcnh6a. (A) Detecção de imagem de dimensões pericárdicas em relação ao tamanho dos olhos usando a ferramenta de polígono no ImageJ. Os limites do saco pericárdico foram marcados pelo usuário a partir de um único quadro de registro com base em alterações na translucidez e pigmentação. Exemplos de dimensões pericárdicas normais e derrame pericárdico são mostrados. Barra da escala = 0,5 mm. (B) Correlação entre as dimensões pericárdicas (em relação à dimensão ocular) e a frequência cardíaca, R2 = 0,33. (C) Exemplo de registro de ECG de um coração de larva de peixe-zebra de 3 dpf (esquerda) e complexos médios (direita). Frequência cardíaca, 131 bpm; intervalo QTc corrigido pela frequência cardíaca, 460 ms. Abreviaturas: dpf = dias pós-adubação; ECG = eletrocardiograma. Por favor, clique aqui para ver uma versão maior desta figura.
Discussão
A engenharia de edições precisas de genes usando CRISPR-Cas9 é desafiada pela baixa eficiência dos mecanismos HDR e sua detecção eficiente. Aqui, uma abordagem de substituição de éxons de dois sgRNA baseada em CRISPR-Cas9 é descrita que produz edições precisas em peixes-zebra com detecção visual direta de edições positivas. A eficácia desta abordagem é demonstrada através da geração de edições precisas no gene zkcnh6a . Este trabalho mostra como a função cardíaca em larvas de peixe-zebra editadas por genes pode ser avaliada usando medidas fenotípicas não invasivas de frequência cardíaca, dimensões pericárdicas e morfologia do ECG. Esta abordagem, desde a introdução de uma edição genética até a avaliação fenotípica, pode ser concluída do início ao fim dentro de aproximadamente 1 semana.
Os benefícios da abordagem de edição e fenotipagem acima são a facilidade do projeto de modificação CRISPR, a ampla aplicabilidade em múltiplos sistemas fisiológicos, a capacidade de inserir grandes genes ou fragmentos de genes e a capacidade de rastrear efeitos variantes longitudinalmente através do desenvolvimento e das gerações. O sucesso de edições precisas nessa abordagem pode estar relacionado à combinação do grande tamanho do molde (devido à inserção do gene repórter e braços longos de homologia), que demonstrou aumentar a eficiência das edições no peixe-zebra14, e à estratégia de guias de dois sgRNA, que tem sido usada efetivamente em edições induzidas por telen de peixe-zebra8.
Uma força particular da abordagem descrita é a capacidade de inserir genes grandes ou fragmentos de genes. Isso pode ser útil, por exemplo, para inserir ortólogos humanos41, permitindo uma caracterização e comparação clinicamente mais traduzíveis entre os ortólogos. Alternativamente, genes que codificam enzimas Cas também podem ser inseridos, permitindo uma linha de peixe-zebra com mecanismos de edição CRISPR in vivo , fornecendo um sistema induzível. Da mesma forma, mecanismos CRISPR alternativos, como a edição principal, poderiam ser integrados e resultar em uma linha de peixe-zebra que são prontamente editados com precisão e eficiência.
Apesar das vantagens dessa abordagem, existem algumas limitações. Primeiro, apenas um único gene e locus foram modificados, e mais testes em outros locais ou em outros genes são necessários para avaliar o quão amplamente aplicável é essa abordagem. Devido aos longos braços de homologia necessários, os custos de design do modelo são mais altos; no entanto, isso pode ser compensado por uma triagem eficiente. Outra limitação é que a abordagem de triagem requer capacidade de detecção de fluorescência. No entanto, os requisitos ópticos são relativamente baixos e podem ser personalizados ou comprados comercialmente a um custo razoavelmente baixo. O uso de uma abordagem de dois sgRNA aumenta o número de potenciais eventos fora do alvo; no entanto, isso provavelmente é mitigado pela menor probabilidade de que os dois guias de sgRNA recozinhem de uma maneira que facilite a incorporação do modelo para produzir a expressão gênica do repórter. Finalmente, o uso de mRNA Cas9 pode levar a mosaicismos, pois o Cas9 não está ativo até estágios posteriores de desenvolvimento. Isso poderia ser explicado pelo sequenciamento de determinados tipos de tecidos; no entanto, dado o tamanho das larvas de peixe-zebra, isso é tecnicamente desafiador.
Em resumo, esta abordagem de edição precisa de dois sgRNA CRISPR-Cas9 em peixe-zebra permite a detecção visual simples de edições positivas e pode ser adaptada para incorporar grandes genes de interesse em qualquer locus. Combinado com medidas fenotípicas, isso permite uma plataforma confiável e de alto rendimento para o estudo de variantes cardíacas clinicamente relevantes.
Divulgações
Os autores não têm conflitos de interesse a divulgar.
Agradecimentos
Esta pesquisa foi apoiada por uma bolsa do Canadian Institutes of Health Research Project (T.W.C.) e pelas bolsas do Conselho de Pesquisa em Ciências Naturais e Engenharia do Canadá Discovery (T.W.C.).
Materiais
| Name | Company | Catalog Number | Comments |
| Program | |||
| CRISPOR | TEFOR Infrastructure | ||
| ENSEMBL | European Bioinformatics Institute | ||
| ImageJ | National Institutes of Health (NIH) | ||
| Micro-Manager | Open Source (Github) | ||
| NEBiocalculator | New England Biolabs (NEB) | ||
| EQUIPMENT | |||
| 24-well Plate | VWR | ||
| 25 mm Petri Dish | VWR | ||
| Blackfly USB3 Camera | Teledyne FLIR | ||
| C1000 Thermal Cycler | Bio-Rad | ||
| Centrifuge 5415C | Eppendorf | ||
| EZNA Gel Extraction Kit | Omega Biotek | ||
| MAXIscript T7 Transcription Kit | Invitrogen | ||
| MaxQ 5000 Incubator | Barnstead Lab Line | ||
| Miniprep Kit | Qiagen | ||
| mMessage mMachine T7 Ultra Transcription Kit | Invitrogen | ||
| ND1000 Spectrophotometer | Nanodrop | ||
| PCR Purification Kit | Qiagen | ||
| PLI 100A Picoinjector | Harvard Apparatus | ||
| PowerPac Basic Power Supply | Bio-Rad | ||
| Stemi 305 Steroscope | Zeiss | ||
| Wide Mini Sub Cell GT Electrophoresis System | Bio-Rad | ||
| ZebTec Zebrafish Housing System | Tecniplast | ||
| SERVICES | |||
| Gene Synthesis | Genewiz | ||
| Sanger Sequencing | Genewiz | ||
| REAGENTS | |||
| 10β Competent Cells | NEB | ||
| 10X PCR Buffer | Qiagen | ||
| 100 mM Nucleotide Mixture | ABM | ||
| Ampicillin | Sigma | ||
| BamHI Endonuclease w/ buffer | NEB | ||
| BsaI Endonuclease w/ buffer | NEB | ||
| DR274 Plasmid (XL1 Blue bacterial agar stab) | Addgene | ||
| EcoRI Endonuclease w/ buffer | NEB | ||
| Glycerol | |||
| HEPES | Sigma | ||
| HindIII Endonuclease w/ buffer | NEB | ||
| Kanamycin | Sigma | ||
| Methylene Blue | Sigma | ||
| MLM3613 Plasmid (XL1 Blue bacterial agar stab) | Addgene | ||
| MS-222 (Tricaine) | Sigma | ||
| pKHR5 Plasmid (DH5α bacterial agar stab) | Addgene | ||
| PmeI Endonuclease w/ buffer | NEB | ||
| SalI Endonuclease w/ buffer | NEB | ||
| Sodium Hydroxide | Sigma | ||
| T4 Ligase w/ buffer | Sigma | ||
| Taq Polymerase | Qiagen | ||
| TE Buffer | Sigma | ||
| Tris Hydrochloride | Sigma | ||
| XhoI Endonuclease w/ buffer | NEB | ||
| RECIPES | |||
| Solution | Component | Supplier | |
| Annealing Buffer (pH 7.5-8.0) | 10 mM Tris | Sigma | |
| 50 mM NaCl | Sigma | ||
| 1 mM EDTA | Sigma | ||
| E3 Media (pH 7.2) | 5 mM NaCl | Sigma | |
| 0.17 mM KCl | Sigma | ||
| 0.33 mM CaCl2 | Sigma | ||
| 0.33 mM MgSO4 | Sigma | ||
| Injection Buffer (pH 7.5) | 20 mM HEPES | Sigma | |
| 150 mM KCl | Sigma |
Referências
- Zarei, A., Razban, V., Hosseini, S. E., Tabei, S. M. B. Creating cell and animal models of human disease by genome editing using CRISPR/Cas9. The Journal of Gene Medicine. 21 (4), 3082(2019).
- Lee, H., Yoon, D. E., Kim, K. Genome editing methods in animal models. Animal Cells and Systems. 24 (1), 8-16 (2020).
- Li, Q., et al. Applications of genome editing technology in animal disease modeling and gene therapy. Computational and Structural Biotechnology Journal. 17, 689-698 (2019).
- Gut, P., Reischauer, S., Stainier, D. Y. R., Arnaout, R. Little fish, big data: Zebrafish as a model for cardiovascular and metabolic disease. Physiological Reviews. 97 (3), 889-938 (2017).
- Kegel, L., et al. Forward genetic screen using zebrafish to identify new genes involved in myelination. Oligodendrocytes: Methods and Protocols. 1936, 185-209 (2019).
- Shah, A. N., Davey, C. F., Whitebirch, A. C., Miller, A. C., Moens, C. B. Rapid reverse genetic screening using CRISPR in zebrafish. Nature Methods. 12 (6), 535-540 (2015).
- González-Rosa, J. M. Zebrafish models of cardiac disease: From fortuitous mutants to precision medicine. Circulation Research. 130 (12), 1803-1826 (2022).
- Hoshijima, K., Jurynec, M. J., Grunwald, D. J. Precise editing of the Zebrafish genome made simple and efficient. Developmental Cell. 36 (6), 654-667 (2016).
- Albadri, S., Del Bene, F., Revenu, C. Genome editing using CRISPR/Cas9-based knock-in approaches in zebrafish. Methods. 121-122, 77-85 (2017).
- Armstrong, G. A. B., et al. Homology directed knockin of point mutations in the zebrafish tardbp and fus genes in ALS using the CRISPR/Cas9 system. PLoS One. 11 (3), 0150188(2016).
- Bai, H., et al. CRISPR/Cas9-mediated precise genome modification by a long ssDNA template in zebrafish. BMC Genomics. 21 (1), 67(2020).
- de Vrieze, E., et al. Efficient generation of knock-in zebrafish models for inherited disorders using CRISPR-Cas9 ribonucleoprotein complexes. International Journal of Molecular Sciences. 22 (17), 9429(2021).
- Eschstruth, A., Schneider-Maunoury, S., Giudicelli, F. Creation of zebrafish knock-in reporter lines in the nefma gene by Cas9-mediated homologous recombination. Genesis. 58 (1), 23340(2020).
- Irion, U., Krauss, J., Nüsslein-Volhard, C. Precise and efficient genome editing in zebrafish using the CRISPR/Cas9 system. Development. 141 (24), 4827-4830 (2014).
- Kimura, Y., Hisano, Y., Kawahara, A., Higashijima, S. Efficient generation of knock-in transgenic zebrafish carrying reporter/driver genes by CRISPR/Cas9-mediated genome engineering. Scientific Reports. 4 (1), 6545(2014).
- Levic, D. S., Yamaguchi, N., Wang, S., Knaut, H., Bagnat, M. Knock-in tagging in zebrafish facilitated by insertion into non-coding regions. Development. 148 (19), (2021).
- Prykhozhij, S. V., et al. Optimized knock-in of point mutations in zebrafish using CRISPR/Cas9. Nucleic Acids Research. 46 (17), 102(2018).
- Wierson, W. A., et al. Efficient targeted integration directed by short homology in zebrafish and mammalian cells. eLife. 9, 53968(2020).
- Boel, A., et al. CRISPR/Cas9-mediated homology-directed repair by ssODNs in zebrafish induces complex mutational patterns resulting from genomic integration of repair-template fragments. Disease Models & Mechanisms. 11 (10), (2018).
- Tessadori, F., et al. Effective CRISPR/Cas9-based nucleotide editing in zebrafish to model human genetic cardiovascular disorders. Disease Models & Mechanisms. 11 (10), (2018).
- Zhang, Y., Zhang, Z., Ge, W. An efficient platform for generating somatic point mutations with germline transmission in the zebrafish by CRISPR/Cas9-mediated gene editing. The Journal of Biological Chemistry. 293 (17), 6611-6622 (2018).
- Vornanen, M., Hassinen, M. Zebrafish heart as a model for human cardiac electrophysiology. Channels. 10 (2), 101-110 (2016).
- Nemtsas, P., Wettwer, E., Christ, T., Weidinger, G., Ravens, U. Adult zebrafish heart as a model for human heart? An electrophysiological study. Journal of Molecular and Cellular Cardiology. 48 (1), 161-171 (2010).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biology. 17 (1), 148(2016).
- Liang, X., Potter, J., Kumar, S., Ravinder, N., Chesnut, J. D. Enhanced CRISPR/Cas9-mediated precise genome editing by improved design and delivery of gRNA, Cas9 nuclease, and donor DNA. Journal of Biotechnology. 241, 136-146 (2017).
- Richardson, C. D., Ray, G. J., DeWitt, M. A., Curie, G. L., Corn, J. E. Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nature Biotechnology. 34 (3), 339-344 (2016).
- Sambrook, J., Fritsch, E. F., Maniatis, T. Precipitation of Large RNAs with Lithium Chloride. Molecular Cloning: A Laboratory Manual, Book 3. E.15. , Cold Spring Harbor Laboratory. Long Island, NY. (1989).
- Sambrook, J., Fritsch, E. F., Maniatis, T. Agarose Gel Electrophoresis. Molecular Cloning: A Laboratory Manual, Book 1. , Cold Spring Harbor Laboratory. Long Island, NY. 3-20 (1989).
- Sorlien, E. L., Witucki, M. A., Ogas, J. Efficient production and identification of CRISPR/Cas9-generated gene knockouts in the model system Danio rerio. Journal of Visualized Experiments. (138), e56969(2018).
- Avdesh, A., et al. Regular care and maintenance of a zebrafish (Danio rerio) laboratory: An introduction. Journal of Visualized Experiments. (69), e4196(2012).
- Rosen, J. N., Sweeney, M. F., Mably, J. D. Microinjection of zebrafish embryos to analyze gene function. Journal of Visualized Experiments. (25), e1115(2009).
- Kossack, M. E., Draper, B. W. Genetic regulation of sex determination and maintenance in zebrafish (Danio rerio). Current Topics in Developmental Biology. 134, 119-149 (2019).
- Tanaka, Y., et al. Functional analysis of KCNH2 gene mutations of type 2 long QT syndrome in larval zebrafish using microscopy and electrocardiography. Heart and Vessels. 34 (1), 159-166 (2019).
- Dhillon, S. S., et al. Optimisation of embryonic and larval ECG measurement in zebrafish for quantifying the effect of QT prolonging drugs. PLoS One. 8 (4), 60552(2013).
- Yu, F., et al. Evolving cardiac conduction phenotypes in developing zebrafish larvae: Implications to drug sensitivity. Zebrafish. 7 (4), 325-331 (2010).
- Hurst, R. M. Development and optimization of tools for embryonic electrocardiograph recording for heart dysfunction in zebrafish. , University of Birmingham. PhD thesis (2018).
- Meeker, N. D., Hutchinson, S. A., Ho, L., Trede, N. S. Method for isolation of PCR-ready genomic DNA from zebrafish tissues. BioTechniques. 43 (5), 610-614 (2007).
- Arnaout, R., et al. Zebrafish model for human long QT syndrome. Proceedings of the National Academy of Sciences of the United States of America. 104 (27), 11316-11321 (2007).
- Milan, D. J., Peterson, T. A., Ruskin, J. N., Peterson, R. T., MacRae, C. A. Drugs that induce repolarization abnormalities cause bradycardia in zebrafish. Circulation. 107 (10), 1355-1358 (2003).
- Langheinrich, U., Vacun, G., Wagner, T. Zebrafish embryos express an orthologue of HERG and are sensitive toward a range of QT-prolonging drugs inducing severe arrhythmia. Toxicology and Applied Pharmacology. 193 (3), 370-382 (2003).
- MacRae, C. A. Cardiac arrhythmia: In vivo screening in the zebrafish to overcome complexity in drug discovery. Expert Opinion on Drug Discovery. 5 (7), 619-632 (2010).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados