Method Article
CRISPR-Cas9-vermittelte präzise Knock-In-Bearbeitungen in Zebrafischherzen
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt einen Ansatz, um präzise Knock-in-Bearbeitungen in Zebrafischembryonen mit der CRISPR-Cas9-Technologie zu ermöglichen. Eine Phänotypisierungspipeline wird vorgestellt, um die Anwendbarkeit dieser Techniken zur Modellierung einer Long-QT-Syndrom-assoziierten Genvariante zu demonstrieren.
Zusammenfassung
Clustered Regular Interspaced Short Palindromic Repeats (CRISPR) in Tiermodellen ermöglichen eine präzise genetische Manipulation zur Untersuchung physiologischer Phänomene. Zebrafische wurden als effektives genetisches Modell verwendet, um zahlreiche Fragen im Zusammenhang mit Erbkrankheiten, Entwicklung und Toxikologie auf der Ebene des gesamten Organs und des Organismus zu untersuchen. Aufgrund des gut annotierten und kartierten Zebrafischgenoms wurden zahlreiche Werkzeuge zur Gen-Editierung entwickelt. Die Wirksamkeit der Erzeugung und Leichtigkeit der Erkennung präziser Knock-In-Bearbeitungen mit CRISPR ist jedoch ein limitierender Faktor. Hier wird ein CRISPR-Cas9-basierter Knock-in-Ansatz mit dem einfachen Nachweis präziser Schnitte in einem Gen beschrieben, das für die kardiale Repolarisation verantwortlich ist und mit der elektrischen Störung Long QT Syndrome (LQTS) assoziiert ist. Dieser Zwei-Single-Guide-RNA-Ansatz (sgRNA) schneidet und ersetzt die Zielsequenz und verknüpft ein genetisch kodiertes Reportergen. Der Nutzen dieses Ansatzes wird durch die Beschreibung nicht-invasiver phänotypischer Messungen der kardialen elektrischen Funktion in Wildtyp- und geneditierten Zebrafischlarven demonstriert. Dieser Ansatz ermöglicht die effiziente Untersuchung krankheitsassoziierter Varianten in einem ganzen Organismus. Darüber hinaus bietet diese Strategie Möglichkeiten zum Einfügen exogener Sequenzen der Wahl, wie Reportergene, Orthologe oder Geneditoren.
Einleitung
CRISPR-basierte Gen-Editing-Strategien in Tiermodellen ermöglichen die Untersuchung genetisch vererbbarer Erkrankungen, Entwicklung und Toxikologie auf der Ebene des gesamten Organismus 1,2,3. Zebrafische bieten ein leistungsfähiges Modell, das in zahlreichen physiologischen Aspekten dem Menschen näher ist als murine oder humane Zellmodelle4. Eine breite Palette genetischer Werkzeuge und Strategien wurde im Zebrafisch sowohl für das Vorwärts-als auch für das umgekehrte genetische Screening6 verwendet. Umfassende genetische Kartierung und Annotation bei Zebrafischen haben Gen-Editing-Ansätze als primäre Technik zur Entwicklung gezielter Gen-Knockouts (KOs) und präziser Knock-Ins (KIs) erleichtert7.
Trotzdem ist die Erzeugung präziser KI-Bearbeitungen im Zebrafisch durch geringe Effizienz und die Schwierigkeit einer genauen Erkennung begrenzt. Obwohl Transkriptionsfaktor-ähnliche Effektornukleasen (TALENs) erfolgreich für KIs8 eingesetzt und optimiert wurden, bietet CRISPR eine verbesserte Gen-Editing-Strategie mit einfacherem sgRNA-Targeting. Zahlreiche Studien haben CRISPR verwendet, um präzise KIs im Zebrafisch 9,10,11,12,13,14,15,16,17,18,19,20 zu erzeugen, obwohl diese Bearbeitungen, die durch CRISPR-vermittelte homologiegesteuerte Reparatur (HDR) erzeugt werden, tendenziell ineffizient mit geringem intrinsischem Erfolg sind. Raten, die eine Genotypisierung als primärer Screen erfordern 9,10,14,21. Dies zeigt die Notwendigkeit eines effizienten KI CRISPR-Systems im Zebrafisch sowie eines zuverlässigen Hochdurchsatzsystems zur Erkennung präziser Bearbeitungen.
Das Ziel dieser Studie war es, eine Plattform zur Erzeugung eines präzisen kardialen Gens KI in Zebrafischherzen mit einfacher und hochdurchsatzartiger Detektion erfolgreicher Bearbeitungen zu beschreiben. Es wird ein CRISPR-Cas9-basierter Zwei-sgRNA-Exonersatzansatz beschrieben, der auf einem TALEN-Ansatz8 basiert. Dieser Ansatz beinhaltet die Exzision der Zielsequenz unter Verwendung von Zwei-sgRNA-Guides und den Ersatz durch eine exogene Vorlagensequenz, die die interessierende KI sowie ein genetisch kodiertes intronisches Reportergen enthält (Abbildung 1). Die Integration eines genetisch kodierten fluoreszierenden Reporters in die intronische Zielgensequenz ermöglicht die effiziente Detektion positiver Edits. Anschließend wird eine Phänotypisierungsplattform zur Beurteilung der kardialen elektrischen Funktion in Zebrafischlarven zur nicht-invasiven Charakterisierung der Genvarianten beschrieben, die mit vererbtem LQTS assoziiert sind, einer kardialen elektrischen Störung, die Personen für den plötzlichen Herztod prädisponiert.
Diese Ansätze werden den Zugang zu und die Nutzung von Zebrafisch-KI-Genbearbeitungen verbessern, um Erbkrankheiten zu modellieren und biologische und physiologische Fragen wie die Kartierung von Genexpressionsmustern und die Entwicklungsregulation anzugehen. Da Zebrafischherzen bessere parallele humane kardiale elektrophysiologische Eigenschaften aufweisen als Mausmodelle, können sie als genetisch beherrschbares System für die Modellierung von Herzerkrankungen besonders attraktiv sein 7,22,23.
Protokoll
Studien mit Zebrafischen wurden in Übereinstimmung mit den Richtlinien und Verfahren des Simon Fraser University Animal Care Committee und des Canadian Council of Animal Care durchgeführt und gemäß Protokoll # 1264K-18 abgeschlossen.
1. Design von CRISPR-Komponenten für präzise Bearbeitungen
- Um die Zwei-sgRNA-Leitfäden zu entwerfen, die zum Exzisieren der Sequenz mit der KI-Zielstelle verwendet werden, identifizieren Sie zunächst den Zebrafisch-Ortholog für das interessierende Gen.
HINWEIS: Abbildung 2 bietet einen zusammenfassenden Überblick über die Schritte zum Entwickeln präziser Bearbeitungen mit dem Zwei-sgRNA-CRISPR-Cas9-Ansatz. - Als nächstes verwenden Sie ein Design-Software-Tool wie CRISPOR24, das die Auswahl für Danio rerio als Spezies und des zu verwendenden Cas-Enzyms beinhaltet.
HINWEIS: Das für diese Studie interessante Gen war zkcnh6a (Ensembl Transcript ID: ENSDART00000090809.6; UniProt Protein ID: B3DJX4), und die Zielmutation war die Aminosäure R56Q.- Für den Zwei-sgRNA-Ansatz wählen Sie eine sgRNA-Position, die dem Zielexon vorausgeht, und eine zweite sgRNA, die sich innerhalb des unmittelbaren Downstream-Introns befindet.
- Stellen Sie sicher, dass die ausgewählten sgRNAs eine hohe Spezifität und eine geringe vorhergesagte Off-Target-Bindung aufweisen. Verwenden Sie CRISPOR-Rankings, um Leitfäden mit minimaler Off-Target-Bindung zu identifizieren. Berücksichtigen Sie keine Leitfäden ohne Diskrepanzen in der Seed-Reihenfolge potenzieller Off-Targets.
- Identifizieren Sie die wahrscheinlichsten potenziellen Off-Target-Sites (basierend auf CRISPOR-Scores, wählen Sie die drei wichtigsten Exon-potenziellen Off-Target-Sites) für die PCR-basierte Sanger-Sequenzierungsgenotypisierung in Schritt 6.1.3.
- Sobald die beiden sgRNAs ausgewählt wurden, erhalten Sie für jede das umgekehrte Komplement, so dass vier Oligonukleotide verwendet werden können: zwei komplementäre Oligos, die der Mutation vorausgehen, und zwei komplementäre Oligos, die stromabwärts liegen.
- Fügen Sie auf jedem der vier Oligos kompatible Restriktionsstellen für den Einbau in ein Leitplasmid Ihrer Wahl hinzu. Für die Integration in das DR274-Plasmid verwenden Sie eine 5'-BsaI-Restriktionsstelle, um einen Überhang zu erzeugen. Stellen Sie sicher, dass die Bsa1-Erkennungsstelle am 5'-Ende der von CRISPOR ausgewählten Führung und die Bsa1-Erkennungsstelle am 5'-Ende der komplementären Stränge konstruiert ist, um die korrekte Ausrichtung der Guides im DR274-Plasmid sicherzustellen (siehe Abbildung 3).
- Um die exogene Vorlage für HDR im Zebrafisch zu entwerfen (Abbildung 1), wählen Sie zwei Sequenzfragmente, die das mVenus YFP-Reportergen flankieren, das im pKHR5-Plasmid untergebracht ist.
HINWEIS: Die beabsichtigte Änderung/Bearbeitung kann entweder im Upstream- oder im Downstream-Fragment enthalten sein.- Lokalisieren Sie mit Ensembl die Zielstelle innerhalb der interessierenden Gensequenz, einschließlich der etwa 2 kb großen flankierenden Sequenz (Homologiearm), die zur Erstellung der Vorlage verwendet wird.
HINWEIS: Homologiearme können symmetrisch oder asymmetrisch25,26 und jeweils etwa 1 kb sein, stromaufwärts und stromabwärts des Zielortes. - Teilen Sie die Vorlage in zwei Segmente auf, die auf beiden Seiten des mVenus YFP-Reportergens eingefügt werden (siehe Abbildung 4). Stellen Sie sicher, dass sich die geteilte Stelle in einem Intron befindet, damit die Kodierungssequenz nicht unterbrochen wird.
HINWEIS: Wenn das Gen gut charakterisiert ist, überprüfen Sie auf funktionelle Rollen im Intron, wie z. B. Spleißstellen oder regulatorische Regionen. Regionen in der Nähe der 5'- oder 3'-Enden sind häufiger am mRNA-Spleißen beteiligt. - Fügen Sie Änderungen in die Vorlagensequenz ein, die i) stille Mutationen im benachbarten Motiv (PAM) oder der Samensequenz des Guide-Protospacers enthalten (beachten Sie alternative PAM-Stellen, auf die das Cas-Enzym abzielen könnte), um ein erneutes Schneiden des Cas-Enzyms zu verhindern; ii) die Änderung des Interesses; iii) die Schaffung von Restriktionsendonuklease-Stellen, um die Klonierung in das pKHR5-Plasmid zu erleichtern, das das mVenus YFP-Reportergen enthält (siehe Abbildung 3).
HINWEIS: In dieser Studie enthielt das erste Template-Segment XhoI stromaufwärts der R56Q-Mutationsstelle und SalI stromabwärts, während das zweite Template-Segment EcoRI stromaufwärts der Guide-Zielsequenz und BamHI stromabwärts hatte. Wenn eine der ausgewählten Restriktionsstellen innerhalb der Vorlagensequenz vorhanden ist, sind Mutationen erforderlich, um diese stumm zu schalten, oder es können alternative Ansätze wie Gibson Assembly verwendet werden.
- Lokalisieren Sie mit Ensembl die Zielstelle innerhalb der interessierenden Gensequenz, einschließlich der etwa 2 kb großen flankierenden Sequenz (Homologiearm), die zur Erstellung der Vorlage verwendet wird.
2. Vorbereitung von CRISPR-Komponenten für die Mikroinjektion von Embryonen
- Bereiten Sie das Cas9 1 Woche vor den Mikroinjektionen für die Mikroinjektion vor. Verwenden Sie Cas9-Protein oder bereiten Sie Cas9-mRNA über In-vitro-Transkription vor.
HINWEIS: In dieser Studie wurde Cas9-mRNA verwendet, da die Effizienz tendenziell höher war.- Amplifizieren Sie Bakterienkulturen von kommerziell erhältlichem XL1 Blue Bakterienagarstich (mit Cas9-Plasmid) unter Verwendung eines geeigneten Antibiotikums wie Ampicillin. Verwenden Sie 675 μL Flüssigkultur (mit 325 μL Glycerin), um einen Backup-Glycerinvorrat für die Langzeitlagerung bei −80 °C zu erstellen.
- Verwenden Sie den Rest der flüssigen Kultur für eine Miniprep-Reinigung gemäß dem mit dem Miniprep-Kit gelieferten Protokoll. Resuspendieren Sie die endgültige gereinigte DNA in 50 μL des bereitgestellten Elutionspuffers. Quantifizieren Sie das Produkt über ein Spektralphotometer, um die Ausbeute und Reinheit zu untersuchen.
- Linearisieren Sie 2 μg der gereinigten DNA durch Restriktionsaufschluss unter Verwendung eines geeigneten Restriktionsenzyms und unter Verwendung des entsprechenden Puffers und der Inkubationszeit, wie für das interessierende Enzym angegeben.
- Reinigen Sie das linearisierte Plasmid mit einem PCR-Aufreinigungskit und resuspendieren Sie es in 30 μL des bereitgestellten Elutionspuffers.
- Nachdem Sie das Produkt quantifiziert haben, verwenden Sie dieses als Vorlage für die In-vitro-Transkription unter Verwendung des geeigneten Transkriptionskits für den interessierenden Promotor. Befolgen Sie das bereitgestellte Protokoll und reinigen Sie über Lithiumchlorid-Fällung27. Resuspendieren Sie die gereinigte RNA in 10 μL nukleasefreiemH2Ound quantifizieren Sie sie, bevor sie bei −20 °C zur Verwendung in der Mikroinjektionsmischung gelagert wird.
- Bereiten Sie die beiden sgRNA-Leitfäden vor.
- Bereiten Sie das sgRNA-Plasmid mit einem Gerüst vor, indem Sie Bakterienkulturen aus dem kommerziell erhältlichen bakteriellen Agarstich XL1 Blue (siehe Materialtabelle für Details) auf die gleiche Weise wie MLM3613 oben (Schritt 2.1.1) amplifizieren, außer Kanamycin anstelle von Ampicillin.
- Die beiden Paare komplementärer einzelsträngiger Oligonukleotide (ssODNs) für die oben entworfenen sgRNA-Guides werden geglüht, indem zunächst die ssODNs in 1x Glühpuffer auf eine Konzentration von 100 μM resuspendiert werden.
- In getrennten Reaktionen für jede der beiden sgRNAs wird das Paar komplementärer ssODNs mit einem Thermocycler geglüht. 2 μg jedes komplementären ssODN-Paares mit 50 μL Glühpuffer mischen und bei 95 °C für 2 min inkubieren, dann auf 25 °C über 45 min abkühlen.
- Verdauen Sie ein kommerziell erhältliches Plasmid, das ein gRNA-Gerüst enthält. 2 μg des DR274-Plasmids werden unter Verwendung von 1 μL BsaI, 2 μL geeignetem Puffer undddH2O bis 20 μL bei 37 °C für 1 h aufgeschlossen. Bestätigen Sie die Linearisierung (optional: Aufreinigung mit einem PCR-Reinigungskit) mittels Gelelektrophorese28.
- In zwei separaten Ligationsreaktionen (eine für jede sgRNA) ligieren Sie die geglühten ssODNs mit dem linearisierten DR274-Plasmid. Verwenden Sie ein molares Insert:Vektor-Verhältnis von 3:1 und berechnen Sie die entsprechende Masse über einen Online-Ligationsrechner. Mischen Sie die erforderliche Masse des Inserts und des Vektors mit 1 μL T4-DNA-Ligase, 2 μL Ligationspuffer und ddH2O bis 12 μL und inkubieren Sie bei Raumtemperatur für 12 h.
- Transformieren Sie 2 μL des ligierten Produkts in geeignete kompetente Zellen (z. B. 10β-Zellen) unter Verwendung von Standardansätzen und amplifizieren und reinigen Sie das Produkt dann mit einem handelsüblichen Miniprep-Kit. Optional: Erstellen Sie einen Glycerinvorrat dieses Produkts.
- Transkribieren Sie die beiden sgRNAs, indem Sie 2 μg jeder Führung unter Verwendung einer 3' nachgeschalteten Restriktionsstelle linearisieren, die so nahe wie möglich am Ende der Raumsequenz liegt. Für das DR274-Plasmid linearisieren Sie mit HindIII und reinigen Sie dann die RNA-Vorlage mit einem PCR-Reinigungskit, wobei 30 μL des Elutionspuffers resuspendiert werden.
- Transkribieren Sie die beiden Guides mit einem RNA-Transkriptionskit. Befolgen Sie das Protokoll des Herstellers und reinigen Sie über Lithiumchlorid-Fällung27. Die beiden gereinigten sgRNA-Guides werden in 10 μL nukleasefreiemH2Oresuspendiert, quantifiziert und bei −20 °C zur Verwendung in der Mikroinjektionsmischung gelagert.
HINWEIS: Das RNA-Transkriptionskit kann keine 5'-Kappe oder einen Poly-A-Schwanz enthalten.
- Bereiten Sie die doppelsträngige, exogene HDR-Reportervorlage vor.
HINWEIS: Die Vorlage wird in zwei Teilen synthetisiert, einem stromaufwärts und einem stromabwärts des mVenus YFP-Reportergens. Diese beiden Segmente sind synthetische Konstrukte, die über einen kommerziellen Anbieter bestellt und dann sequentiell in das pKHR5-Plasmid (das mVenus YFP enthält) ligiert werden.- Das pKHR5-Plasmid wird hergestellt, indem Bakterienkulturen aus dem kommerziell erhältlichen DH5α-Bakterienstamm (siehe Materialtabelle für Details) auf die gleiche Weise wie MLM3613 oben (Schritt 2.1.1) amplifiziert werden.
HINWEIS: Das pKHR5-Plasmid enthält die mVenus YFP-Reportergensequenz. - Resuspendieren Sie die beiden oben entworfenen Vorlagensegmente auf 100 μM im TE-Puffer und transformieren Sie sie dann in 10β-Zellen.
- Verdauen Sie das erste Template-Segment (das dem mVenus YFP-Reportergen vorgelagerte Segment) und das pKHR5-Plasmid unter Verwendung der in Schritt 1.3.3 ausgewählten Restriktionsenzyme.
HINWEIS: Ein sequenzieller Digest von pKHR5 ist aufgrund der Nähe der ausgewählten Restriktionsstellen im MCS erforderlich. - Um das pKHR5-Plasmid für das vorgeschaltete Template-Segment vorzubereiten, werden 4 μg pKHR5 (gemäß Schritt 2.2.4) mit SalI aufgeschlossen und anschließend mit einem PCR-Reinigungskit gereinigt. Resuspendieren Sie in 30 μLddH2Ound verwenden Sie dies als Vorlage für die zweite Verdauungsreaktion mit XhoI. Reinigen Sie das Produkt mit dem PCR-Aufreinigungskit.
- Das erste Template-Segment wird durch Aufschluss von 2 μg des Template-Segments (Schritt 2.3.2), 1 μL XhoI, 1 μL SalI, 2 μL geeignetem Puffer und ddH2O bis 20 μL für 1 h bei 37 °C vorbereitet und das Produkt gelgereinigt.
- Ligieren Sie das vorgeschaltete Template-Segment aus Schritt 2.3.5 unter Verwendung der in Schritt 2.2.5 beschriebenen Reaktionsbedingungen in das vorbereitete pKHR5. Verwandeln Sie das ligierte Produkt in kompetente 10β-Zellen, amplifizieren und reinigen Sie es mit einem Miniprep (optional: Erstellen Sie einen Glycerinvorrat dieses Produkts).
- Verwenden Sie das ligierte Produkt aus Schritt 2.3.6 (das das erste Vorlagensegment enthält, das stromaufwärts des mVenus YFP-Reportergens in pKHR5-Plasmid ligiert ist) und verdauen, um sich auf das zweite (stromabwärts des mVenus-Reporters) Vorlagensegment vorzubereiten. 4 μg des ligierten Produkts (aus Schritt 2.3.6, wie in Schritt 2.2.4) mit BamHI aufgeschlossen und anschließend mit einem PCR-Reinigungskit gereinigt. Resuspendieren Sie in 30 μLddH2Ound verwenden Sie dies als Vorlage für die zweite Aufschlussreaktion mit EcoRI. Reinigen Sie das Produkt mit dem PCR-Aufreinigungskit.
- Das zweite Template-Segment wird durch Aufschluss von 2 μg des Template-Segments (Schritt 2.3.2), 1 μL BamHI, 1 μL EcoRI, 2 μL geeignetem Puffer und ddH2O bis 20 μL für 1 h bei 37 °C vorbereitet und das Produkt gelgereinigt.
- Ligate das nachgeschaltete Template-Segment in das vorbereitete pKHR5 (aus Schritt 2.3.7) unter Verwendung der in Schritt 2.2.5 beschriebenen Reaktionsbedingungen. Verwandeln Sie das ligierte Produkt in kompetente Zellen, amplifizieren und reinigen Sie es mit einem Miniprep-Kit. Erstellen Sie einen Glycerinvorrat dieses Endprodukts, der beide Vorlagensegmente enthält, die auf beiden Seiten des mVenus YFP-Reportergens in pKHR5 ligiert sind.
- Das pKHR5-Plasmid wird hergestellt, indem Bakterienkulturen aus dem kommerziell erhältlichen DH5α-Bakterienstamm (siehe Materialtabelle für Details) auf die gleiche Weise wie MLM3613 oben (Schritt 2.1.1) amplifiziert werden.
- Bereiten Sie die Mikroinjektionsmischung mit der Cas9-mRNA, zwei sgRNAs und der exogenen HDR-Reportervorlage vor.
- Mischen Sie 200 ng/μL Cas9 mRNA, 100 ng/μL jeder sgRNA und 200 ng/μL exogene HDR-Reportervorlage in 1x Injektionspuffer zu einem Endvolumen von 20 μL.
- Lagern Sie die Mikroinjektionsmischung bei −20 °C und entsorgen Sie die nicht verwendete Mischung nach drei Gefrier-Tau-Zyklen.
HINWEIS: Verwenden Sie 4 nL dieser Mikroinjektionsmischung zur Mikroinjektion in den Dottersack jedes Embryos.
3. Zucht von Zebrafischen und Mikroinjektion von Embryonen
ANMERKUNG: Protokolle für die Zebrafischzucht und die Mikroinjektion von einzelligen Embryonen wurden zuvorbeschrieben 29,30,31.
- Verwenden Sie für die Zucht Zebrafische des AB-Stammes, die 6-12 Monate alt sind. Injizieren Sie die Embryonen im Einzellerstadium etwa 40 Minuten nach der Befruchtung (siehe Abbildung 5).
ANMERKUNG: Das biologische Geschlecht der injizierten Embryonen war nicht bekannt; Der Sexualdimorphismus ist erst im Alter von etwa 3 Monaten im Alter von32 Monaten erkennbar.
4. Reportergen-Screening von CRISPR-Cas9-editierten Zebrafischlarven
- Visualisieren Sie die YFP-Integration in Zebrafischlarven nach der Mikroinjektion von CRISPR-Cas9-Komponenten, um nach erfolgreichen HDR-Bearbeitungen zu suchen.
- In einer 25 mm Petrischale 24 Zebrafischlarven 3 Tage nach der Befruchtung (dpf) in 0,3% Tricainmethansulfonat (MS-222, gepuffert auf pH 7,0-7,4 mit HEPES und Natriumhydroxid) anästhesieren, bis sie ihren selbstaufrichtenden Reflex verlieren (typischerweise 1-2 min). Nach der Anästhesie übertragen Sie jede Larve in eine einzelne Vertiefung einer 24-Well-Platte.
- Mit einem Mikroskop, das GFP / YFP nachweisen kann, untersuchen Sie die Augen jeder einzelnen Larve auf Reportergenfluoreszenz.
- Nehmen Sie Bilder jeder Larve auf und dokumentieren Sie das Vorhandensein oder Fehlen einer Reportergenexpression.
5. Phänotypisierung von CRISPR-Cas9-editierten Zebrafischlarven
- Nach dem Reportergen-Screening führen Sie eine kardiale Phänotypisierung (Herzfrequenz, Perikarddimensionen, EKG) an jeder Larve durch. Phänotyp eine gleiche Anzahl von Reportergen-positiven und -negativen Larven.
- Verwenden Sie eine CCD-Kamera (z. B. blackfly USB3) und eine Video- und Bildaufzeichnungssoftware (z. B. Micromanager für ImageJ), um die Herzfrequenz und die Perikarddimensionen zu messen, während die Larven betäubt werden.
- Um die Herzfrequenz zu messen, erstellen Sie mit Micromanager eine Region of Interest (ROI), um das Herz zu erfassen und andere Strukturen auszuschließen.
- Importieren Sie das Video als Bildsequenz in ImageJ und stellen Sie sicher, dass unter Anzahl der Bilder die richtige Anzahl von Bildern eingegeben wird.
- Nachdem die Datei geöffnet ist, verwenden Sie das Rechteckauswahlwerkzeug , um einen ROI innerhalb des Herzens zu zeichnen, aber andere bewegliche Elemente auszuschließen, und speichern Sie den ROI im ROI-Manager (analysieren Sie | Tools | ROI-Manager).
- Klicken Sie auf Plugins | installieren, wählen Sie den Herzfrequenz-Algorithmus aus, um den Code im Standardordner zu installieren, und wählen Sie dann das Plugin unten auf der Registerkarte Plugins aus. Nehmen Sie die Beats pro Minute (bpm) aus dem Popup-Fenster auf.
HINWEIS: Bilderkennungsalgorithmen wurden speziell geschrieben, um die Herzfrequenz zu erkennen, indem einzelne Pixeldichteänderungen im Zusammenhang mit ventrikulärer systolischer Kontraktion gemessen werden. Den Code finden Sie unter https://github.com/dpoburko/zFish_HR.
- Messen Sie Perikarddimensionen mit einem kostenlosen Tool wie ImageJ, um ROIs um den Perikardsack und eines der Augen frei zu zeichnen. Öffnen Sie das Bild in ImageJ und verwenden Sie das Polygonauswahlwerkzeug , um zuerst einen ROI um den Perikardsack zu zeichnen, ihn wie in Schritt 5.1.2 im ROI-Manager zu speichern und für das Auge zu wiederholen. Wählen Sie diese beiden ROIs in der ROI-Verwaltungaus und klicken Sie dann auf Messen. Notieren Sie die Fläche für jeden, um später den Bereich des Perikardsacks zu berechnen, der in jeder Larve auf die Augenpartie normalisiert ist.
- Nach Herzfrequenz- und Perikardmessungen wird das EKG einzelner Larven aufgezeichnet.
HINWEIS: Protokolle zur Aufzeichnung von Zebrafisch-EKG wurden zuvorbeschrieben 33,34,35,36.
6. Genotypisierung von CRISPR-Cas9-editierten Zebrafischlarven
- Führen Sie nach phänotypischen Analysen eine On- und potenzielle Off-Target-Genotypisierung durch, um eine genaue und präzise HDR-Genbearbeitung zu bestätigen.
- Anästhesieren Sie jede 3 dpf Larve in 0,3% MS-222 und Schwanzclip, um gDNA mit der HOTShot-Methode37 zu isolieren. Inkubieren Sie jeden ausgeschnittenen Schwanzclip in 15 μL 25 mM NaOH bei 95 °C für 20 min. Dann mit 1,5 μL Tris-HCl neutralisieren und bei 13.800 x g für 30 s zentrifugieren. Behalten Sie den Überstand bei, der extrahierte gDNA enthält.
- Erholen Sie die Larven in E3-Medien und bringen Sie sie in das Haltungssystem zurück, wenn eine weitere Entwicklung oder Untersuchung beabsichtigt ist.
- Führen Sie unter Verwendung der extrahierten gDNA als Vorlage eine PCR-basierte Sanger-Sequenzierung von On-Target- und potenziellen Off-Target-Stellen durch.
HINWEIS: Optional: Ein verschachtelter PCR-Ansatz kann für einige Genregionen von Vorteil sein. - Stellen Sie sicher, dass das On-Target-Primer-Design die Mutationsstelle und die nächstgelegene sgRNA-Bindungsstelle erfasst. Entwerfen Sie einen separaten Sequenzierungsprimer, um den Übergang vom eingefügten Homologiearm und dem Zielgen zu erkennen, um die Integration in das interessierende Gen zu bestätigen. Entwerfen Sie Primer, um die drei wichtigsten potenziellen Off-Target-Standorte zu sequenzieren, die in Schritt 1.2.3 identifiziert wurden.
HINWEIS: Guide-Design-Softwareprogramme schlagen oft Primer vor, aber Anpassungen können erforderlich sein, um optimale Ergebnisse zu erzielen. - Stellen Sie On- und Off-Target-Genotypisierung, Herzfrequenz, Perikarddimensionen, EKG-Phänotypisierung und Reportergendaten zusammen, die für jeden Zebrafisch identifizierbar sind.
Ergebnisse
Die erfolgreiche Verwendung dieses Zwei-sgRNA-Exonersatz-CRISPR-Ansatzes wird durch die Einführung und einfache Detektion einer präzisen Bearbeitung zur Entwicklung der LQTS-assoziierten Variante R56Q im zkcnh6a-Gen im Zebrafisch unterstrichen. Abbildung 6 zeigt repräsentative 3 dpf-Larven, die im einzelligen Embryostadium wie oben beschrieben mit CRISPR-Komponenten injiziert wurden. Abbildung 6A zeigt das Vorhandensein der YFP mVenus-Reportergenexpression in der Augenlinse als positiver Reporter einer erfolgreichen Template-Integration. Abbildung 6B,C zeigt Sanger-Sequenzierungschromatogramme, die aus genomischer DNA gewonnen wurden, die aus Schwanzclip-Proben von Wildtyp- bzw. Reportergen-positiven Fischen isoliert wurde. Es wurde festgestellt, dass Reporter-Gen-positive Fische die präzise Bearbeitung G bis A haben, die die R56Q-Variante in zkcnh6a einführt. Die Genotypisierung zeigte eine 100%ige Korrelation zwischen der YFP-Reportergenexpression und dem Vorhandensein des präzisen R56Q-Genedits, was dieses Fluoreszenz-Screening-Tool validiert.
Die Phänotypisierung von geneditierten Zebrafischlarven wurde bei 3 dpf durchgeführt. Abbildung 7 zeigt repräsentative Ergebnisse von Wildtyp- und R56Q-Gen-editierten Larven. Die Herzfrequenz wurde wie oben beschrieben per Videoaufnahme erfasst. Ein Beispiel für die Messung der Perikarddimensionen als Verhältnis der Augenfläche ist dargestellt (Abbildung 7A). Abbildung 7B stellt die Herzfrequenz gegen normalisierte Perikarddimensionen dar und hebt einen Trend der Bradykardie mit zunehmendem Perikardödem hervor, der mit Störungen der kardialen Repolarisation imZebrafisch 8,38,39,40 assoziiert ist. Abbildung 7C zeigt ein repräsentatives Beispiel für EKG-Aufnahmen von 3 dpf-Larven. Standardintervalle (QT, QRS) wurden aus gemittelten EKG-Signalen gemessen.

Abbildung 1: Integration der HDR-Vorlage in das Zebrafischgenom. Dunkelgrau, Homologiearme; grüne, sgRNA-Leitziele mit stiller Mutation, um ein erneutes Schneiden von Cas9 zu verhindern; hellgrau, Zielexon von Interesse; rote Linie, Punktmutation; gelbes, mVenus YFP-Reportergen unter einem α-Kristallin-Promotor; Gestrichelte Linien zeigen die Homologie an. Hier war der gezielte präzise Edit R56Q im Exon 2 des zkcnh6a-Gens . Abkürzungen: HDR = homologiegesteuerte Reparatur; sgRNA = Single-Guide-RNA; YFP = gelb fluoreszierendes Protein; DSB = doppelsträngiger Bruch; WT = Wildtyp. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 2: Zusammenfassung der Schritte zur Entwicklung präziser Bearbeitungen in Zebrafischgenen mit dem Zwei-sgRNA-CRISPR-Cas9-Ansatz (zugehörige Protokollschrittnummern sind in Klammern angegeben). Abkürzungen: sgRNA = Single-Guide-RNA; YFP = gelb fluoreszierendes Protein; gDNA = genomische DNA; EKG = Elektrokardiogramm; dpf = Tage nach der Befruchtung; MS-222 = Tricainmethansulfonat. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
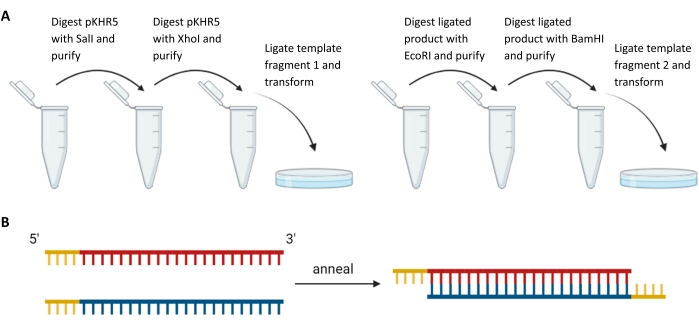
Abbildung 3: Herstellung exogener Template-Fragmente und sgRNA-Guides . (A) Sequentieller Aufschluss und Ligation von Template-Fragmenten strom- und nachgeschaltet der mVenus YFP-Reportergensequenz in pKHR5. (B) Annealing von komplementären sgRNA-Paaren mit Restriktionsüberhang für die Ligation in DR274. Abkürzungen: sgRNA = Single-Guide-RNA; YFP = gelb fluoreszierendes Protein. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
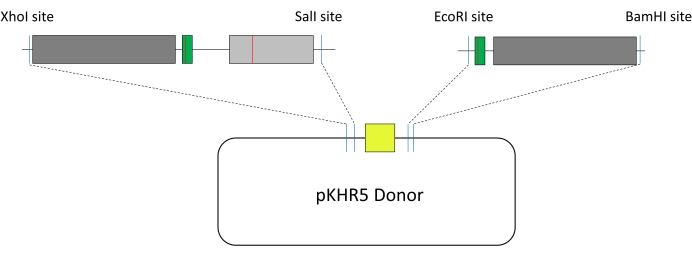
Abbildung 4: Aufbau der HDR-Vorlage. Dunkelgrau, Homologiearme; grüne, sgRNA-Leitziele mit stiller Mutation, um ein erneutes Schneiden von Cas9 zu verhindern; hellgrau, Zielexon von Interesse; rote Linie, Punktmutation; gelbes, mVenus YFP-Reportergen unter einem α-Kristallin-Promotor; dunkelblaue Linie, Einschränkungsstellen hinzugefügt. Die beiden Vorlagenfragmente sind in den pKHR5-Plasmiddonor integriert. Abkürzungen: HDR = homologiegesteuerte Reparatur; sgRNA = Single-Guide-RNA. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 5: Mikroinjektion von einzelligen Zebrafischembryonen mit CRISPR-Cas9-Komponenten. Maßstabsbalken = 0,5 mm. Abkürzungen: HDR = homologiegesteuerte Reparatur; sgRNA = Single-Guide-RNA. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 6: Einfacher Nachweis der mVenus YFP-Reportergenfluoreszenz zeigt eine positive exogene HDR-Template-Integration in das Zielgen. (A) Beispiel für die mVenus YFP-Expression in einem Zebrafischauge (Pfeil) in einer bearbeiteten Zebrafischlarve. (B) Erfolgreiche Bearbeitungen werden durch Sequenzierung von Chromatogrammen bestätigt (links, WT; rechts, R56Q-Bearbeitung). Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 7: Phänotypische Analyse kardialer Konsequenzen in 3 dpf Zebrafischen nach dem präzisen R56Q-Edit im Zielgen zkcnh6a. (A) Bilderkennung der Perikarddimensionen relativ zur Augengröße mit dem Polygonwerkzeug in ImageJ. Die Grenzen des Perikardsacks wurden vom Benutzer aus einem einzigen Aufnahmerahmen anhand von Veränderungen der Transluzenz und Pigmentierung markiert. Beispiele für normale Perikarddimensionen und Perikarderguss werden gezeigt. Maßstabsbalken = 0,5 mm. (B) Korrelation zwischen Perikardmaßen (relativ zur Augendimension) und Herzfrequenz,R2 = 0,33. (C) Beispiel für eine EKG-Aufzeichnung eines 3-dpf-Zebrafischlarvenherzens (links) und gemittelter Komplexe (rechts). Herzfrequenz, 131 Schläge pro Minute; herzfrequenzkorrigiertes QTc-Intervall, 460 ms. Abkürzungen: dpf = Tage nach der Befruchtung; EKG = Elektrokardiogramm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Diskussion
Die Entwicklung präziser Genbearbeitungen mit CRISPR-Cas9 wird durch die geringe Effizienz von HDR-Mechanismen und deren effiziente Detektion herausgefordert. Hier wird ein CRISPR-Cas9-basierter Zwei-sgRNA-Exonersatzansatz beschrieben, der präzise Schnitte im Zebrafisch mit einfacher visueller Erkennung positiver Bearbeitungen erzeugt. Die Wirksamkeit dieses Ansatzes wird durch die Erzeugung präziser Bearbeitungen im zkcnh6a-Gen nachgewiesen. Diese Arbeit zeigt, wie die Herzfunktion in geneditierten Zebrafischlarven anhand nicht-invasiver phänotypischer Messungen der Herzfrequenz, der perikardialen Dimensionen und der EKG-Morphologie beurteilt werden kann. Dieser Ansatz, von der Einführung eines Geneditiers bis zur phänotypischen Bewertung, kann von Anfang bis Ende innerhalb von ca. 1 Woche abgeschlossen werden.
Die Vorteile des obigen Bearbeitungs- und Phänotypisierungsansatzes sind die Leichtigkeit des CRISPR-Modifikationsdesigns, die breite Anwendbarkeit in mehreren physiologischen Systemen, die Fähigkeit, große Gene oder Genfragmente einzufügen, und die Fähigkeit, Varianteneffekte longitudinal über Entwicklung und Generationen hinweg zu verfolgen. Der Erfolg präziser Bearbeitungen in diesem Ansatz kann mit der Kombination der großen Vorlagengröße (aufgrund des Reportergeninserts und der langen Homologiearme), von der gezeigt wurde, dass sie die Effizienz von Bearbeitungen in Zebrafischen14 erhöht, und der Zwei-sgRNA-Guides-Strategie, die effektiv bei Zebrafisch-TALEN-induziertenBearbeitungen 8 eingesetzt wurde, zusammenhängen.
Eine besondere Stärke des beschriebenen Ansatzes ist die Fähigkeit, große Gene oder Genfragmente einzuschleusen. Dies kann beispielsweise nützlich sein, um menschliche Orthologe41 einzufügen, was eine klinisch besser übersetzbare Charakterisierung und einen Vergleich zwischen Orthologen ermöglicht. Alternativ könnten auch Gene, die Cas-Enzyme kodieren, eingefügt werden, was eine Reihe von Zebrafischen mit in vivo CRISPR-Bearbeitungsmechanismen ermöglicht, die ein induzierbares System bieten. In ähnlicher Weise könnten alternative CRISPR-Mechanismen, wie z. B. Prime Editing, integriert werden und zu einer Reihe von Zebrafischen führen, die leicht präzise und effizient bearbeitet werden können.
Trotz der Vorteile dieses Ansatzes gibt es einige Einschränkungen. Erstens wurden nur ein einziges Gen und ein Locus modifiziert, und weitere Tests an anderen Stellen oder in anderen Genen sind notwendig, um zu bewerten, wie breit anwendbar dieser Ansatz ist. Aufgrund der langen erforderlichen Homologiearme sind die Kosten für das Vorlagendesign höher. Dies kann jedoch durch ein effizientes Screening ausgeglichen werden. Eine weitere Einschränkung besteht darin, dass der Screening-Ansatz eine Fluoreszenzdetektion erfordert. Die optischen Anforderungen sind jedoch relativ gering und können zu relativ geringen Kosten kundenspezifisch angefertigt oder kommerziell erworben werden. Die Verwendung eines Zwei-sgRNA-Ansatzes erhöht die Anzahl potenzieller Off-Target-Ereignisse; Dies wird jedoch wahrscheinlich durch die geringere Wahrscheinlichkeit gemildert, dass die beiden sgRNA-Guides beide so geglüht werden, dass die Integration der Vorlage erleichtert wird, um die Reportergenexpression zu erhalten. Schließlich kann die Verwendung von Cas9-mRNA zu Mosaikismen führen, da Cas9 erst in späteren Entwicklungsstadien aktiv ist. Dies könnte durch die Sequenzierung bestimmter Gewebetypen erklärt werden; Angesichts der Größe der Zebrafischlarven ist dies jedoch technisch anspruchsvoll.
Zusammenfassend lässt sich sagen, dass dieser CRISPR-Cas9 Zwei-sgRNA-Präzisions-Editieransatz im Zebrafisch die einfache visuelle Erkennung positiver Schnitte ermöglicht und angepasst werden kann, um große Gene von Interesse an jedem Ort zu integrieren. In Kombination mit phänotypischen Maßnahmen ermöglicht dies eine zuverlässige und hohe Durchsatzplattform für die Untersuchung klinisch relevanter kardialer Varianten.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Diese Forschung wurde durch ein Canadian Institutes of Health Research Project Grant (T.W.C.) und Natural Sciences and Engineering Research Council of Canada Discovery Grants (T.W.C.) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Program | |||
| CRISPOR | TEFOR Infrastructure | ||
| ENSEMBL | European Bioinformatics Institute | ||
| ImageJ | National Institutes of Health (NIH) | ||
| Micro-Manager | Open Source (Github) | ||
| NEBiocalculator | New England Biolabs (NEB) | ||
| EQUIPMENT | |||
| 24-well Plate | VWR | ||
| 25 mm Petri Dish | VWR | ||
| Blackfly USB3 Camera | Teledyne FLIR | ||
| C1000 Thermal Cycler | Bio-Rad | ||
| Centrifuge 5415C | Eppendorf | ||
| EZNA Gel Extraction Kit | Omega Biotek | ||
| MAXIscript T7 Transcription Kit | Invitrogen | ||
| MaxQ 5000 Incubator | Barnstead Lab Line | ||
| Miniprep Kit | Qiagen | ||
| mMessage mMachine T7 Ultra Transcription Kit | Invitrogen | ||
| ND1000 Spectrophotometer | Nanodrop | ||
| PCR Purification Kit | Qiagen | ||
| PLI 100A Picoinjector | Harvard Apparatus | ||
| PowerPac Basic Power Supply | Bio-Rad | ||
| Stemi 305 Steroscope | Zeiss | ||
| Wide Mini Sub Cell GT Electrophoresis System | Bio-Rad | ||
| ZebTec Zebrafish Housing System | Tecniplast | ||
| SERVICES | |||
| Gene Synthesis | Genewiz | ||
| Sanger Sequencing | Genewiz | ||
| REAGENTS | |||
| 10β Competent Cells | NEB | ||
| 10X PCR Buffer | Qiagen | ||
| 100 mM Nucleotide Mixture | ABM | ||
| Ampicillin | Sigma | ||
| BamHI Endonuclease w/ buffer | NEB | ||
| BsaI Endonuclease w/ buffer | NEB | ||
| DR274 Plasmid (XL1 Blue bacterial agar stab) | Addgene | ||
| EcoRI Endonuclease w/ buffer | NEB | ||
| Glycerol | |||
| HEPES | Sigma | ||
| HindIII Endonuclease w/ buffer | NEB | ||
| Kanamycin | Sigma | ||
| Methylene Blue | Sigma | ||
| MLM3613 Plasmid (XL1 Blue bacterial agar stab) | Addgene | ||
| MS-222 (Tricaine) | Sigma | ||
| pKHR5 Plasmid (DH5α bacterial agar stab) | Addgene | ||
| PmeI Endonuclease w/ buffer | NEB | ||
| SalI Endonuclease w/ buffer | NEB | ||
| Sodium Hydroxide | Sigma | ||
| T4 Ligase w/ buffer | Sigma | ||
| Taq Polymerase | Qiagen | ||
| TE Buffer | Sigma | ||
| Tris Hydrochloride | Sigma | ||
| XhoI Endonuclease w/ buffer | NEB | ||
| RECIPES | |||
| Solution | Component | Supplier | |
| Annealing Buffer (pH 7.5-8.0) | 10 mM Tris | Sigma | |
| 50 mM NaCl | Sigma | ||
| 1 mM EDTA | Sigma | ||
| E3 Media (pH 7.2) | 5 mM NaCl | Sigma | |
| 0.17 mM KCl | Sigma | ||
| 0.33 mM CaCl2 | Sigma | ||
| 0.33 mM MgSO4 | Sigma | ||
| Injection Buffer (pH 7.5) | 20 mM HEPES | Sigma | |
| 150 mM KCl | Sigma |
Referenzen
- Zarei, A., Razban, V., Hosseini, S. E., Tabei, S. M. B. Creating cell and animal models of human disease by genome editing using CRISPR/Cas9. The Journal of Gene Medicine. 21 (4), 3082(2019).
- Lee, H., Yoon, D. E., Kim, K. Genome editing methods in animal models. Animal Cells and Systems. 24 (1), 8-16 (2020).
- Li, Q., et al. Applications of genome editing technology in animal disease modeling and gene therapy. Computational and Structural Biotechnology Journal. 17, 689-698 (2019).
- Gut, P., Reischauer, S., Stainier, D. Y. R., Arnaout, R. Little fish, big data: Zebrafish as a model for cardiovascular and metabolic disease. Physiological Reviews. 97 (3), 889-938 (2017).
- Kegel, L., et al. Forward genetic screen using zebrafish to identify new genes involved in myelination. Oligodendrocytes: Methods and Protocols. 1936, 185-209 (2019).
- Shah, A. N., Davey, C. F., Whitebirch, A. C., Miller, A. C., Moens, C. B. Rapid reverse genetic screening using CRISPR in zebrafish. Nature Methods. 12 (6), 535-540 (2015).
- González-Rosa, J. M. Zebrafish models of cardiac disease: From fortuitous mutants to precision medicine. Circulation Research. 130 (12), 1803-1826 (2022).
- Hoshijima, K., Jurynec, M. J., Grunwald, D. J. Precise editing of the Zebrafish genome made simple and efficient. Developmental Cell. 36 (6), 654-667 (2016).
- Albadri, S., Del Bene, F., Revenu, C. Genome editing using CRISPR/Cas9-based knock-in approaches in zebrafish. Methods. 121-122, 77-85 (2017).
- Armstrong, G. A. B., et al. Homology directed knockin of point mutations in the zebrafish tardbp and fus genes in ALS using the CRISPR/Cas9 system. PLoS One. 11 (3), 0150188(2016).
- Bai, H., et al. CRISPR/Cas9-mediated precise genome modification by a long ssDNA template in zebrafish. BMC Genomics. 21 (1), 67(2020).
- de Vrieze, E., et al. Efficient generation of knock-in zebrafish models for inherited disorders using CRISPR-Cas9 ribonucleoprotein complexes. International Journal of Molecular Sciences. 22 (17), 9429(2021).
- Eschstruth, A., Schneider-Maunoury, S., Giudicelli, F. Creation of zebrafish knock-in reporter lines in the nefma gene by Cas9-mediated homologous recombination. Genesis. 58 (1), 23340(2020).
- Irion, U., Krauss, J., Nüsslein-Volhard, C. Precise and efficient genome editing in zebrafish using the CRISPR/Cas9 system. Development. 141 (24), 4827-4830 (2014).
- Kimura, Y., Hisano, Y., Kawahara, A., Higashijima, S. Efficient generation of knock-in transgenic zebrafish carrying reporter/driver genes by CRISPR/Cas9-mediated genome engineering. Scientific Reports. 4 (1), 6545(2014).
- Levic, D. S., Yamaguchi, N., Wang, S., Knaut, H., Bagnat, M. Knock-in tagging in zebrafish facilitated by insertion into non-coding regions. Development. 148 (19), (2021).
- Prykhozhij, S. V., et al. Optimized knock-in of point mutations in zebrafish using CRISPR/Cas9. Nucleic Acids Research. 46 (17), 102(2018).
- Wierson, W. A., et al. Efficient targeted integration directed by short homology in zebrafish and mammalian cells. eLife. 9, 53968(2020).
- Boel, A., et al. CRISPR/Cas9-mediated homology-directed repair by ssODNs in zebrafish induces complex mutational patterns resulting from genomic integration of repair-template fragments. Disease Models & Mechanisms. 11 (10), (2018).
- Tessadori, F., et al. Effective CRISPR/Cas9-based nucleotide editing in zebrafish to model human genetic cardiovascular disorders. Disease Models & Mechanisms. 11 (10), (2018).
- Zhang, Y., Zhang, Z., Ge, W. An efficient platform for generating somatic point mutations with germline transmission in the zebrafish by CRISPR/Cas9-mediated gene editing. The Journal of Biological Chemistry. 293 (17), 6611-6622 (2018).
- Vornanen, M., Hassinen, M. Zebrafish heart as a model for human cardiac electrophysiology. Channels. 10 (2), 101-110 (2016).
- Nemtsas, P., Wettwer, E., Christ, T., Weidinger, G., Ravens, U. Adult zebrafish heart as a model for human heart? An electrophysiological study. Journal of Molecular and Cellular Cardiology. 48 (1), 161-171 (2010).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biology. 17 (1), 148(2016).
- Liang, X., Potter, J., Kumar, S., Ravinder, N., Chesnut, J. D. Enhanced CRISPR/Cas9-mediated precise genome editing by improved design and delivery of gRNA, Cas9 nuclease, and donor DNA. Journal of Biotechnology. 241, 136-146 (2017).
- Richardson, C. D., Ray, G. J., DeWitt, M. A., Curie, G. L., Corn, J. E. Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nature Biotechnology. 34 (3), 339-344 (2016).
- Sambrook, J., Fritsch, E. F., Maniatis, T. Precipitation of Large RNAs with Lithium Chloride. Molecular Cloning: A Laboratory Manual, Book 3. E.15. , Cold Spring Harbor Laboratory. Long Island, NY. (1989).
- Sambrook, J., Fritsch, E. F., Maniatis, T. Agarose Gel Electrophoresis. Molecular Cloning: A Laboratory Manual, Book 1. , Cold Spring Harbor Laboratory. Long Island, NY. 3-20 (1989).
- Sorlien, E. L., Witucki, M. A., Ogas, J. Efficient production and identification of CRISPR/Cas9-generated gene knockouts in the model system Danio rerio. Journal of Visualized Experiments. (138), e56969(2018).
- Avdesh, A., et al. Regular care and maintenance of a zebrafish (Danio rerio) laboratory: An introduction. Journal of Visualized Experiments. (69), e4196(2012).
- Rosen, J. N., Sweeney, M. F., Mably, J. D. Microinjection of zebrafish embryos to analyze gene function. Journal of Visualized Experiments. (25), e1115(2009).
- Kossack, M. E., Draper, B. W. Genetic regulation of sex determination and maintenance in zebrafish (Danio rerio). Current Topics in Developmental Biology. 134, 119-149 (2019).
- Tanaka, Y., et al. Functional analysis of KCNH2 gene mutations of type 2 long QT syndrome in larval zebrafish using microscopy and electrocardiography. Heart and Vessels. 34 (1), 159-166 (2019).
- Dhillon, S. S., et al. Optimisation of embryonic and larval ECG measurement in zebrafish for quantifying the effect of QT prolonging drugs. PLoS One. 8 (4), 60552(2013).
- Yu, F., et al. Evolving cardiac conduction phenotypes in developing zebrafish larvae: Implications to drug sensitivity. Zebrafish. 7 (4), 325-331 (2010).
- Hurst, R. M. Development and optimization of tools for embryonic electrocardiograph recording for heart dysfunction in zebrafish. , University of Birmingham. PhD thesis (2018).
- Meeker, N. D., Hutchinson, S. A., Ho, L., Trede, N. S. Method for isolation of PCR-ready genomic DNA from zebrafish tissues. BioTechniques. 43 (5), 610-614 (2007).
- Arnaout, R., et al. Zebrafish model for human long QT syndrome. Proceedings of the National Academy of Sciences of the United States of America. 104 (27), 11316-11321 (2007).
- Milan, D. J., Peterson, T. A., Ruskin, J. N., Peterson, R. T., MacRae, C. A. Drugs that induce repolarization abnormalities cause bradycardia in zebrafish. Circulation. 107 (10), 1355-1358 (2003).
- Langheinrich, U., Vacun, G., Wagner, T. Zebrafish embryos express an orthologue of HERG and are sensitive toward a range of QT-prolonging drugs inducing severe arrhythmia. Toxicology and Applied Pharmacology. 193 (3), 370-382 (2003).
- MacRae, C. A. Cardiac arrhythmia: In vivo screening in the zebrafish to overcome complexity in drug discovery. Expert Opinion on Drug Discovery. 5 (7), 619-632 (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten