Method Article
CRISPR-Cas9-опосредованные точные правки в сердцах рыбок данио
В этой статье
Резюме
Этот протокол описывает подход к облегчению точного внесения изменений в эмбрионы рыбок данио с использованием технологии CRISPR-Cas9. Представлен конвейер фенотипирования, чтобы продемонстрировать применимость этих методов для моделирования варианта гена, связанного с синдромом удлиненного интервала QT.
Аннотация
Кластеризованные регулярно чередующиеся короткие палиндромные повторы (CRISPR) на животных моделях позволяют проводить точные генетические манипуляции для изучения физиологических явлений. Рыбки данио использовались в качестве эффективной генетической модели для изучения многочисленных вопросов, связанных с наследственными заболеваниями, развитием и токсикологией на уровне всего органа и организма. Благодаря хорошо аннотированному и нанесенному на карту геному рыбки данио были разработаны многочисленные инструменты для редактирования генов. Тем не менее, эффективность генерации и простота обнаружения точных изменений с использованием CRISPR является ограничивающим фактором. Здесь описан подход на основе CRISPR-Cas9 с простым обнаружением точных изменений в гене, ответственном за реполяризацию сердца и связанном с электрическим расстройством, синдромом длинного QT (LQTS). Этот подход с двумя одноруковой РНК (sgRNA) иссекает и заменяет целевую последовательность и связывает генетически закодированный ген-репортер. Полезность этого подхода демонстрируется описанием неинвазивных фенотипических измерений электрической функции сердца у личинок рыбок данио дикого типа и генетически отредактированных. Такой подход позволяет эффективно изучать связанные с заболеванием варианты в целом организме. Кроме того, эта стратегия предлагает возможности для вставки экзогенных последовательностей по выбору, таких как репортерные гены, ортологи или редакторы генов.
Введение
Стратегии редактирования генов на основе CRISPR на животных моделях позволяют изучать генетически наследственные заболевания, развитие и токсикологию на уровне всего организма 1,2,3. Рыбки данио представляют собой мощную модель, которая ближе во многих физиологических аспектах к людям, чем мышиные или человеческие клеточные модели4. Обширный спектр генетических инструментов и стратегий был использован у рыбок данио как для прямого5, так и для обратного генетического скрининга6. Всестороннее генетическое картирование и аннотация рыбок данио облегчили подходы к редактированию генов в качестве основного метода для разработки целевых нокаутов генов (KOs) и точных нокаутов (KIs)7.
Несмотря на это, генерация точных изменений KI у рыбок данио ограничена низкой эффективностью и сложностью точного обнаружения. Хотя транскрипционные эффекторные нуклеазы (TALEN) были успешно использованы и оптимизированы для KIs8, CRISPR обеспечивает улучшенную стратегию редактирования генов с более простым таргетированием sgRNA. Многочисленные исследования использовали CRISPR для получения точных KIs у рыбок данио 9,10,11,12,13,14,15,16,17,18,19,20, хотя эти изменения, полученные с помощью CRISPR-опосредованного гомологического восстановления (HDR), как правило, неэффективны с низким внутренним успехом. показатели, требующие генотипирования в качестве основного экрана 9,10,14,21. Это демонстрирует необходимость эффективной системы KI CRISPR у рыбок данио, а также надежной высокопроизводительной системы для обнаружения точных правок.
Цель этого исследования состояла в том, чтобы описать платформу для генерации точного сердечного гена KI в сердцах рыбок данио с простым и высокопроизводительным обнаружением успешных правок. Описан подход замены экзонов на основе CRISPR-Cas9 с двумя сгРНК, который основан на подходеTALEN 8. Этот подход включает в себя иссечение целевой последовательности с использованием двух сгРНК-направляющих и замену экзогенной шаблонной последовательностью, содержащей интересующий КИ, а также генетически закодированный интронный ген-репортер (рисунок 1). Интеграция генетически закодированного флуоресцентного репортера в интронную последовательность гена-мишени позволяет эффективно обнаруживать положительные изменения. Затем описывается фенотипирующая платформа для оценки электрической функции сердца у личинок рыбок данио для неинвазивной характеристики вариантов генов, связанных с унаследованным LQTS, сердечным электрическим расстройством, которое предрасполагает людей к внезапной сердечной смерти.
Эти подходы расширят доступ и использование изменений генов KI рыбок данио для моделирования наследственных заболеваний и решения биологических и физиологических вопросов, таких как картирование паттернов экспрессии генов и регуляция развития. Поскольку сердца рыбок данио лучше соответствуют сердечным электрофизиологическим характеристикам человека, чем мышиные модели, они могут быть особенно привлекательными в качестве генетически трактуемой системы для моделирования сердечных заболеваний 7,22,23.
протокол
Исследования с использованием рыбок данио были проведены в соответствии с политикой и процедурами Комитета по уходу за животными Университета Саймона Фрейзера и Канадского совета по уходу за животными и были завершены в соответствии с протоколом No 1264K-18.
1. Проектирование компонентов CRISPR для точного редактирования
- Чтобы разработать двух сгРНК-направляющие, которые будут использоваться для иссечения последовательности, содержащей целевой сайт KI, сначала определите ортолог рыбок данио для интересующего гена.
ПРИМЕЧАНИЕ: На рисунке 2 представлен краткий обзор шагов по проектированию точных изменений с использованием подхода CRISPR-Cas9 с двумя сгРНК. - Затем используйте программный инструмент для проектирования, такой как CRISPOR24, который включает в себя выбор для Danio rerio в качестве вида и фермента Cas, который будет использоваться.
ПРИМЕЧАНИЕ: Геном, представляющим интерес для этого исследования , был zkcnh6a (идентификатор транскрипта Ensembl: ENSDART00000090809.6; UniProt Protein ID: B3DJX4), а целевой мутацией была аминокислота R56Q.- Для подхода с двумя сгРНК выберите одно местоположение sgRNA, которое предшествует целевому экзону, и вторую sgRNA, которая расположена в непосредственном нисходящем интроне.
- Убедитесь, что выбранные sgRNAs имеют высокую специфичность и низкое прогнозируемое нецелевое связывание. Используйте рейтинги CRISPOR для определения направляющих с минимальным нецелевым привязкой. Не рассматривайте направляющие без несоответствий в начальной последовательности потенциальных нецелевых целей.
- Определите наиболее вероятные потенциальные нецелевые сайты (на основе показателей CRISPOR, выберите три лучших экзонных потенциальных нецелевых сайта) для генотипирования секвенирования Сэнгера на основе ПЦР на шаге 6.1.3.
- После того, как два sgRNAs были выбраны, получите обратный комплемент для каждого, так что есть четыре олигонуклеотида, которые будут использоваться: два комплементарных олиго, которые предшествуют мутации, и два комплементарных олиго, которые находятся ниже по течению.
- На каждом из четырех олиго добавить совместимые участки ограничения для включения в направляющую плазмиду по выбору; для интеграции в плазмиду DR274 используйте 5-футовый сайт ограничения BsaI для создания свеса. Убедитесь, что сайт распознавания Bsa1 спроектирован на 5-футовом конце направляющего, выбранного из CRISPOR, а сайт распознавания Bsa1 спроектирован на 5-дюймовом конце дополнительных нитей для обеспечения правильной ориентации направляющих в плазмиде DR274 (см. Рисунок 3).
- Чтобы разработать экзогенный шаблон, используемый для HDR у рыбок данио (рисунок 1), выберите два фрагмента последовательности, которые будут фланкировать ген репортера mVenus YFP, размещенный в плазмиде pKHR5.
ПРИМЕЧАНИЕ: Предполагаемая модификация/редактирование может быть включена как в восходящий, так и в нисходящий фрагмент.- Используя Ensembl, найдите целевой сайт в интересующей генной последовательности, включая примерно 2 кб фланкирующей последовательности (гомологическое плечо), которая будет использоваться для создания шаблона.
ПРИМЕЧАНИЕ: Гомологические гербы могут быть симметричными или асимметричными25,26 и приблизительно по 1 кб каждый, вверх и вниз по течению от целевого участка. - Разделите шаблон на два сегмента, которые будут вставлены по обе стороны от гена репортера mVenus YFP (см. рисунок 4). Убедитесь, что разделенный сайт находится в интроне, чтобы последовательность кодирования не прерывалась.
ПРИМЕЧАНИЕ: Если ген хорошо характеризуется, проверьте функциональные роли в интроне, такие как сайты сплайсинга или регуляторные области. Области, близкие к концам 5' или 3', чаще участвуют в сплайсинге мРНК. - Включите изменения в шаблонную последовательность, которые включают в себя i) тихие мутации в направляющем протоспейсере, смежном мотиве (PAM) или последовательности семян (имейте в виду альтернативные сайты PAM, на которые может быть нацелен фермент Cas), чтобы предотвратить резку фермента Cas; ii) изменение интереса; iii) создание участков рестрикции эндонуклеазы для облегчения клонирования в плазмиду pKHR5, которая содержит репортерный ген mVenus YFP (см. рисунок 3).
ПРИМЕЧАНИЕ: В этом исследовании первый сегмент шаблона содержал XhoI вверх по течению от сайта мутации R56Q и SalI вниз по течению, в то время как второй сегмент шаблона имел EcoRI вверх по течению от направляющей целевой последовательности и BamHI вниз по течению. Если какой-либо из выбранных участков ограничения присутствует в последовательности шаблонов, потребуются мутации, чтобы заглушить их, или могут быть использованы альтернативные подходы, такие как сборка Гибсона.
- Используя Ensembl, найдите целевой сайт в интересующей генной последовательности, включая примерно 2 кб фланкирующей последовательности (гомологическое плечо), которая будет использоваться для создания шаблона.
2. Подготовка компонентов CRISPR для микроинъекции эмбриона
- Подготовьте Cas9 к микроинъекции за 1 неделю до микроинъекций. Используйте белок Cas9 или получайте мРНК Cas9 через транскрипцию in vitro .
ПРИМЕЧАНИЕ: В этом исследовании использовалась мРНК Cas9, поскольку эффективность, как правило, была выше.- Амплифицировать бактериальные культуры коммерчески доступного бактериального агарового колба XL1 Blue (содержащего плазмиду Cas9) с использованием соответствующего антибиотика, такого как ампициллин. Используйте 675 мкл жидкой культуры (с 325 мкл глицерина) для создания резервного запаса глицерина для длительного хранения при −80 °C.
- Используйте оставшуюся часть жидкой культуры для очистки минипрепа в соответствии с протоколом, поставляемым с комплектом miniprep. Повторно суспендируют конечную очищенную ДНК в 50 мкл предоставленного буфера элюирования. Количественно оцените продукт с помощью спектрофотометра для изучения выхода и чистоты.
- Линеаризировать 2 мкг очищенной ДНК с помощью рестрикционного переваривания, используя соответствующий фермент рестрикции и используя соответствующий буфер и время инкубации, как указано для интересующего фермента.
- Очистите линеаризированную плазмиду с помощью набора для очистки ПЦР, повторно используя ее в 30 мкл предоставленного буфера элюирования.
- После количественной оценки продукта используйте его в качестве шаблона для транскрипции in vitro с использованием соответствующего набора транскрипции для интересующего промоутера. Следуйте предоставленному протоколу и очищайте с помощью осаждения хлорида лития27. Повторно суспендируют очищенную РНК в 10 мкл не содержащего нуклеазыH2Oи количественно оценивают ее перед хранением при −20 °C для использования в смеси микроинъекций.
- Подготовьте два проводника sgRNA.
- Подготавливают плазмиду sgRNA со каркасом путем амплификации бактериальных культур из коммерчески доступного бактериального агарового колба XL1 Blue (см. Таблицу материалов для деталей) так же, как MLM3613 выше (этап 2.1.1), за исключением использования канамицина вместо ампициллина.
- Отжиг двух пар комплементарных одноцепочечных олигонуклеотидов (ssODNs) для направляющих sgRNA, разработанных выше, путем первого повторного использования ssODNs в 1x буфере отжига до концентрации 100 мкМ.
- В отдельных реакциях для каждой из двух sgRNAs отжиг пары комплементарных ssODN с помощью теплового циклера. Смешайте 2 мкг каждой комплементарной пары ssODN с 50 мкл буфера отжига и инкубируйте при 95 °C в течение 2 мин, затем охладите до 25 °C в течение 45 мин.
- Переваривает коммерчески доступную плазмиду, содержащую каркас гРНК. Переваривайте 2 мкг плазмиды DR274, используя 1 мкл BsaI, 2 мкл соответствующего буфера и ddHот 2O до 20 мкл при 37 °C в течение 1 ч. Подтвердите линеаризацию (необязательно: очистите с помощью набора для очистки ПЦР) с помощью гелевого электрофореза28.
- В двух отдельных реакциях лигирования (по одной для каждой sgRNA) лигальгируйте отожженные ssODNs с линеаризированной плазмидой DR274. Используйте молярное отношение вставки к вектору 3:1, вычисляя соответствующую массу с помощью онлайн-калькулятора лигирования. Смешайте необходимую массу вставки и вектора с 1 мкл ДНК-лигазы Т4, 2 мкл лигационного буфера и ddH2Oдо 12 мкл и инкубируйте при комнатной температуре в течение 12 ч.
- Преобразуйте 2 мкл лигированного продукта в соответствующие компетентные клетки (такие как 10β-клетки) с использованием стандартных подходов, а затем усиливайте и очищайте продукт с помощью коммерчески доступного комплекта Miniprep. По желанию: создайте глицериновый запас этого продукта.
- Транскрибируйте две sgRNAs путем линеаризации 2 мкг каждого направляющего, используя 3'-й нисходящий участок ограничения, который максимально приближен к концу пространственной последовательности. Для плазмиды DR274 линеаризуйте с помощью HindIII, а затем очистите шаблон РНК с помощью набора для очистки ПЦР, повторно используя в 30 мкл буфера элюирования.
- Транскрибируйте два проводника с помощью набора транскрипции РНК. Следуйте протоколу производителя и очищайте с помощью осаждения хлорида лития27. Повторно суспендируют два очищенных проводника сгРНК в 10 мкл не содержащего нуклеазыH2O, количественно оценивают и хранят при −20 °C для использования в смеси микроинъекций.
ПРИМЕЧАНИЕ: Набор для транскрипции РНК не может включать 5-футовый колпачок или поли-А хвост.
- Подготовьте двухцепочечный экзогенный шаблон репортера HDR.
ПРИМЕЧАНИЕ: Шаблон синтезирован в двух частях, одна вверх по течению и одна вниз по течению гена репортера mVenus YFP. Эти два сегмента представляют собой синтетические конструкции, упорядоченные через коммерческого поставщика, а затем последовательно перевязанные в плазмиду pKHR5 (которая содержит mVenus YFP).- Получают плазмиду pKHR5 путем амплификации бактериальных культур из коммерчески доступного бактериального штамма DH5α (см. Таблицу материалов для деталей) таким же образом, как MLM3613 выше (этап 2.1.1).
ПРИМЕЧАНИЕ: Плазмида pKHR5 содержит репортерную последовательность гена mVenus YFP. - Повторно суспендируйте два сегмента шаблона, разработанные выше, до 100 мкМ в буфере TE, а затем преобразуйте в ячейки 10β.
- Переварите первый сегмент шаблона (тот, который находится выше по течению от репортерного гена mVenus YFP) и плазмиду pKHR5, используя ферменты рестрикции, выбранные на этапе 1.3.3.
ПРИМЕЧАНИЕ: Последовательный дайджест pKHR5 необходим в связи с непосредственной близостью выбранных участков ограничения в MCS. - Чтобы подготовить плазмиду pKHR5 для сегмента шаблона, переварите 4 мкг pKHR5 (в соответствии с шагом 2.2.4) с SalI, а затем очистите с помощью набора для очистки ПЦР. Повторно суспендировать в 30 мкл ddH2Oи использовать его в качестве шаблона для второй реакции пищеварения с XhoI. Очистите продукт с помощью набора для очистки ПЦР.
- Подготавливают первый сегмент шаблона путем переваривания 2 мкг сегмента шаблона (этап 2.3.2), 1 мкл XhoI, 1 мкл SalI, 2 мкл соответствующего буфера и ddHот 2Oдо 20 мкл в течение 1 ч при 37 °C и гель-очистки продукта.
- Поместите сегмент восходящего шаблона с этапа 2.3.5 в подготовленный pKHR5, используя условия реакции, описанные на этапе 2.2.5. Преобразуйте лигированный продукт в компетентные 10β-клетки, усиливайте и очищайте с помощью минипрепа (необязательно: создайте глицериновый запас этого продукта).
- Используйте лигированный продукт из шага 2.3.6 (который содержит первый сегмент шаблона, лигированный в плазмиду pKHR5 перед геном репортера mVenus YFP) и дайджест для подготовки ко второму (ниже по потоку репортера mVenus) сегменту шаблона. Переварить 4 мкг лигированного продукта (со стадии 2.3.6, как на стадии 2.2.4) с помощью BamHI, а затем очистить с помощью набора для очистки ПЦР. Повторно суспендировать в 30 мкл ddH2Oи использовать его в качестве шаблона для второй реакции переваривания с EcoRI. Очистите продукт с помощью набора для очистки ПЦР.
- Готовят второй сегмент шаблона путем переваривания 2 мкг сегмента шаблона (этап 2.3.2), 1 мкл BamHI, 1 мкл EcoRI, 2 мкл соответствующего буфера и ddHот 2Oдо 20 мкл в течение 1 ч при 37 °C и гелевой очистки продукта.
- Поместите нисходящий сегмент шаблона в подготовленный pKHR5 (из этапа 2.3.7), используя условия реакции, описанные на этапе 2.2.5. Преобразуйте лигированный продукт в компетентные клетки, усиливайте и очищайте с помощью набора miniprep. Создайте запас глицерина этого конечного продукта, который содержит оба сегмента шаблона, лигированных по обе стороны от репортерного гена mVenus YFP в pKHR5.
- Получают плазмиду pKHR5 путем амплификации бактериальных культур из коммерчески доступного бактериального штамма DH5α (см. Таблицу материалов для деталей) таким же образом, как MLM3613 выше (этап 2.1.1).
- Подготовьте смесь микроинъекций, используя мРНК Cas9, две sgRNAs и экзогенный шаблон репортера HDR.
- Смешайте 200 нг/мкл мРНК Cas9, 100 нг/мкл каждой сгРНК и 200 нг/мкл экзогенного репортерного шаблона HDR в 1x инъекционном буфере с конечным объемом 20 мкл.
- Храните микроинъекционную смесь при температуре −20 °C и выбрасывайте неиспользованную смесь после трех циклов замораживания-оттаивания.
ПРИМЕЧАНИЕ: Используйте 4 нЛ этой микроинъекционной смеси для микроинъекции в желточный мешок каждого эмбриона.
3. Разведение рыбок данио и микроинъекция эмбрионов
ПРИМЕЧАНИЕ: Протоколы по разведению рыбок данио и микроинъекции одноклеточных эмбрионов были описаны ранее 29,30,31.
- Для разведения используют рыбок данио штамма AB и которым 6-12 месяцев. Вводите эмбрионы на одноклеточной стадии примерно через 40 минут после оплодотворения (см. Рисунок 5).
ПРИМЕЧАНИЕ: Биологический пол введенных эмбрионов не был известен; половой диморфизм не проявляется примерно до 3 месяцев в возрасте32 лет.
4. Репортерный генный скрининг отредактированных CRISPR-Cas9 личинок рыбок данио
- Визуализируйте интеграцию YFP в личинках рыбок данио после микроинъекции компонентов CRISPR-Cas9 для скрининга успешного редактирования HDR.
- В чашке Петри 25 мм обезболивают 24 личинки рыбок данио через 3 дня после оплодотворения (dpf) в 0,3% трикаина метансульфоната (MS-222, буферизованный до рН 7,0-7,4 с HEPES и гидроксидом натрия), пока они не потеряют свой самовосстанавливающийся рефлекс (обычно 1-2 мин). После обезболивания перенесите каждую личинку в отдельный колодец из 24-луночной пластины.
- Используя микроскоп, способный обнаруживать GFP/YFP, экран для репортерной флуоресценции генов в глазах каждой отдельной личинки.
- Захват изображений каждой личинки и документирование наличия или отсутствия репортерной экспрессии генов.
5. Фенотипирование CRISPR-Cas9-отредактированных личинок рыбок данио
- После скрининга репортерных генов выполните фенотипирование сердца (частота сердечных сокращений, размеры перикарда, ЭКГ) на каждой личинке. Фенотип равным количеством репортерных ген-положительных и -отрицательных личинок.
- Используйте ПЗС-камеру (например, blackfly USB3) и программное обеспечение для записи видео и изображений (например, Micromanager для ImageJ) для измерения частоты сердечных сокращений и размеров перикарда во время анестезии личинок.
- Чтобы измерить частоту сердечных сокращений, используя Micromanager, создайте область интереса (ROI), чтобы захватить сердце и исключить другие структуры.
- Импортируйте видео в ImageJ в виде последовательности изображений и убедитесь, что в число изображений введено правильное количество кадров.
- После открытия файла используйте инструмент выбора прямоугольника , чтобы нарисовать рентабельность инвестиций в сердце, исключая другие движущиеся элементы, и сохраните рентабельность инвестиций в менеджере ROI (анализируйте | инструменты | Менеджер ROI).
- Нажмите плагины | установить и выберите алгоритм сердечного ритма для установки кода в папку по умолчанию, затем выберите плагин в нижней части вкладки плагинов . Записывайте удары в минуту (bpm) из всплывающего окна.
ПРИМЕЧАНИЕ: Алгоритмы обнаружения изображений были специально написаны для определения частоты сердечных сокращений путем измерения индивидуальных изменений плотности пикселей, связанных с систолическим сокращением желудочков. Код можно найти по адресу https://github.com/dpoburko/zFish_HR.
- Измерьте размеры перикарда с помощью бесплатного инструмента, такого как ImageJ, чтобы свободно рисовать ROI вокруг перикардиального мешка и одного из глаз. Откройте изображение в ImageJ и используйте инструмент выделения полигонов , чтобы сначала нарисовать рентабельность инвестиций вокруг перикардиального мешка, сохранив его в менеджере ROI , как в шаге 5.1.2, и повторите для глаза. Выберите эти два ROI в управлении ROI r, затем нажмите « Измерить». Запишите площадь для каждого, чтобы позже рассчитать площадь перикардиального мешка, нормализованную к области глаз в каждой личинке.
- После измерений частоты сердечных сокращений и перикарда регистрируйте ЭКГ от отдельных личинок.
ПРИМЕЧАНИЕ: Протоколы регистрации ЭКГ рыбок данио были описаны ранее 33,34,35,36.
6. Генотипирование CRISPR-Cas9-отредактированных личинок рыбок данио
- После фенотипического анализа проводят генотипирование на и потенциально вне цели для подтверждения точного и точного редактирования генов HDR.
- Обезболивают каждую личинку 3 dpf в 0,3% MS-222 и хвостовом зажиме для выделения гДНК с помощью методаHOTShot 37. Инкубируйте каждый иссеченный хвостовой зажим в 15 мкл 25 мМ NaOH при 95 °C в течение 20 мин. Затем нейтрализуют 1,5 мкл Tris-HCl и центрифугой при 13 800 х г в течение 30 с. Сохраняют супернатант, который содержит экстрагированную гДНК.
- Восстановите личинок в среде E3 и верните их в жилищную систему, если предполагается дальнейшее развитие или изучение.
- Используя извлеченную гДНК в качестве шаблона, выполните секвенирование Сэнгера на основе ПЦР целевых и потенциальных нецелевых сайтов.
ПРИМЕЧАНИЕ: Необязательно: подход вложенной ПЦР может быть полезен для некоторых областей генов. - Убедитесь, что целевая конструкция праймера захватывает место мутации и ближайший сайт связывания sgRNA. Разработайте отдельный праймер секвенирования для обнаружения перехода от вставленной гомологической руки и гена-мишени для подтверждения интеграции в интересующий ген. Разработка руководств по упорядочению трех наиболее потенциальных нецелевых участков, определенных на этапе 1.2.3.
ПРИМЕЧАНИЕ: Программные программы для проектирования руководств часто предлагают учебники для использования, но для достижения оптимальных результатов может потребоваться настройка. - Соберите генотипирование по и вне цели, частоту сердечных сокращений, размеры перикарда, фенотипирование ЭКГ и репортерные генные данные, идентифицируемые для каждой рыбки данио.
Результаты
Успешное использование этого подхода CRISPR с двумя сгРНК-экзонами подчеркивается введением и простым обнаружением точного редактирования для разработки LQTS-ассоциированного варианта, R56Q, в гене zkcnh6a у рыбок данио. На рисунке 6 показаны репрезентативные личинки 3 dpf, введенные на стадии одноклеточного эмбриона с компонентами CRISPR, как описано выше. На рисунке 6A показано наличие экспрессии гена репортера YFP mVenus в хрусталике глаза в качестве положительного репортера успешной интеграции шаблонов. На рисунке 6B,C показаны секвенирующие хроматограммы Сэнгера, полученные из геномной ДНК, выделенной из образцов хвостового клипа диких и репортерных ген-положительных рыб, соответственно. Было обнаружено, что репортерные генно-положительные рыбы имеют точное редактирование от G до A, которое вводит вариант R56Q в zkcnh6a. Генотипирование показало 100% корреляцию между экспрессией генов репортера YFP и наличием точного редактирования гена R56Q, подтверждая этот инструмент флуоресцентного скрининга.
Фенотипирование генетически отредактированных личинок рыбок данио проводилось при 3 dpf. На рисунке 7 показаны репрезентативные результаты личинок дикого типа и R56Q, отредактированных геном. Частота сердечных сокращений была обнаружена с помощью видеозахвата, как описано выше. Приведен пример измерения размеров перикарда как соотношения площади глаз (рисунок 7А). На рисунке 7B показана частота сердечных сокращений против нормализованных размеров перикарда, выделяя тенденцию брадикардии с увеличением отека перикарда, которая связана с нарушениями реполяризации сердца у рыбок данио 8,38,39,40. На рисунке 7C показан репрезентативный пример записей ЭКГ из 3 личинок dpf. Стандартные интервалы (QT, QRS) измеряли по усредненным сигналам ЭКГ.

Рисунок 1: Интеграция шаблона HDR в геном рыбки данио. Темно-серый, гомологический герб; зеленые, направляющие sgRNA мишени с тихой мутацией для предотвращения перерезания Cas9; светло-серый, целевой экзон, представляющий интерес; красная линия, точечная мутация; желтый, ген-репортер mVenus YFP под α-кристаллинным промотором; пунктирные линии указывают на гомологию. Здесь целевым точным редактированием был R56Q в экзоне 2 гена zkcnh6a . Сокращения: HDR = гомологическое восстановление; sgRNA = однонаправленная РНК; YFP = желтый флуоресцентный белок; DSB = двухцепочечный разрыв; WT = дикий тип. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.

Рисунок 2: Краткое изложение шагов по разработке точных изменений в генах рыбок данио с использованием подхода CRISPR-Cas9 с двумя сгРНК (соответствующие номера шагов протокола указаны в скобках). Сокращения: sgRNA = однонаправленная РНК; YFP = желтый флуоресцентный белок; гДНК = геномная ДНК; ЭКГ = электрокардиограмма; dpf = дни после оплодотворения; MS-222 = трикаин метансульфонат. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
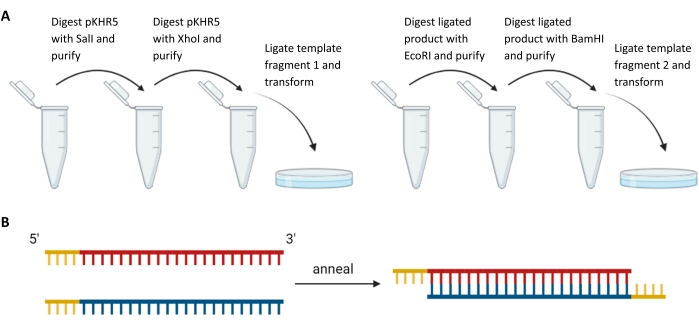
Рисунок 3: Подготовка экзогенных фрагментов шаблона и направляющих sgRNA. (A) Последовательное переваривание и лигирование фрагментов шаблона вверх и вниз по течению от последовательности репортерного гена mVenus YFP в pKHR5. (B) Отжиг комплементарных пар сгРНК с рестрикционным навесом для лигирования в DR274. Сокращения: sgRNA = однонаправленная РНК; YFP = желтый флуоресцентный белок. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
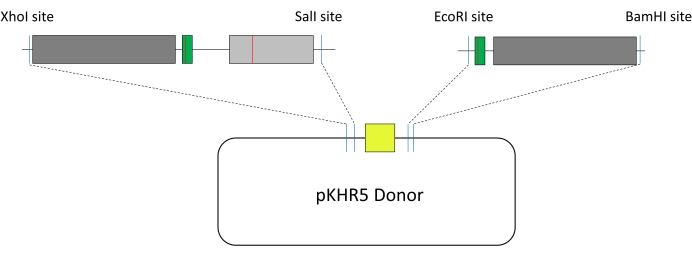
Рисунок 4: Построение шаблона HDR. Темно-серый, гомологический герб; зеленые, направляющие sgRNA мишени с тихой мутацией для предотвращения перерезания Cas9; светло-серый, целевой экзон, представляющий интерес; красная линия, точечная мутация; желтый, ген-репортер mVenus YFP под α-кристаллинным промотором; темно-синяя линия, добавлено ограничение сайтов. Два фрагмента шаблона интегрированы в донор плазмиды pKHR5. Сокращения: HDR = гомологическое восстановление; sgRNA = одноповодная РНК. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.

Рисунок 5: Микроинъекция одноклеточных эмбрионов рыбок данио с компонентами CRISPR-Cas9. Шкала стержня = 0,5 мм. Сокращения: HDR = гомологическое восстановление; sgRNA = одноповодная РНК. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.

Рисунок 6: Простое обнаружение флуоресценции репортерного гена mVenus YFP указывает на положительную интеграцию экзогенного шаблона HDR в ген-мишень. (A) Пример экспрессии mVenus YFP в глазу рыбки данио (стрелка) в отредактированной личинке рыбки данио. (B) Успешные правки подтверждаются секвенированием хроматограмм (слева, WT; справа, редактирование R56Q). Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.

Рисунок 7: Фенотипический анализ сердечных последствий у рыбок данио 3 dpf после точного редактирования R56Q в гене-мишени zkcnh6a . (A) Обнаружение размеров перикарда относительно размера глаз с помощью полигонального инструмента в ImageJ. Границы перикардиального мешка были обозначены пользователем из одного регистрирующего кадра на основе изменения полупрозрачности и пигментации. Приведены примеры нормальных размеров перикарда и перикардиального выпота. Шкала = 0,5 мм. (B) Корреляция между размерами перикарда (относительно размера глаза) и частотой сердечных сокращений,R2 = 0,33. (C) Пример записи ЭКГ из сердца личинки рыбки данио 3 dpf (слева) и усредненных комплексов (справа). Частота сердечных сокращений, 131 уд/мин; интервал QTc с коррекцией частоты сердечных сокращений, 460 мс. Сокращения: dpf = дни после оплодотворения; ЭКГ = электрокардиограмма. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Обсуждение
Разработка точных изменений генов с использованием CRISPR-Cas9 оспаривается низкой эффективностью механизмов HDR и их эффективным обнаружением. Здесь описан подход замены экзонов на основе CRISPR-Cas9 с двумя сгРНК, который производит точные изменения у рыбок данио с простым визуальным обнаружением положительных изменений. Эффективность этого подхода демонстрируется путем генерации точных правок в гене zkcnh6a . В этой статье показано, как функция сердца у генетически отредактированных личинок рыбок данио может быть оценена с использованием неинвазивных фенотипических измерений частоты сердечных сокращений, размеров перикарда и морфологии ЭКГ. Этот подход, от введения редактирования гена до фенотипической оценки, может быть завершен от начала до конца в течение примерно 1 недели.
Преимущества вышеупомянутого подхода к редактированию и фенотипированию заключаются в простоте проектирования модификации CRISPR, широкой применимости в нескольких физиологических системах, способности вставлять большие гены или фрагменты генов и способность отслеживать вариантные эффекты продольно через развитие и поколения. Успех точных правок в этом подходе может быть связан с сочетанием большого размера шаблона (из-за вставки гена репортера и длинных гомологических рычагов), который, как было показано, повышает эффективность правок у рыбок данио14, и стратегии с двумя sgRNA-направляющими, которая была эффективно использована в правках, индуцированных ДАНИОTALEN 8.
Одной из сильных сторон описанного подхода является способность вставлять большие гены или фрагменты генов. Это может быть полезно, например, для вставки человеческих ортологов41, что позволяет получить более клинически переводимую характеристику и сравнение между ортологами. В качестве альтернативы также могут быть вставлены гены, кодирующие ферменты Cas, что позволяет создать линию рыбок данио с механизмами редактирования IN VIVO CRISPR, обеспечивая индуцируемую систему. Аналогичным образом, альтернативные механизмы CRISPR, такие как первичное редактирование, могут быть интегрированы и привести к линии рыбок данио, которые легко редактируются точно и эффективно.
Несмотря на преимущества такого подхода, существуют некоторые ограничения. Во-первых, только один ген и локус были модифицированы, и необходимо дальнейшее тестирование на других участках или в других генах, чтобы оценить, насколько широко применим этот подход. Из-за длинных гомологических рычагов затраты на разработку шаблона выше; однако это может быть компенсировано эффективным скринингом. Другим ограничением является то, что скрининговый подход требует возможности обнаружения флуоресценции. Тем не менее, оптические требования относительно низки и могут быть изготовлены на заказ или коммерчески приобретены по разумно низкой цене. Использование подхода с двумя сгРНК увеличивает количество потенциальных событий, выходящих за рамки цели; однако это, вероятно, смягчается более низкой вероятностью того, что два проводника sgRNA будут отжигаться таким образом, чтобы облегчить включение шаблона для получения экспрессии генов репортера. Наконец, использование мРНК Cas9 может привести к мозаицизму, поскольку Cas9 не активен до более поздних стадий развития. Это может быть объяснено путем секвенирования определенных типов тканей; однако, учитывая размер личинок рыбок данио, это технически сложно.
Таким образом, этот подход CRISPR-Cas9 к точному редактированию двух сгРНК у рыбок данио позволяет легко визуально обнаруживать положительные изменения и может быть адаптирован для включения больших генов, представляющих интерес в любом локусе. В сочетании с фенотипическими мерами это обеспечивает надежную и высокопроизводительную платформу для изучения клинически значимых сердечных вариантов.
Раскрытие информации
У авторов нет конфликта интересов для раскрытия.
Благодарности
Это исследование было поддержано грантом Канадского исследовательского проекта Института здравоохранения (T.W.C.) и грантами Совета по естественным наукам и инженерным исследованиям Канады Discovery (T.W.C.).
Материалы
| Name | Company | Catalog Number | Comments |
| Program | |||
| CRISPOR | TEFOR Infrastructure | ||
| ENSEMBL | European Bioinformatics Institute | ||
| ImageJ | National Institutes of Health (NIH) | ||
| Micro-Manager | Open Source (Github) | ||
| NEBiocalculator | New England Biolabs (NEB) | ||
| EQUIPMENT | |||
| 24-well Plate | VWR | ||
| 25 mm Petri Dish | VWR | ||
| Blackfly USB3 Camera | Teledyne FLIR | ||
| C1000 Thermal Cycler | Bio-Rad | ||
| Centrifuge 5415C | Eppendorf | ||
| EZNA Gel Extraction Kit | Omega Biotek | ||
| MAXIscript T7 Transcription Kit | Invitrogen | ||
| MaxQ 5000 Incubator | Barnstead Lab Line | ||
| Miniprep Kit | Qiagen | ||
| mMessage mMachine T7 Ultra Transcription Kit | Invitrogen | ||
| ND1000 Spectrophotometer | Nanodrop | ||
| PCR Purification Kit | Qiagen | ||
| PLI 100A Picoinjector | Harvard Apparatus | ||
| PowerPac Basic Power Supply | Bio-Rad | ||
| Stemi 305 Steroscope | Zeiss | ||
| Wide Mini Sub Cell GT Electrophoresis System | Bio-Rad | ||
| ZebTec Zebrafish Housing System | Tecniplast | ||
| SERVICES | |||
| Gene Synthesis | Genewiz | ||
| Sanger Sequencing | Genewiz | ||
| REAGENTS | |||
| 10β Competent Cells | NEB | ||
| 10X PCR Buffer | Qiagen | ||
| 100 mM Nucleotide Mixture | ABM | ||
| Ampicillin | Sigma | ||
| BamHI Endonuclease w/ buffer | NEB | ||
| BsaI Endonuclease w/ buffer | NEB | ||
| DR274 Plasmid (XL1 Blue bacterial agar stab) | Addgene | ||
| EcoRI Endonuclease w/ buffer | NEB | ||
| Glycerol | |||
| HEPES | Sigma | ||
| HindIII Endonuclease w/ buffer | NEB | ||
| Kanamycin | Sigma | ||
| Methylene Blue | Sigma | ||
| MLM3613 Plasmid (XL1 Blue bacterial agar stab) | Addgene | ||
| MS-222 (Tricaine) | Sigma | ||
| pKHR5 Plasmid (DH5α bacterial agar stab) | Addgene | ||
| PmeI Endonuclease w/ buffer | NEB | ||
| SalI Endonuclease w/ buffer | NEB | ||
| Sodium Hydroxide | Sigma | ||
| T4 Ligase w/ buffer | Sigma | ||
| Taq Polymerase | Qiagen | ||
| TE Buffer | Sigma | ||
| Tris Hydrochloride | Sigma | ||
| XhoI Endonuclease w/ buffer | NEB | ||
| RECIPES | |||
| Solution | Component | Supplier | |
| Annealing Buffer (pH 7.5-8.0) | 10 mM Tris | Sigma | |
| 50 mM NaCl | Sigma | ||
| 1 mM EDTA | Sigma | ||
| E3 Media (pH 7.2) | 5 mM NaCl | Sigma | |
| 0.17 mM KCl | Sigma | ||
| 0.33 mM CaCl2 | Sigma | ||
| 0.33 mM MgSO4 | Sigma | ||
| Injection Buffer (pH 7.5) | 20 mM HEPES | Sigma | |
| 150 mM KCl | Sigma |
Ссылки
- Zarei, A., Razban, V., Hosseini, S. E., Tabei, S. M. B. Creating cell and animal models of human disease by genome editing using CRISPR/Cas9. The Journal of Gene Medicine. 21 (4), 3082(2019).
- Lee, H., Yoon, D. E., Kim, K. Genome editing methods in animal models. Animal Cells and Systems. 24 (1), 8-16 (2020).
- Li, Q., et al. Applications of genome editing technology in animal disease modeling and gene therapy. Computational and Structural Biotechnology Journal. 17, 689-698 (2019).
- Gut, P., Reischauer, S., Stainier, D. Y. R., Arnaout, R. Little fish, big data: Zebrafish as a model for cardiovascular and metabolic disease. Physiological Reviews. 97 (3), 889-938 (2017).
- Kegel, L., et al. Forward genetic screen using zebrafish to identify new genes involved in myelination. Oligodendrocytes: Methods and Protocols. 1936, 185-209 (2019).
- Shah, A. N., Davey, C. F., Whitebirch, A. C., Miller, A. C., Moens, C. B. Rapid reverse genetic screening using CRISPR in zebrafish. Nature Methods. 12 (6), 535-540 (2015).
- González-Rosa, J. M. Zebrafish models of cardiac disease: From fortuitous mutants to precision medicine. Circulation Research. 130 (12), 1803-1826 (2022).
- Hoshijima, K., Jurynec, M. J., Grunwald, D. J. Precise editing of the Zebrafish genome made simple and efficient. Developmental Cell. 36 (6), 654-667 (2016).
- Albadri, S., Del Bene, F., Revenu, C. Genome editing using CRISPR/Cas9-based knock-in approaches in zebrafish. Methods. 121-122, 77-85 (2017).
- Armstrong, G. A. B., et al. Homology directed knockin of point mutations in the zebrafish tardbp and fus genes in ALS using the CRISPR/Cas9 system. PLoS One. 11 (3), 0150188(2016).
- Bai, H., et al. CRISPR/Cas9-mediated precise genome modification by a long ssDNA template in zebrafish. BMC Genomics. 21 (1), 67(2020).
- de Vrieze, E., et al. Efficient generation of knock-in zebrafish models for inherited disorders using CRISPR-Cas9 ribonucleoprotein complexes. International Journal of Molecular Sciences. 22 (17), 9429(2021).
- Eschstruth, A., Schneider-Maunoury, S., Giudicelli, F. Creation of zebrafish knock-in reporter lines in the nefma gene by Cas9-mediated homologous recombination. Genesis. 58 (1), 23340(2020).
- Irion, U., Krauss, J., Nüsslein-Volhard, C. Precise and efficient genome editing in zebrafish using the CRISPR/Cas9 system. Development. 141 (24), 4827-4830 (2014).
- Kimura, Y., Hisano, Y., Kawahara, A., Higashijima, S. Efficient generation of knock-in transgenic zebrafish carrying reporter/driver genes by CRISPR/Cas9-mediated genome engineering. Scientific Reports. 4 (1), 6545(2014).
- Levic, D. S., Yamaguchi, N., Wang, S., Knaut, H., Bagnat, M. Knock-in tagging in zebrafish facilitated by insertion into non-coding regions. Development. 148 (19), (2021).
- Prykhozhij, S. V., et al. Optimized knock-in of point mutations in zebrafish using CRISPR/Cas9. Nucleic Acids Research. 46 (17), 102(2018).
- Wierson, W. A., et al. Efficient targeted integration directed by short homology in zebrafish and mammalian cells. eLife. 9, 53968(2020).
- Boel, A., et al. CRISPR/Cas9-mediated homology-directed repair by ssODNs in zebrafish induces complex mutational patterns resulting from genomic integration of repair-template fragments. Disease Models & Mechanisms. 11 (10), (2018).
- Tessadori, F., et al. Effective CRISPR/Cas9-based nucleotide editing in zebrafish to model human genetic cardiovascular disorders. Disease Models & Mechanisms. 11 (10), (2018).
- Zhang, Y., Zhang, Z., Ge, W. An efficient platform for generating somatic point mutations with germline transmission in the zebrafish by CRISPR/Cas9-mediated gene editing. The Journal of Biological Chemistry. 293 (17), 6611-6622 (2018).
- Vornanen, M., Hassinen, M. Zebrafish heart as a model for human cardiac electrophysiology. Channels. 10 (2), 101-110 (2016).
- Nemtsas, P., Wettwer, E., Christ, T., Weidinger, G., Ravens, U. Adult zebrafish heart as a model for human heart? An electrophysiological study. Journal of Molecular and Cellular Cardiology. 48 (1), 161-171 (2010).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biology. 17 (1), 148(2016).
- Liang, X., Potter, J., Kumar, S., Ravinder, N., Chesnut, J. D. Enhanced CRISPR/Cas9-mediated precise genome editing by improved design and delivery of gRNA, Cas9 nuclease, and donor DNA. Journal of Biotechnology. 241, 136-146 (2017).
- Richardson, C. D., Ray, G. J., DeWitt, M. A., Curie, G. L., Corn, J. E. Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nature Biotechnology. 34 (3), 339-344 (2016).
- Sambrook, J., Fritsch, E. F., Maniatis, T. Precipitation of Large RNAs with Lithium Chloride. Molecular Cloning: A Laboratory Manual, Book 3. E.15. , Cold Spring Harbor Laboratory. Long Island, NY. (1989).
- Sambrook, J., Fritsch, E. F., Maniatis, T. Agarose Gel Electrophoresis. Molecular Cloning: A Laboratory Manual, Book 1. , Cold Spring Harbor Laboratory. Long Island, NY. 3-20 (1989).
- Sorlien, E. L., Witucki, M. A., Ogas, J. Efficient production and identification of CRISPR/Cas9-generated gene knockouts in the model system Danio rerio. Journal of Visualized Experiments. (138), e56969(2018).
- Avdesh, A., et al. Regular care and maintenance of a zebrafish (Danio rerio) laboratory: An introduction. Journal of Visualized Experiments. (69), e4196(2012).
- Rosen, J. N., Sweeney, M. F., Mably, J. D. Microinjection of zebrafish embryos to analyze gene function. Journal of Visualized Experiments. (25), e1115(2009).
- Kossack, M. E., Draper, B. W. Genetic regulation of sex determination and maintenance in zebrafish (Danio rerio). Current Topics in Developmental Biology. 134, 119-149 (2019).
- Tanaka, Y., et al. Functional analysis of KCNH2 gene mutations of type 2 long QT syndrome in larval zebrafish using microscopy and electrocardiography. Heart and Vessels. 34 (1), 159-166 (2019).
- Dhillon, S. S., et al. Optimisation of embryonic and larval ECG measurement in zebrafish for quantifying the effect of QT prolonging drugs. PLoS One. 8 (4), 60552(2013).
- Yu, F., et al. Evolving cardiac conduction phenotypes in developing zebrafish larvae: Implications to drug sensitivity. Zebrafish. 7 (4), 325-331 (2010).
- Hurst, R. M. Development and optimization of tools for embryonic electrocardiograph recording for heart dysfunction in zebrafish. , University of Birmingham. PhD thesis (2018).
- Meeker, N. D., Hutchinson, S. A., Ho, L., Trede, N. S. Method for isolation of PCR-ready genomic DNA from zebrafish tissues. BioTechniques. 43 (5), 610-614 (2007).
- Arnaout, R., et al. Zebrafish model for human long QT syndrome. Proceedings of the National Academy of Sciences of the United States of America. 104 (27), 11316-11321 (2007).
- Milan, D. J., Peterson, T. A., Ruskin, J. N., Peterson, R. T., MacRae, C. A. Drugs that induce repolarization abnormalities cause bradycardia in zebrafish. Circulation. 107 (10), 1355-1358 (2003).
- Langheinrich, U., Vacun, G., Wagner, T. Zebrafish embryos express an orthologue of HERG and are sensitive toward a range of QT-prolonging drugs inducing severe arrhythmia. Toxicology and Applied Pharmacology. 193 (3), 370-382 (2003).
- MacRae, C. A. Cardiac arrhythmia: In vivo screening in the zebrafish to overcome complexity in drug discovery. Expert Opinion on Drug Discovery. 5 (7), 619-632 (2010).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены