Method Article
CRISPR-Cas9-עריכות מדויקות בתיווך בלבבות דגי זברה
In This Article
Summary
פרוטוקול זה מתאר גישה המאפשרת עריכות מדויקות של דגי זברה באמצעות טכנולוגיית CRISPR-Cas9. צינור פנוטיפ מוצג כדי להדגים את הישימות של טכניקות אלה כדי לדגום וריאנט גן הקשור לתסמונת QT ארוכה.
Abstract
חזרות פלינדרומיות קצרות (CRISPR) מקובצות באופן קבוע במודלים של בעלי חיים מאפשרות מניפולציה גנטית מדויקת לחקר תופעות פיזיולוגיות. דגי זברה שימשו כמודל גנטי יעיל לחקר שאלות רבות הקשורות למחלות תורשתיות, התפתחות וטוקסיקולוגיה ברמת האיברים והאורגניזמים. הודות לגנום של דגי הזברה המבוארים והממופים היטב, פותחו כלים רבים לעריכת גנים. עם זאת, היעילות של יצירה וקלות של זיהוי עריכות מדויקות באמצעות קריספר היא גורם מגביל. המתוארת כאן היא גישת knock-in מבוססת CRISPR-Cas9 עם זיהוי פשוט של עריכות מדויקות בגן האחראי על רה-פולריזציה של הלב וקשור להפרעה החשמלית, תסמונת Long QT (LQTS). גישת RNA (sgRNA) דו-מנחה זו מכניסה ומחליפה את רצף המטרה ומקשרת בין גן מדווח המקודד גנטית. התועלת של גישה זו מודגמת על ידי תיאור מדידות פנוטיפיות לא פולשניות של תפקוד חשמלי לבבי בזחלים מסוג בר ודגי זברה שעברו עריכה גנטית. גישה זו מאפשרת מחקר יעיל של גרסאות הקשורות למחלות באורגניזם שלם. יתר על כן, אסטרטגיה זו מציעה אפשרויות להחדרת רצפים אקסוגניים של בחירה, כגון גנים מדווחים, אורתולוגים או עורכי גנים.
Introduction
אסטרטגיות עריכת גנים מבוססות קריספר במודלים של בעלי חיים מאפשרות לחקור מחלות, התפתחות וטוקסיקולוגיה תורשתיות גנטית ברמת האורגניזם השלם 1,2,3. דגי זברה מספקים מודל רב עוצמה הקרוב יותר בהיבטים פיזיולוגיים רבים לבני אדם מאשר מודלים של תאים שמקורם בבני אדם4. מגוון רחב של כלים ואסטרטגיות גנטיות שימשו בדגי זברה הן לבדיקה גנטית קדימה5 והן לבדיקה גנטית הפוכה6. מיפוי גנטי מקיף וביאור בדגי זברה סייעו לגישות לעריכת גנים כטכניקה עיקרית להנדסת נוקאאוטים של גנים ממוקדים (KOs) ודפיקות מדויקות (KIs)7.
למרות זאת, יצירת עריכות KI מדויקות בדגי זברה מוגבלת על ידי יעילות נמוכה והקושי בזיהוי מדויק. למרות שנוקלאזות משפיעות דמויות גורם שעתוק (TALENs) שימשו בהצלחה ואופטימיזציה עבור KIs8, קריספר מספק אסטרטגיית עריכת גנים משופרת עם מיקוד sgRNA פשוט יותר. מחקרים רבים השתמשו בקריספר כדי ליצור KIs מדויקים בדגי זברה 9,10,11,12,13,14,15,16,17,18,19,20, אם כי עריכות אלה שנוצרו באמצעות תיקון מכוון הומולוגיה בתיווך קריספר (HDR) נוטות להיות לא יעילות עם הצלחה פנימית נמוכה שיעורים הדורשים גנוטיפ כמסך ראשי 9,10,14,21. זה מדגים את הצורך במערכת KI CRISPR יעילה בדגי זברה, כמו גם במערכת אמינה בעלת תפוקה גבוהה לאיתור עריכות מדויקות.
מטרת המחקר הייתה לתאר פלטפורמה ליצירת גן לב מדויק KI בלבבות דגי זברה עם זיהוי פשוט ותפוקה גבוהה של עריכות מוצלחות. מתוארת גישת החלפת אקסון דו-sgRNA מבוססת CRISPR-Cas9, המבוססת על גישת TALEN8. גישה זו כוללת כריתה של רצף המטרה באמצעות מדריכי שני sgRNA והחלפה ברצף תבניות אקסוגני המכיל את ה-KI המעניין וכן גן כתב פנימי מקודד גנטית (איור 1). השילוב של כתב פלואורסצנטי מקודד גנטית בתוך הרצף הפנימי של גן המטרה מאפשר זיהוי יעיל של עריכות חיוביות. לאחר מכן מתוארת פלטפורמה פנוטיפית להערכת התפקוד החשמלי של הלב בזחלים של דגי זברה לצורך אפיון לא פולשני של גרסאות הגנים הקשורות ל-LQTS תורשתי, הפרעה חשמלית לבבית הגורמת לאנשים למוות לבבי פתאומי.
גישות אלה ישפרו את הגישה והשימוש בעריכות גנים של דגי זברה KI כדי ליצור מודלים של מחלות תורשתיות ולתת מענה לשאלות ביולוגיות ופיזיולוגיות, כגון מיפוי דפוסי ביטוי גנים וויסות התפתחותי. מאחר שלבבות דגי זברה מקבילים טוב יותר למאפיינים אלקטרופיזיולוגיים של הלב האנושי מאשר מודלים של מורין, הם עשויים להיות אטרקטיביים במיוחד כמערכת הניתנת למתיחה גנטית עבור מודלים של מחלות לב 7,22,23.
Protocol
מחקרים באמצעות דגי זברה נערכו בהסכמה עם המדיניות והנהלים של הוועדה לטיפול בבעלי חיים של אוניברסיטת סיימון פרייזר והמועצה הקנדית לטיפול בבעלי חיים והושלמו תחת פרוטוקול # 1264K-18.
1. תכנון רכיבי קריספר לעריכות מדויקות
- כדי לתכנן את שני מדריכי ה-sgRNA שישמשו לבלו הרצף המכיל את אתר היעד KI, זהה תחילה את אורתולוג דגי הזברה עבור הגן המעניין.
הערה: איור 2 מספק סקירה מסכמת של השלבים להנדסת עריכות מדויקות באמצעות גישת CRISPR-Cas9 של שני sgRNA. - לאחר מכן, השתמש בכלי תוכנת עיצוב, כגון CRISPOR24, הכולל בחירה עבור Danio rerio כמין ושל אנזים Cas לשימוש.
הערה: הגן המעניין למחקר זה היה zkcnh6a (מזהה תעתיק Ensembl: ENSDART00000090809.6; מזהה חלבון UniProt: B3DJX4), ומוטציית המטרה הייתה חומצת אמינו, R56Q.- עבור גישת שני sgRNA, בחר מיקום sgRNA אחד שמקדים את אקסון המטרה ו- sgRNA שני הממוקם בתוך האינטרון המיידי במורד הזרם.
- ודא של-sgRNAs שנבחרו יש ספציפיות גבוהה ואיגוד חזוי נמוך מחוץ למטרה. השתמש בדירוגי CRISPOR כדי לזהות מדריכים עם כריכה מינימלית מחוץ ליעד. אל תתייחסו למדריכים ללא אי-התאמות ברצף הזרעים של מטרות פוטנציאליות.
- זהה את האתרים הפוטנציאליים הסבירים ביותר מחוץ למטרה (בהתבסס על ציוני CRISPOR, בחר את שלושת האתרים הפוטנציאליים המובילים של אקסון מחוץ ליעד) עבור גנוטיפ ריצוף סנגר מבוסס PCR בשלב 6.1.3.
- לאחר בחירת שני ה-sgRNA, השיגו את המשלים ההפוך עבור כל אחד מהם, כך שיש ארבעה אוליגונוקלאוטידים לשימוש: שני אוליגוסים משלימים שקודמים למוטציה ושני אוליגואים משלימים שנמצאים במורד הזרם.
- על כל אחד מארבעת האוליגוס, הוסף אתרי הגבלה תואמים לשילוב בפלסמיד מדריך לפי בחירה; לשילוב בפלסמיד DR274, השתמש באתר הגבלת BsaI בגודל 5 אינץ' כדי ליצור שלוחה. ודא שאתר הזיהוי של Bsa1 מהונדס בקצה 5' של המדריך שנבחר מתוך CRISPOR ושאתר הזיהוי של Bsa1 מהונדס בקצה 5' של הגדילים המשלימים כדי להבטיח כיוון נכון של המדריכים בפלסמיד DR274 (ראה איור 3).
- כדי לתכנן את התבנית האקסוגנית המשמשת ל-HDR בדגי זברה (איור 1), בחרו שני מקטעי רצף שיגדשו את הגן המדווח mVenus YFP שנמצא בפלסמיד pKHR5.
הערה: ניתן לכלול את השינוי/העריכה המיועדים בקטע במעלה הזרם או במורד הזרם.- באמצעות Ensembl, אתר את אתר המטרה בתוך רצף הגנים המעניין, כולל בערך רצף האגפים של 2 קילובייט (זרוע הומולוגיה), שישמש ליצירת התבנית.
הערה: זרועות הומולוגיה עשויות להיותסימטריות או אסימטריות 25,26 וכ-1 קילובייט כל אחת, במעלה הזרם ובמורד הזרם של אתר היעד. - חלקו את התבנית לשני מקטעים שיוכנסו משני צדי הגן המדווח mVenus YFP (ראו איור 4). ודא שהאתר המפוצל נמצא באינטרון כדי שרצף הקידוד לא יופרע.
הערה: אם הגן מאופיין היטב, בדוק אם קיימים תפקידים פונקציונליים באינטרון, כגון אתרי שחבור או אזורי ויסות. אזורים הקרובים לקצוות 5' או 3' מעורבים לעתים קרובות יותר בשחבור mRNA. - שלבו שינויים ברצף התבניות הכוללים i) מוטציות שקטות במוטיב הסמוך לפרוטו-ספייסר (PAM) או ברצף הזרעים (שימו לב לאתרי PAM חלופיים שהאנזים Cas עשוי להתמקד בהם) כדי למנוע חיתוך מחדש של אנזים Cas; 2) שינוי העניין; 3) יצירת אתרי אנדונוקלאז הגבלה כדי להקל על השיבוט לתוך פלסמיד pKHR5, המכיל את הגן המדווח mVenus YFP (ראו איור 3).
הערה: במחקר זה, מקטע התבנית הראשון הכיל את XhoI במעלה הזרם של אתר המוטציות R56Q ו- SalI במורד הזרם, בעוד שלמקטע התבנית השני היה EcoRI במעלה הזרם של רצף היעד המנחה ו- BamHI במורד הזרם. אם אחד מאתרי ההגבלה שנבחרו נמצא בתוך רצף התבניות, יידרשו מוטציות להשתקת אלה, או שניתן יהיה להשתמש בגישות חלופיות כגון Gibson Assembly.
- באמצעות Ensembl, אתר את אתר המטרה בתוך רצף הגנים המעניין, כולל בערך רצף האגפים של 2 קילובייט (זרוע הומולוגיה), שישמש ליצירת התבנית.
2. הכנת רכיבי קריספר למיקרו הזרקות לעוברים
- הכינו את ה-Cas9 למיקרו-הזרקות שבוע לפני המיקרו-הזרקות. השתמש בחלבון Cas9 או הכן mRNA של Cas9 באמצעות שעתוק במבחנה .
הערה: במחקר זה נעשה שימוש ב-Cas9 mRNA, שכן היעילות נטתה להיות גבוהה יותר.- הגבר תרביות חיידקים של דקירה אגר חיידקית כחולה XL1 הזמינה מסחרית (המכילה פלסמיד Cas9) באמצעות אנטיביוטיקה מתאימה כגון אמפיצילין. השתמש ב-675 μL של תרבית נוזלית (עם 325 μL של גליצרול) כדי ליצור מלאי גליצרול גיבוי לאחסון לטווח ארוך בטמפרטורה של 80°C-.
- השתמשו בשארית התרבית הנוזלית לטיהור מיני-פרפ, בהתאם לפרוטוקול המצורף לערכת ה-miniprep. יש להשעות את הדנ"א המטוהר הסופי ב-50 מיקרו-ליטר של מאגר ה-elution שסופק. כימות המכפלה באמצעות ספקטרופוטומטר כדי לבחון את התפוקה והטוהר.
- ליניאריזציה של 2 מיקרוגרם של הדנ"א המטוהר באמצעות עיכול הגבלה באמצעות אנזים הגבלה מתאים ושימוש בזמן המאגר והדגירה המתאימים כמפורט עבור האנזים המעניין.
- טהרו את הפלסמיד הליניארי באמצעות ערכת טיהור PCR, והחזירו אותו ל-30 מיקרו-ליטר של מאגר ה-elution המסופק.
- לאחר כימות המוצר, השתמש בזה כתבנית לתמלול חוץ גופי באמצעות ערכת התמלול המתאימה למקדם העניין. עקוב אחר הפרוטוקול המסופק וטהר באמצעות משקעי ליתיום כלוריד27. יש להשעות את הרנ"א המטוהר ב-10 μL של H2O נטול נוקלאז ולכמת אותו לפני אחסון בטמפרטורה של 20°C- לשימוש בתערובת המיקרו-הזרקות.
- הכינו את שני מדריכי ה-sgRNA.
- הכן את פלסמיד ה- sgRNA עם פיגום על ידי הגברת תרביות חיידקים מדקירת אגר חיידקית XL1 Blue הזמינה מסחרית (ראה טבלת חומרים לפרטים) באותו אופן כמו MLM3613 לעיל (שלב 2.1.1), למעט שימוש בקנאמיצין במקום באמפיצילין.
- Anneal שני זוגות של אוליגונוקלאוטידים חד-גדיליים משלימים (ssODNs) עבור מדריכי sgRNA שתוכננו לעיל על ידי החייאת ה-ssODNs בחיץ חישול 1x לריכוז של 100 μM.
- בתגובות נפרדות עבור כל אחד משני ה-sgRNAs, ניתן להשתמש בזוג ssODNs משלימים באמצעות מחזור תרמי. יש לערבב 2 מיקרוגרם של כל זוג ssODN משלים עם 50 μL של חיץ חישול ולדגור בטמפרטורה של 95 °C למשך 2 דקות, ואז להתקרר ל-25 °C במשך 45 דקות.
- לעכל פלסמיד זמין מסחרית המכיל פיגום gRNA. יש לעכל 2 מיקרוגרם של פלסמיד DR274 באמצעות 1 μL של BsaI, 2 μL של חיץ מתאים, ו ddH2O עד 20 μL ב 37 °C במשך שעה אחת. אשר ליניאריזציה (אופציונלי: לטהר באמצעות ערכת טיהור PCR) באמצעות אלקטרופורזה בג'ל28.
- בשתי תגובות קשירה נפרדות (אחת לכל sgRNA), ניתן ללקות את ה-ssODN החישול עם פלסמיד DR274 ליניארי. השתמש ביחס טוחן:וקטור של 3:1, חישוב המסה המתאימה באמצעות מחשבון קשירה מקוון. מערבבים את המסה הנדרשת של התוספת והווקטור עם 1 μL של T4 DNA ligase, 2 μL של חיץ ליגציה, ו ddH2O עד 12 μL ודגירה בטמפרטורת החדר במשך 12 שעות.
- הפוך 2 μL של המוצר המקושר לתאים מתאימים (כגון תאים של 10β) באמצעות גישות סטנדרטיות, ולאחר מכן הגבר וטהור את המוצר באמצעות ערכת Miniprep הזמינה מסחרית. אופציונלי: צור מלאי גליצרול של מוצר זה.
- תמלל את שני ה-sgRNAs על ידי ליניאריזציה של 2 מיקרוגרם מכל מדריך באמצעות אתר הגבלה במורד הזרם בגודל 3 אינץ', הקרוב ככל האפשר לסוף רצף המרחב. עבור פלסמיד DR274, ליניארי עם HindIII, ולאחר מכן לטהר את תבנית הרנ"א באמצעות ערכת טיהור PCR, תוך חידוש ב-30 μL של מאגר ה-elution.
- תמלל את שני המדריכים באמצעות ערכת שעתוק RNA. עקוב אחר פרוטוקול היצרן וטהר באמצעות משקעי ליתיום כלוריד27. יש להשעות את שני מדריכי ה-sgRNA המטוהרים ב-10 μL של H2O נטול נוקלאזה, לכמת ולאחסן בטמפרטורה של 20°C- לשימוש בתערובת המיקרו-הזרקות.
הערה: ערכת שעתוק ה-RNA אינה יכולה לשלב כובע 5' או זנב poly-A.
- הכן את תבנית כתב ה- HDR הדו-גדילי והאקסוגני.
הערה: התבנית מסונתזת בשני חלקים, אחד במעלה הזרם ואחד במורד הזרם של הגן המדווח mVenus YFP. שני מקטעים אלה הם מבנים סינתטיים שהוזמנו דרך ספק מסחרי ולאחר מכן נקשרו לפלסמיד pKHR5 (המכיל mVenus YFP) ברצף.- הכן את הפלסמיד pKHR5 על-ידי הגברת תרביות חיידקים מזן החיידקים DH5α הזמין מסחרית (ראה טבלת חומרים לפרטים) באותו אופן כמו MLM3613 לעיל (שלב 2.1.1).
הערה: פלסמיד pKHR5 מכיל את רצף הגנים של כתב mVenus YFP. - חזור על שני מקטעי התבנית שתוכננו לעיל ל- 100 μM במאגר TE ולאחר מכן המר לתאים של 10β.
- עכל את מקטע התבנית הראשון (זה במעלה הזרם של הגן המדווח mVenus YFP) ואת פלסמיד pKHR5 באמצעות אנזימי ההגבלה שנבחרו בשלב 1.3.3.
הערה: יש צורך בתקציר רציף של pKHR5 בשל הקרבה של אתרי ההגבלה שנבחרו ב- MCS. - כדי להכין את פלסמיד pKHR5 למקטע התבנית במעלה הזרם, יש לעכל 4 מיקרוגרם של pKHR5 (לפי שלב 2.2.4) עם SalI ולאחר מכן לטהר באמצעות ערכת טיהור PCR. יש להשתמש ב-30 μL של ddH2O ולהשתמש בזה כתבנית לתגובת העיכול השנייה עם XhoI. טהר את המוצר באמצעות ערכת הטיהור PCR.
- הכן את מקטע התבנית הראשון על ידי עיכול 2 מיקרוגרם של מקטע התבנית (שלב 2.3.2), 1 μL של XhoI, 1 μL של SalI, 2 μL של חיץ מתאים, ו ddH2O עד 20 μL במשך שעה אחת ב 37 °C ו ג'ל לטהר את המוצר.
- השווה את מקטע התבנית במעלה הזרם משלב 2.3.5 ל- pKHR5 המוכן באמצעות תנאי התגובה המתוארים בשלב 2.2.5. הפוך את המוצר המקושר לתאים מוכשרים של 10β, הגבר וטהר באמצעות miniprep (אופציונלי: צור מלאי גליצרול של מוצר זה).
- השתמש במוצר המקשר משלב 2.3.6 (המכיל את מקטע התבנית הראשון המקושר לפלסמיד pKHR5 במעלה הזרם של גן הכתב mVenus YFP) והתעכל כדי להתכונן למקטע התבנית השני (במורד הזרם של כתב mVenus). יש לעכל 4 מיקרוגרם של המוצר המחויב (משלב 2.3.6, כמו בשלב 2.2.4) עם BamHI, ולאחר מכן לטהר באמצעות ערכת טיהור PCR. יש להשעות את ה-30 μL של ddH2O ולהשתמש בזה כתבנית לתגובת העיכול השנייה עם EcoRI. טהר את המוצר באמצעות ערכת הטיהור PCR.
- הכן את מקטע התבנית השני על ידי עיכול 2 מיקרוגרם של מקטע התבנית (שלב 2.3.2), 1 μL של BamHI, 1 μL של EcoRI, 2 μL של חיץ מתאים, ו ddH2O עד 20 μL במשך שעה אחת ב 37 °C ו ג'ל לטהר את המוצר.
- גלגלו את מקטע התבנית במורד הזרם לתוך pKHR5 המוכן (משלב 2.3.7) באמצעות תנאי התגובה המתוארים בשלב 2.2.5. הפוך את המוצר המקושר לתאים מוכשרים, הגבר וטהר באמצעות ערכת miniprep. צור מלאי גליצרול של מוצר סופי זה, המכיל את שני מקטעי התבנית המקושרים משני צדי הגן המדווח mVenus YFP בתוך pKHR5.
- הכן את הפלסמיד pKHR5 על-ידי הגברת תרביות חיידקים מזן החיידקים DH5α הזמין מסחרית (ראה טבלת חומרים לפרטים) באותו אופן כמו MLM3613 לעיל (שלב 2.1.1).
- הכן את תערובת המיקרו-הזרקות באמצעות ה-mRNA של Cas9, שני sgRNA ותבנית כתב HDR אקסוגנית.
- ערבבו 200 ng/μL של Cas9 mRNA, 100 ng/μL של כל sgRNA, ו-200 ng/μL של תבנית כתב HDR אקסוגנית במאגר הזרקה 1x לנפח סופי של 20 μL.
- אחסנו את תערובת המיקרו-הזרקות בטמפרטורה של 20°C- והשליכו את התערובת שאינה בשימוש לאחר שלושה מחזורי הפשרה בהקפאה.
הערה: השתמש ב-4 nL של תערובת מיקרו-הזרקות זו עבור מיקרו-הזרקה לתוך שק החלמון של כל עובר.
3. גידול דגי זברה ומיקרו הזרקות עובריות
הערה: פרוטוקולים לגידול דגי זברה ומיקרו-הזרקה של עוברים חד-תאיים תוארו בעבר 29,30,31.
- לרבייה, השתמש דג זברה של זן AB וכי הם 6-12 חודשים של גיל. להזריק את העוברים בשלב החד-תאי כ-40 דקות לאחר ההפריה ( ראו איור 5).
הערה: המין הביולוגי של העוברים המוזרקים לא היה ידוע; דימורפיזם מיני אינו ניכר עד כ-3 חודשים בגיל32.
4. בדיקת גנים של דג זברה בעריכת CRISPR-Cas9
- דמיינו את שילוב YFP בזחלים של דגי זברה לאחר הזרקה זעירה של רכיבי CRISPR-Cas9 כדי לסנן עריכות HDR מוצלחות.
- בצלחת פטרי בקוטר 25 מ"מ, מרדימים 24 זחלי דגי זברה ב-3 ימים לאחר ההפריה (dpf) ב-0.3% טריקיין מתאן סולפונט (MS-222, חצוי ל-pH 7.0-7.4 עם HEPES ונתרן הידרוקסידי) עד שהם מאבדים את רפלקס התיקון העצמי שלהם (בדרך כלל 1-2 דקות). לאחר ההרדמה, מעבירים כל זחל לבאר בודדת של צלחת בת 24 בארות.
- באמצעות מיקרוסקופ המסוגל לזהות GFP/YFP, לסנן פלואורסצנטיות של גנים מדווחים בעיניים של כל זחל בודד.
- צלם תמונות של כל זחל ותעד את נוכחותו או היעדרו של ביטוי גנים מדווח.
5. פנוטיפ של דג זברה זחלי בעריכת CRISPR-Cas9
- לאחר בדיקת גנים מדווחת, יש לבצע פנוטיפ לבבי (דופק, ממדים קרום הלב, אק"ג) על כל זחל. פנוטיפ מספר שווה של זחלים חיוביים גנטיים ושליליים.
- השתמש במצלמת CCD (לדוגמה, blackfly USB3) ובתוכנה להקלטת וידאו ותמונות (לדוגמה, Micromanager עבור ImageJ) כדי למדוד את הדופק ואת מידות קרום הלב בזמן שהזחלים מורמים.
- כדי למדוד את קצב הלב, באמצעות Micromanager, ליצור אזור עניין (ROI) כדי ללכוד את הלב ולא לכלול מבנים אחרים.
- ייבאו את הסרטון ל-ImageJ כרצף תמונות, וודאו שמספר המסגרות הנכון מוזן תחת מספר התמונות.
- לאחר פתיחת הקובץ, השתמש בכלי בחירת המלבן כדי לצייר ROI בתוך הלב אך לא כולל אלמנטים נעים אחרים, ושמור את ההחזר על ההשקעה במנהל ההחזר על ההשקעה (נתח | כלים | מנהל ROI).
- לחץ על תוספים | ההתקנה, ובחר את אלגוריתם הדופק כדי להתקין את הקוד בתיקיית ברירת המחדל, ולאחר מכן בחר את התוסף בתחתית לשונית התוספים . הקלט את הפעימות לדקה (bpm) מהחלון המוקפץ.
הערה: אלגוריתמים לזיהוי תמונות נכתבו בהתאמה אישית כדי לזהות דופק על-ידי מדידת שינויים בצפיפות הפיקסלים הבודדים הקשורים להתכווצות סיסטולית חדרית. ניתן למצוא את הקוד בכתובת https://github.com/dpoburko/zFish_HR.
- מדוד את מידות קרום הלב באמצעות כלי חינמי כגון ImageJ כדי לצייר באופן חופשי ROIs סביב שק קרום הלב ואחת העיניים. פתחו את התמונה ב- ImageJ והשתמשו בכלי בחירת המצולעים כדי לצייר תחילה החזר השקעה מסביב לשק קרום הלב, תוך שמירה במנהל החזר ההשקעה כמו בשלב 5.1.2, וחזרו על הפעולה לעין. בחר/י את שני ה-ROIs האלה בהחזר ההשקעה על ניהולr, ואז לחץ/י על ״מדוד״. רשום את השטח עבור כל אחד מהם כדי לחשב מאוחר יותר את שטח שק קרום הלב המנורמל לאזור העיניים בכל זחל.
- לאחר מדידות דופק וקרום הלב, הקליטו אק"ג מזחלים בודדים.
הערה: פרוטוקולים לרישום אק"ג של דגי זברה תוארו בעבר33,34,35,36.
6. גנוטיפ של דג זברה זחלי בעריכת CRISPR-Cas9
- לאחר ניתוחים פנוטיפיים, בצע גנוטיפ פוטנציאלי מחוץ למטרה כדי לאשר עריכת גנים HDR מדויקת ומדויקת.
- הרדימו כל זחל 3 dpf ב-0.3% MS-222 וקליפס זנב כדי לבודד gDNA בשיטת HOTShot37. דגירה של כל קליפס זנב שנכרת ב-15 μL של 25 mM NaOH בטמפרטורה של 95 מעלות צלזיוס למשך 20 דקות. לאחר מכן, נטרל עם 1.5 μL של Tris-HCl וצנטריפוגה ב 13,800 x גרם במשך 30 שניות. שמור על הסופרנטנט, המכיל gDNA שחולץ.
- לשחזר את הזחלים בתקשורת E3 ולהחזיר אותם למערכת הדיור אם פיתוח נוסף או מחקר מיועד.
- באמצעות gDNA שחולץ כתבנית, בצע ריצוף סנגר מבוסס PCR של אתרים ממוקדי מטרה ואתרים פוטנציאליים מחוץ למטרה.
הערה: אופציונלי: גישת PCR מקוננת עשויה להועיל לאזורי גנים מסוימים. - ודא שתכנון הפריימר על המטרה לוכד את אתר המוטציה ואת אתר קשירת ה-sgRNA הקרוב ביותר. תכנן פריימר ריצוף נפרד כדי לזהות את המעבר מזרוע ההומולוגיה שהוכנסה ומגן המטרה כדי לאשר את האינטגרציה לגן המעניין. תכנן פריימרים כדי לרצף את שלושת האתרים הפוטנציאליים המובילים מחוץ למטרה שזוהו בשלב 1.2.3.
הערה: תוכנות לעיצוב מדריכים מציעות לעתים קרובות פריימרים לשימוש, אך ייתכן שיהיה צורך בהתאמה אישית כדי להשיג תוצאות מיטביות. - בצע הידור גנוטיפ על המטרה ומחוצה לה, קצב לב, ממדים קרום הלב, פנוטיפ א.ק.ג. ונתוני גנים מדווחים הניתנים לזיהוי עבור כל דג זברה.
תוצאות
השימוש המוצלח בגישת הקריספר להחלפת אקסון דו-sgRNA מודגש על ידי הקדמה וזיהוי פשוט של עריכה מדויקת כדי להנדס את הגרסה הקשורה ל-LQTS, R56Q, בגן zkcnh6a בדגי זברה. איור 6 מראה זחלי 3 dpf מייצגים שהוזרקו בשלב העובר החד-תאי עם רכיבי קריספר כפי שתואר לעיל. איור 6A מראה את נוכחות ביטוי הגנים של כתב YFP mVenus בעדשת העין כדיווח חיובי של שילוב תבניות מוצלח. איור 6B,C מראה את הכרומטוגרמות של ריצוף סנגר המתקבלות מדנ"א גנומי שבודד מדגימות קליפס זנב של דגים מסוג בר ודגים חיוביים לגנים, בהתאמה. דגים חיוביים לגנים נמצאו כבעלי עריכה מדויקת, G ל-A, אשר מציגה את גרסת R56Q לתוך zkcnh6a. גנוטיפ הראה מתאם של 100% בין ביטוי גנים מדווח YFP לבין נוכחות של עריכת הגן R56Q מדויקת, ואישר את כלי הסינון הפלואורסצנטי הזה.
פנוטיפ של זחלי דגי זברה שעברו עריכה גנטית נערך ב-3 dpf. איור 7 מראה תוצאות מייצגות של זחלים מסוג בר ו-R56Q שעברו עריכה גנטית. הדופק זוהה על ידי לכידת וידאו כמתואר לעיל. דוגמה למדידת ממדים קרום הלב כיחס בין שטח העין מוצגת (איור 7A). איור 7B מתווה את קצב הלב כנגד ממדים פריקרדיאליים מנורמלים, ומדגיש מגמה של ברדיקרדיה עם בצקת קרום הלב הגוברת, הקשורה להפרעות של רפולריזציה לבבית בדגי זברה 8,38,39,40. איור 7C מציג דוגמה מייצגת של הקלטות אק"ג מזחלי 3 dpf. מרווחים סטנדרטיים (QT, QRS) נמדדו מאותות אק"ג ממוצעים.

איור 1: שילוב תבנית HDR בגנום של דגי הזברה. אפור כהה, זרועות הומולוגיה; ירוק, sgRNA מדריך מטרות עם מוטציה שקטה כדי למנוע חיתוך מחדש של Cas9; אפור בהיר, אקסון מטרה של עניין; קו אדום, מוטציה נקודתית; צהוב, גן כתב mVenus YFP תחת מקדם קריסטלין α; קווים מקווקווים מציינים הומולוגיה. כאן, העריכה המדויקת הממוקדת הייתה R56Q באקסון 2 של הגן zkcnh6a . קיצורים: HDR = תיקון מכוון הומולוגיה; sgRNA = RNA מדריך יחיד; YFP = חלבון פלואורסצנטי צהוב; DSB = שבר דו-גדילי; WT = סוג פראי. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.

איור 2: סיכום השלבים להנדסה מדויקת של עריכות בגנים של דגי זברה באמצעות גישת CRISPR-Cas9 של שני sgRNA (מספרי שלבים קשורים בפרוטוקול מסומנים בסוגריים). קיצורים: sgRNA = RNA מדריך יחיד; YFP = חלבון פלואורסצנטי צהוב; gDNA = דנ"א גנומי; אק"ג = אלקטרוקרדיוגרמה; dpf = ימים לאחר ההפריה; MS-222 = טריקיין מתאן סולפונט. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
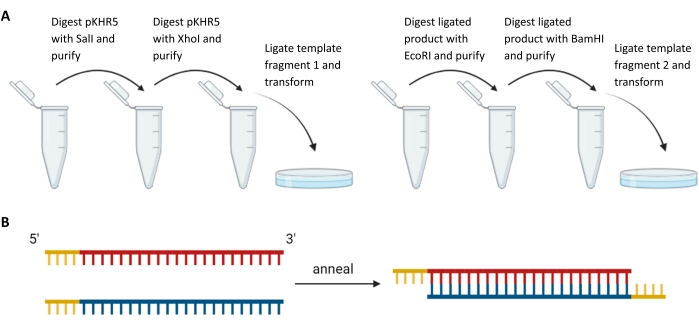
איור 3: הכנת מקטעי תבנית אקסוגניים ומדריכי sgRNA. (A) עיכול וקשירה רציפים של מקטעי תבנית במעלה הזרם ובמורד הזרם של רצף הגנים המדווחים mVenus YFP ב-pKHR5. (B) חישול של זוגות sgRNA משלימים עם מגבלת יתר לקשירת DR274. קיצורים: sgRNA = RNA מדריך יחיד; YFP = חלבון פלואורסצנטי צהוב. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
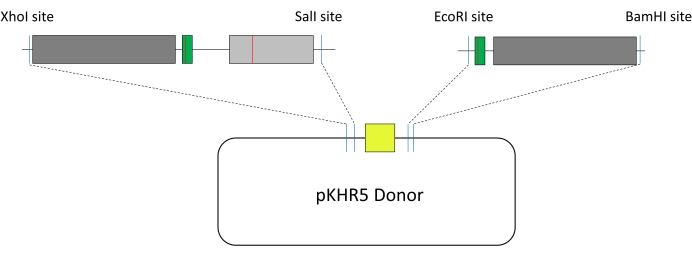
איור 4: בניית תבנית HDR. אפור כהה, זרועות הומולוגיה; ירוק, sgRNA מדריך מטרות עם מוטציה שקטה כדי למנוע חיתוך מחדש של Cas9; אפור בהיר, אקסון מטרה של עניין; קו אדום, מוטציה נקודתית; צהוב, גן כתב mVenus YFP תחת מקדם קריסטלין α; קו כחול כהה, נוספו אתרי הגבלה. שני שברי התבנית משולבים בתורם פלסמיד pKHR5. קיצורים: HDR = תיקון מכוון הומולוגיה; sgRNA = RNA מדריך יחיד. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.

איור 5: מיקרו-הזרקה של עוברי דגי זברה חד-תאיים עם רכיבי CRISPR-Cas9. סרגל קנה מידה = 0.5 מ"מ. קיצורים: HDR = תיקון מכוון הומולוגיה; sgRNA = RNA מדריך יחיד. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.

איור 6: זיהוי קל של פלואורסצנטיות של הגן mVenus YFP מצביע על שילוב חיובי של תבנית אקסוגנית HDR בגן המטרה. (A) דוגמה לביטוי mVenus YFP בעין דג זברה (חץ) בזחל דג זברה ערוך. (B) עריכות מוצלחות מאושרות על ידי רצף כרומטוגרמות (שמאל, WT; ימין, עריכת R56Q). אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.

איור 7: ניתוח פנוטיפי של השלכות לב בדגי זברה 3 dpf בעקבות העריכה המדויקת של R56Q בגן המטרה zkcnh6a. (A) זיהוי תמונה של מידות קרום הלב ביחס לגודל העין באמצעות כלי המצולע ב- ImageJ. גבולות השק הפריקרדיאלי סומנו על ידי המשתמש ממסגרת הקלטה אחת המבוססת על שינויים בשקיפות ובפיגמנטציה. מוצגות דוגמאות לממדים פריקרדיאליים תקינים ולהתפשטות קרום הלב. סרגל קנה מידה = 0.5 מ"מ. (B) מתאם בין ממדי קרום הלב (יחסית לממד העין) לבין קצב הלב, R2 = 0.33. (C) דוגמה לרישום א.ק.ג. מלב זחל דג זברה של 3 dpf (משמאל) ומקומפלקסים ממוצעים (מימין). קצב לב, 131 פעימות לדקה; מרווח QTc מתוקן דופק, 460 אלפיות השנייה. קיצורים: dpf = ימים לאחר ההפריה; אק"ג = אלקטרוקרדיוגרמה. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
Discussion
ההנדסה של עריכות גנים מדויקות באמצעות CRISPR-Cas9 מאותגרת על ידי היעילות הנמוכה של מנגנוני HDR והזיהוי היעיל שלהם. כאן מתוארת גישה להחלפת אקסון דו-sgRNA המבוססת על CRISPR-Cas9 שמייצרת עריכות מדויקות בדגי זברה עם זיהוי חזותי פשוט של עריכות חיוביות. היעילות של גישה זו מודגמת על ידי יצירת עריכות מדויקות בגן zkcnh6a . מאמר זה מראה כיצד ניתן להעריך את תפקוד הלב בזחלי דגי זברה שעברו עריכה גנטית באמצעות מדדים פנוטיפיים לא פולשניים של קצב הלב, ממדי קרום הלב ומורפולוגיה של אק"ג. גישה זו, החל מהכנסת עריכת גנים ועד להערכה פנוטיפית, יכולה להסתיים מתחילתה ועד סופה תוך כשבוע.
היתרונות של גישת העריכה והפנוטיפ הנ"ל הם הקלות של תכנון שינוי קריספר, הישימות הרחבה במערכות פיזיולוגיות מרובות, היכולת להחדיר גנים גדולים או שברי גנים, והיכולת לעקוב אחר השפעות שונות לאורך לאורך התפתחות ודורות. ההצלחה של עריכות מדויקות בגישה זו עשויה להיות קשורה לשילוב של גודל התבנית הגדול (בשל הוספת הגן המדווח וזרועות הומולוגיה ארוכות), שהוכח כמגביר את היעילות של עריכות בדג זברה14, ואסטרטגיית שני מדריכי sgRNA, אשר שימשה ביעילות בעריכות המושרות על ידי דג זברה TALEN8.
חוזקה מסוימת של הגישה המתוארת היא היכולת להחדיר גנים גדולים או שברי גנים. זה עשוי להיות שימושי, למשל, כדי להכניס אורתולוגים אנושיים41, מה שמאפשר אפיון והשוואה קליניים יותר בין אורתולוגים. לחלופין, ניתן להחדיר גם גנים המקודדים אנזימי Cas, מה שמאפשר שורה של דגי זברה עם מנגנוני עריכה in vivo CRISPR, המספקים מערכת אינדוקטיבית. באופן דומה, ניתן לשלב מנגנוני קריספר חלופיים, כגון עריכה ראשונית, וליצור שורה של דגי זברה הניתנים לעריכה מדויקת ויעילה.
למרות היתרונות של גישה זו, יש כמה מגבלות. ראשית, רק גן אחד ולוקוס שונו, ויש צורך בבדיקות נוספות באתרים אחרים או בגנים אחרים כדי להעריך עד כמה גישה זו ישימה באופן נרחב. בשל זרועות ההומולוגיה הארוכות הנדרשות, עלויות עיצוב התבנית גבוהות יותר; עם זאת, זה עשוי להתקזז על ידי סינון יעיל. מגבלה נוספת היא שגישת הסינון דורשת יכולת זיהוי פלואורסצנטית. עם זאת, הדרישות האופטיות נמוכות יחסית וניתן לבנות אותן בהתאמה אישית או לרכוש אותן מסחרית בעלות נמוכה למדי. שימוש בגישה של שני sgRNA מגדיל את מספר האירועים הפוטנציאליים מחוץ למטרה; עם זאת, סביר להניח שהדבר מתמתן על ידי ההסתברות הנמוכה יותר ששני מדריכי ה-sgRNA יתנו שניהם באופן שמקל על שילוב התבנית כדי להניב ביטוי גנים מדווח. לבסוף, שימוש ב-Cas9 mRNA עלול להוביל לפסיפסים מכיוון שה-Cas9 אינו פעיל עד לשלבי התפתחות מאוחרים יותר. ניתן להסביר זאת על ידי ריצוף סוגי רקמות מסוימים; עם זאת, בהתחשב בגודל של זחלי דגי הזברה, זה מאתגר מבחינה טכנית.
לסיכום, גישת העריכה המדויקת של CRISPR-Cas9 two-sgRNA בדגי זברה מאפשרת זיהוי חזותי פשוט של עריכות חיוביות וניתן להתאים אותה לשילוב גנים גדולים בעלי עניין בכל מוקד. בשילוב עם אמצעים פנוטיפיים, זה מאפשר פלטפורמת תפוקה אמינה וגבוהה לחקר גרסאות לב רלוונטיות מבחינה קלינית.
Disclosures
למחברים אין ניגודי עניינים לחשוף.
Acknowledgements
מחקר זה נתמך על ידי מענק פרויקט מחקר של המכונים הקנדיים לבריאות (T.W.C.) ומענקי תגלית של המועצה למחקר מדעי הטבע וההנדסה של קנדה (T.W.C.).
Materials
| Name | Company | Catalog Number | Comments |
| Program | |||
| CRISPOR | TEFOR Infrastructure | ||
| ENSEMBL | European Bioinformatics Institute | ||
| ImageJ | National Institutes of Health (NIH) | ||
| Micro-Manager | Open Source (Github) | ||
| NEBiocalculator | New England Biolabs (NEB) | ||
| EQUIPMENT | |||
| 24-well Plate | VWR | ||
| 25 mm Petri Dish | VWR | ||
| Blackfly USB3 Camera | Teledyne FLIR | ||
| C1000 Thermal Cycler | Bio-Rad | ||
| Centrifuge 5415C | Eppendorf | ||
| EZNA Gel Extraction Kit | Omega Biotek | ||
| MAXIscript T7 Transcription Kit | Invitrogen | ||
| MaxQ 5000 Incubator | Barnstead Lab Line | ||
| Miniprep Kit | Qiagen | ||
| mMessage mMachine T7 Ultra Transcription Kit | Invitrogen | ||
| ND1000 Spectrophotometer | Nanodrop | ||
| PCR Purification Kit | Qiagen | ||
| PLI 100A Picoinjector | Harvard Apparatus | ||
| PowerPac Basic Power Supply | Bio-Rad | ||
| Stemi 305 Steroscope | Zeiss | ||
| Wide Mini Sub Cell GT Electrophoresis System | Bio-Rad | ||
| ZebTec Zebrafish Housing System | Tecniplast | ||
| SERVICES | |||
| Gene Synthesis | Genewiz | ||
| Sanger Sequencing | Genewiz | ||
| REAGENTS | |||
| 10β Competent Cells | NEB | ||
| 10X PCR Buffer | Qiagen | ||
| 100 mM Nucleotide Mixture | ABM | ||
| Ampicillin | Sigma | ||
| BamHI Endonuclease w/ buffer | NEB | ||
| BsaI Endonuclease w/ buffer | NEB | ||
| DR274 Plasmid (XL1 Blue bacterial agar stab) | Addgene | ||
| EcoRI Endonuclease w/ buffer | NEB | ||
| Glycerol | |||
| HEPES | Sigma | ||
| HindIII Endonuclease w/ buffer | NEB | ||
| Kanamycin | Sigma | ||
| Methylene Blue | Sigma | ||
| MLM3613 Plasmid (XL1 Blue bacterial agar stab) | Addgene | ||
| MS-222 (Tricaine) | Sigma | ||
| pKHR5 Plasmid (DH5α bacterial agar stab) | Addgene | ||
| PmeI Endonuclease w/ buffer | NEB | ||
| SalI Endonuclease w/ buffer | NEB | ||
| Sodium Hydroxide | Sigma | ||
| T4 Ligase w/ buffer | Sigma | ||
| Taq Polymerase | Qiagen | ||
| TE Buffer | Sigma | ||
| Tris Hydrochloride | Sigma | ||
| XhoI Endonuclease w/ buffer | NEB | ||
| RECIPES | |||
| Solution | Component | Supplier | |
| Annealing Buffer (pH 7.5-8.0) | 10 mM Tris | Sigma | |
| 50 mM NaCl | Sigma | ||
| 1 mM EDTA | Sigma | ||
| E3 Media (pH 7.2) | 5 mM NaCl | Sigma | |
| 0.17 mM KCl | Sigma | ||
| 0.33 mM CaCl2 | Sigma | ||
| 0.33 mM MgSO4 | Sigma | ||
| Injection Buffer (pH 7.5) | 20 mM HEPES | Sigma | |
| 150 mM KCl | Sigma |
References
- Zarei, A., Razban, V., Hosseini, S. E., Tabei, S. M. B. Creating cell and animal models of human disease by genome editing using CRISPR/Cas9. The Journal of Gene Medicine. 21 (4), 3082(2019).
- Lee, H., Yoon, D. E., Kim, K. Genome editing methods in animal models. Animal Cells and Systems. 24 (1), 8-16 (2020).
- Li, Q., et al. Applications of genome editing technology in animal disease modeling and gene therapy. Computational and Structural Biotechnology Journal. 17, 689-698 (2019).
- Gut, P., Reischauer, S., Stainier, D. Y. R., Arnaout, R. Little fish, big data: Zebrafish as a model for cardiovascular and metabolic disease. Physiological Reviews. 97 (3), 889-938 (2017).
- Kegel, L., et al. Forward genetic screen using zebrafish to identify new genes involved in myelination. Oligodendrocytes: Methods and Protocols. 1936, 185-209 (2019).
- Shah, A. N., Davey, C. F., Whitebirch, A. C., Miller, A. C., Moens, C. B. Rapid reverse genetic screening using CRISPR in zebrafish. Nature Methods. 12 (6), 535-540 (2015).
- González-Rosa, J. M. Zebrafish models of cardiac disease: From fortuitous mutants to precision medicine. Circulation Research. 130 (12), 1803-1826 (2022).
- Hoshijima, K., Jurynec, M. J., Grunwald, D. J. Precise editing of the Zebrafish genome made simple and efficient. Developmental Cell. 36 (6), 654-667 (2016).
- Albadri, S., Del Bene, F., Revenu, C. Genome editing using CRISPR/Cas9-based knock-in approaches in zebrafish. Methods. 121-122, 77-85 (2017).
- Armstrong, G. A. B., et al. Homology directed knockin of point mutations in the zebrafish tardbp and fus genes in ALS using the CRISPR/Cas9 system. PLoS One. 11 (3), 0150188(2016).
- Bai, H., et al. CRISPR/Cas9-mediated precise genome modification by a long ssDNA template in zebrafish. BMC Genomics. 21 (1), 67(2020).
- de Vrieze, E., et al. Efficient generation of knock-in zebrafish models for inherited disorders using CRISPR-Cas9 ribonucleoprotein complexes. International Journal of Molecular Sciences. 22 (17), 9429(2021).
- Eschstruth, A., Schneider-Maunoury, S., Giudicelli, F. Creation of zebrafish knock-in reporter lines in the nefma gene by Cas9-mediated homologous recombination. Genesis. 58 (1), 23340(2020).
- Irion, U., Krauss, J., Nüsslein-Volhard, C. Precise and efficient genome editing in zebrafish using the CRISPR/Cas9 system. Development. 141 (24), 4827-4830 (2014).
- Kimura, Y., Hisano, Y., Kawahara, A., Higashijima, S. Efficient generation of knock-in transgenic zebrafish carrying reporter/driver genes by CRISPR/Cas9-mediated genome engineering. Scientific Reports. 4 (1), 6545(2014).
- Levic, D. S., Yamaguchi, N., Wang, S., Knaut, H., Bagnat, M. Knock-in tagging in zebrafish facilitated by insertion into non-coding regions. Development. 148 (19), (2021).
- Prykhozhij, S. V., et al. Optimized knock-in of point mutations in zebrafish using CRISPR/Cas9. Nucleic Acids Research. 46 (17), 102(2018).
- Wierson, W. A., et al. Efficient targeted integration directed by short homology in zebrafish and mammalian cells. eLife. 9, 53968(2020).
- Boel, A., et al. CRISPR/Cas9-mediated homology-directed repair by ssODNs in zebrafish induces complex mutational patterns resulting from genomic integration of repair-template fragments. Disease Models & Mechanisms. 11 (10), (2018).
- Tessadori, F., et al. Effective CRISPR/Cas9-based nucleotide editing in zebrafish to model human genetic cardiovascular disorders. Disease Models & Mechanisms. 11 (10), (2018).
- Zhang, Y., Zhang, Z., Ge, W. An efficient platform for generating somatic point mutations with germline transmission in the zebrafish by CRISPR/Cas9-mediated gene editing. The Journal of Biological Chemistry. 293 (17), 6611-6622 (2018).
- Vornanen, M., Hassinen, M. Zebrafish heart as a model for human cardiac electrophysiology. Channels. 10 (2), 101-110 (2016).
- Nemtsas, P., Wettwer, E., Christ, T., Weidinger, G., Ravens, U. Adult zebrafish heart as a model for human heart? An electrophysiological study. Journal of Molecular and Cellular Cardiology. 48 (1), 161-171 (2010).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biology. 17 (1), 148(2016).
- Liang, X., Potter, J., Kumar, S., Ravinder, N., Chesnut, J. D. Enhanced CRISPR/Cas9-mediated precise genome editing by improved design and delivery of gRNA, Cas9 nuclease, and donor DNA. Journal of Biotechnology. 241, 136-146 (2017).
- Richardson, C. D., Ray, G. J., DeWitt, M. A., Curie, G. L., Corn, J. E. Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nature Biotechnology. 34 (3), 339-344 (2016).
- Sambrook, J., Fritsch, E. F., Maniatis, T. Precipitation of Large RNAs with Lithium Chloride. Molecular Cloning: A Laboratory Manual, Book 3. E.15. , Cold Spring Harbor Laboratory. Long Island, NY. (1989).
- Sambrook, J., Fritsch, E. F., Maniatis, T. Agarose Gel Electrophoresis. Molecular Cloning: A Laboratory Manual, Book 1. , Cold Spring Harbor Laboratory. Long Island, NY. 3-20 (1989).
- Sorlien, E. L., Witucki, M. A., Ogas, J. Efficient production and identification of CRISPR/Cas9-generated gene knockouts in the model system Danio rerio. Journal of Visualized Experiments. (138), e56969(2018).
- Avdesh, A., et al. Regular care and maintenance of a zebrafish (Danio rerio) laboratory: An introduction. Journal of Visualized Experiments. (69), e4196(2012).
- Rosen, J. N., Sweeney, M. F., Mably, J. D. Microinjection of zebrafish embryos to analyze gene function. Journal of Visualized Experiments. (25), e1115(2009).
- Kossack, M. E., Draper, B. W. Genetic regulation of sex determination and maintenance in zebrafish (Danio rerio). Current Topics in Developmental Biology. 134, 119-149 (2019).
- Tanaka, Y., et al. Functional analysis of KCNH2 gene mutations of type 2 long QT syndrome in larval zebrafish using microscopy and electrocardiography. Heart and Vessels. 34 (1), 159-166 (2019).
- Dhillon, S. S., et al. Optimisation of embryonic and larval ECG measurement in zebrafish for quantifying the effect of QT prolonging drugs. PLoS One. 8 (4), 60552(2013).
- Yu, F., et al. Evolving cardiac conduction phenotypes in developing zebrafish larvae: Implications to drug sensitivity. Zebrafish. 7 (4), 325-331 (2010).
- Hurst, R. M. Development and optimization of tools for embryonic electrocardiograph recording for heart dysfunction in zebrafish. , University of Birmingham. PhD thesis (2018).
- Meeker, N. D., Hutchinson, S. A., Ho, L., Trede, N. S. Method for isolation of PCR-ready genomic DNA from zebrafish tissues. BioTechniques. 43 (5), 610-614 (2007).
- Arnaout, R., et al. Zebrafish model for human long QT syndrome. Proceedings of the National Academy of Sciences of the United States of America. 104 (27), 11316-11321 (2007).
- Milan, D. J., Peterson, T. A., Ruskin, J. N., Peterson, R. T., MacRae, C. A. Drugs that induce repolarization abnormalities cause bradycardia in zebrafish. Circulation. 107 (10), 1355-1358 (2003).
- Langheinrich, U., Vacun, G., Wagner, T. Zebrafish embryos express an orthologue of HERG and are sensitive toward a range of QT-prolonging drugs inducing severe arrhythmia. Toxicology and Applied Pharmacology. 193 (3), 370-382 (2003).
- MacRae, C. A. Cardiac arrhythmia: In vivo screening in the zebrafish to overcome complexity in drug discovery. Expert Opinion on Drug Discovery. 5 (7), 619-632 (2010).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved