Method Article
RNA-seq Kullanarak Moleküler Evrimi ve Gen ekspresyonını Araştırmak için Biyoinformatik Boru Hattı
Bu Makalede
Özet
Bu protokolün amacı, RNA dizileme verilerini kullanarak aday genlerin evrimini ve ekspresyonunun araştırılmasıdır.
Özet
Tüm genom veya transkriptom verileri gibi büyük veri kümelerini damıtmak ve raporlamak genellikle göz korkutucu bir iştir. Sonuçları parçalamanın bir yolu, organizma için önemli olan bir veya daha fazla gen ailesine odaklanmak ve çalışmaktır. Bu protokolde, bir filogeni oluşturmak ve ilgi genlerinin ekspresyonunun ölçülmesi için biyoinformatik adımları özetliriz. Filogenetik ağaçlar, genlerin türler içinde ve arasında nasıl geliştiğine dair fikir verebilir ve ortolojiyi ortaya getirebilir. Bu sonuçlar, farklı bireylerde veya dokularda bu genlerin ekspresyonunun karşılaştırılması için RNA-seq verileri kullanılarak geliştirilebilir. Moleküler evrim ve ifade çalışmaları, türler arasındaki evrim ve gen fonksiyonunun korunması modlarını ortaya çıkarabilir. Bir gen ailesinin karakterizasyonu gelecekteki çalışmalar için bir sıçrama tahtası görevi görebilirsiniz ve yeni bir genom veya transkriptom kağıdında önemli bir gen ailesini vurgulayabilir.
Giriş
Sıralama teknolojilerindeki gelişmeler, model olmayan organizmaların genomlarının ve transkriptomlarının dizilimini kolaylaştırdı. Dna ve RNA'yı birçok organizmadan sıralamanın artan fizibilitesine ek olarak, ilgi genlerini incelemek için çok sayıda veri kamuya açıktır. Bu protokolün amacı, ilgi çekici organizmada önemli bir rol oynayabilecek genlerin moleküler evrimini ve ekspresyonunun araştırılması için biyoinformatik adımlar sağlamaktır.
Bir gen veya gen ailesinin evrimini araştırmak, biyolojik sistemlerin evrimi hakkında fikir verebilir. Bir gen ailesinin üyeleri tipik olarak korunmuş motifler veya homolog gen dizileri tanımlanarak belirlenir. Gen ailesi evrimi daha önce uzaktan ilişkili model organizmaların genomları kullanılarak araştırılmıştır1. Bu yaklaşımın bir sınırlaması, bu gen ailelerinin yakından ilişkili türlerde nasıl evrimleştiğinin ve farklı çevresel seçici baskıların rolünün açık olmamasıdır. Bu protokolde, yakından ilişkili türlerde homologlar için bir arayışa yer verdik. Fitülm düzeyinde bir filogeny üreterek, korunmuş genler veya soyuna özgü çoğaltmalar gibi gen ailesi evrimi eğilimlerini not edebiliriz. Bu seviyede genlerin ortolog mu yoksa paralog mu olduğunu da araştırabiliriz. Birçok homolog muhtemelen birbirine benzer şekilde işlev görürken, bu mutlaka durum2. Filogenetik ağaçların bu çalışmalara dahil edilerek bu homolog genlerin ortolog olup olmadığının çözülmesi önemlidir. Ökaryotlarda, birçok ortez, memeli proteinlerinin maya ortezlerinin işlevini geri kazanma yeteneğinin kanıt ettiği gibi hücre içinde benzer işlevleri korur3. Bununla birlikte, ortezsiz bir genin karakterize bir işlev4gerçekleştirdiği durumlar vardır.
Filogenetik ağaçlar genler ve türler arasındaki ilişkileri tanımlamaya başlar, ancak işlev sadece genetik ilişkilere göre atanamaz. Gen ekspresyon çalışmaları fonksiyonel ek açıklamalar ve zenginleştirme analizi ile birlikte gen fonksiyonu için güçlü destek sağlar. Gen ekspresyonunun ölçülebildiği ve bireyler veya doku tipleri arasında karşılaştırılabildiği durumlar potansiyel işlevin daha fazla anlatılması olabilir. Aşağıdaki protokol Hydra vulgaris7'deopsin genlerinin araştırılmasında kullanılan yöntemleri izler, ancak herhangi bir türe ve herhangi bir gen ailesine uygulanabilirler. Bu tür çalışmaların sonuçları, model olmayan organizmalarda gen fonksiyonu ve gen ağları hakkında daha fazla araştırma için bir temel sağlar. Örnek olarak, fototransdüksiyon basamaklanmasını başlatan proteinler olan opsinlerin filogenisinin araştırılması, gözlerin evrimine ve ışık algılamaya bağlam verir 8,9,10,11. Bu durumda, model olmayan organizmalar özellikle cnidarians veya ctenophores gibi bazal hayvan türlerinin korunması veya fototransdüksiyon kaskadındaki değişiklikler ve clades12 , 13,14. Benzer şekilde, diğer gen ailelerinin filogeni, ifade ve ağlarının belirlenmesi, adaptasyonların altında kalan moleküler mekanizmalar hakkında bizi bilgilendirecektir.
Protokol
Bu protokol UC Irvine hayvan bakım yönergelerini izler.
1. RNA-seq kütüphane hazırlığı
- Aşağıdaki yöntemleri kullanarak RNA'yı yalıtın.
- Örnekleri toplayın. RNA daha sonra çıkarılacaksa, numuneyi flaşla dondurun veya RNA depolama çözeltisi15'e (Malzeme Tablosu) yerleştirin.
- İlgi çekici dokuları ayırmak için organizmayı ötenazi ve diseksiyon.
- Bir ekstraksiyon kiti kullanarak toplam RNA'yı çıkarın ve bir RNA arıtma kiti kullanarakRNA'yı arındırın( Malzeme Masası )
NOT: Farklı türler ve doku tipleri için daha iyi çalışabilecek protokoller ve kitler vardır16,17. Bir kelebek18 ve jelatinli Hydra19'un farklı vücut dokularından RNA çıkardık (tartışmaya bakın). - Her numunenin RNA konsantrasyonu ve kalitesiniölçün( Malzeme Tablosu ). CDNA kitaplıkları oluşturmak için ideal olarak 920'ye daha yakın, RNA bütünlük numaraları (RIN) 8'den yüksek olan örnekleri kullanın.
- cDNA kitaplığını ve sırasını aşağıdaki gibi oluşturun.
- Kitaplık hazırlık kullanım kılavuzuna göre cDNA kitaplıkları oluşturun (tartışmaya bakın).
- cDNA konsantrasyonu ve kalitesini belirleyin (Malzeme Tablosu).
- Kitaplıkları çoklayın ve sıralayın.
2. Bilgisayar kümesine erişme
NOT: RNA-seq analizi büyük dosyaların manipülasyonu gerektirir ve en iyi bilgisayar kümesinde(Malzeme Tablosu)yapılır.
- Terminal (Mac) veya PuTTY (Windows) uygulama penceresinde ssh username@clusterlocation komutunu kullanarak bilgisayar kümesi hesabına giriş yapın.
3. RNA-seq okumaları elde edin
- Sıralama olanaktan veya bir yayında oluşturulan veriler için, yatırıldığı veri deposundan (3.2 veya 3.3) RNA-seq okumaları alın.
- ArrayExpress gibi depolardan veri yüklemek için aşağıdakileri yapın:
- Katılım numarasını kullanarak sitede arama yap.
- Verileri indirmek için bağlantıyı bulun, sonra sol tıklatın ve Bağlantıyı Kopyala 'yıseçin.
- Terminal penceresinde, verileri çözümleme için dizine kopyalamak üzere wget yazın ve Bağlantıyı yapıştır'ı seçin.
- NCBI Kısa Okuma Arşivi (SRA) verilerini indirmek için şu alternatif adımları izleyin:
- Terminalde Wget kullanarak SRA Toolkit v. 2.8.1'i indirin.
NOT: Programları bilgisayar kümesine indirip yüklemek kök erişimi gerektirebilir, yükleme başarısız olursa bilgisayar kümesi yöneticinize başvurun. - Tar -xvf $TARGZFILEyazarak programı yüklemeyi tamamla.
- İndirmek istediğiniz örnekler için SRA katılım numarası için NCBI'yi arayın, SRRXXXXXX biçimine sahip olmalıdır.
- Terminal penceresine [sratoolkit location]/bin/prefetch SRRXXXXXX yazarak RNA-seq verilerini elde edin.
- Eşleştirilmiş uçlu dosyalar için [sratoolkit location]/bin/fastq-dump --split-files SRRXXXXXX iki fastq dosyası (SRRXXXXXX_1.FASTQ ve SRRXXXXXX_2.FASTQ) almak için yazın.
NOT: Trinity de novo derlemesi yapmak için [sratoolkit location]/bin/fastq-dump --defline-seq '@$sn[_$rn]/$ri' --split-files SRRXXXXXX
- Terminalde Wget kullanarak SRA Toolkit v. 2.8.1'i indirin.
4. Adaptörleri ve düşük kaliteli okumaları kırpın (isteğe bağlı)
- Trimmomatic21 v. 0.35'i bilgi işlem kümesine yükleyin veya yükleyin.
- RNA-seq veri dosyalarının bulunduğu dizine, trimmomatik jar dosyasının konumunu, giriş FASTQ dosyalarını, çıkış FASTQ dosyalarını ve okuma uzunluğu ve kalitesi gibi isteğe bağlı parametreleri içeren bir komut yazın.
NOT: Komut, okumaların ham ve istenen kalitesine ve uzunluğuna göre değişir. Nextera primer'ları ile Illumina 43 bp okumaları için şunları kullandık: java -jar /data/apps/trimmomatic/0.35/trimmomatic-0.35.jar PE $READ 1. FASTQ $READ 2. FASTQ paired_READ1. FASTQ unpaired_READ1. FASTQ paired_READ2. FASTQ unpaired_READ2. FASTQ ILLUMINACLIP:adapters.fa:2:30:10 LİDER:20 İZLEYEN:20 SLIDINGWINDOW:4:17 MINLEN:30.
5. Referans montaj elde edin
- İlgi gören türler için bir referans genom veya monte edilmiş transkriptom için google, EnsemblGenomes ve NCBI Genomları ve Nükleotid TSA'yı (Transcriptome Shotgun Assembly) arayın (Şekil 1).
NOT: Bir referans genom veya transkriptom mevcut değilse veya düşük kaliteliyse, bir de novo montajı oluşturmak için ADIM 6'ya geçin. - Bir referans genom veya monte edilmiş transkriptom varsa, aşağıdaki adımları izleyerek analizin gerçekleştirileceği yere fasta dosyası olarak indirin.
- Genom indirmek için bağlantıyı bulun, sol tıklatın ve Bağlantıyı Kopyalayın.
- Terminal penceresine wget yazın ve bağlantı adresini yapıştırın. Varsa, referans genom için GTF dosyasını ve protein FASTA dosyasını da kopyalayın.
6. Bir de novo montajı oluşturun (Adım 5'e Alternatif)
- Cat *READ1 yazarak tüm örnekler için RNA-seq READ1 ve READ2 fastq dosyalarını birleştirin. FASTQ > $all_READ1. FASTQ ve kedi *READ2. FASTQ > all_READ2. FASTQ terminal penceresinde.
- Trinity 22 v.2.8.5'i bilgi işlem kümesine yükleyin veya yükleyin.
- Terminale yazarak oluşturun ve birin: Trinity --seqType fq --max_memory 20G --left $all_READ1. FASTQ --sağ $all_READ2. HıZLıQ.
7. Harita genoma (7.1) veya de novo transkriptome (7.2) okur
- Harita, STAR 23 v.2.6.0c ve RSEM 24 v.1.3.0 kullanarak referans genomunu okur.
- STAR v. 2.6.0c'yi yükleyin veya yükleyin. ve RSEM v. 1.3.0 bilgi işlem kümesine.
- Rsem-prepare-reference --gtf $GENOME yazarak genomun dizinini dizine dizin. GTF -- yıldız -p 16 $GENOME. FASTA $OUTPUT.
- Eşleme, rsem-calculate-expression -p 16 --star --paired-end $READ 1 yazarak her örnek için ifadeyi okur ve hesaplar. FASTQ $READ 2. FASTQ $INDEX $OUTPUT.
- Mv RSEM.genes.results $sample.genes.results kullanarak sonuç dosyasını açıklayıcı bir şeyle yeniden adlandırın.
- rsem-generate-data-matrix *[genes/isoforms.results] > $OUTPUTyazarak tüm sayımların matrisini oluşturun.
- RNE-seq'i RSEM ve bowtie kullanarak Trinity de novo derlemesine eşleyin.
- Trinity 22 v.2.8.5, Bowtie25 v. 1.0.0 ve RSEM v. 1.3.0'ı yükleyin veya yükleyin.
- [trinity_location]/align_and_estimate_abundance.pl --prep-reference --transcripts $TRINITY yazarak her örnek için harita okuma ve hesaplama ifadesi. FASTA --seqType fq --sol $READ 1. FASTQ -- sağ $READ 2. FASTQ --est_method RSEM --aln_method papyon --trinity_mode --output_dir $OUTPUT.
- Mv RSEM.genes.results $sample.genes.results kullanarak sonuç dosyasını açıklayıcı bir şeyle yeniden adlandırın.
- [trinity_location]/abundance_estimates_to_matrix.pl --est_method RSEM *[genes/isoforms].results yazarak tüm sayımların matrisini oluşturun
8. İlgi genlerini tanımlayın
NOT: Aşağıdaki adımlar nükleotid veya protein FASTA dosyaları ile yapılabilir, ancak en iyi şekilde çalışır ve protein dizileri ile daha basittir. Proteinden proteine protein kullanarak yapılan BLAST aramalarının farklı türler arasında arama yaparken sonuç verme olasılığı daha yüksektir.
- Referans genom için STEP 5.2.2'nin FASTA protein dosyasını kullanın veya özel gen özelliği GTF oluşturmak için Ek Malzemeler'e bakın.
- De novo transkriptom için TransDecoder kullanarak bir protein FASTA oluşturun.
- Bilgisayar cluser'ına TransDecoder v. 5.5.0 yükleyin veya yükleyin.
- [Transdecoder location]/TransDecoder.LongOrfs -t $TRINITY yazarak en uzun açık okuma çerçevesini ve tahmin edilen peptit sırasını bulun. FASTA.
- NCBI Genbank'ta yakından ilişkili türlerde homologlar arayın.
- Bir internet tarayıcısı penceresi açın ve https://www.ncbi.nlm.nih.gov/genbank/ gidin.
- Arama çubuğuna, ilgi geninin adını ve sıralanmış veya cins veya fitülar olan yakından ilişkili türlerin adını yazın. Arama çubuğunun solunda proteini seçin ve ara'yı tıklatın.
- Gönder'i tıklatıp Dosya'yı seçerek dizileri ayıklayın. Biçim'in altında FASTA'yı seçin ve Dosya Oluştur'u tıklatın.
- Yerel terminal penceresinde scp $FASTA username@clusterlocation:/$DIR yazarak homologların FASTA dosyasını bilgisayar kümesine taşıyın veya dosyaları bilgisayara ve kümeden aktarmak için FileZilla'yı kullanın.
- BLAST+26kullanarak aday genleri arayın.
- BLAST+ v. 2.8.1'i bilgisayar kümesine yükleyin veya yükleyin.
- Bilgisayar kümesinde, $PEP [BLAST+ location]/makeblastdb -in yazarak genom veya transcriptome çevrilmiş protein FASTA'dan bir BLAST veritabanı oluşturun. FASTA -dbtype prot -out $OUTPUT
- [BLAST+ location]/blastp -db $DATABASE -query $FASTA -evalue 1e-10 -outfmt 6 -max_target_seqs 1 -out-out $OUTPUT yazarak NCBI'denilgi gören türlerin veritabanına homolog gen dizilerini BLAST.
- Daha fazlakomut kullanarak çıktı dosyasını görüntüleyin. İlgi çekici türlerden benzersiz gen kimliklerini yeni bir metin dosyasına kopyalayın.
- Perl -ne 'if(/^>(\S+)/){$c=$i{$1}}$c?print:chomp;$i{$_}=1 if @ARGV' $gene_id.txt $PEP yazarak aday genlerin dizilerini ayıklayın. FASTA > $OUTPUT.
- Karşılıklı BLAST kullanarak gen ek açıklamalarını onaylayın.
- İnternet tarayıcısında https://blast.ncbi.nlm.nih.gov/Blast.cgi gidin.
- Tblastn 'ıseçin, sonra aday sıralarını yapıştırın, Gereksiz olmayan protein dizisi veritabanını seçin ve BLAST 'ıtıklatın.
- Genom veya transkriptomdaki tüm genlere gen ontolojisi (GO) terimleriyle açıklama vererek ek genleri tanımlayın (bkz. tartışma).
- FASTA proteinini yerel bilgisayara aktarın.
- Blast2GO27 , 28,29v. 5.2'yi yerel bilgisayara indirip yükleyin.
- Blast2GO'yuaçın, Dosya'yı tıklatın, Yükle'ye gidin, Sıraları Yükle'ye gidin, Fasta Dosyasını Yükle 'yi (fasta)tıklatın. FASTA dosyasını seçin ve Yükle 'yitıklatın.
- Blast 'ı tıklatın, NCBI Blast'ı seçin ve İleri 'yitıklatın. Parametreleri düzenleyin veya İleri'yi tıklatın, parametreleri düzenleyin ve en benzer gen açıklamasını bulmak için Çalıştır'ı tıklatın.
- Benzer proteinler için Gen Ontoloji ek açıklamalarını aramak için eşleme'yi tıklatın ve Çalıştır'ı tıklatın.
- Sonra interpro'yu tıklatın, EMBL-EBI InterPro'yu seçin ve İleri'yi tıklatın. Parametreleri düzenleyin veya İleri'yi tıklatın ve bilinen gen ailelerinin ve etki alanlarının imzalarını aramak için Çalıştır 'ı tıklatın.
- Dosya'yı tıklatarak ek açıklamaları dışarı aktar , Dışarı Aktar'ı seçin, Tabloyu Dışarı Aktar'ı tıklatın. Gözat 'ıtıklatın, dosyayı adlandırın, Kaydet 'itıklatın, Dışarı Aktar'ı tıklatın.
- Ek aday genlerini tanımlamak için GO ilgi koşulları için ek açıklama tablosunu arayın. Dizileri FASTA dosyasından ayıklama (ADIM 8.4.5)
9. Filogenetik ağaçlar
- MEGA30 v. 7.0.26'yı indirip yerel bilgisayarınıza yükleyin.
- MEGA'yı açın, Hizala 'yıtıklatın, Hizalamayı Düzenle/Oluştur 'utıklatın, Yeni hizalama oluştur 'u tıklatın Tamam 'ıtıklatın, Protein'i seçin.
- Hizalama penceresi açıldığında, Düzenle 'yetıklayın, Dosyadan dizi ekle'ye tıklayın ve aday genlerin protein dizileri ve olası homologlarla FASTA'yı seçin.
- Tüm dizileri seçin. Kol sembolünü bulun ve üzerine gelin. MUSCLE31 algoritmasını kullanarak dizileri hizala demelidir. Kol sembolüne tıklayın ve ardından dizileri hizalamak için Proteini Hizala'ya tıklayın. Parametreleri düzenleyin veya varsayılan parametreleri kullanarak hizalamak için Tamam'ı tıklatın.
- El ile yapılan değişiklikleri görsel olarak inceleyin ve yapın, ardından Hizalama penceresini kaydedin ve kapatın.
- Ana MEGA penceresinde, Modeller'e tıklayın, En İyi DNA / Protein modellerini bul 'a tıklayın (ML),hizalama dosyasını seçin ve aşağıdaki gibi ilgili parametreleri seçin: Analiz: Model Seçimi (ML), Kullanılacak Ağaç: Otomatik (komşu birleştirme ağacı), İstatistiksel Yöntem: Maksimum Olasılık, Değiştirme Türü: Amino Asit, Boşluk / eksik veri tedavisi: Tüm siteleri kullanın, Şube sitesi filtresi: Yok.
- Veriler için en iyi model belirlendikten sonra ana MEGA penceresine gidin. Filogeni'yi tıklatın ve Contruct/Test Maksimum Olasılık Ağacı'nı tıklatın ve gerekirse hizalamayı seçin. Ağaç için uygun parametreleri seçin: İstatistiksel yöntem: Maksimum Olasılık, Filogeni Testi: 100 çoğaltmalı Bootstrap yöntemi, ikame türü: amino asit, model: Freqs ile LG. (+F), siteler arasındaki oranlar: 5 ayrı gama kategorisine sahip gama dağıtılmış (G), boşluk/eksik veri işleme: tüm siteleri kullanın, ML sezgisel yöntem: En Yakın Komşu-Değişim (NNI).
10. TPM kullanarak gen ekspresyonlarını görselleştirin
- Trinity için, bilgisayar kümesinde abundance_estimates_to_matrix.pl çalıştırıldığı dizine gidin ve çıktılardan biri matris olmalıdır. TPM.not_cross_norm. Bu dosyayı yerel bilgisayarınıza aktarın.
NOT: Çapraz numune normalleştirmesi için Ek Malzemeler'e bakın. - Genom analizinden TPM'ler için aşağıdaki adımları izleyin.
- Bilgisayar kümesinde, RSEM yükleme konumuna gidin. scp rsem-generate-data-matrix rsem-generate-generate-TPM-matrix yazarak rsem-generate-data-matrix'ikopyalayın. Yeni dosyayı düzenlemek ve "$offsite = 4" ü TPM için 4'ten 5'e değiştirmek için nano'yu kullanın, şimdi "$offsite = 5" okumalıdır.
- RSEM çıktı dosyalarının bulunduğu dizine gidin .genes.results ve şimdi bir TPM matrisi oluşturmak için rsem-generate-TPM-matrix *[genes/isoforms.results] > $OUTPUT kullanın. Sonuçları yerel bir bilgisayara aktarın.
- Sonuçları ggplot2 ile görselleştirin.
- R v. 4.0.0 ve RStudio v. 1.2.1335'i yerel bir bilgisayara indirin.
- Ekranın sağındaki RStudio'yu açın Paketler sekmesine gidin ve Yükle 'yitıklatın. ggplot2 yazın ve yükle 'yitıklatın.
- TPM tablosunda veri<-read.table("$tpm.txt",üstbilgi = T) yazarak okunan R komut dosyası penceresinde
- Şekil 4'e benzer çubuk grafikler için aşağıdakine benzer bir şey yazın: p<- ggplot() + geom_bar(aes(y=TPM, x=Symbol, fill=Tissue), data=data, stat="identity")
fill<-c("#d7191c","#fdae61", "#ffffbf", "#abd9e9", "#2c7bb6")
p<-p+scale_fill_manual(değerler=dolgu)
p + tema(axis.text.x = element_text(açı = 90))
Sonuçlar
Yukarıdaki yöntemler Şekil 1'de özetlenmiş ve Hydra vulgaris dokularının bir veri kümesine uygulanmıştır. H. vulgaris, mercanlar, denizanaları ve deniz anemonlarını da içeren phylum Cnidaria'ya ait tatlı su omurgasızıdır. H. vulgaris tomurcuklanarak aseksüel olarak üreyebilir ve ikiye ayrıştıklarında başlarını ve ayaklarını yenileyebilirler. Bu çalışmada, Hydra7'deopsin genlerinin evrimini ve ekspresyonunu araştırmayı amaçladık. Hydra göz eksikliği yaparken, ışığa bağlı davranış sergilerler32. Opsin genleri, farklı ışık dalga boylarını tespit etmek ve fototransdüksiyon basamaklanmasına başlamak için görmede önemli olan proteinleri kodlar. Bu gen ailesinin bazal bir türdeki moleküler evrimini ve ekspresyonunun araştırılması, hayvanlarda gözlerin evrimi ve ışık tespiti hakkında fikir verebilir.
Hydra 2.033 referans genomunu ve kamuya açık RNA-seq verilerini (GEO katılım GSE127279) Şekil 1kullanarak rehberli bir montaj oluşturduk. Bu adım yaklaşık 3 gün sürdü. Bu durumda bir de novo transkriptom oluşturmamamıza rağmen, bir Trinity derlemesinin oluşturulması 1 hafta kadar sürebilir ve her kütüphane haritalayıcıya bağlı olarak okuma eşlemesi birkaç saat sürebilir. Birleştirilmiş Hydra montajı (~50.000 transkript) yaklaşık 1 haftalık Şekil 1alan Blast2GO kullanılarak açıklama eklenmiştir. Opsin ile ilişkili genler için diziler fasta dosyasına çıkarıldı. Diğer türlerden opsin genleri için diziler de NCBI GenBank'tan çıkarılmıştır. Biz cnidarians Podocoryna carneaopsins kullandık, Cladonema radiatum, Tripedelia cystophorave Nematostella vectensis, ve biz de outgroups Mnemiopsis leidyi, Trichoplax adhaerens, Drosophila melanogaster ve Homo sapiensdahil. Opsin genleri MEGA7 Şekil 2'dehizalanmıştır. Hizalamayı görüntüleyerek, ışığa duyarlı bir molekülü bağlamak için gerekli korunmuş bir lizin amino asidi eksik olan Hydra opsinlerini tanımlayabildik. Görsel incelemenin ardından model seçim analizi yaparak en iyi modeli belirledik. 100 Şekil 3önyükleme değerine sahip LG + G + F modelini kullanarak maksimum olasılıklı bir ağaç oluşturduk. 149 opsin geni için ağaç yaklaşık 3 günde bitti. Filogeni, opsin genlerinin cnidarians'ta soyuna özgü çoğaltmalarla ve potansiyel olarak H. vulgaris7'detandem çoğaltma ile evrimleştiğinigöstermektedir.
EdgeR'da diferansiyel ekspresyon analizi yaptık ve opsin genlerinin mutlak ekspresyona baktık. Bir veya daha fazla opsin'in kafada (hipostom) yukarı doğru düzene gireceğini ve hipostom ile vücut kolonu, tomurcuklanma bölgesi, ayak ve dokunaçların çift açısından karşılaştırmalarını gerçekleştireceğini varsayıyoruz. Çift açısından bir karşılaştırma örneği olarak, 1.774 transkript hipostom ve vücut sütunu arasında farklı olarak ifade edildi. Birden fazla karşılaştırmada güncellenmiş genleri belirledik ve Blast2GO Tablo 1'deişlevsel bir zenginleştirme yaptık. G-protein bağlantılı reseptör aktivitesinin gruplandırması opsin genlerini içeriyordu. Son olarak, opsin genlerinin farklı dokularda, tomurcuklanma sırasında ve yenilenme sırasında TPM değerlerini ggplot Şekil 4kullanarak çizerek mutlak ifadesine baktık. Burada özetlenen yöntemleri kullanarak filogenendeki diğer opsinlerle gruplaşmayan 2 opsin geni tanımladık, diğerlerinden neredeyse 200 kat daha fazla ifade edilen bir opsin bulduk ve ışık algılama için kullanılabilecek fototransdüksiyon genleriyle birlikte ifade edilen birkaç opsin geni bulduk.
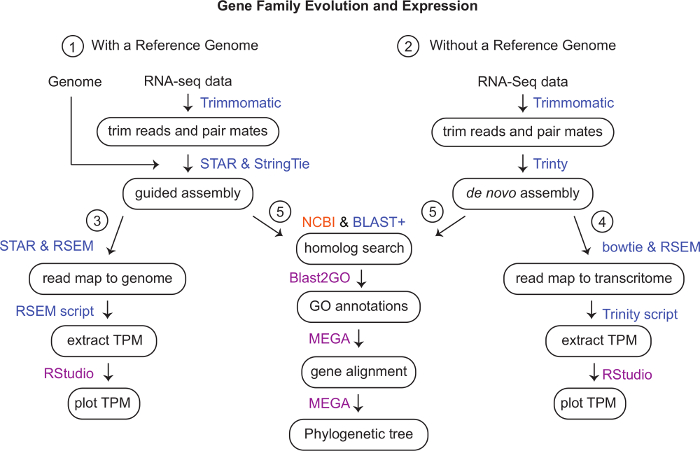
Şekil 1: İş akışı şeması. Bilgisayar kümesindeki verileri analiz etmek için kullanılan programlar mavi, macenta'da yerel bir bilgisayarda kullandığımız programlar ve turuncu web tabanlı bir programdır. (1) Trim RNA-seq okumaları trimmomatic v. 0.35 kullanarak. Bir genom varsa ancak gen modelleri eksikse, STAR v. 2.6.0c ve StringTie v. 1.3.4d kullanarak kılavuzlu bir montaj oluşturun. (İsteğe bağlı bkz: Ek Malzemeler) (2) Referans genom olmadan, Trinity v 2.8.5 kullanarak bir de novo montajı yapmak için kesilmiş okumalar kullanın. (3) Bir referans genom kullanarak gen ekspresyonunu ölçmek için, star kullanarak harita okur ve RSEM v. 1.3.1 kullanarak ölçün. RSEM kullanarak TPM'leri ayıklayın ve RStudio'da görselleştirin. (4) Bowtie ve RSEM, üçleme transkriptom ile eşlenen okumaları haritalamak ve ölçmek için kullanılabilir. Trinity komut dosyası, RStudio'daki sayımları görselleştirmek için bir TPM matrisi oluşturmak için kullanılabilir. (5) Homolog dizileri aramak ve karşılıklı BLAST kullanarak onaylamak için web tabanlı NCBI BLAST ve komut satırı BLAST+ kullanın. Blast2GO kullanarak genlere daha fazla açıklama ekleme. Genleri hizalamak ve en uygun modeli kullanarak filogenetik bir ağaç oluşturmak için MEGA kullanın. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
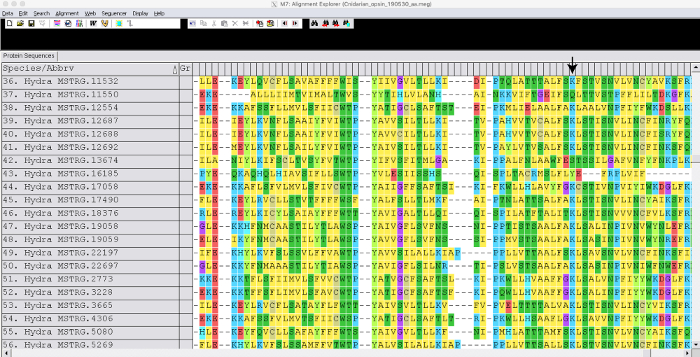
Şekil 2: Hizalanmış gen örneği. Anlık görüntü, HYDRA opsin genlerinin muscle kullanılarak hizalanmış bir kısmını gösterir. Ok, retina ciltli korunmuş bir lizinin yerini gösterir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
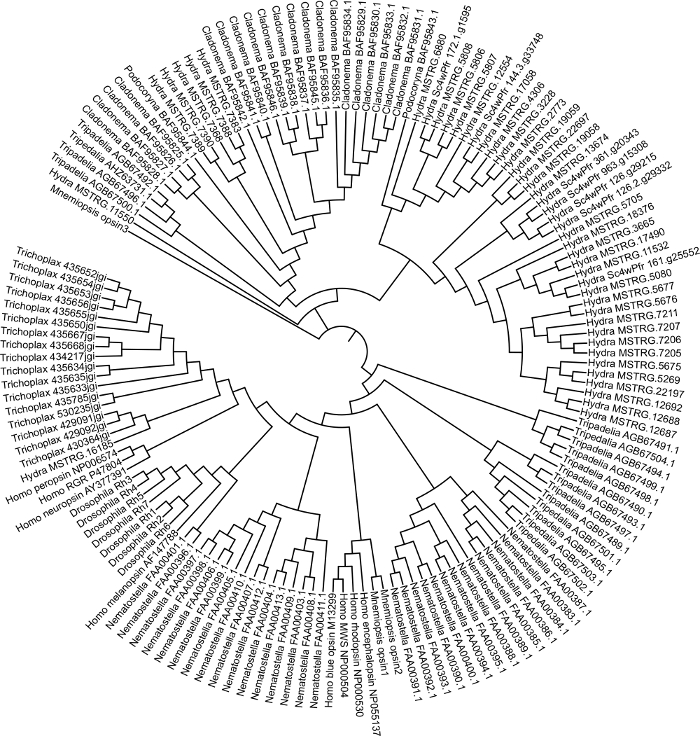
Şekil 3: Cnidarian opsin filogenetik ağaç. Hydra vulgaris, Podocoryna carnea, Cladonema radiatum, Tripedelia cystophora, Nematostella vectensis, Mnemiopsis leidyi, Trichoplax adhaerens , Drosophila melanogaster ve Homo sapiens'tenopsin dizileri kullanılarak MEGA7'de üretilen maksimum olasılıklı ağaç. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
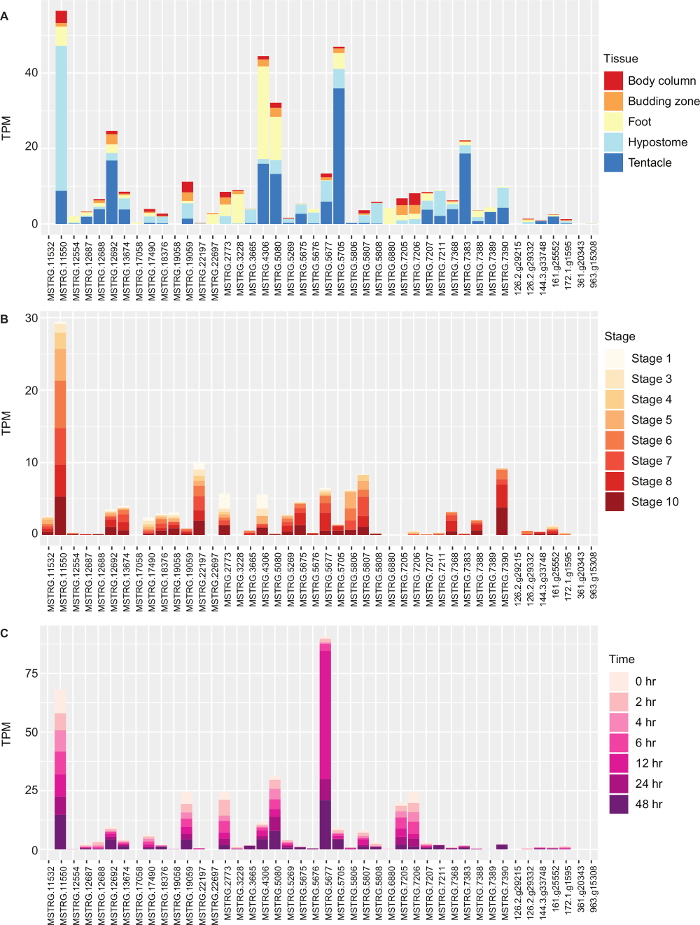
Şekil 4: Hydra vulgaris'teOpsin genlerinin ekspresyosu . (A) Vücut kolonunda Hydra vulgaris opsin genlerinin milyonda bir (TPM) transkriptinde, tomurcuklanma bölgesinde, ayakta, hipostomda ve dokunaçlarda ifade. (B) Hydra tomurcuklanmanın farklı aşamalarında opsin genlerinin ekspresyosu. (C) Farklı rejenerasyon zaman noktalarında Hydra hipostomunun opsin genlerinin ekspresyasyonu. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
| GO KIMLIĞI | GO Adı | GO Kategorisi | Fdr |
| GÖNK:0004930 | G-protein bağlantılı reseptör aktivitesi | MOLEKÜLER FONKSIYON | 0.0000000000704 |
| GÖNK:0007186 | G-protein bağlantılı reseptör sinyal yolu | BIYOLOJIK SÜREÇ | 0.00000000103 |
| GÖNK:0016055 | Wnt sinyal yolu | BIYOLOJIK SÜREÇ | 0.0000358 |
| GÖNK:0051260 | protein homooligomerizasyonu | BIYOLOJIK SÜREÇ | 0.000376 |
| GÖNK:0004222 | metalloendopeptidaz aktivitesi | MOLEKÜLER FONKSIYON | 0.000467 |
| GÖNK:0008076 | gerilim kapılı potasyum kanal kompleksi | HÜCRESEL BILEŞEN | 0.000642 |
| GÖNK:0005249 | gerilim kapılı potasyum kanalı aktivitesi | MOLEKÜLER FONKSIYON | 0.00213495 |
| GÖNK:0007275 | çok hücreli organizma gelişimi | BIYOLOJIK SÜREÇ | 0.00565048 |
| GÖNK:0006813 | potasyum iyon taşımacılığı | BIYOLOJIK SÜREÇ | 0.01228182 |
| GÖNYER:0018108 | peptidyl-tırozin fosforilasyonu | BIYOLOJIK SÜREÇ | 0.02679662 |
Tablo 1: Hipostomede yukarı doğru modüle edilen genlerin fonksiyonel zenginleştirilmesi
Ek Malzemeler. Bu materyalleri indirmek için lütfen tıklayınız.
Tartışmalar
Bu protokolün amacı, RNA-seq verilerini kullanarak bir gen ailesini karakterize etmek için adımların bir özetini sağlamaktır. Bu yöntemlerin çeşitli türler ve veri kümeleri4 , 34,35için çalıştığı kanıtlanmıştır. Burada kurulan boru hattı basitleştirildi ve biyoinformatikte bir acemi tarafından takip edilebilecek kadar kolay olmalıdır. Protokolün önemi, yayımlanabilir bir analizi tamamlamak için tüm adımları ve gerekli programları özetlemesidir. Protokoldeki önemli bir adım, tam uzunlukta transkriptlerin düzgün bir şekilde bir araya gelmesidir, bu yüksek kaliteli genomlardan veya transkriptomlardan gelir. Uygun transkriptleri elde etmek için, yüksek kaliteli RNA ve / veya DNA ve aşağıda tartışılan iyi ek açıklamalar gerekir.
RNA-seq kütüphane hazırlığı için, Hydra19'un küçük vücut parçaları ve kelebekler 18 (Malzeme Masası)için çalışan liste kitlerini dahil ediyoruz. Düşük giriş RNA'sı için değiştirilmiş bir protokol yaklaşımı kullandığımızı not ediyoruz36. RNA ekstraksiyonu yöntemleri maya hücreleri 17 , nöroblastom37, bitkiler38ve böcek larvaları16 dahil olmak üzere birden fazla örnek tipinde karşılaştırılmıştır. Okuyucunun, varsa ilgi alanları için çalışan bir protokol edinmesini veya başlamak için yaygın olarak piyasada bulunan kitleri kullanarak sorun gidermesini öneririz. Uygun gen nicelemesi için RNA örneğinin DNaz ile tedavi edilmesini öneririz. DNA'nın varlığı uygun gen nicelliğini etkileyecektir. Ayrıca, olgun mRNA için seçmek için polyA kuyruk seçimi içeren bir cDNA kütüphanesi hazırlık kiti kullanmanızı öneririz. rRNA tükenmesi daha fazla okuma derinliği ile sonuçlanırken, ekson kapsama yüzdesi polyA + seçimi39kullanılarak RNA'nın ekson kapsama alanından çok daha düşüktür. Son olarak, mümkün olduğunda eşleştirilmiş uçlu ve iplikli40,41kullanmak en iyisidir. Yukarıdaki iletişim kuralında, tek uç okumalar kullanırken okuma eşleme komutlarının değiştirilmesi gerekir.
Yukarıda belirtildiği gibi, ilgi genlerini tanımlayabilmek ve aynı zamanda son gen çoğaltmaları, alternatif birleştirme ve sıralamadaki haplotipler arasında ayrım yapabilmek önemlidir. Bazı durumlarda, bir referans genomuna sahip olmak, genlerin ve eksonların birbirine göre nerede bulunduğunu belirleyerek yardımcı olabilir. Dikkat edilmesi gereken bir şey, bir transkriptom genel bir veritabanından elde edilirse ve yüksek kalitede değilse, Trinity42'yi kullanarak ve RNA-seq kütüphanelerini ilgi çekici dokulardan birleştirerek oluşturmak en iyisi olabilir. Aynı şekilde, bir referans genomun iyi gen modelleri yoksa, RNA-seq kütüphaneleri StringTie43 kullanarak yeni GTF'ler oluşturmak için kullanılabilir (bkz. Ek Malzemeler). Ek olarak, genlerin eksik olduğu ve genoma erişimin olduğu durumlarda, genler homolog dizileri kullanılarak manuel olarak düzenlenebilir ve daha sonra tblastn kullanılarak genoma hizalanabilir. BLAST çıkışı, homologlar kullanılarak yapılan düzeltmeden farklı olabilecek gerçek sırayı belirlemek için kullanılabilir. Eşleşme yoksa, sırayı başlangıçta olduğu gibi bırakın. Çıktıyı kontrol ederken, eksik eksonun gerçekten genin bir parçası olduğundan emin olmak için genom koordinatlarına dikkat edin.
Kullandığımız yazılım ve programlara odaklanmamıza rağmen, farklı veri kümeleri için daha iyi çalışabilecek birçok program nedeniyle bu protokolde değişiklikler vardır. Örnek olarak, bowtie ve RSEM kullanarak transkriptome okumaları eşlemek için komutlar gösteriyoruz, ancak Trinity artık kallisto44 ve somon45gibi çok daha hızlı hizalayıcılar için seçeneğe sahip . Benzer şekilde, Blast2GO (şimdi OmicsBox) kullanarak ek açıklamalar açıklıyoruz, ancak ücretsiz ve çevrimiçi olarak bulunabilecek başka haritalayıcı araçları da var. Denediğimiz bazıları şunlardır: GO FEAT46, eggNOG-mapper47,48ve çok hızlı bir hizalayıcı PANNZER249. Bu web tabanlı ek açıklama araçlarını kullanmak için peptit FASTA'yı yükleyin ve gönderin. PANNZER ve eggNOG eşleyicinin bağımsız sürümleri de bilgisayar kümesine indirilebilir. Başka bir değişiklik, yerel bir bilgisayarda MEGA ve R'yi kullandık ve karşılıklı BLAST'ler yapmak için çevrimiçi NCBI BLAST aracını kullandık, ancak bu programların tümü gerekli programları ve veritabanlarını indirerek bilgisayar kümesinde kullanılabilir. Aynı şekilde, bir kullanıcı yeterli RAM ve depolama alanına sahip olduğu sürece yerel bir bilgisayarda kallisto ve somon hizalayıcıları kullanılabilir. Bununla birlikte, FASTQ ve FASTA dosyaları çok büyük olma eğilimindedir ve kolaylık ve hız için bir bilgisayar kümesi kullanmanızı şiddetle tavsiye ederiz. Buna ek olarak, geliştiricilerinden program indirmek için talimatlar ve bağlantılar sağlarken, birçoğu bioconda' dan yüklenebilir: https://anaconda.org/bioconda.
Biyoinformatik analizler yapılırken karşılaşılan yaygın bir sorun kabuk betiklerinin başarısız olmasıdır. Bunun nedeni çeşitli nedenler olabilir. Bir hata dosyası oluşturulursa, sorun gidermeden önce bu hata dosyası denetlenmelidir. Bir hatanın birkaç yaygın nedeni yazım hataları, eksik anahtar parametreleri ve yazılım sürümleri arasındaki uyumluluk sorunlarıdır. Bu protokolde, veriler için parametreler dahil ediyoruz, ancak yazılım kılavuzları bireysel parametreler için daha ayrıntılı yönergeler sağlayabilir. Genel olarak, yazılımın en güncel sürümlerini kullanmak ve bu sürüme karşılık gelen kılavuza danışmak en iyisidir.
Bu protokolde yapılan geliştirmeler arasında transkriptom çapında diferansiyel ekspresyon analizi ve fonksiyonel zenginleştirme analizi yapmak yer almaktadır. Biyoiletken'de bulunan bir paketi diferansiyel ifade analizi için edgeR50'yi öneririz. Fonksiyonel zenginleştirme analizi için Blast2GO29 ve web tabanlı DAVID51,52. Ayrıca, filogeniyi newick dosyası olarak ayıklayarak ve web tabanlı iTOL53kullanarak daha fazla düzenlemenizi öneririz. Ayrıca, bu protokol genlerin moleküler evrimini ve ifade kalıplarını araştırırken, gen veya protein konumlarını ve işlevlerini doğrulamak için ek deneyler kullanılabilir. mRNA ifadesi RT-qPCR veya in situ hibridizasyon ile doğrulanabilir. Proteinler immünostokimya kullanılarak lokalize edilebilir. Türlere bağlı olarak, gen fonksiyonunu doğrulamak için nakavt deneyleri kullanılabilir. Bu protokol, yukarıda gösterildiği gibi, tipik olarak bazal türlerde fotoreception ile ilişkili bir gen ailesini keşfetmek de dahil olmak üzere çeşitli amaçlar için kullanılabilir7. Bu yöntemlerin bir başka uygulaması, farklı seçici baskılar altında korunmuş bir yoldaki değişiklikleri tanımlamaktır. Örnek olarak, bu yöntemler diurnal kelebekler ve gece güveleri arasındaki görme geçici reseptör potansiyel kanallarının ifadesindeki varyasyonu keşfetmek için kullanılmıştır34.
Açıklamalar
Yazarların açıklayacak bir şeyi yok.
Teşekkürler
Adriana Briscoe, Gil Smith, Rabi Murad ve Aline G. Rangel'e bu adımlardan bazılarını iş akışımıza dahil etme konusunda tavsiye ve rehberlik için teşekkür ederiz. Katherine Williams, Elisabeth Rebboah ve Natasha Picciani'ye de el yazması hakkındaki yorumları için minnettarız. Bu çalışma kısmen George E. Hewitt Tıbbi araştırma vakfı tarafından A.M.M.'ye desteklendi.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Bioanalyzer-DNA kit | Agilent | 5067-4626 | wet lab materials |
| Bioanalyzer-RNA kit | Agilent | 5067-1513 | wet lab materials |
| BLAST+ v. 2.8.1 | On computer cluster* https://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/ | ||
| Blast2GO (on your PC) | On local computer https://www.blast2go.com/b2g-register-basic | ||
| boost v. 1.57.0 | On computer cluster | ||
| Bowtie v. 1.0.0 | On computer cluster https://sourceforge.net/projects/bowtie-bio/files/bowtie/1.3.0/ | ||
| Computing cluster (highly recommended) | NOTE: Analyses of genomic data are best done on a high-performance computing cluster because files are very large. | ||
| Cufflinks v. 2.2.1 | On computer cluster | ||
| edgeR v. 3.26.8 (in R) | In Rstudio https://bioconductor.org/packages/release/bioc/html/edgeR.html | ||
| gcc v. 6.4.0 | On computer cluster | ||
| Java v. 11.0.2 | On computer cluster | ||
| MEGA7 (on your PC) | On local computer https://www.megasoftware.net | ||
| MEGAX v. 0.1 | On local computer https://www.megasoftware.net | ||
| NucleoSpin RNA II kit | Macherey-Nagel | 740955.5 | wet lab materials |
| perl 5.30.3 | On computer cluster | ||
| python | On computer cluster | ||
| Qubit 2.0 Fluorometer | ThermoFisher | Q32866 | wet lab materials |
| R v.4.0.0 | On computer cluster https://cran.r-project.org/src/base/R-4/ | ||
| RNAlater | ThermoFisher | AM7021 | wet lab materials |
| RNeasy kit | Qiagen | 74104 | wet lab materials |
| RSEM v. 1.3.0 | Computer software https://deweylab.github.io/RSEM/ | ||
| RStudio v. 1.2.1335 | On local computer https://rstudio.com/products/rstudio/download/#download | ||
| Samtools v. 1.3 | Computer software | ||
| SRA Toolkit v. 2.8.1 | On computer cluster https://github.com/ncbi/sra-tools/wiki/01.-Downloading-SRA-Toolkit | ||
| STAR v. 2.6.0c | On computer cluster https://github.com/alexdobin/STAR | ||
| StringTie v. 1.3.4d | On computer cluster https://ccb.jhu.edu/software/stringtie/ | ||
| Transdecoder v. 5.5.0 | On computer cluster https://github.com/TransDecoder/TransDecoder/releases | ||
| Trimmomatic v. 0.35 | On computer cluster http://www.usadellab.org/cms/?page=trimmomatic | ||
| Trinity v.2.8.5 | On computer cluster https://github.com/trinityrnaseq/trinityrnaseq/releases | ||
| TRIzol | ThermoFisher | 15596018 | wet lab materials |
| TruSeq RNA Library Prep Kit v2 | Illumina | RS-122-2001 | wet lab materials |
| TURBO DNA-free Kit | ThermoFisher | AM1907 | wet lab materials |
| *Downloads and installation on the computer cluster may require root access. Contact your network administrator. |
Referanslar
- Lespinet, O., Wolf, Y. I., Koonin, E. V., Aravind, L. The role of lineage-specific gene family expansion in the evolution of eukaryotes. Genome Research. 12 (7), 1048-1059 (2002).
- Gabaldón, T., Koonin, E. V. Functional and evolutionary implications of gene orthology. Nature Reviews Genetics. 14 (5), 360-366 (2013).
- Dolinski, K., Botstein, D. Orthology and Functional Conservation in Eukaryotes. Annual Review of Genetics. 41 (1), (2007).
- Macias-Muñoz, A., McCulloch, K. J., Briscoe, A. D. Copy number variation and expression analysis reveals a non-orthologous pinta gene family member involved in butterfly vision. Genome Biology and Evolution. 9 (12), 3398-3412 (2017).
- Cannon, S. B., Mitra, A., Baumgarten, A., Young, N. D., May, G. The roles of segmental and tandem gene duplication in the evolution of large gene families in Arabidopsis thaliana. BMC plant biology. 4, 10(2004).
- Eastman, S. D., Chen, T. H. P., Falk, M. M., Mendelson, T. C., Iovine, M. K. Phylogenetic analysis of three complete gap junction gene families reveals lineage-specific duplications and highly supported gene classes. Genomics. 87 (2), 265-274 (2006).
- Macias-Munõz, A., Murad, R., Mortazavi, A. Molecular evolution and expression of opsin genes in Hydra vulgaris. BMC Genomics. 20 (1), 1-19 (2019).
- Hisatomi, O., Tokunaga, F. Molecular evolution of proteins involved in vertebrate phototransduction. Comparative Biochemistry and Physiology - B Biochemistry and Molecular Biology. 133 (4), 509-522 (2002).
- Arendt, D. Evolution of eyes and photoreceptor cell types. International Journal of Developmental Biology. 47, 563-571 (2003).
- Shichida, Y., Matsuyama, T. Evolution of opsins and phototransduction. Philosophical Transactions of the Royal Society B: Biological Sciences. 364 (1531), 2881-2895 (2009).
- Porter, M. L., et al. Shedding new light on opsin evolution. Proceedings of the Royal Society B: Biological Sciences. 279 (1726), 3-14 (2012).
- Plachetzki, D. C., Degnan, B. M., Oakley, T. H. The origins of novel protein interactions during animal opsin evolution. PLoS ONE. 2 (10), 1054(2007).
- Ramirez, M. D., et al. The last common ancestor of most bilaterian animals possessed at least nine opsins. Genome Biology and Evolution. 8 (12), 3640-3652 (2016).
- Schnitzler, C. E., et al. Genomic organization, evolution, and expression of photoprotein and opsin genes in Mnemiopsis leidyi: a new view of ctenophore photocytes. BMC Biology. 10, 107(2012).
- Pedersen, K. B., Williams, A., Watt, J., Ronis, M. J. Improved method for isolating high-quality RNA from mouse bone with RNAlater at room temperature. Bone Reports. 11, 100211(2019).
- Ridgeway, J. A., Timm, A. E., Fallon, A. Comparison of RNA isolation methods from insect larvae. Journal of Insect Science. 14 (1), 4-8 (2014).
- Scholes, A. N., Lewis, J. A. Comparison of RNA isolation methods on RNA-Seq: Implications for differential expression and meta-Analyses. BMC Genomics. 21 (1), 1-9 (2020).
- Briscoe, A. D., et al. Female behaviour drives expression and evolution of gustatory receptors in butterflies. PLoS genetics. 9 (7), 1003620(2013).
- Murad, R., Macias-Muñoz, A., Wong, A., Ma, X., Mortazavi, A. Integrative analysis of Hydra head regeneration reveals activation of distal enhancer-like elements. bioRxiv. , 544049(2019).
- Gallego Romero, I., Pai, A. A., Tung, J., Gilad, Y. Impact of RNA degradation on measurements of gene expression. BMC Biology. 12, 42(2014).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Trinity. RNA-Seq De novo Assembly Using Trinity. , 1-7 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29, 15-21 (2013).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC bioinformatics. 12, 323(2011).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome biology. 10, 25(2009).
- Camacho, C., et al. BLAST+: architecture and applications. BMC Bioinformatics. 10, 421(2009).
- Conesa, A., Götz, S. Blast2GO: A comprehensive suite for functional analysis in plant genomics. International Journal of Plant Genomics. 619832, (2008).
- Conesa, A., et al. Blast2GO: A universal tool for annotation, visualization and analysis in functional genomics research. Bioinformatics. 21 (18), 3674-3676 (2005).
- Götz, S., et al. High-throughput functional annotation and data mining with the Blast2GO suite. Nucleic Acids Research. 36 (10), 3420-3435 (2008).
- Kumar, S., Stecher, G., Tamura, K. MEGA7: Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets. Molecular biology and evolution. 33 (7), 1870-1874 (2016).
- Edgar, R. C. MUSCLE: Multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Research. 32 (5), 1792-1797 (2004).
- Taddei-Ferretti, C., Musio, C., Santillo, S., Cotugno, A. The photobiology of Hydra's periodic activity. Hydrobiologia. 530, 129-134 (2004).
- Chapman, J. A., et al. The dynamic genome of Hydra. Nature. 464 (7288), 592-596 (2010).
- Macias-Muñoz, A., Rangel Olguin, A. G., Briscoe, A. D. Evolution of phototransduction genes in Lepidoptera. Genome Biology and Evolution. 11 (8), 2107-2124 (2019).
- Macias-Munõz, A., Murad, R., Mortazavi, A. Molecular evolution and expression of opsin genes in Hydra vulgaris. BMC Genomics. 20 (1), (2019).
- Picelli, S., et al. Full-length RNA-seq from single cells using Smart-seq2. Nature Protocols. 9 (1), 171-181 (2014).
- Tavares, L., Alves, P. M., Ferreira, R. B., Santos, C. N. Comparison of different methods for DNA-free RNA isolation from SK-N-MC neuroblastoma. BMC research notes. 4, 3(2011).
- Johnson, M. T. J., et al. Evaluating Methods for Isolating Total RNA and Predicting the Success of Sequencing Phylogenetically Diverse Plant Transcriptomes. PLoS ONE. 7 (11), (2012).
- Zhao, S., Zhang, Y., Gamini, R., Zhang, B., Von Schack, D. Evaluation of two main RNA-seq approaches for gene quantification in clinical RNA sequencing: PolyA+ selection versus rRNA depletion. Scientific Reports. 8 (1), 1-12 (2018).
- Zhao, S., et al. Comparison of stranded and non-stranded RNA-seq transcriptome profiling and investigation of gene overlap. BMC Genomics. 16 (1), 1-14 (2015).
- Corley, S. M., MacKenzie, K. L., Beverdam, A., Roddam, L. F., Wilkins, M. R. Differentially expressed genes from RNA-Seq and functional enrichment results are affected by the choice of single-end versus paired-end reads and stranded versus non-stranded protocols. BMC Genomics. 18 (1), 1-13 (2017).
- Haas, B. J., et al. De novo transcript sequence reconstruction from RNA-seq using the Trinity platform for reference generation and analysis. Nature Protocols. 8 (8), 1494-1512 (2013).
- Pertea, M., et al. StringTie enables improved reconstruction of a transcriptome from RNA-seq reads. Nature biotechnology. 33 (3), 290-295 (2015).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nature Biotechnology. 34 (5), 525-527 (2016).
- Patro, R., Duggal, G., Love, M. I., Irizarry, R. A., Kingsford, C. Salmon provides fast and bias-aware quantification of transcript expression. Nature Methods. 14 (4), 417-419 (2017).
- Araujo, F. A., Barh, D., Silva, A., Guimarães, L., Thiago, R. OPEN GO FEAT a rapid web-based functional annotation tool for genomic and transcriptomic data. , 8-11 (2018).
- Huerta-Cepas, J., et al. Fast genome-wide functional annotation through orthology assignment by eggNOG-mapper. Molecular Biology and Evolution. 34 (8), 2115-2122 (2017).
- Huerta-Cepas, J., et al. EggNOG 5.0: A hierarchical, functionally and phylogenetically annotated orthology resource based on 5090 organisms and 2502 viruses. Nucleic Acids Research. 47, 309-314 (2019).
- Törönen, P., Medlar, A., Holm, L. PANNZER2: A rapid functional annotation web server. Nucleic Acids Research. 46, 84-88 (2018).
- Robinson, M., Mccarthy, D., Chen, Y., Smyth, G. K. edgeR differential expression analysis of digital gene expression data User's Guide. , (2013).
- Huang, D. W., Sherman, B. T., Lempicki, R. A. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources. Nature Protocols. 4 (1), 44-57 (2009).
- Huang, D. W., Sherman, B. T., Lempicki, R. A. Bioinformatics enrichment tools: Paths toward the comprehensive functional analysis of large gene lists. Nucleic Acids Research. 37 (1), 1-13 (2009).
- Letunic, I., Bork, P. Interactive tree of life (iTOL) v3: an online tool for the display and annotation of phylogenetic and other trees. Nucleic acids research. 44, 242-245 (2016).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır