Method Article
RNA-seqを用いた分子進化と遺伝子発現を探るバイオインフォマティクスのパイプライン
要約
このプロトコルの目的は、RNAシーケンシングデータを用いて候補遺伝子の進化と発現を調べるものです。
要約
全ゲノムやトランスクリプトームデータなどの大規模なデータセットを蒸留して報告することは、しばしば困難な作業です。結果を分解する1つの方法は、生物にとって重要な1つ以上の遺伝子ファミリーに焦点を当て、研究することです。このプロトコルでは、系統を生成し、目的の遺伝子の発現を定量化するためのバイオインフォマティクスステップの概要を説明する。系統樹は、遺伝子が種の内外でどのように進化しているかについての洞察を与えるだけでなく、鳥類学を明らかにすることができます。これらの結果は、RNA-seqデータを用いて、異なる個体または組織におけるこれらの遺伝子の発現を比較するために増強することができる。分子進化と発現の研究は、種間の遺伝子機能の進化と保存のモードを明らかにすることができます。遺伝子ファミリーの特徴付けは、将来の研究のスプリングボードとして機能し、新しいゲノムまたはトランスクリプトーム論文で重要な遺伝子ファミリーを強調することができます。
概要
シーケンシング技術の進歩は、非モデル生物のゲノムおよびトランスクリプトムのシーケンシングを促進してきた。多くの生物からのDNAとRNAのシーケンシングの実現可能性の増加に加えて、関心のある遺伝子を研究するために豊富なデータが一般に公開されています。このプロトコルの目的は、目的の生物に重要な役割を果たす可能性のある遺伝子の分子進化と発現を調査するためのバイオインフォマティクスのステップを提供することです。
遺伝子や遺伝子ファミリーの進化を調査することで、生物学的システムの進化に関する洞察を得ることができます。遺伝子ファミリーのメンバーは、通常、保存されたモチーフまたは相同遺伝子配列を同定することによって決定される。遺伝子ファミリーの進化は、以前は遠縁モデル生物1のゲノムを用いて調べられた。このアプローチの制限は、これらの遺伝子ファミリーが密接に関連する種でどのように進化し、異なる環境選択的圧力の役割が明確でないということです。このプロトコルでは、密接に関連する種のホモログの探索を含む。系統レベルで系統を生成することで、保存された遺伝子や系統特異的な重複などの遺伝子ファミリーの進化の傾向に注意することができます。このレベルでは、遺伝子がオルソログかパラログかを調べることもできます。多くのホモログは互いに同様に機能する可能性が高いが、必ずしも2の場合ではない。これらの研究に系統樹を組み込むことは、これらの相同遺伝子がオルソログであるかどうかを解決するために重要である。真核生物において、多くのオルソログは、哺乳類タンパク質が酵母オルソログ3の機能を回復する能力によって証明されるように、細胞内で同様の機能を保持している。しかし、非オーソリンス遺伝子が特徴付き機能4を行う場合があります。
系統樹は遺伝子と種の関係を引き起じ始めますが、遺伝的関係だけに基づいて機能を割り当てることはできません。遺伝子発現解析と機能性アノテーションと濃縮分析を組み合わせることで、遺伝子機能を強力にサポートします。遺伝子発現を定量化し、個人や組織の種類を比較できるケースは、潜在的な機能をより伝えることができます。以下のプロトコルは、Hydra下流7のオプシン遺伝子の調査に用いられる方法に従うが、それらはあらゆる種およびあらゆる遺伝子ファミリーに適用することができる。このような研究の結果は、非モデル生物における遺伝子機能および遺伝子ネットワークのさらなる調査のための基礎を提供する。一例として、光伝達カスケードを開始するタンパク質であるオプシンの系統の調査は、眼および光検出8、9、10、11の進化にコンテキストを与える。この場合、非モデル生物、特にクニダリアンやクテノフォアなどの基底動物種は、クレード12、13、14にわたる光変調カスケードおよび視力の保全または変化を解明することができる。同様に、他の遺伝子ファミリーの系統、発現、ネットワークを決定することは、適応の根底にある分子メカニズムについて私たちに知らせるでしょう。
プロトコル
このプロトコルは、UCアーバイン動物ケアガイドラインに従います。
1. RNA-セクライブラリ調製
- RNAを分離するには、以下の方法を用いた。
- サンプルを収集します。RNAを後で抽出する場合、フラッシュは、サンプルを凍結するか、RNAストレージ溶液15(材料の表)に置く。
- 目的の組織を分離するために生物を安楽死させ、解剖する。
- 抽出キットを用いて全RNAを抽出し、RNA精製キットを用いてRNAを精製する(材料表)
注:異なる種や組織の種類16、17のためにより良く働くプロトコルやキットがあります。我々は、バタフライ18とゼラチン性ヒドラ19の異なる身体組織からRNAを抽出した(議論参照)。 - 各サンプルのRNAの濃度と品質を測定する(材料表)。RNA 完全性番号 (RIN) が 8 より高いサンプルを使用し、理想的には 920 に近い cDNA ライブラリを構築します。
- cDNAライブラリとシーケンスを次のように構築します。
- ライブラリ準備の取扱説明書に従ってcDNAライブラリを構築します(説明を参照)。
- cDNAの濃度と品質を決定する(材料表)。
- ライブラリを多重化し、それらをシーケンスします。
2. コンピュータ クラスタへのアクセス
注: RNA-seq 解析は大きなファイルの操作を必要とし、コンピュータ クラスタ (材料表) で行うのが最善です。
- ターミナル (Mac) または PuTTY (Windows) アプリケーション ウィンドウで コマンド ssh username@clusterlocation を使用して、コンピューター クラスター アカウントにログインします。
3. RNA-seq 読み取りを取得する
- シーケンシング機能から RNA-seq を取得するか、またはパブリケーションで生成されたデータの場合は、それが保管されたデータ リポジトリ (3.2 または 3.3) から読み取ります。
- ArrayExpress などのリポジトリからデータをダウンロードするには、次の操作を行います。
- アクセス番号を使用してサイトを検索します。
- データをダウンロードするリンクを見つけ、左クリックして [ リンクのコピー] を選択します。
- ターミナル ウィンドウで 、wget と入力し、[ リンクを貼り付け] を選択して、データを分析用のディレクトリにコピーします。
- NCBI ショート読取りアーカイブ (SRA) データをダウンロードするには、次の代替手順に従います。
- ターミナルで wgetを使用して SRA ツールキット v. 2.8.1 をダウンロードします。
注 : プログラムをダウンロードしてコンピュータ クラスタにインストールするには、ルート アクセスが必要な場合があります。 - tar -xvf $TARGZFILEと入力してプログラムのインストールを完了します。
- NCBI でダウンロードするサンプルの SRA アクセス番号を検索すると、SRRXXXXXX という形式になります。
- RNA-seq データを取得するには、ターミナル ウィンドウに [sratoolkit 位置]/bin/prefetch SRRXXXXXX と入力します。
- ペアエンドファイルの場合は 、[sratoolkit location]/bin/fastq-dump --split-files SRRXXXXXX を使用して、2 つのファストQファイル(SRRXXXXXX_1.FASTQ と SRRXXXXXX_2.FASTQ)を取得します。
注:トリニティ ・デ・ノボ ・アセンブリを行うには 、コマンド[sratoolkitの場所]/ビン/ファストqダンプ --defline-seq '@$sn[_$rn]/$ri'--スプリットファイルSRRXXXXXXを使用してください
- ターミナルで wgetを使用して SRA ツールキット v. 2.8.1 をダウンロードします。
4.トリムアダプタと低品質の読み取り(オプション)
- コンピューティング クラスターに Trimmomatic21 v. 0.35 をインストールまたはロードします。
- RNA-seq データ ファイルが配置されているディレクトリに、トリミングされた jar ファイルの場所、入力 FASTQ ファイル、出力 FASTQ ファイル、および読み取り長さや品質などのオプションのパラメーターを含むコマンドを入力します。
注: コマンドは、読み取りの生の品質と長さによって異なります。Nexteraプライマーによるイルミナ43 bp読み取りについては 、Java -jar /データ/アプリ/トリミング/0.35/trimmomatic-0.35.jar PE $READ 1を使用しました。ファストク $READ 2.ファストクpaired_READ1。ファストクunpaired_READ1。ファストクpaired_READ2。ファストクunpaired_READ2。ファストク・イルミナクリップ:adapters.fa:2:30:10 先頭:20 トレーリング:20 スライディングウィンドウ:4:17 分:30分の10。
5. 参照アセンブリを取得する
- グーグル、エンセンブルゲノム、NCBIゲノムおよびヌクレオチドTSA(トランスクリプトームショットガンアセンブリ)を検索して、目的の種の参照ゲノムまたは組み立てられたトランスクリプトームを検索します(図1)。
注:参照ゲノムまたはトランスクリプトームが利用できないか、または低品質である場合は、ステップ6に進み、 デノボ アセンブリを生成します。 - 参照ゲノムまたは組み立てられたトランスクリプトームが存在する場合は、以下の手順に従って、高速ファイルとして、分析を実行する場所にダウンロードします。
- ゲノムをダウンロードするリンクを探し、左クリックして リンクをコピーします。
- ターミナルウィンドウで 、wget と入力し、リンクアドレスを貼り付けます。可能であれば、参照ゲノムのGTFファイルとプロテインFASTAファイルもコピーします。
6. デノボ アセンブリを生成する(ステップ5の代替)
- cat *READ1 と入力して、すべてのサンプルに対して RNA-seq READ1 ファイルと READ2 ファストQ ファイルを結合します 。ファストク > $all_READ1.ファストク と 猫 *READ2.ファストク> all_READ2。 ターミナル ウィンドウの FASTQ。
- トリニティ22 v.2.8.5 をコンピューティング クラスタにインストールまたはロードします。
- ターミナルに入力して生成し、アセンブリします: トリニティ --seqType fq --max_memory 20G --左$all_READ1。FASTQ --右$all_READ2。ファストク。
7. ゲノム(7.1)または デノボ トランスクリプトーム(7.2)への読み取りをマップする
- STAR23 v. 2.6.0c および RSEM24 v. 1.3.0 を使用して、参照ゲノムに読み取りをマップします。
- STAR v. 2.6.0c をインストールまたはロードします。そして、RSEM v. 1.3.0 をコンピューティング クラスタに対して行います。
- 「rsem-prepare-reference --gtf $GENOME」と入力してゲノムにインデックスを付けます。GTF --star -p 16 $GENOME。ファスタ$OUTPUT。
- 各サンプルの読み取りと計算を行う場合は 、rsem-calculate-expression -p 16 --star --paired end $READ 1 と入力します。ファストク $READ 2.ファストク$INDEX $OUTPUT。
- mv RSEM.genes.results $sample.genes.results.results.resultsを使用して、結果ファイルの名前をわかりやすい名前に変更します。
- rsem生成データマトリックス*[遺伝子/isoforms.results]> $OUTPUT入力して、すべてのカウントの行列を生成します。
- RSEMとボウタイを使用して、RNA-seqをトリニティ ・デ・ノボ ・アセンブリーにマッピングします。
- トリニティ22 v.2.8.5、ボウタイ25 v. 1.0.0、RSEM v. 1.3.0 をインストールまたはロードします。
- [trinity_location]/align_and_estimate_abundance.pl --prep-reference --transcripts $TRINITYと入力して、各サンプルの読み取りと計算 式をマップします。FASTA --seqType fq --左$READ 1。ファストク --右 $READ 2.FASTQ --est_method RSEM --aln_methodボウタイ --trinity_mode --output_dir $OUTPUT。
- mv RSEM.genes.results $sample.genes.results.results.resultsを使用して、結果ファイルの名前をわかりやすい名前に変更します。
- [trinity_location]/abundance_estimates_to_matrix.pl est_method --rsEM *[遺伝子/アイソフォーム]と入力して、すべてのカウントのマトリックスを生成します。
8. 目的の遺伝子を特定する
注:次の手順は、ヌクレオチドまたはタンパク質FASTAファイルで行うことができますが、最も良く動作し、タンパク質配列でより簡単です。タンパク質からタンパク質へのBLAST検索は、異なる種間を検索する際に結果を与える可能性が高い。
- 参照ゲノムの場合は、STEP 5.2.2のタンパク質FASTAファイルを使用するか、補足材料を参照してカスタム遺伝子特徴GTFを生成します。
- デノボトランスクリプトームの場合は、トランスデコーダーを使用してプロテインFASTAを生成します。
- コンピュータ cluser に TransDecoder v. 5.5.0 をインストールまたはロードします。
- 「トランスデコーダーの位置」/TransDecoder.LongOrfs -t $TRINITYと入力して、最も長いオープンリーディングフレームと予測ペプチドシーケンスを見つけます。ファスト。
- NCBIゲンバンクで密接に関連する種のホモログを検索します。
- インターネットブラウザウィンドウを開き、https://www.ncbi.nlm.nih.gov/genbank/ に移動します。
- 検索バーに、対象遺伝子の名前と、配列または属またはフィラムの密接に関連する種の名前を入力します。検索バーの左側でタンパク質を選択し、[検索]をクリックします。
- [送信]をクリックしてシーケンスを抽出し、[ファイル] を選択します。[フォーマット] で [FASTA] を選択し、[ファイルの作成] をクリックします。
- ローカルターミナルウィンドウで scp $FASTA username@clusterlocation:/$DIR と入力するか、FileZillaを使用してコンピュータとクラスタとの間でファイルを転送することにより、ホモログのFASTAファイルをコンピュータクラスタに移動します。
- BLAST+26を使用して候補遺伝子を検索します。
- BLAST+ v. 2.8.1 をコンピュータ クラスタにインストールまたはロードします。
- コンピュータクラスタでは、ゲノムまたはトランスクリプトーム翻訳タンパク質FASTAからBLASTデータベースを作成し 、[BLAST+ ロケーション]/makeblastdb -in $PEP入力します。FASTA -db 型のプロト -アウト $OUTPUT
- BLASTはNCBIから目的種のデータベースに対する相同遺伝子配列を [BLAST+場所]/blastp-db $DATABASE-query $FASTA -evalue 1e-10 -outfmt 6 -max_target_seqs 1 -out $OUTPUTと入力して目的の種のデータベースにする。
- 詳細コマンドを使用して出力ファイルを表示します。対象の種から新しいテキスト ファイルに、一意の遺伝子 ID をコピーします。
- perl -ne 'if(/^>(\S+)/){$c=$i{$1}}$c?print:chomp;$i{$_}=1'$gene_id.txt $PEPを@ARGVして、候補遺伝子の配列を抽出します。ファスタ> $OUTPUT。
- 相互BLASTを用いて遺伝子注釈を確認する。
- インターネットブラウザで https://blast.ncbi.nlm.nih.gov/Blast.cgi に行きます。
- tblastnを選択し、候補シーケンスを貼り付け、非冗長タンパク質配列データベースを選択してBLASTをクリックします。
- ゲノムまたはトランスクリプトーム内のすべての遺伝子に遺伝子オントロジー(GO)用語を付けて追加遺伝子を同定する(議論参照)。
- プロテイン FASTA をローカル コンピュータに転送します。
- Blast2GO27、28、29v. 5.2 をダウンロードしてローカル コンピュータにインストールします。
- Blast2GOを開き、[ファイル] をクリックし、[読み込み] に移動し、[シーケンスの読み込み]をクリックします。FASTA ファイルを選択し、[ロード] をクリックします。
- ブラストをクリックし、[NCBI ブラスト] を選択して 、[次へ] をクリックします。パラメータを編集するか、[次へ] をクリックしてパラメータを編集し、[実行] をクリックして最も類似した遺伝子の説明を見つけます。
- マッピングをクリックしてから[ 実行 ]をクリックして、類似したタンパク質の遺伝子オントロジーアノテーションを検索します。
- 次に [インタープロ] をクリックし 、[EMBL-EBI インタープロ] を選択して 、[ 次へ] をクリックします。パラメータを編集するか、[ 次へ] をクリックし、[ 実行 ] をクリックして、既知の遺伝子ファミリおよびドメインのシグネチャを検索します。
- [ ファイル] をクリックしてコメントを エクスポートし、[エクスポート] を選択して [ テーブルのエクスポート] をクリックします。[ 参照] をクリックし、ファイル名を 指定して [保存] をクリックし、[ エクスポート] をクリックします。
- 追加の候補遺伝子を特定するために、GO の対象用語の注釈テーブルを検索します。FASTA ファイルからシーケンスを抽出する (STEP 8.4.5)
9. 系統樹
- MEGA30 v. 7.0.26 をダウンロードしてローカルコンピュータにインストールします。
- MEGA を開き、[配置] をクリックし、[配置の編集/作成]をクリックします。
- アライメントウィンドウが開いたら、[ 編集]をクリックし、[ ファイルからシーケンスを挿入 ] をクリックして、候補遺伝子と可能性のあるホモログのタンパク質配列を含む FASTA を選択します。
- すべてのシーケンスを選択します。アームシンボルを見つけて、その上にカーソルを合わせます。それはMUSCLE31 アルゴリズムを使用して配列を整列と言うべきです。アーム記号をクリックし、「 タンパク質の位置合わせ 」をクリックして、配列を整列させます。パラメータを編集するか 、[OK]を クリックしてデフォルト パラメータを使用して整列します。
- 視覚的に検査し、手動で変更を加え、配置ウィンドウを保存して閉じます。
- メイン MEGA ウィンドウで、[ モデル] をクリックし、[ 最適な DNA/タンパク質モデルの検索 (ML)] をクリックし、アライメント ファイルを選択し、次のような対応するパラメーターを選択します: 分析: モデル選択 (ML)、使用するツリー: 自動 (隣接するツリー) 統計方法: 最も高い可能性, 置換タイプ: アミノ酸, ギャップ/欠損データ処理: すべてのサイトを使用, ブランチ サイト フィルター: なし.
- データに最適なモデルが決定されたら、メインの MEGA ウィンドウに移動します。 [Phylogeny] をクリックし、[ コントクト/テスト最大尤度ツリー ] をクリックし、必要に応じて配置を選択します。ツリーに適切なパラメータを選択: 統計法:最大尤度、系統のテスト:100複製のブートストラップ法、置換タイプ:アミノ酸、モデル:Freqsを使用するLG。(+F)、サイト間のレート:5つの離散ガンマガンマカテゴリを持つガンマ分布(G)、ギャップ/欠損データ処理:すべての部位を使用し、MLヒューリスティック法:近傍インターチェンジ(NNI)。
10. TPMを用いた遺伝子発現の可視化
- トリニティの場合、コンピュータクラスタ上で abundance_estimates_to_matrix.pl が実行されたディレクトリに移動し、出力の1つが行列である必要があります。TPM.not_cross_norm。このファイルをローカル コンピュータに転送します。
注: クロス サンプルの正規化については、補足資料を参照してください。 - ゲノム解析のTPMについては、以下の手順に従います。
- コンピュータ クラスタで、RSEM のインストール場所に移動します。scp rsem 生成データマトリックスを入力して、rsem 生成 データマトリックスをコピーします。 nano を使用して新しいファイルを編集し、TPMの場合は「私の$offsite = 4」を4から5に変更すると、「私の$offsite = 5」と表示されます。
- RSEM 出力ファイル .genes.results が存在するディレクトリに移動し 、rsem 生成 TPM-マトリックス *[genes/isoforms.results] > $OUTPUT を使用して TPM 行列を生成します。ローカル コンピューターに結果を転送します。
- ggplot2 で結果を視覚化します。
- R v. 4.0.0 および RStudio v. 1.2.1335 をローカル コンピューターにダウンロードします。
- 画面の右側にある RStudio を開いて [ パッケージ ] タブに移動し、[ インストール] をクリックします。 ggplot2 と入力し、[ インストール] をクリックします。
- データを入力して TPM テーブルに読み取られた R スクリプト ウィンドウで <-read.table("$tpm.txt"、ヘッダー = T)
- 図 4に似た棒グラフの場合、p<- ggplot() + geom_bar(aes(y=TPM、x=シンボル、フィル=ティッシュ)、データ=データ、stat="アイデンティティに似たものを入力します。
fill<-c("#d7191c","#fdae61"、"#ffffbf"、"#abd9e9"、"#2c7bb6")
p<-p+scale_fill_manual(値=塗りつぶし)
p + テーマ(軸.text.x = element_text(角度 = 90))
結果
上記の方法は 図1 に要約され、 ヒドラ下品 組織のデータセットに適用された。 H. 下垂体 は、サンゴ、クラゲ、イソギンチャクを含むフィラム クニダリア に属する淡水無脊椎動物です。 H.下品は 出芽によって無性を再現することができ、二分すると頭と足を再生することができます。本研究では、 ヒドラ7におけるオプチン遺伝子の進化と発現を調べる。 ヒドラ は目を欠いているが、光依存の行動32を示す。オプシン遺伝子は、光の異なる波長を検出し、光伝達カスケードを開始するために視力において重要なタンパク質をコードします。基底種におけるこの遺伝子ファミリーの分子進化と発現を調べれば、動物の目の進化と光検出の洞察を得ることができます。
Hydra 2.033参照ゲノムと一般に入手可能なRNA-seqデータ(GEOアチープションGSE127279)図1を用いてガイド付きアセンブリを生成した。このステップは約3日かかりました。この場合、デノボトランスクリプトームは生成しませんでしたが、トリニティアセンブリは生成に最大1週間かかる場合があり、各ライブラリはマッパーに応じて読み取りマッピングに数時間かかることがあります。合併したヒドラアセンブリ(約50,000のトランスクリプト)に、約1週間の図1を要したBlast2GOを使用して、アポイントトを作成しました。オプシン関連遺伝子の配列をファスタファイルに抽出した。他の種からのオプシン遺伝子の配列もNCBI GenBankから抽出された。クニダリアン・ポドコリーナ・カルネア、クラドネマ・ラディアタム、トリペデリア・シストフォラ、ネマトステラ・ヴェクテンシスのオプシンを使用し、ムネミオシス・レイディ、トリポプラクス・アデエレンス、ドロソフィラ・メラノガスター 、ホモ・サピエンスも含めました。オプシン遺伝子は、MEGA7図2で整列した。アライメントを見ることで、光感受性分子と結合するために必要な保存リジンアミノ酸が欠けているヒドラオプシンを同定することができた。目視検査の後、モデル選択解析を行うことで最適なモデルを決定しました。LG + G + F のモデルを使用して、ブートストラップ値が 100図 3の最尤ツリーを生成しました。149個のオプシン遺伝子について、木は約3日間で完成した。この系統は、オプチン遺伝子がクニダリアンにおける系統特異的複製によって進化し、H.下垂体7におけるタンデム複製によって進化していることを示唆している。
edgeRでの微分発現解析を行い、オプシン遺伝子の絶対発現を調べた。私たちは、1つ以上のオプシンが頭の中でアップレギュレートされると仮定し(ハイブトーム)、ボディカラム、出芽ゾーン、足、触手に対する仮説の対比を行った。対方向比較の一例として、1,774個の転写物が、ハイポトームとボディカラムの間で差異的に発現した。複数の比較でアップレギュレートされた遺伝子を特定し、Blast2GO 表1で機能的な濃縮を行いました。Gタンパク質結合受容体活性のグループ化には、オプシン遺伝子が含まれていた。最後に、ggplot 図4を用いてTPM値をプロットして、異なる組織におけるオプシン遺伝子の絶対発現を、出芽中および再生中に見た。ここで概説した方法を用いて、系統内の他のオプシンとグループ化していない2つのオプシン遺伝子を同定し、他のオプシンのほぼ200倍に発現したオプシンを発見し、光検出に使用できる光増倍遺伝子と共に発現するいくつかのオプチン遺伝子を発見した。
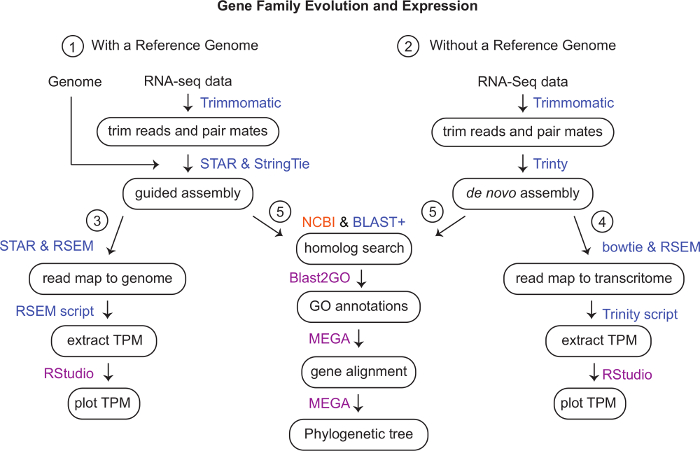
図 1: ワークフローの概略図コンピュータクラスタ上のデータを分析するために使用されるプログラムは青で、マゼンタはローカルコンピュータで使用したプログラムで、オレンジ色はウェブベースのプログラムです。(1) トリムモマティック v. 0.35 を使用して RNA-seq 読み取り値をトリミングします。ゲノムが利用可能で遺伝子モデルが欠落している場合は、STAR v. 2.6.0c および StringTie v. 1.3.4d を使用してガイド付きアセンブリを生成します。(オプションの補足資料を参照)(2) 参照ゲノムがない場合、トリニティ v 2.8.5 を使用して デノボ アセンブリを作成するには、トリミングされた読み取りを使用します。(3) 参照ゲノムを用いて遺伝子発現を定量化するために、マップはSTARを用いて読み取り、RSEM v. 1.3.1を用いて定量化する。RSEM を使用して TPM を抽出し、RStudio で視覚化します。(4) ボウタイとRSEMを使用して、三位一体のトランスクリプトームにマッピングされた読み取りをマッピングして定量化できます。トリニティ スクリプトを使用して、RStudio でカウントを視覚化する TPM マトリックスを生成できます。(5) ウェブベースの NCBI BLAST とコマンドライン BLAST+ を使用して、相同のシーケンスを検索し、相互BLASTを使用して確認します。Blast2GOを使用してさらに遺伝子にアトラセートする。MEGAを使用して遺伝子を整列させ、最も適したモデルを使用して系統樹を生成する。 この図の大きなバージョンを表示するには、ここをクリックしてください。
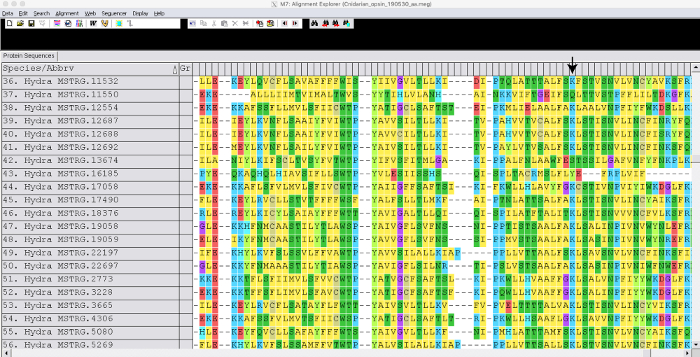
図2:整列遺伝子の例スナップショットは、MUSCLEを使用して整列 したヒドラ オプシン遺伝子の一部を示す。矢印は、レチン結合保存リジンの位置を示す。 この図の大きなバージョンを表示するには、ここをクリックしてください。
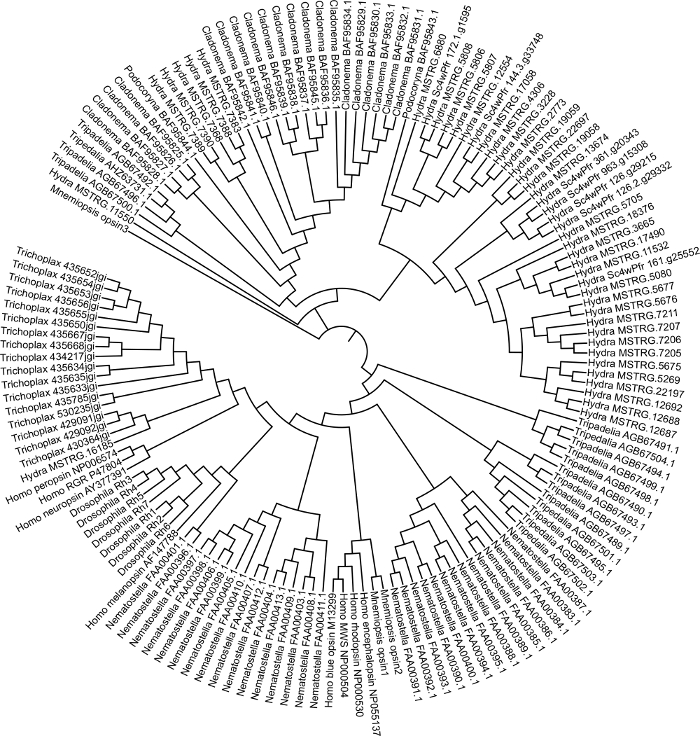
図3:クニダリアンオプシン系統樹.HYDRA下垂体、ポドコリーナカルネア、クラドネマラジウム、トリペデリアシストホラ、ネマトステラvectensis、ムネミオシス・レイディ、トリコプラクアデレンス、ドロソフィラメラノガスター 、ホモサピエンスのオプシン配列を使用してMEGA7で生成される最も可能性の高い木。この図の大きなバージョンを表示するには、ここをクリックしてください。
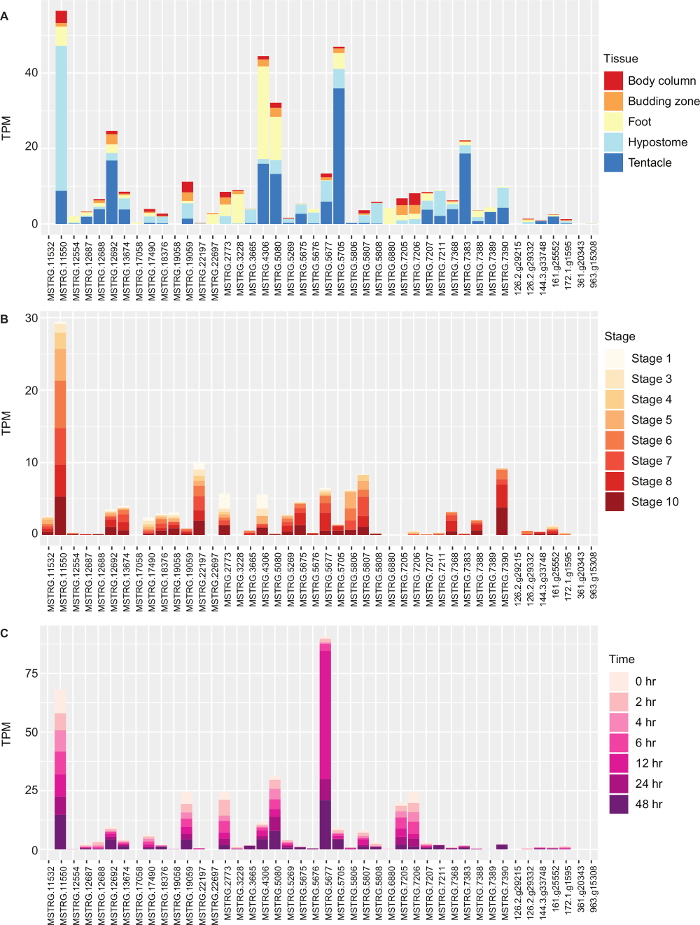
図4 ヒ ドラ下垂のオプシン遺伝子の発現(A)体内列、出芽領域、足、仮説および触手における ヒドラ下垂体 オプシン遺伝子の100万分の1(TPM)の転写物における発現。(B) ヒドラ 出芽の異なる段階におけるオプシン遺伝子の発現。(C)異なる時間の再生点における ヒドラ ハイストームのオプシン遺伝子の発現。 この図の大きなバージョンを表示するには、ここをクリックしてください。
| GO ID | GO名 | GOカテゴリ | FDR |
| 行く:0004930 | Gタンパク質共役受容体活性 | 分子機能 | 0.0000000000704 |
| 行く:0007186 | Gタンパク質共役受容体シグナル伝達経路 | 生物学的プロセス | 0.00000000103 |
| 行く:0016055 | Wnt シグナル経路 | 生物学的プロセス | 0.0000358 |
| 行く:0051260 | タンパク質ホモリゴマー化 | 生物学的プロセス | 0.000376 |
| 行く:0004222 | メタロエンドペプチダーゼ活性 | 分子機能 | 0.000467 |
| 行く:0008076 | 電圧ゲート付きカリウムチャネル複合体 | 細胞成分 | 0.000642 |
| 行く:0005249 | 電圧ゲート付きカリウムチャネル活性 | 分子機能 | 0.00213495 |
| 行く:0007275 | 多細胞生物の開発 | 生物学的プロセス | 0.00565048 |
| 行く:0006813 | カリウムイオン輸送 | 生物学的プロセス | 0.01228182 |
| 行く:0018108 | ペプチジル-チロシンリン酸化 | 生物学的プロセス | 0.02679662 |
表1:ハイポトームでアップレギュレートされた遺伝子の機能濃縮
ディスカッション
このプロトコルの目的は、RNA-seqデータを用いて遺伝子ファミリーを特徴付けるためのステップの概要を提供することにある。これらの方法は、さまざまな種やデータセット4、34、35に対して機能することが証明されています。ここで確立されたパイプラインは簡素化されており、バイオインフォマティクスの初心者が続くのに十分簡単でなければなりません。プロトコルの重要な点は、公開可能な分析を完了するために、すべての手順と必要なプログラムの概要を示すということです。プロトコルの重要なステップは、適切に完全な長さのトランスクリプトを組み立てることです, これは高品質のゲノムまたはトランスクリプトームから来ています.適切なトランスクリプトを得るためには、高品質のRNAおよび/またはDNAおよび以下で議論する良好な注釈が必要です。
RNA-seqライブラリの準備のために、我々は、ヒドラ19と蝶18の小さな体の部分のために働いたリストキットを含む(材料のテーブル)。我々は、低入力RNAのために、我々は、変更されたプロトコルアプローチ36を使用していることに注意してください。RNA抽出方法は、酵母細胞17、神経芽細胞腫37、植物38、昆虫幼虫16を含む複数のサンプルタイプで比較されてきた。読者は、興味のある種に適したプロトコルを取得するか、一般的に市販されているキットを使用してトラブルシューティングを開始することをお勧めします。適切な遺伝子定量を行うため、RNAサンプルをDNaseで処理することを推奨します。DNAの存在は、適切な遺伝子定量に影響を与えます。また、成熟したmRNAを選択するために、ポリAテールセレクションを含むcDNAライブラリの準備キットを使用することをお勧めします。rRNAの枯渇はより深い読み込みをもたらすが、エキソンカバレッジの割合は、ポリA+選択39を使用してRNAのエキソンカバレッジよりもはるかに低い。最後に、可能な場合は、ペアエンドと立ち往生40、41を使用するのが最善です。上記のプロトコルでは、シングルエンド読み取りを使用する場合、読み取りマッピングコマンドを変更する必要があります。
前述のように、関心のある遺伝子を同定できることと、シーケンシングにおける最近の遺伝子重複、代替スプライシング、ハプロタイプを区別できることが重要です。いくつかの例では、参照ゲノムを持つことは、遺伝子とエキソンが互いに相対的に位置する場所を決定することによって助けることができます。注意すべき点の1つは、写音が公的なデータベースから取得され、高品質でない場合、トリニティ42 を使用して、目的の組織からRNA-seqライブラリを組み合わせて生成するのが最善かもしれないということです。同様に、参照ゲノムに良好な遺伝子モデルがない場合、RNA-seq ライブラリを使用して StringTie43を使用して新しい GTF を生成できます (補足材料を参照)。また、遺伝子が不完全でゲノムにアクセスできる場合には、遺伝子をホモログ配列を使用して手動で編集し、tblastnを使用してゲノムに整列させることができます。BLAST出力を使用して実際のシーケンスを決定することができ、これはホモログを使用して行われる補正とは異なる場合があります。一致しない場合は、シーケンスを元のとおりにしておきます。出力をチェックするときは、欠けているエキソンが実際に遺伝子の一部であることを確認するために、ゲノム座標に注意を払ってください。
私たちは使用したソフトウェアとプログラムに焦点を当てていますが、このプロトコルの変更は、さまざまなデータセットに適した多くのプログラムが利用可能なため存在します。例として、ボウタイとRSEMを使用してトランスクリプトームに読み取りをマッピングするためのコマンドを表示しますが、トリニティは今、kallisto44やサーモン45のようなはるかに高速なアライナーのためのオプションを持っています。同様に、Blast2GO(現在のOmicsBox)を使用して注釈を記述していますが、無料でオンラインで見つけることができる他のマッパーツールがあります。私たちが試したいくつかは:GO FEAT46、エッグノグマッパー47、48、および非常に高速アライナーPANNZER249。これらのウェブベースの注釈ツールを使用するには、ペプチドFASTAをアップロードして提出するだけです。PANNZERとeggNOGマッパーのスタンドアロンバージョンもコンピュータクラスタにダウンロードできます。もう一つの変更は、我々はローカルコンピュータ上でMEGAとRを使用し、相互的なBLASTを行うためにオンラインNCBI BLASTツールを使用したが、これらのプログラムのすべてが必要なプログラムやデータベースをダウンロードすることによって、コンピュータクラスタ上で使用することができるということです。同様に、ユーザーが十分な RAM とストレージを持っている限り、アライナ カリストとサーモンをローカル コンピュータで使用できます。ただし、FASTQ ファイルと FASTA ファイルは非常に大きくなる傾向があり、コンピューター クラスタを使って簡単かつ高速に使用することを強くお勧めします。さらに、開発者からプログラムをダウンロードするための指示やリンクを提供していますが、その多くはbioconda:https://anaconda.org/bioconda からインストールすることができます。
バイオインフォマティクス解析を行う際に直面する一般的な問題は、シェルスクリプトの失敗です。これは、さまざまな理由が考えられます。エラー ファイルが作成された場合は、トラブルシューティングの前にこれらのエラー ファイルを確認する必要があります。エラーの一般的な原因としては、誤字、キー パラメータの不足、ソフトウェア バージョン間の互換性の問題があります。このプロトコルでは、データのパラメータを含めますが、ソフトウェアマニュアルでは個々のパラメータに関するより詳細なガイドラインを提供できます。一般的には、最新バージョンのソフトウェアを使用し、そのバージョンに対応するマニュアルを参照することをお勧めします。
このプロトコルの機能強化には、トランスクリプトーム全体の微分発現解析と機能濃縮解析が含まれます。微分発現解析には、バイオコンダクタで利用可能なパッケージをedgeR50に推奨します。機能拡張分析のために、Blast2GO29とウェブベースのDAVID51、52を使用しています。また、新しいファイルとして抽出し、WebベースのiTOL53を使用して、さらに系統を編集することをお勧めします。さらに、このプロトコルは遺伝子の分子進化と発現パターンを調査する一方で、遺伝子またはタンパク質の位置および機能を検証するために追加の実験を使用することができる。mRNA発現は、RT-qPCRまたはその際のハイブリダイゼーションで確認することができる。タンパク質は免疫細胞化学を用いて局在化することができる。種に応じて、ノックアウト実験を使用して遺伝子機能を確認することができます。このプロトコルは、上に示すように、基底種7におけるフォトレセプションに典型的に関連する遺伝子ファミリーを探索することを含む様々な目的に使用することができる。これらの方法の別のアプリケーションは、異なる選択的圧力下で保存された経路の変化を同定することです。一例として、これらの方法は、日経性蝶と夜行性蛾34との間の視覚過渡性受容体電位チャネルの発現の変動を発見するために用いた。
開示事項
著者らは開示するものは何もない。
謝辞
アドリアナ・ブリスコ、ギル・スミス、ラビ・ムラド、アリーン・G・レンゲルに、これらのステップのいくつかをワークフローに組み込む際のアドバイスとガイダンスに感謝します。また、キャサリン・ウィリアムズ、エリザベート・レボア、ナターシャ・ピッチャーニの原稿に対するコメントに感謝しています。この研究の一部は、ジョージ・E・ヒューイット医学研究フェローシップによって支援されました.M.M。
資料
| Name | Company | Catalog Number | Comments |
| Bioanalyzer-DNA kit | Agilent | 5067-4626 | wet lab materials |
| Bioanalyzer-RNA kit | Agilent | 5067-1513 | wet lab materials |
| BLAST+ v. 2.8.1 | On computer cluster* https://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/ | ||
| Blast2GO (on your PC) | On local computer https://www.blast2go.com/b2g-register-basic | ||
| boost v. 1.57.0 | On computer cluster | ||
| Bowtie v. 1.0.0 | On computer cluster https://sourceforge.net/projects/bowtie-bio/files/bowtie/1.3.0/ | ||
| Computing cluster (highly recommended) | NOTE: Analyses of genomic data are best done on a high-performance computing cluster because files are very large. | ||
| Cufflinks v. 2.2.1 | On computer cluster | ||
| edgeR v. 3.26.8 (in R) | In Rstudio https://bioconductor.org/packages/release/bioc/html/edgeR.html | ||
| gcc v. 6.4.0 | On computer cluster | ||
| Java v. 11.0.2 | On computer cluster | ||
| MEGA7 (on your PC) | On local computer https://www.megasoftware.net | ||
| MEGAX v. 0.1 | On local computer https://www.megasoftware.net | ||
| NucleoSpin RNA II kit | Macherey-Nagel | 740955.5 | wet lab materials |
| perl 5.30.3 | On computer cluster | ||
| python | On computer cluster | ||
| Qubit 2.0 Fluorometer | ThermoFisher | Q32866 | wet lab materials |
| R v.4.0.0 | On computer cluster https://cran.r-project.org/src/base/R-4/ | ||
| RNAlater | ThermoFisher | AM7021 | wet lab materials |
| RNeasy kit | Qiagen | 74104 | wet lab materials |
| RSEM v. 1.3.0 | Computer software https://deweylab.github.io/RSEM/ | ||
| RStudio v. 1.2.1335 | On local computer https://rstudio.com/products/rstudio/download/#download | ||
| Samtools v. 1.3 | Computer software | ||
| SRA Toolkit v. 2.8.1 | On computer cluster https://github.com/ncbi/sra-tools/wiki/01.-Downloading-SRA-Toolkit | ||
| STAR v. 2.6.0c | On computer cluster https://github.com/alexdobin/STAR | ||
| StringTie v. 1.3.4d | On computer cluster https://ccb.jhu.edu/software/stringtie/ | ||
| Transdecoder v. 5.5.0 | On computer cluster https://github.com/TransDecoder/TransDecoder/releases | ||
| Trimmomatic v. 0.35 | On computer cluster http://www.usadellab.org/cms/?page=trimmomatic | ||
| Trinity v.2.8.5 | On computer cluster https://github.com/trinityrnaseq/trinityrnaseq/releases | ||
| TRIzol | ThermoFisher | 15596018 | wet lab materials |
| TruSeq RNA Library Prep Kit v2 | Illumina | RS-122-2001 | wet lab materials |
| TURBO DNA-free Kit | ThermoFisher | AM1907 | wet lab materials |
| *Downloads and installation on the computer cluster may require root access. Contact your network administrator. |
参考文献
- Lespinet, O., Wolf, Y. I., Koonin, E. V., Aravind, L. The role of lineage-specific gene family expansion in the evolution of eukaryotes. Genome Research. 12 (7), 1048-1059 (2002).
- Gabaldón, T., Koonin, E. V. Functional and evolutionary implications of gene orthology. Nature Reviews Genetics. 14 (5), 360-366 (2013).
- Dolinski, K., Botstein, D. Orthology and Functional Conservation in Eukaryotes. Annual Review of Genetics. 41 (1), (2007).
- Macias-Muñoz, A., McCulloch, K. J., Briscoe, A. D. Copy number variation and expression analysis reveals a non-orthologous pinta gene family member involved in butterfly vision. Genome Biology and Evolution. 9 (12), 3398-3412 (2017).
- Cannon, S. B., Mitra, A., Baumgarten, A., Young, N. D., May, G. The roles of segmental and tandem gene duplication in the evolution of large gene families in Arabidopsis thaliana. BMC plant biology. 4, 10 (2004).
- Eastman, S. D., Chen, T. H. P., Falk, M. M., Mendelson, T. C., Iovine, M. K. Phylogenetic analysis of three complete gap junction gene families reveals lineage-specific duplications and highly supported gene classes. Genomics. 87 (2), 265-274 (2006).
- Macias-Munõz, A., Murad, R., Mortazavi, A. Molecular evolution and expression of opsin genes in Hydra vulgaris. BMC Genomics. 20 (1), 1-19 (2019).
- Hisatomi, O., Tokunaga, F. Molecular evolution of proteins involved in vertebrate phototransduction. Comparative Biochemistry and Physiology - B Biochemistry and Molecular Biology. 133 (4), 509-522 (2002).
- Arendt, D. Evolution of eyes and photoreceptor cell types. International Journal of Developmental Biology. 47, 563-571 (2003).
- Shichida, Y., Matsuyama, T. Evolution of opsins and phototransduction. Philosophical Transactions of the Royal Society B: Biological Sciences. 364 (1531), 2881-2895 (2009).
- Porter, M. L., et al. Shedding new light on opsin evolution. Proceedings of the Royal Society B: Biological Sciences. 279 (1726), 3-14 (2012).
- Plachetzki, D. C., Degnan, B. M., Oakley, T. H. The origins of novel protein interactions during animal opsin evolution. PLoS ONE. 2 (10), 1054 (2007).
- Ramirez, M. D., et al. The last common ancestor of most bilaterian animals possessed at least nine opsins. Genome Biology and Evolution. 8 (12), 3640-3652 (2016).
- Schnitzler, C. E., et al. Genomic organization, evolution, and expression of photoprotein and opsin genes in Mnemiopsis leidyi: a new view of ctenophore photocytes. BMC Biology. 10, 107 (2012).
- Pedersen, K. B., Williams, A., Watt, J., Ronis, M. J. Improved method for isolating high-quality RNA from mouse bone with RNAlater at room temperature. Bone Reports. 11, 100211 (2019).
- Ridgeway, J. A., Timm, A. E., Fallon, A. Comparison of RNA isolation methods from insect larvae. Journal of Insect Science. 14 (1), 4-8 (2014).
- Scholes, A. N., Lewis, J. A. Comparison of RNA isolation methods on RNA-Seq: Implications for differential expression and meta-Analyses. BMC Genomics. 21 (1), 1-9 (2020).
- Briscoe, A. D., et al. Female behaviour drives expression and evolution of gustatory receptors in butterflies. PLoS genetics. 9 (7), 1003620 (2013).
- Murad, R., Macias-Muñoz, A., Wong, A., Ma, X., Mortazavi, A. Integrative analysis of Hydra head regeneration reveals activation of distal enhancer-like elements. bioRxiv. , 544049 (2019).
- Gallego Romero, I., Pai, A. A., Tung, J., Gilad, Y. Impact of RNA degradation on measurements of gene expression. BMC Biology. 12, 42 (2014).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Trinity. . RNA-Seq De novo Assembly Using Trinity. , 1-7 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29, 15-21 (2013).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC bioinformatics. 12, 323 (2011).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome biology. 10, 25 (2009).
- Camacho, C., et al. BLAST+: architecture and applications. BMC Bioinformatics. 10, 421 (2009).
- Conesa, A., Götz, S. Blast2GO: A comprehensive suite for functional analysis in plant genomics. International Journal of Plant Genomics. 619832, (2008).
- Conesa, A., et al. Blast2GO: A universal tool for annotation, visualization and analysis in functional genomics research. Bioinformatics. 21 (18), 3674-3676 (2005).
- Götz, S., et al. High-throughput functional annotation and data mining with the Blast2GO suite. Nucleic Acids Research. 36 (10), 3420-3435 (2008).
- Kumar, S., Stecher, G., Tamura, K. MEGA7: Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets. Molecular biology and evolution. 33 (7), 1870-1874 (2016).
- Edgar, R. C. MUSCLE: Multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Research. 32 (5), 1792-1797 (2004).
- Taddei-Ferretti, C., Musio, C., Santillo, S., Cotugno, A. The photobiology of Hydra's periodic activity. Hydrobiologia. 530, 129-134 (2004).
- Chapman, J. A., et al. The dynamic genome of Hydra. Nature. 464 (7288), 592-596 (2010).
- Macias-Muñoz, A., Rangel Olguin, A. G., Briscoe, A. D. Evolution of phototransduction genes in Lepidoptera. Genome Biology and Evolution. 11 (8), 2107-2124 (2019).
- Macias-Munõz, A., Murad, R., Mortazavi, A. Molecular evolution and expression of opsin genes in Hydra vulgaris. BMC Genomics. 20 (1), (2019).
- Picelli, S., et al. Full-length RNA-seq from single cells using Smart-seq2. Nature Protocols. 9 (1), 171-181 (2014).
- Tavares, L., Alves, P. M., Ferreira, R. B., Santos, C. N. Comparison of different methods for DNA-free RNA isolation from SK-N-MC neuroblastoma. BMC research notes. 4, 3 (2011).
- Johnson, M. T. J., et al. Evaluating Methods for Isolating Total RNA and Predicting the Success of Sequencing Phylogenetically Diverse Plant Transcriptomes. PLoS ONE. 7 (11), (2012).
- Zhao, S., Zhang, Y., Gamini, R., Zhang, B., Von Schack, D. Evaluation of two main RNA-seq approaches for gene quantification in clinical RNA sequencing: PolyA+ selection versus rRNA depletion. Scientific Reports. 8 (1), 1-12 (2018).
- Zhao, S., et al. Comparison of stranded and non-stranded RNA-seq transcriptome profiling and investigation of gene overlap. BMC Genomics. 16 (1), 1-14 (2015).
- Corley, S. M., MacKenzie, K. L., Beverdam, A., Roddam, L. F., Wilkins, M. R. Differentially expressed genes from RNA-Seq and functional enrichment results are affected by the choice of single-end versus paired-end reads and stranded versus non-stranded protocols. BMC Genomics. 18 (1), 1-13 (2017).
- Haas, B. J., et al. De novo transcript sequence reconstruction from RNA-seq using the Trinity platform for reference generation and analysis. Nature Protocols. 8 (8), 1494-1512 (2013).
- Pertea, M., et al. StringTie enables improved reconstruction of a transcriptome from RNA-seq reads. Nature biotechnology. 33 (3), 290-295 (2015).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nature Biotechnology. 34 (5), 525-527 (2016).
- Patro, R., Duggal, G., Love, M. I., Irizarry, R. A., Kingsford, C. Salmon provides fast and bias-aware quantification of transcript expression. Nature Methods. 14 (4), 417-419 (2017).
- Araujo, F. A., Barh, D., Silva, A., Guimarães, L., Thiago, R. . OPEN GO FEAT a rapid web-based functional annotation tool for genomic and transcriptomic data. , 8-11 (2018).
- Huerta-Cepas, J., et al. Fast genome-wide functional annotation through orthology assignment by eggNOG-mapper. Molecular Biology and Evolution. 34 (8), 2115-2122 (2017).
- Huerta-Cepas, J., et al. EggNOG 5.0: A hierarchical, functionally and phylogenetically annotated orthology resource based on 5090 organisms and 2502 viruses. Nucleic Acids Research. 47, 309-314 (2019).
- Törönen, P., Medlar, A., Holm, L. PANNZER2: A rapid functional annotation web server. Nucleic Acids Research. 46, 84-88 (2018).
- Robinson, M., Mccarthy, D., Chen, Y., Smyth, G. K. . edgeR differential expression analysis of digital gene expression data User's Guide. , (2013).
- Huang, D. W., Sherman, B. T., Lempicki, R. A. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources. Nature Protocols. 4 (1), 44-57 (2009).
- Huang, D. W., Sherman, B. T., Lempicki, R. A. Bioinformatics enrichment tools: Paths toward the comprehensive functional analysis of large gene lists. Nucleic Acids Research. 37 (1), 1-13 (2009).
- Letunic, I., Bork, P. Interactive tree of life (iTOL) v3: an online tool for the display and annotation of phylogenetic and other trees. Nucleic acids research. 44, 242-245 (2016).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved