Method Article
Um Pipeline bioinforático para investigar a evolução molecular e a expressão genética usando RNA-seq
Neste Artigo
Resumo
O objetivo deste protocolo é investigar a evolução e expressão dos genes candidatos usando dados de sequenciamento de RNA.
Resumo
Destilar e relatar grandes conjuntos de dados, como dados de genoma inteiro ou transcriptome, é muitas vezes uma tarefa assustadora. Uma maneira de quebrar resultados é focar em uma ou mais famílias genéticas que são significativas para o organismo e estudar. Neste protocolo, delineamos etapas bioinformáticas para gerar uma filogenia e quantificar a expressão de genes de interesse. As árvores filogenéticas podem dar uma visão de como os genes estão evoluindo dentro e entre espécies, bem como revelar ortologia. Esses resultados podem ser aprimorados usando dados de RNA-seq para comparar a expressão desses genes em diferentes indivíduos ou tecidos. Estudos de evolução molecular e expressão podem revelar modos de evolução e conservação da função genética entre espécies. A caracterização de uma família genética pode servir de trampolim para estudos futuros e pode destacar uma importante família genética em um novo genoma ou papel transcriptome.
Introdução
Os avanços nas tecnologias de sequenciamento facilitaram o sequenciamento de genomas e transcriptomes de organismos não-modelos. Além da maior viabilidade do sequenciamento do DNA e do RNA de muitos organismos, uma abundância de dados está disponível publicamente para estudar genes de interesse. O objetivo deste protocolo é fornecer passos bioinforáticos para investigar a evolução molecular e a expressão de genes que possam desempenhar um papel importante no organismo de interesse.
Investigar a evolução de um gene ou gene familiar pode fornecer uma visão da evolução dos sistemas biológicos. Membros de uma família genética são tipicamente determinados pela identificação de motivos conservados ou sequências genéticas homólogos. A evolução da família genética foi previamente investigada usando genomas de organismos modelos distantes1. Uma limitação para essa abordagem é que não está claro como essas famílias genéticas evoluem em espécies intimamente relacionadas e o papel de diferentes pressões seletivas ambientais. Neste protocolo, incluímos uma busca por homólogos em espécies intimamente relacionadas. Ao gerar uma filogenia a um nível de filogênio, podemos notar tendências na evolução da família genética, como a de genes conservados ou duplicações específicas de linhagem. Neste nível, também podemos investigar se genes são ortologs ou paralogs. Embora muitos homólogos provavelmente funcionem de forma semelhante entre si, isso não é necessariamente o caso2. A incorporação de árvores filogenéticas nesses estudos é importante para resolver se esses genes homólogos são ortologs ou não. Nos eucariotes, muitos ortologos mantêm funções semelhantes dentro da célula, como evidenciado pela capacidade das proteínas mamíferas de restaurar a função dos ortologs de levedura3. No entanto, há casos em que um gene não ortologos realiza uma função caracterizada4.
As árvores filogenéticas começam a delinear relações entre genes e espécies, mas a função não pode ser atribuída apenas com base nas relações genéticas. Estudos de expressão genética combinados com anotações funcionais e análise de enriquecimento fornecem forte suporte para a função genética. Casos em que a expressão genética pode ser quantificada e comparada entre indivíduos ou tipos de tecidos podem ser mais reveladores da função potencial. O protocolo a seguir segue métodos usados na investigação de genes de opsina em Hydra vulgaris7, mas eles podem ser aplicados a qualquer espécie e qualquer família genética. Os resultados desses estudos fornecem uma base para uma investigação mais aprofundada sobre a função genética e redes genéticas em organismos não-modelo. Como exemplo, a investigação da filogenia das opsinas, que são proteínas que iniciam a cascata de fototransdução, dá contexto à evolução dos olhos e da detecção de luz8,9,10,11. Neste caso, organismos não-modelos, especialmente espécies de animais basais, como cnidários ou ctenoforos, podem elucidar a conservação ou alterações na cascata de fototransdução e na visão através de claes12,13,14. Da mesma forma, determinar a filogenia, expressão e redes de outras famílias genéticas nos informará sobre os mecanismos moleculares subjacentes às adaptações.
Protocolo
Este protocolo segue as diretrizes de cuidados com animais da UC Irvine.
1. Preparação da biblioteca RNA-seq
- Isole o RNA usando os seguintes métodos.
- Coletar amostras. Se o RNA for extraído posteriormente, congele a amostra ou coloque na solução de armazenamentoRNA 15 (Tabela de Materiais).
- Eutanize e disseque o organismo para separar tecidos de interesse.
- Extrair RNA total usando um kit de extração e purificar o RNA usando um kit de purificação de RNA(Tabela de Materiais)
NOTA: Existem protocolos e kits que podem funcionar melhor para diferentes espécies e tipos de tecidos16,17. Extraímos RNA de diferentes tecidos corporais de uma borboleta18 e uma Hidragelatinosa 19 (ver discussão). - Meça a concentração e a qualidade do RNA de cada amostra(Tabela de Materiais). Use amostras com números de integridade de RNA (RIN) superiores a 8, idealmente mais próximo de 920 para construir bibliotecas cDNA.
- Construa biblioteca cDNA e sequência da seguinte forma.
- Construa bibliotecas cDNA de acordo com o manual de instruções de preparação da biblioteca (ver discussão).
- Determinar concentração e qualidade do CDNA(Tabela de Materiais).
- Multiplex as bibliotecas e sequenciá-las.
2. Acesse um cluster de computador
NOTA: A análise do RNA-seq requer manipulação de arquivos grandes e é melhor feita em um cluster de computador(Tabela de Materiais).
- Faça login na conta de cluster do computador usando o username@clusterlocation de comando em uma janela de aplicativo terminal (Mac) ou PuTTY (Windows).
3. Obter leituras de RNA-seq
- Obtenha leituras de RNA-seq da instalação de sequenciamento ou, para dados gerados em uma publicação, do repositório de dados onde foi depositado (3.2 ou 3.3).
- Para baixar dados de repositórios como o ArrayExpress faça o seguinte:
- Pesquise no site usando o número de adesão.
- Encontre o link para baixar os dados e clique à esquerda e selecione Copiar link.
- Na janela do terminal, digite wget e selecione Colar link para copiar os dados no diretório para análise.
- Para baixar os dados do NCBI Short Read Archive (SRA) siga estas etapas alternativas:
- No terminal baixe SRA Toolkit v. 2.8.1 usando wget.
NOTA: Baixar e instalar programas no cluster do computador pode exigir acesso raiz, entrar em contato com o administrador do cluster do computador se a instalação falhar. - Termine de instalar o programa digitando tar -xvf $TARGZFILE.
- Pesquise ncbi para obter o número de adesão sra para as amostras que você deseja baixar, ele deve ter o formato SRRXXXXXX.
- Obtenha os dados do RNA-seq digitando [local de srtoolkit]/bin/prefetch SRRXXXXXX NA janela do terminal.
- Para o tipo de arquivos emparelhados [sedetoolkit localização]/bin/fastq-dump -split-files SRRXXXXXX para obter dois arquivos de fastq (SRRXXXXXX_1.FASTQ e SRRXXXXXX_2.FASTQ).
NOTA: Para fazer um conjunto Trinity de novo use o comando [sedetoolkit localização]/bin/fastq-dump --defline-seq '@$sn[_$rn]/$ri' -arquivos divididos SRRXXXXXX
- No terminal baixe SRA Toolkit v. 2.8.1 usando wget.
4. Aparar adaptadores e leituras de baixa qualidade (opcional)
- Instale ou carregue Trimmomatic21 v. 0.35 no cluster de computação.
- No diretório onde os arquivos de dados RNA-seq estão localizados, digite um comando que inclua a localização do arquivo de frasco trimmomatic, os arquivos FASTQ de entrada, arquivos FASTQ de saída e parâmetros opcionais, como comprimento de leitura e qualidade.
NOTA: O comando variará pela qualidade e comprimento brutos e desejados das leituras. Para leituras de Illumina 43 bp com primers Nextera, usamos: java -jar /data/apps/trimmomatic/0.35/trimmomatic-0.35.jar PE $READ 1. FASTQ $READ 2. paired_READ1 FASTQ. unpaired_READ1 FASTQ. paired_READ2 FASTQ. unpaired_READ2 FASTQ. FASTQ ILLUMINACLIP:adapters.fa:2:30:10 LEADING:20 TRAILING:20 SLIDINGWINDOW:4:17 MINLEN:30.
5. Obter montagem de referência
- Pesquise no Google, EnsemblGenomes e NCBI Genomas e Nucleotídeos TSA (Transcriptome Shotgun Assembly) para obter um genoma de referência ou transcriptome montado para as espécies de interesse(Figura 1).
NOTA: Se um genoma de referência ou transcriptome não estiver disponível ou de baixa qualidade, proceda ao PASSO 6 para gerar um conjunto de novo. - Se existir um genoma de referência ou transcriptome montado, baixe-o como um arquivo fasta para onde a análise será realizada seguindo as etapas abaixo.
- Encontre o link para baixar o genoma, clique à esquerda e Copy Link.
- Na janela do terminal digite wget e cole o endereço de link. Se disponível, copie também o arquivo GTF e o arquivo FASTA de proteína para o genoma de referência.
6. Gerar um conjunto de novo (Alternativa ao Passo 5)
- Combine os arquivos RNA-seq READ1 e READ2 fastq para todas as amostras digitando gato *READ1. FASTQ > $all_READ1. FASTQ e gato *READ2. > all_READ2 FASTQ. FASTQ na janela do terminal.
- Instale ou carregue o Trinity22 v.2.8.5 no cluster de computação.
- Gerar e montagem digitando no terminal: Trinity --seqType fq --max_memory 20G --esquerda $all_READ1. FASTQ --direita $all_READ2. O FASTQ.
7. Mapa lê para o genoma (7.1) ou de novo transcriptome (7.2)
- O mapa lê para o genoma de referência usando STAR23 v. 2.6.0c e RSEM24 v. 1.3.0.
- Instale ou carregue STAR v. 2.6.0c. e RSEM v. 1.3.0 para o cluster de computação.
- Indexe o genoma digitando rsem-prepare-referência --gtf $GENOME. GTF -estrela -p 16 $GENOME. $OUTPUT FASTA.
- Mapa lê e calcula a expressão para cada amostra digitando rsem-calculate-expression -p 16 --star --emparelhado-end $READ 1. FASTQ $READ 2. $INDEX $OUTPUT FASTQ.
- Renomeie o arquivo de resultados para algo descritivo usando mv RSEM.genes.results $sample.genes.results.
- Gerar uma matriz de todas as contagens digitando rsem-generate-data-matrix *[genes/isoforms.results] > $OUTPUT.
- Mapeie RNA-seq para o conjunto Trinity de novo usando RSEM e bowtie.
- Instale ou carregue Trinity22 v.2.8.5, Bowtie25 v. 1.0.0 e RSEM v. 1.3.0.
- Mapa lê e calcula a expressão para cada amostra digitando [trinity_location]/align_and_estimate_abundance.pl --prep-reference --transcrições $TRINITY. FASTA --seqType fq --esquerda $READ 1. FASTQ - direita $READ 2. FASTQ --est_method RSEM --aln_method bowtie --trinity_mode --output_dir $OUTPUT.
- Renomeie o arquivo de resultados para algo descritivo usando mv RSEM.genes.results $sample.genes.results.
- Gerar uma matriz de todas as contagens digitando [trinity_location]/abundance_estimates_to_matrix.pl --est_method RSEM *[genes/isoforms].resultados
8. Identificar genes de interesse
NOTA: As seguintes etapas podem ser feitas com arquivos nucleotídeos ou proteínas FASTA, mas funcionam melhor e são mais simples com sequências proteicas. Pesquisas de BLAST usando proteína para proteína são mais propensas a dar resultados na busca entre diferentes espécies.
- Para um genoma de referência, use o arquivo FASTA de proteína do STEP 5.2.2 ou consulte Materiais Suplementares para gerar um recurso genético personalizado GTF.
- Para um de novo transcriptome, gere uma proteína FASTA usando TransDecoder.
- Instale ou carregue TransDecoder v. 5.5.0 no cluser do computador.
- Encontre o quadro de leitura aberto mais longo e a sequência de peptídeos prevista digitando [transdecoder location]/TransDecoder.LongOrfs -t $TRINITY. O FASTA.
- Procure no NCBI Genbank por homólogos em espécies intimamente relacionadas.
- Abra uma janela do navegador de internet e vá para https://www.ncbi.nlm.nih.gov/genbank/.
- Na barra de pesquisa digitam o nome do gene de interesse e o nome de espécies intimamente relacionadas que foram sequenciadas ou gêneros ou filo. À esquerda da barra de pesquisa selecione proteína e clique em pesquisar.
- Extrair sequências clicando em Enviar e, em seguida, selecionar Arquivo. Em Formato, selecione FASTA e clique em Criar Arquivo.
- Mova o arquivo FASTA de homólogos para o cluster do computador digitando scp $FASTA username@clusterlocation:/$DIR em uma janela de terminal local ou use FileZilla para transferir arquivos de e para computador e cluster.
- Procure genes de candidatos usando BLAST+26.
- Instale ou carregue BLAST+ v. 2.8.1 no cluster do computador.
- No cluster do computador, faça um banco de dados BLAST a partir do genoma ou transcriptome proteína traduzida FASTA digitando [localização BLAST+]/makeblastdb -em $PEP. FASTA -dbtype prot -out $OUTPUT
- BLAST as sequências genéticas homólogos do NCBI para o banco de dados da espécie de interesse digitando [localização BLAST+]/blastp -db $DATABASE -query $FASTA -evalue 1e-10 -outfmt 6 -max_target_seqs 1 -out $OUTPUT.
- Exibir o arquivo de saída usando maiso comando . Copie iDs genéticos exclusivos da espécie de interesse para um novo arquivo de texto.
- Extrair as sequências de genes candidatos digitando perl -ne 'se(/^>(\S+)/){$c=$i{$1}}$c?print:chomp;$i{$_}=1 se @ARGV' $gene_id.txt $PEP. > $OUTPUT FASTA.
- Confirme a anotação genética usando BLAST recíproco.
- No navegador da internet vá para https://blast.ncbi.nlm.nih.gov/Blast.cgi.
- Selecione tblastn, em seguida, cole as sequências do candidato, selecione o banco de dados de sequência de proteínas não redundante e clique em BLAST.
- Identifique genes adicionais anotando todos os genes do genoma ou transcriptome com termos de ontologia genética (GO) (ver discussão).
- Transfira a proteína FASTA para o computador local.
- Baixe e instale o Blast2GO27,28,29 v. 5.2 para o computador local.
- Abra o Blast2GO, clique em Arquivo,vá para Carga,vá para Sequências de carga,clique em Load Fasta File (fasta). Selecione o arquivo FASTA e clique em Carregar.
- Clique em Blast, escolha NCBI Blaste clique em Next. Editar parâmetros ou clicar em Next, editar parâmetros e clicar em Executar para encontrar a descrição genética mais semelhante.
- Clique em mapear e clique em Executar para pesquisar anotações de Gene Ontology para proteínas semelhantes.
- Em seguida, clique em interpro, selecione EMBL-EBI InterProe clique em Next. Editar parâmetros ou clicar em Seguire clique em Executar para procurar assinaturas de famílias e domínios de genes conhecidos.
- Exporte as anotações clicando em Arquivo,selecione Exportar,clique em Tabela de Exportação. Clique em Procurar, nomeie o arquivo, clique em Salvar, clique em Exportar.
- Pesquise na tabela de anotação termos de interesse do GO para identificar genes adicionais de candidatos. Extrair as sequências do arquivo FASTA (STEP 8.4.5)
9. Árvores filogenéticas
- Baixe e instale MEGA30 v. 7.0.26 no seu computador local.
- Abra MEGA, clique em Alinhar,clique em Editar/Construir Alinhamento,selecione Criar um novo alinhamento clique em OK, selecione Proteína.
- Quando a janela de alinhamento for aberta, clique em Editar,clique em Inserir sequências do arquivo e selecione o FASTA com sequências proteicas de genes candidatos e prováveis homólogos.
- Selecione todas as sequências. Encontre o símbolo do braço e passe o mouse sobre ele. Deve-se dizer Alinhar sequências usando algoritmo MUSCLE31. Clique no símbolo do braço e clique em Alinhar proteína para alinhar as sequências. Editar parâmetros ou clicar em OK para alinhar usando parâmetros padrão.
- Inspecione visualmente e faça quaisquer alterações manuais e, em seguida, salve e feche a janela de alinhamento.
- Na janela PRINCIPAL MEGA, clique em Modelos,clique em Encontrar os melhores modelos de DNA/proteína (ML), selecione o arquivo de alinhamento e selecione parâmetros correspondentes como: Análise: Seleção de Modelo (ML), Árvore para usar: Automática (árvore de junção vizinha), Método Estatístico: Probabilidade Máxima, Tipo de Substituição: Aminoácido, Tratamento de dados Gap/ausente: Use todos os sites, Filtro do site do ramo: Nenhum.
- Uma vez determinado o melhor modelo para os dados, vá para a janela MEGA principal. Clique em Phylogeny e clique em Árvore de Máxima Probabilidade e selecione o alinhamento, se necessário. Selecione os parâmetros apropriados para a árvore: Método estatístico: Máxima Probabilidade, Teste de Filogenia: Método Bootstrap com 100 réplicas, tipo de substituição: aminoácido, modelo: LG com Freqs. (+F), taxas entre os locais: gama distribuída (G) com 5 categorias gama discretas, tratamento de dados gap/missing: use todos os locais, método heurístico ML: Near-Neighbor-Interchange (NNI).
10. Visualize a expressão genética usando TPM
- Para Trinity, no cluster do computador vá para o diretório onde abundance_estimates_to_matrix.pl foi executado e uma das saídas deve ser matriz. TPM.not_cross_norm. Transfira este arquivo para o seu computador local.
NOTA: Consulte Materiais Suplementares para normalização da amostra cruzada. - Para TPMs de uma análise de genoma siga os passos abaixo.
- No cluster do computador, vá para o local de instalação do RSEM. Copie a matriz rsem-generate-data,digitando scp rsem-generate-data-matrix rsem-generate-TPM-matrix. Use nano para editar o novo arquivo e alterar "meu $offsite = 4" de 4 para 5 para TPM, ele agora deve ler "meu $offsite = 5".
- Vá para o diretório onde os arquivos de saída RSEM .genes.results estão e agora use rsem-generate-TPM-matrix *[genes/isoforms.results] > $OUTPUT para gerar uma matriz TPM. Transferir resultados para um computador local.
- Visualize os resultados em ggplot2.
- Baixe R v. 4.0.0 e RStudio v. 1.2.1335 para um computador local.
- Abra rstudio à direita da tela vá para a guia Pacotes e clique em Instalar. Digite ggplot2 e clique em instalar.
- Na janela do script R lida na tabela TPM digitando dados<-read.table ("$tpm.txt", cabeçalho = T)
- Para gráficos de barra semelhantes à Figura 4 digitam algo semelhante a: p<-ggplot() + geom_bar(aes(y=TPM, x=Symbol, fill=Tissue), data=data, stat="identity")
preenchimento<-c("#d7191c", "#fdae61", "#ffffbf", "#abd9e9", "#2c7bb6")
p<-p+scale_fill_manual(valores=preenchimento)
p + tema (axis.text.x = element_text(ângulo = 90))
Resultados
Os métodos acima são resumidos na Figura 1 e foram aplicados a um conjunto de dados de tecidos hydra vulgaris. H. vulgaris é um invertebrado de água doce que pertence ao filo Cnidaria que também inclui corais, águas-vivas e anêmonas do mar. H. vulgaris pode se reproduzir assexualmente brotando e eles podem regenerar a cabeça e o pé quando bissecto. Neste estudo, buscou-se investigar a evolução e expressão dos genes opsin na Hydra7. Enquanto a Hydra não tem olhos, eles exibem comportamento dependente da luz32. Genes opsina codificam proteínas que são importantes na visão para detectar diferentes comprimentos de onda de luz e iniciar a cascata de fototransdução. Investigar a evolução molecular e a expressão desta família genética em uma espécie basal pode fornecer uma visão sobre a evolução dos olhos e a detecção de luz em animais.
Geramos um conjunto guiado utilizando o genoma de referência Hydra2.0 33 e dados RNA-seq disponíveis publicamente (GEO adesão GSE127279) Figura 1. Este passo levou aproximadamente 3 dias. Embora não tenhamos gerado um transcriptome de novo neste caso, uma montagem trinity pode levar até 1 semana para gerar e cada biblioteca pode levar algumas horas para ler mapeamento dependendo do mapeador. O conjunto Hydra mesclado (~50.000 transcrições) foi anotado usando blast2GO que levou cerca de 1 semana Figura 1. Sequências para genes relacionados com a opsina foram extraídas em um arquivo fasta. Sequências para genes de opsina de outras espécies também foram extraídas do NCBI GenBank. Usamos opsinas dos cnidarianos Podocoryna carnea, Cladonema radiatum, Tripedelia cystophorae Nematostella vectensis,e também incluímos os grupos Mnemiopsis leidyi, Trichoplax adhaerens, Drosophila melanogaster e Homo sapiens. Os genes opsin foram alinhados na Figura 2 MEGA7. Ao visualizar o alinhamento, conseguimos identificar as opsinas hydra que estavam faltando um aminoácido de lisina conservado necessário para ligar uma molécula sensível à luz. Após inspeção visual, determinamos o melhor modelo fazendo uma análise de seleção de modelos. Geramos uma árvore de máxima probabilidade usando o modelo LG + G + F com valor bootstrap de 100 Figura 3. Para 149 genes de opsina, a árvore foi concluída em aproximadamente 3 dias. A filogenia sugere que os genes de opsina estão evoluindo por duplicações específicas de linhagem em cnidários e potencialmente por duplicação em H. vulgaris7.
Realizamos uma análise de expressão diferencial no edgeR e olhamos para a expressão absoluta dos genes opsin. Nós imaginamos que uma ou mais opsinas seriam reguladas na cabeça (hipostome) e realizamos comparações par-wise de hipostome versus coluna corporal, zona de brotação, pé e tentáculos. Como exemplo de comparação par-wise, 1.774 transcrições foram expressas diferencialmente entre o hipostome e a coluna corporal. Determinamos os genes que foram regulados em várias comparações e fizemos um enriquecimento funcional na Tabela Blast2GO 1. O agrupamento da atividade receptora acoplado à proteína G incluiu genes de opsina. Finalmente, analisamos a expressão absoluta dos genes opsin em diferentes tecidos, durante a brotação e durante a regeneração, plotando seus valores TPM usando ggplot Figura 4. Usando os métodos aqui descritos, identificamos 2 genes de opsina que não se agrupavam com as outras opsinas na filogenia, encontramos uma opsina que foi expressa quase 200 vezes mais do que outras, e encontramos alguns genes opsin co-expressos com genes de fototransdução que podem ser usados para detecção de luz.
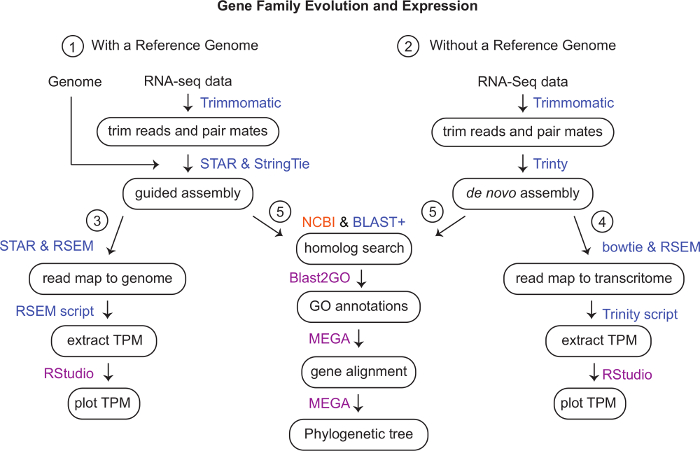
Figura 1: Esquema de fluxo de trabalho. Os programas usados para analisar dados no cluster do computador estão em azul, em magenta são aqueles que usamos em um computador local e em laranja é um programa baseado na Web. (1) Aparar as leituras de RNA-seq utilizando trimmomatic v. 0.35. Se um genoma estiver disponível, mas faltam modelos genéticos, gere um conjunto guiado usando STAR v. 2.6.0c e StringTie v. 1.3.4d. (Opcionais ver Materiais Suplementares) (2) Sem um genoma de referência, use leituras aparadas para fazer um novo conjunto usando Trinity v 2.8.5. (3) Para quantificar a expressão genética usando um genoma de referência, o mapa lê usando STAR e quantifica usando RSEM v. 1.3.1. Extrair TPMs usando RSEM e visualizá-los em RStudio. (4) Bowtie e RSEM podem ser usados para mapear e quantificar leituras mapeadas para um transcriptome de trindade. Um script Trinity pode ser usado para gerar uma matriz TPM para visualizar contagens no RStudio. (5) Use o NCBI BLAST baseado na Web e a linha de comando BLAST+ para procurar sequências homólogas e confirmar usando blast recíproco. Anote ainda mais os genes usando o Blast2GO. Use MEGA para alinhar genes e gerar uma árvore filogenética usando o modelo de melhor ajuste. Clique aqui para ver uma versão maior desta figura.
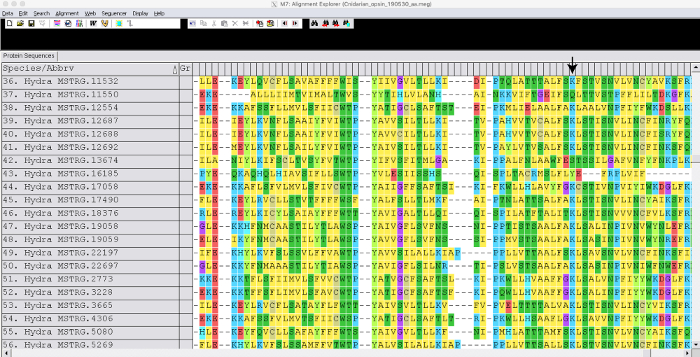
Figura 2: Exemplo de genes alinhados. Snapshot mostra uma porção de genes de operasina Hydra alinhados usando MUSCLE. A seta indica a localização de uma lise conservada de ligação de retina. Clique aqui para ver uma versão maior desta figura.
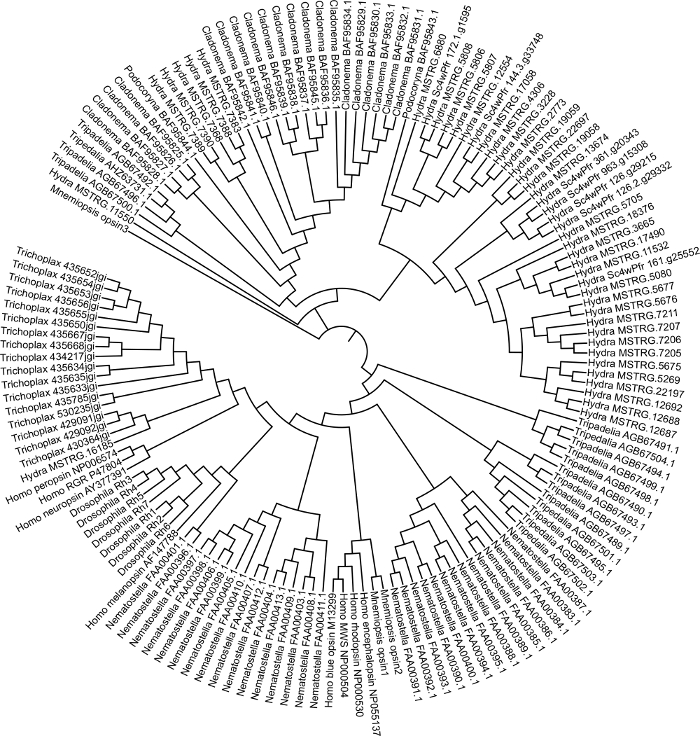
Figura 3: Árvore filogenética de opsina cnidária. Árvore de maior probabilidade gerada em MEGA7 usando sequências de opsina de Hydra vulgaris, Podocoryna carnea, Cladonema radiatum, Tripedelia cystophora, Nematostella vectensis, Mnemiopsis leidyi, Trichoplax adhaerens, Drosophila melanogaster e Homo sapiens. Clique aqui para ver uma versão maior desta figura.
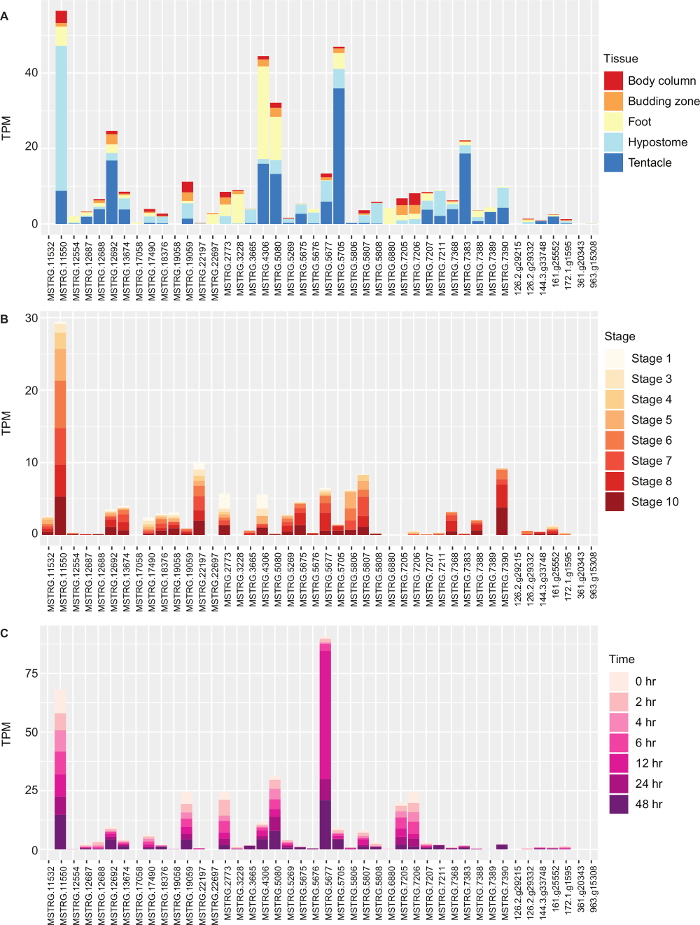
Figura 4: Expressão de genes opsinas em Hydra vulgaris. (A) Expressão em transcrições por milhão (TPM) de genes de hydra vulgaris opsin na coluna corporal, zona brotante, pé, hipostome e tentáculos. (B) Expressão de genes de opsina durante diferentes estágios de brotamento de Hydra. (C) Expressão de genes opsina do hipostome Hydra durante diferentes pontos de tempo de regeneração. Clique aqui para ver uma versão maior desta figura.
| GO ID | GO Name | Categoria GO | Fdr |
| 0004930 | Atividade do receptor acoplado à proteína G | FUNÇÃO MOLECULAR | 0.0000000000704 |
| 0007186 | Caminho de sinalização do receptor acoplado à proteína G | PROCESSO BIOLÓGICO | 0.00000000103 |
| 0016055 | Caminho de sinalização WNT | PROCESSO BIOLÓGICO | 0.0000358 |
| 0051260 | homooligomerização proteica | PROCESSO BIOLÓGICO | 0.000376 |
| 0004222 | atividade metalloendopeptidase | FUNÇÃO MOLECULAR | 0.000467 |
| 0008076 | complexo de canais de potássio fechados por tensão | COMPONENTE CELULAR | 0.000642 |
| 0005249 | atividade do canal de potássio fechado por tensão | FUNÇÃO MOLECULAR | 0.00213495 |
| 0007275 | desenvolvimento de organismos multicelulares | PROCESSO BIOLÓGICO | 0.00565048 |
| 0006813 | transporte de íons de potássio | PROCESSO BIOLÓGICO | 0.01228182 |
| 0018108 | fosforilação peptidyl-tyrosine | PROCESSO BIOLÓGICO | 0.02679662 |
Tabela 1: Enriquecimento funcional de genes regulados no hipostome
Materiais Suplementares. Clique aqui para baixar esses materiais.
Discussão
O objetivo deste protocolo é fornecer um esboço das etapas para caracterizar uma família genética usando dados RNA-seq. Estes métodos têm sido comprovados para funcionar para uma variedade de espécies e conjuntos de dados4,34,35. O gasoduto aqui estabelecido foi simplificado e deve ser fácil o suficiente para ser seguido por um novato em bioinformática. O significado do protocolo é que ele delineia todas as etapas e programas necessários para concluir uma análise publicável. Um passo crucial no protocolo é ter devidamente montado transcrições completas, isso vem de genomas de alta qualidade ou transcriptomes. Para obter transcrições adequadas, é preciso RNA de alta qualidade e/ou DNA e boas anotações discutidas abaixo.
Para a preparação da biblioteca RNA-seq, incluímos kits de lista que funcionaram para pequenas partes do corpo da Hydra19 e borboletas18 (Tabela de Materiais). Notamos que para baixo RNA de entrada usamos uma abordagem de protocolo modificada36. Os métodos de extração de RNA foram comparados em vários tipos de amostra, incluindo células de levedura17,neuroblastoma37,plantas38e larvasde insetos 16 para citar alguns. Recomendamos que o leitor adquira um protocolo que funcione para sua espécie de interesse, se houver, ou solucionar problemas usando kits comumente disponíveis comercialmente para começar. Para quantificação genética adequada, recomendamos tratar a amostra de RNA com DNase. A presença de DNA afetará a quantificação genética adequada. Também recomendamos o uso de um kit de preparação para biblioteca cDNA que inclua uma seleção de cauda polyA para selecionar para mRNA maduro. Enquanto o esgotamento do rRNA resulta em mais profundidade de leitura, a porcentagem de cobertura de exon é muito menor do que a cobertura de exon de RNA usando a seleção polyA+39. Finalmente, quando possível é melhor usar end-end emparelhado e encalhado40,41. No protocolo acima, os comandos de mapeamento de leitura terão de ser modificados ao usar leituras de extremidade única.
Como mencionado acima, é importante ser capaz de identificar genes de interesse e também diferenciar entre duplicações genéticas recentes, emendas alternativas e haplotipos no sequenciamento. Em alguns casos, ter um genoma de referência pode ajudar determinando onde genes e exons estão localizados em relação uns aos outros. Uma coisa a notar é que se um transcriptome é obtido de um banco de dados público e não é de alta qualidade, pode ser melhor gerar usando trinity42 e combinando bibliotecas RNA-seq de tecidos de interesse. Da mesma forma, se um genoma de referência não tiver bons modelos genéticos, bibliotecas RNA-seq podem ser usadas para gerar novos GTFs usando StringTie43 (ver Materiais Suplementares). Além disso, nos casos em que os genes estão incompletos e há acesso a um genoma, os genes podem ser editados manualmente usando sequências de homólogos e então alinhados ao genoma usando tblastn. A saída BLAST pode ser usada para determinar a sequência real, que pode ser diferente da correção feita usando homólogos. Se não houver correspondência, deixe a sequência como era originalmente. Ao verificar a saída preste atenção às coordenadas do genoma para ter certeza de que o exon desaparecido é realmente parte do gene.
Embora nos concentremos em softwares e programas que usamos, existem modificações neste protocolo devido a muitos programas disponíveis que podem funcionar melhor para diferentes conjuntos de dados. Como exemplo, mostramos comandos para mapeamento de leituras para o transcriptome usando bowtie e RSEM, mas Trinity agora tem a opção para alinhadores muito mais rápidos como kallisto44 e salmão45. Da mesma forma, descrevemos anotações usando Blast2GO (agora OmicsBox), mas existem outras ferramentas de mapper que podem ser encontradas gratuitas e online. Alguns que tentamos incluem: GO FEAT46, eggNOG-mapper47,48, e um alinhador muito rápido PANNZER249. Para usar essas ferramentas de anotação baseadas na Web, basta carregar o peptídeo FASTA e enviar. Versões autônomas de PANNZER e eggNOG-mapper também estão disponíveis para serem baixadas no cluster do computador. Outra modificação é que usamos MEGA e R em um computador local e usamos a ferramenta NCBI BLAST on-line para fazer BLASTs recíprocos, no entanto, todos esses programas podem ser usados no cluster de computador baixando os programas e bancos de dados necessários. Da mesma forma, os alinhadores kallisto e salmão podem ser usados em um computador local, desde que um usuário tenha RAM e armazenamento suficientes. No entanto, os arquivos FASTQ e FASTA tendem a ser muito grandes e recomendamos usar um cluster de computador para facilitar e acelerar. Além disso, enquanto fornecemos instruções e links para baixar programas de seus desenvolvedores, muitos deles podem ser instalados a partir de bioconda: https://anaconda.org/bioconda.
Um problema comum enfrentado ao fazer análises bioinformáticas é a falha dos scripts de shell. Isso pode ser devido a uma variedade de razões. Se um arquivo de erro for criado, esses arquivos de erro devem ser verificados antes da solução de problemas. Algumas razões comuns para um erro são erros de digitação, parâmetros-chave ausentes e problemas de compatibilidade entre versões de software. Neste protocolo, incluímos parâmetros para os dados, mas os manuais de software podem fornecer diretrizes mais detalhadas para parâmetros individuais. Em geral, é melhor usar as versões mais atualizadas do software e consultar o manual correspondente a essa versão.
Os aprimoramentos deste protocolo incluem a análise de expressão diferencial em todo o transcriptome e a análise de enriquecimento funcional. Recomendamos edgeR50 para análise de expressão diferencial um pacote disponível no Bioconductor. Para análise de enriquecimento funcional, utilizamos o Blast2GO29 e o DAVID51,52. Também recomendamos a edição adicional do filogenia, extraindo-o como um arquivo newick e usando iTOL53baseado na Web . Além disso, enquanto este protocolo investigará os padrões de evolução molecular e expressão dos genes, experimentos adicionais podem ser usados para validar locais e funções genéticas ou proteicas. a expressão mRNA pode ser confirmada por RT-qPCR ou pela hibridização in situ. As proteínas podem ser localizadas usando imunohistoquímica. Dependendo da espécie, experimentos de nocaute podem ser usados para confirmar a função genética. Este protocolo pode ser usado para uma variedade de objetivos, incluindo, como mostrado acima, explorar uma família genética tipicamente associada à fotorrecepção em uma espécie basal7. Outra aplicação desses métodos é identificar mudanças em um caminho conservado sob diferentes pressões seletivas. Como exemplo, esses métodos foram utilizados para descobrir variação na expressão de canais potenciais de receptores transitórios de visão entre borboletas diurnas e mariposas noturnas34.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Agradecemos a Adriana Briscoe, Gil Smith, Rabi Murad e Aline G. Rangel por conselhos e orientações na incorporação de algumas dessas etapas em nosso fluxo de trabalho. Também somos gratos a Katherine Williams, Elisabeth Rebboah e Natasha Picciani por comentários sobre o manuscrito. Este trabalho foi apoiado em parte por uma bolsa de pesquisa médica da Fundação George E. Hewitt para a A.M.M.
Materiais
| Name | Company | Catalog Number | Comments |
| Bioanalyzer-DNA kit | Agilent | 5067-4626 | wet lab materials |
| Bioanalyzer-RNA kit | Agilent | 5067-1513 | wet lab materials |
| BLAST+ v. 2.8.1 | On computer cluster* https://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/ | ||
| Blast2GO (on your PC) | On local computer https://www.blast2go.com/b2g-register-basic | ||
| boost v. 1.57.0 | On computer cluster | ||
| Bowtie v. 1.0.0 | On computer cluster https://sourceforge.net/projects/bowtie-bio/files/bowtie/1.3.0/ | ||
| Computing cluster (highly recommended) | NOTE: Analyses of genomic data are best done on a high-performance computing cluster because files are very large. | ||
| Cufflinks v. 2.2.1 | On computer cluster | ||
| edgeR v. 3.26.8 (in R) | In Rstudio https://bioconductor.org/packages/release/bioc/html/edgeR.html | ||
| gcc v. 6.4.0 | On computer cluster | ||
| Java v. 11.0.2 | On computer cluster | ||
| MEGA7 (on your PC) | On local computer https://www.megasoftware.net | ||
| MEGAX v. 0.1 | On local computer https://www.megasoftware.net | ||
| NucleoSpin RNA II kit | Macherey-Nagel | 740955.5 | wet lab materials |
| perl 5.30.3 | On computer cluster | ||
| python | On computer cluster | ||
| Qubit 2.0 Fluorometer | ThermoFisher | Q32866 | wet lab materials |
| R v.4.0.0 | On computer cluster https://cran.r-project.org/src/base/R-4/ | ||
| RNAlater | ThermoFisher | AM7021 | wet lab materials |
| RNeasy kit | Qiagen | 74104 | wet lab materials |
| RSEM v. 1.3.0 | Computer software https://deweylab.github.io/RSEM/ | ||
| RStudio v. 1.2.1335 | On local computer https://rstudio.com/products/rstudio/download/#download | ||
| Samtools v. 1.3 | Computer software | ||
| SRA Toolkit v. 2.8.1 | On computer cluster https://github.com/ncbi/sra-tools/wiki/01.-Downloading-SRA-Toolkit | ||
| STAR v. 2.6.0c | On computer cluster https://github.com/alexdobin/STAR | ||
| StringTie v. 1.3.4d | On computer cluster https://ccb.jhu.edu/software/stringtie/ | ||
| Transdecoder v. 5.5.0 | On computer cluster https://github.com/TransDecoder/TransDecoder/releases | ||
| Trimmomatic v. 0.35 | On computer cluster http://www.usadellab.org/cms/?page=trimmomatic | ||
| Trinity v.2.8.5 | On computer cluster https://github.com/trinityrnaseq/trinityrnaseq/releases | ||
| TRIzol | ThermoFisher | 15596018 | wet lab materials |
| TruSeq RNA Library Prep Kit v2 | Illumina | RS-122-2001 | wet lab materials |
| TURBO DNA-free Kit | ThermoFisher | AM1907 | wet lab materials |
| *Downloads and installation on the computer cluster may require root access. Contact your network administrator. |
Referências
- Lespinet, O., Wolf, Y. I., Koonin, E. V., Aravind, L. The role of lineage-specific gene family expansion in the evolution of eukaryotes. Genome Research. 12 (7), 1048-1059 (2002).
- Gabaldón, T., Koonin, E. V. Functional and evolutionary implications of gene orthology. Nature Reviews Genetics. 14 (5), 360-366 (2013).
- Dolinski, K., Botstein, D. Orthology and Functional Conservation in Eukaryotes. Annual Review of Genetics. 41 (1), (2007).
- Macias-Muñoz, A., McCulloch, K. J., Briscoe, A. D. Copy number variation and expression analysis reveals a non-orthologous pinta gene family member involved in butterfly vision. Genome Biology and Evolution. 9 (12), 3398-3412 (2017).
- Cannon, S. B., Mitra, A., Baumgarten, A., Young, N. D., May, G. The roles of segmental and tandem gene duplication in the evolution of large gene families in Arabidopsis thaliana. BMC plant biology. 4, 10 (2004).
- Eastman, S. D., Chen, T. H. P., Falk, M. M., Mendelson, T. C., Iovine, M. K. Phylogenetic analysis of three complete gap junction gene families reveals lineage-specific duplications and highly supported gene classes. Genomics. 87 (2), 265-274 (2006).
- Macias-Munõz, A., Murad, R., Mortazavi, A. Molecular evolution and expression of opsin genes in Hydra vulgaris. BMC Genomics. 20 (1), 1-19 (2019).
- Hisatomi, O., Tokunaga, F. Molecular evolution of proteins involved in vertebrate phototransduction. Comparative Biochemistry and Physiology - B Biochemistry and Molecular Biology. 133 (4), 509-522 (2002).
- Arendt, D. Evolution of eyes and photoreceptor cell types. International Journal of Developmental Biology. 47, 563-571 (2003).
- Shichida, Y., Matsuyama, T. Evolution of opsins and phototransduction. Philosophical Transactions of the Royal Society B: Biological Sciences. 364 (1531), 2881-2895 (2009).
- Porter, M. L., et al. Shedding new light on opsin evolution. Proceedings of the Royal Society B: Biological Sciences. 279 (1726), 3-14 (2012).
- Plachetzki, D. C., Degnan, B. M., Oakley, T. H. The origins of novel protein interactions during animal opsin evolution. PLoS ONE. 2 (10), 1054 (2007).
- Ramirez, M. D., et al. The last common ancestor of most bilaterian animals possessed at least nine opsins. Genome Biology and Evolution. 8 (12), 3640-3652 (2016).
- Schnitzler, C. E., et al. Genomic organization, evolution, and expression of photoprotein and opsin genes in Mnemiopsis leidyi: a new view of ctenophore photocytes. BMC Biology. 10, 107 (2012).
- Pedersen, K. B., Williams, A., Watt, J., Ronis, M. J. Improved method for isolating high-quality RNA from mouse bone with RNAlater at room temperature. Bone Reports. 11, 100211 (2019).
- Ridgeway, J. A., Timm, A. E., Fallon, A. Comparison of RNA isolation methods from insect larvae. Journal of Insect Science. 14 (1), 4-8 (2014).
- Scholes, A. N., Lewis, J. A. Comparison of RNA isolation methods on RNA-Seq: Implications for differential expression and meta-Analyses. BMC Genomics. 21 (1), 1-9 (2020).
- Briscoe, A. D., et al. Female behaviour drives expression and evolution of gustatory receptors in butterflies. PLoS genetics. 9 (7), 1003620 (2013).
- Murad, R., Macias-Muñoz, A., Wong, A., Ma, X., Mortazavi, A. Integrative analysis of Hydra head regeneration reveals activation of distal enhancer-like elements. bioRxiv. , 544049 (2019).
- Gallego Romero, I., Pai, A. A., Tung, J., Gilad, Y. Impact of RNA degradation on measurements of gene expression. BMC Biology. 12, 42 (2014).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Trinity. . RNA-Seq De novo Assembly Using Trinity. , 1-7 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29, 15-21 (2013).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC bioinformatics. 12, 323 (2011).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome biology. 10, 25 (2009).
- Camacho, C., et al. BLAST+: architecture and applications. BMC Bioinformatics. 10, 421 (2009).
- Conesa, A., Götz, S. Blast2GO: A comprehensive suite for functional analysis in plant genomics. International Journal of Plant Genomics. 619832, (2008).
- Conesa, A., et al. Blast2GO: A universal tool for annotation, visualization and analysis in functional genomics research. Bioinformatics. 21 (18), 3674-3676 (2005).
- Götz, S., et al. High-throughput functional annotation and data mining with the Blast2GO suite. Nucleic Acids Research. 36 (10), 3420-3435 (2008).
- Kumar, S., Stecher, G., Tamura, K. MEGA7: Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets. Molecular biology and evolution. 33 (7), 1870-1874 (2016).
- Edgar, R. C. MUSCLE: Multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Research. 32 (5), 1792-1797 (2004).
- Taddei-Ferretti, C., Musio, C., Santillo, S., Cotugno, A. The photobiology of Hydra's periodic activity. Hydrobiologia. 530, 129-134 (2004).
- Chapman, J. A., et al. The dynamic genome of Hydra. Nature. 464 (7288), 592-596 (2010).
- Macias-Muñoz, A., Rangel Olguin, A. G., Briscoe, A. D. Evolution of phototransduction genes in Lepidoptera. Genome Biology and Evolution. 11 (8), 2107-2124 (2019).
- Macias-Munõz, A., Murad, R., Mortazavi, A. Molecular evolution and expression of opsin genes in Hydra vulgaris. BMC Genomics. 20 (1), (2019).
- Picelli, S., et al. Full-length RNA-seq from single cells using Smart-seq2. Nature Protocols. 9 (1), 171-181 (2014).
- Tavares, L., Alves, P. M., Ferreira, R. B., Santos, C. N. Comparison of different methods for DNA-free RNA isolation from SK-N-MC neuroblastoma. BMC research notes. 4, 3 (2011).
- Johnson, M. T. J., et al. Evaluating Methods for Isolating Total RNA and Predicting the Success of Sequencing Phylogenetically Diverse Plant Transcriptomes. PLoS ONE. 7 (11), (2012).
- Zhao, S., Zhang, Y., Gamini, R., Zhang, B., Von Schack, D. Evaluation of two main RNA-seq approaches for gene quantification in clinical RNA sequencing: PolyA+ selection versus rRNA depletion. Scientific Reports. 8 (1), 1-12 (2018).
- Zhao, S., et al. Comparison of stranded and non-stranded RNA-seq transcriptome profiling and investigation of gene overlap. BMC Genomics. 16 (1), 1-14 (2015).
- Corley, S. M., MacKenzie, K. L., Beverdam, A., Roddam, L. F., Wilkins, M. R. Differentially expressed genes from RNA-Seq and functional enrichment results are affected by the choice of single-end versus paired-end reads and stranded versus non-stranded protocols. BMC Genomics. 18 (1), 1-13 (2017).
- Haas, B. J., et al. De novo transcript sequence reconstruction from RNA-seq using the Trinity platform for reference generation and analysis. Nature Protocols. 8 (8), 1494-1512 (2013).
- Pertea, M., et al. StringTie enables improved reconstruction of a transcriptome from RNA-seq reads. Nature biotechnology. 33 (3), 290-295 (2015).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nature Biotechnology. 34 (5), 525-527 (2016).
- Patro, R., Duggal, G., Love, M. I., Irizarry, R. A., Kingsford, C. Salmon provides fast and bias-aware quantification of transcript expression. Nature Methods. 14 (4), 417-419 (2017).
- Araujo, F. A., Barh, D., Silva, A., Guimarães, L., Thiago, R. . OPEN GO FEAT a rapid web-based functional annotation tool for genomic and transcriptomic data. , 8-11 (2018).
- Huerta-Cepas, J., et al. Fast genome-wide functional annotation through orthology assignment by eggNOG-mapper. Molecular Biology and Evolution. 34 (8), 2115-2122 (2017).
- Huerta-Cepas, J., et al. EggNOG 5.0: A hierarchical, functionally and phylogenetically annotated orthology resource based on 5090 organisms and 2502 viruses. Nucleic Acids Research. 47, 309-314 (2019).
- Törönen, P., Medlar, A., Holm, L. PANNZER2: A rapid functional annotation web server. Nucleic Acids Research. 46, 84-88 (2018).
- Robinson, M., Mccarthy, D., Chen, Y., Smyth, G. K. . edgeR differential expression analysis of digital gene expression data User's Guide. , (2013).
- Huang, D. W., Sherman, B. T., Lempicki, R. A. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources. Nature Protocols. 4 (1), 44-57 (2009).
- Huang, D. W., Sherman, B. T., Lempicki, R. A. Bioinformatics enrichment tools: Paths toward the comprehensive functional analysis of large gene lists. Nucleic Acids Research. 37 (1), 1-13 (2009).
- Letunic, I., Bork, P. Interactive tree of life (iTOL) v3: an online tool for the display and annotation of phylogenetic and other trees. Nucleic acids research. 44, 242-245 (2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados