Method Article
Биоинформатический конвейер для исследования молекулярной эволюции и экспрессии генов с использованием RNA-seq
В этой статье
Резюме
Целью этого протокола является исследование эволюции и экспрессии генов-кандидатов с использованием данных секвенирования РНК.
Аннотация
Дистилляция и представление больших наборов данных, таких как данные всего генома или транскриптома, часто является сложной задачей. Один из способов разбить результаты — сосредоточиться на одном или нескольких семействах генов, которые важны для организма и исследования. В этом протоколе мы описываем биоинформативные шаги для создания филогении и количественной оценки экспрессии генов, представляющих интерес. Филогенетические деревья могут дать представление о том, как гены развиваются внутри и между видами, а также выявить орфографию. Эти результаты могут быть улучшены с использованием данных RNA-seq для сравнения экспрессии этих генов у разных людей или тканей. Исследования молекулярной эволюции и экспрессии могут выявить способы эволюции и сохранения функции генов между видами. Характеристика семейства генов может служить трамплином для будущих исследований и может выделить важное семейство генов в новом геноме или транскриптоме.
Введение
Достижения в области технологий секвенирования облегчили секвенирование геномов и транскриптомов немоделированных организмов. В дополнение к повышенной возможности секвенирования ДНК и РНК от многих организмов, обилие данных является общедоступным для изучения генов, представляющих интерес. Целью этого протокола является предоставление биоинформатических шагов для исследования молекулярной эволюции и экспрессии генов, которые могут играть важную роль в интересуемом организме.
Исследование эволюции гена или семейства генов может дать представление об эволюции биологических систем. Члены семейства генов обычно определяются путем идентификации сохраненных мотивов или гомологичных последовательностей генов. Эволюция семейства генов ранее исследовалась с использованием геномов отдаленно связанных модельных организмов1. Ограничением этого подхода является то, что неясно, как эти семейства генов развиваются у близкородственных видов и роль различных селективных давлений окружающей среды. В этот протокол мы включаем поиск гомологов у близкородственных видов. Генерируя филогенез на уровне типа, мы можем отметить тенденции в эволюции семейства генов, такие как сохранение генов или дупликации, специфичные для линии. На этом уровне мы также можем исследовать, являются ли гены ортологами или паралогами. Хотя многие гомологи, вероятно, функционируют аналогично друг другу, это не обязательно так2. Включение филогенетических деревьев в эти исследования важно для решения вопроса о том, являются ли эти гомологичные гены ортологами или нет. У эукариот многие ортологи сохраняют аналогичные функции внутри клетки, о чем свидетельствует способность белков млекопитающих восстанавливать функцию дрожжевых ортологов3. Однако есть случаи, когда неортологичный ген выполняет характерную функцию4.
Филогенетические деревья начинают очертивать отношения между генами и видами, но функция не может быть назначена исключительно на основе генетических связей. Исследования экспрессии генов в сочетании с функциональными аннотациями и анализом обогащения обеспечивают сильную поддержку функции генов. Случаи, когда экспрессия генов может быть количественно оценена и сравнена между людьми или типами тканей, могут быть более показательными для потенциальной функции. Следующий протокол следует методам, используемым при исследовании генов опсина в Hydra vulgaris7,но они могут быть применены к любому виду и любому семейству генов. Результаты таких исследований обеспечивают основу для дальнейшего изучения функции генов и генных сетей в немоделовых организмах. В качестве примера, исследование филогении опсинов, которые являются белками, которые инициируют каскад фототрансдукции, дает контекст эволюции глаз и обнаружения света8,9,10,11. В этом случае немодельные организмы, особенно базальные виды животных, такие как книдарии или гребневицы, могут прояснить сохранение или изменения в каскаде фототрансдукции и зрения черезклады 12,13,14. Точно так же определение филогении, экспрессии и сетей других семейств генов проинформирует нас о молекулярных механизмах, лежащих в основе адаптаций.
протокол
Этот протокол следует рекомендациям по уходу за животными UC Irvine.
1. Подготовка библиотеки РНК-seq
- Изолируйте РНК с помощью следующих методов.
- Соберите образцы. Если РНК должна быть извлечена в более позднее время, флэш-заморозьте образец или поместите в раствор для хранения РНК15 (Таблица материалов).
- Усыпить и препарировать организм, чтобы отделить ткани, представляющие интерес.
- Извлеките общую РНК с помощью экстракционного набора и очистите РНК с помощью набора для очистки РНК (Таблица материалов)
ПРИМЕЧАНИЕ: Существуют протоколы и наборы, которые могут работать лучше для различных видов и типов тканей16,17. Мы извлекли РНК из разных тканей организма бабочки18 и желатиновой гидры19 (см. обсуждение). - Измерьте концентрацию и качество РНК каждого образца(Таблица материалов). Используйте образцы с числами целостности РНК (RIN) выше 8, в идеале ближе к 920 для создания библиотек кДНК.
- Постройте библиотеку и последовательность кДНК следующим образом.
- Создавайте библиотеки кДНК в соответствии с инструкцией по подготовке библиотек (см. обсуждение).
- Определение концентрации и качества кДНК(Таблица материалов).
- Мультиплексирует библиотеки и упорядочивает их.
2. Доступ к кластеру компьютеров
ПРИМЕЧАНИЕ: Анализ RNA-seq требует манипуляций с большими файлами и лучше всего выполняется на компьютерном кластере (Таблица материалов).
- Войдите в учетную запись кластера компьютеров с помощью команды ssh username@clusterlocation в окне приложения терминала (Mac) или PuTTY (Windows).
3. Получение считывания РНК-seq
- Получение считывания РНК-секв из средства секвенирования или, для данных, сгенерированных в публикации, из хранилища данных, где они были депонированы (3.2 или 3.3).
- Чтобы загрузить данные из репозиториев, таких как ArrayExpress, выполните следующие действия.
- Выполните поиск по сайту по номеру присоединения.
- Найдите ссылку для загрузки данных, затем щелкните левой кнопкой мыши и выберите Копировать ссылку.
- В окне терминала введите wget и выберите Вставить ссылку, чтобы скопировать данные в каталог для анализа.
- Чтобы загрузить данные NCBI Short Read Archive (SRA), выполните следующие альтернативные действия:
- На терминале загрузите SRA Toolkit v. 2.8.1 с помощью wget.
ПРИМЕЧАНИЕ: Загрузка и установка программ в кластер компьютеров может потребовать доступа root, обратитесь к администратору кластера компьютера в случае сбоя установки. - Завершите установку программы, набрав tar -xvf $TARGZFILE.
- Выполните поиск в NCBI по номеру присоединения SRA для образцов, которые вы хотите загрузить, он должен иметь формат SRRXXXXXX.
- Получите данные RNA-seq, набрав [расположение sratoolkit]/bin/prefetch SRRXXXXXX в окне терминала.
- Для сопряженных файлов введите [расположение sratoolkit]/bin/fastq-dump --split-files SRRXXXXXX, чтобы получить два файла fastq (SRRXXXXXX_1.FASTQ и SRRXXXXXX_2.FASTQ).
ПРИМЕЧАНИЕ: Для сборки Trinity de novo используйте команду [расположение sratoolkit]/bin/fastq-dump --defline-seq '@$sn[_$rn]/$ri' --split-files SRRXXXXXX
- На терминале загрузите SRA Toolkit v. 2.8.1 с помощью wget.
4. Обрезка адаптеров и некачественные считываемые материалы (опционально)
- Установите или загрузите Trimmomatic21 v. 0.35 в вычислительном кластере.
- В каталоге, где расположены файлы данных RNA-seq, введите команду, включаемую расположение триммоматного jar-файла, входные файлы FASTQ, выходные файлы FASTQ и дополнительные параметры, такие как длина и качество чтения.
ПРИМЕЧАНИЕ: Команда будет варьироваться в зависимости от необработанного и желаемого качества и продолжительности чтения. Для чтения Illumina 43 bp с помощью праймеров Nextera мы использовали: java -jar /data/apps/trimmomatic/0.35/trimmomatic-0.35.jar PE $READ 1. ФАСТК $READ 2. paired_READ1 FASTQ. unpaired_READ1 FASTQ. paired_READ2 FASTQ. unpaired_READ2 FASTQ. FASTQ ILLUMINACLIP:adapters.fa:2:30:10 LEADING:20 TRAILING:20 СКОЛЬЗЯЩЕЕ ВИНО:4:17 MINLEN:30.
5. Получение эталонной сборки
- Поиск в Google, EnsemblGenomes и NCBI Genomes and Nucleotide TSA (Transcriptome Shotgun Assembly) для эталонного генома или собранного транскриптома для интересующих видов(рисунок 1).
ПРИМЕЧАНИЕ: Если эталонный геном или транскриптом недоступны или имеют низкое качество, перейдите к ШАГУ 6 для создания сборки de novo. - Если существует эталонный геном или собранный транскриптом, загрузите его в виде файла fasta, где будет выполнен анализ, выполнив следующие действия.
- Найдите ссылку для загрузки генома, щелкните левой кнопкой мыши и скопируйте ссылку.
- В окне терминала введите wget и вставьте адрес ссылки. Если доступно, также скопируйте файл GTF и файл белка FASTA для эталонного генома.
6. Создание сборки de novo (альтернатива шагу 5)
- Объедините файлы RNA-seq READ1 и READ2 fastq для всех образцов, набрав cat *READ1. FASTQ > $all_READ1. FASTQ и кот *READ2. > all_READ2 FASTQ. FASTQ в окне терминала.
- Установите или загрузите Trinity22 v.2.8.5 в вычислительном кластере.
- Генерация и сборка путем ввода на терминале: Trinity --seqType fq --max_memory 20G --left $all_READ1. FASTQ --правый $all_READ2. ФАСТК.
7. Карта считывается с геномом (7.1) или de novo транскриптомом (7.2)
- Карта считывается с эталонный геном с использованием STAR23 v. 2.6.0c и RSEM24 v. 1.3.0.
- Установите или загрузите STAR v. 2.6.0c. и RSEM v. 1.3.0 для вычислительного кластера.
- Индексировать геном путем типизации rsem-prepare-reference --gtf $GENOME. GTF --звезда -p 16 $GENOME. $OUTPUT ФАСТА.
- Map считывает и вычисляет выражение для каждого образца, вводя rsem-calculate-expression -p 16 --star --paired-end $READ 1. ФАСТК $READ 2. $INDEX $OUTPUT FASTQ.
- Переименуйте файл результатов в описательный, используя mv RSEM.genes.results $sample.genes.results.
- Сгенерируйте матрицу всех счетчиков, набрав rsem-generate-data-matrix *[genes/isoforms.results] > $OUTPUT.
- Нанесите RNA-seq на сборку Trinity de novo с помощью RSEM и галстука-бабочки.
- Установите или загрузите Trinity22 v.2.8.5, Bowtie25 v. 1.0.0 и RSEM v. 1.3.0.
- Map считывает и вычисляет выражение для каждого образца, вводя [trinity_location]/align_and_estimate_abundance.pl --prep-reference --transcripts $TRINITY. FASTA --seqType fq --левый $READ 1. FASTQ --правый $READ 2. FASTQ --est_method RSEM --aln_method галстук-бабочка --trinity_mode --output_dir $OUTPUT.
- Переименуйте файл результатов в описательный, используя mv RSEM.genes.results $sample.genes.results.
- Сгенерируйте матрицу всех счетчиков, набрав [trinity_location]/abundance_estimates_to_matrix.pl --est_method RSEM *[гены/изоформы].results
8. Определите гены, представляющие интерес
ПРИМЕЧАНИЕ: Следующие шаги могут быть выполнены с нуклеотидными или белковыми файлами FASTA, но работают лучше всего и более просты с белковыми последовательностями. Поиск BLAST с использованием белка к белку с большей вероятностью даст результаты при поиске между различными видами.
- Для эталонного генома используйте файл белка FASTA из STEP 5.2.2 или см. Дополнительные материалы для создания пользовательского генного признака GTF.
- Для транскриптома de novo сгенерируйте белок FASTA с помощью TransDecoder.
- Установите или загрузите TransDecoder v. 5.5.0 на компьютер cluser.
- Найдите самый длинный открытый кадр считывания и предсказав последовательность пептидов, набрав [Расположение трансдекодера]/TransDecoder.LongOrfs -t $TRINITY. ФАСТА.
- Поиск омологов близкородственных видов в NCBI Genbank.
- Откройте окно интернет-браузера и перейдите в https://www.ncbi.nlm.nih.gov/genbank/.
- В строке поиска введите название интересуемого гена и название близкородственных видов, которые были секвенированы или род или тип. В левой части строки поиска выберите белок и нажмите кнопку поиска.
- Извлеките последовательности, нажав кнопку Отправить, а затем выберите Файл. В разделе Формат выберите FASTA и нажмите Кнопка Создать файл.
- Переместите файл гомологов FASTA в кластер компьютеров, набрав scp $FASTA username@clusterlocation:/$DIR в окне локального терминала, или используйте FileZilla для передачи файлов на компьютер и кластер и с него.
- Поиск генов-кандидатов с помощью BLAST+26.
- Установите или загрузите BLAST+ v. 2.8.1 в кластере компьютеров.
- В компьютерном кластере сделайте базу данных BLAST из генома или транскриптома, переведенного белка FASTA, набрав [BLAST+ location]/makeblastdb -in $PEP. FASTA -dbtype prot -out $OUTPUT
- BLAST гомологичные последовательности генов из NCBI в базу данных интересующих видов путем ввода [BLAST+ location]/blastp -db $DATABASE -query $FASTA -evalue 1e-10 -outfmt 6 -max_target_seqs 1 -out $OUTPUT.
- Просмотрите выходной файл с помощью команды подробнее. Скопируйте уникальные идентификаторы генов интересующих видов в новый текстовый файл.
- Извлеките последовательности генов-кандидатов, набрав perl -ne 'if(/^>(\S+)/){$c=$i{$1}}$c?print:chomp;$i{$_}=1 if @ARGV' $gene_id.txt $PEP. > $OUTPUT ФАСТА.
- Подтвердите аннотацию гена с помощью реципрокного BLAST.
- В интернет-браузере перейдите в https://blast.ncbi.nlm.nih.gov/Blast.cgi.
- Выберите tblastn, затем вставьте последовательности-кандидаты, выберите базу данных неизбыточных белковых последовательностей и нажмите КНОПКУ BLAST.
- Идентификация дополнительных генов путем аннотирования всех генов в геноме или транскриптоме терминами генной онтологии (GO) (см. обсуждение).
- Перенесите белок FASTA на локальный компьютер.
- Загрузите и установите Blast2GO27,28,29 v. 5.2 на локальный компьютер.
- Откройте Blast2GO, щелкните Файл,перейдите в Загрузить,перейдите в Загрузить последовательности, нажмите Загрузить файл Fasta (fasta). Выберите файл FASTA и нажмите кнопку Загрузить.
- Нажмите «Blast», выберите NCBI Blastи нажмите «Далее». Отредактируйте параметры или нажмите кнопку Далее,отредактируйте параметры и нажмите кнопку Выполнить, чтобы найти наиболее похожее описание гена.
- Щелкните сопоставление, а затем нажмите кнопку Выполнить, чтобы выполнить поиск похожих белков в аннотациях Gene Ontology.
- Затем щелкните interpro, выберите EMBL-EBI InterProи нажмите кнопку Далее. Измените параметры или нажмите кнопку Далееи нажмите кнопку Выполнить для поиска сигнатур известных семейств генов и доменов.
- Экспортируйте аннотации, щелкнув Файл,выбрав Экспорт, нажмите экспорт таблицы. Нажмите кнопку Обзор, присвойте файлу имя, нажмите кнопку Сохранить,нажмите кнопку Экспорт.
- Выполните поиск в таблице аннотаций по интересующих терминов GO, чтобы определить дополнительные гены-кандидаты. Извлеките последовательности из файла FASTA (ШАГ 8.4.5)
9. Филогенетические деревья
- Загрузите и установите MEGA30 v. 7.0.26 на локальный компьютер.
- Откройте MEGA, нажмите «Выровнять»,нажмите «Редактировать/Построить выравнивание»,выберите «Создать новое выравнивание», нажмите OK, выберите «Белок».
- Когда откроется окно выравнивания, нажмите «Редактировать»,нажмите «Вставить последовательности из файла» и выберите FASTA с белковыми последовательностями генов-кандидатов и вероятными гомологами.
- Выберите все последовательности. Найдите символ руки и наведите на него курсо. Следует сказать Выровнять последовательности с помощью алгоритма MUSCLE31. Щелкните символ руки, а затем щелкните Выровнять белок, чтобы выровнять последовательности. Измените параметры или нажмите кнопку ОК, чтобы выровнять параметры по умолчанию.
- Визуально проверьте и внесите любые изменения вручную, затем сохраните и закройте окно выравнивания.
- В главном окне MEGA нажмите на Модели,нажмите Найти лучшие модели ДНК / белка (ML),выберите файл выравнивания и выберите соответствующие параметры, такие как: Анализ: Выбор модели (ML), Дерево для использования: Автоматический (дерево соединения соседей), Статистический метод: Максимальная вероятность, Тип замены: Аминокислота, Обработка пробелов / отсутствующих данных: Использовать все сайты, Фильтр сайта ветвей: Нет.
- После того, как будет определена лучшая модель для данных, перейдите в главное окно MEGA. Щелкните Филогения, щелкните Дерево максимальной вероятности Contruct/Test, а затем при необходимости выберите выравнивание. Выберите подходящие параметры для дерева: Статистический метод: Максимальная вероятность, Тест на филогения: Метод Bootstrap со 100 репликами, Тип подстановки: аминокислота, Модель: LG с Freqs. (+F), показатели среди участков: гамма-распределенный (G) с 5 дискретными гамма-категориями, обработка разрывов/отсутствующих данных: использование всех сайтов, ЭВРИСТИЧЕСКИЙ метод ML: Ближайший-Сосед-Обмен (NNI).
10. Визуализация экспрессии генов с помощью доверенного платформенного модуля
- Для Trinity на компьютере кластера перейдите в каталог, в котором был запущен abundance_estimates_to_matrix.pl и одним из выходов должна быть matrix. TPM.not_cross_norm. Перенесите этот файл на локальный компьютер.
ПРИМЕЧАНИЕ: См. Дополнительные материалы для перекрестной нормализации проб. - Для TPM из анализа генома выполните следующие действия.
- В кластере компьютеров перейдите в папку установки RSEM. Скопируйте rsem-generate-data-matrix, введя scp rsem-generate-data-matrix rsem-generate-TPM-matrix. Используйте nano для редактирования нового файла и измените «my $offsite = 4» с 4 на 5 для TPM, теперь он должен читать «мой $offsite = 5».
- Перейдите в каталог, где находятся выходные файлы RSEM .genes.results, и теперь используйте rsem-generate-TPM-matrix *[genes/isoforms.results] > $OUTPUT для создания матрицы TPM. Передача результатов на локальный компьютер.
- Визуализируйте результаты в ggplot2.
- Загрузите R v. 4.0.0 и RStudio v. 1.2.1335 на локальный компьютер.
- Откройте RStudio в правой части экрана, перейдите на вкладку Пакеты и нажмите Установить. Введите ggplot2 и нажмите кнопку Установить.
- В окне сценария R прочитайте в таблице доверенного платформенного модуля, введя данные<-read.table("$tpm.txt",header = T)
- Для гистограмм, подобных рисунку 4, введите что-то похожее на: p<- ggplot() + geom_bar(aes(y=TPM, x=Symbol, fill=Tissue), data=data, stat="identity")
заполнить<-c("#d7191c","#fdae61", "#ffffbf", "#abd9e9", "#2c7bb6")
p<-p+scale_fill_manual(значения=заливка)
p + тема(ось.текст.x = element_text(угол = 90))
Результаты
Приведенные выше методы обобщены на рисунке 1 и были применены к набору данных тканей Hydra vulgaris. H. vulgaris является пресноводным беспозвоночным, которое принадлежит к типу Cnidaria, который также включает кораллы, медузы и морские анемоны. H. vulgaris может размножаться бесполым путем бутонинга, и они могут регенерировать свою голову и ногу при разделении пополам. В этом исследовании мы стремились исследовать эволюцию и экспрессию генов опсина в Hydra7. В то время как у гидр отсутствуют глаза, они демонстрируют светозависимое поведение32. Гены опсина кодируют белки, которые важны для зрения, чтобы обнаружить различные длины волн света и начать каскад фототрансдукции. Исследование молекулярной эволюции и экспрессии этого семейства генов у базального вида может дать представление об эволюции глаз и обнаружении света у животных.
Мы сгенерировали управляемую сборку с использованием эталонного генома Hydra2.0 33 и общедоступных данных RNA-seq (присоединение к GEO GSE127279) Рисунок 1. Этот шаг занял примерно 3 дня. Хотя в этом случае мы не генерировали транскриптом de novo, сборка Trinity может занять до 1 недели, и каждая библиотека может занять несколько часов для чтения сопоставления в зависимости от картографа. Объединенная сборка Hydra (~ 50 000 расшифровок) была аннотирована с помощью Blast2GO, что заняло около 1 недели на рисунке 1. Последовательности генов, связанных с опсином, были извлечены в файл fasta. Последовательности генов опсина от других видов также были извлечены из NCBI GenBank. Мы использовали опсины из книдарий Podocoryna carnea, Cladonema radiatum, Tripedelia cystophoraи Nematostella vectensis,а также мы включили аутгруппы Mnemiopsis leidyi, Trichoplax adhaerens, Drosophila melanogaster и Homo sapiens. Гены опсина были выровнены на MEGA7 Рисунок 2. Просмотрев выравнивание, мы смогли идентифицировать опсины гидры, в которых отсутствовала сохраненная аминокислота лизина, необходимая для связывания светочувствительной молекулы. После визуального осмотра мы определили лучшую модель, выполнив анализ выбора модели. Мы сгенерировали дерево максимальной вероятности, используя модель LG + G + F со значением начальной загрузки 100 Рисунок 3. Для 149 генов опсина дерево было закончено примерно за 3 дня. Филогенез предполагает, что гены опсина развиваются путем специфических для линии дупликации у книдариев и, возможно, путем тандемной дупликации у H. vulgaris7.
Мы провели дифференциальный экспрессионный анализ в edgeR и рассмотрели абсолютную экспрессию генов опсина. Мы предположили, что один или несколько опсинов будут регулироваться в голове (гипостома), и провели парные сравнения гипостома с колонкой тела, почковой зоной, стопой и щупальцами. В качестве примера парного сравнения 1 774 транскрипта были дифференциально выражены между гипостомой и колонкой тела. Мы определили гены, которые были регулированы в ходе нескольких сравнений, и провели функциональное обогащение в таблице 1Blast2GO. Группировка активности рецепторов, связанных с G-белком, включала гены опсина. Наконец, мы рассмотрели абсолютную экспрессию генов опсина в разных тканях, во время бутонирования и во время регенерации, построив их значения TPM с помощью ggplot Рисунок 4. Используя методы, описанные здесь, мы идентифицировали 2 гена опсина, которые не группировались с другими опсинами в филогении, обнаружили один опсин, который был экспрессирован почти в 200 раз больше, чем другие, и мы обнаружили несколько генов опсина, экспрессируемых совместно с генами фототрансдукции, которые могут быть использованы для обнаружения света.
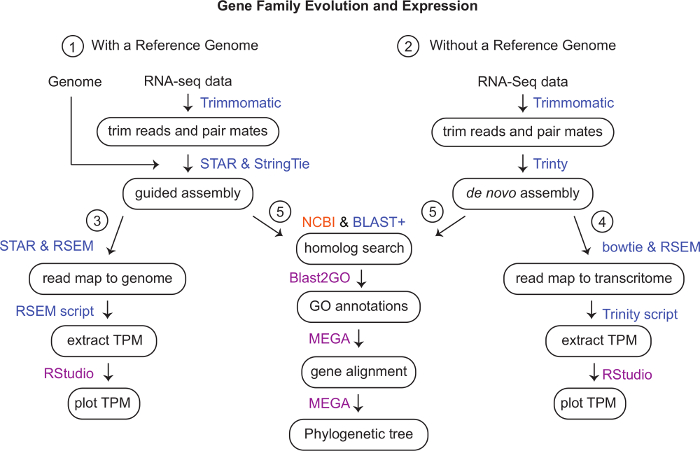
Рисунок 1: Схема рабочего процесса. Программы, используемые для анализа данных в компьютерном кластере, имеют синий цвет, пурпурным цветом - те, которые мы использовали на локальном компьютере, а оранжевым - веб-программу. (1) Тримм Считывает РНК-seq с использованием триммоматики v. 0.35. Если геном доступен, но генные модели отсутствуют, сгенерируйте управляемую сборку с помощью STAR v. 2.6.0c и StringTie v. 1.3.4d. (Факультативно см. Дополнительные материалы) (2) Без эталонного генома используйте обрезанные чтения для создания сборки de novo с использованием Trinity v2.8.5. (3) Для количественной оценки экспрессии генов с использованием эталонного генома карта считывается с использованием STAR и количественно определяется с помощью RSEM v. 1.3.1. Извлеките TPM с помощью RSEM и визуализируйте их в RStudio. (4) Bowtie и RSEM могут использоваться для отображения и количественной оценки считывания, сопоставленного с триечным транскриптомом. Сценарий Trinity можно использовать для создания матрицы доверенного платформенного модуля для визуализации счетчиков в RStudio. (5) Используйте веб-сайт NCBI BLAST и командную строку BLAST+ для поиска гомологичных последовательностей и подтверждения с помощью обратного BLAST. Аннотировать гены далее с помощью Blast2GO. Используйте MEGA для выравнивания генов и генерации филогенетического дерева с использованием наиболее подходящей модели. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
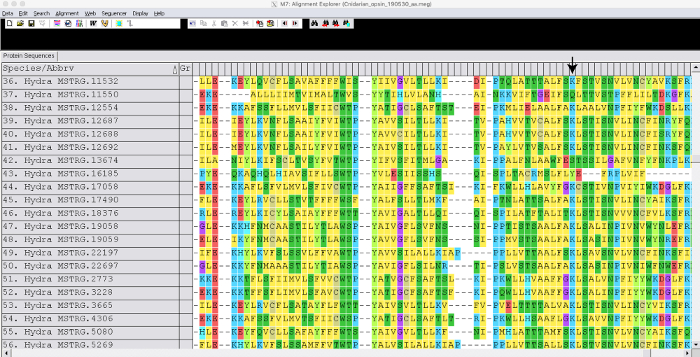
Рисунок 2: Пример выровненных генов. Снимок показывает часть генов опсина гидры, выровненных с помощью MUSCLE. Стрелка указывает местоположение консервированного лизина, связывающего сетчатку. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
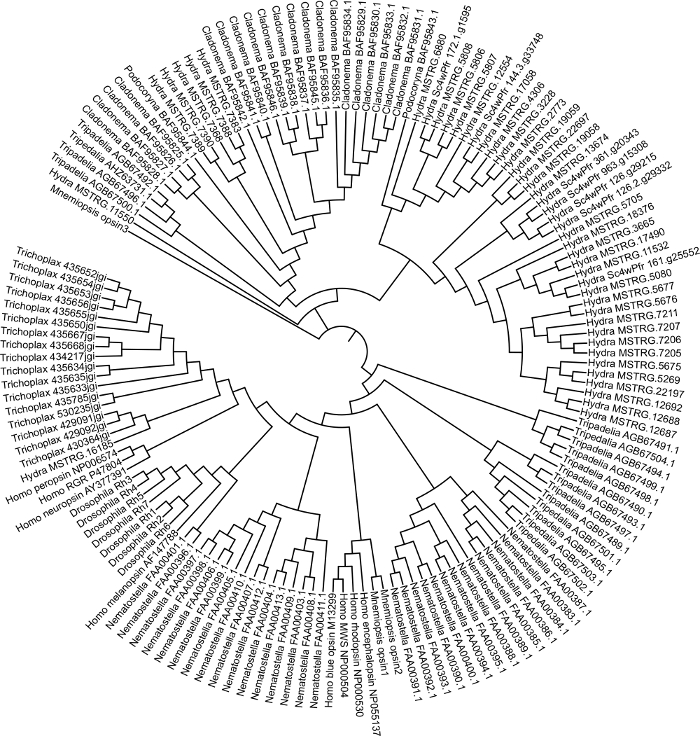
Рисунок 3: Филогенетическое дерево книдариевого опсина. Дерево максимальной вероятности генерируется в MEGA7 с использованием последовательностей опсина из Hydra vulgaris, Podocoryna carnea, Cladonema radiatum, Tripedelia cystophora, Nematostella vectensis, Mnemiopsis leidyi, Trichoplax adhaerens, Drosophila melanogaster и Homo sapiens. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
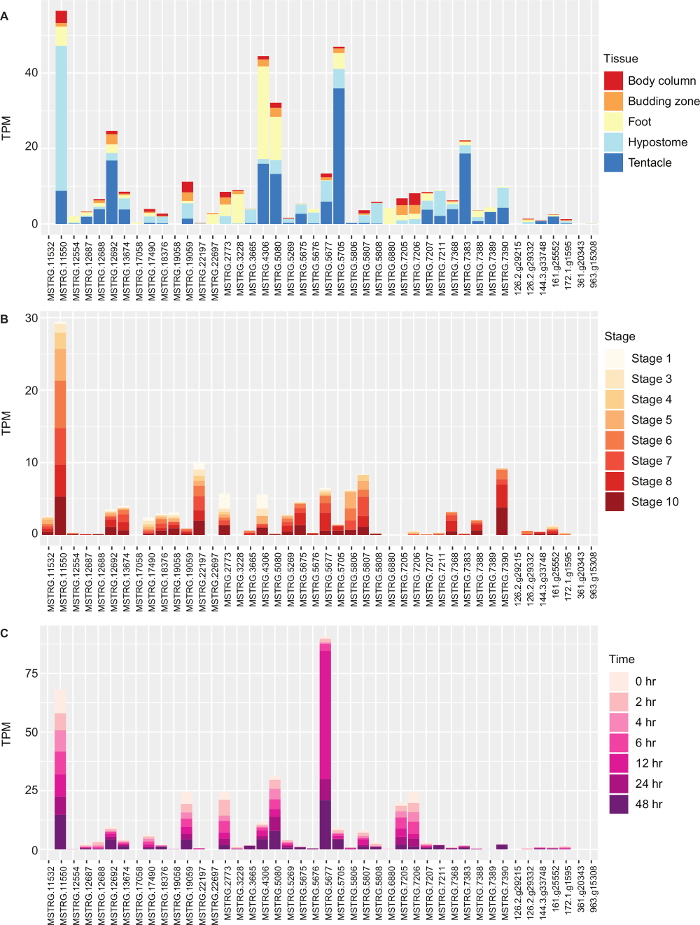
Рисунок 4: Экспрессия генов Opsin у Hydra vulgaris. (A) Экспрессия в транскриптах на миллион (TPM) генов опсина Hydra vulgaris в колонке тела, почковой зоне, стопе, гипостоме и щупальцах. (B) Экспрессия генов опсина на разных стадиях бутонации гидры. (C) Экспрессия генов опсина гипостома гидры в различные временные точки регенерации. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
| ИДЕНТИФИКАТОР GO | Go Имя | Категория GO | Рузвельт |
| ГО:0004930 | Активность рецепторов, связанных с G-белком | МОЛЕКУЛЯРНАЯ ФУНКЦИЯ | 0.0000000000704 |
| ГО:0007186 | Сигнальный путь рецепторов, связанных с G-белком | БИОЛОГИЧЕСКИЙ ПРОЦЕСС | 0.00000000103 |
| ГО:0016055 | Сигнальный путь Wnt | БИОЛОГИЧЕСКИЙ ПРОЦЕСС | 0.0000358 |
| ГО:0051260 | гомолигомеризация белка | БИОЛОГИЧЕСКИЙ ПРОЦЕСС | 0.000376 |
| ГО:0004222 | активность металлоэндопептидазы | МОЛЕКУЛЯРНАЯ ФУНКЦИЯ | 0.000467 |
| ГО:0008076 | комплекс калиевых каналов с напряжением | КЛЕТОЧНЫЙ КОМПОНЕНТ | 0.000642 |
| ГО:0005249 | активность калиевого канала с напряжением | МОЛЕКУЛЯРНАЯ ФУНКЦИЯ | 0.00213495 |
| ГО:0007275 | развитие многоклеточного организма | БИОЛОГИЧЕСКИЙ ПРОЦЕСС | 0.00565048 |
| ГО:0006813 | транспорт ионов калия | БИОЛОГИЧЕСКИЙ ПРОЦЕСС | 0.01228182 |
| ГО:0018108 | пептидил-тирозин фосфорилирование | БИОЛОГИЧЕСКИЙ ПРОЦЕСС | 0.02679662 |
Таблица 1: Функциональное обогащение генов, урегулируемых в гипостоме
Дополнительные материалы. Пожалуйста, нажмите здесь, чтобы загрузить эти материалы.
Обсуждение
Цель этого протокола состоит в том, чтобы дать краткое описание шагов для характеристики семейства генов с использованием данных RNA-seq. Было доказано, что эти методы работают для различных видов и наборов данных4,34,35. Созданный здесь конвейер был упрощен и должен быть достаточно легким, чтобы за ним следовал новичок в биоинформатике. Значение протокола заключается в том, что он описывает все шаги и необходимые программы для завершения публикуемого анализа. Важным шагом в протоколе является правильная сборка полноразмерных транскриптов, которые исходят из высококачественных геномов или транскриптомов. Чтобы получить правильные расшифровки, необходимо высококачественное РНК и / или ДНК и хорошие аннотации, обсуждаемые ниже.
Для подготовки библиотеки RNA-seq мы включаем наборы списков, которые работали для небольших частей тела Гидры19 и бабочек18 (Таблица материалов). Отметим, что для низковходной РНК мы использовали модифицированный протокольный подход36. Методы экстракции РНК сравнивались в нескольких типах образцов, включая дрожжевые клетки17,нейробластому37,растения38и личинки насекомых16, чтобы назвать несколько. Мы рекомендуем читателю приобрести протокол, который работает для интересующих его видов, если таковые существуют, или устранить неполадки с помощью общедоступных коммерчески доступных комплектов для начала. Для правильной количественной оценки генов мы рекомендуем обрабатывать образец РНК ДНКазой. Наличие ДНК повлияет на правильную количественную оценку генов. Мы также рекомендуем использовать набор для подготовки библиотеки кДНК, который включает в себя выбор хвоста polyA для выбора зрелой мРНК. В то время как истощение рРНК приводит к большей глубине считывания, процент покрытия экзона намного ниже, чем покрытие экзона РНК с использованием выбора polyA+39. Наконец, по возможности лучше всего использовать парные концы и мельчие40,41. В приведенном выше протоколе команды сопоставления чтения должны быть изменены при использовании односторонних считываний.
Как упоминалось выше, важно уметь идентифицировать гены, представляющие интерес, а также различать недавние дупликации генов, альтернативное сплайсинг и гаплотипы при секвенировании. В некоторых случаях наличие эталонного генома может помочь, определив, где гены и экзоны расположены относительно друг друга. Следует отметить, что если транскриптом получен из общедоступной базы данных и не является высококачественным, может быть лучше всего генерировать с использованием Trinity42 и объединения библиотек RNA-seq из тканей, представляющих интерес. Аналогичным образом, если эталонный геном не имеет хороших генных моделей, библиотеки RNA-seq могут быть использованы для генерации новых GTF с использованием StringTie43 (см. Дополнительные материалы). Кроме того, в тех случаях, когда гены неполны и есть доступ к геному, гены могут быть вручную отредактированы с использованием гомологических последовательностей, а затем выровнены с геномом с помощью tblastn. Выход BLAST может быть использован для определения фактической последовательности, которая может отличаться от коррекции, выполненной с использованием гомологов. Если совпадений нет, оставьте последовательность как изначально. При проверке выходных данных обратите внимание на координаты генома, чтобы убедиться, что отсутствующий экзон действительно является частью гена.
Хотя мы фокусируемся на программном обеспечении и программах, которые мы использовали, изменения в этом протоколе существуют из-за множества доступных программ, которые могут работать лучше для различных наборов данных. В качестве примера мы показываем команды для сопоставления чтения с транскриптомом с использованием бабочки и RSEM, но у Trinity теперь есть опция для гораздо более быстрых элайнеров, таких как kallisto44 и salmon45. Точно так же мы описываем аннотации с помощью Blast2GO (теперь OmicsBox), но есть и другие инструменты картографа, которые можно найти бесплатно и в Интернете. Некоторые из них, которые мы попробовали, включают: GO FEAT46,eggNOG-mapper47,48и очень быстрый элайнер PANNZER249. Чтобы использовать эти веб-инструменты аннотаций, просто загрузите пептид FASTA и отправьте. Автономные версии PANNZER и eggNOG-mapper также доступны для загрузки в компьютерный кластер. Другая модификация заключается в том, что мы использовали MEGA и R на локальном компьютере и использовали онлайн-инструмент NCBI BLAST для выполнения взаимных BLAST, однако все эти программы могут использоваться в компьютерном кластере путем загрузки необходимых программ и баз данных. Аналогичным образом, элайнеры каллисто и лосось можно использовать на локальном компьютере, если у пользователя достаточно оперативной памяти и хранилища. Тем не менее, файлы FASTQ и FASTA, как правило, очень велики, и мы настоятельно рекомендуем использовать компьютерный кластер для простоты и скорости. Кроме того, хотя мы предоставляем инструкции и ссылки на скачивание программ от их разработчиков, многие из них могут быть установлены из bioconda: https://anaconda.org/bioconda.
Распространенной проблемой, с которой сталкиваются при проведении биоинформатических анализов, является сбой сценариев оболочки. Это может быть связано с самыми разными причинами. Если создается файл ошибки, этот файл ошибки следует проверить перед устранением неполадок. Несколькими распространенными причинами ошибки являются опечатки, отсутствие ключевых параметров и проблемы совместимости между версиями программного обеспечения. В этот протокол мы включаем параметры для данных, но руководства по программному обеспечению могут предоставить более подробные рекомендации по отдельным параметрам. В общем, лучше всего использовать самые последние версии программного обеспечения и обращаться к руководству, соответствующему этой версии.
Усовершенствования этого протокола включают в себя проведение дифференциального анализа выражений в масштабах всего транскриптома и анализа функционального обогащения. Мы рекомендуем edgeR50 для дифференциального экспрессионного анализа пакет, доступный в Bioconductor. Для анализа функционального обогащения мы использовали Blast2GO29 и веб-сайт DAVID51,52. Мы также рекомендуем дополнительно отредактировать филогению, извлекая ее в виде файла newick и используя веб-сайт iTOL53. Кроме того, хотя этот протокол будет исследовать молекулярную эволюцию и паттерны экспрессии генов, дополнительные эксперименты могут быть использованы для проверки местоположения и функций генов или белков. Экспрессия мРНК может быть подтверждена с помощью RT-qPCR или гибридизации in situ. Белки могут быть локализованы с помощью иммуногистохимии. В зависимости от вида, нокаут-эксперименты могут быть использованы для подтверждения функции генов. Этот протокол может быть использован для различных целей, включая, как показано выше, для изучения семейства генов, обычно связанного с фоторецепцией у базального вида7. Другим применением этих методов является выявление изменений в законсервированном пути при различных селективных давлениях. В качестве примера эти методы были использованы для обнаружения вариаций в экспрессии потенциальных каналов транзиторных рецепторов зрения между суточными бабочками и ночными мотыльками34.
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Мы благодарим Адриану Бриско, Гила Смита, Раби Мурада и Алин Г. Рангел за советы и рекомендации по включению некоторых из этих шагов в наш рабочий процесс. Мы также благодарны Кэтрин Уильямс, Элизабет Реббоа и Наташе Пиччиани за комментарии к рукописи. Эта работа была частично поддержана стипендией Фонда Джорджа Э. Хьюитта для медицинских исследований A.M.M.
Материалы
| Name | Company | Catalog Number | Comments |
| Bioanalyzer-DNA kit | Agilent | 5067-4626 | wet lab materials |
| Bioanalyzer-RNA kit | Agilent | 5067-1513 | wet lab materials |
| BLAST+ v. 2.8.1 | On computer cluster* https://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/ | ||
| Blast2GO (on your PC) | On local computer https://www.blast2go.com/b2g-register-basic | ||
| boost v. 1.57.0 | On computer cluster | ||
| Bowtie v. 1.0.0 | On computer cluster https://sourceforge.net/projects/bowtie-bio/files/bowtie/1.3.0/ | ||
| Computing cluster (highly recommended) | NOTE: Analyses of genomic data are best done on a high-performance computing cluster because files are very large. | ||
| Cufflinks v. 2.2.1 | On computer cluster | ||
| edgeR v. 3.26.8 (in R) | In Rstudio https://bioconductor.org/packages/release/bioc/html/edgeR.html | ||
| gcc v. 6.4.0 | On computer cluster | ||
| Java v. 11.0.2 | On computer cluster | ||
| MEGA7 (on your PC) | On local computer https://www.megasoftware.net | ||
| MEGAX v. 0.1 | On local computer https://www.megasoftware.net | ||
| NucleoSpin RNA II kit | Macherey-Nagel | 740955.5 | wet lab materials |
| perl 5.30.3 | On computer cluster | ||
| python | On computer cluster | ||
| Qubit 2.0 Fluorometer | ThermoFisher | Q32866 | wet lab materials |
| R v.4.0.0 | On computer cluster https://cran.r-project.org/src/base/R-4/ | ||
| RNAlater | ThermoFisher | AM7021 | wet lab materials |
| RNeasy kit | Qiagen | 74104 | wet lab materials |
| RSEM v. 1.3.0 | Computer software https://deweylab.github.io/RSEM/ | ||
| RStudio v. 1.2.1335 | On local computer https://rstudio.com/products/rstudio/download/#download | ||
| Samtools v. 1.3 | Computer software | ||
| SRA Toolkit v. 2.8.1 | On computer cluster https://github.com/ncbi/sra-tools/wiki/01.-Downloading-SRA-Toolkit | ||
| STAR v. 2.6.0c | On computer cluster https://github.com/alexdobin/STAR | ||
| StringTie v. 1.3.4d | On computer cluster https://ccb.jhu.edu/software/stringtie/ | ||
| Transdecoder v. 5.5.0 | On computer cluster https://github.com/TransDecoder/TransDecoder/releases | ||
| Trimmomatic v. 0.35 | On computer cluster http://www.usadellab.org/cms/?page=trimmomatic | ||
| Trinity v.2.8.5 | On computer cluster https://github.com/trinityrnaseq/trinityrnaseq/releases | ||
| TRIzol | ThermoFisher | 15596018 | wet lab materials |
| TruSeq RNA Library Prep Kit v2 | Illumina | RS-122-2001 | wet lab materials |
| TURBO DNA-free Kit | ThermoFisher | AM1907 | wet lab materials |
| *Downloads and installation on the computer cluster may require root access. Contact your network administrator. |
Ссылки
- Lespinet, O., Wolf, Y. I., Koonin, E. V., Aravind, L. The role of lineage-specific gene family expansion in the evolution of eukaryotes. Genome Research. 12 (7), 1048-1059 (2002).
- Gabaldón, T., Koonin, E. V. Functional and evolutionary implications of gene orthology. Nature Reviews Genetics. 14 (5), 360-366 (2013).
- Dolinski, K., Botstein, D. Orthology and Functional Conservation in Eukaryotes. Annual Review of Genetics. 41 (1), (2007).
- Macias-Muñoz, A., McCulloch, K. J., Briscoe, A. D. Copy number variation and expression analysis reveals a non-orthologous pinta gene family member involved in butterfly vision. Genome Biology and Evolution. 9 (12), 3398-3412 (2017).
- Cannon, S. B., Mitra, A., Baumgarten, A., Young, N. D., May, G. The roles of segmental and tandem gene duplication in the evolution of large gene families in Arabidopsis thaliana. BMC plant biology. 4, 10(2004).
- Eastman, S. D., Chen, T. H. P., Falk, M. M., Mendelson, T. C., Iovine, M. K. Phylogenetic analysis of three complete gap junction gene families reveals lineage-specific duplications and highly supported gene classes. Genomics. 87 (2), 265-274 (2006).
- Macias-Munõz, A., Murad, R., Mortazavi, A. Molecular evolution and expression of opsin genes in Hydra vulgaris. BMC Genomics. 20 (1), 1-19 (2019).
- Hisatomi, O., Tokunaga, F. Molecular evolution of proteins involved in vertebrate phototransduction. Comparative Biochemistry and Physiology - B Biochemistry and Molecular Biology. 133 (4), 509-522 (2002).
- Arendt, D. Evolution of eyes and photoreceptor cell types. International Journal of Developmental Biology. 47, 563-571 (2003).
- Shichida, Y., Matsuyama, T. Evolution of opsins and phototransduction. Philosophical Transactions of the Royal Society B: Biological Sciences. 364 (1531), 2881-2895 (2009).
- Porter, M. L., et al. Shedding new light on opsin evolution. Proceedings of the Royal Society B: Biological Sciences. 279 (1726), 3-14 (2012).
- Plachetzki, D. C., Degnan, B. M., Oakley, T. H. The origins of novel protein interactions during animal opsin evolution. PLoS ONE. 2 (10), 1054(2007).
- Ramirez, M. D., et al. The last common ancestor of most bilaterian animals possessed at least nine opsins. Genome Biology and Evolution. 8 (12), 3640-3652 (2016).
- Schnitzler, C. E., et al. Genomic organization, evolution, and expression of photoprotein and opsin genes in Mnemiopsis leidyi: a new view of ctenophore photocytes. BMC Biology. 10, 107(2012).
- Pedersen, K. B., Williams, A., Watt, J., Ronis, M. J. Improved method for isolating high-quality RNA from mouse bone with RNAlater at room temperature. Bone Reports. 11, 100211(2019).
- Ridgeway, J. A., Timm, A. E., Fallon, A. Comparison of RNA isolation methods from insect larvae. Journal of Insect Science. 14 (1), 4-8 (2014).
- Scholes, A. N., Lewis, J. A. Comparison of RNA isolation methods on RNA-Seq: Implications for differential expression and meta-Analyses. BMC Genomics. 21 (1), 1-9 (2020).
- Briscoe, A. D., et al. Female behaviour drives expression and evolution of gustatory receptors in butterflies. PLoS genetics. 9 (7), 1003620(2013).
- Murad, R., Macias-Muñoz, A., Wong, A., Ma, X., Mortazavi, A. Integrative analysis of Hydra head regeneration reveals activation of distal enhancer-like elements. bioRxiv. , 544049(2019).
- Gallego Romero, I., Pai, A. A., Tung, J., Gilad, Y. Impact of RNA degradation on measurements of gene expression. BMC Biology. 12, 42(2014).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Trinity. RNA-Seq De novo Assembly Using Trinity. , 1-7 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29, 15-21 (2013).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC bioinformatics. 12, 323(2011).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome biology. 10, 25(2009).
- Camacho, C., et al. BLAST+: architecture and applications. BMC Bioinformatics. 10, 421(2009).
- Conesa, A., Götz, S. Blast2GO: A comprehensive suite for functional analysis in plant genomics. International Journal of Plant Genomics. 619832, (2008).
- Conesa, A., et al. Blast2GO: A universal tool for annotation, visualization and analysis in functional genomics research. Bioinformatics. 21 (18), 3674-3676 (2005).
- Götz, S., et al. High-throughput functional annotation and data mining with the Blast2GO suite. Nucleic Acids Research. 36 (10), 3420-3435 (2008).
- Kumar, S., Stecher, G., Tamura, K. MEGA7: Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets. Molecular biology and evolution. 33 (7), 1870-1874 (2016).
- Edgar, R. C. MUSCLE: Multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Research. 32 (5), 1792-1797 (2004).
- Taddei-Ferretti, C., Musio, C., Santillo, S., Cotugno, A. The photobiology of Hydra's periodic activity. Hydrobiologia. 530, 129-134 (2004).
- Chapman, J. A., et al. The dynamic genome of Hydra. Nature. 464 (7288), 592-596 (2010).
- Macias-Muñoz, A., Rangel Olguin, A. G., Briscoe, A. D. Evolution of phototransduction genes in Lepidoptera. Genome Biology and Evolution. 11 (8), 2107-2124 (2019).
- Macias-Munõz, A., Murad, R., Mortazavi, A. Molecular evolution and expression of opsin genes in Hydra vulgaris. BMC Genomics. 20 (1), (2019).
- Picelli, S., et al. Full-length RNA-seq from single cells using Smart-seq2. Nature Protocols. 9 (1), 171-181 (2014).
- Tavares, L., Alves, P. M., Ferreira, R. B., Santos, C. N. Comparison of different methods for DNA-free RNA isolation from SK-N-MC neuroblastoma. BMC research notes. 4, 3(2011).
- Johnson, M. T. J., et al. Evaluating Methods for Isolating Total RNA and Predicting the Success of Sequencing Phylogenetically Diverse Plant Transcriptomes. PLoS ONE. 7 (11), (2012).
- Zhao, S., Zhang, Y., Gamini, R., Zhang, B., Von Schack, D. Evaluation of two main RNA-seq approaches for gene quantification in clinical RNA sequencing: PolyA+ selection versus rRNA depletion. Scientific Reports. 8 (1), 1-12 (2018).
- Zhao, S., et al. Comparison of stranded and non-stranded RNA-seq transcriptome profiling and investigation of gene overlap. BMC Genomics. 16 (1), 1-14 (2015).
- Corley, S. M., MacKenzie, K. L., Beverdam, A., Roddam, L. F., Wilkins, M. R. Differentially expressed genes from RNA-Seq and functional enrichment results are affected by the choice of single-end versus paired-end reads and stranded versus non-stranded protocols. BMC Genomics. 18 (1), 1-13 (2017).
- Haas, B. J., et al. De novo transcript sequence reconstruction from RNA-seq using the Trinity platform for reference generation and analysis. Nature Protocols. 8 (8), 1494-1512 (2013).
- Pertea, M., et al. StringTie enables improved reconstruction of a transcriptome from RNA-seq reads. Nature biotechnology. 33 (3), 290-295 (2015).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nature Biotechnology. 34 (5), 525-527 (2016).
- Patro, R., Duggal, G., Love, M. I., Irizarry, R. A., Kingsford, C. Salmon provides fast and bias-aware quantification of transcript expression. Nature Methods. 14 (4), 417-419 (2017).
- Araujo, F. A., Barh, D., Silva, A., Guimarães, L., Thiago, R. OPEN GO FEAT a rapid web-based functional annotation tool for genomic and transcriptomic data. , 8-11 (2018).
- Huerta-Cepas, J., et al. Fast genome-wide functional annotation through orthology assignment by eggNOG-mapper. Molecular Biology and Evolution. 34 (8), 2115-2122 (2017).
- Huerta-Cepas, J., et al. EggNOG 5.0: A hierarchical, functionally and phylogenetically annotated orthology resource based on 5090 organisms and 2502 viruses. Nucleic Acids Research. 47, 309-314 (2019).
- Törönen, P., Medlar, A., Holm, L. PANNZER2: A rapid functional annotation web server. Nucleic Acids Research. 46, 84-88 (2018).
- Robinson, M., Mccarthy, D., Chen, Y., Smyth, G. K. edgeR differential expression analysis of digital gene expression data User's Guide. , (2013).
- Huang, D. W., Sherman, B. T., Lempicki, R. A. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources. Nature Protocols. 4 (1), 44-57 (2009).
- Huang, D. W., Sherman, B. T., Lempicki, R. A. Bioinformatics enrichment tools: Paths toward the comprehensive functional analysis of large gene lists. Nucleic Acids Research. 37 (1), 1-13 (2009).
- Letunic, I., Bork, P. Interactive tree of life (iTOL) v3: an online tool for the display and annotation of phylogenetic and other trees. Nucleic acids research. 44, 242-245 (2016).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены