Method Article
Una Tubería Bioinformática Para La Investigación De La Evolución Molecular Y La Expresión Génica Usando RNA-seq
En este artículo
Resumen
El propósito de este protocolo es investigar la evolución y expresión de genes candidatos utilizando datos de secuenciación de ARN.
Resumen
Destilar y reportar grandes conjuntos de datos, como datos de genoma completo o transcriptoma, es a menudo una tarea desalentadora. Una forma de desglosar los resultados es centrarse en una o más familias de genes que son importantes para el organismo y el estudio. En este protocolo, se describen los pasos bioinformáticos para generar una filogenia y cuantificar la expresión de genes de interés. Los árboles filogenéticos pueden dar una idea de cómo los genes están evolucionando dentro y entre las especies, así como revelar la ortología. Estos resultados se pueden mejorar utilizando datos de ARN-seq para comparar la expresión de estos genes en diferentes individuos o tejidos. Los estudios de evolución molecular y expresión pueden revelar modos de evolución y conservación de la función génica entre especies. La caracterización de una familia de genes puede servir como trampolín para futuros estudios y puede destacar una familia de genes importante en un nuevo genoma o transcriptoma.
Introducción
Los avances en las tecnologías de secuenciación han facilitado la secuenciación de genomas y transcriptomas de organismos no modelo. Además de la mayor viabilidad de secuenciar adn y ARN de muchos organismos, una gran cantidad de datos está disponible públicamente para estudiar genes de interés. El propósito de este protocolo es proporcionar pasos bioinformáticos para investigar la evolución molecular y la expresión de genes que pueden desempeñar un papel importante en el organismo de interés.
Investigar la evolución de un gen o familia de genes puede proporcionar información sobre la evolución de los sistemas biológicos. Los miembros de una familia de genes se determinan típicamente mediante la identificación de motivos conservados o secuencias de genes homólogos. La evolución de la familia génica se investigó previamente utilizando genomas de organismos modelo distantemente relacionados1. Una limitación a este enfoque es que no está claro cómo evolucionan estas familias de genes en especies estrechamente relacionadas y el papel de las diferentes presiones selectivas ambientales. En este protocolo, incluimos una búsqueda de homólogos en especies estrechamente relacionadas. Al generar una filogenia a nivel de filo, podemos observar tendencias en la evolución de la familia de genes como la de genes conservados o duplicaciones específicas de linaje. En este nivel, también podemos investigar si los genes son ortólogos o parálogos. Si bien es probable que muchos homólogos funcionen de manera similar entre sí, ese no es necesariamente el caso2. La incorporación de árboles filogenéticos en estos estudios es importante para determinar si estos genes homólogos son ortólogos o no. En los eucariotas, muchos ortólogos conservan funciones similares dentro de la célula, como lo demuestra la capacidad de las proteínas de mamíferos para restaurar la función de los ortólogos de levadura3. Sin embargo, hay casos en los que un gen no ortólogo realiza una función caracterizada4.
Los árboles filogenéticos comienzan a delinear las relaciones entre los genes y las especies, sin embargo, la función no se puede asignar únicamente en función de las relaciones genéticas. Los estudios de expresión génica combinados con anotaciones funcionales y análisis de enriquecimiento proporcionan un fuerte apoyo para la función génica. Los casos en los que la expresión génica se puede cuantificar y comparar entre individuos o tipos de tejidos pueden ser más reveladores de la función potencial. El siguiente protocolo sigue los métodos utilizados en la investigación de los genes de opsina en Hydra vulgaris7,pero se pueden aplicar a cualquier especie y cualquier familia de genes. Los resultados de tales estudios proporcionan una base para la investigación adicional en la función del gen y las redes del gene en organismos no-modelo. A modo de ejemplo, la investigación de la filogenia de las opsinas, que son proteínas que inician la cascada de fototransducción, da contexto a la evolución de los ojos y la detección de la luz8,9,10,11. En este caso, los organismos no modelo, especialmente las especies animales basales como los cnidarios o los ctenophores, pueden dilucidar la conservación o los cambios en la cascada de fototransducción y la visión a través de los clados12,13,14. Del mismo modo, la determinación de la filogenia, expresión y redes de otras familias de genes nos informará sobre los mecanismos moleculares subyacentes a las adaptaciones.
Protocolo
Este protocolo sigue las pautas de cuidado de animales de UC Irvine.
1. Preparación de la biblioteca de ARN-seq
- Aísle el ARN usando los métodos siguientes.
- Recoger muestras. Si el ARN se va a extraer en un momento posterior, congele la muestra o colótese en la solución de almacenamiento de ARN15 (Tabla de Materiales).
- Eutanasiar y diseccionar el organismo para separar tejidos de interés.
- Extraer el ARN total utilizando un kit de extracción y purificar el ARN utilizando un kit de purificación de ARN(Tabla de materiales)
NOTA: Existen protocolos y kits que pueden funcionar mejor para diferentes especies y tipos detejidos 16,17. Hemos extraído ARN de diferentes tejidos corporales de una mariposa18 y una Hidragelatinosa 19 (ver discusión). - Medir la concentración y calidad del ARN de cada muestra (Tabla de Materiales). Utilice muestras con números de integridad de ARN (RIN) superiores a 8, idealmente más cerca de 920 para construir bibliotecas de ADNc.
- Construya la biblioteca y la secuencia de cDNA de la siguiente manera.
- Cree bibliotecas de cDNA de acuerdo con el manual de instrucciones de preparación de bibliotecas (consulte la discusión).
- Determinar la concentración y calidad del ADNc(Tabla de Materiales).
- Multiplexa las bibliotecas y secuenciarlas.
2. Acceder a un clúster de computadoras
NOTA: El análisis de ARN-seq requiere la manipulación de archivos grandes y se realiza mejor en un clúster de computadoras(Tabla de materiales).
- Inicie sesión en la cuenta de clúster de equipos utilizando el comando ssh username@clusterlocation en una ventana de la aplicación de terminal (Mac) o PuTTY (Windows).
3. Obtener lecturas de ARN-seq
- Obtener lecturas de ARN-seq de la instalación de secuenciación o, para los datos generados en una publicación, del repositorio de datos donde se depositaron (3.2 o 3.3).
- Para descargar datos de repositorios como ArrayExpress, haga lo siguiente:
- Busque en el sitio utilizando el número de acceso.
- Busque el vínculo para descargar los datos y, a continuación, haga clic con el botón izquierdo y seleccione Copiar vínculo.
- En la ventana de terminal, escriba wget y seleccione Pegar vínculo para copiar los datos en el directorio para su análisis.
- Para descargar los datos de NCBI Short Read Archive (SRA), siga estos pasos alternativos:
- En el terminal descargue SRA Toolkit v. 2.8.1 usando wget.
Nota : descargar e instalar programas en el clúster de equipos puede requerir acceso de raíz, póngase en contacto con el administrador de clústeres de equipo si se produce un error en la instalación. - Termine de instalar el programa escribiendo tar -xvf $TARGZFILE.
- Busque NCBI para el número de acceso SRA para las muestras que desea descargar, debe tener el formato SRRXXXXXX.
- Obtenga los datos de RNA-seq escribiendo [sratoolkit location]/bin/prefetch SRRXXXXXX en la ventana del terminal.
- Para archivos de extremo emparejado, escriba [sratoolkit location]/bin/fastq-dump --split-files SRRXXXXXX para obtener dos archivos fastq (SRRXXXXXX_1.FASTQ y SRRXXXXXX_2.FASTQ).
NOTA: Para hacer un ensamblaje Trinity de novo utilice el comando [sratoolkit location]/bin/fastq-dump --defline-seq '@$sn[_$rn]/$ri' --split-files SRRXXXXXX
- En el terminal descargue SRA Toolkit v. 2.8.1 usando wget.
4. Adaptadores de recorte y lecturas de baja calidad (opcional)
- Instale o cargue Trimmomatic21 v. 0.35 en el clúster de computación.
- En el directorio donde se encuentran los archivos de datos RNA-seq, escriba un comando que incluya la ubicación del archivo jar trimmomatic, los archivos FASTQ de entrada, los archivos FASTQ de salida y parámetros opcionales como la longitud y la calidad de lectura.
Nota : el comando variará por la calidad sin procesar y deseada y la longitud de las lecturas. Para lecturas illumina 43 bp con cebadores Nextera, usamos: java -jar /data/apps/trimmomatic/0.35/trimmomatic-0.35.jar PE $READ 1. FASTQ $READ 2. FASTQ paired_READ1. fastq unpaired_READ1. fastq paired_READ2. FASTQ unpaired_READ2. FASTQ ILLUMINACLIP:adapters.fa:2:30:10 LEADING:20 TRAILING:20 SLIDINGWINDOW:4:17 MINLEN:30.
5. Obtener el ensamblado de referencia
- Busque en Google, EnsemblGenomes y NCBI Genomes and Nucleotide TSA (Transcriptome Shotgun Assembly) un genoma de referencia o transcriptoma ensamblado para la especie de interés (Figura 1).
NOTA: Si un genoma de referencia o un transcriptoma no están disponibles o son de baja calidad, proceda al PASO 6 para generar un ensamblaje de novo. - Si existe un genoma de referencia o un transcriptoma ensamblado, descárguela como un archivo fasta a donde se realizará el análisis siguiendo los pasos a continuación.
- Encuentre el enlace para descargar el genoma, haga clic con el botón izquierdo y copie el enlace.
- En la ventana del terminal, escriba wget y pegue la dirección del enlace. Si está disponible, copie también el archivo GTF y el archivo FASTA de proteínas para el genoma de referencia.
6. Generar un ensamblaje de novo (Alternativa al Paso 5)
- Combine los archivos RNA-seq READ1 y READ2 fastq para todas las muestras escribiendo cat *READ1. FASTQ > $all_READ1. FASTQ y gato *READ2. FASTQ > all_READ2. FASTQ en la ventana del terminal.
- Instale o cargue Trinity22 v.2.8.5 en el clúster informático.
- Generar y ensamblaje escribiendo en el terminal: Trinity --seqType fq --max_memory 20G --left $all_READ1. FASTQ --right $all_READ2. FASTQ.
7. Mapa de lecturas del genoma (7.1) o transcriptoma de novo (7.2)
- El mapa lee el genoma de referencia usando STAR23 v. 2.6.0c y RSEM24 v. 1.3.0.
- Instale o cargue STAR v. 2.6.0c. y RSEM v. 1.3.0 al clúster de computación.
- Indexe el genoma escribiendo rsem-prepare-reference --gtf $GENOME. GTF --estrella -p 16 $GENOME. FASTA $OUTPUT.
- Asigne y calcule la expresión para cada muestra escribiendo rsem-calculate-expression -p 16 --star --paired-end $READ 1. FASTQ $READ 2. $INDEX $OUTPUT FASTQ.
- Cambie el nombre del archivo de resultados a algo descriptivo utilizando mv RSEM.genes.results $sample.genes.results.
- Generar una matriz de todos los recuentos escribiendo rsem-generate-data-matrix *[genes/isoforms.results] > $OUTPUT.
- Mapee RNA-seq al ensamblaje Trinity de novo usando RSEM y pajarita.
- Instale o cargue Trinity22 v.2.8.5, Bowtie25 v. 1.0.0 y RSEM v. 1.3.0.
- Asigne lecturas y calcule la expresión de cada muestra escribiendo [trinity_location]/align_and_estimate_abundance.pl --prep-reference --transcripts $TRINITY. FASTA --seqType fq --left $READ 1. FASTQ --derecha $READ 2. FASTQ --est_method RSEM --aln_method pajarita --trinity_mode --output_dir $OUTPUT.
- Cambie el nombre del archivo de resultados a algo descriptivo utilizando mv RSEM.genes.results $sample.genes.results.
- Generar una matriz de todos los recuentos escribiendo [trinity_location]/abundance_estimates_to_matrix.pl --est_method RSEM *[genes/isoformas].resultados
8. Identificar genes de interés
NOTA: Los siguientes pasos se pueden hacer con archivos FASTA de nucleótidos o proteínas, pero funcionan mejor y son más sencillos con las secuencias de proteínas. Las búsquedas blast usando proteína a proteína es más probable que den resultados cuando se busca entre diferentes especies.
- Para un genoma de referencia, utilice el archivo FASTA de proteínas del PASO 5.2.2 o consulte Materiales suplementarios para generar una característica genética personalizada GTF.
- Para un transcriptoma de novo, generar una proteína FASTA utilizando TransDecoder.
- Instale o cargue TransDecoder v. 5.5.0 en el cluser del equipo.
- Encuentre el marco de lectura abierto más largo y la secuencia de péptidos predicha escribiendo [Transdecoder location]/TransDecoder.LongOrfs -t $TRINITY. FASTA.
- Busque homólogos en especies estrechamente relacionadas en NCBI Genbank.
- Abra una ventana del navegador de Internet y vaya a https://www.ncbi.nlm.nih.gov/genbank/.
- En la barra de búsqueda escriba el nombre del gen de interés y el nombre de las especies estrechamente relacionadas que han sido secuenciadas o género o filo. A la izquierda de la barra de búsqueda, seleccione proteína y luego haga clic en buscar.
- Extraiga las secuencias haciendo clic en Enviar a y, a continuación, seleccione Archivo. En Formato, seleccione FASTA y, a continuación, haga clic en Crear archivo.
- Mueva el archivo FASTA de homólogos al clúster de equipos escribiendo scp $FASTA username@clusterlocation:/$DIR en una ventana de terminal local o utilice FileZilla para transferir archivos hacia y desde el equipo y el clúster.
- Búsqueda de genes candidatos utilizando BLAST+26.
- Instale o cargue BLAST+ v. 2.8.1 en el clúster de equipos.
- En el clúster de computadoras, haga una base de datos BLAST a partir de la proteína traducida por el genoma o el transcriptoma FASTA escribiendo [BLAST+ location]/makeblastdb -in $PEP. FASTA -dbtype prot -out $OUTPUT
- BLAST las secuencias de genes homólogos de NCBI a la base de datos de la especie de interés escribiendo [BLAST+ location]/blastp -db $DATABASE -query $FASTA -evalue 1e-10 -outfmt 6 -max_target_seqs 1 -out $OUTPUT.
- Vea el archivo de salida utilizando el comando more. Copie los identificadores de genes únicos de las especies de interés en un nuevo archivo de texto.
- Extraiga las secuencias de genes candidatos escribiendo perl -ne 'if(/^>(\S+)/){$c=$i{$1}}$c?print:chomp;$i{$_}=1 if @ARGV' $gene_id.txt $PEP. FASTA > $OUTPUT.
- Confirme la anotación de genes utilizando BLAST recíproco.
- En el navegador de Internet vaya a https://blast.ncbi.nlm.nih.gov/Blast.cgi.
- Seleccione tblastny, a continuación, pegue las secuencias candidatas, seleccione la base de datos de secuencias de proteínas no redundantes y haga clic en BLAST.
- Identifique genes adicionales anotando todos los genes en el genoma o transcriptoma con términos de ontología génica (GO) (ver discusión).
- Transfiera la proteína FASTA a la computadora local.
- Descargue e instale Blast2GO27,28,29 v. 5.2 en el equipo local.
- Abra Blast2GO, haga clic en Archivo, vaya a Cargar, vaya a Cargar secuencias, haga clic en Cargar archivo Fasta (fasta). Seleccione el archivo FASTA y haga clic en Cargar.
- Haga clic en Blast, elija NCBI Blasty haga clic en Next. Edite parámetros o haga clic en Siguiente, editar parámetros y haga clic en Ejecutar para encontrar la descripción genética más similar.
- Haga clic en mapeo y luego haga clic en Ejecutar para buscar anotaciones de ontología génica para proteínas similares.
- A continuación, haga clic en interpro, seleccione EMBL-EBI InterProy haga clic en Siguiente. Edite los parámetros o haga clic en Siguientey haga clic en Ejecutar para buscar firmas de dominios y familias de genes conocidos.
- Exporte las anotaciones haciendo clic en Archivo, seleccione Exportar, haga clic en Exportar tabla. Haga clic en Examinar, asigne un nombre al archivo, haga clic en Guardar, haga clic en Exportar.
- Busque en la tabla de anotaciones los términos de interés de GO para identificar genes candidatos adicionales. Extraer las secuencias del fichero FASTA (STEP 8.4.5)
9. Árboles filogenéticos
- Descargue e instale MEGA30 v. 7.0.26 en su computadora local.
- Abra MEGA, haga clic en Alinear, haga clic en Editar / Construir alineación, seleccione Crear una nueva alineación, haga clic en Aceptar, seleccione Proteína.
- Cuando se abra la ventana de alineación, haga clic en Editar, haga clic en Insertar secuencias desde archivo y seleccione el FASTA con secuencias de proteínas de genes candidatos y homólogos probables.
- Seleccione todas las secuencias. Encuentra el símbolo del brazo y pasa el cursor sobre él. Debería decir Alinear secuencias usando el algoritmo MUSCLE31. Haga clic en el símbolo del brazo y, a continuación, haga clic en Alinear proteína para alinear las secuencias. Edite los parámetros o haga clic en Aceptar para alinearlos con los parámetros predeterminados.
- Inspeccione visualmente y realice los cambios manuales y, a continuación, guarde y cierre la ventana de alineación.
- En la ventana principal de MEGA, haga clic en Modelos, haga clic en Buscar los mejores modelos de ADN / proteína (ML), seleccione el archivo de alineación y seleccione los parámetros correspondientes, tales como: Análisis: Selección de modelo (ML), Árbol a utilizar: Automático (árbol de unión de vecinos), Método estadístico: Máxima verosimilitud, Tipo de sustitución: Aminoácido, Espacio / tratamiento de datos faltantes: Usar todos los sitios, Filtro de sitio de sucursal: Ninguno.
- Una vez que se determina el mejor modelo para los datos, vaya a la ventana principal de MEGA. Haga clic en Filogenia y haga clic en Árbol de máxima verosimilitud de construcción/prueba y, a continuación, seleccione la alineación, si es necesario. Seleccione los parámetros apropiados para el árbol: Método estadístico: Máxima verosimilitud, Prueba de filogenia: Método Bootstrap con 100 réplicas, tipo de sustitución: aminoácido, modelo: LG con Freqs. (+F), tasas entre sitios: gamma distribuida (G) con 5 categorías gamma discretas, tratamiento de datos gap/missing: use all sites, método heurístico ML: Nearest-Neighbor-Interchange (NNI).
10. Visualizar la expresión génica usando TPM
- Para Trinity, en el clúster de computadoras, vaya al directorio donde se ejecutó abundance_estimates_to_matrix.pl y una de las salidas debe ser matrix. TPM.not_cross_norm. Transfiera este archivo al equipo local.
Nota: Consulte materiales suplementarios para la normalización de muestras cruzadas. - Para las MEDIDAS DE PROTECCIÓN de un análisis del genoma, siga los pasos a continuación.
- En el clúster de equipos, vaya a la ubicación de instalación de RSEM. Copie rsem-generate-data-matrix escribiendo scp rsem-generate-data-matrix rsem-generate-TPM-matrix. Use nano para editar el nuevo archivo y cambie "mi $offsite = 4" de 4 a 5 para TPM, ahora debe leer "mi $offsite = 5".
- Vaya al directorio donde están los archivos de salida RSEM .genes.results y ahora use rsem-generate-TPM-matrix *[genes/isoforms.results] > $OUTPUT para generar una matriz TPM. Transferir los resultados a un equipo local.
- Visualice los resultados en ggplot2.
- Descargue R v. 4.0.0 y RStudio v. 1.2.1335 en un equipo local.
- Abra RStudio a la derecha de la pantalla, vaya a la pestaña Paquetes y haga clic en Instalar. Escriba ggplot2 y haga clic en instalar.
- En la ventana de script de R, lea en la tabla tpm escribiendo data<-read.table("$tpm.txt",header = T)
- Para gráficos de barras similares a la Figura 4, escriba algo similar a: p<- ggplot() + geom_bar(aes(y=TPM, x=Symbol, fill=Tissue), data=data, stat="identity")
fill<-c("#d7191c","#fdae61", "#ffffbf", "#abd9e9", "#2c7bb6")
p<-p+scale_fill_manual(values=fill)
p + theme(axis.text.x = element_text(angle = 90))
Resultados
Los métodos anteriores se resumen en la Figura 1 y se aplicaron a un conjunto de datos de tejidos de Hydra vulgaris. H. vulgaris es un invertebrado de agua dulce que pertenece al filo Cnidaria que también incluye corales, medusas y anémonas de mar. H. vulgaris puede reproducirse asexualmente por gemación y pueden regenerar su cabeza y pie cuando están divididos en dos. En este estudio, el objetivo fue investigar la evolución y expresión de los genes de la opsina en Hydra7. Mientras que Hydra carece de ojos, exhiben un comportamiento dependiente de la luz32. Los genes opsina codifican proteínas que son importantes en la visión para detectar diferentes longitudes de onda de luz y comenzar la cascada de fototransducción. La investigación de la evolución molecular y la expresión de esta familia de genes en una especie basal puede proporcionar información sobre la evolución de los ojos y la detección de luz en animales.
Se generó un ensamblaje guiado utilizando el genoma de referencia hydra2.0 33 y datos de ARN-seq disponibles públicamente (geo accesión GSE127279) Figura 1. Este paso tomó aproximadamente 3 días. Aunque no generamos un transcriptoma de novo en este caso, un ensamblaje de Trinity puede tardar hasta 1 semana en generarse y cada biblioteca puede tardar unas horas en leer el mapeo dependiendo del mapeador. El ensamblaje de Hydra fusionado (~ 50,000 transcripciones) se anotó usando Blast2GO, que tomó aproximadamente 1 semana en la Figura 1. Las secuencias para los genes opsin-relacionados fueron extraídas en un archivo del fasta. Las secuencias para los genes de opsina de otras especies también se extrajeron de NCBI GenBank. Utilizamos opsinas de cnidarios Podocoryna carnea, Cladonema radiatum, Tripedelia cystophoray Nematostella vectensis,y también incluimos grupos de salida Mnemiopsis leidyi, Trichoplax adhaerens, Drosophila melanogaster y Homo sapiens. Los genes opsina se alinearon en mega7 figura 2. Al ver la alineación, pudimos identificar las opsinas de Hydra a las que les faltaba un aminoácido de lisina conservado necesario para unirse a una molécula sensible a la luz. Después de la inspección visual, determinamos el mejor modelo haciendo un análisis de selección de modelos. Generamos un árbol de máxima verosimilitud utilizando el modelo LG + G + F con un valor de arranque de 100 Figura 3. Para 149 genes de opsina, el árbol se terminó en aproximadamente 3 días. La filogenia sugiere que los genes de opsina están evolucionando por duplicaciones específicas del linaje en cnidarios y potencialmente por duplicación en tándem en H. vulgaris7.
Realizamos un análisis de expresión diferencial en edgeR y observamos la expresión absoluta de los genes de opsina. Presumimos que una o más opsinas serían upregulated en la cabeza (hypostome) y realizamos comparaciones par-sabias del hypostome contra la columna del cuerpo, la zona de florecimiento, el pie y los tentáculos. Como ejemplo de una comparación par-sabia, 1.774 transcripciones fueron expresadas diferenciado entre el hypostome y la columna del cuerpo. Determinamos los genes que fueron upregulated a través de comparaciones múltiples e hicimos un enriquecimiento funcional en Blast2GO Tabla 1. La agrupación de la actividad acoplada G-proteína del receptor incluyó genes de la opsina. Finalmente, observamos la expresión absoluta de los genes de opsina en diferentes tejidos, durante la brotación y durante la regeneración, trazando sus valores de TPM utilizando ggplot Figura 4. Usando los métodos descritos aquí, identificamos 2 genes de opsina que no se agruparon con las otras opsinas en la filogenia, encontramos una opsina que se expresó casi 200 veces más que otros, y encontramos algunos genes de opsina co-expresados con genes de fototransducción que pueden ser utilizados para la detección de luz.
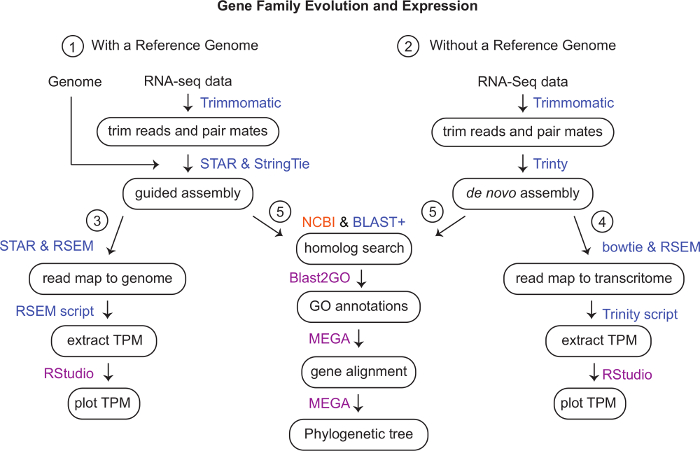
Figura 1: Esquema de flujo de trabajo. Los programas utilizados para analizar datos en el clúster de computadoras están en azul, en magenta son los que usamos en una computadora local y en naranja es un programa basado en la web. (1) Recorte RNA-seq lee usando trimmomatic v. 0.35. Si hay un genoma disponible pero faltan modelos genéticos, genere un ensamblaje guiado usando STAR v. 2.6.0c y StringTie v. 1.3.4d. (Opcional ver Materiales Suplementarios) (2) Sin un genoma de referencia, utilice lecturas recortadas para hacer un ensamblaje de novo usando Trinity v 2.8.5. (3) Para cuantificar la expresión génica utilizando un genoma de referencia, mapear las lecturas utilizando STAR y cuantificar usando RSEM v. 1.3.1. Extraiga los TPM mediante RSEM y visualícelos en RStudio. (4) Bowtie y RSEM se pueden utilizar para mapear y cuantificar las lecturas mapeadas a un transcriptoma de trinidad. A Trinity script can be used to generate a TPM matrix to visualize counts in RStudio. (5) Utilice NCBI BLAST basado en la web y BLAST+ de línea de comandos para buscar secuencias homólogas y confirmar el uso de BLAST recíproco. Anote los genes aún más usando Blast2GO. Utilice MEGA para alinear genes y generar un árbol filogenético utilizando el modelo de mejor ajuste. Haga clic aquí para ver una versión más amplia de esta figura.
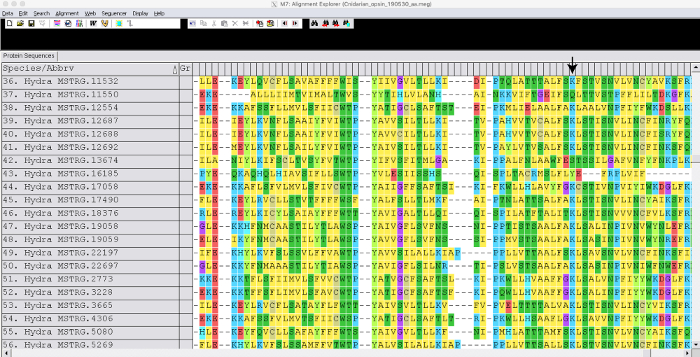
Figura 2: Ejemplo de genes alineados. La instantánea muestra una porción de los genes de la opsina de Hydra alineados usando MUSCLE. La flecha indica la ubicación de una lisina conservada de unión a la retina. Haga clic aquí para ver una versión más amplia de esta figura.
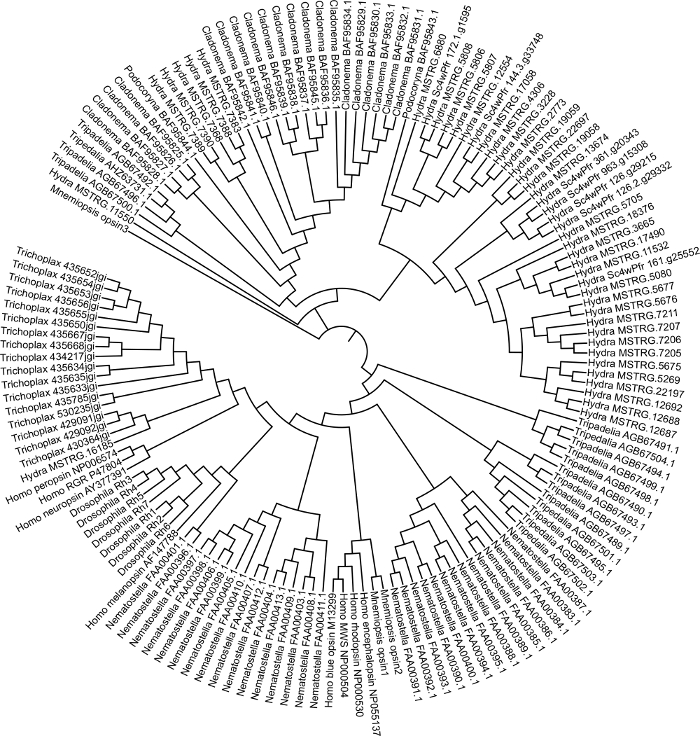
Figura 3: Árbol filogenético de opsina cnidaria. Árbol de máxima verosimilitud generado en MEGA7 utilizando secuencias de opsina de Hydra vulgaris, Podocoryna carnea, Cladonema radiatum, Tripedelia cystophora, Nematostella vectensis, Mnemiopsis leidyi, Trichoplax adhaerens, Drosophila melanogaster y Homo sapiens. Haga clic aquí para ver una versión más amplia de esta figura.
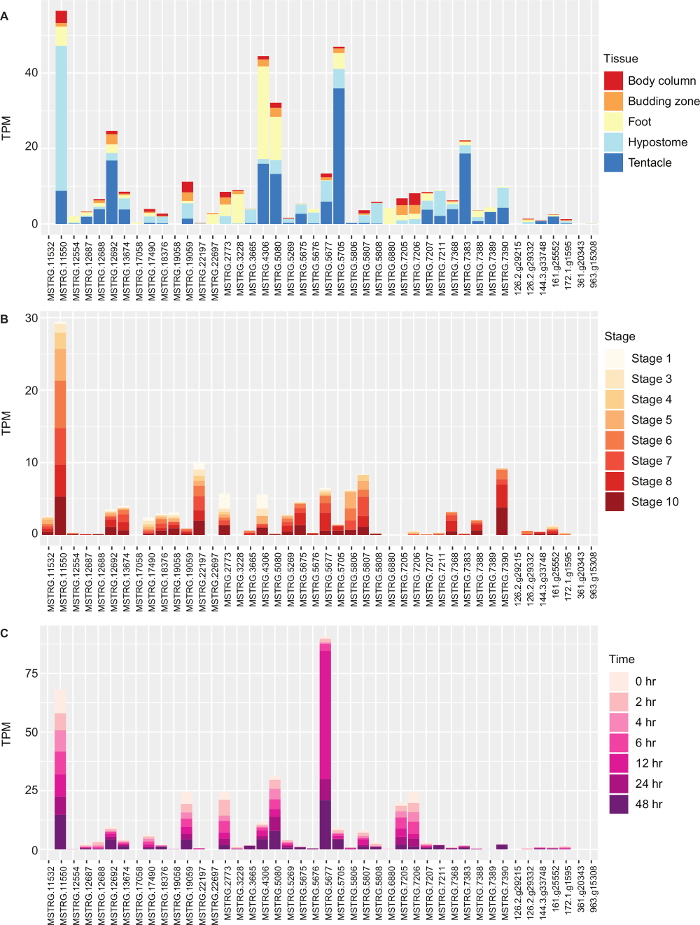
Figura 4: Expresión de los genes opsina en Hydra vulgaris. (A) Expresión en transcripciones por millón (TPM) de los genes de la opsina hydra vulgaris en la columna del cuerpo, zona de gemación, pie, hipostoma y tentáculos. (B) Expresión de genes de opsina durante diferentes etapas de la gemación de Hydra. (C) Expresión de genes opsina del hipostoma de Hydra durante diferentes puntos de tiempo de regeneración. Haga clic aquí para ver una versión más amplia de esta figura.
| Id. de go | Nombre go | Categoría GO | Fdr |
| IR:0004930 | Actividad del receptor acoplado a la proteína G | FUNCIÓN MOLECULAR | 0.0000000000704 |
| IR:0007186 | Vía de señalización del receptor acoplado a proteínas G | PROCESO BIOLÓGICO | 0.00000000103 |
| IR:0016055 | Vía de señalización Wnt | PROCESO BIOLÓGICO | 0.0000358 |
| IR:0051260 | homooligomerización de proteínas | PROCESO BIOLÓGICO | 0.000376 |
| IR:0004222 | actividad metaloendopeptidasa | FUNCIÓN MOLECULAR | 0.000467 |
| IR:0008076 | complejo de canal de potasio bloqueado por voltaje | COMPONENTE CELULAR | 0.000642 |
| IR:0005249 | actividad del canal de potasio bloqueado por voltaje | FUNCIÓN MOLECULAR | 0.00213495 |
| IR:0007275 | desarrollo de organismos multicelulares | PROCESO BIOLÓGICO | 0.00565048 |
| IR:0006813 | transporte de iones de potasio | PROCESO BIOLÓGICO | 0.01228182 |
| IR:0018108 | fosforilación de peptidil-tirosina | PROCESO BIOLÓGICO | 0.02679662 |
Tabla 1: Enriquecimiento funcional de genes upregulated en el hipostoma
Materiales suplementarios. Haga clic aquí para descargar estos materiales.
Discusión
El propósito de este protocolo es proporcionar un contorno de los pasos para caracterizar a una familia del gene usando datos del ARN-seq. Se ha demostrado que estos métodos funcionan para una variedad de especies y conjuntos de datos4,34,35. La tubería establecida aquí se ha simplificado y debería ser lo suficientemente fácil como para ser seguida por un novato en bioinformática. La importancia del protocolo es que describe todos los pasos y programas necesarios para completar un análisis publicable. Un paso crucial en el protocolo es tener transcripciones de longitud completa correctamente ensambladas, esto proviene de genomas o transcriptomas de alta calidad. Para obtener transcripciones adecuadas, se necesita ARN y/o ADN de alta calidad y buenas anotaciones que se analizan a continuación.
Para la preparación de la biblioteca de ARN-seq, incluimos kits de lista que funcionaron para pequeñas partes del cuerpo de Hydra19 y mariposas18 (Tabla de Materiales). Observamos que para el ARN de entrada baja utilizamos un acercamiento modificado del protocolo36. Los métodos para la extracción de ARN se han comparado en múltiples tipos de muestras, incluidas las células de levadura17,el neuroblastoma 37,las plantas38y las larvas de insectos16, por nombrar algunas. Recomendamos al lector adquirir un protocolo que funcione para sus especies de interés, si existe alguna, o solucionar problemas utilizando kits comúnmente disponibles comercialmente para comenzar. Para la cuantificación adecuada de genes, recomendamos tratar la muestra de ARN con DNasa. La presencia de ADN afectará la cuantificación adecuada de los genes. También recomendamos usar un kit de preparación de la biblioteca de ADNc que incluya una selección de cola de poliA para seleccionar el ARNm maduro. Mientras que el agotamiento del ARNr resulta en una mayor profundidad de lectura, el porcentaje de cobertura de exones es mucho menor que la cobertura de exones del ARN utilizando la selección de poliA+39. Por último, cuando sea posible lo mejor es utilizar pareado-extremo y varado40,41. En el protocolo anterior, los comandos read mapping tendrán que modificarse cuando se utilicen lecturas de extremo único.
Como se mencionó anteriormente, es importante poder identificar genes de interés y también diferenciar entre duplicaciones genéticas recientes, empalme alternativo y haplotipos en la secuenciación. En algunos casos, tener un genoma de referencia puede ayudar al determinar dónde se encuentran los genes y los exones en relación entre sí. Una cosa a tener en cuenta es que si un transcriptoma se obtiene de una base de datos pública y no es de alta calidad, puede ser mejor generar usando Trinity42 y combinando bibliotecas de ARN-seq de tejidos de interés. Del mismo modo, si un genoma de referencia no tiene buenos modelos de genes, las bibliotecas de ARN-seq se pueden utilizar para generar nuevos GTF utilizando StringTie43 (ver Materiales Suplementarios). Además, en los casos en que los genes están incompletos y hay acceso a un genoma, los genes se pueden editar manualmente utilizando secuencias homólogas y luego alinearse con el genoma usando tblastn. La salida BLAST se puede utilizar para determinar la secuencia real, que puede ser diferente de la corrección realizada mediante homólogos. Si no hay ninguna coincidencia, deje la secuencia como estaba originalmente. Al comprobar la salida, preste atención a las coordenadas del genoma para asegurarse de que el exón que falta es de hecho parte del gen.
Aunque nos centramos en el software y los programas que utilizamos, existen modificaciones en este protocolo debido a muchos programas disponibles que podrían funcionar mejor para diferentes conjuntos de datos. Como ejemplo, mostramos comandos para mapear lecturas al transcriptoma usando pajarita y RSEM, pero Trinity ahora tiene la opción de alineadores mucho más rápidos como kallisto44 y salmon45. Del mismo modo, describimos las anotaciones usando Blast2GO (ahora OmicsBox) pero hay otras herramientas de mapeador que se pueden encontrar de forma gratuita y en línea. Algunos que hemos probado incluyen: GO FEAT46,eggNOG-mapper47,48,y un alineador muy rápido PANNZER249. Para utilizar estas herramientas de anotación basadas en la web simplemente cargue el péptido FASTA y envíelo. Las versiones independientes de PANNZER y eggNOG-mapper también están disponibles para ser descargadas en el clúster de computadoras. Otra modificación es que usamos MEGA y R en una computadora local y usamos la herramienta EN LÍNEA NCBI BLAST para hacer BLASTs recíprocos, sin embargo, todos estos programas se pueden usar en el clúster de computadoras descargando los programas y bases de datos necesarios. Del mismo modo, los alineadores kallisto y salmon se pueden usar en un equipo local siempre y cuando un usuario tenga suficiente RAM y almacenamiento. Sin embargo, los archivos FASTQ y FASTA tienden a ser muy grandes y recomendamos encarecidamente el uso de un clúster de computadoras para facilitar y acelerar. Además, si bien proporcionamos instrucciones y enlaces para descargar programas de sus desarrolladores, muchos de ellos se pueden instalar desde bioconda: https://anaconda.org/bioconda.
Un problema común que se enfrenta al hacer análisis bioinformáticos es el error de los scripts de shell. Esto puede deberse a una variedad de razones. Si se crea un archivo de error, estos archivos de error deben comprobarse antes de solucionar el problema. Algunas razones comunes para un error son errores tipográficos, falta de parámetros clave y problemas de compatibilidad entre versiones de software. En este protocolo, incluimos parámetros para los datos, pero los manuales de software pueden proporcionar directrices más detalladas para parámetros individuales. En general, lo mejor es utilizar las versiones más actualizadas del software y consultar el manual correspondiente a esa versión.
Las mejoras a este protocolo incluyen hacer un análisis de expresión diferencial de todo el transcriptoma y un análisis de enriquecimiento funcional. Recomendamos edgeR50 para el análisis de expresión diferencial un paquete disponible en Bioconductor. Para el análisis de enriquecimiento funcional, hemos utilizado Blast2GO29 y DAVID51,52basado en la web. También recomendamos seguir editando la filogenia extrayendola como un archivo newick y usando iTOL53basado en la web. Además, si bien este protocolo investigará la evolución molecular y los patrones de expresión de los genes, se pueden utilizar experimentos adicionales para validar las ubicaciones y funciones de genes o proteínas. La expresión del mRNA se puede confirmar por RT-qPCR o el hibridación in situ. Las proteínas se pueden localizar usando immunohistochemistry. Dependiendo de la especie, los experimentos knockout se pueden utilizar para confirmar la función del gen. Este protocolo puede ser utilizado para una variedad de objetivos incluyendo, como se muestra arriba, para explorar una familia de genes típicamente asociada con la fotorrecepción en una especie basal7. Otra aplicación de estos métodos es identificar cambios en una vía conservada bajo diferentes presiones selectivas. Como ejemplo, estos métodos se utilizaron para descubrir la variación en la expresión de los canales potenciales del receptor transitorio de la visión entre las mariposas diurnas y las polillas nocturnas34.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos a Adriana Briscoe, Gil Smith, Rabi Murad y Aline G. Rangel por su asesoramiento y orientación para incorporar algunos de estos pasos en nuestro flujo de trabajo. También estamos agradecidos a Katherine Williams, Elisabeth Rebboah y Natasha Picciani por los comentarios sobre el manuscrito. Este trabajo fue apoyado en parte por una beca de investigación médica de la Fundación George E. Hewitt para A.M.M.
Materiales
| Name | Company | Catalog Number | Comments |
| Bioanalyzer-DNA kit | Agilent | 5067-4626 | wet lab materials |
| Bioanalyzer-RNA kit | Agilent | 5067-1513 | wet lab materials |
| BLAST+ v. 2.8.1 | On computer cluster* https://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/ | ||
| Blast2GO (on your PC) | On local computer https://www.blast2go.com/b2g-register-basic | ||
| boost v. 1.57.0 | On computer cluster | ||
| Bowtie v. 1.0.0 | On computer cluster https://sourceforge.net/projects/bowtie-bio/files/bowtie/1.3.0/ | ||
| Computing cluster (highly recommended) | NOTE: Analyses of genomic data are best done on a high-performance computing cluster because files are very large. | ||
| Cufflinks v. 2.2.1 | On computer cluster | ||
| edgeR v. 3.26.8 (in R) | In Rstudio https://bioconductor.org/packages/release/bioc/html/edgeR.html | ||
| gcc v. 6.4.0 | On computer cluster | ||
| Java v. 11.0.2 | On computer cluster | ||
| MEGA7 (on your PC) | On local computer https://www.megasoftware.net | ||
| MEGAX v. 0.1 | On local computer https://www.megasoftware.net | ||
| NucleoSpin RNA II kit | Macherey-Nagel | 740955.5 | wet lab materials |
| perl 5.30.3 | On computer cluster | ||
| python | On computer cluster | ||
| Qubit 2.0 Fluorometer | ThermoFisher | Q32866 | wet lab materials |
| R v.4.0.0 | On computer cluster https://cran.r-project.org/src/base/R-4/ | ||
| RNAlater | ThermoFisher | AM7021 | wet lab materials |
| RNeasy kit | Qiagen | 74104 | wet lab materials |
| RSEM v. 1.3.0 | Computer software https://deweylab.github.io/RSEM/ | ||
| RStudio v. 1.2.1335 | On local computer https://rstudio.com/products/rstudio/download/#download | ||
| Samtools v. 1.3 | Computer software | ||
| SRA Toolkit v. 2.8.1 | On computer cluster https://github.com/ncbi/sra-tools/wiki/01.-Downloading-SRA-Toolkit | ||
| STAR v. 2.6.0c | On computer cluster https://github.com/alexdobin/STAR | ||
| StringTie v. 1.3.4d | On computer cluster https://ccb.jhu.edu/software/stringtie/ | ||
| Transdecoder v. 5.5.0 | On computer cluster https://github.com/TransDecoder/TransDecoder/releases | ||
| Trimmomatic v. 0.35 | On computer cluster http://www.usadellab.org/cms/?page=trimmomatic | ||
| Trinity v.2.8.5 | On computer cluster https://github.com/trinityrnaseq/trinityrnaseq/releases | ||
| TRIzol | ThermoFisher | 15596018 | wet lab materials |
| TruSeq RNA Library Prep Kit v2 | Illumina | RS-122-2001 | wet lab materials |
| TURBO DNA-free Kit | ThermoFisher | AM1907 | wet lab materials |
| *Downloads and installation on the computer cluster may require root access. Contact your network administrator. |
Referencias
- Lespinet, O., Wolf, Y. I., Koonin, E. V., Aravind, L. The role of lineage-specific gene family expansion in the evolution of eukaryotes. Genome Research. 12 (7), 1048-1059 (2002).
- Gabaldón, T., Koonin, E. V. Functional and evolutionary implications of gene orthology. Nature Reviews Genetics. 14 (5), 360-366 (2013).
- Dolinski, K., Botstein, D. Orthology and Functional Conservation in Eukaryotes. Annual Review of Genetics. 41 (1), (2007).
- Macias-Muñoz, A., McCulloch, K. J., Briscoe, A. D. Copy number variation and expression analysis reveals a non-orthologous pinta gene family member involved in butterfly vision. Genome Biology and Evolution. 9 (12), 3398-3412 (2017).
- Cannon, S. B., Mitra, A., Baumgarten, A., Young, N. D., May, G. The roles of segmental and tandem gene duplication in the evolution of large gene families in Arabidopsis thaliana. BMC plant biology. 4, 10 (2004).
- Eastman, S. D., Chen, T. H. P., Falk, M. M., Mendelson, T. C., Iovine, M. K. Phylogenetic analysis of three complete gap junction gene families reveals lineage-specific duplications and highly supported gene classes. Genomics. 87 (2), 265-274 (2006).
- Macias-Munõz, A., Murad, R., Mortazavi, A. Molecular evolution and expression of opsin genes in Hydra vulgaris. BMC Genomics. 20 (1), 1-19 (2019).
- Hisatomi, O., Tokunaga, F. Molecular evolution of proteins involved in vertebrate phototransduction. Comparative Biochemistry and Physiology - B Biochemistry and Molecular Biology. 133 (4), 509-522 (2002).
- Arendt, D. Evolution of eyes and photoreceptor cell types. International Journal of Developmental Biology. 47, 563-571 (2003).
- Shichida, Y., Matsuyama, T. Evolution of opsins and phototransduction. Philosophical Transactions of the Royal Society B: Biological Sciences. 364 (1531), 2881-2895 (2009).
- Porter, M. L., et al. Shedding new light on opsin evolution. Proceedings of the Royal Society B: Biological Sciences. 279 (1726), 3-14 (2012).
- Plachetzki, D. C., Degnan, B. M., Oakley, T. H. The origins of novel protein interactions during animal opsin evolution. PLoS ONE. 2 (10), 1054 (2007).
- Ramirez, M. D., et al. The last common ancestor of most bilaterian animals possessed at least nine opsins. Genome Biology and Evolution. 8 (12), 3640-3652 (2016).
- Schnitzler, C. E., et al. Genomic organization, evolution, and expression of photoprotein and opsin genes in Mnemiopsis leidyi: a new view of ctenophore photocytes. BMC Biology. 10, 107 (2012).
- Pedersen, K. B., Williams, A., Watt, J., Ronis, M. J. Improved method for isolating high-quality RNA from mouse bone with RNAlater at room temperature. Bone Reports. 11, 100211 (2019).
- Ridgeway, J. A., Timm, A. E., Fallon, A. Comparison of RNA isolation methods from insect larvae. Journal of Insect Science. 14 (1), 4-8 (2014).
- Scholes, A. N., Lewis, J. A. Comparison of RNA isolation methods on RNA-Seq: Implications for differential expression and meta-Analyses. BMC Genomics. 21 (1), 1-9 (2020).
- Briscoe, A. D., et al. Female behaviour drives expression and evolution of gustatory receptors in butterflies. PLoS genetics. 9 (7), 1003620 (2013).
- Murad, R., Macias-Muñoz, A., Wong, A., Ma, X., Mortazavi, A. Integrative analysis of Hydra head regeneration reveals activation of distal enhancer-like elements. bioRxiv. , 544049 (2019).
- Gallego Romero, I., Pai, A. A., Tung, J., Gilad, Y. Impact of RNA degradation on measurements of gene expression. BMC Biology. 12, 42 (2014).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Trinity. . RNA-Seq De novo Assembly Using Trinity. , 1-7 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29, 15-21 (2013).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC bioinformatics. 12, 323 (2011).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome biology. 10, 25 (2009).
- Camacho, C., et al. BLAST+: architecture and applications. BMC Bioinformatics. 10, 421 (2009).
- Conesa, A., Götz, S. Blast2GO: A comprehensive suite for functional analysis in plant genomics. International Journal of Plant Genomics. 619832, (2008).
- Conesa, A., et al. Blast2GO: A universal tool for annotation, visualization and analysis in functional genomics research. Bioinformatics. 21 (18), 3674-3676 (2005).
- Götz, S., et al. High-throughput functional annotation and data mining with the Blast2GO suite. Nucleic Acids Research. 36 (10), 3420-3435 (2008).
- Kumar, S., Stecher, G., Tamura, K. MEGA7: Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets. Molecular biology and evolution. 33 (7), 1870-1874 (2016).
- Edgar, R. C. MUSCLE: Multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Research. 32 (5), 1792-1797 (2004).
- Taddei-Ferretti, C., Musio, C., Santillo, S., Cotugno, A. The photobiology of Hydra's periodic activity. Hydrobiologia. 530, 129-134 (2004).
- Chapman, J. A., et al. The dynamic genome of Hydra. Nature. 464 (7288), 592-596 (2010).
- Macias-Muñoz, A., Rangel Olguin, A. G., Briscoe, A. D. Evolution of phototransduction genes in Lepidoptera. Genome Biology and Evolution. 11 (8), 2107-2124 (2019).
- Macias-Munõz, A., Murad, R., Mortazavi, A. Molecular evolution and expression of opsin genes in Hydra vulgaris. BMC Genomics. 20 (1), (2019).
- Picelli, S., et al. Full-length RNA-seq from single cells using Smart-seq2. Nature Protocols. 9 (1), 171-181 (2014).
- Tavares, L., Alves, P. M., Ferreira, R. B., Santos, C. N. Comparison of different methods for DNA-free RNA isolation from SK-N-MC neuroblastoma. BMC research notes. 4, 3 (2011).
- Johnson, M. T. J., et al. Evaluating Methods for Isolating Total RNA and Predicting the Success of Sequencing Phylogenetically Diverse Plant Transcriptomes. PLoS ONE. 7 (11), (2012).
- Zhao, S., Zhang, Y., Gamini, R., Zhang, B., Von Schack, D. Evaluation of two main RNA-seq approaches for gene quantification in clinical RNA sequencing: PolyA+ selection versus rRNA depletion. Scientific Reports. 8 (1), 1-12 (2018).
- Zhao, S., et al. Comparison of stranded and non-stranded RNA-seq transcriptome profiling and investigation of gene overlap. BMC Genomics. 16 (1), 1-14 (2015).
- Corley, S. M., MacKenzie, K. L., Beverdam, A., Roddam, L. F., Wilkins, M. R. Differentially expressed genes from RNA-Seq and functional enrichment results are affected by the choice of single-end versus paired-end reads and stranded versus non-stranded protocols. BMC Genomics. 18 (1), 1-13 (2017).
- Haas, B. J., et al. De novo transcript sequence reconstruction from RNA-seq using the Trinity platform for reference generation and analysis. Nature Protocols. 8 (8), 1494-1512 (2013).
- Pertea, M., et al. StringTie enables improved reconstruction of a transcriptome from RNA-seq reads. Nature biotechnology. 33 (3), 290-295 (2015).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nature Biotechnology. 34 (5), 525-527 (2016).
- Patro, R., Duggal, G., Love, M. I., Irizarry, R. A., Kingsford, C. Salmon provides fast and bias-aware quantification of transcript expression. Nature Methods. 14 (4), 417-419 (2017).
- Araujo, F. A., Barh, D., Silva, A., Guimarães, L., Thiago, R. . OPEN GO FEAT a rapid web-based functional annotation tool for genomic and transcriptomic data. , 8-11 (2018).
- Huerta-Cepas, J., et al. Fast genome-wide functional annotation through orthology assignment by eggNOG-mapper. Molecular Biology and Evolution. 34 (8), 2115-2122 (2017).
- Huerta-Cepas, J., et al. EggNOG 5.0: A hierarchical, functionally and phylogenetically annotated orthology resource based on 5090 organisms and 2502 viruses. Nucleic Acids Research. 47, 309-314 (2019).
- Törönen, P., Medlar, A., Holm, L. PANNZER2: A rapid functional annotation web server. Nucleic Acids Research. 46, 84-88 (2018).
- Robinson, M., Mccarthy, D., Chen, Y., Smyth, G. K. . edgeR differential expression analysis of digital gene expression data User's Guide. , (2013).
- Huang, D. W., Sherman, B. T., Lempicki, R. A. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources. Nature Protocols. 4 (1), 44-57 (2009).
- Huang, D. W., Sherman, B. T., Lempicki, R. A. Bioinformatics enrichment tools: Paths toward the comprehensive functional analysis of large gene lists. Nucleic Acids Research. 37 (1), 1-13 (2009).
- Letunic, I., Bork, P. Interactive tree of life (iTOL) v3: an online tool for the display and annotation of phylogenetic and other trees. Nucleic acids research. 44, 242-245 (2016).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados