Method Article
使用RNA-seq研究分子进化和基因表达的生物信息学管道
摘要
本协议的目的是使用RNA测序数据调查候选基因的进化和表达。
摘要
蒸馏和报告大型数据集(如全基因组或转录组数据)往往是一项艰巨的任务。分解结果的一种方法是关注一个或多个对生物体和研究具有重要意义的基因家族。在此协议中,我们概述了生物信息学步骤,以生成植物学并量化感兴趣的基因表达。植物遗传树可以深入了解基因在物种内部和物种之间是如何进化的,并揭示正学。这些结果可以使用RNA-seq数据来比较这些基因在不同个体或组织中的表达。分子进化和表达的研究可以揭示物种间基因功能的进化和保存模式。基因家族的特征可以作为未来研究的跳板,并能在新的基因组或转录纸中突出一个重要的基因家族。
引言
测序技术的进步促进了非模型生物基因组和转录组的测序。除了从许多生物体中测序DNA和RNA的可行性增加外,还有大量数据可供公开研究感兴趣的基因。本议定书的目的是提供生物信息学步骤,以研究基因的分子进化和表达,这些基因可能在感兴趣的有机体中发挥重要作用。
研究基因或基因家族的进化可以深入了解生物系统的进化。基因家族的成员通常通过识别保存的图案或同源基因序列来确定。基因家族进化以前是利用来自遥远相关模型生物体1的基因组进行研究的。这种方法的一个局限性是,不清楚这些基因家族是如何在密切相关的物种中进化的,以及不同环境选择性压力的作用。在此协议中,我们包括在密切相关的物种中搜索同源物种。通过在植物水平上生成植物,我们可以注意到基因家族进化的趋势,如保存的基因或特定于血统的复制。在这个水平上,我们也可以调查基因是正石还是对等体。虽然许多同源可能彼此类似,但情况不一定如此。在这些研究中加入植物遗传树对于确定这些同源基因是否是正交者非常重要。在真核生物中,许多矫形器在细胞内保留着类似的功能,哺乳动物蛋白质恢复酵母组织细胞3的功能的能力就证明了这一点。然而,在某些情况下,非正直面基因具有特征功能4。
植物树开始描绘基因和物种之间的关系,但功能不能仅仅根据遗传关系来分配。基因表达研究与功能注释和富集分析相结合,为基因功能提供了强有力的支持。基因表达可以跨个体或组织类型进行量化和比较的案例可以更能说明潜在的功能。以下协议遵循的方法,用于研究在海德拉粗俗7的蛋白基因,但他们可以应用于任何物种和任何基因家族。这些研究的结果为进一步研究非模型生物的基因功能和基因网络奠定了基础。例如,对蛋白的植物学研究,这些蛋白是引发光转移级联的蛋白质,为眼睛和光检测的进化提供了背景。在这种情况下,非模型生物,特别是基础动物物种,如神经元或细胞,可以阐明保护或变化的光转移级联和视觉跨越包12,13,14。同样,确定其他基因家族的植物学、表达和网络将告诉我们适应背后的分子机制。
研究方案
该协议遵循加州大学欧文分校的动物护理指南。
1. RNA-塞克图书馆准备
- 使用以下方法分离RNA。
- 收集样本。如果RNA要在以后提取,闪光冻结样品或放置在RNA存储解决方案15(材料表)。
- 安乐死和解剖生物体,以分离感兴趣的组织。
- 使用提取套件提取总RNA,使用RNA净化套件(材料表)净化RNA
注:有协议和工具包,可能更好地适用于不同的物种和组织类型16,17。我们已经从蝴蝶18和明胶海德拉19的不同身体组织提取RNA(见讨论)。 - 测量每个样品(材料表)的RNA浓度和质量。使用RNA完整性编号(RIN)高于8的样本,最好接近920 来构建cDNA库。
- 构建 cDNA 库并按以下顺序排列。
- 根据库准备说明手册构建 cDNA 库(参见讨论)。
- 确定 cDNA 浓度和质量 (材料表)。
- 将库多路复用并排序。
2. 访问计算机群集
注:RNA-seq 分析需要操作大型文件,最好在计算机集群(材料表)上完成。
- 使用终端 (Mac) 或 PuTTY (Windows) 应用窗口上的命令 ssh username@clusterlocation 登录计算机集群帐户。
3. 获取RNA-塞克读数
- 从测序设施获取RNA-seq读取,或者,对于出版物中生成的数据,从存放数据库(3.2 或 3.3)获取 RNA-seq 读数。
- 要从阵列快报等存储库下载数据,请执行以下工作:
- 使用加入号码搜索网站。
- 查找下载数据的链接,然后左键单击并选择 "复制链接"。
- 在终端窗口上,键入 wget 并选择 粘贴链接 以将数据复制到目录进行分析。
- 要下载 NCBI 短读存档 (SRA) 数据,请遵循以下替代步骤:
- 在终端下载SRA工具包诉2.8.1使用wget。
注意:下载和安装计算机集群的程序可能需要根访问,如果安装失败,请联系计算机集群管理员。 - 通过键入 焦油-xvf$TARGZFILE完成程序的安装。
- 搜索NCBI的SRA加入号码的样品,你想下载,它应该有格式SRRXX。
- 通过在终端窗口中键入 [sratoolkit 位置]/bin/预装 SRRXXXX 来获取 RNA-seq 数据。
- 对于配对端文件类型 [sratoolkit 位置]/bin/快速转储 - 拆分文件 SRRXXXXXX 以获取两个快速文件(SRRXXXXXX_1.FASTQ 和SRRXXXXXX_2.FASTQ)。
注意:要进行三位一体 的 novo 装配,请使用命令 [sratoolkit 位置]/bin/fastq-dump - 定义 -seq "@$sn]]/$ri" - 拆分文件 SRRXXXX
- 在终端下载SRA工具包诉2.8.1使用wget。
4. 修剪适配器和低质量读数(可选)
- 在计算集群上安装或加载修剪21 诉 0.35。
- 在 RNA-seq 数据文件所在的目录中,键入一个命令,包括修剪罐文件的位置、输入 FASTQ 文件、输出 FASTQ 文件以及读取长度和质量等可选参数。
注:命令将因原始和所需的读取质量和长度而异。对于 Illumina 43 bp 读取与 Nextera 引物, 我们使用: java - jar / 数据 / 应用程序 / 修剪 / 0.35 / 修剪 - 0.35.jar PE $READ 1 。快速Q$READ 2。快速paired_READ1。快速unpaired_READ1。快速paired_READ2。快速unpaired_READ2。FASTQ 照明:适配器.fa:2:30:10 领先:20 落后:20 滑动窗口:4:17 分钟:30。
5. 获取参考组件
- 搜索谷歌,恩塞姆布尔基因组,和NCBI基因组和核苷酸TSA(脚本猎枪组装)的参考基因组或组装转录的感兴趣的物种(图1)。
注意:如果参考基因组或转录组不可用或质量低劣,请转到步骤 6 生成 de novo 组装。 - 如果存在参考基因组或组装的转录组,请将其下载为快速文件,以便按照以下步骤进行分析。
- 查找下载基因组的链接,左键单击和 复制链接。
- 在终端窗口类型上 获取 并粘贴链接地址。如果可用,也复制 GTF 文件和蛋白质 FASTA 文件作为参考基因组。
6. 生成 无组 件(第 5 步的替代方案)
- 通过键入猫*READ1,将RNA-seq READ1和READ2快速q文件组合在一起,用于所有样本。快速Q> $all_READ1。快速Q和猫*阅读2。快速> all_READ2。终端窗口上的快速Q。
- 在计算集群上安装或加载三位一 体 22 v.2.8.5。
- 通过在终端上键入生成和组装: 三一 - seqType fq - max_memory 20G - 左$all_READ1。快速Q - 右$all_READ2。快速Q。
7. 地图读取基因组 (7.1) 或 de novo 转录体 (7.2)
- 地图使用 STAR23 v. 2.6.0c 和 RSEM24 v. 1.3.0 读取参考基因组。
- 安装或加载 STAR 诉 2.6.0c.和RSEM诉1.3.0到计算集群。
- 通过键入 rsem 准备参考 - gtf $GENOME来索引基因组。GTF -星-p 16$GENOME。法斯塔$OUTPUT
- 地图通过键入 rsem 计算表达 -p 16 - 星 - 配对端$READ 1 来读取和计算每个示例的表达式。快速Q$READ 2。快速$INDEX $OUTPUT。
- 使用mv RSEM.genes.结果$sample.基因.结果将结果文件重命名为具有描述性的东西。
- 通过键入 rsem 生成数据矩阵 *[基因/等形结果.结果] > $OUTPUT生成所有计数的矩阵。
- 使用 RSEM 和蝴蝶结将 RNA-seq 映射到 三一德诺沃 装配。
- 安装或加载三位一体22 v.2.8.5、鲍蒂25 诉 1.0.0 和 RSEM v. 1.3.0。
- 地图通过键入 [trinity_location] /align_and_estimate_abundance.pl - 准备参考 - 脚本$TRINITY来读取和计算每个示例的表达式。法斯塔 -塞克类型fq-左$READ 1。快速Q -右$READ 2。快速 - est_method Rsem - aln_method蝴蝶结 - trinity_mode - output_dir $OUTPUT。
- 使用mv RSEM.genes.结果$sample.基因.结果将结果文件重命名为具有描述性的东西。
- 通过键入 [trinity_location]/abundance_estimates_to_matrix.pl 生成所有计数的矩阵 - est_method RSEM *[基因/等形]。
8. 识别感兴趣的基因
注意:以下步骤可以与核苷酸或蛋白质FASTA文件一起完成,但工作最好,并且对蛋白质序列更直接。使用蛋白质对蛋白质进行爆炸搜索更有可能在不同物种之间搜索时给出结果。
- 对于参考基因组,请使用 STEP 5.2.2 中的蛋白质 FASTA 文件或查看补充材料生成自定义基因特征 GTF。
- 对于 一个无 转录机,使用转编码器生成蛋白质FASTA。
- 在计算机粘贴机上安装或加载转解编码器 v. 5.5.0。
- 通过键入 [转译器位置]/转译器.LongOrfs-t $TRINITY,找到最长的开放读取帧并预测肽序列。法斯塔
- 搜索NCBI基因库,寻找密切相关物种中的同源物种。
- 打开互联网浏览器窗口,转到 https://www.ncbi.nlm.nih.gov/genbank/。
- 在搜索栏上键入感兴趣的基因的名称和已测序的密切相关物种的名称或属或植物。在搜索栏的左侧选择蛋白质,然后单击搜索。
- 通过单击 "发送"然后 选择 "文件" 提取序列。在格式下,选择 FASTA 然后单击 "创建文件"。
- 通过在本地终端窗口上键入 scp $FASTA username@clusterlocation/$DIR, 或使用 FileZilla 将文件传输到计算机和集群,将同源文件移动到计算机群集。
- 使用 BLAST+26搜索候选基因。
- 在计算机集群上安装或加载 BLAST® v. 2.8.1。
- 在计算机群集上,通过键入 [BLAST]位置]/使blastdb-在$PEP中,从基因组或转录组翻译的蛋白质FASTA中建立一个BLAST数据库。法斯塔 -db 型原型 - 出$OUTPUT
- 通过键入 [BLAST] 位置] / 爆炸 - db $DATABASE - 查询 $FASTA - 价值 1e-10 - outfmt 6 - max_target_seqs 1 - 出$OUTPUT,将 NCBI 的同源基因序列爆炸到感兴趣的物种数据库中。
- 使用更多命令查看输出文件。将感兴趣的物种中的独特基因 ID 复制到新的文本文件中。
- 通过键入 per-ne"如果(//>(\S+)\$c=$i=1}$c打印:chomp;$i==1,如果@ARGV"$gene_id.txt $PEP,提取候选基因的序列。法斯塔> $OUTPUT
- 使用对等爆炸确认基因注释。
- 在互联网上浏览器上转到 https://blast.ncbi.nlm.nih.gov/Blast.cgi。
- 选择tblastn,然后粘贴候选序列,选择非冗余蛋白质序列数据库并单击BLAST。
- 通过用基因本体 (GO) 术语注释基因组或转录组中的所有基因来识别其他基因(参见讨论)。
- 将蛋白质 FASTA 转移到本地计算机。
- 下载并安装 Blast2GO27、28、29诉 5.2 到本地计算机。
- 打开 Blast2GO,单击 文件, 转到 加载, 转到 加载序列, 单击 加载快速文件 (fasta).选择 FASTA 文件并单击 "加载"。
- 点击 爆炸, 选择 NCBI爆炸,并点击 下一步。编辑参数或单击 "下一步",编辑参数并单击 "运行" 以查找最相似的基因描述。
- 单击映射,然后单击 Run 以搜索基因本体论注释中类似的蛋白质。
- 下一个单击 互程序,选择 EMBL-EBI互程序,然后单击 下一个。编辑参数或单击 "下一步",然后单击 "运行" 以搜索已知基因家族和域的签名。
- 通过单击 文件导出注释,选择 导出,单击 "导出表"。单击 "浏览",命名文件,单击 "保存",单击 "导出"。
- 搜索注释表以查找 GO 感兴趣的术语,以确定其他候选基因。从 FASTA 文件中提取序列(STEP 8.4.5)
9. 植物树
- 下载并安装MEGA30 诉7.0.26到您的本地计算机。
- 打开MEGA,点击 对齐,点击 编辑/构建对齐,选择 创建一个新的对齐 点击 确定,选择 蛋白质。
- 当对齐窗口打开时,单击 "编辑",单击 文件中的"插入"序列 ,然后选择具有候选基因和可能同源基因的蛋白质序列的 FASTA。
- 选择所有序列。找到手臂符号并悬停在它上。它应该说使用MUSCLE31 算法对齐序列。单击手臂符号,然后单击 "对齐蛋白 "以对齐序列。编辑参数或单击 "确定" 以使用默认参数对齐。
- 目视检查并进行任何手动更改,然后保存并关闭对齐窗口。
- 在主MEGA窗口中,单击 模型,单击 "查找最佳DNA/蛋白质模型"(ML),选择对齐文件并选择相应的参数,如: 分析:模型选择(ML)、树使用:自动(邻接树)、统计方法:最大可能性、替代类型:氨基酸、间隙/缺失数据处理:使用所有站点、分支站点筛选:无。
- 确定数据的最佳模型后,转到主要的 MEGA 窗口。单击 Phylogeny, 然后单击 "构造/测试最大可能性树 ",然后在必要时选择对齐。为树选择适当的参数: 统计方法:最大可能性,植物学测试:100个复制品的引导方法,替代类型:氨基酸,模型:LG与Freqs。(+F),网站之间的速率:伽马分布(G)与5个离散伽马类别,差距/缺失的数据处理:使用所有站点,ML启发式方法:最近邻居交换(NNI)。
10. 使用 TPM 可视化基因表达
- 对于三一,在计算机群集上转到 运行abundance_estimates_to_matrix.pl 目录,其中一个输出应该是矩阵。TPM.not_cross_norm将此文件传输到本地计算机。
注:有关交叉样本规范化,请参阅补充材料。 - 对于来自基因组分析的 TPM,请遵循以下步骤。
- 在计算机群集上,转到 RSEM 安装位置。通过键入 scp rsem 生成-数据-矩阵-生成-TPM-矩阵,复制 rsem 生成-数据-矩阵。使用 nano 编辑新文件并将 TPM 的"我的$offsite = 4"从 4 更改为 5,现在应读取"我的$offsite = 5"。
- 转到 RSEM 输出文件 .genes.结果所在的目录,现在使用 rsem 生成 -TPM-矩阵 *[基因/等形体.结果] > $OUTPUT 生成 TPM 矩阵。将结果传输到本地计算机。
- 在格普洛特2中可视化结果。
- 将 R 诉 4.0.0 和 RStudio v. 1.2.1335 下载到本地计算机。
- 打开屏幕右侧的 RStudio 转到 "封装 "选项卡并单击 "安装"。键入 ggplot2 并单击 安装。
- 在 TPM 表中通过键入数据读取的 R 脚本窗口 <读.table("$tpm.txt",标题 = T)
- 对于类似于 图 4类型的条形图,类似 :p<- ggplot () = geom_bar(aes(y=TPM,x=符号,填充=组织),数据=数据,统计="身份")
填写
p<-p]scale_fill_manual(值=填充)
p + 主题 (轴. 文本. x = element_text (角度 = 90) )
结果
上述方法在 图1 中总结,并应用于 海德拉粗俗 组织的数据集。 H. 粗俗 是一种淡水无脊椎动物,属于植物 , 其中也包括珊瑚、水母和海葵。 低俗者 可以通过萌芽无性繁殖,在被分割时可以再生头部和脚部。在这项研究中,我们旨在研究 海德拉7号中蛋白基因的进化和表达。虽然 海德拉 缺乏眼睛,他们表现出光依赖行为32。蛋白酶基因编码在视觉中很重要的蛋白质,以检测不同波长的光,并开始光传递级联。研究这种基因家族在基础物种中的分子进化和表达,可以深入了解动物的眼睛和光检测的进化过程。
我们使用Hydra 2.033参考基因组和公开提供的RNA-seq数据(GEO加入GSE127279)图1生成了一个引导组件。这一步骤大约花了3天时间。虽然在这种情况下,我们没有生成de novo转录机,但三位一体组件可能需要长达 1 周的生成时间,每个库可能需要几个小时才能根据地图页进行读取映射。合并后的海德拉集(约50,000份成绩单)使用 Blast2GO 进行注释,该集件大约需要 1 周图 1。蛋白相关基因的序列被提取到一个快速文件中。从NCBI基因银行也提取了其他物种的蛋白基因序列。我们使用了来自胆小板动物波多科里纳卡内亚,克拉多内玛半径,特里佩迪利亚囊肿,和内马托斯特拉兽医的操作,我们也包括出组姆内米奥皮西莱迪,特里乔普拉克斯阿萨伦斯,德罗索菲拉梅拉诺加斯特和智人。蛋白蛋白基因在MEGA7图2中对齐。通过观察对齐,我们能够识别出海德拉蛋白,这些蛋白缺少结合光敏分子所需的保存的氨基氨基酸。经过目视检查,我们通过模型选择分析确定了最佳模型。我们使用型号 LG + G + F 生成了一棵最大可能性树,引导值为 100图 3。对于149个蛋白基因,树在大约3天内完成。植物学表明,蛋白基因正在通过遗传学中特定的复制和H.Vulgaris7的串联复制而进化。
我们在边缘R中进行了微分表达分析,并观察了蛋白基因的绝对表达。我们假设一个或多个手术将在头部(假设体)上调节,并进行假设与身体柱、萌芽区、脚和触角的对比比较。作为对比的一个例子,假设列和身体列之间有1,774个成绩单的差异表达。我们确定了在多个比较中被调节的基因,并在 Blast2GO 表 1中进行了功能丰富。G蛋白耦合受体活性分组包括蛋白基因。最后,我们通过绘制它们的TPM值图 4,观察了不同组织、萌芽期和再生过程中蛋白基因的绝对表达。利用此处概述的方法,我们确定了2个蛋白基因,这些蛋白基因没有与植物学中的其他蛋白组蛋白组,发现了一种表达比其他蛋白多近200倍的蛋白酶,我们发现了一些与光转移基因共同表达的蛋白酶基因,这些蛋白蛋白基因可用于光检测。
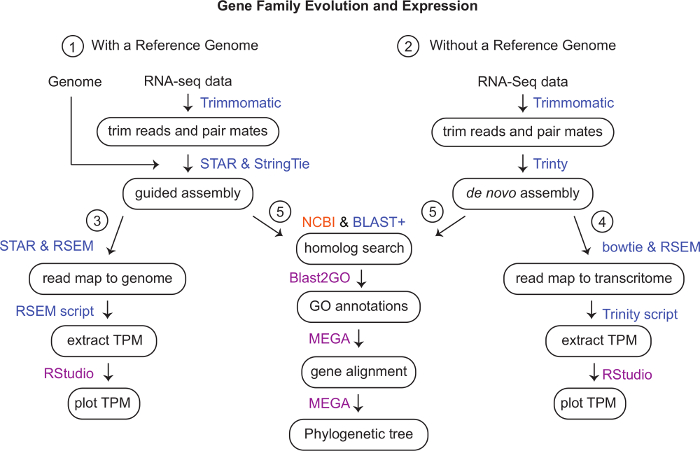
图1:工作流程示意图。用于分析计算机群集数据的程序是蓝色的,品红色是我们在当地计算机上使用的程序,橙色程序是基于网络的程序。(1) 修剪RNA-seq读数使用修剪式 v. 0.35。如果基因组可用,但缺少基因模型,则使用 STAR 诉 2.6.0c 和 StringTie v. 1.3.4d 生成引导组件。(可选见补充材料)(2) 如果没有参考基因组,使用修剪读数使用三一 v 2.8.5 进行 无节制 组装。(3) 要使用参考基因组量化基因表达,地图使用 STAR 读取,使用 RSEM v. 1.3.1 进行量化。使用RSEM提取TPM,并在RSTUDIO中将其可视化。(4) 鲍蒂和RSEM可用于映射和量化映射到三位一体转录组的读数。三位一体脚本可用于生成 TPM 矩阵,以可视化 RStudio 中的计数。(5) 使用基于 Web 的 NCBI 爆炸和命令行 BLAST® 搜索同源序列并使用对等爆炸进行确认。进一步使用 Blast2GO 注释基因。使用 MEGA 来对齐基因,并使用最合适的模型生成植物学树。 请单击此处查看此图的较大版本。
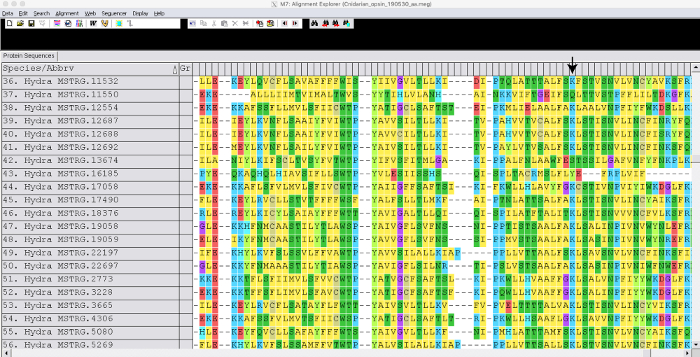
图2:对齐基因示例。快照显示 海德拉 蛋白的一部分使用肌肉对齐。箭头指示视网膜结合保存赖氨酸的位置。 请单击此处查看此图的较大版本。
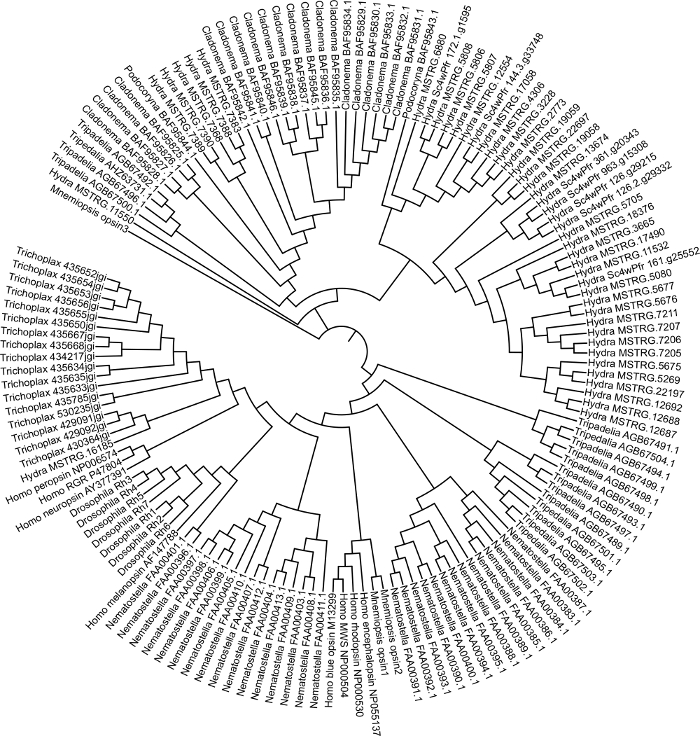
图3:神经蛋白植物学树。在 MEGA7 中产生的最大可能性树使用从 海德拉粗俗, 波多科里纳卡内亚, 克拉多内马半径, 特里佩迪利亚囊肿, 内马托斯特拉兽医, 姆内米奥皮西莱迪, 特里乔普拉克斯阿达伦斯, 德罗索菲拉梅拉诺加斯特 和 智人. 请单击此处查看此图的较大版本。
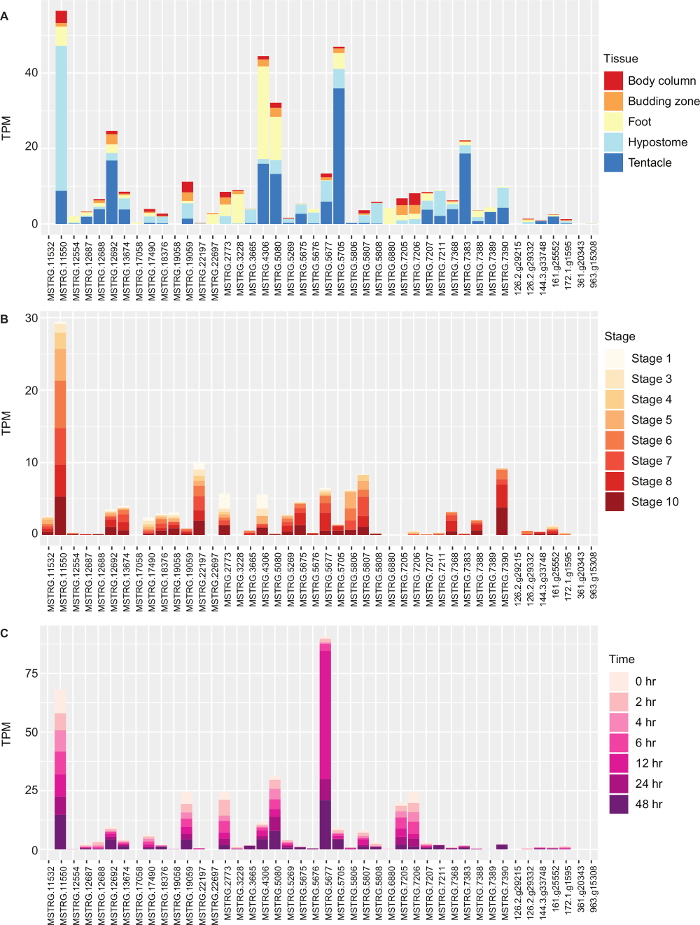
图4: 海德拉粗俗中蛋白基因的表达。(A) 在身体柱、萌芽区、足部、下垂体和触角中,每百万分之一 (TPM) 的海德拉低俗 蛋白基因的表达。(B) 在海德拉 萌芽的不同阶段表达蛋白基因。(C) 在再生的不同时间点表达 海德拉 假设体的蛋白基因。 请单击此处查看此图的较大版本。
| 转到 ID | 去名称 | 去类别 | 罗斯福 |
| 去:0004930 | G蛋白耦合受体活性 | 分子功能 | 0.0000000000704 |
| 去:0007186 | G蛋白耦合受体信号通路 | 生物过程 | 0.00000000103 |
| 去:0016055 | 无信号通路 | 生物过程 | 0.0000358 |
| 去:0051260 | 蛋白质同质化 | 生物过程 | 0.000376 |
| 去:0004222 | 金属循环活动 | 分子功能 | 0.000467 |
| 去:0008076 | 电压门钾通道复合物 | 细胞组件 | 0.000642 |
| 去:0005249 | 电压门钾通道活动 | 分子功能 | 0.00213495 |
| 去:0007275 | 多细胞生物发育 | 生物过程 | 0.00565048 |
| 去:0006813 | 钾离子运输 | 生物过程 | 0.01228182 |
| 去:0018108 | 肽-酪氨酸磷酸化 | 生物过程 | 0.02679662 |
表1:在假设中调节的基因的功能丰富
补充材料。请点击这里下载这些材料。
讨论
本协议的目的是提供使用RNA-seq数据描述基因家族的步骤大纲。这些方法已被证明适用于各种物种和数据集4,34,35。这里建立的管道已经简化,应该很容易,随后是生物信息学的新手。该协议的意义在于,它概述了完成可发布分析的所有步骤和必要程序。协议中的一个关键步骤是正确组装全长成绩单,这来自高质量的基因组或转录组。要获得正确的成绩单,需要高质量的RNA和/或DNA以及下面讨论的良好注释。
对于RNA-seq图书馆的准备,我们包括列表套件,用于海德拉19和蝴蝶18(材料表)的小身体部位。我们注意到,对于低输入RNA,我们使用了修改后的协议方法36。RNA提取方法在多个样本类型中进行了比较,包括酵母细胞17、神经母细胞瘤37、植物38和昆虫幼虫16等。我们建议读者获得一个协议,适用于他们感兴趣的物种,如果有任何存在,或故障排除使用通常商业可用的工具包开始。为了进行适当的基因定量,我们建议使用 DNase 处理 RNA 样本。DNA的存在将影响适当的基因定量。我们还建议使用包含多A尾部选择的cDNA库准备套件来选择成熟的mRNA。虽然 rRNA 耗竭导致更多的读取深度,但使用聚A+选择39的 EXON 覆盖率的百分比远远低于 RNA 的外在覆盖率。最后,在可能的情况下,最好使用配对结束和搁浅40,41。在上面的协议中,使用单端读取时必须修改读取映射命令。
如上所述,重要的是能够识别感兴趣的基因,并区分最近的基因复制,替代拼接,和单体型的测序。在某些情况下,拥有参考基因组可以帮助确定基因和外子相对于对方的位置。需要注意的一件事是,如果转录组是从公共数据库中获取的,质量不高,最好使用三位一体42 生成,并结合感兴趣组织的 RNA-seq 库。同样,如果参考基因组没有良好的基因模型,RNA-seq 库可用于使用 StringTie43生成新的 GTF(参见补充材料)。此外,在基因不完整且无法获得基因组的情况下,基因可以使用同源序列进行手动编辑,然后使用 tblastn 与基因组对齐。BLAST 输出可用于确定实际序列,这可能与使用同源语的校正不同。如果没有匹配,请按原样保留序列。在检查输出时,注意基因组坐标,以确保缺失的外位确实是基因的一部分。
虽然我们专注于我们使用的软件和程序,但由于许多可用的程序可能更好地适用于不同的数据集,因此存在对此协议的修改。例如,我们显示使用蝴蝶结和 RSEM 映射读取到抄本的命令,但三一现在可以选择更快的对齐器,如卡利斯托44和鲑鱼45。同样,我们使用 Blast2GO(现在的 OmicsBox)描述注释,但还有其他的映射工具可以免费和在线找到。一些我们已经尝试过包括:GO FEAT46,蛋鸡映桅杆47,48,和一个非常快速对齐PANNZER249。要使用这些基于 Web 的注释工具,只需上传肽 FASTA 并提交即可。PANNZER 和蛋诺格映点的独立版本也可下载到计算机集群中。另一个修改是,我们使用MEGA和R在当地计算机上,并使用在线NCBI BLAST工具做互惠的BLAST,但所有这些程序都可以在计算机集群上使用下载必要的程序和数据库。同样,只要用户有足够的内存和存储,就可以在当地计算机上使用对齐器卡利斯托和鲑鱼。但是,FASTQ 和 FASTA 文件往往非常大,我们强烈建议使用计算机集群以方便和快速。此外,虽然我们提供指令和链接,从他们的开发人员下载程序,其中许多可以安装从生物康达:https://anaconda.org/bioconda。
在进行生物信息分析时,面临的一个常见问题是外壳脚本失败。这可能是由于各种原因。如果创建了错误文件,则应在排除故障之前检查这些错误文件。出现错误的几个常见原因是打字错误、缺少关键参数以及软件版本之间的兼容性问题。在此协议中,我们包括数据参数,但软件手册可以为单个参数提供更详细的指南。一般来说,最好使用最新的软件版本,并参考该版本对应的手册。
此协议的增强包括进行转录全范围的微分表达分析和功能丰富分析。我们建议 EdgeR50 用于微分表达分析,这是生物导体中可用的一个包。在功能浓缩分析方面,我们使用了 Blast2GO29 和基于 Web 的 DAVID51,52。我们还建议进一步编辑植物学,通过提取它作为一个新的ick文件,并使用基于网络的iTOL53。此外,虽然该协议将研究基因的分子进化和表达模式,但其他实验可用于验证基因或蛋白质的位置和功能。mRNA 表达可以通过 RT-qPCR 或原地混合确认。蛋白质可以通过免疫化学进行本地化。根据物种的不同,淘汰赛实验可用于确认基因功能。此协议可用于各种目标,包括,如上所示,探索一个基因家族通常与光感受在基础物种7。这些方法的另一个应用是识别不同选择性压力下保存路径的变化。例如,这些方法被用来发现视觉瞬态受体潜在通道之间的视觉瞬时受体表达的变化,34号夜间蝴蝶和夜间飞蛾之间。
披露声明
作者没有什么可透露的。
致谢
我们感谢阿德里亚娜·布里斯科、吉尔·史密斯、拉比·穆拉德和艾琳·兰赫尔在将其中一些步骤纳入我们的工作流程方面提供的建议和指导。我们也感谢凯瑟琳·威廉姆斯、伊丽莎白·雷博亚和娜塔莎·皮恰尼对手稿的评论。这项工作部分得到了乔治·休伊特医学研究基金会对A.M.M的支持。
材料
| Name | Company | Catalog Number | Comments |
| Bioanalyzer-DNA kit | Agilent | 5067-4626 | wet lab materials |
| Bioanalyzer-RNA kit | Agilent | 5067-1513 | wet lab materials |
| BLAST+ v. 2.8.1 | On computer cluster* https://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/ | ||
| Blast2GO (on your PC) | On local computer https://www.blast2go.com/b2g-register-basic | ||
| boost v. 1.57.0 | On computer cluster | ||
| Bowtie v. 1.0.0 | On computer cluster https://sourceforge.net/projects/bowtie-bio/files/bowtie/1.3.0/ | ||
| Computing cluster (highly recommended) | NOTE: Analyses of genomic data are best done on a high-performance computing cluster because files are very large. | ||
| Cufflinks v. 2.2.1 | On computer cluster | ||
| edgeR v. 3.26.8 (in R) | In Rstudio https://bioconductor.org/packages/release/bioc/html/edgeR.html | ||
| gcc v. 6.4.0 | On computer cluster | ||
| Java v. 11.0.2 | On computer cluster | ||
| MEGA7 (on your PC) | On local computer https://www.megasoftware.net | ||
| MEGAX v. 0.1 | On local computer https://www.megasoftware.net | ||
| NucleoSpin RNA II kit | Macherey-Nagel | 740955.5 | wet lab materials |
| perl 5.30.3 | On computer cluster | ||
| python | On computer cluster | ||
| Qubit 2.0 Fluorometer | ThermoFisher | Q32866 | wet lab materials |
| R v.4.0.0 | On computer cluster https://cran.r-project.org/src/base/R-4/ | ||
| RNAlater | ThermoFisher | AM7021 | wet lab materials |
| RNeasy kit | Qiagen | 74104 | wet lab materials |
| RSEM v. 1.3.0 | Computer software https://deweylab.github.io/RSEM/ | ||
| RStudio v. 1.2.1335 | On local computer https://rstudio.com/products/rstudio/download/#download | ||
| Samtools v. 1.3 | Computer software | ||
| SRA Toolkit v. 2.8.1 | On computer cluster https://github.com/ncbi/sra-tools/wiki/01.-Downloading-SRA-Toolkit | ||
| STAR v. 2.6.0c | On computer cluster https://github.com/alexdobin/STAR | ||
| StringTie v. 1.3.4d | On computer cluster https://ccb.jhu.edu/software/stringtie/ | ||
| Transdecoder v. 5.5.0 | On computer cluster https://github.com/TransDecoder/TransDecoder/releases | ||
| Trimmomatic v. 0.35 | On computer cluster http://www.usadellab.org/cms/?page=trimmomatic | ||
| Trinity v.2.8.5 | On computer cluster https://github.com/trinityrnaseq/trinityrnaseq/releases | ||
| TRIzol | ThermoFisher | 15596018 | wet lab materials |
| TruSeq RNA Library Prep Kit v2 | Illumina | RS-122-2001 | wet lab materials |
| TURBO DNA-free Kit | ThermoFisher | AM1907 | wet lab materials |
| *Downloads and installation on the computer cluster may require root access. Contact your network administrator. |
参考文献
- Lespinet, O., Wolf, Y. I., Koonin, E. V., Aravind, L. The role of lineage-specific gene family expansion in the evolution of eukaryotes. Genome Research. 12 (7), 1048-1059 (2002).
- Gabaldón, T., Koonin, E. V. Functional and evolutionary implications of gene orthology. Nature Reviews Genetics. 14 (5), 360-366 (2013).
- Dolinski, K., Botstein, D. Orthology and Functional Conservation in Eukaryotes. Annual Review of Genetics. 41 (1), (2007).
- Macias-Muñoz, A., McCulloch, K. J., Briscoe, A. D. Copy number variation and expression analysis reveals a non-orthologous pinta gene family member involved in butterfly vision. Genome Biology and Evolution. 9 (12), 3398-3412 (2017).
- Cannon, S. B., Mitra, A., Baumgarten, A., Young, N. D., May, G. The roles of segmental and tandem gene duplication in the evolution of large gene families in Arabidopsis thaliana. BMC plant biology. 4, 10 (2004).
- Eastman, S. D., Chen, T. H. P., Falk, M. M., Mendelson, T. C., Iovine, M. K. Phylogenetic analysis of three complete gap junction gene families reveals lineage-specific duplications and highly supported gene classes. Genomics. 87 (2), 265-274 (2006).
- Macias-Munõz, A., Murad, R., Mortazavi, A. Molecular evolution and expression of opsin genes in Hydra vulgaris. BMC Genomics. 20 (1), 1-19 (2019).
- Hisatomi, O., Tokunaga, F. Molecular evolution of proteins involved in vertebrate phototransduction. Comparative Biochemistry and Physiology - B Biochemistry and Molecular Biology. 133 (4), 509-522 (2002).
- Arendt, D. Evolution of eyes and photoreceptor cell types. International Journal of Developmental Biology. 47, 563-571 (2003).
- Shichida, Y., Matsuyama, T. Evolution of opsins and phototransduction. Philosophical Transactions of the Royal Society B: Biological Sciences. 364 (1531), 2881-2895 (2009).
- Porter, M. L., et al. Shedding new light on opsin evolution. Proceedings of the Royal Society B: Biological Sciences. 279 (1726), 3-14 (2012).
- Plachetzki, D. C., Degnan, B. M., Oakley, T. H. The origins of novel protein interactions during animal opsin evolution. PLoS ONE. 2 (10), 1054 (2007).
- Ramirez, M. D., et al. The last common ancestor of most bilaterian animals possessed at least nine opsins. Genome Biology and Evolution. 8 (12), 3640-3652 (2016).
- Schnitzler, C. E., et al. Genomic organization, evolution, and expression of photoprotein and opsin genes in Mnemiopsis leidyi: a new view of ctenophore photocytes. BMC Biology. 10, 107 (2012).
- Pedersen, K. B., Williams, A., Watt, J., Ronis, M. J. Improved method for isolating high-quality RNA from mouse bone with RNAlater at room temperature. Bone Reports. 11, 100211 (2019).
- Ridgeway, J. A., Timm, A. E., Fallon, A. Comparison of RNA isolation methods from insect larvae. Journal of Insect Science. 14 (1), 4-8 (2014).
- Scholes, A. N., Lewis, J. A. Comparison of RNA isolation methods on RNA-Seq: Implications for differential expression and meta-Analyses. BMC Genomics. 21 (1), 1-9 (2020).
- Briscoe, A. D., et al. Female behaviour drives expression and evolution of gustatory receptors in butterflies. PLoS genetics. 9 (7), 1003620 (2013).
- Murad, R., Macias-Muñoz, A., Wong, A., Ma, X., Mortazavi, A. Integrative analysis of Hydra head regeneration reveals activation of distal enhancer-like elements. bioRxiv. , 544049 (2019).
- Gallego Romero, I., Pai, A. A., Tung, J., Gilad, Y. Impact of RNA degradation on measurements of gene expression. BMC Biology. 12, 42 (2014).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Trinity. . RNA-Seq De novo Assembly Using Trinity. , 1-7 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29, 15-21 (2013).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC bioinformatics. 12, 323 (2011).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome biology. 10, 25 (2009).
- Camacho, C., et al. BLAST+: architecture and applications. BMC Bioinformatics. 10, 421 (2009).
- Conesa, A., Götz, S. Blast2GO: A comprehensive suite for functional analysis in plant genomics. International Journal of Plant Genomics. 619832, (2008).
- Conesa, A., et al. Blast2GO: A universal tool for annotation, visualization and analysis in functional genomics research. Bioinformatics. 21 (18), 3674-3676 (2005).
- Götz, S., et al. High-throughput functional annotation and data mining with the Blast2GO suite. Nucleic Acids Research. 36 (10), 3420-3435 (2008).
- Kumar, S., Stecher, G., Tamura, K. MEGA7: Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets. Molecular biology and evolution. 33 (7), 1870-1874 (2016).
- Edgar, R. C. MUSCLE: Multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Research. 32 (5), 1792-1797 (2004).
- Taddei-Ferretti, C., Musio, C., Santillo, S., Cotugno, A. The photobiology of Hydra's periodic activity. Hydrobiologia. 530, 129-134 (2004).
- Chapman, J. A., et al. The dynamic genome of Hydra. Nature. 464 (7288), 592-596 (2010).
- Macias-Muñoz, A., Rangel Olguin, A. G., Briscoe, A. D. Evolution of phototransduction genes in Lepidoptera. Genome Biology and Evolution. 11 (8), 2107-2124 (2019).
- Macias-Munõz, A., Murad, R., Mortazavi, A. Molecular evolution and expression of opsin genes in Hydra vulgaris. BMC Genomics. 20 (1), (2019).
- Picelli, S., et al. Full-length RNA-seq from single cells using Smart-seq2. Nature Protocols. 9 (1), 171-181 (2014).
- Tavares, L., Alves, P. M., Ferreira, R. B., Santos, C. N. Comparison of different methods for DNA-free RNA isolation from SK-N-MC neuroblastoma. BMC research notes. 4, 3 (2011).
- Johnson, M. T. J., et al. Evaluating Methods for Isolating Total RNA and Predicting the Success of Sequencing Phylogenetically Diverse Plant Transcriptomes. PLoS ONE. 7 (11), (2012).
- Zhao, S., Zhang, Y., Gamini, R., Zhang, B., Von Schack, D. Evaluation of two main RNA-seq approaches for gene quantification in clinical RNA sequencing: PolyA+ selection versus rRNA depletion. Scientific Reports. 8 (1), 1-12 (2018).
- Zhao, S., et al. Comparison of stranded and non-stranded RNA-seq transcriptome profiling and investigation of gene overlap. BMC Genomics. 16 (1), 1-14 (2015).
- Corley, S. M., MacKenzie, K. L., Beverdam, A., Roddam, L. F., Wilkins, M. R. Differentially expressed genes from RNA-Seq and functional enrichment results are affected by the choice of single-end versus paired-end reads and stranded versus non-stranded protocols. BMC Genomics. 18 (1), 1-13 (2017).
- Haas, B. J., et al. De novo transcript sequence reconstruction from RNA-seq using the Trinity platform for reference generation and analysis. Nature Protocols. 8 (8), 1494-1512 (2013).
- Pertea, M., et al. StringTie enables improved reconstruction of a transcriptome from RNA-seq reads. Nature biotechnology. 33 (3), 290-295 (2015).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nature Biotechnology. 34 (5), 525-527 (2016).
- Patro, R., Duggal, G., Love, M. I., Irizarry, R. A., Kingsford, C. Salmon provides fast and bias-aware quantification of transcript expression. Nature Methods. 14 (4), 417-419 (2017).
- Araujo, F. A., Barh, D., Silva, A., Guimarães, L., Thiago, R. . OPEN GO FEAT a rapid web-based functional annotation tool for genomic and transcriptomic data. , 8-11 (2018).
- Huerta-Cepas, J., et al. Fast genome-wide functional annotation through orthology assignment by eggNOG-mapper. Molecular Biology and Evolution. 34 (8), 2115-2122 (2017).
- Huerta-Cepas, J., et al. EggNOG 5.0: A hierarchical, functionally and phylogenetically annotated orthology resource based on 5090 organisms and 2502 viruses. Nucleic Acids Research. 47, 309-314 (2019).
- Törönen, P., Medlar, A., Holm, L. PANNZER2: A rapid functional annotation web server. Nucleic Acids Research. 46, 84-88 (2018).
- Robinson, M., Mccarthy, D., Chen, Y., Smyth, G. K. . edgeR differential expression analysis of digital gene expression data User's Guide. , (2013).
- Huang, D. W., Sherman, B. T., Lempicki, R. A. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources. Nature Protocols. 4 (1), 44-57 (2009).
- Huang, D. W., Sherman, B. T., Lempicki, R. A. Bioinformatics enrichment tools: Paths toward the comprehensive functional analysis of large gene lists. Nucleic Acids Research. 37 (1), 1-13 (2009).
- Letunic, I., Bork, P. Interactive tree of life (iTOL) v3: an online tool for the display and annotation of phylogenetic and other trees. Nucleic acids research. 44, 242-245 (2016).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。