Method Article
Un pipeline bioinformatique pour étudier l’évolution moléculaire et l’expression génique à l’aide de l’ARN-seq
Dans cet article
Résumé
Le but de ce protocole est d’étudier l’évolution et l’expression des gènes candidats à l’aide de données de séquençage de l’ARN.
Résumé
Distiller et signaler de grands ensembles de données, tels que des données sur le génome entier ou le transcriptome, est souvent une tâche ardue. Une façon de décomposer les résultats est de se concentrer sur une ou plusieurs familles de gènes qui sont importantes pour l’organisme et l’étude. Dans ce protocole, nous décrivons les étapes bioinformatiques pour générer une phylogénie et quantifier l’expression des gènes d’intérêt. Les arbres phylogénétiques peuvent donner un aperçu de l’évolution des gènes au sein des espèces et entre elles, ainsi que révéler l’orthologie. Ces résultats peuvent être améliorés en utilisant des données RNA-seq pour comparer l’expression de ces gènes dans différents individus ou tissus. Les études de l’évolution et de l’expression moléculaires peuvent révéler des modes d’évolution et de conservation de la fonction des gènes entre les espèces. La caractérisation d’une famille de gènes peut servir de tremplin pour de futures études et peut mettre en évidence une famille de gènes importante dans un nouveau génome ou un nouvel article de transcriptome.
Introduction
Les progrès des technologies de séquençage ont facilité le séquençage des génomes et des transcriptomes d’organismes non modèles. En plus de la faisabilité accrue du séquençage de l’ADN et de l’ARN de nombreux organismes, une abondance de données est accessible au public pour étudier les gènes d’intérêt. Le but de ce protocole est de fournir des étapes bioinformatiques pour étudier l’évolution moléculaire et l’expression des gènes qui peuvent jouer un rôle important dans l’organisme d’intérêt.
L’étude de l’évolution d’un gène ou d’une famille de gènes peut donner un aperçu de l’évolution des systèmes biologiques. Les membres d’une famille de gènes sont généralement déterminés en identifiant des motifs conservés ou des séquences de gènes homologues. L’évolution de la famille de gènes a été précédemment étudiée à l’aide de génomes provenant d’organismes modèles lointainement apparentés1. Une limite à cette approche est qu’il n’est pas clair comment ces familles de gènes évoluent chez des espèces étroitement apparentées et le rôle des différentes pressions sélectives environnementales. Dans ce protocole, nous incluons une recherche d’homologues chez des espèces étroitement apparentées. En générant une phylogénie au niveau de l’embranchement, nous pouvons noter des tendances dans l’évolution de la famille de gènes tels que celle des gènes conservés ou des duplications spécifiques à la lignée. À ce niveau, nous pouvons également étudier si les gènes sont des orthologues ou des paralogues. Bien que de nombreux homologues fonctionnent probablement de manière similaire les uns aux autres, ce n’est pas nécessairement le cas2. L’incorporation d’arbres phylogénétiques dans ces études est importante pour déterminer si ces gènes homologues sont des orthologues ou non. Chez les eucaryotes, de nombreux orthologues conservent des fonctions similaires au sein de la cellule, comme en témoigne la capacité des protéines de mammifères à restaurer la fonction des orthologues de levure3. Cependant, il existe des cas où un gène non orthologue effectue une fonction caractérisée4.
Les arbres phylogénétiques commencent à délimiter les relations entre les gènes et les espèces, mais la fonction ne peut pas être attribuée uniquement en fonction des relations génétiques. Les études d’expression génique combinées aux annotations fonctionnelles et à l’analyse de l’enrichissement fournissent un solide soutien à la fonction des gènes. Les cas où l’expression des gènes peut être quantifiée et comparée entre les individus ou les types de tissus peuvent être plus révélateurs de la fonction potentielle. Le protocole suivant suit les méthodes utilisées dans l’étude des gènes de l’opsine dans Hydra vulgaris7, mais ils peuvent être appliqués à n’importe quelle espèce et n’importe quelle famille de gènes. Les résultats de ces études fournissent une base pour une étude plus approfondie de la fonction des gènes et des réseaux de gènes dans les organismes non modèles. A titre d’exemple, l’étude de la phylogénie des opsines, qui sont des protéines qui initient la cascade de phototransduction, donne un contexte à l’évolution de la détection des yeux et de la lumière8,9,10,11. Dans ce cas, des organismes non modèles en particulier des espèces animales basales telles que les cnidaires ou les cténophores peuvent élucider la conservation ou les changements dans la cascade de phototransduction et la vision à travers les clades12,13,14. De même, la détermination de la phylogénie, de l’expression et des réseaux d’autres familles de gènes nous renseignera sur les mécanismes moléculaires sous-jacents aux adaptations.
Protocole
Ce protocole suit les directives de soins aux animaux de l’UC Irvine.
1. Préparation de la bibliothèque RNA-seq
- Isolez l’ARN à l’aide des méthodes suivantes.
- Prélever des échantillons. Si l’ARN doit être extrait ultérieurement, congeler instantanément l’échantillon ou le placer dans une solution de stockage d’ARN15 (Table des matériaux).
- Euthanasier et disséquer l’organisme pour séparer les tissus d’intérêt.
- Extraire l’ARN total à l’aide d’un kit d’extraction et purifier l’ARN à l’aide d’un kit de purificationde l’ARN( Table des matériaux )
REMARQUE: Il existe des protocoles et des kits qui peuvent mieux fonctionner pour différentes espèces et types de tissus16,17. Nous avons extrait l’ARN de différents tissus corporels d’un papillon18 et d’un Hydra gélatineux 19 (voir discussion). - Mesurer la concentration et la qualité de l’ARN de chaque échantillon(Table des matériaux). Utilisez des échantillons dont le nombre d’intégrité de l’ARN (NRI) est supérieur à 8, idéalement plus près de9 20 pour construire des bibliothèques d’ADNc.
- Construisez la bibliothèque et la séquence d’ADNc comme suit.
- Construisez des bibliothèques d’ADNc selon le manuel d’instructions de préparation de la bibliothèque (voir discussion).
- Déterminer la concentration et la qualité del’ADNc (Table des matières).
- Multiplexez les bibliothèques et séquencez-les.
2. Accéder à un cluster d’ordinateurs
REMARQUE: L’analyse de l’ARN-seq nécessite la manipulation de fichiers volumineux et est mieux effectuée sur un cluster informatique(table des matériaux).
- Connectez-vous au compte de cluster d’ordinateurs à l’aide de la commande ssh username@clusterlocation sur une fenêtre d’application de terminal (Mac) ou PuTTY (Windows).
3. Obtenir des lectures RNA-seq
- Obtenir des lectures d’ARN-seq à partir de l’installation de séquençage ou, pour les données générées dans une publication, à partir du dépôt de données où elles ont été déposées (3.2 ou 3.3).
- Pour télécharger des données à partir de référentiels tels que ArrayExpress, procédez comme suit :
- Effectuez une recherche sur le site à l’aide du numéro d’adhésion.
- Recherchez le lien pour télécharger les données, puis cliquez avec le bouton gauche de la souris et sélectionnez Copier le lien.
- Dans la fenêtre du terminal, tapez wget et sélectionnez Coller le lien pour copier les données dans le répertoire à des fins d’analyse.
- Pour télécharger les données NCBI Short Read Archive (SRA), procédez comme suit :
- Sur le terminal, téléchargez SRA Toolkit v. 2.8.1 en utilisant wget.
Remarque : téléchargement et l’installation de programmes sur le cluster d’ordinateurs peut nécessiter un accès root, contactez l’administrateur de cluster de votre ordinateur si l’installation échoue. - Terminez l’installation du programme en tapant tar -xvf $TARGZFILE.
- Recherchez dans NCBI le numéro d’accession SRA pour les échantillons que vous souhaitez télécharger, il devrait avoir le format SRRXXXXXX.
- Obtenez les données RNA-seq en tapant [sratoolkit location]/bin/prefetch SRRXXXXXX dans la fenêtre du terminal.
- Pour les fichiers appariés, tapez [emplacement du sratoolkit]/bin/fastq-dump --split-files SRRXXXXXX pour obtenir deux fichiers fastq (SRRXXXXXX_1.FASTQ et SRRXXXXXX_2.FASTQ).
REMARQUE: Pour faire un assemblage Trinity de novo, utilisez la commande [sratoolkit location]/bin/fastq-dump --defline-seq '@$sn[_$rn]/$ri' --split-files SRRXXXXXX
- Sur le terminal, téléchargez SRA Toolkit v. 2.8.1 en utilisant wget.
4. Trim adaptateurs et lectures de faible qualité (facultatif)
- Installez ou chargez Trimmomatic21 v. 0.35 sur le cluster informatique.
- Dans le répertoire où se trouvent les fichiers de données RNA-seq, tapez une commande qui inclut l’emplacement du fichier jar trimmomatic, les fichiers FASTQ d’entrée, les fichiers FASTQ de sortie et des paramètres facultatifs tels que la longueur et la qualité de lecture.
Remarque : la commande varie en fonction de la qualité brute et souhaitée et la longueur des lectures. Pour illumina 43 bp lit avec les amorces Nextera, nous avons utilisé: java -jar / data / apps / trimmomatic / 0.35 / trimmomatic-0.35.jar PE $READ 1. FASTQ $READ 2. FASTQ paired_READ1. FASTQ unpaired_READ1. FASTQ paired_READ2. FASTQ unpaired_READ2. FASTQ ILLUMINACLIP:adapters.fa:2:30:10 LEADING:20 TRAILING:20 SLIDINGWINDOW:4:17 MINLEN:30.
5. Obtenir l’assemblage de référence
- Recherchez sur Google, EnsemblGenomes et NCBI Genomes and Nucleotide TSA (Transcriptome Shotgun Assembly) un génome de référence ou un transcriptome assemblé pour l’espèce d’intérêt(Figure 1).
REMARQUE : Si un génome de référence ou un transcriptome n’est pas disponible ou de mauvaise qualité, passez à l’ÉTAPE 6 pour générer un assemblage de novo. - S’il existe un génome de référence ou un transcriptome assemblé, téléchargez-le sous forme de fichier fasta où l’analyse sera effectuée en suivant les étapes ci-dessous.
- Trouvez le lien pour télécharger le génome, cliquez avec le bouton gauche de la souris et copiez le lien.
- Dans la fenêtre du terminal, tapez wget et collez l’adresse du lien. Si disponible, copiez également le fichier GTF et le fichier FASTA des protéines pour le génome de référence.
6. Générer un assemblage de novo (alternative à l’étape 5)
- Combinez les fichiers fastq RNA-seq READ1 et READ2 pour tous les échantillons en tapant cat *READ1. FASTQ > $all_READ1. FASTQ et cat *READ2. FASTQ > all_READ2. FASTQ dans la fenêtre du terminal.
- Installez ou chargez Trinity22 v.2.8.5 sur le cluster informatique.
- Générer et assemblage en tapant sur le terminal : Trinity --seqType fq --max_memory 20G --left $all_READ1. FASTQ --droite $all_READ2. FASTQ.
7. Lecture de la carte au génome (7.1) ou au transcriptome de novo (7.2)
- La carte se lit sur le génome de référence en utilisant STAR23 v. 2.6.0c et RSEM24 v. 1.3.0.
- Installez ou chargez STAR v. 2.6.0c. et RSEM v. 1.3.0 au cluster informatique.
- Indexer le génome en tapant rsem-prepare-reference --gtf $GENOME. GTF --star -p 16 $GENOME. FASTA $OUTPUT.
- Mappez l’expression et calculez l’expression pour chaque exemple en tapant rsem-calculate-expression -p 16 --star --paired-end $READ 1. FASTQ $READ 2. FASTQ $INDEX $OUTPUT.
- Renommez le fichier de résultats en quelque chose de descriptif à l’aide de mv RSEM.genes.results $sample.genes.results.
- Générez une matrice de tous les nombres en tapant rsem-generate-data-matrix *[genes/isoforms.results] > $OUTPUT.
- Cartographier l’ARN-seq à l’assemblage de novo Trinity à l’aide de RSEM et de nœud papillon.
- Installez ou chargez Trinity22 v.2.8.5, Bowtie25 v. 1.0.0 et RSEM v. 1.3.0.
- Mappez l’expression de lecture et de calcul pour chaque échantillon en tapant [trinity_location]/align_and_estimate_abundance.pl --prep-reference --transcripts $TRINITY. FASTA --seqType fq --left $READ 1. FASTQ --droite $READ 2. FASTQ --est_method RSEM --nœud papillon aln_method --trinity_mode --output_dir $OUTPUT.
- Renommez le fichier de résultats en quelque chose de descriptif à l’aide de mv RSEM.genes.results $sample.genes.results.
- Générez une matrice de tous les nombres en tapant [trinity_location]/abundance_estimates_to_matrix.pl --est_method RSEM *[gènes/isoformes].results
8. Identifier les gènes d’intérêt
REMARQUE: Les étapes suivantes peuvent être effectuées avec des fichiers FASTA nucléotidiques ou protéiques, mais fonctionnent mieux et sont plus simples avec des séquences de protéines. BLAST recherche en utilisant des protéines à des protéines est plus susceptible de donner des résultats lors de la recherche entre différentes espèces.
- Pour un génome de référence, utilisez le fichier FASTA de protéines de STEP 5.2.2 ou consultez Documents supplémentaires pour générer un GTF de caractéristique de gène personnalisé.
- Pour un transcriptome de novo, générez une protéine FASTA à l’aide de TransDecoder.
- Installez ou chargez TransDecoder v. 5.5.0 sur l’ordinateur cluser.
- Trouvez le cadre de lecture ouvert le plus long et la séquence peptidique prédite en tapant [Emplacement du transdécodeur]/TransDecoder.LongOrfs -t $TRINITY. FASTA.
- Recherchez dans NCBI Genbank des homologues sur des espèces étroitement apparentées.
- Ouvrez une fenêtre de navigateur Internet et accédez à https://www.ncbi.nlm.nih.gov/genbank/.
- Dans la barre de recherche, tapez le nom du gène d’intérêt et le nom des espèces étroitement apparentées qui ont été séquencées ou un genre ou un embranchement. Sur la gauche de la barre de recherche, sélectionnez protéine, puis cliquez sur Rechercher.
- Extrayez les séquences en cliquant sur Envoyer à, puis sélectionnez Fichier. Sous Format, sélectionnez FASTA, puis cliquez sur Créer un fichier.
- Déplacez le fichier FASTA des homologues vers le cluster d’ordinateurs en tapant scp $FASTA username@clusterlocation:/$DIR dans une fenêtre de terminal local ou utilisez FileZilla pour transférer des fichiers vers et depuis l’ordinateur et le cluster.
- Recherchez des gènes candidats à l’aide de BLAST+26.
- Installez ou chargez BLAST+ v. 2.8.1 sur le cluster d’ordinateurs.
- Sur le cluster informatique, faites une base de données BLAST à partir du génome ou du transcriptome traduit la protéine FASTA en tapant [emplacement BLAST+]/makeblastdb -in $PEP. FASTA -dbtype prot -out $OUTPUT
- BLAST les séquences de gènes homologues de NCBI à la base de données de l’espèce d’intérêt en tapant [emplacement BLAST+]/blastp -db $DATABASE -query $FASTA -evalue 1e-10 -outfmt 6 -max_target_seqs 1 -out $OUTPUT.
- Affichez le fichier de sortie à l’aide de la commande plus. Copiez les ID de gènes uniques de l’espèce d’intérêt dans un nouveau fichier texte.
- Extraire les séquences des gènes candidats en tapant perl -ne 'if(/^>(\S+)/){$c=$i{$1}}$c?print:chomp;$i{$_}=1 if @ARGV' $gene_id.txt $PEP. FASTA > $OUTPUT.
- Confirmez l’annotation des gènes à l’aide de BLAST réciproque.
- Sur le navigateur Internet, accédez à https://blast.ncbi.nlm.nih.gov/Blast.cgi.
- Sélectionnez tblastn, puis collez les séquences candidates, sélectionnez la base de données des séquences de protéines non redondantes et cliquez sur BLAST.
- Identifier d’autres gènes en annotant tous les gènes du génome ou du transcriptome avec des termes d’ontologie de gènes (GO) (voir discussion).
- Transférez la protéine FASTA sur l’ordinateur local.
- Téléchargez et installez Blast2GO27,28,29 v. 5.2 sur l’ordinateur local.
- Ouvrez Blast2GO, cliquez sur Fichier, allez dans Charger, allez dans Charger les séquences, cliquez sur Charger le fichier Fasta (fasta). Sélectionnez le fichier FASTA et cliquez sur Charger.
- Cliquez sur Blast, choisissez NCBI Blast, puis cliquez sur Suivant. Modifiez les paramètres ou cliquez sur Suivant, modifiez les paramètres et cliquez sur Exécuter pour trouver la description de gène la plus similaire.
- Cliquez sur mappage, puis cliquez sur Exécuter pour rechercher des annotations d’ontologie génétique pour des protéines similaires.
- Cliquez ensuite sur interpro, sélectionnez EMBL-EBI InterPro, puis cliquez sur Suivant. Modifiez les paramètres ou cliquez sur Suivant, puis sur Exécuter pour rechercher les signatures de familles de gènes et de domaines connus.
- Exportez les annotations en cliquant sur Fichier, sélectionnez Exporter, cliquez sur Exporter la table. Cliquez sur Parcourir, nommez le fichier, cliquez sur Enregistrer, cliquez sur Exporter.
- Recherchez dans le tableau d’annotation les termes d’intérêt go pour identifier d’autres gènes candidats. Extraire les séquences du fichier FASTA (STEP 8.4.5)
9. Arbres phylogénétiques
- Téléchargez et installez MEGA30 v. 7.0.26 sur votre ordinateur local.
- Ouvrez MEGA, cliquez sur Aligner, cliquez sur Modifier / Construire l’alignement, sélectionnez Créer un nouvel alignement cliquez sur OK, sélectionnez Protéine.
- Lorsque la fenêtre d’alignement s’ouvre, cliquez sur Modifier, cliquez sur Insérer des séquences à partir du fichier et sélectionnez le FASTA avec des séquences de protéines de gènes candidats et des homologues probables.
- Sélectionnez toutes les séquences. Trouvez le symbole du bras et survolez-le. Il devrait dire Aligner les séquences en utilisant l’algorithme MUSCLE31. Cliquez sur le symbole du bras, puis sur Aligner la protéine pour aligner les séquences. Modifiez les paramètres ou cliquez sur OK pour les aligner à l’aide des paramètres par défaut.
- Inspectez visuellement et apportez les modifications manuelles, puis enregistrez et fermez la fenêtre d’alignement.
- Dans la fenêtre principale de MEGA, cliquez sur Modèles, cliquez sur Trouver les meilleurs modèles d’ADN / protéines (ML),sélectionnez le fichier d’alignement et sélectionnez les paramètres correspondants tels que: Analyse: Sélection du modèle (ML), Arbre à utiliser: Automatique (arbre voisin), Méthode statistique: Maximum de vraisemblance, Type de substitution: Acide aminé, Traitement des données gap / manquantes: Utiliser tous les sites, Filtre de site de succursale: Aucun.
- Une fois le meilleur modèle pour les données déterminé, accédez à la fenêtre MEGA principale. Cliquez sur Phylogénie, puis sur Contruct/Test Maximum Likelihood Tree (Arbre de vraisemblance de contruct/test), puis sélectionnez l’alignement, si nécessaire. Sélectionnez les paramètres appropriés pour l’arbre: Méthode statistique: Maximum de vraisemblance, Test de phylogénie: Méthode Bootstrap avec 100 répétitions, type de substitution: acide aminé, modèle: LG avec Freqs. (+F), taux parmi les sites : gamma distribué (G) avec 5 catégories gamma discrètes, traitement des données manquantes:utiliser tous les sites, méthode heuristique ML : Échange du plus proche voisin (NNI).
10. Visualiser l’expression des gènes à l’aide de TPM
- Pour Trinity, sur le cluster d’ordinateurs, accédez au répertoire où abundance_estimates_to_matrix.pl a été exécuté et l’une des sorties doit être matrice. TPM.not_cross_norm. Transférez ce fichier sur votre ordinateur local.
Remarque : voir documents supplémentaires pour la normalisation des échantillons croisés. - Pour les MTP d’une analyse génomique, suivez les étapes ci-dessous.
- Sur le cluster d’ordinateurs, accédez à l’emplacement d’installation RSEM. Copiez rsem-generate-data-matrix en tapant scp rsem-generate-data-matrix rsem-generate-TPM-matrix. Utilisez nano pour modifier le nouveau fichier et changer « mon $offsite = 4 » de 4 à 5 pour TPM, il devrait maintenant lire « mon $offsite = 5 ».
- Accédez au répertoire où se trouvent les fichiers de sortie RSEM .genes.results et utilisez maintenant rsem-generate-TPM-matrix *[genes/isoforms.results] > $OUTPUT pour générer une matrice TPM. Transférer les résultats vers un ordinateur local.
- Visualisez les résultats dans ggplot2.
- Téléchargez R v. 4.0.0 et RStudio v. 1.2.1335 sur un ordinateur local.
- Ouvrez RStudio à droite de l’écran, accédez à l’onglet Packages et cliquez sur Installer. Tapez ggplot2 et cliquez sur installer.
- Dans la fenêtre de script R, lisez dans la table TPM en tapant data<-read.table(« $tpm.txt »,header = T)
- Pour les graphiques à barres similaires à la figure 4, tapez quelque chose de similaire à : p<- ggplot() + geom_bar(aes(y=TPM, x=Symbol, fill=Tissue), data=data, stat="identity »)
fill<-c(« #d7191c »,"#fdae61 », « #ffffbf », « #abd9e9 », « #2c7bb6 »)
p<-p+scale_fill_manual(values=fill)
p + thème(axis.text.x = element_text(angle = 90))
Résultats
Les méthodes ci-dessus sont résumées à la figure 1 et ont été appliquées à un ensemble de données de tissus vulgaris Hydra. H. vulgaris est un invertébré d’eau douce qui appartient à l’embranchement Cnidaria qui comprend également des coraux, des méduses et des anémones de mer. H. vulgaris peut se reproduire de manière asexuée par bourgeonnement et ils peuvent régénérer leur tête et leur pied lorsqu’ils sont coupés en deux. Dans cette étude, nous avons cherché à étudier l’évolution et l’expression des gènes de l’opsine dans Hydra7. Alors que Hydra manque d’yeux, ils présentent un comportement dépendant de la lumière32. Les gènes de l’opsine codent des protéines qui sont importantes dans la vision pour détecter différentes longueurs d’onde de la lumière et commencer la cascade de phototransduction. L’étude de l’évolution moléculaire et de l’expression de cette famille de gènes chez une espèce basale peut fournir un aperçu de l’évolution de la détection des yeux et de la lumière chez les animaux.
Nous avons généré un assemblage guidé à l’aide du génome de référence Hydra2.0 33 et des données RNA-seq accessibles au public (geo accession GSE127279) Figure 1. Cette étape a pris environ 3 jours. Bien que nous n’ayons pas généré de transcriptome de novo dans ce cas, un assembly Trinity peut prendre jusqu’à 1 semaine à générer et chaque bibliothèque peut prendre quelques heures pour le mappage de lecture en fonction du mappeur. L’assemblage Hydra fusionné (~ 50 000 transcriptions) a été annoté à l’aide de Blast2GO, ce qui a pris environ 1 semaine Figure 1. Des séquences pour des gènes opsine-connexes ont été extraites dans un fichier fasta. Des séquences pour les gènes d’opsine d’autres espèces ont également été extraites de NCBI GenBank. Nous avons utilisé des opsines de cnidaires Podocoryna carnea, Cladonema radiatum, Tripedelia cystophoraet Nematostella vectensis,et nous avons également inclus les groupes extérieurs Mnemiopsis leidyi, Trichoplax adhaerens, Drosophila melanogaster et Homo sapiens. Les gènes de l’opsine ont été alignés dans MEGA7 Figure 2. En regardant l’alignement, nous avons pu identifier les opsines Hydra qui manquaient un acide aminé de lysine conservé nécessaire pour lier une molécule sensible à la lumière. Après inspection visuelle, nous avons déterminé le meilleur modèle en effectuant une analyse de sélection de modèle. Nous avons généré un arbre de maximum de vraisemblance en utilisant le modèle LG + G + F avec une valeur bootstrap de 100 Figure 3. Pour 149 gènes d’opsine, l’arbre a été terminé en environ 3 jours. La phylogénie suggère que les gènes de l’opsine évoluent par duplications spécifiques à la lignée chez les cnidaires et potentiellement par duplication en tandem chez H. vulgaris7.
Nous avons effectué une analyse différentielle d’expression dans edgeR et examiné l’expression absolue des gènes d’opsine. Nous avons émis l’hypothèse qu’une ou plusieurs opsines seraient régulées à la hausse dans la tête (hypostome) et avons effectué des comparaisons par paires de l’hypostome par rapport à la colonne du corps, à la zone bourgeonnante, au pied et aux tentacules. À titre d’exemple d’une comparaison par paires, 1.774 transcriptions ont été exprimées différemment entre l’hypostome et la colonne de corps. Nous avons déterminé les gènes qui ont été régulés à la hausse à travers de multiples comparaisons et avons fait un enrichissement fonctionnel dans Blast2GO Tableau 1. Le groupement de l’activité couplée de récepteur de G-protéine a inclus des gènes d’opsine. Enfin, nous avons examiné l’expression absolue des gènes de l’opsine dans différents tissus, pendant le bourgeonnement et pendant la régénération en traçant leurs valeurs de TPM à l’aide de ggplot Figure 4. En utilisant les méthodes décrites ici, nous avons identifié 2 gènes d’opsine qui ne se sont pas groupés avec les autres opsines dans la phylogénie, trouvé une opsine qui a été exprimée presque 200 fois plus que d’autres, et nous avons trouvé quelques gènes d’opsine co-exprimés avec des gènes de phototransduction qui peuvent être utilisés pour la détection de la lumière.
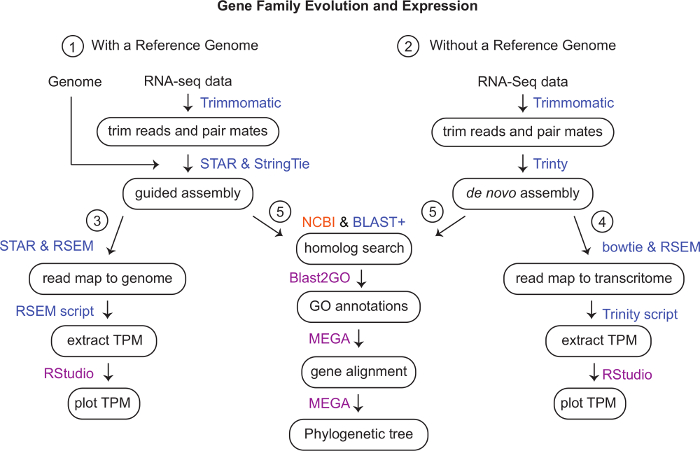
Figure 1 : Schéma de flux de travail. Les programmes utilisés pour analyser les données sur le cluster d’ordinateurs sont en bleu, en magenta sont ceux que nous avons utilisés sur un ordinateur local et en orange est un programme Web. (1) Trim RNA-seq lit en utilisant trimmomatic v. 0.35. Si un génome est disponible mais qu’il manque des modèles de gènes, générez un assemblage guidé en utilisant STAR v. 2.6.0c et StringTie v. 1.3.4d. (Facultatif voir Documents supplémentaires) (2) Sans génome de référence, utilisez des lectures tronquées pour faire un assemblage de novo en utilisant Trinity v 2.8.5. (3) Pour quantifier l’expression des gènes à l’aide d’un génome de référence, la carte se lit à l’aide de STAR et quantifie à l’aide de RSEM v. 1.3.1. Extrayez les TPM à l’aide de RSEM et visualisez-les dans RStudio. (4) Bowtie et RSEM peuvent être utilisés pour cartographier et quantifier les lectures mappées à un transcriptome trinitaire. A Trinity script can be used to generate a TPM matrix to visualize counts in RStudio. (5) Utilisez NCBI BLAST basé sur le Web et LA ligne de commande BLAST+ pour rechercher des séquences homologues et confirmer à l’aide de BLAST réciproque. Annotez davantage les gènes à l’aide de Blast2GO. Utilisez MEGA pour aligner les gènes et générer un arbre phylogénétique en utilisant le modèle le mieux adapté. Veuillez cliquer ici pour voir une version plus grande de cette figure.
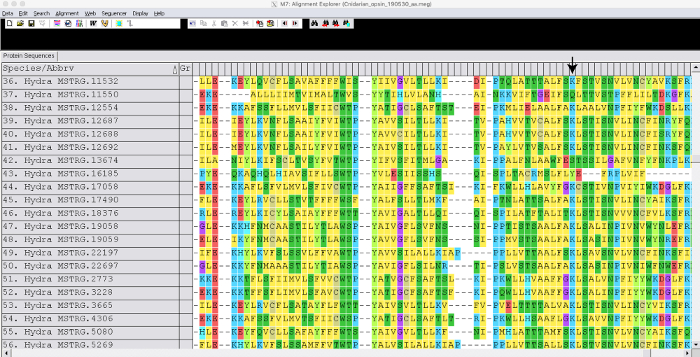
Figure 2 : Exemple de gènes alignés. L’instantané montre une partie des gènes de l’opsine Hydra alignés à l’aide de MUSCLE. La flèche indique l’emplacement d’une lysine conservée de liaison rétinienne. Veuillez cliquer ici pour voir une version plus grande de cette figure.
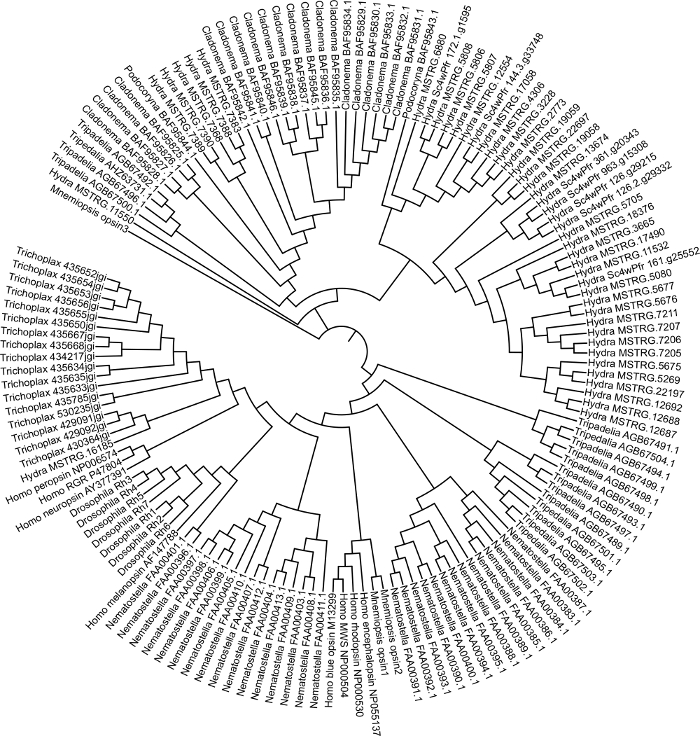
Ill. 3 : Arbre phylogénétique de l’opsine cnidaire. Arbre de vraisemblance maximale généré dans MEGA7 en utilisant des séquences d’opsine de Hydra vulgaris, Podocoryna carnea, Cladonema radiatum, Tripedelia cystophora, Nematostella vectensis , Mnemiopsis leidyi, Trichoplax adhaerens, Drosophila melanogaster et Homo sapiens. Veuillez cliquer ici pour voir une version plus grande de cette figure.
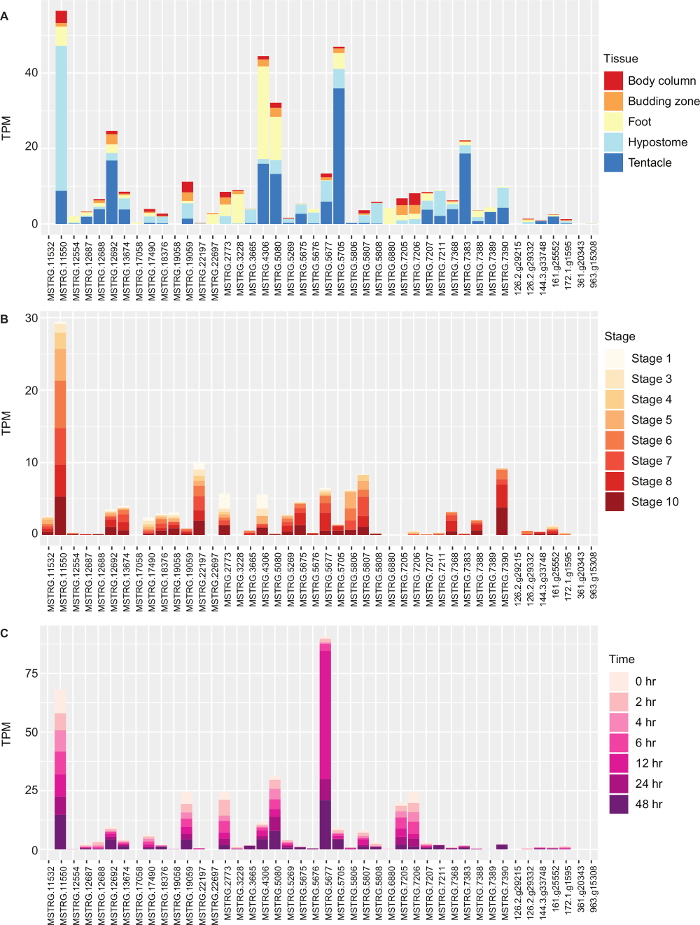
Figure 4 : Expression des gènes de l’opsine dans Hydra vulgaris. (A) Expression en transcriptions par million (TPM) des gènes d’opsine vulgaris d’Hydra dans la colonne du corps, la zone bourgeonnante, le pied, l’hypostome et les tentacules. (B) Expression des gènes de l’opsine au cours des différentes étapes du bourgeonnement de l’hydre. (C) Expression des gènes d’opsine de l’hypostome d’Hydra au cours de différents points de régénération. Veuillez cliquer ici pour voir une version plus grande de cette figure.
| GO ID | Nom GO | Catégorie GO | Fdr |
| GO:0004930 | Activité des récepteurs couplés à la protéine G | FONCTION MOLÉCULAIRE | 0.0000000000704 |
| GO:0007186 | Voie de signalisation des récepteurs couplés à la protéine G | PROCESSUS BIOLOGIQUE | 0.00000000103 |
| GO:0016055 | Voie de signalisation Wnt | PROCESSUS BIOLOGIQUE | 0.0000358 |
| GO:0051260 | homooligomérisation des protéines | PROCESSUS BIOLOGIQUE | 0.000376 |
| GO:0004222 | activité de la métalloendopeptidase | FONCTION MOLÉCULAIRE | 0.000467 |
| GO:0008076 | complexe de canaux potassique voltage-gated | COMPOSANT CELLULAIRE | 0.000642 |
| GO:0005249 | activité du canal potassique voltage-fermée | FONCTION MOLÉCULAIRE | 0.00213495 |
| GO:0007275 | développement d’organismes multicellulaires | PROCESSUS BIOLOGIQUE | 0.00565048 |
| GO:0006813 | transport d’ions potassium | PROCESSUS BIOLOGIQUE | 0.01228182 |
| GO:0018108 | phosphorylation peptidyl-tyrosine | PROCESSUS BIOLOGIQUE | 0.02679662 |
Tableau 1 : Enrichissement fonctionnel des gènes régulés à la hausse dans l’hypostome
Documents supplémentaires. Veuillez cliquer ici pour télécharger ces documents.
Discussion
Le but de ce protocole est de fournir un aperçu des étapes pour caractériser une famille de gènes en utilisant des données RNA-seq. Il a été prouvé que ces méthodes fonctionnent pour une variété d’espèces et d’ensembles de données4,34,35. Le pipeline établi ici a été simplifié et devrait être assez facile à suivre par un novice en bioinformatique. L’importance du protocole est qu’il décrit toutes les étapes et les programmes nécessaires pour effectuer une analyse publiable. Une étape cruciale du protocole est d’avoir correctement assemblé des transcriptions complètes, qui proviennent de génomes ou de transcriptomes de haute qualité. Pour obtenir des transcriptions correctes, il faut de l’ARN et /ou de l’ADN de haute qualité et de bonnes annotations discutées ci-dessous.
Pour la préparation de la bibliothèque RNA-seq, nous incluons des kits de liste qui ont fonctionné pour les petites parties du corps d’Hydra19 et des papillons18 (Table des matériaux). Nous notons que pour l’ARN à faible entrée, nous avons utilisé une approche de protocole modifiée36. Les méthodes d’extraction de l’ARN ont été comparées dans plusieurs types d’échantillons, y compris les cellules de levure17,le neuroblastome37,les plantes38et les larves d’insectes16 pour n’en nommer que quelques-uns. Nous recommandons au lecteur d’acquérir un protocole qui fonctionne pour ses espèces d’intérêt, le cas échéant, ou de dépanner à l’aide de kits couramment disponibles dans le commerce pour commencer. Pour une quantification appropriée des gènes, nous vous recommandons de traiter l’échantillon d’ARN avec de la DNase. La présence d’ADN affectera la quantification des gènes. Nous vous recommandons également d’utiliser un kit de préparation de bibliothèque d’ADNc qui comprend une sélection de queue polyA à sélectionner pour l’ARNm mature. Alors que l’épuisement de l’ARNr entraîne une plus grande profondeur de lecture, le pourcentage de couverture d’exon est beaucoup plus faible que la couverture d’exon de l’ARN en utilisant la sélection polyA +39. Enfin, lorsque cela est possible, il est préférable d’utiliser apparié-extrémité et échoué40,41. Dans le protocole ci-dessus, les commandes de mappage de lecture devront être modifiées lors de l’utilisation de lectures d’extrémité unique.
Comme mentionné ci-dessus, il est important de pouvoir identifier les gènes d’intérêt et de faire la différence entre les duplications récentes de gènes, l’épissage alternatif et les haplotypes dans le séquençage. Dans certains cas, le fait d’avoir un génome de référence peut aider en déterminant où les gènes et les exons sont situés les uns par rapport aux autres. Une chose à noter est que si un transcriptome est obtenu à partir d’une base de données publique et n’est pas de haute qualité, il peut être préférable de générer en utilisant Trinity42 et en combinant des bibliothèques d’ARN-seq à partir de tissus d’intérêt. De même, si un génome de référence n’a pas de bons modèles de gènes, les bibliothèques RNA-seq peuvent être utilisées pour générer de nouveaux GTF à l’aide de StringTie43 (voir Documents supplémentaires). De plus, dans les cas où les gènes sont incomplets et où il y a accès à un génome, les gènes peuvent être modifiés manuellement à l’aide de séquences homologues, puis alignés sur le génome à l’aide de tblastn. La sortie BLAST peut être utilisée pour déterminer la séquence réelle, ce qui peut être différent de la correction effectuée à l’aide d’homologues. S’il n’y a pas de correspondance, laissez la séquence telle quelle à l’origine. Lors de la vérification de la sortie, faites attention aux coordonnées du génome pour vous assurer que l’exon manquant fait bien partie du gène.
Bien que nous nous concentrions sur les logiciels et les programmes que nous avons utilisés, des modifications à ce protocole existent en raison de nombreux programmes disponibles qui pourraient mieux fonctionner pour différents ensembles de données. À titre d’exemple, nous montrons des commandes pour mapper les lectures au transcriptome à l’aide de nœud papillon et RSEM, mais Trinity a maintenant la possibilité pour des aligneurs beaucoup plus rapides tels que kallisto44 et salmon45. De même, nous décrivons les annotations à l’aide de Blast2GO (maintenant OmicsBox), mais il existe d’autres outils de cartographie qui peuvent être trouvés gratuitement et en ligne. Certains que nous avons essayés comprennent: GO FEAT46, eggNOG-mapper47,48, et un aligneur très rapide PANNZER249. Pour utiliser ces outils d’annotation basés sur le Web, téléchargez simplement le peptide FASTA et soumettez-le. Des versions autonomes de PANNZER et eggNOG-mapper sont également disponibles pour être téléchargées sur le cluster d’ordinateurs. Une autre modification est que nous avons utilisé MEGA et R sur un ordinateur local et utilisé l’outil NCBI BLAST en ligne pour faire des BLAST réciproques, mais tous ces programmes peuvent être utilisés sur le cluster d’ordinateurs en téléchargeant les programmes et les bases de données nécessaires. De même, les aligneurs kallisto et salmon peuvent être utilisés sur un ordinateur local tant qu’un utilisateur dispose de suffisamment de RAM et de stockage. Cependant, les fichiers FASTQ et FASTA ont tendance à être très volumineux et nous vous recommandons fortement d’utiliser un cluster d’ordinateurs pour plus de facilité et de vitesse. En outre, bien que nous fournissions des instructions et des liens pour télécharger des programmes de leurs développeurs, beaucoup d’entre eux peuvent être installés à partir de bioconda: https://anaconda.org/bioconda.
Un problème courant rencontré lors de l’analyse bioinformatique est l’échec des scripts shell. Cela peut être dû à diverses raisons. Si un fichier d’erreur est créé, ce fichier d’erreur doit être vérifié avant le dépannage. Quelques raisons courantes d’une erreur sont les fautes de frappe, les paramètres clés manquants et les problèmes de compatibilité entre les versions du logiciel. Dans ce protocole, nous incluons des paramètres pour les données, mais les manuels logiciels peuvent fournir des instructions plus détaillées pour les paramètres individuels. En général, il est préférable d’utiliser les versions les plus récentes du logiciel et de consulter le manuel correspondant à cette version.
Les améliorations apportées à ce protocole incluent l’analyse d’expression différentielle à l’échelle du transcriptome et l’analyse de l’enrichissement fonctionnel. Nous recommandons edgeR50 pour l’analyse d’expression différentielle un package disponible dans Bioconductor. Pour l’analyse de l’enrichissement fonctionnel, nous avons utilisé Blast2GO29 et DAVID51,52basé sur le Web. Nous vous recommandons également d’éditer davantage la phylogénie en l’extrayant sous forme de fichier newick et en utilisant iTOL53basé sur le Web. En outre, bien que ce protocole étudie l’évolution moléculaire et les modèles d’expression des gènes, des expériences supplémentaires peuvent être utilisées pour valider les emplacements et les fonctions des gènes ou des protéines. L’expression d’ADN messagère peut être confirmée par RT-qPCR ou hybridation in situ. Les protéines peuvent être localisées à l’aide de l’immunohistochimie. Selon les espèces, des expériences knockout peuvent être utilisées pour confirmer la fonction du gène. Ce protocole peut être utilisé pour une variété d’objectifs dont, comme indiqué ci-dessus, pour explorer une famille de gènes typiquement associée à la photoréception chez une espèce basale7. Une autre application de ces méthodes consiste à identifier les changements dans une voie conservée sous différentes pressions sélectives. A titre d’exemple, ces méthodes ont été utilisées pour découvrir la variation dans l’expression des canaux potentiels des récepteurs transitoires de la vision entre les papillons diurnes et les papillons nocturnes34.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions Adriana Briscoe, Gil Smith, Rabi Murad et Aline G. Rangel pour leurs conseils et leurs conseils dans l’intégration de certaines de ces étapes dans notre flux de travail. Nous sommes également reconnaissants à Katherine Williams, Elisabeth Rebboah et Natasha Picciani pour leurs commentaires sur le manuscrit. Ce travail a été soutenu en partie par une bourse de recherche médicale de la Fondation George E. Hewitt à A.M.M.
matériels
| Name | Company | Catalog Number | Comments |
| Bioanalyzer-DNA kit | Agilent | 5067-4626 | wet lab materials |
| Bioanalyzer-RNA kit | Agilent | 5067-1513 | wet lab materials |
| BLAST+ v. 2.8.1 | On computer cluster* https://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/ | ||
| Blast2GO (on your PC) | On local computer https://www.blast2go.com/b2g-register-basic | ||
| boost v. 1.57.0 | On computer cluster | ||
| Bowtie v. 1.0.0 | On computer cluster https://sourceforge.net/projects/bowtie-bio/files/bowtie/1.3.0/ | ||
| Computing cluster (highly recommended) | NOTE: Analyses of genomic data are best done on a high-performance computing cluster because files are very large. | ||
| Cufflinks v. 2.2.1 | On computer cluster | ||
| edgeR v. 3.26.8 (in R) | In Rstudio https://bioconductor.org/packages/release/bioc/html/edgeR.html | ||
| gcc v. 6.4.0 | On computer cluster | ||
| Java v. 11.0.2 | On computer cluster | ||
| MEGA7 (on your PC) | On local computer https://www.megasoftware.net | ||
| MEGAX v. 0.1 | On local computer https://www.megasoftware.net | ||
| NucleoSpin RNA II kit | Macherey-Nagel | 740955.5 | wet lab materials |
| perl 5.30.3 | On computer cluster | ||
| python | On computer cluster | ||
| Qubit 2.0 Fluorometer | ThermoFisher | Q32866 | wet lab materials |
| R v.4.0.0 | On computer cluster https://cran.r-project.org/src/base/R-4/ | ||
| RNAlater | ThermoFisher | AM7021 | wet lab materials |
| RNeasy kit | Qiagen | 74104 | wet lab materials |
| RSEM v. 1.3.0 | Computer software https://deweylab.github.io/RSEM/ | ||
| RStudio v. 1.2.1335 | On local computer https://rstudio.com/products/rstudio/download/#download | ||
| Samtools v. 1.3 | Computer software | ||
| SRA Toolkit v. 2.8.1 | On computer cluster https://github.com/ncbi/sra-tools/wiki/01.-Downloading-SRA-Toolkit | ||
| STAR v. 2.6.0c | On computer cluster https://github.com/alexdobin/STAR | ||
| StringTie v. 1.3.4d | On computer cluster https://ccb.jhu.edu/software/stringtie/ | ||
| Transdecoder v. 5.5.0 | On computer cluster https://github.com/TransDecoder/TransDecoder/releases | ||
| Trimmomatic v. 0.35 | On computer cluster http://www.usadellab.org/cms/?page=trimmomatic | ||
| Trinity v.2.8.5 | On computer cluster https://github.com/trinityrnaseq/trinityrnaseq/releases | ||
| TRIzol | ThermoFisher | 15596018 | wet lab materials |
| TruSeq RNA Library Prep Kit v2 | Illumina | RS-122-2001 | wet lab materials |
| TURBO DNA-free Kit | ThermoFisher | AM1907 | wet lab materials |
| *Downloads and installation on the computer cluster may require root access. Contact your network administrator. |
Références
- Lespinet, O., Wolf, Y. I., Koonin, E. V., Aravind, L. The role of lineage-specific gene family expansion in the evolution of eukaryotes. Genome Research. 12 (7), 1048-1059 (2002).
- Gabaldón, T., Koonin, E. V. Functional and evolutionary implications of gene orthology. Nature Reviews Genetics. 14 (5), 360-366 (2013).
- Dolinski, K., Botstein, D. Orthology and Functional Conservation in Eukaryotes. Annual Review of Genetics. 41 (1), (2007).
- Macias-Muñoz, A., McCulloch, K. J., Briscoe, A. D. Copy number variation and expression analysis reveals a non-orthologous pinta gene family member involved in butterfly vision. Genome Biology and Evolution. 9 (12), 3398-3412 (2017).
- Cannon, S. B., Mitra, A., Baumgarten, A., Young, N. D., May, G. The roles of segmental and tandem gene duplication in the evolution of large gene families in Arabidopsis thaliana. BMC plant biology. 4, 10 (2004).
- Eastman, S. D., Chen, T. H. P., Falk, M. M., Mendelson, T. C., Iovine, M. K. Phylogenetic analysis of three complete gap junction gene families reveals lineage-specific duplications and highly supported gene classes. Genomics. 87 (2), 265-274 (2006).
- Macias-Munõz, A., Murad, R., Mortazavi, A. Molecular evolution and expression of opsin genes in Hydra vulgaris. BMC Genomics. 20 (1), 1-19 (2019).
- Hisatomi, O., Tokunaga, F. Molecular evolution of proteins involved in vertebrate phototransduction. Comparative Biochemistry and Physiology - B Biochemistry and Molecular Biology. 133 (4), 509-522 (2002).
- Arendt, D. Evolution of eyes and photoreceptor cell types. International Journal of Developmental Biology. 47, 563-571 (2003).
- Shichida, Y., Matsuyama, T. Evolution of opsins and phototransduction. Philosophical Transactions of the Royal Society B: Biological Sciences. 364 (1531), 2881-2895 (2009).
- Porter, M. L., et al. Shedding new light on opsin evolution. Proceedings of the Royal Society B: Biological Sciences. 279 (1726), 3-14 (2012).
- Plachetzki, D. C., Degnan, B. M., Oakley, T. H. The origins of novel protein interactions during animal opsin evolution. PLoS ONE. 2 (10), 1054 (2007).
- Ramirez, M. D., et al. The last common ancestor of most bilaterian animals possessed at least nine opsins. Genome Biology and Evolution. 8 (12), 3640-3652 (2016).
- Schnitzler, C. E., et al. Genomic organization, evolution, and expression of photoprotein and opsin genes in Mnemiopsis leidyi: a new view of ctenophore photocytes. BMC Biology. 10, 107 (2012).
- Pedersen, K. B., Williams, A., Watt, J., Ronis, M. J. Improved method for isolating high-quality RNA from mouse bone with RNAlater at room temperature. Bone Reports. 11, 100211 (2019).
- Ridgeway, J. A., Timm, A. E., Fallon, A. Comparison of RNA isolation methods from insect larvae. Journal of Insect Science. 14 (1), 4-8 (2014).
- Scholes, A. N., Lewis, J. A. Comparison of RNA isolation methods on RNA-Seq: Implications for differential expression and meta-Analyses. BMC Genomics. 21 (1), 1-9 (2020).
- Briscoe, A. D., et al. Female behaviour drives expression and evolution of gustatory receptors in butterflies. PLoS genetics. 9 (7), 1003620 (2013).
- Murad, R., Macias-Muñoz, A., Wong, A., Ma, X., Mortazavi, A. Integrative analysis of Hydra head regeneration reveals activation of distal enhancer-like elements. bioRxiv. , 544049 (2019).
- Gallego Romero, I., Pai, A. A., Tung, J., Gilad, Y. Impact of RNA degradation on measurements of gene expression. BMC Biology. 12, 42 (2014).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Trinity. . RNA-Seq De novo Assembly Using Trinity. , 1-7 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29, 15-21 (2013).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC bioinformatics. 12, 323 (2011).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome biology. 10, 25 (2009).
- Camacho, C., et al. BLAST+: architecture and applications. BMC Bioinformatics. 10, 421 (2009).
- Conesa, A., Götz, S. Blast2GO: A comprehensive suite for functional analysis in plant genomics. International Journal of Plant Genomics. 619832, (2008).
- Conesa, A., et al. Blast2GO: A universal tool for annotation, visualization and analysis in functional genomics research. Bioinformatics. 21 (18), 3674-3676 (2005).
- Götz, S., et al. High-throughput functional annotation and data mining with the Blast2GO suite. Nucleic Acids Research. 36 (10), 3420-3435 (2008).
- Kumar, S., Stecher, G., Tamura, K. MEGA7: Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets. Molecular biology and evolution. 33 (7), 1870-1874 (2016).
- Edgar, R. C. MUSCLE: Multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Research. 32 (5), 1792-1797 (2004).
- Taddei-Ferretti, C., Musio, C., Santillo, S., Cotugno, A. The photobiology of Hydra's periodic activity. Hydrobiologia. 530, 129-134 (2004).
- Chapman, J. A., et al. The dynamic genome of Hydra. Nature. 464 (7288), 592-596 (2010).
- Macias-Muñoz, A., Rangel Olguin, A. G., Briscoe, A. D. Evolution of phototransduction genes in Lepidoptera. Genome Biology and Evolution. 11 (8), 2107-2124 (2019).
- Macias-Munõz, A., Murad, R., Mortazavi, A. Molecular evolution and expression of opsin genes in Hydra vulgaris. BMC Genomics. 20 (1), (2019).
- Picelli, S., et al. Full-length RNA-seq from single cells using Smart-seq2. Nature Protocols. 9 (1), 171-181 (2014).
- Tavares, L., Alves, P. M., Ferreira, R. B., Santos, C. N. Comparison of different methods for DNA-free RNA isolation from SK-N-MC neuroblastoma. BMC research notes. 4, 3 (2011).
- Johnson, M. T. J., et al. Evaluating Methods for Isolating Total RNA and Predicting the Success of Sequencing Phylogenetically Diverse Plant Transcriptomes. PLoS ONE. 7 (11), (2012).
- Zhao, S., Zhang, Y., Gamini, R., Zhang, B., Von Schack, D. Evaluation of two main RNA-seq approaches for gene quantification in clinical RNA sequencing: PolyA+ selection versus rRNA depletion. Scientific Reports. 8 (1), 1-12 (2018).
- Zhao, S., et al. Comparison of stranded and non-stranded RNA-seq transcriptome profiling and investigation of gene overlap. BMC Genomics. 16 (1), 1-14 (2015).
- Corley, S. M., MacKenzie, K. L., Beverdam, A., Roddam, L. F., Wilkins, M. R. Differentially expressed genes from RNA-Seq and functional enrichment results are affected by the choice of single-end versus paired-end reads and stranded versus non-stranded protocols. BMC Genomics. 18 (1), 1-13 (2017).
- Haas, B. J., et al. De novo transcript sequence reconstruction from RNA-seq using the Trinity platform for reference generation and analysis. Nature Protocols. 8 (8), 1494-1512 (2013).
- Pertea, M., et al. StringTie enables improved reconstruction of a transcriptome from RNA-seq reads. Nature biotechnology. 33 (3), 290-295 (2015).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nature Biotechnology. 34 (5), 525-527 (2016).
- Patro, R., Duggal, G., Love, M. I., Irizarry, R. A., Kingsford, C. Salmon provides fast and bias-aware quantification of transcript expression. Nature Methods. 14 (4), 417-419 (2017).
- Araujo, F. A., Barh, D., Silva, A., Guimarães, L., Thiago, R. . OPEN GO FEAT a rapid web-based functional annotation tool for genomic and transcriptomic data. , 8-11 (2018).
- Huerta-Cepas, J., et al. Fast genome-wide functional annotation through orthology assignment by eggNOG-mapper. Molecular Biology and Evolution. 34 (8), 2115-2122 (2017).
- Huerta-Cepas, J., et al. EggNOG 5.0: A hierarchical, functionally and phylogenetically annotated orthology resource based on 5090 organisms and 2502 viruses. Nucleic Acids Research. 47, 309-314 (2019).
- Törönen, P., Medlar, A., Holm, L. PANNZER2: A rapid functional annotation web server. Nucleic Acids Research. 46, 84-88 (2018).
- Robinson, M., Mccarthy, D., Chen, Y., Smyth, G. K. . edgeR differential expression analysis of digital gene expression data User's Guide. , (2013).
- Huang, D. W., Sherman, B. T., Lempicki, R. A. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources. Nature Protocols. 4 (1), 44-57 (2009).
- Huang, D. W., Sherman, B. T., Lempicki, R. A. Bioinformatics enrichment tools: Paths toward the comprehensive functional analysis of large gene lists. Nucleic Acids Research. 37 (1), 1-13 (2009).
- Letunic, I., Bork, P. Interactive tree of life (iTOL) v3: an online tool for the display and annotation of phylogenetic and other trees. Nucleic acids research. 44, 242-245 (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon