Method Article
Una pipeline bioinformatica per lo studio dell'evoluzione molecolare e dell'espressione genica usando RNA-seq
In questo articolo
Riepilogo
Lo scopo di questo protocollo è quello di indagare l'evoluzione e l'espressione dei geni candidati utilizzando i dati di sequenziamento dell'RNA.
Abstract
Distillare e segnalare set di dati di grandi dimensioni, come i dati sull'intero genoma o sul trascrittame, è spesso un compito scoraggiante. Un modo per abbattere i risultati è concentrarsi su una o più famiglie geniche che sono significative per l'organismo e lo studio. In questo protocollo, delineamo i passaggi bioinformatici per generare una filogenesi e quantificare l'espressione dei geni di interesse. Gli alberi filogenetici possono dare un'idea di come i geni si stanno evolvendo all'interno e tra le specie e rivelare l'ortografia. Questi risultati possono essere migliorati utilizzando i dati RNA-seq per confrontare l'espressione di questi geni in diversi individui o tessuti. Studi sull'evoluzione molecolare e sull'espressione possono rivelare modi di evoluzione e conservazione della funzione genica tra le specie. La caratterizzazione di una famiglia genica può servire come trampolino di lancio per studi futuri e può evidenziare un'importante famiglia genica in un nuovo genoma o carta del trascrittame.
Introduzione
I progressi nelle tecnologie di sequenziamento hanno facilitato il sequenziamento di genomi e trascrittimi di organismi non modello. Oltre alla maggiore fattibilità del sequenziamento di DNA e RNA da molti organismi, è disponibile al pubblico un'abbondanza di dati per studiare i geni di interesse. Lo scopo di questo protocollo è quello di fornire misure bioinformatiche per indagare l'evoluzione molecolare e l'espressione di geni che possono svolgere un ruolo importante nell'organismo di interesse.
Studiare l'evoluzione di un gene o di una famiglia genica può fornire informazioni sull'evoluzione dei sistemi biologici. I membri di una famiglia genica sono tipicamente determinati identificando motivi conservati o sequenze geniche omologhe. L'evoluzione della famiglia genica è stata precedentemente studiata utilizzando genomi di organismi modellolontanamente correlati 1. Una limitazione a questo approccio è che non è chiaro come queste famiglie geniche si evolvano in specie strettamente correlate e il ruolo delle diverse pressioni selettive ambientali. In questo protocollo, includiamo la ricerca di omologhi in specie strettamente correlate. Generando una filogenesi a livello di phylum, possiamo notare tendenze nell'evoluzione della famiglia genica come quella dei geni conservati o duplicazioni specifiche del lignaggio. A questo livello, possiamo anche indagare se i geni sono ortologi o paralogi. Mentre molti omologhi probabilmente funzionano in modo simile l'uno all'altro, questo non è necessariamente ilcaso 2. Incorporare alberi filogenetici in questi studi è importante per risolvere se questi geni omologhi sono ortologi o meno. Negli eucarioti, molti ortologi mantengono funzioni simili all'interno della cellula come evidenziato dalla capacità delle proteine dei mammiferi di ripristinare la funzione degli ortologi di lievito3. Tuttavia, ci sono casi in cui un gene non ortologo svolge una funzione caratterizzata4.
Gli alberi filogenetici iniziano a delineare le relazioni tra geni e specie, ma la funzione non può essere assegnata esclusivamente in base alle relazioni genetiche. Gli studi sull'espressione genica combinati con le annotazioni funzionali e l'analisi dell'arricchimento forniscono un forte supporto per la funzione genica. I casi in cui l'espressione genica può essere quantificata e confrontata tra individui o tipi di tessuto può essere più eloquente della funzione potenziale. Il seguente protocollo segue i metodi utilizzati per studiare i geni opsina in Hydra vulgaris7, ma possono essere applicati a qualsiasi specie e famiglia genica. I risultati di tali studi forniscono una base per ulteriori indagini sulla funzione genica e sulle reti geniche negli organismi non modello. Ad esempio, l'indagine sulla filogenesi delle opsine, che sono proteine che avviano la cascata di fototrasduzione, dà contesto all'evoluzione degli occhi e al rilevamento dellaluce 8,9,10,11. In questo caso, organismi non modello, in particolare specie animali basali come cnidari o ctenofori, possono chiarire la conservazione o i cambiamenti nella cascata di fototraduzione e nella visione attraverso i cladi12,13,14. Allo stesso modo, determinare la filogenesi, l'espressione e le reti di altre famiglie geniche ci informerà sui meccanismi molecolari alla base degli adattamenti.
Protocollo
Questo protocollo segue le linee guida per la cura degli animali uc irvine.
1. Preparazione della libreria RNA-seq
- Isolare l'RNA utilizzando i seguenti metodi.
- Raccogliere campioni. Se l'RNA deve essere estratto in un secondo momento, flash congelare il campione o posizionare nella soluzione distoccaggio dell'RNA 15 (Tabella dei materiali).
- Eutanasia e sezionare l'organismo per separare i tessuti di interesse.
- Estrarre l'RNA totale utilizzando un kit di estrazione e purificare l'RNA utilizzando un kit di purificazione dell'RNA(Table of Materials)
NOTA: Ci sono protocolli e kit che possono funzionare meglio per diverse specie e tipi di tessuto16,17. Abbiamo estratto l'RNA da diversi tessuti corporei di una farfalla 18 e un'Idra gelatinosa19 (vedi discussione). - Misurare la concentrazione e la qualità dell'RNA di ciascun campione (Tabella dei materiali). Utilizzare campioni con numeri di integrità dell'RNA (RIN) superiori a 8, idealmente più vicini a 920 per costruire librerie cDNA.
- Costruire la libreria e la sequenza cDNA come segue.
- Creare librerie cDNA in base al manuale di istruzioni di preparazione della libreria (vedere la discussione).
- Determinare la concentrazione e la qualità del cDNA(Tabella dei materiali).
- Multiplex le librerie e sequenziarle.
2. Accedere a un cluster di computer
NOTA: l'analisi RNA-seq richiede la manipolazione di file di grandi dimensioni ed è meglio eseguita su un cluster di computer (Table of Materials).
- Accedere all'account cluster del computer utilizzando il comando ssh username@clusterlocation in una finestra dell'applicazione terminale (Mac) o PuTTY (Windows).
3. Ottenere letture RNA-seq
- Ottenere letture RNA-seq dalla funzione di sequenziamento o, per i dati generati in una pubblicazione, dal archivio dati in cui è stato depositato (3.2 o 3.3).
- Per scaricare dati da repository come ArrayExpress, eseguire le operazioni seguenti:
- Cercare nel sito utilizzando il numero di adesione.
- Individuare il collegamento per scaricare i dati, quindi fare clic con il pulsante sinistro del mouse e scegliere Copia collegamento.
- Nella finestra terminale digitare wget e selezionare Incolla collegamento per copiare i dati nella directory per l'analisi.
- Per scaricare i dati di NCBI Short Read Archive (SRA), attenersi alla seguente procedura alternativa:
- Nel terminale scaricare SRA Toolkit v. 2.8.1 utilizzando wget.
NOTA: il download e l'installazione di programmi nel cluster di computer potrebbe richiedere l'accesso root, contattare l'amministratore del cluster di computer in caso di esito negativo dell'installazione. - Completare l'installazione del programma digitando tar -xvf $TARGZFILE.
- Cercare in NCBI il numero di adesione SRA per gli esempi che si desidera scaricare, che dovrebbe avere il formato SRRXXXXXX.
- Ottenere i dati RNA-seq digitando [sratoolkit location]/bin/prefetch SRRXXXXXX nella finestra terminale.
- Per i file con estremità accoppiata, digitare [sratoolkit location]/bin/fastq-dump --split-files SRRXXXXXX per ottenere due file fastq (SRRXXXXXX_1.FASTQ e SRRXXXXXX_2.FASTQ).
NOTA: per eseguire un assembly Trinity de novo, utilizzare il comando [sratoolkit location]/bin/fastq-dump --defline-seq '@$sn[_$rn]/$ri' --split-files SRRXXXXXXXX
- Nel terminale scaricare SRA Toolkit v. 2.8.1 utilizzando wget.
4. Tagliare adattatori e letture di bassa qualità (opzionale)
- Installare o caricare Trimmomatic21 v. 0.35 nel cluster ing computing.
- Nella directory in cui si trovano i file di dati RNA-seq digitare un comando che includa la posizione del file jar trimmomatico, i file FASTQ di input, i file FASTQ di output e parametri facoltativi come lunghezza e qualità di lettura.
NOTA: il comando varierà in base alla qualità e alla lunghezza grezze e desiderate delle letture. Per Illumina 43 bp letture con primer Nextera, abbiamo usato: java -jar /data/apps/trimmomatic/0.35/trimmomatic-0.35.jar PE $READ 1. Fastq $READ 2. Fastq paired_READ1. Fastq unpaired_READ1. Fastq paired_READ2. Fastq unpaired_READ2. FASTQ ILLUMINACLIP:adapters.fa:2:30:10 LEADING:20 TRAILING:20 SLIDINGWINDOW:4:17 MINLEN:30.
5. Ottenere l'assieme di riferimento
- Cerca google, EnsemblGenomes e NCBI Genomes and Nucleotide TSA (Transcriptome Shotgun Assembly) per un genoma di riferimento o un trascrittame assemblato per le specie di interesse (Figura 1).
NOTA: Se un genoma di riferimento o un trascritoma non sono disponibili o di bassa qualità, procedere al PASSAGGIO 6 per generare un assieme de novo. - Se esiste un genoma di riferimento o un trascrittame assemblato, scaricarlo come file fasta in cui verrà eseguita l'analisi seguendo i passaggi seguenti.
- Trovare il collegamento per scaricare il genoma, fare clic con il pulsante sinistro del mouse e copiare link.
- Nella finestra terminale digitare wget e incollare l'indirizzo del collegamento. Se disponibile, copiare anche il file GTF e il file FASTA proteico per il genoma di riferimento.
6. Generare un assieme de novo (alternativo al passaggio 5)
- Combina i file fastq RNA-seq READ1 e READ2 per tutti gli esempi digitando cat *READ1. FASTQ > $all_READ1. FASTQ e gatto *READ2. Fastq > all_READ2. FASTQ nella finestra del terminale.
- Installare o caricare Trinity22 v.2.8.5 nel cluster di elaborazione.
- Genera e genera l'assembly digitando sul terminale: Trinity --seqType fq --max_memory 20G --left $all_READ1. FASTQ --right $all_READ2. Funzione FASTQ.
7. La mappa legge sul genoma (7.1) o sul trascrittame de novo (7.2)
- La mappa legge sul genoma di riferimento usando STAR23 v. 2.6.0c e RSEM24 v. 1.3.0.
- Installare o caricare STAR v. 2.6.0c. rsem v. 1.3.0 al cluster ing computing.
- Indicizzare il genoma digitando rsem-prepare-reference --gtf $GENOME. GTF --stella -p 16 $GENOME. Fasta $OUTPUT.
- La mappa legge e calcola l'espressione per ogni campione digitando rsem-calculate-expression -p 16 --star --paired-end $READ 1. Fastq $READ 2. Fastq $INDEX $OUTPUT.
- Rinominare il file dei risultati in qualcosa di descrittivo utilizzando mv RSEM.genes.results $sample.genes.results.
- Generare una matrice di tutti i conteggi digitando rsem-generate-data-matrix *[genes/isoforms.results] > $OUTPUT.
- Mappare l'RNA-seq all'assieme Trinity de novo usando RSEM e bowtie.
- Installare o caricare Trinity22 v.2.8.5, Bowtie25 v. 1.0.0 e RSEM v. 1.3.0.
- La mappa legge e calcola l'espressione per ogni esempio digitando [trinity_location]/align_and_estimate_abundance.pl --prep-reference --transcripts $TRINITY. FASTA --seqType fq --left $READ 1. FASTQ -- $READ 2. FASTQ --est_method RSEM --aln_method bowtie --trinity_mode --output_dir $OUTPUT.
- Rinominare il file dei risultati in qualcosa di descrittivo utilizzando mv RSEM.genes.results $sample.genes.results.
- Generare una matrice di tutti i conteggi digitando [trinity_location]/abundance_estimates_to_matrix.pl --est_method RSEM *[geni/isoforme].results
8. Identificare i geni di interesse
NOTA: I seguenti passaggi possono essere eseguiti con file FASTA nucleotidici o proteici, ma funzionano meglio e sono più semplici con sequenze proteiche. Blast cerca utilizzando proteine e proteine è più probabile che dia risultati durante la ricerca tra diverse specie.
- Per un genoma di riferimento, utilizzare il file FASTA proteico di STEP 5.2.2 o vedere Materiali supplementari per generare una funzione genica personalizzata GTF.
- Per un transcriptome de novo, generare una proteina FASTA usando TransDecoder.
- Installare o caricare TransDecoder v. 5.5.0 nel computer cluser.
- Individuare il fotogramma di lettura aperto più lungo e la sequenza peptidica prevista digitando [Posizione transdecoder]/TransDecoder.LongOrfs -t $TRINITY. Fasta.
- Cerca in NCBI Genbank omologi in specie strettamente correlate.
- Aprire una finestra del browser Internet e passare a https://www.ncbi.nlm.nih.gov/genbank/.
- Sulla barra di ricerca digitare il nome del gene di interesse e il nome di specie strettamente imparentate che sono state sequenziate o genere o phylum. A sinistra della barra di ricerca selezionare proteine, quindi fare clic su cerca.
- Estrarre sequenze facendo clic su Invia a e quindi selezionare File. In Formato selezionare FASTA e quindi fare clic su Crea file.
- Spostare il file FASTA degli omologhi nel cluster di computer digitando scp $FASTA username@clusterlocation:/$DIR in una finestra terminale locale oppure utilizzare FileZilla per trasferire file da e verso computer e cluster.
- Cercare geni candidati utilizzando BLAST+26.
- Installare o caricare BLAST+ v. 2.8.1 nel cluster di computer.
- Nel cluster di computer, creare un database BLAST dal genoma o dalla proteina tradotta del trascritta FASTA digitando [POSIZIONE BLAST+]/makeblastdb -in $PEP. Fasta -dbtype prot -out $OUTPUT
- BLAST le sequenze geniche omologhe da NCBI al database delle specie di interesse digitando [BLAST+ location]/blastp -db $DATABASE -query $FASTA -evalue 1e-10 -outfmt 6 -max_target_seqs 1 -out $OUTPUT.
- Visualizzare il file di output utilizzando il comando more. Copia ID gene univoci dalle specie di interesse in un nuovo file di testo.
- Estrarre le sequenze di geni candidati digitando perl -ne 'if(/^>(\S+)/){$c=$i{$1}}$c?print:chomp;$i{$_}=1 if @ARGV' $gene_id.txt $PEP. Fasta > $OUTPUT.
- Conferma l'annotazione genica usando blast reciproco.
- Sul browser Internet vai a https://blast.ncbi.nlm.nih.gov/Blast.cgi.
- Selezionare tblastn, quindi incollare le sequenze candidate, selezionare il database delle sequenze proteiche non ridondanti e fare clic su BLAST.
- Identificare geni aggiuntivi annotando tutti i geni nel genoma o nel trascritoma con termini di ontologia genica (GO) (vedi discussione).
- Trasferire la proteina FASTA sul computer locale.
- Scaricare e installare Blast2GO27,28,29 v. 5.2 nel computer locale.
- Aprire Blast2GO, fare clic su File, scegliere Carica, Carica sequenze ,fare clic su Carica file Fasta (fasta). Selezionare il file FASTA e fare clic su Carica.
- Fate clic su Esplosione (Blast), selezionate Esplosione NCBI (NCBI Blast)e fate clic su Avanti (Next). Modificare i parametri o fare clic suAvanti , modificare i parametri e fare clic su Esegui per trovare la descrizione genica più simile.
- Fare clic su mapping, quindi su Esegui per cercare proteine simili nelle annotazioni gene ontologiche.
- Fare quindi clic su interpro, selezionare EMBL-EBI InterProe fare clic su Avanti. Modificare i parametri o fare clicsu Avanti e quindi su Esegui per cercare le firme delle famiglie e dei domini genetici noti.
- Esportare le annotazioni facendo clic su File, selezionare Esporta, fare clic su Esporta tabella. Fare clic suSfoglia , assegnare un nome al file, fare clic suSalva , su Esporta.
- Cercare nella tabella di annotazione i termini di interesse GO per identificare altri geni candidati. Estrarre le sequenze dal file FASTA (STEP 8.4.5)
9. Alberi filogenetici
- Scaricare e installare MEGA30 v. 7.0.26 nel computer locale.
- Aprire MEGA, fare clic su Allinea, su Modifica/Allineamento compilazione, selezionare Crea un nuovo allineamento, fare clic su OK, selezionare Proteina.
- All'apertura della finestra di allineamento, fare clic su Modifica, quindi su Inserisci sequenze dal file e selezionare fasta con sequenze proteiche di geni candidati e probabili omologhi.
- Selezionare tutte le sequenze. Trova il simbolo del braccio e passa il mouse su di esso. Dovrebbe dire Allineare le sequenze usando l'algoritmo MUSCLE31. Fare clic sul simbolo del braccio e quindi su Allinea proteine per allineare le sequenze. Modificare i parametri o fare clic su OK per allinearli utilizzando i parametri predefiniti.
- Ispezionare visivamente e apportare eventuali modifiche manuali, quindi salvare e chiudere la finestra di allineamento.
- Nella finestra MEGA principale fare clic su Modelli, fare clic su Trova migliori modelli DNA/Proteine (ML),selezionare il file di allineamento e selezionare i parametri corrispondenti quali: Analisi: Selezione modello (ML), Albero da utilizzare: Automatico (albero adiacente), Metodo statistico: Massima verosimiglianza, Tipo di sostituzione: Amminoacido, Gap/trattamento dati mancanti: Utilizzare tutti i siti, Filtro sito di succursale: Nessuno.
- Una volta determinato il modello migliore per i dati, vai alla finestra MEGA principale. Fate clic su Filogenesi (Phylogeny), quindi selezionate Albero probabilità massima contruct/test (Contruct/Test Maximum Likelihood Tree), quindi selezionate l'allineamento, se necessario. Selezionare i parametri appropriati per l'albero: Metodo statistico: Massima verosimiglianza, Test di Filogenesi: Metodo Bootstrap con 100 repliche, tipo di sostituzione: amminoacido, modello: LG con Freqs. (+F), tassi tra i siti: gamma distribuita (G) con 5 categorie gamma discrete, trattamento gap/dati mancanti: utilizzare tutti i siti, metodo euristico ML: Nearest-Neighbor-Interchange (NNI).
10. Visualizzare l'espressione genica usando TPM
- Per Trinity, nel cluster di computer passare alla directory in cui è stato abundance_estimates_to_matrix.pl e uno degli output deve essere matrix. TPM.not_cross_norm. Trasferire il file nel computer locale.
NOTA: Vedere Materiali supplementari per la normalizzazione del campione incrociato. - Per i TPM da un'analisi del genoma seguire i passaggi seguenti.
- Nel cluster di computer passare al percorso di installazione rsem. Copiare rsem-generate-data-matrix digitando scp rsem-generate-data-matrix rsem-generate-TPM-matrix. Usa nano per modificare il nuovo file e cambiare "my $offsite = 4" da 4 a 5 per TPM, ora dovrebbe leggere "my $offsite = 5".
- Passare alla directory in cui si trovano i file di output RSEM .genes.results e ora utilizzare rsem-generate-TPM-matrix *[genes/isoforms.results] > $OUTPUT per generare una matrice TPM. Trasferire i risultati in un computer locale.
- Visualizzare i risultati in ggplot2.
- Scaricare R v. 4.0.0 e RStudio v. 1.2.1335 in un computer locale.
- Aprire RStudio a destra dello schermo, passare alla scheda Pacchetti e fare clic su Installa. Digitare ggplot2 e fare clic su installa.
- Nella finestra dello script R leggere nella tabella TPM digitando data<-read.table("$tpm.txt",header = T)
- Per i grafici a barre simili alla figura 4 digitare qualcosa di simile a: p<- ggplot() + geom_bar(aes(y=TPM, x=Symbol, fill=Tissue), data=data, stat="identity")
fill<-c("#d7191c","#fdae61", "#ffffbf", "#abd9e9", "#2c7bb6")
p<-p+scale_fill_manual(values=fill)
p + theme(axis.text.x = element_text(angolo = 90))
Risultati
I metodi di cui sopra sono riassunti nella figura 1 e sono stati applicati a un insieme di dati di tessuti Hydra vulgaris. H. vulgaris è un invertebrato di acqua dolce che appartiene al phylum Cnidaria che comprende anche coralli, meduse e anemoni di mare. H. vulgaris può riprodursi asessualmente germogliando e possono rigenerare la testa e il piede quando vengono bisessuali. In questo studio, abbiamo mirato a indagare l'evoluzione e l'espressione dei geni opsina in Hydra7. Mentre Hydra manca di occhi, mostrano un comportamento dipendente dallaluce 32. I geni Opsin codificano proteine importanti nella visione per rilevare diverse lunghezze d'onda della luce e iniziare la cascata di fototrasduzione. Studiare l'evoluzione molecolare e l'espressione di questa famiglia genica in una specie basale può fornire informazioni sull'evoluzione degli occhi e sul rilevamento della luce negli animali.
Abbiamo generato un assemblaggio guidato utilizzando il genoma di riferimento Hydra2.0 33 e i dati RNA-seq disponibili al pubblico (GEO accession GSE127279) Figura 1. Questo passaggio ha richiesto circa 3 giorni. Anche se non abbiamo generato un transcriptome de novo in questo caso, un assembly Trinity può richiedere fino a 1 settimana per essere generato e ogni libreria può richiedere alcune ore per la mappatura di lettura a seconda del mappatore. L'assembly Hydra unito (~50.000 trascrizioni) è stato annotato utilizzando Blast2GO che ha richiesto circa 1 settimana Figura 1. Sequenze per geni correlati all'opsina sono state estratte in un file fasta. Sequenze per geni opsin di altre specie sono state estratte anche da NCBI GenBank. Abbiamo usato opsine di cnidari Podocoryna carnea, Cladonema radiatum, Tripedelia cystophorae Nematostella vectensis,e abbiamo anche incluso gruppi fuori gruppo Mnemiopsis leidyi, Trichoplax adhaerens, Drosophila melanogaster e Homo sapiens. I geni opsin sono stati allineati nella FIGURA 2 delMEGA7 . Osservando l'allineamento, siamo stati in grado di identificare le opsine hydra che mancavano di un amminoacido di lisina conservato necessario per legare una molecola sensibile alla luce. Dopo l'ispezione visiva, abbiamo determinato il modello migliore eseguendo un'analisi di selezione del modello. È stato generato un albero con massima verosimiglianza utilizzando il modello LG + G + F con valore bootstrap pari a 100 Figura 3. Per 149 geni opsina, l'albero è stato terminato in circa 3 giorni. La filogenesi suggerisce che i geni opsin si stanno evolvendo per duplicazioni specifiche del lignaggio nei cnidari e potenzialmente per duplicazione tandem in H. vulgaris7.
Abbiamo eseguito un'analisi dell'espressione differenziale in edgeR e abbiamo esaminato l'espressione assoluta dei geni opsina. Abbiamo ipotizzato che una o più opsine sarebbero state upregolate nella testa (ipostoma) ed eseguito confronti a coppie di ipostoma rispetto alla colonna corporea, alla zona in erba, al piede e ai tentacoli. Come esempio di confronto a coppie, 1.774 trascrizioni sono state espresse in modo differenziato tra l'ipostoma e la colonna corporea. Abbiamo determinato i geni che sono stati upregolamentati attraverso confronti multipli e abbiamo fatto un arricchimento funzionale nella tabella 1 diBlast2GO . Il raggruppamento dell'attività del recettore accoppiato con la proteina G includeva geni opsina. Infine, abbiamo esaminato l'espressione assoluta dei geni opsina nei diversi tessuti, durante lo germogliamento e durante la rigenerazione tracciando i loro valori di TPM usando la figura ggplot 4. Usando i metodi qui descritti, abbiamo identificato 2 geni opsina che non si raggruppavano con le altre opsine nella filogenesi, abbiamo trovato un'opsina che è stata espressa quasi 200 volte di più di altre, e abbiamo trovato alcuni geni opsin co-espressi con geni di fototrasduzione che possono essere utilizzati per il rilevamento della luce.
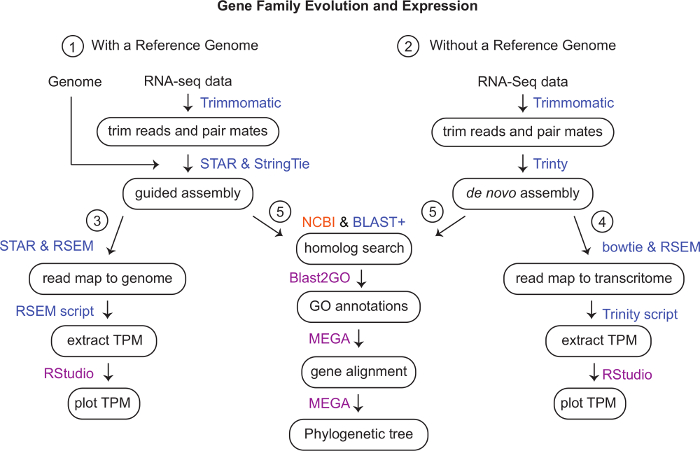
Figura 1: Schema del flusso di lavoro. I programmi utilizzati per analizzare i dati sul cluster di computer sono in blu, in magenta sono quelli che abbiamo usato su un computer locale e in arancione è un programma basato sul web. (1) Tagliare le letture dell'RNA-seq utilizzando trimmomatico v. 0.35. Se un genoma è disponibile ma mancano modelli genetici, generare un assieme guidato utilizzando STAR v. 2.6.0c e StringTie v. 1.3.4d. (Facoltativo vedi Materiali supplementari) (2) Senza genoma di riferimento, utilizzare letture tagliate per effettuare un assemblaggio de novo utilizzando Trinity v 2.8.5. (3) Per quantificare l'espressione genica utilizzando un genoma di riferimento, la mappa legge usando STAR e quantifica utilizzando RSEM v. 1.3.1. Estrarre i TPM utilizzando RSEM e visualizzarli in RStudio. (4) Bowtie e RSEM possono essere utilizzati per mappare e quantificare le letture mappate a un trascrittoma della trinità. Uno script Trinity può essere utilizzato per generare una matrice TPM per visualizzare i conteggi in RStudio. (5) Utilizzare NCBI BLAST basato sul web e blast+ della riga di comando per cercare sequenze omologhe e confermare l'utilizzo di BLAST reciproco. Annotare ulteriormente i geni usando Blast2GO. Usa MEGA per allineare i geni e generare un albero filogenetico utilizzando il modello più adatto. Clicca qui per visualizzare una versione più grande di questa figura.
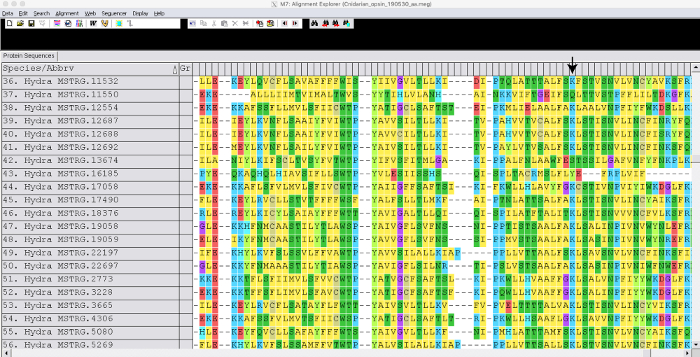
Figura 2: Esempio di geni allineati. L'istantanea mostra una porzione di geni dell'idra opsina allineati usando MUSCLE. La freccia indica la posizione di una llysina conservata legatura retinica. Clicca qui per visualizzare una versione più grande di questa figura.
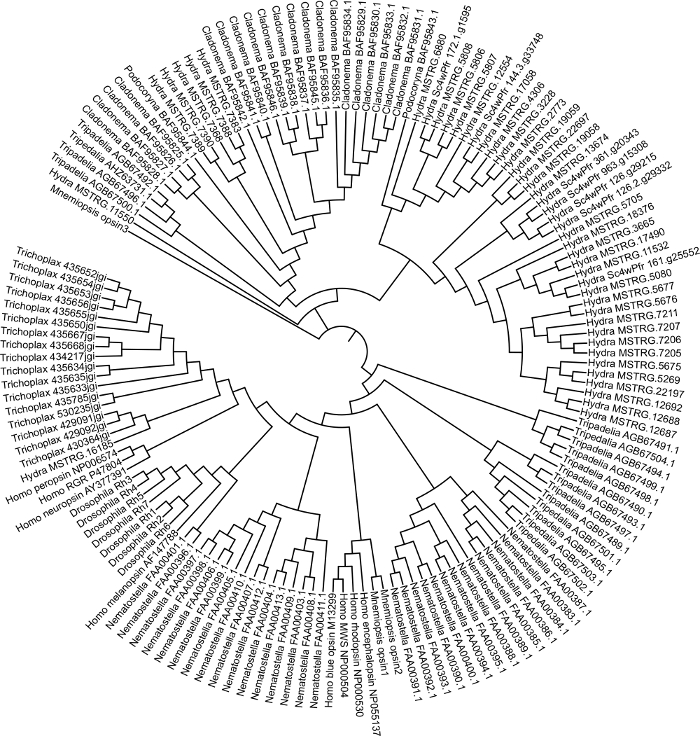
Figura 3: Albero filogenetico dell'opsina cnidaria. Albero ad alta probabilità generato in MEGA7 utilizzando sequenze di opsina da Hydra vulgaris, Podocoryna carnea, Cladonema radiatum, Tripedelia cystophora, Nematostella vectensis, Mnemiopsis leidyi, Trichoplax adhaerens, Drosophila melanogaster e Homo sapiens. Clicca qui per visualizzare una versione più grande di questa figura.
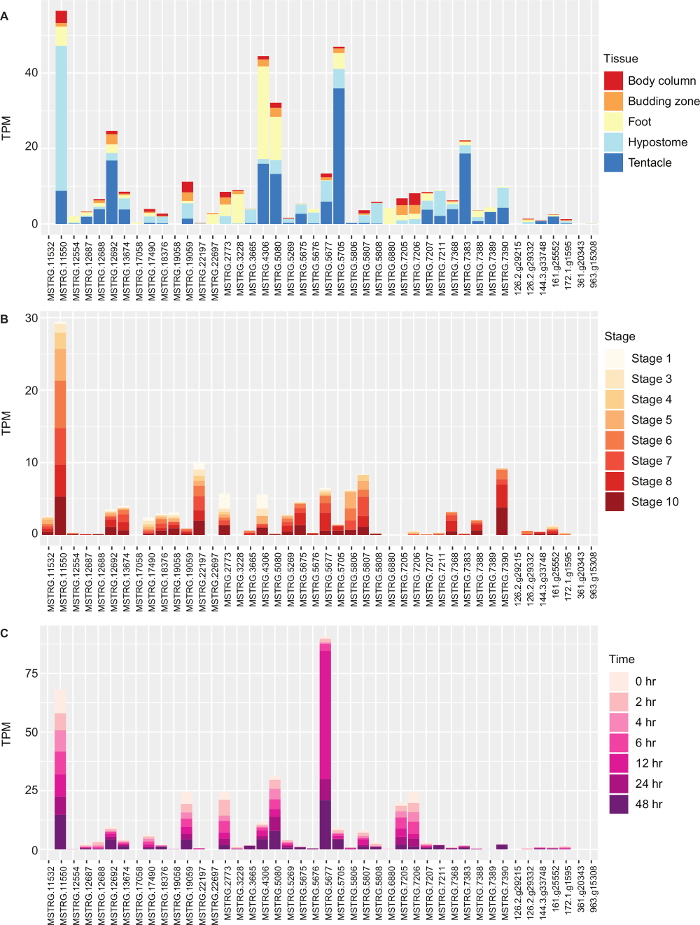
Figura 4: Espressione dei geni Opsin in Hydra vulgaris. (A) Espressione nelle trascrizioni per milione (TPM) dei geni Hydra vulgaris opsin nella colonna corporea, nella zona in erba, nel piede, nell'ipotoma e nei tentacoli. (B) Espressione di geni opsina durante diverse fasi di germogliatura dell'Idra. (C) Espressione dei geni opsina dell'ipostoma dell'Idra durante diversi punti di rigenerazione del tempo. Clicca qui per visualizzare una versione più grande di questa figura.
| GO ID | Nome GO | Categoria GO | Fdr |
| VAI:0004930 | Attività del recettore accoppiato con proteina G | FUNZIONE MOLECOLARE | 0.0000000000704 |
| VAI:0007186 | Via di segnalazione del recettore accoppiato della proteina G | PROCESSO BIOLOGICO | 0.00000000103 |
| VAI:0016055 | Percorso di segnalazione Wnt | PROCESSO BIOLOGICO | 0.0000358 |
| VAI:0051260 | omooligomerizzazione proteica | PROCESSO BIOLOGICO | 0.000376 |
| VAI:0004222 | metalloendopeptidasi | FUNZIONE MOLECOLARE | 0.000467 |
| GO:0008076 | complesso di canali di potassio gated di tensione | COMPONENTE CELLULARE | 0.000642 |
| VAI:0005249 | tensione gated attività del canale del potassio | FUNZIONE MOLECOLARE | 0.00213495 |
| VAI:0007275 | sviluppo di organismi multicellulari | PROCESSO BIOLOGICO | 0.00565048 |
| VAI:0006813 | trasporto ioni di potassio | PROCESSO BIOLOGICO | 0.01228182 |
| VAI:0018108 | fosforilazione peptidil-tirosina | PROCESSO BIOLOGICO | 0.02679662 |
Tabella 1: Arricchimento funzionale dei geni upregolati nell'ipostoma
Materiali supplementari. Clicca qui per scaricare questi materiali.
Discussione
Lo scopo di questo protocollo è quello di fornire una descrizione dei passaggi per caratterizzare una famiglia genica utilizzando i dati RNA-seq. Questi metodi hanno dimostrato di funzionare per una varietà di specie e set di dati4,34,35. Il gasdotto qui stabilito è stato semplificato e dovrebbe essere abbastanza facile da essere seguito da un principiante della bioinformatica. Il significato del protocollo è che delinea tutti i passaggi e i programmi necessari per completare un'analisi pubblicabile. Un passo cruciale nel protocollo è avere trascrizioni a figura intera correttamente assemblate, questo proviene da genomi o trascritti di alta qualità. Per ottenere trascrizioni corrette, è necessario RNA e/o DNA di alta qualità e buone annotazioni discusse di seguito.
Per la preparazione della libreria RNA-seq, includiamo kit di elenco che hanno funzionato per piccole parti del corpo di Hydra19 e farfalle 18 (Tavolo dei materiali). Notiamo che per l'RNA a basso input abbiamo utilizzato un approccio al protocollomodificato 36. I metodi per l'estrazione dell'RNA sono stati confrontati in più tipi di campioni tra cuicellule di lievito 17,neuroblastoma37,piante 38e larve diinsetti 16 per citarne alcuni. Si consiglia al lettore di acquisire un protocollo che funzioni per le proprie specie di interesse, se presenti, o di risolvere i problemi utilizzando kit comunemente disponibili in commercio per iniziare. Per una corretta quantificazione genica, si consiglia di trattare il campione di RNA con la DNasi. La presenza di DNA influenzerà una corretta quantificazione genica. Si consiglia inoltre di utilizzare un kit di preparazione della libreria cDNA che include una selezione di code in poliA da selezionare per l'mRNA maturo. Mentre l'esaurimento dell'rRNA si traduce in una maggiore profondità di lettura, la percentuale di copertura dell'esone è molto inferiore alla copertura esonero dell'RNA utilizzando la selezione polyA +39. Infine, quando possibile è meglio utilizzare l'estremità accoppiata e spiaggiata40,41. Nel protocollo sopra i comandi di mapping di lettura dovranno essere modificati quando si utilizzano letture a singola estremità.
Come accennato in precedenza, è importante essere in grado di identificare i geni di interesse e anche di distinguere tra recenti duplicazioni geniche, giunzione alternativa e aplotipi nel sequenziamento. In alcuni casi, avere un genoma di riferimento può aiutare determinando dove si trovano geni ed esoni l'uno rispetto all'altro. Una cosa da notare è che se un trascritoma è ottenuto da un database pubblico e non è di alta qualità, potrebbe essere meglio generare usando Trinity42 e combinando librerie RNA-seq da tessuti di interesse. Allo stesso modo, se un genoma di riferimento non ha buoni modelli genetici, le librerie RNA-seq possono essere utilizzate per generare nuove GTF utilizzando StringTie43 (vedi Materiali supplementari). Inoltre, nei casi in cui i geni sono incompleti e c'è accesso a un genoma, i geni possono essere modificati manualmente usando sequenze omologhe quindi allineate al genoma usando tblastn. L'uscita BLAST può essere utilizzata per determinare la sequenza effettiva, che può essere diversa dalla correzione eseguita utilizzando gli omologhi. Se non c'è corrispondenza, lasciare la sequenza come era originariamente. Quando si controlla l'output prestare attenzione alle coordinate del genoma per assicurarsi che l'esone mancante sia effettivamente parte del gene.
Sebbene ci concentriamo su software e programmi che abbiamo usato, esistono modifiche a questo protocollo a causa di molti programmi disponibili che potrebbero funzionare meglio per diversi set di dati. Ad esempio, mostriamo i comandi per la mappatura delle letture al transcriptome usando bowtie e RSEM, ma Trinity ora ha la possibilità di allineare molto più velocemente come kallisto44 e salmon45. Allo stesso modo, descriviamo le annotazioni usando Blast2GO (ora OmicsBox) ma ci sono altri strumenti di mapper che possono essere trovati gratuitamente e online. Alcuni che abbiamo provato includono: GO FEAT46, eggNOG-mapper47,48e un allineatore molto veloce PANNZER249. Per utilizzare questi strumenti di annotazione basati sul Web è sufficiente caricare il peptide FASTA e inviarlo. Versioni autonome di PANNZER e eggNOG-mapper sono disponibili anche per il download nel cluster di computer. Un'altra modifica è che abbiamo usato MEGA e R su un computer locale e utilizzato lo strumento NCBI BLAST online per fare BLAST reciproci, tuttavia tutti questi programmi possono essere utilizzati sul cluster di computer scaricando i programmi e i database necessari. Allo stesso modo, gli allineatori kallisto e salmone possono essere utilizzati su un computer locale purché un utente abbia abbastanza RAM e spazio di archiviazione. Tuttavia, i file FASTQ e FASTA tendono ad essere molto grandi e si consiglia vivamente di utilizzare un cluster di computer per facilità e velocità. Inoltre, mentre forniamo istruzioni e link per scaricare programmi dai loro sviluppatori, molti di loro possono essere installati da bioconda: https://anaconda.org/bioconda.
Un problema comune affrontato quando si esvolvano analisi bioinformatiche è il fallimento degli script shell. Ciò può essere dovuto a una serie di motivi. Se viene creato un file di errore, questo file di errore deve essere controllato prima della risoluzione dei problemi. Alcuni motivi comuni di un errore sono errori di battitura, parametri chiave mancanti e problemi di compatibilità tra le versioni del software. In questo protocollo, includiamo parametri per i dati, ma i manuali software possono fornire linee guida più dettagliate per i singoli parametri. In generale, è meglio utilizzare le versioni più aggiornate del software e consultare il manuale corrispondente a quella versione.
I miglioramenti apportati a questo protocollo includono l'analisi dell'espressione differenziale a livello di trascrittame e l'analisi dell'arricchimento funzionale. Si consiglia edgeR50 per l'analisi dell'espressione differenziale un pacchetto disponibile in Bioconductor. Per l'analisi dell'arricchimento funzionale, abbiamo utilizzato Blast2GO29 e DAVID51,52 basato sulweb. Si consiglia inoltre di modificare ulteriormente la filogenesi estraerla come file newick e utilizzando iTOL53basato sul Web. Inoltre, mentre questo protocollo studierà l'evoluzione molecolare e i modelli di espressione dei geni, ulteriori esperimenti possono essere utilizzati per convalidare le posizioni e le funzioni geniche o proteiche. L'espressione mRNA può essere confermata dall'ibridazione RT-qPCR o in situ. Le proteine possono essere localizzate usando l'immunoistochimica. A seconda della specie, gli esperimenti ad eliminazione diretta possono essere usati per confermare la funzione genica. Questo protocollo può essere utilizzato per una varietà di obiettivi tra cui, come mostrato sopra, esplorare una famiglia genica tipicamente associata alla fotorecezione in una specie basale7. Un'altra applicazione di questi metodi è l'identificazione dei cambiamenti in un percorso conservato sotto diverse pressioni selettive. Ad esempio, questi metodi sono stati utilizzati per scoprire variazioni nell'espressione dei canali potenziali del recettore transitorio della vista tra farfalle diurne e falene notturne34.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Ringraziamo Adriana Briscoe, Gil Smith, Rabi Murad e Aline G. Rangel per i consigli e le indicazioni nell'incorporare alcuni di questi passaggi nel nostro flusso di lavoro. Siamo anche grati a Katherine Williams, Elisabeth Rebboah e Natasha Picciani per i commenti sul manoscritto. Questo lavoro è stato sostenuto in parte da una borsa di studio della George E. Hewitt Foundation for Medical research presso A.M.M.
Materiali
| Name | Company | Catalog Number | Comments |
| Bioanalyzer-DNA kit | Agilent | 5067-4626 | wet lab materials |
| Bioanalyzer-RNA kit | Agilent | 5067-1513 | wet lab materials |
| BLAST+ v. 2.8.1 | On computer cluster* https://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/ | ||
| Blast2GO (on your PC) | On local computer https://www.blast2go.com/b2g-register-basic | ||
| boost v. 1.57.0 | On computer cluster | ||
| Bowtie v. 1.0.0 | On computer cluster https://sourceforge.net/projects/bowtie-bio/files/bowtie/1.3.0/ | ||
| Computing cluster (highly recommended) | NOTE: Analyses of genomic data are best done on a high-performance computing cluster because files are very large. | ||
| Cufflinks v. 2.2.1 | On computer cluster | ||
| edgeR v. 3.26.8 (in R) | In Rstudio https://bioconductor.org/packages/release/bioc/html/edgeR.html | ||
| gcc v. 6.4.0 | On computer cluster | ||
| Java v. 11.0.2 | On computer cluster | ||
| MEGA7 (on your PC) | On local computer https://www.megasoftware.net | ||
| MEGAX v. 0.1 | On local computer https://www.megasoftware.net | ||
| NucleoSpin RNA II kit | Macherey-Nagel | 740955.5 | wet lab materials |
| perl 5.30.3 | On computer cluster | ||
| python | On computer cluster | ||
| Qubit 2.0 Fluorometer | ThermoFisher | Q32866 | wet lab materials |
| R v.4.0.0 | On computer cluster https://cran.r-project.org/src/base/R-4/ | ||
| RNAlater | ThermoFisher | AM7021 | wet lab materials |
| RNeasy kit | Qiagen | 74104 | wet lab materials |
| RSEM v. 1.3.0 | Computer software https://deweylab.github.io/RSEM/ | ||
| RStudio v. 1.2.1335 | On local computer https://rstudio.com/products/rstudio/download/#download | ||
| Samtools v. 1.3 | Computer software | ||
| SRA Toolkit v. 2.8.1 | On computer cluster https://github.com/ncbi/sra-tools/wiki/01.-Downloading-SRA-Toolkit | ||
| STAR v. 2.6.0c | On computer cluster https://github.com/alexdobin/STAR | ||
| StringTie v. 1.3.4d | On computer cluster https://ccb.jhu.edu/software/stringtie/ | ||
| Transdecoder v. 5.5.0 | On computer cluster https://github.com/TransDecoder/TransDecoder/releases | ||
| Trimmomatic v. 0.35 | On computer cluster http://www.usadellab.org/cms/?page=trimmomatic | ||
| Trinity v.2.8.5 | On computer cluster https://github.com/trinityrnaseq/trinityrnaseq/releases | ||
| TRIzol | ThermoFisher | 15596018 | wet lab materials |
| TruSeq RNA Library Prep Kit v2 | Illumina | RS-122-2001 | wet lab materials |
| TURBO DNA-free Kit | ThermoFisher | AM1907 | wet lab materials |
| *Downloads and installation on the computer cluster may require root access. Contact your network administrator. |
Riferimenti
- Lespinet, O., Wolf, Y. I., Koonin, E. V., Aravind, L. The role of lineage-specific gene family expansion in the evolution of eukaryotes. Genome Research. 12 (7), 1048-1059 (2002).
- Gabaldón, T., Koonin, E. V. Functional and evolutionary implications of gene orthology. Nature Reviews Genetics. 14 (5), 360-366 (2013).
- Dolinski, K., Botstein, D. Orthology and Functional Conservation in Eukaryotes. Annual Review of Genetics. 41 (1), (2007).
- Macias-Muñoz, A., McCulloch, K. J., Briscoe, A. D. Copy number variation and expression analysis reveals a non-orthologous pinta gene family member involved in butterfly vision. Genome Biology and Evolution. 9 (12), 3398-3412 (2017).
- Cannon, S. B., Mitra, A., Baumgarten, A., Young, N. D., May, G. The roles of segmental and tandem gene duplication in the evolution of large gene families in Arabidopsis thaliana. BMC plant biology. 4, 10(2004).
- Eastman, S. D., Chen, T. H. P., Falk, M. M., Mendelson, T. C., Iovine, M. K. Phylogenetic analysis of three complete gap junction gene families reveals lineage-specific duplications and highly supported gene classes. Genomics. 87 (2), 265-274 (2006).
- Macias-Munõz, A., Murad, R., Mortazavi, A. Molecular evolution and expression of opsin genes in Hydra vulgaris. BMC Genomics. 20 (1), 1-19 (2019).
- Hisatomi, O., Tokunaga, F. Molecular evolution of proteins involved in vertebrate phototransduction. Comparative Biochemistry and Physiology - B Biochemistry and Molecular Biology. 133 (4), 509-522 (2002).
- Arendt, D. Evolution of eyes and photoreceptor cell types. International Journal of Developmental Biology. 47, 563-571 (2003).
- Shichida, Y., Matsuyama, T. Evolution of opsins and phototransduction. Philosophical Transactions of the Royal Society B: Biological Sciences. 364 (1531), 2881-2895 (2009).
- Porter, M. L., et al. Shedding new light on opsin evolution. Proceedings of the Royal Society B: Biological Sciences. 279 (1726), 3-14 (2012).
- Plachetzki, D. C., Degnan, B. M., Oakley, T. H. The origins of novel protein interactions during animal opsin evolution. PLoS ONE. 2 (10), 1054(2007).
- Ramirez, M. D., et al. The last common ancestor of most bilaterian animals possessed at least nine opsins. Genome Biology and Evolution. 8 (12), 3640-3652 (2016).
- Schnitzler, C. E., et al. Genomic organization, evolution, and expression of photoprotein and opsin genes in Mnemiopsis leidyi: a new view of ctenophore photocytes. BMC Biology. 10, 107(2012).
- Pedersen, K. B., Williams, A., Watt, J., Ronis, M. J. Improved method for isolating high-quality RNA from mouse bone with RNAlater at room temperature. Bone Reports. 11, 100211(2019).
- Ridgeway, J. A., Timm, A. E., Fallon, A. Comparison of RNA isolation methods from insect larvae. Journal of Insect Science. 14 (1), 4-8 (2014).
- Scholes, A. N., Lewis, J. A. Comparison of RNA isolation methods on RNA-Seq: Implications for differential expression and meta-Analyses. BMC Genomics. 21 (1), 1-9 (2020).
- Briscoe, A. D., et al. Female behaviour drives expression and evolution of gustatory receptors in butterflies. PLoS genetics. 9 (7), 1003620(2013).
- Murad, R., Macias-Muñoz, A., Wong, A., Ma, X., Mortazavi, A. Integrative analysis of Hydra head regeneration reveals activation of distal enhancer-like elements. bioRxiv. , 544049(2019).
- Gallego Romero, I., Pai, A. A., Tung, J., Gilad, Y. Impact of RNA degradation on measurements of gene expression. BMC Biology. 12, 42(2014).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Trinity. RNA-Seq De novo Assembly Using Trinity. , 1-7 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29, 15-21 (2013).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC bioinformatics. 12, 323(2011).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome biology. 10, 25(2009).
- Camacho, C., et al. BLAST+: architecture and applications. BMC Bioinformatics. 10, 421(2009).
- Conesa, A., Götz, S. Blast2GO: A comprehensive suite for functional analysis in plant genomics. International Journal of Plant Genomics. 619832, (2008).
- Conesa, A., et al. Blast2GO: A universal tool for annotation, visualization and analysis in functional genomics research. Bioinformatics. 21 (18), 3674-3676 (2005).
- Götz, S., et al. High-throughput functional annotation and data mining with the Blast2GO suite. Nucleic Acids Research. 36 (10), 3420-3435 (2008).
- Kumar, S., Stecher, G., Tamura, K. MEGA7: Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets. Molecular biology and evolution. 33 (7), 1870-1874 (2016).
- Edgar, R. C. MUSCLE: Multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Research. 32 (5), 1792-1797 (2004).
- Taddei-Ferretti, C., Musio, C., Santillo, S., Cotugno, A. The photobiology of Hydra's periodic activity. Hydrobiologia. 530, 129-134 (2004).
- Chapman, J. A., et al. The dynamic genome of Hydra. Nature. 464 (7288), 592-596 (2010).
- Macias-Muñoz, A., Rangel Olguin, A. G., Briscoe, A. D. Evolution of phototransduction genes in Lepidoptera. Genome Biology and Evolution. 11 (8), 2107-2124 (2019).
- Macias-Munõz, A., Murad, R., Mortazavi, A. Molecular evolution and expression of opsin genes in Hydra vulgaris. BMC Genomics. 20 (1), (2019).
- Picelli, S., et al. Full-length RNA-seq from single cells using Smart-seq2. Nature Protocols. 9 (1), 171-181 (2014).
- Tavares, L., Alves, P. M., Ferreira, R. B., Santos, C. N. Comparison of different methods for DNA-free RNA isolation from SK-N-MC neuroblastoma. BMC research notes. 4, 3(2011).
- Johnson, M. T. J., et al. Evaluating Methods for Isolating Total RNA and Predicting the Success of Sequencing Phylogenetically Diverse Plant Transcriptomes. PLoS ONE. 7 (11), (2012).
- Zhao, S., Zhang, Y., Gamini, R., Zhang, B., Von Schack, D. Evaluation of two main RNA-seq approaches for gene quantification in clinical RNA sequencing: PolyA+ selection versus rRNA depletion. Scientific Reports. 8 (1), 1-12 (2018).
- Zhao, S., et al. Comparison of stranded and non-stranded RNA-seq transcriptome profiling and investigation of gene overlap. BMC Genomics. 16 (1), 1-14 (2015).
- Corley, S. M., MacKenzie, K. L., Beverdam, A., Roddam, L. F., Wilkins, M. R. Differentially expressed genes from RNA-Seq and functional enrichment results are affected by the choice of single-end versus paired-end reads and stranded versus non-stranded protocols. BMC Genomics. 18 (1), 1-13 (2017).
- Haas, B. J., et al. De novo transcript sequence reconstruction from RNA-seq using the Trinity platform for reference generation and analysis. Nature Protocols. 8 (8), 1494-1512 (2013).
- Pertea, M., et al. StringTie enables improved reconstruction of a transcriptome from RNA-seq reads. Nature biotechnology. 33 (3), 290-295 (2015).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nature Biotechnology. 34 (5), 525-527 (2016).
- Patro, R., Duggal, G., Love, M. I., Irizarry, R. A., Kingsford, C. Salmon provides fast and bias-aware quantification of transcript expression. Nature Methods. 14 (4), 417-419 (2017).
- Araujo, F. A., Barh, D., Silva, A., Guimarães, L., Thiago, R. OPEN GO FEAT a rapid web-based functional annotation tool for genomic and transcriptomic data. , 8-11 (2018).
- Huerta-Cepas, J., et al. Fast genome-wide functional annotation through orthology assignment by eggNOG-mapper. Molecular Biology and Evolution. 34 (8), 2115-2122 (2017).
- Huerta-Cepas, J., et al. EggNOG 5.0: A hierarchical, functionally and phylogenetically annotated orthology resource based on 5090 organisms and 2502 viruses. Nucleic Acids Research. 47, 309-314 (2019).
- Törönen, P., Medlar, A., Holm, L. PANNZER2: A rapid functional annotation web server. Nucleic Acids Research. 46, 84-88 (2018).
- Robinson, M., Mccarthy, D., Chen, Y., Smyth, G. K. edgeR differential expression analysis of digital gene expression data User's Guide. , (2013).
- Huang, D. W., Sherman, B. T., Lempicki, R. A. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources. Nature Protocols. 4 (1), 44-57 (2009).
- Huang, D. W., Sherman, B. T., Lempicki, R. A. Bioinformatics enrichment tools: Paths toward the comprehensive functional analysis of large gene lists. Nucleic Acids Research. 37 (1), 1-13 (2009).
- Letunic, I., Bork, P. Interactive tree of life (iTOL) v3: an online tool for the display and annotation of phylogenetic and other trees. Nucleic acids research. 44, 242-245 (2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon