Method Article
Идентификация альтернативного сплайсинга и полиаденилирования в данных RNA-seq
В этой статье
Резюме
Альтернативное сплайсинг (AS) и альтернативное полиаденилирование (APA) расширяют разнообразие изоформ транскриптов и их продуктов. Здесь мы описываем биоинформационные протоколы для анализа объемных анализов РНК-seq и 3'-концевого секвенирования для обнаружения и визуализации AS и APA, варьирующихся в зависимости от экспериментальных условий.
Аннотация
Наряду с типичным анализом RNA-Seq для измерения дифференциальной экспрессии генов (DGE) в экспериментальных / биологических условиях, данные RNA-seq также могут быть использованы для изучения других сложных регуляторных механизмов на уровне экзонов. Альтернативное сплайсинг и полиаденилирование играют решающую роль в функциональном разнообразии гена, генерируя различные изоформы для регулирования экспрессии генов на посттранскрипционном уровне, и ограничение анализа всем генным уровнем может пропустить этот важный регуляторный слой. Здесь мы демонстрируем подробный пошаговый анализ для идентификации и визуализации использования дифференциального экзона и полиаденилирования в разных условиях с использованием биопроводника и других пакетов и функций, включая DEXSeq, diffSplice из пакета Limma и rMATS.
Введение
RNA-seq широко использовался на протяжении многих лет, как правило, для оценки дифференциальной экспрессии генов и открытия генов1. Кроме того, он также может быть использован для оценки различного использования на уровне экзонов из-за генов, экспрессирующих различные изоформы, что способствует лучшему пониманию регуляции генов на посттранскрипционном уровне. Большинство эукариотических генов генерируют различные изоформы путем альтернативного сплайсинга (AS) для увеличения разнообразия экспрессии мРНК. События AS можно разделить на различные паттерны: пропуск полных экзонов (SE), где («кассетный») экзон полностью удаляется из стенограммы вместе с его фланкирующими интронами; альтернативный (донорский) 5-футовый выбор места сращивания (A5SS) и альтернативный 3-дюймовый (акцепторный) выбор места сращивания (A3SS), когда два или более участков сращивания присутствуют на обоих концах экзона; удержание интронов (RI), когда интрон сохраняется в зрелом транскрипте мРНК, и взаимное исключение использования экзона (MXE), где только один из двух доступных экзонов может быть сохранен за один раз 2,3. Альтернативное полиаденилирование (АПА) также играет важную роль в регулировании экспрессии генов с использованием альтернативных поли(А) сайтов для генерации нескольких изоформ мРНК из одного транскрипта4. Большинство участков полиаденилирования (pAs) расположены в 3' нетранслируемой области (3' UTR), генерируя изоформы мРНК с различными длинами UTR 3'. Поскольку 3' UTR является центральным узлом для распознавания регуляторных элементов, различные длины 3' UTR могут влиять на локализацию, стабильность и трансляцию мРНК5. Существует класс 3'-концевых анализов секвенирования, оптимизированных для обнаружения APA, которые отличаются деталями протокола6. Конвейер, описанный здесь, предназначен для PolyA-seq, но может быть адаптирован для других протоколов, как описано.
В данном исследовании мы представляем конвейер методов дифференциального анализа экзонов 7,8 (рисунок 1), которые можно разделить на две широкие категории: основанные на экзонах (DEXSeq9, diffSplice10) и событийные (реплицированный многомерный анализ сплайсинга транскриптов (rMATS)11). Методы, основанные на экзонах, сравнивают изменение складки в разных условиях отдельных экзонов с мерой общего изменения складки генов, чтобы вызвать дифференциально экспрессированное использование экзонов, и на основе этого вычисляют меру активности АС на уровне гена. Методы, основанные на событиях, используют считывание переходов, охватывающих экзон-интрон, для обнаружения и классификации конкретных событий сплайсинга, таких как пропуск экзона или удержание интронов, и различают эти типы AS в выходных данных3. Таким образом, эти методы обеспечивают взаимодополняющие взгляды для полного анализа AS 12,13. Мы выбрали DEXSeq (на основе пакета DESeq214 DGE) и diffSplice (на основе пакета Limma10 DGE) для исследования, поскольку они являются одними из наиболее широко используемых пакетов для дифференциального анализа сплайсинга. rMATS был выбран в качестве популярного метода для анализа на основе событий. Другим популярным методом, основанным на событиях, является MISO (Смесь изоформ)1. Для APA мы адаптируем подход, основанный на экзонах.
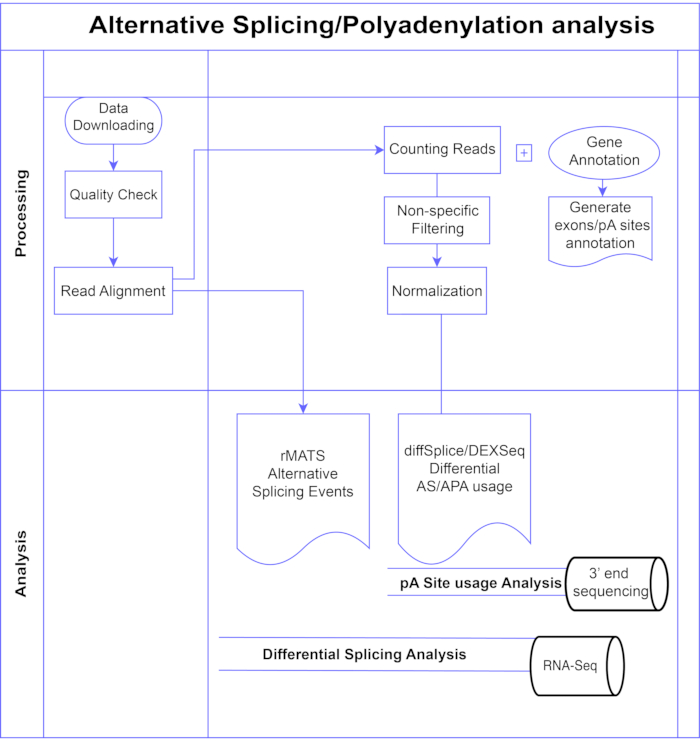
Рисунок 1. Конвейер анализа. Блок-схема шагов, используемых в анализе. Шаги включают в себя: получение данных, выполнение проверок качества и выравнивания считывания с последующим подсчетом считываний с использованием аннотаций для известных экзонов, интронов и участков pA, фильтрацию для удаления низких значений и нормализации. Данные PolyA-seq были проанализированы для альтернативных участков pA с использованием методов diffSplice/DEXSeq, объемная РНК-Seq была проанализирована для альтернативного сплайсинга на уровне экзона методами diffSplice/DEXseq, а события AS анализировались с помощью rMATS. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Данные RNA-seq, использованные в этом исследовании, были получены из Gene Expression Omnibus (GEO) (GSE138691)15. Мы использовали данные RNA-seq мышей из этого исследования с двумя группами состояний: дикий тип (WT) и Muscleblind-подобный нокаут типа 1 (Mbnl1 KO) с тремя репликами в каждой. Чтобы продемонстрировать дифференциальный анализ использования участка полиаденилирования, мы получили данные PolyA-seq о фибробластах эмбриона мыши (MEFs) (GEO Accession GSE60487)16. Данные имеют четыре группы условий: Wild-type (WT), Muscleblind-like type1/type 2 double knockout (Mbnl1/2 DKO), Mbnl 1/2 DKO с Mbnl3 knockdown (KD) и Mbnl1/2 DKO с управлением Mbnl3 (Ctrl). Каждая группа условий состоит из двух реплик.
| Присоединение к ГЭП | Номер запуска SRA | Пример имени | Состояние | Повторять | Ткань | Секвенирование | Длина чтения | |
| РНК-Сек | GSM4116218 | СРР10261601 | Mbnl1KO_Thymus_1 | Нокаут Mbnl1 | Представитель 1 | Тимус | Сопряженный конец | 100 бит/с |
| GSM4116219 | СРР10261602 | Mbnl1KO_Thymus_2 | Нокаут Mbnl1 | Представитель 2 | Тимус | Сопряженный конец | 100 бит/с | |
| GSM4116220 | СРР10261603 | Mbnl1KO_Thymus_3 | Нокаут Mbnl1 | Представитель 3 | Тимус | Сопряженный конец | 100 бит/с | |
| GSM4116221 | СРР10261604 | WT_Thymus_1 | Дикий тип | Представитель 1 | Тимус | Сопряженный конец | 100 бит/с | |
| GSM4116222 | СРР10261605 | WT_Thymus_2 | Дикий тип | Представитель 2 | Тимус | Сопряженный конец | 100 бит/с | |
| GSM4116223 | СРР10261606 | WT_Thymus_3 | Дикий тип | Представитель 3 | Тимус | Сопряженный конец | 100 бит/с | |
| 3П-Сек | GSM1480973 | СРР1553129 | WT_1 | Дикий тип (WT) | Представитель 1 | Мышиные эмбриональные фибробласты (MEF) | Однокомнатный | 40 бит/с |
| GSM1480974 | СРР1553130 | WT_2 | Дикий тип (WT) | Представитель 2 | Мышиные эмбриональные фибробласты (MEF) | Однокомнатный | 40 бит/с | |
| GSM1480975 | СРР1553131 | DKO_1 | Mbnl 1/2 двойной нокаут (DKO) | Представитель 1 | Мышиные эмбриональные фибробласты (MEF) | Однокомнатный | 40 бит/с | |
| GSM1480976 | СРР1553132 | DKO_2 | Mbnl 1/2 двойной нокаут (DKO) | Представитель 2 | Мышиные эмбриональные фибробласты (MEF) | Однокомнатный | 40 бит/с | |
| GSM1480977 | СРР1553133 | DKOsiRNA_1 | Mbnl 1/2 двойной нокаут с Mbnl 3 siRNA (KD) | Представитель 1 | Мышиные эмбриональные фибробласты (MEF) | Однокомнатный | 40 бит/с | |
| GSM1480978 | СРР1553134 | DKOsiRNA_2 | Mbnl 1/2 двойной нокаут с Mbnl 3 siRNA (KD) | Представитель 2 | Мышиные эмбриональные фибробласты (MEF) | Однокомнатный | 36 бит/с | |
| GSM1480979 | СРР1553135 | DKONTsiRNA_1 | Mbnl 1/2 двойной нокаут с нецелевым siRNA (Ctrl) | Представитель 1 | Мышиные эмбриональные фибробласты (MEF) | Однокомнатный | 40 бит/с | |
| GSM1480980 | СРР1553136 | DKONTsiRNA_2 | Mbnl 1/2 двойной нокаут с нецелевым siRNA (Ctrl) | Представитель 2 | Мышиные эмбриональные фибробласты (MEF) | Однокомнатный | 40 бит/с |
Таблица 1. Резюме наборов данных RNA-Seq и PolyA-seq, используемых для анализа.
протокол
1. Установка инструментов и R пакетов, используемых в анализе
- Conda - это популярный и гибкий менеджер пакетов, который позволяет удобно устанавливать пакеты с их зависимостями на всех платформах. Используйте 'Anaconda' (менеджер пакетов conda) для установки 'conda', который можно использовать для установки инструментов/пакетов, необходимых для анализа.
- Скачайте 'Anaconda' в соответствии с системными требованиями от https://www.anaconda.com/products/individual#Downloads и установите его, следуя подсказкам в графическом установщике. Установите все необходимые пакеты с помощью 'conda' , введя следующее в командной строке Linux.
conda install -c daler sratoolkit
conda install -c conda-forge parallel
conda install -c bioconda star bowtie fastqc rmats rmats2sashimiplot samtools fasterq-dump cutadapt bedtools deeptools - Чтобы загрузить все пакеты R, используемые в протоколе, введите следующий код в консоли R (запускается в командной строке Linux, набрав 'R') или консоли Rstudio.
bioc_packages<- c("DEXSeq", "Rsubread", "EnhancedVolcano", "edgeR", "limma", "maser","GenomicRanges")
packages<- c("magrittr", "rtracklayer", "tidyverse", "openxlsx", "BiocManager")
#Install if not already installed
installed_packages<-packages%in% rownames(installed.packages())
installed_bioc_packages<-bioc_packages%in% rownames(installed.packages())
if(any(installed_packages==FALSE)) {
install.packages(packages[!installed_packages],dependencies=TRUE)
BiocManager::install(packages[!installed_bioc_packages], dependencies=TRUE)
}
ПРИМЕЧАНИЕ: В этом вычислительном протоколе команды будут предоставляться в виде файлов R Notebook (файлы с расширением ". Rmd"), файлы кода R (файлы с расширением ". R»),или сценарии оболочки Linux Bash (файлы с расширением ".sh"). Файлы записной книжки R (Rmd) должны быть открыты в RStudio с помощью File| Откройте Файл..., и отдельные фрагменты кода (которые могут быть командами R или командами оболочки Bash), затем запустите в интерактивном режиме, щелкнув зеленую стрелку в правом верхнем углу. Файлы кода R могут быть запущены путем открытия в RStudio или в командной строке Linux путем предварительного преобразования с помощью "Rscript", например, Rscript example.R. Сценарии оболочки выполняются в командной строке Linux путем предварительного преобразования скрипта с помощью команды "sh", например.sh example.sh.
2. Анализ альтернативного сплайсинга (AS) с использованием RNA-seq
- Загрузка и предварительная обработка данных
ПРИМЕЧАНИЕ: Фрагменты кода, аннотированные ниже, доступны в дополнительном файле кода "AS_analysis_RNASeq.Rmd", чтобы следовать отдельным шагам в интерактивном режиме, а также предоставляются в виде bash-скрипта для пакетного запуска в командной строке Linux (ш downloading_data_preprocessing.sh).- Загрузка необработанных данных.
- Загрузите необработанные данные из архива чтения последовательностей (SRA) с помощью команды «предварительная выборка» из инструментария SRA (v2.10.8)17. Присвойте идентификаторам присоединения SRA последовательно в следующей команде, чтобы загрузить их параллельно с помощью утилиты GNU parallel18. Чтобы параллельно загрузить файлы SRA с идентификаторами присоединения из SRR10261601 в SRR10261606, используйте в командной строке Linux следующую команду.
seq 10261601 10261606 | parallel prefetch SRR{} - Извлеките файлы fastq из архива с помощью функции 'fastq-dump' из инструментария SRA. Используйте GNU параллельно и дайте имена всем файлам SRA вместе.
parallel -j 3 fastq-dump --gzip --skip-technical --read-filter pass --dumpbase --split-e --clip --origfmt {} ::: - Загрузите эталонный геном и аннотации для мыши (сборка генома GRCm39) из www.ensembl.org, используя следующее в командной строке Linux.
wget -nv -O annotation.gtf.gz http://ftp.ensembl.org/pub/release-103/gtf/mus_musculus/Mus_musculus.GRCm39.103.gtf.gz \ && gunzip -f annotation.gtf.gz
wget -nv -O genome.fa.gz http://ftp.ensembl.org/pub/release-103/fasta/mus_musculus/dna/Mus_musculus.GRCm39.dna.primary_assembly.fa.gz \ && gunzip -f genome.fa.gz
GTF=$(readlink -f annotation.gtf)
GENOME=$(readlink -f genome.fa)
- Загрузите необработанные данные из архива чтения последовательностей (SRA) с помощью команды «предварительная выборка» из инструментария SRA (v2.10.8)17. Присвойте идентификаторам присоединения SRA последовательно в следующей команде, чтобы загрузить их параллельно с помощью утилиты GNU parallel18. Чтобы параллельно загрузить файлы SRA с идентификаторами присоединения из SRR10261601 в SRR10261606, используйте в командной строке Linux следующую команду.
- Предварительная обработка и картирование чтения в сборку генома
- Контроль качества. Оцените качество необработанных считываемых материалов с помощью FASTQC (FASTQ Quality Check v0.11.9)19. Создайте выходную папку и запустите fastqc параллельно с несколькими входными файлами fasta. На этом шаге будет создан отчет о качестве для каждого образца. Изучите отчеты, чтобы убедиться, что качество чтения является приемлемым, прежде чем проводить дальнейший анализ. (См. руководство пользователя для понимания отчетов в https://www.bioinformatics.babraham.ac.uk/projects/fastqc/)
mkdir fastqc_out
parallel "fastqc {} -o fastqc_out" ::: $RAW_DATA/*.fastq.gz
ПРИМЕЧАНИЕ: При необходимости выполните обрезку адаптера 'cutadapt'20 или 'trimmomatic'21 для удаления секвенирования во фланговые адаптеры, которое варьируется в зависимости от размера фрагмента РНК и длины считывания. В этом анализе мы пропустили этот шаг, так как доля затронутых считываний была минимальной. - Чтение выравнивания. Следующий шаг в предварительной обработке включает в себя сопоставление прочитанных с эталонным геномом. Во-первых, постройте индекс для эталонного генома, используя функцию «genomeGenerate» STAR22 , а затем выровняйте необработанные чтения со ссылкой (альтернативно готовые индексы доступны на веб-сайте STAR и могут использоваться непосредственно для выравнивания). Выполните следующие команды в командной строке Linux.
#Build STAR index
GDIR=STAR_indices
mkdir $GDIR
STAR --runMode genomeGenerate --genomeFastaFiles $GENOME --sjdbGTFfile $GTF --runThreadN 8 --genomeDir $GDIR
ODIR=results/mapping
mkdir -p $ODIR
#Align reads to the genome
for fq1 in $RAW_DATA/*R1.fastq.gz;
do
fq2=$(echo $fq1| sed 's/1.fastq.gz/2.fastq.gz/g');
OUTPUT=$(basename ${fq1}| sed 's/R1.fastq.gz//g');
STAR --genomeDir $GDIR \
--runThreadN 12 \
--readFilesCommand zcat \
--readFilesIn ${fq1}${fq2}\
--outFileNamePrefix $ODIR\/${OUTPUT} \
--outSAMtype BAM SortedByCoordinate \
--outSAMunmapped Within \
--outSAMattributes Standard
Done
ПРИМЕЧАНИЕ: Выравниватель STAR будет генерировать и сортировать файлы BAM (Binary Alignment Map) для каждого образца после прочтения выравнивания. Файлы Bam должны быть отсортированы, прежде чем переходить к дальнейшим шагам.
- Контроль качества. Оцените качество необработанных считываемых материалов с помощью FASTQC (FASTQ Quality Check v0.11.9)19. Создайте выходную папку и запустите fastqc параллельно с несколькими входными файлами fasta. На этом шаге будет создан отчет о качестве для каждого образца. Изучите отчеты, чтобы убедиться, что качество чтения является приемлемым, прежде чем проводить дальнейший анализ. (См. руководство пользователя для понимания отчетов в https://www.bioinformatics.babraham.ac.uk/projects/fastqc/)
- Загрузка необработанных данных.
- Подготовка аннотаций Exon.
- Запустите файл дополнительного кода "prepare_mm_exon_annotation. R" с загруженной аннотацией в формате GTF (Gene transfer format) для подготовки аннотаций. Для запуска введите в командной строке Linux следующую команду.
Rscript prepare_mm_exon_annotation.R annotation.gtf
ПРИМЕЧАНИЕ: Файл GTF содержит несколько записей экзона для различных изоформ. Этот файл используется для «свертывания» нескольких идентификаторов транскриптов для каждого экзона. Это важный шаг для определения контейнеров для подсчета экзонов.
- Запустите файл дополнительного кода "prepare_mm_exon_annotation. R" с загруженной аннотацией в формате GTF (Gene transfer format) для подготовки аннотаций. Для запуска введите в командной строке Linux следующую команду.
- Подсчет прочитанных. Следующим шагом является подсчет количества чтений, сопоставленных с различными расшифровками / экзонами. См. дополнительный файл: "AS_analysis_RNASeq.Rmd".
- Загрузка необходимых библиотек:
packages<- c("Rsubread","tidyverse", "magrittr", "EnhancedVolcano", "edgeR","openxlsx")
invisible(lapply(packages, library, character.only=TRUE)) - Загрузите обработанный файл аннотаций, полученный на предыдущем шаге (2.2).
load("mm_exon_anno.RData") - Прочитайте все файлы bam, полученные на шаге 2.2.2, в качестве входных данных для 'featureCounts' для подсчета прочитанных данных. Прочитайте папку, содержащую файлы bam, сначала перечислив каждый файл из каталога, который заканчивается на .bam. Используйте 'featureCounts' из пакета Rsubread, который принимает файлы bam и обработанную аннотацию GTF (ссылку) в качестве входных данных для создания матрицы счетчиков, связанных с каждым объектом, со строками, представляющими экзоны (объекты), и столбцами, представляющими образцы.
countData <- dir("bams", pattern=".bam$", full.names=T) %>%
featureCounts(annot.ext=anno,
isGTFAnnotationFile=FALSE,
minMQS=0,useMetaFeatures=FALSE,
allowMultiOverlap=TRUE,
largestOverlap=TRUE,
countMultiMappingReads=FALSE,
primaryOnly=TRUE,
isPairedEnd=TRUE,
nthreads=12) - Затем выполните неспецифическую фильтрацию для удаления низко выраженных экзонов («неспецифическая» указывает на то, что информация об экспериментальном состоянии не используется при фильтрации, чтобы избежать смещений выбора). Преобразуйте данные из необработанного масштаба в счетчики на миллион (cpm) с помощью функции cpm из пакетаedgeR 23 и сохраняйте экзоны с количеством, превышающим заданное пороговое значение (для этого набора данных используется один cpm) по крайней мере в трех выборках. Также удаляют гены только с одним экзоном.
# Non-specific filtering: Remove the exons with low counts
isexpr<- rownames(countData$counts)[rowSums(cpm(countData$counts)>1) >=3]
countData$counts<-countData$counts[rownames(countData$counts) %in%isexpr, ]
anno<-anno%>% filter(GeneID%in% rownames(countData$counts))
# Remove genes with only 1 site and NA in geneIDs
dn<-anno%>%group_by(GeneID)%>%summarise(nsites=n())%>% filter(nsites>1&!is.na(GeneID))
anno<-anno%>% filter(GeneID%in%dn$GeneID)
countData$counts<-countData$counts[rownames(countData$counts) %in%anno$GeneID, ]
ПРИМЕЧАНИЕ: Проверьте необходимые параметры для featureCounts при использовании различных данных, например, для одностороннего чтения, установите 'isPairedEnd = FALSE'. Обратитесь к руководству пользователя RSubread, чтобы выбрать параметры для ваших данных, и см. раздел Обсуждение ниже.
- Загрузка необходимых библиотек:
- Дифференциальное сращивание и анализ использования экзонов. Мы описываем две альтернативы для этого шага: DEXSeq и DiffSplice. Любой из них может быть использован и давать аналогичные результаты. Для обеспечения согласованности выберите DEXSeq, если вы предпочитаете пакет DESeq2 для DGE, и используйте DiffSplice для анализа DGE на основе Limma. См. дополнительный файл: "AS_analysis_RNASeq.Rmd".
- Использование пакета DEXSeq для дифференциального анализа экзонов.
- Загрузите библиотеку и создайте образец таблицы для определения экспериментального проекта.
library(DEXSeq)
sampleTable<-data.frame(row.names= c("Mbnl1KO_Thymus_1", "Mbnl1KO_Thymus_2", "Mbnl1KO_Thymus_3", "WT_Thymus_1", "WT_Thymus_2", "WT_Thymus_3"), condition= rep(c("Mbnl1_KO", "WT"),c(3,3)), libType= rep(c("paired-end")))
ПРИМЕЧАНИЕ: Имена строк должны соответствовать именам файлов bam, используемым featureCounts для подсчета прочитанных данных. sampleTable состоит из подробных сведений о каждом образце, который включает в себя: тип библиотеки и условие. Это необходимо для определения контрастов или тестовой группы для обнаружения дифференциального использования. - Подготовьте информационный файл экзона. Информация об экзонах в виде объектов GRanges (геномных диапазонов) (https://bioconductor.org/packages/release/bioc/html/GenomicRanges.html) необходима в качестве входных данных для создания объекта DEXSeq на следующем шаге. Сопоставьте идентификаторы генов с счетчиками чтения, чтобы создать объект exoninfo.
exoninfo<-anno[anno$GeneID%in% rownames(countData$counts),]
exoninfo<-GRanges(seqnames=anno$Chr,
ranges=IRanges(start=anno$Start, end=anno$End, width=anno$Width),strand=Rle(anno$Strand))
mcols(exoninfo)$TranscriptIDs<-anno$TranscriptIDs
mcols(exoninfo)$Ticker<-anno$Ticker
mcols(exoninfo)$ExonID<-anno$ExonID
mcols(exoninfo)$n<-anno$n
mcols(exoninfo)$GeneID<-anno$GeneID
transcripts_l= strsplit(exoninfo$TranscriptIDs, "\\,")
save(countData, sampleTable, exoninfo, transcripts_l, file="AS_countdata.RData") - Создайте объект DEXSeq с помощью функции DEXSeqDataSet. Объект DEXSeq собирает вместе счетчики чтения, сведения об экзонных функциях и образцы. Используйте счетчики чтения, созданные на шаге 3, и информацию об экзоне, полученную на предыдущем шаге, чтобы создать объект DEXSeq из матрицы счетчика. Аргумент sampleData принимает входные данные фрейма данных, определяющие образцы (и их атрибуты: тип библиотеки и условие), 'design' использует sampleData для создания матрицы проектирования для дифференциального тестирования с использованием нотации формул модели. Обратите внимание, что значимый термин взаимодействия, condition:exon, указывает на то, что доля считываний над геном, падающим на конкретный экзон, зависит от экспериментального состояния, т.е. существует AS. Смотрите документацию DEXSeq для полного описания настройки формулы модели для более сложных экспериментальных проектов. Для получения информации о функциях требуются идентификаторы экзонов, соответствующие гены и транскрипты.
dxd<-DEXSeqDataSet(countData$counts,sampleData=sampleTable, design=~sample+exon+condition:exon,featureID=exoninfo$ExonID,groupID=exoninfo$GeneID,featureRanges=exoninfo, transcripts=transcripts_l) - Оценка нормализации и дисперсии. Затем выполните нормализацию между образцами и оцените дисперсию данных, за счет как шума пуассона от дискретной природы РНК-seq, так и биологической изменчивости, используя следующие команды.
dxd %<>% estimateSizeFactors %>% estimateDispersions %T>% plotDispEsts - Тест на дифференциальное использование. После оценки вариации проверьте использование дифференциального экзона для каждого гена и получите результаты.
dxd%<>%testForDEU%>%estimateExonFoldChanges(fitExpToVar=
"condition")#Estimate fold changes
dxr=DEXSeqResults(dxd) - Визуализируйте события сплайсинга для выбранных генов с помощью следующей команды.
plotDEXSeq(dxr,"Wnk1", displayTranscripts=TRUE, splicing=TRUE,legend
=TRUE,cex.axis=1.2,cex=1.3,lwd=2)
Изучите файл R Notebook «AS_analysis_RNASeq.Rmd», чтобы создать дополнительные графики для генов, представляющих интерес, и создать графики вулканов при разных пороговых значениях.
- Загрузите библиотеку и создайте образец таблицы для определения экспериментального проекта.
- Использование diffSplice от Limma для идентификации дифференциального сплайсинга. Следуйте за файлом R Notebook "AS_analysis_RNASeq.Rmd". Убедитесь, что шаги 2.1-2.3 были выполнены для подготовки входных файлов, прежде чем продолжить работу.
- Загрузка библиотек
library(limma)
library(edgeR) - Неспецифическая фильтрация. Извлеките матрицу счетчиков считывания, полученную в 2.3. Создайте список объектов с помощью функции 'DGEList' из пакета edgeR, где строки представляют гены, а столбцы представляют образцы.
mycounts=countData$counts
#Change the rownames of the countdata to exon Ids instead of genes for unique rownames.
rownames(mycounts) = exoninfo$ExonID
dge<-DGEList(counts=mycounts)
#Filtering
isexpr<- rowSums(cpm(dge)>1) >=3
dge<-dge[isexpr,,keep.lib.sizes=FALSE]
#Extract the exon annotations for only transcripts meeting non-specific filter
exoninfo=anno%>% filter(ExonID%in% rownames(dge$counts))
#Convert the exoninfo into GRanges object
exoninfo1<-GRanges(seqnames=exoninfo$Chr,
ranges=IRanges(start=exoninfo$Start, end=exoninfo$End, width=exoninfo$Width),strand=Rle(exoninfo$Strand))
mcols(exoninfo1)$TranscriptIDs<-exoninfo$TranscriptIDs
mcols(exoninfo1)$Ticker<-exoninfo$Ticker
mcols(exoninfo1)$ExonID<-exoninfo$ExonID
mcols(exoninfo1)$n<-exoninfo$n
mcols(exoninfo1)$GeneID<-exoninfo$GeneID
transcripts_l= strsplit(exoninfo1$TranscriptIDs, "\\,")
ПРИМЕЧАНИЕ: В качестве неспецифического этапа фильтрации счетчики фильтруются по cpm < 1 в x из n выборок, где x — минимальное количество реплиц в любом состоянии. n = 6 и x = 3 для данных этого примера. - Нормализуйте счетчики между выборками с помощью функции 'calcNormFactors' из пакета 'edgeR' с использованием усеченного среднего значения M (метод нормализации TMM)24 Он будет вычислять коэффициенты масштабирования для корректировки размеров библиотеки.
dge<-calcNormFactors(dge) - Используйте sampleTable в том виде, в котором он был создан на шаге 2.4.1.1, и создайте матрицу проектирования. Матрица дизайна характеризует дизайн. Смотрите руководство пользователя Limma (https://www.bioconductor.org/packages/devel/bioc/vignettes/limma/inst/doc/usersguide.pdf) главы 8 и 9 для получения подробной информации о матрицах проектирования для более продвинутых экспериментальных конструкций.
Treat<- factor(sampleTable$condition)
design<- model.matrix(~0+Treat)
colnames(design) <- levels(Treat) - Подгонка линейной модели для каждого экзона. Запустите функцию 'voom' пакета 'limma' для обработки данных RNA-seq для оценки дисперсии и генерации прецизионных весов для корректировки шума подсчета Пуассона, а также преобразования счетчиков на уровне экзонов в log2-counts per million (logCPM). Затем запустите линейное моделирование с помощью функции 'lmfit', чтобы подогнать линейные модели к данным выражения для каждого экзона. Вычислите эмпирическую статистику Байеса для встроенной модели, используя функцию 'eBayes' для обнаружения дифференциального выражения экзонов. Затем определите контрастную матрицу для экспериментальных сравнений, представляющих интерес. Используйте 'contrasts.fit' для получения коэффициентов и стандартных погрешностей для каждой пары сравнений.
v<-voom(dge,design,plot=FALSE)
fit<-lmFit(v,design)
fit<-eBayes(fit)
colnames(fit)
cont.matrix<-makeContrasts(
Mbnl1_KO_WT=Mbnl1_KO-WT,
levels=design)
fit2<-contrasts.fit(fit,cont.matrix) - Дифференциальный анализ сращивания. Запустите 'diffSplice' на подходящей модели, чтобы проверить различия в использовании экзонов генов между диким типом и нокаутом и изучить результаты с верхним рейтингом, используя функцию 'topSplice': test="t" дает рейтинг экзонов AS, test="simes" дает ранжирование генов.
ex<-diffSplice(fit2,geneid=exoninfo$GeneID,exonid=exoninfo$ExonID)
ts<-topSplice(ex,n=Inf,FDR=0.1, test="t", sort.by="logFC")
tg<-topSplice(ex,n=Inf,FDR=0.1, test="simes") - Визуализация. Построение результатов с функцией 'plotSplice', придавая гену интерес в генеоидном аргументе. Сохраните лучшие результаты, отсортированные по журналу Fold change в объект и сгенерируйте график вулкана для отображения экзонов.
plotSplice(ex,geneid="Wnk1", FDR=0.1)
#Volcano plot
EnhancedVolcano(ts,lab=ts$ExonID,selectLab= head((ts$ExonID),2000), xlab= bquote(~Log[2]~'fold change'), x='logFC', y='P.Value', title='Volcano Plot', subtitle='Mbnl1_KO vs WT (Limma_diffSplice)', FCcutoff=2, labSize=4,legendPosition="right", caption= bquote(~Log[2]~"Fold change cutoff, 2; FDR 10%"))
- Загрузка библиотек
- Использование rMATS
- Убедитесь, что последняя версия rMATS v4.1.1 (также известная как rMATS turbo из-за сокращения времени обработки и меньших требований к памяти) установлена либо с помощью conda, либо github (https://github.com/Xinglab/rmats-turbo/releases/download/v4.1.1/rmats_turbo_v4_1_1.tar.gz) в рабочем каталоге. Следуйте разделу 4.3 в разделе "AS_analysis_RNASeq.Rmd".
- Перейдите в папку, содержащую файлы bam, полученные после сопоставления, и подготовьте текстовые файлы, как того требует rMATS, для двух условий, скопировав имя bam-файлов (вместе с путем), разделенным ','. В командной строке Linux должны выполняться следующие команды:
mkdir rMATS_analysis
cd bams/
ls -pd "$PWD"/*| grep "WT"| tr '\n'','> Wt.txt
ls -pd "$PWD"/*| grep "Mb"| tr '\n'','> KO.txt
mv *.txt ../rMATS_analysis - Запустите rmats.py с двумя входными файлами, сгенерированными на предыдущем шаге, вместе с файлом GTF, полученным в 2.1.1.3. Это сгенерирует выходную папку 'rmats_out', содержащую текстовые файлы, описывающие статистику (p-значения и уровни включения) для каждого события сращивания отдельно.
python rmats-turbo/rmats.py --b1 KO.txt --b2 Wt.txt --gtf annotation.gtf -t paired --readLength 50 --nthread 8 --od rmats_out/ --tmp rmats_tmp --task pos
ПРИМЕЧАНИЕ: Справочная аннотация в виде файла GTF также обязательна. Проверьте параметры, если данные являются односторонними, и измените параметр -t соответствующим образом. - Изучение результатов rMATS. Используйте биопроводниковый пакет 'maser'25 для изучения результатов rMATS. Загрузите текстовые файлы Junction и exon counts (JCEC) в объект 'maser' и отфильтруйте результат на основе покрытия, включив не менее пяти средних считываний на событие сплайсинга.
library(maser)
mbnl1<-maser("/rmats_out/", c("WT","Mbnl1_KO"), ftype="JCEC")
#Filtering out events by coverage
mbnl1_filt<-filterByCoverage(mbnl1,avg_reads=5) - Визуализация результатов rMATS. Выберите значимые события сращивания с коэффициентом ложного обнаружения (FDR) 10% и минимальным изменением процента сращивания (deltaPSI) на уровне 10% с помощью функции 'topEvents' из пакета 'maser'. Затем проверьте события генов для отдельных генов, представляющих интерес (образец гена Wnk1) и постройте график значений PSI для каждого события сплайсинга этого гена. Создайте график вулкана, указав тип события.
#Top splicing events at 10% FDR
mbnl1_top<-topEvents(mbnl1_filt,fdr=0.1, deltaPSI=0.1)
mbnl1_top
#Check the gene events for a particular gene
mbnl1_wnk1<-geneEvents(mbnl1_filt,geneS="Wnk1", fdr=0.1, deltaPSI=0.1)
maser::display(mbnl1_wnk1,"SE")
plotGenePSI(mbnl1_wnk1,type="SE", show_replicates
=TRUE)
volcano(mbnl1_filt,fdr=0.1, deltaPSI=0.1,type="SE")
+xlab("deltaPSI")+ylab("Log10 Adj. Pvalue")+ggtitle("Volcano Plot of exon skipping events") - Генерация графиков Сашими для результата событий сплайсинга, полученного с помощью rMATS в виде текстовых файлов с помощью пакета 'rmats2shahimiplot'. Запустите скрипт python в командной строке Linux.
python ./src/rmats2sashimiplot/rmats2sashimiplot.py --b1 ../bams/WT_Thymus_1.bam,../bams/WT_Thymus_2.bam,../bams/WT_Thymus_3.bam --b2 ../bams/Mbnl1KO_Thymus_1.bam,../bams/Mbnl1KO_Thymus_2.bam,../bams/Mbnl1KO_Thymus_3.bam -t SE -e ../rMATS_analysis/rmats_out/SE.MATS.JC.txt --l1 WT --l2 Mbnl1_KO --exon_s 1 --intron_s 5 -o ../rMATS_analysis/rmats2shasmi_output
ПРИМЕЧАНИЕ: Этот процесс может занять много времени, так как он сгенерирует график Сашими для всех результатов в файле событий. Выберите лучшие результаты (имена генов и экзоны), отображаемые функцией topEvents из «мазера», и визуализируйте соответствующий график Сашими.
- Использование пакета DEXSeq для дифференциального анализа экзонов.
3. Анализ альтернативного полиаденилирования (APA) с использованием 3' конечного секвенирования
- Загрузка и предварительная обработка данных
ПРИМЕЧАНИЕ: Обратитесь к дополнительному файлу R Notebook "APA_analysis_3PSeq_notebook. Rmd" для выполнения полных команд для загрузки и предварительной обработки данных или запустите дополнительный bash-файл "APA_data_downloading_preprocessing.sh" в командной строке Linux.- Загрузите данные из SRA с идентификаторами присоединения (1553129 по 1553136).
- Обрезка адаптеров и обратное дополнение для получения последовательности чувственных нитей.
ПРИМЕЧАНИЕ: Этот шаг относится к используемому анализу PolyA-seq. - Карта считывает сборку генома мыши с помощью лук-бабочки26.
- Подготовка аннотаций к сайтам pA.
ПРИМЕЧАНИЕ: Обработка файла аннотации сайта pA выполняется сначала с использованием дополнительного файла R Notebook "APA_analysis_3PSeq_notebook. Rmd" (2.1 - 2.6), а затем с помощью bash-файла "APA_annotation_preparation.sh".- Загрузите аннотацию сайтов pA из базы данных PolyASite 2.06.
- Выберите аннотации pA-сайтов, чтобы сохранить 3'-непереведенные области (UTR) pA-сайты, которые аннотируются как терминальный экзон (TE) или 1000 нт после аннотированного терминального экзона (DS) для последующего анализа.
- Получение пиков pA сайта. Встаньте на якорь на каждом участке расщепления pA и визуализируйте среднее покрытие считывания, используя надкроватные и глубокие инструменты27,28. Результаты показали, что пики нанесенных на карту показаний были в основном рассредоточены в пределах ~60 бп вверх по течению от участков расщепления (рисунок 5 и дополнительный рисунок 5). Поэтому координаты участков pA были расширены от файла аннотации до 60 bp вверх по течению от их участков расщепления. В зависимости от конкретного используемого протокола секвенирования 3', этот шаг необходимо будет оптимизировать для анализов, отличных от PolyA-seq.
- Подсчет чтений
- Подготовьте файл аннотации сайтов pA.
anno<- read.table(file= "flanking60added.pA_annotation.bed",
stringsAsFactors=FALSE, check.names=FALSE, header=FALSE, sep="")
colnames(anno) <- c("chrom", "chromStart", "chromEnd", "name", "score", "strand", "rep", "annotation", "gene_name", "gene_id")
anno<- dplyr::select(anno,name,chrom, chromStart,chromEnd, strand,gene_id,gene_name,rep)
colnames(anno) <- c("GeneID", "Chr", "Start", "End", "Strand", "Ensembl", "Symbol", "repID") - Примените 'featureCounts' для получения необработанных счетчиков. Сохраните таблицу счетчиков в виде файла RData "APA_countData.Rdata" для анализа APA с использованием различных инструментов.
countData<- dir("bamfiles", pattern="sorted.bam$", full.names=TRUE) %>%
# Read all bam files as input for featureCounts
featureCounts(annot.ext=anno, isGTFAnnotationFile= FALSE,minMQS=0,useMetaFeatures= TRUE,allowMultiOverlap=TRUE, largestOverlap= TRUE,strandSpecific=1, countMultiMappingReads =TRUE,primaryOnly= TRUE,isPairedEnd= FALSE,nthreads=12)%T>%
save(file="APA_countData.Rdata")
ПРИМЕЧАНИЕ: Не забывайте изменять любой из параметров, перечисленных в функции 'featureCounts'. Модифицируйте параметр «strandSpecific», чтобы убедиться, что он согласуется с направлением секвенирования используемого анализа секвенирования 3', (эмпирически визуализация данных в браузере генома над генами на плюсовых и минусовых нитях прояснит это). - Применение неспецифической фильтрации countData. Фильтрация может значительно повысить статистическую надежность в дифференциальных тестах pA использования сайта. Во-первых, мы удалили те гены, у которых есть только один сайт pA, на котором не может быть определено дифференциальное использование сайта pA. Во-вторых, мы применяем неспецифическую фильтрацию на основе покрытия: количество фильтруется по cpm менее 1 в x из n выборок, где x — минимальное количество реплик в любом состоянии. N = 8 и x = 2 для данных этого примера.
load(file= "APA_countData.Rdata")# Skip this step if already loaded
# Non-specific filtering: Remove the pA sites not differentially expressed in the samples
countData<-countData$counts%>%as.data.frame%>% .[rowSums(edgeR::cpm(.)>1) >=2, ]
anno%<>% .[.$GeneID%in% rownames(countData), ]
# Remove genes with only 1 site and NA in geneIDs
dnsites<-anno%>%group_by(Symbol)%>%summarise(nsites=n())%>% filter(nsites>1&!is.na(Symbol))
anno<-anno%>% filter(Symbol%in%dnsites$Symbol)
countData<-countData[rownames(countData) %in%anno$GeneID, ]
- Подготовьте файл аннотации сайтов pA.
- Анализ использования сайта дифференциального полиаденилирования с использованием конвейеров DEXSeq и diffSplice.
- Использование пакета DEXSeq
ПРИМЕЧАНИЕ: Поскольку контрастная матрица не может быть определена для конвейера DEXSeq, дифференциальный APA-анализ каждого из двух экспериментальных условий должен выполняться отдельно. Дифференциальный APA-анализ состояния WT и условия DKO выполняется в качестве примера для объяснения процедуры. Обратитесь к дополнительному файлу "APA_analysis_3PSeq_notebook. Рмд" для пошагового рабочего процесса этого раздела и дифференциального APA-анализа других контрастов.- Загрузите библиотеку и создайте образец таблицы для определения экспериментального проекта.
c("DEXSeq", "GenomicRanges") %>% lapply(library, character.only=TRUE) %>%invisible
sampleTable1<- data.frame(row.names= c("WT_1","WT_2","DKO_1","DKO_2"),
condition= c(rep("WT", 2), rep("DKO", 2)),
libType= rep("single-end", 4)) - Подготовьте информационный файл участков pA с помощью пакета биопроводников GRanges.
# Prepare the GRanges object for DEXSeqDataSet object construction
PASinfo <- GRanges(seqnames = anno$Chr,
ranges = IRanges(start = anno$Start, end = anno$End),strand = Rle(anno$Strand))
mcols(PASinfo)$PASID<-anno$repID
mcols(PASinfo)$GeneEns<-anno$Ensembl
mcols(PASinfo)$GeneID<-anno$Symbol
# Prepare the new feature IDs, replace the strand information with letters to match the current pA site clusterID
new.featureID <- anno$Strand %>% as.character %>% replace(. %in% "+", "F") %>% replace(. %in% "-", "R") %>% paste0(as.character(anno$repID), .) - Для создания объекта DEXSeq используйте счетчики чтения, созданные на шаге 3.3, и сведения о сайте pA, полученные на предыдущем шаге.
# Select the read counts of the condition WT and DKO
countData1<- dplyr::select(countData, SRR1553129.sorted.bam, SRR1553130.sorted.bam, SRR1553131.sorted.bam, SRR1553132.sorted.bam)
# Rename the columns of countData using sample names in sampleTable
colnames(countData1) <- rownames(sampleTable1)
dxd1<-DEXSeqDataSet(countData=countData1,
sampleData=sampleTable1,
design=~sample+exon+condition:exon,
featureID=new.featureID,
groupID=anno$Symbol,
featureRanges=PASinfo) - Определите контрастную пару путем определения уровней условий в объекте DEXSeq.
dxd1$condition<- factor(dxd1$condition, levels= c("WT", "DKO"))
# The contrast pair will be "DKO - WT" - Оценка нормализации и дисперсии. Подобно данным RNA-seq, для 3' конечных данных секвенирования выполняют нормализацию между выборками (столбичная медиана соотношений для каждой выборки) с помощью функции 'estimateSizeFactors', а также оценивают вариацию данных с помощью функции 'estimateDispersions', а затем визуализируют результат оценки дисперсии с помощью функции 'plotDispEsts'.
dxd1 %<>% estimateSizeFactors %>% estimateDispersions %T>% plotDispEsts - Дифференциальное тестирование использования сайта pA для каждого гена с использованием функции 'testForDEU', затем оцените кратное изменение использования pA сайта с помощью функции 'estimateExonFoldChanges'. Проверьте результаты с помощью функции 'DEXSeqResults' и установите 'FDR < 10%' в качестве критерия для значительно дифференциальных pA сайтов.
dxd1 %<>% testForDEU %>% estimateExonFoldChanges(fitExpToVar = "condition")
dxr1 <- DEXSeqResults(dxd1)
dxr1
mcols(dxr1)$description
table(dxr1$padj<0.1) # Check the number of differential pA sites (FDR < 0.1)
table(tapply(dxr1$padj<0.1, dxr1$groupID, any)) # Check the number of gene overlapped with differential pA site - Визуализация результатов использования дифференциального pA сайта с использованием дифференциальных APA-графиков, генерируемых функцией 'plotDEXSeq' и графика вулкана функцией 'EnhancedVolcano'.
# Select the top 100 significant differential pA sites ranked by FDR
topdiff.PAS<- dxr1%>%as.data.frame%>%rownames_to_column%>%arrange(padj)%$%groupID[1:100]
# Apply plotDEXSeq for the visualization of differential polyA usage
plotDEXSeq(dxr1,"S100a7a", legend=TRUE, expression=FALSE,splicing=TRUE, cex.axis=1.2, cex=1.3,lwd=2)
# Apply perGeneQValue to check the top genes with differential polyA site usage
dxr1%<>% .[!is.na(.$padj), ]
dgene<- data.frame(perGeneQValue= perGeneQValue (dxr1)) %>%rownames_to_column("groupID")
dePAS_sig1<-dxr1%>% data.frame() %>%
dplyr::select(-matches("dispersion|stat|countData|genomicData"))%>%
inner_join(dgene)%>%arrange(perGeneQValue)%>%distinct()%>%
filter(padj<0.1)
# Apply EnhancedVolcano package to visualise differential polyA site usage
"EnhancedVolcano"%>% lapply(library, character.only=TRUE) %>%invisible
EnhancedVolcano(dePAS_sig1, lab=dePAS_sig1$groupID, x='log2fold_DKO_WT',
y='pvalue',title='Volcano Plot',subtitle='DKO vs WT',
FCcutoff=1,labSize=4, legendPosition="right",
caption= bquote(~Log[2]~"Fold change cutoff, 1; FDR 10%"))
- Загрузите библиотеку и создайте образец таблицы для определения экспериментального проекта.
- Использование пакета diffSplice. Обратитесь к дополнительному файлу R Notebook "APA_analysis_3PSeq_notebook. Rmd" для пошагового рабочего процесса этого раздела.
- Определите контрасты, представляющие интерес для дифференциального анализа использования pA.
ПРИМЕЧАНИЕ: Этот шаг должен быть выполнен после построения и обработки объекта DGEList, который включен в файл R Notebook "APA_analysis_3PSeq_notebook. Rmd".contrast.matrix<-makeContrasts(DKO_vs_WT=DKO-
WT,Ctrl_vs_DKO=Ctrl-DKO,
KD_vs_Ctrl=KD-Ctrl,KD_vs_DKO=KD-DKO,levels=design)
fit2<-fit%>%contrasts.fit(contrast.matrix)%>%eBayes
summary(decideTests(fit2))
ex<-diffSplice(fit2,geneid=anno$Symbol,exonid=new.featureID)
topSplice(ex) #Check the top significant results with topSplice - Визуализируйте результат интересующих контрастов (здесь "DKO - WT") с помощью дифференциальных графиков APA по функции 'plotSplice' и графиков вулканов с функцией 'EnhancedVolcano'. Обратитесь к файлу R Notebook "APA_analysis_3PSeq_notebook. Rmd" 4.2.7 - 4.2.9 для визуализации других контрастных пар.
sig1<-topSplice(ex,n=Inf,FDR=0.1,coef=1, test="t", sort.by="logFC")
sig1.genes<-topSplice(ex,n=Inf,FDR=0.1,coef=1, test="simes")
plotSplice(ex, coef=1,geneid="S100a7a", FDR = 0.1)
plotSplice(ex,coef=1,geneid="Tpm1", FDR = 0.1)
plotSplice(ex,coef=1,geneid="Smc6", FDR = 0.1)
EnhancedVolcano(sig1, lab=sig1$GeneID,xlab= bquote(~Log[2]~'fold change'),
x='logFC', y='P.Value', title='Volcano Plot', subtitle='DKO vs WT',
FCcutoff=1, labSize=6, legendPosition="right")
- Определите контрасты, представляющие интерес для дифференциального анализа использования pA.
- Использование пакета DEXSeq
Результаты
После выполнения приведенного выше пошагового рабочего процесса выходные данные анализа AS и APA и репрезентативные результаты представляются в виде таблиц и графиков данных, генерируемых следующим образом.
КАК:
Основные результаты анализа АС (Дополнительная таблица 1 для diffSplice; Таблица 2 для DEXSeq) представляет собой список экзонов, показывающих дифференциальное использование в разных условиях, и список генов, показывающих значительную общую сплайсинговую активность одного или нескольких составляющих его экзонов, ранжированных по статистической значимости. В дополнительной таблице 1, вкладка 2 показаны значимые экзоны, причем столбцы показывают дифференциальный FC экзона по сравнению с остальным, нескорректированное p-значение для каждого экзона и скорректированное p-значение (поправка Бенджамини-Хокберга). Пороговое значение для скорректированных p-значений даст набор экзонов с определенным FDR. Дополнительная таблица 1, вкладка 3 показывает ранжированный список генов, показывающих значимость общей сплайсинговой активности, со столбцом, показывающим скорректированное на уровне генов p-значение, рассчитанное с использованием метода Саймса. Аналогичные данные приведены в таблице 2 для DEXSeq. Дополнительный рисунок 1 и дополнительный рисунок 2 показывают дифференциальную схему сплайсинга в генах Mbnl1, Tcf7 и Lef1, которые были экспериментально подтверждены в опубликованной статье, представленной с данными15. Авторы показали экспериментальную валидацию пяти генов - Mbnl1, Mbnl2, Lef1, Tcf7 и Ncor2. Наш подход обнаружил дифференциальные сплайсинговые паттены во всех этих генах. Здесь мы представляем уровни FDR для каждого гена с использованием DEXSeq, diffSplice и rMATS соответственно, как получено в дополнительных таблицах 1-3: Mbnl1 (0, 6.6E-61 ,0), Mbnl2 (0,0.18,0), Lef1 (1.4E-10, 1.3E-04, 0), Tcf7 (0, 1.1E-6, 0) и Ncor2 (9.2E-11, 1.6E-06, 0).
На рисунке 2 показано сравнение выходных данных, полученных из трех различных инструментов, и показано альтернативное сращивание паттернов в гене Wnk1 . Графики вулканов показаны на рисунке 2A (diffSplice) и рисунке 2B (DEXSeq). Дополнительные три гена с высоким рейтингом показаны на дополнительном рисунке 1 (diffSplice) и дополнительном рисунке 2 (DEXSeq). Ось Y показывает статистическую значимость (-log10 P-values), а ось X показывает размер эффекта (изменение сгиба). Гены, расположенные в верхнем левом или правом квадрантах, указывают на существенный FC и убедительные статистические доказательства подлинных различий.

Рисунок 2. Сравнение результатов альтернативного сплайсинга, полученных с помощью diffSplice, DEXSeq и rMATS. |
(A) Вулканический график (слева) РНК-Seq из анализа Limma diffSplice: ось X показывает изменение логарифмической складки экзона; ось Y показывает значение -log10 p. Каждая точка соответствует экзону. Горизонтальная пунктирная линия при p-значении=1E-5; вертикальные пунктирные линии при двукратном изменении (FC). Красные экзоны показывают существенную FC и статистическую значимость. График дифференциального сплайсинга (справа): Паттерны сплайсинга выставлены для примера гена Wnk1, где ось X показывает идентификатор экзона на транскрипт; ось Y показывает относительное изменение сгиба логарифма экзона (разница между logFC экзона и общим logFC для всех остальных экзонов). Экзоны, выделенные красным цветом, показывают статистически значимую дифференциальную экспрессию (FDR < 0,1).
(B) График вулкана (слева) и дифференциальное использование экзонов (справа) РНК-Seq, полученных из анализа DEXSeq. Ген Wnk1 показывает значительное дифференциальное использование экзонов между нокаутом WT и Mbnl1, выделенных розовым цветом, которые соответствуют тем же дифференциальным экзонам в (A).
(C) График вулкана (слева) и участок Сашими (справа) для Wnk1 , полученный из анализа rMATS. График вулкана, изображающий значительное пропущенное (кассетное) событие экзона (SE) в диком типе по сравнению с нокаутом при 10% FDR с изменением процента сращенных значений (PSI или ΔΨ) > 0,1. Ось x показывает изменение значений PSI в зависимости от условий, а ось Y показывает значение P-значения журнала. График Сашими показывает пропущенное событие экзона в гене Wnk1 , соответствующее значительному дифференциальному экзону в (A) и (B). Каждая строка представляет собой образец RNA-Seq: три реплики дикого типа и нокаут Mbnl1. Высота показывает покрытие считывания в RPKM, а соединительные дуги изображают считывание соединения между экзонами. Аннотированные альтернативные изоформы генной модели показаны в нижней части графика. Нижняя панель C иллюстрирует считывание соединения, используемое для вычисления статистики PSI.
(D) Диаграмма Венна , сравнивающая количество значимых дифференциальных экзонов, полученных различными методами. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Рисунок 2 A (правая панель) показывает диаграммное отображение различий экзонов одного из генов с самым высоким рейтингом, показывая logFC на оси Y и число экзонов на оси x. В этом примере показан кассетный экзон, варьирующийся между условиями для гена Wnk1. График использования дифференциального экзона из DEXSeq показывает доказательства дифференциального сращивания на пяти участках экзонов вблизи Wnk1.6.45. Выделенные экзоны розового цвета, вероятно, будут сращены в образцах Mbnl1 KO по сравнению с WT. Эти экзоны дополняют результаты, полученные с помощью diffSplice, который показывает аналогичную картину в конкретном геномном положении. Другие примеры приведены на дополнительном рисунке 1 и дополнительном рисунке 2. Более подробное представление для подтверждения интересных результатов может быть дано путем сравнения треков покрытия (колебаний) в единицах RPM (чтение на миллион) в браузерах генома UCSC (Университет Санта-Крус) или IGV (Integrative Genomics Viewer) (не показано), наряду с визуальной корреляцией с другими интересными треками, такими как известные модели генов, сохранение и другие анализы генома.
В выходной таблице rMATS перечислены значимые альтернативные события сплайсинга, классифицированные по типу (Дополнительная таблица 3). На рисунке 2C показан вулканический график генов, которые альтернативно сращены, с размером эффекта, измеренным дифференциальной статистикой «процент сращивания» (PSI или ΔΨ)11.
PSI относится к проценту считываний, согласующихся с включением кассетного экзона (т.е. считывает отображение на сам экзон кассеты или соединение считывает, перекрывая экзон) по сравнению со считыванием, совместимым с исключением экзона, т.е. соединение считывает через соседние экзоны вверх и вниз по течению (нижняя панель рисунка 2C). Правая панель рисунка 2C показывает график сашими гена Wnk1 с дифференциальным событием сплайсинга, наложенным на дорожки покрытия для гена, с пропущенным экзоном в Mbnl1 KO. Дуги, соединяющие экзоны, показывают количество считываний соединения (считывает пересечение сращенного интрона). Различные вкладки Дополнительной таблицы 3 показывают значительное считывание каждого типа события, которое охватывает соединения с границами экзонов (счетчики соединений и счетчики экзонов (JCEC)). На рисунке 2D сравниваются значительные дифференциально сращенные экзоны, обнаруженные тремя инструментами.

Рисунок 3. Альтернативные события сплайсинга, полученные с помощью анализа rMATS. A) Типы событий AS. Этот рисунок адаптирован из документации11 rMATS, объясняющей событие сращивания с конститутивными и альтернативно сращенными экзонами. Помечено номером каждого события на FDR 10%. Б) Участок сашими гена Add3, демонстрирующий пропущенный экзон (SE). C) Участок сашими гена Baz2b, демонстрирующий альтернативный 5'-образный участок сращивания (A5SS). D) Участок сашими гена Lsm14b, демонстрирующий альтернативный сайт сращивания 3' (A3SS). E) Участок сашими гена Mta1, демонстрирующий взаимоисключающие экзоны (MXE). F) Участок сашими гена Arpp21 с сохранением интрона (RI). Красные строки представляют собой три реплики дикого типа, а оранжевые строки представляют нокаут-реплики Mbnl1. Ось x соответствует геномным координатам и информации о нити, ось Y показывает покрытие в RPKM. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
На рисунке 3 показаны типы событий сплайсинга SE, A5SS, A3SS, MXE и RI с помощью графиков Сашими главных значимых генов этих событий. При сравнении трех реплик WT и Mbnl1_KO в FDR 10 было обнаружено в общей сложности 1272 события SE, 130 A5SS, 116 A3SS, 215 MXE и 313 RI событий RI. Сюжет сашими иллюстрирует тип события, используя в качестве примера топовые гены.
АПА:
Результат анализа APA аналогичен анализу AS на уровне экзонов. Приведена таблица высших генов, ранжированных по дифференциальной активности АПА в 3'UTR (Дополнительная таблица 4 и Дополнительная таблица 5). На рисунке 4A показаны вулканические графики генов по дифференциальной активности APA в 3'UTR, генерируемых с использованием diffSplice и DEXSeq отдельно. На рисунке 4B показан график Венна, сравнивающий значительно дифференциальные результаты использования pA сайта, полученные от разных конвейеров. На рисунках 4C и 4D показано диаграммное представление дифференциального использования сайта pA в 3'UTR гена Fosl2 (рисунок 4C) и Papola (рисунок 4D), сгенерированное с использованием как diffSplice, так и DEXSeq, которые экспериментально проверены для демонстрации значительного дистального проксимального сдвига (Fosl2) и значительного проксимального сдвига (Papola) использования pA сайта в DKO, соответственно. Другие примеры приведены на дополнительном рисунке 3 и дополнительном рисунке 4.

Рисунок 4. Альтернативные графики полиаденилирования с помощью diffSplice и DEXSeq. A) Вулканические графики данных PolyA-seq, сгенерированные с использованием diffSplice и DEXSeq. Ось X показывает изменение сгиба сайта log pA; Ось y показывает -log10 p-значение. Каждая точка соответствует участку pA. Горизонтальная пунктирная линия при p-значении=1E-5; вертикальные пунктирные линии на 2-х кратном FC. Красные экзоны показывают существенную FC и статистическую значимость. B) График Венна , сравнивающий результаты значительного дифференциального использования pA сайта, полученные от разных трубопроводов. С-Д) Дифференциальные графики APA, полученные с использованием diffSplice и DEXSeq, показывающие проксимальные, внутренние и дистальные участки pA для генов Fosl2 и Papola . Рисунки генерируются той же функцией, что и на рисунке 2 (B), но с сайтами pA, заменяющими экзоны. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Рисунок 5 представляет собой диагностический график для подтверждения ожидаемого распределения считывания вокруг аннотированных участков расщепления pA для используемого анализа PolyA-seq. Он показывает среднее покрытие во фланговых областях, закрепленных на известных участках расщепления рА на уровне всего генома. В этом случае визуализируется ожидаемое накопление считываний выше по течению сайтов. Распределения считывания, закрепленные на участках pA для всех образцов PolyA-seq, показаны на дополнительном рисунке 5.

Рисунок 5. Средний участок покрытия вокруг участков расщепления pA. Участок расщепления для данных PolyA-seq показан вертикальной пунктирной линией. Ось X показывает базовое положение относительно участков расщепления рА, до 100 нуклеотидов вверх и вниз по течению; Ось Y показывает среднее покрытие чтения по всем участкам расщепления pA, нормализованное по размеру библиотеки в CPM. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Дифференциальные результаты APA различных контрастов, генерируемых одним и тем же конвейером, могут быть сопоставлены и проверены путем визуализации считываемого покрытия репрезентативных значительно дифференциальных участков pA в браузере генома. На рисунке 6A показан график Венна, сравнивающий значительно дифференциальное использование pA сайтом различных контрастов, полученных от diffSplice. На рисунке 6B-D представлены снимки IGV считываемого покрытия на участках pA для различных генов, которые показывают закономерности, согласующиеся с теми, которые были обнаружены в анализе APA с использованием diffSplice. Рисунок 6B подтверждает значительное проксимальное и дистальное смещение сайта pA для гена Paip2, которое уникально обнаружено в контрасте DKO против WT, но не в двух других контрастах KD против WT и Ctr против WT. Рисунок 6C подтверждает значительный дистальный к проксимальному сдвигу использования сайта pA для гена Ccl2, обнаруженного уникально в контрасте KD против WT, в то время как рисунок 6D подтверждает значительное дифференциальное использование pA всех контрастов для гена Cacna2d1.
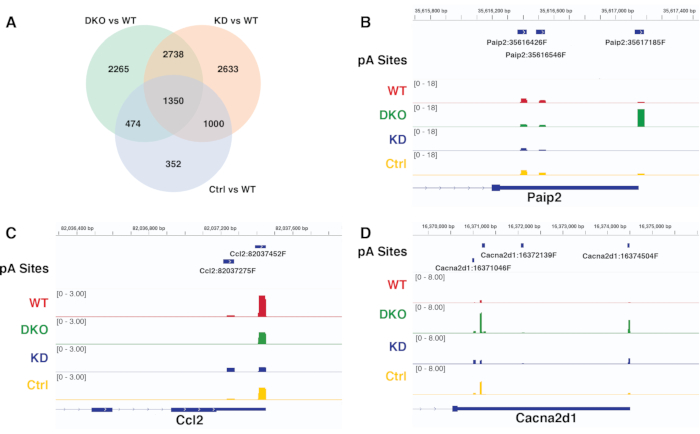
Рисунок 6. Сравнение контрастности и проверка результатов diffSplice. A) Диаграмма Венна, сравнивающая результаты использования сайта со значительным дифференциальным pA различных контрастов, полученных от diffSplice. Б-Д) Снимок IGV , визуализирующий пики pA, охватывают гены Paip2, Ccl2 и Cacna2d1 в разных условиях. Каждый трек представляет собой прочитанное покрытие в определенном состоянии. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Дополнительный рисунок 1. RNA-Seq анализ дифференциального сплайсинга с Limma diffSplice. (A) Вулканический график РНК-Seq из анализа Limma diffSplice: ось X показывает изменение логарифмической складки экзона; ось Y показывает значение -log10 p. Каждая точка соответствует экзону. Горизонтальная пунктирная линия при p-значении=1E-5; вертикальные пунктирные линии при двукратном изменении (FC). Красные экзоны показывают существенную FC и статистическую значимость. (B-D) Графики дифференциального сращивания: Паттерны сплайсинга демонстрируются, например, гены Mbnl1, Tcf7 и Lef1 соответственно, где ось x показывает идентификатор экзона на транскрипт; ось Y показывает относительное изменение сгиба логарифма экзона (разница между logFC экзона и общим logFC для всех остальных экзонов). Экзоны, выделенные красным цветом, показывают статистически значимую дифференциальную экспрессию (FDR < 0,1). Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный рисунок 2. RNA-Seq анализ использования дифференциального экзона с DEXSeq. (А) Вулканический участок. (B-D) Использование дифференциального экзона РНК-Seq, полученных из анализа DEXSeq. Гены Mbnl1, Tcf7 и Lef1, соответственно, показывают значительное дифференциальное использование экзонов между нокаутом WT и Mbnl1, выделенных розовым цветом, которые соответствуют тем же дифференциальным экзонам на дополнительном рисунке 1. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный рисунок 3. Альтернативные участки полиаденилирования по diffSplice. A) Вулканические графики данных PolyA-seq, сгенерированные с помощью diffSplice в трех контрастных парах, полученных из данных PolyA-seq мыши, включая двойной нокаут (DKO) против дикого типа (WT), нокдаун (KD) против WT и контроль (Ctrl) против WT. Ось X показывает изменение сгиба сайта pA журнала; Ось y показывает -log10 p-значение. Каждая точка соответствует участку pA. Горизонтальная пунктирная линия при p-значении=1E-5; вертикальные пунктирные линии на 2-х кратном FC. Красные участки pA показывают существенную FC и статистическую значимость. B) Дифференциальные графики APA, сгенерированные с использованием diffSplice, показывающие проксимальные, внутренние и дистальные участки pA для генов с высоким рейтингом S100a7a, Tpm1 и Smc6. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный рисунок 4. Дифференциальный анализ использования pA конвейером DEXSeq. A) Вулканические графики данных PolyA-seq, сгенерированных с помощью DEXSeq в трех парах, полученных из данных PolyA-seq мыши, включая двойной нокаут (DKO) против дикого типа (WT), нокдаун (KD) против WT и контроль (Ctrl) против WT. Ось X показывает изменение сгиба сайта pA журнала; Ось y показывает -log10 p-значение. Каждая точка соответствует участку pA. Горизонтальная пунктирная линия при p-значении=1E-5; вертикальные пунктирные линии на 2-х кратном FC. Красные участки pA показывают существенную FC и статистическую значимость. B) Дифференциальные графики APA, сгенерированные с использованием DEXSeq, показывающие проксимальные, внутренние и дистальные участки pA для генов с высоким рейтингом S100a7a, Tpm1 и Smc6. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный рисунок 5. График среднего покрытия и тепловые карты вокруг участков расщепления рА. Покрытие показано для четырех условий, с генами на прямой и обратной нитях, показанных отдельно. Ось X показывает базовое положение относительно участков расщепления рА, до 100 нуклеотидов вверх и вниз по течению; ось Y относится к среднему покрытию в соответствующих относительных базовых положениях по всем участкам расщепления рА. Тепловые карты предоставляют альтернативный вид, при этом каждый участок расщепления pA отображается в виде отдельной строки по оси X, упорядоченной по охвату. Интенсивность показывает прочитанное покрытие (см. легенду). Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительная таблица 1. diffСращение выходных данных анализа AS. Первая вкладка определяет имена столбцов для исходных выходных данных, представленных во второй (уровень экзона) и третьей (генный уровень) вкладках. Пожалуйста, нажмите здесь, чтобы загрузить эту таблицу.
Дополнительная таблица 2. Вывод DEXSeq анализа AS. Первая вкладка определяет имена столбцов для исходных выходных данных, представленных во второй (уровень экзона) и третьей (генный уровень) вкладках. Пожалуйста, нажмите здесь, чтобы загрузить эту таблицу.
Дополнительная таблица 3. rMATS результат анализа AS. Первая вкладка определяет имена столбцов для сводного файла (вкладка 2) и файлов JCEC для каждого события (вкладка 3-7). Пожалуйста, нажмите здесь, чтобы загрузить эту таблицу.
Дополнительная таблица 4. diffСращение выходных данных анализа APA. Первая вкладка определяет имена столбцов для исходных выходных данных, представленных на второй (уровень сайта pA) и третьей (уровень гена) вкладках. Пожалуйста, нажмите здесь, чтобы загрузить эту таблицу.
Дополнительная таблица 5. Вывод DEXSeq анализа APA. Первая вкладка определяет имена столбцов для исходных выходных данных, представленных на второй (уровень сайта pA) и третьей (уровень гена) вкладках. Пожалуйста, нажмите здесь, чтобы загрузить эту таблицу.
Дополнительная таблица 6. Сводка о количестве существенно измененных экзонов для сайтов AS и pA для APA. Пожалуйста, нажмите здесь, чтобы загрузить эту таблицу.
Дополнительная таблица 7. Краткое описание инструментов и пакетов, используемых в анализе AS/APA. Пожалуйста, нажмите здесь, чтобы загрузить эту таблицу.
Обсуждение
В этом исследовании мы оценили основанные на экзонах и событиях подходы к обнаружению AS и APA в объемных данных СЕКВЕНИРОВАНИЯ РНК-Seq и 3'. Подходы AS, основанные на экзонах, дают как список дифференциально экспрессированных экзонов, так и ранжирование на уровне генов, упорядоченное по статистической значимости общей дифференциальной сплайсинговой активности на уровне генов (таблицы 1-2, 4-5). Для пакета diffSplice дифференциальное использование определяется путем подгонки взвешенных линейных моделей на уровне экзона для оценки дифференциального изменения логарифмической складки экзона по сравнению со средним изменением логарифмической складки других экзонов в пределах того же гена (называемого per exon FC). Статистическая значимость на уровне генов вычисляется путем объединения отдельных тестов значимости на уровне экзонов в генный тест по методу Саймса10. Это ранжирование по дифференциальной сплайсинговой активности на уровне генов может быть впоследствии использовано для выполнения анализа обогащения набора генов (GSEA) ключевых путей, участвующихв 10. DEXSeq использует аналогичную стратегию, устанавливая обобщенную линейную модель для измерения использования дифференциального экзона, хотя и отличающуюся определенными этапами, такими как фильтрация, нормализация и оценка дисперсии. Сравнивая топ-500 ранжированных экзонов, показывающих активность AS и APA с использованием DEXSeq и DiffSplice, мы обнаружили перекрытие 310 экзонов и 300 pA сайтов соответственно, демонстрируя соответствие двух подходов на основе экзонов, что также было продемонстрировано в предыдущем исследовании 29. Для комплексного обнаружения и классификации AS рекомендуется использовать комбинацию как экзонного (DEXSeq или diffSplice), так и событийно-ориентированного подхода. Для APA пользователи могут выбрать либо DEXSeq, либо diffSplice: было показано, что оба метода хорошо работают в широком диапазоне экспериментов по транскриптомике29.
При подготовке библиотеки RNA-seq для анализа AS важно использовать цепной объемный протокол RNA-seq 8, поскольку большая часть генов в геномах позвоночных перекрывается на разных нитях, а протокол, не специфичный для цепи, не может различать эти перекрывающиеся области, что затрудняет окончательное обнаружение экзонов. Другим соображением является глубина считывания, при этом анализ сплайсинга требует более глубокого секвенирования, чем DGE, например, 30-60 миллионов считываний на выборку по сравнению с 5-25 миллионами считываний на выборку для DGE (https://sapac.support.illumina.com/bulletins/2017/04/considerations-for-rna-seq-read-length-and-coverage-.html). Все инструменты, демонстрируемые в протоколе, поддерживают как односторонние, так и парные данные секвенирования. Если для обнаружения считывания соединений используются только известные аннотации генов, то можно использовать однозначные более короткие считывания (≥ 50 bp), хотя de novo обнаружение новых соединений выигрывает от парных и более длинных (≥ 100bp) считывает30,31. Выбор протокола экстракции РНК - либо выбор полиА, либо истощение рРНК - зависит от качества РНК и экспериментального вопроса - см.31 для обсуждения. В зависимости от деталей построения библиотеки потребуются изменения в приведенных здесь примерах скриптов для параметров выравнивания чтения, подсчета признаков и rMATS. При вычислении начальных счетчиков считывания на уровне экзонов с помощью featureCounts или аналогичных методов необходимо позаботиться о правильной настройке параметров функции для счетчиков и цепкости: в featureCounts мы устанавливаем аргумент «strandSpecific» соответствующим образом для используемого протокола RNA-seq, специфичного для цепи; и для количественной оценки на уровне экзонов ожидается, что чтение будет сопоставляться с соседними экзонами, и поэтому мы устанавливаем для параметра allowMultiOverlap значение TRUE. Для APA существуют различные протоколы 3'-концевого секвенирования6, которые различаются по точному расположению пиков относительно участка pA. Для наших примерных данных мы определяем, что пик составляет 60 bp выше по течению от pA сайта, как показано на рисунке 5, и этот анализ необходимо будет адаптировать для других протоколов 3'-го конечного секвенирования.
В этом протоколе мы ограничиваем сферу охвата обсуждением дифференциального анализа на уровне отдельных экзонов и событий сплайсинга, состоящих из смежных комбинаций экзон-интрон. Мы не обсуждаем класс анализов, основанных на реконструкции изоформ de novo, таких как Cufflinks, Cuffdiff32, RSEM33, Kallisto34 , которые направлены на обнаружение и количественную оценку абсолютной и относительной экспрессии целых альтернативных изоформ. Методы, основанные на экзонах и событиях, более чувствительны для обнаружения отдельных событий сплайсинга30 и во многих случаях предоставляют всю информацию, необходимую для дальнейшего анализа, без необходимости количественной оценки на уровне изоформы.
Последняя версия исходных файлов в этом протоколе доступна по адресу https://github.com/jiayuwen/AS_APA_JoVE
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Это исследование было поддержано Австралийским исследовательским советом (ARC) Future Fellowship (FT16010043) и ANU Futures Scheme.
Материалы
| Name | Company | Catalog Number | Comments |
| Not relevent for computational study |
Ссылки
- Katz, Y., Wang, E. T., Airoldi, E. M., Burge, C. B. Analysis and design of RNA sequencing experiments for identifying isoform regulation. Nature Methods. 7 (12), 1009-1015 (2010).
- Wang, Y., et al. Mechanism of alternative splicing and its regulation. Biomedical Reports. 3 (2), 152-158 (2015).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in Bioinformatics. 21 (6), 2052-2065 (2020).
- Movassat, M., et al. Coupling between alternative polyadenylation and alternative splicing is limited to terminal introns. RNA Biology. 13 (7), 646-655 (2016).
- Tian, B., Manley, J. L. Alternative polyadenylation of mRNA precursors. Nature Reviews Molecular Cell Biology. 18 (1), 18-30 (2017).
- Herrmann, C. J., et al. PolyASite 2.0: a consolidated atlas of polyadenylation sites from 3' end sequencing. Nucleic Acids Research. 48 (1), 174-179 (2020).
- Liu, R., Loraine, A. E., Dickerson, J. A. Comparisons of computational methods for differential alternative splicing detection using RNA-seq in plant systems. BMC Bioinformatics. 15 (1), 364 (2014).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biology. 17 (1), 13 (2016).
- Anders, S., Reyes, A., Huber, W. Detecting differential usage of exons from RNA-seq data. Genome Research. 22 (10), 2008-2017 (2012).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2014).
- Shen, S., et al. rMATS: Robust and flexible detection of differential alternative splicing from replicate RNA-Seq data. Proceedings of the National Academy of Sciences. 111 (51), 5593-5601 (2014).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in bioinformatics. 21 (6), 2052-2065 (2020).
- Kanitz, A., et al. Comparative assessment of methods for the computational inference of transcript isoform abundance from RNA-seq data. Genome biology. 16 (1), 1-26 (2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550 (2014).
- Sznajder, L. J., et al. Loss of MBNL1 induces RNA misprocessing in the thymus and peripheral blood. Nature Communications. 11, 1-11 (2020).
- Batra, R., et al. Loss of MBNL leads to disruption of developmentally regulated alternative polyadenylation in RNA-mediated disease. Molecular Cell. 56 (2), 311-322 (2014).
- Leinonen, R., Sugawara, H., Shumway, M., et al. The sequence read archive. Nucleic acids research. 39, 19-21 (2010).
- Tange, O. . GNU parallel-the command-line power tool. 36, 42-47 (2011).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet journal. 17 (1), 10-12 (2011).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Robinson, M. D., Oshlack, A. A scaling normalization method for differential expression analysis of RNA-seq data. Genome Biology. 11 (3), 25 (2010).
- Veiga, D. F. T. maser: Mapping Alternative Splicing Events to pRoteins. R package version 1.4.0. , (2019).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (13), 25 (2009).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ramírez, F., Dündar, F., Diehl, S., Grüning, B. A., Manke, T. deepTools: a flexible platform for exploring deep-sequencing data. Nucleic acids research. 42 (1), 187-191 (2014).
- Merino, G. A., Conesa, A., Fernández, E. A. A benchmarking of workflows for detecting differential splicing and differential expression at isoform level in human RNA-seq studies. Briefings in bioinformatics. 20 (2), 471-481 (2019).
- Chhangawala, S., Rudy, G., Mason, C. E., Rosenfeld, J. A. The impact of read length on quantification of differentially expressed genes and splice junction detection. Genome biology. 16 (1), 1-10 (2015).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biol. 17, 13 (2016).
- Trapnell, C., et al. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nat Protoc. 7 (3), 562-578 (2012).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC Bioinformatics. 12, 323 (2011).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nat Biotechnol. 34 (5), 525-527 (2016).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены
Мы используем файлы cookie для улучшения качества работы на нашем веб-сайте.
Продолжая пользоваться нашим веб-сайтом или нажимая кнопку «Продолжить», вы соглашаетесь принять наши файлы cookie.