Method Article
RNA-seq数据中替代剪接和聚腺苷酸化的鉴定
摘要
选择性剪接(AS)和替代聚腺苷酸化(APA)扩大了转录本亚型及其产物的多样性。在这里,我们描述了生物信息学协议,以分析批量RNA-seq和3'末端测序测定,以检测和可视化不同实验条件下变化的AS和APA。
摘要
除了对RNA-Seq进行典型分析以测量实验/生物学条件下的差异基因表达(DGE)外,RNA-seq数据还可用于探索外显子水平的其他复杂调控机制。选择性剪接和聚腺苷酸化通过产生不同的亚型来调节转录后水平的基因表达,在基因的功能多样性中起着至关重要的作用,并且将分析限制在整个基因水平上可能会错过这一重要的调控层。在这里,我们演示了详细的分步分析,以使用Bioconductor和其他封装和功能(包括DEXSeq,Limma封装的diffSplice和rMATS)来识别和可视化不同条件下的差异外显子和聚腺苷酸化位点的使用。
引言
多年来,RNA-seq已被广泛用于估计差异基因表达和基因发现1。此外,它还可用于估计由于表达不同亚型的基因而导致的不同外显子水平使用情况,从而有助于更好地了解转录后水平的基因调控。大多数真核基因通过交替剪接(AS)产生不同的亚型,以增加mRNA表达的多样性。AS事件可分为不同的模式:跳过完全外显子(SE),其中("盒式")外显子与其侧翼内含子一起从转录本中完全去除;当外显子两端存在两个或多个剪接位点时,备选(供体)5'剪接位点选择(A5SS)和备选方案3'(受体)剪接位点选择(A3SS);当内含子保留在成熟的mRNA转录本中时保留内含子(RI)和相互排除外显子使用(MXE),其中一次只能保留两个可用外显子中的一个2,3。替代聚腺苷酸化(APA)在使用替代聚(A)位点从单个转录本产生多种mRNA亚型4的基因表达中也起着重要作用。大多数聚腺苷酸化位点(pA)位于3'非翻译区域(3'UTR),产生具有不同3'UTR长度的mRNA亚型。由于 3' UTR 是识别调控元件的中心枢纽,因此不同的 3' UTR 长度会影响 mRNA 的定位、稳定性和翻译5。有一类 3' 末端测序测定经过优化,可检测 APA,这些APA在协议6的细节上有所不同。此处描述的管道是为 PolyA-seq 设计的,但可以适用于所述的其他协议。
在这项研究中,我们提出了一系列差异外显子分析方法7,8 (图1),可分为两大类:基于外显子(DEXSeq9,diffSplice10)和基于事件的(复制转录本剪接的多变量分析(rMATS)11)。基于外显子的方法将单个外显子条件下的倍数变化与整体基因折叠变化的度量进行比较,以调用差异表达的外显子使用情况,并由此计算AS活性的基因水平测量。基于事件的方法使用外显子-内含子跨越结读取来检测和分类特定的剪接事件,例如外显子跳跃或内含子保留,并在输出3中区分这些AS类型。因此,这些方法为AS12,13的完整分析提供了补充观点。我们选择了DEXSeq(基于DESeq214 DGE封装)和diffSplice(基于Limma10 DGE封装)进行研究,因为它们是差分拼接分析中使用最广泛的软件包之一。rMATS被选为基于事件的分析的常用方法。另一种流行的基于事件的方法是MISO(亚型混合物)1。对于APA,我们采用基于外显子的方法。
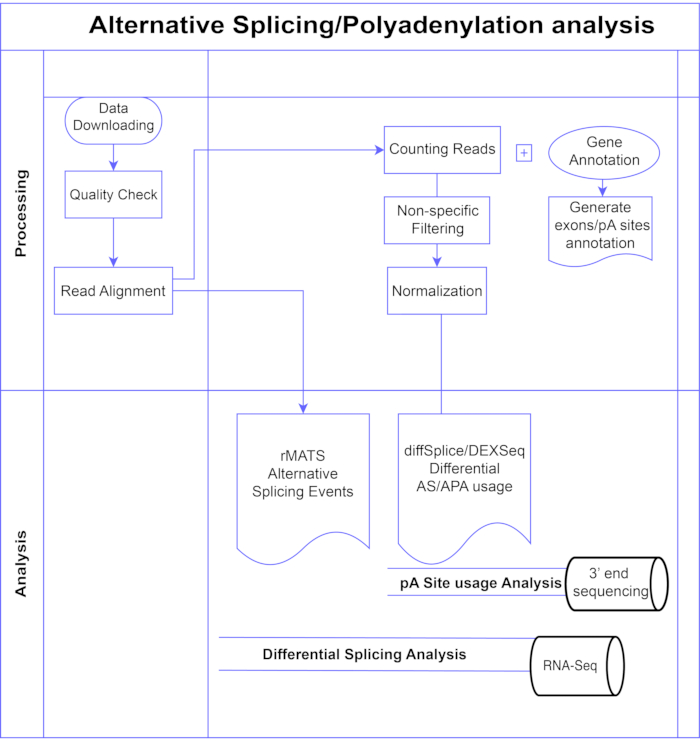
图1.分析管道。 分析中使用的步骤的流程图。步骤包括:获取数据,执行质量检查和读取对齐,然后使用已知外显子,内含子和pA位点的注释对读取进行计数,过滤以去除低计数和标准化。使用diffSplice/DEXSeq方法分析PolyA-seq数据以寻找替代pA位点,使用diffSplice/DEXseq方法分析外显子水平的替代剪接的体RNA-Seq,并使用rMATS分析AS事件。 请点击此处查看此图的大图。
本调查中使用的RNA-seq数据是从基因表达综合(GEO)(GSE138691)15获得的。我们使用本研究的小鼠RNA-seq数据,分为两个条件组:野生型(WT)和肌盲样1型敲除(Mbnl1 KO),每个重复三个。为了证明差异聚腺苷酸化位点的使用分析,我们获得了小鼠胚胎成纤维细胞(MEFs)PolyA-seq数据(GEO加入GSE60487)16。数据有四个条件组:野生型(WT),肌肉盲样1型/2型双敲除(Mbnl1 / 2 DKO),Mbnl 1 / 2 DKO与Mbnl3敲低(KD)和Mbnl1 / 2 DKO与Mbnl3对照(Ctrl)。每个条件组由两个仿行组成。
| 加入全球环境展望 | SRA 运行编号 | 示例名称 | 条件 | 复制 | 组织 | 测 序 | 读取长度 | |
| 核糖核酸序列 | GSM4116218 | SRR10261601 | Mbnl1KO_Thymus_1 | Mbnl1 淘汰赛 | 代表 1 | 胸腺 | 配对端 | 100 基点 |
| GSM4116219 | SRR10261602 | Mbnl1KO_Thymus_2 | Mbnl1 淘汰赛 | 代表 2 | 胸腺 | 配对端 | 100 基点 | |
| GSM4116220 | SRR10261603 | Mbnl1KO_Thymus_3 | Mbnl1 淘汰赛 | 代表 3 | 胸腺 | 配对端 | 100 基点 | |
| GSM4116221 | SRR10261604 | WT_Thymus_1 | 野生型 | 代表 1 | 胸腺 | 配对端 | 100 基点 | |
| GSM4116222 | SRR10261605 | WT_Thymus_2 | 野生型 | 代表 2 | 胸腺 | 配对端 | 100 基点 | |
| GSM4116223 | SRR10261606 | WT_Thymus_3 | 野生型 | 代表 3 | 胸腺 | 配对端 | 100 基点 | |
| 3P-序列 | GSM1480973 | SRR1553129 | WT_1 | 野生型(WT) | 代表 1 | 小鼠胚胎成纤维细胞 (MEF) | 单端 | 40 基点 |
| GSM1480974 | SRR1553130 | WT_2 | 野生型(WT) | 代表 2 | 小鼠胚胎成纤维细胞 (MEF) | 单端 | 40 基点 | |
| GSM1480975 | SRR1553131 | DKO_1 | Mbnl 1/2 双淘汰赛 (DKO) | 代表 1 | 小鼠胚胎成纤维细胞 (MEF) | 单端 | 40 基点 | |
| GSM1480976 | SRR1553132 | DKO_2 | Mbnl 1/2 双淘汰赛 (DKO) | 代表 2 | 小鼠胚胎成纤维细胞 (MEF) | 单端 | 40 基点 | |
| GSM1480977 | SRR1553133 | DKOsiRNA_1 | Mbnl 1/2 双敲除与 Mbnl 3 siRNA (KD) | 代表 1 | 小鼠胚胎成纤维细胞 (MEF) | 单端 | 40 基点 | |
| GSM1480978 | SRR1553134 | DKOsiRNA_2 | Mbnl 1/2 双敲除与 Mbnl 3 siRNA (KD) | 代表 2 | 小鼠胚胎成纤维细胞 (MEF) | 单端 | 36 基点 | |
| GSM1480979 | SRR1553135 | DKONTsiRNA_1 | Mbnl 1/2 双敲除与非靶向 siRNA (Ctrl) | 代表 1 | 小鼠胚胎成纤维细胞 (MEF) | 单端 | 40 基点 | |
| GSM1480980 | SRR1553136 | DKONTsiRNA_2 | Mbnl 1/2 双敲除与非靶向 siRNA (Ctrl) | 代表 2 | 小鼠胚胎成纤维细胞 (MEF) | 单端 | 40 基点 |
表 1.用于分析的RNA-Seq和PolyA-seq数据集摘要。
研究方案
1. 安装分析中使用的工具和 R 包
- Conda 是一个流行且灵活的包管理器,允许在所有平台上方便地安装包及其依赖项。使用"Anaconda"(conda包管理器)安装"conda",可用于安装分析所需的工具/包。
- 根据 https://www.anaconda.com/products/individual#Downloads 的系统要求下载"Anaconda",并按照图形安装程序中的提示进行安装。通过在 Linux 命令行中键入以下内容,使用 'conda' 安装所有必需的软件包。
conda install -c daler sratoolkit
conda install -c conda-forge parallel
conda install -c bioconda star bowtie fastqc rmats rmats2sashimiplot samtools fasterq-dump cutadapt bedtools deeptools - 若要下载协议中使用的所有 R 包,请在 R 控制台(通过键入"R"在 Linux 命令行上启动)或 Rstudio 控制台中键入以下代码。
bioc_packages<- c("DEXSeq", "Rsubread", "EnhancedVolcano", "edgeR", "limma", "maser","GenomicRanges")
packages<- c("magrittr", "rtracklayer", "tidyverse", "openxlsx", "BiocManager")
#Install if not already installed
installed_packages<-packages%in% rownames(installed.packages())
installed_bioc_packages<-bioc_packages%in% rownames(installed.packages())
if(any(installed_packages==FALSE)) {
install.packages(packages[!installed_packages],dependencies=TRUE)
BiocManager::install(packages[!installed_bioc_packages], dependencies=TRUE)
}
注意:在此计算协议中,命令将作为 R 笔记本文件(扩展名为".Rmd"),R 代码文件(扩展名为".R"),或 Linux Bash shell 脚本(扩展名为".sh"的文件)。R 笔记本 (Rmd) 文件应使用 File| 在 RStudio 中打开打开 File...,然后通过单击右上角的绿色箭头以交互方式运行单个代码块(可能是 R 命令或 Bash shell 命令)。R代码文件可以通过在RStudio中打开来运行,或者在Linux命令行上通过前缀"Rscript"来运行,例如Rscript example.R.Shell脚本通过在脚本前面加上"sh"命令在Linux命令行上运行,例如.sh example.sh。
2. 使用 RNA-seq 进行选择性剪接 (AS) 分析
- 数据下载和预处理
注意:下面注释的代码片段可在补充代码文件中找到"AS_analysis_RNASeq.Rmd",以交互方式遵循各个步骤,并且还作为 bash 脚本提供,以便在 Linux 命令行上批量运行(什downloading_data_preprocessing.sh).- 下载原始数据。
- 使用 SRA 工具包 (v2.10.8) 中的"预取"命令从序列读取存档 (SRA) 下载原始数据17。在以下命令中按顺序提供 SRA 入藏 ID,以使用 GNU 并行实用程序18 并行下载它们。要将入藏 ID 的 SRA 文件从 SRR10261601 并行下载到 SRR10261606,请在 Linux 命令行上使用以下方法。
seq 10261601 10261606 | parallel prefetch SRR{} - 使用SRA工具包中的"fastq-dump"功能从存档中提取fastq文件。使用 GNU 并行并一起给出所有 SRA 文件的名称。
parallel -j 3 fastq-dump --gzip --skip-technical --read-filter pass --dumpbase --split-e --clip --origfmt {} ::: - 在 Linux 命令行使用以下方法从 www.ensembl.org 下载小鼠(基因组组装 GRCm39)的参考基因组和注释。
wget -nv -O annotation.gtf.gz http://ftp.ensembl.org/pub/release-103/gtf/mus_musculus/Mus_musculus.GRCm39.103.gtf.gz \ && gunzip -f annotation.gtf.gz
wget -nv -O genome.fa.gz http://ftp.ensembl.org/pub/release-103/fasta/mus_musculus/dna/Mus_musculus.GRCm39.dna.primary_assembly.fa.gz \ && gunzip -f genome.fa.gz
GTF=$(readlink -f annotation.gtf)
GENOME=$(readlink -f genome.fa)
- 使用 SRA 工具包 (v2.10.8) 中的"预取"命令从序列读取存档 (SRA) 下载原始数据17。在以下命令中按顺序提供 SRA 入藏 ID,以使用 GNU 并行实用程序18 并行下载它们。要将入藏 ID 的 SRA 文件从 SRR10261601 并行下载到 SRR10261606,请在 Linux 命令行上使用以下方法。
- 预处理和映射读取到基因组组装
- 质量管理。使用 FASTQC 评估原始读取的质量(FASTQ 质量检查 v0.11.9)19。创建一个输出文件夹,并在多个输入 fasta 文件上并行运行 fastqc。此步骤将为每个样品生成质量报告。在进行进一步分析之前,请检查报告以确保读取质量是可接受的。(请参阅用户手册以了解 https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ 中的报告)
mkdir fastqc_out
parallel "fastqc {} -o fastqc_out" ::: $RAW_DATA/*.fastq.gz
注意:如有必要,使用"cutadapt"20 或"trimmomatic"21 执行适配器修整,以去除侧翼接头的测序,这因RNA片段大小和读取长度而异。在此分析中,我们跳过了此步骤,因为受影响的读取比例很小。 - 读取对齐方式。预处理的下一步包括将读段映射到参考基因组。首先,使用 STAR22 的"genomeGenerate"功能构建参考基因组的索引,然后将原始读数与参考对齐(或者,可从 STAR 网站获得预构建索引,并可直接用于对齐)。在 Linux 命令行运行以下命令。
#Build STAR index
GDIR=STAR_indices
mkdir $GDIR
STAR --runMode genomeGenerate --genomeFastaFiles $GENOME --sjdbGTFfile $GTF --runThreadN 8 --genomeDir $GDIR
ODIR=results/mapping
mkdir -p $ODIR
#Align reads to the genome
for fq1 in $RAW_DATA/*R1.fastq.gz;
do
fq2=$(echo $fq1| sed 's/1.fastq.gz/2.fastq.gz/g');
OUTPUT=$(basename ${fq1}| sed 's/R1.fastq.gz//g');
STAR --genomeDir $GDIR \
--runThreadN 12 \
--readFilesCommand zcat \
--readFilesIn ${fq1}${fq2}\
--outFileNamePrefix $ODIR\/${OUTPUT} \
--outSAMtype BAM SortedByCoordinate \
--outSAMunmapped Within \
--outSAMattributes Standard
Done
注意:读取对齐后,STAR 对准器将为每个样本生成和排序 BAM(二进制对齐图)文件。在继续执行后续步骤之前,必须对 Bam 文件进行排序。
- 质量管理。使用 FASTQC 评估原始读取的质量(FASTQ 质量检查 v0.11.9)19。创建一个输出文件夹,并在多个输入 fasta 文件上并行运行 fastqc。此步骤将为每个样品生成质量报告。在进行进一步分析之前,请检查报告以确保读取质量是可接受的。(请参阅用户手册以了解 https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ 中的报告)
- 下载原始数据。
- 准备外显子注释。
- 运行补充代码文件"prepare_mm_exon_annotation.R",下载的GTF(基因转移格式)格式的注释以准备注释。若要运行,请在 Linux 命令行中键入以下内容。
Rscript prepare_mm_exon_annotation.R annotation.gtf
注意:GTF 文件包含不同亚型的多个外显子条目。此文件用于"折叠"每个外显子的多个转录本 ID。这是定义外显子计数箱的重要步骤。
- 运行补充代码文件"prepare_mm_exon_annotation.R",下载的GTF(基因转移格式)格式的注释以准备注释。若要运行,请在 Linux 命令行中键入以下内容。
- 计数读取。下一步是计算映射到不同转录本/外显子的读取次数。请参阅补充文件:"AS_analysis_RNASeq.Rmd"。
- 加载所需的库:
packages<- c("Rsubread","tidyverse", "magrittr", "EnhancedVolcano", "edgeR","openxlsx")
invisible(lapply(packages, library, character.only=TRUE)) - 加载从上一步 (2.2) 获得的已处理注释文件。
load("mm_exon_anno.RData") - 读取在步骤 2.2.2 中获得的所有 bam 文件作为"featureCounts"的输入以计算读取次数。通过首先列出以 .bam 结尾的目录中的每个文件来读取包含 bam 文件的文件夹。使用 Rsubread 包中的"featureCounts",该包将 bam 文件和处理后的 GTF 注释(参考)作为输入,以生成与每个特征关联的计数矩阵,其中行表示外显子(特征)和列表示样本。
countData <- dir("bams", pattern=".bam$", full.names=T) %>%
featureCounts(annot.ext=anno,
isGTFAnnotationFile=FALSE,
minMQS=0,useMetaFeatures=FALSE,
allowMultiOverlap=TRUE,
largestOverlap=TRUE,
countMultiMappingReads=FALSE,
primaryOnly=TRUE,
isPairedEnd=TRUE,
nthreads=12) - 接下来,执行非特异性过滤以去除低表达的外显子("非特异性"表示过滤中不使用实验条件信息,以避免选择偏差)。使用"edgeR"包23 中的 cpm 函数将数据从原始规模转换为百万分之一 (cpm),并在至少三个样本中保持计数大于可设置阈值的外显子(对于此数据集使用 1 cpm)。同时去除只有一个外显子的基因。
# Non-specific filtering: Remove the exons with low counts
isexpr<- rownames(countData$counts)[rowSums(cpm(countData$counts)>1) >=3]
countData$counts<-countData$counts[rownames(countData$counts) %in%isexpr, ]
anno<-anno%>% filter(GeneID%in% rownames(countData$counts))
# Remove genes with only 1 site and NA in geneIDs
dn<-anno%>%group_by(GeneID)%>%summarise(nsites=n())%>% filter(nsites>1&!is.na(GeneID))
anno<-anno%>% filter(GeneID%in%dn$GeneID)
countData$counts<-countData$counts[rownames(countData$counts) %in%anno$GeneID, ]
注意:使用不同数据时,请检查 featureCount 的必需参数,例如,对于单端读取,请设置"isPairedEnd = FALSE"。请参阅 RSubread 用户指南以选择数据选项,并参阅下面的"讨论"部分。
- 加载所需的库:
- 差分拼接和外显子使用分析。我们描述了此步骤的两种替代方案:DEXSeq 和 DiffSplice。两者都可以使用并给出类似的结果。为了保持一致性,如果您更喜欢用于 DGE 的 DESeq2 软件包,请选择 DEXSeq,并使用 DiffSplice 进行基于 Limma 的 DGE 分析。请参阅补充文件:"AS_analysis_RNASeq.Rmd".
- 使用 DEXSeq 软件包进行差分外显子分析。
- 加载库并创建示例表以定义实验设计。
library(DEXSeq)
sampleTable<-data.frame(row.names= c("Mbnl1KO_Thymus_1", "Mbnl1KO_Thymus_2", "Mbnl1KO_Thymus_3", "WT_Thymus_1", "WT_Thymus_2", "WT_Thymus_3"), condition= rep(c("Mbnl1_KO", "WT"),c(3,3)), libType= rep(c("paired-end")))
注意:行名应与 featureCounts 用于计算读取计数的 bam 文件名一致。样本表包含每个样本的详细信息,其中包括:库类型和条件。这是定义用于检测差异使用情况的对比度或测试组所必需的。 - 准备外显子信息文件。在下一步中创建 DEXSeq 对象时,需要 GRanges(基因组范围)对象 (https://bioconductor.org/packages/release/bioc/html/GenomicRanges.html) 形式的外显子信息作为输入。将基因 ID 与读取计数匹配以创建外显子信息对象。
exoninfo<-anno[anno$GeneID%in% rownames(countData$counts),]
exoninfo<-GRanges(seqnames=anno$Chr,
ranges=IRanges(start=anno$Start, end=anno$End, width=anno$Width),strand=Rle(anno$Strand))
mcols(exoninfo)$TranscriptIDs<-anno$TranscriptIDs
mcols(exoninfo)$Ticker<-anno$Ticker
mcols(exoninfo)$ExonID<-anno$ExonID
mcols(exoninfo)$n<-anno$n
mcols(exoninfo)$GeneID<-anno$GeneID
transcripts_l= strsplit(exoninfo$TranscriptIDs, "\\,")
save(countData, sampleTable, exoninfo, transcripts_l, file="AS_countdata.RData") - 使用 DEXSeqDataSet 函数创建 DEXSeq 对象。DEXSeq 对象将读取计数、外显子特征信息和样本信息收集在一起。使用步骤 3 中生成的读取计数和从上一步获得的外显子信息从计数矩阵创建 DEXSeq 对象。sampleData 参数采用定义样本(及其属性:库类型和条件)的数据帧输入,"design"使用 sampleData 使用模型公式表示法生成用于差异测试的设计矩阵。请注意,一个重要的相互作用项,条件:外显子,表明落在特定外显子上的基因的读取分数取决于实验条件,即存在AS。有关为更复杂的实验设计设置模型公式的完整说明,请参阅 DEXSeq 文档。对于特征信息,需要外显子ID,相应的基因和转录本。
dxd<-DEXSeqDataSet(countData$counts,sampleData=sampleTable, design=~sample+exon+condition:exon,featureID=exoninfo$ExonID,groupID=exoninfo$GeneID,featureRanges=exoninfo, transcripts=transcripts_l) - 归一化和离散估计。接下来,使用以下命令在样品之间执行归一化并估计数据的方差,这是由于 RNA-seq 离散性质和生物变异性的泊松计数噪声。
dxd %<>% estimateSizeFactors %>% estimateDispersions %T>% plotDispEsts - 测试差异使用情况。估计变异后,测试每个基因的差异外显子使用情况并生成结果。
dxd%<>%testForDEU%>%estimateExonFoldChanges(fitExpToVar=
"condition")#Estimate fold changes
dxr=DEXSeqResults(dxd) - 使用以下命令可视化选定基因的剪接事件。
plotDEXSeq(dxr,"Wnk1", displayTranscripts=TRUE, splicing=TRUE,legend
=TRUE,cex.axis=1.2,cex=1.3,lwd=2)
检查 R 笔记本文件"AS_analysis_RNASeq.Rmd"以生成感兴趣的基因的其他图,并生成不同阈值的火山图。
- 加载库并创建示例表以定义实验设计。
- 使用来自 Limma 的差分拼接来识别差分拼接。按照 R 笔记本文件"AS_analysis_RNASeq.Rmd".在继续之前,请确保已按照步骤 2.1-2.3 准备输入文件。
- 加载库
library(limma)
library(edgeR) - 非特定筛选。提取在 2.3 中获得的读取计数矩阵。使用edgeR包中的"DGEList"函数创建特征列表,其中行代表基因,列代表样本。
mycounts=countData$counts
#Change the rownames of the countdata to exon Ids instead of genes for unique rownames.
rownames(mycounts) = exoninfo$ExonID
dge<-DGEList(counts=mycounts)
#Filtering
isexpr<- rowSums(cpm(dge)>1) >=3
dge<-dge[isexpr,,keep.lib.sizes=FALSE]
#Extract the exon annotations for only transcripts meeting non-specific filter
exoninfo=anno%>% filter(ExonID%in% rownames(dge$counts))
#Convert the exoninfo into GRanges object
exoninfo1<-GRanges(seqnames=exoninfo$Chr,
ranges=IRanges(start=exoninfo$Start, end=exoninfo$End, width=exoninfo$Width),strand=Rle(exoninfo$Strand))
mcols(exoninfo1)$TranscriptIDs<-exoninfo$TranscriptIDs
mcols(exoninfo1)$Ticker<-exoninfo$Ticker
mcols(exoninfo1)$ExonID<-exoninfo$ExonID
mcols(exoninfo1)$n<-exoninfo$n
mcols(exoninfo1)$GeneID<-exoninfo$GeneID
transcripts_l= strsplit(exoninfo1$TranscriptIDs, "\\,")
注意:作为非特异性过滤步骤,计数按 cpm 过滤 < n 个样本中的 x 个中的 1 个,其中 x 是任何条件下的最小重复次数。此示例数据的 n = 6 和 x = 3。 - 使用"edgeR"包中的"calcNormFactors"函数使用修剪的 M 值平均值(TMM 归一化方法)24 对样本的计数进行归一化 它将计算比例因子以调整库大小。
dge<-calcNormFactors(dge) - 使用步骤 2.4.1.1 中生成的 sampleTable 并创建设计矩阵。设计矩阵表征了设计。有关更高级实验设计的设计矩阵的详细信息,请参阅 Limma 用户指南 (https://www.bioconductor.org/packages/devel/bioc/vignettes/limma/inst/doc/usersguide.pdf) 第 8 章和第 9 章。
Treat<- factor(sampleTable$condition)
design<- model.matrix(~0+Treat)
colnames(design) <- levels(Treat) - 拟合每个外显子的线性模型。运行"limma"包的"voom"功能来处理RNA-seq数据以估计方差并生成精度权重以校正泊松计数噪声,并将外显子水平计数转换为每百万对数2计数(logCPM)。然后使用"lmfit"函数运行线性建模,将线性模型拟合到每个外显子的表达数据中。使用"eBayes"函数计算拟合模型的经验贝叶斯统计量,以检测微分外显子表达。接下来,为感兴趣的实验比较定义对比矩阵。使用"contrasts.fit"获取每对比较的系数和标准误差。
v<-voom(dge,design,plot=FALSE)
fit<-lmFit(v,design)
fit<-eBayes(fit)
colnames(fit)
cont.matrix<-makeContrasts(
Mbnl1_KO_WT=Mbnl1_KO-WT,
levels=design)
fit2<-contrasts.fit(fit,cont.matrix) - 差分拼接分析。在拟合模型上运行"diffSplice"以测试野生型和敲除型之间基因外显子使用的差异,并使用"topSplice"函数探索排名靠前的结果:test="t"给出AS外显子的排名,test="simes"给出基因的排名。
ex<-diffSplice(fit2,geneid=exoninfo$GeneID,exonid=exoninfo$ExonID)
ts<-topSplice(ex,n=Inf,FDR=0.1, test="t", sort.by="logFC")
tg<-topSplice(ex,n=Inf,FDR=0.1, test="simes") - 可视化。使用"plotSplice"函数绘制结果,在基因参数中给出感兴趣的基因。保存按日志排序的顶部结果 将变化折叠成一个对象并生成火山图以展示外显子。
plotSplice(ex,geneid="Wnk1", FDR=0.1)
#Volcano plot
EnhancedVolcano(ts,lab=ts$ExonID,selectLab= head((ts$ExonID),2000), xlab= bquote(~Log[2]~'fold change'), x='logFC', y='P.Value', title='Volcano Plot', subtitle='Mbnl1_KO vs WT (Limma_diffSplice)', FCcutoff=2, labSize=4,legendPosition="right", caption= bquote(~Log[2]~"Fold change cutoff, 2; FDR 10%"))
- 加载库
- 使用 rMATS
- 确保在工作目录中使用 conda 或 github (https://github.com/Xinglab/rmats-turbo/releases/download/v4.1.1/rmats_turbo_v4_1_1.tar.gz) 安装了最新版本的 rMATS v4.1.1(也称为 rMATS turbo,因为处理时间和内存要求更少)。遵循"AS_analysis_RNASeq.Rmd"中的第 4.3 节。
- 转到包含映射后获得的 bam 文件的文件夹,并根据 rMAT 的要求,通过复制以","分隔的 bam 文件的名称(以及路径)来准备文本文件。应在 Linux 命令行运行以下命令:
mkdir rMATS_analysis
cd bams/
ls -pd "$PWD"/*| grep "WT"| tr '\n'','> Wt.txt
ls -pd "$PWD"/*| grep "Mb"| tr '\n'','> KO.txt
mv *.txt ../rMATS_analysis - 使用上一步中生成的两个输入文件以及 2.1.1.3 中获取的 GTF 文件运行 rmats.py。这将生成一个输出文件夹"rmats_out",其中包含分别描述每个拼接事件的统计数据(p 值和包含水平)的文本文件。
python rmats-turbo/rmats.py --b1 KO.txt --b2 Wt.txt --gtf annotation.gtf -t paired --readLength 50 --nthread 8 --od rmats_out/ --tmp rmats_tmp --task pos
注意:还需要 GTF 文件形式的参考注释。检查参数是否为单端数据,并相应地更改 -t 选项。 - 探索 rMATS 结果。使用Bioconductor包'maser'25 来探索rMATS结果。将结点和外显子计数 (JCEC) 文本文件加载到"激射器"对象中,并通过包括每个拼接事件至少五个平均读取来根据覆盖范围过滤结果。
library(maser)
mbnl1<-maser("/rmats_out/", c("WT","Mbnl1_KO"), ftype="JCEC")
#Filtering out events by coverage
mbnl1_filt<-filterByCoverage(mbnl1,avg_reads=5) - 可视化 rMATS 结果。使用"maser"包中的"topEvents"函数,以 10% 的错误发现率 (FDR) 和拼接百分比 (deltaPSI) 的最小 10% 变化选择显著的拼接事件。接下来,检查感兴趣的单个基因(样本基因-Wnk1)的基因事件,并绘制该基因的每个剪接事件的PSI值。通过指定事件类型生成火山图。
#Top splicing events at 10% FDR
mbnl1_top<-topEvents(mbnl1_filt,fdr=0.1, deltaPSI=0.1)
mbnl1_top
#Check the gene events for a particular gene
mbnl1_wnk1<-geneEvents(mbnl1_filt,geneS="Wnk1", fdr=0.1, deltaPSI=0.1)
maser::display(mbnl1_wnk1,"SE")
plotGenePSI(mbnl1_wnk1,type="SE", show_replicates
=TRUE)
volcano(mbnl1_filt,fdr=0.1, deltaPSI=0.1,type="SE")
+xlab("deltaPSI")+ylab("Log10 Adj. Pvalue")+ggtitle("Volcano Plot of exon skipping events") - 使用"rmats2shahimiplot"包以文本文件的形式为使用rMATS获得的拼接事件结果生成生鱼片图。在 Linux 命令行运行 python 脚本。
python ./src/rmats2sashimiplot/rmats2sashimiplot.py --b1 ../bams/WT_Thymus_1.bam,../bams/WT_Thymus_2.bam,../bams/WT_Thymus_3.bam --b2 ../bams/Mbnl1KO_Thymus_1.bam,../bams/Mbnl1KO_Thymus_2.bam,../bams/Mbnl1KO_Thymus_3.bam -t SE -e ../rMATS_analysis/rmats_out/SE.MATS.JC.txt --l1 WT --l2 Mbnl1_KO --exon_s 1 --intron_s 5 -o ../rMATS_analysis/rmats2shasmi_output
注意:此过程可能非常耗时,因为它将为事件文件中的所有结果生成生鱼片图。从"maser"中选择topEvents函数显示的顶级结果(基因名称和外显子),并可视化相应的生鱼片图。
- 使用 DEXSeq 软件包进行差分外显子分析。
3. 使用 3' 末端测序的替代聚腺苷酸化 (APA) 分析
- 数据下载和预处理
注意: 请参阅补充 R 笔记本文件"APA_analysis_3PSeq_notebook。rmd"表示用于数据下载和预处理步骤的完整命令,或在 Linux 命令行上运行补充 bash 文件 "APA_data_downloading_preprocessing.sh"。- 使用入藏 ID(1553129 至 1553136)从 SRA 下载数据。
- 调整适配器和反向补码以获得检测链序列。
注意:此步骤特定于所使用的PolyA-seq测定。 - 使用领结对齐器26将读取映射到小鼠基因组组装。
- 准备 pA 站点注释。
注意:pA网站注释文件的处理首先使用补充R笔记本文件"APA_analysis_3PSeq_notebook。Rmd"(2.1 - 2.6),然后使用 bash 文件 "APA_annotation_preparation.sh"。- 从 PolyASite 2.0 数据库下载 pA 站点注释6.
- 选择 pA 位点注释以保留 3'-非翻译区域 (UTR) pA 位点,这些位点注释为末端外显子 (TE) 或注释末端外显子 (DS) 下游 1000 nt 以进行下游分析。
- 获得pA位点峰。锚定在每个pA切割位点,并使用bedtools和deeptools27,28可视化平均读取覆盖率。结果表明,映射读段的峰主要分散在切割位点上游~60 bp内(图5和补充图5)。因此,pA位点的坐标从注释文件扩展到其裂解位点上游60 bp。根据所使用的特定3'末端测序方案,此步骤需要针对PolyA-seq以外的检测进行优化。
- 计数读取
- 准备 pA 站点注释文件。
anno<- read.table(file= "flanking60added.pA_annotation.bed",
stringsAsFactors=FALSE, check.names=FALSE, header=FALSE, sep="")
colnames(anno) <- c("chrom", "chromStart", "chromEnd", "name", "score", "strand", "rep", "annotation", "gene_name", "gene_id")
anno<- dplyr::select(anno,name,chrom, chromStart,chromEnd, strand,gene_id,gene_name,rep)
colnames(anno) <- c("GeneID", "Chr", "Start", "End", "Strand", "Ensembl", "Symbol", "repID") - 应用"特征计数"以获取原始计数。将计数表另存为 RData 文件"APA_countData.Rdata",以便使用不同的工具进行 APA 分析。
countData<- dir("bamfiles", pattern="sorted.bam$", full.names=TRUE) %>%
# Read all bam files as input for featureCounts
featureCounts(annot.ext=anno, isGTFAnnotationFile= FALSE,minMQS=0,useMetaFeatures= TRUE,allowMultiOverlap=TRUE, largestOverlap= TRUE,strandSpecific=1, countMultiMappingReads =TRUE,primaryOnly= TRUE,isPairedEnd= FALSE,nthreads=12)%T>%
save(file="APA_countData.Rdata")
注意:请注意更改"功能计数"函数中列出的任何参数。修改"strandSpecific"参数,以确保它与所使用的3'末端测序测定的测序方向一致(根据经验,在基因组浏览器中可视化正负链上的基因数据将阐明这一点)。 - 对计数数据应用非特定筛选。滤波可以显著提高差分pA站点使用测试中的统计鲁棒性。首先,我们去除了那些只有一个pA位点的基因,无法定义差异化的pA位点使用。其次,我们应用基于覆盖率的非特异性过滤:计数通过 cpm 过滤,在 n 个样本中小于 x 中的 1 个,其中 x 是任何条件下的最小重复次数。对于此示例数据,N = 8 和 x = 2。
load(file= "APA_countData.Rdata")# Skip this step if already loaded
# Non-specific filtering: Remove the pA sites not differentially expressed in the samples
countData<-countData$counts%>%as.data.frame%>% .[rowSums(edgeR::cpm(.)>1) >=2, ]
anno%<>% .[.$GeneID%in% rownames(countData), ]
# Remove genes with only 1 site and NA in geneIDs
dnsites<-anno%>%group_by(Symbol)%>%summarise(nsites=n())%>% filter(nsites>1&!is.na(Symbol))
anno<-anno%>% filter(Symbol%in%dnsites$Symbol)
countData<-countData[rownames(countData) %in%anno$GeneID, ]
- 准备 pA 站点注释文件。
- 使用 DEXSeq 和 diffSplice 管道进行差异聚腺苷酸化位点使用分析。
- 使用 DEXSeq 软件包
注意:由于无法为 DEXSeq 管道定义对比矩阵,因此必须分别执行每个两个实验条件的差异 APA 分析。以条件WT和条件DKO的差异APA分析为例进行,以解释该过程。请参阅补充文件"APA_analysis_3PSeq_notebook。马币",用于本节的分步工作流程和其他对比度的差异APA分析。- 加载库并创建示例表以定义实验设计。
c("DEXSeq", "GenomicRanges") %>% lapply(library, character.only=TRUE) %>%invisible
sampleTable1<- data.frame(row.names= c("WT_1","WT_2","DKO_1","DKO_2"),
condition= c(rep("WT", 2), rep("DKO", 2)),
libType= rep("single-end", 4)) - 使用Bioconductor包GRanges准备pA位点信息文件。
# Prepare the GRanges object for DEXSeqDataSet object construction
PASinfo <- GRanges(seqnames = anno$Chr,
ranges = IRanges(start = anno$Start, end = anno$End),strand = Rle(anno$Strand))
mcols(PASinfo)$PASID<-anno$repID
mcols(PASinfo)$GeneEns<-anno$Ensembl
mcols(PASinfo)$GeneID<-anno$Symbol
# Prepare the new feature IDs, replace the strand information with letters to match the current pA site clusterID
new.featureID <- anno$Strand %>% as.character %>% replace(. %in% "+", "F") %>% replace(. %in% "-", "R") %>% paste0(as.character(anno$repID), .) - 使用步骤 3.3 中生成的读取计数和从上一步获得的 pA 站点信息来创建 DEXSeq 对象。
# Select the read counts of the condition WT and DKO
countData1<- dplyr::select(countData, SRR1553129.sorted.bam, SRR1553130.sorted.bam, SRR1553131.sorted.bam, SRR1553132.sorted.bam)
# Rename the columns of countData using sample names in sampleTable
colnames(countData1) <- rownames(sampleTable1)
dxd1<-DEXSeqDataSet(countData=countData1,
sampleData=sampleTable1,
design=~sample+exon+condition:exon,
featureID=new.featureID,
groupID=anno$Symbol,
featureRanges=PASinfo) - 通过定义 DEXSeq 对象中的条件级别来定义对比度对。
dxd1$condition<- factor(dxd1$condition, levels= c("WT", "DKO"))
# The contrast pair will be "DKO - WT" - 归一化和离散估计。与RNA-seq数据类似,对于3'末端测序数据,使用"估计大小因子"函数在样品之间执行归一化(每个样品的比率的列中位数),并使用"估计分散"函数估计数据的变化,然后使用"plotDispEsts"函数可视化色散估计结果。
dxd1 %<>% estimateSizeFactors %>% estimateDispersions %T>% plotDispEsts - 使用函数"testForDEU"对每个基因进行差异pA位点使用测试,然后使用函数"estimateExonFoldChanges"估计pA位点使用率的倍数变化。使用"DEXSeqResults"函数检查结果,并将"FDR < 10%"设置为显著差异 pA 位点的标准。
dxd1 %<>% testForDEU %>% estimateExonFoldChanges(fitExpToVar = "condition")
dxr1 <- DEXSeqResults(dxd1)
dxr1
mcols(dxr1)$description
table(dxr1$padj<0.1) # Check the number of differential pA sites (FDR < 0.1)
table(tapply(dxr1$padj<0.1, dxr1$groupID, any)) # Check the number of gene overlapped with differential pA site - 使用函数"plotDEXSeq"生成的差异APA图和函数"增强火山"生成的火山图可视化差异pA站点使用结果。
# Select the top 100 significant differential pA sites ranked by FDR
topdiff.PAS<- dxr1%>%as.data.frame%>%rownames_to_column%>%arrange(padj)%$%groupID[1:100]
# Apply plotDEXSeq for the visualization of differential polyA usage
plotDEXSeq(dxr1,"S100a7a", legend=TRUE, expression=FALSE,splicing=TRUE, cex.axis=1.2, cex=1.3,lwd=2)
# Apply perGeneQValue to check the top genes with differential polyA site usage
dxr1%<>% .[!is.na(.$padj), ]
dgene<- data.frame(perGeneQValue= perGeneQValue (dxr1)) %>%rownames_to_column("groupID")
dePAS_sig1<-dxr1%>% data.frame() %>%
dplyr::select(-matches("dispersion|stat|countData|genomicData"))%>%
inner_join(dgene)%>%arrange(perGeneQValue)%>%distinct()%>%
filter(padj<0.1)
# Apply EnhancedVolcano package to visualise differential polyA site usage
"EnhancedVolcano"%>% lapply(library, character.only=TRUE) %>%invisible
EnhancedVolcano(dePAS_sig1, lab=dePAS_sig1$groupID, x='log2fold_DKO_WT',
y='pvalue',title='Volcano Plot',subtitle='DKO vs WT',
FCcutoff=1,labSize=4, legendPosition="right",
caption= bquote(~Log[2]~"Fold change cutoff, 1; FDR 10%"))
- 加载库并创建示例表以定义实验设计。
- 使用差异拼接包。请参阅补充 R 笔记本文件"APA_analysis_3PSeq_notebook。Rmd",用于本节的分步工作流。
- 定义差异 pA 使用情况分析的目标对比度。
注意: 此步骤应在构造和处理 DGEList 对象之后执行,该对象包含在 R 笔记本文件"APA_analysis_3PSeq_notebook。啪��contrast.matrix<-makeContrasts(DKO_vs_WT=DKO-
WT,Ctrl_vs_DKO=Ctrl-DKO,
KD_vs_Ctrl=KD-Ctrl,KD_vs_DKO=KD-DKO,levels=design)
fit2<-fit%>%contrasts.fit(contrast.matrix)%>%eBayes
summary(decideTests(fit2))
ex<-diffSplice(fit2,geneid=anno$Symbol,exonid=new.featureID)
topSplice(ex) #Check the top significant results with topSplice - 使用函数"plotSplice"的差分APA图和函数"增强火山"的火山图可视化感兴趣的对比结果(此处为"DKO - WT")。请参阅 R 笔记本文件"APA_analysis_3PSeq_notebook.Rmd" 4.2.7 - 4.2.9 用于其他对比对的可视化。
sig1<-topSplice(ex,n=Inf,FDR=0.1,coef=1, test="t", sort.by="logFC")
sig1.genes<-topSplice(ex,n=Inf,FDR=0.1,coef=1, test="simes")
plotSplice(ex, coef=1,geneid="S100a7a", FDR = 0.1)
plotSplice(ex,coef=1,geneid="Tpm1", FDR = 0.1)
plotSplice(ex,coef=1,geneid="Smc6", FDR = 0.1)
EnhancedVolcano(sig1, lab=sig1$GeneID,xlab= bquote(~Log[2]~'fold change'),
x='logFC', y='P.Value', title='Volcano Plot', subtitle='DKO vs WT',
FCcutoff=1, labSize=6, legendPosition="right")
- 定义差异 pA 使用情况分析的目标对比度。
- 使用 DEXSeq 软件包
结果
运行上述分步工作流程后,AS和APA分析输出和代表性结果以表格和数据图的形式生成,生成如下。
如:
AS分析的主要输出(差异拼接的补充表1;DEXSeq的表2)是显示不同条件差异用法的外显子列表,以及显示其一个或多个组成外显子的显着整体剪接活性的基因列表,按统计学显着性排名。补充表1,选项卡2显示了显着的外显子,列显示外显子与静止的差异FC,每个外显子未调整的p值和调整后的p值(Benjamini-Hockberg校正)。对调整后的 p 值进行阈值设置将给出一组具有定义 FDR 的外显子。补充表1,选项卡3显示了显示整体剪接活性显着性的基因排名列表,其中一列显示了使用Simes方法计算的基因水平调整p值。表 2 中显示了 DEXSeq 的类似数据。补充图1和补充图2显示了Mbnl1,Tcf7和Lef1基因的差异剪接模式,这些模式已在与数据一起提供的已发表文章中进行了实验验证15。作者已经展示了五个基因的实验验证 - Mbnl1,Mbnl2,Lef1,Tcf7和Ncor2。我们的方法在所有这些基因中检测到差异剪接模式。在这里,我们分别使用补充表1-3中获得的DEXSeq,diffSplice和rMATS展示了每个基因的FDR水平:Mbnl1(0,6.6E-61,0),Mbnl2(0,0.18,0),Lef1(1.4E-10,1.3E-04,0),Tcf7(0,1.1E-6,0)和Ncor2(9.2E-11,1.6E-06,0)。
图2显示了从三种不同工具获得的输出之间的比较,并说明了Wnk1基因中的替代剪接模式。火山图如图2A(diffSplice)和图2B(DEXSeq)所示。另外三个高排名基因显示在补充图1(diffSplice)和补充图2(DEXSeq)中。y 轴显示统计显著性(-log10 P 值),x 轴显示效应大小(倍数变化)。位于左上象限或右上象限的基因表明存在实质性的FC和真实差异的有力统计证据。

图2.比较从diffSplice,DEXSeq和rMATS获得的替代剪接结果。|
(A)来自Limma差异剪接分析的RNA-Seq火山图(左):x轴显示对数外显子折叠变化;Y 轴显示 -log10 p 值。每个点对应一个外显子。p 值 = 1E-5 时的水平虚线;两倍变化 (FC) 的垂直虚线。红色外显子显示出实质性的FC和统计学意义。差分剪接图(右):展示了示例基因Wnk1的剪接模式,其中x轴显示每个转录本的外显子ID;y轴显示外显子相对对数折叠变化(外显子的logFC与所有其他外显子的整体logFC之间的差异)。以红色突出显示的外显子显示出统计学上显着的差异表达(FDR < 0.1)。
(B)从DEXSeq分析中获得的RNA-Seq的火山图(左)和差异外显子使用(右)。Wnk1基因显示WT和Mbnl1敲除之间外显子的显着差异使用粉红色,对应于(A)中的相同差异外显子。
(C)从rMATS分析中获得的Wnk1的火山图(左)和生鱼片图(右)。火山图描绘了野生型中显着跳跃(盒式)外显子(SE)事件的火山图,与10%FDR的敲除相比,拼接百分比(PSI或ΔΨ)值的变化>0.1。x 轴显示 PSI 值在条件下的变化,y 轴显示对数 P 值。生鱼片图显示了Wnk1基因中跳过的外显子事件,对应于(A)和(B)中的显着差异外显子。每行代表一个RNA-Seq样品:野生型和Mbnl1敲除的三个重复。高度以RPKM为单位显示读取覆盖率,连接弧表示跨外显子的结读数。带注释的基因模型替代亚型显示在图的底部。C 的底部面板说明了用于计算 PSI 统计数据的结读数。
(D) 维恩图 比较通过不同方法获得的显著差异外显子的数量。请点击此处查看此图的大图。
图2A(右图)显示了排名靠前的基因之一的外显子差异的图表显示,在y轴上显示logFC,在x轴上显示外显子数。这个例子显示了基因 Wnk1的条件之间变化的盒式外显子。来自DEXSeq的差分外显子使用图显示了Wnk1.6.45附近五个外显子位点的差异剪接证据。与WT相比,粉红色突出显示的外显子很可能在Mbnl1 KO样品中被拼接出来。这些外显子与diffSplice获得的结果相辅相成,diffSplice在特定基因组位置显示出类似的模式。更多示例如补充 图1 和 补充图2所示。通过比较UCSC(圣克鲁斯大学)或IGV(综合基因组学查看器)基因组浏览器(未显示)中以RPM(每百万读取数)单位表示的覆盖(摆动)轨迹,可以给出更详细的视图来确认有趣的结果,以及与其他感兴趣的轨迹的视觉相关性,例如已知的基因模型,保护和其他全基因组测定。
rMATS输出表列出了按类型分类的重要替代剪接事件(补充表3)。图 2C 显示了选择性剪接基因的火山图,效应大小由差分"剪接百分比"(PSI或ΔΨ)统计量11测量。
PSI是指与包含盒式外显子一致的读取(即映射到盒式外显子本身的读取或与外显子重叠的连接读取)与与外显子排除一致的读取(即跨相邻上游和下游外显子的连接读取)的百分比(图2C的底图)。图2C的右图显示了Wnk1基因的生鱼片图,差异剪接事件叠加在基因的覆盖轨迹上,Mbnl1 KO中有一个跳跃的外显子。连接外显子的电弧显示连接读数的数量(穿过拼接的内含子的读数)。 补充表3的不同选项卡显示了跨越具有外显子边界的交汇点的每种事件类型(交汇点计数和外显子计数(JCEC))的重要读数。图2D比较了三种工具检测到的显着差异剪接外显子。

图3.通过rMATS分析获得的替代剪接事件。A) 伸缩事件的类型。该图改编自rMATS文档11,该文档解释了本体和交替剪接外显子的剪接事件。标有每个事件的数量为 FDR 10%。B)显示跳跃外显子(SE)的Add3基因的生鱼片图。C)Baz2b基因的生鱼片图显示替代5'剪接位点(A5SS)。D)Lsm14b基因的生鱼片图显示替代3'剪接位点(A3SS)。E) Mta1基因的生鱼片图表现出相互排斥的外显子(MXE)。F) Arpp21基因的生鱼片图显示保留内含子(RI)。红色行表示野生型的三个重复,橙色行表示 Mbnl1 敲除重复。x 轴对应于基因组坐标和链信息,y 轴以 RPKM 为单位显示覆盖范围。请点击此处查看此图的大图。
图3显示了剪接事件SE,A5SS,A3SS,MXE和RI的类型,借助这些事件的顶级重要基因的生鱼片图。在比较WT和Mbnl1_KO的三个重复时,在FDR 10%下共检测到1272个SE事件,130个A5SS,116个A3SS,215个MXE和313个RI事件。生鱼片图以顶级基因为例说明了事件类型。
APA:
APA 分析的输出类似于外显子级 AS 分析。提供了按3'UTR中差异APA活性排名的顶级基因表(补充表4和补充表5)。图4A显示了分别使用diffSplice和DEXSeq生成的3'UTR中差异APA活性的基因火山图。图4B显示了维恩图,比较了从不同管道获得的显著差异的pA站点使用结果。图 4C 和 4D 显示了使用 diffSplice 和 DEXSeq 生成的基因 Fosl2(图 4C)和 Papola (图 4D) 的 3'UTR 中差异 pA 位点使用情况的示意图,经实验验证显示 DKO 中 pA 位点使用率的显著远端到近端移位 (Fosl2) 和显著的近端到远端移位 (Papola), 分别。更多示例如补充图3和补充图4所示。

图4.diffSplice和DEXSeq的替代聚腺苷酸化图。A) 使用 diffSplice 和 DEXSeq 生成的 PolyA-seq 数据的火山图。X 轴显示对数 pA 位点折叠变化;y 轴显示-log 10 p 值。每个点对应一个pA位点。p 值 = 1E-5 时的水平虚线;2 倍 FC 处的垂直虚线。红色外显子显示出实质性的FC和统计学意义。 B)维恩图 比较从不同管道获得的显着差异pA站点使用结果。 C-D) 使用diffSplice和DEXSeq生成的差异APA图显示了 Fosl2 和 Papola 基因的近端,内部和远端pA位点。图由与 图2 (B)相同的功能生成,但用pA位点代替外显子。 请点击此处查看此图的大图。
图5是一个诊断图,用于确认所用PolyA-seq测定的注释pA切割位点周围的预期读数分布。它显示了全基因组水平上锚定在已知pA切割位点的侧翼区域的平均覆盖率。在这种情况下,站点上游的预期读取堆积是可视化的。所有PolyA-seq样品锚定在pA位点的读物分布如补充图5所示。

图5.pA 切割位点周围的均值覆盖率图。 PolyA-seq 数据的解理位点用垂直虚线表示。X轴显示相对于pA裂解位点的碱基位置,上游和下游多达100个核苷酸;y 轴显示所有 pA 切割位点的平均读取覆盖率,按 CPM 中的库大小归一化。 请点击此处查看此图的大图。
通过可视化基因组浏览器中代表性显著差异pA位点的读取覆盖率,可以比较和验证同一管道产生的不同对比度的差异APA结果。图6A是维恩图,比较了从diffSplice获得的不同对比度的显着差异pA位点使用情况。图6B-D是不同基因在pA位点读取覆盖率的IGV快照,其显示的模式与使用diffSplice的APA分析中发现的模式一致。图6B验证了基因Paip2的pA位点使用率的显著近端到远端转移,这在对比DKO与WT中是唯一检测到的,但在其他两种对比中KD与WT和Ctr与WT中没有检测到。 图6C验证了在对比KD与WT中唯一检测到的基因Ccl2的pA位点使用率的显著远端到近端转移, 而图6D验证了基因Cacna2d1的所有对比度的显着差异pA使用情况。
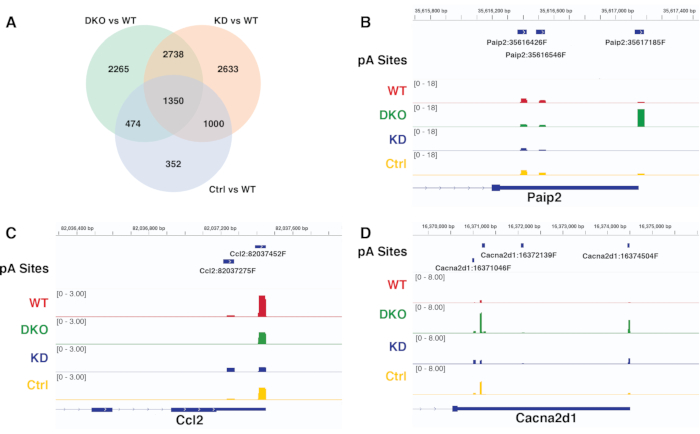
图6.差异拼接结果的对比度比较和验证。A)维恩图比较从diffSplice获得的不同对比度的显着差异pA位点使用结果。 B-D)IGV 快照 可视化了基因 Paip2、Ccl2 和 Cacna2d1 在不同条件下的 pA 峰覆盖率。每个磁道代表特定条件下的读取覆盖率。 请点击此处查看此图的大图。
补充图1。使用Limma差异剪接的RNA-Seq分析。(A)来自Limma差异剪接分析的RNA-Seq火山图:x轴显示对数外显子折叠变化;Y 轴显示 -log10 p 值。每个点对应一个外显子。p 值 = 1E-5 时的水平虚线;两倍变化 (FC) 的垂直虚线。红色外显子显示出实质性的FC和统计学意义。(B-D) 差分拼接图:例如,分别展示了剪接模式,例如基因Mbnl1,Tcf7和Lef1,其中x轴显示每个转录本的外显子ID;y轴显示外显子相对对数折叠变化(外显子的logFC与所有其他外显子的整体logFC之间的差异)。以红色突出显示的外显子显示出统计学上显着的差异表达(FDR < 0.1)。请点击此处下载此文件。
补充图2。使用DEXSeq分析差异外显子使用情况的RNA-Seq。(A)火山图。 (B-D) 差分外显子的使用从 DEXSeq 分析中获得的 RNA-Seq。基因Mbnl1,Tcf7和Lef1分别显示WT和Mbnl1敲除之间外显子的显着差异使用粉红色,对应于补充图1中的相同差异外显子。请点击此处下载此文件。
补充图3。通过diffSplice的替代聚腺苷酸化图。A) 使用 diffSplice 在从小鼠 PolyA-seq 数据中获得的三个对比对中生成的 PolyA-seq 数据的火山图,包括双敲除 (DKO) 与野生型 (WT)、敲低 (KD) 与 WT 以及对照 (Ctrl) 与 WT。 X 轴显示对数 pA 位点折叠变化;y 轴显示-log 10 p 值。每个点对应一个pA位点。p 值 = 1E-5 时的水平虚线;2 倍 FC 处的垂直虚线。红pA位点显示出显著的FC和统计学意义。B) 使用 diffSplice 生成的差异 APA 图,显示高排名基因 S100a7a、Tpm1 和 Smc6 的近端、内部和远端 pA 位点。请点击此处下载此文件。
补充图4。通过 DEXSeq 管道进行差分 pA 使用情况分析。A) 使用 DEXSeq 生成的三对从小鼠 PolyA-seq 数据中获得的 PolyA-seq 数据的火山图,包括双敲除 (DKO) 与野生型 (WT)、敲低 (KD) 与 WT 以及对照 (Ctrl) 与 WT。 X 轴显示对数 pA 位点折叠变化;y 轴显示-log 10 p 值。每个点对应一个pA位点。p 值 = 1E-5 时的水平虚线;2 倍 FC 处的垂直虚线。红pA位点显示出显著的FC和统计学意义。B) 使用 DEXSeq 生成的差异 APA 图,显示高排名基因 S100a7a、Tpm1 和 Smc6 的近端、内部和远端 pA 位点。请点击此处下载此文件。
补充图5。pA 切割位点周围的平均覆盖图和热图。 显示四种情况的覆盖范围,正向和反向链上的基因分别显示。X轴显示相对于pA裂解位点的碱基位置,上游和下游多达100个核苷酸;y 轴是指所有 pA 切割位点上相应相对碱基位置的平均覆盖率。热图提供了另一种视图,每个pA切割位点在x轴上显示为单独的行,按覆盖范围排序。强度显示读取覆盖率(请参阅图例)。请点击此处下载此文件。
补充表1. 差异AS分析的拼接输出。第一个选项卡定义第二(外显子水平)和第三个(基因水平)选项卡中显示的原始输出的列名称。 请按此下载此表格。
补充表2. AS 分析的 DEXSeq 输出。第一个选项卡定义第二(外显子级)和第三(基因级)选项卡中显示的原始输出的列名称。 请按此下载此表格。
补充表3. AS分析的rMATS输出。第一个选项卡定义摘要文件(选项卡 2)的列名和每个事件的 JCEC 文件(选项卡 3-7)。 请按此下载此表格。
补充表4. 差异APA分析的拼接输出。第一个选项卡定义第二(pA 位点级)和第三个(基因级)选项卡中显示的原始输出的列名称。 请按此下载此表格。
补充表5. APA 分析的 DEXSeq 输出。第一个选项卡定义第二(pA 位点级)和第三个(基因级)选项卡中显示的原始输出的列名称。 请按此下载此表格。
补充表6. APA的AS和pA位点的显着改变的外显子数量摘要。 请按此下载此表格。
附表7. AS/APA 分析中使用的工具和软件包的摘要。 请按此下载此表格。
讨论
在这项研究中,我们评估了基于外显子和基于事件的方法,以检测批量RNA-Seq和3'末端测序数据中的AS和APA。基于外显子的AS方法既产生差异表达的外显子列表,又产生按总体基因水平差异剪接活性的统计显着性排序的基因水平排名(表1-2,4-5)。对于diffSplice包,差异用法是通过在外显子水平上拟合加权线性模型来确定的,以估计外显子的差异对数倍数变化与同一基因内其他外显子的平均对数倍数变化(称为每个外显子FC)。通过将单个外显子水平显著性测试组合成Simes方法10的基因智能测试来计算基因水平的统计显着性。该基因水平差异剪接活性的排名随后可用于对涉及的关键途径进行基因集富集分析(GSEA)10。DEXSeq 使用类似的策略,通过拟合广义线性模型来测量差分外显子的使用,尽管在某些步骤(如滤波、归一化和色散估计)上有所不同。在使用DEXSeq和DiffSplice比较显示AS活性和APA的前500个排名的外显子时,我们发现分别有310个外显子和300个pA位点的重叠,这表明两种基于外显子的方法的一致性,这也在之前的研究29中得到证明。建议结合使用基于外显子(DEXSeq 或 diffSplice)和基于事件的方法对 AS 进行全面检测和分类。对于APA,用户可以选择DEXSeq或diffSplice:这两种方法已被证明在广泛的转录组学实验中表现良好29。
在准备用于AS分析的RNA-seq文库时,重要的是使用链特异性批量RNA-seq方案8,因为脊椎动物基因组中的大部分基因在不同的链上重叠,并且非链特异性方案无法区分这些重叠区域,混淆了最终的外显子检测。另一个考虑因素是读取深度,剪接分析需要比DGE更深入的测序,例如每个样本30-6000万次读取,而DGE的每个样本5-2500万次读取(https://sapac.support.illumina.com/bulletins/2017/04/considerations-for-rna-seq-read-length-and-coverage-.html)。协议中演示的所有工具都支持单端和配对测序数据。如果仅使用已知的基因注释来检测连接读数,则可以使用单端较短的读数(≥ 50 bp),尽管新型剪接连接的从头检测受益于配对端和更长(≥ 100bp)读数30,31。RNA提取方案的选择 - polyA选择或rRNA去除 - 取决于RNA的质量和实验问题 - 参见31进行讨论。根据库构建的细节,需要修改此处给出的示例脚本,用于读取对齐、特征计数和 rMATS 的参数。在使用 featureCount 或类似方法计算初始外显子水平读取计数时,必须注意正确配置计数和搁浅的功能选项:在 featureCount 中,我们为所使用的链特异性 RNA-seq 协议适当地设置了"strandSpecific"参数;对于外显子级量化,预计读取将映射到相邻的外显子上,因此我们将 allowMultiOverlap 参数设置为 TRUE。对于APA,有不同的3'末端测序方案6,其峰相对于pA位点的精确位置不同。对于我们的示例数据,我们确定峰位于pA位点上游60 bp,如图5所示,并且该分析需要适用于其他3'末端测序方案。
在该协议中,我们将范围限制为讨论单个外显子水平的差异分析,以及由相邻外显子 - 内含子组合组成的剪接事件。我们不讨论基于亚型从头重建的分析类别,例如袖扣,袖扣32,RSEM33,Kallisto34 ,旨在检测和量化整个替代亚型的绝对和相对表达。外显子和基于事件的方法对于检测单个剪接事件30 更为敏感,并且在许多情况下提供进一步分析所需的所有信息,而无需亚型水平的定量。
此协议中最新版本的源文件可在 https://github.com/jiayuwen/AS_APA_JoVE
披露声明
作者没有什么可透露的。
致谢
这项研究得到了澳大利亚研究委员会(ARC)未来奖学金(FT16010043)和澳大利亚国立大学期货计划的支持。
材料
| Name | Company | Catalog Number | Comments |
| Not relevent for computational study |
参考文献
- Katz, Y., Wang, E. T., Airoldi, E. M., Burge, C. B. Analysis and design of RNA sequencing experiments for identifying isoform regulation. Nature Methods. 7 (12), 1009-1015 (2010).
- Wang, Y., et al. Mechanism of alternative splicing and its regulation. Biomedical Reports. 3 (2), 152-158 (2015).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in Bioinformatics. 21 (6), 2052-2065 (2020).
- Movassat, M., et al. Coupling between alternative polyadenylation and alternative splicing is limited to terminal introns. RNA Biology. 13 (7), 646-655 (2016).
- Tian, B., Manley, J. L. Alternative polyadenylation of mRNA precursors. Nature Reviews Molecular Cell Biology. 18 (1), 18-30 (2017).
- Herrmann, C. J., et al. PolyASite 2.0: a consolidated atlas of polyadenylation sites from 3' end sequencing. Nucleic Acids Research. 48 (1), 174-179 (2020).
- Liu, R., Loraine, A. E., Dickerson, J. A. Comparisons of computational methods for differential alternative splicing detection using RNA-seq in plant systems. BMC Bioinformatics. 15 (1), 364 (2014).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biology. 17 (1), 13 (2016).
- Anders, S., Reyes, A., Huber, W. Detecting differential usage of exons from RNA-seq data. Genome Research. 22 (10), 2008-2017 (2012).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2014).
- Shen, S., et al. rMATS: Robust and flexible detection of differential alternative splicing from replicate RNA-Seq data. Proceedings of the National Academy of Sciences. 111 (51), 5593-5601 (2014).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in bioinformatics. 21 (6), 2052-2065 (2020).
- Kanitz, A., et al. Comparative assessment of methods for the computational inference of transcript isoform abundance from RNA-seq data. Genome biology. 16 (1), 1-26 (2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550 (2014).
- Sznajder, L. J., et al. Loss of MBNL1 induces RNA misprocessing in the thymus and peripheral blood. Nature Communications. 11, 1-11 (2020).
- Batra, R., et al. Loss of MBNL leads to disruption of developmentally regulated alternative polyadenylation in RNA-mediated disease. Molecular Cell. 56 (2), 311-322 (2014).
- Leinonen, R., Sugawara, H., Shumway, M., et al. The sequence read archive. Nucleic acids research. 39, 19-21 (2010).
- Tange, O. . GNU parallel-the command-line power tool. 36, 42-47 (2011).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet journal. 17 (1), 10-12 (2011).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Robinson, M. D., Oshlack, A. A scaling normalization method for differential expression analysis of RNA-seq data. Genome Biology. 11 (3), 25 (2010).
- Veiga, D. F. T. maser: Mapping Alternative Splicing Events to pRoteins. R package version 1.4.0. , (2019).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (13), 25 (2009).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ramírez, F., Dündar, F., Diehl, S., Grüning, B. A., Manke, T. deepTools: a flexible platform for exploring deep-sequencing data. Nucleic acids research. 42 (1), 187-191 (2014).
- Merino, G. A., Conesa, A., Fernández, E. A. A benchmarking of workflows for detecting differential splicing and differential expression at isoform level in human RNA-seq studies. Briefings in bioinformatics. 20 (2), 471-481 (2019).
- Chhangawala, S., Rudy, G., Mason, C. E., Rosenfeld, J. A. The impact of read length on quantification of differentially expressed genes and splice junction detection. Genome biology. 16 (1), 1-10 (2015).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biol. 17, 13 (2016).
- Trapnell, C., et al. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nat Protoc. 7 (3), 562-578 (2012).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC Bioinformatics. 12, 323 (2011).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nat Biotechnol. 34 (5), 525-527 (2016).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。