Method Article
Identificação de Splicing Alternativo e Poliadenilação em Dados de RNA-seq
Neste Artigo
Resumo
O splicing alternativo (AS) e a poliadenilação alternativa (APA) expandem a diversidade de isoformas de transcritos e seus produtos. Aqui, descrevemos protocolos de bioinformática para analisar ensaios de RNA-seq em massa e sequenciamento final de 3' para detectar e visualizar AS e APA variando entre condições experimentais.
Resumo
Assim como a análise típica de RNA-Seq para medir a expressão gênica diferencial (DGE) em condições experimentais / biológicas, os dados de RNA-seq também podem ser utilizados para explorar outros mecanismos regulatórios complexos no nível do éxon. O splicing alternativo e a poliadenilação desempenham um papel crucial na diversidade funcional de um gene, gerando diferentes isoformas para regular a expressão gênica no nível pós-transcricional, e limitar as análises a todo o nível do gene pode perder essa importante camada reguladora. Aqui, demonstramos análises detalhadas passo a passo para identificação e visualização do uso diferencial do éxon e do local de poliadenilação em todas as condições, usando Bioconductor e outros pacotes e funções, incluindo DEXSeq, diffSplice do pacote Limma e rMATS.
Introdução
O RNA-seq tem sido amplamente utilizado ao longo dos anos, tipicamente para estimar a expressão gênica diferencial e a descoberta de genes1. Além disso, também pode ser utilizado para estimar o uso variável do nível de éxons devido ao gene que expressa diferentes isoformas, contribuindo assim para uma melhor compreensão da regulação gênica no nível pós-transcricional. A maioria dos genes eucarióticos gera diferentes isoformas por splicing alternativo (AS) para aumentar a diversidade de expressão de mRNA. Os eventos AS podem ser divididos em diferentes padrões: pulando éxons completos (SE), onde um éxon ("") é completamente removido da transcrição junto com seus íntrons flanqueadores; seleção alternativa (doador) de local de emenda de 5' (A5SS) e seleção de local de emenda alternativa de 3' (aceitador) (A3SS) quando dois ou mais locais de emenda estiverem presentes em cada extremidade de um éxon; retenção de íntrons (IR) quando um íntron é retido dentro do transcrito de mRNA maduro e exclusão mútua do uso de éxons (MXE) onde apenas um dos dois éxons disponíveis pode ser retido de cada vez 2,3. A poliadenilação alternativa (APA) também desempenha um papel importante na regulação da expressão gênica usando sítios alternativos de poli (A) para gerar múltiplas isoformas de mRNA a partir de um único transcrito4. A maioria dos sítios de poliadenilação (pAs) está localizada na região não traduzida de 3' (UTRs de 3'), gerando isoformas de mRNA com diversos comprimentos UTR de 3'. Como a UTR de 3' é o hub central para o reconhecimento de elementos regulatórios, diferentes comprimentos de UTR de 3' podem afetar a localização, a estabilidade e a tradução do mRNA5. Há uma classe de ensaios de sequenciamento final de 3' otimizados para detectar APA que diferem nos detalhes do protocolo6. O pipeline descrito aqui é projetado para PolyA-seq, mas pode ser adaptado para outros protocolos, conforme descrito.
Neste estudo, apresentamos um pipeline de métodos diferenciais de análise de éxons7,8 (Figura 1), que podem ser divididos em duas grandes categorias: baseada em éxons (DEXSeq9, diffSplice 10) e baseada em eventos (replicate Multivariate Analysis of Transcript Splicing (rMATS)11). Os métodos baseados em éxons comparam a mudança de dobra entre as condições de éxons individuais, contra uma medida de mudança geral de dobra gênica para chamar o uso de éxons diferencialmente expressos e, a partir disso, calculam uma medida em nível de gene da atividade EA. Os métodos baseados em eventos usam leituras de junção de abrangência de exon-intron para detectar e classificar eventos de splicing específicos, como pulo de éxon ou retenção de íntrons, e distinguir esses tipos de AS na saída3. Assim, esses métodos fornecem visões complementares para uma análise completa da EA12,13. Selecionamos o DEXSeq (baseado no pacote DESeq214 DGE) e o diffSplice (baseado no pacote Limma10 DGE) para o estudo, pois estão entre os pacotes mais utilizados para análise de splicing diferencial. O rMATS foi escolhido como um método popular para análise baseada em eventos. Outro método popular baseado em eventos é o MISO (Mix of Isoforms)1. Para a APA, adaptamos a abordagem baseada em exons.
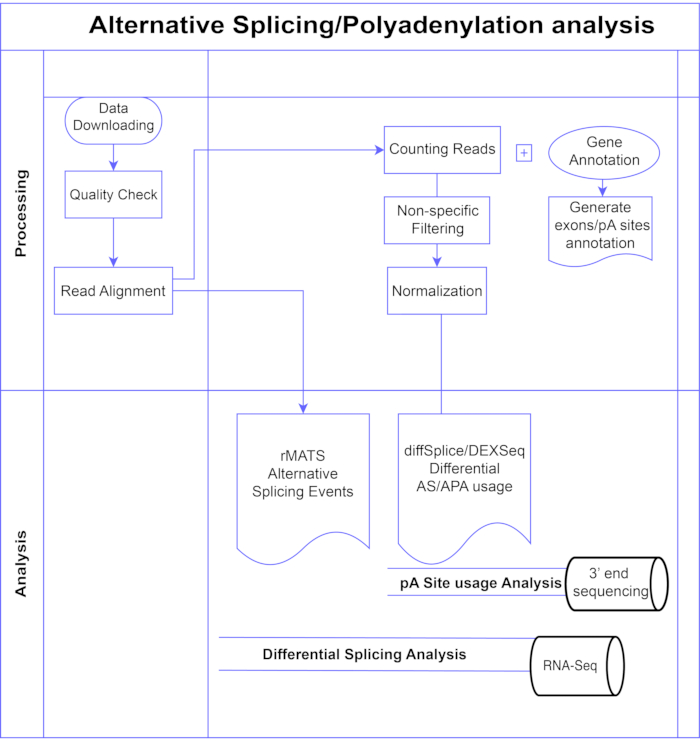
Figura 1. Análise de pipeline. Fluxograma das etapas utilizadas na análise. As etapas incluem: obtenção dos dados, realização de verificações de qualidade e alinhamento de leitura seguidas de contagem de leituras usando anotações para éxons, íntrons e sites pA conhecidos, filtragem para remover contagens baixas e normalização. Os dados de PolyA-seq foram analisados para sítios alternativos de pA usando os métodos diffSplice/DEXSeq, o RNA-Seq em massa foi analisado para splicing alternativo no nível do éxon com os métodos diffSplice/DEXseq e os eventos AS analisados com rMATS. Por favor, clique aqui para ver uma versão maior desta figura.
Os dados de RNA-seq utilizados neste levantamento foram adquiridos do Gene Expression Omnibus (GEO) (GSE138691)15. Utilizamos dados de RNA-seq de camundongos deste estudo com dois grupos de condições: wild-type (WT) e Muscleblind-like type 1 knockout (Mbnl1 KO) com três repetições cada. Para demonstrar a análise diferencial do uso do sítio de poliadenilação, obtivemos dados PolyA-seq de fibroblastos embrionários de camundongos (MEFs) (GEO Accession GSE60487)16. Os dados têm quatro grupos de condições: Wild-type (WT), Muscleblind-like type1/type 2 double knockout (Mbnl1/2 DKO), Mbnl 1/2 DKO com Mbnl3 knockdown (KD) e Mbnl1/2 DKO com controle Mbnl3 (Ctrl). Cada grupo de condições consiste em duas replicações.
| Adesão ao GEO | Número de execução SRA | Nome do exemplo | Condição | Replicar | Tecido | Seqüenciamento | Comprimento de leitura | |
| RNA-Seq | Telemóvel GSM4116218 | SRR10261601 | Mbnl1KO_Thymus_1 | Nocaute Mbnl1 | Rep 1 | Timo | Extremidade emparelhada | 100 pb |
| Telemóvel GSM4116219 | SRR10261602 | Mbnl1KO_Thymus_2 | Nocaute Mbnl1 | Rep 2 | Timo | Extremidade emparelhada | 100 pb | |
| Telemóvel GSM4116220 | SRR10261603 | Mbnl1KO_Thymus_3 | Nocaute Mbnl1 | Rep 3 | Timo | Extremidade emparelhada | 100 pb | |
| Telemóvel GSM4116221 | SRR10261604 | WT_Thymus_1 | Tipo selvagem | Rep 1 | Timo | Extremidade emparelhada | 100 pb | |
| Telemóvel GSM4116222 | SRR10261605 | WT_Thymus_2 | Tipo selvagem | Rep 2 | Timo | Extremidade emparelhada | 100 pb | |
| Telemóvel GSM4116223 | SRR10261606 | WT_Thymus_3 | Tipo selvagem | Rep 3 | Timo | Extremidade emparelhada | 100 pb | |
| 3P-Seq | Mensagem GSM1480973 | SRR1553129 | WT_1 | Tipo selvagem (WT) | Rep 1 | Fibroblastos embrionários de camundongos (MEFs) | Extremidade única | 40 pb |
| Telemóvel 1480974 | SRR1553130 | WT_2 | Tipo selvagem (WT) | Rep 2 | Fibroblastos embrionários de camundongos (MEFs) | Extremidade única | 40 pb | |
| Telemóvel 1480975 | SRR1553131 | DKO_1 | Mbnl 1/2 nocaute duplo (DKO) | Rep 1 | Fibroblastos embrionários de camundongos (MEFs) | Extremidade única | 40 pb | |
| Mensagem GSM1480976 | SRR1553132 | DKO_2 | Mbnl 1/2 nocaute duplo (DKO) | Rep 2 | Fibroblastos embrionários de camundongos (MEFs) | Extremidade única | 40 pb | |
| Telemóvel 1480977 | SRR1553133 | DKOsiRNA_1 | Mbnl 1/2 nocaute duplo com Mbnl 3 siRNA (KD) | Rep 1 | Fibroblastos embrionários de camundongos (MEFs) | Extremidade única | 40 pb | |
| Telemóvel 1480978 | SRR1553134 | DKOsiRNA_2 | Mbnl 1/2 nocaute duplo com Mbnl 3 siRNA (KD) | Rep 2 | Fibroblastos embrionários de camundongos (MEFs) | Extremidade única | 36 pb | |
| Telemóvel 1480979 | SRR1553135 | DKONTsiRNA_1 | Mbnl 1/2 nocaute duplo com siRNA não direcionado (Ctrl) | Rep 1 | Fibroblastos embrionários de camundongos (MEFs) | Extremidade única | 40 pb | |
| Telemóvel 1480980 | SRR1553136 | DKONTsiRNA_2 | Mbnl 1/2 nocaute duplo com siRNA não direcionado (Ctrl) | Rep 2 | Fibroblastos embrionários de camundongos (MEFs) | Extremidade única | 40 pb |
Tabela 1. Resumo dos conjuntos de dados de RNA-Seq e PolyA-seq utilizados para a análise.
Protocolo
1. Instalação de ferramentas e pacotes R utilizados na análise
- Conda é um gerenciador de pacotes popular e flexível que permite a instalação conveniente de pacotes com suas dependências em todas as plataformas. Use 'Anaconda' (gerenciador de pacotes conda) para instalar 'conda', que pode ser usado para instalar as ferramentas/pacotes necessários para a análise.
- Baixe 'Anaconda' de acordo com os requisitos do sistema de https://www.anaconda.com/products/individual#Downloads e instale-o seguindo as instruções no instalador gráfico. Instale todos os pacotes necessários usando 'conda' digitando o seguinte na linha de comando do Linux.
conda install -c daler sratoolkit
conda install -c conda-forge parallel
conda install -c bioconda star bowtie fastqc rmats rmats2sashimiplot samtools fasterq-dump cutadapt bedtools deeptools - Para baixar todos os pacotes R usados no protocolo, digite o seguinte código no console R (iniciado na linha de comando do Linux digitando 'R') ou no console Rstudio.
bioc_packages<- c("DEXSeq", "Rsubread", "EnhancedVolcano", "edgeR", "limma", "maser","GenomicRanges")
packages<- c("magrittr", "rtracklayer", "tidyverse", "openxlsx", "BiocManager")
#Install if not already installed
installed_packages<-packages%in% rownames(installed.packages())
installed_bioc_packages<-bioc_packages%in% rownames(installed.packages())
if(any(installed_packages==FALSE)) {
install.packages(packages[!installed_packages],dependencies=TRUE)
BiocManager::install(packages[!installed_bioc_packages], dependencies=TRUE)
}
NOTA: Neste protocolo computacional, os comandos serão dados como arquivos R Notebook (arquivos com extensão ". Rmd"), arquivos de código R (arquivos com extensão ". R"), ou Linux Bash shell scripts (arquivos com extensão ".sh"). Arquivos R Notebook (Rmd) devem ser abertos no RStudio usando File| Abra Arquivo..., e blocos de código individuais (que podem ser comandos R ou comandos de shell Bash) e execute interativamente clicando na seta verde no canto superior direito. Os arquivos de código R podem ser executados abrindo no RStudio, ou na linha de comando do Linux prefaciando com "Rscript", por exemplo, Rscript example.R. Os scripts Shell são executados na linha de comando do Linux prefaciando o script com o comando "sh", por exemplo.sh example.sh.
2. Análise de splicing alternativo (AS) utilizando RNA-seq
- Download e pré-processamento de dados
Observação : os trechos de código anotados abaixo estão disponíveis no arquivo de código suplementar "AS_analysis_RNASeq.Rmd", para seguir as etapas individuais interativamente, e também são fornecidos como um script bash a ser executado em lote na linha de comando do Linux (sh downloading_data_preprocessing.sh).- Baixando os dados brutos.
- Baixe os dados brutos do SRA (Sequence Read Archive) usando o comando 'prefetch' do SRA toolkit (v2.10.8)17. Dê os IDs de Adesão SRA em sequência no comando a seguir para baixá-los em paralelo usando o utilitário paralelo GNU18. Para baixar arquivos SRA de ids de acesso de SRR10261601 para SRR10261606 em paralelo, use o seguinte na linha de comando do Linux.
seq 10261601 10261606 | parallel prefetch SRR{} - Extraia os arquivos fastq do arquivo usando a função 'fastq-dump' do kit de ferramentas SRA. Use o GNU paralelo e dê os nomes de todos os arquivos SRA juntos.
parallel -j 3 fastq-dump --gzip --skip-technical --read-filter pass --dumpbase --split-e --clip --origfmt {} ::: - Baixe o genoma de referência e as anotações para o Mouse (assembly do genoma GRCm39) do www.ensembl.org usando o seguinte na linha de comando do Linux.
wget -nv -O annotation.gtf.gz http://ftp.ensembl.org/pub/release-103/gtf/mus_musculus/Mus_musculus.GRCm39.103.gtf.gz \ && gunzip -f annotation.gtf.gz
wget -nv -O genome.fa.gz http://ftp.ensembl.org/pub/release-103/fasta/mus_musculus/dna/Mus_musculus.GRCm39.dna.primary_assembly.fa.gz \ && gunzip -f genome.fa.gz
GTF=$(readlink -f annotation.gtf)
GENOME=$(readlink -f genome.fa)
- Baixe os dados brutos do SRA (Sequence Read Archive) usando o comando 'prefetch' do SRA toolkit (v2.10.8)17. Dê os IDs de Adesão SRA em sequência no comando a seguir para baixá-los em paralelo usando o utilitário paralelo GNU18. Para baixar arquivos SRA de ids de acesso de SRR10261601 para SRR10261606 em paralelo, use o seguinte na linha de comando do Linux.
- Pré-processamento e mapeamento de leituras para a montagem do genoma
- Controle de Qualidade. Avalie a qualidade das leituras brutas com o FASTQC (FASTQ Quality Check v0.11.9)19. Crie uma pasta de saída e execute fastqc com paralelo em vários arquivos fasta de entrada. Esta etapa gerará um relatório de qualidade para cada amostra. Examine os relatórios para garantir que a qualidade das leituras seja aceitável antes de fazer uma análise mais aprofundada. (Consulte o manual do usuário para entender os relatórios em https://www.bioinformatics.babraham.ac.uk/projects/fastqc/)
mkdir fastqc_out
parallel "fastqc {} -o fastqc_out" ::: $RAW_DATA/*.fastq.gz
NOTA: Se necessário, execute o corte do adaptador com 'cutadapt'20 ou 'trimmomatic'21 para remover o sequenciamento em adaptadores de flanco, que varia com base no tamanho do fragmento de RNA e no comprimento de leitura. Nesta análise, pulamos essa etapa, pois a fração de leituras afetada foi mínima. - Alinhamento de leitura. A próxima etapa no pré-processamento inclui o mapeamento das leituras para o genoma de referência. Em primeiro lugar, construa o índice para o genoma de referência usando a função 'genomeGenerate' do STAR22 e, em seguida, alinhe as leituras brutas à referência (alternativamente, os índices pré-construídos estão disponíveis no site do STAR e podem ser usados diretamente para alinhamento). Execute os seguintes comandos na linha de comando do Linux.
#Build STAR index
GDIR=STAR_indices
mkdir $GDIR
STAR --runMode genomeGenerate --genomeFastaFiles $GENOME --sjdbGTFfile $GTF --runThreadN 8 --genomeDir $GDIR
ODIR=results/mapping
mkdir -p $ODIR
#Align reads to the genome
for fq1 in $RAW_DATA/*R1.fastq.gz;
do
fq2=$(echo $fq1| sed 's/1.fastq.gz/2.fastq.gz/g');
OUTPUT=$(basename ${fq1}| sed 's/R1.fastq.gz//g');
STAR --genomeDir $GDIR \
--runThreadN 12 \
--readFilesCommand zcat \
--readFilesIn ${fq1}${fq2}\
--outFileNamePrefix $ODIR\/${OUTPUT} \
--outSAMtype BAM SortedByCoordinate \
--outSAMunmapped Within \
--outSAMattributes Standard
Done
Observação : O alinhador STAR irá gerar e classificar arquivos BAM (Mapa de alinhamento binário) para cada exemplo após o alinhamento de leitura. Os arquivos Bam devem ser classificados antes de prosseguir para outras etapas.
- Controle de Qualidade. Avalie a qualidade das leituras brutas com o FASTQC (FASTQ Quality Check v0.11.9)19. Crie uma pasta de saída e execute fastqc com paralelo em vários arquivos fasta de entrada. Esta etapa gerará um relatório de qualidade para cada amostra. Examine os relatórios para garantir que a qualidade das leituras seja aceitável antes de fazer uma análise mais aprofundada. (Consulte o manual do usuário para entender os relatórios em https://www.bioinformatics.babraham.ac.uk/projects/fastqc/)
- Baixando os dados brutos.
- Preparando anotações Exon.
- Execute o arquivo de código suplementar "prepare_mm_exon_annotation. R" com a anotação baixada no formato GTF (Gene transfer format) para preparar as anotações. Para executar, digite o seguinte na linha de comando do Linux.
Rscript prepare_mm_exon_annotation.R annotation.gtf
Observação : O arquivo GTF contém várias entradas de éxon para diferentes isoformas. Esse arquivo é usado para "recolher" as várias IDs de transcrição para cada éxon. É um passo importante definir caixas de contagem de éxons.
- Execute o arquivo de código suplementar "prepare_mm_exon_annotation. R" com a anotação baixada no formato GTF (Gene transfer format) para preparar as anotações. Para executar, digite o seguinte na linha de comando do Linux.
- Contando leituras. O próximo passo é contar o número de leituras mapeadas para diferentes transcrições/éxons. Consulte o arquivo suplementar: "AS_analysis_RNASeq.Rmd".
- Carregar bibliotecas necessárias:
packages<- c("Rsubread","tidyverse", "magrittr", "EnhancedVolcano", "edgeR","openxlsx")
invisible(lapply(packages, library, character.only=TRUE)) - Carregue o arquivo de anotação processado obtido da etapa anterior (2.2).
load("mm_exon_anno.RData") - Leia todos os arquivos bam obtidos na etapa 2.2.2 como entrada para 'featureCounts' para contar leituras. Leia a pasta que contém os arquivos bam listando primeiro cada arquivo do diretório que termina com .bam. Use 'featureCounts' do pacote Rsubread, que usa arquivos bam e anotação GTF processada (referência) como entrada para gerar uma matriz de contagens associadas a cada recurso com linhas representando exons(features) e colunas representando amostras.
countData <- dir("bams", pattern=".bam$", full.names=T) %>%
featureCounts(annot.ext=anno,
isGTFAnnotationFile=FALSE,
minMQS=0,useMetaFeatures=FALSE,
allowMultiOverlap=TRUE,
largestOverlap=TRUE,
countMultiMappingReads=FALSE,
primaryOnly=TRUE,
isPairedEnd=TRUE,
nthreads=12) - Em seguida, execute a filtragem não específica para remover éxons pouco expressos ("não específico" indica que as informações de condição experimental não são usadas na filtragem, para evitar vieses de seleção). Transforme os dados da escala bruta em contagens por milhão (cpm) usando a função cpm do pacote 'edgeR'23 e mantenha os éxons com contagens maiores que um limite definível (para este conjunto de dados um cpm é usado) em pelo menos três amostras. Também remova genes com apenas um éxon.
# Non-specific filtering: Remove the exons with low counts
isexpr<- rownames(countData$counts)[rowSums(cpm(countData$counts)>1) >=3]
countData$counts<-countData$counts[rownames(countData$counts) %in%isexpr, ]
anno<-anno%>% filter(GeneID%in% rownames(countData$counts))
# Remove genes with only 1 site and NA in geneIDs
dn<-anno%>%group_by(GeneID)%>%summarise(nsites=n())%>% filter(nsites>1&!is.na(GeneID))
anno<-anno%>% filter(GeneID%in%dn$GeneID)
countData$counts<-countData$counts[rownames(countData$counts) %in%anno$GeneID, ]
Observação : verifique os parâmetros necessários para featureCounts ao usar dados diferentes, por exemplo, para leituras de extremidade única, defina 'isPairedEnd = FALSE'. Consulte o guia do usuário do RSubread para escolher as opções para seus dados e consulte a seção Discussão abaixo.
- Carregar bibliotecas necessárias:
- Splicing diferencial e análise de uso de éxons. Descrevemos duas alternativas para esta etapa: DEXSeq e DiffSplice. Qualquer um pode ser usado e dar resultados semelhantes. Para obter consistência, selecione DEXSeq se preferir um pacote DESeq2 para DGE e use DiffSplice para uma análise DGE baseada em Limma. Consulte o arquivo suplementar: "AS_analysis_RNASeq.Rmd".
- Usando o pacote DEXSeq para análise diferencial de éxons.
- Carregue a biblioteca e crie uma tabela de exemplo para definir o experimento.
library(DEXSeq)
sampleTable<-data.frame(row.names= c("Mbnl1KO_Thymus_1", "Mbnl1KO_Thymus_2", "Mbnl1KO_Thymus_3", "WT_Thymus_1", "WT_Thymus_2", "WT_Thymus_3"), condition= rep(c("Mbnl1_KO", "WT"),c(3,3)), libType= rep(c("paired-end")))
Observação : os nomes de linha devem ser consistentes com os nomes de arquivo bam usados por featureCounts para contar as leituras. sampleTable consiste em detalhes de cada amostra que inclui: tipo de biblioteca e condição. Isso é necessário para definir os contrastes ou o grupo de teste para detectar o uso diferencial. - Prepare o arquivo de informações do éxon. As informações do Exon na forma de objetos GRanges (intervalos genômicos) (https://bioconductor.org/packages/release/bioc/html/GenomicRanges.html) são necessárias como uma entrada para criar o objeto DEXSeq na próxima etapa. Combine o gene Ids com as contagens de leitura para criar o objeto exoninfo.
exoninfo<-anno[anno$GeneID%in% rownames(countData$counts),]
exoninfo<-GRanges(seqnames=anno$Chr,
ranges=IRanges(start=anno$Start, end=anno$End, width=anno$Width),strand=Rle(anno$Strand))
mcols(exoninfo)$TranscriptIDs<-anno$TranscriptIDs
mcols(exoninfo)$Ticker<-anno$Ticker
mcols(exoninfo)$ExonID<-anno$ExonID
mcols(exoninfo)$n<-anno$n
mcols(exoninfo)$GeneID<-anno$GeneID
transcripts_l= strsplit(exoninfo$TranscriptIDs, "\\,")
save(countData, sampleTable, exoninfo, transcripts_l, file="AS_countdata.RData") - Crie o objeto DEXSeq usando a função DEXSeqDataSet. O objeto DEXSeq coleta contagens de leitura, informações de recurso de éxon e informações de exemplo. Use as contagens de leitura geradas na etapa 3 e as informações de éxon obtidas na etapa anterior para criar o objeto DEXSeq a partir da matriz de contagem. O argumento sampleData usa uma entrada de quadro de dados definindo as amostras (e seus atributos: tipo e condição da biblioteca), 'design' usa sampleData para gerar uma matriz de design para o teste diferencial usando a notação de fórmula do modelo. Note que um termo de interação significativa, condição:éxon, indica que a fração de leituras sobre um gene que cai sobre um éxon particular depende da condição experimental, ou seja, há EA. Consulte a documentação do DEXSeq para obter uma descrição completa da configuração da fórmula do modelo para projetos experimentais mais complexos. Para informações sobre características, são necessários exons ids, genes correspondentes e transcritos.
dxd<-DEXSeqDataSet(countData$counts,sampleData=sampleTable, design=~sample+exon+condition:exon,featureID=exoninfo$ExonID,groupID=exoninfo$GeneID,featureRanges=exoninfo, transcripts=transcripts_l) - Normalização e estimativa de dispersão. Em seguida, realizar a normalização entre as amostras e estimar a variância dos dados, devido tanto ao ruído da contagem de Poisson da natureza discreta do RNA-seq quanto à variabilidade biológica, usando os seguintes comandos.
dxd %<>% estimateSizeFactors %>% estimateDispersions %T>% plotDispEsts - Teste para uso diferencial. Após a estimativa da variação, teste o uso diferencial de éxons para cada gene e gere os resultados.
dxd%<>%testForDEU%>%estimateExonFoldChanges(fitExpToVar=
"condition")#Estimate fold changes
dxr=DEXSeqResults(dxd) - Visualize eventos de splicing para genes selecionados usando o comando a seguir.
plotDEXSeq(dxr,"Wnk1", displayTranscripts=TRUE, splicing=TRUE,legend
=TRUE,cex.axis=1.2,cex=1.3,lwd=2)
Examine o arquivo R Notebook "AS_analysis_RNASeq.Rmd" para gerar gráficos adicionais para genes de interesse e para gerar gráficos de vulcões em diferentes limiares.
- Carregue a biblioteca e crie uma tabela de exemplo para definir o experimento.
- Usando diffSplice de Limma para identificar splicing diferencial. Siga o arquivo R Notebook "AS_analysis_RNASeq.Rmd". Verifique se as etapas 2.1-2.3 foram seguidas para preparar os arquivos de entrada antes de prosseguir.
- Carregar bibliotecas
library(limma)
library(edgeR) - Filtragem não específica. Extrair a matriz de contagens de leitura obtida no ponto 2.3. Crie uma lista de recursos usando a função 'DGEList' do pacote edgeR, onde as linhas representam genes e as colunas representam amostras.
mycounts=countData$counts
#Change the rownames of the countdata to exon Ids instead of genes for unique rownames.
rownames(mycounts) = exoninfo$ExonID
dge<-DGEList(counts=mycounts)
#Filtering
isexpr<- rowSums(cpm(dge)>1) >=3
dge<-dge[isexpr,,keep.lib.sizes=FALSE]
#Extract the exon annotations for only transcripts meeting non-specific filter
exoninfo=anno%>% filter(ExonID%in% rownames(dge$counts))
#Convert the exoninfo into GRanges object
exoninfo1<-GRanges(seqnames=exoninfo$Chr,
ranges=IRanges(start=exoninfo$Start, end=exoninfo$End, width=exoninfo$Width),strand=Rle(exoninfo$Strand))
mcols(exoninfo1)$TranscriptIDs<-exoninfo$TranscriptIDs
mcols(exoninfo1)$Ticker<-exoninfo$Ticker
mcols(exoninfo1)$ExonID<-exoninfo$ExonID
mcols(exoninfo1)$n<-exoninfo$n
mcols(exoninfo1)$GeneID<-exoninfo$GeneID
transcripts_l= strsplit(exoninfo1$TranscriptIDs, "\\,")
NOTA: Como uma etapa de filtragem não específica, as contagens são filtradas por cpm < 1 em x fora de n amostras, onde x é o número mínimo de replicações em qualquer condição. n = 6 e x = 3 para estes dados de exemplo. - Normalize as contagens entre amostras, com a função 'calcNormFactors' do pacote 'edgeR' usando a Média Aparada de valores M (método de normalização TMM)24 Ele calculará fatores de dimensionamento para ajustar os tamanhos das bibliotecas.
dge<-calcNormFactors(dge) - Use sampleTable conforme gerado na etapa 2.4.1.1 e crie a matriz de design. A matriz de design caracteriza o experimento. Consulte os capítulos 8 e 9 do Guia do Usuário do Limma (https://www.bioconductor.org/packages/devel/bioc/vignettes/limma/inst/doc/usersguide.pdf) para obter detalhes sobre matrizes de design para projetos experimentais mais avançados.
Treat<- factor(sampleTable$condition)
design<- model.matrix(~0+Treat)
colnames(design) <- levels(Treat) - Ajustar um modelo linear por éxon. Execute a função 'voom' do pacote 'limma' para processar dados de RNA-seq para estimar a variância e gerar pesos de precisão para corrigir o ruído de contagem de Poisson e transformar as contagens em nível de éxon em contagens log2 por milhão (logCPM). Em seguida, execute a modelagem linear usando a função 'lmfit' para ajustar modelos lineares aos dados de expressão para cada éxon. Calcule estatísticas empíricas de Bayes para o modelo ajustado usando a função 'eBayes' para detectar a expressão diferencial do éxon. Em seguida, defina uma matriz de contraste para as comparações experimentais de interesse. Use 'contrasts.fit' para obter coeficientes e erros-padrão para cada par de comparações.
v<-voom(dge,design,plot=FALSE)
fit<-lmFit(v,design)
fit<-eBayes(fit)
colnames(fit)
cont.matrix<-makeContrasts(
Mbnl1_KO_WT=Mbnl1_KO-WT,
levels=design)
fit2<-contrasts.fit(fit,cont.matrix) - Análise de splicing diferencial. Execute 'diffSplice' no modelo ajustado para testar as diferenças no uso de éxons de genes entre o tipo selvagem e o nocaute e explore os resultados mais bem classificados usando a função 'topSplice': test="t" dá uma classificação de AS exons, test="simes" dá uma classificação de genes.
ex<-diffSplice(fit2,geneid=exoninfo$GeneID,exonid=exoninfo$ExonID)
ts<-topSplice(ex,n=Inf,FDR=0.1, test="t", sort.by="logFC")
tg<-topSplice(ex,n=Inf,FDR=0.1, test="simes") - Visualização. Plote os resultados com a função 'plotSplice', dando o gene de interesse no argumento geneide. Salve os principais resultados classificados por log Fold mude em um objeto e gere um gráfico de vulcão para exibir os éxons.
plotSplice(ex,geneid="Wnk1", FDR=0.1)
#Volcano plot
EnhancedVolcano(ts,lab=ts$ExonID,selectLab= head((ts$ExonID),2000), xlab= bquote(~Log[2]~'fold change'), x='logFC', y='P.Value', title='Volcano Plot', subtitle='Mbnl1_KO vs WT (Limma_diffSplice)', FCcutoff=2, labSize=4,legendPosition="right", caption= bquote(~Log[2]~"Fold change cutoff, 2; FDR 10%"))
- Carregar bibliotecas
- Usando rMATS
- Certifique-se de que a versão mais recente do rMATS v4.1.1 (também conhecido como rMATS turbo devido ao tempo de processamento reduzido e aos menores requisitos de memória) esteja instalada usando conda ou github (https://github.com/Xinglab/rmats-turbo/releases/download/v4.1.1/rmats_turbo_v4_1_1.tar.gz) no diretório de trabalho. Siga a seção 4.3 em "AS_analysis_RNASeq.Rmd".
- Vá para a pasta que contém os arquivos bam obtidos após o mapeamento e prepare os arquivos de texto, conforme exigido pelo rMATS, para as duas condições, copiando o nome dos arquivos bam (juntamente com o caminho) separados por ','. Os seguintes comandos devem ser executados na linha de comando do Linux:
mkdir rMATS_analysis
cd bams/
ls -pd "$PWD"/*| grep "WT"| tr '\n'','> Wt.txt
ls -pd "$PWD"/*| grep "Mb"| tr '\n'','> KO.txt
mv *.txt ../rMATS_analysis - Execute rmats.py com os dois arquivos de entrada gerados na etapa anterior, juntamente com o arquivo GTF obtido em 2.1.1.3. Isso gerará uma pasta de saída 'rmats_out' contendo arquivos de texto descrevendo estatísticas (valores-p e níveis de inclusão) para cada evento de splicing separadamente.
python rmats-turbo/rmats.py --b1 KO.txt --b2 Wt.txt --gtf annotation.gtf -t paired --readLength 50 --nthread 8 --od rmats_out/ --tmp rmats_tmp --task pos
Observação : A anotação de referência na forma de um arquivo GTF também é necessária. Verifique os parâmetros se os dados são de extremidade única e altere a opção -t de acordo. - Explorando os resultados do rMATS. Use o pacote Bioconductor 'maser'25 para explorar os resultados do rMATS. Carregue os arquivos de texto JCEC (Junction and Exon Counts) no objeto 'maser' e filtre o resultado com base na cobertura incluindo pelo menos cinco leituras médias por evento de emenda.
library(maser)
mbnl1<-maser("/rmats_out/", c("WT","Mbnl1_KO"), ftype="JCEC")
#Filtering out events by coverage
mbnl1_filt<-filterByCoverage(mbnl1,avg_reads=5) - Visualizando os resultados do rMATS. Selecione os eventos de splicing significativos em FDR (False Discovery Rate) de 10% e variação mínima de 10% em Percent Spliced In (deltaPSI) usando a função 'topEvents' do pacote 'maser'. Em seguida, verifique os eventos genéticos para genes individuais de interesse (amostra gene-Wnk1) e plote os valores de PSI para cada evento de splicing desse gene. Gere um gráfico de vulcão especificando o tipo de evento.
#Top splicing events at 10% FDR
mbnl1_top<-topEvents(mbnl1_filt,fdr=0.1, deltaPSI=0.1)
mbnl1_top
#Check the gene events for a particular gene
mbnl1_wnk1<-geneEvents(mbnl1_filt,geneS="Wnk1", fdr=0.1, deltaPSI=0.1)
maser::display(mbnl1_wnk1,"SE")
plotGenePSI(mbnl1_wnk1,type="SE", show_replicates
=TRUE)
volcano(mbnl1_filt,fdr=0.1, deltaPSI=0.1,type="SE")
+xlab("deltaPSI")+ylab("Log10 Adj. Pvalue")+ggtitle("Volcano Plot of exon skipping events") - Gere gráficos Sashimi para o resultado de eventos de splicing obtidos com rMATS na forma de arquivos de texto usando o pacote 'rmats2shahimiplot'. Execute o script python na linha de comando do Linux.
python ./src/rmats2sashimiplot/rmats2sashimiplot.py --b1 ../bams/WT_Thymus_1.bam,../bams/WT_Thymus_2.bam,../bams/WT_Thymus_3.bam --b2 ../bams/Mbnl1KO_Thymus_1.bam,../bams/Mbnl1KO_Thymus_2.bam,../bams/Mbnl1KO_Thymus_3.bam -t SE -e ../rMATS_analysis/rmats_out/SE.MATS.JC.txt --l1 WT --l2 Mbnl1_KO --exon_s 1 --intron_s 5 -o ../rMATS_analysis/rmats2shasmi_output
Observação : esse processo pode ser demorado, pois ele irá gerar o gráfico Sashimi para todos os resultados no arquivo de eventos. Escolha os principais resultados (nomes de genes e éxons) conforme exibido pela função topEvents de 'maser' e visualize o gráfico Sashimi correspondente.
- Usando o pacote DEXSeq para análise diferencial de éxons.
3. Análise alternativa de poliadenilação (APA) usando sequenciamento final de 3'
- Download e pré-processamento de dados
Observação : consulte o arquivo suplementar do R Notebook "APA_analysis_3PSeq_notebook. Rmd" para os comandos completos para as etapas de download e pré-processamento de dados, ou execute o arquivo bash suplementar "APA_data_downloading_preprocessing.sh" na linha de comando do Linux.- Baixe os dados do SRA com os Ids de Adesão (1553129 para 1553136).
- Aparar adaptadores e complemento reverso para obter a sequência de sequência de sentido.
Observação : esta etapa é específica para o ensaio PolyA-seq usado. - O mapa lê a montagem do genoma do rato usando o alinhador de gravata borboleta26.
- Preparação de anotações de sites de pA.
Observação : O processamento do arquivo de anotação de site pA é executado em primeiro lugar usando o arquivo suplementar R Notebook "APA_analysis_3PSeq_notebook. Rmd" (2.1 - 2.6) e, em seguida, usando o arquivo bash "APA_annotation_preparation.sh".- Baixe a anotação de sites pA do banco de dados PolyASite 2.06.
- Selecione anotações de site pA para reter sites de pA de região 3'-não traduzida (UTR), que são anotados como Terminal Exon (TE) ou 1000 nt a jusante de um éxon terminal (DS) anotado para análise a jusante.
- Obter picos de pA do site. Ancorar em cada local de clivagem de pA e visualizar a cobertura média de leitura utilizando ferramentas de cama e ferramentas profundas27,28. Os resultados mostraram que os picos das leituras mapeadas foram dispersos principalmente dentro de ~60 pb a montante dos locais de clivagem (figura 5 e figura suplementar 5). Portanto, as coordenadas dos sites de pA foram estendidas do arquivo de anotação para 60 pb a montante de seus sites de clivagem. Dependendo do protocolo de sequenciamento final de 3' específico usado, essa etapa precisará ser otimizada para ensaios diferentes do PolyA-seq.
- Contando leituras
- Prepare o arquivo de anotação de sites pA.
anno<- read.table(file= "flanking60added.pA_annotation.bed",
stringsAsFactors=FALSE, check.names=FALSE, header=FALSE, sep="")
colnames(anno) <- c("chrom", "chromStart", "chromEnd", "name", "score", "strand", "rep", "annotation", "gene_name", "gene_id")
anno<- dplyr::select(anno,name,chrom, chromStart,chromEnd, strand,gene_id,gene_name,rep)
colnames(anno) <- c("GeneID", "Chr", "Start", "End", "Strand", "Ensembl", "Symbol", "repID") - Aplique 'featureCounts' para adquirir contagens brutas. Salve a tabela de contagem como o arquivo RData "APA_countData.Rdata" para análise APA usando diferentes ferramentas.
countData<- dir("bamfiles", pattern="sorted.bam$", full.names=TRUE) %>%
# Read all bam files as input for featureCounts
featureCounts(annot.ext=anno, isGTFAnnotationFile= FALSE,minMQS=0,useMetaFeatures= TRUE,allowMultiOverlap=TRUE, largestOverlap= TRUE,strandSpecific=1, countMultiMappingReads =TRUE,primaryOnly= TRUE,isPairedEnd= FALSE,nthreads=12)%T>%
save(file="APA_countData.Rdata")
NOTA: Esteja consciente de alterar qualquer um dos parâmetros listados na função 'featureCounts'. Modifique o parâmetro 'strandSpecific' para garantir que ele seja consistente com a direção de sequenciamento do ensaio de sequenciamento final de 3' usado (empiricamente, visualizar os dados em um navegador do genoma sobre genes em cadeias de mais e menos esclarecerá isso). - Aplique filtragem não específica de countData. A filtragem pode melhorar significativamente a robustez estatística em testes diferenciais de uso do site de pA. Primeiro, removemos esses genes com apenas um sítio pA, no qual diferencialmente o uso do sítio pA não pode ser definido. Em segundo lugar, aplicamos filtragem não específica com base na cobertura: as contagens são filtradas por cpm menor que 1 em x de n amostras, onde x é o número mínimo de replicações em qualquer condição. N = 8 e x = 2 para estes dados de exemplo.
load(file= "APA_countData.Rdata")# Skip this step if already loaded
# Non-specific filtering: Remove the pA sites not differentially expressed in the samples
countData<-countData$counts%>%as.data.frame%>% .[rowSums(edgeR::cpm(.)>1) >=2, ]
anno%<>% .[.$GeneID%in% rownames(countData), ]
# Remove genes with only 1 site and NA in geneIDs
dnsites<-anno%>%group_by(Symbol)%>%summarise(nsites=n())%>% filter(nsites>1&!is.na(Symbol))
anno<-anno%>% filter(Symbol%in%dnsites$Symbol)
countData<-countData[rownames(countData) %in%anno$GeneID, ]
- Prepare o arquivo de anotação de sites pA.
- Análise diferencial de uso do local de poliadenilação usando pipelines DEXSeq e diffSplice.
- Usando o pacote DEXSeq
NOTA: Como uma matriz de contraste não pode ser definida para o pipeline DEXSeq, a análise diferencial do APA de cada duas condições experimentais deve ser realizada separadamente. A análise diferencial da APA da condição WT e da condição DKO é realizada como exemplo para explicar o procedimento. Consulte o arquivo suplementar "APA_analysis_3PSeq_notebook. Rmd" para o fluxo de trabalho passo-a-passo desta seção e a análise diferencial da APA de outros contrastes.- Carregue a biblioteca e crie uma tabela de exemplo para definir o experimento.
c("DEXSeq", "GenomicRanges") %>% lapply(library, character.only=TRUE) %>%invisible
sampleTable1<- data.frame(row.names= c("WT_1","WT_2","DKO_1","DKO_2"),
condition= c(rep("WT", 2), rep("DKO", 2)),
libType= rep("single-end", 4)) - Prepare o arquivo de informações de sites pA usando o pacote Bioconductor GRanges.
# Prepare the GRanges object for DEXSeqDataSet object construction
PASinfo <- GRanges(seqnames = anno$Chr,
ranges = IRanges(start = anno$Start, end = anno$End),strand = Rle(anno$Strand))
mcols(PASinfo)$PASID<-anno$repID
mcols(PASinfo)$GeneEns<-anno$Ensembl
mcols(PASinfo)$GeneID<-anno$Symbol
# Prepare the new feature IDs, replace the strand information with letters to match the current pA site clusterID
new.featureID <- anno$Strand %>% as.character %>% replace(. %in% "+", "F") %>% replace(. %in% "-", "R") %>% paste0(as.character(anno$repID), .) - Use as contagens de leitura geradas na etapa 3.3 e as informações do site pA obtidas na etapa anterior para criar o objeto DEXSeq.
# Select the read counts of the condition WT and DKO
countData1<- dplyr::select(countData, SRR1553129.sorted.bam, SRR1553130.sorted.bam, SRR1553131.sorted.bam, SRR1553132.sorted.bam)
# Rename the columns of countData using sample names in sampleTable
colnames(countData1) <- rownames(sampleTable1)
dxd1<-DEXSeqDataSet(countData=countData1,
sampleData=sampleTable1,
design=~sample+exon+condition:exon,
featureID=new.featureID,
groupID=anno$Symbol,
featureRanges=PASinfo) - Defina o par de contraste definindo os níveis de condições no objeto DEXSeq.
dxd1$condition<- factor(dxd1$condition, levels= c("WT", "DKO"))
# The contrast pair will be "DKO - WT" - Normalização e estimativa de dispersão. Semelhante aos dados de RNA-seq, para 3' os dados de sequenciamento final realizam a normalização entre amostras (mediana colunal de razões para cada amostra) usando a função 'estimateSizeFactors' e estimam a variação dos dados usando com a função 'estimateDispersions', em seguida, visualizam o resultado da estimativa de dispersão usando a função 'plotDispEsts'.
dxd1 %<>% estimateSizeFactors %>% estimateDispersions %T>% plotDispEsts - Teste diferencial de uso do local de pA para cada gene usando a função 'testForDEU' e, em seguida, estime a mudança de dobra do uso do site de pA usando a função 'estimateExonFoldChanges'. Verifique os resultados usando a função 'DEXSeqResults' e defina 'FDR < 10%' como critério para locais de pA significativamente diferenciais.
dxd1 %<>% testForDEU %>% estimateExonFoldChanges(fitExpToVar = "condition")
dxr1 <- DEXSeqResults(dxd1)
dxr1
mcols(dxr1)$description
table(dxr1$padj<0.1) # Check the number of differential pA sites (FDR < 0.1)
table(tapply(dxr1$padj<0.1, dxr1$groupID, any)) # Check the number of gene overlapped with differential pA site - Visualização dos resultados diferenciais de uso do local de pA usando gráficos APA diferenciais gerados pela função 'plotDEXSeq' e gráfico de vulcão pela função 'EnhancedVolcano'.
# Select the top 100 significant differential pA sites ranked by FDR
topdiff.PAS<- dxr1%>%as.data.frame%>%rownames_to_column%>%arrange(padj)%$%groupID[1:100]
# Apply plotDEXSeq for the visualization of differential polyA usage
plotDEXSeq(dxr1,"S100a7a", legend=TRUE, expression=FALSE,splicing=TRUE, cex.axis=1.2, cex=1.3,lwd=2)
# Apply perGeneQValue to check the top genes with differential polyA site usage
dxr1%<>% .[!is.na(.$padj), ]
dgene<- data.frame(perGeneQValue= perGeneQValue (dxr1)) %>%rownames_to_column("groupID")
dePAS_sig1<-dxr1%>% data.frame() %>%
dplyr::select(-matches("dispersion|stat|countData|genomicData"))%>%
inner_join(dgene)%>%arrange(perGeneQValue)%>%distinct()%>%
filter(padj<0.1)
# Apply EnhancedVolcano package to visualise differential polyA site usage
"EnhancedVolcano"%>% lapply(library, character.only=TRUE) %>%invisible
EnhancedVolcano(dePAS_sig1, lab=dePAS_sig1$groupID, x='log2fold_DKO_WT',
y='pvalue',title='Volcano Plot',subtitle='DKO vs WT',
FCcutoff=1,labSize=4, legendPosition="right",
caption= bquote(~Log[2]~"Fold change cutoff, 1; FDR 10%"))
- Carregue a biblioteca e crie uma tabela de exemplo para definir o experimento.
- Usando o pacote diffSplice. Consulte o arquivo suplementar do R Notebook "APA_analysis_3PSeq_notebook. Rmd" para o fluxo de trabalho passo a passo desta seção.
- Defina contrastes de interesse para análise diferencial de uso de pA.
NOTA: Esta etapa deve ser executada após a construção e processamento do objeto DGEList, que está incluído no arquivo R Notebook "APA_analysis_3PSeq_notebook. Rmd".contrast.matrix<-makeContrasts(DKO_vs_WT=DKO-
WT,Ctrl_vs_DKO=Ctrl-DKO,
KD_vs_Ctrl=KD-Ctrl,KD_vs_DKO=KD-DKO,levels=design)
fit2<-fit%>%contrasts.fit(contrast.matrix)%>%eBayes
summary(decideTests(fit2))
ex<-diffSplice(fit2,geneid=anno$Symbol,exonid=new.featureID)
topSplice(ex) #Check the top significant results with topSplice - Visualize o resultado de contrastes de interesse (aqui "DKO - WT") usando gráficos APA diferenciais pela função 'plotSplice' e gráficos de vulcão com a função 'EnhancedVolcano'. Consulte o arquivo R Notebook "APA_analysis_3PSeq_notebook. Rmd" 4.2.7 - 4.2.9 para a visualização de outros pares de contraste.
sig1<-topSplice(ex,n=Inf,FDR=0.1,coef=1, test="t", sort.by="logFC")
sig1.genes<-topSplice(ex,n=Inf,FDR=0.1,coef=1, test="simes")
plotSplice(ex, coef=1,geneid="S100a7a", FDR = 0.1)
plotSplice(ex,coef=1,geneid="Tpm1", FDR = 0.1)
plotSplice(ex,coef=1,geneid="Smc6", FDR = 0.1)
EnhancedVolcano(sig1, lab=sig1$GeneID,xlab= bquote(~Log[2]~'fold change'),
x='logFC', y='P.Value', title='Volcano Plot', subtitle='DKO vs WT',
FCcutoff=1, labSize=6, legendPosition="right")
- Defina contrastes de interesse para análise diferencial de uso de pA.
- Usando o pacote DEXSeq
Resultados
Depois de executar o fluxo de trabalho passo a passo acima, as saídas de análise AS e APA e os resultados representativos estão na forma de tabelas e gráficos de dados, gerados da seguinte maneira.
COMO:
O principal resultado da análise AS (Tabela Suplementar 1 para diffSplice; Tabela 2 para DEXSeq) é uma lista de éxons mostrando o uso diferencial entre as condições, e uma lista de genes mostrando atividade de splicing global significativa de um ou mais de seus éxons constituintes, classificados por significância estatística. A Tabela Suplementar 1, aba 2, mostra éxons significativos, com colunas mostrando FC diferencial de éxon versus repouso, valor de p não ajustado por éxon e valor de p ajustado (correção de Benjamini-Hockberg). O limiar nos valores de p ajustados dará um conjunto de éxons com FDR definido. A Tabela Suplementar 1, guia 3 mostra uma lista classificada de genes mostrando significância da atividade geral de splicing, com uma coluna mostrando o valor de p ajustado ao nível do gene calculado usando o método de Simes. Dados semelhantes são mostrados na Tabela 2 para DEXSeq. A Figura 1 Suplementar e a Figura Suplementar 2 mostram o padrão de splicing diferencial nos genes Mbnl1, Tcf7 e Lef1 que foram validados experimentalmente no artigo publicado apresentado com os dados15. Os autores mostraram validação experimental de cinco genes - Mbnl1, Mbnl2, Lef1, Tcf7 e Ncor2. Nossa abordagem detectou splicing diferencial em todos esses genes. Aqui apresentamos os níveis de FDR para cada gene usando DEXSeq, diffSplice e rMATS respectivamente como obtidos nas Tabelas Suplementares 1-3: Mbnl1 (0, 6.6E-61 ,0), Mbnl2 (0,0.18,0), Lef1 (1.4E-10, 1.3E-04, 0), Tcf7 (0, 1.1E-6, 0) e Ncor2 (9.2E-11, 1.6E-06, 0).
A Figura 2 mostra uma comparação entre as saídas obtidas de três ferramentas diferentes e ilustra padrões alternativos de splicing no gene Wnk1. Os gráficos vulcânicos são mostrados na Figura 2A (diffSplice) e na Figura 2B (DEXSeq). Outros três genes altamente classificados são mostrados na Figura Suplementar 1 (diffSplice) e na Figura Suplementar 2 (DEXSeq). O eixo y mostra significância estatística (-log10 P-valores) e o eixo x mostra o tamanho do efeito (mudança de dobra). Genes localizados nos quadrantes superior esquerdo ou direito indicam FC substancial e fortes evidências estatísticas de diferenças genuínas.

Figura 2. Comparação dos resultados de splicing alternativo obtidos de diffSplice, DEXSeq e rMATS. |
(A) Gráfico vulcânico (à esquerda) de RNA-Seq da análise de Limma diffSplice: O eixo x mostra a mudança da dobra do éxon logarísta; o eixo y mostra -log10 p-valor. Cada ponto corresponde a um éxon. Linha tracejada horizontal em p-valor=1E-5; linhas tracejadas verticais em duas vezes de mudança (FC). Os éxons vermelhos mostram FC substancial e significância estatística. Gráfico de splicing diferencial (à direita): Padrões de splicing são exibidos para um exemplo de gene Wnk1 onde o eixo x mostra o id do éxon por transcrito; o eixo y mostra a mudança de dobra de log relativa do éxon (a diferença entre o logFC do éxon e o logFC geral para todos os outros éxons). Os éxons destacados em vermelho mostram expressão diferencial estatisticamente significativa (FDR < 0,1).
(B) Gráfico vulcânico (esquerda) e Uso diferencial de éxons (direita) de RNA-Seq obtidos a partir da análise DEXSeq. O gene Wnk1 mostra um uso diferencial significativo de éxons entre WT e Mbnl1 nocaute destacado em rosa, que correspondem aos mesmos éxons diferenciais em (A).
(C) Gráfico de vulcões (esquerda) e gráfico de Sashimi (à direita) para Wnk1 obtidos a partir da análise rMATS. Gráfico de vulcão representando o evento significativo de éxon (SE) ignorado () no tipo selvagem em comparação com o knockout a 10% FDR com mudança na porcentagem emendada em valores (PSI ou ΔΨ) > 0,1. O eixo x mostra a alteração nos valores de PSI entre as condições e o eixo y mostra o valor P do log. O gráfico de Sashimi mostra um evento exônico ignorado no gene Wnk1 , correspondendo a um éxon diferencial significativo em (A) e (B). Cada linha representa uma amostra de RNA-Seq: três réplicas de knock-out do tipo selvagem e Mbnl1. A altura mostra a cobertura de leitura em RPKM e os arcos de conexão retratam leituras de junção através de éxons. Isoformas alternativas do modelo de gene anotado são mostradas na parte inferior do gráfico. O painel inferior de C ilustra as leituras de junção usadas para calcular a estatística PSI.
(D) Diagrama de Venn comparando o número de éxons diferenciais significativos obtidos pelos diferentes métodos. Por favor, clique aqui para ver uma versão maior desta figura.
Figura 2 A (painel direito) mostra uma exibição diagramática das diferenças de éxons de um dos genes mais bem classificados, mostrando logFC no eixo y e número de éxons no eixo x. Este exemplo mostra um éxon de variando entre as condições para o gene Wnk1. O gráfico de uso diferencial de éxons do DEXSeq mostra evidências de splicing diferencial em cinco locais de éxons próximos a Wnk1.6.45. Os éxons destacados em rosa provavelmente serão emendados em amostras de Mbnl1 KO em comparação com o WT. Esses éxons são complementares aos resultados obtidos pelo diffSplice que mostra um padrão semelhante na posição genômica específica. Mais exemplos são mostrados na Figura Suplementar 1 e na Figura Suplementar 2. Uma visão mais detalhada para confirmar resultados interessantes pode ser dada comparando faixas de cobertura (oscilação) em unidades RPM (Leituras por milhão) nos navegadores do genoma UCSC (Universidade de Santa Cruz) ou IGV (Integrative Genomics Viewer) (não mostrado), juntamente com a correlação visual com outras faixas de interesse, como modelos de genes conhecidos, conservação e outros ensaios genômicos amplos.
A tabela de saída rMATS lista eventos de splicing alternativos significativos categorizados por tipo (Tabela Suplementar 3). A Figura 2C mostra um gráfico vulcânico de genes que são emendados alternativos, com o tamanho do efeito medido pela estatística diferencial "porcentagem emendada" (PSI ou ΔΨ) de11.
PSI refere-se à porcentagem de leituras consistentes com a inclusão de um éxon de (ou seja, leituras mapeadas para o próprio éxon de ou leituras de junção sobrepostas ao éxon) em comparação com leituras consistentes com exclusão de éxons, ou seja, leituras de junção através de éxons adjacentes a montante e a jusante (O painel inferior da Figura 2C). O painel direito da Figura 2C mostra o gráfico de sashimi do gene Wnk1 com evento de splicing diferencial sobreposto em faixas de cobertura para o gene, com um éxon ignorado em Mbnl1 KO. Arcos que unem éxons mostram o número de leituras de junção (leituras cruzando um intrão emendado). Diferentes guias da Tabela Suplementar 3 mostram leituras significativas de cada tipo de evento que abrange junções com limites de éxons (contagens de junções e contagens de éxons (JCEC)). A Figura 2D compara os éxons diferencialmente emendados significativos detectados pelas três ferramentas.

Figura 3. Eventos de splicing alternativos adquiridos pela análise rMATS. A) Tipos de eventos AS. Esta figura é adaptada da documentação rMATS11 explicando o evento de splicing com éxons constitutivos e alternativamente emendados. Rotulado com o número de cada evento em FDR 10%. B) Gráfico de Sashimi do gene Add3 exibindo éxon ignorado (SE). C) Gráfico de Sashimi do gene Baz2b exibindo sítio de emenda alternativo de 5' (A5SS). D) Gráfico de Sashimi do gene Lsm14b exibindo sítio de emenda alternativo de 3' (A3SS). E) Gráfico de Sashimi do gene Mta1 exibindo éxons mutuamente exclusivos (MXE). F) Gráfico de Sashimi do gene Arpp21 exibindo íntron retido (IR). As linhas vermelhas representam três réplicas de linhas do tipo selvagem e as linhas laranjas representam as réplicas de nocaute Mbnl1. O eixo x corresponde às coordenadas genômicas e às informações da fita, o eixo y mostra a cobertura em RPKM. Por favor, clique aqui para ver uma versão maior desta figura.
A Figura 3 ilustra os tipos de eventos de splicing SE, A5SS, A3SS, MXE e RI com a ajuda de gráficos Sashimi dos principais genes significativos desses eventos. Ao comparar as três repetições de WT e Mbnl1_KO, um total de 1272 eventos SE, 130 A5SS, 116 A3SS, 215 MXE e 313 eventos IR foram detectados em FDR 10%. O gráfico de Sashimi ilustra o tipo de evento usando os principais genes como exemplo.
APA:
A saída da análise APA é semelhante à análise AS em nível exon. Uma tabela de genes superiores classificados por atividade diferencial da APA na 3'UTR é fornecida (Tabela Suplementar 4 e Tabela Suplementar 5). A Figura 4A mostra os gráficos vulcânicos de genes por atividade diferencial de APA em 3'UTRs gerados usando diffSplice e DEXSeq separadamente. A Figura 4B exibe o gráfico de Venn comparando os resultados de uso do site de pA significativamente diferenciais adquiridos de diferentes pipelines. As Figuras 4C e 4D mostram a representação diagramática do uso diferencial do sítio pA na 3'UTR dos genes Fosl2 (Figura 4C) e Papola (Figura 4D) gerados usando diffSplice e DEXSeq, que são validados experimentalmente para mostrar deslocamento distal para proximal significativo (Fosl2) e deslocamento proximal para distal significativo (Papola) do uso do sítio pA na DKO, respectivamente. Mais exemplos são mostrados na Figura Suplementar 3 e na Figura Suplementar 4.

Figura 4. Gráficos alternativos de poliadenilação por diffSplice e DEXSeq. A) Gráficos vulcânicos de dados PolyA-seq gerados usando diffSplice e DEXSeq. O eixo X mostra a mudança de dobra do local do log pA; O eixo y mostra o valor de p -log10 . Cada ponto corresponde a um site de pA. Linha tracejada horizontal em p-valor=1E-5; linhas tracejadas verticais em FC de 2 vezes. Os éxons vermelhos mostram FC substancial e significância estatística. B) Gráfico de Venn comparando os resultados diferenciais significativos de uso do local de pA adquiridos de diferentes tubulações. C-D) Gráficos diferenciais de APA gerados usando diffSplice e DEXSeq mostrando os sítios pA proximal, interno e distal para os genes Fosl2 e Papola . As figuras são geradas pela mesma função que a Figura 2 (B), mas com sítios pA substituindo éxons. Por favor, clique aqui para ver uma versão maior desta figura.
A Figura 5 é um gráfico de diagnóstico para confirmar a distribuição de leitura esperada em torno de locais de clivagem de pA anotados para o ensaio PolyA-seq usado. Ele mostra a cobertura média em regiões de flanco ancoradas em locais de clivagem de pA conhecidos no nível de todo o genoma. Nesse caso, o acúmulo esperado de leituras a montante dos sites é visualizado. As distribuições de leitura ancoradas em locais de pA para todas as amostras de PolyA-seq são mostradas na Figura 5 Suplementar.

Figura 5. Gráfico de cobertura média em torno dos locais de clivagem de pA. O local de clivagem para dados PolyA-seq é mostrado por linha tracejada vertical. O eixo X mostra a posição da base em relação aos locais de clivagem de pA, até 100 nucleotídeos a montante e a jusante; O eixo y mostra a cobertura média de leitura em todos os locais de clivagem de pA, normalizada pelo tamanho da biblioteca no CPM. Por favor, clique aqui para ver uma versão maior desta figura.
Os resultados diferenciais da APA de diferentes contrastes gerados pelo mesmo pipeline podem ser comparados e verificados visualizando a cobertura de leitura de locais de pA significativamente diferenciais representativos no navegador do genoma. A Figura 6A é o gráfico de Venn comparando o uso significativamente diferencial do sítio pA de diferentes contrastes adquiridos do diffSplice. A Figura 6B-D são os instantâneos IGV da cobertura de leitura em locais de pA para diferentes genes, que mostram os padrões consistentes com os descobertos na análise APA usando diffSplice. A Figura 6B valida o deslocamento proximal para distal significativo do uso do sítio pA para o gene Paip2, que é detectado exclusivamente no contraste DKO vs WT, mas não em outros dois contrastes KD vs WT, e Ctr vs WT. A Figura 6C valida o deslocamento distal para proximal significativo do uso do sítio pA para o gene Ccl2 detectado exclusivamente no contraste KD vs WT, enquanto a Figura 6D valida o uso diferencial significativo de pA de todos os contrastes para o gene Cacna2d1.
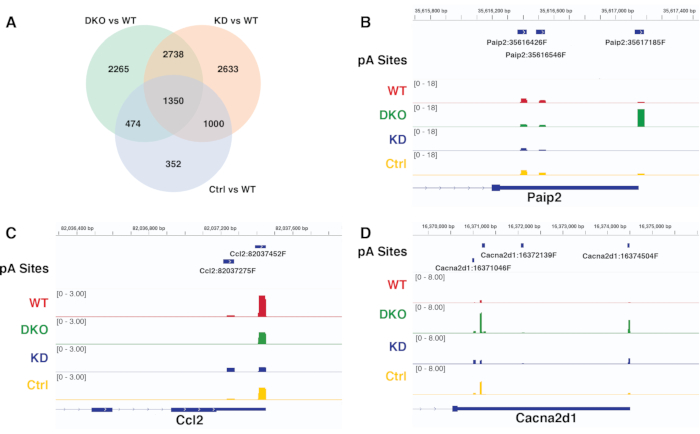
Figura 6. Comparação de contraste e verificação dos resultados do diffSplice. A) Diagrama de Venn comparando resultados diferenciais significativos de uso do sítio pA de diferentes contrastes adquiridos a partir de diffSplice. B-D) Instantâneo de IGV visualizando a cobertura de picos de pA dos genes Paip2, Ccl2 e Cacna2d1 em todas as condições. Cada faixa representa a cobertura de leitura em uma condição específica. Por favor, clique aqui para ver uma versão maior desta figura.
Figura 1 suplementar. Análise de RNA-Seq do splicing diferencial com Limma diffSplice. (A) Gráfico vulcânico de RNA-Seq da análise de Limma diffSplice: O eixo x mostra a mudança da dobra do éxon logarísta; o eixo y mostra -log10 p-valor. Cada ponto corresponde a um éxon. Linha tracejada horizontal em p-valor=1E-5; linhas tracejadas verticais em dupla mudança (FC). Os éxons vermelhos mostram FC substancial e significância estatística. (B-D) Gráficos de splicing diferenciais: Padrões de splicing são exibidos, por exemplo, genes Mbnl1, Tcf7 e Lef1, respectivamente, onde o eixo x mostra o id do éxon por transcrito; o eixo y mostra a mudança de dobra de log relativa do éxon (a diferença entre o logFC do éxon e o logFC geral para todos os outros éxons). Os éxons destacados em vermelho mostram expressão diferencial estatisticamente significativa (FDR < 0,1). Clique aqui para baixar este arquivo.
Figura 2 suplementar. Análise de RNA-Seq do uso diferencial de éxons com DEXSeq. (A) Lote do vulcão. (B-D) Uso diferencial do éxon de RNA-Seq obtido a partir da análise DEXSeq. Os genes Mbnl1, Tcf7 e Lef1, respectivamente, mostram uso diferencial significativo de éxons entre WT e Mbnl1 destacados em rosa, que correspondem aos mesmos éxons diferenciais na figura suplementar 1. Clique aqui para baixar este arquivo.
Figura 3 suplementar. Parcelas alternativas de poliadenilação por diffSplice. A) Gráficos vulcânicos de dados PolyA-seq gerados usando diffSplice em três pares de contraste obtidos a partir dos dados PolyA-seq do mouse, incluindo knockout duplo (DKO) vs wild-type (WT), knock-down (KD) vs WT e controle (Ctrl) vs WT. O eixo X mostra a mudança de dobra do site log pA; O eixo y mostra o valor de p -log10. Cada ponto corresponde a um site de pA. Linha tracejada horizontal em p-valor=1E-5; linhas tracejadas verticais em FC de 2 vezes. Os locais vermelhos de AP apresentam FC substancial e significância estatística. B) Gráficos diferenciais de APA gerados usando diffSplice mostrando os sítios pA proximal, interno e distal para os genes altamente classificados S100a7a, Tpm1 e Smc6. Clique aqui para baixar este arquivo.
Figura 4 suplementar. Análise diferencial de uso de pA por pipeline DEXSeq. A) Gráficos vulcânicos de dados PolyA-seq gerados usando DEXSeq em três pares obtidos a partir dos dados PolyA-seq do rato, incluindo knockout duplo (DKO) vs wild-type (WT), knock-down (KD) vs WT e controle (Ctrl) vs WT. X-axis mostra log pA site fold change; O eixo y mostra o valor de p -log10. Cada ponto corresponde a um site de pA. Linha tracejada horizontal em p-valor=1E-5; linhas tracejadas verticais em FC de 2 vezes. Os locais vermelhos de AP apresentam FC substancial e significância estatística. B) Gráficos diferenciais de APA gerados usando DEXSeq mostrando os sítios pA proximal, interno e distal para os genes altamente classificados S100a7a, Tpm1 e Smc6. Clique aqui para baixar este arquivo.
Figura 5 suplementar. Gráfico de cobertura média e mapas de calor em torno de locais de clivagem de pA. Cobertura mostrada para quatro condições, com genes em cadeias para frente e para trás mostrados separadamente. O eixo X mostra a posição da base em relação aos locais de clivagem de pA, até 100 nucleotídeos a montante e a jusante; O eixo y refere-se à cobertura média nas posições de base relativas correspondentes em todos os locais de clivagem de pA. Os mapas de calor fornecem uma visão alternativa, com cada local de clivagem pA mostrado como uma linha separada no eixo x, ordenada por cobertura. A intensidade mostra a cobertura de leitura (ver legenda). Clique aqui para baixar este arquivo.
Tabela complementar 1. saída diffSplice da análise AS. A primeira guia define os nomes das colunas para as saídas originais apresentadas na segunda (nível exon) e terceira (nível de gene) guias. Por favor, clique aqui para baixar esta Tabela.
Tabela complementar 2. Saída DEXSeq da análise AS. A primeira guia define os nomes das colunas para a saída original apresentada na segunda (nível exon) e terceira (nível de gene) guias. Por favor, clique aqui para baixar esta Tabela.
Tabela complementar 3. rMATS saída da análise AS. A primeira guia define os nomes das colunas para o arquivo de resumo (guia 2) e os arquivos JCEC para cada evento (guia 3-7). Por favor, clique aqui para baixar esta Tabela.
Quadro complementar 4. saída diffSplice da análise APA. A primeira guia define os nomes das colunas para a saída original apresentada na segunda (nível de site pA) e terceira (nível de gene) guias. Por favor, clique aqui para baixar esta Tabela.
Quadro complementar 5. Saída DEXSeq da análise APA. A primeira guia define os nomes das colunas para a saída original apresentada na segunda (nível de site pA) e terceira (nível de gene) guias. Por favor, clique aqui para baixar esta Tabela.
Quadro complementar 6. Um resumo do número de éxons significativamente alterados para sites AS e pA para APA. Por favor, clique aqui para baixar esta Tabela.
Tabela complementar 7. Um resumo das ferramentas e pacotes utilizados na análise AS/APA. Por favor, clique aqui para baixar esta Tabela.
Discussão
Neste estudo, avaliamos abordagens baseadas em éxons e eventos para detectar EA e APA em dados de RNA-Seq e sequenciamento final de 3'. As abordagens AS baseadas em éxons produzem uma lista de éxons diferencialmente expressos e uma classificação em nível de gene ordenada pela significância estatística da atividade de splicing diferencial geral em nível de gene (Tabelas 1-2, 4-5). Para o pacote diffSplice, o uso diferencial é determinado pelo ajuste de modelos lineares ponderados em um nível de éxon para estimar a mudança diferencial de log fold-change de um éxon contra a variação média de log fold-change dos outros éxons dentro do mesmo gene (chamado por exon FC). A significância estatística em nível de gene é calculada pela combinação de testes individuais de significância em nível de éxon em um teste genético pelo método de Simes10. Essa classificação por atividade de splicing diferencial em nível de gene pode ser posteriormente usada para realizar uma análise de enriquecimento de conjunto gênico (GSEA) das principais vias envolvidas10. O DEXSeq usa uma estratégia semelhante, ajustando um modelo linear generalizado para medir o uso diferencial de éxons, embora diferindo em certas etapas, como filtragem, normalização e estimativa de dispersão. Ao comparar os 500 principais éxons classificados mostrando atividade de EA e APA usando DEXSeq e DiffSplice, encontramos uma sobreposição de 310 éxons e 300 sítios de pA, respectivamente, demonstrando a concordância das duas abordagens baseadas em éxons, o que também foi demonstrado em um estudo anterior 29. Recomenda-se usar uma combinação de uma abordagem baseada em exons (DEXSeq ou diffSplice) e uma abordagem baseada em eventos para detecção e classificação abrangentes de EA. Para a APA, os usuários podem escolher DEXSeq ou diffSplice: ambos os métodos demonstraram ter um bom desempenho em uma ampla gama de experimentos transcriptômicos29.
Ao preparar a biblioteca de RNA-seq para uma análise EA, é importante usar um protocolo de RNA-seq em massa específico da fita8, pois uma grande fração de genes em genomas de vertebrados se sobrepõe em diferentes fitas, e um protocolo não específico de fita é incapaz de distinguir essas regiões sobrepostas, confundindo a detecção final de éxons. Outra consideração é a profundidade de leitura, com análises de splicing exigindo sequenciamento mais profundo do que o DGE, por exemplo, 30-60 milhões de leituras por amostra, contra 5-25 milhões de leituras por amostra para DGE (https://sapac.support.illumina.com/bulletins/2017/04/considerations-for-rna-seq-read-length-and-coverage-.html). Todas as ferramentas demonstradas no protocolo suportam dados de sequenciamento de extremidade única e emparelhada. Se apenas anotações de genes conhecidas forem usadas para detectar leituras de junção, leituras mais curtas de extremidade única (≥ 50 pb) podem ser usadas, embora a detecção de novo de novas junções de emenda se beneficie de leituras de extremidade emparelhada e mais longas (≥ 100bp)30,31. A escolha do protocolo de extração de RNA - seja a seleção poliA ou a depleção de rRNA - depende da qualidade do RNA e da questão experimental - veja31 para uma discussão. Dependendo dos detalhes da construção da biblioteca, serão necessárias modificações nos scripts de exemplo fornecidos aqui para os parâmetros de alinhamento de leitura, contagem de recursos e rMATS. Ao calcular as contagens iniciais de leitura no nível do éxon usando featureCounts, ou métodos semelhantes, deve-se tomar cuidado para configurar as opções de função corretamente para contagens e encalhe: em featureCounts, definimos o argumento "strandSpecific" apropriadamente para o protocolo RNA-seq específico da cadeia usado; e para quantificação em nível de exon, espera-se que uma leitura seja mapeada sobre éxons adjacentes e, portanto, definimos o parâmetro allowMultiOverlap como TRUE. Para a APA, existem diferentes protocolos de sequenciamento final de 3' 6 que variam na localização precisa dos picos em relação ao localde pA. Para nossos dados de exemplo, determinamos que o pico é de 60 pb a montante do local de pA, como mostrado pela Figura 5, e essa análise precisará ser adaptada para outros protocolos de sequenciamento final de 3'.
Neste protocolo, limitamos o escopo à discussão de análises diferenciais no nível de éxons individuais e eventos de splicing consistindo de combinações adjacentes de éxons-íntrons. Não discutimos a classe de análises baseadas na reconstrução de isoformas de novo, como Cufflinks, Cuffdiff32, RSEM 33, Kallisto34, que visam detectar e quantificar a expressão absoluta e relativa de isoformas alternativas inteiras. Os métodos exon e baseado em eventos são mais sensíveis para detectar eventos individuais de splicing30 e, em muitos casos, fornecem todas as informações necessárias para uma análise mais aprofundada, sem a necessidade de quantificação em nível de isoforma.
A versão mais recente dos arquivos de origem neste protocolo está disponível em https://github.com/jiayuwen/AS_APA_JoVE
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Este estudo foi apoiado por um Conselho Australiano de Pesquisa (ARC) Future Fellowship (FT16010043) e ANU Futures Scheme.
Materiais
| Name | Company | Catalog Number | Comments |
| Not relevent for computational study |
Referências
- Katz, Y., Wang, E. T., Airoldi, E. M., Burge, C. B. Analysis and design of RNA sequencing experiments for identifying isoform regulation. Nature Methods. 7 (12), 1009-1015 (2010).
- Wang, Y., et al. Mechanism of alternative splicing and its regulation. Biomedical Reports. 3 (2), 152-158 (2015).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in Bioinformatics. 21 (6), 2052-2065 (2020).
- Movassat, M., et al. Coupling between alternative polyadenylation and alternative splicing is limited to terminal introns. RNA Biology. 13 (7), 646-655 (2016).
- Tian, B., Manley, J. L. Alternative polyadenylation of mRNA precursors. Nature Reviews Molecular Cell Biology. 18 (1), 18-30 (2017).
- Herrmann, C. J., et al. PolyASite 2.0: a consolidated atlas of polyadenylation sites from 3' end sequencing. Nucleic Acids Research. 48 (1), 174-179 (2020).
- Liu, R., Loraine, A. E., Dickerson, J. A. Comparisons of computational methods for differential alternative splicing detection using RNA-seq in plant systems. BMC Bioinformatics. 15 (1), 364 (2014).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biology. 17 (1), 13 (2016).
- Anders, S., Reyes, A., Huber, W. Detecting differential usage of exons from RNA-seq data. Genome Research. 22 (10), 2008-2017 (2012).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2014).
- Shen, S., et al. rMATS: Robust and flexible detection of differential alternative splicing from replicate RNA-Seq data. Proceedings of the National Academy of Sciences. 111 (51), 5593-5601 (2014).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in bioinformatics. 21 (6), 2052-2065 (2020).
- Kanitz, A., et al. Comparative assessment of methods for the computational inference of transcript isoform abundance from RNA-seq data. Genome biology. 16 (1), 1-26 (2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550 (2014).
- Sznajder, L. J., et al. Loss of MBNL1 induces RNA misprocessing in the thymus and peripheral blood. Nature Communications. 11, 1-11 (2020).
- Batra, R., et al. Loss of MBNL leads to disruption of developmentally regulated alternative polyadenylation in RNA-mediated disease. Molecular Cell. 56 (2), 311-322 (2014).
- Leinonen, R., Sugawara, H., Shumway, M., et al. The sequence read archive. Nucleic acids research. 39, 19-21 (2010).
- Tange, O. . GNU parallel-the command-line power tool. 36, 42-47 (2011).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet journal. 17 (1), 10-12 (2011).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Robinson, M. D., Oshlack, A. A scaling normalization method for differential expression analysis of RNA-seq data. Genome Biology. 11 (3), 25 (2010).
- Veiga, D. F. T. maser: Mapping Alternative Splicing Events to pRoteins. R package version 1.4.0. , (2019).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (13), 25 (2009).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ramírez, F., Dündar, F., Diehl, S., Grüning, B. A., Manke, T. deepTools: a flexible platform for exploring deep-sequencing data. Nucleic acids research. 42 (1), 187-191 (2014).
- Merino, G. A., Conesa, A., Fernández, E. A. A benchmarking of workflows for detecting differential splicing and differential expression at isoform level in human RNA-seq studies. Briefings in bioinformatics. 20 (2), 471-481 (2019).
- Chhangawala, S., Rudy, G., Mason, C. E., Rosenfeld, J. A. The impact of read length on quantification of differentially expressed genes and splice junction detection. Genome biology. 16 (1), 1-10 (2015).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biol. 17, 13 (2016).
- Trapnell, C., et al. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nat Protoc. 7 (3), 562-578 (2012).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC Bioinformatics. 12, 323 (2011).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nat Biotechnol. 34 (5), 525-527 (2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados