Method Article
Identification d’un épissage alternatif et d’une polyadénylation dans les données de séquençage de l’ARN
Dans cet article
Résumé
L’épissage alternatif (AS) et la polyadénylation alternative (APA) élargissent la diversité des isoformes de transcrits et de leurs produits. Ici, nous décrivons des protocoles bioinformatiques pour analyser les tests de séquençage d’ARN en vrac et de séquençage à 3' pour détecter et visualiser l’AS et l’APA variant selon les conditions expérimentales.
Résumé
En plus de l’analyse typique du RNA-Seq pour mesurer l’expression génique différentielle (DGE) dans des conditions expérimentales / biologiques, les données RNA-seq peuvent également être utilisées pour explorer d’autres mécanismes de régulation complexes au niveau de l’exon. L’épissage alternatif et la polyadénylation jouent un rôle crucial dans la diversité fonctionnelle d’un gène en générant différentes isoformes pour réguler l’expression génique au niveau post-transcriptionnel, et limiter les analyses à l’ensemble du niveau du gène peut manquer cette couche régulatrice importante. Ici, nous démontrons des analyses détaillées étape par étape pour l’identification et la visualisation de l’utilisation différentielle des exons et des sites de polyadénylation dans toutes les conditions, en utilisant Bioconductor et d’autres packages et fonctions, y compris DEXSeq, diffSplice du package Limma et rMATS.
Introduction
Le séquençage de l’ARN a été largement utilisé au fil des ans, généralement pour estimer l’expression différentielle des gènes et la découverte de gènes1. En outre, il peut également être utilisé pour estimer l’utilisation variable du niveau d’exon en raison de l’expression de gènes différentes isoformes, contribuant ainsi à une meilleure compréhension de la régulation des gènes au niveau post-transcriptionnel. La majorité des gènes eucaryotes génèrent différentes isoformes par épissage alternatif (AS) pour augmenter la diversité de l’expression de l’ARNm. Les événements AS peuvent être divisés en différents modèles: saut d’exons complets (SE) où un exon (« cassette ») est complètement retiré de la transcription avec ses introns flanquants; sélection alternative (donneur) du site d’épissage 5' (A5SS) et alternative 3' (accepteur) sélection du site d’épissage (A3SS) lorsque deux sites d’épissage ou plus sont présents à chaque extrémité d’un exon; rétention d’introns (RI) lorsqu’un intron est retenu dans le transcrit d’ARNm mature et exclusion mutuelle de l’utilisation d’exons (MXE) où un seul des deux exons disponibles peut être retenu à la fois 2,3. La polyadénylation alternative (APA) joue également un rôle important dans la régulation de l’expression génique en utilisant des sites poly (A) alternatifs pour générer plusieurs isoformes d’ARNm à partir d’un seul transcrit4. La plupart des sites de polyadénylation (pA) sont situés dans la région 3' non traduite (3' UTR), générant des isoformes d’ARNm avec diverses longueurs 3' UTR. Comme l’UTR 3' est le centre central pour la reconnaissance des éléments régulateurs, différentes longueurs 3' UTR peuvent affecter la localisation, la stabilité et la traduction de l’ARNm5. Il existe une classe de tests de séquençage d’extrémité 3' optimisés pour détecter l’APA qui diffèrent dans les détails du protocole6. Le pipeline décrit ici est conçu pour PolyA-seq, mais peut être adapté à d’autres protocoles comme décrit.
Dans cette étude, nous présentons un pipeline de méthodes d’analyse différentielledes exons 7,8 (Figure 1), qui peuvent être divisées en deux grandes catégories : basées sur les exons (DEXSeq9, diffSplice10) et basées sur les événements (analyse multivariée répliquée de l’épissage des transcriptions (rMATS)11). Les méthodes basées sur les exons comparent le changement de pli entre les conditions des exons individuels, à une mesure du changement global du pli des gènes pour appeler l’utilisation d’exons exprimée différentiellement, et à partir de là, calculez une mesure au niveau du gène de l’activité SA. Les méthodes basées sur les événements utilisent des lectures de jonction couvrant exon-intron pour détecter et classer des événements d’épissage spécifiques tels que le saut d’exon ou la rétention d’introns, et distinguer ces types AS dans la sortie3. Ainsi, ces méthodes fournissent des points de vue complémentaires pour une analyse complète de la SA12,13. Nous avons sélectionné DEXSeq (basé sur le package DESeq214 DGE) et diffSplice (basé sur le package Limma10 DGE) pour l’étude car ils sont parmi les packages les plus largement utilisés pour l’analyse d’épissage différentiel. rMATS a été choisi comme méthode populaire pour l’analyse basée sur les événements. Une autre méthode populaire basée sur les événements est MISO (Mixture of Isoforms)1. Pour l’APA, nous adaptons l’approche basée sur les exons.
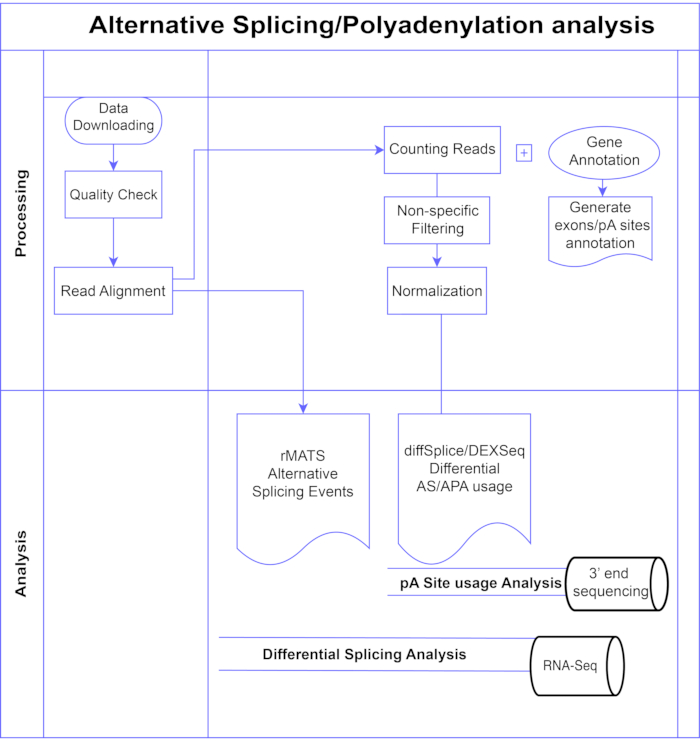
Graphique 1. Pipeline d’analyse. Organigramme des étapes utilisées dans l’analyse. Les étapes comprennent : l’obtention des données, l’exécution de contrôles de qualité et d’alignement des lectures, suivis du comptage des lectures à l’aide d’annotations pour les exons, les introns et les sites pA connus, le filtrage pour supprimer les faibles nombres et la normalisation. Les données PolyA-seq ont été analysées pour d’autres sites pA à l’aide des méthodes diffSplice/DEXSeq, le RNA-Seq en vrac a été analysé pour l’épissage alternatif au niveau de l’exon avec les méthodes diffSplice/DEXseq et les événements AS analysés avec rMATS. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Les données RNA-seq utilisées dans cette enquête ont été acquises à partir de Gene Expression Omnibus (GEO) (GSE138691)15. Nous avons utilisé les données de séquençage de l’ARN de souris de cette étude avec deux groupes de conditions : le type sauvage (WT) et le knockout de type 1 de type Muscleblind (Mbnl1 KO) avec trois réplications chacun. Pour démontrer l’analyse différentielle de l’utilisation du site de polyadénylation, nous avons obtenu des données PolyA-seq sur des fibroblastes embryonnaires de souris (MEF) (GEO Accession GSE60487)16. Les données comportent quatre groupes de conditions : Wild-type (WT), Muscleblind-like type1/type 2 double knockout (Mbnl1/2 DKO), Mbnl 1/2 DKO avec Mbnl3 knockdown (KD) et Mbnl1/2 DKO avec contrôle Mbnl3 (Ctrl). Chaque groupe de conditions se compose de deux répétitions.
| Adhésion GEO | Numéro d’exécution SRA | Nom de l’échantillon | Condition | Répliquer | Tissu | Séquençage | Longueur de lecture | |
| RNA-Seq | GSM4116218 | SRR10261601 | Mbnl1KO_Thymus_1 | Mbnl1 knockout | Rép. 1 | Thymus | Extrémité jumelée | 100 pb |
| GSM4116219 | SRR10261602 | Mbnl1KO_Thymus_2 | Mbnl1 knockout | Rép. 2 | Thymus | Extrémité jumelée | 100 pb | |
| GSM4116220 | SRR10261603 | Mbnl1KO_Thymus_3 | Mbnl1 knockout | Rép. 3 | Thymus | Extrémité jumelée | 100 pb | |
| GSM4116221 | SRR10261604 | WT_Thymus_1 | Type sauvage | Rép. 1 | Thymus | Extrémité jumelée | 100 pb | |
| GSM4116222 | SRR10261605 | WT_Thymus_2 | Type sauvage | Rép. 2 | Thymus | Extrémité jumelée | 100 pb | |
| GSM4116223 | SRR10261606 | WT_Thymus_3 | Type sauvage | Rép. 3 | Thymus | Extrémité jumelée | 100 pb | |
| 3P-Seq | GSM1480973 | SRR1553129 | WT_1 | Type sauvage (WT) | Rép. 1 | Fibroblastes embryonnaires de souris (MEF) | Extrémité unique | 40 pb |
| GSM1480974 | SRR1553130 | WT_2 | Type sauvage (WT) | Rép. 2 | Fibroblastes embryonnaires de souris (MEF) | Extrémité unique | 40 pb | |
| GSM1480975 | SRR1553131 | DKO_1 | Mbnl 1/2 double knockout (DKO) | Rép. 1 | Fibroblastes embryonnaires de souris (MEF) | Extrémité unique | 40 pb | |
| GSM1480976 | SRR1553132 | DKO_2 | Mbnl 1/2 double knockout (DKO) | Rép. 2 | Fibroblastes embryonnaires de souris (MEF) | Extrémité unique | 40 pb | |
| GSM1480977 | SRR1553133 | DKOsiRNA_1 | Mbnl 1/2 double knockout avec Mbnl 3 siRNA (KD) | Rép. 1 | Fibroblastes embryonnaires de souris (MEF) | Extrémité unique | 40 pb | |
| GSM1480978 | SRR1553134 | DKOsiRNA_2 | Mbnl 1/2 double knockout avec Mbnl 3 siRNA (KD) | Rép. 2 | Fibroblastes embryonnaires de souris (MEF) | Extrémité unique | 36 pb | |
| GSM1480979 | SRR1553135 | DKONTsiRNA_1 | Mbnl 1/2 double knockout avec siRNA non ciblé (Ctrl) | Rép. 1 | Fibroblastes embryonnaires de souris (MEF) | Extrémité unique | 40 pb | |
| GSM1480980 | SRR1553136 | DKONTsiRNA_2 | Mbnl 1/2 double knockout avec siRNA non ciblé (Ctrl) | Rép. 2 | Fibroblastes embryonnaires de souris (MEF) | Extrémité unique | 40 pb |
Tableau 1. Résumé des ensembles de données RNA-Seq et PolyA-seq utilisés pour l’analyse.
Protocole
1. Installation des outils et des packages R utilisés dans l’analyse
- Conda est un gestionnaire de paquets populaire et flexible qui permet une installation pratique des paquets avec leurs dépendances sur toutes les plates-formes. Utilisez 'Anaconda' (gestionnaire de paquets conda) pour installer 'conda' qui peut être utilisé pour installer les outils/paquets requis pour l’analyse.
- Téléchargez 'Anaconda' selon la configuration système requise de https://www.anaconda.com/products/individual#Downloads et installez-le en suivant les instructions de l’installateur graphique. Installez tous les paquets requis en utilisant 'conda' en tapant ce qui suit sur la ligne de commande Linux.
conda install -c daler sratoolkit
conda install -c conda-forge parallel
conda install -c bioconda star bowtie fastqc rmats rmats2sashimiplot samtools fasterq-dump cutadapt bedtools deeptools - Pour télécharger tous les paquets R utilisés dans le protocole, tapez le code suivant dans la console R (démarrée sur la ligne de commande Linux en tapant 'R') ou la console Rstudio.
bioc_packages<- c("DEXSeq", "Rsubread", "EnhancedVolcano", "edgeR", "limma", "maser","GenomicRanges")
packages<- c("magrittr", "rtracklayer", "tidyverse", "openxlsx", "BiocManager")
#Install if not already installed
installed_packages<-packages%in% rownames(installed.packages())
installed_bioc_packages<-bioc_packages%in% rownames(installed.packages())
if(any(installed_packages==FALSE)) {
install.packages(packages[!installed_packages],dependencies=TRUE)
BiocManager::install(packages[!installed_bioc_packages], dependencies=TRUE)
}
REMARQUE: Dans ce protocole de calcul, les commandes seront données sous forme de fichiers R Notebook (fichiers avec extension « . Rmd »), Fichiers de code R (fichiers avec extension « . R »), ou des scripts shell Linux Bash (fichiers avec l’extension « .sh »). Les fichiers R Notebook (Rmd) doivent être ouverts dans RStudio à l’aide de Fichier| Ouvrez le fichier..., et les morceaux de code individuels (qui peuvent être des commandes R ou des commandes shell Bash) s’exécutent ensuite de manière interactive en cliquant sur la flèche verte en haut à droite. Les fichiers de code R peuvent être exécutés en ouvrant dans RStudio, ou sur la ligne de commande Linux en préfaçant avec « Rscript », par exemple Rscript example.R. Les scripts Shell sont exécutés sur la ligne de commande Linux en préfaceant le script avec la commande « sh », par exemple.sh example.sh.
2. Analyse d’épissage alternatif (AS) à l’aide du séquençage de l’ARN
- Téléchargement et prétraitement des données
Remarque : Les extraits de code annotés ci-dessous sont disponibles dans le fichier de code supplémentaire »AS_analysis_RNASeq.Rmd« , pour suivre les étapes individuelles de manière interactive, et sont également fournis sous forme de script bash à exécuter par lots sur la ligne de commande Linux (sh downloading_data_preprocessing.sh).- Téléchargement des données brutes.
- Téléchargez les données brutes à partir de Sequence Read Archive (SRA) à l’aide de la commande 'prefetch' de la boîte à outils SRA (v2.10.8)17. Donnez les ID d’accession SRA dans l’ordre dans la commande suivante pour les télécharger en parallèle en utilisant l’utilitaire parallèleGNU 18. Pour télécharger des fichiers SRA d’ID d’accession de SRR10261601 à SRR10261606 en parallèle, utilisez la commande suivante sur la ligne de commande Linux.
seq 10261601 10261606 | parallel prefetch SRR{} - Extrayez les fichiers fastq de l’archive à l’aide de la fonction 'fastq-dump' de la boîte à outils SRA. Utilisez GNU en parallèle et donnez les noms de tous les fichiers SRA ensemble.
parallel -j 3 fastq-dump --gzip --skip-technical --read-filter pass --dumpbase --split-e --clip --origfmt {} ::: - Téléchargez le génome de référence et les annotations pour Mouse (assemblage du génome GRCm39) à partir de www.ensembl.org en utilisant ce qui suit sur la ligne de commande Linux.
wget -nv -O annotation.gtf.gz http://ftp.ensembl.org/pub/release-103/gtf/mus_musculus/Mus_musculus.GRCm39.103.gtf.gz \ && gunzip -f annotation.gtf.gz
wget -nv -O genome.fa.gz http://ftp.ensembl.org/pub/release-103/fasta/mus_musculus/dna/Mus_musculus.GRCm39.dna.primary_assembly.fa.gz \ && gunzip -f genome.fa.gz
GTF=$(readlink -f annotation.gtf)
GENOME=$(readlink -f genome.fa)
- Téléchargez les données brutes à partir de Sequence Read Archive (SRA) à l’aide de la commande 'prefetch' de la boîte à outils SRA (v2.10.8)17. Donnez les ID d’accession SRA dans l’ordre dans la commande suivante pour les télécharger en parallèle en utilisant l’utilitaire parallèleGNU 18. Pour télécharger des fichiers SRA d’ID d’accession de SRR10261601 à SRR10261606 en parallèle, utilisez la commande suivante sur la ligne de commande Linux.
- Prétraitement et mappage des lectures à l’assemblage du génome
- Contrôle qualité. Évaluez la qualité des lectures brutes avec FASTQC (FASTQ Quality Check v0.11.9)19. Créez un dossier de sortie et exécutez fastqc en parallèle sur plusieurs fichiers fasta d’entrée. Cette étape générera un rapport de qualité pour chaque échantillon. Examinez les rapports pour vous assurer que la qualité des lectures est acceptable avant de procéder à une analyse plus approfondie. (Reportez-vous au manuel de l’utilisateur pour comprendre les rapports à https://www.bioinformatics.babraham.ac.uk/projects/fastqc/)
mkdir fastqc_out
parallel "fastqc {} -o fastqc_out" ::: $RAW_DATA/*.fastq.gz
REMARQUE: Si nécessaire, effectuez le découpage de l’adaptateur avec 'cutadapt'20 ou 'trimmomatic'21 pour supprimer le séquençage dans les adaptateurs latéraux, qui varie en fonction de la taille du fragment d’ARN et de la longueur de lecture. Dans cette analyse, nous avons sauté cette étape car la fraction de lectures affectées était minime. - Alignement de lecture. L’étape suivante du prétraitement comprend la mise en correspondance des lectures avec le génome de référence. Tout d’abord, construisez l’index pour le génome de référence en utilisant la fonction 'genomeGenerate' de STAR22 , puis alignez les lectures brutes sur la référence (alternativement, des index prédéfinis sont disponibles sur le site Web de STAR et peuvent être utilisés directement pour l’alignement). Exécutez les commandes suivantes sur la ligne de commande Linux.
#Build STAR index
GDIR=STAR_indices
mkdir $GDIR
STAR --runMode genomeGenerate --genomeFastaFiles $GENOME --sjdbGTFfile $GTF --runThreadN 8 --genomeDir $GDIR
ODIR=results/mapping
mkdir -p $ODIR
#Align reads to the genome
for fq1 in $RAW_DATA/*R1.fastq.gz;
do
fq2=$(echo $fq1| sed 's/1.fastq.gz/2.fastq.gz/g');
OUTPUT=$(basename ${fq1}| sed 's/R1.fastq.gz//g');
STAR --genomeDir $GDIR \
--runThreadN 12 \
--readFilesCommand zcat \
--readFilesIn ${fq1}${fq2}\
--outFileNamePrefix $ODIR\/${OUTPUT} \
--outSAMtype BAM SortedByCoordinate \
--outSAMunmapped Within \
--outSAMattributes Standard
Done
Remarque : L’aligneur STAR génère et trie les fichiers BAM (Binary Alignment Map) pour chaque échantillon après l’alignement en lecture. Les fichiers Bam doivent être triés avant de passer aux étapes suivantes.
- Contrôle qualité. Évaluez la qualité des lectures brutes avec FASTQC (FASTQ Quality Check v0.11.9)19. Créez un dossier de sortie et exécutez fastqc en parallèle sur plusieurs fichiers fasta d’entrée. Cette étape générera un rapport de qualité pour chaque échantillon. Examinez les rapports pour vous assurer que la qualité des lectures est acceptable avant de procéder à une analyse plus approfondie. (Reportez-vous au manuel de l’utilisateur pour comprendre les rapports à https://www.bioinformatics.babraham.ac.uk/projects/fastqc/)
- Téléchargement des données brutes.
- Préparation des annotations Exon.
- Exécutez le fichier de code supplémentaire « prepare_mm_exon_annotation. R » avec l’annotation téléchargée au format GTF (Gene transfer format) pour préparer les annotations. Pour exécuter, tapez ce qui suit sur la ligne de commande Linux.
Rscript prepare_mm_exon_annotation.R annotation.gtf
Remarque : Le fichier GTF contient plusieurs entrées d’exon pour différentes isoformes. Ce fichier est utilisé pour « réduire » les multiples ID de transcription pour chaque exon. C’est une étape importante pour définir les bacs de comptage d’exons.
- Exécutez le fichier de code supplémentaire « prepare_mm_exon_annotation. R » avec l’annotation téléchargée au format GTF (Gene transfer format) pour préparer les annotations. Pour exécuter, tapez ce qui suit sur la ligne de commande Linux.
- Comptage lit. L’étape suivante consiste à compter le nombre de lectures mappées à différentes transcriptions / exons. Voir Fichier supplémentaire: « AS_analysis_RNASeq.Rmd ».
- Chargez les bibliothèques requises :
packages<- c("Rsubread","tidyverse", "magrittr", "EnhancedVolcano", "edgeR","openxlsx")
invisible(lapply(packages, library, character.only=TRUE)) - Chargez le fichier d’annotation traité obtenu à partir de l’étape précédente (2.2).
load("mm_exon_anno.RData") - Lisez tous les fichiers bam obtenus à l’étape 2.2.2 en entrée pour 'featureCounts' pour compter les lectures. Lisez le dossier contenant les fichiers bam en répertoriant d’abord chaque fichier du répertoire qui se termine par .bam. Utilisez 'featureCounts' du paquet Rsubread qui prend les fichiers bam et l’annotation GTF traitée (référence) comme entrée pour générer une matrice de comptages associés à chaque entité avec des lignes représentant des exons (entités) et des colonnes représentant des échantillons.
countData <- dir("bams", pattern=".bam$", full.names=T) %>%
featureCounts(annot.ext=anno,
isGTFAnnotationFile=FALSE,
minMQS=0,useMetaFeatures=FALSE,
allowMultiOverlap=TRUE,
largestOverlap=TRUE,
countMultiMappingReads=FALSE,
primaryOnly=TRUE,
isPairedEnd=TRUE,
nthreads=12) - Ensuite, effectuez un filtrage non spécifique pour éliminer les exons faiblement exprimés (« non spécifique » indique que l’information sur l’état expérimental n’est pas utilisée dans le filtrage, afin d’éviter les biais de sélection). Transformez les données de l’échelle brute en nombres par million (cpm) à l’aide de la fonction cpm du package 'edgeR'23 et conservez les exons avec des nombres supérieurs à un seuil définissable (pour cet ensemble de données, un cpm est utilisé) dans au moins trois échantillons. Supprimez également les gènes avec un seul exon.
# Non-specific filtering: Remove the exons with low counts
isexpr<- rownames(countData$counts)[rowSums(cpm(countData$counts)>1) >=3]
countData$counts<-countData$counts[rownames(countData$counts) %in%isexpr, ]
anno<-anno%>% filter(GeneID%in% rownames(countData$counts))
# Remove genes with only 1 site and NA in geneIDs
dn<-anno%>%group_by(GeneID)%>%summarise(nsites=n())%>% filter(nsites>1&!is.na(GeneID))
anno<-anno%>% filter(GeneID%in%dn$GeneID)
countData$counts<-countData$counts[rownames(countData$counts) %in%anno$GeneID, ]
Remarque : Vérifiez les paramètres requis pour featureCounts lors de l’utilisation de différentes données, par exemple, pour les lectures à une extrémité, définissez 'isPairedEnd = FALSE'. Reportez-vous au guide de l’utilisateur RSubread pour choisir les options pour vos données, et consultez la section Discussion ci-dessous.
- Chargez les bibliothèques requises :
- Épissage différentiel et analyse de l’utilisation des exons. Nous décrivons deux alternatives pour cette étape : DEXSeq et DiffSplice. L’un ou l’autre peut être utilisé et donner des résultats similaires. Pour des raisons de cohérence, sélectionnez DEXSeq si vous préférez un package DESeq2 pour DGE et utilisez DiffSplice pour une analyse DGE basée sur Limma. Voir dossier complémentaire : »AS_analysis_RNASeq.Rmd".
- Utilisation du package DEXSeq pour l’analyse différentielle des exons.
- Chargez la bibliothèque et créez un exemple de table pour définir le plan expérimental.
library(DEXSeq)
sampleTable<-data.frame(row.names= c("Mbnl1KO_Thymus_1", "Mbnl1KO_Thymus_2", "Mbnl1KO_Thymus_3", "WT_Thymus_1", "WT_Thymus_2", "WT_Thymus_3"), condition= rep(c("Mbnl1_KO", "WT"),c(3,3)), libType= rep(c("paired-end")))
Remarque : les noms de ligne doivent être cohérents avec les noms de fichier bam utilisés par featureCounts pour compter les lectures. sampleTable se compose des détails de chaque échantillon, y compris : le type de bibliothèque et l’état. Ceci est nécessaire pour définir les contrastes ou le groupe de test pour détecter l’utilisation différentielle. - Préparez le fichier d’informations sur l’exon. Les informations d’exon sous forme d’objets GRanges (plages génomiques) (https://bioconductor.org/packages/release/bioc/html/GenomicRanges.html) sont requises en tant qu’entrée pour créer l’objet DEXSeq à l’étape suivante. Faites correspondre les ID de gène avec le nombre de lectures pour créer l’objet exoninfo.
exoninfo<-anno[anno$GeneID%in% rownames(countData$counts),]
exoninfo<-GRanges(seqnames=anno$Chr,
ranges=IRanges(start=anno$Start, end=anno$End, width=anno$Width),strand=Rle(anno$Strand))
mcols(exoninfo)$TranscriptIDs<-anno$TranscriptIDs
mcols(exoninfo)$Ticker<-anno$Ticker
mcols(exoninfo)$ExonID<-anno$ExonID
mcols(exoninfo)$n<-anno$n
mcols(exoninfo)$GeneID<-anno$GeneID
transcripts_l= strsplit(exoninfo$TranscriptIDs, "\\,")
save(countData, sampleTable, exoninfo, transcripts_l, file="AS_countdata.RData") - Créez un objet DEXSeq à l’aide de la fonction DEXSeqDataSet. L’objet DEXSeq collecte ensemble le nombre de lectures, les informations sur les entités d’exon et les exemples d’informations. Utilisez les comptes de lecture générés à l’étape 3 et les informations d’exon obtenues à l’étape précédente pour créer l’objet DEXSeq à partir de la matrice de comptage. L’argument sampleData prend une entrée de bloc de données définissant les exemples (et leurs attributs : type et condition de bibliothèque), 'design' utilise sampleData pour générer une matrice de conception pour les tests différentiels à l’aide de la notation de formule de modèle. Notez qu’un terme d’interaction significatif, condition:exon, indique que la fraction de lectures sur un gène tombant sur un exon particulier dépend de la condition expérimentale, c’est-à-dire qu’il y a AS. Consultez la documentation DEXSeq pour une description complète de la définition de la formule du modèle pour les plans expérimentaux plus complexes. Pour les informations sur les caractéristiques, les identifiants d’exon, le gène correspondant et les transcriptions sont requis.
dxd<-DEXSeqDataSet(countData$counts,sampleData=sampleTable, design=~sample+exon+condition:exon,featureID=exoninfo$ExonID,groupID=exoninfo$GeneID,featureRanges=exoninfo, transcripts=transcripts_l) - Normalisation et estimation de la dispersion. Ensuite, effectuez une normalisation entre les échantillons et estimez la variance des données, due à la fois au bruit de comptage de Poisson dû à la nature discrète du séquençage de l’ARN et à la variabilité biologique, à l’aide des commandes suivantes.
dxd %<>% estimateSizeFactors %>% estimateDispersions %T>% plotDispEsts - Testez l’utilisation différentielle. Après l’estimation de la variation, tester l’utilisation différentielle de l’exon pour chaque gène et générer les résultats.
dxd%<>%testForDEU%>%estimateExonFoldChanges(fitExpToVar=
"condition")#Estimate fold changes
dxr=DEXSeqResults(dxd) - Visualisez les événements d’épissage pour les gènes sélectionnés à l’aide de la commande suivante.
plotDEXSeq(dxr,"Wnk1", displayTranscripts=TRUE, splicing=TRUE,legend
=TRUE,cex.axis=1.2,cex=1.3,lwd=2)
Examinez le fichier R Notebook « AS_analysis_RNASeq.Rmd » pour générer des tracés supplémentaires pour les gènes d’intérêt et pour générer des tracés de volcans à différents seuils.
- Chargez la bibliothèque et créez un exemple de table pour définir le plan expérimental.
- Utilisation de diffSplice de Limma pour identifier l’épissage différentiel. Suivez le fichier R Notebook »AS_analysis_RNASeq.Rmd". Assurez-vous que les étapes 2.1 à 2.3 ont été suivies pour préparer les fichiers d’entrée avant de poursuivre.
- Charger des bibliothèques
library(limma)
library(edgeR) - Filtrage non spécifique. Extraire la matrice des comptages de lecture obtenue en 2.3. Créez une liste d’entités à l’aide de la fonction 'DGEList' à partir du package edgeR, où les lignes représentent les gènes et les colonnes représentent les échantillons.
mycounts=countData$counts
#Change the rownames of the countdata to exon Ids instead of genes for unique rownames.
rownames(mycounts) = exoninfo$ExonID
dge<-DGEList(counts=mycounts)
#Filtering
isexpr<- rowSums(cpm(dge)>1) >=3
dge<-dge[isexpr,,keep.lib.sizes=FALSE]
#Extract the exon annotations for only transcripts meeting non-specific filter
exoninfo=anno%>% filter(ExonID%in% rownames(dge$counts))
#Convert the exoninfo into GRanges object
exoninfo1<-GRanges(seqnames=exoninfo$Chr,
ranges=IRanges(start=exoninfo$Start, end=exoninfo$End, width=exoninfo$Width),strand=Rle(exoninfo$Strand))
mcols(exoninfo1)$TranscriptIDs<-exoninfo$TranscriptIDs
mcols(exoninfo1)$Ticker<-exoninfo$Ticker
mcols(exoninfo1)$ExonID<-exoninfo$ExonID
mcols(exoninfo1)$n<-exoninfo$n
mcols(exoninfo1)$GeneID<-exoninfo$GeneID
transcripts_l= strsplit(exoninfo1$TranscriptIDs, "\\,")
REMARQUE : En tant qu’étape de filtrage non spécifique, les comptages sont filtrés par cpm < 1 dans x sur n échantillons, où x est le nombre minimum de répétitions dans n’importe quelle condition. n = 6 et x = 3 pour cet exemple de données. - Normalisez les comptages entre les échantillons, avec la fonction 'calcNormFactors' du package 'edgeR' en utilisant la moyenne tronquée des valeurs M (méthode de normalisation TMM)24 Il calculera les facteurs d’échelle pour ajuster la taille des bibliothèques.
dge<-calcNormFactors(dge) - Utilisez sampleTable tel que généré à l’étape 2.4.1.1 et créez la matrice de conception. La matrice de conception caractérise la conception. Voir les chapitres 8 et 9 du Guide de l’utilisateur de Limma (https://www.bioconductor.org/packages/devel/bioc/vignettes/limma/inst/doc/usersguide.pdf) pour plus de détails sur les matrices de conception pour des conceptions expérimentales plus avancées.
Treat<- factor(sampleTable$condition)
design<- model.matrix(~0+Treat)
colnames(design) <- levels(Treat) - Ajuster un modèle linéaire par exon. Exécutez la fonction 'voom' du package 'limma' pour traiter les données RNA-seq afin d’estimer la variance et de générer des poids de précision pour corriger le bruit de comptage de Poisson, et transformez les comptages au niveau des exons en log2-counts par million (logCPM). Exécutez ensuite la modélisation linéaire à l’aide de la fonction 'lmfit' pour ajuster les modèles linéaires aux données d’expression pour chaque exon. Calculez des statistiques bayésiennes empiriques pour un modèle ajusté à l’aide de la fonction 'eBayes' pour détecter l’expression différentielle des exons. Ensuite, définissez une matrice de contraste pour les comparaisons expérimentales d’intérêt. Utilisez 'contrasts.fit' pour obtenir des coefficients et des erreurs-types pour chaque paire de comparaison.
v<-voom(dge,design,plot=FALSE)
fit<-lmFit(v,design)
fit<-eBayes(fit)
colnames(fit)
cont.matrix<-makeContrasts(
Mbnl1_KO_WT=Mbnl1_KO-WT,
levels=design)
fit2<-contrasts.fit(fit,cont.matrix) - Analyse d’épissage différentiel. Exécutez 'diffSplice' sur le modèle ajusté pour tester les différences dans l’utilisation des gènes d’exons entre le type sauvage et le knockout et explorez les résultats les mieux classés en utilisant la fonction 'topSplice': test="t » donne un classement des exons AS, test="simes » donne un classement des gènes.
ex<-diffSplice(fit2,geneid=exoninfo$GeneID,exonid=exoninfo$ExonID)
ts<-topSplice(ex,n=Inf,FDR=0.1, test="t", sort.by="logFC")
tg<-topSplice(ex,n=Inf,FDR=0.1, test="simes") - Visualisation. Tracez les résultats avec la fonction 'plotSplice', en donnant le gène d’intérêt dans l’argument généide. Enregistrez les meilleurs résultats triés par log Pliez le changement dans un objet et générez un tracé de volcan pour exposer les exons.
plotSplice(ex,geneid="Wnk1", FDR=0.1)
#Volcano plot
EnhancedVolcano(ts,lab=ts$ExonID,selectLab= head((ts$ExonID),2000), xlab= bquote(~Log[2]~'fold change'), x='logFC', y='P.Value', title='Volcano Plot', subtitle='Mbnl1_KO vs WT (Limma_diffSplice)', FCcutoff=2, labSize=4,legendPosition="right", caption= bquote(~Log[2]~"Fold change cutoff, 2; FDR 10%"))
- Charger des bibliothèques
- Utilisation de rMATS
- Assurez-vous que la dernière version de rMATS v4.1.1 (également appelée rMATS turbo en raison du temps de traitement réduit et des besoins en mémoire) est installée à l’aide de conda ou github (https://github.com/Xinglab/rmats-turbo/releases/download/v4.1.1/rmats_turbo_v4_1_1.tar.gz) dans le répertoire de travail. Suivez la section 4.3 dans « AS_analysis_RNASeq.Rmd ».
- Accédez au dossier contenant les fichiers bam obtenus après le mappage et préparez les fichiers texte, comme requis par rMATS, pour les deux conditions en copiant le nom des fichiers bam (ainsi que le chemin) séparés par ','. Les commandes suivantes doivent être exécutées sur la ligne de commande Linux :
mkdir rMATS_analysis
cd bams/
ls -pd "$PWD"/*| grep "WT"| tr '\n'','> Wt.txt
ls -pd "$PWD"/*| grep "Mb"| tr '\n'','> KO.txt
mv *.txt ../rMATS_analysis - Exécutez rmats.py avec les deux fichiers d’entrée générés à l’étape précédente, ainsi que le fichier GTF obtenu dans 2.1.1.3. Cela générera un dossier de sortie 'rmats_out' contenant des fichiers texte décrivant les statistiques (valeurs p et niveaux d’inclusion) pour chaque événement d’épissage séparément.
python rmats-turbo/rmats.py --b1 KO.txt --b2 Wt.txt --gtf annotation.gtf -t paired --readLength 50 --nthread 8 --od rmats_out/ --tmp rmats_tmp --task pos
REMARQUE : L’annotation de référence sous la forme d’un fichier GTF est également requise. Vérifiez les paramètres si les données sont à une extrémité unique et modifiez l’option -t en conséquence. - Exploration des résultats rMATS. Utilisez le package Bioconductor 'maser'25 pour explorer les résultats rMATS. Chargez les fichiers texte JCEC (Junction and exon counts) dans l’objet « maser » et filtrez le résultat en fonction de la couverture en incluant au moins cinq lectures moyennes par événement d’épissage.
library(maser)
mbnl1<-maser("/rmats_out/", c("WT","Mbnl1_KO"), ftype="JCEC")
#Filtering out events by coverage
mbnl1_filt<-filterByCoverage(mbnl1,avg_reads=5) - Visualisation des résultats rMATS. Sélectionnez les événements d’épissage significatifs à un taux de faux découvert (FDR) de 10 % et une variation minimale de 10 % du pourcentage d’épissage entrant (deltaPSI) à l’aide de la fonction « topEvents » du package « maser ». Ensuite, vérifiez les événements génétiques pour les gènes d’intérêt individuels (échantillon de gène - Wnk1) et tracez les valeurs PSI pour chaque événement d’épissage de ce gène. Générez un tracé de volcan en spécifiant le type d’événement.
#Top splicing events at 10% FDR
mbnl1_top<-topEvents(mbnl1_filt,fdr=0.1, deltaPSI=0.1)
mbnl1_top
#Check the gene events for a particular gene
mbnl1_wnk1<-geneEvents(mbnl1_filt,geneS="Wnk1", fdr=0.1, deltaPSI=0.1)
maser::display(mbnl1_wnk1,"SE")
plotGenePSI(mbnl1_wnk1,type="SE", show_replicates
=TRUE)
volcano(mbnl1_filt,fdr=0.1, deltaPSI=0.1,type="SE")
+xlab("deltaPSI")+ylab("Log10 Adj. Pvalue")+ggtitle("Volcano Plot of exon skipping events") - Générez des tracés Sashimi pour le résultat des événements d’épissage obtenu avec rMATS sous forme de fichiers texte en utilisant le package 'rmats2shahimiplot'. Exécutez le script python sur la ligne de commande Linux.
python ./src/rmats2sashimiplot/rmats2sashimiplot.py --b1 ../bams/WT_Thymus_1.bam,../bams/WT_Thymus_2.bam,../bams/WT_Thymus_3.bam --b2 ../bams/Mbnl1KO_Thymus_1.bam,../bams/Mbnl1KO_Thymus_2.bam,../bams/Mbnl1KO_Thymus_3.bam -t SE -e ../rMATS_analysis/rmats_out/SE.MATS.JC.txt --l1 WT --l2 Mbnl1_KO --exon_s 1 --intron_s 5 -o ../rMATS_analysis/rmats2shasmi_output
REMARQUE: Ce processus peut prendre beaucoup de temps car il générera le tracé Sashimi pour tous les résultats du fichier d’événements. Choisissez les meilleurs résultats (noms de gènes et exons) tels qu’affichés par la fonction topEvents de 'maser' et visualisez le diagramme de Sashimi correspondant.
- Utilisation du package DEXSeq pour l’analyse différentielle des exons.
3. Analyse alternative de polyadénylation (APA) utilisant le séquençage de l’extrémité 3'
- Téléchargement et prétraitement des données
REMARQUE : reportez-vous au fichier R Notebook supplémentaire « APA_analysis_3PSeq_notebook. Rmd » pour les commandes complètes pour les étapes de téléchargement et de prétraitement des données, ou exécutez le fichier bash supplémentaire « APA_data_downloading_preprocessing.sh » sur la ligne de commande Linux.- Téléchargez les données de SRA avec les ID d’accession (1553129 à 1553136).
- Ajuster les adaptateurs et inverser le complément pour obtenir la séquence de brin de détection.
REMARQUE: Cette étape est spécifique au test PolyA-seq utilisé. - La carte se lit à l’assemblage du génome de la souris à l’aide de l’aligneur nœud papillon26.
- Préparation des annotations des sites pA.
REMARQUE: Le traitement du fichier d’annotation de site pA est effectué d’abord à l’aide du fichier R Notebook supplémentaire « APA_analysis_3PSeq_notebook. Rmd » (2.1 - 2.6), puis en utilisant le fichier bash « APA_annotation_preparation.sh ».- Téléchargez l’annotation des sites pA à partir de la base de données PolyASite 2.06.
- Sélectionnez les annotations de site pA pour conserver les sites pA de région 3' non traduite (UTR), qui sont annotés comme Terminal Exon (TE) ou 1000 nt en aval d’un exon terminal annoté (DS) pour l’analyse en aval.
- Obtenez les pics de site pA. Ancrer à chaque site de clivage pA et visualiser la couverture moyenne de lecture à l’aide de bedtools et deeptools27,28. Les résultats ont montré que les pics des lectures cartographiées étaient principalement dispersés à ~60 pb en amont des sites de clivage (figure 5 et figure supplémentaire 5). Par conséquent, les coordonnées des sites pA ont été étendues du fichier d’annotation à 60 pb en amont de leurs sites de clivage. En fonction du protocole de séquençage d’extrémité 3' spécifique utilisé, cette étape devra être optimisée pour les tests autres que PolyA-seq.
- Comptage des lectures
- Préparez le fichier d’annotation des sites pA.
anno<- read.table(file= "flanking60added.pA_annotation.bed",
stringsAsFactors=FALSE, check.names=FALSE, header=FALSE, sep="")
colnames(anno) <- c("chrom", "chromStart", "chromEnd", "name", "score", "strand", "rep", "annotation", "gene_name", "gene_id")
anno<- dplyr::select(anno,name,chrom, chromStart,chromEnd, strand,gene_id,gene_name,rep)
colnames(anno) <- c("GeneID", "Chr", "Start", "End", "Strand", "Ensembl", "Symbol", "repID") - Appliquez 'featureCounts' pour acquérir des nombres bruts. Enregistrez la table de comptage en tant que fichier RData « APA_countData.Rdata » pour l’analyse APA à l’aide de différents outils.
countData<- dir("bamfiles", pattern="sorted.bam$", full.names=TRUE) %>%
# Read all bam files as input for featureCounts
featureCounts(annot.ext=anno, isGTFAnnotationFile= FALSE,minMQS=0,useMetaFeatures= TRUE,allowMultiOverlap=TRUE, largestOverlap= TRUE,strandSpecific=1, countMultiMappingReads =TRUE,primaryOnly= TRUE,isPairedEnd= FALSE,nthreads=12)%T>%
save(file="APA_countData.Rdata")
REMARQUE: Soyez conscient de modifier l’un des paramètres répertoriés dans la fonction 'featureCounts'. Modifier le paramètre 'strandSpecific' pour s’assurer qu’il est cohérent avec la direction de séquençage du test de séquençage de l’extrémité 3' utilisé (empiriquement, la visualisation des données dans un navigateur de génome sur les gènes sur les brins plus et moins clarifiera cela). - Appliquez un filtrage non spécifique de countData. Le filtrage peut améliorer considérablement la robustesse statistique dans les tests différentiels d’utilisation du site pA. Tout d’abord, nous avons supprimé les gènes avec un seul site pA, sur lequel l’utilisation différentielle du site pA ne peut pas être définie. Deuxièmement, nous appliquons un filtrage non spécifique basé sur la couverture: les comptages sont filtrés par cpm inférieur à 1 dans x sur n échantillons, où x est le nombre minimum de répétitions dans n’importe quelle condition. N = 8 et x = 2 pour cet exemple de données.
load(file= "APA_countData.Rdata")# Skip this step if already loaded
# Non-specific filtering: Remove the pA sites not differentially expressed in the samples
countData<-countData$counts%>%as.data.frame%>% .[rowSums(edgeR::cpm(.)>1) >=2, ]
anno%<>% .[.$GeneID%in% rownames(countData), ]
# Remove genes with only 1 site and NA in geneIDs
dnsites<-anno%>%group_by(Symbol)%>%summarise(nsites=n())%>% filter(nsites>1&!is.na(Symbol))
anno<-anno%>% filter(Symbol%in%dnsites$Symbol)
countData<-countData[rownames(countData) %in%anno$GeneID, ]
- Préparez le fichier d’annotation des sites pA.
- Analyse différentielle de l’utilisation du site de polyadénylation à l’aide de pipelines DEXSeq et diffSplice.
- Utilisation du package DEXSeq
REMARQUE : Comme une matrice de contraste ne peut pas être définie pour le pipeline DEXSeq, l’analyse différentielle APA de chacune des deux conditions expérimentales doit être effectuée séparément. L’analyse APA différentielle de la condition WT et de la condition DKO est effectuée à titre d’exemple pour expliquer la procédure. Reportez-vous au dossier complémentaire »APA_analysis_3PSeq_notebook. RMD« pour le flux de travail étape par étape de cette section et l’analyse APA différentielle des autres contrastes.- Chargez la bibliothèque et créez un exemple de table pour définir le plan expérimental.
c("DEXSeq", "GenomicRanges") %>% lapply(library, character.only=TRUE) %>%invisible
sampleTable1<- data.frame(row.names= c("WT_1","WT_2","DKO_1","DKO_2"),
condition= c(rep("WT", 2), rep("DKO", 2)),
libType= rep("single-end", 4)) - Préparez le fichier d’information des sites pA à l’aide du package Bioconductor GRanges.
# Prepare the GRanges object for DEXSeqDataSet object construction
PASinfo <- GRanges(seqnames = anno$Chr,
ranges = IRanges(start = anno$Start, end = anno$End),strand = Rle(anno$Strand))
mcols(PASinfo)$PASID<-anno$repID
mcols(PASinfo)$GeneEns<-anno$Ensembl
mcols(PASinfo)$GeneID<-anno$Symbol
# Prepare the new feature IDs, replace the strand information with letters to match the current pA site clusterID
new.featureID <- anno$Strand %>% as.character %>% replace(. %in% "+", "F") %>% replace(. %in% "-", "R") %>% paste0(as.character(anno$repID), .) - Utilisez les comptes de lecture générés à l’étape 3.3 et les informations de site pA obtenues à l’étape précédente pour créer l’objet DEXSeq.
# Select the read counts of the condition WT and DKO
countData1<- dplyr::select(countData, SRR1553129.sorted.bam, SRR1553130.sorted.bam, SRR1553131.sorted.bam, SRR1553132.sorted.bam)
# Rename the columns of countData using sample names in sampleTable
colnames(countData1) <- rownames(sampleTable1)
dxd1<-DEXSeqDataSet(countData=countData1,
sampleData=sampleTable1,
design=~sample+exon+condition:exon,
featureID=new.featureID,
groupID=anno$Symbol,
featureRanges=PASinfo) - Définissez la paire de contrastes en définissant les niveaux de conditions dans l’objet DEXSeq.
dxd1$condition<- factor(dxd1$condition, levels= c("WT", "DKO"))
# The contrast pair will be "DKO - WT" - Normalisation et estimation de la dispersion. Semblable aux données de séquençage de l’ARN, pour les données de séquençage d’extrémité 3', effectuez une normalisation entre les échantillons (médiane des rapports par colonne pour chaque échantillon) en utilisant la fonction 'estimateSizeFactors', et estimez la variation des données en utilisant la fonction 'estimateDispersions', puis visualisez le résultat de l’estimation de la dispersion à l’aide de la fonction 'plotDispEsts'.
dxd1 %<>% estimateSizeFactors %>% estimateDispersions %T>% plotDispEsts - Test différentiel d’utilisation du site pA pour chaque gène à l’aide de la fonction 'testForDEU', puis estimation du changement de pli de l’utilisation du site pA à l’aide de la fonction 'estimateExonFoldChanges'. Vérifiez les résultats à l’aide de la fonction 'DEXSeqResults' et définissez 'FDR < 10%' comme critère pour les sites pA significativement différentiels.
dxd1 %<>% testForDEU %>% estimateExonFoldChanges(fitExpToVar = "condition")
dxr1 <- DEXSeqResults(dxd1)
dxr1
mcols(dxr1)$description
table(dxr1$padj<0.1) # Check the number of differential pA sites (FDR < 0.1)
table(tapply(dxr1$padj<0.1, dxr1$groupID, any)) # Check the number of gene overlapped with differential pA site - Visualisation des résultats différentiels d’utilisation du site pA à l’aide de tracés APA différentiels générés par la fonction 'plotDEXSeq' et de tracé de volcan par la fonction 'EnhancedVolcano'.
# Select the top 100 significant differential pA sites ranked by FDR
topdiff.PAS<- dxr1%>%as.data.frame%>%rownames_to_column%>%arrange(padj)%$%groupID[1:100]
# Apply plotDEXSeq for the visualization of differential polyA usage
plotDEXSeq(dxr1,"S100a7a", legend=TRUE, expression=FALSE,splicing=TRUE, cex.axis=1.2, cex=1.3,lwd=2)
# Apply perGeneQValue to check the top genes with differential polyA site usage
dxr1%<>% .[!is.na(.$padj), ]
dgene<- data.frame(perGeneQValue= perGeneQValue (dxr1)) %>%rownames_to_column("groupID")
dePAS_sig1<-dxr1%>% data.frame() %>%
dplyr::select(-matches("dispersion|stat|countData|genomicData"))%>%
inner_join(dgene)%>%arrange(perGeneQValue)%>%distinct()%>%
filter(padj<0.1)
# Apply EnhancedVolcano package to visualise differential polyA site usage
"EnhancedVolcano"%>% lapply(library, character.only=TRUE) %>%invisible
EnhancedVolcano(dePAS_sig1, lab=dePAS_sig1$groupID, x='log2fold_DKO_WT',
y='pvalue',title='Volcano Plot',subtitle='DKO vs WT',
FCcutoff=1,labSize=4, legendPosition="right",
caption= bquote(~Log[2]~"Fold change cutoff, 1; FDR 10%"))
- Chargez la bibliothèque et créez un exemple de table pour définir le plan expérimental.
- Utilisation du package diffSplice. Reportez-vous au fichier R Notebook supplémentaire « APA_analysis_3PSeq_notebook. Rmd » pour le flux de travail étape par étape de cette section.
- Définir les contrastes d’intérêt pour l’analyse différentielle de l’utilisation des pA.
Remarque : Cette étape doit être effectuée après la construction et le traitement de l’objet DGEList, qui est inclus dans le fichier R Notebook « APA_analysis_3PSeq_notebook. Rmd ».contrast.matrix<-makeContrasts(DKO_vs_WT=DKO-
WT,Ctrl_vs_DKO=Ctrl-DKO,
KD_vs_Ctrl=KD-Ctrl,KD_vs_DKO=KD-DKO,levels=design)
fit2<-fit%>%contrasts.fit(contrast.matrix)%>%eBayes
summary(decideTests(fit2))
ex<-diffSplice(fit2,geneid=anno$Symbol,exonid=new.featureID)
topSplice(ex) #Check the top significant results with topSplice - Visualisez le résultat des contrastes d’intérêt (ici « DKO - WT ») en utilisant des tracés APA différentiels par la fonction 'plotSplice' et des tracés de volcans avec la fonction 'EnhancedVolcano'. Reportez-vous au fichier R Notebook « APA_analysis_3PSeq_notebook. Rmd » 4.2.7 - 4.2.9 pour la visualisation d’autres paires de contraste.
sig1<-topSplice(ex,n=Inf,FDR=0.1,coef=1, test="t", sort.by="logFC")
sig1.genes<-topSplice(ex,n=Inf,FDR=0.1,coef=1, test="simes")
plotSplice(ex, coef=1,geneid="S100a7a", FDR = 0.1)
plotSplice(ex,coef=1,geneid="Tpm1", FDR = 0.1)
plotSplice(ex,coef=1,geneid="Smc6", FDR = 0.1)
EnhancedVolcano(sig1, lab=sig1$GeneID,xlab= bquote(~Log[2]~'fold change'),
x='logFC', y='P.Value', title='Volcano Plot', subtitle='DKO vs WT',
FCcutoff=1, labSize=6, legendPosition="right")
- Définir les contrastes d’intérêt pour l’analyse différentielle de l’utilisation des pA.
- Utilisation du package DEXSeq
Résultats
Après avoir exécuté le flux de travail étape par étape ci-dessus, les résultats d’analyse AS et APA et les résultats représentatifs se présentent sous la forme de tableaux et de diagrammes de données, générés comme suit.
COMME:
Le principal résultat de l’analyse AS (tableau supplémentaire 1 pour diffSplice; Le tableau 2 pour DEXSeq) est une liste d’exons montrant une utilisation différentielle selon les conditions, et une liste de gènes montrant une activité d’épissage globale significative d’un ou plusieurs de ses exons constitutifs, classés par signification statistique. Le tableau supplémentaire 1, onglet 2, montre les exons significatifs, avec des colonnes montrant le différentiel FC de l’exon par rapport au repos, la valeur p non ajustée par exon et la valeur p ajustée (correction de Benjamini-Hockberg). Le seuillage sur les valeurs p ajustées donnera un ensemble d’exons avec un FDR défini. Le tableau supplémentaire 1, onglet 3, montre une liste classée des gènes montrant l’importance de l’activité globale d’épissage, avec une colonne montrant la valeur p ajustée au niveau du gène calculée à l’aide de la méthode Simes. Des données similaires sont présentées dans le tableau 2 pour DEXSeq. La figure supplémentaire 1 et la figure supplémentaire 2 montrent un schéma d’épissage différentiel dans les gènes Mbnl1, Tcf7 et Lef1 qui ont été validés expérimentalement dans l’article publié présenté avec les données15. Les auteurs ont montré la validation expérimentale de cinq gènes - Mbnl1, Mbnl2, Lef1, Tcf7 et Ncor2. Notre approche a détecté des attrapés d’épissage différentiel dans tous ces gènes. Nous présentons ici les niveaux de FDR pour chaque gène en utilisant DEXSeq, diffSplice et rMATS respectivement tels qu’obtenus dans les tableaux supplémentaires 1-3: Mbnl1 (0, 6.6E-61 ,0), Mbnl2 (0,0.18,0), Lef1 (1.4E-10, 1.3E-04, 0), Tcf7 (0, 1.1E-6, 0) et Ncor2 (9.2E-11, 1.6E-06, 0).
La figure 2 montre une comparaison entre les résultats obtenus à partir de trois outils différents et illustre d’autres modèles d’épissage dans le gène Wnk1. Les diagrammes de volcans sont présentés à la figure 2A (diffSplice) et à la figure 2B (DEXSeq). Trois autres gènes hautement classés sont présentés dans la figure supplémentaire 1 (diffSplice) et la figure supplémentaire 2 (DEXSeq). L’axe des y montre la signification statistique (-log10 P-values) et l’axe des x montre l’ampleur de l’effet (changement de pli). Les gènes situés dans les quadrants supérieur gauche ou droit indiquent une FC substantielle et de fortes preuves statistiques de différences réelles.

Graphique 2. Comparaison des résultats d’épissage alternatifs obtenus à partir de diffSplice, DEXSeq et rMATS. |
(A) Tracé du volcan (à gauche) du RNA-Seq à partir de l’analyse de Limma diffSplice: L’axe des x montre le changement de pli de l’exon logarithmique; L’axe des y montre -log10 p-value. Chaque point correspond à un exon. Ligne pointillée horizontale à p-value=1E-5; lignes pointillées verticales à deux plis (FC). Les exons rouges montrent une signification FC et statistique substantielle. Tracé d’épissage différentiel (à droite) : Les modèles d’épissage sont présentés pour un exemple de gène Wnk1 où l’axe des x montre l’exon id par transcription ; l’axe des y montre le changement relatif du pli logarithmique de l’exon (la différence entre le logFC de l’exon et le logFC global pour tous les autres exons). Les exons surlignés en rouge montrent une expression différentielle statistiquement significative (FDR < 0,1).
(B) Tracé du volcan (à gauche) et utilisation différentielle de l’exon (à droite) du RNA-Seq obtenu à partir de l’analyse DEXSeq. Le gène Wnk1 montre une utilisation différentielle significative des exons entre WT et Mbnl1 knock-out mis en évidence en rose, qui correspondent aux mêmes exons différentiels en (A).
(C) Diagramme du volcan (à gauche) et diagramme de Sashimi (à droite) pour Wnk1 obtenu à partir de l’analyse rMATS. Graphique du volcan illustrant un événement significatif d’exon sauté (cassette) (SE) dans le type sauvage par rapport à l’élimination à 10% FDR avec un changement de pourcentage épissé dans les valeurs (PSI ou ΔΨ) > 0,1. L’axe des x montre l’évolution des valeurs PSI selon les conditions et l’axe des y montre le log P-Value. Le diagramme de Sashimi montre un événement d’exon sauté dans le gène Wnk1 , correspondant à un exon différentiel significatif dans (A) et (B). Chaque rangée représente un échantillon d’ARN-Seq : trois répliques de type sauvage et de knock-out Mbnl1. La hauteur montre la couverture de lecture en RPKM et les arcs de connexion représentent les lectures de jonction à travers les exons. Les isoformes alternatives annotées du modèle génétique sont indiquées au bas du graphique. Le panneau inférieur de C illustre les lectures de jonction utilisées pour calculer la statistique PSI.
(D) Diagramme de Venn comparant le nombre d’exons différentiels significatifs obtenus par les différentes méthodes. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Graphique 2 A (panneau de droite) montre un affichage schématique des différences d’exon de l’un des gènes les mieux classés, montrant logFC sur l’axe des y et le nombre d’exons sur l’axe des x. Cet exemple montre un exon de cassette variant selon les conditions du gène Wnk1. Le graphique d’utilisation différentielle des exons de DEXSeq montre des preuves d’épissage différentiel sur cinq sites d’exons près de Wnk1.6.45. Les exons surlignés en rose sont susceptibles d’être épissés dans des échantillons Mbnl1 KO par rapport à WT. Ces exons sont complémentaires aux résultats obtenus par diffSplice qui montre un schéma similaire à la position génomique spécifique. D’autres exemples sont présentés dans la figure supplémentaire 1 et la figure supplémentaire 2. Une vue plus détaillée pour confirmer des résultats intéressants peut être donnée en comparant les pistes de couverture (wiggle) dans les unités RPM (lectures par million) dans les navigateurs génomiques UCSC (Université de Santa Cruz) ou IGV (Integrative Genomics Viewer) (non représenté), ainsi que la corrélation visuelle avec d’autres pistes d’intérêt, telles que les modèles de gènes connus, la conservation et d’autres tests à l’échelle du génome.
Le tableau de sortie rMATS répertorie les événements d’épissage alternatifs significatifs classés par type (tableau supplémentaire 3). La figure 2C montre un diagramme volcanique de gènes qui sont épissés alternativement, avec l’ampleur de l’effet mesurée par la statistique différentielle « pourcentage épissé dans » (PSI ou ΔΨ) de11.
Le PSI fait référence au pourcentage de lectures compatibles avec l’inclusion d’un exon de cassette (c.-à-d. les lectures correspondant à l’exon de la cassette elle-même ou les lectures de jonction chevauchant l’exon) par rapport aux lectures compatibles avec l’exclusion d’exons, c’est-à-dire les lectures de jonction sur les exons adjacents en amont et en aval (panneau inférieur de la figure 2C). Le panneau de droite de la figure 2C montre le tracé en sashimi du gène Wnk1 avec un événement d’épissage différentiel superposé sur des pistes de couverture pour le gène, avec un exon sauté dans Mbnl1 KO. Les arcs joignant les exons indiquent le nombre de lectures de jonction (lectures traversant un intron épissé). Différents onglets du tableau supplémentaire 3 montrent des lectures significatives de chaque type d’événement qui s’étend sur des jonctions avec des limites d’exons (nombre de jonctions et comptage d’exons (JCEC)). La figure 2D compare les exons significativement épissés différentiellement détectés par les trois outils.

Graphique 3. Événements d’épissage alternatifs acquis par l’analyse rMATS. A) Types d’événements SA. Cette figure est adaptée de la documentation rMATS11 expliquant l’événement d’épissage avec des exons constitutifs et alternativement épissés. Étiqueté avec le nombre de chaque événement à FDR 10%. B) Diagramme de sashimi du gène Add3 présentant un exon sauté (SE). C) Diagramme de sashimi du gène Baz2b présentant un site d’épissure alternatif 5' (A5SS). D) Diagramme de sashimi du gène Lsm14b présentant un site d’épissure alternatif 3' (A3SS). E) Tracé de Sashimi du gène Mta1 présentant des exons mutuellement exclusifs (MXE). F) Diagramme de sashimi du gène Arpp21 présentant un intron retenu (RI). Les lignes rouges représentent trois répliques de type sauvage et les lignes orange représentent les répliques knock-out Mbnl1. L’axe des x correspond aux coordonnées génomiques et aux informations sur les brins, l’axe des y montre la couverture en RPKM. Veuillez cliquer ici pour voir une version agrandie de cette figure.
La figure 3 illustre les types d’épissage SE, A5SS, A3SS, MXE et RI à l’aide de diagrammes de Sashimi des gènes les plus significatifs de ces événements. En comparant les trois répétitions de WT et de Mbnl1_KO, un total de 1272 événements SE, 130 événements A5SS, 116 événements A3SS, 215 événements MXE et 313 événements RI ont été détectés à FDR 10%. Le diagramme de Sashimi illustre le type d’événement en utilisant les gènes supérieurs comme exemple.
APA:
Le résultat de l’analyse APA est similaire à l’analyse AS au niveau exon. Un tableau des principaux gènes classés par activité différentielle de l’APA dans le 3'UTR est fourni (tableau supplémentaire 4 et tableau supplémentaire 5). La figure 4A montre les diagrammes volcaniques des gènes par activité différentielle de l’APA dans les 3'UTR générés séparément en utilisant diffSplice et DEXSeq. La figure 4B montre le diagramme de Venn comparant les résultats significativement différentiels d’utilisation du site pA acquis à partir de différents pipelines. Les figures 4C et 4D montrent la représentation schématique de l’utilisation différentielle du site pA dans le 3'UTR du gène Fosl2 (Figure 4C) et Papola (Figure 4D) généré à l’aide de diffSplice et DEXSeq, qui sont validés expérimentalement pour montrer un déplacement distal à proximal significatif (Fosl2) et un déplacement proximal à distal significatif (Papola) de l’utilisation du site pA dans DKO, respectivement. D’autres exemples sont présentés dans la figure supplémentaire 3 et la figure supplémentaire 4.

Graphique 4. Tracés alternatifs de polyadénylation par diffSplice et DEXSeq. A) Tracés de volcans de données PolyA-seq générés à l’aide de diffSplice et DEXSeq. L’axe des X montre le changement de pli du site log pA; L’axe des y affiche -log10 p-value. Chaque point correspond à un site pA. Ligne pointillée horizontale à p-value=1E-5; lignes pointillées verticales à 2 fois FC. Les exons rouges montrent une signification FC et statistique substantielle. B) Diagramme de Venn comparant les résultats différentiels significatifs d’utilisation du site pA acquis à partir de différents pipelines. C-D) Graphiques APA différentiels générés à l’aide de diffSplice et DEXSeq montrant les sites pA proximaux, internes et distaux pour le gène Fosl2 et Tapola . Les figures sont générées par la même fonction que la figure 2 (B) mais avec des sites pA remplaçant les exons. Veuillez cliquer ici pour voir une version agrandie de cette figure.
La figure 5 est un diagramme de diagnostic pour confirmer la distribution de lecture attendue autour des sites de clivage annotés de pA pour le test PolyA-seq utilisé. Il montre la couverture moyenne dans les régions flanquantes ancrées à des sites de clivage pA connus au niveau pangénomique. Dans ce cas, l’empilement attendu des lectures en amont des sites est visualisé. Les distributions de lecture ancrées aux sites pA pour tous les échantillons PolyA-seq sont montrées dans la figure supplémentaire 5.

Graphique 5. Tracé de couverture moyenne autour des sites de clivage pA. Le site de clivage des données PolyA-seq est représenté par une ligne pointillée verticale. L’axe des X montre la position de base par rapport aux sites de clivage pA, jusqu’à 100 nucléotides en amont et en aval; L’axe des y montre la couverture moyenne en lecture sur tous les sites de clivage pA, normalisée par la taille de la bibliothèque en CPM. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Les résultats APA différentiels de différents contrastes générés par le même pipeline peuvent être comparés et vérifiés en visualisant la couverture de lecture de sites pA significativement différentiels représentatifs dans le navigateur génomique. La figure 6A est le diagramme de Venn comparant l’utilisation significativement différentielle du site pA de différents contrastes acquis à partir de diffSplice. Les figures 6B-D sont les instantanés IGV de la couverture de lecture aux sites pA pour différents gènes, qui montrent les modèles cohérents avec ceux découverts dans l’analyse APA à l’aide de diffSplice. La figure 6B valide le déplacement proximal à distal significatif de l’utilisation du site pA pour le gène Paip2, qui est détecté de manière unique dans le contraste DKO vs WT, mais pas dans les deux autres contrastes KD vs WT, et Ctr vs WT. La figure 6C valide le déplacement distal significatif vers proximal de l’utilisation du site pA pour le gène Ccl2 détecté uniquement dans le contraste KD vs WT, tandis que la figure 6D valide l’utilisation différentielle significative de tous les contrastes pour le gène Cacna2d1.
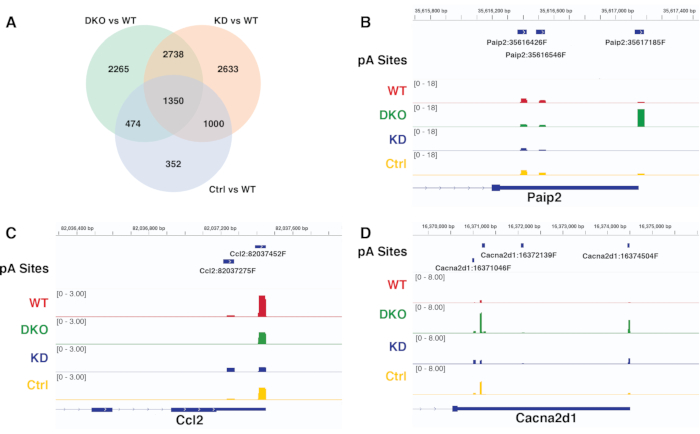
Graphique 6. Comparaison des contrastes et vérification des résultats d’épissure diffSplice. A) Diagramme de Venn comparant les résultats différentiels significatifs d’utilisation du site pA de différents contrastes acquis à partir de diffSplice. B-D) L’instantané IGV visualisant les pics de pA couvre les gènes Paip2, Ccl2 et Cacna2d1 dans toutes les conditions. Chaque piste représente la couverture de lecture dans une condition spécifique. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Figure supplémentaire 1. Analyse RNA-Seq de l’épissage différentiel avec Limma diffSplice. (A) Tracé du volcan de RNA-Seq à partir de l’analyse de Limma diffSplice: L’axe des x montre le changement de pli de l’exon logarithmique; L’axe des y montre -log10 p-value. Chaque point correspond à un exon. Ligne pointillée horizontale à p-value=1E-5; lignes pointillées verticales à double changement (FC). Les exons rouges montrent une signification FC et statistique substantielle. (B-D) Tracés d’épissage différentiel : Les modèles d’épissage sont présentés par exemple les gènes Mbnl1, Tcf7 et Lef1, respectivement, où l’axe des x montre l’exon id par transcrit; l’axe des y montre le changement relatif du pli logarithmique de l’exon (la différence entre le logFC de l’exon et le logFC global pour tous les autres exons). Les exons surlignés en rouge montrent une expression différentielle statistiquement significative (FDR < 0,1). Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 2. Analyse RNA-Seq de l’utilisation différentielle des exons avec DEXSeq. (A) Parcelle volcanique. (B-D) Utilisation différentielle des exons de RNA-Seq obtenu à partir de l’analyse DEXSeq. Les gènes Mbnl1, Tcf7 et Lef1, respectivement, montrent une utilisation différentielle significative des exons entre WT et Mbnl1 knock-out surlignés en rose, qui correspondent aux mêmes exons différentiels dans la figure supplémentaire 1. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 3. Tracés alternatifs de polyadénylation par diffSplice. A) Tracés de volcans de données PolyA-seq générées à l’aide de diffSplice en trois paires de contraste obtenues à partir des données PolyA-seq de souris, y compris double knockout (DKO) vs type sauvage (WT), knock-down (KD) vs WT, et contrôle (Ctrl) vs WT. L’axe X montre le changement de pli du site log pA; L’axe des y affiche -log10 p-value. Chaque point correspond à un site pA. Ligne pointillée horizontale à p-value=1E-5; lignes pointillées verticales à 2 fois FC. Les sites rouges de pA montrent une signification FC et statistique importante. B) Graphiques APA différentiels générés à l’aide de diffSplice montrant les sites pA proximaux, internes et distaux pour les gènes hautement classés S100a7a, Tpm1 et Smc6. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 4. Analyse différentielle de l’utilisation de pA par pipeline DEXSeq. A) Tracés de volcans de données PolyA-seq générées à l’aide de DEXSeq en trois paires obtenues à partir des données PolyA-seq de la souris, y compris le double knockout (DKO) vs le type sauvage (WT), le knock-down (KD) vs WT et le contrôle (Ctrl) vs WT. L’axe X montre le changement de pli du site log pA; L’axe des y affiche -log10 p-value. Chaque point correspond à un site pA. Ligne pointillée horizontale à p-value=1E-5; lignes pointillées verticales à 2 fois FC. Les sites rouges de pA montrent une signification FC et statistique importante. B) Tracés APA différentiels générés à l’aide de DEXSeq montrant les sites pA proximaux, internes et distaux pour les gènes hautement classés S100a7a, Tpm1 et Smc6. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 5. Diagramme de couverture moyenne et cartes thermiques autour des sites de clivage pA. Couverture montrée pour quatre conditions, avec des gènes sur les brins avant et arrière montrés séparément. L’axe des X montre la position de base par rapport aux sites de clivage pA, jusqu’à 100 nucléotides en amont et en aval; L’axe des y fait référence à la couverture moyenne aux positions de base relatives correspondantes sur tous les sites de clivage pA. Les cartes thermiques fournissent une vue alternative, chaque site de clivage pA étant représenté par une ligne distincte sur l’axe des abscisses, ordonnée par couverture. L’intensité montre la couverture en lecture (voir légende). Veuillez cliquer ici pour télécharger ce fichier.
Tableau supplémentaire 1. Résultat diffSplice de l’analyse AS. Le premier onglet définit les noms de colonne des sorties originales présentées dans les deuxième onglets (niveau exon) et troisième (niveau gène). Veuillez cliquer ici pour télécharger ce tableau.
Tableau supplémentaire 2. Sortie DEXSeq de l’analyse AS. Le premier onglet définit les noms de colonne de la sortie d’origine présentée dans les deuxième onglets (niveau exon) et troisième (niveau gène). Veuillez cliquer ici pour télécharger ce tableau.
Tableau supplémentaire 3. Sortie rMATS de l’analyse AS. Le premier onglet définit les noms de colonne du fichier récapitulatif (onglet 2) et les fichiers JCEC pour chaque événement (onglet 3-7). Veuillez cliquer ici pour télécharger ce tableau.
Tableau supplémentaire 4. Résultat diffSplice de l’analyse APA. Le premier onglet définit les noms de colonne de la sortie d’origine présentée dans les deuxième onglets (niveau du site pA) et troisième (niveau du gène). Veuillez cliquer ici pour télécharger ce tableau.
Tableau supplémentaire 5. Sortie DEXSeq de l’analyse APA. Le premier onglet définit les noms de colonne de la sortie d’origine présentée dans les deuxième onglets (niveau du site pA) et troisième (niveau du gène). Veuillez cliquer ici pour télécharger ce tableau.
Tableau supplémentaire 6. Un résumé du nombre d’exons significativement modifiés pour les sites AS et pA pour l’APA. Veuillez cliquer ici pour télécharger ce tableau.
Tableau supplémentaire 7. Un résumé des outils et des packages utilisés dans l’analyse AS/APA. Veuillez cliquer ici pour télécharger ce tableau.
Discussion
Dans cette étude, nous avons évalué des approches basées sur les exons et les événements pour détecter l’AS et l’APA dans les données de séquençage massif de l’ARN-Seq et de l’extrémité 3'. Les approches AS basées sur les exons produisent à la fois une liste d’exons exprimés différentiellement et un classement au niveau du gène ordonné en fonction de la signification statistique de l’activité globale d’épissage différentiel au niveau du gène (tableaux 1-2, 4-5). Pour le paquet diffSplice, l’utilisation différentielle est déterminée en ajustant des modèles linéaires pondérés au niveau de l’exon pour estimer le changement différentiel logarithmique d’un exon par rapport au changement moyen du pli logarithmique des autres exons au sein du même gène (appelé par exon FC). La signification statistique au niveau du gène est calculée en combinant des tests de signification individuels au niveau de l’exon dans un test génique par la méthode Simes10. Ce classement par activité d’épissage différentiel au niveau des gènes peut ensuite être utilisé pour effectuer une analyse d’enrichissement des ensembles de gènes (GSEA) des principales voies impliquées10. DEXSeq utilise une stratégie similaire, en ajustant un modèle linéaire généralisé pour mesurer l’utilisation différentielle des exons, bien qu’il diffère dans certaines étapes telles que le filtrage, la normalisation et l’estimation de la dispersion. En comparant les 500 exons les mieux classés montrant l’activité AS et l’APA en utilisant DEXSeq et DiffSplice, nous avons trouvé un chevauchement de 310 exons et 300 sites pA, respectivement, démontrant la concordance des deux approches basées sur les exons, ce qui a également été démontré dans une étude précédente 29. Il est recommandé d’utiliser une combinaison d’une approche basée sur les exons (DEXSeq ou diffSplice) et d’une approche basée sur les événements pour la détection et la classification complètes de la SA. Pour l’APA, les utilisateurs peuvent choisir DEXSeq ou diffSplice: les deux méthodes se sont avérées performantes dans un large éventail d’expériences transcriptomiques29.
Lors de la préparation de la bibliothèque de séquençage de l’ARN pour une analyse SA, il est important d’utiliser un protocole de séquençage d’ARN en vrac spécifique au brin8, car une grande fraction des gènes dans les génomes des vertébrés se chevauchent sur différents brins, et un protocole non spécifique au brin est incapable de distinguer ces régions qui se chevauchent, confondant la détection finale des exons. Une autre considération est la profondeur de lecture, les analyses d’épissage nécessitant un séquençage plus profond que DGE, par exemple 30 à 60 millions de lectures par échantillon, contre 5 à 25 millions de lectures par échantillon pour DGE (https://sapac.support.illumina.com/bulletins/2017/04/considerations-for-rna-seq-read-length-and-coverage-.html). Tous les outils présentés dans le protocole prennent en charge les données de séquençage à extrémité unique et appariée. Si seules des annotations génétiques connues sont utilisées pour détecter les lectures de jonction, des lectures plus courtes à une extrémité (≥ 50 pb) peuvent être utilisées, bien que la détection de novo de nouvelles jonctions d’épissage bénéficie de lectures appariées et plus longues (≥ 100 pbp)30,31. Le choix du protocole d’extraction de l’ARN - soit la sélection polyA ou la déplétion de l’ARNr - dépend de la qualité de l’ARN et de la question expérimentale - voir31 pour une discussion. En fonction des détails de la construction de la bibliothèque, des modifications seront nécessaires aux exemples de scripts donnés ici pour les paramètres d’alignement de lecture, de comptage de caractéristiques et de rMATS. Lors du calcul du nombre initial de lectures au niveau de l’exon à l’aide de featureCounts, ou de méthodes similaires, il faut veiller à configurer correctement les options de fonction pour les comptages et les brins : dans featureCounts, nous définissons l’argument « strandSpecific » de manière appropriée pour le protocole RNA-seq spécifique au brin utilisé ; et pour la quantification au niveau des exons, on s’attend à ce qu’une lecture soit mappée sur les exons adjacents, et nous définissons donc le paramètre allowMultiOverlap sur TRUE. Pour l’APA, il existe différents protocoles de séquençage d’extrémité3' 6 qui varient dans l’emplacement précis des pics par rapport au site pA. Pour nos exemples de données, nous déterminons que le pic est de 60 pb en amont du site pA, comme le montre la figure 5, et cette analyse devra être adaptée pour d’autres protocoles de séquençage d’extrémité 3'.
Dans ce protocole, nous limitons la portée à la discussion des analyses différentielles au niveau des exons individuels et des événements d’épissage consistant en des combinaisons exon-intron adjacentes. Nous ne discutons pas de la classe d’analyses basées sur la reconstruction isoforme de novo telles que Cufflinks, Cuffdiff32, RSEM 33, Kallisto34 qui visent à détecter et à quantifier l’expression absolue et relative d’isoformes alternatives entières. Les méthodes basées sur les exons et les événements sont plus sensibles pour détecter les événements d’épissage individuels30 et, dans de nombreux cas, fournissent toutes les informations nécessaires pour une analyse plus approfondie, sans qu’il soit nécessaire de les quantifier au niveau de l’isoforme.
La dernière version des fichiers sources de ce protocole est disponible à l’adresse https://github.com/jiayuwen/AS_APA_JoVE
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Cette étude a été financée par une bourse Future Fellowship (FT16010043) du Conseil australien de la recherche (ARC) et un programme ANU Futures.
matériels
| Name | Company | Catalog Number | Comments |
| Not relevent for computational study |
Références
- Katz, Y., Wang, E. T., Airoldi, E. M., Burge, C. B. Analysis and design of RNA sequencing experiments for identifying isoform regulation. Nature Methods. 7 (12), 1009-1015 (2010).
- Wang, Y., et al. Mechanism of alternative splicing and its regulation. Biomedical Reports. 3 (2), 152-158 (2015).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in Bioinformatics. 21 (6), 2052-2065 (2020).
- Movassat, M., et al. Coupling between alternative polyadenylation and alternative splicing is limited to terminal introns. RNA Biology. 13 (7), 646-655 (2016).
- Tian, B., Manley, J. L. Alternative polyadenylation of mRNA precursors. Nature Reviews Molecular Cell Biology. 18 (1), 18-30 (2017).
- Herrmann, C. J., et al. PolyASite 2.0: a consolidated atlas of polyadenylation sites from 3' end sequencing. Nucleic Acids Research. 48 (1), 174-179 (2020).
- Liu, R., Loraine, A. E., Dickerson, J. A. Comparisons of computational methods for differential alternative splicing detection using RNA-seq in plant systems. BMC Bioinformatics. 15 (1), 364 (2014).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biology. 17 (1), 13 (2016).
- Anders, S., Reyes, A., Huber, W. Detecting differential usage of exons from RNA-seq data. Genome Research. 22 (10), 2008-2017 (2012).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2014).
- Shen, S., et al. rMATS: Robust and flexible detection of differential alternative splicing from replicate RNA-Seq data. Proceedings of the National Academy of Sciences. 111 (51), 5593-5601 (2014).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in bioinformatics. 21 (6), 2052-2065 (2020).
- Kanitz, A., et al. Comparative assessment of methods for the computational inference of transcript isoform abundance from RNA-seq data. Genome biology. 16 (1), 1-26 (2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550 (2014).
- Sznajder, L. J., et al. Loss of MBNL1 induces RNA misprocessing in the thymus and peripheral blood. Nature Communications. 11, 1-11 (2020).
- Batra, R., et al. Loss of MBNL leads to disruption of developmentally regulated alternative polyadenylation in RNA-mediated disease. Molecular Cell. 56 (2), 311-322 (2014).
- Leinonen, R., Sugawara, H., Shumway, M., et al. The sequence read archive. Nucleic acids research. 39, 19-21 (2010).
- Tange, O. . GNU parallel-the command-line power tool. 36, 42-47 (2011).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet journal. 17 (1), 10-12 (2011).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Robinson, M. D., Oshlack, A. A scaling normalization method for differential expression analysis of RNA-seq data. Genome Biology. 11 (3), 25 (2010).
- Veiga, D. F. T. maser: Mapping Alternative Splicing Events to pRoteins. R package version 1.4.0. , (2019).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (13), 25 (2009).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ramírez, F., Dündar, F., Diehl, S., Grüning, B. A., Manke, T. deepTools: a flexible platform for exploring deep-sequencing data. Nucleic acids research. 42 (1), 187-191 (2014).
- Merino, G. A., Conesa, A., Fernández, E. A. A benchmarking of workflows for detecting differential splicing and differential expression at isoform level in human RNA-seq studies. Briefings in bioinformatics. 20 (2), 471-481 (2019).
- Chhangawala, S., Rudy, G., Mason, C. E., Rosenfeld, J. A. The impact of read length on quantification of differentially expressed genes and splice junction detection. Genome biology. 16 (1), 1-10 (2015).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biol. 17, 13 (2016).
- Trapnell, C., et al. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nat Protoc. 7 (3), 562-578 (2012).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC Bioinformatics. 12, 323 (2011).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nat Biotechnol. 34 (5), 525-527 (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon