Method Article
تحديد الربط البديل والبولي أدينيل في بيانات RNA-seq
In This Article
Summary
يعمل الربط البديل (AS) والبولي أدينيل البديل (APA) على توسيع تنوع الأشكال المتماثلة ومنتجاتها. هنا ، نصف بروتوكولات المعلوماتية الحيوية لتحليل مقايسات تسلسل RNA-seq السائبة و 3 'للكشف عن وتصور AS و APA المتغيرة عبر الظروف التجريبية.
Abstract
بالإضافة إلى التحليل النموذجي ل RNA-Seq لقياس التعبير الجيني التفاضلي (DGE) عبر الظروف التجريبية / البيولوجية ، يمكن أيضا استخدام بيانات RNA-seq لاستكشاف آليات تنظيمية معقدة أخرى على مستوى exon. يلعب الربط البديل و polyadenylation دورا حاسما في التنوع الوظيفي للجين من خلال توليد أشكال متساوية مختلفة لتنظيم التعبير الجيني على مستوى ما بعد النسخ ، ويمكن أن يؤدي قصر التحليلات على مستوى الجينات بالكامل إلى تفويت هذه الطبقة التنظيمية المهمة. هنا ، نوضح تحليلات مفصلة خطوة بخطوة لتحديد وتصور استخدام موقع exon و polyadenylation التفاضلي عبر الظروف ، باستخدام Bioconductor والحزم والوظائف الأخرى ، بما في ذلك DEXSeq و diffSplice من حزمة Limma و rMATS.
Introduction
تم استخدام RNA-seq على نطاق واسع على مر السنين عادة لتقدير التعبير الجيني التفاضلي واكتشاف الجينات1. بالإضافة إلى ذلك ، يمكن استخدامه أيضا لتقدير الاستخدام المتغير لمستوى إكسون بسبب تعبير الجينات عن أشكال متساوية مختلفة ، وبالتالي المساهمة في فهم أفضل لتنظيم الجينات على مستوى ما بعد النسخ. تولد غالبية الجينات حقيقية النواة أشكالا متساوية مختلفة عن طريق الربط البديل (AS) لزيادة تنوع تعبير mRNA. يمكن تقسيم أحداث AS إلى أنماط مختلفة: تخطي exons كاملة (SE) حيث تتم إزالة exon ("كاسيت") تماما من النص جنبا إلى جنب مع الإنترونات المرافقة له ؛ اختيار موقع لصق بديل (مانح) 5 '(A5SS) واختيار موقع لصق بديل 3 '(متقبل) (A3SS) عند وجود موقعين أو أكثر من مواقع لصق على طرفي إكسون ؛ الاحتفاظ بالإنترونات (RI) عند الاحتفاظ ب intron ضمن نسخة mRNA الناضجة والاستبعاد المتبادل لاستخدام exon (MXE) حيث يمكن الاحتفاظ بواحد فقط من الإكسونات المتاحين في وقت 2,3. يلعب polyadenylation البديل (APA) أيضا دورا مهما في تنظيم التعبير الجيني باستخدام مواقع poly البديلة (A) لتوليد أشكال متماثلة متعددة من mRNA من نسخة واحدة4. تقع معظم مواقع polyadenylation (pAs) في المنطقة غير المترجمة 3 '(3' UTRs) ، مما يولد أشكالا متماثلة من mRNA بأطوال UTR متنوعة 3 بوصات. نظرا لأن 3 'UTR هو المحور المركزي للتعرف على العناصر التنظيمية ، يمكن أن تؤثر أطوال UTR المختلفة 3 'على توطين mRNA واستقراره وترجمته5. هناك فئة من 3 'مقايسات تسلسل نهاية محسنة للكشف عن APA التي تختلف في تفاصيل البروتوكول6. تم تصميم خط الأنابيب الموصوف هنا ل PolyA-seq ، ولكن يمكن تكييفه مع البروتوكولات الأخرى كما هو موضح.
في هذه الدراسة ، نقدم مجموعة من طرق تحليل exon التفاضلية 7,8 (الشكل 1) ، والتي يمكن تقسيمها إلى فئتين عريضتين: القائمة على exon (DEXSeq9 ، diffSplice 10) والقائمة على الحدث (تكرار التحليل متعدد المتغيرات لربط النص (rMATS)11). تقارن الطرق القائمة على الإكسون تغير الطي عبر ظروف الإكسونات الفردية ، مقابل مقياس للتغير الكلي في طية الجينات لاستدعاء استخدام إكسون المعبر عنه بشكل تفاضلي ، ومن ذلك تحسب مقياسا على مستوى الجينات لنشاط AS. تستخدم الطرق المستندة إلى الأحداث قراءات تقاطع exon-intron-spanning لاكتشاف وتصنيف أحداث الربط المحددة مثل تخطي exon أو الاحتفاظ بالإنترونات ، وتمييز أنواع AS هذه في الإخراج3. وبالتالي ، توفر هذه الطرق وجهات نظر تكميلية لتحليل كامل لمعيار المحاسبة12,13. اخترنا DEXSeq (استنادا إلى حزمة DESeq214 DGE) و diffSplice (استنادا إلى حزمة Limma10 DGE) للدراسة لأنهما من بين الحزم الأكثر استخداما لتحليل الربط التفاضلي. تم اختيار rMATS كطريقة شائعة للتحليل القائم على الأحداث. طريقة أخرى شائعة قائمة على الأحداث هي MISO (خليط من الأشكال المتساوية)1. بالنسبة إلى APA ، نقوم بتكييف النهج القائم على exon.
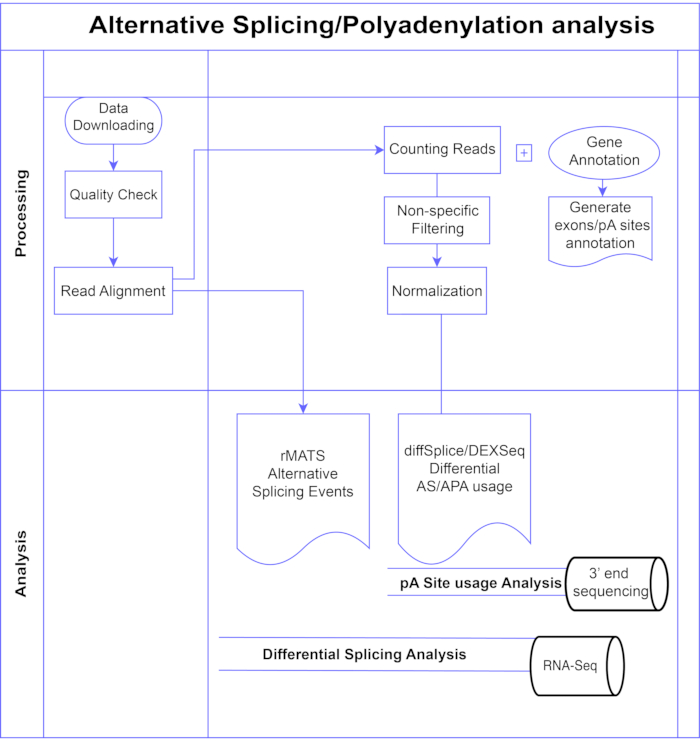
الشكل 1. خط أنابيب التحليل. مخطط انسيابي للخطوات المستخدمة في التحليل. تشمل الخطوات: الحصول على البيانات ، وإجراء فحوصات الجودة ومحاذاة القراءة متبوعة بحساب القراءات باستخدام التعليقات التوضيحية لمواقع exons و introns و pA المعروفة ، والتصفية لإزالة الأعداد المنخفضة والتطبيع. تم تحليل بيانات PolyA-seq لمواقع pA البديلة باستخدام طرق diffSplice / DEXSeq ، وتم تحليل RNA-Seq السائب للربط البديل على مستوى exon باستخدام طرق diffSplice / DEXseq ، وتم تحليل أحداث AS باستخدام rMATS. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
تم الحصول على بيانات RNA-seq المستخدمة في هذا المسح من التعبير الجيني الجامع (GEO) (GSE138691)15. استخدمنا بيانات RNA-seq للفأر من هذه الدراسة مع مجموعتين من الحالات: النوع البري (WT) والضربة القاضية من النوع 1 الشبيه بالعضلات (Mbnl1 KO) مع ثلاث نسخ متماثلة لكل منهما. لإثبات تحليل استخدام موقع polyadenylation التفاضلي ، حصلنا على بيانات الخلايا الليفية الجنينية للفأر (MEFs) PolyA-seq (GEO Accession GSE60487)16. تحتوي البيانات على أربع مجموعات شروط: النوع البري (WT) ، النوع 1 / النوع 2 الشبيه بالعضلات بالضربة القاضية المزدوجة (Mbnl1/2 DKO) ، Mbnl 1/2 DKO مع ضربة قاضية Mbnl3 (KD) و Mbnl1/2 DKO مع التحكم Mbnl3 (Ctrl). تتكون كل مجموعة شرط من نسختين متماثلتين.
| انضمام GEO | رقم تشغيل SRA | اسم العينة | شرط | تكرار | نسيج | التسلسل | طول القراءة | |
| RNA-Seq | جي إس إم 4116218 | SRR10261601 | Mbnl1KO_Thymus_1 | Mbnl1 بالضربة القاضية | ممثل 1 | الغدة الصعترية | نهاية مزدوجة | 100 نقطة أساس |
| جي إس إم 4116219 | SRR10261602 | Mbnl1KO_Thymus_2 | Mbnl1 بالضربة القاضية | ممثل 2 | الغدة الصعترية | نهاية مزدوجة | 100 نقطة أساس | |
| جي إس إم 4116220 | SRR10261603 | Mbnl1KO_Thymus_3 | Mbnl1 بالضربة القاضية | مندوب 3 | الغدة الصعترية | نهاية مزدوجة | 100 نقطة أساس | |
| جي إس إم 4116221 | SRR10261604 | WT_Thymus_1 | نوع البرية | ممثل 1 | الغدة الصعترية | نهاية مزدوجة | 100 نقطة أساس | |
| جي إس إم 4116222 | SRR10261605 | WT_Thymus_2 | نوع البرية | ممثل 2 | الغدة الصعترية | نهاية مزدوجة | 100 نقطة أساس | |
| جي إس إم 4116223 | SRR10261606 | WT_Thymus_3 | نوع البرية | مندوب 3 | الغدة الصعترية | نهاية مزدوجة | 100 نقطة أساس | |
| 3P-Seq | جي إس إم 1480973 | ريال1553129 | WT_1 | النوع البري (WT) | ممثل 1 | الخلايا الليفية الجنينية للفأر (MEFs) | نهاية واحدة | 40 نقطة أساس |
| جي إس إم 1480974 | ريال1553130 | WT_2 | النوع البري (WT) | ممثل 2 | الخلايا الليفية الجنينية للفأر (MEFs) | نهاية واحدة | 40 نقطة أساس | |
| جي إس إم 1480975 | SRR1553131 | DKO_1 | Mbnl 1/2 ضربة قاضية مزدوجة (DKO) | ممثل 1 | الخلايا الليفية الجنينية للفأر (MEFs) | نهاية واحدة | 40 نقطة أساس | |
| جي إس إم 1480976 | SRR1553132 | DKO_2 | Mbnl 1/2 ضربة قاضية مزدوجة (DKO) | ممثل 2 | الخلايا الليفية الجنينية للفأر (MEFs) | نهاية واحدة | 40 نقطة أساس | |
| جي إس إم 1480977 | ريال1553133 | DKOsiRNA_1 | Mbnl 1/2 ضربة قاضية مزدوجة مع Mbnl 3 siRNA (دينار كويتي) | ممثل 1 | الخلايا الليفية الجنينية للفأر (MEFs) | نهاية واحدة | 40 نقطة أساس | |
| جي إس إم 1480978 | ريال1553134 | DKOsiRNA_2 | Mbnl 1/2 ضربة قاضية مزدوجة مع Mbnl 3 siRNA (دينار كويتي) | ممثل 2 | الخلايا الليفية الجنينية للفأر (MEFs) | نهاية واحدة | 36 نقطة أساس | |
| جي إس إم 1480979 | ريال1553135 | DKONTsiRNA_1 | Mbnl 1/2 ضربة قاضية مزدوجة مع siRNA غير مستهدف (Ctrl) | ممثل 1 | الخلايا الليفية الجنينية للفأر (MEFs) | نهاية واحدة | 40 نقطة أساس | |
| جي إس إم 1480980 | SRR1553136 | DKONTsiRNA_2 | Mbnl 1/2 ضربة قاضية مزدوجة مع siRNA غير مستهدف (Ctrl) | ممثل 2 | الخلايا الليفية الجنينية للفأر (MEFs) | نهاية واحدة | 40 نقطة أساس |
الجدول 1. ملخص مجموعات بيانات RNA-Seq و PolyA-seq المستخدمة في التحليل.
Protocol
1. تركيب الأدوات وحزم R المستخدمة في التحليل
- Conda هو مدير حزم شائع ومرن يسمح بالتثبيت المريح للحزم مع تبعياتها عبر جميع الأنظمة الأساسية. استخدم "Anaconda" (مدير حزم conda) لتثبيت "conda" والذي يمكن استخدامه لتثبيت الأدوات / الحزم المطلوبة للتحليل.
- قم بتنزيل "Anaconda" وفقا لمتطلبات النظام من https://www.anaconda.com/products/individual#Downloads وقم بتثبيته باتباع المطالبات في برنامج التثبيت الرسومي. قم بتثبيت جميع الحزم المطلوبة باستخدام "conda" عن طريق كتابة ما يلي في سطر أوامر Linux.
conda install -c daler sratoolkit
conda install -c conda-forge parallel
conda install -c bioconda star bowtie fastqc rmats rmats2sashimiplot samtools fasterq-dump cutadapt bedtools deeptools - لتنزيل جميع حزم R المستخدمة في البروتوكول ، اكتب التعليمة البرمجية التالية في وحدة تحكم R (التي بدأت في سطر أوامر Linux بكتابة "R") أو وحدة تحكم Rstudio.
bioc_packages<- c("DEXSeq", "Rsubread", "EnhancedVolcano", "edgeR", "limma", "maser","GenomicRanges")
packages<- c("magrittr", "rtracklayer", "tidyverse", "openxlsx", "BiocManager")
#Install if not already installed
installed_packages<-packages%in% rownames(installed.packages())
installed_bioc_packages<-bioc_packages%in% rownames(installed.packages())
if(any(installed_packages==FALSE)) {
install.packages(packages[!installed_packages],dependencies=TRUE)
BiocManager::install(packages[!installed_bioc_packages], dependencies=TRUE)
}
ملاحظة: في هذا البروتوكول الحسابي ، سيتم إعطاء الأوامر إما كملفات R Notebook (ملفات ذات امتداد ". Rmd") ، ملفات رمز R (ملفات ذات امتداد ". R") ، أو البرامج النصية Linux Bash shell (الملفات ذات الامتداد ".sh"). يجب فتح ملفات R Notebook (Rmd) في RStudio باستخدام File| افتح ملف...، ثم يتم تشغيل أجزاء التعليمات البرمجية الفردية (التي قد تكون أوامر R أو أوامر Bash shell) بشكل تفاعلي بالنقر فوق السهم الأخضر في الجزء العلوي الأيمن. يمكن تشغيل ملفات التعليمات البرمجية R عن طريق الفتح في RStudio ، أو على سطر أوامر Linux عن طريق التقديم ب "Rscript" ، على سبيل المثال Rscript example.R. يتم تشغيل البرامج النصية Shell على سطر أوامر Linux عن طريق تمهيد البرنامج النصي بالأمر "sh" على سبيل المثال .sh example.sh.
2. تحليل الربط البديل (AS) باستخدام RNA-seq
- تنزيل البيانات ومعالجتها مسبقا
ملاحظة: تتوفر مقتطفات التعليمات البرمجية المشروحة أدناه في ملف التعليمات البرمجية التكميلية "AS_analysis_RNASeq.Rmd"، لاتباع الخطوات الفردية بشكل تفاعلي ، ويتم توفيرها أيضا كبرنامج نصي bash ليتم تشغيله دفعة واحدة على سطر أوامر Linux (ش downloading_data_preprocessing.sh).- تنزيل البيانات الأولية.
- قم بتنزيل البيانات الأولية من أرشيف قراءة التسلسل (SRA) باستخدام الأمر "الجلب المسبق" من مجموعة أدوات SRA (الإصدار 2.10.8)17. أعط معرفات انضمام SRA بالتسلسل في الأمر التالي لتنزيلها بالتوازي باستخدام الأداة المساعدة المتوازيةGNU 18. لتنزيل ملفات SRA لمعرفات الانضمام من SRR10261601 إلى SRR10261606 بالتوازي ، استخدم ما يلي في سطر أوامر Linux.
seq 10261601 10261606 | parallel prefetch SRR{} - استخرج ملفات fastq من الأرشيف باستخدام وظيفة "fastq-dump" من مجموعة أدوات SRA. استخدم غنو بالتوازي وأعط أسماء جميع ملفات SRA معا.
parallel -j 3 fastq-dump --gzip --skip-technical --read-filter pass --dumpbase --split-e --clip --origfmt {} ::: - قم بتنزيل الجينوم المرجعي والتعليقات التوضيحية للماوس (تجميع الجينوم GRCm39) من www.ensembl.org باستخدام ما يلي في سطر أوامر Linux.
wget -nv -O annotation.gtf.gz http://ftp.ensembl.org/pub/release-103/gtf/mus_musculus/Mus_musculus.GRCm39.103.gtf.gz \ && gunzip -f annotation.gtf.gz
wget -nv -O genome.fa.gz http://ftp.ensembl.org/pub/release-103/fasta/mus_musculus/dna/Mus_musculus.GRCm39.dna.primary_assembly.fa.gz \ && gunzip -f genome.fa.gz
GTF=$(readlink -f annotation.gtf)
GENOME=$(readlink -f genome.fa)
- قم بتنزيل البيانات الأولية من أرشيف قراءة التسلسل (SRA) باستخدام الأمر "الجلب المسبق" من مجموعة أدوات SRA (الإصدار 2.10.8)17. أعط معرفات انضمام SRA بالتسلسل في الأمر التالي لتنزيلها بالتوازي باستخدام الأداة المساعدة المتوازيةGNU 18. لتنزيل ملفات SRA لمعرفات الانضمام من SRR10261601 إلى SRR10261606 بالتوازي ، استخدم ما يلي في سطر أوامر Linux.
- تتم قراءة المعالجة المسبقة ورسم الخرائط لتجميع الجينوم
- مراقبة الجودة. قم بتقييم جودة القراءات الأولية باستخدام FASTQC (فحص جودة FASTQ v0.11.9)19. قم بإنشاء مجلد إخراج وتشغيل fastqc بالتوازي على ملفات fasta متعددة الإدخال. ستنشئ هذه الخطوة تقرير جودة لكل عينة. افحص التقارير للتأكد من أن جودة القراءات مقبولة قبل إجراء مزيد من التحليل. (راجع دليل المستخدم لفهم التقارير في https://www.bioinformatics.babraham.ac.uk/projects/fastqc/)
mkdir fastqc_out
parallel "fastqc {} -o fastqc_out" ::: $RAW_DATA/*.fastq.gz
ملاحظة: إذا لزم الأمر ، قم بإجراء تشذيب المحول باستخدام "cutadapt'20 أو "trimmomatic"21 لإزالة التسلسل إلى محولات جانبية ، والتي تختلف بناء على حجم جزء RNA وطول القراءة. في هذا التحليل ، تخطينا هذه الخطوة لأن جزء القراءات المتأثرة كان ضئيلا. - قراءة المحاذاة. تتضمن الخطوة التالية في المعالجة المسبقة تعيين القراءات إلى الجينوم المرجعي. أولا ، قم ببناء فهرس الجينوم المرجعي باستخدام وظيفة "genomeGenerate" ل STAR22 ثم قم بمحاذاة القراءات الأولية مع المرجع (بدلا من ذلك ، تتوفر الفهارس المعدة مسبقا من موقع STAR ويمكن استخدامها مباشرة للمحاذاة). قم بتشغيل الأوامر التالية في سطر أوامر Linux.
#Build STAR index
GDIR=STAR_indices
mkdir $GDIR
STAR --runMode genomeGenerate --genomeFastaFiles $GENOME --sjdbGTFfile $GTF --runThreadN 8 --genomeDir $GDIR
ODIR=results/mapping
mkdir -p $ODIR
#Align reads to the genome
for fq1 in $RAW_DATA/*R1.fastq.gz;
do
fq2=$(echo $fq1| sed 's/1.fastq.gz/2.fastq.gz/g');
OUTPUT=$(basename ${fq1}| sed 's/R1.fastq.gz//g');
STAR --genomeDir $GDIR \
--runThreadN 12 \
--readFilesCommand zcat \
--readFilesIn ${fq1}${fq2}\
--outFileNamePrefix $ODIR\/${OUTPUT} \
--outSAMtype BAM SortedByCoordinate \
--outSAMunmapped Within \
--outSAMattributes Standard
Done
ملاحظة: سيقوم محاذاة STAR بإنشاء وفرز ملفات BAM (خريطة المحاذاة الثنائية) لكل عينة بعد محاذاة القراءة. يجب فرز ملفات Bam قبل المتابعة إلى خطوات أخرى.
- مراقبة الجودة. قم بتقييم جودة القراءات الأولية باستخدام FASTQC (فحص جودة FASTQ v0.11.9)19. قم بإنشاء مجلد إخراج وتشغيل fastqc بالتوازي على ملفات fasta متعددة الإدخال. ستنشئ هذه الخطوة تقرير جودة لكل عينة. افحص التقارير للتأكد من أن جودة القراءات مقبولة قبل إجراء مزيد من التحليل. (راجع دليل المستخدم لفهم التقارير في https://www.bioinformatics.babraham.ac.uk/projects/fastqc/)
- تنزيل البيانات الأولية.
- إعداد شروح إكسون.
- قم بتشغيل ملف التعليمات البرمجية التكميلية "prepare_mm_exon_annotation. R" مع التعليق التوضيحي الذي تم تنزيله بتنسيق GTF (تنسيق نقل الجينات) لإعداد التعليقات التوضيحية. للتشغيل، اكتب ما يلي في سطر أوامر Linux.
Rscript prepare_mm_exon_annotation.R annotation.gtf
ملاحظة: يحتوي ملف GTF إدخالات إكسون متعددة لأشكال isoforms مختلفة. يستخدم هذا الملف "لطي" معرفات النصوص المتعددة لكل exon. إنها خطوة مهمة لتحديد صناديق عد إكسون.
- قم بتشغيل ملف التعليمات البرمجية التكميلية "prepare_mm_exon_annotation. R" مع التعليق التوضيحي الذي تم تنزيله بتنسيق GTF (تنسيق نقل الجينات) لإعداد التعليقات التوضيحية. للتشغيل، اكتب ما يلي في سطر أوامر Linux.
- العد يقرأ. الخطوة التالية هي حساب عدد القراءات المعينة إلى نصوص / إكسونات مختلفة. انظر الملف التكميلي: "AS_analysis_RNASeq.Rmd".
- تحميل المكتبات المطلوبة:
packages<- c("Rsubread","tidyverse", "magrittr", "EnhancedVolcano", "edgeR","openxlsx")
invisible(lapply(packages, library, character.only=TRUE)) - قم بتحميل ملف التعليقات التوضيحية الذي تمت معالجته والذي تم الحصول عليه من الخطوة السابقة (2.2).
load("mm_exon_anno.RData") - اقرأ جميع ملفات bam التي تم الحصول عليها في الخطوة 2.2.2 كإدخال ل "featureCounts" لحساب القرعات. اقرأ المجلد الذي يحتوي على ملفات bam عن طريق سرد كل ملف أولا من الدليل الذي ينتهي ب .bam. استخدم "featureCounts" من حزمة Rsubread التي تأخذ ملفات bam والتعليق التوضيحي GTF المعالج (المرجع) كمدخل لإنشاء مصفوفة من الأعداد المرتبطة بكل ميزة مع صفوف تمثل exons (الميزات) والأعمدة التي تمثل العينات.
countData <- dir("bams", pattern=".bam$", full.names=T) %>%
featureCounts(annot.ext=anno,
isGTFAnnotationFile=FALSE,
minMQS=0,useMetaFeatures=FALSE,
allowMultiOverlap=TRUE,
largestOverlap=TRUE,
countMultiMappingReads=FALSE,
primaryOnly=TRUE,
isPairedEnd=TRUE,
nthreads=12) - بعد ذلك ، قم بإجراء تصفية غير محددة لإزالة exons المعبر عنها بشكل متواضع ("غير محدد" يشير إلى أن معلومات الحالة التجريبية لا تستخدم في التصفية ، لتجنب تحيزات الاختيار). قم بتحويل البيانات من مقياس أولي إلى عدد لكل مليون (cpm) باستخدام وظيفة cpm من حزمة 'edgeR'23 واحتفظ ب exons بأعداد أكبر من عتبة قابلة للتعيين (يتم استخدام نسخة واحدة لكل ألف ظهور لمجموعة البيانات هذه) في ثلاث عينات على الأقل. أيضا إزالة الجينات مع إكسون واحد فقط.
# Non-specific filtering: Remove the exons with low counts
isexpr<- rownames(countData$counts)[rowSums(cpm(countData$counts)>1) >=3]
countData$counts<-countData$counts[rownames(countData$counts) %in%isexpr, ]
anno<-anno%>% filter(GeneID%in% rownames(countData$counts))
# Remove genes with only 1 site and NA in geneIDs
dn<-anno%>%group_by(GeneID)%>%summarise(nsites=n())%>% filter(nsites>1&!is.na(GeneID))
anno<-anno%>% filter(GeneID%in%dn$GeneID)
countData$counts<-countData$counts[rownames(countData$counts) %in%anno$GeneID, ]
ملاحظة: تحقق من المعلمات المطلوبة ل featureCounts عند استخدام بيانات مختلفة ، على سبيل المثال ، للقراءات أحادية الطرف ، قم بتعيين 'isPairedEnd = FALSE'. ارجع إلى دليل مستخدم RSubread لاختيار خيارات بياناتك، وراجع قسم المناقشة أدناه.
- تحميل المكتبات المطلوبة:
- الربط التفاضلي وتحليل استخدام إكسون. وصفنا بديلين لهذه الخطوة: DEXSeq و DiffSplice. إما يمكن استخدامها وإعطاء نتائج مماثلة. للتناسق ، حدد DEXSeq إذا كنت تفضل حزمة DESeq2 ل DGE واستخدم DiffSplice لتحليل DGE المستند إلى Limma. انظر الملف التكميلي: "AS_analysis_RNASeq.Rmd".
- استخدام حزمة DEXSeq لتحليل إكسون التفاضلي.
- قم بتحميل المكتبة وإنشاء جدول عينة لتحديد التصميم التجريبي.
library(DEXSeq)
sampleTable<-data.frame(row.names= c("Mbnl1KO_Thymus_1", "Mbnl1KO_Thymus_2", "Mbnl1KO_Thymus_3", "WT_Thymus_1", "WT_Thymus_2", "WT_Thymus_3"), condition= rep(c("Mbnl1_KO", "WT"),c(3,3)), libType= rep(c("paired-end")))
ملاحظة: يجب أن تكون أسماء الصفوف متناسقة مع أسماء ملفات bam المستخدمة من قبل featureCounts لحساب القروءات. يتكون جدول العينة من تفاصيل كل عينة والتي تشمل: نوع المكتبة والحالة. هذا مطلوب لتحديد التباين أو مجموعة الاختبار للكشف عن الاستخدام التفاضلي. - قم بإعداد ملف معلومات exon. معلومات Exon في شكل كائنات GRanges (النطاقات الجينومية) (https://bioconductor.org/packages/release/bioc/html/GenomicRanges.html) مطلوبة كمدخل لإنشاء كائن DEXSeq في الخطوة التالية. قم بمطابقة معرفات الجينات مع أعداد القراءة لإنشاء كائن exoninfo.
exoninfo<-anno[anno$GeneID%in% rownames(countData$counts),]
exoninfo<-GRanges(seqnames=anno$Chr,
ranges=IRanges(start=anno$Start, end=anno$End, width=anno$Width),strand=Rle(anno$Strand))
mcols(exoninfo)$TranscriptIDs<-anno$TranscriptIDs
mcols(exoninfo)$Ticker<-anno$Ticker
mcols(exoninfo)$ExonID<-anno$ExonID
mcols(exoninfo)$n<-anno$n
mcols(exoninfo)$GeneID<-anno$GeneID
transcripts_l= strsplit(exoninfo$TranscriptIDs, "\\,")
save(countData, sampleTable, exoninfo, transcripts_l, file="AS_countdata.RData") - قم بإنشاء كائن DEXSeq باستخدام وظيفة DEXSeqDataSet. يجمع كائن DEXSeq عدد مرات القراءة ومعلومات معالم exon ومعلومات العينة. استخدم أعداد القراءة التي تم إنشاؤها في الخطوة 3 ومعلومات exon التي تم الحصول عليها من الخطوة السابقة لإنشاء كائن DEXSeq من مصفوفة العد. تأخذ الوسيطة sampleData إدخال إطار بيانات يحدد العينات (وسماتها: نوع المكتبة وشرطها) ، ويستخدم "التصميم" sampleData لإنشاء مصفوفة تصميم للاختبار التفاضلي باستخدام تدوين صيغة النموذج. لاحظ أن مصطلح التفاعل المهم ، condition: exon ، يشير إلى أن جزء القراءات على جين يسقط على إكسون معين يعتمد على الحالة التجريبية ، أي أن هناك AS. راجع وثائق DEXSeq للحصول على وصف كامل لإعداد صيغة النموذج للتصميمات التجريبية الأكثر تعقيدا. للحصول على معلومات الميزة ، يلزم وجود معرفات exon والجينات المقابلة والنسخ.
dxd<-DEXSeqDataSet(countData$counts,sampleData=sampleTable, design=~sample+exon+condition:exon,featureID=exoninfo$ExonID,groupID=exoninfo$GeneID,featureRanges=exoninfo, transcripts=transcripts_l) - التطبيع وتقدير التشتت. بعد ذلك ، قم بإجراء التطبيع بين العينات وتقدير تباين البيانات ، بسبب كل من ضوضاء حساب بواسون من الطبيعة المنفصلة ل RNA-seq والتباين البيولوجي ، باستخدام الأوامر التالية.
dxd %<>% estimateSizeFactors %>% estimateDispersions %T>% plotDispEsts - اختبار الاستخدام التفاضلي. بعد تقدير التباين ، اختبر استخدام الإكسون التفاضلي لكل جين وقم بإنشاء النتائج.
dxd%<>%testForDEU%>%estimateExonFoldChanges(fitExpToVar=
"condition")#Estimate fold changes
dxr=DEXSeqResults(dxd) - تصور أحداث الربط للجينات المحددة باستخدام الأمر التالي.
plotDEXSeq(dxr,"Wnk1", displayTranscripts=TRUE, splicing=TRUE,legend
=TRUE,cex.axis=1.2,cex=1.3,lwd=2)
افحص ملف R Notebook "AS_analysis_RNASeq.Rmd" لإنشاء قطع إضافية للجينات ذات الأهمية ولإنشاء قطع أراضي بركانية عند عتبات مختلفة.
- قم بتحميل المكتبة وإنشاء جدول عينة لتحديد التصميم التجريبي.
- استخدام diffSplice من Limma لتحديد الربط التفاضلي. اتبع ملف R Notebook "AS_analysis_RNASeq.Rmd". تأكد من اتباع الخطوات 2.1-2.3 لإعداد ملفات الإدخال قبل المضي قدما.
- تحميل المكتبات
library(limma)
library(edgeR) - تصفية غير محددة. استخراج مصفوفة عدد القراءة التي تم الحصول عليها في 2.3. قم بإنشاء قائمة بالميزات باستخدام وظيفة "DGEList" من حزمة edgeR ، حيث تمثل الصفوف الجينات وتمثل الأعمدة العينات.
mycounts=countData$counts
#Change the rownames of the countdata to exon Ids instead of genes for unique rownames.
rownames(mycounts) = exoninfo$ExonID
dge<-DGEList(counts=mycounts)
#Filtering
isexpr<- rowSums(cpm(dge)>1) >=3
dge<-dge[isexpr,,keep.lib.sizes=FALSE]
#Extract the exon annotations for only transcripts meeting non-specific filter
exoninfo=anno%>% filter(ExonID%in% rownames(dge$counts))
#Convert the exoninfo into GRanges object
exoninfo1<-GRanges(seqnames=exoninfo$Chr,
ranges=IRanges(start=exoninfo$Start, end=exoninfo$End, width=exoninfo$Width),strand=Rle(exoninfo$Strand))
mcols(exoninfo1)$TranscriptIDs<-exoninfo$TranscriptIDs
mcols(exoninfo1)$Ticker<-exoninfo$Ticker
mcols(exoninfo1)$ExonID<-exoninfo$ExonID
mcols(exoninfo1)$n<-exoninfo$n
mcols(exoninfo1)$GeneID<-exoninfo$GeneID
transcripts_l= strsplit(exoninfo1$TranscriptIDs, "\\,")
ملاحظة: كخطوة تصفية غير محددة، تتم تصفية الأعداد بواسطة cpm < 1 في x من أصل n عينات، حيث x هو الحد الأدنى لعدد النسخ المتماثلة في أي شرط. n = 6 و x = 3 لبيانات المثال هذه. - تطبيع الأعداد عبر العينات ، باستخدام وظيفة "calcNormFactors" من حزمة "edgeR" باستخدام المتوسط المقتطع لقيم M (طريقة تسوية TMM)24 سيحسب عوامل القياس لضبط أحجام المكتبة.
dge<-calcNormFactors(dge) - استخدم sampleTable كما تم إنشاؤه في الخطوة 2.4.1.1 وقم بإنشاء مصفوفة التصميم. مصفوفة التصميم تميز التصميم. راجع دليل مستخدم Limma (https://www.bioconductor.org/packages/devel/bioc/vignettes/limma/inst/doc/usersguide.pdf) الفصلين 8 و 9 للحصول على تفاصيل حول مصفوفات التصميم للحصول على تصميمات تجريبية أكثر تقدما.
Treat<- factor(sampleTable$condition)
design<- model.matrix(~0+Treat)
colnames(design) <- levels(Treat) - تناسب نموذج خطي لكل إكسون. قم بتشغيل وظيفة "voom" لحزمة "limma" لمعالجة بيانات RNA-seq لتقدير التباين وإنشاء أوزان دقيقة لتصحيح ضوضاء عدد Poisson ، وتحويل الأعداد على مستوى exon إلى log2-counts لكل مليون (logCPM). ثم قم بتشغيل النمذجة الخطية باستخدام وظيفة "lmfit" لملاءمة النماذج الخطية مع بيانات التعبير لكل إكسون. احسب إحصائيات بايز التجريبية للنموذج المجهز باستخدام وظيفة "eBayes" للكشف عن تعبير إكسون التفاضلي. بعد ذلك ، حدد مصفوفة تباين للمقارنات التجريبية ذات الأهمية. استخدم "contrasts.fit" للحصول على المعاملات والأخطاء القياسية لكل زوج من المقارنة.
v<-voom(dge,design,plot=FALSE)
fit<-lmFit(v,design)
fit<-eBayes(fit)
colnames(fit)
cont.matrix<-makeContrasts(
Mbnl1_KO_WT=Mbnl1_KO-WT,
levels=design)
fit2<-contrasts.fit(fit,cont.matrix) - تحليل الربط التفاضلي. قم بتشغيل "diffSplice" على النموذج المجهز لاختبار الاختلافات في استخدام exon للجينات بين النوع البري والضربة القاضية واستكشاف النتائج الأعلى مرتبة باستخدام وظيفة "topSplice": test = "t" يعطي ترتيبا ل AS exons ، test = "simes" يعطي ترتيبا للجينات.
ex<-diffSplice(fit2,geneid=exoninfo$GeneID,exonid=exoninfo$ExonID)
ts<-topSplice(ex,n=Inf,FDR=0.1, test="t", sort.by="logFC")
tg<-topSplice(ex,n=Inf,FDR=0.1, test="simes") - التصور. ارسم النتائج باستخدام وظيفة "plotSplice" ، مع إعطاء الجين محل الاهتمام في حجة geneid. احفظ أفضل النتائج التي تم فرزها حسب السجل قم بطي التغيير إلى كائن وقم بإنشاء مخطط بركان لعرض exons.
plotSplice(ex,geneid="Wnk1", FDR=0.1)
#Volcano plot
EnhancedVolcano(ts,lab=ts$ExonID,selectLab= head((ts$ExonID),2000), xlab= bquote(~Log[2]~'fold change'), x='logFC', y='P.Value', title='Volcano Plot', subtitle='Mbnl1_KO vs WT (Limma_diffSplice)', FCcutoff=2, labSize=4,legendPosition="right", caption= bquote(~Log[2]~"Fold change cutoff, 2; FDR 10%"))
- تحميل المكتبات
- باستخدام rMATS
- تأكد من تثبيت أحدث إصدار من rMATS v4.1.1 (المعروف أيضا باسم rMATS turbo نظرا لانخفاض وقت المعالجة ومتطلبات الذاكرة الأقل) إما باستخدام conda أو github (https://github.com/Xinglab/rmats-turbo/releases/download/v4.1.1/rmats_turbo_v4_1_1.tar.gz) في دليل العمل. اتبع القسم 4.3 في "AS_analysis_RNASeq.Rmd".
- انتقل إلى المجلد الذي يحتوي على ملفات bam التي تم الحصول عليها بعد التعيين وقم بإعداد الملفات النصية ، كما هو مطلوب بواسطة rMATS ، للشرطين عن طريق نسخ اسم ملفات bam (جنبا إلى جنب مع المسار) مفصولة ب ','. يجب تشغيل الأوامر التالية في سطر أوامر Linux:
mkdir rMATS_analysis
cd bams/
ls -pd "$PWD"/*| grep "WT"| tr '\n'','> Wt.txt
ls -pd "$PWD"/*| grep "Mb"| tr '\n'','> KO.txt
mv *.txt ../rMATS_analysis - قم بتشغيل rmats.py باستخدام ملفي الإدخال اللذين تم إنشاؤهما في الخطوة السابقة ، جنبا إلى جنب مع ملف GTF الذي تم الحصول عليه في 2.1.1.3. سيؤدي هذا إلى إنشاء مجلد إخراج "rmats_out" يحتوي على ملفات نصية تصف الإحصائيات (قيم p ومستويات التضمين) لكل حدث ربط على حدة.
python rmats-turbo/rmats.py --b1 KO.txt --b2 Wt.txt --gtf annotation.gtf -t paired --readLength 50 --nthread 8 --od rmats_out/ --tmp rmats_tmp --task pos
ملاحظة: التعليق التوضيحي المرجعي في شكل ملف GTF مطلوب أيضا. تحقق من المعلمات إذا كانت البيانات أحادية الطرف ، وقم بتغيير الخيار -t وفقا لذلك. - استكشاف نتائج rMATS. استخدم حزمة الموصلات الحيوية "maser'25 لاستكشاف نتائج rMATS. قم بتحميل الملفات النصية ل Junction و exon counts (JCEC) في كائن "maser" وقم بتصفية النتيجة بناء على التغطية من خلال تضمين خمسة قراءات متوسطة على الأقل لكل حدث ربط.
library(maser)
mbnl1<-maser("/rmats_out/", c("WT","Mbnl1_KO"), ftype="JCEC")
#Filtering out events by coverage
mbnl1_filt<-filterByCoverage(mbnl1,avg_reads=5) - تصور نتائج rMATS. حدد أحداث الربط المهمة بمعدل الاكتشاف الخاطئ (FDR) 10٪ والحد الأدنى للتغيير بنسبة 10٪ في النسبة المئوية للتقسيم (deltaPSI) باستخدام وظيفة "topEvents" من حزمة "maser". بعد ذلك ، تحقق من أحداث الجينات للجينات الفردية ذات الأهمية (عينة الجين Wnk1) وارسم قيم PSI لكل حدث ربط لهذا الجين. قم بإنشاء مخطط بركان عن طريق تحديد نوع الحدث.
#Top splicing events at 10% FDR
mbnl1_top<-topEvents(mbnl1_filt,fdr=0.1, deltaPSI=0.1)
mbnl1_top
#Check the gene events for a particular gene
mbnl1_wnk1<-geneEvents(mbnl1_filt,geneS="Wnk1", fdr=0.1, deltaPSI=0.1)
maser::display(mbnl1_wnk1,"SE")
plotGenePSI(mbnl1_wnk1,type="SE", show_replicates
=TRUE)
volcano(mbnl1_filt,fdr=0.1, deltaPSI=0.1,type="SE")
+xlab("deltaPSI")+ylab("Log10 Adj. Pvalue")+ggtitle("Volcano Plot of exon skipping events") - قم بإنشاء مخططات Sashimi لنتائج أحداث الربط التي تم الحصول عليها باستخدام rMATS في شكل ملفات نصية باستخدام حزمة "rmats2shahimiplot". قم بتشغيل البرنامج النصي python في سطر أوامر Linux.
python ./src/rmats2sashimiplot/rmats2sashimiplot.py --b1 ../bams/WT_Thymus_1.bam,../bams/WT_Thymus_2.bam,../bams/WT_Thymus_3.bam --b2 ../bams/Mbnl1KO_Thymus_1.bam,../bams/Mbnl1KO_Thymus_2.bam,../bams/Mbnl1KO_Thymus_3.bam -t SE -e ../rMATS_analysis/rmats_out/SE.MATS.JC.txt --l1 WT --l2 Mbnl1_KO --exon_s 1 --intron_s 5 -o ../rMATS_analysis/rmats2shasmi_output
ملاحظة: يمكن أن تستغرق هذه العملية وقتا طويلا لأنها ستنشئ مؤامرة الساشيمي لجميع النتائج في ملف الأحداث. اختر أفضل النتائج (أسماء الجينات والإكسونات) كما هو معروض بواسطة وظيفة topEvents من "maser" وتصور مؤامرة الساشيمي المقابلة.
- استخدام حزمة DEXSeq لتحليل إكسون التفاضلي.
3. تحليل polyadenylation البديل (APA) باستخدام تسلسل نهاية 3 '
- تنزيل البيانات ومعالجتها مسبقا
ملاحظة: راجع ملف R Notebook التكميلي "APA_analysis_3PSeq_notebook. Rmd" للأوامر الكاملة لتنزيل البيانات وخطوات المعالجة المسبقة ، أو قم بتشغيل ملف bash التكميلي "APA_data_downloading_preprocessing.sh" في سطر أوامر Linux.- قم بتنزيل البيانات من SRA باستخدام معرفات الانضمام (1553129 إلى 1553136).
- تقليم المحولات وتكملة عكسية للحصول على تسلسل حبلا الإحساس.
ملاحظة: هذه الخطوة خاصة بمقايسة PolyA-seq المستخدمة. - تقرأ الخريطة لتجميع جينوم الماوس باستخدام مصفف ربطة العنق26.
- إعداد التعليقات التوضيحية لمواقع pA.
ملاحظة: تتم معالجة ملف التعليقات التوضيحية لموقع pA أولا باستخدام ملف R Notebook التكميلي "APA_analysis_3PSeq_notebook. Rmd" (2.1 - 2.6) ، ثم استخدام ملف bash "APA_annotation_preparation.sh".- قم بتنزيل التعليق التوضيحي لمواقع pA من قاعدة بيانات PolyASite 2.06.
- حدد التعليقات التوضيحية لموقع pA للاحتفاظ بمواقع pA للمنطقة غير المترجمة (UTR) ، والتي يتم شرحها ك Terminal Exon (TE) أو 1000 nt في اتجاه مجرى النهر من exon الطرفي المشروح (DS) لتحليل المصب.
- الحصول على قمم موقع pA. مرساة في كل موقع انشقاق pA ، وتصور متوسط تغطية القراءة باستخدام أدوات السرير والأدوات العميقة27,28. أظهرت النتائج أن قمم القراءات المعينة كانت مشتتة بشكل أساسي داخل ~ 60 نقطة أساس في المنبع لمواقع الانقسام (الشكل 5 والشكل التكميلي 5). لذلك ، تم توسيع إحداثيات مواقع pA من ملف التعليقات التوضيحية إلى 60 نقطة أساس في المنبع لمواقع الانقسام الخاصة بها. اعتمادا على بروتوكول التسلسل النهائي المحدد 3 'المستخدم ، ستحتاج هذه الخطوة إلى التحسين للمقايسات بخلاف PolyA-seq.
- يقرأ العد
- قم بإعداد ملف التعليقات التوضيحية لمواقع pA.
anno<- read.table(file= "flanking60added.pA_annotation.bed",
stringsAsFactors=FALSE, check.names=FALSE, header=FALSE, sep="")
colnames(anno) <- c("chrom", "chromStart", "chromEnd", "name", "score", "strand", "rep", "annotation", "gene_name", "gene_id")
anno<- dplyr::select(anno,name,chrom, chromStart,chromEnd, strand,gene_id,gene_name,rep)
colnames(anno) <- c("GeneID", "Chr", "Start", "End", "Strand", "Ensembl", "Symbol", "repID") - قم بتطبيق "featureCounts" للحصول على الأعداد الأولية. احفظ جدول العد كملف RData "APA_countData.Rdata" لتحليل APA باستخدام أدوات مختلفة.
countData<- dir("bamfiles", pattern="sorted.bam$", full.names=TRUE) %>%
# Read all bam files as input for featureCounts
featureCounts(annot.ext=anno, isGTFAnnotationFile= FALSE,minMQS=0,useMetaFeatures= TRUE,allowMultiOverlap=TRUE, largestOverlap= TRUE,strandSpecific=1, countMultiMappingReads =TRUE,primaryOnly= TRUE,isPairedEnd= FALSE,nthreads=12)%T>%
save(file="APA_countData.Rdata")
ملاحظة: كن واعيا لتغيير أي من المعلمات المدرجة في الدالة 'featureCounts'. قم بتعديل المعلمة "strandSpecific" للتأكد من توافقها مع اتجاه التسلسل لمقايسة التسلسل النهائي 3 'المستخدمة (تجريبيا ، فإن تصور البيانات في متصفح الجينوم على الجينات على خيوط زائد وناقص سيوضح ذلك). - تطبيق تصفية غير محددة ل countData. يمكن أن تؤدي التصفية إلى تحسين المتانة الإحصائية بشكل كبير في اختبارات استخدام موقع pA التفاضلية. أولا ، أزلنا تلك الجينات مع موقع pA واحد فقط ، والذي لا يمكن تحديد استخدام موقع pA بشكل تفاضلي. ثانيا ، نطبق ترشيحا غير محدد بناء على التغطية: يتم تصفية الأعداد بواسطة cpm أقل من 1 في x من عينات n ، حيث x هو الحد الأدنى لعدد النسخ المتماثلة في أي حالة. N = 8 و x = 2 لبيانات المثال هذه.
load(file= "APA_countData.Rdata")# Skip this step if already loaded
# Non-specific filtering: Remove the pA sites not differentially expressed in the samples
countData<-countData$counts%>%as.data.frame%>% .[rowSums(edgeR::cpm(.)>1) >=2, ]
anno%<>% .[.$GeneID%in% rownames(countData), ]
# Remove genes with only 1 site and NA in geneIDs
dnsites<-anno%>%group_by(Symbol)%>%summarise(nsites=n())%>% filter(nsites>1&!is.na(Symbol))
anno<-anno%>% filter(Symbol%in%dnsites$Symbol)
countData<-countData[rownames(countData) %in%anno$GeneID, ]
- قم بإعداد ملف التعليقات التوضيحية لمواقع pA.
- تحليل استخدام موقع البولي أدينيل التفاضلي باستخدام خطوط أنابيب DEXSeq و diffSplice.
- باستخدام حزمة DEXSeq
ملاحظة: نظرا لأنه لا يمكن تحديد مصفوفة تباين لخط أنابيب DEXSeq ، يجب إجراء تحليل APA التفاضلي لكل شرطين تجريبيين بشكل منفصل. يتم إجراء تحليل APA التفاضلي للحالة WT والحالة DKO كمثال لشرح الإجراء. الرجوع إلى الملف التكميلي "APA_analysis_3PSeq_notebook. رمد" لسير العمل خطوة بخطوة لهذا القسم وتحليل APA التفاضلي للتناقضات الأخرى.- قم بتحميل المكتبة وإنشاء جدول عينة لتحديد التصميم التجريبي.
c("DEXSeq", "GenomicRanges") %>% lapply(library, character.only=TRUE) %>%invisible
sampleTable1<- data.frame(row.names= c("WT_1","WT_2","DKO_1","DKO_2"),
condition= c(rep("WT", 2), rep("DKO", 2)),
libType= rep("single-end", 4)) - قم بإعداد ملف معلومات مواقع pA باستخدام حزمة الموصلات الحيوية GRanges.
# Prepare the GRanges object for DEXSeqDataSet object construction
PASinfo <- GRanges(seqnames = anno$Chr,
ranges = IRanges(start = anno$Start, end = anno$End),strand = Rle(anno$Strand))
mcols(PASinfo)$PASID<-anno$repID
mcols(PASinfo)$GeneEns<-anno$Ensembl
mcols(PASinfo)$GeneID<-anno$Symbol
# Prepare the new feature IDs, replace the strand information with letters to match the current pA site clusterID
new.featureID <- anno$Strand %>% as.character %>% replace(. %in% "+", "F") %>% replace(. %in% "-", "R") %>% paste0(as.character(anno$repID), .) - استخدم عدد مرات القراءة التي تم إنشاؤها في الخطوة 3.3 ومعلومات موقع pA التي تم الحصول عليها من الخطوة السابقة لإنشاء كائن DEXSeq.
# Select the read counts of the condition WT and DKO
countData1<- dplyr::select(countData, SRR1553129.sorted.bam, SRR1553130.sorted.bam, SRR1553131.sorted.bam, SRR1553132.sorted.bam)
# Rename the columns of countData using sample names in sampleTable
colnames(countData1) <- rownames(sampleTable1)
dxd1<-DEXSeqDataSet(countData=countData1,
sampleData=sampleTable1,
design=~sample+exon+condition:exon,
featureID=new.featureID,
groupID=anno$Symbol,
featureRanges=PASinfo) - حدد زوج التباين من خلال تحديد مستويات الظروف في كائن DEXSeq.
dxd1$condition<- factor(dxd1$condition, levels= c("WT", "DKO"))
# The contrast pair will be "DKO - WT" - التطبيع وتقدير التشتت. على غرار بيانات RNA-seq ، بالنسبة لبيانات التسلسل النهائي 3 بوصات ، قم بإجراء التطبيع بين العينات (متوسط النسب حسب العمود لكل عينة) باستخدام وظيفة "estimateSizeFactors" ، وتقدير تباين البيانات باستخدام وظيفة "estimateDispersions" ، ثم تصور نتيجة تقدير التشتت باستخدام وظيفة "plotDispEsts".
dxd1 %<>% estimateSizeFactors %>% estimateDispersions %T>% plotDispEsts - اختبار استخدام موقع pA التفاضلي لكل جين باستخدام الوظيفة "testForDEU" ، ثم تقدير تغيير أضعاف استخدام موقع pA باستخدام الوظيفة "estimateExonFoldChanges". تحقق من النتائج باستخدام الوظيفة "DEXSeqResults" وقم بتعيين "FDR < 10٪" كمعيار لمواقع pA التفاضلية بشكل كبير.
dxd1 %<>% testForDEU %>% estimateExonFoldChanges(fitExpToVar = "condition")
dxr1 <- DEXSeqResults(dxd1)
dxr1
mcols(dxr1)$description
table(dxr1$padj<0.1) # Check the number of differential pA sites (FDR < 0.1)
table(tapply(dxr1$padj<0.1, dxr1$groupID, any)) # Check the number of gene overlapped with differential pA site - تصور نتائج استخدام موقع pA التفاضلي باستخدام مخططات APA التفاضلية الناتجة عن الوظيفة "plotDEXSeq" ومخطط البركان بواسطة الوظيفة "EnhancedVolcano".
# Select the top 100 significant differential pA sites ranked by FDR
topdiff.PAS<- dxr1%>%as.data.frame%>%rownames_to_column%>%arrange(padj)%$%groupID[1:100]
# Apply plotDEXSeq for the visualization of differential polyA usage
plotDEXSeq(dxr1,"S100a7a", legend=TRUE, expression=FALSE,splicing=TRUE, cex.axis=1.2, cex=1.3,lwd=2)
# Apply perGeneQValue to check the top genes with differential polyA site usage
dxr1%<>% .[!is.na(.$padj), ]
dgene<- data.frame(perGeneQValue= perGeneQValue (dxr1)) %>%rownames_to_column("groupID")
dePAS_sig1<-dxr1%>% data.frame() %>%
dplyr::select(-matches("dispersion|stat|countData|genomicData"))%>%
inner_join(dgene)%>%arrange(perGeneQValue)%>%distinct()%>%
filter(padj<0.1)
# Apply EnhancedVolcano package to visualise differential polyA site usage
"EnhancedVolcano"%>% lapply(library, character.only=TRUE) %>%invisible
EnhancedVolcano(dePAS_sig1, lab=dePAS_sig1$groupID, x='log2fold_DKO_WT',
y='pvalue',title='Volcano Plot',subtitle='DKO vs WT',
FCcutoff=1,labSize=4, legendPosition="right",
caption= bquote(~Log[2]~"Fold change cutoff, 1; FDR 10%"))
- قم بتحميل المكتبة وإنشاء جدول عينة لتحديد التصميم التجريبي.
- باستخدام حزمة diffSplice. راجع ملف R Notebook التكميلي "APA_analysis_3PSeq_notebook. Rmd" لسير العمل خطوة بخطوة لهذا القسم.
- تحديد تباينات الاهتمام لتحليل استخدام pA التفاضلي.
ملاحظة: يجب تنفيذ هذه الخطوة بعد إنشاء ومعالجة كائن DGEList ، والذي تم تضمينه في ملف R Notebook "APA_analysis_3PSeq_notebook. رمد".contrast.matrix<-makeContrasts(DKO_vs_WT=DKO-
WT,Ctrl_vs_DKO=Ctrl-DKO,
KD_vs_Ctrl=KD-Ctrl,KD_vs_DKO=KD-DKO,levels=design)
fit2<-fit%>%contrasts.fit(contrast.matrix)%>%eBayes
summary(decideTests(fit2))
ex<-diffSplice(fit2,geneid=anno$Symbol,exonid=new.featureID)
topSplice(ex) #Check the top significant results with topSplice - تصور نتيجة تناقضات الاهتمام (هنا "DKO - WT") باستخدام مخططات APA التفاضلية بواسطة الوظيفة "plotSplice" ومؤامرات البركان مع الوظيفة "EnhancedVolcano". راجع ملف R Notebook "APA_analysis_3PSeq_notebook. Rmd" 4.2.7 - 4.2.9 لتصور أزواج التباين الأخرى.
sig1<-topSplice(ex,n=Inf,FDR=0.1,coef=1, test="t", sort.by="logFC")
sig1.genes<-topSplice(ex,n=Inf,FDR=0.1,coef=1, test="simes")
plotSplice(ex, coef=1,geneid="S100a7a", FDR = 0.1)
plotSplice(ex,coef=1,geneid="Tpm1", FDR = 0.1)
plotSplice(ex,coef=1,geneid="Smc6", FDR = 0.1)
EnhancedVolcano(sig1, lab=sig1$GeneID,xlab= bquote(~Log[2]~'fold change'),
x='logFC', y='P.Value', title='Volcano Plot', subtitle='DKO vs WT',
FCcutoff=1, labSize=6, legendPosition="right")
- تحديد تباينات الاهتمام لتحليل استخدام pA التفاضلي.
- باستخدام حزمة DEXSeq
النتائج
بعد تشغيل سير العمل خطوة بخطوة أعلاه ، تكون مخرجات تحليل AS و APA والنتائج التمثيلية في شكل جداول ومخططات بيانات ، يتم إنشاؤها على النحو التالي.
مثل:
الناتج الرئيسي لتحليل AS (الجدول التكميلي 1 ل diffSplice; الجدول 2 ل DEXSeq) عبارة عن قائمة بالإكسونات التي تظهر الاستخدام التفاضلي عبر الظروف ، وقائمة بالجينات التي تظهر نشاط الربط الكلي الكبير لواحد أو أكثر من الإكسونات المكونة لها ، مرتبة حسب الدلالة الإحصائية. الجدول التكميلي 1 ، علامة التبويب 2 تظهر exons كبيرة ، مع أعمدة تظهر FC التفاضلي ل exon مقابل الباقي ، وقيمة p غير المعدلة لكل exon ، والقيمة p المعدلة (تصحيح Benjamini-Hockberg). سيعطي العتبة على قيم p المعدلة مجموعة من exons مع FDR المحدد. الجدول التكميلي 1 ، علامة التبويب 3 تظهر قائمة مرتبة من الجينات التي تظهر أهمية نشاط الربط الكلي ، مع عمود يوضح القيمة p المعدلة على مستوى الجينات المحسوبة باستخدام طريقة Simes. يتم عرض بيانات مماثلة في الجدول 2 ل DEXSeq. يوضح الشكل التكميلي 1 والشكل التكميلي 2 نمط الربط التفاضلي في جينات Mbnl1 و Tcf7 و Lef1 التي تم التحقق من صحتها تجريبيا في المقالة المنشورة المقدمة مع البيانات15. أظهر المؤلفون التحقق التجريبي من خمسة جينات - Mbnl1 و Mbnl2 و Lef1 و Tcf7 و Ncor2. اكتشف نهجنا فطائر الربط التفاضلية في كل هذه الجينات. نقدم هنا مستويات FDR لكل جين باستخدام DEXSeq و diffSplice و rMATS على التوالي كما تم الحصول عليها في الجداول التكميلية 1-3: Mbnl1 (0 ، 6.6E-61 ، 0) ، Mbnl2 (0،0.18،0) ، Lef1 (1.4E-10 ، 1.3E-04 ، 0) ، Tcf7 (0 ، 1.1E-6 ، 0) و Ncor2 (9.2E-11 ، 1.6E-06 ، 0).
يعرض الشكل 2 مقارنة بين المخرجات التي تم الحصول عليها من ثلاث أدوات مختلفة ويوضح أنماط الربط البديلة في جين Wnk1. تظهر مخططات البركان في الشكل 2A (diffSplice) والشكل 2B (DEXSeq). يتم عرض ثلاثة جينات إضافية عالية التصنيف في الشكل التكميلي 1 (diffSplice) والشكل التكميلي 2 (DEXSeq). يظهر المحور ص دلالة إحصائية (-log10 قيم P) ويظهر المحور السيني حجم التأثير (تغيير الطي). تشير الجينات الموجودة في الأرباع العلوية اليسرى أو اليمنى إلى FC كبير وأدلة إحصائية قوية على وجود اختلافات حقيقية.

الشكل 2. مقارنة نتائج الربط البديلة التي تم الحصول عليها من diffSplice و DEXSeq و rMATS. |
(A) مخطط بركان (يسار) ل RNA-Seq من تحليل Limma diffSplice: يظهر المحور x تغير طية logon exon ؛ يظهر المحور ص -log10 قيمة p. كل نقطة تقابل إكسون. خط متقطع أفقي عند قيمة p = 1E-5 ؛ خطوط متقطعة عمودية عند تغيير أضعاف (FC). تظهر الإكسونات الحمراء أهمية كبيرة ودلالة إحصائية. مخطط الربط التفاضلي (يمين): يتم عرض أنماط الربط لمثال الجين Wnk1 حيث يظهر المحور السيني معرف exon لكل نسخة ؛ يظهر المحور ص تغير طية السجل النسبي ل exon (الفرق بين logFC الخاص ب exon والسجل الكلي لجميع exons الأخرى). تظهر Exons المميزة باللون الأحمر تعبيرا تفاضليا ذا دلالة إحصائية (FDR < 0.1).
(ب) مخطط البركان (يسار) واستخدام إكسون التفاضلي (يمين) ل RNA-Seq تم الحصول عليها من تحليل DEXSeq. يظهر جين Wnk1 استخداما تفاضليا كبيرا للإكسونات بين ضربة قاضية WT و Mbnl1 مظللة باللون الوردي ، والتي تتوافق مع نفس الإكسونات التفاضلية في (A).
(ج) مخطط البركان (يسار) ومخطط الساشيمي (يمين) ل Wnk1 تم الحصول عليها من تحليل rMATS. مخطط بركان يصور حدثا كبيرا تم تخطيه (كاسيت) إكسون (SE) في النوع البري مقارنة بالضربة القاضية عند 10٪ FDR مع تغير في النسبة المئوية المقسمة في قيم (PSI أو ΔΨ) > 0.1. يظهر المحور السيني التغيير في قيم PSI عبر الظروف ، ويعرض المحور ص قيمة P للسجل. يظهر مخطط الساشيمي حدث إكسون تم تخطيه في جين Wnk1 ، يتوافق مع إكسون تفاضلي كبير في (A) و (B). يمثل كل صف عينة RNA-Seq: ثلاث نسخ مكررة من النوع البري و Mbnl1 بالضربة القاضية. يظهر الارتفاع تغطية القراءة في RPKM وتصور الأقواس المتصلة قراءات التقاطع عبر exons. يتم عرض الأشكال البديلة لنموذج الجينات المشروحة في الجزء السفلي من قطعة الأرض. توضح اللوحة السفلية من C قراءات التقاطع المستخدمة لحساب إحصائية PSI.
د: مخطط فن الذي يقارن عدد الإكسونات التفاضلية الهامة التي حصلت عليها الطرق المختلفة. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
الشكل 2 A (اللوحة اليمنى) يعرض عرضا تخطيطيا لاختلافات exon لأحد الجينات الأعلى مرتبة ، ويظهر logFC على المحور y ورقم exon على المحور x. يوضح هذا المثال إكسون كاسيت متغير بين شروط الجين Wnk1. يظهر مخطط استخدام إكسون التفاضلي من DEXSeq دليلا على الربط التفاضلي في خمسة مواقع إكسون بالقرب من Wnk1.6.45. من المحتمل أن يتم تقسيم الإكسونات المميزة باللون الوردي في عينات Mbnl1 KO مقارنة ب WT. هذه الإكسونات مكملة للنتائج التي تم الحصول عليها بواسطة diffSplice والتي تظهر نمطا مشابها في الموقع الجينومي المحدد. يتم عرض المزيد من الأمثلة في الشكل التكميلي 1 والشكل التكميلي 2. يمكن تقديم عرض أكثر تفصيلا لتأكيد النتائج المثيرة للاهتمام من خلال مقارنة مسارات التغطية (التذبذب) في RPM (يقرأ لكل مليون) وحدة في UCSC (جامعة سانتا كروز) أو IGV (عارض الجينوم التكاملي) متصفحات الجينوم (غير معروضة) ، جنبا إلى جنب مع الارتباط البصري مع المسارات الأخرى ذات الأهمية ، مثل نماذج الجينات المعروفة ، والحفظ ، وغيرها من المقايسات على مستوى الجينوم.
يسرد جدول إخراج rMATS أحداث الربط البديلة المهمة المصنفة حسب النوع (الجدول التكميلي 3). يوضح الشكل 2C قطعة بركان من الجينات التي هي مقسمة بديلة ، مع قياس حجم التأثير من خلال إحصائية "النسبة المئوية المقسمة" (PSI أو ΔΨ) التفاضلية البالغة11.
يشير PSI إلى النسبة المئوية للقراءات المتسقة مع تضمين إكسون كاسيت (أي تعيين القراءات إلى إكسون الكاسيت نفسه أو قراءات التقاطع المتداخلة مع إكسون) مقارنة بالقراءات المتوافقة مع استبعاد إكسون ، أي يقرأ التقاطع عبر إكسونات المنبع والمصب المجاورة (اللوحة السفلية من الشكل 2 ج). تظهر اللوحة اليمنى من الشكل 2C مخطط الساشيمي لجين Wnk1 مع حدث الربط التفاضلي المتراكب على مسارات التغطية للجين ، مع تخطي إكسون في Mbnl1 KO. تظهر الأقواس التي تربط exons عدد قراءات الوصلات (تقرأ عبور إنترون مقسم). تظهر علامات التبويب المختلفة في الجدول التكميلي 3 قراءات مهمة لكل نوع من الأحداث التي تمتد عبر التقاطعات مع حدود exon (أعداد الوصلات وأعداد exon (JCEC)). يقارن الشكل 2D الإكسونات المقسمة تفاضليا المهمة التي اكتشفتها الأدوات الثلاث.

الشكل 3. أحداث الربط البديلة المكتسبة عن طريق تحليل rMATS. أ) أنواع أحداث AS. تم تكييف هذا الشكل من وثائق rMATS11 التي تشرح حدث الربط مع exons التأسيسية والمقسمة بدلا من ذلك. المسمى برقم كل حدث في FDR 10٪. ب) مؤامرة الساشيمي من جين Add3 تظهر تخطي إكسون (SE). ج) مؤامرة الساشيمي لجين Baz2b الذي يعرض موقع لصق البديل 5 '(A5SS). د) مؤامرة الساشيمي لجين Lsm14b الذي يعرض موقع لصق البديل 3 '(A3SS). ه) مؤامرة الساشيمي لجين Mta1 الذي يعرض إكسونات حصرية متبادلة (MXE). F) مؤامرة الساشيمي من جين Arpp21 تظهر intron المحتفظ به (RI). تمثل الصفوف الحمراء ثلاث نسخ متماثلة من النوع البري وتمثل الصفوف البرتقالية النسخ المتماثلة للضربة القاضية Mbnl1. يتوافق المحور السيني مع الإحداثيات الجينومية ومعلومات الشريط ، ويظهر المحور ص التغطية في RPKM. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
يوضح الشكل 3 أنواع أحداث الربط SE و A5SS و A3SS و MXE و RI بمساعدة مخططات الساشيمي لأهم الجينات لتلك الأحداث. عند مقارنة النسخ المتماثلة الثلاثة لكل من WT و Mbnl1_KO ، تم اكتشاف ما مجموعه 1272 حدثا SE و 130 A5SS و 116 A3SS و 215 MXE و 313 حدث RI عند FDR 10٪. توضح مؤامرة الساشيمي نوع الحدث باستخدام الجينات العليا كمثال.
ابا:
يشبه الناتج من تحليل APA تحليل AS على مستوى exon. يتم توفير جدول للجينات العليا مرتبة حسب نشاط APA التفاضلي في 3'UTR (الجدول التكميلي 4 والجدول التكميلي 5). يوضح الشكل 4A مخططات البراكين للجينات عن طريق نشاط APA التفاضلي في 3'UTRs التي تم إنشاؤها باستخدام diffSplice و DEXSeq بشكل منفصل. يعرض الشكل 4B مخطط Venn الذي يقارن نتائج استخدام موقع pA التفاضلية بشكل كبير التي تم الحصول عليها من خطوط أنابيب مختلفة. يوضح الشكلان 4C و 4D التمثيل التخطيطي لاستخدام موقع pA التفاضلي في 3'UTR للجين Fosl2 (الشكل 4C) وبابولا (الشكل 4D) الذي تم إنشاؤه باستخدام كل من diffSplice و DEXSeq ، والتي تم التحقق من صحتها تجريبيا لإظهار تحول كبير من البعيد إلى القريب (Fosl2) وتحول كبير من القريب إلى البعيد (Papola) لاستخدام موقع pA في DKO ، على التوالي. يتم عرض المزيد من الأمثلة في الشكل التكميلي 3 والشكل التكميلي 4.

الشكل 4. مؤامرات polyadenylation البديلة بواسطة diffSplice و DEXSeq. أ) مخططات بركان لبيانات PolyA-seq التي تم إنشاؤها باستخدام diffSplice و DEXSeq. يظهر المحور X تغيير أضعاف موقع السجل pA ؛ يظهر المحور ص -log10 قيمة p. كل نقطة تتوافق مع موقع pA. خط متقطع أفقي عند قيمة p = 1E-5 ؛ خطوط متقطعة عمودية في 2 أضعاف FC. تظهر الإكسونات الحمراء أهمية كبيرة ودلالة إحصائية. ب) مخطط Venn الذي يقارن نتائج استخدام موقع pA التفاضلية الهامة التي تم الحصول عليها من خطوط أنابيب مختلفة. C-D) مخططات APA التفاضلية التي تم إنشاؤها باستخدام diffSplice و DEXSeq تظهر مواقع pA القريبة والداخلية والبعيدة لجين Fosl2 و Papola . يتم إنشاء الأرقام بنفس وظيفة الشكل 2 (ب) ولكن مع استبدال مواقع pA بالإكسونات. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
الشكل 5 عبارة عن مخطط تشخيصي لتأكيد توزيع القراءة المتوقع حول مواقع انقسام pA المشروحة لمقايسة PolyA-seq المستخدمة. يظهر متوسط التغطية في المناطق المحيطة الراسية في مواقع انقسام pA المعروفة على مستوى الجينوم. في هذه الحالة ، يتم تصور التراكم المتوقع للقراءات في المنبع للمواقع. يتم عرض توزيعات القراءة الراسية في مواقع pA لجميع عينات PolyA-seq في الشكل التكميلي 5.

الشكل 5. يعني مؤامرة التغطية حول مواقع الانقسام pA. يتم عرض موقع الانقسام لبيانات PolyA-seq بواسطة خط متقطع عمودي. يظهر المحور X موضع القاعدة بالنسبة لمواقع الانقسام pA ، حتى 100 نيوكليوتيد في المنبع والمصب ؛ يظهر المحور ص متوسط تغطية القراءة على جميع مواقع الانقسام pA ، والتي تم تسويتها حسب حجم المكتبة في CPM. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
يمكن مقارنة نتائج APA التفاضلية للتباينات المختلفة الناتجة عن نفس خط الأنابيب والتحقق منها من خلال تصور تغطية القراءة لمواقع pA التفاضلية التمثيلية بشكل كبير في متصفح الجينوم. الشكل 6A هو مخطط Venn الذي يقارن استخدام موقع pA التفاضلي بشكل كبير للتناقضات المختلفة المكتسبة من diffSplice. الشكل 6B-D هي لقطات IGV لتغطية القراءة في مواقع pA للجينات المختلفة ، والتي تظهر الأنماط المتسقة مع تلك المكتشفة في تحليل APA باستخدام diffSplice. يتحقق الشكل 6B من صحة التحول القريب إلى البعيد الكبير لاستخدام موقع pA للجين Paip2 ، والذي يتم اكتشافه بشكل فريد في التباين DKO مقابل WT ، ولكن ليس في تباينين آخرين KD مقابل WT ، و Ctr مقابل WT. يتحقق الشكل 6C من صحة التحول الكبير البعيد إلى القريب لاستخدام موقع pA للجين Ccl2 المكتشف بشكل فريد في التباين KD مقابل WT ، بينما يتحقق الشكل 6D من صحة استخدام pA التفاضلي الكبير لجميع التباينات للجين Cacna2d1.
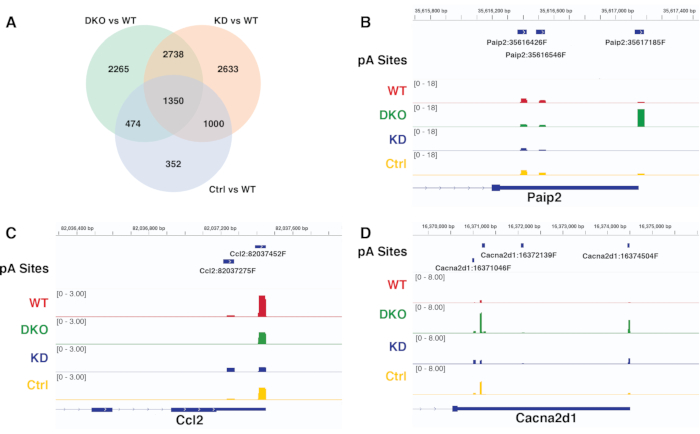
الشكل 6. مقارنة التباين والتحقق من نتائج الاختلافات. أ) مخطط Venn يقارن نتائج استخدام موقع pA التفاضلية الهامة للتناقضات المختلفة المكتسبة من diffSplice. ب-د) لقطة IGV تصور pA قمم تغطية الجينات Paip2 و Ccl2 و Cacna2d1 عبر الظروف. يمثل كل مسار تغطية القراءة في حالة معينة. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
الشكل التكميلي 1. تحليل RNA-Seq للربط التفاضلي مع Limma diffSplice. (أ) قطعة أرض بركان ل RNA-Seq من تحليل Limma diffSplice: يظهر المحور x تغير طية logon exon ؛ يظهر المحور ص -log10 قيمة p. كل نقطة تقابل إكسون. خط متقطع أفقي عند قيمة p = 1E-5 ؛ خطوط متقطعة عمودية عند تغيير مزدوج (FC). تظهر الإكسونات الحمراء أهمية كبيرة ودلالة إحصائية. (B-D) قطع الربط التفاضلية: يتم عرض أنماط الربط على سبيل المثال الجينات Mbnl1 و Tcf7 و Lef1 ، على التوالي ، حيث يظهر المحور السيني معرف exon لكل نسخة. يظهر المحور ص تغير طية السجل النسبي ل exon (الفرق بين logFC الخاص ب exon والسجل الكلي لجميع exons الأخرى). تظهر Exons المميزة باللون الأحمر تعبيرا تفاضليا ذا دلالة إحصائية (FDR < 0.1). الرجاء الضغط هنا لتنزيل هذا الملف.
الشكل التكميلي 2. تحليل RNA-Seq لاستخدام إكسون التفاضلي مع DEXSeq. (أ) قطعة أرض البركان. (B-D) استخدام إكسون التفاضلي من RNA-Seq التي تم الحصول عليها من تحليل DEXSeq. تظهر الجينات Mbnl1 و Tcf7 و Lef1 ، على التوالي ، استخداما تفاضليا كبيرا للإكسونات بين WT و Mbnl1 المظللة باللون الوردي ، والتي تتوافق مع نفس الإكسونات التفاضلية في الشكل التكميلي 1. الرجاء الضغط هنا لتنزيل هذا الملف.
الشكل التكميلي 3. مؤامرات polyadenylation البديلة بواسطة diffSplice. أ) مخططات البركان لبيانات PolyA-seq التي تم إنشاؤها باستخدام diffSplice في ثلاثة أزواج تباين تم الحصول عليها من بيانات الماوس PolyA-seq ، بما في ذلك الضربة القاضية المزدوجة (DKO) مقابل النوع البري (WT) ، والضربة القاضية (KD) مقابل WT ، والتحكم (Ctrl) مقابل WT. يظهر المحور X تغيير أضعاف موقع log pA ؛ يظهر المحور ص -log10 قيمة p. كل نقطة تتوافق مع موقع pA. خط متقطع أفقي عند قيمة p = 1E-5 ؛ خطوط متقطعة عمودية في 2 أضعاف FC. تظهر مواقع PA الحمراء أهمية كبيرة ودلالة إحصائية. ب) مخططات APA التفاضلية التي تم إنشاؤها باستخدام diffSplice والتي تظهر مواقع pA القريبة والداخلية والبعيدة للجينات ذات التصنيف العالي S100a7a و Tpm1 و Smc6. الرجاء الضغط هنا لتنزيل هذا الملف.
الشكل التكميلي 4. تحليل استخدام pA التفاضلي بواسطة خط أنابيب DEXSeq. أ) مخططات بركان لبيانات PolyA-seq التي تم إنشاؤها باستخدام DEXSeq في ثلاثة أزواج تم الحصول عليها من بيانات PolyA-seq للماوس ، بما في ذلك الضربة القاضية المزدوجة (DKO) مقابل النوع البري (WT) ، والضربة القاضية (KD) مقابل WT ، والتحكم (Ctrl) مقابل WT. يظهر المحور X تغيير طي موقع log pA ؛ يظهر المحور ص -log10 قيمة p. كل نقطة تتوافق مع موقع pA. خط متقطع أفقي عند قيمة p = 1E-5 ؛ خطوط متقطعة عمودية في 2 أضعاف FC. تظهر مواقع PA الحمراء أهمية كبيرة ودلالة إحصائية. ب) مخططات APA التفاضلية التي تم إنشاؤها باستخدام DEXSeq والتي تظهر مواقع pA القريبة والداخلية والبعيدة للجينات ذات التصنيف العالي S100a7a و Tpm1 و Smc6. الرجاء الضغط هنا لتنزيل هذا الملف.
الشكل التكميلي 5. يعني مخطط التغطية والخرائط الحرارية حول مواقع الانقسام pA. تظهر التغطية لأربعة شروط، مع عرض الجينات على الخيوط الأمامية والخلفية بشكل منفصل. يظهر المحور X موضع القاعدة بالنسبة لمواقع الانقسام pA ، حتى 100 نيوكليوتيد في المنبع والمصب ؛ يشير المحور ص إلى متوسط التغطية في مواضع القاعدة النسبية المقابلة عبر جميع مواقع الانقسام pA. توفر خرائط الحرارة عرضا بديلا ، حيث يظهر كل موقع انشقاق pA كصف منفصل على المحور السيني ، مرتبة حسب التغطية. تظهر الكثافة تغطية القراءة (انظر وسيلة الإيضاح). الرجاء الضغط هنا لتنزيل هذا الملف.
الجدول التكميلي 1. diffSplice الناتج من تحليل AS. تحدد علامة التبويب الأولى أسماء الأعمدة للمخرجات الأصلية المعروضة في علامتي التبويب الثانية (مستوى exon) والثالثة (مستوى الجينات). الرجاء الضغط هنا لتحميل هذا الجدول.
الجدول التكميلي 2. إخراج DEXSeq لتحليل AS. تحدد علامة التبويب الأولى أسماء الأعمدة للإخراج الأصلي المقدم في علامات التبويب الثانية (مستوى exon) والثالثة (مستوى الجينات). الرجاء الضغط هنا لتحميل هذا الجدول.
الجدول التكميلي 3. ناتج rMATS لتحليل AS. تحدد علامة التبويب الأولى أسماء الأعمدة لملف الملخص (علامة التبويب 2) وملفات JCEC لكل حدث (علامة التبويب 3-7). الرجاء الضغط هنا لتحميل هذا الجدول.
الجدول التكميلي 4. diffSplice الناتج من تحليل APA. تحدد علامة التبويب الأولى أسماء الأعمدة للإخراج الأصلي المقدم في علامتي التبويب الثانية (على مستوى موقع pA) والثالثة (مستوى الجينات). الرجاء الضغط هنا لتحميل هذا الجدول.
الجدول التكميلي 5. ناتج DEXSeq لتحليل APA. تحدد علامة التبويب الأولى أسماء الأعمدة للإخراج الأصلي المقدم في علامتي التبويب الثانية (على مستوى موقع pA) والثالثة (مستوى الجينات). الرجاء الضغط هنا لتحميل هذا الجدول.
الجدول التكميلي 6. ملخص لعدد الإكسونات التي تم تغييرها بشكل كبير لمواقع AS و pA ل APA. الرجاء الضغط هنا لتحميل هذا الجدول.
الجدول التكميلي 7. ملخص للأدوات والحزم المستخدمة في تحليل AS/APA. الرجاء الضغط هنا لتحميل هذا الجدول.
Discussion
في هذه الدراسة ، قمنا بتقييم النهج القائمة على exon والقائمة على الأحداث للكشف عن AS و APA في بيانات تسلسل RNA-Seq السائبة و 3 'التسلسل النهائي. تنتج مناهج AS القائمة على exon كلا من قائمة الإكسونات المعبر عنها بشكل تفاضلي وترتيب على مستوى الجينات مرتبة حسب الأهمية الإحصائية لنشاط الربط التفاضلي على مستوى الجينات الإجمالي (الجداول 1-2 ، 4-5). بالنسبة لحزمة diffSplice ، يتم تحديد الاستخدام التفاضلي عن طريق تركيب نماذج خطية مرجحة على مستوى exon لتقدير تغير طية اللوغاريتم التفاضلية ل exon مقابل متوسط تغير طية اللوغاريتم للإكسونات الأخرى داخل نفس الجين (يسمى لكل exon FC). يتم حساب الدلالة الإحصائية على مستوى الجينات من خلال الجمع بين اختبارات الدلالة الفردية على مستوى إكسون في اختبار جيني بواسطة طريقة سايمز10. يمكن لاحقا استخدام هذا الترتيب حسب نشاط الربط التفاضلي على مستوى الجينات لإجراء تحليل إثراء مجموعة الجينات (GSEA) للمسارات الرئيسية المعنية10. تستخدم DEXSeq استراتيجية مماثلة ، من خلال تركيب نموذج خطي معمم لقياس استخدام إكسون التفاضلي ، على الرغم من اختلافها في خطوات معينة مثل التصفية والتطبيع وتقدير التشتت. عند مقارنة أفضل 500 إكسون مصنف يظهر نشاط AS و APA باستخدام DEXSeq و DiffSplice ، وجدنا تداخلا بين 310 مواقع exons و 300 pA ، على التوالي ، مما يدل على توافق النهجين القائمين على exon ، والذي تم توضيحه أيضا في دراسة سابقة 29. يوصى باستخدام مزيج من كل من exon القائم (إما DEXSeq أو diffSplice) والنهج القائم على الأحداث للكشف الشامل وتصنيف AS. بالنسبة إلى APA ، يمكن للمستخدمين اختيار إما DEXSeq أو diffSplice: لقد ثبت أن كلتا الطريقتين تؤديان أداء جيدا عبر مجموعة واسعة من تجارب النسخ29.
عند إعداد مكتبة RNA-seq لتحليل AS ، من المهم استخدام بروتوكول RNA-seq السائب الخاص بالشريط8 ، حيث يتداخل جزء كبير من الجينات في جينومات الفقاريات على خيوط مختلفة ، والبروتوكول غير الخاص بالشريط غير قادر على التمييز بين هذه المناطق المتداخلة ، مما يربك الكشف النهائي عن exon. هناك اعتبار آخر هو عمق القراءة ، حيث تتطلب تحليلات الربط تسلسلا أعمق من DGE ، على سبيل المثال 30-60 مليون قراءة لكل عينة ، مقابل 5-25 مليون قراءة لكل عينة ل DGE (https://sapac.support.illumina.com/bulletins/2017/04/considerations-for-rna-seq-read-length-and-coverage-.html). تدعم جميع الأدوات الموضحة في البروتوكول كلا من بيانات التسلسل أحادية الطرف والطرف المزدوج. إذا تم استخدام التعليقات التوضيحية الجينية المعروفة فقط للكشف عن قراءات الوصلات ، فيمكن استخدام قراءات أقصر أحادية النهاية (≥ 50 نقطة أساس) ، على الرغم من أن الكشف الجديد عن تقاطعات لصق جديدة يستفيد من النهاية المزدوجة والأطول (≥ 100 نقطة أساس) يقرأ30,31. يعتمد اختيار بروتوكول استخراج الحمض النووي الريبي - إما اختيار polyA أو استنفاد rRNA - على جودة الحمض النووي الريبي والسؤال التجريبي - انظر31 للمناقشة. اعتمادا على تفاصيل إنشاء المكتبة ، ستكون هناك حاجة إلى تعديلات على أمثلة البرامج النصية الواردة هنا لمعلمات محاذاة القراءة وعد الميزات و rMATS. عند حساب أعداد القراءة الأولية على مستوى إكسون باستخدام featureCounts ، أو طرق مماثلة ، يجب توخي الحذر لتكوين خيارات الوظيفة بشكل صحيح للأعداد والشريط: في featureCounts ، قمنا بتعيين وسيطة "strandSpecific" بشكل مناسب لبروتوكول RNA-seq الخاص بالشريط المستخدم ؛ وبالنسبة للقياس الكمي على مستوى exon ، من المتوقع أن يتم تعيين القراءة فوق exons المجاورة ، ولذا قمنا بتعيين المعلمة allowMultiOverlap على TRUE. بالنسبة ل APA ، هناك بروتوكولات تسلسل نهاية 3 'مختلفة 6 والتي تختلف في الموقع الدقيق للقمم بالنسبة لموقع pA. بالنسبة لبيانات المثال الخاصة بنا ، نحدد أن الذروة هي 60 نقطة أساس في المنبع لموقع pA كما هو موضح في الشكل 5 ، وسيحتاج هذا التحليل إلى تكييفه مع بروتوكولات التسلسل النهائي الأخرى ل 3 بوصات.
في هذا البروتوكول ، نقصر النطاق على مناقشة التحليلات التفاضلية على مستوى الإكسونات الفردية ، وأحداث الربط التي تتكون من مجموعات exon-intron المجاورة. نحن لا نناقش فئة التحليلات القائمة على إعادة بناء isoform de novo مثل Cufflinks و Cuffdiff32 و RSEM 33 و Kallisto34 والتي تهدف إلى اكتشاف وتحديد التعبير المطلق والنسبي للأشكال المتماثلة البديلة بأكملها. تعد الطرق القائمة على exon والأحداث أكثر حساسية للكشف عن أحداث الربط الفردية30 وفي كثير من الحالات توفر جميع المعلومات اللازمة لمزيد من التحليل ، دون الحاجة إلى القياس الكمي على مستوى isoform.
يتوفر أحدث إصدار من الملفات المصدر في هذا البروتوكول في https://github.com/jiayuwen/AS_APA_JoVE
Disclosures
ليس لدى المؤلفين ما يكشفون عنه.
Acknowledgements
تم دعم هذه الدراسة من قبل زمالة المستقبل لمجلس البحوث الأسترالي (ARC) (FT16010043) ومخطط ANU Futures.
Materials
| Name | Company | Catalog Number | Comments |
| Not relevent for computational study |
References
- Katz, Y., Wang, E. T., Airoldi, E. M., Burge, C. B. Analysis and design of RNA sequencing experiments for identifying isoform regulation. Nature Methods. 7 (12), 1009-1015 (2010).
- Wang, Y., et al. Mechanism of alternative splicing and its regulation. Biomedical Reports. 3 (2), 152-158 (2015).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in Bioinformatics. 21 (6), 2052-2065 (2020).
- Movassat, M., et al. Coupling between alternative polyadenylation and alternative splicing is limited to terminal introns. RNA Biology. 13 (7), 646-655 (2016).
- Tian, B., Manley, J. L. Alternative polyadenylation of mRNA precursors. Nature Reviews Molecular Cell Biology. 18 (1), 18-30 (2017).
- Herrmann, C. J., et al. PolyASite 2.0: a consolidated atlas of polyadenylation sites from 3' end sequencing. Nucleic Acids Research. 48 (1), 174-179 (2020).
- Liu, R., Loraine, A. E., Dickerson, J. A. Comparisons of computational methods for differential alternative splicing detection using RNA-seq in plant systems. BMC Bioinformatics. 15 (1), 364 (2014).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biology. 17 (1), 13 (2016).
- Anders, S., Reyes, A., Huber, W. Detecting differential usage of exons from RNA-seq data. Genome Research. 22 (10), 2008-2017 (2012).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2014).
- Shen, S., et al. rMATS: Robust and flexible detection of differential alternative splicing from replicate RNA-Seq data. Proceedings of the National Academy of Sciences. 111 (51), 5593-5601 (2014).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in bioinformatics. 21 (6), 2052-2065 (2020).
- Kanitz, A., et al. Comparative assessment of methods for the computational inference of transcript isoform abundance from RNA-seq data. Genome biology. 16 (1), 1-26 (2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550 (2014).
- Sznajder, L. J., et al. Loss of MBNL1 induces RNA misprocessing in the thymus and peripheral blood. Nature Communications. 11, 1-11 (2020).
- Batra, R., et al. Loss of MBNL leads to disruption of developmentally regulated alternative polyadenylation in RNA-mediated disease. Molecular Cell. 56 (2), 311-322 (2014).
- Leinonen, R., Sugawara, H., Shumway, M., et al. The sequence read archive. Nucleic acids research. 39, 19-21 (2010).
- Tange, O. . GNU parallel-the command-line power tool. 36, 42-47 (2011).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet journal. 17 (1), 10-12 (2011).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Robinson, M. D., Oshlack, A. A scaling normalization method for differential expression analysis of RNA-seq data. Genome Biology. 11 (3), 25 (2010).
- Veiga, D. F. T. maser: Mapping Alternative Splicing Events to pRoteins. R package version 1.4.0. , (2019).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (13), 25 (2009).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ramírez, F., Dündar, F., Diehl, S., Grüning, B. A., Manke, T. deepTools: a flexible platform for exploring deep-sequencing data. Nucleic acids research. 42 (1), 187-191 (2014).
- Merino, G. A., Conesa, A., Fernández, E. A. A benchmarking of workflows for detecting differential splicing and differential expression at isoform level in human RNA-seq studies. Briefings in bioinformatics. 20 (2), 471-481 (2019).
- Chhangawala, S., Rudy, G., Mason, C. E., Rosenfeld, J. A. The impact of read length on quantification of differentially expressed genes and splice junction detection. Genome biology. 16 (1), 1-10 (2015).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biol. 17, 13 (2016).
- Trapnell, C., et al. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nat Protoc. 7 (3), 562-578 (2012).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC Bioinformatics. 12, 323 (2011).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nat Biotechnol. 34 (5), 525-527 (2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved