Method Article
RNA-seqデータにおける選択的スプライシングおよびポリアデニル化の同定(英語)
要約
選択的スプライシング(AS)および選択的ポリアデニル化(APA)は、転写産物アイソフォームとその産物の多様性を拡大します。ここでは、バルクRNA-seqを分析するためのバイオインフォマティクスプロトコルと、実験条件によって変化するASおよびAPAを検出および視覚化するための3'エンドシーケンシングアッセイについて説明します。
要約
実験的/生物学的条件にわたる差次的遺伝子発現(DGE)を測定するためのRNA-Seqの典型的な分析に加えて、RNA-seqデータを利用して、エクソンレベルで他の複雑な調節メカニズムを探索することもできます。選択的スプライシングとポリアデニル化は、転写後レベルで遺伝子発現を調節するための異なるアイソフォームを生成することにより、遺伝子の機能的多様性に重要な役割を果たし、解析を遺伝子レベル全体に限定すると、この重要な調節層を見逃す可能性があります。ここでは、BioconductorおよびDEXSeq、LimmaパッケージのdiffSplice、rMATSなどの他のパッケージと機能を使用して、条件全体でのエクソンおよびポリアデニル化部位の使用状況を識別および視覚化するための詳細なステップバイステップ分析を示します。
概要
RNA-seqは、通常、差次的遺伝子発現の推定および遺伝子発見1に広く使用されてきました。また、異なるアイソフォームを発現する遺伝子によるエクソンレベルの使用状況の推定にも利用でき、転写後レベルでの遺伝子制御の理解に貢献します。真核生物遺伝子の大部分は、選択的スプライシング(AS)によって異なるアイソフォームを生成し、mRNA発現の多様性を高めます。ASイベントは、異なるパターンに分けることができる:完全エクソン(SE)のスキップ(カセット)エクソンが、その隣接するイントロンと共に転写物から完全に除去される。エクソンの両端に2つ以上のスプライス部位が存在する場合の代替(ドナー)5'スプライス部位選択(A5SS)および代替3'(アクセプター)スプライス部位選択(A3SS);イントロンが成熟mRNA転写物内に保持される場合のイントロンの保持(RI)、および一度に2つの利用可能なエクソンのうちの1つだけを保持できるエクソン使用の相互排除(MXE)2,3。代替ポリアデニル化(APA)はまた、単一の転写産物から複数のmRNAアイソフォームを生成する代替ポリ(A)部位を用いて遺伝子発現を調節する上で重要な役割を果たす4。ほとんどのポリアデニル化部位(pA)は3'非翻訳領域(3' UTR)に位置し、多様な3' UTR長のmRNAアイソフォームを生成します。3' UTRは調節要素を認識するための中心的なハブであるため、異なる3' UTR長はmRNAの局在、安定性および翻訳に影響を与える可能性があります5。プロトコル6の詳細が異なるAPAを検出するために最適化された3'エンドシーケンシングアッセイのクラスがあります。ここで説明するパイプラインはPolyA-seq用に設計されていますが、説明されているように他のプロトコルにも適合させることができます。
本研究では、エクソンベース(DEXSeq9、diffSplice10)とイベントベース(転写産物スプライシングの反復多変量解析(rMATS)11)の2つの大きなカテゴリに分類できる、差動エクソン解析法7,8(図1)のパイプラインを提示します。エクソンベースの方法は、個々のエクソンの条件にわたるフォールド変化を、発現差のあるエクソン使用量を呼び出すための全体的な遺伝子フォールド変化の測定値と比較し、そこからAS活性の遺伝子レベルの測定値を計算します。イベントベースの方法は、エクソン-イントロン-スパニング接合リードを使用して、エクソンスキップやイントロンの保持などの特定のスプライシングイベントを検出および分類し、出力3でこれらのASタイプを区別します。したがって、これらの方法は、AS12,13の完全な分析のための補完的なビューを提供します。DEXSeq(DESeq214 DGEパッケージに基づく)とdiffSplice(Limma10 DGEパッケージに基づく)は、差動スプライシング解析に最も広く使用されているパッケージであるため、研究に選択しました。rMATSは、イベントベースの分析の一般的な方法として選択されました。別の一般的なイベントベースの方法は、MISO(アイソフォームの混合)1です。APAでは、エクソンベースのアプローチを採用しています。
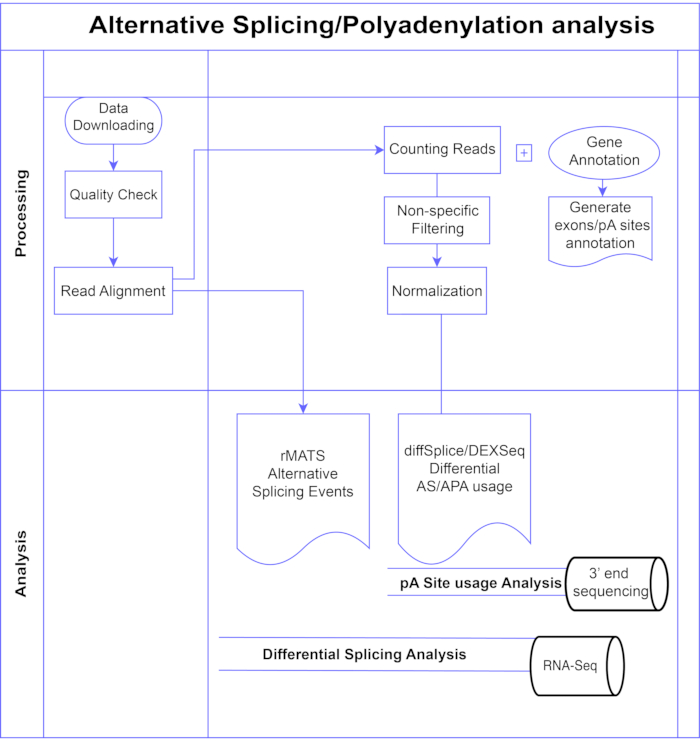
図 1.分析パイプライン。 分析で使用されるステップのフローチャート。手順には、データの取得、品質チェックとリードアライメントの実行、その後の既知のエクソン、イントロン、pAサイトのアノテーションを使用したリードのカウント、低カウントを削除するためのフィルタリング、正規化が含まれます。PolyA-seqデータはdiffSplice/DEXSeq法を用いて代替pA部位について解析し、バルクRNA-SeqはdiffSplice/DEXseq法を用いてエクソンレベルでの選択的スプライシングについて解析し、ASイベントはrMATSを用いて解析した。 この図の拡大版を表示するには、ここをクリックしてください。
本調査で用いたRNA-seqデータは、Gene Expression Omnibus(GEO)(GSE138691)15から取得したものである。この研究のマウスRNA-seqデータを、野生型(WT)とマッスルブラインド様タイプ1ノックアウト(Mbnl1 KO)の2つの条件群で使用し、それぞれ3回の反復を行いました。差的ポリアデニル化部位利用分析を実証するために、マウス胚線維芽細胞(MEF)PolyA-seqデータ(GEOアクセッションGSE60487)16を得た。データには、野生型(WT)、マッスルブラインド様タイプ1/タイプ2ダブルノックアウト(Mbnl1/2 DKO)、Mbnl3ノックダウン(KD)を備えたMbnl 1/2 DKO、およびMbnl3コントロール(Ctrl)を備えたMbnl1/2DKOの4つの条件グループがあります。各条件グループは、2 つの反復で構成されます。
| ゲオアクセッション | SRA 実行番号 | サンプル名 | 条件 | レプリケート | 組織 | シークエンシング | 読み取り長 | |
| RNA-シークエンス | GSM4116218 | SRR10261601 | Mbnl1KO_Thymus_1 | Mbnl1ノックアウト | 担当者 1 | 胸腺 | ペアエンド | 100 bp |
| GSM4116219 | SRR10261602 | Mbnl1KO_Thymus_2 | Mbnl1ノックアウト | 担当者 2 | 胸腺 | ペアエンド | 100 bp | |
| GSM4116220 | SRR10261603 | Mbnl1KO_Thymus_3 | Mbnl1ノックアウト | 担当者 3 | 胸腺 | ペアエンド | 100 bp | |
| GSM4116221 | SRR10261604 | WT_Thymus_1 | 野生型 | 担当者 1 | 胸腺 | ペアエンド | 100 bp | |
| GSM4116222 | SRR10261605 | WT_Thymus_2 | 野生型 | 担当者 2 | 胸腺 | ペアエンド | 100 bp | |
| GSM4116223 | SRR10261606 | WT_Thymus_3 | 野生型 | 担当者 3 | 胸腺 | ペアエンド | 100 bp | |
| 3P-シーケンセック | GSM1480973 | SRR1553129 | WT_1 | 野生型(WT) | 担当者 1 | マウス胚性線維芽細胞(MEF) | シングルエンド | 40 bp |
| GSM1480974 | SRR1553130 | WT_2 | 野生型(WT) | 担当者 2 | マウス胚性線維芽細胞(MEF) | シングルエンド | 40 bp | |
| GSM1480975 | SRR1553131 | DKO_1 | Mbnl 1/2 ダブルノックアウト (DKO) | 担当者 1 | マウス胚性線維芽細胞(MEF) | シングルエンド | 40 bp | |
| GSM1480976 | SRR1553132 | DKO_2 | Mbnl 1/2 ダブルノックアウト (DKO) | 担当者 2 | マウス胚性線維芽細胞(MEF) | シングルエンド | 40 bp | |
| GSM1480977 | SRR1553133 | DKOsiRNA_1 | Mbnl 1/2 ダブルノックアウト Mbnl 3 siRNA (KD) | 担当者 1 | マウス胚性線維芽細胞(MEF) | シングルエンド | 40 bp | |
| GSM1480978 | SRR1553134 | DKOsiRNA_2 | Mbnl 1/2 ダブルノックアウト Mbnl 3 siRNA (KD) | 担当者 2 | マウス胚性線維芽細胞(MEF) | シングルエンド | 36 bp | |
| GSM1480979 | SRR1553135 | DKONTsiRNA_1 | Mbnl 1/2 ダブルノックアウトとノンターゲティング siRNA (Ctrl) | 担当者 1 | マウス胚性線維芽細胞(MEF) | シングルエンド | 40 bp | |
| GSM1480980 | SRR1553136 | DKONTsiRNA_2 | Mbnl 1/2 ダブルノックアウトとノンターゲティング siRNA (Ctrl) | 担当者 2 | マウス胚性線維芽細胞(MEF) | シングルエンド | 40 bp |
表 1.解析に使用したRNA-SeqおよびPolyA-Seqデータセットの概要。
プロトコル
1. 分析で使用するツールと R パッケージのインストール
- Condaは、すべてのプラットフォーム間で依存関係を持つパッケージを簡単にインストールできる、人気のある柔軟なパッケージマネージャーです。「アナコンダ」(コンマパッケージマネージャー)を使用して、分析に必要なツール/パッケージをインストールするために使用できる「コンダ」をインストールします。
- https://www.anaconda.com/products/individual#Downloads からシステム要件に従って「Anaconda」をダウンロードし、グラフィカルインストーラーのプロンプトに従ってインストールします。Linuxコマンドラインで次のように入力して 、「conda」 を使用して必要なすべてのパッケージをインストールします。
conda install -c daler sratoolkit
conda install -c conda-forge parallel
conda install -c bioconda star bowtie fastqc rmats rmats2sashimiplot samtools fasterq-dump cutadapt bedtools deeptools - プロトコルで使用されるすべての R パッケージをダウンロードするには、R コンソール (Linux コマンド ラインで「R」と入力して開始) または Rstudio コンソールで次のコードを入力します。
bioc_packages<- c("DEXSeq", "Rsubread", "EnhancedVolcano", "edgeR", "limma", "maser","GenomicRanges")
packages<- c("magrittr", "rtracklayer", "tidyverse", "openxlsx", "BiocManager")
#Install if not already installed
installed_packages<-packages%in% rownames(installed.packages())
installed_bioc_packages<-bioc_packages%in% rownames(installed.packages())
if(any(installed_packages==FALSE)) {
install.packages(packages[!installed_packages],dependencies=TRUE)
BiocManager::install(packages[!installed_bioc_packages], dependencies=TRUE)
}
注:この計算プロトコルでは、コマンドはR Notebookファイル(拡張子が「.のファイル」)として与えられます。Rmd")、Rコードファイル(拡張子が「」のファイル)R")、または Linux Bash シェルスクリプト (拡張子 ".sh" のファイル)。R ノートブック (Rmd) ファイルは、ファイル|ファイルを開く...、および個々のコードチャンク(RコマンドまたはBashシェルコマンド)は、右上の緑色の矢印をクリックしてインタラクティブに実行されます。R コード ファイルは、RStudio で開くか、Linux コマンド ラインで "Rscript" で始まる (例: Rscript example.R) を実行して実行できます。シェル スクリプトは、Linux コマンド ラインでスクリプトの先頭に "sh" コマンドを付けることで実行できます (例: .sh example.sh)。
2. RNA-seqを用いた選択的スプライシング(AS)解析
- データのダウンロードと前処理
注:以下に注釈が付けられたコードスニペットは、補足コードファイル「AS_analysis_RNASeq.Rmd」を使用して、個々の手順をインタラクティブに実行し、Linuxコマンドラインでバッチで実行するbashスクリプトとしても提供されます(sh downloading_data_preprocessing.sh).- 生データのダウンロード。
- SRA ツールキット (v2.10.8)17 から 'prefetch' コマンドを使用して、シーケンス読み取りアーカイブ (SRA) から生データをダウンロードします。次のコマンドでSRAアクセッションIDを順番に指定し、GNU並列ユーティリティ18を使用して並行してダウンロードします。アクセッション ID の SRA ファイルを SRR10261601 から SRR10261606 に並行してダウンロードするには、Linux コマンドラインで以下を使用します。
seq 10261601 10261606 | parallel prefetch SRR{} - SRAツールキットから「fastqダンプ」機能を使用してアーカイブからfastqファイルを抽出します。GNU並列を使用し、すべてのSRAファイルの名前を一緒に指定します。
parallel -j 3 fastq-dump --gzip --skip-technical --read-filter pass --dumpbase --split-e --clip --origfmt {} ::: - マウス(ゲノムアセンブリGRCm39)のリファレンスゲノムとアノテーションを www.ensembl.org からダウンロードするには、Linuxコマンドラインで以下を使用します。
wget -nv -O annotation.gtf.gz http://ftp.ensembl.org/pub/release-103/gtf/mus_musculus/Mus_musculus.GRCm39.103.gtf.gz \ && gunzip -f annotation.gtf.gz
wget -nv -O genome.fa.gz http://ftp.ensembl.org/pub/release-103/fasta/mus_musculus/dna/Mus_musculus.GRCm39.dna.primary_assembly.fa.gz \ && gunzip -f genome.fa.gz
GTF=$(readlink -f annotation.gtf)
GENOME=$(readlink -f genome.fa)
- SRA ツールキット (v2.10.8)17 から 'prefetch' コマンドを使用して、シーケンス読み取りアーカイブ (SRA) から生データをダウンロードします。次のコマンドでSRAアクセッションIDを順番に指定し、GNU並列ユーティリティ18を使用して並行してダウンロードします。アクセッション ID の SRA ファイルを SRR10261601 から SRR10261606 に並行してダウンロードするには、Linux コマンドラインで以下を使用します。
- ゲノムアセンブリへの前処理とマッピングリード
- 品質管理。FASTQC (FASTQ 品質チェック v0.11.9)19 を使用して、生の読み取りの品質を評価します。出力フォルダを作成し、複数の入力fastaファイルに対して並行してfastqcを実行します。この手順では、各サンプルの品質レポートが生成されます。レポートを調べて、読み取りの品質が許容範囲内であることを確認してから、さらに分析を行います。( https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ のレポートを理解するには、ユーザーマニュアルを参照してください)
mkdir fastqc_out
parallel "fastqc {} -o fastqc_out" ::: $RAW_DATA/*.fastq.gz
注:必要に応じて、「cutadapt」20 または「trimmomatic」21 を使用してアダプタートリミングを実行し、RNAフラグメントサイズとリード長に基づいて変化する隣接アダプターへのシーケンスを削除します。この分析では、影響を受ける読み取りの割合が最小限であったため、このステップをスキップしました。 - 読み取りアライメント。前処理の次のステップには、リードを参照ゲノムにマッピングすることが含まれます。まず、STAR22 の「genomeGenerate」機能を使用して参照ゲノムのインデックスを構築し、次に生のリードをリファレンスにアライメントします(または、事前に構築されたインデックスはSTARのWebサイトから入手でき、アライメントに直接使用できます)。Linux コマンド ラインで次のコマンドを実行します。
#Build STAR index
GDIR=STAR_indices
mkdir $GDIR
STAR --runMode genomeGenerate --genomeFastaFiles $GENOME --sjdbGTFfile $GTF --runThreadN 8 --genomeDir $GDIR
ODIR=results/mapping
mkdir -p $ODIR
#Align reads to the genome
for fq1 in $RAW_DATA/*R1.fastq.gz;
do
fq2=$(echo $fq1| sed 's/1.fastq.gz/2.fastq.gz/g');
OUTPUT=$(basename ${fq1}| sed 's/R1.fastq.gz//g');
STAR --genomeDir $GDIR \
--runThreadN 12 \
--readFilesCommand zcat \
--readFilesIn ${fq1}${fq2}\
--outFileNamePrefix $ODIR\/${OUTPUT} \
--outSAMtype BAM SortedByCoordinate \
--outSAMunmapped Within \
--outSAMattributes Standard
Done
注: STAR アライナーは、読み取りアライメント後に各サンプルの BAM (バイナリ アライメント マップ) ファイルを生成して並べ替えます。Bamファイルは、次の手順に進む前にソートする必要があります。
- 品質管理。FASTQC (FASTQ 品質チェック v0.11.9)19 を使用して、生の読み取りの品質を評価します。出力フォルダを作成し、複数の入力fastaファイルに対して並行してfastqcを実行します。この手順では、各サンプルの品質レポートが生成されます。レポートを調べて、読み取りの品質が許容範囲内であることを確認してから、さらに分析を行います。( https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ のレポートを理解するには、ユーザーマニュアルを参照してください)
- 生データのダウンロード。
- エクソンアノテーションの準備。
- 補足コード ファイル "prepare_mm_exon_annotation.R」をGTF(遺伝子導入フォーマット)形式でダウンロードしたアノテーションでアノテーションを準備します。実行するには、Linux コマンド ラインで次のように入力します。
Rscript prepare_mm_exon_annotation.R annotation.gtf
注:GTFファイルには、異なるアイソフォームの複数のエクソンエントリが含まれています。このファイルは、各エクソンの複数の転写産物IDを「折りたたむ」ために使用されます。エクソン計数ビンを定義することは重要なステップです。
- 補足コード ファイル "prepare_mm_exon_annotation.R」をGTF(遺伝子導入フォーマット)形式でダウンロードしたアノテーションでアノテーションを準備します。実行するには、Linux コマンド ラインで次のように入力します。
- 読み取りをカウントします。次のステップは、異なる転写物/エクソンにマッピングされたリードの数をカウントすることです。補足ファイル: "AS_analysis_RNASeq.Rmd" を参照してください。
- 必要なライブラリをロードします。
packages<- c("Rsubread","tidyverse", "magrittr", "EnhancedVolcano", "edgeR","openxlsx")
invisible(lapply(packages, library, character.only=TRUE)) - 前の手順(2.2)で取得した処理済みのアノテーションファイルを読み込みます。
load("mm_exon_anno.RData") - 手順 2.2.2 で取得したすべての bam ファイルを 'featureCounts' の入力として読み取り、読み取りをカウントします。最初に .bam で終わるディレクトリから各ファイルを一覧表示して、bam ファイルを含むフォルダーを読み取ります。バムファイルと処理されたGTFアノテーション(参照)を入力として受け取るRsubreadパッケージの 'featureCounts'を使用して、エクソン(特徴)を表す行とサンプルを表す列を持つ各特徴に関連付けられたカウントのマトリックスを生成します。
countData <- dir("bams", pattern=".bam$", full.names=T) %>%
featureCounts(annot.ext=anno,
isGTFAnnotationFile=FALSE,
minMQS=0,useMetaFeatures=FALSE,
allowMultiOverlap=TRUE,
largestOverlap=TRUE,
countMultiMappingReads=FALSE,
primaryOnly=TRUE,
isPairedEnd=TRUE,
nthreads=12) - 次に、非特異的フィルタリングを実行して、発現の低いエクソンを除去する(「非特異的」は、選択バイアスを避けるために、実験条件情報がフィルタリングに使用されないことを示す)。「edgeR」パッケージ23 のcpm関数を使用して、データを生のスケールから100万個あたりのカウント(cpm)に変換し、少なくとも3つのサンプルで設定可能なしきい値(このデータセットでは1つのcpmを使用)を超えるカウントのエクソンを保持します。また、エクソンが1つだけの遺伝子も削除します。
# Non-specific filtering: Remove the exons with low counts
isexpr<- rownames(countData$counts)[rowSums(cpm(countData$counts)>1) >=3]
countData$counts<-countData$counts[rownames(countData$counts) %in%isexpr, ]
anno<-anno%>% filter(GeneID%in% rownames(countData$counts))
# Remove genes with only 1 site and NA in geneIDs
dn<-anno%>%group_by(GeneID)%>%summarise(nsites=n())%>% filter(nsites>1&!is.na(GeneID))
anno<-anno%>% filter(GeneID%in%dn$GeneID)
countData$counts<-countData$counts[rownames(countData$counts) %in%anno$GeneID, ]
注: シングルエンド読み取りなど、異なるデータを使用する場合は、featureCount に必要なパラメーターを確認し、「isPairedEnd = FALSE」を設定します。RSubreadユーザーガイドを参照してデータのオプションを選択し、以下の「ディスカッション」セクションを参照してください。
- 必要なライブラリをロードします。
- 差動スプライシングとエクソン使用分析。このステップでは、DEXSeq と DiffSplice という 2 つの選択肢について説明します。どちらも使用でき、同様の結果が得られます。一貫性を保つために、DGE に DESeq2 パッケージを使用する場合は DEXSeq を選択し、Limma ベースの DGE 解析には DiffSplice を使用します。補足ファイルを参照してください: "AS_analysis_RNASeq.Rmd".
- 差動エクソン分析にDEXSeqパッケージを使用。
- ライブラリをロードし、実験計画を定義するサンプルテーブルを作成します。
library(DEXSeq)
sampleTable<-data.frame(row.names= c("Mbnl1KO_Thymus_1", "Mbnl1KO_Thymus_2", "Mbnl1KO_Thymus_3", "WT_Thymus_1", "WT_Thymus_2", "WT_Thymus_3"), condition= rep(c("Mbnl1_KO", "WT"),c(3,3)), libType= rep(c("paired-end")))
注 : 行名は、読み取りをカウントするために featureCount が使用する bam ファイル名と一致している必要があります。sampleTable は、ライブラリの種類と条件を含む各サンプルの詳細で構成されます。これは、差分使用を検出するためのコントラストまたはテストグループを定義するために必要です。 - エクソン情報ファイルを準備します。GRanges(ゲノム範囲)オブジェクト(https://bioconductor.org/packages/release/bioc/html/GenomicRanges.html)の形式のエクソン情報は、次のステップでDEXSeqオブジェクトを作成するための入力として必要です。遺伝子IDをリードカウントと照合して、exoninfoオブジェクトを作成します。
exoninfo<-anno[anno$GeneID%in% rownames(countData$counts),]
exoninfo<-GRanges(seqnames=anno$Chr,
ranges=IRanges(start=anno$Start, end=anno$End, width=anno$Width),strand=Rle(anno$Strand))
mcols(exoninfo)$TranscriptIDs<-anno$TranscriptIDs
mcols(exoninfo)$Ticker<-anno$Ticker
mcols(exoninfo)$ExonID<-anno$ExonID
mcols(exoninfo)$n<-anno$n
mcols(exoninfo)$GeneID<-anno$GeneID
transcripts_l= strsplit(exoninfo$TranscriptIDs, "\\,")
save(countData, sampleTable, exoninfo, transcripts_l, file="AS_countdata.RData") - DEXSeqDataSet 関数を使用して DEXSeq オブジェクトを作成します。DEXSeq オブジェクトは、読み取りカウント、エクソン機能情報、およびサンプル情報をまとめて収集します。ステップ 3 で生成された読み取りカウントと前のステップで取得したエクソン情報を使用して、カウントマトリックスから DEXSeq オブジェクトを作成します。sampleData引数は、サンプル(およびその属性:ライブラリタイプと条件)を定義するデータフレーム入力を受け取り、「design」はsampleDataを使用して、モデル式表記を使用して差分テスト用の設計マトリックスを生成します。有意な相互作用項であるcondition:exonは、特定のエクソンに当てはまる遺伝子に対するリードの割合が実験条件に依存する、すなわちASが存在することを示すことに留意されたい。より複雑な実験計画のモデル式の設定の詳細については、DEXSeqのドキュメントを参照してください。機能情報については、エクソンID、対応する遺伝子および転写産物が必要です。
dxd<-DEXSeqDataSet(countData$counts,sampleData=sampleTable, design=~sample+exon+condition:exon,featureID=exoninfo$ExonID,groupID=exoninfo$GeneID,featureRanges=exoninfo, transcripts=transcripts_l) - 正規化と分散推定。次に、次のコマンドを使用して、サンプル間の正規化を実行し、RNA-seqの離散的な性質と生物学的変動性の両方によるポアソンカウントノイズによるデータの分散を推定します。
dxd %<>% estimateSizeFactors %>% estimateDispersions %T>% plotDispEsts - 差分使用をテストします。変動の推定後、各遺伝子のエクソン使用量の違いを検定し、結果を生成します。
dxd%<>%testForDEU%>%estimateExonFoldChanges(fitExpToVar=
"condition")#Estimate fold changes
dxr=DEXSeqResults(dxd) - 次のコマンドを使用して、選択した遺伝子のスプライシングイベントを視覚化します。
plotDEXSeq(dxr,"Wnk1", displayTranscripts=TRUE, splicing=TRUE,legend
=TRUE,cex.axis=1.2,cex=1.3,lwd=2)
R Notebook ファイル "AS_analysis_RNASeq.Rmd" を調べて、目的の遺伝子の追加プロットを生成し、さまざまなしきい値で火山プロットを生成します。
- ライブラリをロードし、実験計画を定義するサンプルテーブルを作成します。
- LimmaのdiffSpliceを使用して、差動スプライシングを識別します。Rノートブックファイルに従ってください」AS_analysis_RNASeq.Rmd".先に進む前に、入力ファイルを準備するための手順 2.1 から 2.3 に従っていることを確認してください。
- ライブラリのロード
library(limma)
library(edgeR) - 非特異的フィルタリング。2.3で得られた読み取りカウントの行列を抽出します。edgeRパッケージの「DGEList」関数を使用して、行が遺伝子を表し、列がサンプルを表す特徴のリストを作成します。
mycounts=countData$counts
#Change the rownames of the countdata to exon Ids instead of genes for unique rownames.
rownames(mycounts) = exoninfo$ExonID
dge<-DGEList(counts=mycounts)
#Filtering
isexpr<- rowSums(cpm(dge)>1) >=3
dge<-dge[isexpr,,keep.lib.sizes=FALSE]
#Extract the exon annotations for only transcripts meeting non-specific filter
exoninfo=anno%>% filter(ExonID%in% rownames(dge$counts))
#Convert the exoninfo into GRanges object
exoninfo1<-GRanges(seqnames=exoninfo$Chr,
ranges=IRanges(start=exoninfo$Start, end=exoninfo$End, width=exoninfo$Width),strand=Rle(exoninfo$Strand))
mcols(exoninfo1)$TranscriptIDs<-exoninfo$TranscriptIDs
mcols(exoninfo1)$Ticker<-exoninfo$Ticker
mcols(exoninfo1)$ExonID<-exoninfo$ExonID
mcols(exoninfo1)$n<-exoninfo$n
mcols(exoninfo1)$GeneID<-exoninfo$GeneID
transcripts_l= strsplit(exoninfo1$TranscriptIDs, "\\,")
注:非特異的フィルタリングステップとして、カウントはn個のサンプルのうちx個のcpm<1でフィルタリングされます(xは任意の条件での最小反復数です)。この例のデータでは、n = 6 および x = 3 です。 - M 値のトリミング平均 (TMM 正規化法) を使用して 'edgeR' パッケージの 'calcNormFactors' 関数を使用して、サンプル全体のカウントを正規化します 24 スケーリング係数を計算してライブラリのサイズを調整します。
dge<-calcNormFactors(dge) - ステップ 2.4.1.1 で生成された sampleTable を使用して、計画行列を作成します。設計マトリックスは設計を特徴付けます。より高度な実験計画の計画行列の詳細については、Limma User Guide (https://www.bioconductor.org/packages/devel/bioc/vignettes/limma/inst/doc/usersguide.pdf) の第 8 章と第 9 章を参照してください。
Treat<- factor(sampleTable$condition)
design<- model.matrix(~0+Treat)
colnames(design) <- levels(Treat) - エクソンごとに線形モデルを当てはめます。「limma」パッケージの「voom」関数を実行してRNA-seqデータを処理して分散を推定し、ポアソンカウントノイズを補正するための精密な重みを生成し、エクソンレベルのカウントをlog2カウント/百万(logCPM)に変換します。次に、「lmfit」関数を使用して線形モデリングを実行し、線形モデルを各エクソンの式データに適合させます。「eBayes」関数を使用して適合モデルの経験的ベイズ統計を計算し、エクソン発現の差を検出します。次に、対象の実験比較のためのコントラスト行列を定義します。'contrasts.fit' を使用して、比較の各ペアの係数と標準誤差を取得します。
v<-voom(dge,design,plot=FALSE)
fit<-lmFit(v,design)
fit<-eBayes(fit)
colnames(fit)
cont.matrix<-makeContrasts(
Mbnl1_KO_WT=Mbnl1_KO-WT,
levels=design)
fit2<-contrasts.fit(fit,cont.matrix) - 差動スプライシング解析。適合モデルで「diffSplice」を実行して、野生型とノックアウト型の間の遺伝子のエクソン使用の違いをテストし、「topSplice」関数を使用して上位ランクの結果を調べます:test="t"はASエクソンのランキングを与え、test="simes"は遺伝子のランキングを与えます。
ex<-diffSplice(fit2,geneid=exoninfo$GeneID,exonid=exoninfo$ExonID)
ts<-topSplice(ex,n=Inf,FDR=0.1, test="t", sort.by="logFC")
tg<-topSplice(ex,n=Inf,FDR=0.1, test="simes") - 視覚化。'plotSplice'関数で結果をプロットし、geneid引数に目的の遺伝子を与えます。ログでソートされた上位の結果を保存する 変化をオブジェクトに折り畳み、エクソンを示す火山プロットを生成します。
plotSplice(ex,geneid="Wnk1", FDR=0.1)
#Volcano plot
EnhancedVolcano(ts,lab=ts$ExonID,selectLab= head((ts$ExonID),2000), xlab= bquote(~Log[2]~'fold change'), x='logFC', y='P.Value', title='Volcano Plot', subtitle='Mbnl1_KO vs WT (Limma_diffSplice)', FCcutoff=2, labSize=4,legendPosition="right", caption= bquote(~Log[2]~"Fold change cutoff, 2; FDR 10%"))
- ライブラリのロード
- rMATS の使用
- 最新バージョンのrMATS v4.1.1(処理時間が短縮され、メモリの要件が少ないため、rMAT turboとも呼ばれます)が、作業ディレクトリのcondaまたはgithub(https://github.com/Xinglab/rmats-turbo/releases/download/v4.1.1/rmats_turbo_v4_1_1.tar.gz)を使用してインストールされていることを確認します。「AS_analysis_RNASeq.Rmd」のセクション 4.3 に従ってください。
- マッピング後に取得した bam ファイルを含むフォルダーに移動し、 rMATS で要求されているように、bam ファイルの名前を (パスと共に) ',' で区切ってコピーして、2 つの条件のテキスト ファイルを準備します。次のコマンドは、Linux コマンドラインで実行する必要があります。
mkdir rMATS_analysis
cd bams/
ls -pd "$PWD"/*| grep "WT"| tr '\n'','> Wt.txt
ls -pd "$PWD"/*| grep "Mb"| tr '\n'','> KO.txt
mv *.txt ../rMATS_analysis - 前のステップで生成された 2 つの入力ファイルと、2.1.1.3 で取得した GTF ファイルを使用して rmats.py を実行します。これにより、各スプライシングイベントの統計(p値と包含レベル)を個別に記述するテキストファイルを含む出力フォルダー「rmats_out」が生成されます。
python rmats-turbo/rmats.py --b1 KO.txt --b2 Wt.txt --gtf annotation.gtf -t paired --readLength 50 --nthread 8 --od rmats_out/ --tmp rmats_tmp --task pos
注: GTF ファイル形式の参照注釈も必要です。データがシングルエンドの場合はパラメータを確認し、それに応じて -t オプションを変更します。 - rMATSの結果を調べる。バイオコンダクターパッケージ「maser」25 を使用して、rMATSの結果を調べます。ジャンクションおよびエクソンカウント(JCEC)テキストファイルを「maser」オブジェクトにロードし、スプライシングイベントごとに少なくとも5つの平均読み取りを含めることにより、カバレッジに基づいて結果をフィルタリングします。
library(maser)
mbnl1<-maser("/rmats_out/", c("WT","Mbnl1_KO"), ftype="JCEC")
#Filtering out events by coverage
mbnl1_filt<-filterByCoverage(mbnl1,avg_reads=5) - rMATSの結果を視覚化する。「maser」パッケージの「topEvents」機能を使用して、偽検出率(FDR)10%およびスプライス率(deltaPSI)の最小10%の変化で重要なスプライシングイベントを選択します。次に、目的の個々の遺伝子(サンプル遺伝子-Wnk1)の遺伝子イベントを確認し、その遺伝子の各スプライシングイベントのPSI値をプロットします。イベントタイプを指定して火山プロットを生成します。
#Top splicing events at 10% FDR
mbnl1_top<-topEvents(mbnl1_filt,fdr=0.1, deltaPSI=0.1)
mbnl1_top
#Check the gene events for a particular gene
mbnl1_wnk1<-geneEvents(mbnl1_filt,geneS="Wnk1", fdr=0.1, deltaPSI=0.1)
maser::display(mbnl1_wnk1,"SE")
plotGenePSI(mbnl1_wnk1,type="SE", show_replicates
=TRUE)
volcano(mbnl1_filt,fdr=0.1, deltaPSI=0.1,type="SE")
+xlab("deltaPSI")+ylab("Log10 Adj. Pvalue")+ggtitle("Volcano Plot of exon skipping events") - rMATSで得られたスプライシングイベント結果の刺身プロットを「rmats2shahimiplot」パッケージを使用してテキストファイルの形式で生成します。Linux コマンドラインで python スクリプトを実行します。
python ./src/rmats2sashimiplot/rmats2sashimiplot.py --b1 ../bams/WT_Thymus_1.bam,../bams/WT_Thymus_2.bam,../bams/WT_Thymus_3.bam --b2 ../bams/Mbnl1KO_Thymus_1.bam,../bams/Mbnl1KO_Thymus_2.bam,../bams/Mbnl1KO_Thymus_3.bam -t SE -e ../rMATS_analysis/rmats_out/SE.MATS.JC.txt --l1 WT --l2 Mbnl1_KO --exon_s 1 --intron_s 5 -o ../rMATS_analysis/rmats2shasmi_output
注:このプロセスは、イベントファイル内のすべての結果の刺身プロットを生成するため、時間がかかる場合があります。topEvents 関数によって表示される上位の結果 (遺伝子名とエクソン) を 'maser' から選択し、対応する刺身プロットを視覚化します。
- 差動エクソン分析にDEXSeqパッケージを使用。
3. 3'末端シーケンシングを用いた代替ポリアデニル化(APA)解析
- データのダウンロードと前処理
注: 補足の R ノートブック ファイル "APA_analysis_3PSeq_notebook.Rmd」を使用して、データのダウンロードと前処理の手順の完全なコマンドを使用するか、Linuxコマンドラインで補足bashファイル「APA_data_downloading_preprocessing.sh」を実行します。- アクセッション ID (1553129 から 1553136) を使用して SRA からデータをダウンロードします。
- アダプターと逆相補をトリミングして、センス鎖シーケンスを取得します。
注:このステップは、使用するPolyA-seqアッセイに固有のものです。 - マップリードは、ボウタイアライナー26を用いたマウスゲノムアセンブリへ。
- pAサイトアノテーションの準備。
注:pAサイトアノテーションファイルの処理は、まず補足Rノートブックファイル「APA_analysis_3PSeq_notebookを使用して実行されます。Rmd" (2.1 - 2.6) を使用し、bash ファイル "APA_annotation_preparation.sh" を使用します。- PolyASite 2.0 データベースから pA サイトのアノテーションをダウンロードする6.
- pAサイトアノテーションを選択して、3'-非翻訳領域(UTR)pAサイトを保持し、ダウンストリーム分析のために末端エクソン(TE)またはアノテーション付き末端エクソン(DS)の1000 nt下流にアノテーションを付けます。
- pAサイトのピークを取得します。各pA切断部位にアンカーし、ベッドツールとディープツールを使用して平均読み取りカバレッジを視覚化します27,28。結果は、マッピングされたリードのピークが主に切断部位の上流~60 bp以内に分散していることを示しました(図5および補足図5)。したがって、pA部位の座標は、アノテーションファイルから切断部位の60 bp上流まで拡張されました。使用する特定の3'末端シーケンシングプロトコルに応じて、このステップはPolyA-seq以外のアッセイ用に最適化する必要があります。
- 読み取りのカウント
- pA サイト注釈ファイルを準備します。
anno<- read.table(file= "flanking60added.pA_annotation.bed",
stringsAsFactors=FALSE, check.names=FALSE, header=FALSE, sep="")
colnames(anno) <- c("chrom", "chromStart", "chromEnd", "name", "score", "strand", "rep", "annotation", "gene_name", "gene_id")
anno<- dplyr::select(anno,name,chrom, chromStart,chromEnd, strand,gene_id,gene_name,rep)
colnames(anno) <- c("GeneID", "Chr", "Start", "End", "Strand", "Ensembl", "Symbol", "repID") - 'featureCounts' を適用して、生のカウントを取得します。カウント テーブルを RData ファイル "APA_countData.Rdata" として保存し、さまざまなツールを使用して APA 分析を行います。
countData<- dir("bamfiles", pattern="sorted.bam$", full.names=TRUE) %>%
# Read all bam files as input for featureCounts
featureCounts(annot.ext=anno, isGTFAnnotationFile= FALSE,minMQS=0,useMetaFeatures= TRUE,allowMultiOverlap=TRUE, largestOverlap= TRUE,strandSpecific=1, countMultiMappingReads =TRUE,primaryOnly= TRUE,isPairedEnd= FALSE,nthreads=12)%T>%
save(file="APA_countData.Rdata")
注: 'featureCounts' 関数にリストされているパラメーターのいずれかを変更するように注意してください。「strandSpecific」パラメータを変更して、使用する3'エンドシーケンシングアッセイのシーケンシング方向と一致するようにします(経験的に、プラス鎖とマイナス鎖の遺伝子をゲノムブラウザーで視覚化すると、これが明確になります)。 - カウントデータの非特定のフィルタリングを適用します。フィルタリングにより、差分pAサイト使用テストの統計的堅牢性を大幅に向上させることができます。まず、pA部位が1つしかない遺伝子を除去し、その上でpA部位の使用法を定義できない。次に、カバレッジに基づいて非特異的フィルタリングを適用します:カウントは、n個のサンプルのうちx個で1未満のcpmでフィルタリングされ、xは任意の条件での最小反復数です。このサンプルデータでは、N = 8 および x = 2 です。
load(file= "APA_countData.Rdata")# Skip this step if already loaded
# Non-specific filtering: Remove the pA sites not differentially expressed in the samples
countData<-countData$counts%>%as.data.frame%>% .[rowSums(edgeR::cpm(.)>1) >=2, ]
anno%<>% .[.$GeneID%in% rownames(countData), ]
# Remove genes with only 1 site and NA in geneIDs
dnsites<-anno%>%group_by(Symbol)%>%summarise(nsites=n())%>% filter(nsites>1&!is.na(Symbol))
anno<-anno%>% filter(Symbol%in%dnsites$Symbol)
countData<-countData[rownames(countData) %in%anno$GeneID, ]
- pA サイト注釈ファイルを準備します。
- DEXSeqおよびdiffSpliceパイプラインを使用した差分ポリアデニル化サイト利用解析。
- DEXSeq パッケージの使用
注:DEXSeqパイプラインにはコントラストマトリックスを定義できないため、2つの実験条件の差分APA分析を個別に実行する必要があります。条件WTと条件DKOの微分APA解析を例に、手順を説明する。補足ファイル「APA_analysis_3PSeq_notebook。ティッカー」このセクションのステップバイステップのワークフローと、他のコントラストの差分APA分析について説明します。- ライブラリをロードし、実験計画を定義するサンプルテーブルを作成します。
c("DEXSeq", "GenomicRanges") %>% lapply(library, character.only=TRUE) %>%invisible
sampleTable1<- data.frame(row.names= c("WT_1","WT_2","DKO_1","DKO_2"),
condition= c(rep("WT", 2), rep("DKO", 2)),
libType= rep("single-end", 4)) - バイオコンダクターパッケージGRangesを使用してpAサイト情報ファイルを準備します。
# Prepare the GRanges object for DEXSeqDataSet object construction
PASinfo <- GRanges(seqnames = anno$Chr,
ranges = IRanges(start = anno$Start, end = anno$End),strand = Rle(anno$Strand))
mcols(PASinfo)$PASID<-anno$repID
mcols(PASinfo)$GeneEns<-anno$Ensembl
mcols(PASinfo)$GeneID<-anno$Symbol
# Prepare the new feature IDs, replace the strand information with letters to match the current pA site clusterID
new.featureID <- anno$Strand %>% as.character %>% replace(. %in% "+", "F") %>% replace(. %in% "-", "R") %>% paste0(as.character(anno$repID), .) - 手順 3.3 で生成された読み取りカウントと、前の手順で取得した pA サイト情報を使用して、DEXSeq オブジェクトを作成します。
# Select the read counts of the condition WT and DKO
countData1<- dplyr::select(countData, SRR1553129.sorted.bam, SRR1553130.sorted.bam, SRR1553131.sorted.bam, SRR1553132.sorted.bam)
# Rename the columns of countData using sample names in sampleTable
colnames(countData1) <- rownames(sampleTable1)
dxd1<-DEXSeqDataSet(countData=countData1,
sampleData=sampleTable1,
design=~sample+exon+condition:exon,
featureID=new.featureID,
groupID=anno$Symbol,
featureRanges=PASinfo) - DEXSeqオブジェクトの条件のレベルを定義して、コントラストペアを定義します。
dxd1$condition<- factor(dxd1$condition, levels= c("WT", "DKO"))
# The contrast pair will be "DKO - WT" - 正規化と分散推定。RNA-seqデータと同様に、3'末端シーケンシングデータの場合、「推定サイズ要因」関数を使用してサンプル間の正規化(各サンプルの比の列方向の中央値)を実行し、「推定分散」関数を使用してデータの変動を推定し、「plotDispEsts」関数を使用して分散推定結果を視覚化します。
dxd1 %<>% estimateSizeFactors %>% estimateDispersions %T>% plotDispEsts - 関数「testForDEU」を使用して各遺伝子の差分pAサイト使用をテストし、次に関数「推定ExonFoldChanges」を使用してpAサイト使用のフォールド変化を推定します。関数「DEXSeqResults」を使用して結果を確認し、有意に異なるpAサイトの基準として「FDR < 10%」を設定します。
dxd1 %<>% testForDEU %>% estimateExonFoldChanges(fitExpToVar = "condition")
dxr1 <- DEXSeqResults(dxd1)
dxr1
mcols(dxr1)$description
table(dxr1$padj<0.1) # Check the number of differential pA sites (FDR < 0.1)
table(tapply(dxr1$padj<0.1, dxr1$groupID, any)) # Check the number of gene overlapped with differential pA site - 関数「plotDEXSeq」によって生成された微分APAプロットと関数「拡張火山」による火山プロットを使用して、差分pAサイト使用結果の視覚化。
# Select the top 100 significant differential pA sites ranked by FDR
topdiff.PAS<- dxr1%>%as.data.frame%>%rownames_to_column%>%arrange(padj)%$%groupID[1:100]
# Apply plotDEXSeq for the visualization of differential polyA usage
plotDEXSeq(dxr1,"S100a7a", legend=TRUE, expression=FALSE,splicing=TRUE, cex.axis=1.2, cex=1.3,lwd=2)
# Apply perGeneQValue to check the top genes with differential polyA site usage
dxr1%<>% .[!is.na(.$padj), ]
dgene<- data.frame(perGeneQValue= perGeneQValue (dxr1)) %>%rownames_to_column("groupID")
dePAS_sig1<-dxr1%>% data.frame() %>%
dplyr::select(-matches("dispersion|stat|countData|genomicData"))%>%
inner_join(dgene)%>%arrange(perGeneQValue)%>%distinct()%>%
filter(padj<0.1)
# Apply EnhancedVolcano package to visualise differential polyA site usage
"EnhancedVolcano"%>% lapply(library, character.only=TRUE) %>%invisible
EnhancedVolcano(dePAS_sig1, lab=dePAS_sig1$groupID, x='log2fold_DKO_WT',
y='pvalue',title='Volcano Plot',subtitle='DKO vs WT',
FCcutoff=1,labSize=4, legendPosition="right",
caption= bquote(~Log[2]~"Fold change cutoff, 1; FDR 10%"))
- ライブラリをロードし、実験計画を定義するサンプルテーブルを作成します。
- diffSplice パッケージを使用します。補足の R ノートブック ファイル "APA_analysis_3PSeq_notebook.このセクションのステップバイステップのワークフローの「Rmd」を参照してください。
- 差分pA使用分析の対象となるコントラストを定義します。
注: この手順は、R ノートブック ファイル "APA_analysis_3PSeq_notebook" に含まれている DGEList オブジェクトの構築と処理の後に実行する必要があります。Rmd」を参照してください。contrast.matrix<-makeContrasts(DKO_vs_WT=DKO-
WT,Ctrl_vs_DKO=Ctrl-DKO,
KD_vs_Ctrl=KD-Ctrl,KD_vs_DKO=KD-DKO,levels=design)
fit2<-fit%>%contrasts.fit(contrast.matrix)%>%eBayes
summary(decideTests(fit2))
ex<-diffSplice(fit2,geneid=anno$Symbol,exonid=new.featureID)
topSplice(ex) #Check the top significant results with topSplice - 関数「plotSplice」による微分APAプロットと関数「拡張火山」による火山プロットを使用して、関心のあるコントラスト(ここでは「DKO-WT」)の結果を視覚化します。R ノートブック ファイル "APA_analysis_3PSeq_notebook.Rmd" 4.2.7 - 4.2.9 (他のコントラストペアの視覚化用)。
sig1<-topSplice(ex,n=Inf,FDR=0.1,coef=1, test="t", sort.by="logFC")
sig1.genes<-topSplice(ex,n=Inf,FDR=0.1,coef=1, test="simes")
plotSplice(ex, coef=1,geneid="S100a7a", FDR = 0.1)
plotSplice(ex,coef=1,geneid="Tpm1", FDR = 0.1)
plotSplice(ex,coef=1,geneid="Smc6", FDR = 0.1)
EnhancedVolcano(sig1, lab=sig1$GeneID,xlab= bquote(~Log[2]~'fold change'),
x='logFC', y='P.Value', title='Volcano Plot', subtitle='DKO vs WT',
FCcutoff=1, labSize=6, legendPosition="right")
- 差分pA使用分析の対象となるコントラストを定義します。
- DEXSeq パッケージの使用
結果
上記のステップバイステップのワークフローを実行した後、ASおよびAPA分析出力と代表的な結果は、次のように生成されたテーブルとデータプロットの形式になります。
として:
AS分析の主な出力(diffSpliceの補足表1;DEXSeq)の表2)は、条件間で異なる使用を示すエクソンのリスト、およびその構成エクソンの1つ以上の有意な全体的なスプライシング活性を示す遺伝子のリストであり、統計的有意性によってランク付けされています。補足表1のタブ2は有意なエクソンを示し、列はエクソン対休止の差FC、エクソンごとの未調整p値、および調整済みp値(ベンジャミニ-ホックバーグ補正)を示しています。調整されたp値の閾値化は、定義されたFDRを持つエクソンのセットを与えます。補足表1のタブ3は、全体的なスプライシング活性の有意性を示す遺伝子のランク付けされたリストを示し、列はSimes法を用いて計算された遺伝子レベルの調整されたp値を示す。DEXSeqについても同様のデータを表2に示します。補足図1および補足図2は、データ15とともに提示された公開論文で実験的に検証されたMbnl1、Tcf7およびLef1遺伝子における差動スプライシングパターンを示す。著者らは、Mbnl1、Mbnl2、Lef1、Tcf7、Ncor2の5つの遺伝子の実験的検証を示しました。私たちのアプローチは、これらすべての遺伝子で差動スプライシングパテンを検出しました。ここでは、補足表1〜3で得られたDEXSeq、diffSplice、およびrMATSを使用して、各遺伝子のFDRレベルを示します:Mbnl1(0、6.6E-61、0)、Mbnl2(0,0.18,0)、Lef1(1.4E-10、1.3E-04、0)、Tcf7(0、1.1E-6、0)およびNcor2(9.2E-11、1.6E-06、0)。
図2は、3つの異なるツールから得られた出力の比較を示し、Wnk1遺伝子の選択的スプライシングパターンを示しています。火山プロットを図2A(差分スプライス)と図2B(DEXSeq)に示します。さらに3つの高ランクの遺伝子を補足図1(diffSplice)および補足図2(DEXSeq)に示します。Y 軸は統計的有意性 (-log10 p 値)、X 軸は効果サイズ (倍率変化) を示します。左上または右上の象限に位置する遺伝子は、実質的なFCと真の違いの強力な統計的証拠を示しています。

図 2.diffSplice、DEXSeq、およびrMATSから得られた選択的スプライシング結果の比較。|
(A)Limma diffSplice解析からのRNA-Seqの火山プロット(左):x軸は対数エクソンフォールドの変化を示しています。Y 軸は -log10 p 値を示します。各点はエクソンに対応する。p値= 1E-5の水平破線。2倍変化時の縦破線(FC)。赤色エクソンは、実質的なFCおよび統計的有意性を示す。差動スプライシングプロット(右):サンプル遺伝子Wnk1のスプライシングパターンが示され、x軸は転写物ごとのエクソンIDを示しています。y軸は、エクソンの相対対数フォールドの変化(エクソンのlogFCと他のすべてのエクソンの全体的なlogFCとの差)を示す。赤で強調表示されたエクソンは、統計的に有意な差次的発現を示す(FDR < 0.1)。
(B)DEXSeq解析から得られたRNA-Seqの火山プロット(左)とエクソン使用量の差(右)。Wnk1遺伝子は、ピンク色で強調表示されたWTとMbnl1ノックアウトとの間のエクソンの有意な差のある使用を示し、これらは(A)の同じ差動エクソンに対応する。
(C)rMATS分析から得られたWnk1の火山プロット(左)と刺身プロット(右)。10%FDRでのノックアウトと比較した野生型の有意なスキップ(カセット)エクソン(SE)イベントを示し、スプライス率の変化(PSIまたはΔΨ)値が0.1>。X 軸は条件間の PSI 値の変化を示し、Y 軸は対数値 P 値を示します。刺身プロットは、Wnk1遺伝子におけるスキップエクソン事象を示しており、(A)および(B)における有意な差動エクソンに対応する。各行はRNA-Seqサンプル(野生型およびMbnl1ノックアウトの3回の反復)を表す。高さはRPKMでの読み取りカバレッジを示し、接続アークはエクソン間のジャンクション読み取りを示します。アノテーションされた遺伝子モデル代替アイソフォームは、プロットの下部に示されています。C の下部パネルは、PSI 統計の計算に使用されるジャンクション読み取りを示しています。
(D)異なる方法により得られた有意な差動エクソンの数を比較する ベン図 。この図の拡大版を表示するには、ここをクリックしてください。
図 2A(右パネル)は、上位遺伝子のエクソン差を図式で表示したもので、y軸にlogFC、x軸にエクソン数を示しています。この実施例は、遺伝子 Wnk1の条件間で変化するカセットエクソンを示す。DEXSeqの差動エクソン使用プロットは、Wnk1.6.45付近の5つのエクソン部位での差動スプライシングの証拠を示しています。ピンク色で強調表示されたエクソンは、WTと比較してMbnl1 KOサンプルでスプライスアウトされている可能性があります。これらのエクソンは、特定のゲノム位置で同様のパターンを示すdiffSpliceによって得られた結果と相補的です。その他の例を補足図 1 および 補足図2に示します。興味深い結果を確認するためのより詳細なビューは、UCSC(サンタクルーズ大学)またはIGV(統合ゲノミクスビューア)ゲノムブラウザ(図示せず)のRPM(Reads per Million)単位のカバレッジ(小刻みに動く)トラックを、既知の遺伝子モデル、保存、その他のゲノムワイドアッセイなどの他の関心のあるトラックとの視覚的な相関関係と比較することによって提供できます。
rMATS出力表には、タイプ別に分類された重要な選択的スプライシングイベントがリストされています(補足表3)。 図2C は、選択的スプライスされた遺伝子の火山プロットを示し、効果サイズは11の微分「スプライス率」(PSIまたはΔΨ)統計量によって測定されます。
PSIは、カセットエクソンの包含と一致するリード(すなわち、カセットエクソン自体へのマッピングの読み取り、またはエクソンと重なるジャンクションリード)を、エクソン排除と一致するリード、すなわち隣接する上流および下流のエクソンにわたるジャンクションリードと比較する割合を指す(図2Cの下部パネル)。図2Cの右側のパネルは、Wnk1遺伝子の刺身プロットを示しており、Mbnl1 KOのエクソンがスキップされた、遺伝子のカバレッジトラックに差分スプライシングイベントが重ねられています。エクソンを結合するアークは、ジャンクションリード(スプライスアウトされたイントロンを横切るリード)の数を示します。 補足表3のさまざまなタブは、エクソン境界を持つジャンクションにまたがる各タイプのイベントの有意な読み取りを示しています(ジャンクションカウントとエクソンカウント(JCEC))。図2Dは、3つのツールによって検出された有意な差動スプライスされたエクソンを比較しています。

図 3.rMATS分析によって取得された選択的スプライシングイベント。A) AS イベントの種類。この図は、構成的および代替的にスプライスされたエクソンによるスプライシングイベントを説明するrMATS文書11から適応されています。FDR 10%で各イベントの番号でラベル付けされています。B)スキップエクソン(SE)を呈するAdd3遺伝子の刺身プロット。C)代替5'スプライス部位(A5SS)を示すBaz2b遺伝子の刺身プロット。D)代替3'スプライス部位(A3SS)を示すLsm14b遺伝子の刺身プロット。E)相互に排他的なエクソン(MXE)を示すMta1遺伝子の刺身プロット。F)保持イントロン(RI)を示すArpp21遺伝子の刺身プロット。赤い行は野生型の3回の反復を表し、オレンジ色の行はMbnl1ノックアウト反復を表します。x軸はゲノム座標と鎖情報に対応し、y軸はRPKMでのカバレッジを示す。この図の拡大版を表示するには、ここをクリックしてください。
図3は、スプライシングイベントSE、A5SS、A3SS、MXE、およびRIのタイプを、これらのイベントの上位の有意遺伝子の刺身プロットの助けを借りて示しています。WTとMbnl1_KOの両方の3つの反復を比較すると、FDR 10%で合計1272のSEイベント、130のA5SS、116のA3SS、215のMXE、および313のRIイベントが検出されました。刺身プロットは、例として上位遺伝子を使用してイベントの種類を示しています。
アパ:
APA分析からの出力は、エクソンレベルのAS分析に似ています。3'UTRにおける差別APA活性によってランク付けされた上位遺伝子の表が提供される(補足表4および補足表5)。図4Aは、diffSpliceとDEXSeqを別々に使用して生成された3'UTRの異なるAPA活性による遺伝子の火山プロットを示しています。図4Bは、異なるパイプラインから取得した有意差pAサイト使用結果を比較したベンプロットを示しています。図4Cおよび4Dは、DKOにおけるpA部位使用の有意な遠位から近位へのシフト(Fosl2)および有意な近位から遠位へのシフト(Papola)を示すことが実験的に検証された、diffSpliceとDEXSeqの両方を使用して生成された遺伝子Fosl2(図4C)およびPapola(図4D)の3'UTRにおける異なるpAサイト使用量の図式図を示しています。 それぞれ。その他の例を補足図3および補足図4に示します。

図 4.diffSpliceとDEXSeqによる代替ポリアデニル化プロット。A) diffSplice と DEXSeq を使用して生成された PolyA-seq データの火山プロット。X軸は、ログpAサイトのフォールド変化を示す。Y 軸は -log10 の p 値を示します。各ポイントは pA サイトに対応します。p値= 1E-5の水平破線。2倍のFCの縦破線。赤色エクソンは、実質的なFCおよび統計的有意性を示す。 B) 異なるパイプラインから得られた有意差pAサイト使用結果を比較したベンプロット。 C-D) diffSpliceおよびDEXSeqを使用して生成された差動APAプロットは、 Fosl2 および Papola 遺伝子の近位、内部および遠位pA部位を示す。図は 図2 (B)と同じ機能で生成されますが、pAサイトがエクソンに置き換わります。 この図の拡大版を表示するには、ここをクリックしてください。
図5は、使用したPolyA−seqアッセイについてアノテーション付きpA切断部位周辺の予想されるリード分布を確認するための診断プロットである。既知のpA切断部位に固定された隣接領域の平均カバレッジをゲノムワイドレベルで示しています。この場合、サイトのアップストリームで予想される読み取りの積み上げが視覚化されます。すべてのPolyA-seqサンプルのpAサイトに固定されたリード分布を 補足図5に示します。

図 5.pA切断部位周辺の平均被覆率プロット。 PolyA-seqデータの切断部位を縦破線で示しています。X軸は、pA切断部位に対する塩基位置を示し、上流および下流の最大100ヌクレオチド。y軸は、すべてのpA切断部位の平均リードカバレッジを、CPMのライブラリサイズで正規化したものです。 この図の拡大版を表示するには、ここをクリックしてください。
同じパイプラインによって生成された異なるコントラストの差動APA結果は、ゲノムブラウザで代表的な有意差pAサイトの読み取りカバレッジを視覚化することによって比較および検証できます。図6Aは、diffSpliceから取得した異なるコントラストの有意差pAサイト使用率を比較したベンプロットです。図6B-Dは、異なる遺伝子のpA部位におけるリードカバレッジのIGVスナップショットであり、diffSpliceを用いたAPA解析で発見されたものと一致するパターンを示しています。図6Bは、遺伝子Paip2のpAサイト使用の有意な近位から遠位へのシフトを検証し、これはコントラストDKO対WTで一意に検出されるが、他の2つのコントラストKD対WT、およびCtr対WTでは検出されない。 図6Cは、コントラストKD対WTで一意に検出された遺伝子Ccl2のpAサイト使用の有意な遠位から近位へのシフトを検証し、 一方、図6Dは、遺伝子Cacna2d1のすべてのコントラストの有意な差pA使用率を検証しています。
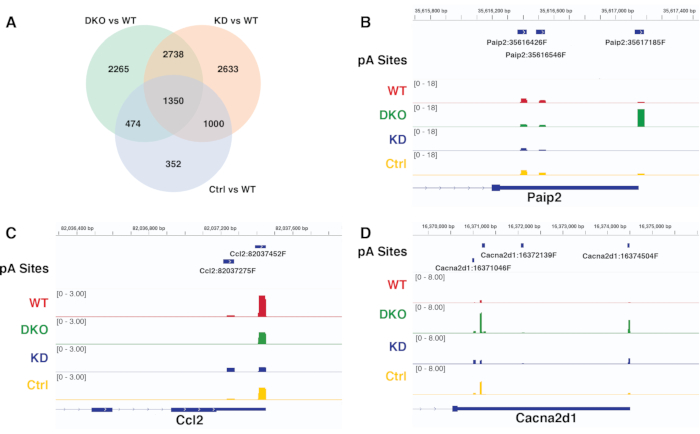
図 6.コントラストの比較と差分スプライスの結果の検証。A) diffSpliceから取得した異なるコントラストの有意な差分pAサイト使用結果を比較したベン図。B-D)条件全体で遺伝子Paip2、Ccl2、およびCacna2d1のpAピークカバレッジを視覚化するIGVスナップショット。各トラックは、特定の条件での読み取りカバレッジを表します。この図の拡大版を表示するには、ここをクリックしてください。
補足図1.Limma差分スプライスを用いた差次的スプライシングのRNA-Seq解析(A) Limma diffSplice解析からのRNA-Seqの火山プロット:x軸は対数エクソンフォールドの変化を示す。Y 軸は -log10 p 値を示します。各点はエクソンに対応する。p値= 1E-5の水平破線。2 倍変化時の縦破線 (FC)。赤色エクソンは、実質的なFCおよび統計的有意性を示す。 (B-D) 差動スプライシングプロット: スプライシングパターンは、例えば遺伝子 Mbnl1、 Tcf7、 および Lef1をそれぞれ示し、x軸は転写物あたりのエクソンIDを示す。y軸は、エクソンの相対対数フォールドの変化(エクソンのlogFCと他のすべてのエクソンの全体的なlogFCとの差)を示す。赤で強調表示されたエクソンは、統計的に有意な差次的発現を示す(FDR < 0.1)。 このファイルをダウンロードするには、ここをクリックしてください。
補足図2.DEXSeqによるエクソン使用の違いのRNA-Seq解析。(A)火山プロット。 (B-D) エクソン使用差DEXSeq分析から得られたRNA-Seqの。遺伝子Mbnl1、Tcf7、およびLef1は、それぞれピンク色で強調表示されたWTとMbnl1ノックアウトの間でエクソンの有意な異なる使用を示し、補足図1の同じ差動エクソンに対応します。このファイルをダウンロードするには、ここをクリックしてください。
補足図3.diffSpliceによる代替ポリアデニル化プロット。A)マウスPolyA-seqデータから得られた3つのコントラストペアでdiffSpliceを使用して生成されたPolyA-seqデータの火山プロット(ダブルノックアウト(DKO)対野生型(WT)、ノックダウン(KD)対WT、およびコントロール(Ctrl)対WTを含む。 X軸は、ログpAサイトのフォールド変化を示します。Y 軸は -log10 の p 値を示します。各ポイントは pA サイトに対応します。p値= 1E-5の水平破線。2倍のFCの縦破線。赤色pAサイトは、実質的なFCおよび統計的有意性を示す。B)diffSpliceを使用して生成された、高ランクの遺伝子S100a7a、Tpm1、およびSmc6の近位、内部、および遠位のpAサイトを示す差動APAプロット。このファイルをダウンロードするには、ここをクリックしてください。
補足図4。DEXSeqパイプラインによる差分pA使用量分析A)マウスPolyA-seqデータから得られた3つのペアでDEXSeqを使用して生成されたPolyA-seqデータの火山プロット(ダブルノックアウト(DKO)対野生型(WT)、ノックダウン(KD)対WT、およびコントロール(Ctrl)対WTを含む。 X軸は、ログpAサイトのフォールド変化を示します。Y 軸は -log10 の p 値を示します。各ポイントは pA サイトに対応します。p値= 1E-5の水平破線。2倍のFCの縦破線。赤色pAサイトは、実質的なFCおよび統計的有意性を示す。B)DEXSeqを使用して生成された、高ランクの遺伝子S100a7a、Tpm1、およびSmc6の近位、内部、および遠位pAサイトを示す差動APAプロット。このファイルをダウンロードするには、ここをクリックしてください。
補足図5.pA切断部位周辺の平均カバレッジプロットとヒートマップ。 カバレッジは4つの条件で示され、正鎖と逆鎖の遺伝子は別々に示されています。X軸は、pA切断部位に対する塩基位置を示し、上流および下流の最大100ヌクレオチド。y軸は、すべてのpA切断部位にわたる対応する相対塩基位置での平均カバレッジを指します。ヒートマップは代替ビューを提供し、各pA切断部位はx軸上に個別の行として表示され、カバレッジ順に並べられます。強度は読み取りカバレッジを示します(凡例を参照)。このファイルをダウンロードするには、ここをクリックしてください。
附則表1. AS 解析の差分スプライス出力。最初のタブは、2番目(エクソンレベル)と3番目(遺伝子レベル)のタブに表示される元の出力の列名を定義します。 この表をダウンロードするには、ここをクリックしてください。
別表2. AS解析のDEXSeq出力。最初のタブは、2番目(エクソンレベル)と3番目(遺伝子レベル)のタブに表示される元の出力の列名を定義します。 この表をダウンロードするには、ここをクリックしてください。
補足表3。 AS分析のrMATS出力。最初のタブでは、要約ファイル (タブ 2) の列名と各イベントの JCEC ファイル (タブ 3-7) を定義します。 この表をダウンロードするには、ここをクリックしてください。
補足表4。 APA 分析の差分スプライス出力。最初のタブは、2番目(pAサイトレベル)および3番目(遺伝子レベル)のタブに表示される元の出力の列名を定義します。 この表をダウンロードするには、ここをクリックしてください。
別表5. APA 分析の DEXSeq 出力。最初のタブは、2番目(pAサイトレベル)および3番目(遺伝子レベル)のタブに表示される元の出力の列名を定義します。 この表をダウンロードするには、ここをクリックしてください。
補足表6。 APAのASおよびpA部位の有意に変化したエクソンの数の要約。 この表をダウンロードするには、ここをクリックしてください。
補足表7。 AS/APA 分析で使用されるツールとパッケージの概要。 この表をダウンロードするには、ここをクリックしてください。
ディスカッション
この研究では、バルクRNA-Seqおよび3'末端シーケンシングデータからASおよびAPAを検出するためのエクソンベースおよびイベントベースのアプローチを評価しました。エクソンベースのASアプローチは、発現差のあるエクソンのリストと、全体的な遺伝子レベルの差次的スプライシング活性の統計的有意性によって順序付けられた遺伝子レベルのランク付けの両方を生成します(表1-2、4-5)。diffSpliceパッケージの場合、差分使用量は、エクソンレベルで重み付き線形モデルをフィッティングして、同じ遺伝子内の他のエクソンの平均対数フォールド変化(パーエクソンFCと呼ばれる)に対するエクソンの微分対数フォールド変化を推定することによって決定されます。遺伝子レベルの統計的有意性は、個々のエクソンレベルの有意性検定をSimes法10による遺伝子ごとの検定に組み合わせることによって計算されます。遺伝子レベルの差次的スプライシング活性によるこのランキングは、続いて、関与する主要な経路の遺伝子セット濃縮分析(GSEA)を実行するために使用することができる10。DEXSeqは、フィルタリング、正規化、分散推定などの特定のステップでは異なりますが、一般化線形モデルを当てはめてエクソン使用量を測定することで、同様の戦略を使用します。DEXSeqとDiffSpliceを使用してAS活性とAPAを示す上位500位のエクソンを比較すると、それぞれ310のエクソンと300のpAサイトの重複が見つかり、以前の研究でも実証された2つのエクソンベースのアプローチの一致が実証されました29。ASの包括的な検出と分類のために、エクソンベース(DEXSeqまたはdiffSpliceのいずれか)とイベントベースのアプローチの両方を組み合わせて使用することをお勧めします。APAの場合、ユーザーはDEXSeqまたはdiffSpliceのいずれかを選択できます:どちらの方法も、幅広いトランスクリプトミクス実験で良好に機能することが示されています29。
AS解析用のRNA-seqライブラリを調製する際には、脊椎動物ゲノムの遺伝子の大部分が異なる鎖上で重複しており、非鎖特異的プロトコルではこれらの重複領域を区別できず、最終的なエクソン検出が交絡するため、鎖特異的バルクRNA-seqプロトコル8を使用することが重要です。別の考慮事項はリード深度であり、スプライシング分析ではDGEよりも深いシーケンスが必要です(たとえば、サンプルあたり3,000万〜6,000万リード、DGEのサンプルあたり5〜2,500万リード(https://sapac.support.illumina.com/bulletins/2017/04/considerations-for-rna-seq-read-length-and-coverage-.html)。プロトコルで示されているすべてのツールは、シングルエンドとペアエンドの両方のシーケンシングデータをサポートしています。既知の遺伝子アノテーションのみを使用してジャンクションリードを検出する場合は、シングルエンドの短いリード(≥ 50 bp)を使用できますが、新規スプライスジャンクションのde novo検出は、ペアエンドおよびより長いリード(≥ 100 bp)の恩恵を受けます30,31。RNA抽出プロトコルの選択(ポリA選択またはrRNA枯渇のいずれか)は、RNAの質と実験上の質問に依存します-議論については31を参照してください。ライブラリ構築の詳細に応じて、読み取りアライメント、特徴カウント、およびrMATSのパラメーターについて、ここに示すサンプルスクリプトを変更する必要があります。featureCountsまたは同様の方法を使用して初期エクソンレベルのリードカウントを計算する際には、カウントと座礁性に対して関数オプションを正しく構成するように注意する必要があります:featureCountsでは、使用する鎖特異的RNA-seqプロトコルに対して「strandSpecific」引数を適切に設定します。エクソンレベルの定量化では、リードが隣接するエクソンにマッピングされることが予想されるため、allowMultiOverlapパラメーターをTRUEに設定します。APAの場合、pAサイトに対するピークの正確な位置が異なる、異なる3'末端シーケンシングプロトコル6があります。サンプルデータでは、図5に示すように、ピークはpAサイトの60 bp上流にあると判断し、この分析は他の3'末端シーケンシングプロトコルに適合させる必要があります。
このプロトコルでは、個々のエクソンのレベルでの差分解析、および隣接するエクソンとイントロンの組み合わせからなるスプライシングイベントの議論に限定します。カフリンクス、カフディフ32、RSEM 33、カリスト34など、代替アイソフォーム全体の絶対的および相対的発現を検出および定量化することを目的とした、アイソフォームデノベーションに基づく分析のクラスについては説明しません。エクソンおよび事象ベースの方法は、個々のスプライシング事象を検出するためにより感度が高く30、多くの場合、アイソフォームレベルの定量を必要とせずに、さらなる分析に必要なすべての情報を提供する。
このプロトコルのソースファイルの最新バージョンは 、https://github.com/jiayuwen/AS_APA_JoVE
開示事項
著者は開示するものは何もありません。
謝辞
この研究は、オーストラリア研究評議会(ARC)のフューチャーフェローシップ(FT16010043)およびANUフューチャースキームの支援を受けました。
資料
| Name | Company | Catalog Number | Comments |
| Not relevent for computational study |
参考文献
- Katz, Y., Wang, E. T., Airoldi, E. M., Burge, C. B. Analysis and design of RNA sequencing experiments for identifying isoform regulation. Nature Methods. 7 (12), 1009-1015 (2010).
- Wang, Y., et al. Mechanism of alternative splicing and its regulation. Biomedical Reports. 3 (2), 152-158 (2015).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in Bioinformatics. 21 (6), 2052-2065 (2020).
- Movassat, M., et al. Coupling between alternative polyadenylation and alternative splicing is limited to terminal introns. RNA Biology. 13 (7), 646-655 (2016).
- Tian, B., Manley, J. L. Alternative polyadenylation of mRNA precursors. Nature Reviews Molecular Cell Biology. 18 (1), 18-30 (2017).
- Herrmann, C. J., et al. PolyASite 2.0: a consolidated atlas of polyadenylation sites from 3' end sequencing. Nucleic Acids Research. 48 (1), 174-179 (2020).
- Liu, R., Loraine, A. E., Dickerson, J. A. Comparisons of computational methods for differential alternative splicing detection using RNA-seq in plant systems. BMC Bioinformatics. 15 (1), 364 (2014).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biology. 17 (1), 13 (2016).
- Anders, S., Reyes, A., Huber, W. Detecting differential usage of exons from RNA-seq data. Genome Research. 22 (10), 2008-2017 (2012).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2014).
- Shen, S., et al. rMATS: Robust and flexible detection of differential alternative splicing from replicate RNA-Seq data. Proceedings of the National Academy of Sciences. 111 (51), 5593-5601 (2014).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in bioinformatics. 21 (6), 2052-2065 (2020).
- Kanitz, A., et al. Comparative assessment of methods for the computational inference of transcript isoform abundance from RNA-seq data. Genome biology. 16 (1), 1-26 (2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550 (2014).
- Sznajder, L. J., et al. Loss of MBNL1 induces RNA misprocessing in the thymus and peripheral blood. Nature Communications. 11, 1-11 (2020).
- Batra, R., et al. Loss of MBNL leads to disruption of developmentally regulated alternative polyadenylation in RNA-mediated disease. Molecular Cell. 56 (2), 311-322 (2014).
- Leinonen, R., Sugawara, H., Shumway, M., et al. The sequence read archive. Nucleic acids research. 39, 19-21 (2010).
- Tange, O. . GNU parallel-the command-line power tool. 36, 42-47 (2011).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet journal. 17 (1), 10-12 (2011).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Robinson, M. D., Oshlack, A. A scaling normalization method for differential expression analysis of RNA-seq data. Genome Biology. 11 (3), 25 (2010).
- Veiga, D. F. T. maser: Mapping Alternative Splicing Events to pRoteins. R package version 1.4.0. , (2019).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (13), 25 (2009).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ramírez, F., Dündar, F., Diehl, S., Grüning, B. A., Manke, T. deepTools: a flexible platform for exploring deep-sequencing data. Nucleic acids research. 42 (1), 187-191 (2014).
- Merino, G. A., Conesa, A., Fernández, E. A. A benchmarking of workflows for detecting differential splicing and differential expression at isoform level in human RNA-seq studies. Briefings in bioinformatics. 20 (2), 471-481 (2019).
- Chhangawala, S., Rudy, G., Mason, C. E., Rosenfeld, J. A. The impact of read length on quantification of differentially expressed genes and splice junction detection. Genome biology. 16 (1), 1-10 (2015).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biol. 17, 13 (2016).
- Trapnell, C., et al. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nat Protoc. 7 (3), 562-578 (2012).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC Bioinformatics. 12, 323 (2011).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nat Biotechnol. 34 (5), 525-527 (2016).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved