Method Article
זיהוי שחבור חלופי ופוליאדנילציה בנתוני RNA-seq
In This Article
Summary
שחבור חלופי (AS) ופוליאדנילציה חלופית (APA) מרחיבים את מגוון האיזופורמים של שעתוק ותוצרים. כאן אנו מתארים פרוטוקולים ביואינפורמטיים לניתוח מבחני RNA-seq בתפזורת ו-3' של ריצוף קצה כדי לזהות ולהמחיש AS ו-APA המשתנים בין תנאי ניסוי.
Abstract
בנוסף לניתוח הטיפוסי של RNA-Seq למדידת ביטוי גנים דיפרנציאלי (DGE) בתנאים ניסיוניים/ביולוגיים, ניתן להשתמש בנתוני RNA-seq גם כדי לחקור מנגנוני ויסות מורכבים אחרים ברמת האקסון. שחבור חלופי ופוליאדנילציה ממלאים תפקיד מכריע במגוון התפקודי של גן על ידי יצירת איזופורמים שונים כדי לווסת את ביטוי הגנים ברמה שלאחר השעתוק, והגבלת הניתוחים לרמת הגן כולה עלולה לפספס את שכבת הוויסות החשובה הזו. כאן, אנו מדגימים ניתוחים מפורטים צעד אחר צעד לזיהוי והדמיה של שימוש באקסון דיפרנציאלי ובאתר פוליאדנילציה בתנאים שונים, תוך שימוש ב- Bioconductor ובחבילות ופונקציות אחרות, כולל DEXSeq, diffSplice מחבילת לימה ו- rMATS.
Introduction
RNA-seq היה בשימוש נרחב לאורך השנים בדרך כלל להערכת ביטוי גנים דיפרנציאליים וגילוי גנים1. בנוסף, ניתן להשתמש בו גם כדי להעריך שימוש משתנה ברמת אקסון עקב גנים המבטאים איזופורמים שונים, ובכך לתרום להבנה טובה יותר של ויסות גנים ברמה שלאחר השעתוק. רוב הגנים האאוקריוטים מייצרים איזופורמים שונים על ידי שחבור חלופי (AS) כדי להגדיל את המגוון של ביטוי mRNA. ניתן לחלק את אירועי AS לדפוסים שונים: דילוג על אקסונים שלמים (SE) שבהם אקסון ("קלטת") מוסר לחלוטין מהתמליל יחד עם האינטרונים האגפים שלו; בחירת אתר שחבור חלופי (תורם) 5' (A5SS) וחלופה 3' (מקבל) בחירת אתר שחבור (A3SS) כאשר שני אתרי שחבור או יותר נמצאים משני קצותיו של אקסון; שמירה של אינטרונים (RI) כאשר אינטרון נשמר בתוך תעתיק mRNA בוגר והדרה הדדית של שימוש באקסון (MXE) כאשר רק אחד משני האקסונים הזמינים יכול להישמר בכל פעם 2,3. פוליאדנילציה חלופית (APA) ממלאת גם תפקיד חשוב בוויסות ביטוי גנים באמצעות אתרי פולי (A) חלופיים ליצירת איזופורמים מרובים של mRNA מתעתיק יחיד4. רוב אתרי הפוליאדנילציה (pAs) ממוקמים באזור 3' לא מתורגם (3' UTRs), ומייצרים איזופורמים של mRNA עם אורכי UTR מגוונים של 3'. מכיוון שה-UTR 3' הוא הרכזת המרכזית לזיהוי אלמנטים רגולטוריים, אורכי UTR שונים של 3' יכולים להשפיע על לוקליזציה, יציבות ותרגוםmRNA 5. ישנם סוגים של מבחני רצף קצה 3 'הממוטבים לזיהוי APA השונים בפרטי הפרוטוקול6. הצינור המתואר כאן מיועד ל- PolyA-seq, אך ניתן להתאים אותו לפרוטוקולים אחרים כמתואר.
במחקר זה אנו מציגים צינור של שיטות ניתוח אקסון דיפרנציאליות7,8 (איור 1), שניתן לחלק לשתי קטגוריות רחבות: מבוססות אקסון (DEXSeq9, diffSplice 10) ומבוססות אירועים (ניתוח רב-משתני משוכפל של שחבור תעתיק (rMATS)11). השיטות המבוססות על אקסון משוות את שינוי הקיפול על פני תנאים של אקסונים בודדים, כנגד מידה של שינוי קיפול גנים כולל כדי לקרוא לשימוש באקסון המתבטא באופן דיפרנציאלי, ומתוך כך מחשבים מדידה ברמת הגן של פעילות AS. שיטות מבוססות אירועים משתמשות בקריאות צומת exon-intron-spanning כדי לזהות ולסווג אירועי שחבור ספציפיים כגון דילוג אקסון או שמירה של אינטרונים, ולהבחין בין סוגי AS אלה בפלט3. לפיכך, שיטות אלה מספקות תצוגות משלימות לניתוח מלא של AS12,13. בחרנו ב- DEXSeq (בהתבסס על חבילת DESeq214 DGE) ו- diffSplice (בהתבסס על חבילת Limma10 DGE) למחקר מכיוון שהם בין החבילות הנפוצות ביותר לניתוח שחבור דיפרנציאלי. rMATS נבחרה כשיטה פופולרית לניתוח מבוסס אירועים. שיטה פופולרית נוספת המבוססת על אירועים היא MISO (תערובת של איזופורמים)1. עבור APA אנו מתאימים את הגישה מבוססת exon.
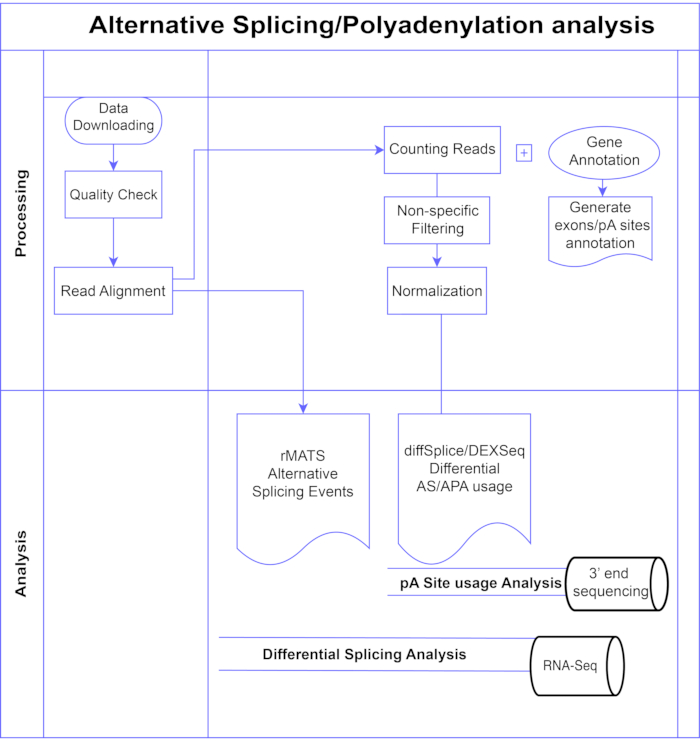
איור 1. צינור ניתוח. תרשים זרימה של השלבים המשמשים בניתוח. השלבים כוללים: קבלת הנתונים, ביצוע בדיקות איכות ויישור קריאה ולאחר מכן ספירת קריאות באמצעות ביאורים עבור exons, introns ואתרי pA ידועים, סינון כדי להסיר ספירות נמוכות ונורמליזציה. נתוני PolyA-seq נותחו עבור אתרי pA חלופיים באמצעות שיטות diffSplice/DEXSeq, RNA-Seq בתפזורת נותח עבור שחבור חלופי ברמת האקסון בשיטות diffSplice/DEXseq, ואירועי AS נותחו עם rMATS. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
נתוני ה-RNA-seq ששימשו בסקר זה נרכשו מביטוי גנים אומניבוס (GEO) (GSE138691)15. השתמשנו בנתוני RNA-seq של עכברים ממחקר זה עם שתי קבוצות מצב: נוקאאוט מסוג פראי (WT) ונוקאאוט דמוי שריר מסוג 1 (Mbnl1 KO) עם שלושה שכפולים כל אחד. כדי להדגים ניתוח שימוש באתר פוליאדנילציה דיפרנציאלית, השגנו נתוני פוליA-seq של עוברי עכברים (MEFs) (GEO Accession GSE60487)16. לנתונים יש ארבע קבוצות מצבים: סוג פראי (WT), דמוי שרירים מסוג 1/סוג 2 נוקאאוט כפול (Mbnl1/2 DKO), Mbnl 1/2 DKO עם נוקאאוט Mbnl3 (KD) ו- Mbnl1/2 DKO עם בקרת Mbnl3 (Ctrl). כל קבוצת תנאים מורכבת משני עותקים משוכפלים.
| הצטרפות גיאוגרפית | מספר הפעלה של SRA | שם לדוגמה | תנאי | לשכפל | טישו | רצף | אורך קריאה | |
| רנ"א-סק | GSM4116218 | SRR10261601 | Mbnl1KO_Thymus_1 | נוקאאוט Mbnl1 | נציג 1 | התימוס | סוף מזווג | 100 כ"ס |
| GSM4116219 | SRR10261602 | Mbnl1KO_Thymus_2 | נוקאאוט Mbnl1 | חזרה 2 | התימוס | סוף מזווג | 100 כ"ס | |
| GSM4116220 | SRR10261603 | Mbnl1KO_Thymus_3 | נוקאאוט Mbnl1 | חזרה 3 | התימוס | סוף מזווג | 100 כ"ס | |
| GSM4116221 | SRR10261604 | WT_Thymus_1 | סוג פראי | נציג 1 | התימוס | סוף מזווג | 100 כ"ס | |
| GSM4116222 | SRR10261605 | WT_Thymus_2 | סוג פראי | חזרה 2 | התימוס | סוף מזווג | 100 כ"ס | |
| GSM4116223 | SRR10261606 | WT_Thymus_3 | סוג פראי | חזרה 3 | התימוס | סוף מזווג | 100 כ"ס | |
| 3P-Seq | GSM1480973 | SRR1553129 | WT_1 | סוג פראי (WT) | נציג 1 | פיברובלסטים עובריים לעכבר (MEFs) | קצה יחיד | 40 bp |
| GSM1480974 | SRR1553130 | WT_2 | סוג פראי (WT) | חזרה 2 | פיברובלסטים עובריים לעכבר (MEFs) | קצה יחיד | 40 bp | |
| GSM1480975 | SRR1553131 | DKO_1 | Mbnl 1/2 נוקאאוט כפול (DKO) | נציג 1 | פיברובלסטים עובריים לעכבר (MEFs) | קצה יחיד | 40 bp | |
| GSM1480976 | SRR1553132 | DKO_2 | Mbnl 1/2 נוקאאוט כפול (DKO) | חזרה 2 | פיברובלסטים עובריים לעכבר (MEFs) | קצה יחיד | 40 bp | |
| GSM1480977 | SRR1553133 | DKOsiRNA_1 | Mbnl 1/2 נוקאאוט כפול עם Mbnl 3 siRNA (KD) | נציג 1 | פיברובלסטים עובריים לעכבר (MEFs) | קצה יחיד | 40 bp | |
| GSM1480978 | SRR1553134 | DKOsiRNA_2 | Mbnl 1/2 נוקאאוט כפול עם Mbnl 3 siRNA (KD) | חזרה 2 | פיברובלסטים עובריים לעכבר (MEFs) | קצה יחיד | 36 bp | |
| GSM1480979 | SRR1553135 | DKONTsiRNA_1 | Mbnl 1/2 נוקאאוט כפול עם siRNA ללא מיקוד (Ctrl) | נציג 1 | פיברובלסטים עובריים לעכבר (MEFs) | קצה יחיד | 40 bp | |
| GSM1480980 | SRR1553136 | DKONTsiRNA_2 | Mbnl 1/2 נוקאאוט כפול עם siRNA ללא מיקוד (Ctrl) | חזרה 2 | פיברובלסטים עובריים לעכבר (MEFs) | קצה יחיד | 40 bp |
טבלה 1. סיכום של מערכי נתונים של RNA-Seq ו- PolyA-seq המשמשים לניתוח.
Protocol
1. התקנת כלים וחבילות R המשמשים בניתוח
- קונדה הוא מנהל חבילות פופולרי וגמיש המאפשר התקנה נוחה של חבילות עם התלות שלהן בכל הפלטפורמות. השתמש ב- 'Anaconda' (מנהל חבילות conda) כדי להתקין 'conda' שניתן להשתמש בו כדי להתקין את הכלים / החבילות הדרושים לניתוח.
- הורד את 'Anaconda' בהתאם לדרישות המערכת מ- https://www.anaconda.com/products/individual#Downloads והתקן אותו על ידי ביצוע ההנחיות במתקין הגרפי. התקן את כל החבילות הנדרשות באמצעות 'conda' על-ידי הקלדת הפרטים הבאים בשורת הפקודה של Linux.
conda install -c daler sratoolkit
conda install -c conda-forge parallel
conda install -c bioconda star bowtie fastqc rmats rmats2sashimiplot samtools fasterq-dump cutadapt bedtools deeptools - כדי להוריד את כל חבילות R המשמשות בפרוטוקול, הקלד את הקוד הבא במסוף R (שהתחיל בשורת הפקודה של Linux על-ידי הקלדת 'R') או במסוף Rstudio.
bioc_packages<- c("DEXSeq", "Rsubread", "EnhancedVolcano", "edgeR", "limma", "maser","GenomicRanges")
packages<- c("magrittr", "rtracklayer", "tidyverse", "openxlsx", "BiocManager")
#Install if not already installed
installed_packages<-packages%in% rownames(installed.packages())
installed_bioc_packages<-bioc_packages%in% rownames(installed.packages())
if(any(installed_packages==FALSE)) {
install.packages(packages[!installed_packages],dependencies=TRUE)
BiocManager::install(packages[!installed_bioc_packages], dependencies=TRUE)
}
הערה: בפרוטוקול חישובי זה, הפקודות יינתנו כקבצי מחברת R (קבצים עם סיומת ". Rmd"), קבצי קוד R (קבצים עם סיומת ". R"), או לינוקס Bash מעטפת סקריפטים (קבצים עם סיומת ".sh"). יש לפתוח קבצי מחברת R (Rmd) ב- RStudio באמצעות קובץ | פתח קובץ..., וגושי קוד בודדים (שעשויים להיות פקודות R או פקודות מעטפת Bash) ולאחר מכן הפעל באופן אינטראקטיבי על-ידי לחיצה על החץ הירוק בפינה השמאלית העליונה. ניתן להפעיל קבצי קוד R על ידי פתיחה ב- RStudio, או בשורת הפקודה של לינוקס על ידי הקדמה עם "Rscript", למשל Rscript example.R. סקריפטים של Shell מופעלים בשורת הפקודה של לינוקס על ידי הקדמת הסקריפט עם הפקודה "sh" למשל.sh example.sh.
2. ניתוח שחבור חלופי (AS) באמצעות RNA-seq
- הורדה ועיבוד מראש של נתונים
הערה: מקטעי הקוד המבוארים להלן זמינים בקובץ הקוד המשלים "AS_analysis_RNASeq.RMD", כדי לבצע את השלבים הבודדים באופן אינטראקטיבי, והם מסופקים גם כסקריפט bash להיות מופעל באצווה על שורת הפקודה לינוקס (SH downloading_data_preprocessing.sh).- הורדת הנתונים הגולמיים.
- הורד את הנתונים הגולמיים מארכיון קריאת רצף (SRA) באמצעות הפקודה 'prefetch' מתוך ערכת הכלים של SRA (v2.10.8)17. תן את מזהי הגישה של SRA ברצף בפקודה הבאה כדי להוריד אותם במקביל באמצעות כלי השירות המקבילי של גנו18. כדי להוריד קבצי SRA של מזהי גישה מ- SRR10261601 ל- SRR10261606 במקביל, השתמש בקבצים הבאים בשורת הפקודה של Linux.
seq 10261601 10261606 | parallel prefetch SRR{} - חלץ את קבצי fastq מהארכיון באמצעות הפונקציה 'fastq-dump' מתוך ערכת הכלים של SRA. השתמש במקביל לגנו ותן את השמות של כל קבצי SRA יחד.
parallel -j 3 fastq-dump --gzip --skip-technical --read-filter pass --dumpbase --split-e --clip --origfmt {} ::: - הורד את גנום הייחוס וביאורים עבור עכבר (מכלול גנום GRCm39) מתוך www.ensembl.org באמצעות הדברים הבאים בשורת הפקודה של לינוקס.
wget -nv -O annotation.gtf.gz http://ftp.ensembl.org/pub/release-103/gtf/mus_musculus/Mus_musculus.GRCm39.103.gtf.gz \ && gunzip -f annotation.gtf.gz
wget -nv -O genome.fa.gz http://ftp.ensembl.org/pub/release-103/fasta/mus_musculus/dna/Mus_musculus.GRCm39.dna.primary_assembly.fa.gz \ && gunzip -f genome.fa.gz
GTF=$(readlink -f annotation.gtf)
GENOME=$(readlink -f genome.fa)
- הורד את הנתונים הגולמיים מארכיון קריאת רצף (SRA) באמצעות הפקודה 'prefetch' מתוך ערכת הכלים של SRA (v2.10.8)17. תן את מזהי הגישה של SRA ברצף בפקודה הבאה כדי להוריד אותם במקביל באמצעות כלי השירות המקבילי של גנו18. כדי להוריד קבצי SRA של מזהי גישה מ- SRR10261601 ל- SRR10261606 במקביל, השתמש בקבצים הבאים בשורת הפקודה של Linux.
- עיבוד מקדים ומיפוי של קריאות למכלול הגנום
- בקרת איכות. הערך את איכות הקריאות הגולמיות באמצעות FASTQC (בדיקת איכות FASTQ v0.11.9)19. צור תיקיית פלט והפעל fastqc עם מקביל על קבצי fasta קלט מרובים. שלב זה יפיק דוח איכות עבור כל מדגם. בדוק את הדוחות כדי לוודא שאיכות הקריאות מקובלת לפני ביצוע ניתוח נוסף. (עיין במדריך למשתמש להבנת הדוחות ב- https://www.bioinformatics.babraham.ac.uk/projects/fastqc/)
mkdir fastqc_out
parallel "fastqc {} -o fastqc_out" ::: $RAW_DATA/*.fastq.gz
הערה: במידת הצורך, בצע חיתוך מתאם עם 'cutadapt'20 או 'trimmomatic'21 כדי להסיר את הריצוף למתאמי אגף, המשתנה בהתאם לגודל מקטע ה- RNA ואורך הקריאה. בניתוח זה דילגנו על שלב זה מכיוון ששבריר הקריאות שהושפעו היה מינימלי. - יישור קריאה. השלב הבא בעיבוד מקדים כולל מיפוי הקריאות לגנום הייחוס. ראשית, בנה את האינדקס עבור גנום הייחוס באמצעות הפונקציה 'genomeGenerate' של STAR22 ולאחר מכן יישר את הקריאות הגולמיות לייחוס (לחלופין אינדקסים בנויים מראש זמינים מאתר STAR וניתן להשתמש בהם ישירות ליישור). הפעל את הפקודות הבאות בשורת הפקודה של Linux.
#Build STAR index
GDIR=STAR_indices
mkdir $GDIR
STAR --runMode genomeGenerate --genomeFastaFiles $GENOME --sjdbGTFfile $GTF --runThreadN 8 --genomeDir $GDIR
ODIR=results/mapping
mkdir -p $ODIR
#Align reads to the genome
for fq1 in $RAW_DATA/*R1.fastq.gz;
do
fq2=$(echo $fq1| sed 's/1.fastq.gz/2.fastq.gz/g');
OUTPUT=$(basename ${fq1}| sed 's/R1.fastq.gz//g');
STAR --genomeDir $GDIR \
--runThreadN 12 \
--readFilesCommand zcat \
--readFilesIn ${fq1}${fq2}\
--outFileNamePrefix $ODIR\/${OUTPUT} \
--outSAMtype BAM SortedByCoordinate \
--outSAMunmapped Within \
--outSAMattributes Standard
Done
הערה: יישור ה-STAR ייצור וימיין קבצי BAM (מפת יישור בינארית) עבור כל דגימה לאחר קריאת היישור. יש למיין את קבצי Bam לפני שתמשיך לשלבים נוספים.
- בקרת איכות. הערך את איכות הקריאות הגולמיות באמצעות FASTQC (בדיקת איכות FASTQ v0.11.9)19. צור תיקיית פלט והפעל fastqc עם מקביל על קבצי fasta קלט מרובים. שלב זה יפיק דוח איכות עבור כל מדגם. בדוק את הדוחות כדי לוודא שאיכות הקריאות מקובלת לפני ביצוע ניתוח נוסף. (עיין במדריך למשתמש להבנת הדוחות ב- https://www.bioinformatics.babraham.ac.uk/projects/fastqc/)
- הורדת הנתונים הגולמיים.
- הכנת ביאורים של אקסון.
- הפעל את קובץ הקוד המשלים "prepare_mm_exon_annotation. R" עם הביאור שהורדת בפורמט GTF (פורמט העברת גנים) כדי להכין את הביאורים. כדי להפעיל, הקלד את הפרטים הבאים בשורת הפקודה של Linux.
Rscript prepare_mm_exon_annotation.R annotation.gtf
הערה: קובץ GTF מכיל ערכי אקסון מרובים עבור איזופורמים שונים. קובץ זה משמש כדי "לכווץ" את מזהי התמלילים המרובים עבור כל אקסון. זהו צעד חשוב להגדרת פחי ספירת אקסון.
- הפעל את קובץ הקוד המשלים "prepare_mm_exon_annotation. R" עם הביאור שהורדת בפורמט GTF (פורמט העברת גנים) כדי להכין את הביאורים. כדי להפעיל, הקלד את הפרטים הבאים בשורת הפקודה של Linux.
- הספירה קוראת. השלב הבא הוא לספור את מספר הקריאות הממופות לתמלילים/אקסונים שונים. ראה קובץ משלים: "AS_analysis_RNASeq.Rmd".
- טען ספריות נדרשות:
packages<- c("Rsubread","tidyverse", "magrittr", "EnhancedVolcano", "edgeR","openxlsx")
invisible(lapply(packages, library, character.only=TRUE)) - טען את קובץ הביאור המעובד שהתקבל מהשלב הקודם (2.2).
load("mm_exon_anno.RData") - קרא את כל קבצי ה-bam שהתקבלו בשלב 2.2.2 כקלט עבור 'featureCounts' כדי לספור קריאות. קרא את התיקיה המכילה קבצי bam על-ידי רישום תחילה של כל קובץ מהספריה המסתיימת ב- .bam. השתמש ב-'featureCounts' מחבילת Rsubread שלוקחת קבצי BAM ומעובדת ביאור GTF (הפניה) כקלט כדי ליצור מטריצה של ספירות המשויכות לכל תכונה עם שורות המייצגות אקסונים (תכונות) ועמודות המייצגות דוגמאות.
countData <- dir("bams", pattern=".bam$", full.names=T) %>%
featureCounts(annot.ext=anno,
isGTFAnnotationFile=FALSE,
minMQS=0,useMetaFeatures=FALSE,
allowMultiOverlap=TRUE,
largestOverlap=TRUE,
countMultiMappingReads=FALSE,
primaryOnly=TRUE,
isPairedEnd=TRUE,
nthreads=12) - לאחר מכן, בצע סינון לא ספציפי כדי להסיר אקסונים בעלי ביטוי נמוך ("לא ספציפי" מציין שמידע מצב הניסוי אינו משמש בסינון, כדי למנוע הטיות בחירה). המר את הנתונים מקנה מידה גולמי לספירה למיליון (cpm) באמצעות הפונקציה cpm מחבילת 'edgeR'23 ושמור אקסונים עם ספירות הגדולות מסף ניתן להגדרה (עבור ערכת נתונים זו נעשה שימוש ב- cpm אחד) בשלוש דגימות לפחות. גם להסיר גנים עם אקסון אחד בלבד.
# Non-specific filtering: Remove the exons with low counts
isexpr<- rownames(countData$counts)[rowSums(cpm(countData$counts)>1) >=3]
countData$counts<-countData$counts[rownames(countData$counts) %in%isexpr, ]
anno<-anno%>% filter(GeneID%in% rownames(countData$counts))
# Remove genes with only 1 site and NA in geneIDs
dn<-anno%>%group_by(GeneID)%>%summarise(nsites=n())%>% filter(nsites>1&!is.na(GeneID))
anno<-anno%>% filter(GeneID%in%dn$GeneID)
countData$counts<-countData$counts[rownames(countData$counts) %in%anno$GeneID, ]
הערה: בדוק את הפרמטרים הנדרשים עבור featureCounts בעת שימוש בנתונים שונים, לדוגמה, עבור קריאות חד-צדדיות, הגדר 'isPairedEnd = FALSE'. עיין במדריך למשתמש של RSubread כדי לבחור את האפשרויות עבור הנתונים שלך, ועיין בסעיף הדיון להלן.
- טען ספריות נדרשות:
- שחבור דיפרנציאלי וניתוח שימוש באקסון. אנו מתארים שתי חלופות לשלב זה: DEXSeq ו- DiffSplice. כל אחד מהם יכול לשמש ולתת תוצאות דומות. לקבלת עקביות, בחר DEXSeq אם אתה מעדיף חבילת DESeq2 עבור DGE והשתמש ב- DiffSplice לניתוח DGE מבוסס לימה. ראה קובץ משלים: "AS_analysis_RNASeq.RMD".
- שימוש בחבילת DEXSeq לניתוח אקסון דיפרנציאלי.
- טען ספרייה וצור טבלה לדוגמה כדי להגדיר את התכנון הניסיוני.
library(DEXSeq)
sampleTable<-data.frame(row.names= c("Mbnl1KO_Thymus_1", "Mbnl1KO_Thymus_2", "Mbnl1KO_Thymus_3", "WT_Thymus_1", "WT_Thymus_2", "WT_Thymus_3"), condition= rep(c("Mbnl1_KO", "WT"),c(3,3)), libType= rep(c("paired-end")))
הערה: שמות השורות צריכים להיות עקביים עם שמות הקבצים של bam המשמשים את featureCounts כדי לספור את הקריאות. טבלת דוגמאות מורכבת מפרטים של כל מדגם הכולל: סוג ספרייה ומצב. זה נדרש כדי להגדיר את הניגודים או קבוצת הבדיקה לאיתור שימוש דיפרנציאלי. - הכן את קובץ המידע של exon. מידע אקסון בצורה של GRanges (טווחים גנומיים) אובייקטים (https://bioconductor.org/packages/release/bioc/html/GenomicRanges.html) נדרש כקלט כדי ליצור את האובייקט DEXSeq בשלב הבא. התאם את מזהי הגנים לספירות הקריאה כדי ליצור אובייקט exoninfo.
exoninfo<-anno[anno$GeneID%in% rownames(countData$counts),]
exoninfo<-GRanges(seqnames=anno$Chr,
ranges=IRanges(start=anno$Start, end=anno$End, width=anno$Width),strand=Rle(anno$Strand))
mcols(exoninfo)$TranscriptIDs<-anno$TranscriptIDs
mcols(exoninfo)$Ticker<-anno$Ticker
mcols(exoninfo)$ExonID<-anno$ExonID
mcols(exoninfo)$n<-anno$n
mcols(exoninfo)$GeneID<-anno$GeneID
transcripts_l= strsplit(exoninfo$TranscriptIDs, "\\,")
save(countData, sampleTable, exoninfo, transcripts_l, file="AS_countdata.RData") - צור אובייקט DEXSeq באמצעות הפונקציה DEXSeqDataSet. אובייקט DEXSeq אוסף יחד ספירות קריאה, מידע על תכונות exon ומידע לדוגמה. השתמש בספירות הקריאה שנוצרו בשלב 3 ובמידע exon המתקבל מהשלב הקודם כדי ליצור את האובייקט DEXSeq ממטריצת הספירה. הארגומנט sampleData מקבל קלט מסגרת נתונים המגדיר את הדוגמאות (ואת התכונות שלהן: סוג ספרייה ותנאי), 'design' משתמש ב- sampleData כדי ליצור מטריצת עיצוב לבדיקה הדיפרנציאלית באמצעות סימון נוסחת מודל. שימו לב שמונח אינטראקציה משמעותי, condition:exon, מציין ששבריר הקריאות מעל גן שנופל על אקסון מסוים תלוי בתנאי הניסוי, כלומר יש AS. עיין בתיעוד של DEXSeq לקבלת תיאור מלא של הגדרת נוסחת המודל עבור עיצובים ניסיוניים מורכבים יותר. לקבלת מידע על תכונות, יש צורך במזהי exon, גנים מתאימים ותעתיקים.
dxd<-DEXSeqDataSet(countData$counts,sampleData=sampleTable, design=~sample+exon+condition:exon,featureID=exoninfo$ExonID,groupID=exoninfo$GeneID,featureRanges=exoninfo, transcripts=transcripts_l) - נורמליזציה ואומדן פיזור. לאחר מכן, לבצע נורמליזציה בין דגימות ולהעריך את השונות של הנתונים, בשל שני רעש ספירת פואסון מן האופי הבדיד של RNA-seq ושונות ביולוגית, באמצעות הפקודות הבאות.
dxd %<>% estimateSizeFactors %>% estimateDispersions %T>% plotDispEsts - בדיקה לשימוש דיפרנציאלי. לאחר הערכת השונות, בדוק את השימוש באקסון דיפרנציאלי עבור כל גן והפק את התוצאות.
dxd%<>%testForDEU%>%estimateExonFoldChanges(fitExpToVar=
"condition")#Estimate fold changes
dxr=DEXSeqResults(dxd) - הצג באופן חזותי אירועי שחבור עבור גנים נבחרים באמצעות הפקודה הבאה.
plotDEXSeq(dxr,"Wnk1", displayTranscripts=TRUE, splicing=TRUE,legend
=TRUE,cex.axis=1.2,cex=1.3,lwd=2)
בחן את קובץ מחברת R "AS_analysis_RNASeq.Rmd" כדי ליצור חלקות נוספות עבור גנים מעניינים וליצור חלקות הר געש בסף שונה.
- טען ספרייה וצור טבלה לדוגמה כדי להגדיר את התכנון הניסיוני.
- שימוש ב-diffSplice מלימה לזיהוי שחבור דיפרנציאלי. עקוב אחר קובץ המחברת R "AS_analysis_RNASeq.RMD". ודא ששלבים 2.1-2.3 בוצעו כדי להכין קבצי קלט לפני שתמשיך הלאה.
- טעינת ספריות
library(limma)
library(edgeR) - סינון לא ספציפי. חלץ את מטריצת ספירות הקריאה המתקבלות ב- 2.3. צור רשימה של תכונות באמצעות הפונקציה 'DGEList' מחבילת edgeR, שבה שורות מייצגות גנים ועמודות מייצגות דגימות.
mycounts=countData$counts
#Change the rownames of the countdata to exon Ids instead of genes for unique rownames.
rownames(mycounts) = exoninfo$ExonID
dge<-DGEList(counts=mycounts)
#Filtering
isexpr<- rowSums(cpm(dge)>1) >=3
dge<-dge[isexpr,,keep.lib.sizes=FALSE]
#Extract the exon annotations for only transcripts meeting non-specific filter
exoninfo=anno%>% filter(ExonID%in% rownames(dge$counts))
#Convert the exoninfo into GRanges object
exoninfo1<-GRanges(seqnames=exoninfo$Chr,
ranges=IRanges(start=exoninfo$Start, end=exoninfo$End, width=exoninfo$Width),strand=Rle(exoninfo$Strand))
mcols(exoninfo1)$TranscriptIDs<-exoninfo$TranscriptIDs
mcols(exoninfo1)$Ticker<-exoninfo$Ticker
mcols(exoninfo1)$ExonID<-exoninfo$ExonID
mcols(exoninfo1)$n<-exoninfo$n
mcols(exoninfo1)$GeneID<-exoninfo$GeneID
transcripts_l= strsplit(exoninfo1$TranscriptIDs, "\\,")
הערה: כשלב סינון לא ספציפי, ספירות מסוננות לפי cpm < 1 ב- x מתוך n דגימות, כאשר x הוא המספר המינימלי של עותקים משוכפלים בכל תנאי. n = 6 ו- x = 3 עבור נתונים לדוגמה זו. - נרמל את הספירות בין דגימות, עם הפונקציה 'calcNormFactors' מחבילת 'edgeR' באמצעות ממוצע חתוך של ערכי M (שיטת הנורמליזציה של TMM)24 היא תחשב גורמי קנה מידה כדי להתאים את גודל הספרייה.
dge<-calcNormFactors(dge) - השתמש בטבלה לדוגמה כפי שנוצרה בשלב 2.4.1.1 וצור את מטריצת העיצוב. מטריצת העיצוב מאפיינת את העיצוב. עיין במדריך למשתמש של Limma (https://www.bioconductor.org/packages/devel/bioc/vignettes/limma/inst/doc/usersguide.pdf) פרקים 8 ו-9 לקבלת פרטים על מטריצות תכנון עבור עיצובים ניסיוניים מתקדמים יותר.
Treat<- factor(sampleTable$condition)
design<- model.matrix(~0+Treat)
colnames(design) <- levels(Treat) - התאם מודל ליניארי לכל אקסון. הפעל פונקציית 'voom' של חבילת 'לימה' כדי לעבד נתוני RNA-seq כדי להעריך שונות וליצור משקלים מדויקים לתיקון רעש ספירת פואסון, ולהפוך את הספירות ברמת אקסון ל- log2-counts למיליון (logCPM). לאחר מכן הפעל מידול ליניארי באמצעות פונקציית 'lmfit' כדי להתאים מודלים ליניאריים לנתוני הביטוי עבור כל אקסון. חישוב נתונים סטטיסטיים אמפיריים של בייס עבור מודל מותאם באמצעות פונקציית 'eBayes' כדי לזהות ביטוי אקסון דיפרנציאלי. לאחר מכן, הגדר מטריצת ניגודיות להשוואות הניסוייות המעניינות. השתמש ב- 'contrasts.fit' כדי להשיג מקדמים ושגיאות תקן עבור כל זוג של השוואה.
v<-voom(dge,design,plot=FALSE)
fit<-lmFit(v,design)
fit<-eBayes(fit)
colnames(fit)
cont.matrix<-makeContrasts(
Mbnl1_KO_WT=Mbnl1_KO-WT,
levels=design)
fit2<-contrasts.fit(fit,cont.matrix) - ניתוח שחבור דיפרנציאלי. הפעל את 'diffSplice' על המודל המותאם כדי לבחון את ההבדלים בשימוש באקסון של גנים בין סוג בר לנוקאאוט ולחקור את התוצאות המדורגות ביותר באמצעות פונקציית 'topSplice': test="t" נותן דירוג של אקסונים AS, test="simes" נותן דירוג של גנים.
ex<-diffSplice(fit2,geneid=exoninfo$GeneID,exonid=exoninfo$ExonID)
ts<-topSplice(ex,n=Inf,FDR=0.1, test="t", sort.by="logFC")
tg<-topSplice(ex,n=Inf,FDR=0.1, test="simes") - ויזואליזציה. התווה את התוצאות באמצעות הפונקציה 'plotSplice', המעניקה גן בעל עניין בארגומנט הגנטי. שמור את התוצאות העליונות ממוינות לפי יומן שינוי קיפול לאובייקט וצור חלקת הר געש כדי להציג את האקסונים.
plotSplice(ex,geneid="Wnk1", FDR=0.1)
#Volcano plot
EnhancedVolcano(ts,lab=ts$ExonID,selectLab= head((ts$ExonID),2000), xlab= bquote(~Log[2]~'fold change'), x='logFC', y='P.Value', title='Volcano Plot', subtitle='Mbnl1_KO vs WT (Limma_diffSplice)', FCcutoff=2, labSize=4,legendPosition="right", caption= bquote(~Log[2]~"Fold change cutoff, 2; FDR 10%"))
- טעינת ספריות
- שימוש ב-rMATS
- ודא שהגרסה העדכנית ביותר של rMATS v4.1.1 (הידועה גם בשם rMATS turbo בשל זמן העיבוד המופחת ופחות דרישות זיכרון) מותקנת באמצעות conda או github (https://github.com/Xinglab/rmats-turbo/releases/download/v4.1.1/rmats_turbo_v4_1_1.tar.gz) בספריית העבודה. עקוב אחר סעיף 4.3 ב-"AS_analysis_RNASeq.Rmd".
- עבור אל התיקייה המכילה קבצי bam שהתקבלו לאחר מיפוי והכן קבצי טקסט, כנדרש על ידי rMATS, עבור שני התנאים על ידי העתקת השם של קבצי bam (יחד עם הנתיב) מופרדים על ידי ','. יש להפעיל את הפקודות הבאות בשורת הפקודה של Linux:
mkdir rMATS_analysis
cd bams/
ls -pd "$PWD"/*| grep "WT"| tr '\n'','> Wt.txt
ls -pd "$PWD"/*| grep "Mb"| tr '\n'','> KO.txt
mv *.txt ../rMATS_analysis - הפעל rmats.py עם שני קבצי הקלט שנוצרו בשלב הקודם, יחד עם קובץ GTF שהושג ב- 2.1.1.3. פעולה זו תיצור תיקיית פלט 'rmats_out' המכילה קבצי טקסט המתארים נתונים סטטיסטיים (ערכי p ורמות הכללה) עבור כל אירוע שחבור בנפרד.
python rmats-turbo/rmats.py --b1 KO.txt --b2 Wt.txt --gtf annotation.gtf -t paired --readLength 50 --nthread 8 --od rmats_out/ --tmp rmats_tmp --task pos
הערה: נדרש גם ביאור הפניה בצורה של קובץ GTF. בדוק את הפרמטרים אם הנתונים הם חד-צדדיים, ושנה את האפשרות -t בהתאם. - בחינת תוצאות rMATS. השתמש בחבילת המוליכים הביולוגיים 'maser'25 כדי לחקור את תוצאות rMATS. טען את קבצי הטקסט של Junction ו- exon counts (JCEC) לאובייקט 'maser' וסנן את התוצאה בהתבסס על כיסוי על-ידי הכללת לפחות חמש קריאות ממוצעות לכל אירוע שחבור.
library(maser)
mbnl1<-maser("/rmats_out/", c("WT","Mbnl1_KO"), ftype="JCEC")
#Filtering out events by coverage
mbnl1_filt<-filterByCoverage(mbnl1,avg_reads=5) - הדמיה של תוצאות rMATS. בחר את אירועי השחבור המשמעותיים בשיעור גילוי כוזב (FDR) 10% ושינוי מינימלי של 10% באחוז שחבור ב- (deltaPSI) באמצעות הפונקציה 'topEvents' מחבילת 'maser'. לאחר מכן, בדוק את אירועי הגן עבור גנים בודדים בעלי עניין (מדגם גן-Wnk1) והתווה ערכי PSI עבור כל אירוע שחבור של אותו גן. צור תרשים הר געש על-ידי ציון סוג האירוע.
#Top splicing events at 10% FDR
mbnl1_top<-topEvents(mbnl1_filt,fdr=0.1, deltaPSI=0.1)
mbnl1_top
#Check the gene events for a particular gene
mbnl1_wnk1<-geneEvents(mbnl1_filt,geneS="Wnk1", fdr=0.1, deltaPSI=0.1)
maser::display(mbnl1_wnk1,"SE")
plotGenePSI(mbnl1_wnk1,type="SE", show_replicates
=TRUE)
volcano(mbnl1_filt,fdr=0.1, deltaPSI=0.1,type="SE")
+xlab("deltaPSI")+ylab("Log10 Adj. Pvalue")+ggtitle("Volcano Plot of exon skipping events") - צור עלילות סשימי עבור תוצאת אירועי השחבור המתקבלת עם rMATS בצורה של קבצי טקסט באמצעות חבילת 'rmats2shahimiplot'. הפעל את סקריפט הפייתון בשורת הפקודה של לינוקס.
python ./src/rmats2sashimiplot/rmats2sashimiplot.py --b1 ../bams/WT_Thymus_1.bam,../bams/WT_Thymus_2.bam,../bams/WT_Thymus_3.bam --b2 ../bams/Mbnl1KO_Thymus_1.bam,../bams/Mbnl1KO_Thymus_2.bam,../bams/Mbnl1KO_Thymus_3.bam -t SE -e ../rMATS_analysis/rmats_out/SE.MATS.JC.txt --l1 WT --l2 Mbnl1_KO --exon_s 1 --intron_s 5 -o ../rMATS_analysis/rmats2shasmi_output
הערה: תהליך זה עשוי לגזול זמן רב מכיוון שהוא ייצור את עלילת סשימי עבור כל התוצאות בקובץ האירועים. בחר את התוצאות המובילות (שמות גנים ואקסונים) כפי שהן מוצגות על-ידי הפונקציה topEvents מתוך 'maser' והצג באופן חזותי את עלילת הסשימי המתאימה.
- שימוש בחבילת DEXSeq לניתוח אקסון דיפרנציאלי.
3. ניתוח פוליאדנילציה חלופית (APA) באמצעות ריצוף קצה 3'
- הורדה ועיבוד מראש של נתונים
הערה: עיין בקובץ מחברת R המשלים "APA_analysis_3PSeq_notebook. Rmd" עבור הפקודות המלאות להורדת נתונים ושלבי עיבוד מראש, או הפעל את קובץ bash המשלים "APA_data_downloading_preprocessing.sh" בשורת הפקודה של לינוקס.- הורד נתונים מ-SRA עם מזהי ההצטרפות (1553129 עד 1553136).
- חתוך מתאמים והשלמה הפוכה כדי לקבל את רצף גדילי החישה.
הערה: שלב זה הוא ספציפי לבדיקת PolyA-seq שבה נעשה שימוש. - המפה קוראת להרכבת גנום העכבר באמצעות קשת26.
- הכנת ביאורים לאתרי pA.
הערה: העיבוד של קובץ הביאור של אתר ה- pA מתבצע תחילה באמצעות קובץ מחברת R משלים "APA_analysis_3PSeq_notebook. Rmd" (2.1 - 2.6), ולאחר מכן באמצעות קובץ bash "APA_annotation_preparation.sh".- הורד ביאור אתרי pA ממסד הנתונים PolyASite 2.06.
- בחר ביאורים של אתרי pA כדי לשמור על אתרי pA של 3'-אזור לא מתורגם (UTR), המבוארים כ- Terminal Exon (TE) או 1000 nt במורד הזרם של אקסון מסוף מבואר (DS) לניתוח במורד הזרם.
- השג פסגות אתר pA. עוגן בכל אתר מחשוף pA, והצג באופן חזותי את כיסוי הקריאה הממוצע באמצעות מצעיםו-deeptools 27,28. התוצאות הראו כי הפסגות של הקריאות הממופות פוזרו בעיקר בטווח של ~60 bp במעלה הזרם של אתרי הבקיעה (איור 5 ואיור משלים 5). לכן, הקואורדינטות של אתרי pA הורחבו מקובץ הביאור ל-60 bp במעלה הזרם של אתרי הבקיעה שלהם. בהתאם לפרוטוקול הריצוף הסופי הספציפי בגודל 3 אינץ' שבו נעשה שימוש, יהיה צורך למטב שלב זה למבחנים שאינם PolyA-seq.
- ספירת קריאות
- הכן את קובץ הביאור של אתרי pA.
anno<- read.table(file= "flanking60added.pA_annotation.bed",
stringsAsFactors=FALSE, check.names=FALSE, header=FALSE, sep="")
colnames(anno) <- c("chrom", "chromStart", "chromEnd", "name", "score", "strand", "rep", "annotation", "gene_name", "gene_id")
anno<- dplyr::select(anno,name,chrom, chromStart,chromEnd, strand,gene_id,gene_name,rep)
colnames(anno) <- c("GeneID", "Chr", "Start", "End", "Strand", "Ensembl", "Symbol", "repID") - החל 'featureCounts' כדי לרכוש ספירות גולמיות. שמור את טבלת הספירה כקובץ RData "APA_countData.Rdata" לניתוח APA באמצעות כלים שונים.
countData<- dir("bamfiles", pattern="sorted.bam$", full.names=TRUE) %>%
# Read all bam files as input for featureCounts
featureCounts(annot.ext=anno, isGTFAnnotationFile= FALSE,minMQS=0,useMetaFeatures= TRUE,allowMultiOverlap=TRUE, largestOverlap= TRUE,strandSpecific=1, countMultiMappingReads =TRUE,primaryOnly= TRUE,isPairedEnd= FALSE,nthreads=12)%T>%
save(file="APA_countData.Rdata")
הערה: שים לב לשנות כל אחד מהפרמטרים המפורטים בפונקציה 'featureCounts'. שנה את הפרמטר 'strandSpecific' כדי להבטיח שהוא תואם את כיוון הריצוף של בדיקת הריצוף הסופית של 3' המשמשת (באופן אמפירי, הדמיה של הנתונים בדפדפן גנום על פני גנים על גדילי פלוס ומינוס תבהיר זאת). - החל סינון לא ספציפי של countData. סינון יכול לשפר משמעותית את החוסן הסטטיסטי במבחני שימוש דיפרנציאליים באתר pA. ראשית, הסרנו את הגנים האלה עם אתר pA אחד בלבד, שבו לא ניתן להגדיר באופן דיפרנציאלי את השימוש באתר pA. שנית, אנו מיישמים סינון לא ספציפי המבוסס על כיסוי: ספירות מסוננות לפי cpm פחות מ-1 ב-x מתוך n דגימות, כאשר x הוא המספר המינימלי של שכפולים בכל מצב. N = 8 ו- x = 2 עבור נתונים לדוגמה זו.
load(file= "APA_countData.Rdata")# Skip this step if already loaded
# Non-specific filtering: Remove the pA sites not differentially expressed in the samples
countData<-countData$counts%>%as.data.frame%>% .[rowSums(edgeR::cpm(.)>1) >=2, ]
anno%<>% .[.$GeneID%in% rownames(countData), ]
# Remove genes with only 1 site and NA in geneIDs
dnsites<-anno%>%group_by(Symbol)%>%summarise(nsites=n())%>% filter(nsites>1&!is.na(Symbol))
anno<-anno%>% filter(Symbol%in%dnsites$Symbol)
countData<-countData[rownames(countData) %in%anno$GeneID, ]
- הכן את קובץ הביאור של אתרי pA.
- ניתוח שימוש דיפרנציאלי באתר פוליאדנילציה באמצעות צינורות DEXSeq ו- diffSplice.
- שימוש בחבילת DEXSeq
הערה: מכיוון שלא ניתן להגדיר מטריצת ניגודיות עבור צינור DEXSeq, ניתוח APA דיפרנציאלי של כל שני תנאי ניסוי צריך להתבצע בנפרד. ניתוח APA דיפרנציאלי של התנאי WT והתנאי DKO מבוצע כדוגמה כדי להסביר את ההליך. עיין בקובץ המשלים "APA_analysis_3PSeq_notebook. RMD" עבור זרימת העבודה שלב אחר שלב של סעיף זה וניתוח APA דיפרנציאלי של ניגודים אחרים.- טען ספרייה וצור טבלה לדוגמה כדי להגדיר את התכנון הניסיוני.
c("DEXSeq", "GenomicRanges") %>% lapply(library, character.only=TRUE) %>%invisible
sampleTable1<- data.frame(row.names= c("WT_1","WT_2","DKO_1","DKO_2"),
condition= c(rep("WT", 2), rep("DKO", 2)),
libType= rep("single-end", 4)) - הכן את קובץ המידע של אתרי pA באמצעות חבילת Bioconductor GRanges.
# Prepare the GRanges object for DEXSeqDataSet object construction
PASinfo <- GRanges(seqnames = anno$Chr,
ranges = IRanges(start = anno$Start, end = anno$End),strand = Rle(anno$Strand))
mcols(PASinfo)$PASID<-anno$repID
mcols(PASinfo)$GeneEns<-anno$Ensembl
mcols(PASinfo)$GeneID<-anno$Symbol
# Prepare the new feature IDs, replace the strand information with letters to match the current pA site clusterID
new.featureID <- anno$Strand %>% as.character %>% replace(. %in% "+", "F") %>% replace(. %in% "-", "R") %>% paste0(as.character(anno$repID), .) - השתמש בספירות הקריאה שנוצרו בשלב 3.3 ובפרטי אתר ה- pA שהתקבלו מהשלב הקודם כדי ליצור את האובייקט DEXSeq.
# Select the read counts of the condition WT and DKO
countData1<- dplyr::select(countData, SRR1553129.sorted.bam, SRR1553130.sorted.bam, SRR1553131.sorted.bam, SRR1553132.sorted.bam)
# Rename the columns of countData using sample names in sampleTable
colnames(countData1) <- rownames(sampleTable1)
dxd1<-DEXSeqDataSet(countData=countData1,
sampleData=sampleTable1,
design=~sample+exon+condition:exon,
featureID=new.featureID,
groupID=anno$Symbol,
featureRanges=PASinfo) - הגדר את זוג הניגודיות באמצעות הגדרת רמות התנאים באובייקט DEXSeq.
dxd1$condition<- factor(dxd1$condition, levels= c("WT", "DKO"))
# The contrast pair will be "DKO - WT" - נורמליזציה ואומדן פיזור. בדומה לנתוני RNA-seq, עבור נתוני ריצוף קצה של 3' בצע נורמליזציה בין דגימות (חציון מבחינת עמודה של יחסים עבור כל דגימה) באמצעות הפונקציה 'estimateSizeFactors', והערך את השונות של הנתונים באמצעות הפונקציה 'estimateDispersions', ולאחר מכן הצג באופן חזותי את תוצאת אומדן הפיזור באמצעות הפונקציה 'plotDispEsts'.
dxd1 %<>% estimateSizeFactors %>% estimateDispersions %T>% plotDispEsts - בדיקת שימוש דיפרנציאלית באתר pA עבור כל גן באמצעות הפונקציה 'testForDEU', ולאחר מכן להעריך את שינוי הקיפול של השימוש באתר pA באמצעות הפונקציה 'estimateExonFoldChanges'. בדוק את התוצאות באמצעות הפונקציה 'DEXSeqResults' והגדר 'FDR < 10%' כקריטריון לאתרי pA דיפרנציאליים משמעותיים.
dxd1 %<>% testForDEU %>% estimateExonFoldChanges(fitExpToVar = "condition")
dxr1 <- DEXSeqResults(dxd1)
dxr1
mcols(dxr1)$description
table(dxr1$padj<0.1) # Check the number of differential pA sites (FDR < 0.1)
table(tapply(dxr1$padj<0.1, dxr1$groupID, any)) # Check the number of gene overlapped with differential pA site - הדמיה של תוצאות השימוש באתר pA דיפרנציאלי באמצעות חלקות APA דיפרנציאליות שנוצרו על ידי הפונקציה 'plotDEXSeq' ומגרש הר געש על ידי הפונקציה 'EnhancedVolcano'.
# Select the top 100 significant differential pA sites ranked by FDR
topdiff.PAS<- dxr1%>%as.data.frame%>%rownames_to_column%>%arrange(padj)%$%groupID[1:100]
# Apply plotDEXSeq for the visualization of differential polyA usage
plotDEXSeq(dxr1,"S100a7a", legend=TRUE, expression=FALSE,splicing=TRUE, cex.axis=1.2, cex=1.3,lwd=2)
# Apply perGeneQValue to check the top genes with differential polyA site usage
dxr1%<>% .[!is.na(.$padj), ]
dgene<- data.frame(perGeneQValue= perGeneQValue (dxr1)) %>%rownames_to_column("groupID")
dePAS_sig1<-dxr1%>% data.frame() %>%
dplyr::select(-matches("dispersion|stat|countData|genomicData"))%>%
inner_join(dgene)%>%arrange(perGeneQValue)%>%distinct()%>%
filter(padj<0.1)
# Apply EnhancedVolcano package to visualise differential polyA site usage
"EnhancedVolcano"%>% lapply(library, character.only=TRUE) %>%invisible
EnhancedVolcano(dePAS_sig1, lab=dePAS_sig1$groupID, x='log2fold_DKO_WT',
y='pvalue',title='Volcano Plot',subtitle='DKO vs WT',
FCcutoff=1,labSize=4, legendPosition="right",
caption= bquote(~Log[2]~"Fold change cutoff, 1; FDR 10%"))
- טען ספרייה וצור טבלה לדוגמה כדי להגדיר את התכנון הניסיוני.
- באמצעות חבילת diffSplice. עיין בקובץ מחברת R המשלים "APA_analysis_3PSeq_notebook. Rmd" עבור זרימת העבודה שלב אחר שלב של סעיף זה.
- הגדר ניגודים מעניינים לניתוח שימוש דיפרנציאלי ב- pA.
הערה: שלב זה צריך להתבצע לאחר בנייה ועיבוד של אובייקט DGEList, אשר כלול בקובץ מחברת R "APA_analysis_3PSeq_notebook. רמ"ד".contrast.matrix<-makeContrasts(DKO_vs_WT=DKO-
WT,Ctrl_vs_DKO=Ctrl-DKO,
KD_vs_Ctrl=KD-Ctrl,KD_vs_DKO=KD-DKO,levels=design)
fit2<-fit%>%contrasts.fit(contrast.matrix)%>%eBayes
summary(decideTests(fit2))
ex<-diffSplice(fit2,geneid=anno$Symbol,exonid=new.featureID)
topSplice(ex) #Check the top significant results with topSplice - דמיינו את התוצאה של ניגודים מעניינים (כאן "DKO - WT") באמצעות חלקות APA דיפרנציאליות על ידי הפונקציה 'plotSplice' וחלקות הר געש עם הפונקציה 'EnhancedVolcano'. עיין בקובץ מחברת R "APA_analysis_3PSeq_notebook. Rmd" 4.2.7 - 4.2.9 להדמיה של זוגות ניגודיות אחרים.
sig1<-topSplice(ex,n=Inf,FDR=0.1,coef=1, test="t", sort.by="logFC")
sig1.genes<-topSplice(ex,n=Inf,FDR=0.1,coef=1, test="simes")
plotSplice(ex, coef=1,geneid="S100a7a", FDR = 0.1)
plotSplice(ex,coef=1,geneid="Tpm1", FDR = 0.1)
plotSplice(ex,coef=1,geneid="Smc6", FDR = 0.1)
EnhancedVolcano(sig1, lab=sig1$GeneID,xlab= bquote(~Log[2]~'fold change'),
x='logFC', y='P.Value', title='Volcano Plot', subtitle='DKO vs WT',
FCcutoff=1, labSize=6, legendPosition="right")
- הגדר ניגודים מעניינים לניתוח שימוש דיפרנציאלי ב- pA.
- שימוש בחבילת DEXSeq
תוצאות
לאחר הפעלת זרימת העבודה שלב אחר שלב לעיל, פלטי הניתוח AS ו- APA והתוצאות המייצגות הם בצורה של טבלאות ומתווי נתונים, שנוצרו כדלקמן.
כפי:
התפוקה העיקרית של ניתוח AS (טבלה משלימה 1 עבור diffSplice; טבלה 2 עבור DEXSeq) היא רשימה של אקסונים המציגים שימוש דיפרנציאלי בין תנאים, ורשימת גנים המציגים פעילות שחבור כוללת משמעותית של אחד או יותר מהאקסונים המרכיבים אותו, המדורגים לפי מובהקות סטטיסטית. טבלה משלימה 1, לשונית 2 מציגה אקסונים משמעותיים, עם עמודות המציגות FC דיפרנציאלי של אקסון לעומת מנוחה, ערך p לא מותאם לכל אקסון וערך p מותאם (תיקון בנימיני-הוקברג). סף על ערכי ה- p המותאמים ייתן קבוצה של אקסונים עם FDR מוגדר. טבלה משלימה 1, לשונית 3 מציגה רשימה מדורגת של גנים המציגים משמעות של פעילות שחבור כוללת, עם עמודה המציגה ערך p מותאם ברמת הגן המחושב באמצעות שיטת סימס. נתונים דומים מוצגים בטבלה 2 עבור DEXSeq. איור משלים 1 ואיור משלים 2 מראים דפוס שחבור דיפרנציאלי בגנים Mbnl1, Tcf7 ו-Lef1 אשר אומתו בניסוי במאמר שפורסם עם הנתונים15. החוקרים הראו אימות ניסיוני של חמישה גנים - Mbnl1, Mbnl2, Lef1, Tcf7 ו- Ncor2. הגישה שלנו זיהתה פטנים של שחבור דיפרנציאלי בכל הגנים האלה. כאן אנו מציגים את רמות FDR עבור כל גן באמצעות DEXSeq, diffSplice ו- rMATS בהתאמה כפי שמתקבל בטבלאות משלימות 1-3: Mbnl1 (0, 6.6E-61 ,0), Mbnl2 (0,0.18,0), Lef1 (1.4E-10, 1.3E-04, 0), Tcf7 (0, 1.1E-6, 0) ו- Ncor2 (9.2E-11, 1.6E-06, 0).
איור 2 מציג השוואה בין יציאות המתקבלות משלושה כלים שונים וממחיש דפוסי שחבור חלופיים בגן Wnk1 . חלקות הר הגעש מוצגות באיור 2A (diffSplice) ובאיור 2B (DEXSeq). שלושה גנים נוספים בעלי דירוג גבוה מוצגים באיור משלים 1 (diffSplice) ובאיור משלים 2 (DEXSeq). ציר ה-y מראה מובהקות סטטיסטית (-log10 P-values) וציר ה-x מראה את גודל האפקט (שינוי קיפול). גנים הממוקמים ברביע השמאלי או הימני העליון מצביעים על FC משמעותי וראיות סטטיסטיות חזקות להבדלים אמיתיים.

איור 2. השוואה של תוצאות שחבור חלופיות המתקבלות מ- diffSplice, DEXSeq ו- rMATS. |
(A) חלקת הר געש (משמאל) של RNA-Seq מאנליזת Limma diffSplice: ציר ה-x מראה שינוי בקיפול אקסון יומן; ציר ה-y מציג -log10-p-value . כל נקודה מתאימה לאקסון. קו מקווקו אופקי בערך p = 1E-5; קווים מקווקווים אנכיים בשינוי פי שניים (FC). אקסונים אדומים מראים FC משמעותי ומובהקות סטטיסטית. עלילת שחבור דיפרנציאלית (מימין): תבניות שחבור מוצגות עבור גן לדוגמה Wnk1 שבו ציר ה-x מציג מזהה אקסון לכל תעתיק; ציר ה-y מציג את שינוי קיפול יומן הרישום היחסי של אקסון (ההפרש בין logFC של האקסון לבין ה-logFC הכולל עבור כל האקסונים האחרים). אקסונים המודגשים באדום מראים ביטוי דיפרנציאלי מובהק סטטיסטית (FDR < 0.1).
(B) תרשים הר געש (משמאל) ושימוש באקסון דיפרנציאלי (מימין) של RNA-Seq המתקבל מניתוח DEXSeq. הגן Wnk1 מראה שימוש דיפרנציאלי משמעותי באקסונים בין WT ל-Mbnl1 המודגשים בוורוד, המתאימים לאותם אקסונים דיפרנציאליים ב-(A).
(C) עלילת הר געש (משמאל) ומגרש סשימי (מימין) עבור Wnk1 שהתקבלו מניתוח rMATS. עלילת הר געש המתארת אירוע משמעותי של דילוג (קלטת) אקסון (SE) בסוג פראי בהשוואה לנוקאאוט ב-10% FDR עם שינוי באחוזים שחבורים בערכים (PSI או ΔΨ) > 0.1. ציר ה- x מציג שינוי בערכי PSI בין תנאים, וציר ה- y מציג ערך P של יומן. עלילת סשימי מראה אירוע אקסון מדלג בגן Wnk1, המתאים לאקסון דיפרנציאלי משמעותי ב-(A) ו-(B). כל שורה מייצגת דגימת RNA-Seq: שלושה שכפולים של סוג פראי ונוק-אאוט Mbnl1. הגובה מראה כיסוי קריאה ב- RPKM והקשתות המחברות מתארות קריאות צומת על פני אקסונים. איזופורמים חלופיים של מודל גנים מבואר מוצגים בתחתית העלילה. הפאנל התחתון של C ממחיש קריאות צומת המשמשות לחישוב סטטיסטיקת PSI.
(D) דיאגרמת Venn המשווה את מספר האקסונים הדיפרנציאליים המשמעותיים המתקבלים בשיטות השונות. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
תרשים 2 A (לוח ימני) מציג תצוגה דיאגרמתית של הבדלי אקסון של אחד הגנים המדורגים ראשונים, ומציג logFC על ציר y ומספר אקסון על ציר x. דוגמה זו מציגה אקסון קסטה המשתנה בין התנאים לגן Wnk1. עלילת השימוש באקסון דיפרנציאלי מ- DEXSeq מראה עדויות לשחבור דיפרנציאלי בחמישה אתרי אקסון ליד Wnk1.6.45. האקסונים המודגשים בוורוד צפויים להיות מתפצלים בדגימות Mbnl1 KO בהשוואה ל- WT. אקסונים אלה משלימים את התוצאות המתקבלות על ידי diffSplice אשר מראה דפוס דומה במיקום הגנומי הספציפי. דוגמאות נוספות מוצגות באיור משלים 1 ובאיור משלים 2. תצוגה מפורטת יותר כדי לאשר תוצאות מעניינות יכולה להינתן על ידי השוואת מסלולי כיסוי (נדנדה) ביחידות RPM (קריאות למיליון) בדפדפני הגנום של UCSC (אוניברסיטת סנטה קרוז) או IGV (מציג גנומיקה אינטגרטיבי) (לא מוצג), יחד עם מתאם חזותי עם מסלולים אחרים של עניין, כגון מודלים ידועים של גנים, שימור ומבחנים אחרים ברחבי הגנום.
טבלת פלט rMATS מפרטת אירועי שחבור חלופיים משמעותיים המסווגים לפי סוג (טבלה משלימה 3). איור 2C מראה חלקת הר געש של גנים שהם שחבורים חלופיים, כאשר גודל ההשפעה נמדד על ידי הסטטיסטיקה הדיפרנציאלית של "אחוז שחבור פנימה" (PSI או ΔΨ) של11.
PSI מתייחס לאחוז הקריאות העקביות עם הכללת אקסון קלטת (כלומר, קריאות מיפוי לאקסון הקלטת עצמה או קריאות צומת החופפות את האקסון) בהשוואה לקריאות התואמות את אי הכללת האקסון, כלומר קריאות צומת על פני אקסונים סמוכים במעלה הזרם ובמורד הזרם (הפאנל התחתון של איור 2C). הפאנל הימני של איור 2C מראה חלקת סשימי של גן Wnk1 עם אירוע שחבור דיפרנציאלי על מסלולי כיסוי של הגן, עם אקסון מדלג ב-Mbnl1 KO. קשתות המצטרפות לאקסונים מציגות את מספר קריאות הצמתים (קריאות חוצות אינטרון שחבור). כרטיסיות שונות של טבלה משלימה 3 מציגות קריאות משמעותיות של כל סוג של אירוע המשתרע על פני צמתים עם גבולות אקסון (ספירות צמתים וספירות אקסון (JCEC)). איור 2D משווה את האקסונים השחבורים הדיפרנציאליים המשמעותיים שזוהו על-ידי שלושת הכלים.

איור 3. אירועי שחבור חלופיים שנרכשו על ידי ניתוח rMATS. א) סוגי אירועי AS. נתון זה מותאם מתיעוד rMATS11 המסביר את אירוע השחבור עם אקסונים מכוננים ולחילופין שחבורים. מסומן עם המספר של כל אירוע ב- FDR 10%. ב) עלילת סשימי של הגן Add3 המציגה אקסון מדלג (SE). ג) חלקת סשימי של גן Baz2b המציגה אתר שחבור חלופי 5' (A5SS). D) חלקת סשימי של הגן Lsm14b המציגה אתר שחבור חלופי 3' (A3SS). E) עלילת סשימי של גן Mta1 המציגה אקסונים בלעדיים הדדית (MXE). ו) עלילת סשימי של גן Arpp21 המציגה אינטרון שמור (RI). שורות אדומות מייצגות שלושה שכפולים של שורות מסוג פראי ושורות כתומות מייצגות שכפולים של Mbnl1 נוק-אאוט. ציר ה-x מתאים לקואורדינטות גנומיות ולמידע על גדילים, ציר y מראה כיסוי ב-RPKM. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
איור 3 ממחיש סוגים של אירועי שחבור SE, A5SS, A3SS, MXE ו-RI בעזרת חלקות סשימי של הגנים המשמעותיים ביותר של אותם אירועים. בהשוואה בין שלושת המשכפלים של WT ו- Mbnl1_KO, זוהו בסך הכל 1272 אירועי SE, 130 A5SS, 116 A3SS, 215 MXE ו- 313 אירועי RI ב- FDR 10%. עלילת סשימי ממחישה את סוג האירוע באמצעות גנים עליונים כדוגמה.
APA:
הפלט מניתוח APA דומה לניתוח AS ברמת אקסון. טבלה של גנים מובילים המדורגים לפי פעילות APA דיפרנציאלית ב- 3'UTR מסופקת (טבלה משלימה 4 וטבלה משלימה 5). איור 4A מראה את חלקות הר הגעש של גנים על-ידי פעילות APA דיפרנציאלית ב-3'UTRs שנוצרו באמצעות diffSplice ו-DEXSeq בנפרד. איור 4B מציג את תרשים Venn המשווה את תוצאות השימוש באתר pA דיפרנציאלי משמעותי שנרכשו מצינורות שונים. איורים 4C ו-4D מראים את הייצוג הדיאגרמתי של שימוש באתר pA דיפרנציאלי ב-3'UTR של הגן Fosl2 (איור 4C) ו-Papola (איור 4D) שנוצר באמצעות diffSplice ו-DEXSeq, אשר מאומתים באופן ניסיוני כדי להראות תזוזה דיסטלית פרוקסימלית לפרוקסימלית משמעותית (Papola) של שימוש באתר pA ב-DKO, בהתאמה. דוגמאות נוספות מוצגות באיור משלים 3 ובאיור משלים 4.

תרשים 4. חלקות פוליאדנילציה חלופיות על ידי diffSplice ו- DEXSeq. A) חלקות הר געש של נתוני PolyA-seq שנוצרו באמצעות diffSplice ו- DEXSeq. ציר X מציג יומן pA שינוי קיפול אתר; ציר y מציג -log10-ערך p. כל נקודה מתאימה לאתר pA. קו מקווקו אופקי בערך p = 1E-5; קווים מקווקווים אנכיים ב- FC פי 2. אקסונים אדומים מראים FC משמעותי ומובהקות סטטיסטית. ב) עלילת Venn המשווה את תוצאות השימוש באתר pA דיפרנציאלי משמעותי שנרכשו מצינורות שונים. ג-ד) עלילות APA דיפרנציאליות שנוצרו באמצעות diffSplice ו- DEXSeq המציגות את אתרי ה- pA הפרוקסימליים, הפנימיים והדיסטליים עבור הגן Fosl2 ו - Papola . דמויות נוצרות על-ידי אותה פונקציה כמו איור 2 (B) אך עם אתרי pA שמחליפים אקסונים. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
איור 5 הוא עלילת אבחון המאשרת את התפלגות הקריאה הצפויה סביב אתרי ביקוע pA מבוארים עבור בדיקת PolyA-seq שבה נעשה שימוש. הוא מראה את הכיסוי הממוצע באזורי אגף המעוגנים באתרי ביקוע pA ידועים ברמה הרחבה של הגנום. במקרה זה, הערימה הצפויה של קריאות במעלה הזרם של האתרים הוא דמיה. התפלגויות הקריאה המעוגנות באתרי pA עבור כל דגימות PolyA-seq מוצגות באיור משלים 5.

איור 5. מגרש כיסוי ממוצע סביב אתרי מחשוף pA. אתר המחשוף עבור נתוני PolyA-seq מוצג על ידי קו מקווקו אנכי. ציר X מראה את מיקום הבסיס ביחס לאתרי ביקוע pA, עד 100 נוקלאוטידים במעלה הזרם ובמורד הזרם; ציר y מציג את כיסוי הקריאה הממוצע מעל כל אתרי המחשוף של pA, מנורמל לפי גודל הספרייה ב- CPM. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
ניתן להשוות ולאמת את תוצאות ה- APA הדיפרנציאליות של ניגודים שונים שנוצרו על ידי אותו צינור על ידי הדמיה של כיסוי הקריאה של אתרי pA דיפרנציאליים מייצגים בדפדפן הגנום. איור 6A הוא מתווה Venn המשווה את השימוש באתר pA דיפרנציאלי משמעותי של ניגודים שונים שנרכשו מ-diffSplice. איור 6B-D הם תצלומי IGV של כיסוי הקריאה באתרי pA עבור גנים שונים, אשר מראים את הדפוסים התואמים לאלה שהתגלו בניתוח APA באמצעות diffSplice. איור 6B מאמת את התזוזה הפרוקסימלית עד הדיסטלית המשמעותית של השימוש באתר pA עבור הגן Paip2, אשר מזוהה באופן ייחודי בניגודיות DKO לעומת WT, אך לא בשני הניגודים האחרים KD לעומת WT, ו- Ctr לעומת WT. איור 6C מאמת את התזוזה הדיסטלית המשמעותית עד הפרוקסימלית של השימוש באתר pA עבור הגן Ccl2 שזוהה באופן ייחודי בניגוד KD לעומת WT, ואילו איור 6D מאמת את השימוש המשמעותי ב-pA דיפרנציאלי של כל הניגודים עבור הגן Cacna2d1.
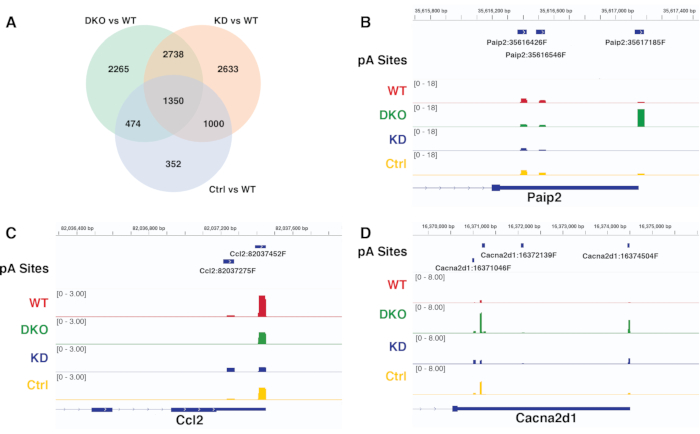
תרשים 6. השוואת ניגודיות ואימות של תוצאות diffSplice. א) דיאגרמת Venn המשווה תוצאות שימוש דיפרנציאליות משמעותיות באתר pA של ניגודים שונים שנרכשו מ- diffSplice. ב-ד) תמונת מצב של IGV הממחישה את הכיסוי של הגנים Paip2, Ccl2 ו- Cacna2d1 בתנאים שונים. כל רצועה מייצגת את כיסוי הקריאה במצב מסוים. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
איור משלים 1. ניתוח RNA-Seq של שחבור דיפרנציאלי עם Limma diffSplice. (A) תרשים הר געש של RNA-Seq מניתוח Limma diffSplice: ציר ה-x מראה שינוי בקיפול אקסון יומן; ציר ה-y מציג -log10-p-value . כל נקודה מתאימה לאקסון. קו מקווקו אופקי בערך p = 1E-5; קווים מקווקווים אנכיים בשינוי כפול (FC). אקסונים אדומים מראים FC משמעותי ומובהקות סטטיסטית. (ב-ד) חלקות שחבור דיפרנציאליות: תבניות שחבור מוצגות למשל בגנים Mbnl1, Tcf7 ו- Lef1, בהתאמה, כאשר ציר ה-x מראה מזהה אקסון לכל תעתיק; ציר ה-y מציג את שינוי קיפול יומן הרישום היחסי של אקסון (ההפרש בין logFC של האקסון לבין ה-logFC הכולל עבור כל האקסונים האחרים). אקסונים המודגשים באדום מראים ביטוי דיפרנציאלי מובהק סטטיסטית (FDR < 0.1). אנא לחץ כאן כדי להוריד קובץ זה.
איור משלים 2. ניתוח RNA-Seq של שימוש באקסון דיפרנציאלי עם DEXSeq. (א) חלקת הר געש. (ב-ד) שימוש באקסון דיפרנציאלי של RNA-Seq המתקבל מניתוח DEXSeq. גנים Mbnl1, Tcf7 ו- Lef1, בהתאמה, מראים שימוש דיפרנציאלי משמעותי באקסונים בין WT ל- Mbnl1 נוק-אאוט מודגש בוורוד, התואמים את אותם אקסונים דיפרנציאליים באיור משלים 1. אנא לחץ כאן כדי להוריד קובץ זה.
איור משלים 3. חלקות פוליאדנילציה חלופיות על ידי diffSplice. A) חלקות הר געש של נתוני PolyA-seq שנוצרו באמצעות diffSplice בשלושה זוגות ניגודיות שהתקבלו מנתוני PolyA-seq של העכבר, כולל נוקאאוט כפול (DKO) לעומת סוג פראי (WT), נוק-דאון (KD) לעומת WT, ובקרה (Ctrl) לעומת WT. ציר X מראה שינוי בקיפול אתר pA יומן; ציר y מציג -log10-ערך p. כל נקודה מתאימה לאתר pA. קו מקווקו אופקי בערך p = 1E-5; קווים מקווקווים אנכיים ב- FC פי 2. אתרי pA אדומים מראים FC משמעותי ומובהקות סטטיסטית. B) חלקות APA דיפרנציאליות שנוצרו באמצעות diffSplice המציגות את אתרי ה- pA הפרוקסימליים, הפנימיים והדיסטליים עבור הגנים המדורגים ביותר S100a7a, Tpm1 ו- Smc6. אנא לחץ כאן כדי להוריד קובץ זה.
איור משלים 4. ניתוח שימוש דיפרנציאלי ב- pA על ידי צינור DEXSeq. A) חלקות הר געש של נתוני PolyA-seq שנוצרו באמצעות DEXSeq בשלושה זוגות המתקבלים מנתוני PolyA-seq של העכבר, כולל נוקאאוט כפול (DKO) לעומת סוג פראי (WT), נוק-דאון (KD) לעומת WT, ובקרה (Ctrl) לעומת WT. ציר X מראה שינוי בקיפול אתר pA יומן; ציר y מציג -log10-ערך p. כל נקודה מתאימה לאתר pA. קו מקווקו אופקי בערך p = 1E-5; קווים מקווקווים אנכיים ב- FC פי 2. אתרי pA אדומים מראים FC משמעותי ומובהקות סטטיסטית. ב) חלקות APA דיפרנציאליות שנוצרו באמצעות DEXSeq המציגות את אתרי ה- pA הפרוקסימליים, הפנימיים והדיסטליים עבור הגנים המדורגים ביותר S100a7a, Tpm1 ו- Smc6. אנא לחץ כאן כדי להוריד קובץ זה.
איור משלים 5. מגרש כיסוי ממוצע ומפות חום סביב אתרי ביקוע pA. הכיסוי מוצג עבור ארבעה מצבים, כאשר גנים על גדילים קדימה ואחורה מוצגים בנפרד. ציר X מראה את מיקום הבסיס ביחס לאתרי ביקוע pA, עד 100 נוקלאוטידים במעלה הזרם ובמורד הזרם; ציר y מתייחס לכיסוי הממוצע במיקומי בסיס יחסיים מקבילים בכל אתרי המחשוף של pA. מפות חום מספקות תצוגה חלופית, כאשר כל אתר ביקוע pA מוצג כשורה נפרדת על ציר ה-x, מסודר לפי כיסוי. אינטנסיביות מראה סיקור קריאה (ראה מקרא). אנא לחץ כאן כדי להוריד קובץ זה.
טבלה משלימה 1. פלט diffSplice של ניתוח AS. הכרטיסייה First מגדירה את שמות העמודות עבור הפלטים המקוריים המוצגים בכרטיסיות השנייה (רמת exon) והשלישית (ברמת הגן). אנא לחץ כאן כדי להוריד טבלה זו.
טבלה משלימה 2. פלט DEXSeq של ניתוח AS. הכרטיסייה First מגדירה את שמות העמודות עבור הפלט המקורי המוצג בכרטיסיות השנייה (רמת exon) והשלישית (ברמת הגן). אנא לחץ כאן כדי להוריד טבלה זו.
טבלה משלימה 3. פלט rMATS של ניתוח AS. הכרטיסייה הראשונה מגדירה את שמות העמודות עבור קובץ הסיכום (לשונית 2) ואת קבצי JCEC עבור כל אירוע (כרטיסיה 3-7). אנא לחץ כאן כדי להוריד טבלה זו.
טבלה משלימה 4. פלט diffSplice של ניתוח APA. הכרטיסייה First מגדירה את שמות העמודות עבור הפלט המקורי המוצג בכרטיסיות השנייה (ברמת אתר pA) והשלישית (ברמת הגן). אנא לחץ כאן כדי להוריד טבלה זו.
טבלה משלימה 5. פלט DEXSeq של ניתוח APA. הכרטיסייה First מגדירה את שמות העמודות עבור הפלט המקורי המוצג בכרטיסיות השנייה (ברמת אתר pA) והשלישית (ברמת הגן). אנא לחץ כאן כדי להוריד טבלה זו.
טבלה משלימה 6. סיכום של מספר האקסונים שהשתנו באופן משמעותי עבור אתרי AS ו- pA עבור APA. אנא לחץ כאן כדי להוריד טבלה זו.
טבלה משלימה 7. סיכום של כלים וחבילות המשמשים בניתוח AS/APA. אנא לחץ כאן כדי להוריד טבלה זו.
Discussion
במחקר זה, הערכנו גישות מבוססות אקסון ומבוססות אירועים לזיהוי AS ו-APA בנתוני RNA-Seq בתפזורת ובנתוני ריצוף קצה של 3'. גישות ה-AS המבוססות על אקסון מייצרות הן רשימה של אקסונים המבוטאים באופן דיפרנציאלי והן דירוג ברמת הגן המסודר לפי המובהקות הסטטיסטית של פעילות השחבור הדיפרנציאלית הכוללת ברמת הגן (טבלאות 1-2, 4-5). עבור חבילת diffSplice, השימוש הדיפרנציאלי נקבע על ידי התאמת מודלים ליניאריים משוקללים ברמת אקסון כדי להעריך את שינוי קיפול היומן הדיפרנציאלי של אקסון כנגד שינוי קיפול היומן הממוצע של האקסונים האחרים בתוך אותו גן (הנקרא per exon FC). המובהקות הסטטיסטית ברמת הגן מחושבת על ידי שילוב מבחני מובהקות בודדים ברמת אקסון למבחן מבחינת גנים בשיטת סימס10. דירוג זה על ידי פעילות שחבור דיפרנציאלית ברמת הגן יכול לשמש לאחר מכן לביצוע ניתוח העשרת קבוצת גנים (GSEA) של מסלולי מפתח המעורבים10. DEXSeq משתמש באסטרטגיה דומה, על ידי התאמת מודל ליניארי כללי למדידת שימוש באקסון דיפרנציאלי, אם כי שונה בשלבים מסוימים כגון סינון, נורמליזציה והערכת פיזור. כאשר השווינו את 500 האקסונים המדורגים הראשונים המציגים פעילות AS ו-APA באמצעות DEXSeq ו-DiffSplice, מצאנו חפיפה של 310 אקסונים ו-300 אתרי pA, בהתאמה, המדגימה את הקונקורדנציה של שתי הגישות מבוססות האקסון, שהודגמה גם במחקר קודם 29. מומלץ להשתמש בשילוב של גישה מבוססת אקסון (DEXSeq או diffSplice) וגישה מבוססת אירועים לזיהוי וסיווג מקיפים של AS. עבור APA, משתמשים יכולים לבחור ב- DEXSeq או ב- diffSplice: שתי השיטות הוכחו כבעלות ביצועים טובים במגוון רחב של ניסויי תעתיק29.
בהכנת ספריית RNA-seq לניתוח AS, חשוב להשתמש בפרוטוקול RNA-seq בתפזורת ספציפית לגדיל 8, שכן חלק גדול מהגנים בגנומים של בעלי חוליות חופפים על גדילים שונים, ופרוטוקול שאינו ספציפי לגדילים אינו מסוגל להבחין בין אזורים חופפים אלה, מה שמבלבל את זיהוי האקסון הסופי. שיקול נוסף הוא עומק הקריאה, כאשר ניתוחי שחבור דורשים ריצוף עמוק יותר מאשר DGE, למשל 30-60 מיליון קריאות לדגימה, לעומת 5-25 מיליון קריאות לכל דגימה עבור DGE (https://sapac.support.illumina.com/bulletins/2017/04/considerations-for-rna-seq-read-length-and-coverage-.html). כל הכלים המודגמים בפרוטוקול תומכים הן בנתוני רצף חד-צדדיים והן בנתוני רצף זוגיים. אם משתמשים רק בביאורים ידועים של גנים כדי לזהות קריאות צומת, ניתן להשתמש בקריאות קצרות יותר חד-צדדיות (≥ 50 bp), אם כי זיהוי דה נובו של צמתי שחבור חדשים נהנה מקישור קצה וארוך יותר (≥ 100bp) קורא30,31. הבחירה בפרוטוקול מיצוי RNA - בחירת polyA או דלדול rRNA - תלויה באיכות הרנ"א ובשאלת הניסוי - ראה31 לדיון. בהתאם לפרטי בניית הספרייה, יידרשו שינויים בסקריפטים לדוגמה שניתנו כאן עבור הפרמטרים של יישור קריאה, ספירת תכונות ו- rMATS. בחישוב ספירות הקריאה הראשוניות ברמת אקסון באמצעות featureCounts, או שיטות דומות, יש להקפיד להגדיר את אפשרויות הפונקציה בצורה נכונה עבור ספירות וגדילות: ב- featureCounts, אנו מגדירים את הארגומנט "strandSpecific" כראוי עבור פרוטוקול RNA-seq ספציפי לגדיל המשמש; ועבור כימות ברמת האקסון צפוי שקריאה תמפה מעל אקסונים סמוכים, ולכן הגדרנו את הפרמטר allowMultiOverlap ל- TRUE. עבור APA, ישנם פרוטוקולי ריצוף קצהשונים של 3' 6 אשר משתנים במיקום המדויק של פסגות ביחס לאתר pA. עבור הנתונים לדוגמה שלנו אנו קובעים שהשיא הוא 60 bp במעלה הזרם של אתר ה-pA כפי שמוצג באיור 5, וניתוח זה יצטרך להיות מותאם לפרוטוקולי ריצוף קצה אחרים של 3'.
בפרוטוקול זה אנו מגבילים את ההיקף לדיון בניתוחים דיפרנציאליים ברמה של אקסונים בודדים, ואירועי שחבור המורכבים מצירופים סמוכים של אקסון-אינטרון. איננו דנים בסוג האנליזות המבוססות על שחזור איזופורם דה נובו כגון חפתים, Cuffdiff32, RSEM33, Kallisto34 שמטרתן לזהות ולכמת את הביטוי המוחלט והיחסי של איזופורמים חלופיים שלמים. שיטות האקסון והאירועים רגישות יותר לאיתור אירועי שחבור בודדים30 ובמקרים רבים מספקות את כל המידע הדרוש לניתוח נוסף, ללא צורך בכימות ברמת האיזופורם.
הגרסה העדכנית ביותר של קבצי המקור בפרוטוקול זה זמינה בכתובת https://github.com/jiayuwen/AS_APA_JoVE
Disclosures
למחברים אין מה לחשוף.
Acknowledgements
מחקר זה נתמך על ידי מלגת העתיד של מועצת המחקר האוסטרלית (ARC) (FT16010043) ותוכנית החוזים העתידיים של ANU.
Materials
| Name | Company | Catalog Number | Comments |
| Not relevent for computational study |
References
- Katz, Y., Wang, E. T., Airoldi, E. M., Burge, C. B. Analysis and design of RNA sequencing experiments for identifying isoform regulation. Nature Methods. 7 (12), 1009-1015 (2010).
- Wang, Y., et al. Mechanism of alternative splicing and its regulation. Biomedical Reports. 3 (2), 152-158 (2015).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in Bioinformatics. 21 (6), 2052-2065 (2020).
- Movassat, M., et al. Coupling between alternative polyadenylation and alternative splicing is limited to terminal introns. RNA Biology. 13 (7), 646-655 (2016).
- Tian, B., Manley, J. L. Alternative polyadenylation of mRNA precursors. Nature Reviews Molecular Cell Biology. 18 (1), 18-30 (2017).
- Herrmann, C. J., et al. PolyASite 2.0: a consolidated atlas of polyadenylation sites from 3' end sequencing. Nucleic Acids Research. 48 (1), 174-179 (2020).
- Liu, R., Loraine, A. E., Dickerson, J. A. Comparisons of computational methods for differential alternative splicing detection using RNA-seq in plant systems. BMC Bioinformatics. 15 (1), 364 (2014).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biology. 17 (1), 13 (2016).
- Anders, S., Reyes, A., Huber, W. Detecting differential usage of exons from RNA-seq data. Genome Research. 22 (10), 2008-2017 (2012).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2014).
- Shen, S., et al. rMATS: Robust and flexible detection of differential alternative splicing from replicate RNA-Seq data. Proceedings of the National Academy of Sciences. 111 (51), 5593-5601 (2014).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in bioinformatics. 21 (6), 2052-2065 (2020).
- Kanitz, A., et al. Comparative assessment of methods for the computational inference of transcript isoform abundance from RNA-seq data. Genome biology. 16 (1), 1-26 (2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550 (2014).
- Sznajder, L. J., et al. Loss of MBNL1 induces RNA misprocessing in the thymus and peripheral blood. Nature Communications. 11, 1-11 (2020).
- Batra, R., et al. Loss of MBNL leads to disruption of developmentally regulated alternative polyadenylation in RNA-mediated disease. Molecular Cell. 56 (2), 311-322 (2014).
- Leinonen, R., Sugawara, H., Shumway, M., et al. The sequence read archive. Nucleic acids research. 39, 19-21 (2010).
- Tange, O. . GNU parallel-the command-line power tool. 36, 42-47 (2011).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet journal. 17 (1), 10-12 (2011).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Robinson, M. D., Oshlack, A. A scaling normalization method for differential expression analysis of RNA-seq data. Genome Biology. 11 (3), 25 (2010).
- Veiga, D. F. T. maser: Mapping Alternative Splicing Events to pRoteins. R package version 1.4.0. , (2019).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (13), 25 (2009).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ramírez, F., Dündar, F., Diehl, S., Grüning, B. A., Manke, T. deepTools: a flexible platform for exploring deep-sequencing data. Nucleic acids research. 42 (1), 187-191 (2014).
- Merino, G. A., Conesa, A., Fernández, E. A. A benchmarking of workflows for detecting differential splicing and differential expression at isoform level in human RNA-seq studies. Briefings in bioinformatics. 20 (2), 471-481 (2019).
- Chhangawala, S., Rudy, G., Mason, C. E., Rosenfeld, J. A. The impact of read length on quantification of differentially expressed genes and splice junction detection. Genome biology. 16 (1), 1-10 (2015).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biol. 17, 13 (2016).
- Trapnell, C., et al. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nat Protoc. 7 (3), 562-578 (2012).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC Bioinformatics. 12, 323 (2011).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nat Biotechnol. 34 (5), 525-527 (2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved