Method Article
Истощение рибосомальной РНК для Mosquito метагеномных Gut РНК-сл
В этой статье
Резюме
Рибосомной РНК (рРНК) истощение протокол был разработан для обогащения РНК (мРНК), РНК-след от комаров metatranscriptome кишечника. Примеры специфических зондов рРНК, которые были использованы для удаления рРНК через вычитание, были созданы из комара и его микробами кишечника. Выполнение протокола может привести к удалению примерно 90-99% рРНК.
Аннотация
Кишечнике комара вмещает динамической микробных сообществ на различных этапах жизненного цикла насекомого. Характеристика генетического потенциала и функциональности кишечника сообщество обеспечит понимание последствий кишечник микрофлоры на комаров черты жизни. Метагеномных РНК-Seq стала важным инструментом для анализа transcriptomes от различных микробов, присутствующих в микробного сообщества. РНК обычно содержит всего 1-3% от общего числа РНК, а РНК составляет около 90%. Это является сложной задачей для обогащения РНК из образцов метагеномных микробной РНК, потому что большинство видов мРНК прокариот отсутствует стабильная поли (А) хвост. Это предотвращает олиго D (T) опосредованное мРНК изоляции. Здесь мы описываем протокол, который использует образцы, полученные рРНК захвата зонда для удаления рРНК из метагеномных общей выборке РНК. Для начала, как комар и микробных мелких и крупных фрагментов субъединицы рРНК усиливаются от метагеномных образца ДНК сообщества. Тогда, коммубесконечности конкретных биотинилированного антисмысловых рибосомных РНК-зондов синтезированы в пробирке использованием T7 РНК-полимеразы. Биотинилированные зонды рРНК гибридизуют к общей РНК. Гибридов захвачен покрытых стрептавидином шарики и удалить из общей РНК. Это вычитание протокола на основе эффективно удаляет как комар и микробных рРНК из общей выборки РНК. Образец мРНК обогащенный дальнейшей обработке для усиления РНК и РНК-Seq.
Введение
Следующее поколение технологии секвенирования значительно продвинулись метагеномика исследования, позволяющие оценить таксономического состава и генетической функциональности микробной сборки. РНК-последовательности (РНК-Seq) 1 может обойти культуры на основе методов для исследования микробных metatranscriptomes в различных контекстах 2-5. Основным препятствием к микробной РНК-след является трудность в обогащении мРНК, как прокариотических видов мРНК не стабильно полиаденилированных. Таким образом, олиго D (T) опосредованное посланник обогащения не применяется. Удаление обильные рРНК является альтернативным подходом к обогащению мРНК. Коммерческая истощения рРНК комплекты, такие как бактериальный комплект Microexpress обогащению мРНК (Ambion), RiboMinus транскриптома изоляции Kit (бактерии) (Life Technologies), и мРНК-ONLY Прокариотические комплект изоляции мРНК (Эпицентр), которые преимущественно деградирует рРНК с экзонуклеаза, были использованы для удаления рРНК 6-8. Тем не менее, захват зондов в Microexpressили RiboMinus хорошо для удаления известных рРНК из типичных грамположительных и грамотрицательных бактерий (см. технические характеристики производителей), но меньше, совместимый с рРНК от неизвестных микробов. Следовательно, удаление может быть менее эффективным для метагеномных 8-10 образцов. Кроме того, верности мРНК изобилие была сомнительной, когда экзонуклеаза лечения был применен 11. В целом, вычитание на основе рРНК истощения была менее предвзято и более эффективным в мРНК обогащения в метагеномных настройки 10-13.
Кишечнике комара вмещает динамической микробного сообщества 14. Мы заинтересованы в том характеризующих функцию комаров микробиомом кишечника с помощью РНК-Seq. В образце РНК изолирован от комаров кишки, как комар и микробной РНК присутствуют. Здесь мы описываем изменения протокола, используемого сообщества специфических зондов рРНК эффективно разрушают комаров и микробных рРНК субтрактивный гибридизации. Полученные мРНКобогащенной образцы подходят для РНК-Seq. В целом рабочий процесс изображен на рисунке 1.
протокол
Процедура
1. Mosquito Выращивание
- Задняя комара Anopheles gambiae G3 напряжение в insectary на 27,5 ° C при 80% влажности и 12:12 цикле ч свет / темнота.
- Поток личинки с земли корм для кошек с пивными дрожжами в соотношении 1:1.
- Поток взрослых комаров на крови мышей на 3-й день после появления всходов для производства яиц.
2. Mosquito Dissection Gut
- Автоклав инструменты диссекции (слайды и щипцы).
- Соберите 50 комары использованием аспиратора и разместить их на поток CO 2 кровати (Ultimate Flypad). Промыть комаров образца последовательно в трех чашках Петри, содержащей 70% этанола для очистки комаров поверхности тела.
- Поместите образец на предметное стекло под стереомикроскопия. Убрать живот.
- Соберите 50 кишки в условиях метагеномных для выделения ДНК. Соберите 50 кишки в условия метагеномных выделения РНК.
3. Метагеномных выделения ДНК
Примечание: метагеномных ДНК выделяли с использованием мета-G-Ном выделения ДНК Kit (Эпицентр биотехнологии) с модификацией описанных ниже.
- Место 50 комара кишки в 300 мкл TE. Однородный их в течение 1 мин с использованием био-Gen PRO200 гомогенизатора (PRO Научно Inc, США) со скоростью 2000 оборотов в минуту на льду.
- Добавить 2 мкл готового Lyse Лизоцим решение и 1 мкл РНКазы к клеточной суспензии. Смешайте встряхиванием и инкубировать при 37 ° С в течение 30 мин.
- Добавить 300 мкл Мета-Лизис решение (2X) и 1 мкл протеиназы К в трубке. Смешайте встряхиванием. Коротко импульсные трубки центрифуги обеспечить, чтобы все решения находится в нижней части трубки, и инкубировать при 65 ° С в течение 15 мин, охладить до комнатной температуры, затем место на льду в течение 3-5 мин.
- Добавить 350 мкл реагента MPC осадков белка в трубку и перемешать встряхиванием энергично в течение 10 сек.
- Пелле тОн мусора путем центрифугирования в течение 10 мин при 12000 мкг при 4 ° C.
- Передача супернатант в чистую 2-мл трубки, и отказаться от гранул.
- Добавить 570 мкл изопропанола в супернатант. Смешать путем обращения трубки в несколько раз.
- Пелле ДНК центрифугированием в течение 10 мин при 12000 мкг при 4 ° C. Снимите изопропанола без выбивании осадок ДНК.
- Добавить 500 мкл 75% этанола мыть гранул. Центрифуга в течение 5 мин при 12000 мкг при 4 ° C. Снимите этанола, не нарушая осадок ДНК. Воздушно-сухой осадок при комнатной температуре в течение 2 мин. Примечание: не более сухой осадок ДНК.
- Ресуспендируют осадок ДНК в 50 мкл буфера ТЕ.
- Подтвердить размер выделенной ДНК по сравнению с Fosmid ДНК управления (40 кб; 100 нг / мкл), входящий в комплект, с помощью гель-электрофореза в 1% агарозном геле.
4. Всего выделения РНК
Примечание: очистить рабочую областьс RNaseZap чтобы свести к минимуму загрязнение РНКазы. Метагеномных РНК выделяли из образцов кишечнике комара использованием реагентов TriPure изоляции (Roche) с незначительными изменениями.
- Положите 50 комара кишки в 2-мл пробирку с 500 мкл реагента изоляции TriPure. Однородный течение 1 мин при скорости 2000 оборотов в минуту на льду, используя гомогенизатор. Пусть сидят при комнатной температуре в течение 5 мин, чтобы диссоциации нуклеопротеида комплексов.
- Добавить 100 мкл хлороформа и вихрь в течение 12 секунд, и оставьте при комнатной температуре в течение 2 мин.
- Центрифуга при 10000 х г в течение 10 мин. Осторожно вынуть трубку, не нарушая разделенных фаз.
- Передача водной фазы в новую пробирку, добавляют 250 мкл изопропанола, хорошо перемешать и положить при температуре -20 ° С в течение 20 мин.
- Центрифуга при 12000 х г в течение 10 мин при 4 ° С для осаждения РНК.
- Удалите супернатант без выбивании гранул. Вымойте гранул с 750 мкл 75% этанола.
- Внесите от супернатант, не нарушая гранул.ИК высушить трубку в течение 2 мин.
- Ресуспендируют РНК с 30 мкл воды нуклеазы бесплатно.
- Относитесь к общей РНК ДНКазой при 37 ° С в течение 20 мин, а затем инактивации ДНКазы путем инкубации при температуре 75 ° С в течение 15 мин. Осадок РНК этанолом и ресуспендируют РНК в 30 мкл воды нуклеазы бесплатно.
- Определить количество общей РНК с использованием NanoDrop.
5. Рибосомных РНК истощения
Примечание: Протокол был разработан на основе ранее описанных методов 12,13,15.
- ПЦР-амплификации гена рибосомной фрагментов РНК
Этот шаг создает образец конкретного рРНК бассейна, который будет использоваться в качестве шаблонов в пробирке транскрипции для получения антисмысловой рибосомальной РНК (arRNA) зонды, которые являются бесплатными для рРНК в общей выборке РНК.- Разработка и синтез праймеров наборы для рРНК фрагмент ПЦР (табл. 1).
- Запуск ПЦР в 50мкл реакции с использованием Taq ДНК-полимеразы (Qiagen) с 50 нг ДНК-матрицы, 1 х ПЦР-буфера, 1,5 Mg 2 Cl, 0,2 мкМ праймеров с 35 циклов денатурации при 94 ° С в течение 10 сек, 15 сек отжигу при температуре 40 ° С в течение бактериальных 23S ампликона и 50 ° C для других ампликонов и расширения при 72 ° С в течение 1 мин (окончательное удлинение при 72 ° С в течение 5 мин).
- Просмотр продуктов ПЦР на 1% агарозном геле.
- Очищают ПЦР продуктов с использованием очистки QIAquick ПЦР комплекте с элюирования в 50 мкл буфера для элюции. Количественно концентрация использованием NanoDrop.
- В пробирке транскрипции биотин-меченных анти-смысл рРНК зондов (arRNA). Примечание: На данном этапе образцы специфических зондов из различных ампликонов рРНК синтезируются помощью MEGAscript T7 комплект.
- Настройка 20 мкл реакции для каждого образца конкретных ампликона рРНК путем смешивания ингредиентов ниже (табл. 2). Выдержите в течение ночи при 37 ° C. Примечание: Обычно реакция дает> 50 мкг РНК.
- Добавить 1 мкл ДНКазы I (входит в комплект) к реакционному и инкубировать при 37 ° С в течение 20 мин для удаления ДНК.
- Добавить 100 мкл 100% этанола к реакции, центрифуги при 12000 х г в течение 10 мин при 4 ° С для осаждения синтезированы arRNA. Удалите супернатант и мыть гранул с 750 мкл 75% этанола. Ресуспендируют РНК-зондов в 50 мкл воды нуклеазы бесплатно.
- Количественная концентрации РНК использованием NanoDrop. Магазин зондов при температуре -80 ° C.
- рРНК вычитание с биотинилированным arRNA. Примечание: Этот шаг использует биотинилированного arRNA для гибридизации с рРНК в общей выборке РНК. РРНК-arRNA гибриды захвачен покрытых стрептавидином магнитных шариков. Процедура основана на протокол, описанный в ссылке 13 с незначительными изменениями.
- Реагенты
- Подготовка 0,1 N NaOH, 20X натрий хлорIDE-цитрата (SSC) и 1 х SSC с 20% формамида.
- Гибридизация
- Смешайте 1 мкг общей РНК с бактериальной 16S и 23S зондов (0,75 мкг каждая), москитные 18S и 28S зондов (0,75 мкг каждая), 1 мкл ингибитора РНКазы, 2,5 мкл 20Х SSC буфера, 10 мкл 100% формамида в 200 мкл ПЦР-пробирку , добавить нуклеазы без воды до 50 мкл.
- Поместите реакционную трубку в термоциклер и инкубировать при 70 ° C в течение 5 минут, чтобы открыть вторичной структуры и нарастить до 25 ° C с использованием 5 ° C шагом в течение 1 мин каждый ° C, чтобы гибридизация рРНК и arRNA зондов.
- Удаление рРНК
- Передача 300 мкл покрытых стрептавидином шарики в 1,7 мл трубки. Положите трубку на магнитной стойке отделение для иммобилизации бисера. Удалить супернатант. Ресуспендируют бисером в 300 мкл 0,1 н NaOH и милих хорошо. Положите трубку обратно на магнитной стойке и пипетировать от супернатанта. Ресуспендируют бисером в 300 мкл 1X SSC, хорошо перемешать, обездвижить бисером и пипетировать от супернатанта. Повторите мыть с 300 мкл 1X SSC еще раз.
- Сделайте 3 аликвоты из бисера, 100 мкл, в 1,7 мл пробирок. Положите трубку на магнитной стойке, и удалите супернатант с помощью пипетки. Примечание: аликвоты бусин используются для 3-х раундов захвата биотинилированного рРНК-arRNA гибридов.
- Добавить 50 мкл 1X SSC с 20% формамида в 50 мкл реакции гибридизации. Передача реакции в первое борта трубы аликвоты, и хорошо перемешать. Выдержите при комнатной температуре в течение 10 мин, чтобы позволить биотинилированного зонда-рРНК гибриды быть захвачены стрептавидином шарики. Флик трубку каждые 2 минуты, чтобы смешивать во время инкубации.
- Положите трубку на магнитной стойке для иммобилизации бисера, передает супернатанта на второй АликUOT из бисера, хорошо перемешать и инкубировать как указано выше. Проведение третьего захвата путем передачи супернатант в третьей трубы из бисера. Смешать и инкубировать.
- Передача не-рРНК супернатант в новую пробирку и проведении очистки с использованием RNeasy MinElute комплект для удаления формамида после эксплуатации завода-изготовителя.
- Проверьте эффективность рРНК истощения с помощью РНК-6000 Agilent Pico чип набор на Bioanalyzer (рис. 4).
- Реагенты
Примечание: рРНК обедненного образцов РНК могут быть мРНК усиления при необходимости 8.
Результаты
Протокол включает в себя три раздела: (1) подготовка метагеномных шаблонов ДНК для ПЦР-рРНК, (2) создание образцов специфических зондов рРНК захвата; (3) истощение рРНК от общей РНК субтрактивный гибридизации. Выделение высокого качества метагеномных ДНК и РНК имеет важное значение для всего процесса. Изменение мета-G-Ном выделения ДНК протокол дает высокое качество метагеномных ДНК комара кишки, как показано на рисунке 2. Это может быть сложным, чтобы изолировать высокого качества общей РНК из комара кишки. Это, вероятно, связано с наличием различных ферментов, в том числе РНКазы, в комара кишки. Мы сравнили общей добычи РНК производительность Qiagen выделения РНК комплект для Roche TriPure. Electropherogram анализа Agilent Bioanalyzer показал, что общей РНК выделяли с помощью TriPure содержит четкие пики рРНК (рис. 3), которая не видела в РНК, полученные с использованием Qiagen комплект. Возможно, комаров производных РНКазы эффективно инактивироватьд к фенола в TriPure. Как правило, 50 комаров выход кишки ~ 20 мкг общей РНК. Идеальное количество от общего числа входных РНК для текущего процесса истощения рРНК составляет ~ 1 мкг, которая оптимизирует эффективность рРНК вычитание и дает ~ 150 нг очищенной РНК. Рисунке 4 показано сравнение электрофореграммы РНК до и после рРНК вычитание. РРНК эффективно удаляются в то время как не-рРНК вида были значительно обогатилась в остальных РНК. Наличие большого размера РНК (> 2000 б.п.) указывает на хорошее качество мРНК обогащенного (рис. 4В). Протокол может быть расширена, чтобы вместить больший вклад РНК для обработки. Обычно 5 мкг РНК, необходимых для РНК-след процесса. Поэтому мы использовали MessageAmp II-РНК бактерий Усиление Kit для создания достаточного количества рРНК обедненного РНК на РНК-Seq. Мы рекомендуем использовать 150 нг или более очищенной РНК для амплификации РНК для обеспечения точности усиления. Этот протокол был заявлСВУ в кишечнике комара РНК-Seq проекта. Истощение рРНК эффективно достигается с помощью образцов, полученных зондами захвата. Таблице 3 приведены доли рРНК говорится в РНК-Seq выходе из четырех образцов. Два сахарных кормили и два крови подачей образцов кишки были обработаны истощения и РНК-Seq. Illumina последовательность генерируется 23-24М читает для каждого образца. РРНК вычитания эффективно удаляется 90-99% рРНК как из ткани комаров и микробов в кишечнике образцов. Истощение была почти завершена в сахарной подачей образцов кишечника, и 6.12-10.98% рРНК остается в крови подачей образцов (табл. 3).
| Amplicon | Нападающий 5'-3 ' | Обратный 5'-3 ' |
| Бактериальные 16S | 27F | 803R_T7 |
| AGAGTTTGATCCTGGCTCAG | TAATACGACTCACTATAGG NCTACCTGGGTATCTAATCC | |
| 347F | 1492R_T7 | |
| GGAGGCAGCAGTRRGGAAT | TAATACGACTCACTATAGG GACGGCTACCTTGTTACGACTT | |
| Бактериальные 23S | 189F | 2490R_T7 |
| GAASTGAAACATCTHAGTA | TAATACGACTCACTATAGG GCGACATCGAGGTGCCAAAC | |
| 1075F | 2241R_T7 | |
| GTTGGCTTRGARGCAGC | TAATACGACTCACTATAG GGACCGCCCCAGTHAAACT | |
| Mosquito 18S | 18SF1 | 18SR1_T7 |
| GGTTGATCCTGCCAGTAGTAT | TAATACGACTCACTATAGG CAAACGCTTTCGCTTCTGT | |
| 18SF2 | 18SR2_T7 | |
| GGGCCGGCGTTGGCCGAGAAT | TAATACGACTCACTATAGG TTCACTTACGGAAACCTTG | |
| Mosquito 28S | 28SF1 | 28SR1_T7 |
| AGG AGG AAC CAC GGA TAC CC | TAATACGACTCACTATAGG ACCACCAAGCATGGGTCGCC | |
| 28SF2 | 28SR2_T7 | |
| AGGAACCACAGGTACGGACC | TAATACGACTCACTATAG GTCCCGGAGGTGCCTCAA |
Таблица 1. Primer наборы для рРНК фрагмент усиления бактериальной 16S и 23 праймеров разработаны на основе 20,19. Последовательностей промотора T7 выделены жирным шрифтом.
10 х буфера| 2 мкл | |
| ПЦР-продукты (0.5-1 мкг) | 1,5 мкл |
| ATP (75 мм) | 2 мкл |
| GTP (75 мм) | 2 мкл |
| CTP (75) | 1,5 мкл |
| UTP (75мм) | 1,5 мкл |
| Биотин-11-CTP (10 мм) | 3,75 мкл |
| Биотин-16-UTP (10мм) | 3,75 мкл |
| T7 РНК-полимеразы | 2 мкл |
Таблица 2. Компоненты в пробирке синтеза зондов рРНК.
| Образец | Всего читает | Mosquito рРНКчитает (%) | Микробная 16S читает (%) | Микробная 23S читает (%) | Всего% рРНК читает * |
| SM1 | 24341850 | 121,987 (0.50) | 3,717 (0.02) | 6,810 (0.03) | 0,54 |
| SM2 | 23487202 | 114,430 (0.49) | 30,271 (0.13) | 38,261 (0.16) | 0,78 |
| BM1 | 23438304 | 265,433 (1.13) | 2,054,777 (8.77) | 253,051 (1.08) | 10,98 |
| BM2 | 24212240 | 110,240 (0.46) | 1,186,423 (4.90) | 185,074 (0.76) | 6,12 |
Таблица 3. Statристик рРНК говорится в РНК-след выходных. SM: сахар кормили кишки; BM: кровь кишки кормили. *: Сумма процентов комаров рРНК и микробных рРНК говорится в сообщении.

Рисунок 1. Блок-схема процедурных шагов метагеномных ДНК и РНК изоляции, рРНК захвата зонда синтез, гибридизация, и захватить бисера на основе истощения.
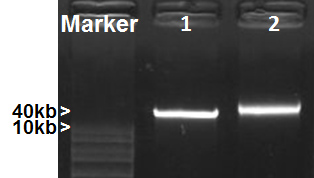
Рисунок 2. Метагеномных выделения ДНК. Образцы ДНК были разделены на 1% агарозном геле. 1, Fosmid ДНК (40kb), 2, Москитная кишечника метагеномных ДНК.

Рисунок 3.Сравнение двух полной изоляции реагентов РНК для их эффективности в добыче качества РНК из комара кишки. Наложение электрофореграммы показывает, что общая РНК из TriPure изоляции (в синем) была рРНК пики и широкого распространения РНК, в то время как от Qiagen изоляции (в красном) не было четкого пика рРНК.

Рисунок 4. Эффективность рРНК истощения. Электрофореграммы показать общую РНК до (А) и после (б) истощение рРНК. РРНК пики эффективно удаляют. РНК больше, чем 4kb остается в образце рРНК исчерпан. Концентрация РНК 107,6 нг / мкл (А) и 8,6 нг / мкл в (B).
Обсуждение
Комплекс микробного сообщества находится в кишечнике комара экосистемы 14,16,17. Metatranscriptomic секвенирования (RNA-след) может выявить зависит от контекста функциональной информации путем опроса всей микробной транскриптом 4,18. Технически, олиго-D (T) опосредованное обогащения прокариотических мРНК не применяется в связи с отсутствием стабильной поли (А) хвост из посланников. Кроме того, рРНК истощения была использована для мРНК обогащения. Здесь мы разработали на основе вычитания протокол к истощению рРНК использованием рРНК зондов из собственного метагеномных ДНК в организме комара микробного сообщества кишечника. Эффективность удаления рРНК зависит от зоны захвата рРНК зондов, которые создаются рРНК ПЦР. Отжига эффективности различных наборов праймеров таргетинга консервативных регионах варьируется в зависимости от различных таксонов 19. Таким образом, мы рекомендуем попробовать различные комбинации праймеров на метагеномных образцы, а затем объединениярРНК продукции для максимального охвата. В нашей процедуре, мы создаем захвата зондов путем объединения рРНК продуктов ПЦР двух передних и двух обратных праймеров, которые могут образовывать 4 комбинации наборов праймеров (табл. 1). Кроме того, рРНК намерен сформировать вторичную структуру. По сравнению с полным рРНК в качестве зондов длиной 13 мелких фрагментов рРНК имеют меньшую склонность к образованию вторичной структуры, которые могут способствовать гибридизации захвата зонда к образцу рРНК. Биотинилированные зонды эффективно гибридизации с рРНК в общей выборке РНК и гибридов удаляют, захватив с покрытых стрептавидином шарики. Процедура удаляет большую часть рРНК (рис. 4). В четырех образцах мы обработали, 90-99% рРНК эффективно обедненного (табл. 3). Эта процедура может быть изменен для различных насекомых связанные исследований микробиомом.
Раскрытие информации
Авторы заявляют, что у них нет конкурирующих финансовых интересов.
Благодарности
Эта работа была поддержана NIH грант 1SC2GM092789-01A1 и MS было исследование ученый NMSU Howard Hughes Medical учреждение Бакалавриат исследовательских программ. Видео был режиссером и продюсером Эми Lanasa и координируется д-р Филип Льюис с Creative Media института NMSU.
Материалы
| Name | Company | Catalog Number | Comments |
| Reagent/Material | |||
| Meta-G-Nome DNA isolation kit | Epicentre | MGN0910 | Metagenomic DNA isolation |
| TriPure | Roche | 11667165001 | Mdetagenomic RNA isolation |
| MEGAscript T7 kit | Ambion | AM1334 | In vitro synthesis of RNA probes |
| RNaseZap | Ambion | AM9780 | RNase free working area |
| Biotin-16-UTP | Roche | 11388908910 | In vitro synthesis of RNA |
| Biotin-11-CTP | Roche | 4739205001 | In vitro synthesis of RNA |
| Streptavidin magnetic beads | NEB | S1420S | Capture of rRNA hybrids |
| Magnetic separation rack | NEB | S1506S | Capture of rRNA hybrids |
| RNeasy mini kit | QIAGEN | 74104 | Purification of subtracted RNA |
| RNase-Free DNase Set | QIAGEN | 79254 | Removal DNA contamination |
| Agilent RNA 6000 Pico Kit | Agilent Technologies Inc. | 5067-1513 | Electropherogram of RNA |
| Equipment | |||
| Bio-Gen PRO200 Homogenizer | PRO Scientific | 01-01200 | Mosquito gut tissue homogenization |
| NanoDrop 1000 Spectrophotometer | Thermo Scientific | DNA & RNA quantitation | |
| 2100 Bioanalyzer | Agilent Technologies Inc. | G2940CA | Electropherogram of RNA |
Ссылки
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nat. Rev. Genet. 10, 57-63 (2009).
- Xie, W., et al. Pyrosequencing the Bemisia tabaci Transcriptome Reveals a Highly Diverse Bacterial Community and a Robust System for Insecticide Resistance. PLoS One. 7, e35181 (2012).
- Xie, L., et al. Profiling the metatranscriptome of the protistan community in Coptotermes formosanus with emphasis on the lignocellulolytic system. Genomics. 99, 246-255 (2012).
- Gosalbes, M. J., et al. Metatranscriptomic approach to analyze the functional human gut microbiota. PLoS One. 6, e17447 (2011).
- Urich, T., et al. Simultaneous assessment of soil microbial community structure and function through analysis of the meta-transcriptome. PLoS One. 3, e2527 (2008).
- Gilbert, J. A., et al. Detection of large numbers of novel sequences in the metatranscriptomes of complex marine microbial communities. PLoS One. 3, e3042 (2008).
- Gifford, S. M., Sharma, S., Rinta-Kanto, J. M., Moran, M. A. Quantitative analysis of a deeply sequenced marine microbial metatranscriptome. ISME J. 5, 461-472 (2011).
- Poretsky, R. S., et al. Comparative day/night metatranscriptomic analysis of microbial communities in the North Pacific subtropical gyre. Environmental microbiology. 11, 1358-1375 (2009).
- Shrestha, P. M., Kube, M., Reinhardt, R., Liesack, W. Transcriptional activity of paddy soil bacterial communities. Environmental Microbiology. 11, 960-970 (2009).
- Mettel, C., Kim, Y., Shrestha, P. M., Liesack, W. Extraction of mRNA from soil. Applied and Environmental Microbiology. 76, 5995-6000 (2010).
- He, S., et al. Validation of two ribosomal RNA removal methods for microbial metatranscriptomics. Nat. Meth. 7, 807-812 (2010).
- Pang, X., et al. Bacterial mRNA purification by magnetic capture-hybridization method. Microbiol. Immunol. 48, 91-96 (2004).
- Stewart, F. J., Ottesen, E. A., DeLong, E. F. Development and quantitative analyses of a universal rRNA-subtraction protocol for microbial metatranscriptomics. ISME J. 4, 896-907 (2010).
- Wang, Y., Gilbreath, T. M., Kukutla, P., Yan, G., Xu, J. Dynamic gut microbiome across life history of the malaria mosquito Anopheles gambiae in Kenya. PLoS One. 6, e24767 (2011).
- Su, C., Sordillo, L. M. A simple method to enrich mRNA from total prokaryotic RNA. Mol. Biotechnol. 10, 83-85 (1998).
- Gusmao, D. S., et al. Culture-dependent and culture-independent characterization of microorganisms associated with Aedes aegypti (Diptera: Culicidae) (L.) and dynamics of bacterial colonization in the midgut. Acta Trop. 115, 275-281 (2010).
- Lindh, J. M., Terenius, O., Faye, I. 16S rRNA gene-based identification of midgut bacteria from field-caught Anopheles gambiae sensu lato and A. funestus mosquitoes reveals new species related to known insect symbionts. Appl. Environ. Microbiol. 71, 7217-7223 (2005).
- Simon, C., Daniel, R. Metagenomic analyses: past and future trends. Appl. Environ. Microbiol. 77, 1153-1161 (2011).
- Wang, Y., Qian, P. Y. Conservative fragments in bacterial 16S rRNA genes and primer design for 16S ribosomal DNA amplicons in metagenomic studies. PLoS One. 4, e7401 (2009).
- Hunt, D. E., et al. Evaluation of 23S rRNA PCR primers for use in phylogenetic studies of bacterial diversity. Appl. Environ. Microbiol. 72, 2221-2225 (2006).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены