Method Article
El agotamiento de ARN ribosomal de metagenómica intestino del mosquito RNA-seq
En este artículo
Resumen
Un ARN ribosómico (ARNr) de protocolo agotamiento fue desarrollado para enriquecer el ARN mensajero (ARNm) de ARN-ss de la metatranscriptome intestino del mosquito. Sondas de muestreo específicos rRNA, que fueron utilizados para extraer el rRNA mediante sustracción, se crea a partir del mosquito y sus microbios intestinales. Rendimiento del protocolo puede resultar en la eliminación de aproximadamente el 90-99% de rRNA.
Resumen
El intestino del mosquito acomoda las comunidades microbianas dinámicas a través de las diferentes etapas del ciclo de vida del insecto. Caracterización de la capacidad genética y la funcionalidad de la comunidad intestino proporcionará información sobre los efectos de la microbiota intestinal en los hábitos de vida de mosquitos. Metagenómica RNA-Seq se ha convertido en una herramienta importante para analizar transcriptomes de diversos microbios presentes en una comunidad microbiana. El ARN mensajero comprende normalmente sólo el 1-3% del total de ARN, mientras que rRNA constituye aproximadamente el 90%. Es un reto para enriquecer ARN mensajero a partir de una muestra de ARN microbiano metagenomic porque la mayoría de las especies de ARNm procariótico carecen de poli (A) las colas. Esto evita oligo d (T) de aislamiento de mRNA mediada. Aquí se describe un protocolo que emplea muestra derivada rRNA sondas de captura para eliminar el rRNA de una muestra metagenomic ARN total. Para empezar, tanto los mosquitos y microbianos fragmentos de subunidades pequeñas y grandes rRNA se amplificó a partir de ADN de una muestra comunitaria metagenómica. Entonces, la comunidadcomunitarias específicas sondas biotiniladas antisentido ARN ribosomal se sintetizan in vitro usando ARN polimerasa de T7. Las sondas biotiniladas rRNA se hibridan con el ARN total. Los híbridos son capturados por las perlas recubiertas con estreptavidina y se retira del ARN total. Este protocolo sustracción basado elimina eficazmente tanto mosquito y rRNA microbiana del total de la muestra de RNA. La muestra de ARNm enriquecido se procesa adicionalmente para la amplificación del ARN y el ARN Seq-.
Introducción
La próxima generación de la tecnología de secuenciación ha avanzado mucho estudio metagenómica, permitiendo evaluar la composición taxonómica y la funcionalidad de un conjunto genético microbiano. La secuenciación de ARN (RNA-Seq) 1 puede pasar por alto métodos de cultivo para investigar metatranscriptomes microbianos en el 2-5 contextos diferentes. Un obstáculo importante para microbiana RNA-seq es la dificultad en el enriquecimiento de ARNm, como las especies de ARNm procariótico no están establemente poliadenilado. Por lo tanto, oligo d (T) mediada por enriquecimiento mensajero no es aplicable. La eliminación del rRNA abundante es un enfoque alternativo para enriquecer ARNm. Los kits comerciales para el agotamiento de rRNA, como Microexpress kit bacteriana Enriquecimiento ARNm (Ambion), RiboMinus Transcriptome Isolation Kit (bacterias) (Life Technologies), y el ARNm de SÓLO kit de aislamiento de mRNA procariótico (Epicentre) que preferentemente degrada rRNA con una exonucleasa, se han utilizado para retirar rRNA 6-8. Sin embargo, las sondas de captura en Microexpresso RiboMinus son buenos para quitar rRNA conocido desde los típicos bacterias Gram-positivas y Gram-negativas-(ver especificaciones de los fabricantes), pero menos compatible con rRNA de microbios desconocidos. Por consiguiente, la supresión puede ser menos eficiente para las muestras 8-10 metagenómicas. Además, la fidelidad mRNA abundancia era cuestionable cuando el tratamiento se aplicó exonucleasa 11. En general, resta basado agotamiento rRNA fue menos sesgada y más eficaz en el enriquecimiento de mRNA en entornos metagenómicas 10-13.
El intestino del mosquito se adapta a una comunidad microbiana dinámica 14. Estamos interesados en la caracterización de la función del microbioma intestinal mosquito mediante el uso de RNA-Seq. En una muestra de ARN aislado de las entrañas de mosquitos, tanto de ARN de mosquitos y microbianas presentes. Aquí se describe un protocolo modificado para usar sondas específicas de la comunidad rRNA para agotar de manera eficiente rRNA mosquito y microbiana por hibridación sustractiva. El ARNm resultantemuestras enriquecidas son apropiados para RNA-Seq. El flujo de trabajo general se representa en la figura 1.
Protocolo
Procedimiento
1. Mosquito Crianza
- Rear mosquito Anopheles gambiae cepa G3 en un insectario en 27,5 ° C con 80% de humedad y ciclo de 12:12 horas de luz / oscuridad.
- Las larvas se alimentan con la comida del suelo gato con levadura de cerveza en una proporción 1:1.
- Alimentar a los mosquitos adultos en sangre de ratones en el día 3 post-emergencia para la producción de huevos.
2. Disección intestino del mosquito
- Autoclave herramientas de disección y pinzas (diapositivas).
- Reúne 50 mosquitos utilizando un aspirador y colocarlos en la cama de CO 2 de flujo (Ultimate Flypad). Enjuague un espécimen mosquito secuencialmente en tres placas de Petri que contienen 70% de etanol para limpiar la superficie del cuerpo del mosquito.
- Colocar la muestra sobre un portaobjetos de vidrio bajo una estereomicroscopía. Retire el intestino.
- Reúne 50 agallas por condición de aislamiento de ADN metagenómica. Reúne 50 agallas por condición metagenómica para el aislamiento de ARN.
3. Aislamiento de ADN metagenómica
Nota: ADN metagenómico se aisló utilizando el Meta-G-Nome DNA Isolation Kit (Biotecnologías Epicentre) con la modificación descrita a continuación.
- Coloque 50 tripas de mosquitos en 300 l de TE. Homogeneizar ellos por 1 min utilizando Bio-Gen PRO200 homogeneizador (PRO Scientific Inc, EE.UU.) a la velocidad de 2.000 rpm en hielo.
- Añadir 2 l de solución de lisozima Ready-Lyse y 1 l de RNasa A a la suspensión celular. Mezclar por agitación e incubar a 37 ° C durante 30 min.
- Añadir 300 l de solución de lisis Meta-(2X) y 1 l de proteinasa K al tubo. Mezcle por vortex. Brevemente pulso a centrifugar el tubo para asegurar que toda la solución se encuentra en la parte inferior del tubo, y se incuba a 65 ° C durante 15 min, se enfría a temperatura ambiente, luego colocar en hielo durante 3-5 min.
- Añadir 350 l de reactivo de precipitación de proteínas MPC al tubo y se mezcla mediante agitación en vórtex vigorosamente durante 10 segundos.
- Pellet tque los desechos por centrifugación durante 10 min a 12.000 xg a 4 ° C.
- Transferir el sobrenadante a un limpio de 2 ml tubo, y desechar el sedimento.
- Añadir 570 l de isopropanol al sobrenadante. Mezclar mediante veces invirtiendo el tubo varias.
- Pellet el ADN por centrifugación durante 10 min a 12.000 xg a 4 ° C. Eliminar el isopropanol sin desalojar el sedimento de ADN.
- Añadir 500 l de etanol al 75% para lavar el sedimento. Centrifugar durante 5 min a 12.000 xg a 4 ° C. Eliminar el etanol sin perturbar el sedimento de ADN. Aire secar el sedimento a temperatura ambiente durante 2 min. Nota: No se exceda en seco el precipitado de ADN.
- Resuspender el sedimento de ADN en 50 l de tampón TE.
- Validar el tamaño del ADN aislado en comparación con el ADN de control fosmid (40 kb; 100 ng / l) proporcionado en el kit, a través de electroforesis en gel en un gel de agarosa 1%.
4. Aislamiento de ARN total
Nota: Limpie el área de trabajocon RNaseZap para minimizar la contaminación de ARNasa. Metagenomic ARN se aisló de muestras de intestino del mosquito utilizando el TriPure Isolation Reagent (Roche) con una modificación menor.
- Ponga 50 tripas de mosquitos en un tubo de 2 ml con 500 l de reactivo de aislamiento TriPure. Homogeneizar durante 1 min a velocidad de 2.000 rpm en hielo usando un homogeneizador. Deje reposar a temperatura ambiente durante 5 min para permitir la disociación de los complejos de nucleoproteínas.
- Añadir 100 l de cloroformo y agitar durante 12 segundos, y dejar reposar a temperatura ambiente durante 2 min.
- Centrifugar a 10.000 xg durante 10 min. Con cuidado, saque el tubo sin molestar fases separadas.
- Transferir la fase acuosa a un nuevo tubo, añadir 250 l de isopropanol, se mezcla bien, y poner a -20 º C durante 20 min.
- Centrifugar a 12.000 xg durante 10 min a 4 ° C para precipitar el ARN.
- Descartar el sobrenadante sin desalojar pellet. Lavar el pellet con 750 l de etanol al 75%.
- Pipetear el sobrenadante sin perturbar el sedimento. LaIR secar el tubo durante 2 min.
- Resuspender el RNA con 30 l de agua libre de nucleasas.
- Tratar el ARN total con DNasa a 37 ° C durante 20 min y, a continuación inactivar la DNasa mediante la incubación a 75 ° C durante 15 min. ARN precipitado con etanol y se resuspende ARN en 30 l de agua libre de nucleasas.
- Determinar la cantidad de RNA total utilizando un NanoDrop.
5. Ribosomal RNA agotamiento
Nota: El protocolo fue desarrollado en base a los métodos descritos anteriormente 12,13,15.
- La amplificación por PCR de fragmentos de genes ribosomales de RNA
Este paso crea muestra específica de piscinas rRNA, que se utilizan como plantillas para la transcripción in vitro para producir anticuerpos anti-sentido ARN ribosómico (arRNA) sondas que son complementarias a rRNA en el total de la muestra de ARN.- Diseñar y sintetizar conjuntos de cebadores para el ARNr fragmento de PCR (Tabla 1).
- Realizar la PCR en 50l de reacción utilizando Taq ADN polimerasa (Qiagen) con 50 ng de DNA plantilla, 1 x tampón de PCR, 1,5 mM de Mg 2 Cl, 0,2 m con cebadores de 35 ciclos de desnaturalización a 94 ° C durante 10 seg, 15 seg de recocido a 40 ° C para amplicón bacteriana 23S, y 50 ° C para los amplicones de otros, y extensión a 72 ° C durante 1 min (extensión final a 72 ° C durante 5 min).
- Ver productos de PCR en gel de agarosa al 1%.
- Purificar los productos de PCR utilizando el kit de purificación de PCR QIAquick con elución en 50 l de tampón de elución. Cuantificar la concentración usando NanoDrop.
- La transcripción in vitro de sondas marcadas con biotina rRNA anti-sentido (arRNA). Nota: En este paso, las sondas específicas de la muestra de diferentes amplicones de ARNr se sintetizan usando el kit MEGAscript T7.
- Configurar una reacción de 20 l para cada amplicón muestra rRNA específico mezclando los ingredientes indicados a continuación (Tabla 2). Incubar durante una noche a 37 ° C. Nota: por lo general los rendimientos de la reacción> 50 microgramos de ARN.
- Añadir 1 l de DNasa I (incluido en el kit) a la reacción y se incuba a 37 ° C durante 20 min para eliminar la plantilla de ADN.
- Añadir 100 l de etanol 100% a la reacción, se centrifuga a 12.000 xg durante 10 min a 4 ° C para precipitar sintetizado arRNA. Desechar el sobrenadante y lavar el precipitado con 750 l de etanol al 75%. Resuspender las sondas de ARN de cada 50 l de agua libre de nucleasas.
- Cuantificar la concentración de ARN utilizando un NanoDrop. Las sondas se almacena a -80 ° C.
- rRNA resta con biotina arRNA. Nota: Este paso emplea la arRNA biotinilado para hibridarse a ARNr en el total de la muestra de ARN. Los híbridos rRNA-arRNA son capturados por estreptavidina con perlas magnéticas recubiertas. El procedimiento se basa en el protocolo descrito en la referencia 13 con modificaciones menores.
- Reactivos
- Preparar 0,1 N NaOH, 20X cloruros de sodioide-citrato (SSC) y 1 X SSC con 20% de formamida.
- Hibridación
- Mezclar 1 RNA total de microgramos con 16S y 23S sondas (0,75 g cada una), 18S mosquitos y sondas 28S (0,75 g cada uno), 1 l inhibidor de RNasa, 2,5 l de 20X tampón SSC, formamida 10 l 100% en un tubo de 200 l PCR , añadir agua libre de nucleasa a 50 l.
- Colocar el tubo de reacción en un termociclador e incubar a 70 ° C durante 5 minutos para abrir la estructura secundaria y la deceleración a 25 ° C usando incrementos de 5 º C por 1 min cada ° C para permitir la hibridación de sondas de rRNA y arRNA.
- La eliminación del rRNA
- Transferir 300 mu l perlas recubiertas con estreptavidina en un tubo de 1,7 ml. Colocar el tubo en un estante de separación magnética para inmovilizar las perlas. Eliminar el sobrenadante. Volver a suspender las bolas en 300 l de NaOH 0,1 N y mix también. Coloque el tubo de nuevo a la rejilla magnética y pipetear el sobrenadante. Volver a suspender las bolas en 300 l de 1X SSC, mezclar bien, inmovilizar el talón y pipetear el sobrenadante. Repita el lavado con 300 l de 1X SSC una vez más.
- Haga 3 alícuotas de perlas, 100 mu l cada uno, en tubos de 1,7 ml. Se coloca el tubo en la gradilla magnética, y eliminar el sobrenadante por pipeteo. Nota: Las alícuotas de perlas se utiliza durante 3 rondas de captura biotinilada rRNA arRNA híbridos.
- Añadir 50 l SSC 1X con 20% de formamida en la reacción de hibridación 50 l. Transferir la reacción en el tubo alícuota talón primero, y mezclar bien. Incubar a TA durante 10 min para permitir que los biotinilados rRNA sonda-híbridos para ser capturado por perlas de estreptavidina. Ponga el tubo cada 2 minutos para mezclar durante la incubación.
- Se coloca el tubo en la gradilla magnética para inmovilizar las perlas, transferir el sobrenadante a la segunda Aliquot de perlas, se mezcla bien y se incuba como anteriormente. Llevar a cabo la captura tercero mediante la transferencia de sobrenadante en el tubo tercio de perlas. Mezclar e incubar.
- Transferencia no rRNA sobrenadante a un tubo nuevo y purificación conducta usando el kit RNeasy MinElute para eliminar formamida siguiendo el manual del fabricante.
- Comprobar la eficacia agotamiento rRNA utilizando un Agilent 6000 RNA kit Pico chip en un Bioanalyzer (Figura 4).
- Reactivos
Nota: Los rRNA depleción de las muestras de ARN están sujetos a ARNm de amplificación si es necesario 8.
Resultados
El protocolo incluye tres secciones: (1) preparación de los moldes de ADN metagenómica para rRNA PCR, (2) la creación de muestreo específicos rRNA sondas de captura, (3) el agotamiento de rRNA de ARN total mediante hibridación sustractiva. Aislamiento de alta calidad metagenomic ADN y ARN es esencial para el proceso. El modificada Meta-G-Nome protocolo de aislamiento de ADN se obtiene de alta calidad de ADN metagenómico de tripas de mosquitos, como se muestra en la Figura 2. Puede ser difícil de aislar de alta calidad ARN total de las entrañas de mosquitos. Esto es probablemente debido a la presencia de diversas enzimas, incluyendo la RNasa, en los intestinos de los mosquitos. Se comparó el rendimiento total de la extracción de RNA del kit Qiagen aislamiento de ARN a Roche TriPure. Electroferograma de Agilent Bioanalyzer análisis mostró que el ARN total aislado utilizando TriPure contiene claros picos de rRNA (Figura 3), que no se ve en el ARN obtenido usando el kit de Qiagen. Quizás mosquito derivado de RNasa se inactivan eficazmented por el fenol en TriPure. Típicamente, rendimiento 50 tripas mosquito ~ 20 microgramos de ARN total. La cantidad ideal del total de ARN de entrada para el proceso de reducción actual rRNA es de ~ 1 g, lo que optimiza la eficiencia de rRNA resta y los rendimientos de ~ 150 ng RNA purificado. Figura 4 muestra la comparación de electroferogramas ARN antes y después de rRNA resta. El rRNA se eliminó de manera eficiente, mientras que las especies no rRNA fueron muy enriquecida en el ARN restantes. La presencia de RNA de gran tamaño (> 2.000 pb) indica una buena calidad de mRNA enriquecido (Figura 4B). El protocolo puede ser ampliado para dar cabida a un mayor aporte de ARN para su procesamiento. Por lo general, 5 g de ARN se requiere para el proceso de RNA-seq. Por lo tanto, hemos utilizado MessageAmp II-Bacteria kit de amplificación de ARN para generar una cantidad adecuada de rRNA empobrecido ARN para RNA-Seq. Se recomienda utilizar 150 ng o más ARN purificado para la amplificación del ARN para asegurar la fidelidad de la amplificación. Este protocolo ha sido aplIED a un intestino del mosquito RNA-Seq proyecto. El agotamiento rRNA se logró eficazmente mediante el uso de sondas de captura derivados de la muestra. Tabla 3 muestra el porcentaje de rRNA se lee en la salida de RNA-Seq de cuatro muestras. Dos azúcar alimentados y alimentados con dos muestras de intestino de sangre fueron procesadas por el agotamiento y la RNA Seq-. La secuenciación de Illumina generado 23-24M lee para cada muestra. La resta eficacia rRNA rRNA eliminado 90-99% del tejido mosquito ambos y los microbios en las muestras intestinales. El agotamiento estaba casi completa en las muestras de intestino alimentados con azúcar, y 6.12-10.98% rRNA permanencia en la sangre alimentados muestras (Tabla 3).
| Amplicon | Adelante 5'-3 ' | Revertir 5'-3 ' |
| 16S bacteriano | 27F | 803R_T7 |
| AGAGTTTGATCCTGGCTCAG | TAATACGACTCACTATAGG NCTACCTGGGTATCTAATCC | |
| 347F | 1492R_T7 | |
| GGAGGCAGCAGTRRGGAAT | TAATACGACTCACTATAGG GACGGCTACCTTGTTACGACTT | |
| 23S bacteriana | 189F | 2490R_T7 |
| GAASTGAAACATCTHAGTA | TAATACGACTCACTATAGG GCGACATCGAGGTGCCAAAC | |
| 1075F | 2241R_T7 | |
| GTTGGCTTRGARGCAGC | TAATACGACTCACTATAG GGACCGCCCCAGTHAAACT | |
| 18S Mosquito | 18SF1 | 18SR1_T7 |
| GGTTGATCCTGCCAGTAGTAT | TAATACGACTCACTATAGG CAAACGCTTTCGCTTCTGT | |
| 18SF2 | 18SR2_T7 | |
| GGGCCGGCGTTGGCCGAGAAT | TAATACGACTCACTATAGG TTCACTTACGGAAACCTTG | |
| Mosquito 28S | 28SF1 | 28SR1_T7 |
| AGG AGG AAC CAC TAC GGA CC | TAATACGACTCACTATAGG ACCACCAAGCATGGGTCGCC | |
| 28SF2 | 28SR2_T7 | |
| AGGAACCACAGGTACGGACC | TAATACGACTCACTATAG GTCCCGGAGGTGCCTCAA |
Conjuntos Tabla 1. Cebadores para amplificación del fragmento 16S rRNA bacterianas y cebadores 23 están diseñados sobre la base de 20,19. Secuencias del promotor T7 están en negrita.
| 10 x tampón | 2 l |
| PCR productos (0.5-1 mg) | L 1,5 |
| ATP (75 mM) | 2 l |
| GTP (75 mM) | 2 l |
| CTP (75 mM) | L 1,5 |
| UTP (75 mM) | L 1,5 |
| Biotina-11-CTP (10 mM) | 3,75 l |
| Biotina-16-UTP (10 mM) | 3,75 l |
| T7 ARN polimerasa | 2 l |
Tabla 2. Componentes de la síntesis in vitro de sondas de rRNA.
| Muestra | Total de lecturas | Mosquito rRNAlee (%) | 16S microbianos lee (%) | 23S microbiana lee (%) | % Total de lecturas rRNA * |
| SM1 | 24341850 | 121,987 (0.50) | 3,717 (0.02) | 6,810 (0.03) | 0,54 |
| SM2 | 23487202 | 114,430 (0.49) | 30,271 (0.13) | 38,261 (0.16) | 0,78 |
| BM1 | 23438304 | 265,433 (1.13) | 2,054,777 (8.77) | 253,051 (1.08) | 10,98 |
| BM2 | 24212240 | 110,240 (0.46) | 1,186,423 (4.90) | 185,074 (0.76) | 6,12 |
Tabla 3. Estadísticaterísticas de rRNA lee en RNA-seq salida. SM: azúcar intestino gruesos: BM: gut sangre materna. *: La suma de los porcentajes de mosquito rRNA rRNA y microbiana lee.

Figura 1. Diagrama de flujo que muestra las etapas de procedimiento de ADN metagenómico y el aislamiento de ARN, síntesis de la sonda de captura de rRNA, la hibridación y la captura de perlas de agotamiento base.
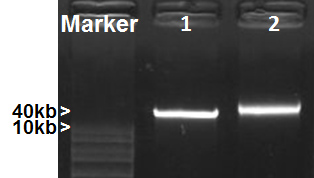
Figura 2. Aislamiento de ADN metagenómico. Muestras de ADN fueron separados en gel de agarosa al 1%. 1, fosmid ADN (40kb) 2, el intestino del mosquito ADN metagenómica.

Figura 3.Comparación de los dos reactivos de aislamiento de ARN total por su eficacia en la extracción de un ARN de calidad de intestino del mosquito. Superposición de electroferogramas que muestra total de ARN de aislamiento TriPure (en azul) tenía picos rRNA y amplia distribución de ARN, mientras que el aislamiento de Qiagen (en rojo) no tenía picos rRNA claras.

Figura 4. Eficiencia de rRNA agotamiento. Electroferogramas mostrar el ARN total antes (A) y después (B) el agotamiento de rRNA. Los picos de rRNA fueron retirados de manera eficiente. ARN superior a 4 kb restos en el rRNA muestra empobrecido. La concentración de ARN fue 107,6 ng / l en (A) y 8,6 ng / l en (B).
Discusión
Una comunidad microbiana compleja reside en el ecosistema intestinal mosquito 14,16,17. Metatranscriptomic secuenciación (RNA-seq) puede revelar información de contexto funcional dependiente interrogando a todo el transcriptoma 4,18 microbiana. Técnicamente, oligo-d (T) el enriquecimiento de ARNm mediada procariota no es aplicable debido a la ausencia de de poli (A) las colas de los mensajeros. Alternativamente, el agotamiento de rRNA se ha utilizado para el enriquecimiento del ARNm. Aquí, hemos desarrollado un protocolo de sustracción basado para agotar rRNA utilizando sondas de rRNA hechas a partir del ADN metagenómico muy propia en la comunidad microbiana intestinal mosquito. La eficacia de la eliminación rRNA depende de la cobertura de las sondas de captura de ARNr que son generados por la PCR rRNA. La eficiencia de la hibridación de cebadores diferentes conjuntos de objetivos regiones conservadas varía en los diferentes taxones 19. Por lo tanto, recomendamos probar diferentes combinaciones de cebadores en muestras metagenómica, a continuación, poner en común losproductos rRNA para maximizar la cobertura. En nuestro procedimiento, se crea sondas de captura mediante la combinación de los productos de PCR rRNA de dos adelante y dos cebadores inversos, que pueden formar 4 combinaciones de conjuntos de cebadores (Tabla 1). Además, se propone rRNA para formar una estructura secundaria. En comparación con rRNA de longitud completa como sondas 13, rRNA fragmentos más pequeños tienen una menor tendencia a formar la estructura secundaria, lo que puede facilitar la hibridación de sondas de captura para el rRNA muestra. Las sondas biotiniladas eficientemente hibridar con ARNr en la muestra total de ARN y los híbridos se eliminan mediante la captura con perlas recubiertas con estreptavidina. El procedimiento elimina la mayor parte del rRNA (Figura 4). En las cuatro muestras que han procesado, 90-99% de ARNr se agotó eficazmente (Tabla 3). Este procedimiento puede ser modificado adicionalmente para una variedad de estudios microbioma insectos asociados.
Divulgaciones
Los autores declaran que no tienen intereses en conflicto financieros.
Agradecimientos
Este trabajo fue apoyado por el NIH subvención 1SC2GM092789-01A1 y MS fue un becario de investigación de NMSU Howard Hughes Medical Research Programs institución universitaria. El video fue dirigido y producido por Amy lanasa y coordinado por el Dr. Philip Lewis con el Creative Media Institute en NMSU.
Materiales
| Name | Company | Catalog Number | Comments |
| Reagent/Material | |||
| Meta-G-Nome DNA isolation kit | Epicentre | MGN0910 | Metagenomic DNA isolation |
| TriPure | Roche | 11667165001 | Mdetagenomic RNA isolation |
| MEGAscript T7 kit | Ambion | AM1334 | In vitro synthesis of RNA probes |
| RNaseZap | Ambion | AM9780 | RNase free working area |
| Biotin-16-UTP | Roche | 11388908910 | In vitro synthesis of RNA |
| Biotin-11-CTP | Roche | 4739205001 | In vitro synthesis of RNA |
| Streptavidin magnetic beads | NEB | S1420S | Capture of rRNA hybrids |
| Magnetic separation rack | NEB | S1506S | Capture of rRNA hybrids |
| RNeasy mini kit | QIAGEN | 74104 | Purification of subtracted RNA |
| RNase-Free DNase Set | QIAGEN | 79254 | Removal DNA contamination |
| Agilent RNA 6000 Pico Kit | Agilent Technologies Inc. | 5067-1513 | Electropherogram of RNA |
| Equipment | |||
| Bio-Gen PRO200 Homogenizer | PRO Scientific | 01-01200 | Mosquito gut tissue homogenization |
| NanoDrop 1000 Spectrophotometer | Thermo Scientific | DNA & RNA quantitation | |
| 2100 Bioanalyzer | Agilent Technologies Inc. | G2940CA | Electropherogram of RNA |
Referencias
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nat. Rev. Genet. 10, 57-63 (2009).
- Xie, W., et al. Pyrosequencing the Bemisia tabaci Transcriptome Reveals a Highly Diverse Bacterial Community and a Robust System for Insecticide Resistance. PLoS One. 7, e35181 (2012).
- Xie, L., et al. Profiling the metatranscriptome of the protistan community in Coptotermes formosanus with emphasis on the lignocellulolytic system. Genomics. 99, 246-255 (2012).
- Gosalbes, M. J., et al. Metatranscriptomic approach to analyze the functional human gut microbiota. PLoS One. 6, e17447 (2011).
- Urich, T., et al. Simultaneous assessment of soil microbial community structure and function through analysis of the meta-transcriptome. PLoS One. 3, e2527 (2008).
- Gilbert, J. A., et al. Detection of large numbers of novel sequences in the metatranscriptomes of complex marine microbial communities. PLoS One. 3, e3042 (2008).
- Gifford, S. M., Sharma, S., Rinta-Kanto, J. M., Moran, M. A. Quantitative analysis of a deeply sequenced marine microbial metatranscriptome. ISME J. 5, 461-472 (2011).
- Poretsky, R. S., et al. Comparative day/night metatranscriptomic analysis of microbial communities in the North Pacific subtropical gyre. Environmental microbiology. 11, 1358-1375 (2009).
- Shrestha, P. M., Kube, M., Reinhardt, R., Liesack, W. Transcriptional activity of paddy soil bacterial communities. Environmental Microbiology. 11, 960-970 (2009).
- Mettel, C., Kim, Y., Shrestha, P. M., Liesack, W. Extraction of mRNA from soil. Applied and Environmental Microbiology. 76, 5995-6000 (2010).
- He, S., et al. Validation of two ribosomal RNA removal methods for microbial metatranscriptomics. Nat. Meth. 7, 807-812 (2010).
- Pang, X., et al. Bacterial mRNA purification by magnetic capture-hybridization method. Microbiol. Immunol. 48, 91-96 (2004).
- Stewart, F. J., Ottesen, E. A., DeLong, E. F. Development and quantitative analyses of a universal rRNA-subtraction protocol for microbial metatranscriptomics. ISME J. 4, 896-907 (2010).
- Wang, Y., Gilbreath, T. M., Kukutla, P., Yan, G., Xu, J. Dynamic gut microbiome across life history of the malaria mosquito Anopheles gambiae in Kenya. PLoS One. 6, e24767 (2011).
- Su, C., Sordillo, L. M. A simple method to enrich mRNA from total prokaryotic RNA. Mol. Biotechnol. 10, 83-85 (1998).
- Gusmao, D. S., et al. Culture-dependent and culture-independent characterization of microorganisms associated with Aedes aegypti (Diptera: Culicidae) (L.) and dynamics of bacterial colonization in the midgut. Acta Trop. 115, 275-281 (2010).
- Lindh, J. M., Terenius, O., Faye, I. 16S rRNA gene-based identification of midgut bacteria from field-caught Anopheles gambiae sensu lato and A. funestus mosquitoes reveals new species related to known insect symbionts. Appl. Environ. Microbiol. 71, 7217-7223 (2005).
- Simon, C., Daniel, R. Metagenomic analyses: past and future trends. Appl. Environ. Microbiol. 77, 1153-1161 (2011).
- Wang, Y., Qian, P. Y. Conservative fragments in bacterial 16S rRNA genes and primer design for 16S ribosomal DNA amplicons in metagenomic studies. PLoS One. 4, e7401 (2009).
- Hunt, D. E., et al. Evaluation of 23S rRNA PCR primers for use in phylogenetic studies of bacterial diversity. Appl. Environ. Microbiol. 72, 2221-2225 (2006).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados