Method Article
L'esaurimento delle RNA ribosomiale per metagenomiche Gut Mosquito RNA-seq
In questo articolo
Riepilogo
Un RNA ribosomiale (rRNA), il protocollo è stato sviluppato per esaurimento arricchire RNA messaggero (mRNA) per RNA-seq del metatranscriptome intestino zanzara. Sonde rRNA di esempio specifici, che sono stati utilizzati per rimuovere rRNA mediante sottrazione, sono stati creati dalla zanzara e dei suoi microbi intestinali. Prestazioni del protocollo può comportare la rimozione di circa il 90-99% di rRNA.
Abstract
L'intestino zanzara ospita comunità microbiche dinamiche in diverse fasi del ciclo di vita dell'insetto. Caratterizzazione della capacità genetica e la funzionalità della comunità intestino si forniscono informazioni sugli effetti del microbiota intestinale su tratti della vita di zanzara. Metagenomiche RNA-Seq è diventato uno strumento importante per analizzare trascrittomi da vari microbi presenti in una comunità microbica. RNA messaggero costituito normalmente solo 1-3% di RNA totale, mentre rRNA costituisce circa il 90%. E 'difficile per arricchire RNA messaggero da un campione di RNA metagenomica microbica, perché la maggior parte delle specie di mRNA procariote mancano stabili poli (A) code. Questo impedisce oligo d (T) isolamento dell'mRNA mediata. Qui, descriviamo un protocollo che utilizza campione derivato rRNA catturare sonde per rimuovere rRNA da un metagenomica campione di RNA totale. Per iniziare, sia zanzara e microbiche piccoli e grandi frammenti di rRNA subunità sono amplificati da un campione di DNA metagenomica comunità. Poi, la comunitàcomunitari specifici biotinilati sonde antisenso RNA ribosomiale sono sintetizzati in vitro usando T7 RNA polimerasi. Le sonde biotinilate rRNA sono ibridate l'RNA totale. Gli ibridi sono catturati da streptavidina perle rivestite e rimossi dal RNA totale. Questa sottrazione protocollo basato rimuove efficacemente sia zanzara e rRNA microbica dal campione di RNA totale. Il campione di mRNA arricchito ulteriormente trattato per l'amplificazione di RNA e RNA-Seq.
Introduzione
Di prossima generazione di tecnologia di sequenziamento ha notevolmente avanzato studio metagenomica, consentendo di valutare la composizione tassonomica e la funzionalità genetico di un assemblaggio microbica. RNA-Sequencing (RNA-Seq) 1 può ignorare la cultura a base di metodi per indagare metatranscriptomes microbiche in diversi contesti 2-5. Un notevole ostacolo alla microbica RNA-seq è la difficoltà di arricchire mRNA, come le specie di mRNA procariotici non sono stabilmente poliadenilato. Pertanto, oligo d (T) l'arricchimento mediato messenger non è applicabile. La rimozione di rRNA abbondante è un approccio alternativo per arricchire mRNA. Kit commerciali deplezione rRNA, come Microexpress kit batterica Enrichment mRNA (Ambion), RiboMinus Transcriptome Isolation Kit (batteri) (Life Technologies), e mRNA-ONLY Prokaryotic kit di isolamento di mRNA (Epicentre) che degrada preferenzialmente rRNA con una esonucleasi, sono stati utilizzati per la rimozione di rRNA 6-8. Tuttavia, le sonde di cattura in Microexpresso RiboMinus sono buoni per la rimozione di rRNA noto dalle tipiche dei batteri Gram-positivi e Gram-negativi (vedere le specifiche dei costruttori), ma meno compatibile con rRNA da microbi sconosciuti. Conseguentemente, la rimozione può essere meno efficiente per metagenomiche 8-10 campioni. Inoltre, la fedeltà abbondanza mRNA era discutibile quando il trattamento è stato applicato esonucleasi 11. Nel complesso, la sottrazione a base di esaurimento rRNA era meno di parte e più efficace di arricchimento mRNA nelle impostazioni metagenomiche 10-13.
L'intestino zanzara ospita una comunità dinamica microbica 14. Siamo interessati a caratterizzare la funzione del microbioma intestinale zanzara utilizzando RNA-Seq. In un campione di RNA isolato da il coraggio di zanzare, sia RNA zanzara e microbiche sono presenti. Qui, descriviamo un protocollo modificato per utilizzare sonde rRNA specifici della comunità di esaurire in modo efficiente rRNA zanzara e microbica mediante ibridazione sottrattiva. L'mRNA risultantecampioni arricchiti sono adatti per RNA-Seq. Il flusso di lavoro complessivo è rappresentato in figura 1.
Protocollo
Procedura
1. Mosquito allevamento
- Posteriore zanzara Anopheles gambiae ceppo G3 in un insettario a 27,5 ° C con 80% di umidità e ciclo ore 12:12 del chiaro / scuro.
- Larve si nutrono con cibo per gatti terra con lievito di birra con rapporto 1:1.
- Pasci le zanzare adulte su topi di sangue al giorno 3 post-emergenza per la produzione di uova.
2. Gut Dissection Mosquito
- Strumenti di dissezione autoclave (diapositive e pinze).
- Colleziona 50 zanzare utilizzando un aspiratore e metterli sul letto di flusso di CO 2 (Ultimate Flypad). Risciacquare zanzara sequenzialmente un campione in tre piastre di Petri contenenti 70% di etanolo per pulire la superficie zanzara corpo.
- Posizionare il campione su un vetrino sotto un stereomicroscopia. Rimuovere l'intestino.
- Colleziona 50 budella per condizione di isolamento del DNA metagenomica. Colleziona 50 budella per condizione per metagenomica isolamento dell'RNA.
3. DNA Isolation metagenomiche
Nota: DNA metagenomiche è isolato con il Meta-G-Nome DNA Isolation Kit (Biotecnologie Epicentre) con la modifica descritta di seguito.
- Mettere 50 budella di zanzara in 300 microlitri TE. Omogeneizzare loro per 1 min con Bio-Gen PRO200 omogeneizzatore (PRO Scientific Inc, USA) a 2.000 giri velocità su ghiaccio.
- Aggiungere 2 microlitri di Ready-Lyse soluzione di lisozima e 1 ml di RNasi A alla sospensione cellulare. Mescolare nel vortex e incubare a 37 ° C per 30 min.
- Aggiungere 300 pl di Meta-Lysis Solution (2X) e 1 ml di proteinasi K al tubo. Miscelare su vortex. Brevemente impulsi centrifugare la provetta per assicurare che tutta la soluzione è nel fondo del tubo, e incubare a 65 ° C per 15 minuti, raffreddare a temperatura ambiente, poi posto in ghiaccio per 3-5 min.
- Aggiungere 350 microlitri di Reagente Precipitation proteine MPC per il tubo e mescolare nel vortex energicamente per 10 sec.
- Pellet tegli detriti mediante centrifugazione per 10 minuti a 12.000 xg a 4 ° C.
- Trasferire il surnatante in un ambiente pulito da 2 ml tubo, e scartare il pellet.
- Aggiungere 570 ml di isopropanolo al supernatante. Miscelare per inversione della provetta diverse.
- Pellet il DNA mediante centrifugazione per 10 minuti a 12.000 xg a 4 ° C. Rimuovere il isopropanolo senza staccare il pellet di DNA.
- Aggiungere 500 ml di etanolo al 75% per lavare il pellet. Centrifugare per 5 minuti a 12.000 xg a 4 ° C. Rimuovere l'etanolo senza disturbare il pellet di DNA. Asciugare il pellet a temperatura ambiente per 2 min. Nota: Non troppo secco il pellet di DNA.
- Risospendere il pellet di DNA in 50 microlitri di tampone TE.
- Convalidare la dimensione del DNA isolato rispetto al DNA di controllo Fosmid (40 kb; 100 ng / pl) fornito nel kit, tramite elettroforesi su gel di agarosio 1% gel.
4. L'RNA totale isolamento
Nota: Pulire l'area di lavorocon RNaseZap per minimizzare la contaminazione RNasi. Metagenomiche RNA è stato isolato da campioni di budello di zanzara con il reagente di isolamento TriPure (Roche) con una piccola modifica.
- Mettere 50 budella di zanzara in un tubo da 2 ml con 500 Reagente isolamento TriPure. Omogeneizzare per 1 min a velocità di 2000 rpm su ghiaccio utilizzando un omogeneizzatore. Lasciare riposare a temperatura ambiente per 5 minuti per consentire la dissociazione dei complessi nucleoproteici.
- Aggiungere 100 microlitri cloroformio e vortex per 12 sec, e lasciate riposare a temperatura ambiente per 2 min.
- Centrifugare a 10.000 xg per 10 min. Estrarre con attenzione il tubo senza disturbare fasi separate.
- Trasferire fase acquosa in una nuova provetta, aggiungere 250 microlitri isopropanolo, mescolare bene, e mettere a -20 ° C per 20 min.
- Centrifugare a 12.000 xg per 10 min a 4 ° C a RNA precipitato.
- Scartare il sopranatante senza staccare pellet. Lavare il pellet con 750 microlitri di etanolo al 75%.
- Pipettare il sopranatante senza disturbare il pellet. Lair asciugare la provetta per 2 min.
- Risospendere l'RNA con 30 microlitri di acqua priva di nucleasi.
- Trattare gli RNA totali con DNasi a 37 ° C per 20 min, quindi inattivare la DNasi incubando a 75 ° C per 15 min. RNA precipitato con etanolo e risospendere l'RNA in 30 microlitri di acqua priva di nucleasi.
- Determinare la quantità di RNA totale usando un NanoDrop.
5. RNA ribosomiale esaurimento
Nota: Il protocollo è stato sviluppato sulla base di metodi descritti in precedenza 12,13,15.
- Amplificazione PCR di frammenti genici RNA ribosomiale
Questo passaggio crea campioni specifici per piscine rRNA, che saranno utilizzati come modelli per la trascrizione in vitro per la produzione di anti-senso RNA ribosomiale (arRNA) sonde che sono gratuiti per rRNA nel campione di RNA totale.- Progettare e sintetizzare set di primer per rRNA frammento di PCR (Tabella 1).
- Eseguire PCR in 50reazione microlitri utilizzando DNA polimerasi Taq (Qiagen) con 50 ng di DNA template, 1 x tampone PCR, Mg 1.5mm 2 Cl, 0,2 pM primer con 35 cicli di denaturazione a 94 ° C per 10 sec, 15 sec ricottura a 40 ° C per batterica amplicone 23S, e 50 ° C per gli ampliconi altri, e si estendono a 72 ° C per 1 min (estensione finale a 72 ° C per 5 min).
- Visualizza i prodotti di PCR su gel di agarosio all'1%.
- Purificare prodotti PCR utilizzando il kit di purificazione PCR QIAquick con eluizione in 50 microlitri tampone di eluizione. Quantificare la concentrazione utilizzando NanoDrop.
- Trascrizione in vitro di biotina-sonde marcate rRNA anti-senso (arRNA). Nota: In questo passaggio, le sonde del campione specifici di varie ampliconi rRNA sono sintetizzati utilizzando il MEGAscript T7 kit.
- Impostare una reazione di 20 microlitri per ogni amplicone specifico rRNA campione mescolando gli ingredienti indicati (Tabella 2). Incubare per una notte a 37 ° C. Nota: in genere le rese di reazione> 50 mcg di RNA.
- Aggiungere 1 ml DNasi I (incluso nel kit) per la reazione e incubare a 37 ° C per 20 minuti per rimuovere il DNA stampo.
- Aggiungere 100 pl di etanolo 100% per la reazione, centrifugare a 12.000 xg per 10 min a 4 ° C per precipitare sintetizzato arRNA. Gettare il surnatante e lavare il pellet con 750 microlitri di etanolo al 75%. Risospendere le sonde di RNA in 50 microlitri di acqua priva di nucleasi.
- Quantificare concentrazione di RNA utilizzando un NanoDrop. Sonde Conservare a -80 ° C.
- rRNA sottrazione con biotinilato arRNA. Nota: Questa fase utilizza la arRNA biotinilato di ibridare al rRNA nel campione di RNA totale. Gli rRNA-arRNA ibridi vengono catturati da biglie magnetiche rivestite con streptavidina. La procedura si basa sul protocollo descritto nel riferimento 13 con lievi modifiche.
- Reagenti
- Preparare NaOH 0,1 N, 20X cloruri di sodioide-citrato (SSC) e 1 X SSC con il 20% formammide.
- Ibridazione
- Mescolare 1 RNA mcg totali con 16S 23S batteriche e sonde (0,75 mg ciascuno), 18S 28S zanzare e sonde (0,75 mg ciascuna), 1 inibitore microlitri RNasi, 2,5 microlitri tampone SSC 20X, 10 microlitri formammide 100% in un tubo da 200 microlitri PCR , aggiungere acqua priva di nucleasi a 50 microlitri.
- Posizionare il tubo di reazione in un termociclatore ed incubare a 70 ° C per 5 minuti per aprire struttura secondaria e rampa di discesa a 25 ° C con 5 ° C con incrementi di 1 min per ogni ° C per consentire l'ibridazione di sonde rRNA e arRNA.
- Rimozione di rRNA
- Trasferire 300 ul perline rivestite con streptavidina in una provetta 1,7 ml. Posizionare il tubo su un rack di separazione magnetica per immobilizzare le perline. Rimuovere il surnatante. Risospendere le sfere in 300 ml di 0,1 N NaOH e mix bene. Posizionare il tubo di ritorno al rack magnetico e pipettare il surnatante. Risospendere le sfere in 300 ml di 1X SSC, mescolare bene, immobilizzare le perline e pipettare il surnatante. Ripetere il lavaggio con 300 ml di 1X SSC ancora una volta.
- Fai 3 aliquote di perline, 100 pl ciascuno, in 1,7 ml provette. Posizionare il tubo sulla griglia magnetica, e rimuovere il surnatante pipettando. Nota: Le aliquote di perline vengono utilizzate per 3 turni di catturare biotinilati rRNA-arRNA ibridi.
- Aggiungere 50 microlitri 1X SSC con il 20% di formammide nella reazione di 50 microlitri di ibridazione. Trasferire la reazione nel primo tubo aliquota tallone, e mescolare bene. Incubare a temperatura ambiente per 10 min per permettere alle sonde biotinilate-rRNA ibridi da acquisire con perline streptavidina. Flick il tubo ogni 2 minuti per mescolare durante l'incubazione.
- Posizionare il tubo sulla griglia magnetica per immobilizzare le perline, trasferire il sopranatante secondo aliquot di perline, mescolare bene e incubare come sopra. Eseguire la cattura terzo trasferendo supernatante nel terzo tubo di perline. Miscelare e incubare.
- Trasferimento non rRNA supernatante in un nuovo tubo e purificazione condotta utilizzando RNeasy kit MinElute per rimuovere formammide seguendo le istruzioni del produttore.
- Verificare l'efficienza deplezione rRNA utilizzando un kit RNA Agilent 6000 Pico chip su un Bioanalyzer (Figura 4).
- Reagenti
Nota: I rRNA impoverito campioni di RNA sono soggette a mRNA amplificazione, se necessario 8.
Risultati
Il protocollo prevede tre sezioni: (1) la preparazione di modelli di DNA metagenomiche per rRNA PCR, (2) la creazione di sonde di cattura specifici rRNA di esempio, (3) esaurimento di rRNA di RNA totale di ibridazione sottrattiva. Isolamento del DNA di alta qualità metagenomica e RNA è essenziale per l'intero processo. La modifica Meta-G-Nome protocollo di isolamento del DNA produce DNA di alta qualità metagenomica di intestini zanzara, come mostrato in Figura 2. Può essere difficile per isolare l'RNA totale di alta qualità, di intestini di zanzara. Questo è probabilmente dovuto alla presenza di vari enzimi, tra RNasi, in budella zanzara. Abbiamo confrontato le prestazioni estrazione dell'RNA totale di kit Qiagen isolamento dell'RNA alla Roche TriPure. Elettroferogramma di analisi Agilent Bioanalyzer dimostrato che l'RNA totale isolato utilizzando TriPure contiene chiari picchi rRNA (Figura 3), che non si vede nel RNA ottenuto utilizzando il kit Qiagen. Forse zanzara-RNasi è derivata disattivare efficacemented dal fenolo in TriPure. In genere, 50 resa zanzara coraggio ~ 20 mcg di RNA totale. La quantità ideale di input RNA totale per l'attuale processo di esaurimento rRNA è ~ 1 ug, che ottimizza l'efficienza di rRNA sottrazione e rese ~ 150 ng di RNA. Figura 4 mostra il confronto di elettroferogrammi RNA prima e dopo sottrazione rRNA. L'rRNA è stata eliminata in modo efficace, mentre i non-rRNA specie sono state notevolmente arricchito di RNA rimanenti. La presenza di RNA grandi dimensioni (> 2000 bp) indica una buona qualità di mRNA arricchito (Figura 4B). Il protocollo può essere scalata fino ad accogliere un ingresso maggiore di RNA per l'elaborazione. Solitamente 5 microgrammi di RNA è richiesto per il processo di RNA-seq. Pertanto, abbiamo utilizzato MessageAmp II-Bacteria RNA Amplification Kit per generare una adeguata quantità di rRNA impoverito RNA per RNA-Seq. Si consiglia di utilizzare 150 ng o più purificato RNA per l'amplificazione di RNA per garantire la fedeltà di amplificazione. Questo protocollo è stato applIED a una zanzara intestino RNA-Seq progetto. L'esaurimento rRNA è stato realizzato, utilizzando sonde di cattura campione derivato. Tabella 3 elenca la percentuale di rRNA legge nel RNA-Seq uscita di quattro campioni. Due zucchero-fed e due campioni di sangue alimentati intestinali sono stati processati per esaurimento e RNA-Seq. Il sequenziamento Illumina generato 23-24M legge per ogni campione. La sottrazione rRNA efficacemente rimossi 90-99% rRNA sia da tessuti zanzara e microbi nei campioni intestinali. L'esaurimento era quasi completa nei zucchero alimentati campioni intestinali, e 6,12-10,98% rRNA rimasto nelle nutriti con campioni di sangue (Tabella 3).
| Amplicon | Forward 5'-3 ' | Reverse 5'-3 ' |
| 16S batteriche | 27F | 803R_T7 |
| AGAGTTTGATCCTGGCTCAG | TAATACGACTCACTATAGG NCTACCTGGGTATCTAATCC | |
| 347F | 1492R_T7 | |
| GGAGGCAGCAGTRRGGAAT | TAATACGACTCACTATAGG GACGGCTACCTTGTTACGACTT | |
| 23S batteriche | 189F | 2490R_T7 |
| GAASTGAAACATCTHAGTA | TAATACGACTCACTATAGG GCGACATCGAGGTGCCAAAC | |
| 1075F | 2241R_T7 | |
| GTTGGCTTRGARGCAGC | TAATACGACTCACTATAG GGACCGCCCCAGTHAAACT | |
| 18S Mosquito | 18SF1 | 18SR1_T7 |
| GGTTGATCCTGCCAGTAGTAT | TAATACGACTCACTATAGG CAAACGCTTTCGCTTCTGT | |
| 18SF2 | 18SR2_T7 | |
| GGGCCGGCGTTGGCCGAGAAT | TAATACGACTCACTATAGG TTCACTTACGGAAACCTTG | |
| Mosquito 28S | 28SF1 | 28SR1_T7 |
| AGG AAC CAC AGG TAC GGA CC | TAATACGACTCACTATAGG ACCACCAAGCATGGGTCGCC | |
| 28SF2 | 28SR2_T7 | |
| AGGAACCACAGGTACGGACC | TAATACGACTCACTATAG GTCCCGGAGGTGCCTCAA |
Set di primer Tabella 1. Frammento di amplificazione per 16S rRNA batterici e 23 primer sono stati progettati sulla base di 20,19. Sequenze del promotore T7 sono in grassetto.
| 10 x tampone | 2 microlitri |
| Prodotti di PCR (0,5-1 mg) | 1,5 pl |
| ATP (75mm) | 2 microlitri |
| GTP (75mm) | 2 microlitri |
| CTP (75mm) | 1,5 pl |
| UTP (75mm) | 1,5 pl |
| Biotina-11-CTP (10mM) | 3,75 pl |
| Biotina-16-UTP (10mM) | 3,75 pl |
| T7 RNA polimerasi | 2 microlitri |
Tabella 2. Componenti di sintesi in vitro di sonde rRNA.
| Campione | Totale letture | Mosquito rRNAletture (%) | 16S microbiche legge (%) | 23S microbica legge (%) | % Totale di letture rRNA * |
| SM1 | 24341850 | 121,987 (0.50) | 3,717 (0.02) | 6,810 (0.03) | 0,54 |
| SM2 | 23487202 | 114,430 (0.49) | 30,271 (0.13) | 38,261 (0.16) | 0,78 |
| BM1 | 23438304 | 265,433 (1.13) | 2,054,777 (8.77) | 253,051 (1.08) | 10,98 |
| BM2 | 24212240 | 110,240 (0.46) | 1,186,423 (4.90) | 185,074 (0.76) | 6,12 |
Tabella 3. Statstiche di rRNA legge in RNA-seq uscita. SM: zucchero gut fed; BM: gut sangue nutriti. *: La somma delle percentuali di zanzara rRNA e microbiche rRNA legge.

Figura 1. Diagramma che mostra le fasi procedurali del DNA metagenomica e isolamento di RNA, sintesi rRNA cattura della sonda, l'ibridazione, e catturare perline esaurimento based.
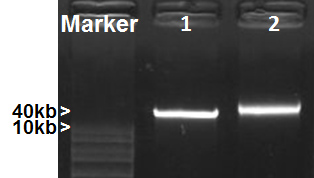
Figura 2. Isolamento del DNA metagenomiche. Campioni di DNA sono stati separati su gel di agarosio all'1%. 1, Fosmid DNA (40kb), 2, Mosquito intestino DNA metagenomica.

Figura 3.Confronto di due reagenti totale isolamento di RNA per la loro efficienza a estrarre un RNA di qualità da intestino di zanzara. Sovrapposizione di elettroferogrammi mostra che l'RNA totale da isolamento TriPure (in blu) ha avuto picchi di rRNA e distribuzione a livello di RNA, mentre quello dall'isolamento Qiagen (in rosso) non ha avuto picchi di rRNA chiare.

Figura 4. Efficienza di rRNA esaurimento. Elettroferogrammi mostrano gli RNA totali prima (A) e dopo (B) deplezione rRNA. I picchi rRNA sono stati rimossi efficientemente. RNA superiore a 4kb rimane nel campione rRNA impoverito. La concentrazione di RNA era 107,6 ng / mL in (A) e 8,6 ng / mL in (B).
Discussione
Una comunità microbica complessa risiede nel ecosistema intestinale zanzara 14,16,17. Metatranscriptomic sequenziamento (RNA-seq) può rivelare le informazioni di contesto dipendente funzionale interrogando l'intero trascrittoma microbica 4,18. Tecnicamente, oligo-d (T) di arricchimento mediata mRNA procariotici non è applicabile a causa dell'assenza di stabile poly (A) code dei messaggeri. In alternativa, deplezione rRNA è stato utilizzato per l'arricchimento mRNA. Qui, abbiamo sviluppato un protocollo basato su sottrazione di esaurire rRNA con sonde rRNA a base di DNA molto proprio metagenomica nella comunità microbica intestinale zanzara. L'efficienza di rimozione rRNA dipende dalla copertura delle sonde di cattura dell'rRNA generati dalla PCR rRNA. L'efficienza ricottura di primer diversi set di mira le regioni conservate varia da diversi taxa 19. Pertanto, si consiglia di provare diverse combinazioni di primer su campioni metagenomiche, e poi mettere in comune leprodotti rRNA per massimizzare la copertura. Nel nostro procedimento, creiamo sonde di cattura combinando rRNA prodotti di PCR di due avanti e due reverse primer, che possono formare 4 combinazioni di set di primer (Tabella 1). Inoltre, rRNA intende formare una struttura secondaria. Rispetto al rRNA lunghezza come sonde 13, frammenti rRNA piccole hanno una minore tendenza a formare la struttura secondaria, che può facilitare l'ibridazione delle sonde di cattura per l'rRNA campione. Le sonde biotinilate efficiente ibridizzare rRNA nel campione di RNA totale e gli ibridi sono rimossi da catturare con perline rivestite con streptavidina. La procedura rimuove la maggior parte del rRNA (Figura 4). Nei quattro campioni abbiamo trasformati, 90-99% di rRNA stato effettivamente impoverito (Tabella 3). Questa procedura può essere ulteriormente modificato per una varietà di insetti associati studi microbioma.
Divulgazioni
Gli autori dichiarano di non avere conflitto di interessi finanziari.
Riconoscimenti
Questo lavoro è stato sostenuto da NIH sovvenzione 1SC2GM092789-01A1, e MS è stato uno studioso di ricerca di NMSU Howard Hughes Medical Institution di laurea Programmi di Ricerca. Il video è stato diretto e prodotto da Amy Lanasa e coordinato dal Dr. Philip Lewis con il Creative Media Institute NMSU.
Materiali
| Name | Company | Catalog Number | Comments |
| Reagent/Material | |||
| Meta-G-Nome DNA isolation kit | Epicentre | MGN0910 | Metagenomic DNA isolation |
| TriPure | Roche | 11667165001 | Mdetagenomic RNA isolation |
| MEGAscript T7 kit | Ambion | AM1334 | In vitro synthesis of RNA probes |
| RNaseZap | Ambion | AM9780 | RNase free working area |
| Biotin-16-UTP | Roche | 11388908910 | In vitro synthesis of RNA |
| Biotin-11-CTP | Roche | 4739205001 | In vitro synthesis of RNA |
| Streptavidin magnetic beads | NEB | S1420S | Capture of rRNA hybrids |
| Magnetic separation rack | NEB | S1506S | Capture of rRNA hybrids |
| RNeasy mini kit | QIAGEN | 74104 | Purification of subtracted RNA |
| RNase-Free DNase Set | QIAGEN | 79254 | Removal DNA contamination |
| Agilent RNA 6000 Pico Kit | Agilent Technologies Inc. | 5067-1513 | Electropherogram of RNA |
| Equipment | |||
| Bio-Gen PRO200 Homogenizer | PRO Scientific | 01-01200 | Mosquito gut tissue homogenization |
| NanoDrop 1000 Spectrophotometer | Thermo Scientific | DNA & RNA quantitation | |
| 2100 Bioanalyzer | Agilent Technologies Inc. | G2940CA | Electropherogram of RNA |
Riferimenti
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nat. Rev. Genet. 10, 57-63 (2009).
- Xie, W., et al. Pyrosequencing the Bemisia tabaci Transcriptome Reveals a Highly Diverse Bacterial Community and a Robust System for Insecticide Resistance. PLoS One. 7, e35181(2012).
- Xie, L., et al. Profiling the metatranscriptome of the protistan community in Coptotermes formosanus with emphasis on the lignocellulolytic system. Genomics. 99, 246-255 (2012).
- Gosalbes, M. J., et al. Metatranscriptomic approach to analyze the functional human gut microbiota. PLoS One. 6, e17447(2011).
- Urich, T., et al. Simultaneous assessment of soil microbial community structure and function through analysis of the meta-transcriptome. PLoS One. 3, e2527(2008).
- Gilbert, J. A., et al. Detection of large numbers of novel sequences in the metatranscriptomes of complex marine microbial communities. PLoS One. 3, e3042(2008).
- Gifford, S. M., Sharma, S., Rinta-Kanto, J. M., Moran, M. A. Quantitative analysis of a deeply sequenced marine microbial metatranscriptome. ISME J. 5, 461-472 (2011).
- Poretsky, R. S., et al. Comparative day/night metatranscriptomic analysis of microbial communities in the North Pacific subtropical gyre. Environmental microbiology. 11, 1358-1375 (2009).
- Shrestha, P. M., Kube, M., Reinhardt, R., Liesack, W. Transcriptional activity of paddy soil bacterial communities. Environmental Microbiology. 11, 960-970 (2009).
- Mettel, C., Kim, Y., Shrestha, P. M., Liesack, W. Extraction of mRNA from soil. Applied and Environmental Microbiology. 76, 5995-6000 (2010).
- He, S., et al. Validation of two ribosomal RNA removal methods for microbial metatranscriptomics. Nat. Meth. 7, 807-812 (2010).
- Pang, X., et al. Bacterial mRNA purification by magnetic capture-hybridization method. Microbiol. Immunol. 48, 91-96 (2004).
- Stewart, F. J., Ottesen, E. A., DeLong, E. F. Development and quantitative analyses of a universal rRNA-subtraction protocol for microbial metatranscriptomics. ISME J. 4, 896-907 (2010).
- Wang, Y., Gilbreath, T. M. 3rd, Kukutla, P., Yan, G., Xu, J. Dynamic gut microbiome across life history of the malaria mosquito Anopheles gambiae in Kenya. PLoS One. 6, e24767(2011).
- Su, C., Sordillo, L. M. A simple method to enrich mRNA from total prokaryotic RNA. Mol. Biotechnol. 10, 83-85 (1998).
- Gusmao, D. S., et al. Culture-dependent and culture-independent characterization of microorganisms associated with Aedes aegypti (Diptera: Culicidae) (L.) and dynamics of bacterial colonization in the midgut. Acta Trop. 115, 275-281 (2010).
- Lindh, J. M., Terenius, O., Faye, I. 16S rRNA gene-based identification of midgut bacteria from field-caught Anopheles gambiae sensu lato and A. funestus mosquitoes reveals new species related to known insect symbionts. Appl. Environ. Microbiol. 71, 7217-7223 (2005).
- Simon, C., Daniel, R. Metagenomic analyses: past and future trends. Appl. Environ. Microbiol. 77, 1153-1161 (2011).
- Wang, Y., Qian, P. Y. Conservative fragments in bacterial 16S rRNA genes and primer design for 16S ribosomal DNA amplicons in metagenomic studies. PLoS One. 4, e7401(2009).
- Hunt, D. E., et al. Evaluation of 23S rRNA PCR primers for use in phylogenetic studies of bacterial diversity. Appl. Environ. Microbiol. 72, 2221-2225 (2006).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon