Method Article
Depletion der ribosomalen RNA für Mosquito Gut metagenomische RNA-seq
In diesem Artikel
Zusammenfassung
A ribosomalen RNA (rRNA) Depletion-Protokoll wurde entwickelt, um Boten-RNA (mRNA) für die RNA-seq der Darm der Mücke metatranscriptome bereichern. Probe spezifischen rRNA-Sonden, die verwendet werden, um über rRNA Subtraktion zu entfernen waren, wurden aus der Mücke und dessen Darm Mikroben erstellt. Leistung des Protokoll kann bei der Entfernung von etwa 90-99% von rRNA führen.
Zusammenfassung
Darm der Mücke nimmt dynamischen mikrobiellen Gemeinschaften in den verschiedenen Phasen des Insekts Lebenszyklus. Charakterisierung des genetischen Kapazität und Funktionalität des Darms Gemeinschaft wird einen Einblick in die Auswirkungen der Darmflora sorgen für mosquito life Züge. Metagenomische RNA-Seq ist ein wichtiges Werkzeug, um Transkriptomen aus verschiedenen Mikroben in einer mikrobiellen Gemeinschaft zu analysieren. Messenger RNA umfasst in der Regel nur 1-3% der Gesamt-RNA, während rRNA stellt ca. 90%. Es ist eine Herausforderung, Boten-RNA aus einer metagenomische mikrobiellen RNA-Probe zu bereichern, weil die meisten prokaryotischen mRNA-Spezies stabilen Poly (A) Schwanz fehlt. Dadurch wird verhindert, Oligo-d (T) vermittelten mRNA Isolation. Hier beschreiben wir ein Protokoll, das Probe abgeleitet rRNA Fängersonden an rRNA von einem metagenomische RNA-Probe zu entfernen beschäftigt. Um zu beginnen, sind beide Mücke und mikrobielle kleinen und großen Untereinheit rRNA-Fragmente aus einem metagenomische Community DNA-Probe verstärkt. Dann wird die Kommunischaft spezifischen biotinylierten Antisense ribosomalen RNA-Sonden werden in vitro mit T7-RNA-Polymerase synthetisiert. Die biotinylierten rRNA-Sonden an der Gesamt-RNA hybridisiert. Die Hybride werden durch Streptavidin-beschichteten Kügelchen eingefangen und entfernt aus der Gesamt-RNA. Diese Subtraktion-basiertes Protokoll effizient entfernt sowohl Mücke und mikrobielle rRNA aus der Gesamt-RNA Probe. Die mRNA angereicherte Probe ist ferner für die RNA-Amplifikation und RNA-Seq verarbeitet.
Einleitung
Next-Generation-Sequencing-Technologie hat Metagenomik Studie, indem sie die taxonomische Zusammensetzung und die genetische Funktionalität eines mikrobiellen Assemblage beurteilen fortgeschritten. RNA-Sequenzierung (RNA-Seq) 1 umgehen kann Kultur-basierten Methoden, um mikrobielle metatranscriptomes in verschiedenen Kontexten 2-5 untersuchen. Ein Haupthindernis für mikrobielle RNA-seq ist die Schwierigkeit bei der Anreicherung mRNA, da die mRNA-Spezies prokaryotischen nicht stabil polyadenyliert sind. Daher ist Oligo-d (T)-vermittelten messenger Bereicherung nicht anwendbar. Entfernung von reichlich rRNA ist ein alternativer Ansatz zur mRNA bereichern. Kommerzielle rRNA Depletion-Kits, wie Microexpress bakterielle mRNA Enrichment Kit (Ambion), RiboMinus Transkriptom Isolation Kit (Bakterien) (Life Technologies), und mRNA-ONLY Prokaryotische mRNA Isolation Kit (Epicentre), die bevorzugt abgebaut rRNA mit einer Exonuklease, sind verwendet worden, zum Entfernen rRNA 6-8. Jedoch sind die Fangsonden im Microexpressoder RiboMinus sind gut zum Entfernen von bekannten rRNA von typischen Gram-positive und Gram-negative Bakterien (siehe Herstellerangaben), aber weniger kompatibel mit rRNA von unbekannten Mikroben. Folglich kann die Entfernung weniger effizient für metagenomische Proben 8-10. Außerdem war die mRNA Wiedergabetreue bedenklich, wenn die Exonucleasebehandlung 11 aufgebracht wurde. Insgesamt war Subtraktion-basierte rRNA Verarmung weniger voreingenommen und effektiver mRNA Anreicherung in metagenomische Einstellungen 10-13.
Darm der Mücke nimmt eine dynamische mikrobiellen Gemeinschaft 14. Wir sind daran interessiert in der Charakterisierung der Funktion von Darm der Mücke microbiome durch RNA-Seq. In einer RNA-Probe aus den Moskito-guts isoliert sind beide Mücke und mikrobieller RNA vorhanden. Hier beschreiben wir eine modifizierte Protokoll Gemeinschaft spezifische rRNA-Sonden zur effizienten Abbau Mücke und mikrobielle rRNA durch subtraktive Hybridisierung. Die resultierende mRNAangereicherte Proben sind für RNA-Seq. Der gesamte Workflow ist in Abbildung 1 dargestellt.
Protokoll
Verfahren
Ein. Mosquito Aufzucht
- Rear Mücke Anopheles gambiae G3 Stamm in Insektarium bei 27,5 ° C mit 80% Luftfeuchtigkeit und 00.12 hr Zyklus von hell / dunkel.
- Führen Larven mit Boden Katzenfutter mit Bierhefe bei 1:1.
- Führen erwachsenen Mücken an Mäusen Blut am Tag 3 nach dem Auflaufen für die Eiererzeugung.
2. Mosquito Gut Dissection
- Autoclave Dissektion Werkzeuge (Dias und Pinzette).
- Sammle 50 Mücken mit einem Aspirator und legen Sie sie auf CO 2-Fluss Bett (Ultimate Flypad). Spülen eine Mücke Probe nacheinander in drei Petrischalen mit 70% Ethanol, um die Mücke Körperoberfläche reinigen.
- Legen Sie die Probe auf einen Objektträger aus Glas unter einem Stereomikroskopie. Entfernen Sie den Darm.
- Sammle 50 guts pro Bedingung für Metagenom DNA-Isolierung. Sammle 50 guts pro Bedingung für metagenomische RNA-Isolierung.
3. Metagenom DNA Isolation
Hinweis: Metagenom DNA wird unter Verwendung des Meta-G-Nome DNA Isolation Kit (Epicentre Biotechnologies) mit der nachstehend beschriebenen Änderungen.
- Zeigen 50 Moskito-guts in 300 ul TE. Homogenisieren sie für 1 min mit Bio-Gen PRO200 Homogenisator (PRO Scientific Inc, USA) bei Drehzahl 2.000 min auf Eis.
- Fügen Sie 2 ul Ready-Lyse Lysozym-Lösung und 1 ul RNase A zur Zellsuspension. Mix durch Vortexen und bei 37 ° C für 30 min.
- Fügen Sie 300 ul Meta-Lysis Solution (2X) und 1 ul Proteinase K auf das Rohr. Mix durch Vortexen. Kurz-Impuls zentrifugieren das Rohr zu gewährleisten, dass alle von der Lösung in dem Boden des Rohres ist, und Inkubieren bei 65 ° C für 15 min, auf Raumtemperatur abkühlen lassen, dann auf Eis für 3-5 min.
- Fügen Sie 350 ul MPC Protein Precipitation Reagent auf das Rohr und mischen durch Vortexen kräftig für 10 Sekunden.
- Pellet ter Debris durch Zentrifugation für 10 min bei 12.000 × g bei 4 ° C.
- Den Überstand in ein sauberes 2-ml-Tube, und entsorgen Sie die Pellets.
- Fügen Sie 570 ul Isopropanol zum Überstand. Mix durch Invertieren des Röhrchens mehrmals.
- Pellet die DNA durch Zentrifugation für 10 min bei 12.000 × g bei 4 ° C. Entfernen Sie die Isopropanol ohne Lösen des DNA-Pellet.
- Add 500 ul 75% Ethanol, um das Pellet zu waschen. Zentrifuge 5 min bei 12.000 × g bei 4 ° C. Entfernen Sie die Ethanol, ohne die DNA-Pellet. Luft trocknen das Pellet bei Raumtemperatur für 2 min. Hinweis: Nicht zu trocken das DNA-Pellet.
- Resuspendieren des DNA-Pellet in 50 ul TE-Puffer.
- Überprüfen der Größe der isolierten DNA durch Vergleich mit der Kontroll-DNA Fosmid (40 kb; 100 ng / ul) in dem Kit vorgesehen, über Gelelektrophorese auf einem 1% Agarosegel.
4. Gesamt-RNA Isolierung
Hinweis: Reinigen Sie den Arbeitsbereichmit RNaseZap RNase Kontamination zu minimieren. Metagenomische RNA wurde aus Moskito Darm Proben unter Verwendung des Tripure Isolation Reagent (Roche) mit einer geringfügigen Modifikation isoliert.
- Legen Sie 50 mosquito Mut in einer 2-ml-Tube mit 500 ul Tripure Isolation Reagent. Homogenisieren für 1 min bei Drehzahl 2.000 min auf Eis mit einem Homogenisator. Lassen Sie sitzen bei RT für 5 min zur Dissoziation des Nucleoproteinkomplexe ermöglichen.
- Fügen Sie 100 ul Chloroform und Vortex für 12 sec, und lassen Sie sich bei RT für 2 min.
- Zentrifugieren bei 10.000 × g für 10 min. Entfernen Sie vorsichtig das Rohr ohne störende getrennten Phasen.
- Übertragen wässrige Phase in ein neues Röhrchen, fügen Sie 250 ul Isopropanol, gut mischen, und legte bei -20 ° C für 20 min.
- Zentrifugieren bei 12.000 × g für 10 min bei 4 ° C zur Ausfällung von RNA.
- Überstand verwerfen, ohne Verdrängen Pellet. Waschen des Pellets mit 750 ul 75% Ethanol.
- Abpipettieren den Überstand ohne das Pellet. Air trocknen das Rohr für 2 min.
- Die RNA mit 30 ul Nuklease freiem Wasser.
- Behandlung der Gesamt-RNA mit DNase bei 37 ° C für 20 min, anschließend die DNase zu inaktivieren durch Inkubation bei 75 ° C für 15 min. Niederschlag RNA mit Ethanol und resuspendieren RNA in 30 ul Nuklease freiem Wasser.
- Bestimmen Sie die Menge der Gesamt-RNA mit einem NanoDrop.
5. Ribosomalen RNA Depletion
Hinweis: Das Protokoll wurde auf der Grundlage zuvor beschriebenen Methoden 12,13,15 entwickelt.
- PCR-Amplifikation der ribosomalen RNA-Genfragmente
Dieser Schritt erstellt Probe-spezifische rRNA-Pools, die als Vorlagen für die in vitro Transkription verwendet werden, um Anti-Sense-ribosomalen RNA (arRNA) Sonden, die komplementär zu der RNA-Probe rRNA produzieren wird.- Entwerfen und zu synthetisieren Primer-Sets für rRNA-Fragment PCR (Tabelle 1).
- Führen PCR in 50ul Reaktion unter Verwendung von Taq DNA-Polymerase (Qiagen) mit 50 ng DNA-Template, 1 x PCR-Puffer, 1,5 mM Mg 2 Cl, 0,2 pM Primer mit 35 Zyklen von Denaturierung bei 94 ° C für 10 sec, Annealing 15 sec bei 40 ° C für bakteriellen 23S Amplikon und 50 ° C für die anderen Amplikons, und sich bei 72 ° C für 1 min (Endverlängerung bei 72 ° C für 5 min).
- Erfahren PCR-Produkte auf 1% Agarosegel.
- Läutern PCR-Produkte mit dem QIAquick-PCR mit Elution in 50 ul Elutionspuffer. Quantifizierung der Konzentration mit NanoDrop.
- In vitro-Transkription von Biotin-markierten Antisense-rRNA-Sonden (arRNA). Anmerkung: In diesem Schritt Probe spezifischen Sonden rRNA aus verschiedenen Amplifikate werden synthetisiert unter Verwendung des T7 MEGAscript Kit.
- Einrichten einer 20 ul Reaktionsgemisch für jede Probe spezifischen rRNA Amplicon durch Vermischen der nachstehenden Bestandteile (Tabelle 2). Inkubieren über Nacht bei 37 ° C. Anmerkung: in der Regel die Ausbeuten> 50 ug RNA.
- Fügen Sie 1 ul DNase I (im Kit enthalten), um die Reaktion und bei 37 ° C für 20 min, um die DNA-Template zu entfernen.
- Je 100 ul 100% Ethanol zum Reaktionsgemisch, Zentrifuge bei 12.000 g für 10 min bei 4 ° C zur Ausfällung synthetisiert arRNA. Überstand verwerfen und waschen das Pellet mit 750 ul 75% Ethanol. Resuspendieren RNA-Sonden in 50 ul Nuklease freiem Wasser.
- Quantifizierung RNA-Konzentration mit einem NanoDrop. Shop-Sonden bei -80 ° C.
- rRNA Subtraktion mit biotinylierten arRNA. Anmerkung: Dieser Schritt setzt das biotinylierte arRNA zu hybridisieren in der RNA-Probe rRNA. Die rRNA-arRNA Hybride werden durch Streptavidin-beschichteten magnetischen Kügelchen eingefangen. Das Verfahren basiert auf dem Protokoll in Referenz 13 beschrieben mit geringen Modifikationen basiert.
- Reagenzien
- Bereiten 0,1 N NaOH, 20X Natrium-Chloride-Citrat (SSC) und 1 X SSC mit 20% Formamid.
- Hybridisation
- Mix 1 ug Gesamt-RNA mit bakteriellen 16S-und 23S-Sonden (0,75 pg pro Stück), Moskito-18S und 28S-Sonden (0,75 pg jedes), 1 ul RNase-Inhibitor, 2,5 ul 20X SSC-Puffer, 10 ul 100% Formamid in einem 200 ul PCR-Röhrchen , Add Nuklease-freiem Wasser auf 50 ul.
- Legen Sie das Reaktionsrohr in einem Thermocycler und inkubieren bei 70 ° C für 5 Minuten zur sekundären Struktur zu öffnen und Rampe auf 25 ° C mit 5 ° C-Schritten für je 1 min ° C, um die Hybridisierung von rRNA und arRNA Sonden ermöglichen.
- Entfernung von rRNA
- Übertragen 300 ul Streptavidin beschichtete Kügelchen in ein 1,7-ml-Tube. Legen Sie das Rohr auf einer magnetischen Trennung Rack, um die Perlen zu immobilisieren. Überstand entfernen. Resuspendieren der Beads in 300 ul 0,1 N NaOH und mix gut. Platzieren Sie den Schlauch wieder auf die magnetische Rack und abpipettieren den Überstand. Resuspendieren der Beads in 300 ul 1X SSC, gut mischen, immobilisieren die Perlen und abpipettieren den Überstand. Wiederholen Sie die Wäsche mit 300 ul 1X SSC noch einmal.
- Machen Sie 3 Aliquots von Perlen, 100 ul jeweils in 1,7 ml Röhrchen. Legen Sie das Rohr auf der magnetischen Rack, und entfernen Sie den Überstand mit einer Pipette. Hinweis: Die Aliquots Perlen sind für 3 Runden zu erfassen biotinylierten rRNA-arRNA Hybriden verwendet.
- Fügen Sie 50 ul 1X SSC mit 20% Formamid in den 50 ul Hybridisierungsreaktion. Übertragen Sie die Reaktion in der ersten Raupe Aliquotierröhrchen und gut mischen. Inkubieren bei Raumtemperatur für 10 min, damit die biotinylierte Sonde rRNA-Hybriden durch Streptavidinbeads eingefangen werden. Flick das Rohr alle 2 Minuten während der Inkubationszeit zu mischen.
- Legen Sie das Rohr auf der magnetischen Rack, um die Perlen zu fixieren, den Überstand der zweiten Aliquot von Perlen, gut mischen und inkubieren wie oben. Durchführung der dritten Aufnahme durch Übertragung Überstand in den dritten Schlauch von Kügelchen. Mischen und inkubieren.
- Übertragen non-rRNA Überstand in ein neues Röhrchen und Durchführung Reinigung mittels RNeasy MinElute Kit Formamid nach der Anleitung des Herstellers zu entfernen.
- Überprüfen Sie die rRNA Erschöpfung Effizienz durch die Verwendung eines Agilent RNA 6000 Pico-Chip-Kit auf einem Bioanalyzer (Abbildung 4).
- Reagenzien
Hinweis: Die rRNA abgereichertem RNA-Proben unterliegen Verstärkung mRNA ggf. 8.
Ergebnisse
Das Protokoll umfasst drei Abschnitte: (1) Herstellung von DNA-templates für metagenomische rRNA PCR, (2) Schaffung Probe spezifischen Capture-rRNA-Sonden, (3) Abreicherung von rRNA aus Gesamt-RNA durch subtraktive Hybridisierung. Isolation von hoher Qualität Metagenom DNA und RNA ist wichtig für den gesamten Prozess. Das modifizierte Meta-G-DNA-Isolierung Nome Protokoll ergibt hochwertige metagenomische DNA aus Moskito Eingeweide, wie in Abbildung 2 dargestellt. Es kann schwierig sein, qualitativ hochwertige Gesamt-RNA aus mosquito Mut zu isolieren. Dies ist wahrscheinlich auf die Anwesenheit von verschiedenen Enzymen, darunter RNase, in Moskito Eingeweide. Wir verglichen die Gesamt-RNA-Extraktion Leistung von Qiagen RNA Isolation Kit von Roche Tripure. Elektropherogramm von Agilent Bioanalyzer Analyse zeigte, dass die Gesamt-RNA isoliert und benutzt Tripure klare rRNA Peaks (Abbildung 3), die nicht an der RNA unter Verwendung des Qiagen-Kits gesehen enthält. Vielleicht Moskito-abgeleitete RNase wird wirksam abgetötetd durch die Phenol in Tripure. Typischerweise 50 Moskito-guts Ausbeute ~ 20 ug Gesamt-RNA. Die optimale Menge an Gesamt-RNA-Eingang für den aktuellen Prozess ist rRNA Depletion ~ 1 pg, was die Effizienz der rRNA Subtraktion und Ausbeuten ~ 150 ng gereinigte RNA optimiert. Abbildung 4 zeigt den Vergleich von RNA Elektropherogramme vor und nach rRNA Subtraktion. Das rRNA wurde effizient entfernt werden, während die nicht-rRNA Spezies wurden stark in den verbleibenden RNA angereichert. Die Anwesenheit großer Größe RNA (> 2.000 bp) zeigt eine gute Qualität der angereicherte mRNA (4B). Das Protokoll kann skaliert werden, um eine größere RNA-Eingang zur Verarbeitung unterzubringen. Regel 5 ug RNA für die RNA-seq Prozess erforderlich. Daher verwendeten wir MessageAmp II-Bakterien RNA Amplification Kit, um eine ausreichende Menge an rRNA abgereicherten RNA zur RNA-Seq erzeugen. Wir empfehlen die Verwendung von 150 ng oder mehr gereinigte RNA für die RNA-Amplifikation, um die Treue der Verstärkung zu gewährleisten. Dieses Protokoll wurde applIED zu einer Mücke gut RNA-Seq Projekt. Die rRNA Depletion wurde effektiv durch Probe abgeleitet Fangsonden erreicht. Tabelle 3 sind der Prozentsatz der rRNA liest die RNA-Seq Leistung von vier Proben. Zwei Zucker gefüttert und zwei Blut-fed gut Proben wurden für Erschöpfung und RNA-Seq verarbeitet. Die Illumina-Sequenzierung erzeugten 23-24M liest für jede Probe. Die rRNA Subtraktion effektiv entfernt 90-99% rRNA sowohl mosquito Gewebe und Mikroben im Darm Proben. Depletion war fast komplett in den Zucker-fed gut Proben und 6,12-10,98% rRNA blieb in den Blut-fed Proben (Tabelle 3).
| Amplicon | Vorwärts 5'-3 ' | Umzukehren 5'-3 ' |
| Bakteriellen 16S | 27F | 803R_T7 |
| AGAGTTTGATCCTGGCTCAG | TAATACGACTCACTATAGG NCTACCTGGGTATCTAATCC | |
| 347F | 1492R_T7 | |
| GGAGGCAGCAGTRRGGAAT | TAATACGACTCACTATAGG GACGGCTACCTTGTTACGACTT | |
| Bakteriellen 23S | 189f | 2490R_T7 |
| GAASTGAAACATCTHAGTA | TAATACGACTCACTATAGG GCGACATCGAGGTGCCAAAC | |
| 1075F | 2241R_T7 | |
| GTTGGCTTRGARGCAGC | TAATACGACTCACTATAG GGACCGCCCCAGTHAAACT | |
| Mosquito 18S | 18SF1 | 18SR1_T7 |
| GGTTGATCCTGCCAGTAGTAT | TAATACGACTCACTATAGG CAAACGCTTTCGCTTCTGT | |
| 18SF2 | 18SR2_T7 | |
| GGGCCGGCGTTGGCCGAGAAT | TAATACGACTCACTATAGG TTCACTTACGGAAACCTTG | |
| Mosquito 28S | 28SF1 | 28SR1_T7 |
| AGG AAC CAC AGG TAC GGA CC | TAATACGACTCACTATAGG ACCACCAAGCATGGGTCGCC | |
| 28SF2 | 28SR2_T7 | |
| AGGAACCACAGGTACGGACC | TAATACGACTCACTATAG GTCCCGGAGGTGCCTCAA |
Tabelle 1. Primer-Sets für rRNA-Fragment-Amplifikation bakterieller 16S und 23 Primer basieren auf 20,19 konzipiert. T7 Promotor-Sequenzen sind fett gedruckt.
| 10 x Puffer | 2 ul |
| PCR-Produkte (0.5-1 ug) | 1,5 ul |
| ATP (75 mM) | 2 ul |
| GTP (75 mM) | 2 ul |
| CTP (75mm) | 1,5 ul |
| UTP (75mm) | 1,5 ul |
| Biotin-11-CTP (10 mM) | 3,75 ul |
| Biotin-16-UTP (10 mM) | 3,75 ul |
| T7-RNA-Polymerase | 2 ul |
Tabelle 2. Bestandteile zur in vitro Synthese von rRNA-Sonden.
| Probe | Insgesamt liest | Mosquito rRNAliest (%) | Mikrobielle 16S liest (%) | Mikrobielle 23S liest (%) | Gesamt% der rRNA gelesen * |
| SM1 | 24.341.850 | 121,987 (0.50) | 3,717 (0.02) | 6,810 (0.03) | 0,54 |
| SM2 | 23.487.202 | 114,430 (0.49) | 30,271 (0.13) | 38,261 (0.16) | 0,78 |
| BM1 | 23.438.304 | 265,433 (1.13) | 2,054,777 (8.77) | 253,051 (1.08) | 10,98 |
| BM2 | 24.212.240 | 110,240 (0.46) | 1,186,423 (4.90) | 185,074 (0.76) | 6,12 |
Tabelle 3. Statschaften der rRNA liest RNA-seq-Ausgang. SM: Zucker gefütterten gut; BM: Blut gefüttert gut. *: Die Summe der Prozentsätze von Moskito rRNA und mikrobielle rRNA liest.

Abbildung 1. Flussdiagramm, das die Verfahrensschritte der metagenomische DNA und RNA-Isolation, rRNA Fangsonde Synthese, Hybridisierung und einzufangen Kügelchen basierten Abreicherung.
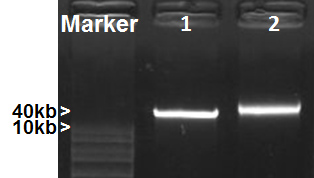
Abbildung 2. Metagenomische DNA-Isolierung. DNA-Proben wurden auf 1% Agarosegel aufgetrennt. 1, Fosmid DNA (40kb); 2, Mosquito gut Metagenom DNA.

Abbildung 3.Vergleich von zwei Gesamt-RNA isoliert Reagenzien für ihre Effizienz bei der Gewinnung eine Qualität von RNA aus Mücke gut. Overlay von Elektropherogrammen zeigt, dass Gesamt-RNA aus Tripure Isolation (in blau) rRNA Peaks und breite RNA-verteilung hatten, während die von Qiagen isoliert (in rot) hatte keine klare rRNA Peaks.

Abbildung 4. Effizienz der rRNA Erschöpfung. Elektropherogramme zeigen die Gesamt-RNA vor (A) und nach (B) rRNA Erschöpfung. Die rRNA Peaks wurden effizient entfernt. RNA größer als 4kb Überreste in rRNA verarmten Probe. Die RNA-Konzentration betrug 107,6 ng / ul in (A) und 8,6 ng / ul in (B).
Diskussion
Eine komplexe mikrobielle Gemeinschaft befindet sich in Darm der Mücke Ökosystems 14,16,17. Metatranscriptomic Sequenzierung (RNA-seq) können kontextabhängig funktionelle Informationen durch Abfragen des gesamten mikrobiellen Transkriptom 4,18 offenbaren. Technisch ist Oligo-d (T) vermittelten Anreicherung prokaryontischer mRNA nicht anwendbar auf das Fehlen von stabilen Poly (A) Schwanz der Boten. Alternativ wurde für mRNA-rRNA-Depletion Anreicherung verwendet worden. Hier haben wir eine Subtraktion-basiertes Protokoll zur rRNA Abbau mit rRNA-Sonden aus der eigenen Metagenom DNA in Darm der Mücke mikrobiellen Gemeinschaft gemacht. Die Effizienz der rRNA Entfernung hängt von der Übertragungsqualität der rRNA Fangsonden, die von der rRNA-PCR generiert werden. Das Glühen Effizienz der verschiedenen Primer-Sets Targeting konservierten Regionen variiert in den verschiedenen Taxa 19. Deshalb empfehlen wir, versuchen verschiedene Kombinationen von Primern, die auf metagenomische Proben und dann die Bündelung derrRNA-Produkte zur Maximierung der Reichweite. In unserem Verfahren, erzeugen wir Erfassungssonden durch Kombinieren rRNA PCR-Produkte von zwei Vorwärts-und zwei Rückwärts-Primern, die 4 Kombinationen von Primersets (Tabelle 1) bilden kann. Darüber hinaus beabsichtigt rRNA, um eine sekundäre Struktur zu bilden. Verglichen mit voller Länge rRNA als Sonden 13, weisen kleinere Fragmente rRNA eine geringere Neigung zur sekundären Struktur zu bilden, die die Hybridisierung der Einfang-Sonden zu der Probe rRNA erleichtern kann. Die biotinylierten Sonden effizient hybridisieren in der RNA-Probe rRNA und die Hybride werden durch die Erfassung mit Streptavidin beschichtete Kügelchen entfernt. Das Verfahren entfernt den größten Teil der rRNA (Abbildung 4). In den vier Proben haben wir verarbeitet wurde 90-99% der rRNA effektiv abgereichert (Tabelle 3). Dieses Verfahren kann weiterhin für eine Vielzahl von Insekten-assoziierten Mikrobioms Studien modifiziert werden.
Offenlegungen
Die Autoren erklären, dass sie keine finanziellen Interessen haben.
Danksagungen
Diese Arbeit wurde vom NIH 1SC2GM092789-01A1 unterstützt und MS war ein Forschungsaufenthalt von NMSU Howard Hughes Medical Institution Undergraduate Research Programs. Das Video wurde inszeniert und von Amy Lanasa produziert und koordiniert von Dr. Philip Lewis mit der Creative Media Institut NMSU.
Materialien
| Name | Company | Catalog Number | Comments |
| Reagent/Material | |||
| Meta-G-Nome DNA isolation kit | Epicentre | MGN0910 | Metagenomic DNA isolation |
| TriPure | Roche | 11667165001 | Mdetagenomic RNA isolation |
| MEGAscript T7 kit | Ambion | AM1334 | In vitro synthesis of RNA probes |
| RNaseZap | Ambion | AM9780 | RNase free working area |
| Biotin-16-UTP | Roche | 11388908910 | In vitro synthesis of RNA |
| Biotin-11-CTP | Roche | 4739205001 | In vitro synthesis of RNA |
| Streptavidin magnetic beads | NEB | S1420S | Capture of rRNA hybrids |
| Magnetic separation rack | NEB | S1506S | Capture of rRNA hybrids |
| RNeasy mini kit | QIAGEN | 74104 | Purification of subtracted RNA |
| RNase-Free DNase Set | QIAGEN | 79254 | Removal DNA contamination |
| Agilent RNA 6000 Pico Kit | Agilent Technologies Inc. | 5067-1513 | Electropherogram of RNA |
| Equipment | |||
| Bio-Gen PRO200 Homogenizer | PRO Scientific | 01-01200 | Mosquito gut tissue homogenization |
| NanoDrop 1000 Spectrophotometer | Thermo Scientific | DNA & RNA quantitation | |
| 2100 Bioanalyzer | Agilent Technologies Inc. | G2940CA | Electropherogram of RNA |
Referenzen
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nat. Rev. Genet. 10, 57-63 (2009).
- Xie, W., et al. Pyrosequencing the Bemisia tabaci Transcriptome Reveals a Highly Diverse Bacterial Community and a Robust System for Insecticide Resistance. PLoS One. 7, e35181(2012).
- Xie, L., et al. Profiling the metatranscriptome of the protistan community in Coptotermes formosanus with emphasis on the lignocellulolytic system. Genomics. 99, 246-255 (2012).
- Gosalbes, M. J., et al. Metatranscriptomic approach to analyze the functional human gut microbiota. PLoS One. 6, e17447(2011).
- Urich, T., et al. Simultaneous assessment of soil microbial community structure and function through analysis of the meta-transcriptome. PLoS One. 3, e2527(2008).
- Gilbert, J. A., et al. Detection of large numbers of novel sequences in the metatranscriptomes of complex marine microbial communities. PLoS One. 3, e3042(2008).
- Gifford, S. M., Sharma, S., Rinta-Kanto, J. M., Moran, M. A. Quantitative analysis of a deeply sequenced marine microbial metatranscriptome. ISME J. 5, 461-472 (2011).
- Poretsky, R. S., et al. Comparative day/night metatranscriptomic analysis of microbial communities in the North Pacific subtropical gyre. Environmental microbiology. 11, 1358-1375 (2009).
- Shrestha, P. M., Kube, M., Reinhardt, R., Liesack, W. Transcriptional activity of paddy soil bacterial communities. Environmental Microbiology. 11, 960-970 (2009).
- Mettel, C., Kim, Y., Shrestha, P. M., Liesack, W. Extraction of mRNA from soil. Applied and Environmental Microbiology. 76, 5995-6000 (2010).
- He, S., et al. Validation of two ribosomal RNA removal methods for microbial metatranscriptomics. Nat. Meth. 7, 807-812 (2010).
- Pang, X., et al. Bacterial mRNA purification by magnetic capture-hybridization method. Microbiol. Immunol. 48, 91-96 (2004).
- Stewart, F. J., Ottesen, E. A., DeLong, E. F. Development and quantitative analyses of a universal rRNA-subtraction protocol for microbial metatranscriptomics. ISME J. 4, 896-907 (2010).
- Wang, Y., Gilbreath, T. M. 3rd, Kukutla, P., Yan, G., Xu, J. Dynamic gut microbiome across life history of the malaria mosquito Anopheles gambiae in Kenya. PLoS One. 6, e24767(2011).
- Su, C., Sordillo, L. M. A simple method to enrich mRNA from total prokaryotic RNA. Mol. Biotechnol. 10, 83-85 (1998).
- Gusmao, D. S., et al. Culture-dependent and culture-independent characterization of microorganisms associated with Aedes aegypti (Diptera: Culicidae) (L.) and dynamics of bacterial colonization in the midgut. Acta Trop. 115, 275-281 (2010).
- Lindh, J. M., Terenius, O., Faye, I. 16S rRNA gene-based identification of midgut bacteria from field-caught Anopheles gambiae sensu lato and A. funestus mosquitoes reveals new species related to known insect symbionts. Appl. Environ. Microbiol. 71, 7217-7223 (2005).
- Simon, C., Daniel, R. Metagenomic analyses: past and future trends. Appl. Environ. Microbiol. 77, 1153-1161 (2011).
- Wang, Y., Qian, P. Y. Conservative fragments in bacterial 16S rRNA genes and primer design for 16S ribosomal DNA amplicons in metagenomic studies. PLoS One. 4, e7401(2009).
- Hunt, D. E., et al. Evaluation of 23S rRNA PCR primers for use in phylogenetic studies of bacterial diversity. Appl. Environ. Microbiol. 72, 2221-2225 (2006).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten