Method Article
Esgotamento de RNA ribossômico para metagenomico Gut Mosquito RNA-seq
Neste Artigo
Resumo
A ribosomal RNA protocolo de depleção (rRNA) foi desenvolvido para enriquecer o RNA mensageiro (mRNA) de RNA-seq do intestino metatranscriptome mosquito. Sondas de amostras específicas rRNA, que foram usados para remover rRNA via subtração, foram criados a partir do mosquito e seus micróbios intestinais. Desempenho do protocolo pode resultar na remoção de aproximadamente 90-99% do rRNA.
Resumo
O intestino do mosquito acomoda comunidades microbianas dinâmicas em diferentes estágios do ciclo de vida do inseto. Caracterização da capacidade genética e funcionalidade da comunidade intestino irá fornecer informações sobre os efeitos da microbiota intestinal em traços de vida do mosquito. Metagenomico RNA-Seq tornou-se uma importante ferramenta para analisar transcriptomas de micróbios vários presentes em uma comunidade microbiana. RNA mensageiro compreende geralmente apenas 1-3% de ARN total, enquanto rRNA constitui cerca de 90%. É um desafio para o enriquecimento de RNA mensageiro a partir de uma amostra de metagenômico microbiana RNA pois a maioria das espécies de ARNm procariótico falta estáveis poli (A) tails. Isto evita que o oligo d (T) de isolamento de mRNA mediada. Aqui, nós descrevemos um protocolo que emprega amostra derivada de rRNA capturar sondas para remover rRNA a partir de uma amostra de RNA metagenômico total. Para começar, os dois fragmentos de mosquito e microbianas rRNA pequenas e grandes subunidades são amplificados a partir de uma amostra de DNA metagenômico comunidade. Então, a comunicaçãonitárias específicas biotinilados anti-sentido de sondas de RNA ribossomal são sintetizados in vitro usando T7 ARN-polimerase. As sondas biotiniladas rRNA são hibridizadas para o RNA total. Os híbridos são capturados por esferas revestidas com estreptavidina e removido a partir do ARN total. Este protocolo de subtração baseada remove eficientemente tanto mosquito e rRNA microbiano a partir da amostra de RNA total. O mRNA da amostra enriquecida é adicionalmente processado para o ARN e amplificação de RNA-Seq.
Introdução
Próxima geração de tecnologia de seqüenciamento tem muito avançado estudo metagenômica, permitindo avaliar a composição taxonômica e funcionalidade genética de um conjunto microbiana. RNA-Sequencing (RNA-Seq) 1 pode ignorar os métodos baseados em cultura para investigar metatranscriptomes microbianas em 2-5 contextos diferentes. Um dos principais obstáculos à microbiana RNA-seq é a dificuldade no enriquecimento do mRNA, como as espécies de ARNm procariótico não são estavelmente polyadenylated. Portanto, oligo d (T) enriquecimento mensageiro mediada não é aplicável. Remoção de rRNA abundante é uma abordagem alternativa para o enriquecimento de mRNA. Kits comerciais de depleção de rRNA, tais como kit Enriquecimento bacteriana mRNA Microexpress (Ambion), RiboMinus Isolation Kit Transcriptoma (bactérias) (Life Technologies), e-mRNA Isolation kit APENAS Prokaryotic mRNA (Epicentre) que preferencialmente degrada rRNA com uma exonuclease, têm sido usados para a remoção de rRNA 6-8. No entanto, as sondas de captura em Microexpressou RiboMinus são bons para a remoção de rRNA a partir de conhecido típicos bactérias Gram-positivas e Gram-negativas (ver as especificações dos fabricantes), mas menos compatível com o rRNA de micróbios desconhecidos. Por conseguinte, a remoção pode ser menos eficiente para 8-10 metagenoma amostras. Além disso, a fidelidade abundância de ARNm era questionável, quando o tratamento com exonuclease foi aplicado 11. No geral, subtração baseada esgotamento rRNA foi menos tendencioso e mais eficaz no enriquecimento de mRNA em ambientes metagenômico 10-13.
O intestino do mosquito acomoda uma comunidade microbiana dinâmica 14. Estamos interessados em caracterizar a função do intestino microbiome mosquito usando RNA-Seq. Numa amostra de RNA isolado a partir dos intestinos de mosquitos, ambos os ARN de mosquito e microbianos estão presentes. Aqui, descrevemos um protocolo modificado para usar sondas específicas da comunidade rRNA de forma eficiente esgotar rRNA mosquito e microbiana por hibridização subtrativa. O mRNA resultanteamostras enriquecidas são apropriados para RNA-Seq. O fluxo de trabalho geral é mostrada na Figura 1.
Protocolo
Procedimento
1. Elevando o mosquito
- Rear mosquito Anopheles gambiae estirpe G3 em insetário de 27,5 ° C com uma humidade de 80% e no ciclo de 12:12 horas luz / escuridão.
- Alimentar as larvas com alimentos chão gato com levedura de cerveja na proporção de 1:1.
- Alimentar os mosquitos adultos em sangue de camundongos no dia 3 pós-emergência para a produção de ovos.
2. Dissecção Gut mosquito
- Autoclave instrumentos de dissecação (slides e fórceps).
- Colete 50 mosquitos usando um aspirador e colocá-los em CO cama fluxo 2 (Ultimate Flypad). Enxaguar um espécime mosquito sequencialmente em três placas de Petri contendo 70% de etanol para limpar a superfície do corpo do mosquito.
- Coloque a amostra sobre uma lâmina de vidro sob uma lupa estereoscópica. Remover o intestino.
- Colete 50 coragem por condição de isolamento de DNA metagenômico. Colete 50 coragem por condição de isolamento do RNA metagenomico.
3. Isolamento de DNA metagenomico
Nota: DNA metagenômico é isolado utilizando o Meta-G-Nome DNA Isolation Kit (Epicentre Biotechnologies), com a modificação descrita abaixo.
- Coloque 50 entranhas de mosquito em 300 TE ul. Homogeneizar durante 1 min, utilizando Bio-Gen PRO200 homogeneizador (PRO Scientific Inc, EUA) a 2000 rpm de velocidade sobre o gelo.
- Adicionam-se 2 jil de Ready-Lyse solução de lisozima e 1 ul de ARNase A à suspensão de células. Mistura por agitação em vórtex e incubar a 37 ° C durante 30 min.
- Adicionar 300 ul de Meta-Lysis Solution (2X) e 1 ml de Proteinase K para o tubo. Misture por vórtex. Resumidamente pulso-centrifugar o tubo para assegurar que a totalidade da solução está no fundo do tubo, e incubar a 65 ° C durante 15 minutos, arrefecer até à temperatura ambiente, e depois colocar em gelo durante 3-5 min.
- Adicionar 350 ul de reagente de precipitação de proteínas MPC para o tubo e misturar vigorosamente em vortex durante 10 seg.
- Pellet tele detritos por centrifugação durante 10 min a 12000 xg a 4 ° C.
- Transferir o sobrenadante para um tubo limpo de 2 ml, e rejeitar o sedimento.
- Adicionar 570 uL de isopropanol ao sobrenadante. Misture por vezes invertendo o tubo várias.
- Pellet o DNA por centrifugação durante 10 min a 12000 xg a 4 ° C. Remover o isopropanol sem desalojar o sedimento de DNA.
- Adicionar 500 ul de etanol a 75% para lavar o sedimento. Centrifugar durante 5 minutos a 12000 xg a 4 ° C. Remover o etanol, sem perturbar o sedimento de DNA. Ar-secar o sedimento à temperatura ambiente durante 2 min. Nota: Não excesso de seca, o pellet de DNA.
- Ressuspender o sedimento de ADN em 50 ul de tampão TE.
- Validar o tamanho do DNA isolado em comparação com o ADN de controlo Fosmid (40 kb, 100 ng / ul) fornecido no kit, através de electroforese em gel num gel de agarose a 1%.
4. Isolamento de RNA total
Nota: Limpe a área de trabalhocom RNaseZap para minimizar a contaminação com RNase. RNA metagenoma foi isolado a partir de amostras intestinais de mosquitos usando o Reagente de Isolamento TriPure (Roche) com uma pequena modificação.
- Coloque 50 entranhas de mosquito em um tubo de 2 ml de reagente com 500 ul TriPure isolamento. Homogeneizar durante 1 min a 2000 rpm de velocidade em gelo usando um homogeneizador. Deixe sentar à temperatura ambiente durante 5 min para permitir que a dissociação de complexos nucleoproteicos.
- Adicionam-se 100 ul de clorofórmio e agitar em vortex por 12 segundos, e deixe sentar à temperatura ambiente durante 2 min.
- Centrifugar a 10.000 xg durante 10 min. Retire cuidadosamente o tubo sem perturbar fases separadas.
- Transferir fase aquosa para um novo tubo, adicionar 250 uL de isopropanol, misturar bem e colocou-se a -20 ° C durante 20 min.
- Centrifugar a 12.000 xg durante 10 min a 4 ° C para precipitar o RNA.
- Desprezar o sobrenadante sem desalojar pelota. Lavar o sedimento com 750 ul de etanol a 75%.
- Pipetar fora o sobrenadante sem perturbar o sedimento. Ari secar o tubo durante 2 min.
- Ressuspender o RNA com 30 ul de água livre de nuclease.
- Tratar o ARN total com ADNase a 37 ° C durante 20 min e, em seguida, desactivar a DNase por incubação a 75 ° C durante 15 min. RNA precipitado com etanol e RNA ressuspender em 30 ul de água livre de nuclease.
- Determinar a quantidade de ARN total utilizando um NanoDrop.
5. Esgotamento RNA ribossomal
Nota: O protocolo foi desenvolvido com base em métodos descritos anteriormente 12,13,15.
- A amplificação por PCR de fragmentos de genes de RNA ribossômico
Este passo cria pools de amostras específicas do rRNA, que serão utilizadas como moldes para a transcrição in vitro para a produção de anti-sentido de RNA ribossomal (arRNA) sondas que são complementares ao rRNA na amostra de RNA total.- Projetar e sintetizar conjuntos de primers para rRNA fragmento de PCR (Tabela 1).
- Executar PCR em 50reacção ul usando DNA-polimerase Taq (Qiagen) com 50 ng de ADN molde, 1 x tampão de PCR, 1,5 mM Mg 2 Cl, 0,2 uM primários com 35 ciclos de desnaturação a 94 ° C durante 10 seg, 15 seg de recozimento a 40 ° C para amplicon 23S bacteriano, e 50 ° C para os amplicons outros, e que se estendem a 72 ° C durante 1 min (extensão final a 72 ° C durante 5 min).
- Ver os produtos de PCR em gel de agarose a 1%.
- Purifica-se os produtos de PCR utilizando o kit de purificação PCR QIAquick com eluição em 50 ul de tampão de eluição. Quantificar a concentração usando NanoDrop.
- Transcrição in vitro de sondas marcadas com biotina-anti-sentido de rRNA (arRNA). Nota: Neste passo, as sondas específicas de amostras de vários amplicons de rRNA são sintetizados utilizando o kit MEGAscript T7.
- Defina-se uma reacção de 20 uL de cada amostra de rRNA amplicon específico misturando os ingredientes a seguir (Tabela 2). Incubar durante a noite a 37 ° C. Nota: normalmente os rendimentos de reação> 50 ug de RNA.
- Adicionar 1 ml de DNase I (incluso no kit) à reacção e incuba-se a 37 ° C durante 20 min para remover o molde de DNA.
- Adicionam-se 100 ul de etanol a 100% para a reacção, de centrifugação a 12.000 xg durante 10 min a 4 ° C para precipitar sintetizado arRNA. Elimine o sobrenadante e lavar o sedimento com 750 ul de etanol a 75%. Ressuspender sondas de RNA em 50 ul de água livre de nuclease.
- Quantificar RNA usando uma concentração NanoDrop. Sondas de armazenar a -80 ° C.
- rRNA subtração com arRNA biotinilado. Nota: Este passo utiliza o arRNA biotinilado para hibridar com rRNA na amostra de RNA total. Os híbridos rRNA-arRNA são capturados por estreptavidina-pérolas magnéticas revestidas. O procedimento baseia-se no protocolo descrito na referência 13, com pequenas modificações.
- Reagentes
- Prepare NaOH 0,1 N, 20X cloro sódioide-citrato (SSC) e 1 X SSC com formamida a 20%.
- Hibridização
- Misturar 1 ug de RNA total com 16S e 23S sondas (0,75 ug de cada), e sondas de mosquito 18S 28S (0,75 ug de cada), 1 ul de inibidor de RNase, 2,5 ul de tampão 20X SSC, 10 ul de formamida a 100% em um tubo de PCR 200 ul , adicione a água livre de nuclease a 50 ul.
- Colocar o tubo de reacção num termociclador e incubar a 70 ° C durante 5 minutos, para abrir a estrutura secundária e rampa para baixo a 25 ° C, utilizando incrementos de 5 ° C durante 1 min cada ° C para permitir a hibridação das sondas de rRNA e arRNA.
- Remoção de rRNA
- Transferir 300 uL de estreptavidina pérolas revestidas dentro de um tubo ml 1.7. Colocar o tubo em uma cremalheira de separação magnética para imobilizar as contas. Remover o sobrenadante. Voltar a suspender as pérolas em 300 ul de NaOH 0,1 N e mix bem. Colocar o tubo de volta para a cremalheira magnético e pipetar fora o sobrenadante. Voltar a suspender as pérolas em 300 ul de SSC 1X, misturar bem, imobilizar as contas e pipetar fora o sobrenadante. Repetir a lavagem com 300 ul de 1X SSC mais uma vez.
- Fazer 3 aliquotas de grânulos, 100 ul de cada, em 1,7 ml tubos. Colocar o tubo no suporte magnético, e remover o sobrenadante por pipetagem. Nota: As alíquotas de contas são utilizadas para a captura de três rodadas de biotinilados rRNA-arRNA híbridos.
- Adicionar 50 ul de SSC 1X, com formamida a 20% da reacção de hibridação em 50 ul. Transferir a mistura reaccional para o tubo de primeira alíquota do grânulo, e misturar bem. Incubar à temperatura ambiente durante 10 min para permitir que os biotinilados sonda de rRNA-híbridos para ser capturado por esferas de estreptavidina. Flick o tubo a cada 2 minutos para misturar, durante a incubação.
- Colocar o tubo no suporte magnético para imobilizar as contas, transferir o sobrenadante para o segundo aliquot dos grânulos, misturar bem e incubar como acima. Realizar a captura terceira transferindo sobrenadante para o terceiro tubo de esferas. Misturar e incubar.
- Transferência não-rRNA sobrenadante para um novo tubo e a purificação utilizando o kit MinElute conduta RNeasy para remover formamida seguindo o manual do fabricante.
- Verificar a eficiência depleção rRNA usando um Agilent 6000 kit RNA chip de Pico em um Bioanalyzer (Figura 4).
- Reagentes
Nota: As amostras de rRNA depleção de RNA são sujeitos a amplificação de mRNA se necessário 8.
Resultados
O protocolo inclui três secções: (1) a preparação de moldes de ADN para metagenoma rRNA PCR, (2) a criação de sondas de amostra de rRNA de captura específicos, (3) a depleção de rRNA a partir de ARN total por hibridação subtractiva. Isolamento de alta qualidade metagenômico ADN e ARN são essenciais para o processo. A modificação Meta-G-Nome protocolo de isolamento de ADN de alta qualidade produz DNA metagenômico de vísceras de mosquitos, conforme mostrado na Figura 2. Ele pode ser um desafio para isolar alta qualidade RNA total de vísceras de mosquitos. Isto é provavelmente devido à presença de várias enzimas, incluindo a RNase, em intestinos de mosquitos. Nós comparamos o desempenho total extracção de RNA da Qiagen kit de isolamento de RNA para Roche TriPure. Electroferograma da Agilent Bioanalyzer análise mostrou que o RNA total isolado utilizando TriPure contém picos claros rRNA (Figura 3), o que não é observado no RNA obtido usando o kit Qiagen. Talvez mosquito derivado de ARNase é efectivamente inactivard pelo fenol em TriPure. Tipicamente, um rendimento de 50 tripas mosquito ~ 20 ug de RNA total. A quantidade ideal de RNA total de entrada para o processo actual de depleção rRNA é ~ 1 ug, que optimiza a eficiência do rRNA subtração e rendimentos ~ 150 ng de RNA purificado. Figura 4 mostra a comparação de eletroferogramas RNA antes e depois de rRNA subtração. O rRNA foi removido eficientemente enquanto que as espécies não-rRNA foram grandemente enriquecido nos RNA restantes. A presença de grande tamanho RNA (> 2.000 pb) indica uma boa qualidade de mRNA enriquecido (Figura 4B). O protocolo pode ser dimensionada para acomodar uma maior entrada de RNA para processamento. 5 ug de RNA geralmente é necessário para o processo de RNA-seq. Portanto, utilizou-se MessageAmp II-As bactérias Kit de Amplificação de RNA para gerar uma quantidade adequada de rRNA empobrecido para RNA RNA-Seq. Recomendamos utilizando 150 ng de RNA purificado ou mais para a amplificação de RNA para assegurar a fidelidade da amplificação. Este protocolo tem sido applIED para um projeto de mosquito intestino RNA-Seq. A depleção de rRNA foi efectivamente conseguida utilizando sondas derivadas de amostras de captura. Tabela 3 lista a percentagem de rRNA lê a saída do RNA-Seq de quatro amostras. Dois-de-açúcar e alimentados duas amostras de sangue alimentados intestino foram processados para a exaustão e RNA-Seq. O sequenciamento Illumina gerado 23-24M lê para cada amostra. A subtração rRNA eficazmente removido rRNA 90-99% de ambos os tecidos de mosquito e micróbios nas amostras de intestino. Depleção quase total nas amostras de açúcar alimentados intestino, e rRNA 6,12-10,98% permaneceu nas amostras de sangue alimentados (Tabela 3).
| Amplicon | Encaminhar 5'-3 ' | Reverso 5'-3 ' |
| 16S | 27F | 803R_T7 |
| AGAGTTTGATCCTGGCTCAG | TAATACGACTCACTATAGG NCTACCTGGGTATCTAATCC | |
| 347F | 1492R_T7 | |
| GGAGGCAGCAGTRRGGAAT | TAATACGACTCACTATAGG GACGGCTACCTTGTTACGACTT | |
| 23S bacterianas | 189F | 2490R_T7 |
| GAASTGAAACATCTHAGTA | TAATACGACTCACTATAGG GCGACATCGAGGTGCCAAAC | |
| 1075F | 2241R_T7 | |
| GTTGGCTTRGARGCAGC | TAATACGACTCACTATAG GGACCGCCCCAGTHAAACT | |
| 18S mosquito | 18SF1 | 18SR1_T7 |
| GGTTGATCCTGCCAGTAGTAT | TAATACGACTCACTATAGG CAAACGCTTTCGCTTCTGT | |
| 18SF2 | 18SR2_T7 | |
| GGGCCGGCGTTGGCCGAGAAT | TAATACGACTCACTATAGG TTCACTTACGGAAACCTTG | |
| Mosquito 28S | 28SF1 | 28SR1_T7 |
| AGG AAC CAC AGG TAC GGA CC | TAATACGACTCACTATAGG ACCACCAAGCATGGGTCGCC | |
| 28SF2 | 28SR2_T7 | |
| AGGAACCACAGGTACGGACC | TAATACGACTCACTATAG GTCCCGGAGGTGCCTCAA |
Conjuntos de mesa 1. Primer para 16S rRNA fragmento de amplificação Bacterianas e 23 primers são projetados com base em 20,19. Sequências do promotor T7 estão em negrito.
| 10 x tampão | 2 ul |
| Produtos de PCR (0,5-1 ug) | 1,5 ul |
| ATP (75 mM) | 2 ul |
| GTP (75 mM) | 2 ul |
| CTP (75 mM) | 1,5 ul |
| UTP (75 mM) | 1,5 ul |
| Biotina-11-CTP (10 mM) | 3,75 ul |
| Biotina-16-UTP (10 mM) | 3,75 ul |
| T7 RNA polimerase | 2 ul |
Tabela 2. Componentes da síntese in vitro de sondas de rRNA.
| Amostra | Total de leituras | Mosquito rRNAlê (%) | 16S microbianas lê (%) | 23S microbiana lê (%) | % Total de rRNA lê * |
| SM1 | 24341850 | 121,987 (0.50) | 3,717 (0.02) | 6,810 (0.03) | 0,54 |
| SM2 | 23487202 | 114,430 (0.49) | 30,271 (0.13) | 38,261 (0.16) | 0,78 |
| BM1 | 23438304 | 265,433 (1.13) | 2,054,777 (8.77) | 253,051 (1.08) | 10,98 |
| BM2 | 24212240 | 110,240 (0.46) | 1,186,423 (4.90) | 185,074 (0.76) | 6,12 |
Tabela 3. Statterísticas de rRNA lê em RNA-seq saída. SM: açúcar intestino alimentado; BM: gut alimentados sangue. *: A soma dos percentuais de mosquito rRNA e rRNA microbiana lê.

Figura 1. Fluxograma que mostra as etapas processuais de DNA metagenômico e isolamento de RNA, rRNA síntese da sonda de captura, a hibridação, e capturar grânulos depleção baseado.
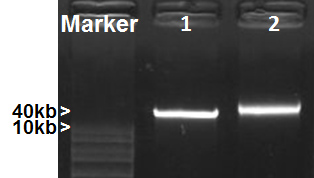
Figura 2. Isolamento de DNA metagenômico. Amostras de DNA foram separados sobre gel de agarose a 1%. 1, Fosmid DNA (40kb); 2, Mosquito DNA metagenomico intestino.

Figura 3.Comparação entre dois reagentes de isolamento de ARN totais para a sua eficiência na extracção de um RNA de qualidade a partir do intestino do mosquito. Sobreposição de eletroferogramas mostra que o RNA total a partir de TriPure isolamento (em azul), teve picos de rRNA e de distribuição de RNA de largura, enquanto que a partir da Qiagen isolamento (a vermelho) não tinha picos rRNA claras.

Figura 4. Eficiência de rRNA depleção. Eletroferogramas mostrar o RNA total, antes (A) e após a depleção de rRNA (B). Os picos de rRNA foram eficientemente removidos. RNA maior do que 4KB permanece em rRNA amostra empobrecido. A concentração de RNA foi 107,6 ng / ul em (A) e de 8,6 ng / uL, em (B).
Discussão
A comunidade microbiana complexa reside no ecossistema estômago do mosquito 14,16,17. Metatranscriptomic seqüenciamento (RNA-seq) pode revelar informações dependente do contexto funcional interrogando o microbiana todo transcriptoma 4,18. Tecnicamente, oligo-d enriquecimento (T) de mRNA mediada procariótico não é aplicável devido à ausência de poli estáveis (A) tails dos mensageiros. Alternativamente, a depleção de rRNA foi usado para o enriquecimento de mRNA. Aqui, foi desenvolvido um protocolo de subtração baseada esgotar rRNA utilizando sondas feitas a partir do rRNA o DNA próprio metagenômico na comunidade microbiana intestinal mosquito. A eficiência da remoção de rRNA depende da cobertura das sondas de captura de rRNA que são gerados por PCR do rRNA. A eficiência de hibridação de iniciador diferente define a segmentação das regiões conservadas varia entre diferentes taxa 19. Portanto, recomendamos tentar diferentes combinações de primers em amostras metagenômico, e depois reunir oprodutos de rRNA para maximizar a cobertura. No nosso processo, criamos sondas de captura, combinando os produtos de PCR de rRNA de dois em frente e dois iniciadores inversos, que podem formar quatro combinações de conjuntos de iniciadores (Tabela 1). Além disso, o rRNA pretende formar uma estrutura secundária. Comparado com rRNA de comprimento completo como sondas 13, fragmentos menores de rRNA têm uma menor tendência para a formação da estrutura secundária, que podem facilitar a hibridação das sondas de captura para o rRNA de amostra. As sondas biotiniladas eficientemente hibridar com rRNA na amostra de RNA total e os híbridos são removidos por captura com contas revestidas com estreptavidina. O procedimento remove a maior parte do rRNA (Figura 4). Nas quatro amostras que tenham transformado, 90-99% de rRNA foi efectivamente esgotado (Tabela 3). Este procedimento pode ser adicionalmente modificado por uma variedade de estudos de insectos associados microbioma.
Divulgações
Os autores declaram que não têm interesses conflitantes financeiros.
Agradecimentos
Este trabalho foi financiado pelo NIH concessão 1SC2GM092789-01A1, e MS era um estudioso de pesquisa de NMSU Médico Howard Hughes Instituição Programas de Pesquisa de Graduação. O vídeo foi dirigido e produzido por Amy Lanasa e coordenado pelo Dr. Philip Lewis com o Creative Media Institute NMSU.
Materiais
| Name | Company | Catalog Number | Comments |
| Reagent/Material | |||
| Meta-G-Nome DNA isolation kit | Epicentre | MGN0910 | Metagenomic DNA isolation |
| TriPure | Roche | 11667165001 | Mdetagenomic RNA isolation |
| MEGAscript T7 kit | Ambion | AM1334 | In vitro synthesis of RNA probes |
| RNaseZap | Ambion | AM9780 | RNase free working area |
| Biotin-16-UTP | Roche | 11388908910 | In vitro synthesis of RNA |
| Biotin-11-CTP | Roche | 4739205001 | In vitro synthesis of RNA |
| Streptavidin magnetic beads | NEB | S1420S | Capture of rRNA hybrids |
| Magnetic separation rack | NEB | S1506S | Capture of rRNA hybrids |
| RNeasy mini kit | QIAGEN | 74104 | Purification of subtracted RNA |
| RNase-Free DNase Set | QIAGEN | 79254 | Removal DNA contamination |
| Agilent RNA 6000 Pico Kit | Agilent Technologies Inc. | 5067-1513 | Electropherogram of RNA |
| Equipment | |||
| Bio-Gen PRO200 Homogenizer | PRO Scientific | 01-01200 | Mosquito gut tissue homogenization |
| NanoDrop 1000 Spectrophotometer | Thermo Scientific | DNA & RNA quantitation | |
| 2100 Bioanalyzer | Agilent Technologies Inc. | G2940CA | Electropherogram of RNA |
Referências
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nat. Rev. Genet. 10, 57-63 (2009).
- Xie, W., et al. Pyrosequencing the Bemisia tabaci Transcriptome Reveals a Highly Diverse Bacterial Community and a Robust System for Insecticide Resistance. PLoS One. 7, e35181(2012).
- Xie, L., et al. Profiling the metatranscriptome of the protistan community in Coptotermes formosanus with emphasis on the lignocellulolytic system. Genomics. 99, 246-255 (2012).
- Gosalbes, M. J., et al. Metatranscriptomic approach to analyze the functional human gut microbiota. PLoS One. 6, e17447(2011).
- Urich, T., et al. Simultaneous assessment of soil microbial community structure and function through analysis of the meta-transcriptome. PLoS One. 3, e2527(2008).
- Gilbert, J. A., et al. Detection of large numbers of novel sequences in the metatranscriptomes of complex marine microbial communities. PLoS One. 3, e3042(2008).
- Gifford, S. M., Sharma, S., Rinta-Kanto, J. M., Moran, M. A. Quantitative analysis of a deeply sequenced marine microbial metatranscriptome. ISME J. 5, 461-472 (2011).
- Poretsky, R. S., et al. Comparative day/night metatranscriptomic analysis of microbial communities in the North Pacific subtropical gyre. Environmental microbiology. 11, 1358-1375 (2009).
- Shrestha, P. M., Kube, M., Reinhardt, R., Liesack, W. Transcriptional activity of paddy soil bacterial communities. Environmental Microbiology. 11, 960-970 (2009).
- Mettel, C., Kim, Y., Shrestha, P. M., Liesack, W. Extraction of mRNA from soil. Applied and Environmental Microbiology. 76, 5995-6000 (2010).
- He, S., et al. Validation of two ribosomal RNA removal methods for microbial metatranscriptomics. Nat. Meth. 7, 807-812 (2010).
- Pang, X., et al. Bacterial mRNA purification by magnetic capture-hybridization method. Microbiol. Immunol. 48, 91-96 (2004).
- Stewart, F. J., Ottesen, E. A., DeLong, E. F. Development and quantitative analyses of a universal rRNA-subtraction protocol for microbial metatranscriptomics. ISME J. 4, 896-907 (2010).
- Wang, Y., Gilbreath, T. M. 3rd, Kukutla, P., Yan, G., Xu, J. Dynamic gut microbiome across life history of the malaria mosquito Anopheles gambiae in Kenya. PLoS One. 6, e24767(2011).
- Su, C., Sordillo, L. M. A simple method to enrich mRNA from total prokaryotic RNA. Mol. Biotechnol. 10, 83-85 (1998).
- Gusmao, D. S., et al. Culture-dependent and culture-independent characterization of microorganisms associated with Aedes aegypti (Diptera: Culicidae) (L.) and dynamics of bacterial colonization in the midgut. Acta Trop. 115, 275-281 (2010).
- Lindh, J. M., Terenius, O., Faye, I. 16S rRNA gene-based identification of midgut bacteria from field-caught Anopheles gambiae sensu lato and A. funestus mosquitoes reveals new species related to known insect symbionts. Appl. Environ. Microbiol. 71, 7217-7223 (2005).
- Simon, C., Daniel, R. Metagenomic analyses: past and future trends. Appl. Environ. Microbiol. 77, 1153-1161 (2011).
- Wang, Y., Qian, P. Y. Conservative fragments in bacterial 16S rRNA genes and primer design for 16S ribosomal DNA amplicons in metagenomic studies. PLoS One. 4, e7401(2009).
- Hunt, D. E., et al. Evaluation of 23S rRNA PCR primers for use in phylogenetic studies of bacterial diversity. Appl. Environ. Microbiol. 72, 2221-2225 (2006).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados