Method Article
דלדול של רנ"א ריבוזומלי לMetagenomic גוט יתוש RNA-seq
In This Article
Summary
פרוטוקול ריבוזומלי רנ"א (rRNA) דלדול פותח כדי להעשיר RNA שליח (mRNA) לRNA-seq של metatranscriptome בטן היתוש. בדיקות דגימה ספציפיות rRNA, ששמשו להסרת rRNA דרך חיסור, נוצרו מהיתושים וחיידקי המעיים שלה. ביצועים של הפרוטוקול יכולים להוביל להסרה של כ 90-99% מrRNA.
Abstract
בטן היתוש להכיל קהילות מיקרוביאלי דינמיות על פני שלבים שונים של מחזור החיים של החרק. אפיון של היכולות ותפקודים הגנטיים של קהילת המעיים יספק תובנה לתוך ההשפעות של חיידקי מעיים על תכונות חי יתושים. Metagenomic RNA-Seq הפך כלי חשוב לנתח transcriptomes מחיידקים המצויים בקהילת חיידקים שונים. RNA השליח בדרך כלל מהווה רק 1-3% מכלל רנ"א, בעוד rRNA מהווה כ 90%. זה מאתגר להעשרת RNA שליח ממדגם רנ"א מיקרוביאלי metagenomic כי מיני mRNA פרוקריוטים רוב חסרים (A) זנבות פולי יציבים. זה מונע בידוד Oligo ד (ט) בתיווך-mRNA. כאן, אנו מתארים פרוטוקול שמעסיק מדגם נגזר rRNA ללכוד בדיקות כדי להסיר rRNA ממדגם RNA סך metagenomic. כדי להתחיל, גם יתושים וברי rRNA של חיידקים קטנים וגדולים למקטע הם מוגברים מדגימת DNA קהילת metagenomic. ואז, commuבדיקות nity ספציפיות biotinylated antisense RNA ריבוזומלי מסונתזות במבחנה באמצעות RNA פולימראז T7. בדיקות rRNA biotinylated הם הכלאה לרנ"א בסך הכל. הכלאיים נתפסו על ידי חרוזי streptavidin מצופים והוצאו מכלל רנ"א. פרוטוקול חיסור מבוסס ביעילות מסיר גם יתושים וrRNA של חיידקים מדגימת RNA המוחלטת. המדגם המועשר mRNA עובר עיבוד נוסף להגברת RNA ו-RNA-Seq.
Introduction
טכנולוגיית הדור הבא של רצף התקדמה מחקר metagenomics מאוד על ידי המאפשר להעריך את ההרכב הטקסונומי והפונקציונלי גנטיים של מכלול של חיידקים. RNA-רצף (RNA-Seq) 1 יכול לעקוף שיטות תרבות מבוססת לחקור metatranscriptomes מיקרוביאלי ב2-5 הקשרים שונים. מכשול עיקרי לחיידקי RNA-seq הוא הקושי בהעשרה-mRNA, כמין mRNA פרוקריוטים אינו polyadenylated ביציבות. לכן, העשרת Oligo ד (ט) בתיווך שליח אינה ישימה. הסרת rRNA שפע היא גישה חלופית להעשרה-mRNA. ערכות מסחריות rRNA דלדול, כגון ערכת Microexpress קטריאלי mRNA העשרה (Ambion), RiboMinus transcriptome בידוד קיט (חיידקים) (חי טכנולוגיות), ורק mRNA-ערכה פרוקריוטים mRNA בידוד (Epicentre) שמעדיפים מדרדרת rRNA עם exonuclease, היו בשימוש להסרת rRNA 6-8. עם זאת, בדיקות ללכוד בMicroexpressאו RiboMinus טוב להסרת rRNA הידוע מחיידקים גראם חיוביים וגראם שליליים טיפוסיים (ראה מפרטים של יצרנים), אבל פחות מתאים עם rRNA מחיידקים לא ידועים. כתוצאה מכך, ההסרה עשויה להיות פחות יעילה עבור 8-10 דגימות metagenomic. חוץ מזה, נאמנות שפע mRNA הייתה מוטלת בספק, כאשר טיפול exonuclease יושם 11. בסך הכל, דלדול rRNA חיסור מבוסס היה פחות מוטה ויעיל יותר בהעשרת mRNA בהגדרות metagenomic 10-13.
בטן היתוש להכיל חיידקי קהילה דינמית 14. אנו מעוניינים באפיון הפונקציה של microbiome בטן היתוש באמצעות RNA-Seq. במדגם RNA המבודד מאומץ היתושים, שני RNA היתושים וחיידקים הם הווה. כאן, אנו מתארים פרוטוקול הותאם לשימוש בדיקות rRNA ספציפיות קהילתיות יעילות כדי לרוקן rRNA יתושים וחיידקים על ידי הכלאת חיסור. MRNA כתוצאהדגימות מועשרות מתאימות לRNA-Seq. העבודה הכוללת מתוארת באיור 1.
Protocol
נוהל
1. חקר גידול יתושים
- מתח אחורי יתוש האנופלס gambiae G3 בinsectary ב27.5 מעלות צלזיוס עם לחות 80% ומחזור שעות 12:12 אור / חושך.
- להאכיל את הזחלים במזון חתולי קרקע עם שמרי בירה ביחס 1:1.
- להאכיל את היתושים בוגרים בדם עכברים ביום 3 לאחר הופעתה לייצור ביצים.
2. Dissection גוט יתושים
- כלי נתיחת חיטוי (שקופיות ומלקחיים).
- אסוף 50 יתושים באמצעות aspirator ומניח אותם על מיטת זרימה 2 CO (Flypad האולטימטיבי). שטוף דגימת יתושים ברצף בשלוש צלחות פטרי המכילים 70% אתנול לניקוי משטח גוף היתוש.
- הנח את הדגימה בשקופית זכוכית תחת stereomicroscopy. הסר את המעיים.
- אסוף 50 אומץ לתנאים לבידוד דנ"א metagenomic. אסוף 50 אומץ לתנאים לבידוד RNA metagenomic.
3. בידוד דנ"א Metagenomic
הערה: ה-DNA Metagenomic מבודדת באמצעות Meta-G-DNA נום בידוד הקיט (יוטכנולוגיות Epicentre) עם השינוי המתואר להלן.
- הנח 50 קרבים נגד יתושים ב300 TE μl. הומוגני לדקות 1 באמצעות Bio-Gen PRO200 homogenizer (PRO המדעית Inc, ארה"ב) בשעת 2000 סל"ד מהירות על קרח.
- הוסף 2 μl של פתרון Lysozyme המוכן lyse וμl 1 מתוך RNase להשעית התא. מיקס ידי vortexing ולדגור על 37 מעלות צלזיוס למשך 30 דקות.
- הוסף 300 μl של פתרון Meta-תמוגה (2X) וμl 1 מתוך proteinase K לצינור. מערבב על ידי vortexing. בקצרה דופק סרכזת הצינור כדי להבטיח שכל הפתרון הוא בתחתית של תחתית, ולדגור על 65 מעלות צלזיוס למשך 15 דקות, מצנן לטמפרטורת חדר, ואז מקום על קרח במשך 3-5 דקות.
- הוסף 350 μl של מגיב משקעי חלבון MPC לצינור והתמהיל ידי vortexing נמרץ למשך 10 שניות.
- גלולה לאהוא הפסולת ידי צנטריפוגה למשך 10 דקות ב12000 XG ב 4 ° C.
- העברת supernatant לצינור נקי 2-מ"ל, וזורק את הכדור.
- הוסף 570 μl של isopropanol לsupernatant. מערבב על ידי היפוך כמה פעמי צינור.
- גלולת דנ"א ידי צנטריפוגה למשך 10 דקות ב12000 XG ב 4 ° C. הסר את isopropanol בלי להינתק DNA הגלולה.
- הוסף 500 μl של אתנול 75% כדי לשטוף את הגלולה. צנטריפוגה למשך 5 דקות ב12000 XG ב 4 ° C. הסר אתנול מבלי להפריע גלולת דנ"א. מזגן מייבש את הגלולה בטמפרטורת חדר למשך 2 דקות. הערה: אל על יבש DNA הגלולה.
- Resuspend DNA גלול ב 50 μl של מאגר TE.
- לאמת את גודלו של ה-DNA המבודד בהשוואה ל-DNA בקרת Fosmid (40 ק"ג; 100 ng / μl) המסופק בערכה, באמצעות ג'ל אלקטרופורזה על 1% agarose ג'ל.
4. בידוד RNA סה"כ
שימו לב: יש לנקות את שטח העבודהעם RNaseZap כדי למזער את זיהום RNase. רנ"א Metagenomic היה מבודד מדגימות מעי יתושים באמצעות מגיב בידוד TriPure (רוש) עם שינויים קלים.
- שים 50 קרבים נגד יתושים בצינור 2-מ"ל עם מגיב הבידוד TriPure μl 500. הומוגני ל1 דקות במהירות 2000 סל"ד על קרח באמצעות homogenizer. תן לו לשבת בRT למשך 5 דקות כדי לאפשר ניתוק של קומפלקסי nucleoprotein.
- הוסף כלורופורם 100 μl ומערבולת עבור 12 שניות, ותן לו לשבת בRT עבור 2 דקות.
- צנטריפוגה XG ב -10,000 למשך 10 דקות. זהירות להוציא את הצינורית ללא שלבים מופרדים מטרידים.
- העברת שלב מימי לצינור חדש, להוסיף 250 isopropanol μl, מערבב היטב, ולשים ב -20 ° C למשך 20 דקות.
- צנטריפוגה ב XG 12000 עבור 10 דקות ב 4 ° C כדי רנ"א משקע.
- בטל supernatant מבלי להינתק גלולה. שטוף את הכדור עם אתנול 75% 750 μl.
- פיפטה את supernatant מבלי להפריע הגלולה.IR ייבוש הצינור עבור 2 דקות.
- Resuspend RNA עם מים חינם nuclease μl 30.
- פנק את רנ"א הסך הכל עם DNase על 37 מעלות צלזיוס למשך 20 דקות, ולאחר מכן לנטרל את DNase ידי הדגרה ב 75 מעלות צלזיוס למשך 15 דקות. רנ"א הנחפז ואתנול וResuspend RNA במים חופשיים nuclease μl 30.
- לקבוע את הכמות של RNA כלל באמצעות NanoDrop.
5. דלדול רנ"א ריבוזומלי
הערה: הפרוטוקול פותח על בסיס שיטות שתוארה קודם לכן 12,13,15.
- הגברת PCR של שברי RNA ריבוזומלי גנים
צעד זה יוצר ברכות מדגם ספציפיות rRNA, אשר ישמש כתבניות לשעתוק במבחנה כדי לייצר חלליות אנטי תחושת ריבוזומלי RNA (arRNA) כי הם מחמיאים לrRNA במדגם RNA המוחלט.- לתכנן ולסנתז סטי פריימר לrRNA בר PCR (1 טבלה).
- הפעל PCR ב 50תגובת μl באמצעות DNA פולימראז תקי (Qiagen) בתבנית 50 ng-DNA, 1 x-PCR חיץ, Mg 1.5mm 2 Cl, 0.2 פריימרים מיקרומטר עם 35 מחזורים של denaturing ב 94 ° C עבור 10 שניות, 15 שניות בחישול 40 ° C עבור amplicon חיידקים 23s, ו50 ° C לamplicons האחרים, והארכה ב 72 מעלות צלזיוס במשך דקות 1 (רחבה סופית ב 72 מעלות צלזיוס למשך 5 דקות).
- צפו במוצרי PCR ב% agarose ג'ל 1.
- לטהר מוצרי PCR באמצעות ערכת טיהור PCR QIAquick עם elution במאגר elution 50 μl. לכמת את הריכוז באמצעות NanoDrop.
- בשעתוק במבחנה של בדיקות rRNA אנטי תחושת ביוטין שכותרת-(arRNA). הערה: בשלב זה, בדיקות ספציפיות לדוגמה מתוך amplicons rRNA השונה מסונתזות באמצעות MEGAscript T7 הערכה.
- הגדרת תגובת μl 20 עבור כל amplicon rRNA מדגם מסוים על ידי ערבוב את המרכיבים הבאים (טבלה 2). דגירת הלילה בשעת 37 ° C. הערה: בדרך כלל תשואות התגובה> 50 RNA מיקרוגרם.
- הוסף 1 DNase אני μl (כלול בערכה) לתגובה ולדגור על 37 מעלות צלזיוס למשך 20 דקות כדי להסיר את תבנית ה-DNA.
- הוסף אתנול 100 μl 100% לתגובה, בצנטריפוגה XG 12000 עבור 10 דקות ב 4 ° C כדי לזרז מסונתז arRNA. בטל supernatant ולשטוף את הגלולה עם אתנול 75% 750 μl. בדיקות resuspend RNA במים חופשיים nuclease μl 50.
- לכמת ריכוז RNA באמצעות NanoDrop. בדיקות בחנויות ב-80 ° C.
- rRNA חיסור עם arRNA biotinylated. הערה: בשלב זה מעסיק arRNA biotinylated להכליא לrRNA במדגם RNA המוחלט. כלאי rRNA-arRNA נתפסו על ידי חרוזים מגנטיים streptavidin מצופים. ההליך מבוסס על הפרוטוקול מתואר בהתייחסות 13 עם שינויים קלים.
- ריאגנטים
- הכן 0.1 N NaOH, chlor נתרן 20XIDE-ציטרט (SSC) ו-X SSC 1 עם פוראמיד 20%.
- הכלאה
- מערבבי 1 RNA מיקרוגרם כלל עם 16S חיידקים ובדיקות 23s (0.75 מיקרוגרם כל אחד), 18S יתושים ובדיקות -28 (0.75 מיקרוגרם כל אחד), מעכב 1 μl RNase, 2.5 μl 20X SSC חיץ, 10 פוראמיד 100% μl בצינור PCR 200 μl , מי nuclease ללא תוספת ל 50 μl.
- הנח את הצינור בתגובת Cycler ולדגור תרמית על 70 המעלות צלזיוס למשך 5 דקות כדי לפתוח מבנה משני וכבשו עד 25 מעלות צלזיוס באמצעות 5 ° C מרווחים לכל 1 דקות C ° לאפשר ההכלאה של בדיקות rRNA וarRNA.
- הסרת rRNA
- העברת 300 חרוזים מצופים streptavidin μl לתוך צינור מ"ל 1.7. הנח את השפופרת על מדף הפרדה מגנטית, כדי לשתק את החרוזים. הסרת supernatant. Resuspend את החרוזים ב300 μl של 0.1N NaOH ומייליםx כן. הנח את הצינור בחזרה למדף המגנטי ופיפטה את supernatant. Resuspend את החרוזים ב300 μl של 1X SSC, מערבב היטב, לשתק את החרוזים ופיפטה את supernatant. חזור על לשטוף עם 300 μl זמן 1X SSC אחד יותר.
- לעשות 3 aliquots של חרוזים, 100 μl כל אחד, בצינורות 1.7 מ"ל. הנח את הצינור על המתלה המגנטית, ולהסיר את supernatant ידי pipetting. הערה: aliquots של חרוזים משמשים ל3 סיבובים של לכידת כלאי biotinylated rRNA-arRNA.
- הוסף 50 1X SSC μl עם פוראמיד 20% לתגובת ההכלאה 50 μl. להעביר את התגובה לתוך צינור aliquot החרוז הראשון, ומערבב היטב. לדגור על RT למשך 10 דקות על מנת לאפשר למכוניות ההיברידיות biotinylated חללית-rRNA שנכבשו בידי חרוזי streptavidin. פליק הצינור כל 2 דקות כדי לערבב במהלך הדגירה.
- הנח את הצינור על המתלה המגנטית, כדי לשתק את החרוזים, להעביר את supernatant לaliq 2uot של חרוזים, מערבב היטב והדגירה כאמורה לעיל. לנהל לכידה השלישית על ידי העברת supernatant לתוך הצינור השלישי של חרוזים. לערבב ודגירה.
- העברת supernatant הלא rRNA לתוך צינור חדש וטיהור התנהלות באמצעות ערכת MinElute RNeasy להסיר פוראמיד בעקבות ההוראות של היצרן.
- בדקו את יעילות דלדול rRNA באמצעות ערכת Agilent RNA 6000 פיקו שבב על Bioanalyzer (איור 4).
- ריאגנטים
הערה: דוגמאות rRNA המדולדלות RNA כפופות לmRNA הגברה במידת הצורך 8.
תוצאות
הפרוטוקול כולל שלושה חלקים: (1) הכנת תבניות DNA metagenomic לrRNA PCR, (2) יצירת בדיקות rRNA לכידה ספציפיות לדוגמה: (3) דלדול של rRNA מרנ"א כלל על ידי הכלאת חיסור. בידוד של metagenomic DNA ו-RNA באיכות הגבוהה הוא חיוני לכל התהליך. פרוטוקול בידוד דנ"א Meta-G-נום השונה מניב דנ"א באיכות גבוהה metagenomic מבטן יתושים, כפי שמוצג באיור 2. זה יכול להיות מאתגר לבודד RNA כלל באיכות גבוהה מבטן יתושים. סביר להניח שסיבה לנוכחותם של אנזימים שונים, לרבות RNase, באומץ נגד יתושים. אנחנו לעומת סך ביצועי מיצוי RNA של ערכת בידוד RNA Qiagen לרוש TriPure. Electropherogram ניתוח Bioanalyzer Agilent הראה כי רנ"א כלל מבודד באמצעות TriPure מכיל פסגות ברורות rRNA (איור 3), שלא ראה ברנ"א המתקבל באמצעות Qiagen הערכה. אולי RNase יתושים המופקים ביעילות להשביתד על ידי פנול בTriPure. בדרך כלל, 50 תשואת אומץ יתוש ~ 20 RNA מיקרוגרם בסך הכל. הכמות האידיאלית של קלט RNA הכולל לתהליך דלדול rRNA הנוכחי היא ~ מיקרוגרם 1, אשר מייעל את היעילות של rRNA חיסור ותשואות ~ 150 ng מטוהרי RNA. איור 4 מראה את ההשוואה של electropherograms RNA לפני ואחרי rRNA החיסור. RRNA הוסר ביעילות ואילו מינים לא rRNA התעשרו מאוד ברנ"א שנותר. הנוכחות של הגודל RNA הגדול (> 2000 נ"ב) מציינת איכות טובה של mRNA המועשר (איור 4 ב). הפרוטוקול ניתן לשנותם כדי להתאים קלט גדול יותר של רנ"א לעיבוד. בדרך כלל 5 מיקרוגרם רנ"א נדרש לתהליך RNA-seq. לכן, השתמש ערכת הגברת MessageAmp השני-RNA חיידקים כדי לייצר כמות מספקת של rRNA המדולדל רנ"א לRNA-Seq. אנו ממליצים להשתמש ב150 ng או יותר מטוהרי RNA להגברת רנ"א כדי להבטיח את הנאמנות של הגברה. פרוטוקול זה כבר Applמטען לפרויקט בטן יתוש RNA-Seq. דלדול rRNA הושג ביעילות באמצעות בדיקות ללכוד נגזרות לדוגמה. לוח 3 רשימות אחוז rRNA קורא בתפוקת RNA-Seq מתוך ארבע דוגמאות. שניים סוכר האכיל ושתי דגימות מעי דם הניזונים עובדו לדלדול וRNA-Seq. רצף Illumina נוצר 23-24M קורא לכל דגימה. RRNA החיסור ביעילות להסיר rRNA 90-99% משני רקמות יתושים וחיידקים בדגימות המעי. הדלדול היה כמעט מלא בדגימות מעי סוכר דשן, וrRNA 6.12-10.98% נשאר בדגימות הדם שהוזן (לוח 3).
| Amplicon | קדימה 5'-3 ' | הפוך 5'-3 ' |
| 16S חיידקים | 27F | 803R_T7 |
| AGAGTTTGATCCTGGCTCAG | TAATACGACTCACTATAGG NCTACCTGGGTATCTAATCC | |
| 347F | 1492R_T7 | |
| GGAGGCAGCAGTRRGGAAT | TAATACGACTCACTATAGG GACGGCTACCTTGTTACGACTT | |
| 23s חיידקים | 189F | 2490R_T7 |
| GAASTGAAACATCTHAGTA | TAATACGACTCACTATAGG GCGACATCGAGGTGCCAAAC | |
| 1075F | 2241R_T7 | |
| GTTGGCTTRGARGCAGC | TAATACGACTCACTATAG GGACCGCCCCAGTHAAACT | |
| 18S יתושים | 18SF1 | 18SR1_T7 |
| GGTTGATCCTGCCAGTAGTAT | TAATACGACTCACTATAGG CAAACGCTTTCGCTTCTGT | |
| 18SF2 | 18SR2_T7 | |
| GGGCCGGCGTTGGCCGAGAAT | TAATACGACTCACTATAGG TTCACTTACGGAAACCTTG | |
| יתוש -28 | 28SF1 | 28SR1_T7 |
| AGG AAC CAC AGG TAC GGA CC | TAATACGACTCACTATAGG ACCACCAAGCATGGGTCGCC | |
| 28SF2 | 28SR2_T7 | |
| AGGAACCACAGGTACGGACC | TAATACGACTCACTATAG GTCCCGGAGGTGCCTCAA |
סטי פריימר טבלת 1. ל16S rRNA בר ההגברה חיידקיים ו23 primers נועדו מבוססים על 20,19. רצפי פרומוטר T7 הם באותיות מודגשות.
10 x חיץ| 2 μl | |
| מוצרי ה-PCR (0.5-1 מיקרוגרם) | 1.5 μl |
| ATP (75mm) | 2 μl |
| GTP (75mm) | 2 μl |
| CTP (75mm) | 1.5 μl |
| UTP (75mm) | 1.5 μl |
| ביוטין-11-CTP (10 מ"מימ) | 3.75 μl |
| ביוטין-16-UTP (10 מ"מימ) | 3.75 μl |
| פולימראז T7-RNA | 2 μl |
טבלת 2. רכיבים בסינתזת מבחנה של בדיקות rRNA.
| מדגם | סה"כ קורא | היתוש rRNAקורא (%) | 16S מיקרוביאלי קורא (%) | 23s מיקרוביאלית קורא (%) | % סה"כ של כניסות rRNA * |
| SM1 | 24341850 | 121,987 (0.50) | 3,717 (0.02) | 6,810 (0.03) | 0.54 |
| SM2 | 23487202 | 114,430 (0.49) | 30,271 (0.13) | 38,261 (0.16) | 0.78 |
| BM1 | 23438304 | 265,433 (1.13) | 2.054.777 (8,77) | 253,051 (1.08) | 10.98 |
| BM2 | 24212240 | 110,240 (0.46) | 1,186,423 (4.90) | 185,074 (0.76) | 6.12 |
לוח 3. Statistics של rRNA קורא בתפוקת RNA-seq. SM: מעי האכיל סוכר; BM: מעי האכיל דם. *: סכום אחוזי היתוש rRNA rRNA של חיידקים וקורא.

איור 1. תרשים זרימה המציג את הצעדים פרוצדורליים של DNA metagenomic ובידוד RNA, rRNA סינתזת לכידת חללית, הכלאה, וללכוד את חרוזי דלדול מבוסס.
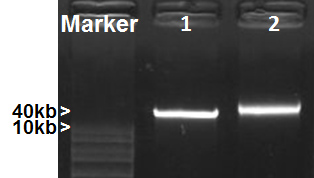
איור 2. בידוד דנ"א Metagenomic. דגימות DNA הופרד ב% agarose ג'ל 1. 1, Fosmid דנ"א (40KB); 2, ה-DNA metagenomic בטן היתוש.

איור 3.השוואה בסך הכל 2 ריאגנטים בידוד RNA ליעילותם בחילוץ של RNA איכותי מבטן יתוש. כיסוי של electropherograms מראה כי רנ"א מוחלט מבידוד TriPure (בכחול) הייתה פסגות rRNA והפצה רחבה רנ"א, בעוד שמבידוד Qiagen (באדום) לא היה לי פסגות rRNA ברורות.

איור 4. יעילות של דלדול rRNA. Electropherograms להראות את רנ"א כלל לפני () ואחרי (ב ') דלדול rRNA. פסגות rRNA הוסרו ביעילות. RNA גדול מ4KB שרידים מדולדלים במדגם rRNA. ריכוז רנ"א היה 107.6 ng / μl ב() ו -8.6 ng / μl ב( B).
Discussion
קהילת חיידקים מורכבת מתגוררת במערכת האקולוגית בטן יתוש 14,16,17. Metatranscriptomic רצף (RNA-seq) יכול לחשוף מידע פונקציונלי תלוי בהקשר של חקירה כל חיידקי transcriptome 4,18. מבחינה טכנית, העשרת Oligo-ד (ט) בתיווך של mRNA פרוקריוטי אינה ישימה בשל העדר (A) זנבות של השליחים פולי היציבים. לחלופין, דלדול rRNA כבר בשימוש להעשרה-mRNA. הנה, אנחנו פתחנו פרוטוקול חיסור מבוסס כדי לרוקן rRNA rRNA באמצעות בדיקות דנ"א שנעשו מmetagenomic עצמו מאוד בקהילת חיידקי מעי היתוש. היעילות של הסרת rRNA תלוי בכיסוי של הבדיקות ללכוד rRNA המופקים על ידי rRNA PCR. יעילות החישול של פריימר השונה קובעת מיקוד משתנה אזורים משתמרים פני מינים אחרים 19. לכן, אנו ממליצים לנסות שילובים שונים של פריימרים על דגימות metagenomic, ולאחר מכן poolingמוצרי rRNA למקסם את הכיסוי. בנוהל שלנו, אנו יוצרים בדיקות ללכוד על ידי שילוב מוצרי rRNA PCR של שני קדימה ושניים פריימרים הפוכים, שיכול להיוצר 4 שילובים של סטי פריימר (טבלה 1). בנוסף, rRNA מתכוון ליצור מבנה שניוני. בהשוואה לrRNA אורך מלא כמו בדיקות 13, שברים קטנים יותר rRNA יש נטייה נמוכה יותר ליצירת מבנה משני, שיכול להקל על ההכלאה של בדיקות ללכידת rRNA המדגם. בדיקות biotinylated יעילות להכליא לrRNA במדגם RNA המוחלט והכלאיים יוסרו על ידי לכידה עם חרוזים מצופים streptavidin. ההליך מסיר את רוב rRNA (איור 4). בארבע הדגימות שעובדנו, 90-99% מrRNA היו מרוקנים ביעילות (לוח 3). הליך זה יכול להיות שונה יותר עבור מגוון רחב של מחקרי Microbiome חרקים נלווים.
Disclosures
החוקרים מצהירים כי אין להם אינטרסים כלכליים מתחרים.
Acknowledgements
עבודה זו נתמכה על ידי מענק NIH 1SC2GM092789-01A1, והטרשת הנפוצה הייתה תלמיד מחקר של תוכניות NMSU הווארד יוז רפואי מכון מחקר לתואר ראשונות. הסרטון ביים והפיק על ידי איימי Lanasa ומתואם על ידי ד"ר פיליפ לואיס עם מדיה מכון קריאייטיב ב NMSU.
Materials
| Name | Company | Catalog Number | Comments |
| Reagent/Material | |||
| Meta-G-Nome DNA isolation kit | Epicentre | MGN0910 | Metagenomic DNA isolation |
| TriPure | Roche | 11667165001 | Mdetagenomic RNA isolation |
| MEGAscript T7 kit | Ambion | AM1334 | In vitro synthesis of RNA probes |
| RNaseZap | Ambion | AM9780 | RNase free working area |
| Biotin-16-UTP | Roche | 11388908910 | In vitro synthesis of RNA |
| Biotin-11-CTP | Roche | 4739205001 | In vitro synthesis of RNA |
| Streptavidin magnetic beads | NEB | S1420S | Capture of rRNA hybrids |
| Magnetic separation rack | NEB | S1506S | Capture of rRNA hybrids |
| RNeasy mini kit | QIAGEN | 74104 | Purification of subtracted RNA |
| RNase-Free DNase Set | QIAGEN | 79254 | Removal DNA contamination |
| Agilent RNA 6000 Pico Kit | Agilent Technologies Inc. | 5067-1513 | Electropherogram of RNA |
| Equipment | |||
| Bio-Gen PRO200 Homogenizer | PRO Scientific | 01-01200 | Mosquito gut tissue homogenization |
| NanoDrop 1000 Spectrophotometer | Thermo Scientific | DNA & RNA quantitation | |
| 2100 Bioanalyzer | Agilent Technologies Inc. | G2940CA | Electropherogram of RNA |
References
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nat. Rev. Genet. 10, 57-63 (2009).
- Xie, W., et al. Pyrosequencing the Bemisia tabaci Transcriptome Reveals a Highly Diverse Bacterial Community and a Robust System for Insecticide Resistance. PLoS One. 7, e35181(2012).
- Xie, L., et al. Profiling the metatranscriptome of the protistan community in Coptotermes formosanus with emphasis on the lignocellulolytic system. Genomics. 99, 246-255 (2012).
- Gosalbes, M. J., et al. Metatranscriptomic approach to analyze the functional human gut microbiota. PLoS One. 6, e17447(2011).
- Urich, T., et al. Simultaneous assessment of soil microbial community structure and function through analysis of the meta-transcriptome. PLoS One. 3, e2527(2008).
- Gilbert, J. A., et al. Detection of large numbers of novel sequences in the metatranscriptomes of complex marine microbial communities. PLoS One. 3, e3042(2008).
- Gifford, S. M., Sharma, S., Rinta-Kanto, J. M., Moran, M. A. Quantitative analysis of a deeply sequenced marine microbial metatranscriptome. ISME J. 5, 461-472 (2011).
- Poretsky, R. S., et al. Comparative day/night metatranscriptomic analysis of microbial communities in the North Pacific subtropical gyre. Environmental microbiology. 11, 1358-1375 (2009).
- Shrestha, P. M., Kube, M., Reinhardt, R., Liesack, W. Transcriptional activity of paddy soil bacterial communities. Environmental Microbiology. 11, 960-970 (2009).
- Mettel, C., Kim, Y., Shrestha, P. M., Liesack, W. Extraction of mRNA from soil. Applied and Environmental Microbiology. 76, 5995-6000 (2010).
- He, S., et al. Validation of two ribosomal RNA removal methods for microbial metatranscriptomics. Nat. Meth. 7, 807-812 (2010).
- Pang, X., et al. Bacterial mRNA purification by magnetic capture-hybridization method. Microbiol. Immunol. 48, 91-96 (2004).
- Stewart, F. J., Ottesen, E. A., DeLong, E. F. Development and quantitative analyses of a universal rRNA-subtraction protocol for microbial metatranscriptomics. ISME J. 4, 896-907 (2010).
- Wang, Y., Gilbreath, T. M. 3rd, Kukutla, P., Yan, G., Xu, J. Dynamic gut microbiome across life history of the malaria mosquito Anopheles gambiae in Kenya. PLoS One. 6, e24767(2011).
- Su, C., Sordillo, L. M. A simple method to enrich mRNA from total prokaryotic RNA. Mol. Biotechnol. 10, 83-85 (1998).
- Gusmao, D. S., et al. Culture-dependent and culture-independent characterization of microorganisms associated with Aedes aegypti (Diptera: Culicidae) (L.) and dynamics of bacterial colonization in the midgut. Acta Trop. 115, 275-281 (2010).
- Lindh, J. M., Terenius, O., Faye, I. 16S rRNA gene-based identification of midgut bacteria from field-caught Anopheles gambiae sensu lato and A. funestus mosquitoes reveals new species related to known insect symbionts. Appl. Environ. Microbiol. 71, 7217-7223 (2005).
- Simon, C., Daniel, R. Metagenomic analyses: past and future trends. Appl. Environ. Microbiol. 77, 1153-1161 (2011).
- Wang, Y., Qian, P. Y. Conservative fragments in bacterial 16S rRNA genes and primer design for 16S ribosomal DNA amplicons in metagenomic studies. PLoS One. 4, e7401(2009).
- Hunt, D. E., et al. Evaluation of 23S rRNA PCR primers for use in phylogenetic studies of bacterial diversity. Appl. Environ. Microbiol. 72, 2221-2225 (2006).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved