Análise de RNA de amostras ambientais usando RT-PCR
Visão Geral
Fonte: Laboratórios do Dr. Ian Pepper e Dr. Charles Gerba - Universidade do Arizona
Autor de Demonstração: Bradley Schmitz
A reação em cadeia de transcrição reversa-polimerase (RT-PCR) envolve o mesmo processo que o PCR convencional — temperatura de ciclismo para amplificar ácidos nucleicos. No entanto, enquanto o PCR convencional só amplifica ácidos desoxiribonucleicos (DNA), o RT-PCR permite a amplificação dos ácidos ribonucleicos (RNA) através da formação de DNA complementar (cDNA). Isso permite que organismos baseados em RNA encontrados dentro do ambiente sejam analisados utilizando métodos e tecnologias que são projetados para o DNA.
Muitos vírus encontrados no ambiente usam o RNA como seu material genético. Vários patógenos virais baseados em RNA, como o Norovirus,e organismos indicadores, como o vírus da mísola de pimenta (PMMoV), não possuem métodos de detecção baseados em cultura para quantificação. Para detectar a presença desses vírus RNA em amostras ambientais do solo, água, agricultura, etc., os ensaios moleculares dependem do RT-PCR para converter RNA em DNA. Sem o RT-PCR, os microbiologistas não seriam capazes de avaliar e pesquisar inúmeros vírus baseados em RNA que representam riscos à saúde humana e ambiental.
O RT-PCR também pode ser empregado como uma ferramenta para medir a atividade microbiana no ambiente. Messenger RNA (mRNA) é o modelo de fixação única para tradução de proteínas, e medir os níveis de diferentes mRNAs indica quais genes dos quais micróbios estão sendo expressos dentro do ambiente. Analisar a expressão genética dá pistas de quais caminhos biológicos são usados pelos organismos para sobreviver em diferentes condições ambientais. Em alguns casos, a expressão genética pode ser utilizada para determinar quais organismos podem sobreviver melhor em condições adversas e ter capacidades para bioremediação de solo ou água contaminados.
Princípios
O PCR baseia-se na amplificação de modelos de DNA, o que limita seu uso na detecção de RNA de organismos. No entanto, o RT-PCR fornece um meio de usar o RNA para produzir cDNA usando enzimas especializadas, conhecidas como transcriptase reversa (RT). Este cDNA pode então ser usado como o modelo inicial para amplificação subsequente com PCR convencional (Figura 1).
A transcrição reversa pode ser controlada para amplificar apenas produtos desejados ou toda uma comunidade de ácidos nucleicos encontrados dentro de uma amostra ambiental, dependendo dos primers que são usados para modelar a síntese de cDNA. Isso é importante, pois as amostras de solo e água são frequentemente saturadas com vários ácidos nucleicos que não são desejados para análises específicas. Primers aleatórios, que podem se ligar a sequências de RNA encontradas em qualquer tipo de micróbios, podem ser usados no RT-PCR para detectar a maioria do RNA, para que a amostra possa ser analisada para a presença e abundância relativa de múltiplos organismos no ambiente. Por outro lado, primers específicos de sequência iniciam a síntese de CDNA para sequências precisas encontradas em apenas um ou poucos organismos. Isso permite que uma amostra ambiental seja testada para um propósito específico, como determinar se o Norovírus, que pode causar doenças gastrointestinais em humanos, está presente na água.

Figura 1. Processo passo a passo da análise RT-PCR de amostras de RNA ambiental.
Procedimento
1. Coleta de Amostras: Amostra de Solo
- Encontre uma amostra através de GPS, coordenadas ou visão.
- Para amostragem aleatória, escolha pontos aleatórios dentro de uma área para obter um censo geral de habitats microbianos. A amostragem transect coleta de pontos ao longo de uma linha reta, por exemplo,adjacente a um córrego. As amostras de grade são sistematicamente retiradas de pontos em intervalos regulares e são úteis para mapear comunidades microbianas em uma área desconhecida ou variável.
- A amostragem dentro de uma profundidade de 7,5-15 cm fornece acesso à atividade microbiana mais abundante que está próxima, mas não dentro, da rizosfera (a região estreita do solo diretamente afetada pelas raízes das plantas e seus microrganismos associados).
- Para coletar a amostra do solo, empurre e gire uma mão no chão até a profundidade predeterminada.
- Levante o auger. O solo é encontrado dentro da haste oca do auger.
- Raspe o solo no fundo do auger em um saco de coleta de solo. Certifique-se de não tocar ou contaminar o solo.
- Rotule a bolsa corretamente com localização, nome, data e hora.
- No laboratório, passe o solo através de uma peneira de 2 mm para remover qualquer cascalho e rocha.
- Analise uma parte do solo para obter o teor de umidade do solo. Para obter detalhes desta etapa, consulte o vídeo da JoVE Science Education sobre a umidade do solo.
2. Coleta de Amostras: Amostra de Água
- Encontre a localização da amostra via GPS, coordenadas ou visão.
- Pegue a água em uma garrafa de armazenamento. Registrar o volume de água coletado; se os micróbios da amostra forem avaliados quantitativamente, a concentração microbiana pode ser determinada com base no volume coletado.
- Teste imediatamente a água para quaisquer parâmetros necessários para o experimento (temperatura, pH, condutividade, salinidade, nitrogênio e teor de fósforo, etc.) utilizando as sondas eletrônicas apropriadas.
- Coloque a garrafa contendo a amostra de água em um refrigerador com gelo. Transfira o refrigerador para o laboratório.
3. Extração de RNA
- Para coletar e concentrar microrganismos da amostra ambiental, consulte o vídeo da JoVE Science Education sobre a extração de ácido nucleico comunitário.
- Extrair RNA de vírus usando um kit de extração comercial de acordo com as instruções do fabricante.
- Brevemente, misture primeiro as amostras com tampão de lise complementado com etanol e, em seguida, adicione as amostras em colunas de spin.
- Centrifugar as colunas a aproximadamente 12.000 x g, descartar fluxo através. Adicione o tampão de lavagem às colunas e centrífugas novamente.
- Adicione água livre de RNase às colunas e centrífuga por 30 s para elute RNA em tubos de microfuge de baixa adesão estéreis de 1,5 mL.
4. Transcrição reversa - PCR
- Recupere os seguintes reagentes do congelador -20 °C: dNTP, concentrado(por exemplo,10x) tampão de transcrição reversa, primers (neste exemplo, primers aleatórios). Descongele isso no gelo ou à temperatura ambiente dentro de um capô limpo, e mantenha-os no gelo uma vez descongelados. Também recupere a transcriptase reversa e o inibidor de RNase e mantenha-os no gelo.
- Calcule os volumes de reagente necessários para fazer uma "mistura mestre" que combina todos os reagentes constantes entre cada reação (ver Tabela 1 para uma reação amostral). Prepare mix mestre suficiente para cada amostra, bem como um controle positivo (usando um modelo de transcrição conhecido) e controle negativo (por exemplo,sem transcrição reversa, com apenas água como modelo, etc.) reações. Inclua um volume extra de 10%.
- Monte a mistura mestre em tubos de microfuça de baixa adesão de 1,5 mL. Isso minimiza a ligação das moléculas de reagente às paredes plásticas dos tubos.
- Quando os reagentes forem descongelados, adicione volumes calculados ao tubo de microfuça. Vórtice suavemente e centrífuga cada tubo antes da adição. Certifique-se de alterar as pontas da pipeta entre a adição de cada reagente para evitar contaminação.
- Depois de todos os reagentes terem sido adicionados, vórtice e centrífuga a mistura mestre para garantir uma mistura homogênea. Coloque os reagentes de volta ao armazenamento a -20 °C.
- Prepare e rotule tiras PCR de 8 tubos, designando um tubo para cada amostra ou reação de controle.
- Alíquota igual de mistura mestre em cada tubo. Em seguida, adicione componentes específicos de reação, como os extratos de RNA.
- Coloque a tampa da tampa da tira com segurança na tira PCR e centrífuga em um minicentrifuge com um adaptador de tubo de tira para garantir que todo o líquido seja coletado na parte inferior de cada tubo(Figura 2).
- Coloque a tira PCR com segurança no termociclador. Pressione para baixo para garantir que os tubos estejam protegidos.
- Defina o termociclador para executar o programa apropriado para o RT que está sendo usado (consulte tabela 2 para um protocolo de amostra). Quando o programa estiver concluído, os tubos conterão produtos cDNA, que podem então ser submetidos à amplificação pcr. Para obter detalhes, consulte os vídeos da JoVE Science Education sobre PCR e PCR quantitativo. Armazene cDNA a -20 °C até usar.

Figura 2. Tira de 8 tubos tampada contendo mistura e extrato mestre.
| Reagente | Volume por reação 1 (μL) |
| 10x tampão RT | 2.0 |
| 25x dNTPs | 0.8 |
| Primer aleatório de 10x | 2.0 |
| Multiscrito | 1.0 |
| Inibidor de Rnase | 1.0 |
| Grau Molecular H2O | 3.2 |
| Total Volume | 10 |
Mesa 1. RT Master Mix Ingredientes.
| Passo 1 | Passo 2 | Passo 3 | Passo 4 |
| 25 °C, 10 min | 37 °C , 120 min | 85 °C , 5 min | 4 °C , ∞ |
Mesa 2. Programa de Thermocicr de Reação RT.
Resultados
Quando o RT-PCR estiver completo, parte do produto PCR pode ser separado e visualizado em um gel de agarose(Figura 3). Neste exemplo, uma cartilha específica de genes foi usada para detectar a presença de um vírus RNA. Faixas do tamanho esperado são obtidas a partir de duas das amostras e da reação de controle positivo, mas não do controle negativo, indicando a presença desse vírus em duas das amostras de água que estão sendo testadas.
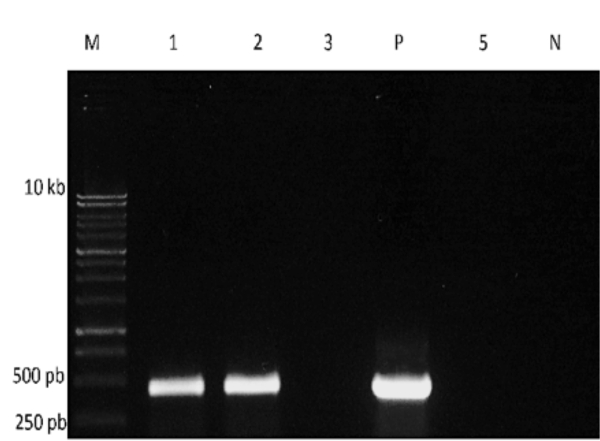
Figura 3. Eletroforese de gel de produtos RT-PCR. M: marcador de tamanho de DNA; P: controle positivo; N: controle negativo. As reações utilizando RNA de quatro amostras de água foram realizadas nas faixas 1, 2, 3 e 5.
Aplicação e Resumo
O RT-PCR é necessário para criar cDNA a partir de um modelo RNA. Isso permite que microrganismos baseados em RNA sejam analisados utilizando ensaios moleculares desenvolvidos para DNA. Uma vez sintetizado o CDNA, os ensaios do PCR podem determinar a presença ou ausência de microrganismos baseados em RNA dentro de uma amostra ambiental. Isso permite uma análise mais a jusante para determinar a ecologia microbiana, os riscos à saúde e os riscos ambientais.
O RT-PCR também pode ser utilizado para avaliar o mRNA como um meio de observar quais genes estão sendo expressos em um ambiente. Isso fornece informações sobre quais proteínas e caminhos os micróbios dependem para sobreviver em condições ambientais particulares. Análises de expressão genética podem identificar vias microbianas que despreensam contaminantes ambientais, como hidrocarbonetos ou solventes clorados, e micróbios com essas vias podem ser aproveitados para a bioremediação.
A avaliação de riscos incorpora o RT-PCR para analisar os riscos à saúde humana e ambiental. A combinação da TR com o PCR quantitativo permite que os vírus RNA sejam enumerados dentro das amostras, para que a exposição humana e ambiental possa ser calculada para fins de Avaliação quantitativa de Risco Microbiano (QMRA).
Pular para...
Vídeos desta coleção:

Now Playing
Análise de RNA de amostras ambientais usando RT-PCR
Environmental Microbiology
40.9K Visualizações

Determinação do teor de umidade no solo
Environmental Microbiology
359.9K Visualizações

Técnica Asséptica em Ciências Ambientais
Environmental Microbiology
126.7K Visualizações

Coloração de Gram de bactérias de fontes ambientais
Environmental Microbiology
100.8K Visualizações

Visualização de Microrganismos do Solo por Meio do Ensaio de Lâmina de Contato e Microscopia
Environmental Microbiology
42.7K Visualizações

Fungos Filamentosos
Environmental Microbiology
58.0K Visualizações

Extração de DNA de comunidades de colônias bacterianas
Environmental Microbiology
29.1K Visualizações

Detecção de Microrganismos Ambientais com Reação em Cadeia da Polimerase e Eletroforese em Gel
Environmental Microbiology
44.8K Visualizações

Quantificando Microorganismos e Vírus Ambientais Usando qPCR
Environmental Microbiology
48.2K Visualizações

Análise da Qualidade da Água por Organismos Indicadores
Environmental Microbiology
30.0K Visualizações

Isolamento de bactérias fecais de amostras de água por filtração
Environmental Microbiology
39.7K Visualizações

Detecção de Bacteriófagos em Amostras Ambientais
Environmental Microbiology
40.9K Visualizações

Cultivo e Enumeração de Bactérias de Amostras de Solo
Environmental Microbiology
185.1K Visualizações

Análise da Curva de Crescimento Bacteriano e suas Aplicações Ambientais
Environmental Microbiology
296.6K Visualizações

Contagem de Algas em Métodos de Cultura
Environmental Microbiology
14.1K Visualizações
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados