Analyse d'ARN d'échantillons environnementaux à l'aide de la RT-PCR
Vue d'ensemble
Source : Laboratoires du Dr Ian poivre et Dr Charles Gerba - Université de l’Arizona
Auteur mettant en évidence : Bradley Schmitz
Réaction en chaîne polymérase transcription inverse (RT-PCR) implique le même processus que la PCR classique — cyclisme température d’amplification des acides nucléiques. Toutefois, alors que la PCR classique ne fait qu’amplifier acides désoxyribonucléiques (ADN), RT-PCR permet l’amplification des acides ribonucléiques (ARN) par le biais de la formation de l’ADN complémentaire (ADNc). Cela permet aux organismes de base d’ARN trouvées dans l’environnement d’être analysés utilisant méthodes et technologies qui sont conçus pour l’ADN.
Beaucoup de virus présents dans l’environnement utilise RNA comme leur matériel génétique. Plusieurs base d’ARN virus pathogènes, tels que les Noroviruset micro-organismes indicateurs, tels que les virus doux de marbrure de poivre (PMMoV), n’ont pas de méthodes de détection basée sur la culture de quantification. Afin de détecter la présence de ces virus à ARN dans des échantillons environnementaux de sol, eau, agriculture, etc., essais moléculaires dépendent de RT-PCR pour convertir RNA dans l’ADN. Sans la RT-PCR, microbiologistes ne serait pas en mesure de dosage et de nombreux virus à base d’ARN qui posent des risques pour la santé humaine et l’environnementale de recherche.
RT-PCR peut également être employé comme un outil pour mesurer l’activité microbienne dans l’environnement. ARN messager (ARNm) est le modèle simple brin pour la traduction des protéines, et la mesure des niveaux d’ARNm différents indique quels gènes dont les microbes sont exprimées au sein de l’environnement. Analyse de l’expression des gènes donne des indices à quelles voies biologiques sont utilisés par les organismes pour survivre dans des conditions environnementales différentes. Dans certains cas, l’expression des gènes peut être utilisée pour déterminer quels organismes peuvent survivent mieux dans des conditions difficiles et ont des capacités pour la biorestauration des sols contaminés ou d’eau.
Principles
PCR est basé sur l’amplification des gabarits d’ADN, ce qui limite son utilisation dans la détection de RNA d’organismes. Toutefois, la RT-PCR fournit un moyen d’utilisant l’ARN pour produire le cDNA utilisant des enzymes spécialisées, appelées la transcriptase inverse (RT). Cet ADNc utilisable alors que le modèle de départ pour l’amplification ultérieure avec la PCR classique (Figure 1).
La transcription inverse peut être contrôlée pour amplifier uniquement les produits désirés ou une communauté entière d’acides nucléiques, trouvé dans un échantillon environnemental, selon les amorces qui sont utilisés pour modèle la synthèse de cDNA. C’est important, car les échantillons de sol et l’eau sont souvent saturées avec de divers acides nucléiques qui ne sont pas souhaitées pour des analyses spécifiques. Amorces aléatoires, ce qui peuvent lier des séquences d’ARN dans n’importe quel type de microbes, peuvent être utilisés par RT-PCR pour détecter l’ARN plus, donc l’échantillon peut être analysé pour la présence et l’abondance relative de plusieurs organismes dans l’environnement. En revanche, amorces spécifiques de séquence initient la synthèse de cDNA pour des séquences précises dans seulement un ou plusieurs organismes. Cela permet un échantillon environnemental à tester dans un but précis, par exemple déterminer si Norovirus, qui peuvent causer des maladies gastro-intestinales chez l’homme, est présent dans l’eau.

La figure 1. Étape par étape de l’analyse d’échantillons environnementaux de RNA RT-PCR.
Procédure
1. prélèvement : Échantillon de sol
- Trouvez un emplacement échantillon via GPS, coordonnées ou vue.
- Pour l’échantillonnage aléatoire, choisir des points aléatoires au sein d’une région pour obtenir un recensement général des habitats microbiens. Transect d’échantillonnage perçoit des points le long de la ligne droite, par exemple, adjacent à un lit. Grille échantillons proviennent systématiquement des points à intervalles réguliers et sont utiles pour les communautés microbiennes de cartographie dans une région inconnue ou variable.
- Profondeur d’échantillonnage dans un 3-6 pouces (7,5 à 15 cm) permet d’accéder à l’activité microbienne plus abondante qui est proche, mais pas dans la rhizosphère (la région étroite du sol directement touché par les racines des plantes et les microorganismes associés).
- Recueillir l’échantillon de sol, pousser et tourner une tarière à main dans le sol à la profondeur prédéterminée.
- Soulevez la tarière. Le sol se trouve dans la tige creuse de la tarière.
- Racler le sol au bas de la vis sans fin dans un sac de ramassage de sol. Être sûr de ne pas toucher ou contaminer le sol.
- Le sac correctement avec l’emplacement, nom, date et heure de l’étiquette.
- Au laboratoire, passez le sol à travers un tamis de 2 mm pour éliminer tout gravier et la roche.
- Analyser une partie du sol pour la teneur en humidité du sol. Pour plus de détails de cette étape, consultez JoVE Science Education vidéo sur l’humidité du sol.
2. prélèvement : Échantillon de l’eau
- Recherchez l’emplacement de l’échantillon via GPS, coordonnées ou vue.
- Recueillez l’eau dans un flacon. Noter le volume d’eau recueillie ; Si les microbes dans l’échantillon sont quantitativement analysés, puis la concentration microbienne peut être déterminée compte tenu du volume collecté.
- Tester immédiatement l’eau pour tous les paramètres requis pour l’expérience (température, pH, conductivité, salinité, azote et phosphore contenu, etc) en utilisant les sondes électroniques appropriés.
- Placer le flacon contenant l’échantillon d’eau dans une glacière avec de la glace. Transférer la glacière au laboratoire.
3. Extraction de l’ARN
- Pour rassembler et concentrer les micro-organismes provenant de l’échantillon environnemental, veuillez vous reporter à la vidéo sur l’extraction des acides nucléiques communauté JoVE Science Education.
- Extrait RNA virus à l’aide d’un kit d’extraction commerciale conformément aux instructions du fabricant.
- En bref, tout d’abord mélanger les échantillons avec tampon de lyse complété avec de l’éthanol, puis ajoutez les échantillons dans les colonnes à centrifuger.
- Centrifuger les colonnes à environ 12 000 x g, redistribution de défausse. Tampon de lavage s’ajoute les colonnes et centrifuger à nouveau.
- Ajouter de l’eau exempte de RNase aux colonnes et centrifuger pendant 30 s à éluer l’ARN dans des tubes stériles microtubes de faible adhérence de 1,5 mL.
4. reverse Transcription - PCR
- Récupérer les réactifs suivants du congélateur-20 ° C : dNTP, concentré (par exemple, x 10) inverser un tampon transcription amorces (dans cet exemple, amorces aléatoires). Décongeler celles-ci sur la glace ou à température ambiante à l’intérieur d’une hotte propre et gardez-les sur la glace après décongelé. Aussi récupérer la transcriptase inverse et l’inhibiteur de RNase et mettez-les sur la glace.
- Calculer les volumes de réactifs nécessaires pour faire un « mix master » qui combine tous les réactifs constant entre chaque réaction (voir le tableau 1 pour une réaction de l’échantillon). Préparer suffisamment mélange principal pour chaque échantillon, comme un contrôle positif (à l’aide d’un modèle connu de transcription) et négatif (par exemple, sans la transcriptase inverse, avec seulement de l’eau comme modèle, etc.) des réactions de contrôle. Inclure une remise de 10 % en volume.
- Assembler le mélange maître dans des microtubes 1,5 mL faible adhérence. Cela minimise la liaison de molécules de réactifs sur les parois des tubes en plastique.
- Lorsque les réactifs sont décongelés, ajouter les volumes calculés pour le tube à centrifuger. Doucement de vortex et centrifuger chaque tube avant l’addition. Veillez à modifier les pointes de pipette entre l’ajout de chaque réactif pour empêcher la contamination.
- Après tous les réactifs ont été ajoutées, vortex et centrifuger master mix pour assurer un mélange homogène. Remettre les réactifs en conservation à-20 ° C.
- Préparer et étiqueter de 8 tubes PCR bandes, désignant un tube pour chaque réaction d’échantillon ou de contrôle.
- Aliquote un volume égal de mélange maître dans chaque tube. Ensuite, ajouter des composants de réaction spécifiques, tels que les extraits de RNA.
- Placez le couvercle de la bande sur la bande de PCR et centrifuger dans un minicentrifuge avec un adaptateur de tube de bande pour assurer tout le liquide est recueilli au bas de chaque tube (Figure 2).
- Bande de PCR place solidement dans le thermocycleur. Appuyez pour s’assurer que les tubes sont sécurisés.
- Définissez le thermocycleur pour exécuter le programme approprié pour le RT utilisé (voir le tableau 2 pour un protocole d’échantillonnage). Lorsque le programme est terminé, les tubes contiendra cDNA produits, qui peuvent alors être soumis à l’amplification par PCR. Pour plus de détails, veuillez consulter les vidéos de l’enseignement des sciences JoVE sur PCR et PCR quantitative. Stocker les ADNc à-20 ° C jusqu'à l’utilisation.

La figure 2. Plafonné à bande de 8-tube contenant l’extrait et mélange principal.
| Réactif | Volume 1 (μL) de la réaction |
| mémoire tampon RT 10 x | 2.0 |
| 25 x dNTPs | 0,8 |
| 10 x amorce aléatoire | 2.0 |
| Multiscribe | 1.0 |
| Inhibiteur de RNase | 1.0 |
| Niveau moléculaire H2O | 3.2 |
| Volume total | 10 |
Le tableau 1. Ingrédients de Mix Master RT.
| Étape 1 | Étape 2 | Étape 3 | Étape 4 |
| 25 ° C, 10 min | 37 ° C, 120 min | 85 ° C, 5 min | 4 ° C, ∞ |
Le tableau 2. Programme RT réaction thermocycleur.
Résultats
Lorsque RT - PCR est terminée, certains des produits PCR peuvent être séparés et visualisés sur gel d’agarose (Figure 3). Dans cet exemple, une amorce de gène-spécifique a été utilisée pour détecter la présence d’un virus à ARN. Bandes de la taille attendue sont obtenus de deux des échantillons et la réaction de contrôle positif, mais pas de contrôle négatif, indiquant la présence de ce virus chez deux des échantillons d’eau mis à l’essai.
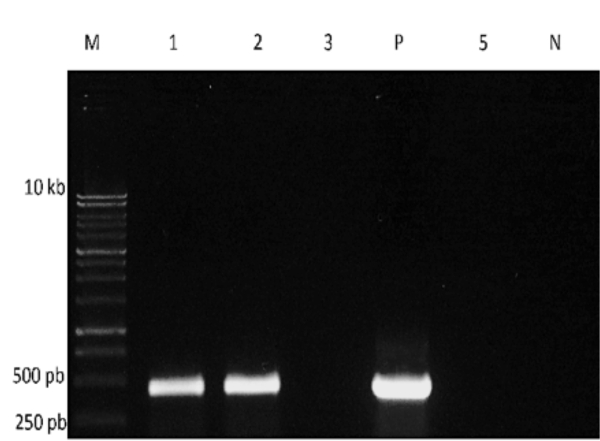
Figure 3. Électrophorèse sur gel des produits RT-PCR. M: marqueur de taille ADN ; P: contrôle positif ; N : contrôle négatif. Les réactions utilisant l’ARN de quatre échantillons d’eau ont été exécutées dans les voies 1, 2, 3 et 5.
Applications et Résumé
RT-PCR est nécessaire pour la création d’ADNc d’un descripteur d’ARN. Cela permet à base d’ARN des micro-organismes pour être analysés utilisant essais moléculaires mis au point pour l’ADN. Une fois que l’ADNc est synthétisé, tests de PCR peuvent déterminer la présence ou l’absence de base d’ARN de micro-organismes dans un échantillon environnemental. Cette mesure plus loin en aval analyse afin de déterminer l’écologie microbienne, risques pour la santé et les risques environnementaux.
RT-PCR peut également être utilisé pour doser les ARNm comme un moyen d’observer quels gènes sont exprimés dans un environnement. Il fournit des informations dont les microbes les protéines et les voies dépendent pour survivre à des conditions environnementales en particulier. Analyses d’expression génique peuvent identifier des voies microbiennes que contaminants environnementaux de ventilation tels que des hydrocarbures ou des solvants chlorés, et microbes avec ces voies peuvent être exploitées pour la biorestauration.
Évaluation des risques intègre la RT-PCR afin d’analyser les risques pour la santé humaine et l’environnement. Combinant la RT avec PCR quantitative permet de virus à ARN soit énumérée dans les échantillons, afin que l’exposition humaine et environnementale peut être calculée aux fins quantitatives microbienne risque évaluation (AMCQ).
Passer à...
Vidéos de cette collection:

Now Playing
Analyse d'ARN d'échantillons environnementaux à l'aide de la RT-PCR
Environmental Microbiology
40.4K Vues

Détermination de la teneur en humidité du sol
Environmental Microbiology
359.3K Vues

Technique aseptique en sciences de l'environnement
Environmental Microbiology
126.4K Vues

Coloration de Gram des bactéries provenant de sources environnementales
Environmental Microbiology
100.2K Vues

Visualisation des micro-organismes du sol grâce aux lames de contact et à la microscopie
Environmental Microbiology
42.2K Vues

Champignons filamenteux
Environmental Microbiology
57.3K Vues

Extraction d'ADN communautaire à partir de colonies bactériennes
Environmental Microbiology
28.8K Vues

Détection des micro-organismes environnementaux avec la réaction en chaîne par polymérase et l'électrophorèse sur gel
Environmental Microbiology
44.5K Vues

Quantification des micro-organismes et des virus environnementaux à l'aide de la qPCR
Environmental Microbiology
47.8K Vues

Analyse de la qualité de l'eau via des organismes indicateurs
Environmental Microbiology
29.5K Vues

Isolement des bactéries fécales à partir des échantillons d'eau par filtration
Environmental Microbiology
39.3K Vues

Détection de bactériophages dans des échantillons environnementaux
Environmental Microbiology
40.7K Vues

Culture et dénombrement des bactéries dans un échantillon de sol
Environmental Microbiology
184.3K Vues

Analyse de la courbe de croissance bactérienne et ses applications environnementales
Environmental Microbiology
295.9K Vues

Dénombrement des algues via une méthodologie de culture
Environmental Microbiology
13.8K Vues