Análisis de muestras ambientales mediante RT-PCR del ARN
Visión general
Fuente: Laboratorios del Dr. Ian Pepper y el Dr. Charles Gerba - Universidad de Arizona
Demostrando autor: Bradley Schmitz
Reacción en cadena reversa de la transcripción-polimerasa (RT-PCR) implica el mismo proceso que la PCR convencional — temperatura ciclismo para amplificar ácidos nucleicos. Sin embargo, mientras que la PCR convencional sólo amplifica los ácidos desoxirribonucleico (ADN), RT-PCR permite la amplificación de los ácidos ribonucleicos (RNA) mediante la formación de ADN complementario (cDNA). Esto permite que organismos basados en RNA dentro del entorno para ser analizados utilizando métodos y tecnologías que están diseñados para el ADN.
Muchos de los virus encontrados en el ambiente usan RNA como su material genético. Varias basadas en RNA patógenos virales, como el Norovirusy organismos indicadores, tales como virus de moteado suave del pimiento (PMMoV), no tienen métodos de detección basados en la cultura para la cuantificación. Para detectar la presencia de estos virus de RNA en muestras ambientales de suelo, agua, agricultura, etc., ensayos moleculares dependen de RT-PCR para convertir el RNA en DNA. Sin RT-PCR, microbiólogos no sería capaces de análisis y de investigación de numerosos virus RNA-basado que plantean riesgos para la salud humana y ambiental.
RT-PCR se puede también emplear como una herramienta para medir la actividad microbiana en el medio ambiente. ARN mensajero (ARNm) es la sola plantilla para la traducción de la proteína, y medición de los niveles de los mRNAs diferentes indica que genes que microbios se expresan en el entorno. Análisis de expresión génica da pistas de qué vías biológicas son utilizadas por los organismos para sobrevivir en diferentes condiciones ambientales. En algunos casos, la expresión génica puede ser utilizada para determinar que organismos pueden sobrevivir mejores en duras condiciones y tienen las capacidades para la biorremediación de suelos contaminados o de agua.
Principios
PCR se basa en la amplificación de plantillas de la DNA, que limita su uso en la detección de RNA de organismos. Sin embargo, RT-PCR proporciona un medio para que el uso de RNA para producir cDNA utilizando enzimas especializadas, conocidas como transcriptasa inversa (RT). Este cDNA puede utilizarse entonces como la plantilla de partida para la posterior amplificación con PCR convencional (figura 1).
Reversa de la transcripción puede ser controlada amplificar sólo productos o una comunidad entera de los ácidos nucleicos en una muestra ambiental, dependiendo de los iniciadores que son utilizados a plantilla de la síntesis de cDNA. Esto es importante, como muestras de suelo y agua son a menudo saturadas con varios ácidos nucleicos que no desea para análisis específicos. Cartillas al azar, que pueden atar a las secuencias de RNA encontradas en cualquier tipo de microbios, pueden utilizarse en RT-PCR para detectar ARN más, por lo que la muestra puede ser analizada para la presencia y abundancia relativa de varios organismos en el medio ambiente. Por otro lado, primers específicos de secuencia inician la síntesis de cDNA para secuencias precisas se encuentran en sólo uno o unos pocos organismos. Esto permite una muestra ambiental a probarse para un propósito específico, tales como determinar si Norovirus, que pueden causar enfermedades gastrointestinales en humanos, está presente en el agua.

Figura 1. Proceso paso a paso del análisis de RT-PCR de muestras ambientales de RNA.
Procedimiento
1. muestra Colección: Muestra de suelo
- Encuentre una muestra ubicación vía GPS, coordenadas o vista.
- Para el muestreo al azar, escoger puntos al azar dentro de un área para obtener un censo general de los hábitats microbianos. Recoge muestras de puntos a lo largo de una línea recta, por ejemplo, junto a un cauce de transectos. Muestras de cuadrícula se toman sistemáticamente de puntos a intervalos regulares y son útiles para las comunidades microbianas de la cartografía en una zona desconocida o variable.
- De muestreo dentro de un 3-6 pulgadas (7.5-15 cm) profundidad proporciona acceso a la actividad microbiana más abundante que está cerca, pero no dentro de la rizosfera (región estrecha de suelo afectado directamente por las raíces de la planta y sus microorganismos asociados).
- Para recolectar la muestra de suelo, empuje y gire un taladro de mano en el suelo a la profundidad predeterminada.
- Levantar la barrena. El suelo se encuentra dentro del tallo hueco de la barrena.
- Raspar el suelo en la parte inferior de la barrena en una bolsa de recolección de suelo. Asegúrese de no tocar o contaminar el suelo.
- Etiquetar la bolsa con ubicación, nombre, fecha y hora.
- En el laboratorio, pasa del suelo a través de un tamiz de 2 mm para eliminar cualquier grava y roca.
- Analizar una porción del suelo para contenido de humedad del suelo. Para más detalles de este paso, consulte JoVE Ciencias de la educación video sobre humedad del suelo.
2. muestra Colección: Muestra de agua
- Encontrar la ubicación de la muestra vía GPS, coordenadas o vista.
- Recoger el agua en una botella de almacenamiento. Registrar el volumen de agua recolectada; Si los microbios en la muestra se analizan cuantitativamente, entonces la concentración microbiana se puede determinar basándose en el volumen recogido.
- Inmediatamente probar el agua para los parámetros requeridos para el experimento (temperatura, pH, conductividad, salinidad, nitrógeno y fósforo contenido, etc.) utilizando las sondas electrónicas adecuadas.
- Coloque la botella que contiene la muestra de agua en una hielera con hielo. Transferencia de la hielera al laboratorio.
3. extracción de RNA
- Para recoger y concentrar los microorganismos de la muestra ambiental, refiera por favor a la enseñanza de las Ciencias JoVE video de extracción de ácidos nucleicos de comunidad.
- Extracto de RNA de virus utilizando un kit de extracción comercial según las instrucciones del fabricante.
- Brevemente, primero se mezclan las muestras con tampón de lisis con etanol, después añadir las muestras en las columnas de la vuelta.
- Centrifugue las columnas a aproximadamente 12.000 x g, flujo de descarte. Añadir tampón de lavado a las columnas y centrifugue de nuevo.
- Añadir agua libre de ARNasa a las columnas y centrifugue durante 30 s para eluir el RNA en los tubos del microfuge de baja adherencia de 1,5 mL estéril.
4. Invierta la transcripción - PCR
- Recuperar los siguientes reactivos en el congelador de-20 ° C: dNTP, concentrado (por ejemplo, 10 x) inversa buffer de transcripción, cartillas (en este ejemplo, primers al azar). Descongelar estos en hielo o a temperatura ambiente dentro de una campana limpia y mantenerse en hielo descongelado. También recuperar la transcriptasa reversa y un inhibidor de RNasa y mantener en hielo.
- Calcular los volúmenes de reactivo necesarios para hacer una "mezcla maestra" que combina todos los reactivos constantes entre cada reacción (ver tabla 1 para una reacción de la muestra). Preparar suficiente mezcla maestra para cada muestra, así como un control positivo (con una plantilla de transcripción conocido) y negativas (p. ej., sin transcriptasa inversa, con agua como plantilla, etc.) reacciones de control. Incluyen un 10% adicional en el volumen.
- Montar la mezcla principal en tubos de microcentrífuga de 1,5 mL baja adherencia. Esto minimiza el atascamiento de las moléculas de reactivo para paredes de los tubos de plástico.
- Cuando los reactivos son descongelados, añadir volúmenes calculados para el tubo de microcentrífuga. Suavemente el vortex y centrifugar cada tubo antes de la adición. Asegúrese de cambiar las puntas de pipeta entre adición de cada reactivo para evitar la contaminación.
- Después de todos los reactivos se han añadido, vortex y centrifugar la mezcla principal para asegurar una mezcla homogénea. Vuelva a colocar los reactivos en almacenamiento a-20 ° C.
- Preparar y etiquetar el tubo 8 tiras PCR, que designa un tubo para cada muestra o control de la reacción.
- Parte alícuota de un volumen igual de la master mix a cada tubo. Luego, añadir componentes de reacción específicos, tales como los extractos de RNA.
- Coloque la tapa de la tira firmemente en la franja de PCR y centrifugar en una minicentrifuge con un adaptador de tubo tira para asegurar todo el líquido se recoge en la parte inferior de cada tubo (figura 2).
- Tira de lugar PCR en termociclador. Presione hacia abajo para que los tubos estén asegurados.
- Establecer el termociclador para ejecutar el programa apropiado para el RT se utiliza (ver tabla 2 para un protocolo de muestra). Cuando el programa es completo, los tubos contienen productos de cDNA, que luego pueden ser sometidos a la amplificación de la polimerización en cadena. Para obtener más información, consulte los videos de JoVE Ciencias de la educación en PCR y PCR cuantitativa. Tienda cDNA a-20 ° C hasta su uso.

Figura 2. Tira de 8-tubo tapada que contiene extracto y mezcla principal.
| Reactivo de | Volumen por 1 reacción (μL) |
| 10 x Buffer de RT | 2.0 |
| 25 x dNTPs | 0,8 |
| 10 x cartilla al azar | 2.0 |
| Multiscribe | 1.0 |
| Inhibidor de Rnasa | 1.0 |
| Grado molecular H2O | 3.2 |
| Volumen total | 10 |
Tabla 1. RT amo mezcla los ingredientes.
| Paso 1 | Paso 2 | Paso 3 | Paso 4 |
| 25 ° C, 10 min. | 37 ° C, 120 min | 85 ° C, 5 min. | 4 ° C, ∞ |
Tabla 2. RT reacción termociclador programa.
Resultados
Cuando RT - PCR es completa, algunos de los productos PCR pueden separados y visualizados en un gel de agarosa (figura 3). En este ejemplo, se utilizó una cartilla gene-específica para detectar la presencia de un virus de ARN. Bandas del tamaño esperado se obtienen de dos de las muestras y la reacción de control positivo, pero no del control negativo, indicando la presencia de este virus en dos de las muestras de agua poniendo a prueba.
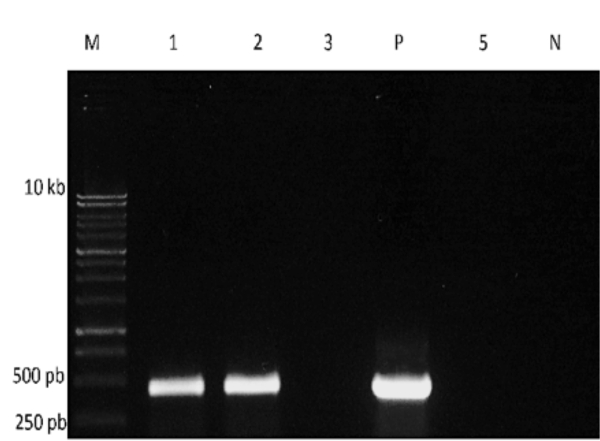
Figura 3. Electroforesis en gel de los productos de RT-PCR. M: marcador de tamaño de ADN; P: control positivo; N: control negativo. Reacciones con RNA de cuatro muestras de agua fueron funcionadas en los carriles 1, 2, 3 y 5.
Aplicación y resumen
RT-PCR es necesario para la creación de cDNA de una plantilla de RNA. Esto permite que microorganismos basado en RNA ser analizados utilizando análisis moleculares desarrollados por ADN. Una vez que es sintetizado el ADNc, análisis PCR pueden determinar la presencia o ausencia de microorganismos basados en RNA dentro de una muestra ambiental. Esto permite más análisis aguas abajo para determinar la ecología microbiana, riesgos para la salud y los riesgos ambientales.
RT-PCR también puede ser utilizado para análisis de mRNA como un medio para observar qué genes se expresan en un ambiente. Esto proporciona información acerca de que proteínas y vías de microbios dependen para sobrevivir, en particular, las condiciones ambientales. Análisis de expresión génica pueden identificar vías microbianas contaminantes ambientales de degradación tales como hidrocarburos o solventes clorados, y microbios con estas vías pueden aprovecharse para la biorremediación.
Evaluación de riesgos incorpora RT-PCR para analizar riesgos para la salud humana y ambiental. La combinación de RT con PCR cuantitativa permite virus del RNA a enumerar dentro de muestras, para que la exposición humana y ambiental puede ser calculada con el propósito de evaluación cuantitativa de riesgo microbiano (QMRA).
Saltar a...
Vídeos de esta colección:

Now Playing
Análisis de muestras ambientales mediante RT-PCR del ARN
Environmental Microbiology
40.4K Vistas

Determinación del contenido de humedad en el suelo
Environmental Microbiology
359.3K Vistas

Técnica aséptica en ciencias ambientales
Environmental Microbiology
126.4K Vistas

Tinción de Gram de la Bacteria de fuentes ambientales
Environmental Microbiology
100.2K Vistas

Visualizarse los microorganismos del suelo mediante el contacto Deslice análisis y microscopia
Environmental Microbiology
42.2K Vistas

Hongos filamentosos
Environmental Microbiology
57.3K Vistas

Extracción de ADN de la comunidad de colonias bacterianas
Environmental Microbiology
28.8K Vistas

Detección de microorganismos ambientales con la reacción en cadena de polimerasa y la electroforesis en Gel de
Environmental Microbiology
44.5K Vistas

Cuantificación de microorganismos ambientales y virus utilizando qPCR
Environmental Microbiology
47.8K Vistas

Análisis de la calidad del agua a través de organismos indicadores
Environmental Microbiology
29.5K Vistas

Aislamiento de bacterias fecales de muestras de agua por filtración
Environmental Microbiology
39.3K Vistas

Detección de bacteriófagos en muestras ambientales
Environmental Microbiology
40.7K Vistas

Cultivo y enumeración de bacterias a partir de muestras de suelo
Environmental Microbiology
184.3K Vistas

Análisis de la curva de crecimiento bacteriano y sus aplicaciones ambientales
Environmental Microbiology
295.9K Vistas

Enumeración de las algas mediante metodología cultivable
Environmental Microbiology
13.8K Vistas
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados